i
IDENTIFIKASI KERAGAMAN D-LOOP DNA MITOKONDRIA
PADA SAPI POTONG DI INDONESIA
SKRIPSI SRI RAHAYU
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
INSTITUT PERTANIAN BOGOR 2012
ii RINGKASAN
SRI RAHAYU. D14080212. 2012. Identifikasi Keragaman D-loop DNA Mitokondria pada Sapi Potong Di Indonesia. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Dr. Jakaria, S.Pt., M.Si. Pembimbing Anggota : Prof. Dr. Ir. Muladno, MSA.
Sapi Bali merupakan salah satu sumber daya genetik asli Indonesia, sumber daya genetik lain yang dimiliki oleh Indonesia pada ternak sapi yaitu hasil silangan sapi asli dan sapi luar yang masuk ke Indonesia yang telah menjadi sapi lokal seperti sapi Madura, Pesisir, Aceh, dan PO. Persilangan/perkembangan jenis sapi tersebut menyebabkan muncul keragaman genetik dan keterkaitan/hubungan kekerabatan (filogenetik) pada sapi asli dan sapi lokal Indonesia, termasuk keragaman di DNA mitokondria (mtDNA) di daerah D-loop. Keragaman pada mtDNA penting untuk diketahui, yaitu sebagai informasi dasar tentang mtDNA khususnya di daerah D-loop (data base) pada sapi-sapi Indonesia, mengingat sapi-sapi tersebut sudah diakui oleh dunia.
Penelitian ini bertujuan untuk mendapatkan sekuen mtDNA daerah D-loop pada sapi dan mengetahui keragaman daerah D-loop mtDNA pada sapi Bali, Madura, Pesisir, Aceh dan PO di Indonesia. Tujuan lain yaitu untuk mengetahui hubungan genetik (filogenetik) antara sapi Bali, Madura, Pesisir, Aceh, dan PO.
Sampel darah sapi yang digunakan sebanyak 18 sampel yaitu dua sampel dari sapi Bali yang berasal dari BIB dan empat sampel dari BPTU sapi Bali pulau Bali, masing-masing dua sampel sapi Madura yang berasal dari Sapudi dan Kabupaten Sampang, dua sampel sapi Pesisir dari Kabupaten Pesisir Selatan, dua sampel sapi Aceh dari Kabupaten Aceh Besar, dan masing-masing dua sampel sapi PO yang berasal dari Kab. Kebumen dan kandang A Fakultas Peternakan IPB. Sampel darah tersebut diamplifikasi dengan PCR (Polymerase Chain Reaction) dan dilanjutkan dengan sekuen. Data sekuen D-loop disejajarkan berganda dengan sekuen acuan Bos
indicus dari GenBank (kode akses AY126697) menggunakan program Clustal W
yang terdapat dalam program MEGA 5.0. Hitungan jarak genetik antara sapi penelitian menggunakan perhitungan pairwise distance dan analisis filogeni dengan menggunakan metode bootstrapped Neighboor-Joining (NJ) dengan 1000 kali pengulangan.
Hasil PCR pada suhu 60 oC menghasilkan produk amplifikasi sebesar 1145 bp, namun yang dapat dianalisis sepanjang 584 bp. Hasil amplifikasi tersebut didapatkan sekuen basa nukleotida sebesar 22 bp (GTACATAATATTAATGTAATAA)
sebagai motif ulangan dan ditemukan pada sapi Bali, Madura, dan PO. Motif ulangan lain sepanjang 22 bp yaitu GTACATAATATTAATGTAATAA juga ditemukan pada sapi Pesisir, Aceh, Bali 1, dan Bali 2. Hasil analisis D-loop mtDNA dengan sekuen acuan Bos indicus, menunjukkan bahwa sapi Aceh satu klaster dengan sapi Pesisir, mempunyai jarak genetik yang lebih dekat dengan sapi Bos
indicus, sedangkan sapi Bali, Madura, dan PO membentuk klaster sendiri yang
memiliki jarak genetik lebih dekat dengan sapi Bali.
iii ABSTRACT
Identification of Genetic Variation D-loop of mtDNA in Indonesian Beef Cattles Rahayu, S., Jakaria, Muladno
Indonesia have indigenouse genetic resources especially in Bali cattle, in addition there is others local cattle genetic resource which is the cross breeding from indigenouse cattle and foreign cattle like Madura, Aceh, and Pesisir cattle. The impact from cross breedingare necessary to know the genealogy and their relationships (phylogenetic) of the cattle. The aims of this study were to determine the diversity from the D-loop of mtDNA sequences and phylogenetic relationship from Bali, Madura, Pesisir, and Aceh cattle through GenBank. A total of 18 sampels from the 6 Bali cattles (Bali island), 4 Madura cattles (Madura island), 2 Pesisir cattles (south Pesisir), 2 Aceh cattles (Aceh Besar), and from 4 PO cattles as out group were analyzed by Polimerase Chain Reaction (PCR) amplification and subsequently sequenced. The data were aligned refer to Bos indicus sequences from GenBank using Cluscal W programme and analyzed by MEGA5 programme. The result of PCR product of D-loop sequences by 60 oC annealing temperature were 1145 bp length which could be analyzed 584 bp. On the other hand, amplification nukeotide sequence was 22 bp length as repeat tandem which found in Bali, Madura, Pesisir, Aceh, and PO cattle. From the D-loop mtDNA analysis indicated that Aceh cattle had the same cluster as Pesisir cattle which have closer genetic distance to Bos
indicus, on the other hand Bali, Madura and PO cattle had the same cluster in
different group which have closer genetic distance to Bali cattle.
iv
IDENTIFIKASI KERAGAMAN D-LOOP DNA MITOKONDRIA
PADA SAPI POTONG DI INDONESIA
SRI RAHAYU D14080212
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
INSTITUT PERTANIAN BOGOR 2012
v Judul : Identifikasi Keragaman D-Loop DNA Mitokondria pada Sapi
Potong di Indonesia Nama : Sri Rahayu
NIM : D14080212 Menyetujui, Pembimbing Utama, (Dr. Jakaria, S.Pt., M. Si.) NIP : 19660105 199303 1 001 Pembimbing Anggota,
(Prof. Dr. Ir. Muladno, MSA) NIP : 19610824 198603 1 001
Mengetahui: Ketua Departemen,
Ilmu Produksi dan Teknologi Peternakan
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc.) NIP: 19591212 198603 1 004
vi RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 20 Juli 1990 di Banjarnegara. Penulis merupakan anak keempat dari empat bersaudara dari pasangan Bapak H. Mohammad Sodiq dan Ibu Hj. Sutariyah.
Penulis menyelesaikan pendidikan dasar pada tahun 2002 di SDN 6 Batur. Pendidikan lanjutan tingkat pertama diselesaikan pada tahun 2005 di MTs. Muhammadiyah Batur, Banjarnegara dan pendidikan menengah atas diselesaikan pada tahun 2008 di SMA Negeri 1 Bawang, Banjarnegara.
Penulis diterima di Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB USMI pada tahun 2008. Penulis diterima sebagai mahasiswa Mayor Teknologi Peternakan di Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan Institut Pertanian Bogor pada tahun 2009. Selama mengikuti pendidikan, Penulis aktif di Himpunan Mahasiswa Produksi Ternak Himaproter periode 2011 dan Majalah Peduli Pangan dan Gizi EMULSI periode 2010-2011, Institut Pertanian Bogor. Penulis juga berkesempatan menjadi asisten mata kuliah Genetika Ternak pada tahun 2011-2012. Penulis berkesempatan mengikuti magang di Balai Penelitian Ternak (Balitnak) Bogor pada tahun 2009 dan Balai Embrio Ternak (BET) Bogor pada tahun 2010. Penulis pernah lolos dalam PKM penelitian yang didanai oleh DIKTI pada tahun 2011.
vii KATA PENGANTAR
Alhamdulillahirrabil’alamin,
Puji dan syukur Penulis panjatkan ke hadirat Allah SWT yang telah memberikan rahmat, karunia, rizki dan nikmat iman dan Islam yang telah diberikan sehingga Penulis memperoleh kemudahan dalam menyusun dan menyelesaikan skripsi ini yang berjudul ”Identifikasi Keragaman D-loop DNA Mitokondria pada Sapi Potong di Indonesia”. Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan di Fakultas Peternakan, Institut Pertanian Bogor. Shalawat dan salam semoga selalu kita curahkan kepada Nabi Muhammad
SAW.
Sapi Bali merupakan salah satu sumber daya genetik asli Indonesia yang sudah didomestikasi dan termasuk aset dunia yang sangat bernilai. Indonesia juga memiliki bangsa-bangsa sapi lokal yang sudah diakui dunia seperti sapi Madura, Pesisir, Aceh, dan PO. Hingga saat ini informasi genetik sapi asli atau sapi lokal di Indonesia secara umum masih bersifat parsial terbatas pada salah satu jenis sapi. Informasi genetik sangat menunjang untuk program pemuliaan sapi Indonesia terutama dalam upaya pelestarian, pengembangan dan pemanfaatan secara berkelanjutan. Oleh karena itu, maka perlu dilakukan penelitian-penelitian di bidang ini yaitu dengan melakukan studi keragaman D-loop mtDNA pada sapi asli dan sapi lokal Indonesia.
Penulis menyadari bahwa dalam penulisan ini masih banyak kekurangan. Kritik dan saran yang membangun sangat diharapkan demi kesempurnaan tulisan ini. Semoga tulisan ini dapat bermanfaat bagi pembaca dan menjadi pedoman dasar untuk penelitian serupa pada masa yang akan datang. Amin.
Bogor, Desember 2012
viii DAFTAR ISI Halaman RINGKASAN ... ii ABSTRACT ... iii LEMBAR PERNYATAAN ... iv LEMBAR PENGESAHAN ... v RIWAYAT HIDUP ... vi
KATA PENGANTAR ... vii
DAFTAR ISI ... viii
DAFTAR TABEL ... x
DAFTAR GAMBAR ... xi
DAFTAR LAMPIRAN ... xii
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan ... 3
TINJAUAN PUSTAKA ... 4
Klasifikasi Sapi ... 4
Bangsa Sapi di Indonesia ... 4
Sapi Bali ... 5 Sapi Madura ... 6 Sapi Pesisir ... 6 Sapi Aceh ... 7 Keragaman Genetik ... 8 DNA Mitokondria ... 9
Kekerabatan Sapi di Indonesia ... 12
MATERI DAN METODE ... 15
Lokasi dan Waktu ... 15
Materi ... 15
Sampel Darah ... 15
Alat dan Bahan ... 15
Primer DNA Mitokondria Daerah D-loop ... 16
Prosedur ... 16
Pengambilan Sampel Darah ... 16
Isolasi DNA ... 16
Amplifikasi DNA Mitokondria Daerah D-loop ... 17
Elektroforesis ... 18
ix
Rancangan dan Analisis Data ... 18
Jarak Genetik dan Pohon Filogeni ... 18
HASIL DAN PEMBAHASAN ... 20
Amplifikasi Daerah D-loop ... 20
Keragaman D-loop ... 22
Komposisi Basa Nukleotida ... 22
Sekuen Minisatelit ... 25
Jarak Genetik Sapi Asli dan Sapi Lokal Indonesia ... 27
Analisis Filogenetik ... 31
KESIMPULAN DAN SARAN ... 35
Kesimpulan ... 35
Saran ... 35
UCAPAN TERIMA KASIH ... 36
DAFTAR PUSTAKA ... 37
x DAFTAR TABEL
Nomor Halaman
1. Jumah Sampel Darah Ternak Sapi Indonesia... 15 2. Rataan Komposisi Nukleotida Daerah D-loop Parsial Sapi Bali,
Madura, Aceh, Pesisir, PO Setelah Disejajarkan dengan B. indicus Dari Genbank (Ukuran 584 bp) ... 23 3. Lokasi Sekuen Berulang pada Setiap Individu Sapi Bali, Madura,
PO, Pesisir, dan Aceh ... 26 4. Jumlah Motif Ulangan Basa Nukleotida yang Ditemukan pada
Bangsa Sapi Bali, Madura, PO, Pesisir, dan Aceh ... 26 5. Jarak Genetik Berdasarkan Metode Pairwise Distance Daerah
D-loop yang Dilakukan Pengelompokan pada Sapi Bali, Sapi Madura
dan Sapi PO dengan Sapi B. indicus dan B.taurus dari Genbank ... 28 6. Jarak Genetik Berdasarkan Metode Pairwise Distance Daerah
D-loop Setiap Individu pada Sapi Bali, Sapi Madura dan Sapi PO
xi DAFTAR GAMBAR Nomor Halaman 1. Sapi Bali ... 5 2. Sapi Madura ... 6 3. Sapi Pesisir ... 7 4. Sapi Aceh ... 8
5. Genom Mitokondria pada B. indicus ... 10
6. Skema DNA Mitokondria Daerah D-loop ... 11
7. Komposisi Genetik Populasi Sapi di Beberapa Wilayah di Indonesia ... 13
8. Konstruksi Pohon Filogenik Sapi Asli dan Sapi Lokal Indonesia Berdasarkan DNA Daerah CO I ... 13
9. Konstruksi Pohon Filogenik Sapi Asli dan Sapi Lokal Indonesia ... 14
10. Situs Penempelan Primer Sekuen D-Loop DNA Mitokondria Sapi 17 11. Hasil Ektroforesis Produk PCR Daerah D-loop mtDNA ... 20
12. Sketsa Letak Penempelan Primer Forward dan Reverse untuk Mengamplifikasi pada Daerah D-loop Sapi Bali, Madura, Pesisir, Aceh, dan PO ... 22
13. Frekuensi A+T dan G+C Daerah D-loop mtDNA Parsial Berukuran 584 bp pada Sapi Bali, Madura, Aceh, Pesisir, dan PO . 24 14. Konstruksi Pohon Filogeni Antar Individu Sapi Berdasarkan Metode Neighbor-Joining ... 33
15. Konstruksi Pohon Filogeni Kelompok Sapi Berdasarkan Metode Neighbor-Joining ... 34
xii DAFTAR LAMPIRAN
Nomor Halaman
1. Modifikasi Metode Isolasi DNA Menggunakan Genomic DNA
Mini Kit (Geneaid) ... 43
2. Elektroforesis Produk PCR sebelum Dilakukan Sekuensing ... 44
3. Lokasi Penempelan Primer Forward dan Reverse pada Sekuen Basa Nukleotida Gen D-loop parsial Sapi Asli dan Sapi Lokal Indonesia ... 45
4. Contoh Hasil Sekuensing pada Sapi Bali ... 46
5. Contoh Hasil Sekuensing pada Sapi Madura ... 47
6. Contoh Hasil Sekuensing pada Sapi Pesisir ... 48
7. Contoh Hasil Sekuensing pada Sapi Aceh ... 49
8. Contoh Hasil Sekuensing pada Sapi PO ... 50
9. Situs-situs Delesi dan Insersi Basa-basa Nukleotida D-loop parsial 584 bp ... 51
10. Jumlah Nukleotida Sapi Bali, Madura, Aceh, Pesisir, dan PO dan Sapi Pembanding dari GenBank ... 59
11. Motif Ulangan pada sapi Bali, Madura, Pesisir, Aceh, dan PO... 60
12. Nomor Akses Sekuen Daerah D-loop Utuh B. indicus dan B. taurus dari Genbank pada Situs NBCI yang Digunakan untuk Membentuk Pohon Filogeni ... 61
1 PENDAHULUAN
Latar Belakang
Sapi Bali merupakan salah satu sumber daya genetik ternak asli Indonesia yang sudah didomestikasi (MacHugh, 1996) dan termasuk salah satu aset dunia yang sangat berharga (Noor et al., 2000). Sementara, bangsa sapi lokal Indonesia yaitu Madura, Aceh, dan Pesisir. Sapi Sumba-Ongole (SO) dan Java-Ongole (PO) juga dianggap sebagai sapi lokal Indonesia (Martojo, 2003). Noor (2010) menyatakan bahwa ternak asli maupun lokal memiliki keunggulan yaitu mampu beradaptasi dengan lingkungan lokal seperti kualitas pakan berkualitas rendah, ketersediaan air yang kurang, iklim tropis, manajemen yang kurang baik, dan ketahanan terhadap penyakit. Dengan demikian, ternak-ternak inilah yang paling cocok untuk dipelihara dan dikembangkan di Indonesia khususnya bagi peternak kecil (small holder farmer), walaupun produksinya relatif lebih rendah dari ternak impor.
Sapi Bali merupakan sapi yang berasal dari domestikasi banteng (Bos
javanicus) yang pada awalnya termasuk banteng liar asli dari Pulau Bali (Hayashi et al., 1980). Proses domestikasi tersebut mengakibatkan terdapat beberapa perbedaan
dan kesamaan antara ternak domestikasi dan ternak liar sebagai nenek moyang (Vaisanen dan Jensen, 2003). Sementara sapi lokal (Madura, Pesisir, dan Aceh) merupakan persilangan banteng dan sapi luar yang masuk ke Indonesia. Namun, telah cukup lama berada di Indonesia sehingga berkembang biak sesuai dengan lingkungan lokal (Abdullah et al., 2008), hal tersebut menyebabkan kemungkinan adanya keragaman genetik seperti keragaman di DNA mitokondria (mtDNA) di daerah D-loop antara sapi-sapi lokal Indonesia dan sapi Bali. Adanya dugaan keterkaitan/hubungan kekerabatan (filogenetik) antar bangsa sapi yang ada di Indonesia menjadi hal penting untuk diketahui.
Keterkaitan hubungan antar sapi asli dan sapi lokal Indonesia telah dilakukan berdasarkan ukuran tubuh (Otsuka et al., 1980; Abdullah, 2008), ukuran tengkorak (Hayashi et al., 1980), protein darah (Namikawa et al., 1980), DNA mikrosatelit (Mohamad et al., 2009) dan DNA mitokondria daerah CO I (Febriana, 2011). Beberapa hasil penelitian tersebut masih bersifat parsial dan terbatas pada salah satu bangsa sapi lokal. Di sisi lain, Indonesia memiliki beberapa bangsa sapi lokal Indonesia yang merupakan sapi-sapi yang sudah diakui oleh dunia (sapi Bali,
2 Madura, Pesisir, Aceh, dan PO). Semakin berkembangnya kajian ilmu genetika molekuler saat ini, maka penelitian terhadap bangsa sapi lokal Indonesia pada tingkat molekuler khususnya pada DNA mitokondria sangat diperlukan, terutama DNA mitokondria di daerah D-loop karena DNA mitokondria mempunyai jumlah turunan yang lebih tinggi (high copy number), yaitu mempunyai jumlah salinan sebesar 103 -104 molekul DNA mitokondria/sel somatik (Randi, 2000). Keunggulan lain dari mtDNA yaitu ukuran mtDNA kecil sehingga dapat dipelajari secara utuh. Genom DNA mitokondria mempunyai laju evolusi 5-10 kali lebih cepat dari DNA inti (Clayton, 1992).
Nijman et al. (2003) menyatakan bahwa penentuan daerah D-loop mtDNA pada sapi dapat digunakan untuk menunjukkan proses hibridisasi pada bangsa sapi Indonesia termasuk sapi asli Indonesia (sapi Bali). Penanda atau marker DNA mitokondria adalah penanda genetik berdasarkan garis maternal yang hanya diwariskan melalui induk tanpa mengalami rekombinasi. Penciri yang berbasis pada DNA mitokondria dapat digunakan untuk merekonstruksi pohon kekerabatan (filogenetik) pada berbagai bangsa/spesies yang saling berdekatan (Duryadi, 1994).
Penelitian di daerah D-loop sudah sangat spesifik dan sudah digunakan sangat luas, penelitian mtDNA daerah D-loop pernah dilakukan untuk mengetahui variasi DNA mitokondria dan evolusi sapi Hitam Jepang (Bos taurus) (Mannen et
al., 1998), memposisikan sapi Zebu Amerika berasal dari Bos indicus (Meirelles et al., 1999), menentukan perbedaan genetik dan variasi sekuen daerah D-loop mtDNA
sapi asli Cina (Lai, 2005), mengetahui hibridisasi banteng (Bos javanicus) dan zebu (Bos indicus) (Nijman et al., 2003), menetukan keragaman genetik dan pohon filogenetik kerbau sungai di Mesir (Hassan et al., 2009). Hal ini menjadikan penelitian identifikasi keragaman D-loop mtDNA penting untuk dilakukan, mengingat sapi-sapi tersebut merupakan sumber daya genetik yang dimiliki Indonesia. Hasil penelitian ini diharapkan dapat memberikan informasi dasar tentang DNA mitokondria di daerah D-loop (data base) pada sapi-sapi Indonesia. Selain itu hasil penelitian ini diharapkan dapat digunakan sebagai salah satu dasar dalam menentukan kebijakan program pemuliaan sapi Indonesia terutama dalam upaya pelestarian, pengembangan dan pemanfaatannya secara berkelanjutan.
3 Tujuan
Penelitian ini bertujuan untuk mendapat sekuen daerah D-loop mtDNA dan mengetahui keragaman daerah D-loop mtDNA pada sapi Bali, Madura, Pesisir, Aceh dan PO di Indonesia. Tujuan lain yaitu untuk mengetahui hubungan genetik (filogenetik) antara sapi Bali, Madura, Pesisir, Aceh, dan PO.
4 TINJAUAN PUSTAKA
Klasifikasi Sapi
Penggolongan sapi ke dalam suatu bangsa (breed) sapi, didasarkan atas sekumpulan persamaan karakteristik tertentu yang sama. Atas dasar karakteristik tersebut, mereka dapat dibedakan dari ternak lainnya meskipun masih dalam spesies yang sama. Karakteristik yang dimiliki tersebut akan diturunkan ke generasi berikutnya. Menurut Blakely dan Bade (1992) bangsa sapi mempunyai klasifikasi taksonomi sebagai berikut :
Phylum : Chordata Subphylum : Vertebrata Class : Mamalia Sub class : Theria Infra class : Eutheria Ordo : Artiodactyla Sub ordo : Ruminantia Infra ordo : Pecora Famili : Bovidae Genus : Bos (cattle)
Spesies : Bos taurus (sapi Eropa)
Bos indicus (sapi India/sapi zebu) Bos javanicus (banteng/sapi Bali)
Bangsa Sapi di Indonesia
Berdasarkan taksonomi sapi diklasifikasikan menjadi tiga, yaitu Bos
indicus, Bos taurus, dan Bos javanicus (Payne dan Hodges, 1997). Sapi Bali,
Madura, Padang, Aceh, Peranakan Ongole, dan Grati dikenal sebagai sapi-sapi yang terdapat di Indonesia. Sapi-sapi tersebut memiliki karakteristik warna kulit maupun ukuran tubuh yang berbeda. Kondisi seperti ini, dimungkinkan sebagai refleksi introgresi sapi Bos indicus dari India dan Bos taurus dari Eropa (Otsuka et al., 1980). Berdasarkan uji jarak genetik, sapi Madura mempunyai hubungan terdekat dengan sapi Bali. Sapi Madura, Jawa dan Peranakan Ongole diklasifikasikan sebagai
5 maupun Eropa (Namikawa et al., 1980; Mohamad et al., 2009; Febriana, 2011). Lebih lanjut dijelaskan bahwa sapi Bali merupakan sebagian besar tetua sapi-sapi yang terdapat di Indonesia (Payne dan Hodges, 1997).
Sapi Bali
Sapi Bali merupakan sapi yang berasal dari domestikasi banteng (Bos
banteng javanicus) (Nijman et al., 2003) yang termasuk banteng liar asli yang
berasal dari Pulau Bali (Hayashi et al., 1980). Sapi-sapi tersebut berasal dari pegunungan yang terdapat di Bali dan kemudian pergi ke daratan pada tahun 1856. Sapi Bali tersebut kemudian menyebar ke pulau Sulawesi, Lombok, dan Timor dan sebagian kecil pulau di Indonesia (Payne dan Rollinson, 1973).
Gambar 1. Sapi Bali
Sumber : Hartaningsih (2008)
Sapi Bali termasuk sapi kecil dengan ukuran bobot yaitu 150-300 kg pada jantan bobot badan dewasa (Talib et al., 2002). Sapi Bali memiliki karakteristik yang seragam. Ternak ini berukuran sedang, berdada dalam, kaki yang bagus. Warna bulu sapi Bali yaitu merah, keemasan, dan coklat tua dikenal juga walaupun tidak umum. Sapi Bali memiliki Bibir, kaki, dan ekor hitam dan kaki berwarna putih dari lutut ke bawah, dan terdapat warna putih di bawah paha dan bagian oval yang putih yang jelas pada bagian pantat. Pada bagian punggung selalu terdapat garis hitam yang jelas, dari bahu dan berakhir di atas ekor. Warna pada ternak jantan lebih gelap daripada betina, warna bulu menjadi coklat tua sampai hitam pada saat
6 mencapai dewasa. Sapi Bali memiliki bulu pendek, halus, dan licin, kulit berpigmen dan halus, dan kepala lebar dan pendek (Williamson dan Payne, 1993). Gambar sapi Bali dapat dilihat pada Gambar 1.
Sapi Madura
Sapi Madura yaitu sapi yang banyak ditemukan di Pulau Madura. Sapi jantan mempunyai ciri-ciri ukuran gumba sedang, namun lebih kecil daripada gumba sapi Aceh jantan. Sapi Madura betina hanya ditemukan jejak-jejak gumba. Secara umum sapi Madura yang terdapat di pulau Madura memiliki warna coklat, tetapi beberapa penelitian menemukan warna sapi Madura mirip dengan sapi Bali yaitu memiliki kaki berwarna putih, pantat berwarna putih atau hitam, dan memiliki garis hitam di bagian punggung (Otsuka et al., 1980).
Gambar 2. Sapi Madura
Sumber : Direktorat Jenderal Peternakan, (2012a)
Sapi Pesisir
Sapi pesisir merupakan salah satu sapi lokal Indonesia yang terdapat di Sumatera Barat yang berbeda dari sapi lokal lain yang terdapat di Indonesia yaitu memiliki bentuk dan ukuran yang kecil (Jakaria, 2008). Sapi ini yang berasal dari Kabupaten Pesisir Selatan merupakan sapi terkecil kedua di dunia setelah sapi dwarf
west Afrika Shorthorn yang berasal dari Wilayah pantai Afrika Barat (Sarbaini,
7 satu keunggulan komparatif yang tidak dimiliki oleh bangsa sapi lain (Jakaria, 2008). Keunggulan lain yang dimiliki sapi pesisir yaitu variasi bulu yang beragam, sehingga menjadikan identitas suatu bangsa, yaitu putih, kuning muda, kuning tua, merah bata, cokelat, dan hitam (Sarbaini, 2004).
Sapi Pesisir memiliki rataan tinggi pundak 114 cm dan betina 109 cm pada umur 4 tahun (Saladin, 1983). Sementara Sarbaini (2004) mendapatkan rataan tinggi pundak pada sapi jantan dewasa pada setiap sub populasi sapi pesisir, yaitu di daerah Kabupaten Pesisir Selatan, Padang Pariaman, Kabupaten Agam masing-masing 99,9 cm; 108, 7 cm; dan 101,8 cm; sedangkan pada betina masing-masing 99,2 cm; 108,2 cm; dan 101,7 cm.
Gambar 3. Sapi Pesisir
Sumber : Direktorat Jenderal Peternakan (2012b)
Sapi Aceh
Sapi Aceh merupakan bangsa sapi tipe kecil yang ditemukan khusus di daerah Aceh (Abdullah et al., 2008). Hasil penelitian Abdullah (2008) menunjukkan bobot badan dan ukuran tubuh sapi Aceh mengalami penurunan dibandingkan dengan bobot badan dan ukuran tubuh yang dilaporkan pada tahun 1926. Ukuran-ukuran tubuh sapi Aceh mempunyai Ukuran-ukuran-Ukuran-ukuran tubuh yang lebih kecil pada tingkat umur yang sama, apabila dibandingkan sapi Bali, Madura dan PO. Namun masih berada di atas rataan ukuran-ukuran tubuh dan bobot badan sapi Pesisir di
8 Sumatera Barat. Rata-rata bobot hidup sapi Aceh dewasa betina 161,19 kg dan jantan 191,78 kg (Abdullah, 2008).
Abdullah et al. (2008) menemukan hampir seluruh populasi sapi Aceh mempunyai garis muka yang cekung dan sebagian (4,5%) memiliki garis muka yang lurus. Secara umum sapi Aceh mempunyai garis punggung yang cekung (89,25%), sebagian mempunyai garis punggung cembung (6,25%) dan sebagian kecil mempunyai garis punggung lurus (4,5%). Secara kualitatif, sapi Aceh mempunyai warna dominan merah bata dan cokelat muda serta pola warna beragam mulai warna gelap sampai terang. Bentuk pertumbuhan tanduk sapi betina mengarah ke samping melengkung ke atas kemudian ke depan dan pada jantan mengarah ke samping melengkung ke atas.
Gambar 4. Sapi Aceh
Sumber : Nangroe Aceh Darusalam. Litbang (2012)
Keragaman Genetik
Keragaman genetik yaitu perbedaan genotip diantara ternak-ternak yang tidak memiliki hubungan keluarga (Noor, 2010). Keragaman genetik timbul akibat proses pembelahan meiosis saat pembentukan gamet, yang disebabkan salah satunya karena terdapat peluang terjadinya rekombinasi kromosom yang berasal dari kedua tetua. Sumber keragaman lain yaitu mutasi gen yang terjadi secara alami, di mana frekuensi kejadiaanya relatif rendah. Namun dalam skala masa evolusi perkembangan berbagai jenis ternak evolusi merupakan keragaman yang cukup penting, karena mutasi
9 tersebut dapat diwariskan. Hal tersebut dikarenakan hanya terjadi pada sel germinal yang terdapat pada ovarium dan testis (Martojo, 1992; Jusuf, 2001; King, 2002b).
Keanekaragaman pada berbagai populasi ternak yang ada pada saat ini merupakan akumulasi dari mutasi-mutasi yang terjadi selama evolusi pada masa lampau (Martojo, 1992). Keragaman genetik dapat dilihat dengan menggunakan penanda morfologi dan penanda molekuler (King, 2002a).
Penanda morfologi merupakan penanda yang telah banyak digunakan dalam program genetika dasar maupun dalam program praktis pemuliaan, karena penanda ini paling mudah untuk diamati dan dibedakan. Namun, penanda ini memiliki beberapa kelemahan dalam aplikasi lapang yaitu ketelitian dan ketepatan penentuan mutu genetik hewan penanda morfologi sangat rendah. Dengan demikian untuk kegiatan pemuliaan tidak cukup hanya berdasarkan pada informasi karakteristik morfologi saja, tetapi dengan kemajuan ilmu pengetahuan saat ini, maka dapat digunakan alternatif penanda lain yaitu penanda molekuler yang telah relatif lebih mudah untuk dikerjakan (Muladno, 2006). Keragaman nukleotida merupakan parameter yang akurat untuk menggambarkan keragaman genetik. Unsur positif dengan menggunakan keragaman nukleotida tidak tergantung besar sampel dan panjang DNA (Hartl dan Clark, 1989).
Sekarang telah ada beberapa penanda DNA untuk menganalisis latar belakang genetik hibrida pada sapi (Nijman et al., 2003) yaitu penanda mtDNA. Penanda mtDNA menunjukkan introgresi melalui silsilah maternal (Randi, 2000), yang pada sapi menunjukkan sejarah kekerabatannya (Nijman et al., 2003). Penelitian Lai et al. (2005) berdasarkan pengujian keragaman sekuen pada daerah kontrol DNA mitokondria yaitu daerah D-loop menunjukkan bahwa sapi asli Cina merupakan keturunan dari maternal sapi Asia Timur.
DNA Mitokondria
Material DNA yang secara umum digunakan dalam analisa genetik yaitu DNA yang berasal dari inti, tetapi untuk organisme eukariot sumber DNA dapat pula diperoleh dari organel-organel sitoplasmik. Salah satu organel yang dapat menjadi sumber bahan genetik adalah mitokondria (Duryadi, 1994; King, 2002a). Mitokondria adalah organel yang bertanggung jawab di dalam metabolisme aerobik pada sel-sel eukariot. Mitokondria mempunyai molekul DNA tersendiri dengan
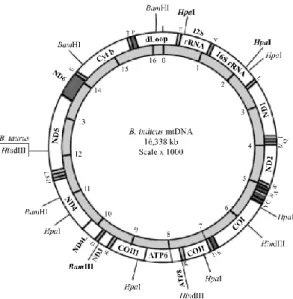
10 ukuran kecil dan sederhana yang memiliki susunan yang berbeda dengan DNA inti (Randi, 2000). Ukuran dari DNA mitokondria pada sapi yaitu sebesar 16.338 pasang basa (Duryadi, 1994).
Sekitar 1% dari material genetik organisme eukariot terdapat dalam mitokondria yang dibentuk dari untai-ganda sirkular yang mengandung 2-10 salinan molekul DNA. DNA mitokondria memiliki fungsi penting antara lain sebagai penyandi 2 RNA ribosom (RNA ribosom besar 16S dan kecil 12S), 22 molekul RNA tranfer, dan 13 protein (tujuh sub unit NADH dehidrogenase (kompleks I), sitokrom
b kompleks III, tiga sub unit sitokrom oksidase (kompleks IV), dan dua sub unit ATP
sintase) yang berperan penting dalam rantai respiratorik (Randi, 2009). Adapun genom mitokonria Bos indicus dapat dilihat pada Gambar 5.
Gambar 5. Genom Mitokondria pada B. indicus
Sumber : Anderson et al. (1982)
Kelebihan menggunakan DNA mitokondria sebagai penanda genetik dalam studi variabilitas genetik intraspesifik (inter populasi) dapat memberikan informasi secara kualitatif maupun kuantitatif. Hal tersebut dikarenakan laju mutasi DNA mitokondria lebih tinggi dibandingkan DNA nukleus yaitu 5-10 kali lebih sering (Randi, 2000). Kelebihan tersebut menyebabkan mtDNA berevolusi sangat cepat, sehingga dapat digunakan untuk melacak kejadian yang relatif baru seperti pada studi hibridisasi alami antara dua sub spesies seperti hibridisasi pada Bos javanicus dan
11 DNA mitokondria memiliki sifat khusus yaitu diturunkan melalui induk tanpa mengalami rekombinasi. Sifat tersebut sehingga dapat digunakan untuk suatu rekonstitusi historik dari genealogi matrilinier suatu spesies maupun antar populasi. Studi keragaman genetik interspesifik berdasarkan perbedaan dan persamaan mtDNA dapat menghasilkan rekonstruksi filogenik dari beberapa spesies yang satu terhadap yang lain (Duryadi, 1994).
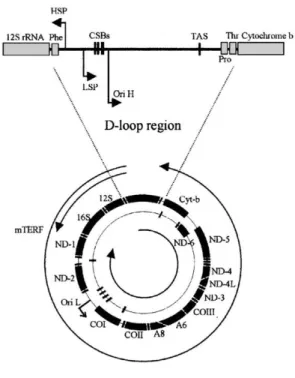
Kajian keragaman genetik yang berdasarkan DNA mitokondria saat ini sangat berkembang karena DNA mitokondria mempunyai jumlah turunan yang tinggi (high copy number), mempunyai jumlah salinan sebesar 103-104 molekul DNA mitokondria/sel somatik, tergantung jaringan dan status fisiologis. Ukuran DNA mitokondria kecil sehingga dapat dipelajari secara utuh. Daerah DNA mitokondria paling bervariasi yaitu daerah D-loop (Randi, 2000). D-loop DNA mitokondria adalah control region, yaitu tempat yang mengatur replikasi dan transkripsi mtDNA yaitu awal dari replikasi rantai berat (Ho). Dinamakan D-loop karena pada fragmen tersebut terdapat fragmen DNA dengan sruktur 3-rantai (membentuk hairpin), terbentuk akibat terciptanya rantai berat (H-strand) yang menggantikan rantai induk dan membentuk struktur tripleks D-loop (3-strand) (Clayton, 1992).
Gambar 6. Skema DNA Mitokondria Daerah D-loop
12 Daerah yang memb)entuk hairpin/D-loop berdekatan dengan gen tRNAphe dan terdapat promotor (Heavy Strand Promotor/HSP dan Light Strand Promotor/ LSP) yang berfungsi sebagai transkripsi genom mitokondria juga terdapat daerah OH (Origin of Replication) untuk rantai berat yang berfungsi awal replikasi (Clayton, 1992). Daerah D-loop yang hipervariabel (mempunyai variasi basa yang cukup tinggi) terletak di luar segmen yang mempunyai fungsi transkripsi dan replikasi tersebut (Wood dan Phua, 1996), sehingga dapat digunakan untuk mengetahui silsilah dari suatu ternak dan hubungan kekerabatan (filogenetik) (Mannen et al., 1998). Daerah D-loop mtDNA disajikan pada Gambar 6.
Kekerabatan Sapi di Indonesia
Berdasarkan ukuran tubuh sapi-sapi asli Indonesia yaitu sapi Bali, Madura, Aceh dan Padang memiliki kesamaan ukuran tubuh. Variasi dari sapi tersebut yaitu mempunyai warna bulu yang berbeda, karena sapi Padang memiliki variasi bulu yang beragam. Sementara pada sapi Madura betina memiliki ukuran tubuh yang lebih besar dari pada sapi Aceh dan sapi Padang, namun lebih kecil daripada sapi Bali. Apabila dibandingkan dengan sapi asli yang terdapat di Asia Tenggara seperti Thailand dan Malaysia Barat, sapi-sapi Indonesia yang terdapat di Sumatra (sapi Aceh dan sapi Padang) memiliki hubungan yang dekat. Sementara sapi Madura sangat dekat dengan sapi asli Thailand, sedangkan ukuran tubuh sapi asli Filipina dan Taiwan memiliki kesamaan dengan sapi Bali pada beberapa bagian (Otsuka et al., 1980).
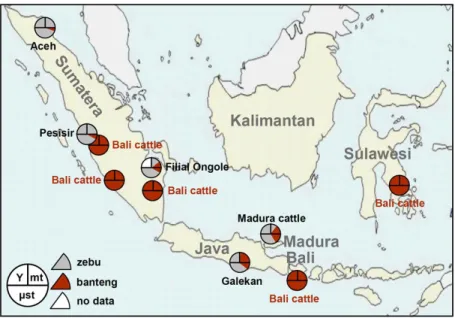
Berdasarkan penelitian Hayashi et al. (1980) ukuran tengkorak sapi Aceh mempunyai lebar dan tinggi tengkorak yang lebih pendek dibanding panjang tengkorak sapi tersebut, dan bagian cerebral tengkorak lebih besar dibanding bagian wajah, tingkat keseragaman sapi Aceh juga lebih rendah dari pada sapi Bali. Sementara sapi Madura mempunyai lebar dan tinggi tengkorak yang memiliki ukuran diantara sapi Aceh dan sapi Bali, sapi Madura juga memiliki tingkat keseragaman yang rendah seperti pada sapi Aceh. Berbeda dengan sapi Bali yang lebih menyerupai banteng dari pada sapi Aceh dan sapi Madura. Namun tidak ditemukan tonjolan intercornual di tengkorak sapi Bali. Karakteristik ini tidak ditemukan pada sapi asli lain maupun banteng. Sebaran komposisi genetik populasi sapi di beberapa wilayah Indonesia disajikan pada Gambar 7.
13 Gambar 7. Komposisi Genetik Populasi Sapi di Beberapa Wilayah di Indonesia
Sumber : Mohamad et al. (2009)
Analisis hubungan kekerabatan pada sapi lokal Indonesia berdasarkan sekuen DNA Mitokondria Gen Cytochrome Oxidase I (Gen COI) menunjukkan bahwa sapi Bali terpisah dari pengelompokan keempat bangsa sapi Indonesia lainnya. Sapi Aceh, Pesisir, PO, dan Madura terletak dalam kelompok yang sama dengan Bos
indicus. Dari kedua pengelompokan tersebut menunjukkan bahwa sapi Bali
mempunyai asal-usul tersendiri sedangkan keempat sapi lain berasal dari kelompok sapi Zebu (Gambar 8) (Febriana, 2011). Hal tersebut juga didukung oleh pendapat Mohamad et al. (2009) melalui analisis DNA mitokondria, Y-kromosom, dan mikrosatelit (Gambar 9).
Gambar 8. Konstruksi Pohon Filogenik Sapi Asli dan Sapi Lokal Indonesia Berdasarkan DNA Daerah CO I
14 Gambar 9. Konstruksi Pohon Filogenik Sapi Asli dan Sapi Lokal Indonesia
15 MATERI DAN METODE
Lokasi dan Waktu
Penelitian dilaksanakan di Laboratorium Genetika Molekuler Ternak, Bagian Pemuliaan dan Genetika Ternak, Fakultas Peternakan, Institut Pertanian Bogor. penelitian ini dilakukan pada bulan Juni - Agustus 2012
Materi
Sampel Darah
Sampel darah sapi yang digunakan yaitu sapi asli dan sapi lokal sebanyak 18 sampel dari lima bangsa sapi yang yaitu sapi Bali, Madura, Pesisir, Aceh, dan PO. Jumlah masing-masing sampel yang digunakan dalam penelitian ini disajikan pada Tabel 1.
Tabel 1. Jumah Sampel Darah Ternak Sapi Indonesia
Ternak n Asal
Sapi Bali 2
4
BIB Sapi Bali, Bali BPTU Sapi Bali, Bali
Sapi Madura 2
2
Sapudi
Kab. Sampang
Sapi Aceh 2 Kab. Aceh Besar
Sapi Pesisir 2 Kab. Pesisir Selatan
Sapi PO 2
2
Kab. Kebumen
Kandang A Fakultas Peternakan IPB
Total 18
Keterangan : n = jumlah individu sampel Alat dan Bahan
Bahan yang digunakan pada pengambilan sampel darah adalah alkohol 70%, es, dan kapas. Alat yang digunakan antara lain jarum venojact, tabung vacutainer 10 ml, dan termos. Sampel darah yang digunakan merupakan koleksi dari laboratorium Pemuliaan dan Genetika Molekuler, Bagian Pemuliaan dan Genetika Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan.
16 Amplifikasi daerah D-loop dilakukan melalui Polymerase Chain Reaction (PCR). Bahan-bahan yang digunakan pada proses amplifikasi DNA adalah adalah DW, sampel DNA, 10 × buffer, MgCl2, primer forward dan reverse, enzim taq,dan
dNTPs. Alat yang digunakan adalah satu set pipet mikro, alat sentrifugasi, tabung PCR, mesin PCR, vortex, dan freezer.
Bahan yang digunakan untuk elektroforesis terdiri atas gel elektroforesis,
loading dye, dan marker 100 pb. Bahan-bahan yang digunakan untuk membuat gel
elektroforesis yaitu agarose, 0,5 × TBE, Ethidium Bromide. Bahan-bahan yang digunakan untuk membuat larutan loading dye yaitu bromothymol blue, xylene
cyanol, gliserol. Sementara, alat yang digunakan antara lain microwave, stearer, magnet stearer, gelas ukur, tabung erlenmeyer, gel tray, pencetak untuk sumur
(comb), power supply 100 volt, gelas ukur, pipet makro dan mikro, serta
UV-Transiluminator. Buffer elektroda yang digunakan terdiri dari Tris, Glycine dan aquadestilata.
Primer DNA Mitokondria Daerah D-loop
Primer merupakan molekul oligonukleotida yang berukuran pendek (sekitar 18-24 pasang basa) yang akan menempel pada DNA cetakan di tempat spesifik. Pasangan primer yang digunakan untuk mengamplifikasi mtDNA daerah D-loop sapi dengan runutan primer forward5′-TAGTGCTAATACCAACGGCC-3′ dan primer
reverse5′-AGGCATTTTCAGTGCCTTGC-3′ (Hassan et al., 2009).
Prosedur
Pengambilan Sampel Darah
Pengambilan sampel darah sapi sebanyak sekitar 3 ml melalui vena jugularis dengan menggunakan venojact lalu segera dimasukkan ke dalam tabung vaccutainer yang dimasukkan kedalam termos es dan disimpan dalam suhu 4 ºC.
Isolasi DNA
Isolasi DNA dilakukan dari sampel darah dengan menggunakan Genomic
DNA mini kit Geneaid (Lampiran 2) yang dimodifikasi untuk penggunaan sampel
17 Amplifikasi DNA Mitokondria Daerah D-loop
Amplifikasi lengkap fragmen D-loop DNA mitokondria menggunakan primer seperti yang digunakan Hassan et al. (2009), yaitu primer Forward
5′-TAGTGCTAATACCAACGGCC-3′ dan primer Reverse
5′-AGGCATTTTCAGTGCCTTGC-3′ dengan panjang produk PCR 1145 bp (Gambar 10). Sekuen D-loop mtDNA diperoleh dari NBCI (http://www.ncbi.nlm.nih.gov/). Nomor akses standardnya adalah Bos indicus dengan kode akses AY126697.
Keterangan :
: Primer forward : Primer reverse
Warna hijau menunjukkan basa nukleotida yang berbeda
Volume setiap reaksi PCR sebanyak 25 µl yang terdiri atas 2 µl sampel DNA; 0,3 µl primer forward dan reverse; 0,2 µl dNTPs; 1 µl MgCl2; 2,5 µl 10 ×
buffer0,1 µl enzim Taq Polymerase; dan 18,9 µl destilation water (DW) yang
dilakukan dengan menggunakan mesin PCR model Applied Biosystems. Kondisi PCR yang digunakan adalah: predenaturasi pada suhu 95 °C selama lima menit, dilanjutkan dengan siklus utama yaitu tahap denaturasi pada suhu 95 °C selama 30 detik, tahap penempelan primer (annealing) pada suhu 60 °C selama 45 detik, dan tahap polimerasi (extension) pada suhu 72 °C selama satu menit diulang sebanyak 35 siklus. Reaksi PCR diakhiri dengan polimerasi (final exstension) pada suhu 72 °C selama lima menit.
5’−TAGTACTAATACCAACAGCCGGCACAGTTGAAAACAAATTACTAAAATGAAGACAGGTCTTTGTAGTACATCTAA TATACTGGTCTTGTAAACCAGAGAAGGAGAACAACTAACCTCCCTAAGACTCAAGGAAGAAACTGTAGTCTCACCGTC AACCCCCAAAGCTGAAGTTCTATTTAAACTATTCCCTGAACACTATTAATATAGTTCCATAAATGCAAAGAGCCTTAT CAGTATTAAATTTATCAAAAATCCCAATAACTCAACACAGAATTTGCACCCTAACCAAATATTACAAACACCACTAGC TAACATAACACGCCCATACACAGACCACAGAATGAATTACCCAGGCAAGAGGTAATGTACATAACATTAATGTAATAA AGACATGATATGTATATAGTACATTAAATTATATACCCCATGCATATAAGCAAGTACATGATCTCTATAATAGTACAT AATACATACAATTATTAATTGTACATAGTACATTATATCAAATCCATCCTCAACAACATATCTACTATATACCCCTTC CACTAGATCACGAGCTTAATTACCATGCCGCGTGAAACCAGCAACCCGCTAAGCAGAGGATCCCTCTTCTCGCTCCGG GCCCATAGACCGTGGGGGTCGCTATTTAATGAATTTTACCAGGCATCTGGTTCTTTCTTCAGGGCCATCTCATCTAAA GTGGTCCATTCTTTCCTCTTAAATAAGACATCTCGATGGACTAATGACTAATCAGCCCATGCTCACACATAACTGTGC TGTCATACATTTGGTATTTTTTTATTTTGGGGGATGCTTGGACTCAGCTATGGCCGTCAAAGGCCCCGACCCGGAGCA TCTATTGTAGCTGGACTTAACTGCATCTTGAGCACCAGCATAATGATAGGCATGGGCATTACAGTCAATGGTCACAGG ACATAAATACATTATATATCCCCCCCTTCATAAAAACCTCCCCCTTAAATATTCACCACCACTTTTAACAGACTTTTC CCTAGATACTTATTTAAATTTTCCACACTTTCAATACTCAATTTAGCACTCCAAACAAAGTCAATATATAAACGCAGG CCCCCCCCCCCCCCGTTGATGTAGCTTAACCCAAAGCAAGGCACTGAAAATGCCT-3’
18 Elektroforesis
Elektroforesis menggunakan agarose 1,5% dilakukan dengan cara 0,45 g agarose dilarutkan dalam larutan 0,5 x TBE sebanyak 30 ml lalu dipanaskan dalam
microwave selama sekitar lima menit kemudian dilakukan stirer dan ditambahkan
2,5 µl EtBr. Larutan dituang ke dalam cetakan gel, diberi sisiran pada tepi gel dan gel dibiarkan mengeras. Sisir dicabut setelah gel mengeras sehingga terbentuk sumur-sumur.
Produk PCR sebanyak lima µl dicampurkan dengan loading dye sebanyak satu µl dengan menggunakan mikropipet lalu dimasukkan dalam sumur-sumur gel dan satu sumur gel dimasukkan marker sebanyak 2 µl yang digunakan sebagai penanda. Kemudian gel ditempatkan ke dalam gel tray elektroforesis yang sudah berisi larutan buffer dan dialiri listrik 100 volt selama 30 menit, molekul DNA yang bermuatan negatif pada pH netral akan bergerak (bermigrasi) ke arah positif. Gel agarose yang telah selesai dilakukan elektoforesis kemudian diambil untuk melihat panjang pita DNA dengan menggunakan sinar ultraviolet dalam trans illuminator. Panjang pita DNA dapat diketahui dengan cara menarik garis lurus masing-masing pita sampel DNA dengan posisi pita DNA marker.
Perunutan Produk PCR (Sekuensing)
Pebacaan sekuen menjadi alat penting dan utama dalam biologi molekular karena dapat mengetahui komposisi nukleotida dan asam amino suatu gen, juga digunakan untuk menganalisis kekerabatan dan jalur evolusi (Albert et al., 1994). Produk PCR daerah D-loop dari penelitian ini berupa pita tunggal yang berukuran 1145 pb. Analisis perunutan dilakukan di Macrogen, Korea Selatan.
Rancangan dan Analisis Data
Jarak Genetik dan Pohon Filogeni
Sekuen DNA dilakukan manual editing dengan menggunakan BioEdit. Runutan nukleotida yang telah diedit disejajarkan dengan runutan baku nukleotida B.
indicus dari Genbank (kode akses AY126697; Mirreti et al., 2002). Proses
pensejajaran (alignment) dilakukan dengan menggunakan program Clustal W yang terdapat pada program MEGA 5.0 (Molecular Evolutionary Genetics Analysis 5.0;
19 Tamura et a., 2007). Perhitungan komposisi basa nukleotida, jarak genetik dan konstruksi pohon filogeni dilakukan dengan menggunakan program MEGA 5.0. Jarak genetik digunakan untuk melihat kedekatan hubungan genetik antara sampel yang dianalisis. Nilai jarak genetik diperoleh dengan membagi jumlah nukleotida yang berbeda degan jumlah total nukleotida. Perhitungan pairwise distance digunakan untuk melihat jarak rata-rata p-distance dari basa nukleotida daerah
D-loop. Pohon filogeni direkrontruksi dengan menggunakan metode bootsraped Neighboor-Joining (NJ) dengan 1000 kali pengulangan (Nei dan Kumar, 2000).
20 HASIL DAN PEMBAHASAN
Amplifikasi Daerah D-loop
Amplifikasi daerah D-loop DNA mitokondria (mtDNA) pada sampel DNA sapi Bali, Madura, Pesisir, Aceh, dan PO dilakukan dengan menggunakan mesin PCR Applied Biosystem. Hasil optimal fragmen D-loop mtDNA berhasil dilakukan amplifikasi pada kondisi annealing dengan suhu 60 °C selama 45 detik, dan diperoleh produk PCR dengan panjang 1145 bp (Gambar 11).
M B1 B2 B3 M1 M2 P1 P2 (+) Keterangan : M (marker) = ladder 100 bp B = Bali M = Madura P = PO
Gambar 11. Hasil Ektroforesis Produk PCR Daerah D-loop mtDNA
Keberhasilan amplifikasi daerah D-loop sangat ditentukan dengan kondisi penempelan primer pada DNA genom, selain faktor-faktor bahan pereaksi PCR dan mesin PCR yang digunakan. Weissensteiner (2004) menyatakan bahwa suhu penempelan (annealing) primer yang sesuai merupakan hal yang paling penting untuk keberhasilan PCR disamping kosentrasi MgCl2. Berdasarkan hasil amplifikasi
yang dilakukan oleh Hassan et al. (2009) penempelan (annealing) primer daerah
D-loop mtDNA pada kerbau sungai dengan primer yang sama, yaitu pada suhu 59 °C
selama 45 detik akan menghasilkan produk PCR yang baik. Berbeda pada suhu
annealing yang optimal pada penelitian ini, yaitu lebih tinggi dibandingkan dengan
1000 bp bp
21 suhu annealing yang digunakan oleh Hassan et al. (2009) yaitu 60 °C selama 45 detik. Hal tersebut dikarenakan melting temperature (Tm) akan turun sebesar 5 °C setiap terdapat 1% ketidakcocokan pada basa dalam untai ganda (Carter, 2000a).
Amplifikasi daerah D-loop mtDNA ini dilakukan dengan menggunakan pasangan primer forward 5′-TAGTGCTAATACCAACGGCC-3′ dan primer reverse 5′-AGGCATTTTCAGTGCCTTGC-3′ sesuai dengan desain yang digunakan oleh Hassan et al. (2009), yaitu primer yang digunakan untuk amplifikasi daerah D-loop kerbau sungai. Hasil analisis menunjukkan bahwa primer tersebut dapat digunakan untuk mengamplifikasi D-loop mtDNA pada sapi, yang menunjukkan bahwa primer yang didesain memiliki high similarity. Hal tersebut diduga karena sapi dan kerbau masih berkerabat dekat terdapat dalam satu rumpun yaitu Bovini (Lenstra dan Bradley, 2006), yang menyebabkan keduanya masih berkerabat secara taksonomi, sehingga memiliki kemiripan basa nukleotida yang tinggi. Namun, pada primer
forward terdapat dua nukleotida yang berbeda antara sapi dan kerbau. Perbedaan
nukleotida tersebut terdapat pada nukleotida ke-5 dan ke-17, pada kedua urutan tersebut sapi memiliki nukleotida A dan pada kerbau sungai memiliki nukleotida G. Hal tersebut diduga karena sapi dan kerbau terdapat pada genus yang berbeda, yaitu
Bos (sapi) dan Bubalus (kerbau) (Lenstra dan Bradley, 2006). Dawkin (2000)
menyatakan bahwa secara taksonomi hubungan kekerabatan (filogenetik) akan memisah ketika terjadi perbedaan atau perubahan dalam basa nukleotida, semakin banyak perbedaan tersebut maka hubungan kekerabatan akan semakin jauh.
Berdasarkan runutan genom utuh DNA mitokondria Bos indicus (sapi Nellore) dengan kode akses (AY126697) dari GenBank. Produk PCR hasil amplifikasi pasangan primer forward 5′-TAGTGCTAATACCAACGGCC-3′ dan primer reverse 5′-AGGCATTTTCAGTGCCTTGC-3′ menghasilkan pita tunggal yang jelas berukuran 1145 pb (Gambar 11). Munculnya satu pita ini menunjukkan bahwa pasangan primer yang digunakan bersifat spesifik hanya menempel pada posisi yang diharapkan (pada kondisi annealing yang digunakan) (Ratnayani et al, 2007). Hasil amplifikasi tersebut terdiri atas 53 bp fragmen gen CYT B pada posisi ke-1087 sampai dengan 1140 (15604-15656), 69 bp tRNAThr pada posisi ke-1 sampai dengan 69 (15661-15729), 66 bp tRNAPro pada posisi ke-1 sampai dengan 66 (15729-15794), 913 bp fragmen utuh daerah D-loop pada posisi 1 sampai dengan
22 913 (15795-16341, 1-366), dan 41 bp fragmen tRNAPhe pada posisi basa ke-1 sampai dengan 41 (367-407) (Lampiran 3). Ilustrasi letak penempelan pasangan primer tersebut pada daerah D-loop sapi penelitian disajikan pada Gambar 12.
Hasil amplifikasi pada produk PCR primer forward menempel pada posisi basake-88 sampai dengan 108 (15604-15623) pada bagian CYT B dan primer
reverse menempel pada tRNAPhe posisi ke-22 sampai dengan 41 (367-407) (Lampiran 3). Ilustrasi letak penempelan pasangan primer forward dan primer
reverse pada daerah D-loop sapi terdapat dalam Gambar 12.
Gambar 12. Sketsa Letak Penempelan Primer Forward dan Reverse untuk Mengamplifikasi pada Daerah D-loop Sapi Bali, Madura, Pesisir, Aceh, dan PO
Keragaman D-loop
Komposisi Basa Nukleotida
Setelah dilakukan sekuensing pada produk PCR dari dua arah yaitu primer
forward dan primer reverse didapatkan hasil sekuen sepanjang 1145 bp dengan
sekuen standardnya adalah sekuen Bos indicus dari GenBank (kode akses AY126697; Mirreti et al., 2002). Sekuen tersebut terdiri dari sekuen D-loop sepanjang 913 bp.
Analisis keragaman runutan nukleotida dilakukan setelah runutan DNA sapi Bali, Madura, Aceh, Pesisir, dan PO disejajarkan dengan Cluscal W dengan acuan utama pada runutan nuleotida sapi B. indicus (Nellore) dari GenBank (kode akses AY126697; Mirreti et al., 2002). Hasil pensejajaran sekuen tersebut diperoleh bahwa jumlah basa nukleotida pada setiap individu sapi yang diteliti tidak sama (Gambar 11). Hal tersebut dikarenakan terdapat mutasi yang menghilangkan (delesi)
Primer F
20 bp tRNA Thr tRNA Pro Primer R 20 bp
69 bp 66 bp
Teramplifikasi 1145 bp
( CYT B 53 bp, 69 bp tRNAThr,66 bp tRNAPro, D-loop utuh, 41 bptRNAPhe)
CYT B 53 bp
tRNAPhe 41 bp D-loop (GenBank) 913 bp
23 dan menyisipkan (insersi) beberapa nukleotida pada sapi Bali, Madura, Aceh, Pesisir, dan PO yang setiap individu jumlahnya tidak sama (Lampiran 4).
Hasil pensejajaran runutan nukleotida sapi Bali, Madura, Aceh, Pesisir, dan PO sepanjang 584 bp yang dapat dianalisis dengan acuan runutan B. indicus (Nellore) dari GenBank, maka setiap bangsa sapi yang diteliti memiliki rataan komposisi basa nukleotida yang berbeda (Tabel 2). Perbedaan jumlah basa nukleotida daerah D-loop parsial untuk masing-masing sapi dapat dilihat pada Lampiran 5.
Tabel 2. Rataan Komposisi Nukleotida Daerah D-loop Parsial Sapi Bali, Madura, Aceh, Pesisir, PO Setelah Disejajarkan dengan B. indicus Dari Genbank (Ukuran 584 bp) Bangsa Sapi % n T(U) C A G A+T C+G B. indicus 3 27,3 24,2 35,7 12,8 63,0 37,0 B. taurus 4 28,2 23,5 34,9 13,4 63,1 36,9 Bali 6 26,9 25,3 34,8 13,0 61,7 38,3 Madura 4 26,9 25,1 34,9 13,1 61,8 38,2 Aceh 2 27,3 24,2 35,7 12,8 63,0 37,0 Pesisir 2 26,7 24,4 35,6 13,3 62,3 37,7 PO 4 27,0 25,4 34,8 12,9 61,8 38,3
Keterangan : n = jumlah individu sampel
Rataan nukleotida T daerah D-loop tertinggi sampai terendah berturut-turut dimiliki oleh sapi Aceh 27,3%; PO 27,0%; Bali dan Madura masing-masing 26,9%; serta Pesisir 26,7%. Rataan nukleotida C daerah D-loop tertinggi sampai terendah berturut-turut dimiliki oleh sapi PO 25,4%; Bali 25,3%; Madura 25,1%; Pesisir 24,4%; dan Aceh 24,2%. Rataan nukleotida A daerah D-loop tertinggi sampai terendah berturut-turut dimiliki oleh sapi Aceh 35,7%; Pesisir 35,6%; Madura 34,9%; serta Bali dan PO masing-masing 34,8%. Rataan nukleotida G daerah D-loop tertinggi sampai terendah berturut-turut dimiliki oleh sapi Pesisir 13,3%; Madura 13,1%; Bali 13,0%; PO 12,9%; dan Aceh 12,8% (Tabel 2). Perbedaan komposisi
24 nukleotida A, T, G, dan C menunjukan perbedaan komposisi asam amino yang dikandungnya (Ridley, 1991).
Gambar 13. Frekuensi A+T dan G+C Daerah D-loop mtDNA Parsial Berukuran 584 bp pada Sapi Bali, Madura, Aceh, Pesisir, dan PO
Nukleotida A+T pada semua sapi yang diteliti yaitu sapi Bali, Madura, Pesisir, Aceh, dan PO memiliki rataan yang lebih tinggi dibandingkan dengan rataan nukleotida G+C. Rataan nukleotida A+T dari paling tinggi hingga paling rendah berturut-turut yaitu Aceh 63,0%; Pesisir 62,3%; Madura dan PO masing-masing 61,8%; serta Bali 61,7%. Rataan nukleotida G+C dari paling tinggi hingga paling rendah berturut-turut yaitu Bali dan PO masing-massing 38,3%; Madura 38,2%, Pesisir 37,7%; dan Aceh 37,0%. Keragaman frekuensi basa nukleotida pada sapi Bali, Madura, Aceh, Pesisir, dan PO tersebut dikarenakan mtDNA memiliki laju mutasi lima sampai sepuluh kali lebih cepat dari pada DNA inti (Wilson et al., 1985) dan pada daerah kontrol memiliki kecepatan evolusi 10-20 kali lebih cepat dibandingkan dengan daerah mtDNA lainnya (Randi, 2000). Komposisi basa nukleotida A+T memiliki frekuensi yang lebih tinggi dibadingkan dengan komposisi G+C pada hasil penelitian ini, karena pada daerah tersebut merupakan daerah
noncoding. Hal tersebut diduga penyebabkan daerah noncoding memiliki laju
evolusi lebih tinggi.
Komposisi G+C dari mulai tertinggi hingga terendah ditunjukkan berturut-turut dimiliki oleh sapi Bali 38,3%; PO 38,3%; Madura 38,2%; Pesisir 37,7%; dan
0 10 20 30 40 50 60 70
Bali Madura Aceh Pesisir PO
F re kue ns i Bangsa Sapi A+T C+G
25 Aceh 37,0%. Pada bakteri yang mampu hidup pada suhu termofilik memiliki komposisi G+C yang tinggi (Yuwono, 2009). Hal tersebut karena ikatan antara G+C lebih stabil dari pada ikatan pada A+T. Oleh karena itu, sapi-sapi Indonesia terutama pada sapi Bali memiliki kemampuan ketahanan yang lebih baik pada lingkungan tropis dibanding sapi-sapi Bos indicus maupun Bos taurus.
Sekuen Minisatelit
Minisatelit DNA merupakan urutan sekuen berulang yang berurutan (Nakamura et al., 1987). Minisatelit ini terjadi karena pada DNA tersebut terdapat pasangan basa nukleotida dalam satu untaian DNA yang sama (intra-strand pairing) (Yuwono, 2009). Subtipe ini terdiri atas DNA yang berukuran kecil hingga sedang dari unit yang berulang. Secara umum terdiri atas panjang sekuen berulang yang polimorfik. Pengulangan tersebut dapat terjadi 1%-60% per genom, dengan salinan setiap sekuen individu berkisar antara 1-106 per genom. Sekuen berulang yang memiliki frekuensi tinggi biasanya ditemukan pada DNA noncoding (Carter, 2000b).
Berdasarkan hasil analisis sekuen dari seluruh sapi yang diteliti telah disejajarkan dengan menggunakan metode Cluscal W, maka ditemukan sekuen berulang atau minisatelit yang memiliki panjang 22 bp. Hal tersebut sesuai dengan pendapat Carter (2000b) yang menyatakan bahwa panjang sekuen ulangan berurutan antara 9 bp sampai 250 bp.
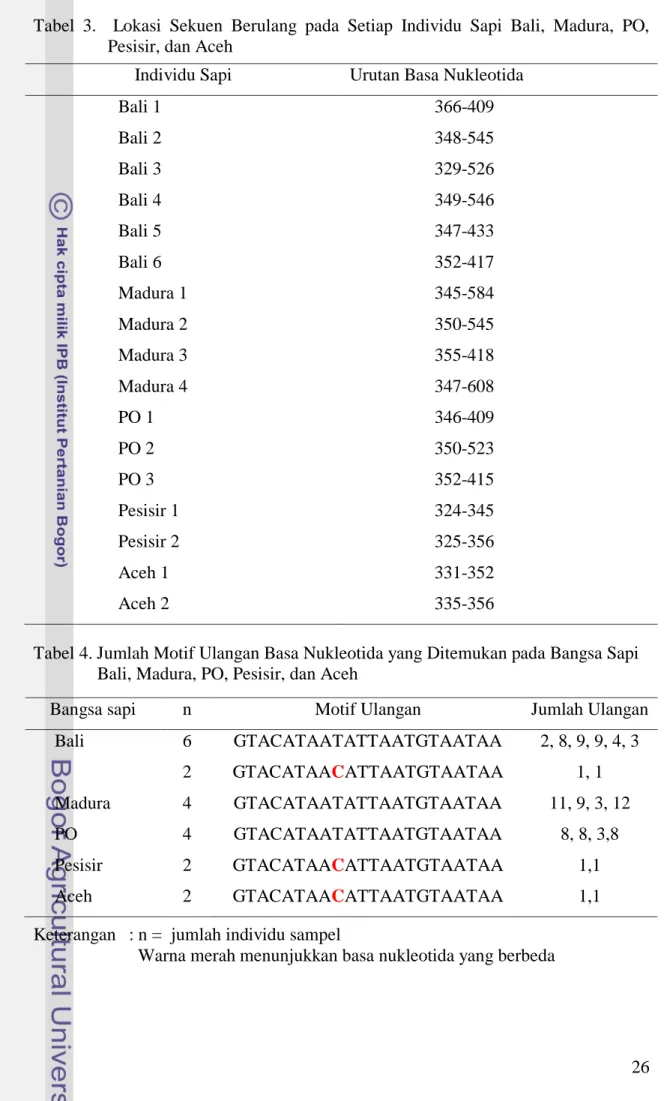
Motif ulangan yang ditemukan pada penelitian ini terdapat antara basa nukleotida ke-324 sampai dengan 608. Sekuen berulang pada sapi Bali terletak pada urutan basa nukleotida ke-329 sampai dengan 546, sapi Madura urutan basa nukleotida ke-345 sampai dengan 608, sapi PO urutan basa nukleotida ke-346 sampai dengan 523, sapi Pesisir urutan basa nukleotida ke-324 sampai dengan 356, dan sapi Aceh urutan basa nukleotida ke-331 sampai dengan 356 (Tabel 3). Alves et
al. (2009) juga menemukan sekuen berulang pada daerah D-loop. Sekuen tersebut
terdapat pada urutan basa nukleotida ke-380 sampai dengan 500, dari bagian yang dilakukan amplifikasi mulai dari basa nukleotida ke-230 sampai dengan 520.
26 Tabel 3. Lokasi Sekuen Berulang pada Setiap Individu Sapi Bali, Madura, PO,
Pesisir, dan Aceh
Individu Sapi Urutan Basa Nukleotida
Bali 1 366-409 Bali 2 348-545 Bali 3 329-526 Bali 4 349-546 Bali 5 347-433 Bali 6 352-417 Madura 1 345-584 Madura 2 350-545 Madura 3 355-418 Madura 4 347-608 PO 1 346-409 PO 2 350-523 PO 3 352-415 Pesisir 1 324-345 Pesisir 2 325-356 Aceh 1 331-352 Aceh 2 335-356
Tabel 4. Jumlah Motif Ulangan Basa Nukleotida yang Ditemukan pada Bangsa Sapi Bali, Madura, PO, Pesisir, dan Aceh
Bangsa sapi n Motif Ulangan Jumlah Ulangan
Bali 6 2 GTACATAATATTAATGTAATAA GTACATAACATTAATGTAATAA 2, 8, 9, 9, 4, 3 1, 1 Madura 4 GTACATAATATTAATGTAATAA 11, 9, 3, 12 PO 4 GTACATAATATTAATGTAATAA 8, 8, 3,8 Pesisir 2 GTACATAACATTAATGTAATAA 1,1 Aceh 2 GTACATAACATTAATGTAATAA 1,1
Keterangan : n = jumlah individu sampel
27 Semua sapi yang diteliti yaitu sapi Bali, Madura, Pesisir, Aceh, dan PO ditemukan motif ulangan. Motif ulangan yang ditemukan pada penelitian ini yang terdapat di daerah D-loop DNA mitokondria yaitu sekuen dengan basa nukleotida GTACATAATATTAATGTAATAA dan GTACATAACATTAATGTAATAA yang masing-masing memiliki panjang 22 bp (Tabel 4).
Motif ulangan dengan sekuen GTACATAATATTAATGTAATAA ditemukan pada sapi Bali, Madura, dan PO (Tabel 4). Setiap Bangsa sapi memiliki jumlah motif ulangan yang berbeda-beda. Motif ulangan pada sapi Bali berkisar antara 2-9 ulangan, pada sapi Madura berkisar antara 3-12, dan pada sapi PO berkisar antara 3-8. Alves et al. (2009) menemukan jumlah ulangan daerah D-loop mtDNA pada babi yang memiliki jumlah ulangan antara 18-30 ulangan. Jumlah motif ulangan pada sapi Bali, Madura, Pesisir, dan Aceh disajikan pada Tabel 4.
Motif ulangan lain yang ditemukan pada penelitian ini juga memiliki panjang 22 bp yaitu GTACATAACATTAATGTAATAA ditemukan pada sapi Pesisir, Aceh, Bali 1, dan Bali 2 (Tabel 3). Motif ulangan ini memiliki perbedaan dengan motif ulangan sebelumnya yaitu terdapat satu basa nukleotida yang mengalami subtitusi transisi yaitu pada basa nukleotida ke-9 minisatelit atau pada basa nukleotida yang ke-438 D-loop mtDNA setelah disejajarkan. Transisi tersebut dari basa nukleotida T menjadi C. Motif ulangan ini pada sapi Pesisir, Aceh, dan Bali ditemukan sebanyak satu ulangan.
Hasil motif ulangan tersebut dapat digunakan untuk membedakan bangsa sapi yang berasal dari kelompok sapi Bali dengan kelompok yang lain yaitu B. indicus dan B. taurus. Hal tersebut sesuai dengan pendapat Carter (2000b) dan King (2002c) bahwa motif ulangan dapat digunakan untuk fingerprinting. Carter (2000b) juga menyatakan bahwa motif ulangan dapat digunakan untuk mengestimasi heterozigositas 90%-99% marker genetik yang sangat bervariabel.
Jarak Genetik Sapi Asli dan Sapi Lokal Indonesia
Kedekatan hubungan genetik antara lima bangsa sapi yaitu Bali, Madura, Pesisir, Aceh, dan PO dengan sapi B. indicus dilihat dengan mengukur jarak genetik. Jarak genetik diukur dengan menggunakan analisis Pairwise Distance Calculation yang ditunjukkan dengan matriks perbedaan genetik antara lima bangsa sapi (Bali, Madura, Pesisir, Aceh, dan PO) dan sapi B. indicus yang diambil dari GenBank.
28 Jarak genetik model ini digunakan untuk melihat tingkat subtitusi transisi dan tranversi melalui banyaknya perbedaan nukleotida per pasangan (Abdullah, 2008). Sapi-sapi yang memiliki nilai jarak genetik semakin rendah, maka ternak tersebut memiliki hubungan kekerabatan semakin dekat. Sebaliknya ternak yang memiliki jarak genetik tinggi, maka hubungan kekerabatannya semakin jauh.
Hasil perhitungan nilai jarak genetik antara individu pada lima bangsa sapi berkisar antara 0,000 sampai 0,146. Jarak genetik tertinggi dari sapi yang diteliti yaitu pada sapi Madura 1 dan Pesisir 1 maupun sapi Pesisir 2 yaitu dengan nilai 0,146. Sementara jarak genetik terendah yaitu dengan nilai 0,000 ditemukan pada sapi Aceh 1 maupun sapi Aceh 2 dan B. indicus, antara sapi Pesisir 1 dan sapi Pesisir 2, antara sapi Bali 2 dan sapi Bali 3 maupun sapi Bali 4, antara sapi Madura 2 dan sapi Bali 2, 3 , dan 4, antara sapi Madura 3 dan sapi Bali 5, antara sapi PO 1 dan sapi Bali 5, antara sapi PO 3 dan sapi Bali 5, antara PO 3 dan Madura 5, dan antara PO 1 dan PO 3 (Tabel 6).
Tabel 5. Jarak Genetik Berdasarkan Metode Pairwise Distance Daerah D-loop yang Dilakukan Pengelompokan pada Sapi Bali, Sapi Madura dan Sapi PO dengan Sapi B. indicus dan B.taurus dari Genbank
No. Bangsa 1 2 3 4 5 6 7 1 B. indicus - 2 B. taurus 0,056 - 3 Aceh 0,005 0,055 - 4 Pesisir 0,012 0,061 0,007 - 5 Bali 0,129 0,151 0,128 0,136 - 6 Madura 0,134 0,155 0,133 0,140 0,009 - 7 PO 0,132 0,153 0,131 0,138 0,007 0,009 -
Hal yang sama juga terlihat pada perhitungan jarak genetik yang dilakukan pengelompokan. Jarak genetik tertinggi ditemukan pada sapi Madura dan sapi Pesisir, sedangkan nilai jarak genetik terendah dimiliki oleh sapi Aceh dan B.
indicus. Jarak antar individu antara sapi Bali dengan sapi Bali yang lain berkisar
0,000-0,012; jarak individu antara sapi Madura dengan sapi Madura yang lain berkisar 0,003-0,022; jarak antara individu sapi Pesisir dengan sapi Pesisir yang lain
29 yaitu 0,000; dan jarak antara individu sapi Aceh dengan sapi Aceh yang lain yaitu 0,000.
Nilai pada jarak genetik yang dilakukan pengelompokan berkisar antara 0,005 sampai 0,140. Berdasarkan nilai jarak genetik sapi Aceh memiliki jarak genetik yang lebih tinggi dengan B. taurus yaitu 0,055 bila dibandingkan dengan B. indicus yaitu 0,005, dan nilai jarak genetik sangat tinggi bila dibandingkan dengan sapi Bali yaitu 0, 128. Sementara pada sapi Pesisir juga menyatakan hal yang sama yaitu memiliki jarak genetik yang lebih dekat dengan B. indicus, namun nilai jarak genetik tersebut berbeda. Nilai jarak genetik dengan B. indicus yaitu 0,012; B. taurus yaitu 0,061; dan sapi Bali yaitu 0, 136. Berbeda dengan sapi Madura yang memiliki nilai jarak genetik yang lebih rendah terhadap sapi Bali (0,009), dibandingkan dengan B.
indicus (0,134) dan B. taurus (0,155). Hal serupa juga ditunjukkan pada sapi PO,
yaitu dengan nilai jarak genetik terhadap sapi Bali memiliki nilai yang lebih rendah yaitu 0,007 bila dibandingkan dengan B. indicus (0,132) dan B. taurus (0,153). Nilai-nilai jarak genetik tersebut disajikan pada Tabel 5.
Jarak genetik pada sapi Aceh memiliki nilai yang lebih rendah dengan sapi Pesisir, yang menunjukkan bahwa sapi Aceh dan Pesisir memiliki hubungan yang dekat. Sementara sapi Bali nilai jarak genetik yang rendah dengan sapi Madura dan PO, yang menunjukkan hubungan sapi-sapi tersebut memiliki kedekatan. Kedekatan hubungan beberapa sapi lokal tersebut diduga karena adanya perkawinan tidak acak dan aliran gen yang terjadi akibat letak geografis yang berdekatan antara sapi-sapi tersebut, sehingga akan mengurangi perbedaan antar populasi yang telah terakumulasi akibat seleksi alam maupun genetic drift.
30
Tabel 6. Jarak Genetik Berdasarkan Metode Pairwise Distance Daerah D-loop Setiap Individu pada Sapi Bali, Sapi Madura dan Sapi PO dengan Sapi
B. indicus dan B.taurus dari Genbank
No. Sampel 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 1 Nellore_(GB) - 2 Aceh_1 0,000 - 3 Aceh_2 0,000 0,000 - 4 Pesisir_1 0,007 0,007 0,007 - 5 Pesisir_2 0,007 0,007 0,007 0,000 - 6 Bali_1 0,137 0,137 0,137 0,145 0,145 - 7 Bali_2 0,123 0,123 0,123 0,131 0,131 0,012 - 8 Bali_3 0,121 0,121 0,121 0,128 0,128 0,011 0,000 - 9 Bali_4 0,126 0,126 0,126 0,133 0,133 0,012 0,000 0,000 - 10 Bali_5 0,131 0,131 0,131 0,139 0,139 0,007 0,008 0,005 0,008 - 11 Bali_6 0,131 0,131 0,131 0,139 0,139 0,009 0,010 0,007 0,010 0,002 - 12 Madura_1 0,139 0,139 0,139 0,146 0,146 0,021 0,019 0,016 0,022 0,013 0,015 - 13 Madura_2 0,126 0,126 0,126 0,133 0,133 0,012 0,000 0,000 0,000 0,008 0,010 0,022 - 14 Madura_3 0,131 0,131 0,131 0,139 0,139 0,007 0,008 0,005 0,008 0,000 0,002 0,013 0,008 - 15 Madura_4 0,135 0,135 0,135 0,142 0,142 0,010 0,012 0,009 0,012 0,003 0,005 0,017 0,012 0,003 - 16 PO_1 0,134 0,134 0,134 0,141 0,141 0,007 0,009 0,006 0,009 0,000 0,002 0,014 0,009 0,000 0,003 - 17 PO_2 0,135 0,135 0,135 0,142 0,142 0,012 0,013 0,011 0,013 0,005 0,007 0,018 0,013 0,005 0,008 0,005 - 18 PO_3 0,131 0,131 0,131 0,139 0,139 0,007 0,008 0,005 0,008 0,000 0,002 0,013 0,008 0,000 0,003 0,000 0,005 - 19 PO_4 0,124 0,124 0,124 0,131 0,131 0,016 0,003 0,004 0,003 0,012 0,013 0,022 0,003 0,012 0,015 0,012 0,017 0,012 -
31 Analisis Filogenetik
Pohon filogenetik atau pohon evolusi adalah pohon yang menunjukkan hubungan evolusi antara berbagai spesies yang diyakini memiliki nenek moyang yang sama diantara beberapa spesies. Pohon filogenetik dapat dilakukan dengan mengidentifikasi urutan nukleotida yang homolog pada mtDNA (Dawkin, 2000).
Rekonstruksi pohon filogenetik sapi asli dan sapi lokal dilakukan berdasarkan urutan nukleotida daerah kontrol dari genom DNA mitokondria yang dianalisis secara parsial. Panjang sekuen D-loop yang dapat dianalisis hanya sepanjang 584 bp. Hal tersebut dikarenakan pada beberapa sapi yang diteliti terdapat variasi ulangan minisatelit dan adanya delesi pada sekuen basa nukleotida pada beberapa sapi. Selain itu juga dikarenakan mtDNA memiliki laju mutasi lima sampai sepuluh kali lebih cepat dari pada DNA inti (Wilson et al., 1985) dan pada daerah kontrol memiliki kecepatan evolusi 10-20 kali lebih cepat dibandingkan dengan daerah mtDNA lainnya (Randi, 2000), sehingga segmen ini lebih banyak situs polimorfik yang berguna dalam rekonstruksi pohon filogeni intraspesifik. Pohon filogenetik ini dibentuk dengan menggunakan metoda Neighbor-joining yang termasuk dalam metoda jarak dengan prinsip pengelompokkan taksa berdasarkan nilai jarak evolusioner pasangan-pasangan operational taxonomyunit dimana setiap percabangan yang terdapat dalam pohon filogeni berevolusi pada kecepatan yang tidak sama (Hartl, 2000).
Konstruksi pohon filogeni bangsa-bangsa sapi asli dan sapi lokal Indonesia berdasarkan jarak genetik p-distance dari susunan dan komposisi basa-basa nukleotida daerah D-loop menunjukkan adanya perbedaan materi-materi genetik diantara kelompok maupun individu sapi-sapi tersebut. Berdasarkan analisis perbedaan genetik antar individu untuk membentuk pohon filogeni berdasarkan metode 2 parameter Kimura dalam uji bootsrap 1000 kali pengulangan, diperoleh dua klaster sapi lokal Indonesia, yaitu klaster pertama klaster A yang terdiri dari sapi Bali, Madura, serta PO dan klaster yang kedua yaitu klaster B yang terdiri dari sapi Aceh dan Pesisir (Gambar 14).
Klaster A yaitu sapi Aceh dan Pesisir termasuk dalam kelompok B. indicus dari GenBank dengan nilai uji bootstrap 98%. Pengklasteran sapi Aceh dan Pesisir terpisah terhadap sapi Bali (B. javanicus). Nilai uji bootstrap 100% yang