ISOLASI cDNA SUCROSE TRANSPORTER (SUT) DARI BATANG
TANAMAN TEBU (Saccharum officinarum L.)
Slameto
1), Bambang Sugiharto
2)1)
Teks penuh
1)
Gambar
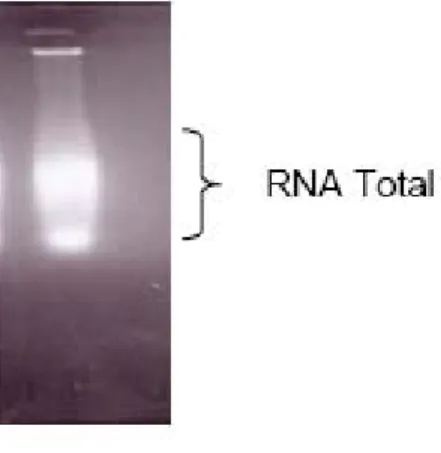

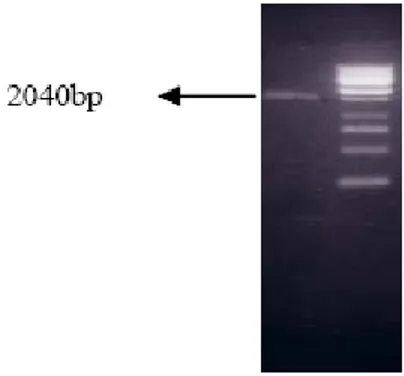
Dokumen terkait
Kesimpulan : Terdapat perbedaan pada jumlah eosinofil sputum maupun VEP 1 % yang bermakna, dan neutrofil sputum yang tidak bermakna sebelum dan sesudah pemberian
Rinitis medicamentosa adalah obstruksi nasal yang terjadi pada pasien yang menggunakan vasokonstriktor intranasal secara kronis. Belum diketahui dengan jelas penyebabnya,
Perancangan Video Promosi Taman Nasional Karimunjawa dengan tema “Beautiful Experience of Karimunjawa” ini adalah judul tugas akhir yang diambil oleh penulis karena
Helaian bunga pita membentang ke luar (straight), bertekstur lunak, berbentuk lonjong, berwarna putih, memiliki tepi yang rata, ujung bunga pita meruncing, pada permukaan
Dari hasil observasi yang peneliti peroleh pada tanggal saat proses pembelajaran yang dilakukan oleh Bu Izzatul Banati, S.Pd.I, Beliau terlihat
Evaluasi dan tindak lanjut pembelajaran Ilmu Pengetahuan Sosial (IPS) di SMK Negeri 1 Lawa berdasarkan data yang diperoleh, dapat dijelaskan bahwa evaluasi pembelajaran
Rencana Pelaksanaan Anggaran Belanja Langsung Perangkat Daerah per Triwulan. Uraian Triwulan
Ketidakpastian dari jenis risiko dan penyebab risiko dominan yang terjadi pada aktivitas supply chain IKM intip menjadikan sejumlah pihak pemilik IKM Intip cenderung berhati