IDENTIFIKASI JATI SUNGU MENGGUNAKAN PENANDA MIKROSATELIT
(Identification of Sungu teak using microsatellite markers)
I.L.G. Nurtjahjaningsih dan A. Rimbawanto
Balai Besar Penelitian Bioteknologi dan Pemuliian Tanaman Hutan
ABSTRACT
DNA analysis could be applied to distinguish varieties of teak. Aim of this study was to identify teak Sungu at seedling level. Twelve leaf samples of Sungu teak were collected from different origins i.e. Biotrop, Kutoarjo, Gunung Kidul and four samples of local teak from Jember, were analyzed using 7 microsatellite/simple sequence repeat (SSR) primers. The results showed that 6 out of 7 microsatellite primers were polymorphic, the number of detected allele (Na) per locus ranged from 2 to 8. Values of gene diversity (HE) per locus
ranged between 0.469 and 0.819. Two out of 6 polymorphic microsatellite primers were excess homozygote; the values were significant deviated from Hardy Weinberg Equilibrium (HWE). Eight private alleles represented specific alleles for Sungu teak, while 6 private alleles identified local teak from Jember. A dendrogram showed 2 clusters that separated very well between Sungu teak and local teak. Moreover Sungu teak from Biotrop and Kutoarjo were clustered at a sub cluster. Effectiveness DNA analysis to identify variety was briefly discussed.
Key words: Sungu teak, variety identification, microsatellite primer
ABSTRAK
Analisa DNA dapat diaplikasikan untuk membedakan varietas jati. Tujuan penelitian ini adalah untuk mengidentifikasi jati Sungu pada tingkat bibit. Dua belas sampel daun jati Sungu diperoleh dari Biotrop, Kutoarjo dan Gunung Kidul, dan empat sampel daun jati lokal diperoleh dari Jember. Tujuh penanda mikrosatelit/simple sequence repeat (SSR) digunakan dalam penelitian ini. Hasil analisa menunjukkan 6 dari 7 penanda SSR bersifat polymorphik. Jumlah allele per lokus (Na) yang terdeteksi berkisar dari 2 sampai dengan 8. Nilai keragaman genetik (HE) per lokus berkisar antara 0,469 dan
0,819. Dua dari 6 penanda SSR yang polymorphik menunjukkan homozygote yang berlebih dan nilainya secara signifikan menyimpang dari hukum keseimbangan Hardy Weinberg. Delapan allele privat merupakan allele spesifik pada jati Sungu, dan 6 allele privat menunjukkan allele spesifik pada jati lokal. Dendrogarm menunjukkan perbedaan kluster antara jati Sungu dan jati lokal. Selain itu, jati Sungu asal Biotrop dan Kutoarjo mengelompok menjadi satu sub kluster. Efektivitas analisa DNA untuk identifikasi varietas jati diuraikan dalam tulisan ini.
Kata kunci: Jati Sungu, identifikasi varietas, penanda mikrosatelit.
I. PENDAHULUAN
Jati (Tectona grandis Linn f.) tersebar secara alami di India, Thailand, Myanmar dan Laos (Verhaegen dkk. 2005). Meskipun Jati bukan jenis tanaman asli Indonesia, namun jenis ini sudah diintroduksi ke Indonesia sejak lama (jaman kolonial Belanda) sehingga penyebarannya cukup luas. Daerah sebaran jati yang utama berada di Jawa dan Muna, kemudian meluas hingga Madura, Bali dan Sumbawa.
Sifat awet kayu dan teksturnya yang bagus menyebabkan jati banyak dikembangkan secara vegetatif baik oleh pengusaha bibit skala besar maupun kecil bahkan masyarakat. Dalam proses budidaya dan penyebarannya, jati mengalami perubahan karakter morphologi sehingga menurut sifat kayu dan bentuk pohonnya ditemukan beberapa varietas jati di Indonesia diantaranya jati Lengo/Malam, jati Sungu, jati Werut, jati Doreng, jati Kembang dan jati Kapur (Mahfudz dkk. t.t.). Selain itu, lembaga penelitian seperti LIPI juga mengembangkan jati cepat tumbuh dan cepat panen sehingga muncul merk dagang jati yang belum diuji asal-usulnya secara genetik, misalnya jati emas, jati berlian dan jati platinum. Varietas maupun merk dagang jati ini dibudidayakan dan dipasarkan sehingga banyak varietas jati tersebar di masyarakat. Identitas morphologi varietas jati ini mudah dikenali saat tanaman sudah besar namun sangat sulit pada tahap bibit, sehingga banyak pengguna bibit jati harus berhati-hati untuk tidak salah memilih varietas jati yang diinginkan.
Identifikasi varietas tanaman menggunakan analisa DNA merupakan metode penting di banyak bidang pertanian/kehutanan dan penelitian biologi tanaman. Analisa DNA mampu membedakan varietas pada taraf gen, sehingga memberikan hasil yang lebih akurat dan lebih cepat dibandingkan dengan pengamatan secara morphologi (Morell dkk. 1995). Aplikasi analisa DNA untuk identifikasi varietas telah banyak dilakukan pada tanaman semusim seperti padi (Rahman dkk. 2009), kentang (Novakova dkk. 2009), gandum (Fernandez dkk. 2002) dan anggur (Tessier dkk. 1999). Sedangkan pada tanaman kehutanan, identifikasi varietas telah dilakukan pada jenis Populus
(Rahman dan Rajora 2002). Pendekatan metode ini dilakukan dengan cara mengidentifikasi kedekatan secara genetik antar varietas. Ukuran amplifikasi allele maupun frekuensi allele berbeda-beda untuk setiap varietas sehingga bisa digunakan sebagai analisa sidik jari (fingerprinting) pada tanaman. Varietas yang mempunyai struktur allele yang sama akan mengelompok, sebaliknya varietas dengan struktur allele yang berbeda akan terpisah.
Ada beberapa penanda DNA yang dapat digunakan untuk mengidentifikasi varietas, diantaranya Random Amplification
Polymorphic DNA (RAPD) dan Simple Sequence Repeat
(SSR)/mikrosatelit. Penanda RAPD mempunyai 10 basa dan bersifat dominan. Pengembangan penanda ini dilakukan dengan cara screening dan relatif mudah dilakukan. Penanda ini mempunyai keragaman genetik cukup tinggi. Aplikasi penanda RAPD telah digunakan untuk mengidentifikasi varietas gandum (Fernandez dkk. 2002). Namun demikian kelemahan penanda ini salah satunya adalah rendahnya nilai kestabilan amplifikasi allele (low reproducibility). Penanda mikrosatelit mempunyai sepasang sekuense primer sehingga amplifikasi allele lebih stabil dan lebih powerful. Penanda mikrosatelit bersifat kodominan dan mempunyai level keragaman genetik dan pembeda genotype yang sangat tinggi (Polymorphic Information Content=PIC) sehingga mampu membedakan genotype antar varietas (Karakousis dkk. 2003). Penanda mikrosatelit telah digunakan untuk mengidentifikasi varietas gandum liar (Feng dkk. 2006). Tujuan dari penelitian ini adalah untuk mengidentifikasi jati Sungu pada tingkat bibit menggunakan penanda mikrosatelit.
II. BAHAN DAN METODE Koleksi Materi Genetik
Materi genetik berupa sampel daun jati Sungu berasal dari 4 daerah yaitu Biotrop, Kutoarjo dan Gunung Kidul, dan sampel daun jati lokal dari Jember sebagai outgrup (kontrol) dengan total sampel sebanyak16 sampel daun. Sampel daun tersebut dikemas menggunakan amplop dan disimpan dalam deep freezer pada suhu -20oC sampai dilakukan ekstraksi DNA.
Ekstraksi DNA dan Analisa Mikrosatelit
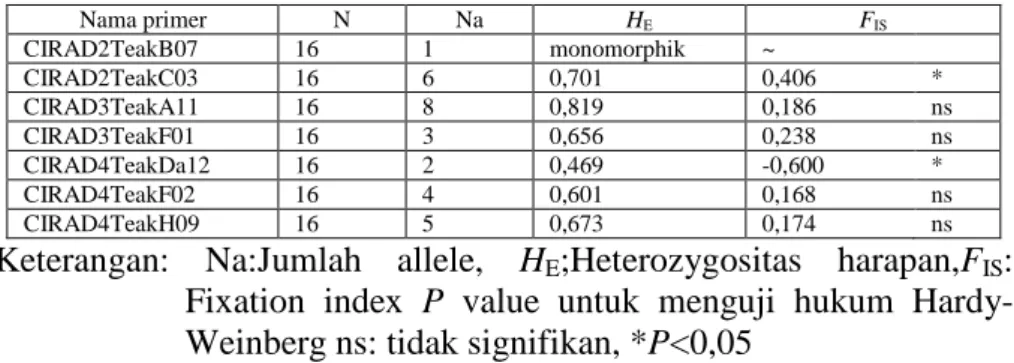
Sampel daun diekstraksi menggunakan metode CTAB (Cetyltrimethyl ammonium bromide) (Shiraishi and Watanabe, 1995) dan digunakan sebagai template DNA. Analisa identifikasi jati Sungu menggunakan 7 penanda mikrosatelit pada Tectona grandis yaitu CIRAD2TeakB07, CIRAD2TeakC03, CIRAD3TeakA11, CIRAD3TeakF01, CIRAD4TeakDa12, CIRAD4TeakF02 dan CIRAD4TeakH09 (Verhaegen dkk.2005). Adapun sekuen basa 7 penanda tersebut disajikan pada Tabel 1.
Tabel 1. Sekuen primer dan motif ulangan mikrosatelit pada Tectona grandis
Nama lokus Sekuen primer Motif ulangan Ta (oC) CIRAD3TeakA11 F:AAACCATGACAGAAACGAATC (GA)16 51
R:TTGGGAATGGGAGGAGAAGT
CIRAD2TeakB07 F:GGGTGCTGATGATTTTGAGTT (TC)14 51 R:CTAAGGAGTGAGTGGAGTTTT
CIRAD2TeakC03 F:AGGTGGGATGTGGTTAGAAGC (GA)17 51 R:AAATGGTCATCAGTGTCAGAA
CIRAD4TeakDa12 F:CGCACACCAGTAGCAGTAGCC (GA)4(N)5(GA)11A(GA)4 51 R:GCCGGAAAAAGAAAAACCAAA
CIRAD3TeakF01 F:GCTCTCCACCAACCTAAACAA (TC)16 51 R:AAAACGTCTCACCTTCTCACT
CIRAD4TeakF02 F:CCGGTAAAAAGGTGTGTCA (TC)4(AC)3(N)16(TC)11 51 R:GAGTGGAAGTGCTAATGGA
CIRAD4TeakH09 F:GCAAACCAACCTTACT (GA)14 51 R:CCGTTAGCACTCCATT
Proses PCR (Polymorphism Chain Reaction) menggunakan mesin thermal cycler GeneAmp PCR system 9700 (Applied Biosystem), dalam larutan reaksi 10 μL terdiri dari 25 ng/μL DNA, masing-masing
pasangan primer (Tabel 1), 1 x buffer, 2,5 mM MgCl2, 2,5 mM dNTP dan AmplitaqGold polymerase (Applied Biosystem). Kondisi PCR meliputi step denaturasi yaitu 4 menit pada suhu 94oC diikuti dengan step annealing dalam 30 siklus selama 30 detik pada suhu 94oC, 45 detik pada masing-masing spesifik suhu annealing, 45 detik pada suhu 72oC dan step akhir pemanjangan 30 menit pada suhu 72oC. Amplifikasi hasil PCR dideteksi menggunakan ABI 3100 gene analyzer avant (Applied Biosystem). Fragment DNA dianalisa menggunakan genemapper (Applied Biosystem).
Analisa Data
Untuk mengetahui keragaman genetik per lokus, menggunakan parameter jumlah allele yang terdeteksi (Na), nilai keragaman genetik (HE) dan koefiesien inbreeding (FIS). Untuk mengidentifikasi varietas menggunakan parameter private allele per daerah asal per varietas jati. Private allele adalah allele yang muncul hanya pada satu populasi saja dan menunjukkan sidik jari per varietas jati per daerah asal. Nilai Na,
HE, FIS dan allele private dihitung menggunakan program GENALEX 6 (Peakall and Smouse, 2006). Dendrogram yang menunjukkan hubungan kekerabatan antar daerah asal Jati dihitung menggunakan program POPTREE2 (Takezaki dkk. 2010).
III. HASIL DAN PEMBAHASAN
A. Keragaman Genetik Penanda Mikrosatelit
Satu dari 7 primer bersifat monomorphik yaitu primer CIRAD2TeakB07, sedangkan 6 primer yang lain menunjukkan amplifikasi allele yang polymorphik selanjutnya digunakan untuk analisa identifikasi varietas jati. Jumlah allele yang terdeteksi per lokus (Na) pada seluruh daerah asal jati berkisar antara 2 sampai 10 (Tabel 2). Nilai keragaman genetik (HE) per primer cukup tinggi yaitu berkisar antara 0,469 (CIRAD4TeakDa12) sampai 0,819 (CIRAD3TeakA11). Dua primer secara signifikan menyimpang dari segregasi hukum Hardy-Weinberg yaitu CIRAD2TeakC03 dan CIRAD4TeakDa12, sedangkan 4 primer mikrosatelit polymorphik yang lain tidak signifikan.
Tabel 2. Parameter keragaman genetik per lokus menggunakan 16 sampel daun Jati
Nama primer N Na HE FIS CIRAD2TeakB07 16 1 monomorphik ~ CIRAD2TeakC03 16 6 0,701 0,406 * CIRAD3TeakA11 16 8 0,819 0,186 ns CIRAD3TeakF01 16 3 0,656 0,238 ns CIRAD4TeakDa12 16 2 0,469 -0,600 * CIRAD4TeakF02 16 4 0,601 0,168 ns CIRAD4TeakH09 16 5 0,673 0,174 ns Keterangan: Na:Jumlah allele, HE;Heterozygositas harapan,FIS:
Fixation index P value untuk menguji hukum Hardy-Weinberg ns: tidak signifikan, *P<0,05
Banyaknya jumlah allele yang terdeteksi dan tingginya nilai keragaman genetik merupakan kelebihan penanda mikrosatelit dibandingkan dengan penanda DNA lainnya. Dengan menggunakan penanda mikrosatelit yang sama pada jati, jumlah allele yang terdeteksi berkisar antara 13 sampai 20 dengan nilai keragaman genetik berkisar antara 0,550 sampai 0,852 (Verhaegen dkk.2005). Banyaknya jumlah allele yang terdeteksi disebabkan karena jumlah sampel yang digunakan lebih banyak dan berasal dari sebaran yang luas yaitu India, Thailand, Laos, Indonesia dan Afrika. Sebaliknya pada penelitian ini menggunakan daerah asal sampel sangat terbatas yaitu Biotrop, Kutoarjo, Gunung Kidul dan Jember, sehingga allele yang terdeteksi berjumlah sedikit. Meskipun menggunakan sampel yang terbatas, nilai keragaman genetik pada penelitian ini menunjukkan nilai yang sebanding dengan sampel yang digunakan pada penelitian yang dilaporkan oleh Verhaegen dkk.(2005).
Amplifikasi null allele menyebabkan sebuah primer menyimpang secara signifikan dari segregasi hukum Hardy Weinberg, sehingga menunjukkan level homozygote berlebih/kekurangan level heterozygositas (Moriguchi dkk. 2003). Null allele merupakan amplifikasi allele heterozygote walaupun sebenarnya homozygote. Karakter yang nyata pada primer yang mempunyai null allele adalah amplifikasi allele yang tidak stabil, kadang muncul, kadang tidak muncul. Null allele terjadi pada saat proses PCR dan disebabkan oleh mutasi sekuen DNA (Moriguchi dkk. 2003). Homozygote berlebih ditunjukkan oleh primer CIRAD2TeakC03 dan CIRAD4TeakDa12 pada penelitian pengembangan penanda mikrosatelit jati (Verhaegen
dkk.2005). Selain null allele, jenis sampel juga menyebabkan sebuah primer mempunyai level homozygote yang berlebih. Sampel yang digunakan dalam penelitian ini berasal dari bibit yang diperbanyak secara vegetatif yang berasal dari beberapa klon saja. Sampel yang berkerabat secara genetik ini menyebabkan tingginya level homozygositas.
B. Identifikasi Jati Sungu Menggunakan Penanda Mikrosatelit
Tabel 3 menunjukkan empat penanda SSR pada jati yang mempunyai private allele per populasi untuk setiap varietas jati. Delapan privat allele muncul pada varietas jati Sungu yaitu allele 258, 270 (CIRAD2TeakC03), 252, 257, 259 (CIRAD3TeakA11), 208 (CIRAD4TeakF02), 134, 136 (CIRAD4TeakH09), sedangkan 6 privat allele muncul pada varietas jati lokal Jember yaitu allele 241, 245, 263 (CIRAD3TeakA11), 204 (CIRAD4TeakF02), 198, 204 (CIRAD4TeakH09). Tabel 3. Privat allele dan frekuensi private allele per varietas jati dari setiap daerah asal yang ditunjukkan menggunakan empat penanda mikrosatelit
Nama penanda SSR Private allele
Frekuensi private allele
Jati Sungu Jati lokal Biotrop Kutoarjo Gn Kidul Jember CIRAD2TeakC03 258 0,000 0,000 0,167 0,000 270 0,000 0,167 0,000 0,000 CIRAD3TeakA11 241 0,000 0,000 0.000 0.167 245 0,000 0,000 0.000 0.167 252 0,500 0,000 0.000 0.000 257 0,000 0,167 0.000 0.000 259 0,000 0,167 0.000 0.000 263 0,000 0,000 0.000 0.167 CIRAD4TeakF02 204 0,000 0,000 0,000 0,667 208 0,500 0,000 0,000 0,000 CIRAD4TeakH09 134 0,000 0,000 0,500 0,000 136 0,500 0,000 0,000 0,000 198 0,000 0,000 0,000 0,333 204 0,000 0,000 0,000 0,667
Angka dengan garis-bawah: frekuensi privat allele
Pada penelitian ini untuk membedakan varietas jati Sungu dan jati lokal menggunakan sampel masing-masing 12 dan 4 sampel. Sedangkan untuk membedakan Populus deltoides var. deltoides dan var occidentalis menggunakan jumlah sampel masing-masing 18 dan 2
sampel dan pada varietas P. nigra var nigra dan var italica masing-masing menggunakan 16 dan 9 sampel (Rahman dan Rajora 2002). Apabila dibandingkan dengan referensi tersebut, maka sampel yang digunakan untuk membedakan varietas jati masih dalam kisaran jumlah sampel yang digunakan pada penelitian varietas Populus.
Varietas tanaman dapat dibedakan satu dengan yang lain berdasarkan unique/spesifik allele yaitu ukuran allele atau privat allele. Pada penelitian varietas Populus, ukuran allele (bp) digunakan sebagai unique/spesifik allele untuk membedakan varietas dan hanya menggunakan 3 penanda mikrosatelit untuk menunjukkan spesifik allele tersebut (Rahman dan Rajora 2002). Pada penelitian ini menggunakan privat allele sebagai unique allele dapat membedakan varietas jati dari 4 penanda mikrosatelit. Private allele yang dimiliki berbeda pada masing-masing varietas jati dan dapat membedakan secara jelas jenis varietas jati Sungu dan jati lokal sehingga allele tersebut dapat digunakan sebagai sidik jari dan membedakan varietas jati.
Gambar 1. Dendrogram menunjukkan hubungan kekerabatan antar daerah asal jati
Dendrogram disusun berdasarkan nilai jarak genetik (data tidak ditampilkan) dan menunjukkan hubungan kekerabatan antar daerah asal jati (Gambar 1). Dendrogram membentuk 2 kluster yaitu kluster Jati lokal Jember dan kluster jati Sungu asal Biotrop, Kutoarjo dan Gunung Kidul. Jati Sungu asal Biotrop dan Kutoarjo membentuk satu sub kluster dengan tingkat kepercayaan 63%, sedangkan Jati asal Gunung Kidul membentuk sub kluster lain, meskipun masih dalam kluster yang sama.
jati Sungu dan lokal. Jati Sungu asal Biotrop dan Kutoarjo mempunyai kedekatan yang lebih tinggi dibandingkan jati Sungu asal Gunung Kidul. Hal ini tidak mengherankan karena bibit jati Sungu Biotrop merupakan hasil pembiakan vegetatif klon asal Kutoarjo (Peneliti PT Murakabi, Komunikasi Pribadi).
IV. KESIMPULAN
Hasil penelitian diatas menunjukkan bahwa analisa DNA memberikan hasil yang akurat dan cepat terhadap identifikasi varietas jati. Private allele yang merupakan allele spesifik pada masing-masing varietas dapat membedakan jenis jati Sungu dan jati lokal asal Jember. Analisa dendrogram memperjelas bahwa jati Sungu terpisah dengan jati lokal asal Jember. Identifikasi varietas jati menggunakan penanda DNA ini dapat diaplikasikan untuk memberikan perlindungan terhadap pengusaha dan pengguna bibit agar tidak salah memilih varietas jati yang diinginkan, terutama ketika tanaman jati masih pada tingkat bibit.
V. UCAPAN TERIMA KASIH
Ucapan terima kasih ditujukan kepada PT Murakabi yang telah menyediakan sampel daun bibit jati Sungu asal Biotrop dan Kutoarjo, dan kepada Dr. Mahfudz yang telah menyediakan sampel jati Sungu dari Gunung Kidul. Ucapan terima kasih juga ditujukan kepada Bapak Y. Triyanta yang telah banyak membantu kegiatan penelitian di Laboratorium Genetika Molekuler.
VI. DAFTAR PUSTAKA
Feng, Z.Y., Liu, X.J., Zhang, Y.Z., Ling, H.Q. 2006. Genetic diversity analysis of Tibetan wild barley using SSR markers. Acta Genetica Sinica 33 (10): 917-928
Fernandez, M.E. Figueiras, A.M., Benito, C. 2002. The use of ISSR and RAPD markers for detecting DNA polymorphism,
genotype identification and genetic diversity among barley cultivars with known origin. Theor. Appl.Genet. 104:845-851 Karakousis, A., Barr, AR.,Chalmers, KJ, Ablett, GA., Holton, TA,
Henry,RJ, Lim,P, Langridge, P. 2003. Potential of SSR markers for plant breeding and variety identification in Australian barley germplasm. Australian Journal of Agricultural Research 54 (12): 1197-1210
Mahfudz, Fauzi, M.A.,Yuliah, Herawan, T.,Prastyono, Supriyanto, H. t.t. Sekilas tentang Jati (Tectona grandis). Pusat Penelitian dan Pengembangan Bioteknologi dan Pemuliaan Tanaman Hutan Yogyakarta. 73 hal.
Morell, M., Peakall R., Appels,R., Preston, L.R., Lloyd, H.L. 1995. DNA profiling techniques for variety identification. Australian Journal of Experimental Agriculture 35 (6): 807-819
Moriguchi, Y., Iwata, H., Ujino-Ihara, T., Yoshimura, K., Taira, H., Tsmura, Y. 2003. Development and characterization of microsatellite markers for Cryptomeria japonica D.Don. Theor. Appl. Genet. 106: 751-758
Novakova, A., Simackova, K., Barta, J., Curn, V. 2009. Potato variety identification by molecular markers based on retrotransposon analyses. Czech J. Genet. Plant Breed 45 (1): 1-10
Peakall, R., Smouse PE. 2006. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes 6: 288-295
Rahman, M.H., Rajora, O.P. 2002. Microsatellite DNA fingerprinting, differentiation, and genetic relationship of clones, cultivars, and varieties of six poplar species from three sections of the genus Populus. Genome 45: 1083-1094
Rahman, M.S., Rezwan Molla, Md., Samsul Alam, Md., Rahman, L. 2009. DNA fingerprinting of rice (Oriza sativa L.) cultivars using microsatellite markers. Australian Journal of Crop Science 3(3): 122-128
Shiraishi, S., Watanabe, A. 1995. Identification of chloroplast genome between Pinus densiflora Sieb et Zucc and P. thumbergii Parl based on the polymorphism in rbcL gene. Journal of Japanese Forestry Society 77: 429-436
Takezaki, N., Nei, M., and Tamura, K. 2010. POPTREE2: Software for constructing population trees from allele frequency data and computing other population statistics with windows interface. Molecular Biology Evolution 27(4): 747-752
Tessier, C., David, J., This, P., Boursiqout, J.M., Charrier, A. 1999. Optimization of the choice of molecular markers for varietal identification in Vitis vinifera L. Theor Appl Genet 98: 171-177
Verhaegen, D., Ofori, D., Foana, I., Poitel, M., Vaillant, A. 2005. Development and characterization of microsatellite markers in