TAKSONOMI MOLEKULER ‘DNA BARCODING’ DAN
ANALISIS FILOGENETIK IKAN HIU DI PELABUHAN
PERIKANAN PALABUHANRATU BERDASARKAN MARKA
MITOKONDRIA
RAHMAD
SKRIPSI
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi yang berjudul Taksonomi Molekuler „ DNA Barcoding‟ dan Analisis Filogenetik Ikan Hiu di Pelabuhan Perikanan Palabuhanratu berdasarkan Marka Mitokondria adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, April 2013
Rahmad NIM C54090075
ABSTRAK
RAHMAD. Taksonomi Molekuler „DNA barcoding‟ dan Analisis Filogenetik Ikan Hiu di Pelabuhan Perikanan Palabuhanratu Berdasarkan Marka Mitokondria. Dibimbing oleh HAWIS MADDUPPA dan BEGINER SUBHAN.
Perdagangan ikan hiu umumnya hanya dalam bentuk sirip sehingga sulit untuk diidentifikasi secara konvensional. Taksonomi molekuler DNA barcoding dapat membantu proses identifikasi ini karena hanya membutuhkan sedikit jaringan tubuh dari ikan hiu tersebut. Penelitian ini mengumpulkan sampel di PPN Palabuhanratu yang bertujuan untuk mengidentifikasi ikan hiu secara molekuler dan status konservasinya, serta menentukan hubungan filogenetik antar spesies ikan hiu. Hasil penelitian ini menunjukkan dari 28 sampel yang teramplifikasi terdapat 3 ekor hiu jenis Rhynchobatus australiae, 7 ekor hiu jenis Alopias superciliosus, 12 ekor hiu jenis Carcharhinus falciformis, 2 ekor hiu jenis Isurus paucus, 2 ekor hiu jenis Carcharhinus limbatus dan 2 ekor hiu jenis Carcharhinus longimanus. Hiu tersebut termasuk kedalam kategori vulnerable (R.australiae, A.superciliosus, C.Longimanus dan I.paucus) dan near threatened (C.falciformis dan C.limbatus). Konstruksi pohon filogenetik menunjukkan bahwa hiu yang didaratkan di Palabuhanratu terbagi kedalam 4 kelompok besar dengan 4 famili yang berbeda yaitu Rhynchobatidae, Alopiidae, Lamnidae dan Carcharhinidae. Kata kunci: hiu, DNA, barcoding, filogenetik, famili
ABSTRACT
RAHMAD. Molecular Taxonomy „Barcoding DNA‟ and Phylogenetic Analysis of Shark at Fisheries Port Palabuhanratu Based on Marka Mitochondria. Under Advisory by HAWIS MADDUPPA and BEGINER SUBHAN.
Sharks are generally traded only in the form of fins and therefore it is difficult to identify them conventionally. Molecular taxonomy of DNA barcoding is a tool that can help identification process because it only requires a few tissues from the shark. The samples were collected from fisheries port of Palabuhanratu in order to identified sharks species molecularly and confirm their conservation status, as well as building phylogenetic tree among sharks species landed in this market. The result showed that of 28 sample amplified were belonged to 3 species of sharks Rhyncobatus australiae, 7 species Alopias superciliosus, 12 species Carcharhinus falciformis, 2 species Isurus paucus, 2 species Carcharhinus limbatus and 2 species Carcharhinus longimanus. The identified sharks were included in to the category of vulnerable (R.australiae, A.superciliosus, C.longimanus and I.paucus) and near threatened (C.falciformis and C.limbatus) of IUCN redlist. The construction of phylogenetic tree showed that sharks were divided into 4 big groups with 4 different families which are Rhynchobatidae, Alopiidae, Lamnidae and Carcharhinidae.
© Hak Cipta Milik IPB, Tahun 2013 Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB.
Dilarang mengumumkan dan memperbanyak sebagian atau seluruh karya tulis ini dalam bentuk apapun tanpa izin IPB.
TAKSONOMI MOLEKULER ‘DNA BARCODING’ DAN
ANALISIS FILOGENETIK IKAN HIU DI PELABUHAN
PERIKANAN PALABUHANRATU BERDASARKAN MARKA
MITOKONDRIA
RAHMAD
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Kelautan
Pada
Departemen Ilmu dan Teknologi Kelautan
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR BOGOR
Judul Skripsi : Taksonomi Molekuler „DNA Barcoding‟ dan Analisis Filogenetik Ikan Hiu di Pelabuhan Perikanan
Palabuhanratu berdasarkan Marka Mitokondria
Nama : Rahmad NIM : C54090075 Disetujui oleh, Diketahui oleh Dr Ir I Wayan Nurjaya, MSc Ketua Departemen
Tanggal lulus : 07 Mei 2013 Dr Hawis Madduppa, SPi MSi Pembimbing I
Beginer Subhan, SPi MSi Pembimbing II
PRAKATA
Puji dan syukur penulis panjatkan kepada ALLAH subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan September ini ialah DNA barcoding dengan judul Taksonomi Molekuler „DNA barcoding’ dan Analisis Filogenetik Ikan hiu di Pelabuhan Perikanan Palabuhanratu berdasarkan Marka Mitokondria.
Terima kasih penulis ucapkan kepada Bapak Dr Hawis Madduppa SPi MSi dan Bapak Beginer Subhan SPi MSi selaku pembimbing, serta Bapak Prof Dr Drh G Ngurah Mahardika yang telah banyak memberikan saran. Di samping itu, penghargaan penulis sampaikan kepada Laboratorium Indonesian Biodiversity Research Center, Bali dan seluruh staff pegawai yang telah mendukung dan membantu dalam pelaksanaan penelitian ini. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, teman-teman, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, April 2013
DAFTAR ISI
DAFTAR TABEL viii
DAFTAR GAMBAR ix DAFTAR LAMPIRAN x PENDAHULUAN 1 Latar Belakang 1 Tujuan Penelitian 2 METODOLOGI . 2
Waktu dan Tempat Penelitian 2
Koleksi Sampel 2
Ekstraksi DNA dan PCR 2
Elektroforesis 3
Siklus Pengurutan Nukleotida 3
Analisis Data 3
Identifikasi spesies dan Status konservasi 3
Analisis filogenetik 4
HASIL DAN PEMBAHASAN 4
Identifikasi Spesies dan Status Konservasi 4
Hubungan Filogenetik 9
KESIMPULAN DAN SARAN 11
Kesimpulan 11
Saran 11
DAFTAR PUSTAKA 11
LAMPIRAN 13
DAFTAR TABEL
1.Hasil identifikasi spesies ikan hiu secara molekuler 4 2. Daftar harga sirip ikan hiu berdasarkan ukuran dan pengolahannya 6
DAFTAR GAMBAR
1. Hasil analisis DNA barcoding berdasarkan pohon filogenetik 10
DAFTAR LAMPIRAN
1. Prosedur kerja PCR 14
2. Hasil pengurutan basa nukleotida 15
3. Hasil analisis BLAST dalam NCBI 22
1
PENDAHULUAN
Latar Belakang
Ikan hiu mengalami eksploitasi secara besar-besaran di berbagai negara di dunia, termasuk di Indonesia. Indonesia merupakan eksportir utama sirip ikan hiu untuk pasar internasional misalnya Hongkong, Cina dan Singapura (Dharmadi dan Fahmi 2005). Pada tahun 1987, produksi perikanan hiu di Indonesia tercatat sebesar 36.884 ton, kemudian pada tahun 2000, produksi perikanan hiu meningkat hingga hampir dua kali lipat sebesar 68.366 ton (Dharmadi dan Fahmi 2003). Kondisi seperti ini dapat menyebabkan beberapa spesies hiu akan mengalami kepunahan. Perdagangan ikan hiu umumnya hanya dalam bentuk sirip sehingga sulit untuk diidentifikasi secara konvensional.
Taksonomi molekuler DNA barcoding dapat membantu proses identifikasi ini karena hanya membutuhkan sedikit jaringan tubuh dari ikan hiu tersebut. Selama beberapa dekade terakhir, teknik molekuler DNA Barcoding telah diperkenalkan untuk identifikasi spesies hiu, khususnya ketika metode taksonomi tradisional gagal karena tidak mempunyai informasi morfologi yang cukup. Hebert et al. (2003) memperkenalkan DNA barcode sebagai sarana untuk mengidentifikasi semua spesies hewan. Kemampuan barcode DNA untuk mengidentifikasi spesies bergantung pada degenerasi kode genetik. Hal ini diperkuat oleh Ward dan Holmes (2007), yang menganalisis region barcode DNA di 388 spesies ikan, termasuk 4 holocephali dan 61 elasmobranchs. Kajian ini menunjukkan bahwa DNA barcode mampu mendeskriminasikan hingga 98% - 99% dari spesies ikan yang diteliti sejauh ini.
Penggunaan DNA mitokondria (mtDNA) sebagai gen target semakin banyak di lakukan untuk identifikasi suatu spesies (Kyle dan Wilson 2007). Kelebihan yang dimiliki oleh mtDNA sebagai target dalam identifikasi spesies, diantaranya ialah berevolusi lebih cepat dibandingkan DNA inti, berukuran lebih kecil dibandingkan DNA inti, terdapat beberapa salinan didalam sel dan sekuen lengkap DNA mitokondria beberapa organisme perairan telah diketahui. Oleh karena itu, penggunaan mtDNA sangat efektif untuk penentuan dan pengidentifikasian keragaman genetik suatu makhluk hidup. Di samping itu, hal yang mendukung penggunaan mtDNA sebagai penanda genetik salah satunya adalah karena mtDNA terdapat dalam jumlah copy yang tinggi, sehingga memudahkan dalam pengisolasian dan purifikasi untuk berbagai keperluan analisa (Duryadi 1994)
Filogenetik adalah salah satu sistem klasifikasi yang didasarkan pada keeratan hubungan nenek moyang (Purnomo dan Pudjoarinto 1999). Tujuan dari penyusunan filogenetik adalah untuk mengkontruksi dengan tepat hubungan antara organisme dan mengestimasi perbedaan yang terjadi dari satu nenek moyang kepada keturunannya (Li et al. 1999). Pohon filogenetik akan menyajikan hasil analisis BLAST sesuai dengan karakteristik cabang yang terbentuk sehingga sekuen hiu yang sama atau berdekatan akan membentuk satu kelompok dan sekuen hiu yang berbeda akan membentuk kelompok tersendiri.
2
Oleh karena itu, sistem klasifikasi ini sangat penting digunakan dalam penelusuran kekerabatan evolusioner diantara berbagai takson (Mabrouk et al. 2006). Hasil penelitian ini diharapkan dapat dijadikan database dasar dalam manajemen populasi dan usaha konservasi ikan hiu.
Tujuan Penelitian
1. Identifikasi jenis ikan hiu secara molekuler dan status konservasi ikan hiu yang didaratkan di Pelabuhan Perikanan Palabuhanratu, Sukabumi, Jawa Barat
2. Menentukan hubungan filogenetik antar spesies ikan hiu yang didaratkan di Pelabuhan Perikanan Palabuhanratu, Sukabumi, Jawa Barat.
METODOLOGI
Waktu dan Tempat Penelitian
Pengambilan sampel dilakukan pada Bulan September dan November 2012 bertempat di Pelabuhan Perikanan Palabuhanratu, Sukabumi, Jawa Barat. Pengolahan data sampel dilakukan di Laboratorium Biologi Molekuler, Indonesian Biodiversity Research Center, Bali dan Laboratorium Biodiversitas dan Biosistematika Kelautan, Departemen Ilmu dan Teknologi Kelautan, Fakultas Perikanan dan Ilmu Kelautan, Institut Pertanian Bogor.
Koleksi Sampel
Sampel berupa daging/sirip hiu sebanyak 39 sampel, kemudian dimasukkan kedalam tabung reaksi yang telah diisi ethanol 96% sebanyak 1 ml, dan diberi label menurut masing-masing sampel individu. Tabung yang telah berisi sampel dan ethanol disimpan rapi di laboratorium. Informasi tambahan berupa lokasi penangkapan hiu dan harga sirip hiu diperoleh dari hasil wawancara studi lapang.
Ekstraksi DNA dan PCR
Ekstraksi DNA bertujuan untuk menghancurkan sel dan mengambil jaringan pada sampel. Pada penelitian ini metode ekstraksi yang digunakan adalah metode Chelex 10% (Walsh et al. 1991).
Polymerase Chain Reaction (PCR) merupakan suatu teknik perbanyakan DNA secara enzimatik. Komponen utama dalam PCR adalah 1 µL DNA template, 2,5 µL dNTP, 1,5 µL buffer PCR, 2 µL MgCL2, 1,25 µL primer dan 0,125 µL tag
polymerase. Primer yang digunakan untuk hiu adalah fish BCH: 5'-TAA ACT TCA GGG TGA CCA AAA AAT CA-3' atau fish BCL: 5'-TCA ACY AAT CAY AAA GAT ATY GGC AC-3' (Former et al. 1994).
Secara prinsip, PCR merupakan proses yang diulang-ulang sebanyak 39 kali siklus. Setiap siklus terdiri atas tiga tahapan yaitu tahap peleburan (denaturasi) yang berlansung pada suhu 94-96° C, tahap penempelan (annealing) yang berlansung pada suhu 50-64° C dan tahap pemanjangan (ekstensi) yang berlansung pada suhu 72° C dimana DNA polimerase memperpanjang rantai DNA primer yang komplemen dengan rantai cetakan DNA.
Proses amplifikasi dimulai dengan mengisi lembar kerja PCR dengan tanggal, jumlah sampel, tipe ekstraksi dan catatan lainnya (Lampiran 1). Pengisian lembar kerja ini dilakukan untuk menghitung berapa banyak master mix (MM) yang dibutuhkan dan enzim tag polimerase serta jumlah ekstrak yang digunakan (Lampiran 1).
Elektroforesis
Elektroforesis bertujuan untuk mengetahui kualitas DNA dalam produk PCR. Tahap awal dalam elektroforesis adalah membuat gel agarosa 1%, yaitu dengan mencampurkan 0,75 gram bubuk agarosa dengan 75 mL buffer ke dalam tabung erlemenyer. Panaskan dengan mesin microwave sampai agarosa benar-benar terlarut, kemudian tambahkan 4 µL cyber green atau EtBr (sebagai pewarna molekul), dan tuang dalam cetakan agarosa. Pasang sisir pada cetakan dan tunggu hingga 15-20 menit hingga agarosa mengeras. Setelah gel agarosa mengeras, masukkan gel tersebut kedalam tangki elektroforesis yang berisi buffer TBE 0,5X Selanjutnya siapkan sampel yang akan dielektroforesis. Hamparkan parafilm dan totolkan sekitar 1µL loading dye untuk 1 sampel.
Ambil 4 µL PCR produk dan campurkan dengan loading dye kemudian masukkan dalam sumur gel. Jalankan mesin elektroforesis pada 200 V dan arus 400 mA selama 15 menit, angkat gel dan lihat hasilnya dengan menggunakan lampu UV pada panjang gelombang 254 nm dan foto menggunakan kamera.
Siklus Pengurutan Nukleotida (cycle sequencing)
Siklus pengurutan nukleotida (Sequencing DNA) adalah metode untuk menentukan urutan nukleotida yang terdapat dalam DNA. Urutan DNA berhubungan dengan informasi genetik turunan dalam nukleus (inti), plasmid, mitokondria, dan kloroplas yang membentuk dasar pengembangan semua makhluk hidup. DNA sequencing menggunakan metode PCR sebagai pijakannya. DNA yang akan ditentukan urutan basa ACGT-nya dijadikan sebagai cetakan untuk kemudian diamplifikasi menggunakan enzim dan bahan-bahan yang mirip dengan reaksi PCR, namun ada penambahan beberapa pereaksi tertentu. Proses sequencing DNA dikirim ke Sequencing Facility UC Berkeley, California untuk melakukan pengurutan nukleotida.
Analisis Data
Identifikasi Spesies dan Status Konservasi
Analisis data dilakukan dengan menggunakan software Mega 5.05 (Molecular Evolutionary Genetic Analysis) untuk pembacaan urutan nukleotida
4
dan penjajaran dengan menggunakan ClustalW pada program tersebut untuk melihat adanya keragaman nukleotida (Tamura et al. 2007). Data hasil penjajaran nukleotida yang diperoleh kemudian dicocokkan dengan data yang tersedia pada GeneBank di NCBI (National Center for Biotechnology Information) dengan menggunakan BLAST (Basic Local Alignment Search Tool). Hasil yang diperoleh dari GeneBank akan dianalisis status konservasinya dengan menggunakan IUCN redlist (International Union for Conservation of Nature and Natural Resources).
Analisis Filogenetik
Pembuatan pohon filogenetik pada penelitian ini menggunakan metode Neighbour-joining tree dengan nilai boostrap 100 dan model kimura 2- parameter. Outgroup yang digunakan yaitu Rhinobatus penggali atau pari penggali muncar dengan tujuan untuk mengetahui sejauh mana keakuratan cabang pohon filogenetik yang terbentuk.
HASIL DAN PEMBAHASAN
Identifikasi Spesies dan Status Konservasi
Urutan nukleotida DNA yang telah teramplifikasi menunjukkan bahwa dari 39 sampel daging ikan hiu yang teramplifikasi hanya terdapat 28 sekuen yang bisa teridentifikasi spesiesnya. Hasil penelitian ini menunjukkan terdapat 4 famili ikan hiu yang didaratkan di PPN Palabuhanratu yaitu Rhynchobatidae, Alopiidae, Lamnidae dan Carcharhinidae dengan beberapa spesies yang memiliki urutan basa nukleotida yang berbeda. Hiu yang didaratkan di PPN Palabuhanratu, diantaranya 3 ekor hiu jenis Rhynchobatus australiae (white spotted guitarfish), 7 ekor hiu jenis Alopias superciliosus (bigeye thresher shark), 12 ekor hiu jenis Carcharhinus falciformis (silky shark), 2 ekor hiu jenis Isurus paucus (longfin mako), 2 ekor hiu jenis Carcharhinus limbatus (blacktip shark) dan 2 ekor hiu jenis Carcharhinus longimanus (oceanic whitetip shark) (Tabel 1)
Tabel 1. Hasil identifikasi spesies ikan hiu yang didaratkan di Pelabuhan Perikanan Palabuhanratu dengan menggunakan BLAST
No Famili Analisis BLAST Jumlah (ekor)
IUCN Red list 1 Rhynchobatidae Rhynchobatus australiae 3 Vulnerable
2 Alopiidae Alopias superciliosus 7 Vulnerable
3 Carcharhinidae Carcharhinus falciformis 12 Near Threatened
Carcharhinus limbatus 2 Near Threatened
Carcharhinus longimanus 2 Vulnerable
4 Lamnidae Isurus paucus 2 Vulnerable Hiu jenis ini (Tabel 1) banyak terdapat di Pelabuhan Perikanan Palabuhanratu disebabkan oleh wilayah fishing ground nelayan yang hanya mencapai Pantai Ujung Genteng, Sukabumi, Jawa Barat. Selain itu,
tidak-terdapatnya alat tangkap yang khusus menangkap hiu di Palabuhanratu menyebabkan hiu di daerah ini hanya tertangkap sebagai hasil tangkapan sampingan dari alat tangkap pancing, jaring insang dan rawai tuna. Kapal-kapal perikanan atau armada penangkapan hiu yang khusus menangkap hiu tidak ada yang berdomisili di PPN Palabuhanratu. Oleh karena itu, kapal-kapal perikanan yang tersedia hanya menangkap hiu sebagai hasil tangkapan sampingan dan sesuai dengan jenis alat tangkap yang dapat menangkap hiu.
Ikan hiu yang didaratkan di PPN Palabuhanratu pada tahun 2004 mencapai 87.296 kg yang terdiri dari berbagai jenis seperti hiu aron (Carcharhiunus amblyrhynchos), hiu lanjam (Carcharhinus melanopterus), hiu anjing (Isurus oxirynchus), hiu kebo (Carcharhinus brachyurus), hiu koboy (Carcharhiunus longimanus), hiu buas (Carcharhinus plumbeus), hiu caping (Sphyrna lewini), hiu laek (Prionance glaucu), hiu lutung (Alopias superciliosus), hiu omas (Galeocerdo cuvier) dan hiu monyet (Alopias superciliosus) (DJPT 2004).
Teluk Pelabuhanratu merupakan teluk terbesar di pantai Selatan Pulau Jawa yang berhadapan lansung dengan Samudera Hindia. Secara geografik teluk Pelabuhanratu terletak pada posisi 6°57‟ LS dan 106°22‟-106°33‟ BT (Rahayu 1999). Topografi perairan daerah ini memperlihatkan perairan yang dangkal sampai berjarak 300 m dari garis pantai dengan kedalaman kurang lebih 600 m (Pariwono et al. 1998). Menurut Sanusi (1994), topografi dasar laut (bathymetri) perairan teluk Pelabuhanratu adalah curam dengan kedalaman antara 3-4 m (perairan pantai) sampai lebih dari 200 m, dibagian teluk merupakan lereng kontinental (continental shelf). Profil demikian mengakibatkan adanya fenomena arus menyusur pantai (longshore current) di beberapa lokasi perairan teluk.
Ikan hiu merupakan jenis ikan pelagis dan juga demersal yang bersifat “euryhalin” artinya ikan hiu memiliki derajat toleransi yang tinggi terhadap salinitas, sehingga dapat hidup diperairan payau, tawar dan laut sebagai habitat utamanya. Habitat ikan hiu berbeda-beda untuk setiap jenisnya, biasanya ikan hiu menyenangi air yang jernih dengan substrat pasir, batu, kerikil atau terumbu karang. Menurut Allen (1997), ikan hiu terdapat di semua laut, dari laut yang dangkal sampai laut lepas (oseanik) pada berbagai kedalaman, hingga 3000 m dari permukaan. Tetapi kehidupan ikan hiu terpusat pada daerah neritik (dekat pantai) dan oseanik sampai kedalaman 200 m yang kaya akan makanan (DJPT 1992). Secara umum ikan hiu dapat ditangkap dengan menggunakan alat tangkap seperti pukat ikan dan pancing (Campagno 1984a).
Direktorat Perikanan Tangkap, Departemen Kelautan dan Perikanan 2004 menyatakan “ hiu dapat tertangkap dengan menggunakan alat-alat tangkap seperti rawai tuna (tuna longline), rawai hanyut (drift longline), jaring insang tetap (set gillnet), jaring insang lingkar (encircling gillnet), pancing ulur (hand line), trammel net dan purse seine.
Berdasarkan hasil studi ini, harga sirip ikan hiu yang diperdagangkan relatif sama untuk setiap spesies tergantung ukuran dan proses pengolahannya. Harga sirip hiu sebelum proses dengan ukuran 6-14 cm dijual dengan harga mencapai Rp 450.000/kg dan ukuran > 45 cm mencapai Rp 1.600.000/kg sedangkan harga sirip ikan hiu setelah proses mencapai Rp 950.000/kg untuk ukuran 6-14 cm dan 2.100.000/kg untuk ukuran > 45 cm (Tabel 2). Hal ini menunjukkan
bahwa-6
semakin besar ukuran sirip ikan hiu maka nilai jualnya akan semakin tinggi dan semakin kecil sirip ikan hiu maka nilainya jualnya akan semakin rendah.
Tabel 2. Harga sirip ikan hiu berdasarkan ukuran dan pengolahannya No Ukuran Sirip (cm) Harga Sirip Hiu
(Rp/Kg))
Belum Proses Sudah Proses
1 > 45 1.600.000 2.100.000 2 40 – 44 1.550.000 2.000.000 3 35 – 39 1.450.000 1.950.000 4 30 – 34 1.350.000 1.850.000 5 25 – 29 1.100.000 1.700.000 6 20 – 24 850.000 1.350.000 7 15 – 19 675.000 1.170.000 8 6 – 14 450.000 950.000
Famili hiu Rhynchobatidae hanya teridentifikasi satu spesies yaitu Rhynchobatus australiae (white-spotted guitarfish) dengan jumlah 3 individu dan memiliki pasangan basa nukleotida tertentu (Lampiran 2). Pada penelitian ini hiu spesies Rhynchobatus australiae memiliki panjang basa nukleotida yang teramplifikasi sebesar 612bp dan tingkat kemiripan mencapai 99-100% dengan spesies yang ada pada GeneBank (Lampiran 3). Menurut IUCN redlist (IUCN-SSC 2001) spesies Rhynchobatus australiae merupakan spesies ikan hiu dengan status vulnerable artinya hiu spesies ini memiliki resiko yang tinggi terhadap kepunahan di alam sehingga penangkapannya perlu diatur dengan baik. Rhynchobatus australiae memiliki sirip ekor bagian bawah dengan cuping ekor yang jelas, bentuk moncong seperti botol dan semakin mengecil ke daerah ujung, terdapat bintik - bintik putih di tubuhnya dengan dua bintik hitam pada sirip dada (Lampiran 4). Hiu Rhynchobatus australiae hidup di dasar perairan yang bersubstrat lunak dan dekat dengan terumbu karang dengan panjang tubuhnya hanya mencapai 300 cm (White et al. 1997).
Famili hiu Alopiidae hanya teridentifikasi satu spesies yaitu Alopias superciliosus (bigeyes thresher shark) dengan jumlah 7 individu dan memiliki pasangan basa nukleotida tertentu (Lampiran 2). Hasil penelitian ini menunjukkan ikan hiu jenis Alopias superciliosus memiliki panjang basa nukleotida mencapai 655bp dan tingkat kemiripan mencapai 100% dengan spesies yang ada pada GeneBank (Lampiran 3). Menurut IUCN redlist tahun 2001 hiu jenis Alopias superciliosus termasuk kedalam kategori vulnerable atau rawan.
Penangkapan hiu Alopias superciliosus pada tahun 2004 mencapai 2.023 kg sehingga apabila tidak dilakukan pengaturan stock penangkapan secara teratur maka hiu tersebut akan mengalami penurunan hasil tangkapan atau bahkan kepunahan. Ciri-ciri hiu jenis Alopias superciliosus atau hiu lutung adalah ekor bagian atas hampir sepanjang ukuran tubuhnya, memiliki mata yang sangat besar dengan bagian atasnya hampir mencapai bagian atas kepala, dan memiliki warna putih di bagian perut (Lampiran 4).
7
Hiu lutung dapat tumbuh mencapai 461 cm dan sering dijumpai dekat pantai hingga laut lepas dari permukaan hingga kedalaman 600 m. Makanannya berupa ikan-ikan dasar dan pelagis serta kelompok cepalopoda (White et al. 1997).
Famili hiu Carcharhinidae merupakan famili hiu yang paling mendominasi di PPN Palabuhanratu, bahkan mencapai 16 individu dari total 28 sampel dengan 3 spesies yang berbeda yaitu Carcharhinus falciformis (silky shark) sebanyak 12 individu, Carcharhinus limbatus (blacktip shark) sebanyak 2 individu dan Carcharhinus longimanus (oceanic whitetip shark) sebanyak 2 individu dengan kode pasangan basa nukleotida tertentu (Lampiran 2). Hasil penelitian ini menunjukkan bahwa hiu jenis Carcharhinus falciformis memiliki urutan panjang basa nukleotida mencapai 570-652bp dan tingkat kemiripan spesies dengan spesies GeneBank mencapai 99-100% (Lampiran 3). Menurut IUCN redlist tahun 2001 hiu dengan jenis Carcharhinus falciformis termasuk kedalam kategori near threatened artinya kategori ini diyakini akan terancam keberadaannya di masa mendatang, apabila tidak ada usaha pengelolaan terhadap jenis tersebut.
Carcharhinus falciformis memiliki moncong panjang dan bulat menyempit, gigi atas kecil dengan lekukan di satu sisinya, gigi bawah kecil ramping dan tegak (Lampiran 4). Habitatnya di oseanik dan pelagis, tetapi lebih banyak terdapat di lepas pantai dekat dengan daratan; biasanya dekat dengan permukaan, tetapi kadang dijumpai hingga kedalaman 500 m. Hiu jenis Carcharhinus falciformis tersebar di seluruh perairan tropis dan biasanya memiliki panjang tubuh hingga mencapai 350 cm (White et al. 1997).
Hiu jenis Carcharhinus limbatus memiliki urutan panjang basa nukleotida mencapai 652bp dengan persentase kemiripan spesies dengan spesies yang ada pada GeneBank mencapai 100% (Lampiran 3). Menurut IUCN redlist 2001 hiu jenis Carcharhinus limbatus termasuk kedalam kategori near threatened. Ciri-ciri hiu Carcharhinus limbatus adalah memiliki mocong panjang dan lancip, sirip punggung, dada dan bagian bawah sirip ekor bewarrna polos pada hiu dewasa dan berujung hitam pada hiu muda, memiliki gigi atas dan bawah yang sama, hampir simetris dengan bentuk taring tegak dan ramping (Lampiran 4).
Carcharhinus limbatus memiliki panjang tubuh mencapai 255 cm dan merupakan hiu pelagis yang hidup di paparan benua dan kepulauan; kadang dijumpai di perairan lepas pantai tetapi jarang terdapat pada kedalaman lebih dari 30 m. Makanan utama hiu jenis ini berupa ikan-ikan kecil, termasuk pari kecil, krustasea dan kelompok cumi-cumi (White et al. 1997).
Ikan hiu jenis Carcharhinus longimanus memiliki urutan panjang basa nukleotida mencapai 652bp dengan persentase kemiripan spesies dengan spesies yang ada pada GeneBank mencapai 99% (Lampiran 3). Menurut IUCN redlist (IUCN SSC 2001) hiu jenis Carcharhinus longimanus termasuk kedalam kategori vulnerable. Tingkat penangkapan hiu jenis Carcharhinus longimanus pada tahun 2003 di PPN Palabuhanratu hanya mencapai 303 kg akan tetapi hasil penangkapan hiu jenis ini meningkat pada tahun 2004 mencapai 847 kg (DJPT 2004). Hiu koboy atau hiu Carcharhinus longimanus memiliki ciri-ciri moncong yang pendek, bulat melebar, gigi atas berbentuk segitiga lebar, tegak, bergerigi di tepinya, gigi bawah kecil, dan ramping.
Hiu jenis Carcharhinus longimanus memiliki ujung sirip bewarna putih pada hiu dewasa dan berujung hitam pada juvenil, tergolong kedalam hiu oseanik dan pelagis dari permukaan sampai kedalaman 152 m dan biasanya terdapat jauh di lepas pantai (Lampiran 4). Hiu jenis ini mampu tumbuh hingga mencapai 395 cm. Makanan hiu Carcharhinus longimanus berupa ikan-ikan kecil, cumi-cumi, penyu, udang dan merupakan salah satu jenis hiu yang paling berbahaya bagi manusia (White et al. 1997).
Famili hiu Lamnidae hanya teridentifikasi satu spesies yaitu Isurus paucus (longfin mako) dengan 2 individu dan memiliki kode pasangan basa nukleotida tertentu (Lampiran 2). Hasil penelitian ini menunjukkan bahwa hiu spesies Isurus paucus memiliki urutan panjang basa nukleotida mencapai 645-652bp dan tingkat kemiripan spesies dengan spesies yang ada pada Genebank mencapai 99% (Lampiran 3). Menurut IUCN redlist tahun 2001 hiu spesies Isurus paucus termasuk kedalam kategori vulnerable. Ciri-ciri hiu jenis ini yaitu memiliki moncong lebar meruncing dengan bagian bawah berwarna abu-abu hingga gelap, gigi depan memiliki tepi halus dan lurus, tidak melengkung pada ujungnya dan biasanya dapat tumbuh hingga 417 cm (Lampiran 4). Makanan hiu ini dapat berupa ikan-ikan kecil, udang, dan cumi-cumi, kadang-kadang dijumpai bagian tubuh lumba-lumba dalam pencernaannya dan dapat membahayakan manusia (White et al. 1997).
Hiu yang didaratkan di PPN Palabuhanratu termasuk kedalam kategori vulnerable dan near threatened sehingga pemerintah perlu melakukan usaha pengelolaan perdagangan dengan baik agar kelestarian hiu tersebut tetap terlindungi. Ada beberapa faktor yang menyebabkan beberapa jenis hiu terancam mengalami kepunahan, diantaranya tingkat kematian hiu sangat tinggi akibat tangkapan yang tidak disengaja oleh nelayan, hiu memiliki siklus hidup yang panjang dan relatif tinggi akan tetapi hiu memiliki kemampuan reproduksi yang rendah, serta membutuhkan waktu yang lama sehingga mengakibatkan mudah terjadinya eksploitasi pada hiu. Ikan hiu seperti great white shark, blue shark, longfin mako, shortfin mako dan tiger shark hanya memiliki 2-3 keturunan per tahun dan sangat lambat untuk mencapai usia matang bahkan sampai 10 tahun (Dharmadi dan Fahmi 2005).
Menurut Camhi et al. (1998) menjelaskan selain faktor yang disebutkan diatas ada faktor utama yang menyebabkan hiu terancam mengalami kepunahan yaitu terjadinya penurunan kualitas areal pembesaran ikan daerah-daerah pantai, estuaria maupun air tawar akibat pembangunan, eksploitasi yang berlebihan, dan pencemaran.
Tingkat kesamaan (homogenitas) yang diperoleh dari analisis BLAST cukup tinggi yaitu mencapai 99-100%. Hal ini menunjukkan bahwa pengidentifikasian spesies dengan menggunakan DNA barcoding sangat sesuai. Selain itu, kemampuan teknik DNA barcoding untuk membedakan spesies dapat juga dilihat dengan analisis pohon filogenetik yang dilakukan setelah penentuan urutan nukleotida untuk menunjukkan kedekatan antar spesies sampel.
9
Hubungan Filogenetik
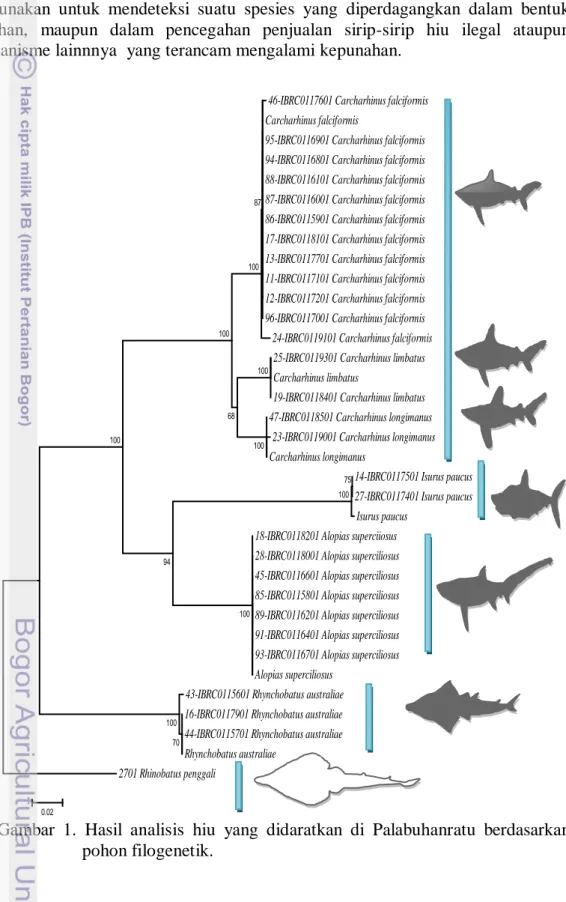
Pohon filogenetik memberikan informasi tentang pengklasifikasian populasi berdasarkan hubungan evolusionernya (Purnomo dan Pudjoarinto 1999). Hasil analisis pohon filogenetik menunjukkan bahwa hiu yang didaratkan di PPN Palabuhanratu terbagi kedalam 4 famili yaitu Rhynchobatidae, Alopiidae, Lamnidae dan Carcharhinidae yang membentuk 4 kelompok besar (Cluster) sesuai dengan familinya. Pohon filogenetik memperlihatkan bahwa hasil analisis BLAST sesuai dengan karakteristik cabang yang dibentuk oleh pohon filogenetik. Ikan hiu yang memiliki sekuen (urutan basa nukleotida) yang sama atau berdekatan akan membentuk kelompok (Cluster) sendiri (berada dalam satu kelompok besar) dan hiu yang memiliki urutan basa nukleotida yang berbeda akan terpisah dan membentuk kelompok tersendiri.
Berdasarkan konstruksi pohon filogenetik diketahui bahwa kelompok pertama ditempati oleh famili Carcharhinidae dengan hiu jenis Carcharhinus falciformis, Carcharhinus limbatus dan Carcharhinus longimanus. Ikan hiu jenis Carcharhinus limbatus dan Carcharhinus longimanus membentuk cabang pohon filogenetik yang terpisah akan tetapi masih berada dalam satu kelompok besar. Pengelompokkan pohon filogenetik dalam satu kelompok untuk famili Carcharhinidae yang sama menggambarkan kedekatan kekerabatannya. Semakin banyak urutan nukleotida (sekuen) yang sama maka nilai similaritasnya akan semakin tinggi sehingga posisinya di dalam percabangan pohon filogenetik akan semakin berdekatan (Gambar 1).
Kelompok yang kedua ditempati oleh famili Lamnidae yaitu hiu dengan jenis Isurus paucus. Hiu jenis Isurus paucus terletak pada satu pohon karena memiliki sekuen yang sama sehingga memiliki hubungan kekerabatan yang tinggi dengan nomor kode akses pada GeneBank FJ519011.1. Kelompok yang ketiga pada pohon filogenetik ditempati oleh famili Alopiidae yaitu hiu jenis Alopias superciliosus. Famili Alopiidae memiliki sekuen yang sama sehingga terletak pada satu kelompok dan memiliki hubungan kekerabatan yang tinggi dengan nomor akses pada GeneBank DQ108329.1 (Gambar 1).
Kelompok terakhir yaitu kelompok keempat ditempati oleh anggota famili Rhyncobatidae yaitu hiu jenis Rhynchobatus australiae. Famili Rhynchobatidae memiliki urutan basa nukleotida yang sama dan berdekatan sehingga terletak dalam satu pohon filogenetika. Famili Rhynchobatidae memiliki nilai similaritas yang tinggi dengan kode akses GeneBank JN022595.1 (Gambar 1).
Grup yang berada paling jauh merupakan sekuen dari ikan pari jenis Rhinobatus penggali yang digunakan sebagai outgroup. Sekuen dari outgroup semestinya berkorelasi dekat dengan sekuen-sekuen yang dianalisis, tetapi juga mempunyai perbedaan yang signifikan antara outgroup dengan sekuen yang lain. Nilai boostrap ditunjukkan pada angka-angka yang terletak pada cabang-cabang pohon filogenetik. Boostrap merupakan alat bantu umum yang biasa digunakan untuk mencari pendekatan dalam distribusi statistik yang dikehendaki, dengan kata lain nilai boostrap bertujuan untuk mengetahui dan menguji seberapa baik set data model yang digunakan. Jika nilai boostrap rendah, sekuen seharusnya dikeluarkan dari analisis untuk mendapatkan sebuah pohon filogenetika yang dapat di percaya.
10
Penelitian ini menunjukkan rata-rata nilai boostrap melebihi 90, perhitungan nilai boostrap yang lebih dari 90 menunjukkan tingkat kesamaan spesies yang tinggi dengan database yang ada dan rekontsruksi pohon filogenetika dapat dipercaya keakuratannya. Tingginya kemampuan untuk mengidentifikasi hubungan antar spesies dengan analisis pohon filogentik ini membuktikan bahwa teknik DNA barcoding dan rekonstruksi pohon filogenetik sangat cocok digunakan untuk mendeteksi suatu spesies yang diperdagangkan dalam bentuk olahan, maupun dalam pencegahan penjualan sirip-sirip hiu ilegal ataupun organisme lainnnya yang terancam mengalami kepunahan.
Gambar 1. Hasil analisis hiu yang didaratkan di Palabuhanratu berdasarkan pohon filogenetik.
46-IBRC0117601 Carcharhinus falciformis Carcharhinus falciformis
95-IBRC0116901 Carcharhinus falciformis 94-IBRC0116801 Carcharhinus falciformis 88-IBRC0116101 Carcharhinus falciformis 87-IBRC0116001 Carcharhinus falciformis 86-IBRC0115901 Carcharhinus falciformis 17-IBRC0118101 Carcharhinus falciformis 13-IBRC0117701 Carcharhinus falciformis 11-IBRC0117101 Carcharhinus falciformis 12-IBRC0117201 Carcharhinus falciformis 96-IBRC0117001 Carcharhinus falciformis 24-IBRC0119101 Carcharhinus falciformis 25-IBRC0119301 Carcharhinus limbatus Carcharhinus limbatus
19-IBRC0118401 Carcharhinus limbatus 47-IBRC0118501 Carcharhinus longimanus
23-IBRC0119001 Carcharhinus longimanus Carcharhinus longimanus
14-IBRC0117501 Isurus paucus 27-IBRC0117401 Isurus paucus Isurus paucus
18-IBRC0118201 Alopias superciiosus 28-IBRC0118001 Alopias superciliosus 45-IBRC0116601 Alopias superciliosus 85-IBRC0115801 Alopias superciliosus 89-IBRC0116201 Alopias superciliosus 91-IBRC0116401 Alopias superciliosus 93-IBRC0116701 Alopias superciliosus Alopias superciliosus
43-IBRC0115601 Rhynchobatus australiae 16-IBRC0117901 Rhynchobatus australiae 44-IBRC0115701 Rhynchobatus australiae Rhynchobatus australiae 2701 Rhinobatus penggali 70 100 75 100 100 94 100 100 100 68 100 100 87 0.02
11
KESIMPULAN DAN SARAN Kesimpulan
Spesies ikan hiu yang teridentifikasi secara molekuler yaitu 3 ekor hiu jenis Rhyncobatus australiae, 7 ekor hiu jenis Alopias superciliosus, 12 ekor hiu jenis Carcharhinus falciformis, 2 ekor hiu jenis Isurus paucus, 2 ekor hiu jenis Carcharhinus limbatus dan 2 ekor hiu jenis Carcharhinus longimanus. Hiu yang didaratkan di Pelabuhan Perikanan Palabuhanratu termasuk kedalam kategori vulnerable (rawan) dan near threatened (hampir terancam).
Konstruksi pohon filogenetik menunjukkan bahwa hiu yang didaratkan terbagi kedalam 4 kelompok besar dengan 4 famili yang berbeda yaitu Rhynchobatidae, Alopiidae, Lamnidae dan Carcharhinidae. Tingkat kesamaan (homogenitas) yang diperoleh dari hasil analisis BLAST cukup tinggi, berkisar antara 99-100%. Hal ini membuktikan bahwa hasil analisis DNA barcoding dan pohon filogenetik dapat digunakan untuk mengidentifikasi spesies secara molekuler yang diperdagangkan secara tidak utuh atau berupa produk olahan misalnya identifikasi jenis ikan hiu dalam bentuk sirip.
Saran
Perlu penelitian lanjutan untuk mengetahui variasi keragaman genetik ikan hiu dengan sampel yang lebih banyak dan lokasi penelitian di seluruh Indonesia sehingga pemerintah Indonesia memiliki data dasar dan status hiu yang di perdagangkan di Pelabuhan Perikanan. Hal ini dapat menjadi acuan bagi pengelolaan perdagangan hiu di Indonesia, sehingga kelestarian hiu tetap terlindungi.
DAFTAR PUSTAKA
Allen G. 1997. Marine Fishes of Tropical Australia and South-East Asia : A Field Guide For Anglers And Divers. Western Australian Museum, 292 pp.
Camhi M, Flowler S, Musick J, Brautigam A and Fordham S. 1998. Sharks and their relatives, ecology and conservation. Occasional Paper of the IUCN Spesies Survival Commission No.20. IUCN, Gland, Switzerland and Cambridge, UK.39p.
Campagno LJV. 1984a. FAO Spesies Catalogue. Vol 4. Sharks of the World. An Annoted and Illustrate Catalogue of Sharks Spesies Known to Date. Part 1. “ Hexanchiformes to Lamniformes”. FAO Fish. Synop (125) Vol 4, Pt 1:249p.
Dharmadi dan Fahmi. 2003. Fisheries characteristic of artisanal sharks and rays in the Indonesian waters. Proc. Seminar on Marine and Fisheries Jakarta. Agency for Marine and Fisheries Research. MMAF. p.122-129.
Dharmadi dan Fahmi. 2005. Status Perikanan Hiu dan Aspek Pengelolaannya. Oseana. 30(1): 1-8.
[DJPT] Direktorat Perikanan Tangkap. 1992. Statistik Perikanan Indonesia Tahun 2000. Direktorat Perikanan Tangkap. Jakarta. Hal 38-43.
12
[DJPT] Direktorat Perikanan Tangkap. 2004. Statistik Perikanan Tahun 2004 Pelabuhan Perikanan Nusantara Palabuhanratu. Palabuhanratu. 78 hal. Duryadi D. 1994. Peran DNA mitokondria (mtDNA) dalam studi keragaman
genetik dan biologi populasi pada hewan. Hayati 1(1):1-4.
Folmer O, Black M, Hoeh W, Lutz R and Vrijenhoek R. 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Molecular Marine Biology and Biotecnology 3:294-297.
Hebert PDN, Cywinska A, Ball SL, deWaard JR. 2003. Biological identifications through DNA barcodes, Proc, R.Soc.B 270, 313-321.
IUCN-SSC. 2001. IUCN Red list categories and criteria IUCN-The World Conservation Union. Gland. Swizerland and Cambridge, UK.34p.
Kyle CJ, Wilson CC. 2007. Mitochondrial DNA identification of game and harvested freshwater fish spesies. Forensic Sciece Internasional 166(1): 68-76.
Li S, Pearl DK and Doss H. 1999. Phylogenetic tree construction using Markov ChainMonte Carlo.Fred Huntchinson Cancer Research Center Washington. http:// www.gwu.edu/~clade/faculty/lipscomb/Cladistic.pdf (14 February 2013).
Mabrouk MS, Hamdy M, Mamdouh M, Aboelfotoh M, and Kadah YM. 2006. BIOINFTool: Bioinformatics and sequence data analysis in molecular biology using Matlab. Proc. Cairo International Biomedical Engineering Conference.
Pariwono JI, Eidman M, Santoso R, Purba M, Prartono T, Widodo, Djuariah U dan Hutapea JA. 1988. Studi upwelling di Perairan Pulau Jawa. Fakultas Perikanan. IPB. Bogor.
Purnomo dan Pudjoarinto. 1999. Struktur Perkembangan I Morfologi Tumbuhan. Fakultas Biologi UGM. Yogyakarta, hal. 1-3.
Rahayu DS. 1999. Studi Tentang Komposisi Hasil Tangkapan Rawai layur pada siang dan malam hari di Perairan Pelabuhan Ratu, Sukabumi. Jawa Barat [Skripsi]. Bogor (ID): Institut Pertanian Bogor.
Sanusi HS. 1994. Karakteristik Kimia dan Kesuburan Perairan Teluk Pelabuhan Ratu (Tahap II-Musim Timur). Ilmu-Ilmu Perairan dan Perikanan Indonesia 11(2): 93-100.
Tamura K, Dudley J, Nei M, Kumar S. 2007. MEGA: Molecular Evolutionary Genetic Analysis (MEGA) software version 5.05. Advance Access published May 7. Oxford University Press. Mol Bio 10. 1093/molbev/msm092.
Ward RD, Holmes BH. 2007. An analysis of Nucleotide and Amino Acid
Variability in the Barcode Region of Cytochrome C Oxidase (CO 1) in Fishes. Mol. Ecol. Notes (7): 899-907.
Walsh PS, Mezger DA, Higuchi R. 1991. Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material. Biotecniques. 1991 Apr 10(4):506.
White WT, Last PR, Stevens JD, Yearsley GK, Fahmi, Dharmadi. 1997. Economically important sharks and rays of Indonesia. National Library of Australia Catalouguing in Publication entry. ACIAR monograph series ; no. 124:1-338.
14
Lampiran 1. Prosedur Kerja PCR
1. Gunakan Gloves (sarung tangan) kemudian keluarkan reagen: Air, dNTP, buffer PCR, MgCl2, Primer 1 dan Primer 2 dari freezer untuk mencairkan. 2. Isi lembar kerja PCR dengan tanggal, jumlah sampel, tipe ekstraksi dan
catatan lainnya.
3. Tandai dan nomori tabung PCR dalam rack
4. Setelah bahan cair, jentikkan setiap tabung dengan jari untuk mencampur, kocok isi hingga dasar tabung.
5. Buat campuran MM 1 : gunakan pipet NO DNA, tambahkan bahan sesuai dengan volume yang telah dihitung dalam daftar di lembar PCR di tabung 1,5mL. Gunakan tip berbeda untuk setiap penambahan reagen. Pipet naik turun untuk mencampur reagen sepenuhnya.
6. Gunakan pipet NO DNA, buat MM 2 dalam tabung 1,5mL terpisah, tetapi jangan menambahkan taq. Gunakan tip berbeda untuk setiap penambahan reagen. Pipet naik turun untuk mencampur reagen sepenuhnya.
7. Gunakan pipet NO DNA, bagi 14 μL MM1 ke dalam setiap tabung PCR. 8. Pindahkan DNA ekstra Chelex dari ruang pendingin dan jika perlu,
sentrifugasi singkat untuk menghilangkan kondensasi. Gunakan pipet DNA rendah, tambahkan 1 μL DNA ekstrak untuk setiap tabung.
9. Ambil taq sesuai yang di inginkan dari freezer kemudian tambahkan kedalam MM2 dan pipet naik turun untuk mencampur.
10. Pilih dan mulai program hot-star PCR. Biarkan penutup panas dan tahan sebentar sampai suhu mencapai 800 C. Kemudian tempatkan strip tabung ke dalam mesin PCR.
11. Atur hingga 10μL pipetman, pipet naik turun MM2 untuk mencampur, kemudian tambahkan MM2 ke dalam masing-masing tabung dan ganti tip untuk setiap sampel.
12. Unpause program dan lihat layar mesin PCR untuk menjamin bahwa mesin PCR sedang bekerja.
13. Bersihkan tempat kerja, letakkan reagen ke dalam freezer dan ekstrak DNA ke dalam lemari pendingin.
15
Lampiran 2. Hasil pengurutan basa nukleotida (sequencing) pada sampel ikan hiu di Pelabuhan Perikanan Palabuhanratu, Sukabumi, Jawa Barat.
Famili Rhynchobatidae
Rhynchobatus australiae _ IBRC01.156.01
ATTCGAACAGAACTTAGTCAACCAGGAACACTTCTTGGAGATGACCAAATCTACAAT GTGGTTGTAACAGCCCATGCCTTTGTGATAATCTTTTTCATGGTTATACCAATCATGAT CGGAGGCTTTGGGAATTGACTAGTCCCCCTAATAATTGGTGCTCCAGACATAGCATTC CCACGAATAAATAACATAAGCTTCTGATTATTACCTCCATCATTTCTTCTTTTATTGGC CTCTGCCGGAGTTGAAGCTGGAGCTGGAACAGGTTGGACTGTGTATCCTCCCCTTGCT GGCAATCTTGCCCATGCCGGAGCCTCTGTAGACTTACCCATCTTTTCATTACATTTAGC AGGAATTTCCTCAATTTTAGCATCTATTAATTTTATCACAACTATTATCAACATGAAAC CCCCAGCAATTTCCCAATACCAAACACCTCTATTCGTATGATCAATTCTTGTAACAACT ATCTTACTCTTACTATCACTACCAGTTCTAGCAGCAGGCATTACTATACTCCTCACAGA TCGTAATCTCAATACAACCTTCTTTGATCCAGCGGGGGGAGGAGATCCAATCCTTTAC CAACACTTATTCTGATTT
Rhynchobatus australiae _ IBRC01.157.01
ACCCTATACTTGATTTTCGGTGCTTGAGCAGGAATAGTTGGTACTGGTCTGAGTCTGCT CATTCGAACAGAACTTAGTCAACCAGGAACACTTCTTGGAGATGACCAAATCTACAAT GTGGTTGTAACAGCCCATGCCTTTGTGATAATCTTTTTCATGGTTATACCAATCATGAT CGGAGGCTTTGGGAATTGACTAGTCCCCCTAATAATTGGTGCTCCAGACATAGCATTC CCACGAATAAATAACATAAGCTTCTGATTATTACCTCCATCATTTCTTCTTTTATTGGC CTCTGCCGGAGTTGAAGCTGGAGCTGGAACAGGTTGGACTGTGTATCCACCCCTTGCT GGCAATCTTGCCCATGCCGGAGCCTCTGTAGACTTAGCCATCTTTTCATTACATTTAGC AGGAATTTCCTCAATTTTAGCATCTATTAATTTTATCACAACTATTATCAACATGAAAC CCCCAGCAATTTCCCAATACCAAACACCTCTATTCGTATGATCAATTCTTGTAACAACT ATCTTACTCTTACTATCACTACCAGTTCTAGCAGCAGGCATTACTATACTCCTCACAGA TCGTAATCTCAATACAACCTTCTTTGATCCAGCGGGGGGAGGAGATCCAATCCTTTAC CAACACTTATTCTGATTTT
Rhynchobatus australiae _ IBRC01.179.01
ACCCTATACTTGATTTTCGGTGCTTGAGCAGGAATAGTTGGTACTGGTCTGAGTCTGCT CATTCGAACAGAACTTAGTCAACCAGGAACACTTCTTGGAGATGACCAAATCTACAAT GTGGTTGTAACAGCCCATGCCTTTGTGATAATCTTTTTCATGGTTATACCAATCATGAT CGGAGGCTTTGGGAATTGACTAGTCCCCCTAATAATTGGTGCTCCAGACATAGCATTC CCACGAATAAATAACATAAGCTTCTGATTATTACCTCCATCATTTCTTCTTTTATTGGC CTCTGCCGGAGTTGAAGCTGGAGCTGGAACAGGTTGGACTGTGTATCCACCCCTTGCT GGCAATCTTGCCCATGCCGGAGCCTCTGTAGACTTAGCCATCTTTTCATTACATTTAGC AGGAATTTCCTCAATTTTAGCATCTATTAATTTTATCACAACTATTATCAACATGAAAC CCCCAGCAATTTCCCAATACCAAACACCTCTATTCGTATGATCAATTCTTGTAACAACT ATCTTACTCTTACTATCACTACCAGTTCTAGCAGCAGGCATTACTATACTCCTCACA GATCGTAATCTCAATACAACCTTCTTTGATCCAGCGGGGGGAGGAGATCCAATCCTTT ACCAACACTTATTCTGATTT Famili Alopiidae
Alopias superciliosus _ IBRC01.158.01
ACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCCTCAGCCTTC TAATTCGAGCCGAGTTAGGCCAGCCCGGATCACTCCTAGGGGATGATCAGGTCTATAA TGTTATCGTAACCGCCCATGCATTTGTAATAATCTTCTTCATGGTTATACCCGTAATAA TTGGGGGATTTGGAAACTGATTAGTACCCTTAATAATTGGTGCACCAGACATGGCCTT CCCGCGAATAAATAACATAAGCTTTTGACTCCTTCCCCCTTCTTTTCTCTTACTCCTAG CTTCAGCTGGGGTTGAAGCTGGAGCTGGCACTGGTTGAACAGTTTATCCCCCCTTAGC TGGCAACTTAGCACATGCTGGGGCATCTGTTGACTTGGCCATTTTCTCGCTTCATTTAG CAGGTATCTCATCAATTTTAGCTTCAATTAACTTTATTACAACTATCATTAATATAAAA CCACCAGCCATCTCTCAATATCAAACACCATTATTTGTATGATCAATCCTAGTAACAA CCATCCTCCTCCTCTTATCCCTCCCAGTACTCGCAGCCGGCATCACAATATTATTAACT
16
GATCGAAACCTAAACACAACATTCTTTGACCCAGCAGGAGGAGGAGATCCAATTCTTT ACCAACATCTATTTTGATTTTTT
Alopias superciliosus _ IBRC01.162.01
ACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCCTCAGCCTTC TAATTCGAGCCGAGTTAGGCCAGCCCGGATCACTCCTAGGGGATGATCAGGTCTATAA TGTTATCGTAACCGCCCATGCATTTGTAATAATCTTCTTCATGGTTATACCCGTAATAA TTGGGGGATTTGGAAACTGATTAGTACCCTTAATAATTGGTGCACCAGACATGGCCTT CCCGCGAATAAATAACATAAGCTTTTGACTCCTTCCCCCTTCTTTTCTCTTACTCCTAG CTTCAGCTGGGGTTGAAGCTGGAGCTGGCACTGGTTGAACAGTTTATCCCCCCTTAGC TGGCAACTTAGCACATGCTGGGGCATCTGTTGACTTGGCCATTTTCTCGCTTCATTTAG CAGGTATCTCATCAATTTTAGCTTCAATTAACTTTATTACAACTATCATTAATATAAAA CCACCAGCCATCTCTCAATATCAAACACCATTATTTGTATGATCAATCCTAGTAACAA CCATCCTCCTCCTCTTATCCCTCCCAGTACTCGCAGCCGGCATCACAATATTATTAACT GATCGAAACCTAAACACAACATTCTTTGACCCAGCAGGAGGAGGAGATCCAATTCTTT ACCAACATCTATTTTGATTTTTTGGTCAC
Alopias superciliosus _ IBRC01.164.01
ACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCCTCAGCCTTC TAATTCGAGCCGAGTTAGGCCAGCCCGGATCACTCCTAGGGGATGATCAGGTCTATAA TGTTATCGTAACCGCCCATGCATTTGTAATAATCTTCTTCATGGTTATACCCGTAATAA TTGGGGGATTTGGAAACTGATTAGTACCCTTAATAATTGGTGCACCAGACATGGCCTT CCCGCGAATAAATAACATAAGCTTTTGACTCCTTCCCCCTTCTTTTCTCTTACTCCTAG CTTCAGCTGGGGTTGAAGCTGGAGCTGGCACTGGTTGAACAGTTTATCCCCCCTTAGC TGGCAACTTAGCACATGCTGGGGCATCTGTTGACTTGGCCATTTTCTCGCTTCATTTAG CAGGTATCTCATCAATTTTAGCTTCAATTAACTTTATTACAACTATCATTAATATAAAA CCACCAGCCATCTCTCAATATCAAACACCATTATTTGTATGATCAATCCTAGTAACAA CCATCCTCCTCCTCTTATCCCTCCCAGTACTCGCAGCCGGCATCACAATATTATTAA CTGATCGAAACCTAAACACAACATTCTTTGACCCAGCAGGAGGAGGAGATCCAATTC TTTACCAACATCTATTTTGATTTTTTGGTCAC
Alopias superciliosus _ IBRC01.166.01
ACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCCTCAGCCTTC TAATTCGAGCCGAGTTAGGCCAGCCCGGATCACTCCTAGGGGATGATCAGGTCTATAA TGTTATCGTAACCGCCCATGCATTTGTAATAATCTTCTTCATGGTTATACCCGTAATAA TTGGGGGATTTGGAAACTGATTAGTACCCTTAATAATTGGTGCACCAGACATGGCCTT CCCGCGAATAAATAACATAAGCTTTTGACTCCTTCCCCCTTCTTTTCTCTTACTCCTAG CTTCAGCTGGGGTTGAAGCTGGAGCTGGCACTGGTTGAACAGTTTATCCCCCCTTAGC TGGCAACTTAGCACATGCTGGGGCATCTGTTGACTTGGCCATTTTCTCGCTTCATTTAG CAGGTATCTCATCAATTTTAGCTTCAATTAACTTTATTACAACTATCATTAATATAAAA CCACCAGCCATCTCTCAATATCAAACACCATTATTTGTATGATCAATCCTAGTAACAA CCATCCTCCTCCTCTTATCCCTCCCAGTACTCGCAGCCGGCATCACAATATTATTAACT GATCGAAACCTAAACACAACATTCTTTGACCCAGCAGGAGGAGGAGATCCAATTCTTT ACCAACATCTATTTTGATTTT
Alopias superciliosus _ IBRC01.167.01
ACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCCTCAGCCTTC TAATTCGAGCCGAGTTAGGCCAGCCCGGATCACTCCTAGGGGATGATCAGGTCTATAA TGTTATCGTAACCGCCCATGCATTTGTAATAATCTTCTTCATGGTTATACCCGTAATAA TTGGGGGATTTGGAAACTGATTAGTACCCTTAATAATTGGTGCACCAGACATGGCCTT CCCGCGAATAAATAACATAAGCTTTTGACTCCTTCCCCCTTCTTTTCTCTTACTCCTAG CTTCAGCTGGGGTTGAAGCTGGAGCTGGCACTGGTTGAACAGTTTATCCCCCCTTAGC TGGCAACTTAGCACATGCTGGGGCATCTGTTGACTTGGCCATTTTCTCGCTTCATTTAG CAGGTATCTCATCAATTTTAGCTTCAATTAACTTTATTACAACTATCATTAATATAAAA CCACCAGCCATCTCTCAATATCAAACACCATTATTTGTATGATCAATCCTAGTAACAA CCATCCTCCTCCTCTTATCCCTCCCAGTACTCGCAGCCGGCATCACAATATTATTAACT GATCGAAACCTAAACACAACATTCTTTGACCCAGCAGGAGGAGGAGATCCAATTCTTT ACCAACATCTATTTTGATTTTTTGGTCAC
17
Alopias superciliosus _ IBRC01.180.01
ACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCCTCAGCCTTC TAATTCGAGCCGAGTTAGGCCAGCCCGGATCACTCCTAGGGGATGATCAGGTCTATAA TGTTATCGTAACCGCCCATGCATTTGTAATAATCTTCTTCATGGTTATACCCGTAATAA TTGGGGGATTTGGAAACTGATTAGTACCCTTAATAATTGGTGCACCAGACATGGCCTT CCCGCGAATAAATAACATAAGCTTTTGACTCCTTCCCCCTTCTTTTCTCTTACTCCTAG CTTCAGCTGGGGTTGAAGCTGGAGCTGGCACTGGTTGAACAGTTTATCCCCCCTTAGC TGGCAACTTAGCACATGCTGGGGCATCTGTTGACTTGGCCATTTTCTCGCTTCATTTAG CAGGTATCTCATCAATTTTAGCTTCAATTAACTTTATTACAACTATCATTAATATAAAA CCACCAGCCATCTCTCAATATCAAACACCATTATTTGTATGATCAATCCTAGTAACAA CCATCCTCCTCCTCTTATCCCTCCCAGTACTCGCAGCCGGCATCACAATATTATTAA CTGATCGAAACCTAAACACAACATTCTTTGACCCAGCAGGAGGAGGAGATCCAATTC TTTACCAACATCTATTTTGATTT
Alopias superciliosus _ IBRC01.182.01
ACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCCTCAGCCTTC TAATTCGAGCCGAGTTAGGCCAGCCCGGATCACTCCTAGGGGATGATCAGGTCTATAA TGTTATCGTAACCGCCCATGCATTTGTAATAATCTTCTTCATGGTTATACCCGTAATAA TTGGGGGATTTGGAAACTGATTAGTACCCTTAATAATTGGTGCACCAGACATGGCCTT CCCGCGAATAAATAACATAAGCTTTTGACTCCTTCCCCCTTCTTTTCTCTTACTCCTAG CTTCAGCTGGGGTTGAAGCTGGAGCTGGCACTGGTTGAACAGTTTATCCCCCCTTAGC TGGCAACTTAGCACATGCTGGGGCATCTGTTGACTTGGCCATTTTCTCGCTTCATTTAG CAGGTATCTCATCAATTTTAGCTTCAATTAACTTTATTACAACTATCATTAATATAAAA CCACCAGCCATCTCTCAATATCAAACACCATTATTTGTATGATCAATCCTAGTAACAA CCATCCTCCTCCTCTTATCCCTCCCAGTACTCGCAGCCGGCATCACAATATTATTAACT GATCGAAACCTAAACACAACATTCTTTGACCCAGCAGGAGGAGGAGATCCAATTCTTT ACCAACATCTATTTTGATTT Famili Carcharhinidae
Carcharhinus falciformis _ IBRC01.159.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGAATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTTTTT
Carcharhinus falciformis _ IBRC01.160.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGGATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTTTTTGGTCAC
18
Carcharhinus falciformis _ IBRC01.161.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGAATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTTTTTGGT
Carcharhinus falciformis _ IBRC01.168.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGGATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTT
Carcharhinus falciformis _IBRC01.169.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGAATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTTTTTGGT
Carcharhinus falciformis _IBRC01.170.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTT
Carcharhinus falciformis _IBRC01.171.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC
19 CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGAATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTT
Carcharhinus falciformis _IBRC01.172.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGGATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTTTTTGGTCAC
Carcharhinus falciformis _IBRC01.177.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGGATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTTTTT
Carcharhinus falciformis _IBRC01.181.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGGATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTTTTT
Carcharhinus falciformis _IBRC01.176.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGTTATGCCAATCATAAT
TGGTGGTTTCGGAAATTGACTAGTTCCTTTAATAATTGGTGCACCAAATATAGCCTTCC CACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTTCTCCTCGCC TCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCTA GTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGCC GGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAACC ACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACTA TTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGAATTACAATATTACTTACAGAT CGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTTT
Carcharhinus falciformis _IBRC01.191.01
ACCCTTTACGTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTC TAATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAA TGTAATCGTAACCGCCCACGCTTTTGTAATAATATTTTTTATGGTTATGCCAATCATAA TTGGTGGTTTCGGAAATTGATTAGTTCCTTTAATAATTGGTGCACCAGATATAGCCTTC CCACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTATTCTCCTCGC CTCTGCTGGAGTAGAAGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCT AGTAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGC CGGTGTGTCATCTATTCTAGCTTCAATTAATTTTATTACAACTATTATCAATATAAAAC CACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACT ATTCTTCTCCTCCTATCACTTCCAGTTCTTGCAGCAGGGATTACAATATTACTTACAGA TCGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTATC AACATTTATTTTGATTTTTTGGTCACCCT
Carcharhinus limbatus _ IBRC01.184.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTCGGAACAGCCCTAAGTCTCC TAATTCGAGCTGAACTTGGGCAACCTGGATCACTTTTAGGGGATGATCAGATTTATAA TGTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATAGTTATACCAATTATAA TTGGTGGTTTCGGAAATTGACTAGTTCCTCTAATAATTGGTGCACCAGACATAGCCTTT CCACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTCCTTCTCGC CTCTGCTGGAGTAGAAGCTGGGGCAGGTACTGGTTGAACAGTGTATCCTCCATTAGCT AGCAACTTAGCACATGCTGGACCATCTGTTGATTTAGCCATTTTCTCTCTTCACTTAGC TGGTGTTTCATCAATTTTAGCTTCAATTAATTTTATTACAACTATTATTAATATAAAAC CACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTGACCACT ATTCTTCTTCTCCTTTCACTTCCAGTCCTTGCAGCAGGGATTACAATATTACTTACAGA TCGTAACCTCAATACCACATTCTTTGATCCTGCAGGTGGAGGAGATCCAATCCTTTAT CAACATTTATTTTGATTTTTT
Carcharhinus limbatus _ IBRC01.193.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTCGGAACAGCCCTAAGTCTCC TAATTCGAGCTGAACTTGGGCAACCTGGATCACTTTTAGGGGATGATCAGATTTATAA TGTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATAGTTATACCAATTATAA TTGGTGGTTTCGGAAATTGACTAGTTCCTCTAATAATTGGTGCACCAGACATAGCCTTT CCACGAATAAATAACATAAGTTTCTGACTTCTTCCACCATCATTTCTTCTCCTTCTCGC CTCTGCTGGAGTAGAAGCTGGGGCAGGTACTGGTTGAACAGTGTATCCTCCATTAGCT AGCAACTTAGCACATGCTGGACCATCTGTTGATTTAGCCATTTTCTCTCTTCACTTAGC TGGTGTTTCATCAATTTTAGCTTCAATTAATTTTATTACAACTATTATTAATATAAAAC CACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTGACCACT ATTCTTCTTCTCCTTTCACTTCCAGTCCTTGCAGCAGGGATTACAATATTACTTACAGA TCGTAACCTCAATACCACATTCTTTGATCCTGCAGGTGGAGGAGATCCAATCCTTTAT CAACATTTATTTTGATTT
Carcharhinus longimanus _ IBRC01.185.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAACTTGGGCAACCTGGATCACTTTTAGGAGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTCATGGTTATACCAATTATAAT
TGGTGGTTTCGGAAATTGATTAGTCCCTTTAATAATTGGAGCACCAGATATAGCCTTC CCACGAATAAATAACATAAGTTTCTGACTTCTTCCACCGTCATTTCTTCTCCTTCTCGC CTCTGCTGGAGTAGAGGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCT AGCAACCTAGCACATGCTGGACCATCTGTTGATTTAGCTATTTTCTCTCTTCACTTAGC CGGTGTTTCATCAATTTTAGCTTCAATTAATTTTATCACAACCATTATTAATATAAAAC CACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACT ATTCTTCTTCTCCTCTCACTTCCAGTTCTTGCAGCAGGGATTACAATATTACTTACAGA CCGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTAT CAACATTTATTTTGATTTTTTG
Carcharhinus longimanus _ IBRC01.190.01
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTCTT AATTCGAGCTGAACTTGGGCAACCTGGATCACTTTTAGGAGATGATCAGATTTATAAT GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTCATGGTTATACCAATTATAAT TGGTGGTTTCGGAAATTGATTAGTCCCTTTAATAATTGGAGCACCAGATATAGCCTTC CCACGAATAAATAACATAAGTTTCTGACTTCTTCCACCGTCATTTCTTCTCCTTCTCGC CTCTGCTGGAGTAGAGGCTGGAGCAGGTACTGGTTGAACAGTTTATCCTCCATTAGCT AGCAACCTAGCACATGCTGGACCATCTGTTGATTTACCTATTTTCTCTCTTCACTTAGC CGGTGTTTCATCAATTTTAGCTTCAATTAATTTTATCACAACCATTATTAATATAAAAC CACCAGCCATTTCCCAATATCAAACACCATTATTTGTTTGATCTATTCTTGTAACCACT ATTCTTCTTCTCCTCTCACTTCCAGTTCTTGCAGCAGGGATTACAATATTACTTACAGA CCGTAACCTTAATACTACATTCTTTGATCCTGCAGGTGGAGGAGACCCAATCCTTTAT CAACATTTATTTTGATTTTTT Famili Lamnidae
Isurus paucus _ IBRC01.174.01
ACCCTGTATTTAATCTTTGGTGCATGAGCAGGGATAGTGGGAACAGCCCTAAGCCTTC TAATTCGCGCCGAGCTGGGTCAGCCAGGTTCTCTTCTAGGGGACGATCAGATTTATAA TGTTATTGTAACCGCCCATGCATTTGTAATGATTTTCTTCATGGTAATGCCCGTGATAA TTGGGGGCTTTGGGAACTGACTGGTGCCTTTAATGATCGGTGCACCCGATATGGCCTT CCCCCGAATAAACAACATGAGCTTCTGACTCCTCCCCCCTTCTTTTCTCTTACTGCTAG CCTCAGCCGGGGTTGAATCAGGGGCTGGAACTGGCTGGACAGTTTACCCTCCCCTAGC TGGTAACTTAGCACATGCTGGGGCATCTGTTGACTTAGCTATCTTCTCCCTTCACCTAG CAGGTATCTCGTCAATTCTGGCCTCTATTAACTTCATCACGACAATCATCAACATAAA ACCACCAGCAATTTCTCAGTACCAAACACCCCTATTTGTGTGATCCATCCTAGTAACA ACTATCCTGCTCCTTCTAGCCCTCCCAGTACTCGCCGCCGGCATTACAATACTACTTAC GGACCGAAACCTGAACACAACATTCTTTGACCCGGCAGGAGGGGGAGATCCTATCCT CTACCAACATCTATTCTGATTT
Isurus paucus _ IBRC01.175.01
ACCCTGTATTTAATCTTTGGTGCATGAGCAGGGATAGTGGGAACAGCCCTAAGCCTTC TAATTCGCGCCGAGCTGGGTCAGCCAGGTTCTCTTCTAGGGGACGATCAGATTTATAA TGTTATTGTAACCGCCCATGCATTTGTAATGATTTTCTTCATGGTAATGCCCGTGATAA TTGGGGGCTTTGGGAACTGACTGGTGCCTTTAATGATCGGTGCACCCGATATGGCCTT CCCCCGAATAAACAACATGAGCTTCTGACTCCTCCCCCCTTCTTTTCTCTTACTGCTAG CCTCAGCCGGGGTTGAATCAGGGGCTGGAACTGGCTGGACAGTTTACCCTCCCCTAGC TGGTAACTTAGCACATGCTGGGGCATCTGTTGACTTAGCTATCTTCTCCCTTCACCTAG CAGGTATCTCGTCAATTCTGGCCTCTATTAACTTCATCACGACAATCATCAACATAAA ACCACCAGCAATTTCTCAGTACCAAACACCCCTATTTGTGTGATCCATCCTAGTAACA ACTATCCTGCTCCTTCTAGCCCTCCCAGTACTCGCCGCCGGCATTACAATACTACTTAC GGACCGAAACCTGAACACAACATTCTTTGACCCGGCAGGAGGGGGAGATCCTATCCT CTACCAACATCTATTCTGA
Lampiran 3. Tabel hasil identifikasi ikan hiu yang didaratkan di Pelabuhan Perikanan Palabuhanratu dengan menggunakan BLAST.
No Kode Sampel Famili Analisis
BLAST IUCN red list Simililarity (%) GeneBank Accession
1 IBRC01.156.01 Rhynchobatidae Rhynchobatus australiae
Vulnerable 99 JN022595.1
2 IBRC01.157.01 Rhynchobatidae R.australiae Vulnerable 100 JN022595.1 3 IBRC01.179.01 Rhynchobatidae R.australiae Vulnerable 100 JN022595.1
4 IBRC01.158.01 Alopiidae Alopias
superciliosus
Vulnerable 100 DQ108329.1
5 IBRC01.162.01 Alopiidae A.superciliosus Vulnerable 100 DQ108329.1
6 IBRC01.164.01 Alopiidae A.superciliosus Vulnerable 100 DQ108329.1
7 IBRC01.166.01 Alopiidae A.superciliosus Vulnerable 100 DQ108329.1
8 IBRC01.167.01 Alopiidae A.superciliosus Vulnerable 100 DQ108329.1
9 IBRC01.180.01 Alopiidae A.superciliosus Vulnerable 100 DQ108329.1
10 IBRC01.182.01 Alopiidae A.superciliosus Vulnerable 100 DQ108329.1
11 IBRC01.159.01 Carcharhinidae Carcharhinus falciformis
Near Threatened
99 EU398611.1
12 IBRC01.160.01 Carcharhinidae C.falciformis Near Threatened
100 EU398611.1
13 IBRC01.161.01 Carcharhinidae C.falciformis Near Threatened
99 EU398611.1
14 IBRC01.168.01 Carcharhinidae C.falciformis Near Threatened
100 EU398611.1
15 IBRC01.169.01 Carcharhinidae C. falciformis Near Threatened
99 EU398611.1
16 IBRC01.170.01 Carcharhinidae C.falciformis Near Threatened
100 EU398611.1
17 IBRC01.171.01 Carcharhinidae C. falciformis Near Threatened
99 EU398611.1
18 IBRC01.172.01 Carcharhinidae C.falciformis Near Threatened
100 EU398611.1
19 IBRC01.176.01 Carcharhinidae C. falciformis Near Threatened
99 EU398611.1
20 IBRC01.177.01 Carcharhinidae C.falciformis Near Threatened
100 EU398611.1
21 IBRC01.181.01 Carcharhinidae C. falciformis Near Threatened
100 EU398611.1
22 IBRC01.191.01 Carcharhinidae C. falciformis Near Threatened
99 EU398611.1
23 IBRC01.184.01 Carcharhinidae C.limbatus Near Threatened
100 DQ885079.1
24 IBRC01.193.01 Carcharhinidae C.limbatus Near Threatened
100 DQ885079.1
25 IBRC01.185.01 Carcharhinidae C.longimanus Vulnerable 99 JQ654709.1 26 IBRC01.190.01 Carcharhinidae C.longimanus Vulnerable 99 JQ654709.1
27 IBRC01.174.01 Lamnidae Isurus paucus Vulnerable 99 FJ519011.1
23
Lampiran 4. Ikan-ikan hiu yang teridentifikasi di Pelabuhan Perikanan Palabuhanratu, Sukabumi, Jawa Barat.
Gambar 1. Isurus paucus Gambar 2. Carcharhinus falciformis
Gambar 3. Carcharhinus limbatus Gambar 4. Carcharhinus longimanus
Gambar 5. Rhyncobatus australiae Gambar 6. Alopias superciliosus
Sumber : White WT. et al 1997 (Economically important sharks and rays of