DNA
BARCODING
DAN ANALISIS FILOGENETIK IKAN
HIU YANG DIDARATKAN DI PELABUHAN
PERIKANAN SAMUDERA CILACAP
EKA MAYA KURNIASIH
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi yang berjudul DNA Barcoding dan Analisis Filogenetik Ikan Hiu yang Didaratkan di Pelabuhan Perikanan Samudera Cilacap adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, September 2013
ABSTRAK
EKA MAYA KURNIASIH. DNA Barcoding dan Analisis Filogenetik Ikan Hiu yang Didaratkan di Pelabuhan Perikanan Samudera Cilacap. Dibimbing oleh HAWIS MADDUPPA dan BEGINER SUBHAN.
Perairan Indonesia diketahui lebih dari 75 spesies hiu yang ada, yang saat ini banyak tertangkap dan diperdagangkan bagian-bagian tubuh tertentu seperti sirip, minyak hati, daging, tulang, kulit dan mata. Status konservasi CITES dan IUCN telah menyatakan bahwa sebagian besar spesies hiu terancam dan menghadapi kepunahan. Mengidentifikasi spesies dari potongan tubuh organisme tidak dapat dilakukan, oleh karena itu DNA Barcoding dan filogenetik dapat digunakan. Tujuan dari penelitian ini adalah untuk mengidentifikasi hiu spesies yang diperdagangkan di pelabuhan perikanan Cilacap dan menentukan hubungan filogenetiknya. Hasil yang telah berhasil diamplifikasi dari lokus COI (Cytochrome Oxidase I) sebagai barcoding dalam pembuatan pohon filogeni yang menunjukkan kekerabatan terdapat 6 kelompok famili hiu yang berbeda yang diperdagangkan di Cilacap. Hasil ini juga menunjukkan tiga status konservasi spesies hiu yang tertangkap, yaitu status rawan (Sphyrna zygaena, Alopias superciliosus, Carcharhinus obscurus, Isurus paucus, dan Isurus oxyrinchus), status hampir terancam (Pseudocarcharias kamoharai, Carcharhinus falciformis, Prionace glauca, dan Squalus hemipinnis) dan dalam status terancam (Sphyrna lewini). Hasil identifikasi ini penting membantu pemerintah untuk membentuk perlindungan hiu dan kebijakan perdagangan.
ABSTRACT
EKA MAYA KURNIASIH. DNA Barcoding and Phylogenetic Analysis of Shark Landed in Fisheries Port Cilacap. Under Supervision of HAWIS MADDUPPA and BEGINER SUBHAN.
Indonesian waters are known to be lived by more than 75 species of shark. Most shark species are now being caught and traded for their body parts, including their fins, liver oil, meat, bones, skin, and eyes. However, trade data on shark in Indonesia is very lack. The identification of a shark fin to species level is very problematic, as morphologically similar species and specimens, which are poorly preserved or have had key diagnostic features removed, can be difficult to identify. Therefore this research was conducted to identify shark species being traded in Cilacap fisheries port, to confirm their conservation status with IUCN redlist and to determine their phylogenetic relationship. The PCR using COI (Cytochrome Oxidase I) was successfully amplified for 32 samples. A total of 10 spesies was identified and 6 clusters of different shark family was revealed. These identified shark were categorized in three IUCN conservation status : vulnerable status (Sphyrna zygaena, Alopias superciliosus, Carcharhinus obscurus, Isurus paucus, and Isurus oxyrinchus), near threatened status (Pseudocarcharias kamoharai, Carcharhinus falciformis, Prionace glauca, and Squalus hemipinnis) and one species in endangered status (Sphyrna lewini). The identification result is important to help government to build shark conservation and trading policy. Keywords: DNA Barcoding, phylogenetic, shark, species identification,
DNA
BARCODING
DAN ANALISIS FILOGENETIK IKAN
HIU YANG DIDARATKAN DI PELABUHAN
PERIKANAN SAMUDERA CILACAP
EKA MAYA KURNIASIH
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Kelautan pada
Departemen Ilmu dan Teknologi Kelautan
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
Judul Skripsi : DNA Barcoding dan Analisis Filogenetik Ikan Hiu yang Didaratkan di Pelabuhan Perikanan Samudera Cilacap Nama : Eka Maya Kurniasih
NIM : C54090040
Disetujui oleh
Diketahui oleh
Dr Ir I Wayan Nurjaya MSc Ketua Departemen
Tanggal Lulus: 26 September 2013 Dr Hawis Madduppa SPi, MSi Pembimbing I
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan September 2012 ini ialah genetika hiu dengan judul DNA Barcoding dan Analisis Filogenetik Ikan Hiu yang Didaratkan di Pelabuhan Perikanan Samudera Cilacap.
Terima kasih penulis ucapkan kepada Bapak Dr Hawis Madduppa SPi, MSi dan Bapak Beginer Subhan SPi, MSi selaku pembimbing, serta Bapak Prof Dr Drh G Ngurah Mahardika yang telah mendukung penelitian ini. Di samping itu, penghargaan penulis sampaikan kepada Laboratorium Indonesian Biodiversity Research Center, Bali beserta seluruh staff dan pegawai yang telah banyak memberi saran dan membantu dalam pelaksanaan penelitian ini. Ungkapan terima kasih juga disampaikan angkatan Ilmu dan Teknologi Kelautan tahun 2009 atas dukungannya dan kepada ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, September 2013
DAFTAR ISI
DAFTAR TABEL………. ix
DAFTAR GAMBAR………. ix
DAFTAR LAMPIRAN……….. ix
PENDAHULUAN………1
Latar Belakang………..1
Tujuan Penelitian………. 2
METODE……… 2
Waktu dan Tempat………... 2
Bahan……… 2
Alat………... 2
Koleksi Sampel……… 2
Ekstraksi dan Amplifikasi DNA ……… 3
Elektroforesis………... 3
Sekuensing DNA……… 3
Analisis Data……… 4
Identifikasi Spesies dan Status Konservasi……….. 4
Analisis Filogenetik……….. 4
HASIL DAN PEMBAHASAN……… 4
Identifikasi Spesies dan Status Konservasi……….. 4
Hubungan Filogenetik……….. 8
SIMPULAN DAN SARAN………... 12
Simpulan……… 11
Saran……….. 12
DAFTAR PUSTAKA……… 12
LAMPIRAN……….. 14
DAFTAR TABEL
1. Hasil identifikasi spesies hiu dan status konservasinya…………. 5
2. Hasil tangkapan ikan hiu di PPS Cilacap…...……… 8
3. Matriks jarak genetik spesies ikan hiu di PPS Cilacap... 11
DAFTAR GAMBAR
1. Konstruksi pohon filogeni berdasarkan DNA Barcoding hiu yang didaratkan di PPS Cilacap……….…… 11DAFTAR LAMPIRAN
1. Prosedur kerja PCR………. 152. Komposisi Master Mix pada PCR……… 15
3. Hasil analisis BLAST dalam NCBI……….. 16
4. Hasil pengurutan basa nukleotida………...17
1
PENDAHULUAN
Latar Belakang
Kegiatan penangkapan sumber daya perikanan yang cenderung bersifat eksploitatif dan pengelolaan dengan menggunakan pendekatan produksi saat ini sudah pesat yang telah berdampak pada penurunan populasi beberapa jenis komoditas perikanan yang tergolong andalan komoditas ekspor seperti ikan hiu. Ikan hiu merupakan ikan bertulang rawan yang termasuk ke dalam kelas Chondrichthyes yang dapat dijumpai hampir diseluruh wilayah perairan Indonesia, baik di perairan territorial, perairan samudera maupun Zona Ekonomi Eksklusif Indonesia (ZEE). Spesies hiu yang ditemukan lebih dari 75 jenis hiu di perairan Indonesia dan sebagian besar dari jenis tersebut sudah banyak dimanfaatkan dan diperdagangkan (Wibowo dan Susanto 1995). Hampir seluruh bagian tubuh ikan hiu dapat dimanfaatkan, mulai dari sirip, minyak hati sampai daging, tulang, kulit dan mata. Indonesia memasok sekitar 15% dari total kebutuhan sirip hiu dunia, sedangkan negara-negara lainnya hanya sekitar 1% (Stevens et al. 2000); (Traffic 2002). Badan Pusat Statistik (BPS) mencatat sekitar 434 ton sirip ikan hiu diekspor sepanjang tahun 2012. Nilai perdagangan tersebut mencapai US$ 6 juta atau mencapai Rp 57 miliar.
Perkembangan perdagangan ikan hiu yang telah meningkat serta semakin intensifnya penangkapan ikan hiu menyebabkan ikan hiu rentan terhadap jumlah populasinya di perairan Indonesia terutama di perairan selatan Indonesia. Hampir sebagian besar spesies hiu yang ada termasuk ke dalam daftar merah (IUCN) sebagai spesies yang terancam punah seperti Sphyrna lewini, Alopias superciliosus, Carcharhinus obscurus, dan Isurus oxyrinchus.
Perdagangan ikan hiu umumnya hanya dalam bentuk sirip sehingga sulit untuk diidentifikasi secara konvensional Hebert et al. (2003) memperkenalkan DNA barcode sebagai sarana untuk mengidentifikasi semua spesies hewan. Kemampuan barcode DNA untuk mengidentifikasi spesies bergantung pada degenerasi kode genetik. Taksonomi molekuler DNA barcoding dapat membantu proses identifikasi ini karena hanya membutuhkan sedikit jaringan tubuh dari ikan hiu tersebut. Pengkajian keragaman genetik melalui penandaan molekuler menggunakan DNA (Deoxyribo Nucleid Acid) baik pada DNA inti dan DNA mitokondria (mtDNA) akan didapatkan hasil yang dapat mengungkapkan perbedaan dengan lebih teliti dalam membedakan intra dan interspesies yang menyangkut tentang struktur, komposisi dan organisasi genom pada tingkat DNA.
2
Pengetahuan struktur genetik ini penting untuk konservasi dan proteksi ikan hiu sebagai spesies langka dan terancam serta menduga pengaruh perubahan terhadap populasi alami. Kondisi yang seperti telah dijelaskan sebelumnya yang membuat analisis keanekaragaman genetik ikan hiu dengan penentuan marka mitokondria dalam mengukur kekerabatan spesies ikan hiu dalam populasi dengan hasil yang ingin dicapai dapat mengidentifikasi spesies ikan hiu yang banyak ditangkap atau diperdagangkan serta keanekaragaman genetik ikan hiu di perairan selatan Indonesia, khususnya yang didaratkan di Pelabuhan Perikanan Samudera Cilacap, Jawa Tengah.
Tujuan Penelitian
1. Mengidentifikasi spesies ikan hiu secara molekuler dan status konservasinya dari ikan hiu yang didaratkan di Pelabuhan Perikanan Samudera Cilacap, Jawa Tengah.
2. Menentukan hubungan filogenetik antar spesies ikan hiu yang didaratkan di Pelabuhan Perikanan Samudera Cilacap, Jawa Tengah.
METODE
Waktu dan Tempat
Pengambilan sampel hiu dilakukan pada Bulan November 2012 bertempat di Pelabuhan Perikanan Samudera Cilacap, Jawa Tengah. Pengolahan data sampel hiu dilakukan di Laboratorium Biologi Molekuler, Indonesian Biodiversity Research Center, Bali dan Laboratorium Biodiversitas dan Biosistematika Kelautan, Departemen Ilmu dan Teknologi Kelautan, Fakultas Perikanan dan Ilmu Kelautan, Institut Pertanian Bogor.
Alat
Alat yang digunakan dalam penelitian ini adalah vortex, mikrosentrifus, heating block, forceps, mikropipet, tip pipet, thermo cyler, kalkulator, alat tulis, timbangan, tabung erlemenyer, gelas ukur, microwave, serta Perangkat Lunak Mega 5.2.
Bahan
Bahan yang digunakan dalam penelitian adalah sampel daging hiu, larutan ekstraksi Chelex 10%, sarung tangan, etanol, ddH2O, buffer PCR, dNTP, enzim taq polymerase, MgCl2, primer, Agarosa, EtBr, loading dye, serta low mass leader.
Koleksi Sampel
3
sampel yang dimasukkan kedalam tabung reaksi yang telah diisi etanol 96% sebanyak 1 ml, dan diberi label menurut masing-masing sampel individu. Tabung yang telah berisi sampel dan ethanol disimpan rapi di Laboratorium.
Ekstraksi dan Amplifikasi DNA
Ekstraksi DNA bertujuan untuk menghancurkan sel dan mengambil jaringan pada sampel. Metode ekstraksi yang digunakan adalah metode Chelex 10% (Walsh et al. 1991). Proses amplifikasi DNA menggunakan metode Polymerase Chain Reaction (PCR) Hotstart merupakan suatu teknik atau metode perbanyakan (replikasi) DNA secara enzimatik. Komponen utama dalam PCR adalah DNA template, dNTPs, buffer PCR, MgCl2, primer, dan enzimpolymerase. Primer yang digunakan untuk hiu adalah primer modifikasi yang dipakai menggu-nakan "Matt Craig, Pers. Comm" untuk lokus COI fish BCH: 5'-TAA ACT TCA GGG TGA CCA AAA AAT CA-3' atau fish BCL: 5'-TCA ACY AAT CAY AAA GAT ATY GGC AC3' (Baldwin et al. 2009)
Proses PCR pada penelitian ini dilakukan sebanyak 38 kali siklus setiap siklus terdiri atas tiga tahap. PCR dilakukan dengan menggunakan mesin PCR (thermo cyler) yang terdiri dari beberapa proses yaitu pemisahan DNA utas ganda (denaturation) pada suhu 94 oC selama 30 detik, penempelan primer (anneling) pada suhu 48 oC selama 30 detik dan pemanjangan segmen DNA (ekstention) pada suhu 72 oC selama 45 detik.
Proses amplifikasi ini dimulai dengan mengisi lembar kerja PCR dengan tanggal, jumlah sampel, tipe ekstraksi dan catatan lainnya (Lampiran 1). Pengisian lembar kerja ini dilakukan untuk menghitung berapa banyak master mix (MM) yang dibutuhkan dan enzim taq Polimerase serta jumlah ekstrak yang digunakan (Lampiran 2).
Elektroforesis
Elektroforesis adalah teknik pemisahan komponen atau molekul bermuatan berdasarkan perbedaan tingkat migrasinya dalam sebuah medan listrik. Elektroforesis bertujuan untuk mengetahui kualitas DNA dalam produk PCR. Senyawa dengan muatan elektronnya akan berpindah tempat dalam satu bidang elektrik sebanding dengan laju kerapatan muatan mereka. Fragmen DNA dengan panjang yang berbeda divisualisasikan menggunakan pewarna fluorescent spesifik untuk DNA, seperti bromida etidium. Jenis gel yang digunakan adalah agarosa yang dapat menunjukkan band atau ukuran fragmen pasangan basa yang dapat dilihat dengan sinar ultraviolet.
Sekuensing DNA
4
Proses sekuen DNA dikirim ke Berkeley Sequencing Facility di Amerika untuk disekuen.
Analisis Data
Identifikasi Spesies dan Status Konservasi
Analisis data dilakukan dengan menggunakan software Mega 5.2 (Molecular Evolutionary Genetic Analysis) untuk pembacaan urutan nukleotida dan penjajaran (alignment) menggunakan ClustalW pada program tersebut untuk melihat adanya keragaman nukleotida (Tamura et al 2007). Data hasil penjajaran nukleotida yang diperoleh kemudian dicocokkan dengan data yang tersedia pada GeneBank di NCBI (National Center for Biotechnology Information) dengan menggunakan BLAST (Basic Local Alignment Search Tool).
Hasil yang diperoleh dari GeneBank dianalisis status konservasinya dengan menggunakan IUCN (International Union for Conservation of Nature and Natural Resources). Tujuan didirikannya IUCN adalah untuk membantu komunitas di seluruh dunia dalam konservasi alam.Kategori status IUCN Red List merupakan kategori yang digunakan oleh IUCN dalam melakukan klasifikasi terhadap spesies-spesies berbagai makhuk hidup yang terancam kepunahan.Menurut Camhi et al tahun 1998 ada beberapa kategori yang termasuk kedalam daftar IUCN yaitu punah (Extinct), punah di alam (Extinct in the wild), sangat terancam (Critically endangered), terancam (Endangered), rawan (Vulnerable), hampir terancam (Near threatened), tidak mengkhawatirkan (Least concern), minim informasi (Data deficient) dan belum di evaluasi (Not evaluated).
Analisis Filogenetik
Filogenetik adalah salah satu sistem klasifikasi yang didasarkan pada hubungan kekerabatan (evolusi) antara takson satu dengan lainnya (Purnomo dan Pudjoarinto 1999). Oleh karena itu sistem klasifikasi ini sangat penting untuk digunakan dalam penelusuran kekerabatan evolusioner diantara berbagai takson yang ada (Mabrouk et al 2006). Pembuatan rekostruksi filogenetik pada penelitian ini menggunakan metode maximum likelihood tree dengan nilaiboostrap 1000, dan Tamura Nei (TN93) model. Outgroup yang digunakan yaitu Rhinobatus penggali atau pari penggali muncar untuk mengetahui sejauh mana keakuratan yang diperoleh dari bentukan cabang pohon filogenetik.
HASIL DAN PEMBAHASAN
DNA Barcoding dan Status Konservasi
5
teramplifikasi sebanyak 32 individu, hal ini dapat disebabkan saat pengolahan sampel pada proses ekstraksi atau PCR tidak berhati-hati sehingga didapatkan data sekuen yang bukan merupakan jenis hiu tetapi spesies lain, yaitu bakteri, sehingga dipastikan bahwa sampel ikan hiu mengalami kotaminasi.
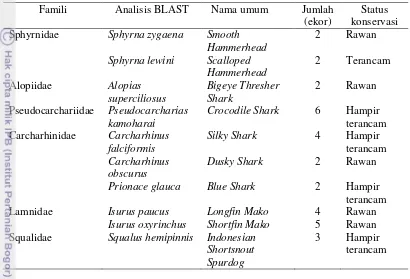
Tabel 1. Hasil identifikasi spesies hiu yang didaratkan di PPS Cilacap menggunakan BLAST dan status konservasi berdasarkan IUCN
Famili Analisis BLAST Nama umum Jumlah
(ekor)
Status konservasi
Sphyrnidae Sphyrna zygaena Smooth
Hammerhead
Crocodile Shark 6 Hampir terancam
Prionace glauca Blue Shark 2 Hampir terancam
Lamnidae Isurus paucus Longfin Mako 4 Rawan
Isurus oxyrinchus Shortfin Mako 5 Rawan
Squalidae Squalus hemipinnis Indonesian
Shortsnout Spurdog
3 Hampir
terancam
Hasil BLAST (Tabel 1) memberikan informasi bahwa individu yang terdapat di Pelabuhan Perikanan Samudera Cilacap teridentifikasi sebagai 6 famili ikan hiu yang terdapat di Pelabuhan Perikanan Samudera Cilacap yaitu Sphyrnidae, Alopiidae, Pseudocarchariidae, Lamnidae, Squalidae dan Carcharhinidae. Famili terbanyak yang ditemukan adalah famili Carcharhinidae dalam status pada IUCN termasuk hampir teracam. Jenis hiu yang didaratkan di Pelabuhan Perikanan Samudera Cilacap ada 10 spesies dari 32 individu.
Ada dua spesies dari Famili Sphyrnidae (hammerhead shark) yaitu Sphyrna zygaena dan Sphyrna lewini (Lampiran 5). Pada penelitian ini hiu spesies Sphyrna zygaena dan Sphyrna lewini memiliki tingkat kemiripan mencapai 100% dengan jenis yang ada pada GeneBank (www.ncbi.nlm.nih.gov/blast/)(Lampiran 2).
6
Spesies lain dari famili Sphyrnidae adalah spesies Sphyrna lewini (Scalloped hammerhead). Hiu Sphyrna lewini atau hiu martil atau caping dijumpai dari lapisan permukaan hingga kedalaman 275 m dan jumlah anak yang dilahirkan 12-41 ekor dengan masa kandungan 9-10 bulan (White et al. 1997). Menurut IUCN (IUCN-SSC 2001) spesies ini termasuk terancam yaitu memiliki resiko kepunahan di alam yang sangat tinggi dan hiu spesies ini sering tertangkap oleh pancing rawai hiu dan jaring insang tuna.
Famili Alopiidae hanya teridentifikasi satu spesies yaitu Alopias superciliosus (bigeye thresher shark ) (Lampiran 5) dengan jumlah 2 individu. Hasil penelitian ini menunjukkan ikan hiu spesies dari Alopias superciliosus dengan tingkat kemiripan mencapai 99-100% dengan spesies yang ada di GeneBank (Lampiran 1). Menurut IUCN tahun 2001 hiu spesies Alopias superciliosus juga termasuk kedalam kategori rawan sehingga pengelolaan penangkapan sangat penting dilakukan untuk menjaga kelestarian hiu spesies ini. Hiu spesies Alopias superciliosus atau dikenal didaerah cilacap sebagai hiu paitan yang dapat tumbuh mencapai 461 cm dan sering dijumpai mulai dekat pantai hingga laut lepas, dari permukaan hingga kedalaman 600 m. Makanannya berupa ikan-ikan dasar dan pelagis serta kelompok cepalopoda. Hiu spesies ini melahirkan 2-4 ekor anak dengan periode waktu memijah tidak diketahui dan sering tertangkap oleh pancing rawai tuna dan rawai hiu (White et al. 1997).
Famili Pseudocarchariidae (Lampiran 5) juga teridentifikasi hanya satu spesies yaitu Pseudocarcharias kamoharai atau hiu tongar dengan jumlah 6 individu. Hiu spesies ini dijumpai mulai dari permukaan hingga pada kedalaman 590 m dan melahirkan 4 ekor anak (White et al. 1997). Status pada IUCN termasuk hampir teracam.
Famili Carcharhinidae (Lampiran 5) merupakan famili hiu yang teridentifikasi memiliki jumlah spesies yang paling banyak yaitu 3 spesies hiu dengan total 8 individu yaitu Carcharhinus falciformis (silky shark) sebanyak 4 individu, Carcharhinus obscurus (Dusky Shark) sebanyak 2 individu dan Prionace glauca (Blue Shark) sebanyak 2 individu.
Famili dari Carcharhinidae yaitu Prionace glauca (Blue Shark). Status pada IUCN termasuk hampir teracam artinya kategori ini diyakini akan terancam keberadaannya di masa mendatang, apabila tidak ada usaha pengelolaan terhadap spesies tersebut. Hiu ini jumlah anak yang dilahirkan 4-135 ekor dalam setahun atau dua tahun dengan lama kandungan 9-12 bulan (White et al. 1997). Hiu ini dapat dijumpai pada lapisan permukaan hingga kedalaman 800 m.
Hiu spesies Carcharhinus falciformis memiliki tingkat kemiripan spesies dengan spesies GeneBank mencapai 99-100% (Lampiran 3). Menurut IUCN tahun 2001 hiu dengan jenis Carcharhinus falciformis termasuk kedalam kategori atau hampir terancam artinya kategori ini diyakini akan terancam keberadaannya di masa mendatang, apabila tidak ada usaha pengelolaan terhadap spesies tersebut. Carcharhinus falciformis atau hiu lanyam biasanya dapat dijumpai dekat dengan permukaan dijumpai hingga kedalaman 500 m (White et al. 1997).
7
anak yang dilahirkan 3-14 ekor dengan lama kandungan 16 bulan (White et al. 1997).
Famili Lamnidae (Lampiran 5) hanya teridentifikasi 2 spesies yaitu Isurus paucus (longfin mako) dan Isurus oxyrinchus (Shortfin Mako) dengan total jumlah individu paling banyak yaitu 9 individu. Hasil penelitian ini menunjukkan bahwa hiu spesies Isurus paucus atau hiu tenggiri memiliki tingkat kemiripan spesies dengan spesies yang ada pada Gene Bank sebesar 100 % (Lampiran 2). Menurut IUCN tahun 2001 hiu dengan spesies Isurus paucus dengan jumlah 4 individu termasuk kedalam kategori rawan. Hiu spesies ini melahirkan anak 2-8 ekor dalam satu kali masa reproduksi dengan periode waktu yang belum diketahui (White et al. 1997).
Hiu spesies Isurus oxyrinchus (Shortfin Mako) terdapat 5 individu dengan bentuk tubuhnya langsing sehingga mampu berenang cepat. Hidup di laut terbuka lepas pantai tetapi sering ditemukan di lapisan permukaan hingga kedalaman 600 m. IUCN tahun 2001 hiu dengan spesies Isurus oxyrinchus dengan jumlah 4 individu termasuk kedalam kategori rawan dapat dilihat dari jumlah melahirkan 4-25 anak dalam satu kali masa memijah pada periode 15-18 bulan atau reproduksi yang terjadi setiap 3 tahun (White et al. 1997).
Famili Squalidae (Lampiran 5) teridentifikasi satu spesies yaitu Squalus hemipinnis (Indonesian Shortsnout Spurdog) dengan jumlah 3 individu. Pada IUCN termasuk hampir teracam artinya kategori ini diyakini akan terancam keberadaannya di masa mendatang, apabila tidak ada usaha pengelolaan terhadap jenis tersebut. Hiu ini kemungkinan terdapat di perairan yang lebih dangkal daripada jenis lain dari genus ini serta dapat melahirkan 3-10 ekor anak dalam satu kali masa memijah dengan periode waktu yang belum diketahui (White et al. 1997).
Ditinjau dari segi ekonomis, ikan hiu memegang peranan penting. Dagingnya dapat dijadikan olahan makanan atau dibuat tepung ikan, kulit dan hatinya dibuat minyak ikan yang merupakan sumber vitamin A, dari kelenjar pankreas dapat dibuat insulin untuk obat kencing manis, siripnya dikeringkan dan dijual untuk dibuat sirip ikan hiu yang istimewa.
Berdasarkan dari informasi dari PPS Cilacap satu sirip hiu dapat mencapai harga Rp 100.000. Dari harga tersebut terlihat bahwa sirip hiu sangat bernilai ekonomis tinggi sehingga nelayan lebih tertarik dengan penangkapan hiu. Investasi secara besar-besaran akan berdampak penangkapan ikan hiu secara besar-besaran sehingga menyebabkan beberapa spesies hiu mengalami penurunan jumlah populasi bahkan kepunahan sehingga dapat berpengaruh pada sistem ekologi terutama rantai makanan.
8
(vulnerable), dan hampir terancam (near threatened). Kondisi ini merupakan kondisi dengan risiko tinggi kepunahan alaminya di habitat alaminya.
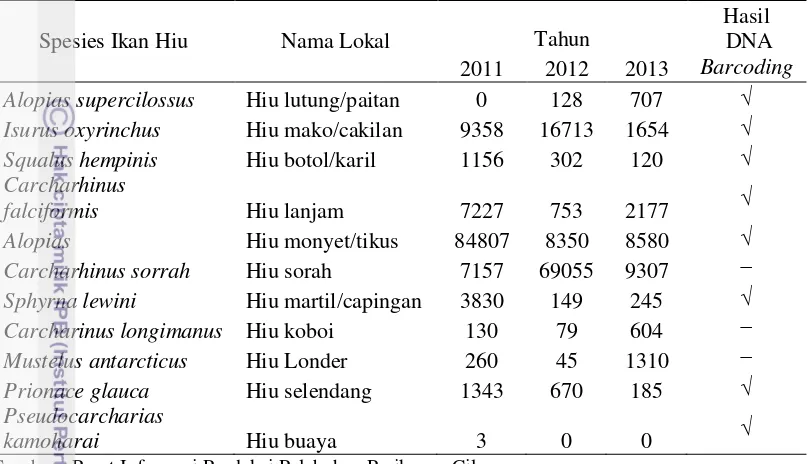
Tabel 2. Perbandingan hasil tangkapan ikan hiu di PPS Cilacap dengan hasil DNA Barcoding
Alopias supercilossus Hiu lutung/paitan 0 128 707 √
Isurus oxyrinchus Hiu mako/cakilan 9358 16713 1654 √
Squalus hempinis Hiu botol/karil 1156 302 120 √
Carcharhinus
falciformis Hiu lanjam 7227 753 2177 √
Alopias Hiu monyet/tikus 84807 8350 8580 √
Carcharhinus sorrah Hiu sorah 7157 69055 9307 −
Sphyrna lewini Hiu martil/capingan 3830 149 245 √
Carcharinus longimanus Hiu koboi 130 79 604 −
Mustelus antarcticus Hiu Londer 260 45 1310 −
Prionace glauca Hiu selendang 1343 670 185 √
Pseudocarcharias
kamoharai Hiu buaya 3 0 0 √
Sumber : Pusat Informasi Produksi Pelabuhan Perikanan Cilacap
Berdasarkan data produksi ikan di PPS Cilacap pada tahun 2011, 2012 dan 2013 (Tabel 2) dari 10 spesies hiu yang teridentifikasi berdasarkan hasil analisis BLAST terdapat kesamaan dari 11 spesies hiu yang tercatat di PPS Cilacap seperti spesies Alopias supercilossus, Carcharhinus falciformis, Isurus oxyrinchus, Squalus hempinis, Sphyrna lewini, Prionace glauca, dan Pseudocarcharias kamoharai. Pada tahun 2011 hasil tangkapan ikan hiu paling banyak pada spesies ikan hiu Alopias spp. (hiu monyet/tikus), pada tahun 2012 hasil tangkapan ikan hiu paling banyak pada spesies ikan hiu Carcharhinus sorrah (hiu sorah) dan pada tahun 2013 hasil tangkapan ikan hiu paling banyak pada spesies ikan hiu
Carcharhinus sorrah (hiu sorah). Hiu-hiu tersebut menjadi target penangkapan karena harga jual (terutama siripnya) yang cukup tinggi dibanding dengan hiu lainnya.
Pada tahun 2012 dan 2013 spesies Pseudocarcharias kamoharai tidak ada yang didaratkan di PPS Cilacap. Hal ini dapat dijadikan indikasi bahwa spesies ini sudah semakin terancam.Tren ukuran hiu hasil tangkapan nelayan juga menunjukan gejala penurunan panjang ukuran ikan berdasarkan dari hasil observasi di pendaratan ikan Cilacap terdapat banyak ditemukan hiu masih berukuran kecil anak (<0.5 kg atau < 60 cm). Selain itu beberapa spesies hiu sudah jarang didaratkan di PPS Cilacap seperti hiu buas (Galeocerdo cuvier) dan hiu martil (Sphyrna lewini) dan bahkan hiu gergaji (Pristis microdon) sudah tidak pernah didaratkan di pelabuhan ini (PPS Cilacap 2009).
9
sehingga kegiatan penangkapan ikan sering tumpang tindih dengan daerah berkembang biaknya hiu. Kegiatan penangkapan ikan dapat menyebabkan mereka hilang dari lautan sebelum mencapai masa reproduksi. Demikian pula degradasi lingkungan laut tersebut dapat mengancam daerah asuhan mereka (Camhi 1998). Hal ini yang membuat ikan hiu terkadang mudah tertangkap yang merupakan hasil sampingan dari jenis ikan lain yang menjadi tujuan utama penangkapan seperti ikan tuna, tongkol dan udang. Walaupun demikian, hasil tangkapan hiu terus meningkat setiap tahunnya (Compagno 1984). Penggunaan alat tangkap didaerah Cilacap yang mengunakan jaring liong bun atau disebut dengan bottom gillnet serta menggunakan rawai cucut yang banyak digunakan masyarakat sekitar Cilacap. Walaupun sebagian besar produksi perikanan hiu di Indonesia dihasilkan dari hasil tangkap sampingan, akan tetapi menurut data FAO sudah sangat cukup menempatkan posisi Indonesia sebagai negara yang mempunyai produksi perikanan hiu terbesar di dunia dengan menguasai 12,1% tangkapan ikan hiu di dunia selama kurun waktu 1990-2004 (Lack dan Sant 2006).
Sebagian besar spesies hiu tumbuh dan berkembang sangat lambat serta memerlukan waktu bertahun-tahun hingga mencapai usia dewasa (Hoeve 1988). Pada hiu berukuran besar, biasanya memerlukan waktu enam hingga delapan belas tahun atau lebih untuk mencapai usia dewasa (Last dan Stevens 1994). Para ilmuwan kesulitan untuk menentukan umur hidup hiu. Spesies hiu tertentu yang berukuran besar dapat hidup hingga umur 40 tahun lebih (White etal. 2002). Hiu mempunyai daur reproduksi yang panjang (satu atau dua tahun untuk beberapa jenis hiu) serta waktu pengeraman yang lama juga (Compagno 1984). Proses pengeraman untuk hiu berukuran kecil mencapai tiga hingga empat bulan sedangkan untuk hiu berukuran besar bisa mencapai dua tahun atau lebih. Hiu mempunyai tingkat fekunditas (fecundity rate) yang rendah. Jumlah embrio yang dilahirkan serta membutuhkan waktu lama mengakibatkan mudah terjadi eksploitasi pada sumberdaya hiu karena kemampuan pulihnya yang rendah. Upaya menjaga kualitas habitat dan ekosistem laut juga menjadi keharusan untuk mempertahankan keseimbangan dan keberlangsungan ikan hiu.
Hubungan Filogenetik
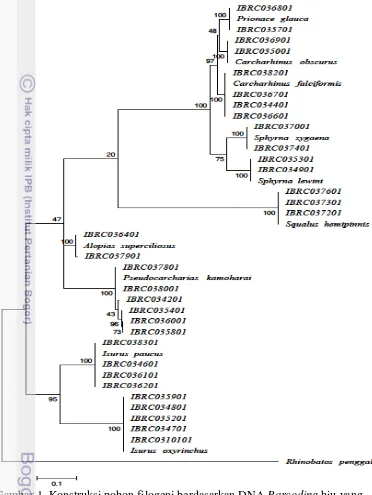
Kekerabatan antar jenis dianalisis dengan topologi pohon filogenetik yang menunjukkan ada 10 spesies yang termasuk dalam 6 famili yang berbeda yang terdapat di Pelabuhan Perikanan Samudera Cilacap. Tingkat kepercayaan dari topologi pohon filogeni dilihat dari pengujian dengan metode bootstrapping yaitu dengan melihat nilai bootstrap support yang muncul dari konstruksi percabangan pohon suatu clade yang diulang sebanyak 1000 kali pengulangan. Hasil pengujian dengan metode bootstraping ditampilkan dalam bentuk diagram kladogram disertai nilai penghitungan boostrap di setiap percabangannya (Gambar 1). Semakin tinggi nilai bootstrap (mencapai nilai 100%), maka akurasi dari percabangan pohon filogeni yang terbentuk semakin tinggi.
10
taksa yang berkerabat jauh dengan taksa ingroup; sementara ingroup merupakan spesies atau organisme yang digunakan dalam penelitian ini.
Gambar 1. Konstruksi pohon filogeni berdasarkan DNA Barcoding hiu yang didaratkan di PPS Cilacap
Pada topologi pohon filogenetik terdapat skala 0.1 yang menunjukkan bahwa dari 10 urutan nukleotida ada 1 basa yang berubah di setiap percabangan. Pada setiap percabangan yang membentuk clade atau kelompok yang didukung oleh nilai boostrap juga didukung oleh nilai jarak genetik. Jarak genetik digunakan dalam melihat kedekatan hubungan antara spesies ikan hiu di Cilacap.
11
Spesies 4 : Squalus hemipinnis Spesies 10: Carcharhinus obscurus Spesies 5 : Isurus oxyrinchus Spesies 11: Rhinobatus penggali Spesies 6 : Isurus paucus
Semakin dekat jarak genetik, maka semakin dekat hubungan kekerabatan antar clade dalam pohon filogeni. Pada matrik jarak genetik dalam Tabel 3, nilai jarak genetik terjauh adalah antara Rhinobatus penggali dan Squalus hemipinnis dengan nilai 0.284 yang dijadikan sebagai pembanding antara jarak genetik outgroup dan ingroup. Sementara jarak genetik antar ingroup dalam masing-masing clade berkisar antara 0.040 – 0.284 %.
SIMPULAN DAN SARAN
Simpulan
12
Saran
Perlu dilakukan penelitian dalam bidang genetika yang lebih lanjut untuk membuat database yang lebih luas mengenai jumlah, tempat, serta spesies hiu yang ditangkap di seluruh wilayah Indonesia. Selain itu, perlu juga dilakukan penelitian mengenai perdagangan hiu dalam segi ekonomi dan sosial untuk mengetahui peranan perdagangan hiu bagi sentra ekonomi dan sosial masyarakat pesisir. Dimana database dan hasil sosial ekonomi ini dapat digunakan sebagai dasar dalam penentuan kebijakan konservasi dan perdagangan hasil laut, khususnya hiu di kawasan Indonesia.
DAFTAR PUSTAKA
Ayotte, L. 2005. Sharks-Educator’s Guide. 3D Entertaintment ltd. And United Nations Environtment Program.
Baldwin CC, Mounts JH, Smith DG, Weigt LA. 2009. Genetic identification and color descriptions of early life-history stages of Belizean Phaeoptyx and Astrapogon (Teleostei: Apogonidae) with comments on identification of adult Phaeoptyx. Zootaxa 2008: 1–22.
Camhi M, Fowler, J. Musick, A. Brautigam and S. Fordham 1998. Sharks and their relatives, ecology and conservation.Occasional Paper of the IUCN Species Survival Commission No.20.IUCN, Gland, Switzerland and Cambridge, UK. 39p.
Compagno.I.J. 1984. “FAO species catalogue.Vol 4.Sharks of the world. An annoted and illustrate catalogue of shark species known to date Part 2. Carcharniformes”.FAO ful, Synop.(125) vol 4. 655p.
Duryadi D. 1994. Peranan DNA Mitokondria (mtDNA) dalam Studi Keragaman Genetik dan Biologi Populasi pada Hean hayati 1(1) : 1-4
Hoeve, U. W. 1988. Ensiklopedi Indonesia Serial Ikan. P.T. Dai Nippon Printing Indonesia. Jakarta. 252.
International Union for Conservation of Nature and Natural Resources [IUCN]. 2001. IUCN Red list categories and criteria IUCN-The World Conservation Union. Gland. Swizerland and Cambridge, UK.34p
Kyle CJ, Wilson CC. 2007. Mitochondrial DNA identification of game and harvested freshwater fish spesies.Forensic Sciece Internasional 166(1): 68-76.
Lack, M. & Sant, G. 2006. Confronting Shark Conservation Head On!. Cambridge: TRAFFIC International. iv+29 hal.
Last, P. R. & J. D. Stevens. 1994. Sharks and Rays of Australia. Fisheries Research and Development Corporation. 513 p.
Mabrouk, M.S., M. Hamdy, M. Mamdouh, M. Aboelfotoh, and Y.M. Kadah. 2006. BIOINFTool: Bioinformatics and sequence data analysis in molecular biology using Matlab. Proc. Cairo International Biomedical Engineering Conference.
13
Stevens J.D., Bonfil, R, Dulvy, and Walker, P.A. 2000. The effects of fishing on sharks, rays and chiemaeras (chondrinhtyans), and the implications for marine ecosistem. ICES Journal of Marine Science, 57:476-494.
Tamura K, Dudley J, Nei M, Kumar S. 2007. Mega 4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Mol Biol Evol .24:1596-1599.
Traffic. (2002). A CITES priorities: Sharks and the twelfth meeting of the conference of the parties to CITES. Retrieved 6 February, 2004, from (http://www.traffic.org/news/Sharks_CoP12.pdf)
Walsh PS, Mezger DA, Higuchi R. 1991. Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material. Biotecniques. 1991 Apr 10(4):506.
Wibowo S. dan H. Susanto. 1995. Sumberdaya dan Pemanfaatan Hiu. Penebar Swadaya. Jakarta. 156 hal.
White, W. T., N. G. Hall and I. C. Potter. 2002. Reproductive biology and growth
during pre and postanal life of T. personata and T. mucosa. Marine Biology
140: 699-712.
14
15
Lampiran 1. Prosedur Kerja PCR
1. Gunakan Gloves(sarung tangan) kemudian keluarkan reagen: Air, dNTP, buffer PCR, MgCl2, Primer 1 dan Primer 2 dari freezer untuk mencairkan. 2. Isi lembar kerja PCR dengan tanggal, jumlah sampel, tipe ekstraksi dan
catatan lainnya.
3. Tandai dan nomori tabung PCR dalam rack
4. Setelah bahan cair, jentikkan setiap tabung dengan jari untuk mencampur, kocok isi hingga dasar tabung.
5. Buat campuran MM 1 : gunakan pipet NO DNA, tambahkan bahan sesuai dengan volume yang telah dihitung dalam daftar di lembar PCR di tabung 1,5mL. Gunakan tip berbeda untuk setiap penambahan reagen. Pipet naik turun untuk mencampur reagen sepenuhnya.
6. Gunakan pipet NO DNA, buat MM 2 dalam tabung 1,5mL terpisah, tetapi jangan menambahkan taq.Gunakan tip berbeda untuk setiap penambahan reagen. Pipet naik turun untuk mencampur reagen sepenuhnya.
7. Gunakan pipet NO DNA, bagi 14 μL MM1 ke dalam setiap tabung PCR. 8. Pindahkan DNA ekstra Chelex dari ruang pendingin dan jika perlu,
sentrifugasi singkat untuk menghilangkan kondensasi. Gunakan pipet DNA rendah, tambahkan 1 μL DNA ekstrak untuk setiap tabung.
9. Ambil taq sesuai yang di inginkan dari freezer kemudian tambahkan kedalam MM2 dan pipet naik turun untuk mencampur.
10.Pilih dan mulai program hot-star PCR. Biarkan penutup panas dan tahan sebentar sampai suhu mencapai 800 C. Kemudian tempatkan strip tabung ke dalam mesin PCR.
11.Atur hingga 10μL pipetman, pipet naik turun MM2 untuk mencampur, kemudian tambahkan MM2 ke dalam masing-masing tabung dan ganti tip untuk setiap sampel.
12.Unpause program dan lihat layar mesin PCR untuk menjamin bahwa mesin PCR sedang bekerja.
13.Bersihkan tempat kerja, letakkan reagen ke dalam freezer dan ekstrak DNA ke dalam lemari pendingin.
Lampiran 2. Komposisi Master Mix pada PCR Master mix ... tabung
STANDAR PROTOCOL ( 1μL DNA template)
16
Lampiran 3.Tabel hasil identifikasi ikan hiu yang didaratkan di Pelabuhan Perikanan Cilacap dengan menggunakan BLAST.
No Sample Code
BLAST analysis Geberal Name IUCN red list Simililarity (%) GeneBank Accession
Carcharhinus falciformis Silky Shark Near Threatened
100 EU398611.1
3 IBRC.03.46. 01
Isurus paucus Longfin Mako Vulnerable 100 FJ519011.1
4 IBRC.03.47. 01
Isurus oxyrinchus Shortfin Mako Vulnerable 100 FJ518947.1
5 IBRC.03.48. 01
Isurus oxyrinchus Shortfin Mako Vulnerable 99 JQ654705.1
6 IBRC.03.49. 01
Sphyrna lewini Scalloped Hammerhead
Endangered 100 FJ519428.1
7 IBRC.03.50. 01
Carcharhinus obscurus Dusky Shark Vulnerable 100 KC470543.1
8 IBRC.03.52. 01
Isurus oxyrinchus Shortfin Mako Vulnerable 100 JQ654705.1
9 IBRC.03.53.
Prionace glauca Blue Shark Near
Threatened
Isurus oxyrinchus Shortfin Mako Vulnerable 99 JQ654713.1
14 IBRC.03.60. 01
Alopias superciliosus Crocodile Shark Near Threatened
99 FJ519579.1
15 IBRC.03.61. 01
Isurus paucus Longfin Mako Vulnerable 100 FJ519011.1
16 IBRC.03.62. 01
Isurus paucus Longfin Mako Vulnerable 100 FJ519011.1
17 IBRC.03.64. 01
Alopias superciliosus Bigeye Thresher Shark
Vulnerable 99 DQ108329.1
18 IBRC.03.66. 01
Carcharhinus falciformis Silky Shark Near Threatened
100 EU398611.1
19 IBRC.03.67. 01
Carcharhinus falciformis Silky Shark Near Threatened
100 EU398611.1
20 IBRC.03.68. 01
Prionace glauca Blue Shark Near
Threatened
100 JQ654713.1
21 IBRC.03.69. 01
Carcharhinus obscurus Dusky Shark Vulnerable 100 KC470543.1
22 IBRC.03.70. 01
Sphyrna zygaena Smooth Hammerhead
Vulnerable 100 EU399018.1
23 IBRC.03.72.
Vulnerable 100 EF539308.1
25 IBRC.03.74. 01
Sphyrna zygaena Smooth Hammerhead
Vulnerable 100 EU399018.1
26 IBRC.03.76.
Alopias superciliosus Bigeye Thresher Shark
Vulnerable 100 DQ108329.1
29 IBRC.03.80. 01
Isurus oxyrinchus Shortfin Mako Vulnerable 100 FJ518947.1
30 IBRC.03.82.
Isurus paucus Longfin Mako Vulnerable 100 FJ519011.1
32 IBRC.03.10 1.01
17
Lampiran 4.Hasil pengurutan basa nukleotida (sequencing) pada sampel ikan hiu di Pelabuhan Perikanan Samudera Cilacap, Jawa Tengah.
18
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT AATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAAT
GTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTA……….
#IBRC035901_Isurus oxyrinchus 664
CCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCTA ATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAATG
TAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGG……….
#IBRC034801_Isurus oxyrinchus 660
CCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCTA ATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAATG
TAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTTATGGT………
#IBRC035201_Isurus oxyrinchus 665
CACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTT CTAATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATA
ATGTAATCGTAACCGCCCACGCTTTTGTAATAATCTTT…..………
#IBRC034701_Isurus oxyrinchus 667
ACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCT
#IBRC036201_Isurus paucus 585
TGGGTCAGCCAGGTTCTCTTCTAGGGGACGATCAGATTTATAATGTTATTGTAACCGC CCATGCATTTGTAATGATTTTCTTCATGGTAATGCCCGTGATAATTGGGGGCTTTGGGA
ACTGACTGGTGCCTTTAATGATCGGTGCACCCGATATGGCC……….
#IBRC036101_Isurus paucus 558
TTCGCGCCGAACTGGGTCAGCCAGGTTCTCTTCTAGGGGACGATCAGATTTATAATGT TATTGTAACCGCCCATGCATTTGTAATGATTTTCTTCATGGTAATGCCCGTGATAATTG
GGGGCTTTGGGAACTGACTGGTGCCTTTAATGATCGGTGCA……….
#IBRC034601_Isurus paucus 665
ACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCTTAAGCCTTT TAATTCGAGCTGAACTGGGACAACCTGGATCACTTTTAGGAGATGACCAAATCTATAA
TGTAATTGTAACCGCCCATGCATTTGTAATAATCTTCTTCATGGT………
#IBRC038301_Isurus paucus 663
TGTATTTAATCTTTGGTGCATGAGCAGGAATGGTAGGGACAGCCCTAAGCCTTTTAAT TCGTGCCGAACTGGGTCAGCCTGGTTCCCTCCTAGGGGATGATCAGATTTATAATGTT
ATTGTAACCGCCCATGCATTTGTAATAATTTTCTTTATG……….
#IBRC037901_Alopias superciliosus 667
ACCCTTTACCTGATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTCC TAATTCGAGCTGAACTTGGGCAACCTGGATCACTTTTAGGAGATGATCAGATTTATAA
TGTAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTCATG………..
#IBRC036401_Alopias superciliosus 668
CACCCTGTATTTAATCTTTGGTGCATGAGCAGGGATAGTGGGAACAGCCCTAAGCCTT CTAATTCGCGCCGAACTGGGTCAGCCAGGTTCTCTTCTAGGGGACGATCAGATTTATA
19
#IBRC036001_Pseudocarcharias kamoharai 666
CACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCCTAAGCCTT TTAATTCGAGCTGAACTGGGTCAACCTGGATCACTTTTAGGAGATGACCAAATCTATA
ATGTGATTGTAACCGCCCATGCATTCGTAATAATCTTCTTCATGGT……….
#IBRC035801_Pseudocarcharias kamoharai 661
CACCCTTTATTTAATCTTTGGTGCATGAGCAGGAATAGTGGGAACAGCCCTAAGCCTT TTAATTCGAGCTGAACTGGGTCAACCTGGATCACTTTTAGGAGATGACCAAATCTATA
ATGTAATTGTAACCGCCCATGCATTCGTAATAATC………..
#IBRC035401_Pseudocarcharias kamoharai 663
CCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTCCTA ATTCGAGCTGAACTTGGGCAACCTGGATCACTTTTAGGAGATGATCAGATTTATAATG
TAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTTCA……….
#IBRC034201_Pseudocarcharias kamoharai 574
TAGGAGATGACCAGATTTATAATGTAATTGTAACCGCCCACGCTTTCGTAATAATCTT TTTTATAGTTATGCCAATCATAATTGGTGGCTTCGGGAATTGATTAGTTCCTTTAATAA
TTGGTGCACCAGACATGGCCTTCCCACGAATA………..
#IBRC038001_Isurus oxyrinchus 673
GGCACCCTTTACCTGATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTC TCCTAATTCGAGCTGAACTTGGGCAACCTGGATCACTTTTAGGAGATGATCAGATTTA
TAATGTAATCGTAACCGCCCACGCTTTTGTAATAATCT………
#IBRC037801_Pseudocarcharias kamoharai 664
CCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGCCCTAAGTCTTCTA ATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGATCAGATTTATAATG
TAATCGTAACCGCCCACGCTTTTGTAATAATCTTTTT………
#IBRC035701_Prionace glauca 662
GCCCTAAGCCTTCTAATTCGCGCCGAACTGGGTCAGCCAGGTTCTCTTCTAGGGGACG
ATCAGATTTATAATGTTATTGTAACCGCCCATGCATTTGTAATGATTT……….
#IBRC036801_Prionace glauca 406
CCACCATCATTTCTTCTTCTCCTCGCCTCTGCTGGAGTAGAAGCTGGAGCAGGTACTG GTTGAACAGTTTATCCTCCATTAGCTAGTAACCTAGCACATGCTGGACCATCTGTTGA
TTTAGCTATTTTCTCTCTTCACTTAGCCGGTGTGTC………..
#IBRC036901_Carcharhinus obscurus 689
AAGATATTGGCACCCTTTACCTAATTTTTGGTGCATGAGCAGGTATAGTTGGAACAGC CCTAAGTCTTCTAATTCGAGCTGAGCTTGGACAACCTGGATCACTTTTAGGGGATGAT
CAGATTTATAATGTAATCGTAACCGCCCACGCTTTTGT………
#IBRC035001_Carcharhinus obscurus 671
20
Lampiran 5. Spesies hiu yang teridentifikasi di Pelabuhan Perikanan Samudera Cilacap,JawaTengah.
Sumber : White et al 1997(Economically important sharks and rays of Indonesia
Alopias superciliosus (Hiu Paitan/Lutung)
Sphyrna lewini ( Hiu Caping/ Martil)
Sphyrna zygaena ( Hiu Caping/Martil)
Squalus hempinis (HiuBotol/Karil)
Pseudocarcharias kamoharai (Hiu Buaya/Tongar)
Isurus paucus (Hiu Tenggiri)
Isurus oxyrinchus
(Hiu Mako/Cakilan Air) Carcharhinus falciformis (Hiu Lanjam)
Carcharhinus obscurus (Hiu Lanyam)
21
RIWAYAT HIDUP
Penulis dilahirkan di Jambi pada tanggal 23 Mei 1991 dari ayah yang bernama Desuryawarman dan ibu Yeni Hartinah. Penulis merupakan anak pertama dari dua bersaudara. Lulus dari Sekolah Menengah Atas (SMA) Negeri 6 Jambipada tahun 2009, penulis diterima sebagai mahasiswa Institut Pertanian Bogor, Fakultas Perikanan dan Ilmu Kelautan, Departemen Ilmu dan Teknologi Kelautan melalui jalur Undangan Seleksi Masuk IPB (USMI).
Selama kuliah di Institut Pertanian Bogor penulis pernah menjadi asisten mata kuliah Biologi Laut periode 2011-2012 dan periode 2012-2013 dan asisten mata kuliah Keanekaragaman Hayati Laut periode 2012-2013. Penulis juga pernah mengikuti Program Kreatifitas Mahasiswa yang didanai oleh DIKTI tahun 2012. Penulis aktif dalam organisasi Badan Eksekutif Mahasiswa FPIK sebagai anggota Biro Corporation periode 2011-2012 dan sebagai sekretaris Advokasi dan Kesejahteraan Mahasiswa periode 2012-2013. Selain itu, penulis juga aktif sebagai sekretaris dalam organisasi Marine Biologi Club periode 2011-2012 dan periode 2012-2013 dan Organisasi Mahasiswa Daerah Himpunan Mahasiswa Jambi (HIMAJA) pada periode 2011-2012. Sejak awal tahun 2013 penulis juga bergabung di Indonesian Biodiversity Research Center (IBRC) Bali sebagai Junior Research Fellow.