DESAIN PRIMER
LAPORAN PRAKTIKUM
disusun untuk memenuhi salah satu tugas mata kuliah Biologi Molekuler
oleh : Dhaifan Diza A 1303790 Anisa Suci S 1300904 Novia Rahayu A 1302152 Riani Ulfah 1300952 Shabrina Ulfa 1302152
DEPARTEMEN PENDIDIKAN BIOLOGI
FAKULTAS PENDIDIKAN MATEMATIKA DAN ILMU PENGETAHUAN ALAM UNIVERSITAS PENDIDIKAN INDONESIA
BAB I PENDAHULUAN
1.1 Latar Belakang
Penemuan yang berpengaruh terhadap perkembangan ilmu biologi molekuler salah satunya adalah PCR (Polymerase Chain Reaction). Teknik ini mempunyai fungsi utama untuk memperbanyak DNA target. Proses yang terjadi melibatkan suatu primer yang berfungsi sebagai cetakan untuk membuat DNA baru.
Desain primer dilakukan untuk memperoleh primer yang dapat digunakan dalam amplifikasi DNA dengan metode Polymerase Chain Reaction (PCR). Keberhasilan amplifikasi DNA tergantung dari ketepatan primer yang digunakan. Primer yang digunakan dalam proses PCR harus dapat membatasi daerah yang akan diamplifikasi. Primer berfungsi sebagai pembatas fragmen DNA target yang akan diamplifikasi.
Amplifikasi DNA dengan metode PCR terdiri dari tiga tahap. Tahap awal dari proses amplifikasi yaitu denaturasi untai DNA, selanjutnya dilakukan penempelan primer pada fragmen DNA target (annealing) dan tahap akhir merupakan proses extension yaitu proses pemanjangan sekuen DNA. Perancangan untuk memperoleh suatu primer yang memenuhi kriteria primer yang baik untuk amplifikasi dilakukan secara in silico, yaitu merancang/mendesain primer dengan bantuan suatu program dalam komputer.
1.2 Tujuan
Praktikum ini bertujuan untuk mengetahui pelaksanaan teknik PCR dan cara mendesain primer menggunakan aplikasi Clustalx 1.83
BAB II
2.1. Desain Primer
Tahapan PCR yang paling menentukan adalah penempelan primer. Sepasang primer oligonukleotida (primer maju dan primer mundur) yang akan dipolimerisasi masing-masing harus menempel pada sekuens target, tepatnya pada kedua ujung fragmen yang akan diamplifikasi. Untuk itu urutan basanya harus komplementer atau setidak-tidaknya memiliki homologi cukup tinggi dengan urutan basa kedua daerah ujung fragmen yang akan diamplifikasi itu. Padahal, kita belum mengetahui dengan pasti urutan basa sekuens target. Oleh karena itu, diperlukan cara tertentu untuk merancang urutan basa kedua primer yang akan digunakan.
Dasar yang digunakan adalah urutan basa yang diduga mempunyai kemiripan dengan urutan basa sekuens target. Urutan ini adalah urutan serupa dari sejumlah spesies/strain organisme lainnya yang telah diketahui/dipublikasikan. Sebagai contoh, untuk merancang sepasang primer yang diharapkan dapat mengamplifikasi sebagian gen lipase pada isolat Bacillus termofilik tertentu dapat digunakan informasi urutan basa gen lipase dari strain-strain Pseudomonas fluorescens, P. mendocina , dan sebagainya, yang sebelumnya telah diketahui.
Urutan-urutan basa fragmen tertentu dari berbagai strain tersebut kemudian dijajarkan dan dicari satu daerah atau lebih yang memperlihatkan homologi tinggi antara satu strain dan lainnya. Daerah ini dinamakan daerah lestari (conserved area). Sebagian/seluruh urutan basa pada daerah lestari inilah yang akan menjadi urutan basa primer.Sebenarnya, daerah lestari juga dapat ditentukan melalui penjajaran urutan asam amino pada tingkat protein. Urutan asam amino ini kemudian diturunkan ke urutan basa DNA. Dari satu urutan asam amino sangat mungkin akan diperoleh lebih dari satu urutan basa DNA karena setiap asam amino dapat disandi oleh lebih dari satu triplet kodon. Dengan demikian, urutan basa primer yang disusun dapat merupakan kombinasi beberapa kemungkinan. Primer dengan urutan basa semacam ini dinamakan primer degenerate. Selain itu, primer yang disusun melalui penjajaran urutan basa DNA pun dapat merupakan primer degenerate karena urutan basa pada daerah lestari di tingkat DNA pun tidak selamanya memperlihatkan homologi sempurna (100%).
Urutan basa pasangan primer yang telah disusun kemudian dianalisis menggunakan program komputer untuk mengetahui kemungkinan terjadinya
primer-dimer akibat homologi sendiri(self-homology) atau homologi. silang (cross-homology). Selain itu, juga perlu dilihat kemungkinan terjadinya salah tempel (mispriming), yaitu penempelan primer di luar sekuens target. Analisis juga dilakukan untuk mengetahui titik leleh (Tm) masing-masing primer dan kandungan GC-nya. Sepasang primer yang baik harus mempunyai Tm yang relatif sama dengan kandungan GC yang cukup tinggi.
Desain primer yang baik sangat penting untuk keberhasilan reaksi PCR. Pertimbangan desain yang penting yang diuraikan di bawah ini sebagai kunci untuk amplifikasi spesifik dengan hasil tinggi.
1. Panjang Primer.
Hal ini secara umum diterima bahwa panjang optimal primer PCR adalah 18-22 mer (basa).
2. Primer Melting Temperature.
Primer Melting Temperature (Tm) merupakan temperatur yang diperlukan oleh separoh primer dupleks mengalami disosiasi/lepas ikatan. Primer dengan Tm berkisar antara 52-58oC sangat ideal, sedangkan Tm diatas 65oC akan mengurangi efektifitas anealing sehingga proses amplifikasi DNA kurang berjalan baik. Tm ini sangat ditentukan oleh jumlah basa GC (GC contains). Tm primer dapat dihitung dengan formula: Tm (oC) = ((G+C) x4) + ((A+T) x2))……….secara kasar (kurang akurat) atau Tm(oC) = {ÄH/ ÄS + R ln(C)} - 273.15……..secara akurat.
3. Primer annealing temperatur.
Temperatur annealing (Ta) merupakan suhu yang diperkirakan primer dapat berikatan dengan template (DNA) dengan stabil (DNA-DNA hybrid stability). Jika suhu annealing tinggi akan menyulitkan terjadinya ikatan primer dengan DNA template sehingga akan menghasilkan produk PCR yang rendah (kurang efisien). Namun jika Ta terlalu rendah akan menyebabkan terjadinya penempelan primer pada DNA tempalt yang tidak spesifik. Ta dapat dihitung dengan menggunakan formula: Ta = 0.3 x Tm(primer) + 0.7
Tm (product) – 14.9, dengan Tm(primer) = Tm primer dan Tm(product) = Tm produk PCR.
4. GC Content
Jumlah Basa G dan C (GC content) di dalam primer yang ideal sekitar 40-60%.
5. GC Clamp
Jumlah basa G dan C yang terdapat pada 5 basa terakhir (3 ) disebut‟ dengan GC clamp. GC clamp yang baik sekitar 3 basa G/C dan tidak melebihi 5 basa G/C. keberadaan G/C di ujung 3 primer sangat membantu‟ terjadinya stabilitas iktan antara primer dengan DNA templat yang diperlukan untuk inisiasi polymerase DNA (proses PCR).
6. Primer Secondary Structures
6.1. Hairpins : terbentuknya struktur loop/hairpin pada primer sebaiknya dihindari, namun sangat sulit untuk memperoleh primer tanpa memiliki struktur hairpin. Hairpin pada ujung 3' dengan ÄG(energy yang diperlukan untuk memecah struktur hairpin) = -2 kcal/mol dan hairpin internal dengan ÄG = -3 kcal/mol masih dapat ditoleransi.
6.2. Self Dimer : primer dapat beriktan dengan primer lainnya yang sejenis disebut dengan self-dimer. Self-dimer pada ujung 3' dengan ÄG = -5 kcal/mol dan self-dimer pada bagian internal dengan ÄG= -6 kcal/mol masih dapat ditoleransi.
6.3. Cross Dimer: Primer dapat berikatan dengan primer pasangannya (reverse dan forward) sehingga disebut cross dimmers. Secara optimal, cross dimerinternal pada ujung 3' dengan ÄG = -5 kcal/mol dan self- dimer pada bagian internal dengan ÄG= -6 kcal/mol masih dapat ditoleransi.
7. Repeats
Primer sebaiknya tidak memiliki urutan pengulangan dari 2 basa dan maksimum pengulangan 2 basa sebanyak 4 kali masih dapat di toleransi. Misalnya ATATATAT.
Primer sebaiknya tidak memiliki urutan basa yang di ulang terus menerus. Pengulangan basa berurutan sampai 4 kali masih dapat di toleransi. Misalnya AGCGGGGGATGGGG memiliki urutan basa G diulang 5 kali berturut-turut.
9. Avoid Cross homology
Untuk menghindari cross homologi dapat dilakukan dengan cara menganalisis homologi primer dengan DNA genome melalui BLAST-NCBI. (10) Amplicon Length. Panjang PCR produk yang ideal berkisar antara 100-500 pasang basa.
10. Optimum Annealing temperature (Ta Opt)
Suhu annealing optimum sangat mempengaruhi hasil pcr. TaOpt ini dapat dihitung dengan cara Ta Opt = 0.3 x(Tm of primer) + 0.7 x(Tm of product) – 25. (12) Primer Pair Tm Mismatch. Perbedaan Tm sepasang primer sebaiknya tidak lebih dari 5oC.
2.2 Penelusuran Data Gen
Sekuen gen diperoleh dari database NCBI (National Center for Biotechnology Information). Langkah pertama yang dilakukan adalah mengakses laman NCBI melalui http://www.ncbi.nlm.nih.gov/,sebagaimana tampilan yang disajikan pada gambar berikut ini.
Gambar 2.2.1 Laman NCBI
NCBI merupakan sumber informasi bagi perkembangan biologi molekuler, yang membuat database bagi data genomik, proteomik maupun metabolomik dari berbagai organisme. Data sekuen nukleotida yang disajikan dalam NCBI dapat berupa bentuk fasta maupun bentuk sekuen seperti lazimnya.
BAB III
METODE PENELITIAN 3.1 Jenis Praktikum
Praktikum kali ini merupakan praktikum desain primer dengan cara manual dan online. Digunakan beberapa aplikasi pendukung seperti Clustax, dan beberapa situs web guna mendukung praktikum kali ini, seperti NCBI gene bank.
3.2 Waktu dan Tempat
Hari, tanggal : Senin, 11 April 2016 Waktu : 13.00 – 15.00 WIB
Tempat: Laboratorium Mikrobiologi FPMIPA UPI 3.3 Alat dan Bahan
Tabel 1.1 Alat
No Nama Alat Jumlah
1. Laptop 1 buah
2. Aplikasi Cluxtas
-3.4 Langkah Kerja
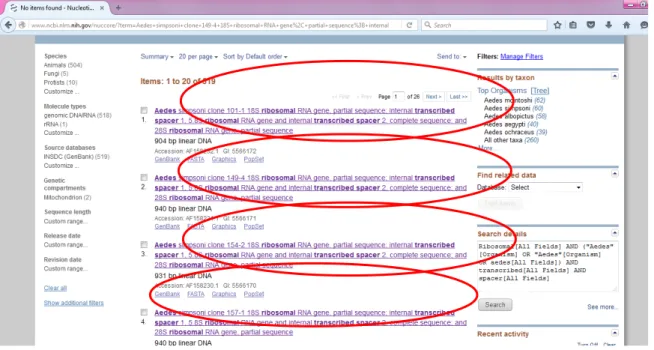
Dalam melakukan desain primer dapat dilakukan secara online dan manual kedua cara ini memiliki tahapan yang berbeda untuk mendesain primer secara online perlu digunakan beberapa aplikasi pendukung, dan beberapa situs web guna mendapatkan primer yang diinginkan. Tahapan pertama dalam mendesain primer secara online yaitu dengan mencari sekuens primer yang diinginkan dengan membuka situs gene bank http: // www.ncbi.nlm.nih.gov dan memasukan key word gen yang akan dicari, sebagai contoh pada gambar gen Aedes sp.
Gambar 3.4.1 Pemasukan Key Word Sekuens Gen yang Diinginkan (Dokumentasi Kelompok, 2016)
Setelah gen yang dianginkan didapatkan maka dipilih beberapa gen minimal tiga macam sekuens berbeda yang akan digunakan sebagai kandidat desain primer, dengan syarat memiliki karakteristik yang tidak jauh berbeda, seperti spesies atau genus yang sama, panjang sekuens yang sama, dan penggunaan primer yang sama sebagai contoh pada praktikum kali ini adalah gen 16s RNA dan penggunaan sekuens dari spesies Aedes simpsoni.
Gambar 3.4.2 Pemilihan Kandidat Sekuens Primer (Dokumentasi Kelompok, 2016)
Setelah beberapa kandidat sekuens primer dipilih berdasarkan kemiripan karakteristik, maka masing – masing urutan sekuens dipindahkan pada aplikasi notepad yang ada pada komputer, dalam contoh keempat kandidat yang terpilih di copy urutan sekuen kemudian keempat sekuen di pasta pada aplikasi notepad. Untuk mendapatkan urutan sekuens maka, klik terlebih dahulu sekuen yang diinginkan, kemudian klik fasta, maka urutan sekuens muncul, setelah muncul maka sekuen dapat dicopy kemudian di pasta dalam notepad, lakukan hal yang sama pada keempat kandidat yang terpilih.
Gambar 3.4.3 Klik Fasta untuk Mendapatkan Urutan Sekuen (Dokumentasi Kelompok, 2016)
Gambar 3.4.4 Sekuen yang akan di Copy pada Notepad (Dokumentasi Kelompok, 2016)
Gambar 3.4.5 Sekuen yang di Copy pada Notepad (Dokumentasi Kelompok, 2016)
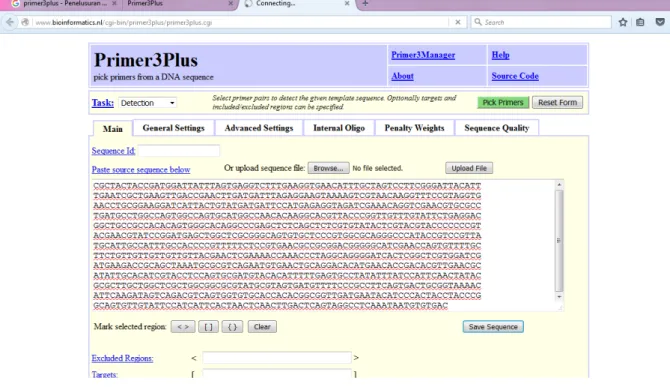
Setelah keempat urutan sekuen dipindahkan pada notepad maka pengujian secara online dapat dilakukan dengan memasuki alamat website
www.bioinformatics.nl/ primer3plus. Pada halaman website ini maka akan diketahui bagaimana kualitas primer yang kita design dengan memasukan urutan sekuen gen yang sudah dikandidatkan sebagai primer kemudian klik pick primers.
Gambar 3.4.6 Pengujian kriteria Primer menggunakan Aplikasi Clustax (Dokumentasi Kelompok, 2016)
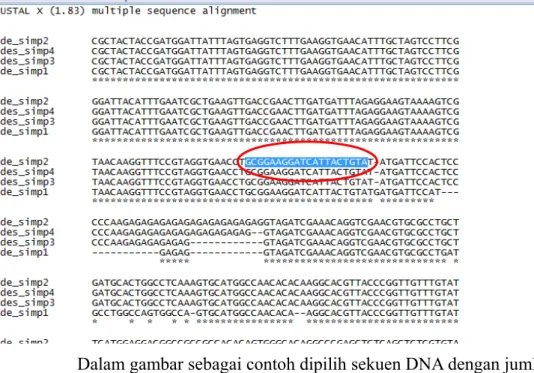
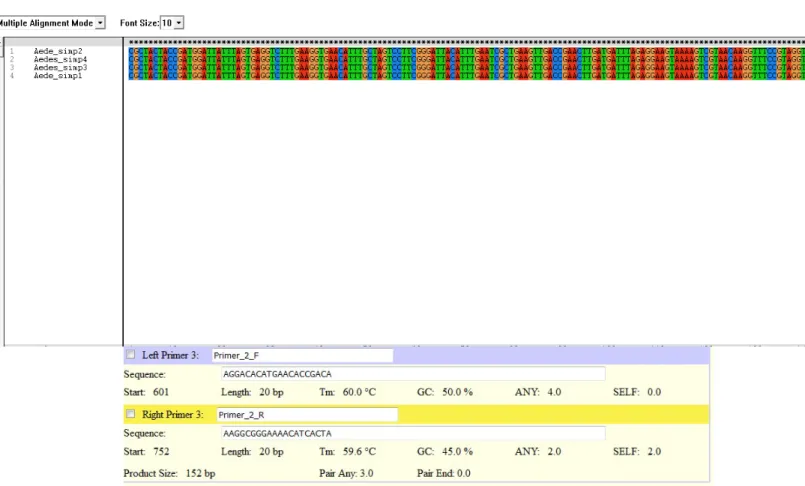
Sekuen DNA yang terpilih kemudian diuji, untuk menentukan apakah keempat kandidat primer yang terpilh memilki kriteria yang sama dan baik atau tidak. Pengujian ini menggunakan aplikasi Clustax. Seluruh sekuen yang terpilih dimasukan kedalam aplikasi tersebut kemudian akan muncul bintang pada bar bagian atas. Jika bintang yang muncul banyak maka hal tersebut menunjukan keempat kandidat memilki kriteria yang sama untuk dijadikan primer.
Gambar 3.4.7 Sekuen yang akan di uji Kualitas secara Online (Dokumentasi Kelompok, 2016)
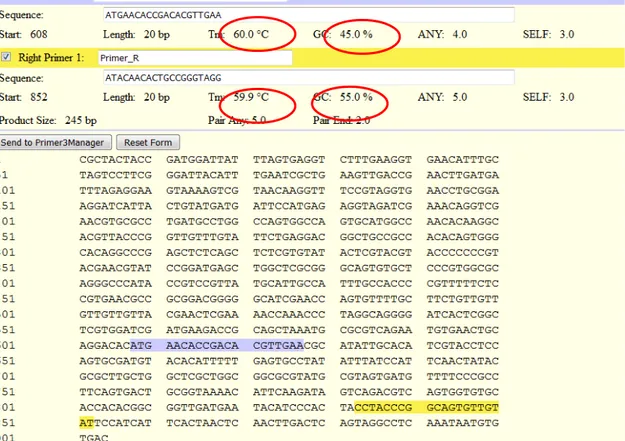
Setelah pick primers diklik maka akan didapatkan hasil identifikasi primer yang digunakan, hasil akan memperlihatkan kandungan GC, suhu melting (Tm), suhu annealing (Ta). Dari hasil tersebut maka akan menggambarkan bagaimana kualitas primer yang akan digunaan, jika primer memiliki karakter yang sesuai dengan syarat, mak primer dapat digunakan. Namun apabila tidak sesuai dengan syarat yang ada maka dapat digunakan sekuen DNA yang lain. Sebagai contoh dari keempat kandidat primer yang terpilih maka dapat dipilih kandidat lainnya yang sesuai dengan syarat primer.
Gambar 3.4.7 Hasil Uji Kualitas Primer secara Online (Dokumentasi Kelompok, 2016)
Tahap yang berbeda dilakukan apabila desain primer dengan cara manual hal yang membedakan adalah perhitungan kualitas primer secara manual, seperti perhitungan kandungan GC, suhu melting (Tm) dan suhu annealing (Ta). Hal pertama yang perlu dilakukan adalah dengan memilih sekuen dengan panjang base pairs yang ditentukan dari salah satu kandidat primer yang terpilih.
Dalam gambar sebagai contoh dipilih sekuen DNA dengan jumlah 20 bp, dari sekuen yang terpilih maka kualitas primer diuji secara manual dengan rumus :
1. Rumus Jumlah GC :
Jumlah Panjang Sekuens
¿
Jumlah GC
¿
x 100 % 2. Rumus Suhu Melting (TM) : {( G + C ) x 4 } + { ( A + T ) x 2 ) 3. Rumus Suhu Annealing (TA) : Tm - 5◦C
Dari hasil perhitungan maka akan diketahui kualitas primer yang akan digunakan.
BAB IV
HASIL PENGAMATAN DAN PEMBAHASAN 4.1 Hasil Pengamatan
Gambar 4.1.2 Hasil data sekuens primer secara online
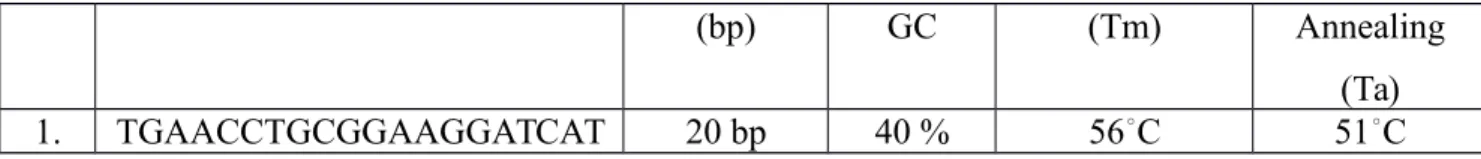
Gambar 4.1.3 Sekuens yang Dipilih sebagai Kandidat Primer Tabel 4.1.1 Hasil Pengamatan Perhitungan Manual
No. Sekuens yang Terpilih Panjang Jumlah Suhu Melting Suhu
Sekuens kandidat primer terpilih
(bp) GC (Tm) Annealing (Ta)
1. TGAACCTGCGGAAGGATCAT 20 bp 40 % 56◦C 51◦C
Keterangan :
4. Rumus Jumlah GC :
Jumlah Panjang Sekuens
¿
Jumlah GC
¿
x 100 % 5. Rumus Suhu Melting (TM) : {( G + C ) x 4 } + { ( A + T ) x 2 ) 6. Rumus Suhu Annealing (TA) : Tm - 5◦C
4.2 Pembahasan
Praktikum ini menggunakan software Clustalx1.83 yang bertujuan untuk membuat alignment sekuens. Suatu primer harus dialignment itu karena untuk memilih kandidat primer yang akan digunakan. Dapat dilihat dari hasil data sekuens yang telah dibuat dari software Clustalx1.83 bahwa terdapat bintang yang penuh dibagian atas itu menunjukkan bahwa sekuens yang didapatkan itu baik.
Gambar 4.2.1 Hasil data sekuens menggunakan aplikasi Clustalx yang baik Setelah didapatkan hasil sekuens maka dilakukan pengujian secara online pada web www.bioinformatics.nl/primer3plus yang dilakukan untuk mengetahui titik leleh (Tm) masing-masing primernya dan kandungan GC-nya. Sepasang primer yang baik harus mempunyai Tm yang relatif sama dengan kandungan GC yang cukup tinggi. Desain primer yang baik juga sangat penting untuk keberhasilan reaksi PCR. Sesuai dengan hasil sekuens Bintang yang penuh
secara online tersebut dari primer 1 kanan dan primer 2 kanan Tm nya terlihat lebih baik karena primer 1 kanan Tm yaitu 59,9 oC dan primer 2 kanan Tm nya yaitu 59,6 oC sedangkan primer 1 kiri dan primer 2 kiri Tm yang didapatkannya yaitu primer 1 kiri 60,2 oC dan primer 2 kiri 60,0 oC hal tersebut memperlihatkan terlihat jauh dari skala optimal suhu meltingnya (Tm) karena optimal suhu melting tersebut yaitu sekitar 52-59 oC.
Hasil dari kandungan GC yang optimal itu sekitar 40-60%. Sesuai hasil sekuens primer 1 kanan dan kiri telah baik karena primer 1 kanan terdapat kandungan GCnya 55,0% dan primer 1 kirinya 50,0%. Dapat terlihat pula untuk primer 2 baik kanan maupun kiri terlihat bahwa kandungan GCnya optimal untuk primer 2 kanan terdapat kandungan GCnya 45,0% dan primer 2 kiri 50,0%. Jadi dapat terlihat bahwa hasil kandungan GC dari kedua primer tersebut baik.
Sedangkan hasil yang dihitung menggunakan manual itu dengan sekuens yang terpilihnya TGAACCTGCGGAAGGATCAT yang memiliki panjang 20bp dapat dilihat pada hasil pengamatan bahwa kandungan GCnya 40% hal tersebut sudah dapat terlihat baik karena sudah dijelaskan tadi diatas optimal kandungan GC itu sekitar 40-60%. Sedangkan suhu meltingnya (Tm) sebesar 56 oC suhu tersebut baik sesuai dengan syarat primer. Jika dilihat dari primer 1 dan 2 yang dihhitung menggunakan aplikasi online tersebut dapat dilihat bahwa suhu meltingnya lebih baik primer yang dihitung menggunakan manual karena suhunya itu disekitar 52-58 oC.
BAB V PENUTUP 5.1 Kesimpulan
Desain primer dapat dilakukan dengan menggunakan Clustalx 1.83. Program tersebut memiliki kelebihan dan kekurangan tersendiri dalam mendesain primer. Primer yang didapatkan dari software tersebut telah sesuai dengan persyaratan primer yang baik.
5.2 Saran
Mahasiswa dapat membandingkannya dengan aplikasi pembuat design primer lainnya seperti GENETYCX ataupun Express 3.01 sehingga lebih paham dengan kelebihan dan kekurangan sesuai dengan kebutuhan peneliti.
DAFTAR PUSTAKA
Widowati, 2013. Desain Primer Sitokrom B Sebagai salah satu Komponen PCR untuk deteksi DNA. Yogyakarta: Lembaga Penelitian Universitas Islam Negeri Sunan Kalijaga. Tersedia [Online]: http://digilib.uin-suka.ac.id/11908/1/BOPTN-7%20Esti%20W%20Widowati.pdf. 17 April 2016