Pusat Studi Lingkungan dan Kependudukan (PSLK)
Universitas Muhammadiyah Malang, tanggal 29 April 2017
Lestari et al., Konfirmasi Penurunan Alel
1
KONFIRMASI PENURUNAN ALEL TETUA PERSILANGAN KEDELAI
PADA GENERASI F1 BERDASARKAN MARKA SSR
Confirmation of Alleles from Soybean Parental Lines Inherited to F1 Generation Based on SSR Markers
Puji Lestari1, Sustiprijatno2, Asadi3
1,2,3Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian-
Badan Litbang Pertanian,
Jl. Tentara Pelajar No. 3A Bogor 16111, Telp. 0251-8337975 e-mail korespondensi: plestari129@yahoo.com
ABSTRAK
Pemuliaan konvensional melalui persilangan merupakan upaya yang sering dilakukan di Indonesia untuk merakit varietas unggul baru (VUB) kedelai. Keberhasilan persilangan sangat ditentukan oleh kebenaran penurunan alel secara genetik pada generasi F1dari tetua persilangannya. Marka molekuler diketahui banyak dimanfaatkan untuk membantu program pemuliaan kedelai. Karena itu tujuan penelitian ini adalah melakukan konfirmasi penurunan alel pada generasi F1 dari tetua persilangan kedelai menggunakan marka SSR (simple sequence repeat). Sebanyak 10 marka SSR digunakan untuk analisis molekuler melalui amplifikasi DNA beberapa progeni generasi F1 dari 7 populasi persilangan kedelai melalui PCR. Total 9 SSR menunjukkan polimorfisme pada paling sedikit satu pasang tetua, kecuali Satt002. Marka yang menunjukkan polimorfisme paling banyak pada kombinasi tetua (7 kombinasi) adalah Satt294 dan Satt191. Jumlah individu F1 yang benar-benar merupakan hasil persilangan berdasarkan penurunan alel dari 7 populasi adalah antara 40-100%, dan sebaliknya individu dengan alel hanya berasal dari salah satu tetua menunjukkan kegagalan persilangan karena selfing. Populasi yang menunjukkan keberhasilan persilangan 100% dengan segregasi alel dari tetuanya adalah B4641 X B57, B4621 X B57, dan B4545 X B62.Indvidu-individu F1 berhasil diseleksi dari tiap populasi berdasarkan pola penurunan alel kedua tetuanya. Karena itu, marka 9 SSR tersebut membuktikan dapat menelusuri penurunan alel dari kedua tetua pada progeni F1 yang berimplikasi pada percepatan seleksi progeni hasil persilangan kedelai.
Kata kunci: alel, SSR, kedelai, generasi F1, persilangan
ABSTRACT
Conventional breeding through crossing is considerable effort frequently undertaken to improve soybean variety in Indonesia. Successful crossing could be achieved when true alleles from both parents inherited to F1 generation. On other side, molecular markers are well known to be applied to assist breeding program of soybean. Therefore, this study aimed to confirm alleles of both parents truly inherited to the F1 generation revealed by SSR (simple sequence repeat) markers. A total of 10 SSR markers were used for molecular analysis by DNA amplification of several F1 progenies of seven populations derived from crossing based on PCR approach. Nine SSRs showed polymorphism in at least one pair of parent, in exception of Satt002. Markers which showed polymorphism in all populations (7 combinations) were Satt294 and Satt191. Number of true individuals F1 from crossing based on allele inheritance ranged 40-100%, in contrast, individual with allele from one of parent indicated crossing failure as a result of self-pollination. The population demonstrating 100% success of crossing with segregated alleles from both parents were B4641 X B57, B57 X B4621, and B4545 X B62. FI Individuals were successfully selected from each population based on allele inheritance pattern from both parents. Therefore, 9 SSR markers proved their ability to trace the alleles from both parents to F1 progenies, implicating for the acceleration of progeny selection in soybean breeding. Keywords: allele, SSR, soybean, F1 generation, crossing
Kedelai (Glycine max [L]. Merrill) merupakan salah satu komoditas pangan dari Legumioceae dengan kandungan protein nabati yang tinggi (Erliana, 2010) dan banyak dikonsumsi masyarakat. Permintaan kedelai Indonesia sebesar 2,3-2,5 juta ton (Adie & Krisnawati, 2013), sedangkan produktivitas kedelai Indonesia sebesar 14,16 kw/ha (BPS, 2014). Produksi kedelai tahun 2015 sebanyak 998,87 ribu ton biji kering atau meningkat sebanyak 43,87 ribu ton (4,59%) (BPS, 2015). Meskipun produksi kedelai meningkat, namun belum mampu memenuhi kebutuhan nasional dikarenakan pertambahan jumlah penduduk. Cekaman abiotik dan biotik akibat iklim tropis Indonesia semakin memicu permasalahan dalam upaya meningkatkan produktivitas kedelai.
Pengembangan varietas unggul baru (VUB) kedelai dengan dengan karakter penting lainnya harus diupayakan. Peningkatan produktivitas kedelai dan kualitas sesuai permintaan di pasaran dapat dilakukan melalui perbaikan genetik tanaman khususnya pemuliaan tanaman. Pemuliaan konvensional di Indonesia telah cukup lama dilakukan dan menghasilkan varietas unggul yang dilepas. Beberapa VUB hasil persilangan dan pendekatan lain seperti mutasi dan pemutihan varietas lokal telah banyak dilepas, dan merupakan varietas populer diantaranya Grobogan, Wilis, Dega-1 dan lainnya (Balitbangtan, 2016). Potensi hasil VUB terbaru Dega-1 yang merupakan hasil persilangan antara Grobogan dan Malabar, mencapai 3 ton/ha pada kondisi optimal, berbiji
besar, umur genjah dan beradaptasi luas. Meskipun telah diperoleh sejumlah VUB, program pemuliaan kedelai di Indonesia masih harus terus dilakukan guna menunjang usaha swasembada kedelai.
Keberhasilan pemuliaan tanaman ditentukan oleh keberadaan keragaman genetik yang luas pada populasi plasma nutfah kedelai. Khususnya varietas sangat berperan penting dalam produksi kedelai, karena untuk mencapai hasil yang tinggi sangat ditentukan oleh potensi genetiknya (Marliah et al., 2012). Untuk memperoleh varietas unggul, evaluasi dan karakterisasi morfologi selama ini telah sering dilakukan oleh para pemulia dalam melakukan persilangan. Namun karakterisasi morfologi sangat dipengaruhi lingkungan dan memerlukan waktu di lapang sehingga pendekatan marka molekuler perlu dipertimbangkan.
Penggunaan marka molekuler dapat memberikan hasil yang lebih cepat, efektif dan akurat dibandingkan dengan karakterisasi secara morfologi. Karakterisasi molekuler tidak hanya ditujukan untuk pemetaan, analisis keragaman genetik, studi evolusi, identifikasi varietas dan studi genetik lainnya, namun juga bermanfaat untuk menguji kebenaran progeni hasil persilangan. Setiap generasi persilangan dapat dievaluasi penurunan alel dari kedua tetua persilangannya berdasar marka molekuler kodominan seperti single sequence repeat (SSR) (Bredemeijer et al., 2002; Jun et al., 2011; Kuroda et al., 2013).
Marka SSR telah banyak digunakan pada spesies kedelai. Lokus SSR diapit oleh urutan nukleotida yang terkonservasi. Sekuen pengapit tersebut dapat dijadikan primer spesifik yang dapat diamplifikasi menggunakan
polymerase chain reaction (PCR). Rata-rata kecepatan
mutasi SSR berkisar dari 10-6 sampai 10-2 kejadian per lokus per generasi, lebih tinggi dibandingkan dengan rata-rata mutasi pada gen yang mengkodekan lokus (Li et al., 2002). Marka SSR banyak digunakan untuk berbagai analisis genetik, analisis sidik jari DNA dan sebagai marka seleksi dalam pemuliaan. Selain itu, marka ini sangat berguna karena pewarisan mengikuti hukum Mendel, mudah diaplikasikan dengan teknik PCR (Handoyo & Rudiretna, 2001) dan mempunyai
polymorphic information content (PIC) atau tingkat
heterosigositas tinggi (Santosa et al., 2006; Chaerani et
al., 2011). SSR telah banyak diaplikasikan di kedelai dan
menunjukkan kelebihannya (Kuroda et al., 2013; Santana
et al., 2014) dalam membantu program pemuliaan.
Karena itu studi ini bertujuan untuk melakukan konfirmasi F1 hasil persilangan masing-masing tetua varietas kedelai menggunakan marka SSR.
METODE Materi tanaman
Materi genetik yang digunakan dalam penelitian ini terdiri atas tujuh populasi F1 hasil persilangan dari
masing-masing dua tetua persilangan. Tujuh tetua meliputi Grobogan, B4545, Wilis, B62, B4641, B57, dan B4621 disilangkan yang menghasilkan kombinasi populasi sebagai berikut Grobogan X B57, Grobogan X B62, B4641 X B57, B4621 X B62, B4621 X B57, Wilis X B57 dan B4545 X B62.
Isolasi DNA
Daun kedelai sekitar 100 mg digerus menjadi bubuk halus dalam eppendorf dengan nitrogen cair. Ekstraksi DNA dilakukan dengan menggunakan GeneExTM Plant Kit (GeneAll, Korea). Larutan DNA stok yang diperoleh kemudian diuji secara kuantitatif dan kualitatif untuk mengetahui tingkat kemurnian dan konsentrasinya. Uji kuantitatif dilakukan dengan menggunakan nanodrop spektrofotometer (Thermo Scientific, USA) sedangkan uji kualitatif dilakukan dengan teknik elektroforesis pada gel agarosa 1%. Produk PCR divisualisasikan di bawah sinar UV (GelDoc). Larutan DNA stok kemudian diencerkan menjadi konsentrasi 10 ng/ul untuk digunakan dalam kegiatan PCR.
Amplifikasi PCR
Total sekitar 10 pasang primer simple sequence
repeat (SSR) digunakan untuk analisis PCR dalam
penelitian ini (Tabel 1). Sekuen primer SSR ini adalah adopsi dari penelitian Cregan et al. (1999) dan dapat diakses di http://soybean.org/resources/ssr.php.
Untuk analisis PCR, setiap sampel diamplifikasi dalam total reaksi 10 µl yang mengandung 10 ng DNA
template sebanyak 1 µl, Kappa 2G fast ready mix (Kappa
Biosystems, USA) sebanyak 5 µl, primer forward dan
reverse 10 µM masing-masing sebanyak 0,5 µl dan
ddH2O steril sebanyak 2 µl. Reaksi PCR dilakukan dengan mesin PCR T1 thermocycler (Biometra, Germany) dengan kondisi PCR sebagai berikut : denaturasi awal dilalukan pada suhu 95oC selama 5 menit, diikuti oleh sebanyak 35 siklus proses denaturasi pada suhu 94oC selama 30 detik, tahap penempelan primer pada suhu 55oC selama 1 menit, tahap perpanjangan basa pasa suhu 72 oC selama 1 menit dan perpanjangan akhir pada suhu 60oC selama 15 menit serta pendinginan dengan dengan suhu 10 oC . Hasil PCR dielektroforesis pada gel poliakrilamid 8% dan distaining dalam larutan etidium bromide. Visualisasi pita DNA menggunakan DelDoc.
Analisis Data
Analisis data dilakukan berdasarkan skoring pita DNA yang muncul polimorfik pada tetua persilangan. Selanjutnya skoring dilakukan dengan membandingkan hasil persilangan F1 dengan tetuanya. Hasil skoring dibuat dalam persentase untuk mengetahui validasi dari hasil persilangan F1.
Pusat Studi Lingkungan dan Kependudukan (PSLK)
Universitas Muhammadiyah Malang, tanggal 29 April 2017
Lestari et al., Konfirmasi Penurunan Alel
3
Tabel 1. Daftar primer yang digunakan dalam penelitian ini
Nama Primer Forward Reverse Motif
SATT009 CCAACTTGAAATTACTAGAGAAA CTTACTAGCGTATTAACCCTT (AAAT)3 (AAT)13 SATT144 CGTCGCCATCACTATGAGAA CCATCTTGAGCAGAGTTTGAAGTT (TAA)18 SATT147 CCATCCCTTCCTCCAAATAGAT CTTCCACACCCTAGTTTAGTGACAA (ATA)15
SATT191 CGCGATCATGTCTCTG GGGAGTTGGTGTTTTCTTGTG (TAT)19
SATT294 GCGGGTCAAATGCAAATTATTTTT GCGCTCAGTGTGAAAGTTGTTTCTAT (TAT)23 SATT308 GCGTTAAGGTTGGCAGGGTGGAAGTG GCGCAGCTTTATACAAAAATCAACAA (TTA)22 SATT431 GCGTGGCACCCTTGATAAATAA GCGCACGAAAGTTTTTCTGTAACA (AAT)21 SATT002 TGTGGGTAAAATAGATAAAAAT TCATTTTGAATCGTTGAA (TA)5(AT)5
SATT030 AAAAAGTGAACCAAGCC TCTTAAATCTTATGTTGATGC (ATA)21
SATT045 TGGTTTCTACTTTCTATAATTATTT ATGCCTCTCCCTCCT (AAT)18
*
Cregan et al. (1999)HASIL DAN PEMBAHASAN
Berdasarkan polimorfisme antara dua tetua persilangan dari tujuh kombinasi (Grobogan X B57, Grobogan X B62, B4641 X B57, B4621 X B62, B4621 X B57, Wilis X B57, dan B4545 X B62), terseleksi 9 primer SSR yang menunjukkan polimorfisme dari total 10 primer yang disurvei. Sembilan primer tersebut meliputi Satt009, Satt144, Satt147, Satt191, Satt294, Satt308, Satt431, Satt030 dan Satt045, dan selanjutnya dilanjutkan analisisnya ke progeni F1. Ukuran amplikon target dari sembilan primer berkisar antara 200-300 pb (pasang basa) dan dapat diskoring. SSR dimer sampai tetra-mer dengan jumlah motif dari tiga sampai 23 pada primer SSR tersebut menunjukkan polimorfisme yang tinggi pada tetua-tetua persilangan sesuai harapan. Polimorfisme ini
dapat direpresentasikan dengan nilai polymorphic
information content (PIC), yang merupakan refleksi dari
nilai frekuensi dan keragaman alel (Mandal et al., 2016). Nilai PIC dari 9 primer berkisar dari 0,39 (Satt144) sampai 0,91 (Satt294) dengan rata-rata 0,75. Sebanyak 7 marka menunjukkan nilai PIC > 0,7 dan sebanyak 2 marka menunjukkan nilai PIC < 0,5. Hal ini menunjukkan bahwa marka SSR tersebut merupakan marka yang informatif dan bermanfaat untuk membedakan aksesi kedelai F1 hasil persilangan.
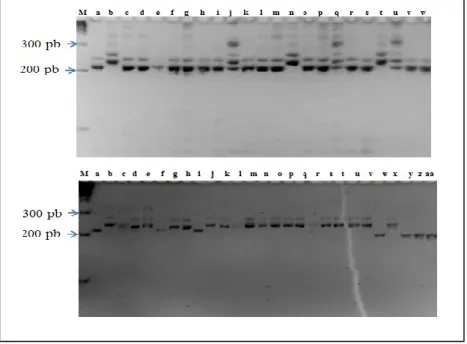
Pola polimorfisme tiap primer menunjukkan validitas yang dapat digunakan sebagai dasar analisis konfirmasi penurunan alel progeni F1 dari tetua persilangannya. Contoh pola pita DNA polimorfik yang dihasikan oleh primer SSR Satt191 pada individu F1 dari tujuh kombinasi persilangan ditampilkan pada Gambar 1.
Gambar 1. Pola pita DNA yang dihasilkan primer Satt191 pada generasi F1 dari tujuh kombinasi persilangan kedelai. (Keterangan Gambar: M= DNA Ladder, a= Grobogan, b= 57, c= 1, d= 2, e= 3, f= 4, g= 5, h= 6, i= 7, j= 8, k= 9, l= 10, m= Grobogan, n= 62, o= 11, p=12, q= 13, r= 14, s= 4641, t= 57, u= 15, v= 16 dan w= 17. Gambar bawah: M= DNA Ladder, a= B4621, b= B62, c= 1, d= 2, e= 3, f= 4, g= 5, h= 6, i= B4621, j= B57, k= 7, l= 8, m= Wilis, n= B57, o= 9, p=10, q= 11, r= 12, s=
Sebagian besar primer SSR yang menunjukkan polimorfisme dalam studi ini tidak lepas dari sifat SSR yang variabilitasnya tinggi pada tiap lokus di genom yang tinggi, multialel dan kodominan sehingga sesuai sebagai
marker-assisted selection (MAS) di kedelai (Santana et al., 2014). Peluang mendapatkan alel unik dan spesifik
dalam suatu koleksi juga besar (Chaerani et al., 2011; Lestari et al., 2016) sehingga pemilihan marka SSR ini sudah sesuai untuk studi ini.
Diantara 9 primer SSR yang polimorfik, sebanyak 2 primer yaitu Satt191 dan Satt291 menunjukkan polimorfisme pada semua populasi, 3 SSR polimorfik
(Satt045, Satt147 dan Satt431) pada enam populasi, 3 SSR (Satt009, Satt308 dan Satt030) polimorfik pada lima populasi, dan satu SSR hanya polimorfik pada 2 populasi (Satt144). Sedangkan berdasarkan latar belakang genetik tetua persilangan, hanya dua populasi (Grobogan XB57 dan Grobogan X B62) yang menunjukkan polimorfisme pada semua 9 primer SSR. Dua kombinasi tetua persilangan yaitu B4641 X B57 dan B4621 XB 57 hanya monomorfik pada Satt144, sementara B4621 X B62 menunjukkan polimorfisme pada 6 SSR kecuali Satt1441, Satt308 dan Satt431 (Tabel 2).
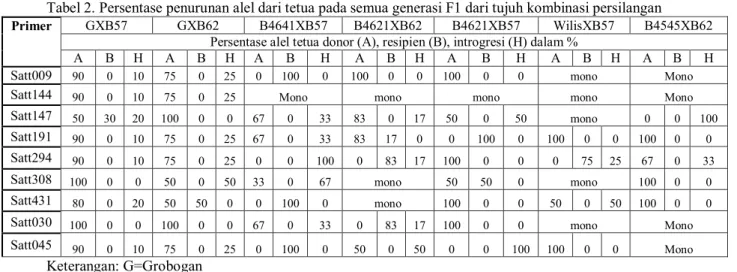
Tabel 2. Persentase penurunan alel dari tetua pada semua generasi F1 dari tujuh kombinasi persilangan
Primer GXB57 GXB62 B4641XB57 B4621XB62 B4621XB57 WilisXB57 B4545XB62
A B H A B Persentase alel tetua donor (A), resipien (B), introgresi (H) dalam % H A B H A B H A B H A B H A B H
Satt009 90 0 10 75 0 25 0 100 0 100 0 0 100 0 0 mono Mono
Satt144 90 0 10 75 0 25 Mono mono mono mono Mono
Satt147 50 30 20 100 0 0 67 0 33 83 0 17 50 0 50 mono 0 0 100
Satt191 90 0 10 75 0 25 67 0 33 83 17 0 0 100 0 100 0 0 100 0 0
Satt294 90 0 10 75 0 25 0 0 100 0 83 17 100 0 0 0 75 25 67 0 33
Satt308 100 0 0 50 0 50 33 0 67 mono 50 50 0 mono 100 0 0
Satt431 80 0 20 50 50 0 0 100 0 mono 100 0 0 50 0 50 100 0 0
Satt030 100 0 0 100 0 0 67 0 33 0 83 17 100 0 0 mono Mono
Satt045 90 0 10 75 0 25 0 100 0 50 0 50 0 0 100 100 0 0 Mono
Keterangan: G=Grobogan
Dua kombinasi persilangan lainnya yaitu B4545 X B62 dan Wilis X B57 masing-masing polimorfik hanya pada lima dan empat SSR. Sebagian besar populasi menunjukkan polimorfisme lebih dari dua SSR, kecuali pada tetua Wilis X B57. Berdasarkan pola polimorfisme dengan alel SSR tersebut, B57, dan Grobogan-B62 cukup jauh secara genetik, kontras dengan Wilis-B57 maupun B4545-B62.
Dari survei polimorfisme pada semua kombinasi tetua persilangan terlihat jelas bahwa marka SSR sangat berguna dalam menganalisis keragaman genetik dari genotipe kedelai baik berkerabat dekat maupun jauh. Hasil ini mendukung informasi sebelumnya sebagai pengembangan marka SSR dalam studi ini (Diwan & Cregan, 1997; Cregan et al., 1999), bahwa marka SSR ini bermanfaat untuk seleksi dalam populasi persilangan dan dapat diaplikasikan pada beberapa populasi kedelai. Tetua persilangan seperti Grobogan, Wilis, B4545 dan genotipe lainnya memiliki karakter morfo-agronomi berlainan terkait produksi, umur pembungaan dan umur panen, karakter biji dan lainnya. Jadi, penggunaan marka ini juga membuktikan keberhasilan evaluasi genotipe termasuk tetua persilangan yang memiliki karakter morfo-agronomi spesifik (Santana et al., 2014).
Pola kesamaan alel ditunjukkan antara F1 dengan tiap tetuanya pada 9 marka SSR yang digunakan. Berdasarkan penelusuran alel tiap SSR, diidentifikasi bahwa tidak semua nomor individu F1 adalah hasil persilangan dari kedua tetua pada tiap populasi. Beberapa
individu merupakan hasil selfing dari masing-masing tetua, sesuai dengan hasil Kuroda et al. (2013) yang menyeleksi progeni hasil persilangan kedelai liar dan kedelai budidaya dan ditemukan selfing. Dimana ada alel yang tidak diekspresikan secara fenotipik sehingga pendekatan molekuler diperlukan. Dalam studi ini, secara SSR tunggal belum dapat meyakinkan kebenaran progeni F1 namun ada kecenderungan konsistensi beberapa marka. Sebagai contoh lima SSR (Satt009, Satt144, Satt191, Satt294 dan Satt045) berhasil mengkonfirmasi bahwa 10% dan 25% F1, dan 90% dan 75% masing-masing adalah introgresi kedua tetua dan selfing Grobogan pada populasi Grobogan X B57 dan Grobogan X B62. Pada populasi lain bahkan terlihat jelas bahwa progeni yang dihasilkan adalah selfing dari salah satu persilangan. Hal penting, gabungan dari alel SSR tersebut berhasil mengkonfirmasi kebenaran individu generasi F1 dari alel yang diturunkan dari kedua tetua persilangannya. Informasi ini memperlihatkan bahwa marka-marka universal (Cregan et al., 1999) bermanfaat untuk seleksi pada generasi awal dan dapat untuk generasi lanjutan terkait karakter tertentu misalnya ketahanan terhadap cyst nematoda (Santana et al., 2014).
Sebanyak tiga populasi F1 menunjukkan 100% keberhasilan persilangan dari tetuanya (B4541 X B57, B4621 XB57 dan B4545 X B62), dan dua populasi hanya 50% keberhasilannya yaitu Grobogan X B62 dan Wilis X B57. Persilangan hanya berhasil 40% ditemukan pada F1 dari persilangan Grobogan X B57 (Tabel 3). Secara detil,
Pusat Studi Lingkungan dan Kependudukan (PSLK)
Universitas Muhammadiyah Malang, tanggal 29 April 2017
Lestari et al., Konfirmasi Penurunan Alel
5
individu-individu F1 yang benar-benar hasil persilangan telah diidentifikasi (Tabel 4).
Tabel 3. Persentase keberhasilan persilangan yang diperoleh pada generasi F1 dari tujuh populasi kedelai
Progeni F1 GXB57 GXB62 B4641XB57 B4621XB62 B4621XB57 WilisXB57 B4545XB62
Total progeni 10.0 4.0 3.0 6 2 8 3
Jumlah F1 benar 4.0 2.0 3.0 3.0 2.0 4.0 3.0
% F1 benar 40% 50% 100% 67 100 50 100
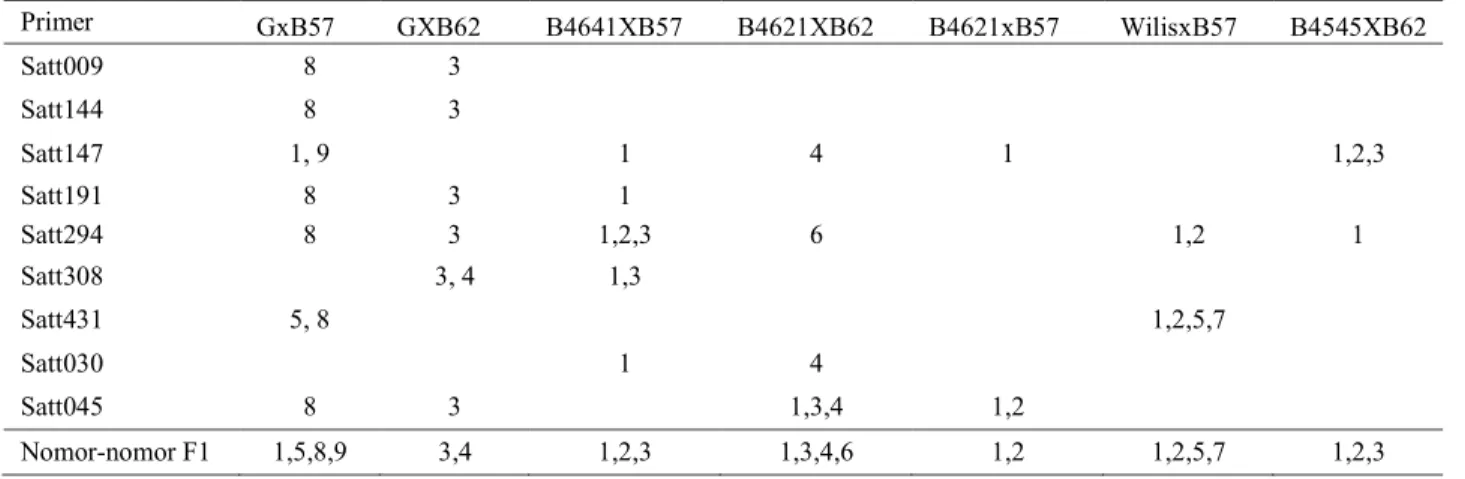
Tabel 4. Nomor-nomor individu yang merupakan progeni F1 hasil persilangan dari masing-masing tetua varietas kedelai Primer GxB57 GXB62 B4641XB57 B4621XB62 B4621xB57 WilisxB57 B4545XB62 Satt009 8 3 Satt144 8 3 Satt147 1, 9 1 4 1 1,2,3 Satt191 8 3 1 Satt294 8 3 1,2,3 6 1,2 1 Satt308 3, 4 1,3 Satt431 5, 8 1,2,5,7 Satt030 1 4 Satt045 8 3 1,3,4 1,2 Nomor-nomor F1 1,5,8,9 3,4 1,2,3 1,3,4,6 1,2 1,2,5,7 1,2,3
Dari total 10 nomor F1 Grobogan X B57, diidentifikasi bahwa nomor 1,5,8 dan 9 membawa alel Grobogan dan B57, demikian juga nomor 3 dan 4 yang membawa alel Grobogan dan B62. Hasil analisis SSR ini terbukti secara cepat dan efisien mampu menyeleksi individu-individu hasil persilangan tanpa harus melakukan karakterisasi morfologi di lapang yang memerlukan waktu lama, sesuai dengan laporan sebelumnya (Wu et al., 2011; Khanh et al., 2013). Namun demikian, konfirmasi karakterisasi moroflogi di lapang dari individu-individu F1 terseleksi yang merupakan hasil persilangan dari kedua tetuanya diperlukan. Selanjutnya, dalam studi ini 9 marka SSR cukup untuk konfirmasi kebenaran progeny F1 dan sebagian besar genotipe sudah dapat dibedakan. Namun agar tingkat diskriminatif lebih tinggi, jumlah marka dianjurkan ditambah untuk analisis selanjutnya dalam membantu seleksi progeni dalam pemuliaan kedelai.
KESIMPULAN
Sebanyak 9 dari 10 marka SSR menunjukkan polimorfisme dan nilai PIC tujuh marka lebih dari 0,7 yang merefleksikan kemampuannya mendiskriminasi genotipe dan seleksi individu F1. Gabungan dari alel SSR tersebut berhasil mengkonfirmasi kebenaran individu generasi F1 dari alel yang diturunkan dari kedua tetua persilangannya pada tujuh populasi. Keberhasilan persilangan kedelai telah diidentifikasi, dimana 3 populasi menunjukkan keberhasilan 100%. Nomor-nomor individu F1 yang benar-benar merupakan hasil persilangan telah diidentifikasi dan perlu dikonfirmasi di lapang melalui karakterisasi morfologinya.
UCAPAN TERIMA KASIH
Penulis mengucapkan terima kasih kepada Della Suciyanti dan Mufti Alfiansyah yang telah membantu dalam pengerjaan analisis laboratorium, serta Kristianto Nugroho yang telah membantu analisis data.
DAFTAR RUJUKAN
Adie, M.M., & Krisnawati, A. (2013). Keragaman dan seleksi hasil biji dari galur-galur kedelai generasi lanjut. Dalam: Patiwi H & Winarto A (Eds.).
Prosiding Seminar Nasional Hasil penelitian Tanaman Aneka Kacang dan Umbi. pp. 19-26.
Pusat Penelitian dan Pengembangan Tanaman Pangan. Badan Penelitian dan Pengembangan Pertanian, Bogor.
Balitbangtan. (2016). Dega-1, VUB untuk menunjang
swasembada kedelai. Retrieved from
www.litbang.pertanian.go.id/berita/one/2531 BPS. (2014). Produksi padi, jagung, dan kedelai. Berita
Resmi Statistik. No 22/03/Th XVII, 3 Maret 2014. BPS. (2015). Pertanian. Badan Pusat Statistik. (Diakses
pada 30 November 2015).
Bredemeijer, M., Cooke, J., Ganal, W., Peeters, R., Isaac, P., Noordijk, Y., Rendell, S. Jackson, J., Roder, S., Wendehake, K., Dijcks, Amelaine, M., Wickaert, V., Bertrand, L, & Vosman B. (2002). Construction and testing of a microsatellite containing more than 500 tomato varieties. Theor.
Chaerani, Hidayatun, N., &. Utami, D.W. (2011). Keragaman genetik 50 aksesi plasma nutfah kedelai berdasarkan sepuluh penanda mikrosatelit.
Jurnal AgroBiogen, 7(2), 96-105.
Cregan, P.B., Jarvik, T., Bush, A.L., Shoemaker, R.C., Lark, K.G., Kahler, A.L., Kaya, N., Vantoai, T.T., Lohnes, D.G., Chung, J., & Specht, J.E. (1999). An integrated genetic linkage map of the soybean.
Crop Science, 39, 1464-1490.
Diwan, N., & Cregan, P.B. (1997). Automated sizing of flourescen-labeled simple sequence repeat (SSR) markers to assay genetic variation in soybeans.
Theor. Appl. Genet., 95, 723-733.
Erliana G. (2010). Mutu kedelai nasional lebih baik dari kedelai impor. Berita Puslitbangtan 45. November 2010. Hal. 17.
Handoyo, D., & Rudiretna, A. (2001). Prinsip umum dan pelaksanaan Polymerase Chain Reaction (PCR).
Unitas, 9(1), 17-29.
Jun, T.H., Minchel, A.P., & Mian, M.A.R. (2011). Development of soybean aphid genomic SSR markers using next generation sequencing.
Genome, 54(6), 360-367.
Khanh, T.D., Anh, T.Q., Buu, B.C., & Xuan, T.D. (2013). Applying molecular breeding to improve soybean rust resistance in Vietnamese elite soybean.
American J. Plant Sci., 4,1-6.
Kuroda, Y., Kaga, A., Tomooka, N., Yano, H., Takada, Y., Kato, S., & Vuaghan, D. (2013). QTL affecting fitness of hybrids between wild and cultivated soybeans in experimental fields. Ecology and
Evolution, 3(7), 2150-2168.
Lestari, P., Risliawati, A., Utami, D.W., Hidayatun, N., Santoso, T.J., & Chaerani. (2016). Pengembangan identitas spesifik berbasis marka SSR pada 29
varietas kedelai lokal Indonesia. Jurnal Biologi
Indonesia, 12(2), 219-229.
Li, Y.C., Korol, A.B., Fahima, T., Beiles, A., & Nevo, E. (2002). Microsatellites: genomic distribution, putative functions and mutational mechanisms: a review. Molecular Ecology, 11(12), 2453-2465. Mandal, R., Nag, S., Tarafdar, J., & Mitra, S., (2016). A
comparison of efficieny parameters of SSR markers and genetic diversity analysis in
Amaorphophallus paeniifolius (Dennst.) Nicolson. Braz. Arch.Biol.Tchnol., 59, e160492.
Marliah, A., Hidayat. T., & Husna, N. (2012). Pengaruh varietas dan jarak tanaman terhadap pertumbuhan dan hasil tanaman kedelai (Glycine max L.). Universitas Syiah Kuala, Banda Aceh. Jurnal
Agrista, 16(1), 22-28.
Santana, F.A., da Silva, M.F., Guimares, J.K.F., Ferreira, M.F.S., Pereira, W.D. Piovesan, N.D., & de Barros, E.G. (2014). Marker-assisted selection strategies for developing resistant soybean plants to cyst nematode. Crop Breeding and Applied
Biotechnology, 14(3), 180-186.
Santoso, T.J., Dwinita, W. U., & Septiningsih, E.M. (2006). Analisis sidik jari DNA plasma nutfah kedelai menggunakan markah SSR. Jurnal
Agrobiogen, 2(1), 1-7.
Van Eeuwijk, F.A., & Baril, C.P. (2001). Conceptual and statistical issues related to the use of molecular markers for distinctness and essential derivation. Proc. Int. Symp. on Molecular Markers. Eds. Doré, Dosba & Baril. Acta Hort., 546, 35-53.
Wu, X., Vuong, T.D, Leroy, J.A., Shannon, J.G., Sleper, D.A., & Nguyen, H.T. (2011). Selection of a core set of RILs from Forrest X William 82 to develop a framework map in soybean. Theor. Appl. Genet., 122(6), 1179-1187.