100
Keragaman Genetik Populasi Tetua Saudara Kandung (Sibs) Kelapa Sawit Dura
Deli Berdasarkan Penanda DNA Mikrosatelit
NIDA WAFIQAH NABILA M. SOLIN
1, SOBIR
1, DAN NURITA TOURAN-MATHIUS
21Departemen Agronomi dan Hortikultura, Fakultas Pertanian, IPB Jalan Meranti, Kampus IPB Dramaga Bogor 16680 2Plant Production and Biotechnology Division, PT. SMART TBK Jalan Raya Cijayanti KP. Pasar Maung RT.004 / 006, Bogor 168810
E-mail: [email protected].
Diterima 27 Mei 2013 / Direvisi 5 Agustus 2013 / Disetujui 25 Nopember 2013
ABSTRAK
Mikrosatelit merupakan penanda yang sangat efisien untuk mengevaluasi struktur keragaman genetik pada genus Elaeis. Dura Deli merupakan materi genetik yang digunakan sebagai tetua dalam program pemuliaan kelapa sawit yang perlu diketahui potensi genetiknya sebelum digunakan dalam perakitan varietas baru maupun produksi benih. Penelitian ini bertujuan untuk mendapatkan informasi keragaman genetik tetua saudara kandung (sibs) Dura Deli kelapa sawit berdasarkan penanda DNA mikrosatelit. Dua populasi Dura Deli hasil selfing dengan 30 sampel per populasi digunakan untuk dianalisis dengan 27 primer mikrosatelit. Pola pita DNA menunjukkan bahwa terdapat 20 primer yang polimorfik dan 7 primer yang monomorfik pada sampel yang diuji, dengan jumlah alel 2 sampai 5. Persentase lokus polimorfik yang diperoleh sebesar 96,30% dengan rata-rata nilai PIC 0,50. Analisis keragaman genetik menggunakan analisis PCoA, struktur, maupun NTSys, memisahkan populasi tetua Dura Deli menjadi dua kelompok besar sesuai asal usul tetuanya. Hasil klaster analisis menunjukkan formasi kedua grup berdasarkan keragaman genetiknya. Grup A, dengan keragaman genetik ~20%, merupakan hasil selfing Dura Deli 1. Sedangkan Grup B, dengan keragaman genetik ~ 28% merupakan hasil selfing Dura Deli 2. Kedua bahan genetik ini memiliki keragaman genetik yang rendah di dalam kelompok, namun keragaman genetik cukup tinggi antar kelompok (~32%), sebagaimana dikonfirmasi dengan menggunakan PCoA dan Struktur. Keragaman genetik yang rendah intra populasi tetua tersebut memungkinkan untuk digunakannya sebagai materi persilangan, dalam perakitan varietas baru maupun produksi benih sawit.
Kata kunci: Kelapa Sawit, Dura Deli, lokus polimorfik, analisis kluster, keragaman genetik.
ABSTRACT
Genetic Diversity of Deli Dura Oil Palm’s Elder Siblings Population based on
Microsatellite Markers
Microsatellite is a marker that highly efficient to evaluate the genetic diversity structure of Elaeis genus. Dura Deliis the genetic material that is used as a parent in breeding programs palm, before is used as the crosses material for new varieties’s assembly or oil production’s seed production. This study is aimed to elucidate genetic diversity information. Two populations of Dura Deli selfing with 30 samples per population used to be analyzed with 27 microsatellites. Deoxyribose Nucleic Acid banding pattern indicates that there are 20 polymorphic and 7 monomorphic markers on samples tested, with the number of alleles from 2 to 5. The percentage of polymorphic loci obtained at 96.30 % with an average PIC value of 0.48. Analysis of genetic diversity using PCoA, structure or NTSys separating the Dura Deli parental populations into two major groups corresponding to the origin. The results of cluster analysis showed the formation into two groups based on their genetic diversity. Group A, with ~ 20% genetic diversity, is the result of Dura Deli 1 selfing. While Group B, with ~28% genetic diversity is the result of Dura Deli 2 selfing. The genetic material has a low genetic diversity within the group but higher genetic diversity between group (~32 %), as well as confirmed by PCoA and Structure. Those less genetic diversity intra population allows to be used as the crosses material for new varieties’s assembly or oil production’s seed production.
Keywords: Oil palm, Dura Deli, polymorphic locus, cluster analysis, genetic diversity.
PENDAHULUAN
Kelapa sawit (Elaeis guineensis Jacq.) merupakan komoditi perkebunan yang mempunyai peran penting dalam berbagai aspek kehidupan di
Indonesia, khususnya aspek perekonomian dalam negeri. Indonesia memiliki beberapa keunggulan sebagai salah satu negara yang sangat potensial untuk menanamkan investasi di bidang perkebunan kelapa sawit dan industri hilirnya. Hal tersebut ditunjang
101
dengan meningkatnya permintaan minyak sawitdunia untuk kebutuhan pangan (edible oil), industri (oleochemical), dan sumber energi alternatif berbasis biodiesel (Wahyono, 2006). Secara konsisten pen-dapatan nasional dari kelapa sawit terus menunjuk-kan peningkatan dan berpeluang menjadi komoditi yang paling menguntungkan dibandingkan dengan komoditi lainnya (Sugema 2007).
Luas areal tanaman kelapa sawit di Indonesia, hanya sekitar 7.363.847 pada tahun 2008, kemudian berkembang sangat cepat hingga mencapai 9.074.621 pada tahun 2012. Hal ini seiring dengan mening-katnya produksi CPO, yaitu 17.539.788 pada tahun 2008 hingga mencapai 23.521.071 ton pada tahun 2012 (Direktorat Jenderal Perkebunan, 2012). Indonesia sampai saat ini merupakan negara penghasil Crude
Palm Oil (CPO) terbesar di dunia.
Pengembangan industri kelapa sawit memer-lukan beberapa upaya untuk mencapai peningkatan produktivitas nasional, salah satunya adalah peman-faatan benih unggul bermutu didukung oleh ketersediaan sumber daya genetik (plasma nutfah) yang mempunyai tingkat keragaman genetik yang tinggi (Direktorat Jenderal Perkebunan, 2010). Benih unggul bermutu dapat dihasilkan dari program pemuliaan yang efisien dan terarah. Peningkatan produksi melalui penyediaan bibit unggul berdaya hasil tinggi merupakan salah satu upaya strategis, yang terus dilakukan untuk memenuhi kebutuhan minyak sawit yang semakin meningkat dari tahun ke tahun (Asmono et al., 1998).
Program pemuliaan untuk mendapatkan benih unggul bermutu kelapa sawit terus dilakukan oleh para pemulia, melalui perakitan varietas baru yang dievaluasi berdasarkan karakter fenotipenya. Dura Deli merupakan salah satu materi genetik yang digu-nakan sebagai tetua betina dalam merakit varietas unggul dan produksi benih bermutu kelapa sawit. Informasi potensi genetik Dura Deli perlu diketahui untuk memastikan keunggulannya. Seleksi tetua untuk perakitan varietas baru dapat dilakukan melalui identifikasi karakter morfologi dan produksi secara langsung pada tanaman kelapa sawit. Namun, fenotipik sangat dipengaruhi oleh lingkungan, sehingga diperlukan bantuan metode lain yang lebih akurat untuk mendapatkan informasi bahan genetik. Oleh karena itu, pendekatan molekuler yang mena-ngani variasi genetik dari populasi alami tanpa referensi karakter fenotipik sedang diadopsi secara luas. Penggunaan penanda DNA dapat meningkatkan ketepatan dan efisiensi seleksi, yang mengarah ke percepatan pengembangan bahan genetik baru dengan hasil yang tinggi. Mikrosatelit menyediakan alat untuk Marker Assisted Selection (MAS) dalam berbagai aplikasi (Collard et al., 2008).
Mikrosatelit merupakan penanda genetik yang telah terbukti dapat digunakan sebagai alat untuk analisis genetik tanaman. Beberapa studi telah mem-bandingkan polimorfisme dari RAPD, RFLP AFLP danan SSR (Antonio et al., 2004). Pada banyak kasus, SSR paling polimorfik dalam setiap lokus. Polimor-fisme ini dapat digunakan untuk membedakan varietas yang berjarak genetik dekat dan untuk memperkirakan hubungan genetik antar individual. Billotte et al. (2001) melaporkan hasil pengembangan penanda SSR kelapa sawit yang dilakukan secara bertahap, mulai dari penapisan pustaka SSR yang diperkaya dengan unit pengulangan (GA)n, (GT)n, dan (CCG)n, sampai kepada karakterisasi akhir lokus SSR. Saat ini, SSR merupakan sistem penanda molekuler paling menjanjikan untuk memahami struktur populasi genetik kelapa sawit (Singh et al., 2009). Selain itu, Thongthawee et al. (2010) telah menggunakan penanda mikrosatelit untuk analisis tetua pada kelapa sawit. Analisis data multivariat menunjukkan kemampuan penanda SSR yang sangat efisien untuk mengevaluasi struktur keragaman genetik pada genus Elaeis. Keberadaan keragaman alel yang tinggi mengindikasikan bahwa penggunaan SSR pada E. guineensis akan menjadi perangkat yang sangat bermanfaat untuk kajian genetik.
Teknik yang merupakan kombinasi seleksi berbasis penanda molekuler dengan teknik pemuliaan konvensional merupakan alat bantu strategis yang dapat mempersingkat waktu seleksi, sehingga dapat mempercepat pencapaian tujuan pemuliaan tanaman, yaitu untuk menyediakan sumberdaya genetik, khususnya tetua yang memiliki karakter unggul dalam waktu yang singkat. Penelitian ini bertujuan untuk mendapatkan informasi keragaman genetik tetua saudara kandung Dura Deli kelapa sawit berdasarkan penanda DNA mikrosatelit.
BAHAN DAN METODE
Penelitian dilaksanakan di Laboratorium
Genomic and Traanscriptomic, Plant Production and Biotechnology Division, PT SMART Tbk., Sentul.
Pengambilan sampel tanaman dilaksanakan di Kebun Percobaan Indra Sakti Estate, Pekanbaru. Waktu pelaksanaan penelitian dimulai dari bulan Agustus 2012 hingga November 2013.
Bahan Tanaman dan Isolasi DNA
Tanaman yang digunakan adalah sampel daun muda kelapa sawit yang terdiri dari 30 tetua Dura Deli 1 dan 30 tetua Dura Deli 2 terseleksi milik PT. SMART Tbk. Ke-60 tetua merupakan saudara kandung (sibs), yang tersedia sebagai sampel tanaman di kebun percobaan Indra Sakti Estate, Pekanbaru.
102
DNA genom total diperoleh dengan menggerus sampel menggunakan Nitrogen cair, kemudian sampel diisolasi menggunakan GenElute Plant Genomic DNA Miniprep Kit (Sigma-Aldrich). Kualitas DNA diketahui melalui elektroforesis meng-gunakan gel agarose 1%, sedangkan kuantitas DNA ditentukan menggunakan NanoDrop HND-1000 spektrofotometer (NanoDrop Technologies Inc.). Konsentrasi DNA yang digunakan untuk reaksi PCR sebanyak 20 ng μL-1.
Amplifikasi PCR
Evaluasi parameter Polymerase Chain Reaction (PCR) untuk amplifikasi mikrosatelit menggunakan DNA bahan tanaman tetua yang dianalisis. Sebanyak 60 sampel tetua dievaluasi dengan 27 penanda mikro-satelit sesuai dengan kondisi amplifikasi yang di-laporkan oleh Billotte et al. (2001; 2005). Polyacrylamide
Gel Electrophoresis (PAGE) 6%w/v digunakan untuk
visualisasi produk hasil amplifikasi PCR dengan metode pewarnaan mengikuti Benbouza et al. (2006). Pita DNA yang diperoleh dari lokus SSR diskor secara manual sesuai metode Allou et al. (2008).
Analisis Data
Analisis data berdasarkan hasil skoring pita DNA pada gel akrilamid. Pita diskor secara manual sebagai data biner dengan skor 1 untuk yang ada pita atau skor 0 untuk yang tidak ada pita. Selanjutnya data disusun berdasarkan format data perangkat lunak yang digunakan.
Penghitungan jumlah alel dan Polymorphism
Information Content (PIC) menggunakan software
PowerMarker V3.25 (Liu dan Muse 2005). Untuk menghitung jumlah alel efektif per lokus (Ne), heterozigositas teramati (Ho), heterozigositas harapan (He), dan analisis koordinat utama (PCoA), diguna-kan GenAlex ver. 6.501 (Peakall dan Smouse, 2012). Pembuatan pohon filogeni dan penentuan jarak genetik antar tetua digunakan NTSyspc2.1 (Rohlf, 2000), dan untuk melihat struktur populasi digunakan
Structure (Pritchard et al., 2000).
HASIL DAN PEMBAHASAN
Analisis Mikrosatelit dan Polimorfisme
Hasil yang diperoleh dari 27 primer yang dievaluasi menunjukkan bahwa terdapat 20 primer yang polimorfik dan 7 primer yang monomorfik pada sampel yang digunakan, dengan jumlah alel berkisar antara 2 sampai 5 alel, seperti yang disajikan padaTabel 1.
Persentase lokus polimorfik yang diperoleh adalah sebesar 96,30% baik pada populasi Dura Deli 1 maupun Dura Deli 2 untuk lokus mikrosatelit yang digunakan. Polimorfisme yang tinggi ini menjelaskan kemampuan penanda untuk mengamplifikasi target sekuen dan mendeteksi polimorfisme antar tetua yang diuji. Proporsi lokus polimorfik yang sangat tinggi ini juga menunjukkan bahwa terdapat hetero-zigositas dan heterogenitas genetik yang tinggi antara individu-individu di dalam populasi (Ajambang et al., 2012). Polimorfisme dalam populasi tertentu sering disebabkan adanya varian genetik yang diwakili oleh jumlah alel pada lokus dan frekuensi distribusinya dalam suatu populasi (Dangi et al., 2004).
Jumlah Alel Efektif (Ne), Heterozigositas Harapan (He), Heterozigositas Teramati (Ho) dan Polymor-phism Information Content (PIC)
Jumlah alel efektif adalah sebuah ukuran dari jumlah alel efektif yang diperoleh dari masing-masing karakter. Nilai ini adalah nilai resiprok atau nilai kebalikan dari homozigositas. Semakin tinggi nilai Ne, maka semakin banyak individu yang heterozigot. Rata-rata jumlah alel efektif yang diperoleh pada populasi yang diuji adalah 2,35. Jumlah ini lebih rendah dari yang didapatkan Zulhermana et al. (2010) (3,67), Agustina et al. (2010)(3,85), Ajambang et al. (2012) (4,71), Billotte et al. (2001) (5,3), tetapi lebih tinggi dari yang diperoleh Maizura et al. (2006) (1,9) dan Allou et al. (2008) (1,75).
Rendahnya jumlah alel pada analisis ini ke-mungkinan disebabkan materi genetik yang diguna-kan merupadiguna-kan populasi tetua Dura saudara kandung (sibs), yang memiliki kedekatan genetik dan semakin menurun keragaman genetiknya akibat
selfing yang telah dilakukan. Upaya-upaya perbaikan
genetik kelapa sawit menyebabkan keragaman genetik menyempit, sehingga alel yang diperoleh juga semakin sedikit. Bakoume et al. (2007) menemukan bahwa alel pada populasi liar kelapa sawit menunjuk-kan penurunan yang disebabmenunjuk-kan oleh seleksi ber-tahun-tahun pada bahan genetik. Arias et al. (2012) menggambarkan hasil ini sebagai kecenderungan umum penurunan keragaman genetik yang disebab-kan oleh perbaidisebab-kan genetik pada kelapa sawit.
Nilai heterozigositas teramati (Ho) berkisar antara 0,35-1,00 dan nilai heterozigositas harapan (He) berkisar antara 0,21-0,74. Secara umum nilai rata-rata heterozigositas teramati lebih tinggi daripada nilai rata-rata heterozigositas harapan. Hal ini berarti bahwa setiap karakter memiliki heterozigositas yang tinggi. Nilai heterosigositas ini sesuai dengan probabilitas bahwa dua alel yang diambil secara acak dari suatu populasi dapat dibedakan dengan menggunakan penanda yang diuji (Dangi et al., 2004).
103
Tabel 1. Daftar mikrosatelit yang dievaluasi, dengan jumlah alel dan polimorfisme teramati.Table 1. List of microsatellite evaluated, with the number of allele sand the polymorphism observed.
Lokus Forward Primer Reverse Primer Alel Jlh. Polimor-fisme
mEgCIR3847 CGTTAGTGTCGCTTATTATG AAATGAGGAAGCGTAAC 4 P
mEgCIR3574 AGAGACCCTATTTGCTTGAT GACAAAGAGCTTGTCACAC 4 P
mEgCIR0878 CAAAGCAACAAAGCTAGTTAGTA CAAGCAACCTCCATTTAGAT 3 P
mEgCIR2518 GATCCCATGGTAAAGACT AAGCCTCAAAAGAAGACC 2 M
mEgCIR2569 TAGCCGCACTCCCACGAAGC CCAGAATCATCAGACTCGGACAG 2 M
mEgCIR2427 GAAGGGGCATTGGATTT TACCTATTACAGCGAGAGTG 4 P
mEgCIR0783 GAATGTGGCTGTAAATGCTGAGTG AAGCCGCATGGACAACTCTAGTAA 3 P
mEgCIR0059 TGCAGGGGATGCTTTTATT CCCTTAATTCCTGCCTTATT 3 P
mEgCIR3869 CCAATGCAGGGGACATT GAAGCCAGTGGAAAGATAGT 3 P
mEgCIR3519 CCACTGCTTCAAATTTACTAG GCGTCCAAAACATAAATCAC 3 P
mEgCIR3639 ACGTTTTGGCAACTCTC ACTCCCCTCTTTGACAT 4 P
mEgCIR0134 AGTTTGGGGCTTACCTG CTTCCACGCACCCTCTC 2 M
mEgCIR0046 AGCCTTAGTATTTTGTTGAT CCTCTGATTTGTCCTTTTGG 4 P
mEgCIR3890 GTGCAGATGCAGATTATATG CCTTTAGAATTGCCGTATC 3 P
mEgCIR3389 GTCCATGTGCATAAGAGAG CTCTTGGCATTTCAGATAC 3 P
mEgCIR3543 GTTCCCTGACCATCTTTGAG GTCGGCGATTGATTAGATTC 3 P
mEgCIR0779 AATGCAGACCAAGCTAATCATATAC GTTCAGGTGATGGTGACTCAGATAG 2 M
mEgCIR2144 ACAAGGCTCTTCAAGAGAT CCACTGCCAACACTAGTAC 4 P
mEgCIR3555 CATCAGAGCCTTCAAACTAC AGCCTGAATTGCCTCTC 4 P
mEgCIR3569 AAGGCTTGGAGTTGAGGTAT CACCATTGCATCATTATTCC 1 M
mEgCIR3809 CCTTGCATTCCACTATT AGTTCTCAAGCCTCACA 2 M
mEgCIR3275 GAAGCCTGAGACCGCATAGA TTCGGTGATGAAGATTGAAG 2 M
mEgCIR2590 GTAGTTAAGGGACTTGTAGTC AAGTCTCTTGTGCTGATACA 2 P
mEgCIR3260 AGGGCAAGTCATGTTTC TATAAGGGCGAGGTATT 2 P
mEgCIR0801 TGGTTGGCAGGTATTATTAG TTAGAGGCTGTGATGAGTTG 5 P
mEgCIR0521 GTGACTTTGGGCTGAAT ACAGCATCTCCAACTCTATC 3 P
mEgCIR0326 GCTAACCACAGGCAAAAACA AAGCCGCACTAACATACACATC 3 P Keterangan: P = Polimorfik; M = monomorfik.
Note : P = Polymorphism; M = Monomprphism.
Nilai PIC tertinggi yang diperoleh pada studi ini adalah 0,70 pada lokus mEgCIR2144 dan terendah adalah 0,18 pada lokus mEgCIR3260, dengan rata-rata nilai PIC sebesar 0,50 (Tabel 2). Nilai ini meng-indikasikan bahwa penanda SSR tersebut cukup informatif. Botstein et al. (1980) mengklasifikasikan nilai PIC menjadi 3 tiga kelas, yaitu PIC>0.5 adalah sangat informatif, 0.25>PIC<0.5 termasuk sedang dan
PIC<0.25 memiliki nilai informatif yang rendah. Menurut Missio et al. (2010), semakin besar nilai PIC dalam suatu penanda, maka semakin baik penanda tersebut digunakan sebagai penanda molekuler. Dalam penelitian ini, terdapat 11 dari 27 penanda yang memiliki nilai PIC > 0.5, yang berarti penanda tersebut dapat digunakan untuk studi pemuliaan selanjutnya pada kelapa sawit.
104
Tabel 2. Jumlah alel efektif (Ne), heterozigositas harapan (He), heterozigositas teramati (Ho) dan Polymorphism Information Content (PIC) pada 20 lokus SSR yang polimorfik.
Table 2. The number of effective alleles (Ne), heterozygosity expectations (He), observed heterozygosity (Ho) and Polymorphism Information Content(PIC) at 20SSR loci were polymorphic. Locus Ne Ho He PIC mEgCIR3847 2,06 0,93 0,51 0,40 mEgCIR3574 2,94 1,00 0,66 0,60 mEgCIR0878 2,18 0,72 0,54 0,44 mEgCIR2427 3,85 1,00 0,74 0,69 mEgCIR0783 2,07 0,73 0,52 0,45 mEgCIR0059 2,67 1,00 0,63 0,55 mEgCIR3869 2,58 1,00 0,61 0,54 mEgCIR3519 2,20 0,95 0,55 0,44 mEgCIR3639 2,79 0,47 0,64 0,57 mEgCIR0046 2,89 1,00 0,65 0,60 mEgCIR3890 2,29 1,00 0,56 0,47 mEgCIR3389 2,02 0,35 0,51 0,44 mEgCIR3543 2,23 0,73 0,55 0,48 mEgCIR2144 4,00 1,00 0,75 0,70 mEgCIR3555 2,44 1,00 0,59 0,50 mEgCIR2590 1,64 0,53 0,39 0,31 mEgCIR3260 1,26 0,23 0,21 0,18 mEgCIR0801 3,12 0,88 0,68 0,62 mEgCIR0521 2,67 1,00 0,63 0,55 mEgCIR0326 2,51 1,00 0,60 0,52
Keragaman Genetik Antar dan Intra Populasi
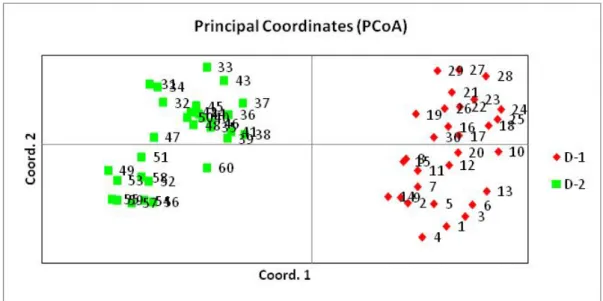
Hasil analisis Principle Coordinate Analysis (PCoA) menunjukkan pemisahan genotipe hasil selfing Dura Deli 1 (1-30), dari genotipe hasil selfing Dura Deli 2 (31-60) (Gambar 1). Hal ini membuktikan bahwa pengelompokan hasil analisis molekuler kedua selfing pohon induk Dura tersebut berbanding lurus dengan data fenotipik di lapang. Hasil pengamatan karakter morfologi dan produksi di lapang menun-jukkan perbedaan yang cukup signifikan antara kedua populasi yang diuji. Hasil ini juga menun-jukkan bahwa masing-masing populasi tetua yang diuji memiliki tingkat keragaman yang rendah antar individu dalam populasi dan memiliki perbedaan yang cukup tinggi dengan populasi lainnya.
Arias et al. (2012) mendapatkan hasil PCoA yang menunjukkan distribusi setiap individu di dalam setiap grup dengan tingkat kesamaan genetik yang tinggi. Hasil ini konsisten dengan hasil yang dilaporkan oleh studi keragaman genetik yang me-libatkan bahan genetik tipe Deli Dura sebagai referensi, dan dapat disimpulkan bahwa populasi
yang telah diperbaiki genetiknya memiliki keragaman genetik yang lebih rendah. Melchinger (1993) menga-takan bahwa tujuan PcoA, yaitu untuk menghasilkan plot grafis dengan dimensi rendah dari data, sehingga jarak geomatris antara individu-individu dalam plot mencerminkan jarak genetik antar individu dengan distorsi minimal.
Struktur populasi data dianalisis meng-gunakan Structure (Pritchard et al., 2000) dengan nilai
burn-in period 100000 dan MCMC 100000. Sebanyak 25
analisis independen dilakukan untuk setiap nilai K (dari 1 sampai 5) dengan 5 kali perulangan. Nilai K sebenarnya kemudian dianalisis menggunakan
Structure Harvester (Earl et al., 2012), sebuah program
untuk memvisualisasi hasil structure dan meng-implementasikan metode Evanno et al. (2005). Hasil dari Structure Harvester menunjukkan bahwa nilai K tertinggi terdapat pada K=2. Hasil analisis struktur populasi juga mengindikasikan bahwa ke-60 pohon tetua dapat dikelompokkan menjadi dua grup.
105
Gambar 1. Principal Coordinate Analysis (PCoA) pada 60 sampel kelapa sawit yang digunakan.Figure 1. Principal Coordinate Analysis (PCoA) on 60 samples of oil palm used
Cluster D1 Cluster D2
Gambar 2. Kesamaan genetik populasi tetua kelapa sawit dengan hasil klastering terbaik (K=2) dari 60 pohon tetua.
Figure 2. Genetic similarity of the parental population clustering oil palm with the best results (K =2) of 60 elder tree.
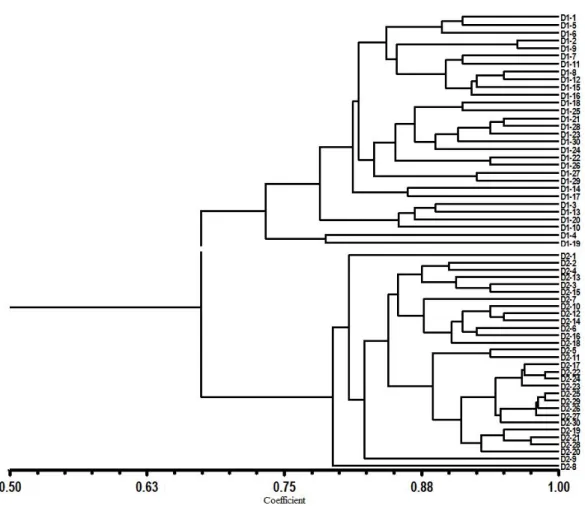
Hasil analisis klaster menggunakan NTSys, menghasilkan dendogram yang menunjukkan for-masi ke dua grup berdasarkan kemiripan genetiknya (Gambar 3). Grup A dengan keragaman genetik ~20%, merupakan bahan genetik hasil selfing Dura Deli 1. Sedangkan Grup B dengan keragaman genetik ~28% merupakan bahan genetik hasil selfing Dura Deli 2. Kedua bahan genetik ini memiliki keragaman genetik yang rendah di dalam kelompok, namun memiliki keragaman genetik yang lebih tinggi antar kelompok (~32%).
Nilai keragaman genetik yang rendah di dalam dua kelompok tetua yang dianalisis ini dapat dijelaskan oleh beberapa faktor, antara lain (a) fakta bahwa populasi tersebut merupakan hasil selfing dari
tetua Dura yang berasal dari Papua New Guinea, yang diseleksi menjadi Dura Deli 1 dan Dura Deli 2, kemudian di selfing untuk dijadikan bahan genetik dalam penelitian ini, (b) pertukaran global dari sumber tanaman yang digunakan dalam program pemuliaan yang dikembangkan oleh perusahaan-perusahaan dalam waktu lama, dan (c) sumber yang terbatas dari populasi yang dijelaskan oleh Bakoume
et al., 2007. Tingkat keragaman genetik yang rendah
ini konsisten dengan hasil yang dilaporkan oleh beberapa studi keragaman genetik yang melibatkan bahan genetik Dura Deli sebagai referensi Zaki et al., 2012).
106
Gambar 3. Dendogram keragaman genetik 60 sampel populasi tetua saudara kandung (sibs) Dura Deli berdasarkan marka mikrosatelit.
Figure 3. Dendogram of 60 siblings (sibs) Dura Deli population base on microsatellite marker.
KESIMPULAN
1. Aplikasi 27 primer mikrosatelit terhadap 2 populasi tetua Dura Deli menghasilkan 20 primer polimorfik dengan persentase lokus polimorfik 96,30% dan rata-rata nilai PIC 0,50.
2. Analisis keragaman genetik menggunakan analisis PCoA, struktur, maupun NTSys, me-misahkan populasi tetua Dura Deli menjadi dua kelompok besar sesuai asal usul tetuanya.
3. Keragaman genetik yang rendah di dalam tetua Dura Deli memungkinkan untuk digunakannya sebagai materi persilangan dalam perakitan varietas baru maupun produksi benih sawit selanjutnya.
UCAPAN TERIMA KASIH
Ucapan terima kasih disampaikan kepada PT. SMART Tbk yang telah menyediakan bahan tanaman dan biaya pelaksanaan penelitian ini, serta Ir. Ismail Maskromo, M.Si dan Roberdi, SP, M.Si yang telah membantu dalam penulisan artikel ini.
DAFTAR PUSTAKA
Agustina, L., R. Rivallan, Zulhermanna, Puspitaningrum, Y. Sudarsono, X. Perrier, D. Asmono, and N. Billotte. 2010. Allelic diversity of 22 Sampoerna Agro’s oil palm pisifera based on microsatellite markers. In Proc. Int. Oil Palm Conf, Indonesia.
Ajambang, W., Sudarsono, D. Asmono, and N. Toruan. 2012. Microsatellite markers reveal Cameroon’s wild oil palm population as a possible solution to broaden the genetic base in the Indonesia-Malaysia oil palm breeding programs. Afr. J. of Biotechnol., 11(69): 13244-13249.
Allou, D., B. Adon, and A. Sangare. 2008. Molecular variability from two selections of BRT10 population in an inbreeding program of oil palm (Elaeis guineensis Jacq.) in Cote d’Ivoire. Afr. J. Biotechnol., 7(20): 3550-3553.
Anderson, J.A., G.A. Churchill, J.E. Autrique, S.D. Tanksley, and M.E. Sorrells. 1993. Optimizing parental selection for genetic linkage maps. Genome 36 : 181 – 186.
107
Antonio A.F. Garcia, L. L. Benchimol, A.M.M.Barbosa, Isaias O. Geraldi, C. L.Souza Jr. and Anete P. de Souza. 2004. Comparison of RAPD, RFLP, AFLP and SSR markers for diversity studies in tropical maize inbred lines. Genetics and Molecular Biology. 27 (4) 579-588.
Arias, D., C. Montoya, L. Rey and H. Romero. 2012. Genetic similarity among commercial oil palm materials based on microsatellite markers. Agronomía Colombiana: 30(2), 188-195.
Asmono, D., A.R. Purba and K. Pamin. 1998. Current assesment of IOPRI oil palm improvement after the second generation of reciprocal recurrent selection. 1998. Int. Oil Palm Conf., 564-572. Bakoume, C., R. Wickneswari, N. Raijanaidu, A.
Kushairi, P. Amblard, and N. Billotte. 2007. Allelic diversity of natural oil palm (Elaeis
guineensis Jacq.) populations detected by
microsatellite markers. Implication in conservation. Rev. Palmas 28(1), 149-158.
Benbouza, H., Jacquemin J.M., J.P. Baudoin, and G. Mergeai. 2006. Optimization of a reliable, fast, cheap and sensitive silver staining method to detect SSR markers in polyacrylamide gels. Biotechnol. Agron. Soc. Environ.10(2): 77 – 81. Billotte, N., A.M. Risterucci, E. Barcelos, J.L. Noyer,
P. Amblard, and F.C. Baurens. 2001. Development, characterization, and across-taxa utility of oil palm (Elais guineensis Jacq.) microsatelitte markers. Genome 44: 413 – 425. Billotte, N., N. Marseillac, A.M. Risterucci, B. Adon, P.
Brottier, F.C. Baurens, R. Singh, A. Herrán, H. Asmadi, C. Billot, P. Amblard, T. Durrand-Gasselin, B. Courtois, D. Asmono, S.C. Cheah, W. Rohde, E, Ritter and A. Charrier. 2005. Microsatellite-based high density linkage map in oil palm (Elaeis guineensisJacq.). Theor. Appl. Genet. 110: 754-765.
Botstein, D., R.L. White, M. Skolnick, and R.W. Davis. 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. Am. J. Hum. Genet. 32:314– 331.
Collard, B.C.Y., C.M.V. Cruz, K.L. McNally, P.S. Vick and D.J. Mackill. 2008. Rice moleculer reeding laboratories in the genomics era: current and future considerations. Int. J. Of Plant Genom. Vol 2008 25p.
Dangi, R.K., M.D. Lagu, L.B. Choudhary, P.K. Ranjekar and V.S. Gupta. 2004. Assessment of genetic diversity in Trigonella foenum-graecum and Trigonella caerulea using ISSR nd RAPD markers. BMC Plant Biol. 4:13.
Direktorat Jenderal Perkebunan. 2010. Kebun koleksi nasional sumberdaya genetik (KKN SDG)
kelapa sawit, penyelamat plasma nutfah kelapa di Indonesia. http://ditjenbun. deptan.go.id/ bbp2tpsu. [diakses tanggal 26 Agustus 2012]. Direktorat Jenderal Perkebunan. 2012. Produksi, luas
areal dan produktivitas perkebunan di Indonesia.http://www.pertanian.go.id/Indika-tor/ tabel-3-prod-lsareal-prodvitas-bun.pdf. [diakses tanggal 12 Juni 2014].
Earl, Dent A. and vonHoldt, M. Bridgett. 2012. Structure Harvester: a website and program for visualizing Structure output and implementing Evanno method. Conserv. Genet. Res. Vol. 4(2) pp. 359-361.
Evanno, G., S. Regnaut and J. Goudet. 2005. Detecting the number of clusters of individuals using the software structure: a simulation study. Mol. Ecol. 14, 2611-2620.
Liu, K. and S.V. Muse. 2005. Power Marker: An integrated analysis environment for genetic marker analysis. Bioinformatics. 1:21(9):2128-9. Maizura, I., N. Rajanaidu, A. Zakri, and S. Cheah.
2006. Assessment of Genetic diversity in oil palm (Elaeis guineensis Jacq) using restriction fragment length polymorphism (RFLP). Genet. Resourc. Crop Evol. 53(1), 187-195.
Melchinger, A.E. 1993. Use of RFLP markers for analyses of genetic relationships among breeding materials and prediction of hybrid performance. P. 6211-628 dalam D.R. Buxton (ed.) Proc. Of the Int. Crop Sci. Congress 1st, Ames, 1A, July 1992. CSSA, Madison, WI. Missio, F.R, E.T. Caixeta, E.M Zambolim, L.
Zambolim, C.D. Cruz, and N.S. Sakiyama. Polymorphic information content of SSR markers for Coffeaspp. 2010. Crop Breeding and Applied Biotechnology 10: 89-94
Peakall, R. and P.E. Smouse. 2012. GenAlEx 6.5: Genetic Analysis in Excel. Population genetic software for teaching and research – an update. 28(19): 2537–2539. Downloaded from http://bioinformatics.oxfordjournals.org/ on November 18, 2013.
Pritchard, J.K., M. Stephens and P. Donnelly. 2000. Inference of population structure using multilocus genotype data. Genetics 155:945– 959.
Rohlf, F. 2000. NTSYS pc: Numerical taxonomy and multivariate system, ver. 2.1, computer program, Exeter Publishing, Setauket, New York, NY.
Singh, R., M.Z. Noorhariza, N.C. Ting, R. Rozana, S.G. Tan, L.E.T. Low, M. Ithnin, S.C. Cheah. 2009. Exploiting an oil palm EST the development of gene-derived and their exploitation for assess-ment of genetic diversity. Biologia. 63:1–9. Sugema. 2007. Strategi pengembangan industri hilir
kelapa sawit [terhubung berkala]. http:// www.indef.or.id.
108
Thongthawee, S., P. Tittinutchanon and H. Volkaert. 2010. Microsatellites for parentage analysis in an oil palm breeding population. Thai J. of Genet. 2010. 3(2): 172-181.
Wahyono, T. 2006. Kondisi terkini pasar global minyak sawit. PPKS, Medan.
Zaki, N.M., R. Singh, R. Rosli and I. Ismail. 2012. Elaeis oleifera Genomic-SSR Markers: Exploitation in Oil Palm Germplasm Diversity and Cross-mplification in Arecaceae. Int. J. Mol. Sci.13: 4069-4088.
Zulhermana, Sudarsono, D. Asmono, and Yulismawati. 2010. Intra and interpopulation genetic diversity of Oil Palm (Elaeis guineensis Jacq.) pisifera clones originated from Nigeria based on SSR markers analysis. In Proc. Int. Oil Palm Conf, Indonesia.