KARAKTERISASI MOLEKULER ENAM SUB POPULASI
KAMBING LOKAL INDONESIA BERDASARKAN ANALISIS
SEKUEN DAERAH D-LOOP DNA MITOKONDRIA
Pendahuluan
Analisis DNA mitokondria (mtDNA) merupakan salah satu metode yang banyak digunakan untuk mempelajari asal-usul ternak domestikasi (MacHugh & Bradley 2001). Pada mamalia DNA mitokondria hanya diturunkan lewat jalur induk (maternal) tanpa rekombinasi. Sekuen nukleotida genom mitokondria telah digunakan untuk mempelajari asal-usul sapi (Troy et al. 2001), babi (Giuffra et al. 2000), domba (Hiendleder et al. 2002), kuda (Villa et al. 2001), anjing (Savolainen et al. 2002), keledai (Beja-Pereira et al. 2004) dan kambing (Joshi et
al. 2004; Luikart et al. 2001; Mannen et al. 2001; Sultana et al. 2003; Chen et al.
2005; Naderi et al. 2007; Royoet al. 2009; Zhao et al. 2011).
Kambing domestik dapat dikelompokkan menjadi 4 garis kelompok keturunan utama atau Lineage (Joshi et al. 2004). Lineage A merupakan kelompok kambing domestikasi yang paling beragam dan luas penyebarannya di dunia. Lineage B menyebar di daerah Asia Timur dan Asia Selatan termasuk Cina, Mongolia, Afrika Selatan, Afrika Utara, Laos, Malaysia, Pakistan dan India. Lineage C menyebar di sekitar Mongolia, Swiss, Slovenia, Pakistan dan India. Lineage D di daerah Pakistan dan kambing lokal di India. Selain itu Naderi et al. (2007) mengelompokkan kambing menjadi enam haplogroup yaitu menambahkan haplogroup F dan G dengan 4 haplogroup yang dilaporkan oleh Joshi et al. (2004). Lineage F menyebar di daerah Sicilia dan lineage G menyebar di daerah Timur Tengah dan Afrika Utara. Kombinasi pengelompokan molekuler dengan temuan arkeologi menunjukkan bahwa kambing domestikasi mempunyai asal usul beberapa garis keturunan ibu (maternal origins) (Zeeder & Hesse 2000). Dengan penambahan sampel terutama di daerah yang mungkin merupakan asal garis keturunan, maka analisis mtDNA akan mendukung pemahaman pengelompokan kambing lokal di berbagai wilayah (MacHugh & Bradley 2001; Chen et al. 2005).
Bahan dan Metode
Sampel Darah Kambing Penelitian
Pengambilan sampel darah sekitar 2 ml dari setiap ekor kambing penelitian dilakukan pada bagian vena jugularis dengan menggunakan jarum
venoject yang disambungkan ke tabung vakum dengan EDTA 5 ml. Semua
sampel darah selama di lapangan dengan etanol absolut 2x volume darah dikocok hingga homogen.
Ekstraksi DNA
Ekstraksi DNA dilakukan dengan memodifikasi metode Sambrook et al. (1989) menggunakan bufer lisis sel 350 µl 1xSTE, dan 40 µl 10% SDS, 20 µl 5 mg/ml proteinase-K. DNA dimurnikan dengan metode fenol-kloroform, yaitu dengan menambahkan 40 µl 5 M NaCl dan 400 µl fenol dan kloroform: iso amil alkohol (24:1). DNA diendapkan dengan menambahkan 40 µl 5 M NaCl dan 800 µl etanol absolute. Endapan dicuci dengan menambahkan 400 µl etanol 70%, Selanjutnya sisa etanol dibuang dan diuapkan dengan menggunakan pompa vakum. DNA kemudian disuspensikan dengan 80 µl 80% bufer TE.
Amplifikasi DNA
Sebanyak 543 sampel DNA yang telah diekstraksi dipilih sebanyak 60 sampel yang masing-masing 10 sampel DNA per sub populasi. Amplifikasi ruas D-loop genom mitokondria menggunakan primer AF23 (forward) 5’GCGTACGCAAT CTTACGATCA-3’ dan AF22 (reverse) 5’ATGCAGTTAAGTCCAGCTAC-3’. Primer AF23 menempel pada basa ke 14979-14999 dan AF22 menempel pada basa ke 16379-16398 dari mtDNA
Capra hircus (GenBank no akses AF 533441). Pasangan primer AF23 dan AF22
mengapit ruas tengah hingga akhir Cyt b, tRNA Pro, tRNA Thr dan juga bagian awal hingga tengah daerah D-loop dengan panjang 1420 pb (Gambar 9).
Komposisi reaksi PCR dalam volume 25µl adalah sampel DNA 2µl (10-100ng), RBC Bioscience taq polymerasi 1.25 unit beserta sistim bufernya, dNTP mix 0.4 nmol, MgCl2 0.2 mM, primer AF22 dan AF23 masing-masing 1 nmol.
Kondisi PCR yang digunakan untuk proses amplifikasi adalah tahap denaturasi awal pada suhu 94oC selama 3 menit, tahap denaturasi pada suhu 94oC selama
45 detik, tahap penempelan primer (annealing) pada suhu 58oC selama 50 menit, dan tahap polimerasi (extension) pada suhu 72oC selama 1 menit yang diulang selama 30 siklus, kemudian reaksi PCR diakhiri dengan polimerasi akhir pada suhu 72oC selama 5 menit. Visualisasi produk PCR dilakukan menggunakan teknik elektroforesis gel poliakrilamid (PAGE) 6% dalam buffer 1xTBE (Tris-HCl 10 Mm, asam borat 1 M, EDTA 0.1 mM). Elektroforesis dijalankan pada kondisi 200 mV selama 50 menit. Proses dilanjutkan dengan pewarnaan sensitif perak (Tegelstrom 1986) yang dimodifikasi.
Perunutan DNA
Produk amplifikasi yang menunjukkan pita tunggal (sekitar 1400 pb) dimurnikan dan dijadikan cetakan dalam reaksi PCR untuk perunutan nukleotida. Masing-masing kelompok kambing dipilih 5 sampel yang saling berjauhan lokalitasnya. Primer yang digunakan dalam proses penentuan runutan nukleotida sama dengan primer yang digunakan untuk amplifikasi. Reaksi PCR tersebut dilakukan dengan menggunakan metode dideoxi terminator dengan dNTP berlabel (big dye terminator). Perunutan nukleotida menggunakan mesin ABI
Prism 3700-Avant Genetic Analyzer di PT Charoen Pockphan dan PT Genetika
Sains Indonesia Jakarta.
Analisis Data
Runutan nukleotida yang diperoleh kemudian diedit secara manual dengan bantuan program Bio Edit versi 6.0.7. Urutan DNA yang telah diedit disejajarkan dengan beberapa runutan DNA dari kelompok Capra hircus yang dipublikasikan dalam GenBank (http://ncbi.nlm.nih.gov). Proses pensejajaran menggunakan ClustalW versi 8.1 yang ada dalam program MEGA 4 (Tamura et
al. 2007) yang kemudian diedit lagi secara manual terutama untuk ruas-ruas
DNA berulang (Lampiran 4).
Analisis yang dilakukan meliputi penghitungan komposisi nukleotida, laju subsitusi, jarak genetik menggunakan program MEGA versi 4.0 (Tamura et al. 2007). Perhitungan nilai jarak genetik dilakukan berdasarkan model subsitusi Kimura 2 parameter (K2P). Rekonstruksi pohon filogeni dilakukan berdasarkan semua nukleotida yang bersifat parsimoni menggunakan metode Neigbour
Joining (NJ) dengan uji bootstrap 1000 kali dengan program NETWORK versi 4.6
(Fluxus TechnologyLtd. 2010).
Hasil dan Pembahasan
Polimorfisme Segmen daerah D-loop DNA Mitokondria
Panjang ruas D-loop yang saling sejajar antar sampel adalah 879 pb. Dari situs-situs ruas D-loop tersebut ditemukan 50 situs polimorfik yang terdiri dari 21 mutasi insersi dan 29 transversi (Gambar 10). Berdasarkan 50 situs nukleotida yang bersifat polimorfik ke-6 sub populasi kambing lokal bisa dibagi menjadi 19 haplotip. Dari 19 jumlah haplotip, masing-masing sub populasi kambing lokal mempunyai jumlah haplotip khas yang bervariasi antara 2-4 haplotip. Variasi haplotip yang khas tersebut dapat digunakan sebagai penciri genetik pada setiap sub populasi kambing lokal Indonesia (Tabel 11).
Tabel 11 Jumlah haplotip berdasarkan runutan nukleotida D-loop mtDNA setiap sub populasi kambing lokal Indonesia
Kambing lokal n sample n haplotipe Haplotipe
Marica (M) 5 3 1, 2, 3 Kacang (K) 5 4 4, 5, 6, 7 Samosir (S) 5 2 8, 9 Benggala (B) 5 3 10, 11, 12 Jawarandu (J) 5 4 13, 14, 15, 16 Muara (R) 5 3 17, 18, 19
Gambar 9 Struktur genom mitokondria yang diapit oleh primer AF23 dan AF22. Nomor mengacu pada Capra hircus (GenBank no. akses AF 533441)
14.153 14.981 15.431 16.643pb 16.309 CYTB Thr Pro t-RNA
D-loop Kambing lokal (879 pb)
Panjang D-loop (1.212pb)
15.296 15.431 15.365
Nukleotida DNA mt kambing lokal (1.420 pb)
15.292
AF23 AF22
Gambar 10 Polimorfisme runutan nukleotida daerah D-loop DNA mitokondria pada 6 sub populasi kambing lokal mengacu pada Capra hircus (GenBank no. akses AF 533441) (tiga baris pertama dibaca secara vertikal merupakan posisi nukleotida)
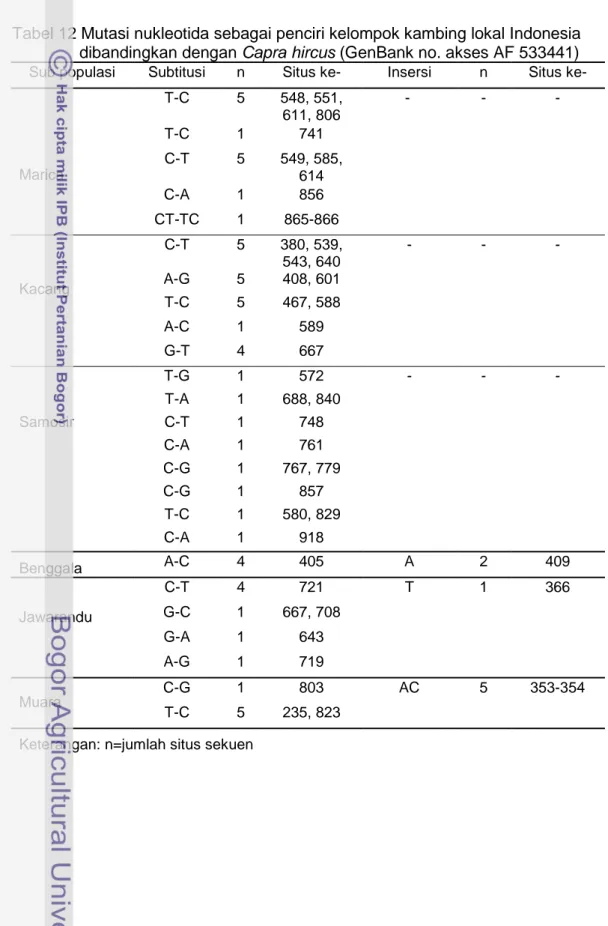
Setiap kelompok kambing juga ditemukan perbedaan antara satu kelompok sub populasi kambing lokal dengan kelompok yang lain, sehingga runutan nukleotida yang khas dapat digunakan sebagai penciri dari setiap sub populasi kambing lokal tersebut (Tabel 12). Perubahan (mutasi) susunan nukleotida berupa subtitusi ditemukan pada situs nukleotida Kambing Kacang, Marica, dan Kambing Muara, sedangkan pada Kambing Benggala, Jawarandu dan Kambing Muara dijumpai mutasi berupa subtitusi dan insersi. Mutasi insersi nukleotida yang ditemukan pada 3 sub populasi yaitu insersi nukleotida AC pada 2333344444 4445555555 5555555555 566666666 6777777788 888888 3556800014 6890223444 5556788888 901144456 8014466700 224566 5346058977 7744459389 0166204589 311403977 8891817936 390656 C.hircus_NC_005044 T---CAA-AA TAACCCCCTC CTAGTTCCTA AATCCGGCG TGATCCCCCT TTCCCT Marica1 .---...-.G .G..T-..CT TCGA..TT.. ..CT... ...C ... Marica2 .---...-.G .G..T-..CT TCGA..TT.. ..CT... ...C ... Marica3 .---...-.G .G..T-..CT TCGA..TT.. ..CT... ...C ...A.. Marica4 .---...-.G .G..T-..CT TCGA..TT.. ..CT... ...C...C ....TC Marica5 .---...-.G .G..T-..CT TCGA..TT.. ..CT... ...C ... Kacang1 .---T.G-GG CGGTT.TT.. ..GA..T... GG..T..T. ... ... Kacang2 .---T.G-GG CGGTT.TT.. ..GA..T.CC ... ... ... Kacang3 .---T.G-GG CGGTT.TT.. ..GA..T... G...T. ... ... Kacang4 .---T.G-GG CGGTT.TT.. ..GA..T... G...T. ... ... Kacang5 .---T.G-GG CGGTT.TT.. ..GA..T... GG...T. ... ... Samosir1 .---...-.G .G..T-.... ....GCT... ... A...TAGG.. .CA... Samosir2 .---...-.G .G..T-.... ...T... ... ... ... Samosir3 .---...-.G .G..T-.... ...T... ... ... ... Samosir4 .---...-.G .G..T-.... ...T... ... ... ... Samosir5 .---...-.G .G..T-.... ...T... ... ... ... Benggala1 .---.C.-.. ... ... G... ... ... Benggala2 .---.C.A.. ... ... G... ... ... Benggala3 .---...-.. ... ... G... ... ... Benggala4 .---.C.A.. ... ... G... ... ... Benggala5 .---.C.-.. ... ... G... ... ... Jawarandu1 .---...-.. ... ... ...T.A... ... ... Jawarandu2 .--T...-.. .G... ... ... ... ... Jawarandu3 .---...-.. .G... ... ...A... ... ... Jawarandu4 .---...-.. .G... ... ...A... ... ... Jawarandu5 .---...-.. .G... ... ...AA.C .CG... ... Muara1 .AC-...-.. ... ... ... ... C... Muara2 .AC-...-.. ... ... ... ...G. C... Muara3 .AC-...-.. ... ... ... ... C... Muara4 CAC-...-.. ... ... ... ... C... Muara5 CAC-...-.. ... ... ... ... C...
situs ke 353, 354 pada Kambing Muara, insersi nukleotida T pada situs ke 366 pada Kambing Jawarandu dan insersi A pada situs ke 409 pada Kambing Benggala, sedangkan mutasi nukleotida pada 3 sub polpulasi lainnya merupakan mutasi substitusi.
Tabel 12 Mutasi nukleotida sebagai penciri kelompok kambing lokal Indonesia dibandingkan dengan Capra hircus (GenBank no. akses AF 533441)
Sub populasi Subtitusi n Situs ke- Insersi n Situs ke-
Marica T-C 5 548, 551, 611, 806 - - - T-C 1 741 C-T 5 549, 585, 614 C-A 1 856 CT-TC 1 865-866 Kacang C-T 5 380, 539, 543, 640 - - - A-G 5 408, 601 T-C 5 467, 588 A-C 1 589 G-T 4 667 Samosir T-G 1 572 - - - T-A 1 688, 840 C-T 1 748 C-A 1 761 C-G 1 767, 779 C-G 1 857 T-C 1 580, 829 C-A 1 918 Benggala A-C 4 405 A 2 409 Jawarandu C-T 4 721 T 1 366 G-C 1 667, 708 G-A 1 643 A-G 1 719 Muara C-G 1 803 AC 5 353-354 T-C 5 235, 823
Keragaman Runutan Nukleotida
Urutan frekuensi nukleotida paling tinggi terdapat pada nukleotida A(33.7), kemudian T (27.2%), C (26.4) dan G (12.6) secara berurutan. Perbandingan rataan frekuensi A dan T (60.9%) lebih tinggi dibandingkan C dan G (39.8%). Perbedaan susunan basa nukleotida paling rendah ditemukan antara Kambing Kacang dengan C. hircus AF 533441 (0.5%), dan tertinggi dijumpai antara Kambing Kacang dengan Muara (10.30%). Rataan keragaman susunan nukleotida secara keseluruhan adalah 0.014±0.002. Keragaman susunan nukleotida antar individu dalam kelompok kambing penelitian yaitu; Marica (0.001), Kacang (0.005), Samosir (0.008), Jawarandu (0.004), Muara (0.001) dan Kambing Benggala tidak terdapat perbedaan antar individu dalam kelompok (0.000). Perbedaan keragaman susunan nukleotida antar kelompok kambing lokal berkisar antara 0.004 – 0.103. Keragaman susunan nukleotida paling tinggi adalah antara Kambing Kacang dengan Muara (0.1030) dan paling rendah dijumpai antara Kambing Samosir dengan Marica (0.004) dan nilai yang sama dijumpai antara Kambing Jawarandu dan Kambing Muara (Tabel 13).
Tabel 13 Keragaman nukleotida D-loop mtDNA pada 6 kambing lokal Indonesia
Genotip C. hircus Marica Kacang Samosir Benggala J.randu Muara
Capra hircus* 0 Marica (M) 0.022 0 Kacang (K) 0.005 0.041 0 Samosir (S) 0.025 0.004 0.043 0 Benggala (B) 0.049 0.008 0.072 0.013 0 Jawarandu (J) 0.061 0.011 0.089 0.016 0.005 0 Muara (R) 0.073 0.016 0.103 0.021 0.004 0.004 0
*GenBank no. akses AF 533441
Jarak Genetik Kambing Penelitian dengan Kambing Lainnya
Jarak genetik digunakan untuk melihat kedekatan hubungan genetik ternak yang dipelihara di daerah yang berbeda. Selanjutnya informasi jarak genetik dapat digunakan sebagai petunjuk awal dari stuktur populasi dan diferensiasi suatu rumpun di dalam membuat keputusan program konservasi (Ponzoni 1997).Jarak genetik antar individu dalam kelompok kambing penelitian antara lain; nilai jarak genetik Kambing Samosir 0.004, Kacang dan Jawarandu 0.003,
Marica 0.002, Muara 0.001 dan Kambing Benggala tidak ada perbedaan (0.000). Nilai jarak genetik antar individu dan sub populasi ini menunjukkan sampel Kambing Benggala relatif homogen walaupun diambil dari tempat berbeda kabupaten.
Nilai jarak genetik antar kelompok kambing lokal berkisar 0.004-0.044 dan rataan jarak genetik keseluruhan adalah 0.014. Jarak genetik tertinggi antara kambing lokal dengan pembanding Capra hircus (AF 533441) ditemukan pada Kambing Muara (0.044) dan terendah dengan Kambing Kacang (0.028).
Nilai jarak genetik antar kelompok kambing lokal terkecil antara Kambing Muara dan Benggala (0.004), sedangkan tertinggi merupakan jarak genetik antara Kambing Kacang dan Marica (0.004) (Tabel 16). Jarak genetik Kambing Marica paling dekat dengan Kambing Samosir (0.015) dan paling jauh dengan Kambing Kacang (0.023). Jarak genetik Kambing Kacang paling dekat hubungannya dengan Kambing Samosir (0.017) dan paling jauh hubungannya dengan Kambing Jawarandu dan Muara (0.021). Jarak genetik Kambing Samosir paling dekat hubungannya dengan Kambing Jawarandu (0.008) dan paling jauh hubungannya dengan Kambing Muara dan Benggala (0.009). Jarak genetik Kambing Benggala lebih dekat hubungannya dengan Kambing Muara (0.004) dibandingkan dengan Kambing Jawarandu (0.005). Jarak Kambing Jawarandu dengan Kambing Muara adalah 0.005 (Tabel 14). Hal ini menunjukkan bahwa Kambing Jawarandu masih dekat hubungan kekerabatan dengan Kambing Muara.
Pohon filogeni yang dibentuk berdasarkan metode 2 parameter Kimura dalam uji bootstrap 1000 kali pengulangan, diperoleh enam klaster kambing yaitu masing-masing sub populasi membentuk klaster tersendiri yaitu klaster Kacang, Marica, Samosir, Jawarandu, Muara dan Benggala. Kambing Kacang merupakan klaster yang relatif dekat dengan Capra hircus dengan nilai uji
bootstrap (65%). Kambing Kacang dikenal sebagai kambing asli Indonesia yang
tersebar secara luas hampir di seluruh kepulauan yang ada penduduknya.
Kambing Jawarandu, Muara dan Benggala menunjukkan terjadi mutasi perubahan susunan basa nukleotida dalam bentuk subtitusi dan insersi (Tabel 14). Perubahan susunan nukleotida dalam bentuk insersi pada ketiga kambing ini diduga karena merupakan hasil persilangan pejantan kambing dari luar (outgroup) dengan induk Kambing Kacang (hibridisasi) yang telah
beradaptasi (berevolusi) dengan kondisi agro-ekosistim lokal dimana kambing tersebut berada.
Tabel 14 Jarak genetik berdasarkan runutan nukleotida pada 6 sub populasi kambing lokal Indonesia
Sub populasi C. hircus Marica Kacang Samosir Benggala J.randu Muara
Capra hircus* 0 Marica (M) 0.03 0 Kacang (K) 0.028 0.023 0 Samosir (S) 0.041 0.015 0.017 0 Benggala (B) 0.041 0.019 0.02 0.009 0 Jawarandu (J) 0.042 0.018 0.021 0.008 0.005 0 Muara (R) 0.044 0.019 0.021 0.009 0.004 0.005 0
*GenBank no. akses AF 533441
Mutasi subtitusi ditemukan pada Kambing Kacang, Marica dan Samosir diduga merupakan tanda adanya proses adaptasi dari Kambing Kacang (sebagai kambing asli) dengan kondisi lokasi baru yang berbeda dengan kondisi di Sumatera, Jawa dan Bali, dimana perubahan susunan basa nukleotida terjadi dalam bentuk substitusi sebagai akibat proses adaptasi terhadap kondisi lingkungan yang sumber pakannya terbatas dan diduga akibat adanya seleksi yang berhubungan dengan tujuan produksi yang diinginkan oleh peternak.
Seperti Kambing Samosir, tujuan utama pemeliharaan kambing ditujukan untuk menghasilkan kambing yang berwarna putih, karena nilai ekonomi (harga jual) yang tinggi. Pengguna atau konsumen menggunakan kambing berwarna putih untuk keperluan acara ritual bagi penganut agama/aliran kepercayaan Parmalim di daerah Gunung Pusuk Buhit dan sekitar Kabupaten Samosir.
Perubahan mutasi nukleotida pada Kambing Marica diduga disebabkan proses adaptasi dengan kondisi iklim yang berbeda dan ketersediaan bahan pakan terutama pada saat musim kemarau yang rata-rata diatas 6-9 bulan per tahun membuat ketersediaan rumput sangat terbatas. Pada musim pertengahan dan akhir musim kemarau umumnya rumput sudah layu dan kering diakibatkan musim kering yang berkepanjangan seperti pada umumnya di daerah kepulauan Indonesia Bagian Timur, sehingga ketersediaan rumput sangat terbatas. Kemungkinan dalam jangka waktu yang lama Kambing Marica mengalami proses adaptasi dengan kondisi setempat, maka terjadilah proses mutasi
subtitusi nukleotida yang secara fenotip Kambing Marica mempunyai performans tubuh yang lebih kecil jika dibandingkan dengan Kambing Kacang.
Jika digabungkan dengan sekuen nukleotida daerah D-loop DNA mitokondria kambing dari luar negeri yang terdapat pada GenBank, kelompok kambing lokal yang diamati mempunyai jarak genetik yang jauh dari Capra hircus (AF 533441) karena sampel kambing tersebut berasal dari Eropa (Italia) (Parma
et al. 2003) dengan nilai uji bootstrap sangat tinggi (99%) (Gambar 11).
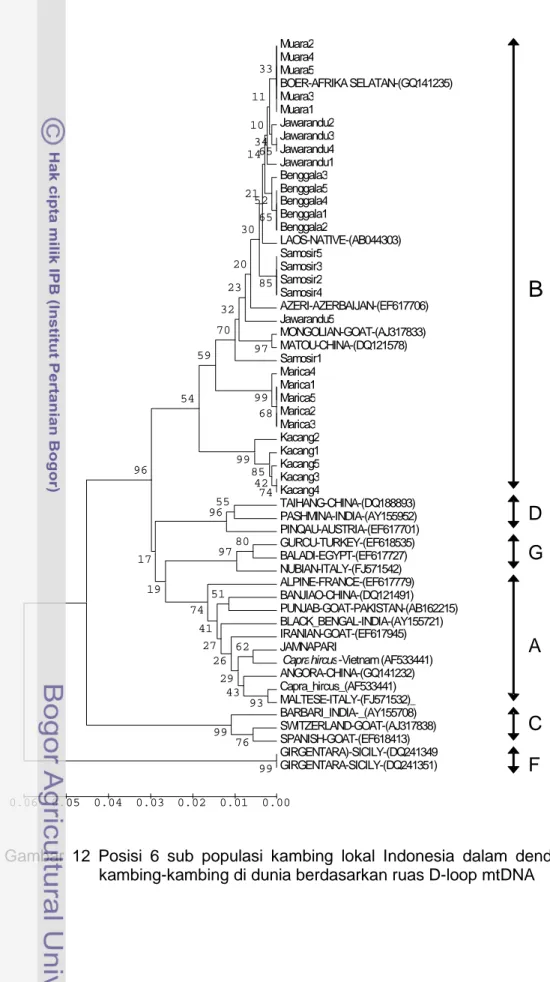
Sekuen DNA daerah D-loop kambing lokal yang diteliti, jika disejajarkan dan dibandingkan dengan 26 sekuen DNA yang berasosiasi dari GenBank yang mewakili 6 kelompok utama (haplogroup) kambing di dunia berdasarkan asal-usulnya secara maternal (Naderi et al. 2007; Joshi et al. 2004) menunjukkan bahwa keenam sub populasi kambing lokal Indonesia termasuk kedalam kelompok haplotip Lineage B (Gambar 12). Kambing yang termasuk dalam haplogroup B adalah kambing yang berdasarkan garis keturunan ibu (maternal) dan telah dilaporkan menyebar di daerah Asia Timur dan Asia Selatan termasuk Cina, Mongolia, Afrika Selatan, Afrika Utara, Laos, Malaysia, Pakistan dan India.
Dari Gambar 12 terdapat nilai uji bootstrap dengan pengulangan 1000 kali pada Kambing Boer sangat rendah (30%) pada Kambing Boer dan (38-65%) Kambing Jawarandu menunjukkan bahwa kedua jenis kambing tersebut masih dekat kekerabatannya, karena Kambing Boer adalah merupakan persilangan Kambing Jamnapari dengan kambing lokal di Afrika Selatan.
Nilai uji bootstrap Kambing Kacang dan Marica merupakan paling tinggi (99%) kemudian disusul oleh Kambing Jawarandu (92%), Muara (78%), Samosir (66%) dan Benggala (64-65%). Hasil uji bootstrap 1000 kali ulangan pada
analisis Neighbour Joining dengan metode 2 parameter Kimura menunjukkan
bahwa keenam sub populasi kambing lokal yang diteliti terbagi kedalam 6 kelompok atau bisa dikelompokkan menjadi 6 rumpun yang berbeda yaitu rumpun kambing pertama Kambing Kacang, kedua Kambing Marica, ketiga Kambing Samosir, keempat Kambing Jawarandu, kelima Kambing Muara dan keenam Kambing Benggala (Gambar 11) dengan nilai uji bootstrap di atas 60 % antara satu kelompok sub populasi kambing lokal yang satu dengan yang lainnya.
Hampir sama dengan nilai uji bootstrap Kambing Muara juga rendah (46%), hal ini juga memperkuat dugaan bahwa Kambing Muara juga merupakan hasil persilangan kambing lokal dengan Peranakan Etawah (PE) di Indonesia,
saat ini Kambing Muara sudah mempunyai karakteristik morfologi tersendiri dan susunan basa nukleotida mempunyai kekhasan tersendiri.
Gambar 11 Dendogram 6 sub populasi kambing lokal Indonesia berdasarkan ruas D-loop mtDNA (bootstrap 1000x)
Kambing Muara bentuk ukuran tubuh hampir sama dengan PE, tetapi lebar dada relatif lebih panjang dan panjang telinga lebih pendek dan pola warna lebih bervariasi. Kambing Jawarandu bentuk tubuh lebih kecil, telinga pendek
Muara Benggala Jawarandu Samosir Marica Kacang Muara4 Muara5 Muara2 Muara3 Muara1 Benggala3 Benggala5 Benggala2 Benggala1 Benggala4 Jawarandu1 Jawarandu3 Jawarandu4 Jawarandu5 Jawarandu2 Samosir2 Samosir3 Samosir4 Samosir1 Samosir5 Marica1 Marica2 Marica5 Marica3 Marica4 Kacang2 Kacang1 Kacang5 Kacang3 Kacang4 C.hircus NC_0050441 71 49 72 63 99 68 67 99 65 63 67 64 93 78 0.000 0.005 0.010 0.015 AF 533441
dan warna bulu tubuh relatip lebih bervariasi jika dibandingkan dengan Kambing Etawah dan Kambing Peranakan Etawah.
Gambar 12 Posisi 6 sub populasi kambing lokal Indonesia dalam dendogram kambing-kambing di dunia berdasarkan ruas D-loop mtDNA
Muara2 Muara4 Muara5 BOER-AFRIKA SELATAN-(GQ141235) Muara3 Muara1 Jawarandu2 Jawarandu3 Jawarandu4 Jawarandu1 Benggala3 Benggala5 Benggala4 Benggala1 Benggala2 LAOS-NATIVE-(AB044303) Samosir5 Samosir3 Samosir2 Samosir4 AZERI-AZERBAIJAN-(EF617706) Jawarandu5 MONGOLIAN-GOAT-(AJ317833) MATOU-CHINA-(DQ121578) Samosir1 Marica4 Marica1 Marica5 Marica2 Marica3 Kacang2 Kacang1 Kacang5 Kacang3 Kacang4 TAIHANG-CHINA-(DQ188893) PASHMINA-INDIA-(AY155952) PINQAU-AUSTRIA-(EF617701) GURCU-TURKEY-(EF618535) BALADI-EGYPT-(EF617727) NUBIAN-ITALY-(FJ571542) ALPINE-FRANCE-(EF617779) BANJIAO-CHINA-(DQ121491) PUNJAB-GOAT-PAKISTAN-(AB162215) BLACK_BENGAL-INDIA-(AY155721) IRANIAN-GOAT-(EF617945) JAMNAPARI ANGORA-CHINA-(GQ141232) Capra_hircus_(AF533441) MALTESE-ITALY-(FJ571532)_ BARBARI_INDIA-_(AY155708) SWITZERLAND-GOAT-(AJ317838) SPANISH-GOAT-(EF618413) GIRGENTARA)-SICILY-(DQ241349 GIRGENTARA-SICILY-(DQ241351) 65 74 85 65 99 68 33 52 34 42 99 97 85 11 93 10 14 21 30 99 76 62 80 20 23 43 32 29 97 70 55 99 26 51 96 27 41 59 74 54 19 17 96 0.00 0.01 0.02 0.03 0.04 0.05 0.06
B
D
G
A
C
F
Kambing Benggala mempunyai nilai bootstrap cukup jauh (65%) dari Kambing Kacang. Berdasarkan performans karakteristik tubuh dan warna bulu Kambing Benggala diduga merupakan persilangan Kambing Black Bengal dengan kambing lokal yang telah beradaptasi dengan kondisi agro-ekosistim di Pulau Timor dan Pulau Flores (Propinsi Nusa Tenggara Timur). Kambing Jawarandu, Muara dan Benggala merupakan persilangan kambing dari luar dengan kambing lokal, yang kemudian terjadi proses adaptasi dengan kondisi agro-ekosistim lokal sehingga penampilan produksi dan karakteristik kambing yang diteliti telah berubah jika dibandingkan dengan karakteristik rumpun asal kambing itu sendiri. Seperti Kambing Benggala jika dibandingkan dengan Kambing Black Bengal tubuh Kambing Benggala relatip lebih kecil, tetapi bentuk telinga dan warna bulu sama-sama warna hitam dan coklat tua pada umumnya.
Berdasarkan analisis Median joining network dari 30 sekuen nukleotida daerah D-loop kambing yang diamati terdapat 50 situs yang bersifat polimorfik, terdapat sebanyak 19 haplotipe runutan DNA yang unik. Aliran gen berasal dari Kambing Kacang sebagai kambing asli di Indonesia, yang kemudian mengalami adaptasi sesuai dengan kondisi agro-ekosistem dan perlakuan manajemen dan terjadi perubahan susunan nukleotida dan perubahan fenotipik yang dikenal dengan Kambing Marica di daerah Sulawesi Selatan dan Kambing Samosir di daerah Kabupaten Samosir. Mutasi susunan nukleotida paling banyak di jumpai antara Kambing Kacang dengan Kambing Marica dan Samosir (Gambar 13).
Perubahan mutasi ini terjadi diduga akibat adanya proses adaptasi dari Kambing Kacang sebagai kambing asli Indonesia dengan kondisi lingkungan yang sangat berbeda seperti lamanya musim kering yang tinggi di daerah Sulawesi Selatan yang mengakibatkan pakan hijauan layu dan mengering sehingga sangat terbatas jumlahnya di ujung musim kemarau.
Hal ini bisa dilihat dari perubahan (mutasi) susunan nukleotida dalam bentuk subtitusi pada situs–situs tertentu (Tabel 14). Pada Kambing Jawarandu, Benggala dan Muara ditemukan mutasi subtitusi dan insersi nukleotida diduga akibat proses adaptasi lingkungan dan merupakan hasil persilangan (proses hibridisasi) dengan rumpun kambing di luar kambing lokal yang terdapat di daerah setempat.
Gambar 13 Median joining network dari 19 haplotip nukleotida daerah D-loop DNA mitokondria pada 6 sub populasi kambing lokal Indonesia
Simpulan
Keragaman genetik 6 kambing lokal Indonesia yang diamati adalah 0.014±0.002 dan ditemukan 50 situs susunan nukleotida yang polimorfik dan terdiri dari 19 haplotip yang unik. Keenam sub populasi kambing lokal Indonesia menunjukkan keragaman susunan nukleotida yang berbeda antara setiap kelompok dengan kelompok sub populasi kambing lainnya. Perbedaan susunan nukleotida yang khas pada setiap rumpun kambing dapat dipakai sebagai penciri DNA antar rumpun kambing. Berdasarkan analisis keragaman genetik pada sekuen kambing penelitian dan sekuen nukleotida dari GenBank diduga asal usul tetua secara maternal kambing lokal yang diamati termasuk kedalam kelompok utama (haplogroup) lineage B.
KARAKTERISASI MOLEKULER PADA ENAM SUB
POPULASI KAMBING LOKAL INDONESIA BERDASARKAN
ANALISIS DNA KROMOSOM Y
Pendahuluan
Variasi genetik dari rumpun ternak lokal sangat penting untuk mempertahankan sumberdaya genetik yang tidak tergantikan dan juga bermanfaat untuk membentuk bibit ternak yang baru. Gen SRY terletak pada kromosom Y yang terdiri dari ekson tunggal. Gen ini bertanggungjawab pada penentuan jenis kelamin pada ternak mamalia (Sinclair et al. 1990, Prashant et
al. 2008).
Sampai saat ini hasil penelitian tentang keragaman genetik kromosom Y pada kambing lokal Indonesia masih sangat terbatas. Tujuan penelitian ini adalah untuk mempelajari keragaman genetik Kromosom Y bagian gen SRY dan hubungan genetik antar sub populasi menurut garis keturunan secara paternal pada 6 sub-populasi kambing lokal Indonesia.
Bahan dan Metode
Sampel Darah Kambing Penelitian
Sebanyak 18 sampel dipilih dari 77 sampel darah kambing jantan yang dikoleksi yang terdiri masing-masing 3 sampel per kelompok 6 sub populasi kambing.
Ekstraksi DNA
Perlakuan sama seperti yang dilakukan pada Ekstraksi DNA untuk analisis mitokondria.
Amplifikasi DNA
Amplifikasi genom mitokondria menggunakan primer AF126 (forward) 5’CCAGATCGATGTAGAGACAT-3’ dan AF127 (reverse) 5’TGCAATTTA CAAAGAGGTGGAA-3’. Primer AF126 menempel pada basa ke 3 925 - 3 947 nt dan AF127 menempel pada basa ke 4 675 - 4 696 nt dari runutan DNA kromosom Y pada sekuen lengkap Capra hircus dengan no akses GenBank EU
581862. Komposisi reaksi PCR dalam volume 25µl adalah sampel DNA 2µl (10-100ng), RBC Bioscience taq polymerase 1.25 unit beserta sistim bufernya, dNTP 0.4 nmol, MgCl2 0.2 mM, primer AF22 dan AF23 masing-masing 1 nmol.
Kondisi PCR yang digunakan untuk proses amplifikasi adalah tahap denaturasi awal pada suhu 94oC selama 3 menit, tahap denaturasi pada suhu 94oC selama 45 detik, tahap penempelan primer (annealing) pada suhu 62oC selama 50 menit, dan tahap polimerasi (extension) pada suhu 72oC selama 1 menit yang diulang selama 30 siklus, kemudian reaksi PCR diakhiri dengan polimerasi akhir pada suhu 72oC selama 5 menit. Visualisasi produk PCR dilakukan menggunakan tehnik elektroforesis gel poliakrilamid (PAGE) 6% dalam bufer 1xTBE (Tris-HCl 10 Mm, asam borat 1 M, EDTA 0.1 mM). Elektroforesis dijalankan pada kondisi 200 mV selama 50 menit, kemudian proses dilanjutkan dengan pewarnaan sensitive perak (Tegelstrom 1986) yang di modifikasi.
Perunutan DNA
Produk amplifikasi yang menunjukkan pita tunggal kemudian dimurnikan dan dijadikan cetakan dalam reaksi PCR. Primer yang digunakan dalam proses PCR untuk perunutan nukleotida sama dengan primer yang digunakan untuk amplifikasi. Reaksi PCR dilakukan dengan menggunakan metode dideoxi
terminator dengan dNTP berlabel (big dye terminator). Perunutan nukleotida
menggunakan mesin ABI Prism 3700-Avant Genetic Analyzer.
Analisis Data
Runutan nukleotida yang diperoleh kemudian diedit dengan menggunakan program Bio Edit versi 6.0.7. Urutan DNA yang telah diedit disejajarkan dengan beberapa runutan DNA dari kelompok Capra hircus yang dipublikasikan dalam GenBank (http://ncbi.nlm.nih.gov). Data yang diambil sebagai pembanding adalah gen SRY kromosom Y Capra hircus dengan no.akses EU 581862. Proses pensejajaran menggunakan ClustalW versi 8.1 yang tertanam dalam program MEGA 4 (Tamura et al. 2007) yang kemudian diedit lagi secara manual. Pengeditan hasil pensejajaran dilakukan dengan unit-unit ruas DNA berulang.
Analisis yang dilakukan meliputi penghitungan komposisi nukleotida, laju subsitusi, jarak genetik berdasarkan ruas DNA kromosom Y pada segmen SRY. Proses analisis dilakukan dengan menggunakan program MEGA versi 4 (Tamura
et al. 2007). Perhitungan nilai jarak genetik dilakukan berdasarkan model
subsitusi Kimura 2 parameter (K2P). Rekonstruksi pohon filogeni dilakukan berdasarkan ruas daerah gen SRY untuk semua nukleotida yang bersifat parsimoni. Rekonstruksi pohon filogeni keduanya dilakukan menggunakan metode Neigbour Joining (NJ) dengan bootstrap 1000 kali. Untuk mengetahui jumlah haplotip dan penyebaran mutasi nukleotida digunakan analisis
Median-Joining Network versi 4.6 (Fluxus Technology Ltd., 2005).
Hasil dan Pembahasan
Polimorfisme DNA Kromosom Y
Amplifikasi gen SRY pada daerah Kromosom Y menggunakan pasangan primer AF126 dan AF127 yang didesain sendiri berdasarkan sekuen komplit gen SRY pada Capra hircus dari GenBank dengan nomor akses EU 581862. Dari hasil analisis sekuensing setelah diedit secara manual menggunakan program Bio Edit dan kemudian digabungkan antara hasil sekuen forward dengan hasil
sekuen reverse maka diperoleh hasil keseluruhan sekuen nukleotida sepanjang
773 pb dengan letak posisi sekuen diperkirakan antara situs ke 3 925 – 4 697 pb (Gambar 14).
Setelah saling disejajarkan antara hasil sekuen nukleotida dari keenam sub populasi kambing lokal yang diamati maka ditemukan 6 situs polimorfik (variable site) yang terdiri atas 1 singleton variable site yang ditandai dengan adanya insersi nukleotida T pada sub populasi Kambing Benggala, dan 5
parsimony informative sites yang ditandai dengan adanya mutasi substitusi pada
Gambar 14 Struktur ruas gen SRY yang diapit oleh primer AF126 dan AF127 pada 6 sub populasi kambing lokal Indonesia
Jika dibandingkan dengan sekuen nukleotida gen SRY pada Capra hircus (EU 581862) dari GenBank disini dapat dilihat terdapat mutasi yang sama pada keenam sub populasi kambing lokal Indonesia yaitu perubahan mutasi nukleotida dari G berubah menjadi A pada situs ke 398 pada penelitian ini. Runutan sekuen nukleotida yang bersifat polimorfisme pada Kromosom Y keenam sub populasi kambing lokal dibandingkan dengan Capra hircus (EU 581862) dapat dilihat pada Gambar 15.
Terdapat 4 haplotipe berdasarkan 6 situs nukleotida yang bersifat polimorfik, dimana situs polimorfisme Kambing Kacang sama dengan Kambing Jawarandu, situs polimorfisme Kambing Marica sama dengan Kambing Samosir, sedangkan Kambing Muara dan Benggala berbeda satu sama lain. Runutan basa-basa nukleotida yang polimorfik terletak pada situs ke 330 - 754 berupa subtitusi dan insersi.
Berdasarkan runutan nukleotida yang disejajarkan ditemukan substitusi nukleotida khas yang bisa digunakan sebagai penciri 6 kambing lokal jika dibandingkan dengan Capra hircus (EU 581862), akan tetapi Kambing Shiba dari Jepang sama dengan situs nukleotida C. hircus. Perubahan susunan (mutasi) nukleotida berupa subtitusi ditemukan pada situs nukleotida Kambing Kacang dan Jawarandu di situs ke 398 (G-A), Marica dan Samosir di situs ke 330 (A-T), 398 (G-A), 701 (T-C), Muara di situs 330 (A-T), 398(G-A), 701 (T-C), 718 (A-G), 754 (C-T) dan Benggala di situs 330 (A-T), 1398(G-A), 701(T-C), 718 (A-G), 754 (C-T). Mutasi insersi nukleotida T hanya ditemukan di situs ke 369 pada Kambing Benggala.
Gambar 15 Polimorfisme nukleotida gen SRY pada 6 sub populasi kambing lokal Indonesia (tiga baris pertama dibaca secara vertikal merupakan posisi nukleotida yang mengacu pada C. hircus GenBank no. akses EU581862 dan Shiba Goat no. akses D82963
Frekuensi Nukleotida dan Jarak Genetik
Berdasarkan hasil sekuen DNA gen SRY analisis jarak genetik antara 6 sub populasi kambing lokal yang diamati dibandingkan dengan sekuen nukleotida pada Capra hircus (EU 581862) dari GenBank, maka jarak genetik paling tinggi adalah pada Kambing Muara (0.001), kemudian diikuti Kambing Benggala (0.008), Kambing Marica sama dengan Kambing Samosir (0.006), dan paling rendah terdapat pada Kambing Kacang sama dengan Kambing Jawarandu (0.002).
Perbandingan jarak genetik antar sub populasi kambing lokal ditemukan bahwa tidak ada jarak perbedaan antara Kambing Kacang dengan Kambing Jawarandu (0.000), demikian juga antara Kambing Marica sama dengan Kambing Samosir (0.000). Jarak genetik paling tinggi dijumpai perbedaan antara Kambing Muara terhadap Kambing Kacang dan Jawarandu (0.008), kemudian dikuti jarak perbedaan antara Kambing Benggala terhadap Kambing Kacang dan Jawarandu (0.006). Perbedaan jarak genetik Kambing Marica dan Samosir terhadap semua sub populasi kambing lainnya sama (0.004), kecuali terhadap
3333333333 3333333333 3333333334 7777777777 7777777777 7777777777 2222222223 6666666667 9999999990 0000000001 1111111112 5555555556 1234567890 1234567890 1234567890 1234567890 1234567890 1234567890 C.hircus_EU581862 TAATTTTAAA AGAATTTG-G CTCTGTTGAT TTCTAAAGCA CTTTCTGATA TTTCCACCTC Benggala1 ...T ...T. ...A.. C... ... ...T... Benggala ...T ...T. ...A.. C... ... ...T... Benggala3 ...T ...T. ...A.. C... ... ...T... Jawarandu1 ... ...-. ...A.. ... ... ... Jawarandu2 ... ...-. ...A.. ... ... ... Jawarandu3 ... ...-. ...A.. ... ... ... Kacang1 ... ...-. ...A.. ... ... ... Kacang2 ... ...-. ...A.. ... ... ... Marica1 ...T ...-. ...A.. C... ... ... Marica2 ...T ...-. ...A.. C... ... ... Muara1 ...T ...-. ...A.. C... ...G.. ...T... Muara2 ...T ...-. ...A.. C... ...G.. ...T... Samosir ...T ...-. ...A.. C... ... ... Shiba_goat_D82963 ... ...-. ... ... ... ...
Kambing Benggala (0.002). Jarak genetik berdasarkan susunan nukleotida DNA Kromosom Y Kambing Kacang, Benggala, Jawarandu, Muara, Samosir, Marica dan Capra hircus (EU 581862) dari GenBank dapat dilihat pada Tabel 16.
Tabel 15 Matrik keragaman nukleotida gen SRY kromosom Y pada kambing lokal Indonesia
Sub populasi C.hircus Kacang Samosir Marica Jawarandu Muara Benggala
Capra hircus* Kacang 0.002 Samosir 0.006 0.004 Marica 0.006 0.004 0.000 Jawarandu 0.002 0.000 0.004 0.004 Muara 0.010 0.008 0.004 0.004 0.008 Benggala 0.008 0.006 0.002 0.002 0.006 0.002
*GenBank no. akses EU 581862
Keragaman genetik antar individu dalam kelompok sub populasi kambing penelitian yaitu sub populasi Kambing Samosir 0.004, Kambing Kacang dan Kambing Jawarandu 0.003, Kambing Marica 0.002, Kambing Muara 0.001 dan Kambing Benggala tidak ada perbedaan (0.000).
Berdasarkan hasil analisis philogenik runutan nukleotida DNA kromoson Y kambing lokal Indonesia dengan nilai bootstrap 89% menunjukkan bahwa Kambing Kacang masih satu kelompok dengan Kambing Jawarandu dan Kambing Marica masih satu kelompok dengan Samosir (21%), sedangkan Kambing Benggala (65%) dan Muara (69%) membentuk kelompok terpisah satu sama lain (Gambar 16).
Berdasarkan hasil analisis metode Neighbour Joining pada keenam sub populasi kambing yang diamati dapat dikelompokkan berdasarkan keragaman genetik secara paternal menjadi 4 kelompok, yaitu kelompok Kambing Kacang dan Jawarandu, kelompok Kambing Marica dan Samosir, kelompok Kambing Benggala dan kelompok Kambing Muara.
Gambar 16 Dendogram Neighbour Joining berdasarkan runutan nukleotida gen SRY antara 6 sub populasi kambing lokal Indonesia
Jika digabungkan dengan rumpun kambing out group dari beberapa situs nukleotida gen SRY di GenBank ditemukan bahwa keenam sub populasi kambing lokal Indonesia membentuk kelompok tersendiri terpisah dari kelompok kambing lainnya. Situs nukleotida Capra hircus (EU 581862) satu kelompok dengan Kambing Shiba dari Jepang dengan jarak genetik dengan kelompok kambing lokal Indonesia 63%, sedangkan jarak kambing lokal dengan Kambing Sangamneri dan Capra hircus (D0845) sangat jauh yaitu 100 % berbeda dengan kambing lokal Indonesia (diduga karenan hasil sekuen kromosom Y yang didapat lebih pendek). Sampai saat ini hasil penelitian gen SRY pada ternak kambing yang dilaporkan dan bisa diakses di GenBank masih sangat terbatas, sehingga belum bisa leluasa membandingkan dengan jenis rumpun kambing lainnya.
Berdasarkan analisis Median-joining network terdapat 6 situs yang bersifat polimorfik, dan terdapat sebanyak 4 haplotip runutan DNA yang unik (Gambar 17) yaitu Kambing Marica dan Samosir (1 haplotip), Kambing Kacang dan Jawarandu (1 haplotip), Kambing Muara (1 haplotip) dan Kambing Benggala (1 haplotip). Muara1 Muara2 Benggala1 Benggala3 Benggala2 Marica1 Marica2 Samosir Jawarandu3 Jawarandu2 Kacang1 Kacang2 Jawarandu1 C.hircus EU581862 Shiba goat D82963 68 66 21 19 64 23 22 87 0.0005
Gambar 17 Median-joining network dari 4 haplotip gen SRY dari 6 sub populasi kambing lokal Indonesia: Kacang (K1), Marica (M1), Samosir (S1),
Jawarandu (J1), Muara (R1) dan Benggala (B1)
Diduga Kambing Marica dan Kambing Samosir berasal dari pejantan Kambing Kacang yang telah mengalami proses adaptasi terhadap lingkungan dan campur tangan perlakuan peternak yang diarahkan untuk tujuan produksi tertentu. Hal ini bisa dilihat dari perubahan (mutasi) susunan nukleotida dalam bentuk substitusi pada situs tertentu (Tabel 15). Sedangkan pada Kambing Jawarandu, Benggala dan Muara ditemukan mutasi subtitusi dan insersi nukleotida diduga akibat proses adaptasi lingkungan dan merupakan hasil persilangan (proses hibridisasi) dengan rumpun kambing diluar kambing lokal yang terdapat di daerah setempat.
Simpulan
Keragaman genetik 6 kambing lokal Indonesia yang diamati adalah 0.004 ± 0.002 dan ditemukan 6 situs susunan nukleotida yang polimorfik dan terdiri dari 4 haplotip yang unik, yaitu pada Kambing Kacang dan Jawarandu (1 haplotip), Marica dan Samosir (1 haplotip), Muara (1 haplotip) dan Benggala (1 haplotip). Keenam sub populasi kambing yang diamati dikelompokkan menjadi 4 haplotip kambing yang berbeda, dimana terdapat perbedaan susunan nukleotida yang khas sebagai penciri DNA antara satu genotip dengan genotip yang lainnya. Asal usul tetua secara paternal Kambing Jawarandu, Marica, Samosir diduga berasal dari Kambing Kacang.
IDENTIFIKASI GEN GDF9 DAN HUBUNGANNYA DENGAN
SIFAT PROLIFIK PADA KAMBING LOKAL INDONESIA
Pendahuluan
Sifat prolifik adalah kemampuan untuk melahirkan dua atau lebih anak sekaligus per periode kelahiran pada induk ternak. Sifat prolifik dikendalikan oleh gen-gen yang dikenal sebagai keluarga gen kesuburan (fecundity genes), yaitu bone morphogenetic protein receptor type 1B (BMPR1B) yang disebut juga dengan nama Fecundity Boorola (FecB) (Souza et al. 2001; Davis 2005, Davis et
al. 2006); growth differentiation factor 9 (GDF9), disebut FecG (Hanrahan et al.
2004); bone morphogenetic protein 15 (BMP15) disebut dengan FecX (Hanrahan
et al. 2004; Galloway et al. 2000). Tiga gen fekunditas diatas dikelompokkan
sebagai TGF-β super family yang telah diidentifikasi pada mamalia.
Beberapa mutasi pada gen GDF9 dilaporkan berhubungan dengan peningkatan laju tingkat ovulasi dan litter size pada ternak ruminansia kecil (Gilchrist et al. 2005) Gen GDF9 ini diekspresikan pada oosit dan sel-sel granulosa ovarium sejak tahap folikel primer sampai oosit diovulasikan. Gen GDF9 yang terdapat pada kromosom 5 pada domba, kambing dan sapi telah berhasil dipetakan yang terdiri atas dua ekson dan satu intron. Selain itu, gen GDF9 ini diketahui menyandikan prepropeptida sepanjang 453 residu asam amino. Polipeptida aktif adalah sepanjang 135 residu asam amino (Bodensteiner
et al. 1999; Hanrahan et al. 2004). Pengaturan ekpresi GDF9 dan proses
pematangan prepropeptida menjadi polipeptida aktif melibatkan ruas-ruas pengaturan ekspresi gen yang terdapat dalam promotor dan intron (Gilchrist et
al. 2005).
Secara alami ternak ruminansia kecil cenderung bersifat prolifik, termasuk kambing dan domba lokal Indonesia. Kambing Kacang mempunyai rata-rata anak sekelahiran (litter size) antara 1.56 – 1.98 ekor (Sodiq et al. 2003; Hoda 2008), kambing PE sekitar 1.3-1.7 anak per kelahiran (Sodiq et al. 2003), sedangkan Kambing Samosir dan Kambing Muara belum ada laporan yang menyebutkan kemampuan prolifikasinya. Beberapa program pemuliaan saat ini aktif untuk mengembangkan kambing yang mengarah pada peningkatan produksi daging, peningkatan pertumbuhan, daya adaptasi terhadap kondisi lingkungan yang sulit dan peningkatan sifat prolifik.
Hasil penelitian identifikasi keragaman gen BMPR1B dan BMP15 di ruas ekson 1, ekson 2 dan intron pada induk Kambing Kacang, PE, Muara dan Samosir dengan sampel yang sama dengan penelitian menunjukkan bahwa sekuen antara kelompok induk beranak kembar hampir sama dengan kelompok induk beranak tunggal atau bersifat monomorfik (Hasan et al. 2011). Pada saat ini informasi yang berhubungan dengan keragaman gen GDF9 pada ruas promotor masih belum ada laporan penelitian.
Tujuan penelitian ini adalah untuk mengidentifikasi keragaman gen GDF9 pada dua kelompok induk, yaitu induk yang rata-rata beranak tunggal (non-prolifik) dan kelompok induk yang rata-rata beranak kembar ((non-prolifik) pada Kambing Kacang, PE, Samosir dan Muara.
Bahan dan Metode
Penelitian ini dilaksanakan mulai bulan Mei 2010 sampai dengan Maret 2011 di Laboratorium Fungsi Hayati dan Perilaku Hewan, Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Pengambilan Sampel Darah Kambing
Pengambilan sampel darah Kambing Kacang dan Kambing Peranakan Etawah (PE) dilakukan di Kandang Percobaan Loka Penelitian Kambing Potong Sei Putih, Sumatera Utara. Sedangkan sampel darah Kambing Samosir diambil dari peternakan rakyat di Kabupaten Samosir dan Kambing Muara di peternakan rakyat di Kabupaten Tapanuli Utara, Propinsi Sumatera Utara. Sampel darah kambing yang diperoleh dari lapangan adalah 149 sampel antara DNA kelompok induk yang rata-rata beranak tunggal (wild type atau normal) disebut dengan kelompok non-prolifik dan dengan kelompok induk yang beranak kembar (mutant
type atau mutan) yang disebut dengan kelompok induk prolifik (Tabel 17).
Tabel 16 Distribusi kambing prolifik dan non prolifik
No. Rumpun kambing Prolifik Non-prolifik Jumlah
1 Kacang 14 11 25
2 Peranakan Etawah (PE) 14 15 29
3 Muara 9 26 35
4 Samosir 24 36 60
Sampel darah diambil dari vena jugularis sekitar 2 ml menggunakan jarum venoject yang dihubungkan dengan tabung vakum. Darah yang diperoleh di lapangan langsung diawetkan dalam alkohol absolut 2x volume darah, kemudian darah dikocok dengan kuat hingga larut dalam alkohol.
Ekstraksi DNA
Ekstraksi genom DNA dilakukan menggunakan Genomic DNA Mini Kit for
Fresh Blood (GeneAid) yang dimodifikasi. Modifikasi yang dilakukan bertujuan
untuk menghilangkan alkohol sebelum dilakukan proses ekstraksi DNA. Sampel darah dalam alkohol sebanyak 1mL disentrifugasi 5 000 rpm selama 5 menit. Endapan sel dicuci dengan menambahkan aquades hingga volume total 1,5 mL dan didiamkan selama 20 menit. Pencucian dilakukan sebanyak dua kali. Sel-sel darah yang telah dicuci disuspensikan dengan bufer pelisis (GT bufer) 100µL, kemudian dilisis lebih lanjut dengan enzim Proteinase K 0.01-0.5 µg/mL dan diinkubasi pada suhu 600C selama 30 menit. Langkah selanjutnya yaitu, pemisahan bahan organik non-DNA dan pemurnian molekul DNA dilakukan sesuai dengan prosedur dari perusahaan.
Amplifikasi Gen GDF9
Amplifikasi gen GDF9 pada ruas promotor (5’flanking region) dilakukan dengan mesin TaKaRa Thermal Cycler menggunakan primer AF 211 forward CCTCAGTCTTCTCCTCGGTTCC dan AF 212 reverse CTGGAAGTGG GAGAAGTGG yang mengacu pada Dong et al. (2005). Amplifikasi gen GDF9 menghasilkan ruas DNA dengan panjang 1972 pb berdasarkan penempelan primer pada sekuen DNA Capra hircus dengan kode accession number EF446168.
Reaksi PCR dilakukan dalam volume 12 µl, yang terdiri atas sampel DNA sekitar 10 ng, primer forward dan reverse masing-masing 1 ng, dan KAPA Taq Ready Mix DNA polymerase (KAPATaq DNA polymerase 1 unit, bufer polimerase dengan Mg2+.25 mM dan setiap dNTP masing-masing 0,4 mM). Kondisi PCR, yaitu predenaturasi 940C selama 5 menit, (denaturasi 940C selama 60 detik, penempelan primer 58 0C selama 90 detik, pemanjangan 720C 90 detik) sebanyak 30 siklus, pemanjangan akhir pada suhu 720 C selama 10 menit, dan penyimpanan dilakukan pada suhu 40C. Amplikon dideteksi dengan
elektroforesis gel poliakrilamida 6% yang dilanjutkan dengan pewarnaan perak (Byun et al. 2009).
Perunutan DNA
Untuk mengetahui posisi dan jenis nukleotida yang mengalami mutasi maka dilakukan perunutan DNA (sekuensing). Beberapa amplikon gen GDF9 pada setiap sub populasi dicampur menjadi satu berdasarkan sifat prolifikasi induk kambing. Campuran amplikon selanjutnya disekuensing menggunakan primer yang sama dengan amplifikasi awal. Teknik ini dilakukan untuk memindai adanya mutasi dengan lebih cepat. Hal ini dilakukan atas dasar laju mutasinya sangat rendah.
Analisis Data
Runutan nukleotida yang diperoleh kemudian diedit secara manual dengan bantuan program Bio Edit versi 6.0.7. Urutan DNA yang telah diedit disejajarkan dengan beberapa runutan DNA dari kelompok Capra hircus yang dipublikasikan dalam GenBank (http://ncbi.nlm.nih.gov). Proses pensejajaran menggunakan Clustal W versi 8.1 yang ada dalam program MEGA 4 (Tamura et
al. 2007) yang kemudian diedit lagi secara manual. Analisis yang dilakukan
meliputi penghitungan komposisi nukleotida, laju mutasi delesi dan substitusi. Proses menentukan struktur gen yang diperoleh untuk mencari situs yang homolog dengan program BLASTN versi 2.2.25 dan dianalisis dengan menggunakan program MEGA versi 4.
Hasil dan Pembahasan
Amplifikasi gen GDF9 menggunakan primer AF 211 dan AF 212 menghasilkan fragmen DNA dengan panjang 1296 pb. Mutasi pada gen GDF9 dapat meningkatkan laju tingkat ovulasi dan litter size. Ruminansia kecil dengan genotip heterozigot carier akan meningkatkan laju ovulasinya rata-rata 1.5 dengan rata-rata litter size 1.0. Sedangkan genotip homozigot carrier akan meningkatkan laju ovulasi rata-rata 3.0 dengan rataan litter size 1.5 (Davis 2005). Jika hasil amplikon gen GDF9 sudah dipotong dengan enzim masih meragukan, dapat diverifikasi dengan metode sekuensing.
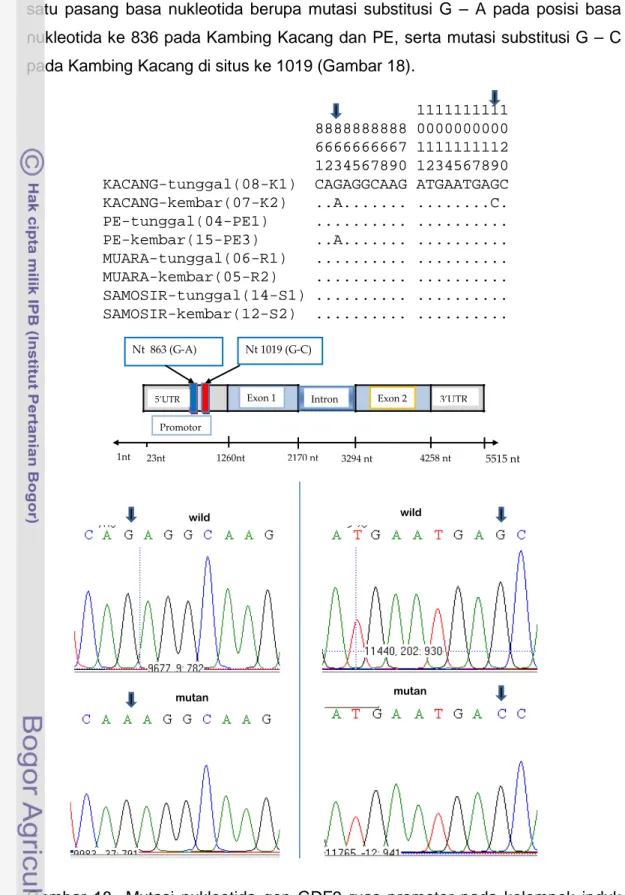
Hasil sekuensing pada gen GDF9 menunjukkan adanya polimorfisme satu pasang basa nukleotida berupa mutasi substitusi G – A pada posisi basa nukleotida ke 836 pada Kambing Kacang dan PE, serta mutasi substitusi G – C pada Kambing Kacang di situs ke 1019 (Gambar 18).
Gambar 18 Mutasi nukleotida gen GDF9 ruas promotor pada kelompok induk prolifik (mutan) dan induk non-prolifik (wild) pada Kambing Kacang dan Peranakan Etawah (Tiga baris pertama dibaca secara vertikal merupakan posisi nukleotida).
1nt 1260nt 2170 nt 4258 nt Exon 1 Exon 2 5515 nt Nt 863 (G-A) Nt 1019 (G-C) Intron 3294 nt Promotor 3’UTR 5’UTR 23nt wild wild 1111111111 8888888888 0000000000 6666666667 1111111112 1234567890 1234567890 KACANG-tunggal(08-K1) CAGAGGCAAG ATGAATGAGC KACANG-kembar(07-K2) ..A... ...C. PE-tunggal(04-PE1) ... ... PE-kembar(15-PE3) ..A... ... MUARA-tunggal(06-R1) ... ... MUARA-kembar(05-R2) ... ... SAMOSIR-tunggal(14-S1) ... ... SAMOSIR-kembar(12-S2) ... ... mutan mutan
Keragaman gen GDF9 dengan sifat prolifik pada kambing bervariasi dan dipengaruhi oleh rumpun ternak dan posisi ruas DNA yang diidentifikasi. GDF9 ruas ekson 1 dan ekson 2 pada Kambing Black Bengal yang dikenal prolifik adalah monomorfik, tetapi pada Kambing Jining Grey dilaporkan polimorfik (Feng
et al. 2010).
Hasil penelitian ini hampir sama dengan penelitian Polley et al. (2009). Adanya alel-alel mutasi nukleotida gen GDF9 di ruas promotor pada Kambing Kacang dan PE dapat menambah temuan identifikasi keragaman gen GDF9 untuk keperluan seleksi calon induk yang prolifik pada ternak domba dan kambing, seperti yang tertera pada Tabel 18.
Tabel 17 Jenis-jenis mutan gen GDF9 pada ternak domba dan kambing
Mutasi DNA
Ekson Mutasi asam amino Jenis ternak Referensi
G 260 A 2 Arginin-Histidin Domba Hanrahan et al. 2004
G 471 T Tidak ada mutasi Domba Hanrahan et al. 2004
G 477 A Tidak ada mutasi Domba Hanrahan et al. 2004
G 721 A Glutamin-Lys Domba Hanrahan et al. 2004
G 978 G Tidak ada mutasi Domba Hanrahan et al. 2004
G 994 A Val - Ile Domba Hanrahan et al. 2004
G 1111 A Val - Met Domba Hanrahan et al. 2004
G 1184 T Ser - Phe Domba Hanrahan et al. 2004
A 562 C 2 Glutamin-Prolin Kambing Qianbei pockmarked
Ren et al. 2010
G 26 A 1 Tidak ada mutasi Kambing Jining Grey Wu et al. 2010 G 792 A 2 Valine - Isoleusine Kambing Jining Grey Wu et al. 2010 A 183 C 1 Tidak ada mutasi Kambing Jining Grey Chu et al. 2011 C 336 T 2 Valine - Isoleusine Kambing Jining Grey Chu et al. 2011 Kambing Jining Grey Feng et al. 2011
Runutan nukleotida gen GDF9 bersifat polimorfik dan diduga berhubungan dengan pengaturan sifat prolifikasi pada Kambing White goat (Xu-qin et al. 2009), Jining Grey (Feng et al. 2010), sementara gen ini juga bersifat monomorfik pada Kambing Black Bengal (Polley et al. 2009), Boer dan Huanghuai Goats (He 2010). Fenomena tersebut juga dijumpai pada penelitian ini, yaitu fenomena keragaman mutasi gen GDF9 di ruas promotor pada keempat sub populasi kambing lokal yang diamati bervariasi. Berdasarkan hasil penelitian tersebut dapat ditarik kesimpulan bahwa ada atau tidaknya polimorfisme nukleotida gen-gen fekunditas pada kambing masih sangat bervariasi antar rumpun-rumpun kambing dan hubungan gen tersebut dengan pengaturan tingkat prolifikasi pada induk kambing. Temuan alel-alel mutasi gen ini diduga dapat
digunakan sebagai salah satu parameter atau metode dalam upaya seleksi calon induk bibit kambing yang prolifik untuk meningkatkan produktivitas dan efisiensi pemeliharaan ternak kambing.
Berdasarkan kejadian mutasi yang bersifat parsimony runutan nukleotida gen GDF9 ruas promotor (Lampiran 7) menunjukkan bahwa kambing lokal Indonesia berada dalam satu klaster dan lebih dekat dengan Kambing Jining grey dari Cina (Gambar 19).
Gambar 19 Dendogram kambing lokal Indonesia berdasarkan runutan nukleotida ruas promotor gen GDF9 metode NJ bootstrap 1000x
Simpulan
Mutasi gen GDF9 ruas promotor bersifat polimorfik yang mengekpresikan adanya hubungan mutasi gen GDF9 dengan sifat prolifik pada Kambing Kacang dan PE. Keragaman gen GDF9 pada induk Kambing Muara dan Samosir bersifat monomorfik. KACANG-tunggal KACANG-kembar C.hircus PE-kembar MUARA-tunggal MUARA-kembar PE-tunggal SAMOSIR-tunggal SAMOSIR-kembar 100 91 62 60 29 26 0.01
PEMBAHASAN UMUM
Sesuai dengan tujuan penelitian untuk mengidentifikasi karakteristik morphometrik dan genetik pada 6 sub populasi kambing lokal Indonesia dan strategi pemanfaatannya secara berkelanjutan, kambing lokal pada 6 sub populasi yang diamati memiliki karakteristik fenotipik (ukuran-ukuran tubuh, bobot badan dan warna tubuh) yang beragam. Rataan ukuran-ukuran tubuh dan bobot badan berbeda antara satu sub populasi dengan sub populasi kambing lokal lainnya. Karakter panjang badan, tinggi pundak, tinggi pinggul, lebar dada, lingkar dada, lingkar kanon, lebar tengkorak, tinggi tengkorak pada kambing lokal yang diamati menunjukkan nilai koefisien keragaman tinggi.
Seleksi juga dapat dilakukan pada warna dan pola warna tubuh kambing lokal yang beragam apabila diinginkan untuk meningkatkan pola warna tertentu. Menurut Astuti (2004) seleksi ternak lokal dengan memanfaatkan keragaman karakteristik sifat produksi dan reproduksi akan meningkatkan produktifitas, disamping memiliki dampak penting terhadap pelestarian sumber daya genetik ternak. Dari keenam sub populasi kambing yang diamati terdapat tiga sub populasi yang sudah menunjukkan pola warna yang spesifik yaitu Kambing Benggala cenderung berwarna hitam, Kambing Samosir sebagian besar berwarna putih, sedangkan Kambing Muara sebagian besar berwarna putih dengan belang hitam ataupun coklat kemerahan (merah bata). Pada Kambing Kacang, Marica dan Jawarandu pola warna masih sangat beragam, sehingga peluang untuk mengarah ke pola warna tertentu untuk membentuk pola warna yang dinginkan masih sangat terbuka.
Adanya perbedaan susunan dan komposisi basa-basa nukleotida antara kambing-kambing lokal Indonesia (Kacang, Jawarandu, Marica, Muara, Samosir dan Benggala) berdasarkan analisis D-loop parsial (957 bp) DNA mitokondria, menunjukkan ada perbedaan materi genetik antara kambing-kambing tersebut. Demikian juga apabila dibandingkan keenam sub populasi kambing lokal Indonesia yang diamati dengan kambing-kambing Eropa, Afrika, Cina dan kambing dari data GenBank, maka jelas terdapat perbedaan materi genetik Kambing Kacang, Jawarandu, Marica, Muara, Samosir dan Benggala dengan kambing dari kelompok tersebut. Menurut Naderi et al. (2007) berdasarkan keragaman genetik DNA mitokondria pendugaan asal-usul ternak kambing dapat dkelompokkan menjadi enam kelompok utama (haplogroup
lineage). Berdasarkan hasil analisis sekuen yang diperoleh keenam sub populasi
kambing lokal yang diamati termasuk kedalam lineage B yaitu kelompok kambing yang menyebar di Asia Timur dan Asia Selatan, Cina, Mongolia, Afrika Selatan, Afrika Utara, Laos, Malaysia, Pakistan dan India. Selain 6 sub populasi kambing lokal Indonesia yang diamati dalam penelitian ini, di Indonesia juga dijumpai jenis rumpun kambing lainnya yang termasuk dalam kelompok utama (haplogroup
lineage) A yang berupa rumpun-rumpun kambing yang daerah penyebarannya
dari Eropa seperti Kambing Saanen, Kambing Nubian dan Jamnapari (Etawah). Susunan nukleotida yang khas (unik) ini dapat dipakai sebagai salah satu penciri untuk mengidentifikasi kambing lokal Indonesia. Penciri khas kambing lokal Indonesia dapat diperoleh setelah dibandingkan dengan nukleotida
Capra hircus (AF533441) dari GenBank yaitu perubahan nukleotida pada situs ke
201, 386, 625 (A-G); 113, 633, 697 (C-T); 298 (G-A); 537 (G-C); 597 (T-C) dan 812 (C-A). Sedangkan setiap sub populasi susunan basa DNA yang unik yang dapat dipakai sebagai penciri khas pada Kambing Kacang, Marica, Jawarandu, Muara, Samosir dan Kambing Benggala.
Berbeda dengan keragaman susunan genetik yang tinggi pada metode analisis DNA mitokondria, pada analisis DNA kromosom Y pada segmen gen SRY tingkat keragaman susunan genetik relatif lebih rendah. Berdasarkan primer yang didesain sendiri diperoleh hasil analisis sekuen susunan basa nukleotida sepanjang 499 bp dan terdiri dari 6 situs yang bersifat polimorfik dan ditemukan 4 haplotip susunan nukleotida yang khas yang dapat dipakai sebagai salah satu acuan untuk mengetahui asal-usul berdasarkan keturunan secara paternal (menurut garis keturunan ayah, tetua jantan). Berdasarkan haplotip tersebut ditemukan bahwa Kambing Kacang satu kelompok dengan Kambing Jawarandu, disini memperkuat dugaan Kambing Jawarandu merupakan keturunan dari persilangan pejantan Kambing Kacang dengan induk Kambing Peranakan Etawa (PE) atau Etawah, hal ini juga bisa dilihat dari penampilan ciri-ciri fenotipik yang berbeda dengan Kambing PE yang antara lain panjang dan lebar telinga lebih pendek, tinggi pundak lebih rendah sehingga performan Kambing Jawarandu lebih mengarah ke Kambing Kacang dari pada ke arah Kambing Etawah (Jamnapari).
Kambing Marica dan Kambing Samosir satu kelompok tetapi keragaman genetiknya tidak berbeda nyata dengan kelompok Kambing Kacang dan Kambing Jawarandu. Diduga Kambing ini berasal dari Kambing Kacang yang
telah mengalami adaptasi dengan lingkungan setempat dalam jangka waktu yang lama. Kambing Marica diduga mengalami adaptasi dengan kondisi lingkungan di Sulawesi Selatan dengan masa musim kemarau yang panjang (6-9 bulan per tahun), sehingga mulai dari pertengahan sampai akhir musim kering rumput sudah mengering dan jumlahnya relatif terbatas sehingga kambing beradaptasi, secara fenotip dapat dilihat ukuran tubuh mengecil dan bulu relatif coklat kemerahan dan mengkilat.
Pada Kambing Samosir terjadi karena seleksi campur tangan manusia, dimana peternak ingin memelihara kambing yang berwarna putih untuk keperluan ritual keagamaan, sehingga kambing yang berwarna putih dapat dijual dengan harga yang lebih tinggi. Tetapi di beberapa tempat karakteristik ukuran tubuh dan pola warna Kambing Samosir ada yang agak mirip dengan Kambing Saanen, diduga Kambing Samosir mungkin juga merupakan hasil persilangan Kambing lokal dengan Kambing Saanen, tetapi performan tubuh dan bobot tubuh relatif lebih kecil jika dibandingkan dengan Kambing Saanen. Jika ditinjau dari runutan nukleotida daerah D-loop DNA mitokondria dan DNA kromosom Y segmen gen SRY lebih dekat dengan Kambing Kacang dan Kambing Marica.
Kambing Muara diduga merupakan kambing persilangan Kambing Peranakan Etawah dengan kambing lokal di daerah Kabupaten Tapanuli Utara yang telah beradaptasi dengan lingkungan setempat. Dengan topografi lingkungan dataran tinggi yang berbukit-bukit, ketersediaan pakan relatif cukup membentuk postur tubuh Kambing Muara relatif besar dan kompak, sangat cocok untuk ternak kambing penghasil daging. Juga berdasarkan pengamatan dari jumlah anak perkelahiran, beberapa induk beranak empat dapat hidup sampai lepas sapih di tingkat petani. Hal ini diduga disamping suplay makanan cukup juga di dukung produksi susu yang relatif baik untuk mendukung pertumbuhan anak kambing sejak lahir.
Kambing Benggala umumnya berwarna hitam dan ada coklat tua, telinga separuh di ujung jatuh, tinggi pundak relatif pendek . Kambing ini terpisah jauh dari Kambing Kacang (0.006). Diduga kambing ini merupakan hasil persilangan Kambing Black Bengal dengan kambing lokal di daerah Nusa Tenggara Timur. Diduga ternak tersebut masuk ke daerah NTT melalui para pedagang dari India, Pakistan atau Sri Lanka yang di bawa serta untuk menghasilkan susu dan daging. Masyarakat menyebutnya Kambing Benggala (mungkin berasal dari
India dari kata Benggali) atau karena warna kambing tersebut berwarna hitam pada umumnya.
Berdasarkan jarak genetik pada ukuran-ukuran tubuh, penampilan, pola warna dominan, keragaman genetik berdasarkan D-loop DNA mitokondria dan kromosom Y segmen gen SRY, diduga setiap populasi kambing lokal Indonesia yang diamati telah membentuk karakteristik morfologi dan karakteristik genetik tersendiri yang khas sehingga bisa dikelompokkan menjadi 6 rumpun atau galur kambing yaitu Kambing Kacang, Kambing Jawarandu, Kambing Marica, Kambing Muara, Kambing Samosir dan Kambing Benggala. Berdasarkan hasil penelitian ini kambing lokal Indonesia dapat dipetakan keragaman genetiknya untuk tujuan konservasi sumberdaya genetik ternak lokal dan pengembangan ternak lokal guna mendukung ketahanan pangan dan peningkatan pendapatan masyarakat peternak kambing. Dengan diperolehnya status dan karakteristik morfologi kambing lokal ini, perlu dilanjutkan karakterisasi produksi, reproduksi dan daya tahannya terhadap penyakit dan perubahan lingkungan di daerah tropis.
Sampai saat ini laporan hasil penelitian pendahuluan tentang hubungan ketiga gen fekunditas tersebut masih sangat bervariasi, sebagian dilaporkan ada polimorfisme dan sebagian lagi melaporkan tidak terdapat polimorfisme. Beberapa hasil penelitian juga melaporkan bahwa keragaman Gen GDF9 bersifat monomorfik pada Kambing Boer, Haimen, Huanghuai, Nubi, dan Kambing Matou (Guo-Hua et al. 2008). Hasil penelitian menunjukkan tingkat keragaman gen GDF9 pada kelompok induk yang rata-rata beranak kembar dan kelompok induk yang rata-rata beranak tunggal pada Kambing Kacang, PE, Samosir dan Muara bersifat monomorfik. Alternatif lain yang mungkin dilakukan adalah dengan mengidentifikasi gen-gen lain yang diduga berhubungan dengan sifat fekunditas pada induk kambing yang beranak kembar, seperti beberapa jenis gen yang diduga berhubungan dengan sifat beranak kembar pada kambing antara lain ; gen FSHB yang berhubungan dengan superovulasi pada Kambing Matou (Zhang et al. 2011), gen KiSS-1 diduga berhubungan dengan sifat prolifik pada Kambing Guanzhong dan Xinong Saanen (Huo et al. 2011), gen INHA masih diduga mengontrol sifat prolifik pada Kambing Matou dan Kambing Haimen (Guo-Huo et al. 2007). Fenogram berdasarkan data karakter morfometrik, dendogram berdasarkan mitokondria menunjukkan bahwa masing-masing sub populasi berbeda, sehingga dapat dikelompokkan menjadi 6 rumpun kambing lokal. Sedangkan dendogram berdasarkan nukleotida kromosom Y dan gen GDF9 ruas
promotor menunjukkan bahwa secara paternal Kambing Kacang dan Jawarandu termasuk satu kelompok, serta Kambing Muara dengan PE satu kelompok. Ini memperkuat dugaan bahwa Kambing PE merupakan tetua jantan dari Kambing Muara.
Hasil penelitian ini memperoleh data dan informasi pendahuluan tentang pemetaan keragaman sumberdaya genetik kambing lokal Indonesia khususnya untuk rumpun atau galur Kambing Kacang, Marica, Muara, Jawarandu, Samosir dan Kambing Benggala. Berdasarkan Peraturan Menteri Pertanian Nomor 35/Permentan/OT.140/8/2006, tentang Pedoman Pelestarian dan Pemanfaatan Sumberdaya Genetik, upaya pelestarian dan pengembangan kambing lokal Indonesia penting dilakukan untuk mengantisipasi kehilangan sumberdaya genetik plasma nuftah kambing akibat adanya persilangan dan pemotongan ternak yang tidak seimbang dengan produksinya.
Untuk menghindari kehilangan sumberdaya genetik kambing lokal Indonesia perlu dilanjutkan karakterisasi produktivitas dan keunggulan potensi genetiknya dan penelitian pengembangan, kemudian disertai dengan sosialisasi pengenalan ternak kambing lokal tersebut terutama di wilayah dimana populasi aslinya berada (in-situ), sehingga timbul minat untuk memelihara dan mengembangkannya. Sampai saat ini pada umumnya sistim pemeliharaan ternak kambing lokal masih diserahkan kepada kebaikan alam, dengan pemeliharaan secara tradisional (kearifan lokal). Disadari atau tidak ternak kambing lokal tersebut berperan menambah pendapatan keluarga petani di daerah pedesaan dengan sistim pemeliharaan dengan biaya relatip rendah (low
input system), sesuai dengan kondisi agro-ekosistem setempat dan ternak dapat
hidup dan berproduksi.
Jika berhasil ditetapkan secara ilmiah rumpun-rumpun atau galur kambing lokal berdasarkan hasil-hasil penelitian dan sudah dapat ditetapkan, diharapkan kemudian akan dilanjutkan dengan penetapan wilayah pengembangan rumpun atau galur kambing lokal tertentu. Dengan begitu sumberdaya genetik kambing lokal tersebut dapat dipertahankan dan dikembangkan secara berkelanjutkan untuk meningkatkan pendapatan petani dan ikut mendukung ketahanan pangan di daerah-daerah pedesaan. Hal ini mengacu kepada Undang-Undang Republik Indonesia Nomor 18 tahun 2009, tentang Peternakan dan Kesehatan Hewan yang telah mengakomodir kekuatiran pemerintah akan terjadinya penggerusan materi genetik ternak lokal dengan melaksanakan program pewilayahan yang
dibagi atas: a) wilayah sumber bibit, yang bersifat mengembangkan secara murni, dengan mempertimbangkan jenis ternak dan rumpun, agroklimat, kepadatan penduduk, sosial ekonomi, budaya serta ilmu pengetahuan dan teknologi, b) wilayah produksi, pengembangbiakan dengan tujuan komersil yang memungkinkan menggunakan teknik-teknik perkawinan silang dan penggemukan, dan c) wilayah konservasi, dengan melakukan penangkaran hewan/ternak asli yang masih ada atau mengembangbiakkan hasil dari suatu wilayah sumber bibit (Departemen Pertanian 2006). Di Indonesia umumnya kambing lokal mempunyai potensi reproduksi yang sangat baik, maka introduksi melalui persilangan merupakan salah satu cara untuk menggabungkan sifat adaptabilitas dengan keunggulan genetik pertumbuhan rumpun kambing yang diintroduksi. Alternatif lain adalah dengan melakukan program seleksi untuk pengembangan ternak kambing lokal yang dianggap cukup potensial untuk menjadi bibit unggul yang berbasis rumpun kambing lokal, seperti galur Kambing PE Kaligesing yang sudah ditetapkan oleh pemerintah.
Berdasarkan jumlah sampel selama pengamatan di lapangan jumlah data pejantan sangat terbatas dan sulit dijumpai di daerah pedesaan. Diduga hal ini terjadi karena peternak lebih sering menjual kambing jantan karena harga jual yang lebih tinggi dan kurang memperhatikan kualitas pejantan yang akan digunakan. Hal tersebut akan menyebabkan erosi genetik dan penurunan kualitas genetik ternak. Pada umumnya pengetahuan tentang jenis rumpun dan tingkat kesadaran menjaga kelestarian ternak kambing lokal masih sangat terbatas, sehingga perlu dilakukan sosialisasi dan penyuluhan tentang perlunya mengenal dan mempertahankan sumberdaya genetik kambing lokal Indonesia.