ABSTRAK
Kanker payudara merupakan penyakit yang terjadi akibat pertumbuhan sel yang tidak terkendali pada payudara. Salah satu penyebab kanker payudara adalah ekspresi yang berlebihan dari reseptor estrogen alfa (RE-α). Emodin merupakan salah satu senyawa fitoestrogen. Berdasarkan penelitian Liliana (2015) secara in silico menggunakan protokol penambatan yang telah divalidasi Setiawati, Riswanto, Yuliani dan Istyastono (2014) dan dilanjutkan dengan post-docking analysis oleh Istyastono (2015) senyawa emodin bukan ligan aktif dalam ikatan RE-α.
Pada penelitian ini, dilakukan desain teoretis berbantukan komputer untuk mendapatkan desain modifikasi struktur emodin sebagai ligan aktif RE-α dengan uji in silico menggunakan protokol penambatan yang telah divalidasi oleh Setiawati et al. (2014) dan dilanjutkan dengan post-docking analysis oleh Istyastono (2015). Desain modifikasi yang aktif kemudian divisualisasikan posenya pada kantung ikatan RE-α dan dilakukan analisis diskoneksi untuk menentukan rute sintesisnya.
Hasil penelitian yang dilakukan terdapat enam desain modifikasi struktur emodin yang merupakan ligan aktif terhadap REα yaitu desain modifikasi kode Fito11, Fito12, Fito14, Fito17, Fito24, dan Fito25. Berdasarkan hasil analisis diskoneksi hanya desain modifikasi kode Fito11, Fito14, Fito24, dan Fito25 yang dapat dilanjutkan dengan usulan mekanisme sintesis yaitu dengan menggunakan reaksi substitusi nukleofilik aromatis.
ABSTRACT
Breast cancer is a disease caused by uncontrolled cell growth at breast tissue. One of the causes of breast cancer is the over-expression of estrogen receptor alpha (ER-α) by the estrogen hormone. Emodin is one of the phytoestrogen compound. Based on in silico research by Liliana (2015), using docking protocol by Setiawati, Riswanto, Yuliani and Istyastono (2014) and continued with post-docking analysis by Istyastono (2015) shown that emodin was not an active ligand on ERα.
In this study, theoretical computer-aided design was conducted to obtain the design of structural modification of emodin as active ligand on ERα based in silico research using Setiawati et al. (2014) docking protocol and Istyastono (2015) post-docking analysis protocol. Active design was visualized on ER-α binding pocket and continued with disconection analysis to determine synthesis route.
In this research, six designs of structural modification of emodin which were active ligands on ER-α. Those are Fito11, Fito12, Fito14, Fito17, Fito24, and Fito25. Based on disconnection analysis only Fito11, Fito14, Fito24, and Fito25 were proceed with the proposed mechanism of synthesis. The synthesis is using an aromatic nucleophilic substitution reaction.
Keywords: Emodin, breast cancer, estrogen receptor alpha, design of structural modification of emodin
DESAIN TEORETIS BERBANTUKAN KOMPUTER MODIFIKASI
STRUKTUR EMODIN SEBAGAI LIGAN RESEPTOR ESTROGEN ALFA
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat
Memperoleh Gelar Sarjana Farmasi (S.Farm.)
Program Studi Farmasi
Oleh :
Pius Pradana Bekti Indramawan
NIM : 128114001
FAKULTAS FARMASI
UNIVERSITAS SANATA DHARMA
YOGYAKARTA
i
DESAIN TEORETIS BERBANTUKAN KOMPUTER MODIFIKASI
STRUKTUR EMODIN SEBAGAI LIGAN RESEPTOR ESTROGEN ALFA
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat
Memperoleh Gelar Sarjana Farmasi (S.Farm.)
Program Studi Farmasi
Oleh :
Pius Pradana Bekti Indramawan
NIM : 128114001
FAKULTAS FARMASI
UNIVERSITAS SANATA DHARMA
YOGYAKARTA
iv
HALAMAN PERSEMBAHAN
“Tuhan adalah kekuatan dan perisaiku” – Mazmur 28:7
“If you are affraid to fail, then you‘re probably going to fail” – Kobe Bryant
“Setiap orang yang berusaha dan bekerja keras, suatu saat pasti akan melakukan kesalahan, sedangkan mereka yang hanya duduk berdiam diri serta berpangku
tangan, tidak akan pernah melakukan kesalahan.” – Bambang Pamungkas
“Everybody is a genius. But if you judge a fish by its ability to climb a tree, it will spends its whole life believing that it is stupid.” – Albert Einstein
“Cukup satu langkah awal. Ada kerikil saya singkirkan. Melangkah lagi. Bertemu duri saya sibakkan. Melangkah lagi. Terhadang lubang saya lompati. Melangkah lagi. Berjumpa api saya mundur. Melangkah lagi. Berjalan terus dan mengatasi masalah.” – Bob Sadino
Saya persembahkan untuk: Tuhan Yang Maha Esa
Kedua orang tua dan adik saya (Albertus Pradananto Haryo Budi Wicaksono) Teman-teman seperjuangan dalam penyusunan skripsi (Ave, Berto, dan Rina) Teman-teman Farmasi USD 2012 selama perjalanan kuliah penulis
Diri saya sendiri
vi PRAKATA
Puji dan syukur kepada Tuhan Yang Maha Esa atas berkat dan rahmat-Nya sehingga penyusunan skripsi dengan judul “Desain Teoretis berbantukan Komputer Modifikasi Struktur Emodin sebagai Ligan Reseptor Estrogen Alfa” dapat diselesaikan sebagai salah satu syarat untuk memperoleh gelar Sarjana Farmasi (S.Farm.). Dalam penyusunan skripsi, penulis mendapat banyak bantuan dari berbagai pihak. Oleh karena itu, penulis mengucapkan terima kasih melalui prakata ini kepada:
1. Enade Perdana Istyastono, Ph.D., Apt. selaku dosen pembimbing yang telah memberikan pengetahuan melalui bimbingan, pengarahan, dan saran dari selama proses skripsi.
2. Jeffry Julianus, M.Si. dan Florentinus Dika Octa Riswanto, M.Sc., selaku dosen penguji yang telah memberikan saran dari ujian proposal sampai penyusunan naskah skripsi ini.
3. Kedua orang tua atas usaha, doa, dan dukungan selama perjalanan kuliah penulis hingga penyusunan skripsi.
4. Adikku (Albertus Pradananto Haryo Budi Wicaksono) atas doa, dukungan, dan hiburannya.
5. Ave, Berto, dan Rina selaku teman seperjuangan sejak awal perkuliahan hingga penyusunan skripsi atas semangat dan dukungannya.
6. Feli, Feli, Nana, Karla dan Keket yang senantiasa memberikan bantuan dan bimbingan yang dibutuhkan selama pengerjaan skripsi ini.
7. Ridho yang telah membantu dalam proses instalasi program yang digunakan selama penyusunan skripsi.
8. Abal, Keket, Irest, Resta, Ella, Edo, dan Mike, teman sepermainan yang selalu memberikan semangat dan mendoakan suksesnya skripsi ini.
9. Seluruh teman-teman Farmasi USD 2012 selaku almamater dan teman seperjuangan.
vii
Akhir kata, penulis menyadari bahwa masih terdapat banyak kekurangan dalam skripsi yang sudah disusun oleh penulis mengingat keterbatasan kemampuan penulis. Oleh karena itu, penulis sangat mengharapakan saran dan kritik yang membangun. Semoga karya ini dapat bermanfaat bagi perkembangan penelitian senyawa ligan reseptor estrogen alfa dan kanker payudara.
ix DAFTAR ISI
HALAMAN JUDUL ... i
HALAMAN PERSETUJUAN PEMBIMBING ... ii
HALAMAN PENGESAHAN ... iii
HALAMAN PERSEMBAHAN ... iv
LEMBAR PERNYATAAN PERSETUJUAN PUBLIKASI... v
PRAKATA ... vi
PERNYATAAN KEASLIAN KARYA ... viii
DAFTAR ISI ... ix
2.3.1. Preparasi Desain Modifikasi Struktur Emodin ... 4
2.3.2. Penambatan Desain Modifikasi Struktur Emodin ... 5
2.3.3. Post-Docking Analysis ... 5
2.3.4. Analisis Hasil ... 5
2.3.5. Analisis Diskoneksi dan Penentuan Jalur Sintesis ... 6
3. HASIL DAN PEMBAHASAN ... 7
4. KESIMPULAN ... 26
DAFTAR PUSTAKA ... 27
LAMPIRAN ... 29
x
DAFTAR TABEL
Tabel I. Aktivitas bitstring penting senyawa emodin... 3 Tabel II. PLIF bitstring penting menurut decision tree... 6 Tabel III. Nama IUPAC desain modifikasi struktur emodin kode
Fito1-Fito10... 9 Tabel IV. Luaran penambatan desain modifikasi emodin kode
Fito1-Fito10 berdasarkan bitstring penting dalam decision
tree... 9 Tabel V. Nama IUPAC desain modifikasi struktur emodin kode
Fito11-Fito27... 12 Tabel VI. Luaran penambatan desain modifikasi emodin kode
Fito11-Fito27 berdasarkan bitstring penting dalam decision
tree... 13 Tabel VII. Skor ChemPLP lima replikasi desain modifikasi struktur aktif
xi
DAFTAR GAMBAR
Gambar 1. Struktur emodin... 2
Gambar 2. Pose emodin dalam kantung ikatan RE-α... 2
Gambar 3. Alur emodin dalam decision tree... 3
Gambar 4. Alur tahapan prosedur... 7
Gambar 5. Desain modifikasi struktur emodin kode Fito1-Fito10... 8
Gambar 6. Desain modifikasi struktur emodin kode Fito11-Fito14... 10
Gambar 7. Desain modifikasi struktur emodin kode Fito15-Fito27... 11
Gambar 8. Alur decision tree yang dilalui desain kode Fito11, Fito12, Fito14, Fito17, Fito24, dan Fito25... 14
Gambar 9. (a) Visualisasi replikasi pose desain Fito11 pada kantung ikatan RE-α. (b) Visualisasi pose desain Fito11 pada kantung ikatan RE-α... 17
Gambar 10. (a) Visualisasi replikasi pose desain Fito12 pada kantung ikatan RE-α. (b) Visualisasi pose desain Fito12 pada kantung ikatan RE-α... 17
Gambar 11. (a) Visualisasi replikasi pose desain Fito14 pada kantung ikatan RE-α. (b) Visualisasi pose desain Fito14 pada kantung ikatan RE-α... 18
Gambar 12. (a) Visualisasi replikasi pose desain Fito17 pada kantung ikatan RE-α. (b) Visualisasi pose desain Fito17 pada kantung ikatan RE-α... 18
Gambar 13. (a) Visualisasi replikasi pose desain Fito24 pada kantung ikatan RE-α. (b) Visualisasi pose desain Fito24 pada kantung ikatan RE-α... 19
Gambar 14. (a) Visualisasi replikasi pose desain Fito25 pada kantung ikatan RE-α. (b) Visualisasi pose desain Fito25 pada kantung ikatan RE-α... 19
Gambar 15. Mekanisme diskoneksi Fito11... 20
xii
Gambar 17. Mekanisme diskoneksi Fito12... 21
Gambar 18. Mekanisme diskoneksi Fito14... 21
Gambar 19. Mekanisme reaksi sintesis desain Fito14... 22
Gambar 20. Mekanisme diskoneksi Fito17... 22
Gambar 21. Mekanisme diskoneksi Fito24... 23
Gambar 22. Mekanisme reaksi sintesis desain Fito24... 24
Gambar 23. Mekanisme diskoneksi Fito25... 24
xiii
DAFTAR LAMPIRAN
Lampiran 1. Desain modifikasi struktur emodin menggunakan
BKChem0.13.0... 30
Lampiran 2. Penambahan atom hidrogen pada pH 7,4 dan generate 3D desain modifikasi struktur emodin menggunakan Open Babel... 30
Lampiran 3. Perintah penambatan berupa file shell script dengan nama file clin.sh... 31
Lampiran 4. Pembuatan folder dengan nama file emod di dalam server.... 31
Lampiran 5. Upload file shell script dan struktur 3D desain modifikasi struktur emodin... 32
Lampiran 6. Menjalankan perintah penambatan di server... 32
Lampiran 7. Proses penambatan dalam PLANTS1.2... 33
Lampiran 8. Download luaran hasil dari penambatan pada PLANT1.2... 34
Lampiran 9. Analisis hasil penambatan dengan aplikasi statistik R... 35
Lampiran 10. File Data_R.csv untuk melihat ikatan dengan bitstring penting dan memilih satu replikasi dengan skor ChemPLP terendah... 35
Lampiran 11. Input ligan pada PyMOL... 36
Lampiran 12. Input struktur 3D Reseptor Estrogen Alfa pada PyMOL... 36
Lampiran 13. Input protein binding site pada PyMOL... 37
xiv ABSTRAK
Kanker payudara merupakan penyakit yang terjadi akibat pertumbuhan sel yang tidak terkendali pada payudara. Salah satu penyebab kanker payudara adalah ekspresi yang berlebihan dari reseptor estrogen alfa (RE-α). Emodin merupakan salah satu senyawa fitoestrogen. Berdasarkan penelitian Liliana (2015) secara in silico menggunakan protokol penambatan yang telah divalidasi Setiawati, Riswanto, Yuliani dan Istyastono (2014) dan dilanjutkan dengan post-docking analysis oleh Istyastono (2015) senyawa emodin bukan ligan aktif dalam ikatan RE-α.
Pada penelitian ini, dilakukan desain teoretis berbantukan komputer untuk mendapatkan desain modifikasi struktur emodin sebagai ligan aktif RE-α dengan uji in silico menggunakan protokol penambatan yang telah divalidasi oleh Setiawati et al. (2014) dan dilanjutkan dengan post-docking analysis oleh Istyastono (2015). Desain modifikasi yang aktif kemudian divisualisasikan posenya pada kantung ikatan RE-α dan dilakukan analisis diskoneksi untuk menentukan rute sintesisnya.
Hasil penelitian yang dilakukan terdapat enam desain modifikasi struktur emodin yang merupakan ligan aktif terhadap REα yaitu desain modifikasi kode Fito11, Fito12, Fito14, Fito17, Fito24, dan Fito25. Berdasarkan hasil analisis diskoneksi hanya desain modifikasi kode Fito11, Fito14, Fito24, dan Fito25 yang dapat dilanjutkan dengan usulan mekanisme sintesis yaitu dengan menggunakan reaksi substitusi nukleofilik aromatis.
xv ABSTRACT
Breast cancer is a disease caused by uncontrolled cell growth at breast tissue. One of the causes of breast cancer is the over-expression of estrogen receptor alpha (ER-α) by the estrogen hormone. Emodin is one of the phytoestrogen compound. Based on in silico research by Liliana (2015), using docking protocol by Setiawati, Riswanto, Yuliani and Istyastono (2014) and continued with post-docking analysis by Istyastono (2015) shown that emodin was not an active ligand
on ERα.
In this study, theoretical computer-aided design was conducted to obtain
the design of structural modification of emodin as active ligand on ERα based in silico research using Setiawati et al. (2014) docking protocol and Istyastono (2015) post-docking analysis protocol. Active design was visualized on ER-α binding pocket and continued with disconection analysis to determine synthesis route.
In this research, six designs of structural modification of emodin which were active ligands on ER-α. Those are Fito11, Fito12, Fito14, Fito17, Fito24, and Fito25. Based on disconnection analysis only Fito11, Fito14, Fito24, and Fito25 were proceed with the proposed mechanism of synthesis. The synthesis is using an aromatic nucleophilic substitution reaction.
1
1. Pendahuluan
Kanker merupakan penyakit peringkat teratas penyebab kematian di seluruh dunia, pada tahun 2012, angka kematian akibat kanker mencapai 8,2 juta orang dan 521.000 orang diantaranya disebabkan oleh kanker payudara (World Health Organization, 2015). Kanker payudara merupakan tumor ganas yang dimulai dari sel-sel payudara kemudian tumbuh dan menyerang jaringan di sekitarnya serta organ lainnya (American Cancer Society, 2014). Pada tahun 2014, terdapat 48,998 kasus kanker payudara pada wanita di Indonesia dengan kematian sebesar 19.730 kasus (21,4%) dari seluruh kasus kematian yang disebabkan oleh kanker (World Health Organization, 2014).
Estrogen dan reseptor estrogen (RE) berperan penting dalam genesis dan pertumbuhan malignan pada kanker payudara. Tingkat estrogen yang tinggi dan ekspresi reseptor estrogen alfa secara berlebih sering terobservasi pada sebagian besar kanker payudara (Hayashi et al., 2003). Hal tersebut menyebabkan inhibisi reseptor estrogen alfa menjadi pendekatan utama dalam pencegahan dan terapi kanker (Suganya, Radha, Naorem, dan Nishandhini, 2014).
Salah satu obat yang digunakan untuk terapi kanker payudara adalah tamoxifen. Tamoxifen bekerja sebagai parsial antagonis, tamoxifen berkompetisi dengan estrogen untuk berikatan dengan reseptor sehingga dapat merusak fungsi reseptor estrogen (Salazar, Ratnam, Patki, Kisovic, Trumbly, dan Iman, 2011). Obat tersebut ternyata menyebabkan berbagai efek samping seperti penggumpalan darah, stroke, kanker rahim, dan katarak, sehingga perlu dilakukan pengembangan obat untuk terapi kanker payudara, salah satunya dengan melakukan pengembangan ke arah obat alternatif atau tradisional dari tanaman seperti fitoestrogen untuk menemukan senyawa baru yang dapat mengobati kanker payudara dengan efek samping yang kecil (Suganya et al., 2014).
2
Gambar 1. Struktur emodin (Pubchem, 2016)
Emodin merupakan senyawa golongan anthraquinone yang terdapat dalam Aloe vera, Rheum officinale, Polygonum cuspidatum, Rheum palmate, dan tanaman genus lain seperti Polygonum, Rhamnaceae, Leguminosae, dan Liliaceae. (Huang et al., 2013; Izhaki, 2002; Xing et al., 2015; Yaoxian, Hui, Yunyan, Yangin, Xin, dan Xiaoke, 2013). Berdasarkan penelitian Liliana (2015), emodin telah diteliti
secara in silico menggunakan protokol Setiawati, Riswanto, Yuliani, dan Istyastono (2014) dan post-docking analysis Istyastono (2015). Hasil dari penelitian tersebut emodin berupa skor ChemPLP terendah yaitu -75,292 dari 1000 kali replikasi dan pose emodin dalam kantung ikatan RE-α.
Gambar 2. Pose emodin dalam kantung ikatan RE-α (Liliana, 2015)
3
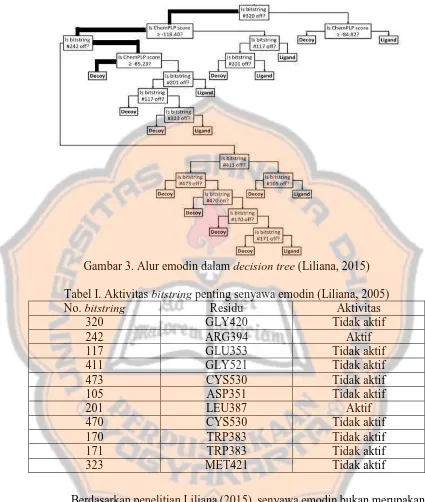
Gambar 3. Alur emodin dalam decision tree (Liliana, 2015)
Tabel I. Aktivitas bitstring penting senyawa emodin (Liliana, 2005)
No. bitstring Residu Aktivitas
320 GLY420 Tidak aktif
242 ARG394 Aktif
117 GLU353 Tidak aktif
411 GLY521 Tidak aktif
473 CYS530 Tidak aktif
105 ASP351 Tidak aktif
201 LEU387 Aktif
470 CYS530 Tidak aktif
170 TRP383 Tidak aktif
171 TRP383 Tidak aktif
323 MET421 Tidak aktif
4
bertindak sebagai ligan aktif pada RE-α menggunakan protokol Setiawati et al. (2014) dan protokol post-docking analysis Istyastono (2015).
2. Metode Penelitian
Penelitian ini termasuk jenis penelitian observasional berbantukan komputer untuk mendesain senyawa modifikasi struktur emodin yang mampu menjadi ligan aktif RE-α.
2.1. Alat penelitian
Alat yang digunakan adalah server Fakultas Farmasi Universitas Sanata Dharma dengan nomor alamat IP 103.247.10.66 (pharcomp.web.id), Toshiba Satelite C640 dengan spesifikasi: Prosesor Intel Pentium P6200 @2,13GHz, RAM 2,00 GB, 32-bit Operating System, Linux Ubuntu 12.04.
2.2. Bahan penelitian
Bahan yang digunakan adalah protokol yang telah dikembangkan dari protokol Anita et al. (2012) dan divalidasi oleh Setiawati et al. (2014) dan protokol post-docking analysis yang dikembangkan Istyastono (2015), struktur tiga dimensi senyawa modifikasi emodin yang diperoleh dari luaran Open Babel, BKChem0.13.0 (Istyastono, Anita, dan Sundowo, 2014) untuk mendesain struktur, Open Babel (Anita et al., 2012) untuk menambahkan atom hidrogen pada pH 7,4 dan mengubah struktur ke bentuk 3D. SPORES (Brink dan Exner, 2009) untuk menyiapkan senyawa yang akan ditambatkan ke PLANTS1.2 (Korb, Stutzel, dan Exner, 2009), Docking Software PLANTS1.2 (Korb et al., 2009) untuk mensimulasikan penambatan molekuler sehingga didapatkan skor ChemPLP, PyPLIF (Radifar et al., 2013) untuk mengidentifikasi protein-ligan interaction fingerprint, PyMOL1.2 (Lill dan Danielson, 2011) untuk
menghasilkan gambar molekuler, dan R 3.2.1 (R Foundation, 2015) untuk analisis statistik.
2.3. Prosedur penelitian
2.3.1.Preparasi desain modifikasi struktur emodin
5
struktur dari senyawa emodin dengan menggunakan BKChem0.13.0 dan didapatkan file .mol. Struktur .mol ditambahkan atom hidrogen pada pH 7,4 menggunakan Open Babel, kemudian dilakukan generate 3D dengan menggunakan Open Babel yang akan mendapatkan file berupa .mol2. Struktur .mol2 disiapkan untuk ditambatkan pada perangkat lunak PLANTS1.2 (Korb et al., 2009) menggunakan aplikasi SPORES (Brink dan Exner, 2009).
2.3.2.Penambatan desain modifikasi struktur emodin
Luaran dari SPORES ditambatkan dengan menggunakan PLANTS1.2 dengan konfigurasi mengacu pada Setiawati et al. (2014). Iterasi penambatan molekuler dilakukan tiga kali dengan luaran berupa 3 x 50 pose kemudian dipilih satu skor ChemPLP terbaik (terendah). Prosedur penambatan senyawa modifikasi struktur emodin direplikasi lima kali, sehingga diperoleh lima pose terbaik.
2.3.3.Post-docking analysis
Hasil penambatan berupa skor ChemPLP dan PLIF bitstring yang dimasukan pada decision tree melalui metode RPART dengan aplikasi statistik R versi 3.2.1 (R Foundation, 2015). Data hasil analisis dari decision tree akan memperlihatkan senyawa modifikasi struktur emodin aktif sebagai
ligan atau tidak aktif. Jika hasil analisis senyawa modifikasi emodin adalah ligan aktif, maka akan dilanjutkan dengan analisis diskoneksi, tetapi jika ligan inaktif, maka prosedur diulang dari inspeksi visual dan preparasi desain senyawa modifikasi struktur emodin hingga didapatkan ligan aktif. Pose dari senyawa modifikasi emodin dilihat interaksinya dengan RE-α menggunakan PyMOL1.2 (Lill dan Danielson, 2011).
2.3.4.Analisis hasil
6
tidaknya suatu pose. Dengan taraf kepercayaan 95%, data bitstring diolah untuk menentukan apakah senyawa uji aktif sebagai ligan pada RE-α.
Visualisasi pose dengan PyMOL1.2 (Lill dan Danielson, 2011) dilakukan dengan memilih pose dengan kriteria:
1. Pose dengan bitstring 320 aktif dan skor ChemPLP terkecil
2. Pose dengan skor ChemPLP terendah
3. Tidak ada pose yang aktif pada bitstring 320, sehingga dipilih pose dengan ChemPLP terkecil saja
Tabel II. PLIF bitstring yang penting menurut decision tree (Istyastono, 2015)
Nomor Bitstring
Residu
Terkait Jenis Interaksi
320 GLY420 Ikatan hidrogen (protein sebagai akseptor) 242 ARG394 Ikatan hidrogen (protein sebagai donor) 117 GLU353 Ikatan hidrogen (protein sebagai akseptor) 411 GLY521 Ikatan hidrogen (protein sebagai akseptor) 473 CYS530 Ikatan hidrogen (protein sebagai donor) 105 ASP351 Interaksi elektrostatik (protein sebagai anion) 201 LEU387 Ikatan hidrogen (protein sebagai akseptor) 470 CYS530 Interaksi non polar
170 TRP383 Aromatik face-to-face 171 TRP383 Aromatik edge-to-face 323 MET421 Interaksi non polar
2.3.5.Analisis diskoneksi dan penentuan jalur sintesis
7
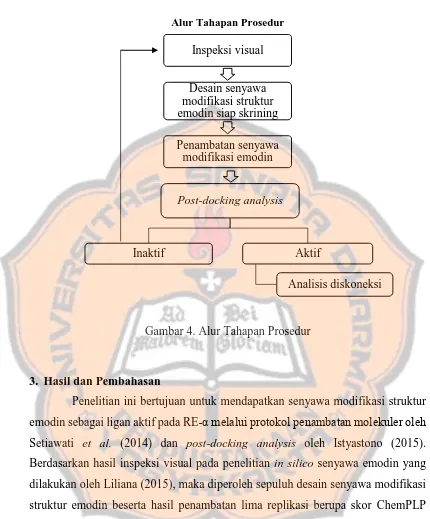
Alur Tahapan Prosedur
Gambar 4. Alur Tahapan Prosedur
3. Hasil dan Pembahasan
Penelitian ini bertujuan untuk mendapatkan senyawa modifikasi struktur emodin sebagai ligan aktif pada RE-α melalui protokol penambatan molekuler oleh Setiawati et al. (2014) dan post-docking analysis oleh Istyastono (2015). Berdasarkan hasil inspeksi visual pada penelitian in silico senyawa emodin yang dilakukan oleh Liliana (2015), maka diperoleh sepuluh desain senyawa modifikasi struktur emodin beserta hasil penambatan lima replikasi berupa skor ChemPLP yang ditampilkan pada Gambar 5 dan PLIF bitstring pada tabel IV.
Inspeksi visual
Desain senyawa modifikasi struktur emodin siap skrining
Penambatan senyawa modifikasi emodin
Post-docking analysis
Inaktif Aktif
8
9
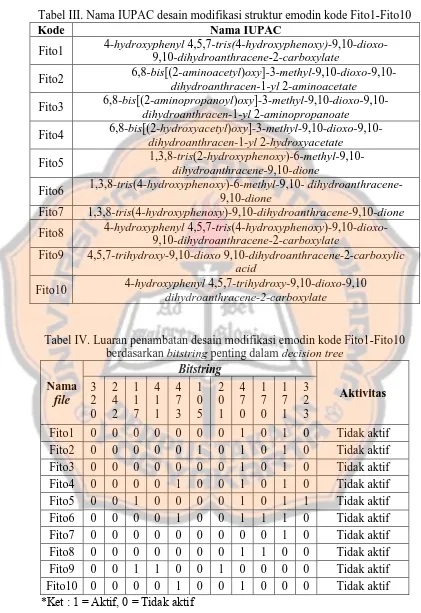
Tabel III. Nama IUPAC desain modifikasi struktur emodin kode Fito1-Fito10
Kode Nama IUPAC
Tabel IV. Luaran penambatan desain modifikasi emodin kode Fito1-Fito10 berdasarkan bitstring penting dalam decision tree
10
Hasil penambatan kesepuluh desain modifikasi struktur emodin menggunakan protokol Setiawati et al. (2014) dan post-docking analysis oleh Istyastono (2015) dengan taraf kepercayaan 95% menggunakan metode RPART menunjukkan desain tersebut inaktif terhadap RE-α.
Berdasarkan penelitian Herlynda (2016), diperoleh desain senyawa ligan aktif terhadap RE-α dengan memodifikasi senyawa 2,6-dihydroxyanthraquinone yang juga menggunakan protokol Setiawati et al. (2014) dan post-docking analysis oleh Istyastono (2015). Pada penelitian tersebut, gugus hidroksil dari senyawa 2,6-dihydroxyanthraquinone diganti dengan gugus eter yang berikatan dengan fenol.
Struktur senyawa 2,6-dihydroxyanthraquinone merupakan golongan yang sama dengan emodin yaitu golongan anthraquinone. Oleh karena itu, mengacu pada penelitian Herlynda (2016) dibuat tujuh belas desain modifikasi struktur dengan mengganti gugus fenol dengan gugus aromatis heterosiklik yang berikatan dengan hidroksil yang ditunjukkan pada Gambar 6 dan Gambar 7 beserta hasil penambatan berupa skor ChemPLP dan PLIF bitstring pada tabel VI. Senyawa aromatis mempunyai interaksi yang lebih baik dari senyawa alifatis karena bentuk aromatis yang flat sehingga mempunyai interaksi yang baik dengan hydrophobic binding region sedangkan senyawa alifatis berbentuk bulky sehingga mempunyai interaksi
yang buruk dengan hydrophobic binding region.
11
12
Tabel V. Nama IUPAC desain modifikasi struktur emodin kode Fito11-Fito27
13
Tabel VI. Luaran penambatan desain modifikasi emodin II berdasarkan bitstring penting dalam decision tree
Hasil penambatan tujuh belas desain modifikasi struktur emodin menggunakan protokol Setiawati et al. (2014) dan post-docking analysis oleh Istyastono (2015) dengan taraf kepercayaan 95% menggunakan metode RPART terdapat enam desain senyawa yang merupakan ligan aktif pada kantung ikatan
RE-α. Desain senyawa yang merupakan ligan aktif adalah desain kode Fito11, Fito12,
14
Desain Fito11, Fto12, Fito14, Fito17, Fito24, Fito25 mempunyai skor
ChemPLP terendah secara berturut-turut yaitu -94,6361; -93,1833; -89,3156; -91,4631; -92,2775; dan -92,7708. Seluruh desain tersebut juga mempunyai alur
pada decision tree yang sama yaitu aktif pada bitstring 242 (ARG394) dan 201 (LEU387) serta mempunyai skor ChemPLP <-85,23 yang ditunjukkan pada gambar 8.
Gambar 8. Alur decision tree yang dilalui desain kode Fito11, Fito12, Fito14, Fito17, Fito24, dan Fito25
15
Tabel VII. Skor ChemPLP lima replikasi desain modifikasi struktur aktif pada RE-α
Replikasi Skor ChemPLP
Fito11 Fito12 Fito14 Fito17 Fito24 Fito25 1 -94,6361 -93,0925 -89,2704 -91,4571 -92,1898 -92,7270 2 -94,6071 -92,7575 -89,2766 -91,4631 -92,2041 -92,7071 3 -94,6210 -93,1833 -89,3156 -91,4428 -92,2510 -92,6420 4 -94,5906 -92,5864 -89,3055 -91,4588 -91,6777 -91,7708 5 -94,6067 -92,7191 -89,2977 -91,4533 -92,2775 -92,7385
Visualisasi pose Fito11 dalam kantung ikatan RE-α menunjukkan ikatan hidrogen dengan residu ARG394 yang bertindak sebagai donor hidrogen. Ikatan hidrogen dengan backbone tampak yaitu dengan LEU387 yang bertindak sebagai akseptor hidrogen. Fito11 berinteraksi non polar dengan CYS530, TRP383, dan LEU387. Fito11 juga berinteraksi secara aromatik edge-to-face dengan TRP383. Pose Fito11 dari lima replikasi dalam kantung ikatan RE-α menunjukkan interaksi yang sama (Gambar 9).
Visualisasi pose Fito12 dalam kantung ikatan RE-α menunjukkan ikatan hidrogen dengan residu ARG394 yang bertindak sebagai donor hidrogen dan GLU353 yang bertindak sebagai akseptor hidrogen. Ikatan hidrogen dengan backbone tampak yaitu dengan LEU387 yang bertindak sebagai akseptor hidrogen.
Fito12 berinteraksi secara non polar dengan TRP383 dan LEU387. Fito12 juga berinteraksi secara aromatik edge-to-face dengan TRP383. Pose Fito12 dari lima replikasi dalam kantung ikatan RE-α menunjukkan interaksi yang sama (Gambar10).
16
Visualisasi pose Fito17 dalam kantung ikatan RE-α menunjukkan ikatan hidrogen dengan residu ARG394 yang bertindak sebagai donor hidrogen. Ikatan hidrogen backbone tampak yaitu dengan LEU387 yang bertindak sebagai akseptor hidrogen dan MET528 yang bertindak sebagai akseptor hidrogen. Interaksi non polar juga tampak yaitu dengan LEU387, TRP383, dan CYS530. Fito17 juga berinteraksi secara aromatik edge-to-face dengan TRP383. Pose Fito17 dari lima replikasi dalam kantung ikatan RE-α menunjukkan interaksi yang sama (Gambar12).
Pose Fito24 dalam kantung ikatan RE-α menunjukkan ikatan hidrogen dengan residu ARG394 yang bertindak sebagai donor hidrogen. Ikatan hidrogen dengan backbone tampak yaitu dengan LEU387 dan LEU525 yang keduanya bertindak sebagai akseptor hidrogen. Interaksi non polar juga tampak yaitu dengan LEU387, CYS530, dan TRP383. Fito24 juga berinteraksi secara aromatik edge-to-face dengan TRP383. Pose Fito24 dari lima replikasi dalam kantung ikatan RE-α
menunjukkan interaksi yang sama (Gambar13).
Pose Fito25 dalam kantung ikatan RE-α menunjukkan ikatan hidrogen dengan residu ARG394 yang bertindak sebagai donor hidrogen. Ikatan hidrogen dengan backbone juga tampak yaitu dengan LEU387 dan MET528 yang keduanya bertindak sebagai akseptor hidrogen. Interaksi non polar juga tampak yaitu dengan LEU387, CYS530, dan TRP383. Fito25 juga berinteraksi secara aromatik edge-to-face dengan TRP383. Pose Fito25 dari lima replikasi dalam kantung ikatan RE-α
17
(a) (b)
Gambar 9. (a) Visualisasi replikasi pose desain Fito11 pada kantung ikatan RE-α. (b) Visualisasi pose desain Fito11 pada kantung ikatan RE-α
(a) (b)
Gambar 10. (a) Visualisasi replikasi pose desain Fito12 pada kantung ikatan
18
(a) (b)
Gambar 11. (a) Visualisasi replikasi pose desain Fito14 pada kantung ikatan
RE-α. (b) Visualisasi pose desain Fito14 pada kantung ikatan RE-α
(a) (b)
Gambar 12. (a) Visualisasi replikasi pose desain Fito17 pada kantung ikatan
19
(a) (b)
Gambar 13. (a) Visualisasi replikasi pose desain Fito24 pada kantung ikatan
RE-α. (b) Visualisasi pose desain Fito24 pada kantung ikatan RE-α
(a) (b)
Gambar 14. (a) Visualisasi replikasi pose desain Fito25 pada kantung ikatan
RE-α. (b) Visualisasi pose desain Fito25 pada kantung ikatan RE-α
20
didiskoneksi dengan memotong ikatan antara atom O gugus eter dengan atom C dari piridin sehingga diperoleh 2,6-dihydroxyanthraquinone dan 2-chloro-5-hydroxypiridine sebagai starting material yang tersedia di Sigma Aldrich (Gambar
15). Berdasarkan analisis diskoneksi tersebut, maka dibuat usulan rute sintesis senyawa Fito11 dengan reaksi substitusi nukleofilik aromatis antara 2,6-dihydroxyanthraquinone dan 2-chloro-5-hydroxypiridine menggunakan katalis
NaOH (Gambar16).
Gambar 15. Mekanisme diskoneksi desain Fito11
Gambar 16. Mekanisme reaksi sintesis desain Fito11
21
dan 4-chloro-1H-pyrrol-2-ol sebagai starting material (Gambar17). Berdasarkan analisis diskoneksi tersebut, tidak ditemukan starting material yang tersedia, maka tidak ada usulan jalur sintesis Fito12.
Gambar 17. Mekanisme diskoneksi desain Fito12
Struktur Fito14 didiskoneksi dengan memotong ikatan antara atom O gugus eter dengan atom C dari pyrrazine sehingga diperoleh 2,6-dihydroxyanthraquinone dan 2-bromo-5-hydroxypyrrazine sebagai starting material yang tersedia di Sigma Aldrich (Gambar 18). Usulan rute sintesis senyawa Fito14 yaitu dengan reaksi substitusi nukleofilik aromatis antara 2,6-dihydroxyanthraquinone dan 2-bromo-5-hydroxypirazine dengan katalis NaOH (Gambar19).
22
Gambar 19. Mekanisme reaksi sintesis desain Fito14
Struktur Fito17 didiskoneksi dengan memotong ikatan antara atom O gugus eter dengan atom C dari thiophene sehingga diperoleh 2,6-dihydroxyanthraquinone dan 5-Chlorothiophene-2-ol sebagai starting material (Gambar20). Berdasarkan analisis diskoneksi tersebut, tidak ditemukan starting material yang tersedia, maka tidak ada usulan jalur sintesis Fito17.
23
Struktur Fito24 didiskoneksi dengan memotong ikatan antara atom O gugus eter dengan atom C dari thiophene sehingga diperoleh 2,6-dihydroxyanthraquinone dan 5-Chlorothiophene-2-carboxylic acid sebagai starting material yang tersedia di Sigma Aldrich (Gambar 21). Usulan rute sintesis senyawa Fito24 menggunakan dua tahap yaitu reaksi reduksi 5-Chlorothiophene-2-carboxylic acid menjadi (5-chlorothiophen-2-yl)methanol kemudian dilanjutkan substitusi nukleofilik aromatis
antara 2,6-dihydroxyanthraquinone dan (5-chlorothiophen-2-yl)methanol dengan katalis NaOH. (Gambar22).
24
Gambar 22. Mekanisme reaksi sintesis desain Fito24
Struktur Fito25 didiskoneksi dengan memotong ikatan antara atom O gugus eter dengan atom C dari thiazole sehingga diperoleh sinton 2,6-dihydroxyanthraquinone dan 2-Chlorothiazole-4-carboxylic acid sebagai starting
material yang tersedia di Sigma Aldrich (Gambar 23). Usulan rute sintesis senyawa
Fito24 menggunakan dua tahap yaitu reaksi reduksi 2-Chlorothiazole-4-carboxylic acid menjadi (2-chloro-1,3-thiazol-4-yl)methanol kemudian dilanjutkan substitusi
25
Gambar 23. Mekanisme diskoneksi desain Fito25
26
4. Kesimpulan
Berdasarkan hasil penambatan dan post-docking analysis terdapat enam desain modifikasi struktur emodin yang merupakan ligan aktif pada RE-α menggunakan protokol Setiawati et al. (2014) dan protokol post-docking analysis Istyastono (2015) yaitu desain Fito11 (2,6-bis[(5-hydroxypyridin-2yl)]-9,10-dihydroanthracene-9,10-dione), Fito12
(2,6-bis[(5-hydroxy-1H-pyrrol-3-yl)oxy]-9,10-dihydroanthracene-9,10-dione), Fito14 (2,6-bis[(5-hydroxypyrazin-2-yl)oxy]-9,10-dihydroanthracene-9,10-dione), Fito17 (2,6-bis[(5-hydroxythiophen-2-yl)oxy]-9,10-dihydroanthracene-9,10-dione), Fito24 (2,6-bis({[5-(hydroxymethyl)
thiophen-2-yl]oxy})-9,10-dihydroanthracene-9,10-dione), dan Fito25
27
DAFTAR PUSTAKA
American Cancer Society, 2014, Breast Cancer, American Cancer Society, USA. Anita, Y., Radifar, M., Kardono, L. B. S., Hanafi, M., dan Istyastono, E. P., 2012,
Structure-Based Design of Eugenol Analogs as Potential Estrogen Receptors Antagonist, Biomedical Informatics, 8 (19), pp. 901-906. Brink, T. T., dan Exner, T. E., 2009, Influence of Protonation, Tautomeric, and
Stereoisomeric States on Protein-ligand Docking Result, Journal of Chemical Information and Modeling, 55 (9), pp. 1962 – 1972.
Hayashi, S., Eguchi, H., Tanimoto, K., Yoshida, T., Omoto, Y., Inoue, A., et al., 2003, The Expression and Function of Estrogen Receptor α and β in Human Breast Cancer and Its Clinical Application, Endocrine-Related Cancer, 10, pp. 193-202.
Herlynda, K. S., 2016, Desain Teoretis Berbantukan Komputer Modifikasi Struktur 2,6-dihydroxyanthraquinone sebagai Ligan Reseptor Estrogen Alfa, Skripsi. Hal. 8-11.
Huang, P. H., Huang. C. Y., Chen, M. C., Lee, Y. T., Yue, C. H., Wang, H. Y., et al., 2013, Emodin and Aloe-Emodin Suppress Breast Cancer Cell
Proliferation through ERα Inhibition, Evidence-Based Complementary and Alternative Medicine. pp. 89-91.
Istyastono, E. P., 2015, Employing Recursive Partition and Regression Tree Method to Increase the Quality of Structure-Based Virtual Screening in the Estrogen Receptor Alpha Ligands Identification, Asian Journal of Pharmaceutical and Clinical Research, 8 (6), pp. 207-209.
Istyastono, E. P., Anita, Y., dan Sundowo, A., 2014, Computer-aided Structure-based Design of 3,3'-Diallyl-[1,1'-biphenyl]-4,4'-diol Analogs of Eugenol as Potential Ligands for Estrogen Receptor Alpha, 3rd International Conference on Computation for Science and Technology, pp. 89-91. Izhaki, dan Ida., 2002, Emodin – a Secondary Metabolite with Multiple Ecological
Functions in Higher, New Phytologist, 155, pp. 205-217.
Korb, O., Stutzle, T., dan Exner, T.E., 2009, Empirical Scoring Function for Advanced Protein-ligand Docking with PLANTS, Journal of Chemical Information and Modeling, 49 (1), pp. 84-98.
Liliana, 2015, Uji In Silico Senyawa Emodin sebagai Ligan Reseptor Estrogen Alfa, Skripsi, hal. 15-17.
Lill, M. A., dan Danielson, M. L., 2011, Computer-aided Drug Design Platform Using PyMOL, Journal of Computer-Aided Molecuular Design, 25 (1), pp. 13-19.
Pubchem, 2016, Emodin, https://pubchem.ncbi.nlm.nih.gov/compound/emodin, diakses tanggal 10 Januari 2016.
R Foundation, 2015, R 3.2.1 is released, http://www.r-statistics.com/ diakses tanggal 14 Januari 2016.
28
Salazar, M.D., Ratnam, M., Patki, M., Kisovic, I., Trumbly, R., dan Iman, M., 2011, During Hormone Depletion or Tamoxifen Treatment of Breast Cancer Cells the Estrogen Receptor Apoprotein Supports Cell Cycling through The Retinoic Acid Receptor a1 Apoprotein, Breast Camcer Research. Setiawati, A., Riswanto, F.D.O., Yuliani, S.H., dan Istyastono, E.P., 2014,
Retrospective Validation of Structure-Based Virtual Screening Protocol to Identify Ligands for Estrogen Receptor Alpha and Its Application to Identify the Alpha-Mangostin Binding Pose, Indonesian Journal of Chemistry, 14 (2), pp. 103-108.
Suganya, J., Radha, M., Naorem, D. L., dan Nishandhini, M., 2014, In Silico Docking Studies of Selected Flavonoids – Natural Healing Agents againts Breast Cancer, Asian Pacific Journal of Cancer Prevention, 15 (19), pp. 8155-8159.
World Health Organization, 2014, Indonesia, World Health Organization, http://www.who.int/cancer/country-profiles/idn_en.pdf?ua=1, diakses tanggal 10 Januari 2016.
World Health Organization, 2015, Cancer, World Health Organization, http://www.who.int/mediacentre/factsheets/fs297/en/, diakses tanggal 10 Januari 2016.
Xing, J.Y., Song, G.P., Deng, J.P., Jiang, L.Z., Xiong, P., Yang, B.J., et al., 2015, Antitumor Effects and Mechanism of Novel Emodin Rhamnoside Derivates against Human Cancer Cells In Vitro, PLOS ONE, 10 (12). Yaoxian, W., Hui, Y., Yunyan, Z., Yangin, L., Xin, G., dan Xiaoke, W., 2013,
29
30
Lampiran1. Desain modifikasi struktur emodin menggunakan BKChem0.13.0
31
Lampiran3. Perintah penambatan berupa file shell script dengan nama file clin.sh
32
Lampiran5. Upload file shell script dan struktur 3D desain modifikasi struktur emodin
33
34
35
Lampiran 9. Analisis hasil penambatan dengan aplikasi statistik R
36
Lampiran 11. Input ligan pada PyMOL
37
Lampiran13. Input protein binding site
38
BIOGRAFI PENULIS
Penulis skripsi berjudul “Desain Teoretis berbantukan
Komputer Modifikasi Struktur Emodin sebagai Ligan Reseptor Estrogen Alfa” bernama Pius Pradana Bekti
Indramawan. Lahir di Purworejo pada 21 Agustus 1994 dari pasangan FX. Budi Susanto dan Th. Endartri Kusuma Astuti sebagai anak pertama dari dua bersaudara. Penulis menempuh pendidikan formal yang dimulai dari TK Strada Bhakti Utama Jakarta (1999-2000), SD Strada Bhakti Utama Jakarta (2000-2006), SMP Strada Bhakti Utama Jakarta (2006-2009), SMA Bruderan Purworejo (2009-2012).
Dalam masa kuliah, penulis berperan aktif dalam kegiatan Herbal Garden Team (2012-2013), PSF Veronica (2012), Desa Mitra IV (2012),
Pelepasan Wisuda Program Studi Farmasi (2012), Pekan Suci Paskah Campus Ministry (2013), Latihan Kepemimpinan (2013), UKF Squadra Viola (2012-2015),