HUBUNGAN METILASI DNA DAN EKSPRESI GEN
MADS BOX DENGAN BUAH MANTEL PADA TANAMAN
KLONAL KELAPA SAWIT (Elaeis guineensis Jacq.)
MAHARANI ANISCHAN
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2013
Dengan ini saya menyatakan bahwa Tesis yang berjudul Hubungan Metilasi DNA dan Ekspresi Gen MADS-box dengan Buah Mantel pada Tanaman Klonal Kelapa Sawit (Elaeis guineensis Jacq.) adalah karya saya bersama komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir Tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, November 2013
Maharani Anischan NRP P051100161
RINGKASAN
MAHARANI ANISCHAN. Hubungan Metilasi DNA dan Ekspresi Gen MADS-box dengan Buah Mantel pada Tanaman Klonal Kelapa Sawit (Elaeis guineensis Jacq.). Dibimbing oleh SUHARSONO dan NURITA TORUAN-MATHIUS.
Buah mantel pada tanaman klonal kelapa sawit skala besar sangat berpengaruh terhadap produktivitas minyak. Fenotip mantel disebabkan oleh perubahan epigenetik yang melibatkan metilasi DNA dan gen MADS-box yang menyandikan transformasi homeotik organ reproduksi. Penelitian ini bertujuan untuk memanfaatkan teknik Ultra Performance Liquid Chromatography (UPLC) untuk mengetahui tingkat metilasi yang menentukan abnormalitas buah dan Quantitative Real Time PCR (qPCR) untuk mengetahui hubungan abnormalitas buah dengan ekspresi gen MADS-box.
Penelitian ini terdiri dari dua percobaan, yaitu analisis metilasi DNA dan analisis ekspresi gen MADS-box. Bahan yang digunakan adalah buah muda normal dan buah muda mantel dengan abnormalitas sangat berat. Buah mantel dikelompokkan menjadi dua, yaitu buah mantel yang terdapat di dalam tandan (Abn m) dan buah mantel yang terdapat di luar tandan (Abn).
Analisis ekspresi gen dilakukan dengan melakukan analisis mRNA dan analisis metilasi dilakukan terhadap DNA genom total. Untuk analisis ekspresi gen, mRNA digunakan sebagai cetakan untuk mensintesis cDNA. Masing-masing percobaan dilaksanakan dengan dua kali ulangan, untuk setiap gen dan cDNA. Ekspresi dari gen target dibandingkan dengan EF1-α1 sebagai gen pembanding.
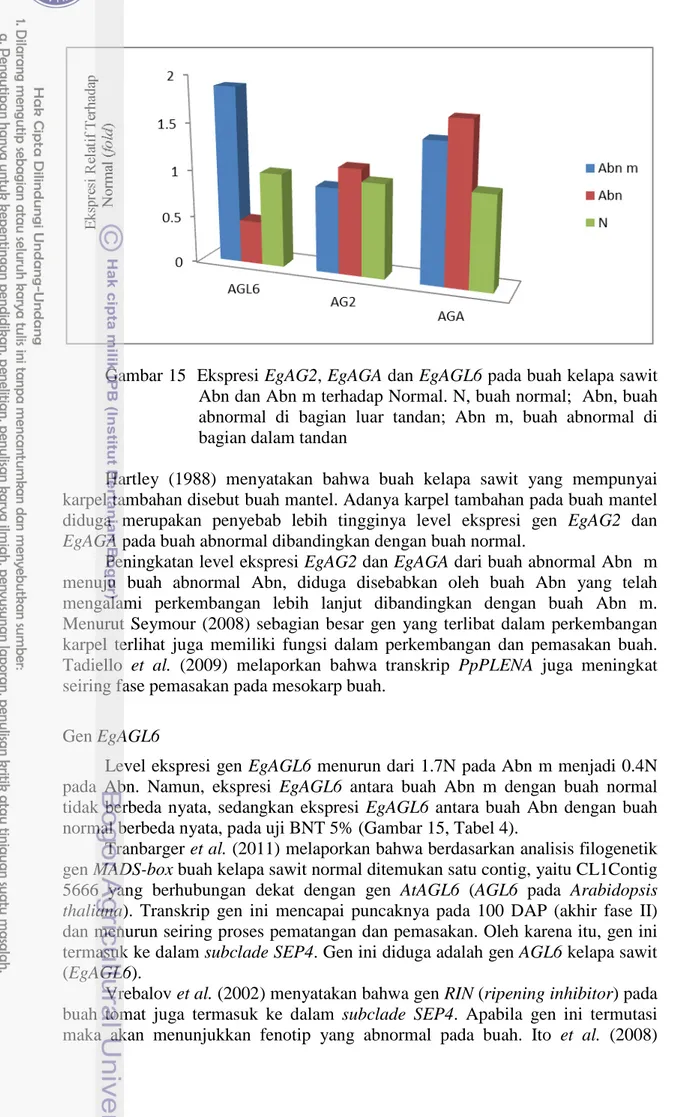
Berdasarkan efisiensi qPCR dengan membandingkan gen target dan gen pembanding, dari 11 gen yang diuji, yaitu: EgSQUA1, EgSQUA3, EgFUL, EgGLO1, EgGLO2, EgDEF1, EgAG2, EgAGA, EgAGL6, EgF-box dan EgRING, hanya tiga gen, yaitu EgAG2, EgAGA dan EgAGL6 yang memenuhi syarat untuk analisis qPCR. Ekspresi gen EgAG2 tidak berbeda antara buah mantel dan buah normal, sedangkan ekspresi gen EgAGA dan EgAGL6 antara buah mantel dan buah normal berbeda. Ekspresi gen EgAGA lebih tinggi di buah mantel daripada di buah normal, sedangkan ekspresi gen EgAGL6 lebih rendah di buah mantel tua daripada di buah mantel muda dan buah normal.
Pemotongan DNA genom dengan enzim S1 Nuklease dan UPLC menunjukkan adanya hipermetilasi pada buah mantel sebesar 18.33-19.55% dibandingkan dengan buah normal sebesar 5.67%. Peningkatan level metilasi salah satunya ditunjukkan oleh penurunan yang nyata dari level ekspresi gen EgAGL6 pada buah mantel. Gen ini diduga terlibat dalam perkembangan buah mantel.
Kata kunci: metilasi DNA, gen MADS-box, buah mantel, quantitative real-time PCR
MAHARANI ANISCHAN. Correlation between DNA Methylation and Expression of the MADS-box Genes on Mantled Fruit of Oil Palm (Elaeis guineensis Jacq.) Clonal Plants. Supervised by SUHARSONO and NURITA TORUAN-MATHIUS.
The presence of abnormal mantled fruit on large scale clonal production of oil palm had trully affected the oil productivity. Mantled phenotype is likely to be that of an epigenetic change involving DNA methylation and the MADS-box transcription factor genes which encoded floral organ homeotic transformation. We developed an Ultra Performance Liquid Chromatography (UPLC) to quantify the degree of methylation related to fruit abnormality and Quantitative Real-Time PCR (qPCR) to know the correlation between the expression of the MADS-box genes with mantled fruit abnormality.
This study were consist of two experiments, the DNA methylation analysis and the MADS-box genes expression analysis. Materials used in this study were the young fruit of both normal and mantled with very heavy type of abnormality. Abnormal mantled fruit were grouped into two types, one is taken from inside (Abn m) and other is from outside the bunch (Abn).
In gene expression analysis, cDNA used as the template for qPCR were obtained from the reverse transcription of the mRNA, while methylation analysis performed on total DNA genomic. The research for each gene and cDNA were conducted in two replications. The expression of the target genes were compared to EF1-α1 as the reference gene.
Based on qPCR efficiency by comparing the target genes with the reference gene, from 11 genes that have been tested, i.e EgSQUA1, EgSQUA3, EgFUL, EgGLO1, EgGLO2, EgDEF1, EgAG2, EgAGA, EgAGL6, EgF-box and EgRING, only three genes are qualified for qPCR analysis, i.e EgAG2, EgAGA and EgAGL6. EgAG2 expression was not different in both mantled and normal fruit, while EgAGA expression was higher in mantled and EgAGL6 expression was significantly lower in outside-bunched mantled fruit (Abn).
Nuclease S1 digestion and UPLC revealed the genome-wide increasing in DNA methylation on mantled fruit (18.33-19.55%) compared to its normal counterparts (5.67%). This increased in global DNA methylation showed by the significant decreased in EgAGL6 transcript level on mantled fruit. This gene is assumed to be involved in the development of mantled fruit.
Key words: DNA methylation, MADS-box genes, mantled fruit, quantitative real-time PCR.
© Hak Cipta milik IPB, tahun 2013
Hak Cipta dilindungi Undang-undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; pengutipan tersebut tidak merugikan kepentingan IPB
Dilarang mengumumkan dan memperbanyak sebagian atau seluruh Karya Tulis dalam bentuk apapun tanpa izin IPB
HUBUNGAN METILASI DNA DAN EKSPRESI GEN
MADS-BOX DENGAN BUAH MANTEL PADA TANAMAN
KLONAL KELAPA SAWIT (Elaeis guineensis Jacq.)
MAHARANI ANISCHAN
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Bioteknologi
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN
BOGOR
2013
Nama : Maharani Anischan
NIM : P051100161
Disetujui oleh, Komisi Pembimbing
Prof Dr Ir Suharsono, DEA
Ketua Anggota
Dr Ir Nurita Toruan-Mathius, MS
Diketahui oleh,
Ketua Program Studi Dekan Sekolah Pascasarjana Bioteknologi
Prof Dr Ir Suharsono, DEA Dr Ir Dahrul Syah, MSc Agr Tanggal Ujian:23 Agustus 2013 Tanggal Lulus:
Judul Penelitian Hubungan Metilasi DNA dan Ekspresi Oen MADS-box dengan Buah Mantel pada Tanaman Kional Kelapa Sawit (Elaeis guineensis Jacq.)
Nama Maharani Anischan
NIM P051100161
Disetujui oIeh, Komisi Pembimbing
rita Toruan-Mathius MS Anggota
Prof Dr Ir Suharsono, DEA Ketua
Diketahui oIeh,
Ketua Program Studi Bioteknologi
Prof Dr Ir Suharsono, DEA
Tanggal Ujian:23 Agustus 2013 Tanggal Lulus:
2
2 NO
J1013
Puji serta syukur penulis ucapkan kehadirat Allah SWT karena berkat Rahmat dan Hidayah-Nya penulis dapat menyelesaikan penelitian dengan judul “Hubungan Metilasi DNA dan Ekspresi Gen MADS-box dengan Buah Mantel pada Tanaman Klonal Kelapa Sawit (Elaeis guineensis Jacq.)”.
Ucapan terimakasih penulis sampaikan kepada:
1. Prof Dr Ir Suharsono, DEA sebagai ketua komisi pembimbing dan Dr Nurita Toruan-Mathius, MS sebagai anggota komisi pembimbing yang telah memberikan bimbingan, arahan dan ilmunya kepada penulis.
2. Dr Ir Aris Tjahjoleksono, DEA selaku dosen penguji luar komisi yang telah memberikan saran dan masukan demi kesempurnaan tesis ini.
3. Bakrie Center Foundation yang telah memberikan beasiswa pendidikan, hingga penulis dapat menyelesaikan studi pada program Pascasarjana Bioteknologi di IPB.
4. PT. SMART, Tbk. yang telah memberikan dana dan fasilitas untuk pelaksanaan penelitian ini.
5. Andree Sunanjaya Kusnandar, S.Si yang telah memberikan kesempatan kepada penulis untuk melaksanakan sebagian dari proyek penelitiannya. 6. Keluarga besar laboratorium Genomik dan Transkriptomik, PT. SMART,
Tbk. di Bogor yang telah banyak memberikan bantuan atas kelancaran penelitian ini.
7. Kedua orangtua beserta kakak-kakak yang telah memberikan begitu banyak perhatian, dukungan dan do’anya kepada penulis.
8. Segenap sahabat mahasiswa S2 Bioteknologi IPB angkatan 2010 yang telah banyak memberikan motivasi dan bantuannya.
9. Segenap karyawan serta staff administrasi Program Studi Bioteknologi IPB, yang telah banyak membantu penulis dalam proses administrasi.
Serta semua pihak yang telah banyak memberikan motivasi, dan dukungannya serta menjadi inspirasi bagi penulis.
Semoga karya ilmiah ini bermanfaat.
Bogor, November 2013 Maharani Anischan
DAFTAR ISI Halaman DAFTAR TABEL ix DAFTAR GAMBAR x DAFTAR LAMPIRAN x 1 PENDAHULUAN 1 Latar Belakang 1 Tujuan Penelitian 2 2 TINJAUAN PUSTAKA 3
Tanaman Kelapa Sawit 3
Faktor Transkripsi 7
Gen MADS-box 8
Metilasi DNA dan Ekspresi Gen 10
3 METODOLOGI 15
Waktu dan Tempat Penelitian 15
Bahan 15
Prosedur Kerja 15
4 HASIL DAN PEMBAHASAN 23
Isolasi RNA Total 23
Sintesis cDNA Utas Tunggal 24
Penentuan Ekspresi Gen dengan qPCR 25
Isolasi DNA Genomik 32
Pencacahan DNA Genomik dengan Enzim S1 Nuklease 33
Kuantifikasi Metilasi dengan UPLC 34
Hubungan Metilasi DNA dengan Ekspresi Gen 35
5 SIMPULAN DAN SARAN 37
Simpulan 37
Saran 37
DAFTAR PUSTAKA 38
LAMPIRAN 45
DAFTAR RIWAYAT HIDUP 51
DAFTAR TABEL
Halaman 1 Komposisi buah Dura, Tenera dan Pisifera 4 2 Rasio absorbansi RNA total serta Rasio 28 S:18S rRNA 24
3 Validasi qPCR Gen MADS-box 25
4 Perhitungan BNT EgAGL6, EgAG2 dan EgAGA 29
1 Buah kelapa sawit 4 2 Potongan melintang pada buah kelapa sawit regeneran 6
3 Domain faktor transkripsi 7
4 Struktur domain MIKC pada gen MADS-box 8
5 Model penentuan identitas struktur bunga berdasarkan ekspresi gen
MADS-box yang saling tumpang-tindih 10
6 Metilasi pada basa sitosin 11
7 Proses qPCR yang diamatidari peningkatan sinyal fluoresen 13
8 Alur Penelitian 16
9 RNA Total buah kelapa sawit hasil elektroforesis Qiaxcel 23
10 Hasil amplifikasi EgACT1 24
11 Posisi Primer ActF dan ActR yang didisain dari gen aktin kelapa sawit EgACT1 pada ekson 1 dan ekson 2 dari gen aktin kedelai pSAc3
(Shah et al. 1982) 25
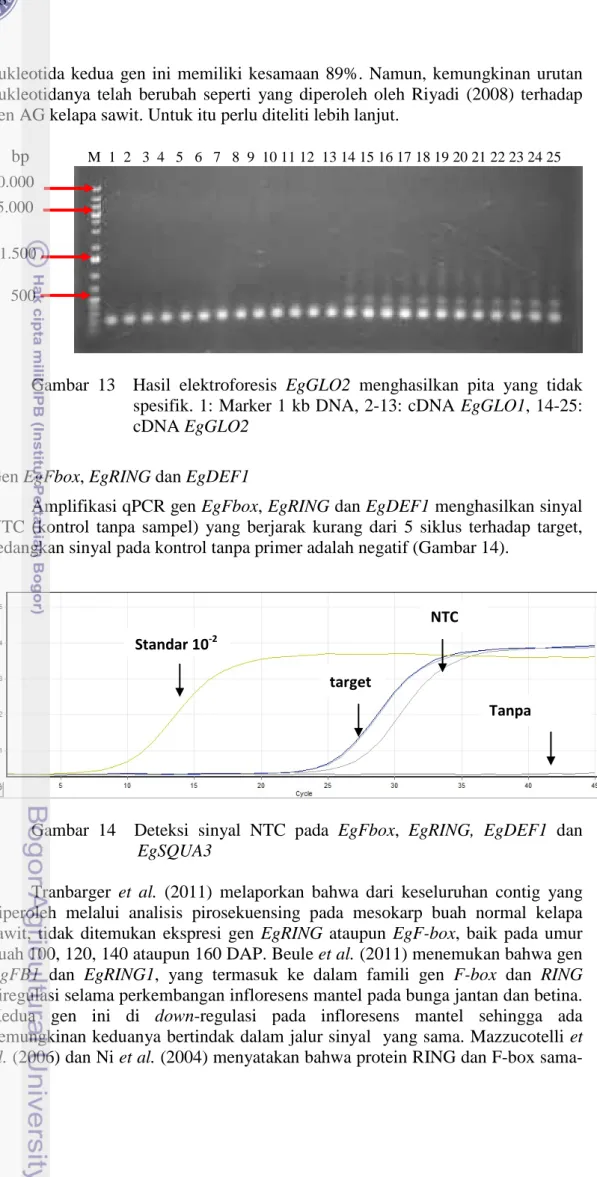
12 Deteksi sinyal EgSQUA1 pada buah kelapa sawit. 26 13 Hasil elektroforesis EgGLO2 menghasilkan pita yang tidak spesifik 28 14 Deteksi sinyal NTC pada EgFbox, EgRING dan EgDEF1 28 15 Ekspresi gen EgAG2, EgAGA dan EgAGL6 pada buah kelapa sawit
Abn dan Abn m terhadap Normal 30
16 Hasil elektroforesis DNA genomik buah kelapa sawit 33 17 Hasil pencacahan DNA genomik oleh enzim Nuclease S1 33 18 Pola peak kandungan metilsitosin (5-mC) pada buah normal dan
abnormal 34
19 Perbandingan kandungan 5-metil-sitidin antara buah normal dan
abnormal 34
20 Hasil Pembacaan Metilasi DNA genom dengan UPLC 50
DAFTAR LAMPIRAN
Halaman
1 Data kurva standar 45
2 Plot ∆CT melawan log amount template kurva standar 47 3 Kesamaan cDNA beberapa gen MADS-box
(Sumber: Tranbarger et al. 2011) 49
PENDAHULUAN
Latar Belakang
Kelapa sawit merupakan tanaman utama perkebunan di Indonesia. Hal ini ditunjukkan oleh peningkatan luasan areal perkebunan dan jumlah ekspor komoditas ini dari tahun ke tahun (BPS 2009). Indonesia juga merupakan negara penghasil minyak sawit terbesar dunia saat ini karena hampir 9 Mha luas areal perkebunan kelapa sawit Indonesia menghasilkan lebih dari 80% total minyak sawit dunia (FAO Statistik 2013).
Salah satu upaya untuk memenuhi kebutuhan produksi adalah penyediaan bibit berkualitas dengan produktivitas tinggi. Hal tersebut dapat dilakukan melalui perbanyakan klonal dari varietas unggul kelapa sawit dengan teknik kultur jaringan. Kendala yang dihadapi dalam pemanfaatan teknologi kultur jaringan adalah timbulnya abnormalitas pada organ reproduktif diantaranya buah mantel yang disebabkan oleh adanya variasi somaklonal (Corley et al. 1986). Pada bunga ditemukan suatu struktur menyerupai karpel yang menggantikan stamen dan berbentuk seperti “pembungkus” pada buah (Adam et al. 2005). Menurut Hetharie (2008) pengaruh buah mantel pada produksi minyak dapat sangat besar, yaitu menurun sekitar 20-30% dibandingkan dengan buah normal. Hal ini disebabkan oleh akumulasi minyak yang sangat sedikit pada mesokarp buah mantel. Bahkan pada kasus buah mantel berat, mesokarp buah berubah menjadi jaringan yang sangat keras.
Variasi somaklonal diartikan sebagai variasi genetik dan fenotipik di antara tanaman-tanaman yang diperbanyak secara klonal dari satu klon donor tunggal. Variasi ini salah satunya melibatkan perubahan ploidi (Kaeppler et al. 2000), namun Rival et al. (1997) tidak menemukan adanya perubahan pada level ploidi antara regeneran tanaman kelapa sawit normal maupun abnormal.
Variasi sekuens juga dapat muncul melalui proses kultur (Kaeppler et al. 2000). Pencarian penanda molekuler untuk fenotip mantel kelapa sawit telah dilakukan melalui analisis RAPD (Rival et al. 1998), AFLP (Matthes et al. 2001) dan RFLP (Jaligot et al. 2002), namun juga belum berhasil menemukan pita DNA spesifik yang dapat membedakan tanaman yang berbuah normal dengan abnormal secara universal.
Rao & Donought (1990) mengamati adanya suatu derajat yang signifikan dari pembalikan fenotip abnormal menjadi normal. Hal ini mengindikasikan adanya epigenetik. Menurut Kaeppler et al. (2000) epigenetik melibatkan perubahan ekspresi gen yang dapat balik (reversible), namun bukan disebabkan oleh aberasi kromosom atau perubahan pada sekuens DNA.
Bollati & Baccarelli (2010) menyatakan bahwa metilasi DNA merupakan salah satu mekanisme yang terlibat dalam epigenetik. Hal ini dibuktikan oleh terdeteksinya global dan sekuens-spesifik hipometilasi DNA melalui analisis HPLC pada kalus (Jaligot et al. 2000) dan MSAP (Methylation-Sensitive Amplified Polymorphism) pada daun (Matthes et al. 2001) regeneran kelapa sawit yang menunjukkan fenotip abnormal. Tregear et al. (2002) menduga bahwa terdapat sejumlah kecil gen tertentu yang sangat sensitif terhadap perubahan metilasi pada kelapa sawit mantel.
Rival et al. (2000) melaporkan bahwa penurunan dalam metilasi DNA ini tidak satupun dapat dijelaskan oleh perubahan dalam level trankripsi dari tiga gen utama Metiltransferase DNA, sehingga gen-gen ini dapat dikatakan tidak terlibat langsung dalam pembentukan fenotip abnormal. Kubis et al. (2003) juga menyatakan bahwa perubahan dalam status metilasi elemen transposable pada regeneran kelapa sawit juga tidak terdeteksi. Oleh sebab itu diperlukan metode lain untuk menerangkan status metilasi DNA.
Fenotip mantel disebabkan oleh modifikasi epigenetik yang mengubah pola ekspresi dari sejumlah gen yang terlibat dalam pembungaan dan perkembangan buah (Shearman et al. 2012). Proses organogenesis bunga (Adam et al. 2007a) dan perkembangan buah (Tranbarger et al. 2011) kelapa sawit didasarkan pada peranan faktor transkripsi, yaitu gen MADS-box. Menurut Elliot & Elliot (2009) metilasi salah satunya dapat terjadi di level faktor transkripsi.
Penelitian ini ditujukan untuk mengetahui hubungan antara abnormalitas buah kelapa sawit akibat metilasi dengan ekspresi beberapa gen MADS-box. Hasil penelitian ini diharapkan dapat memberi informasi mengenai kuantitas metilasi yang menentukan tingkat abnormalitas pada buah serta hubungannya dengan gen MADS-box yang terekspresi pada buah abnormal. Informasi tersebut dapat dimanfaatkan untuk mendapatkan pemecahan permasalahan abnormalitas pada kelapa sawit.
Qualitative Real Time Polymerase Chain Reaction (qPCR) adalah suatu teknik yang paling sensitif untuk deteksi dan kuantifikasi ekspresi gen yang ada pada saat ini (Kubista et al. 2006). Yang et al. (2011) menggunakan metode Ultra Performance Liquid Chromatography (UPLC) untuk menetapkan status metilasi DNA.
Tujuan
Tujuan penelitian ini adalah (i) mengkuantifikasi level ekspresi beberapa gen MADS-box, (ii) menghitung level metilasi DNA genom dan (iii) menerangkan hubungan antara tingkat abnormalitas akibat metilasi dengan ekspresi gen MADS-box yang ada pada buah mantel.
3
TINJAUAN PUSTAKA
Tanaman Kelapa Sawit
Kelapa sawit adalah tanaman monokotil yang termasuk ke dalam famili Arecaceae, tribe Cocoseae, subtribe Elaeidinae dan genus Elaeis. Nama Elaeis diambil dari bahasa Yunani, elaia, untuk olive (buah zaitun), sedangkan nama spesifik guineensis untuk kelapa sawit merujuk kepada pohon yang ditemukan oleh Jacquin di Teluk Guinea (Jacquemard 1998). Saat ini ada tiga spesies Elaeis, yaitu Elaeis guineensis dari Afrika yang dikenal sebagai kelapa sawit, dan dua spesies asli dari Amerika Selatan dan Amerika Tengah, yaitu E. oleifera dan E. odora. Dari ketiga spesies ini E. guineensis merupakan spesies ekonomi paling utama (Corley & Tinker 2003).
Kelapa sawit merupakan penghasil minyak nabati. Minyak dihasilkan dari dua bagian buah, yaitu dari mesokarp disebut crude palm oil (CPO) atau minyak sawit, dan dari kernel disebut kernel palm oil (KPO) (Hartley 1988). Menurut Koh et al. (2009) kelapa sawit Afrika (E. guineensis Jacq.) adalah sumber utama minyak nabati dunia. Hal ini disebabkan oleh minyak yang dihasilkan mencapai lebih dari 6.5 ton per hektar (tertinggi dari semua tanaman oleaginous) serta luasnya areal yang diperuntukkan bagi perkebunannya.
Buah kelapa sawit termasuk jenis buah sesil drupe atau buah berbiji satu yang dibungkus oleh daging buah dari berbagai bentuk mulai dari bulat hingga ovoid atau memanjang. Panjang buah berkisar dari 2 sampai 7 cm. Buah terdiri atas eksokarp (kulit) yang tipis, mesokarp (daging) yang berminyak, endokarp (cangkang) yang keras dan suatu endosperm berminyak (kernel). Endokarp dan kernel merupakan bagian dari biji (Ng et al. 2003). Selain mengandung minyak yang jumlahnya mencapai 80% berat kering buah (Murphy 2009), mesokarp juga mengandung 11-21% materi berserat (Ng et al.2003) serta kaya akan karotenoid. CPO adalah sumber provitamin A terbanyak (Sambanthamurthi et al. 2000).
Secara anatomi, buah didefinisikan sebagai ovari yang telah matang dan terkadang membawa sebagian atau bahkan keseluruhan dari jaringan karpel yang ada pada bunganya (Giovannoni 2004). Perkembangan buah terjadi setelah pembuahan seiring dengan pematangan biji. Proses ini diawali dengan pembesaran buah melalui pembelahan dan penambahan volume sel. Embrio kemudian matang dan biji mengakumulasi cadangan makanan, mengalami desikasi dan kehilangan air, sampai akhirnya buah matang. Proses pematangan disertai dengan perubahan dalam hal rasa, tekstur, warna dan aroma (White 2002). Tranbarger et al. (2011) menyatakan bahwa terdapat lima fase dalam perkembangan mesokarp buah kelapa sawit, yaitu:
1. Pembelahan sel antiklinal dan ekspansinya seiring dengan peningkatan awal dalam berat serta ukuran buah. Fase ini terjadi antara 30-60 DAP (Day After Pollination),
2. Periode transisi yang dikarakterisasi oleh suatu fase lag dalam pengakumulasian berat segar serta puncak akumulasi IAA dan konjugatnya. Fase ini terjadi antara 60-100 DAP,
3. Akhir periode transisi yang ditandai oleh penurunan dalam metabolit auksin, gibberelin dan sitokinin. Fase ini terjadi antara 100-120 DAP,
4. Peningkatan berat segar mesokarp dan awal pengakumulasian lipid yang terjadi seiring dengan permulaan pematangan pada 120 DAP,
5. Akumulasi lipid dan karotenoid serta sejumlah kecil metabolit hormon (auksin, gibberelin, sitokinin atau etilen) yang terjadi antara 120-140 DAP, 6. Merupakan fase pemasakan yang ditandai dengan peningkatan hormon ABA dan etilen serta berat segar dan berat kering buah, pelepasan dinding sel terkait proses pemasakan dalam mesokarp, serta akumulasi lipid dan karotenoid dalam mesokarp. Puncaknya adalah pada 160 DAP, yaitu ketika akumulasi lipid dalam organel subseluler mengisi volume sel dan sel mesokarp telah diisi oleh bagian-bagian yang jelas, seperti kromoplast yang kaya akan karotenoid.
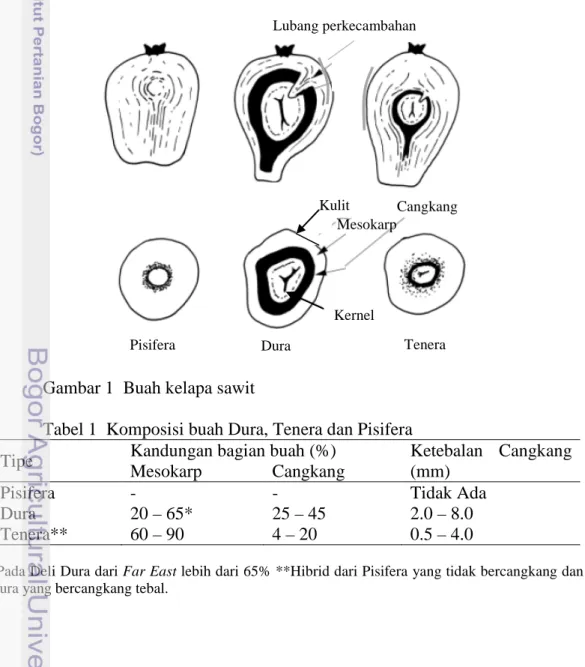
Struktur internal dari buah bervariasi terutama dalam hal ketebalan cangkang, seperti ditunjukkan pada Gambar 1 dan Tabel 1 (Hardon 1955). Menurut Paranjothy (1984) ketebalan cangkang dikendalikan oleh gen tunggal. Tiga tipe buah yang berada di bawah kontrol monogenik dan menjadi dasar untuk pengelompokan minyak sawit, yaitu (1) Dura yang bersifat homozigous (ShSh) dengan cangkang tebal (2-8 mm), (2) Tenera yang bersifat heterozigous (Shsh) dengan cangkang tipis (0.5-4.0 mm) dan (3) Pisifera yang bersifat homozigous (shsh) dengan tanpa cangkang.
Gambar 1 Buah kelapa sawit
Tabel 1 Komposisi buah Dura, Tenera dan Pisifera
Tipe Kandungan bagian buah (%) Ketebalan Cangkang (mm)
Mesokarp Cangkang
Pisifera - - Tidak Ada
Dura 20 – 65* 25 – 45 2.0 – 8.0
Tenera** 60 – 90 4 – 20 0.5 – 4.0
*Pada Deli Dura dari Far East lebih dari 65% **Hibrid dari Pisifera yang tidak bercangkang dan Dura yang bercangkang tebal.
Penampang melintang
Lubang perkecambahan
Pisifera Dura Tenera
Kernel Cangkang E m b Emb E m Mesokarp Kulit
5
Berdasarkan warna buah maka spesies E. guineensis dikelompokkan atas (1) Nigrescens, yaitu buah berwarna ungu hingga hitam di bagian apex dan tidak berwarna di bagian dasar saat umur buah masih muda dan menjadi merah kekuningan (orange) saat matang, (2) Virescens, yaitu buah berwarna hijau saat muda dan menjadi orange saat matang, (3) Albescens, yaitu buah berwarna kuning pucat dan tembus cahaya, karena mengandung sedikit karoten pada mesokarp, dan (4) Poissoni, yaitu sering disebut buah mantel atau buah dengan karpel tambahan atau mempunyai lebih dari satu biji dalam buah (Hartley 1988).
Karakteristik biologis dari kelapa sawit berupa (1) siklus hidup yang panjang (Lubis 1992), (2) tidak memiliki bentuk reproduksi vegetatif alami (Khoo et al. 1999) serta (3) tingginya tingkat heterogenitas di antara hibrid-hibrid kelapa sawit (Asmono et al. 2000). Hal ini menyebabkan strategi pembibitan konvensional akan memakan waktu lama dan membutuhkan perlakuan yang intensif. Propagasi klonal dari tanaman kelapa sawit unggul melalui kultur jaringan telah dikembangkan untuk bibit atau sumber bahan tanam (Khaw et al. 1999).
Menurut Khoo et al. (1999) bibit yang berasal dari klon hasil kultur jaringan menunjukkan hasil lebih baik dari bibit yang berasal dari biji. Lubis (1992) menyatakan bahwa tanaman hasil kultur jaringan menghasilkan jumlah tandan buah lebih banyak, berat tandan lebih tinggi dan waktu produksi yang lebih cepat. Duran-Gasselin et al. (1993) melaporkan bahwa kelapa sawit hasil kultur jaringan meningkatkan produksi minyak sawit mentah (MSM) sebesar 12% sampai 30% dibandingkan dengan tanaman yang berasal dari benih hibrida. Soh et al. (2001) juga menyatakan bahwa minyak yang dihasilkan mengalami peningkatan 20% sampai 30% dibandingkan dengan bahan tanam hasil perbanyakan secara generatif.
Protokol kultur jaringan pada dasarnya menyebabkan penghentian aktivitas secara tiba-tiba saat jaringan eksplan diambil dari tanaman donor. Selain itu juga terjadi perubahan kondisi lingkungan eksplan. Penggunaan zat pengatur tumbuh pada level tinggi juga dapat memicu proses redifferensiasi atau bahkan dedifferensiasi dari jaringan eksplan (Rival 2000). Istilah variasi somaklonal diberikan pada fenotip baru yang muncul secara acak dalam suatu lini sel somatis, khususnya setelah propagasi klonal in-vitro (Jaligot et al. 2010).
Corley et al. (1986) mengungkapkan bahwa 5 sampai 10% proporsi kelapa sawit yang berasal dari embrio somatik memperlihatkan fenotip varian somaklon bersayap (mantled). Beberapa klon asal kultur jaringan menghasilkan bunga abnormal dengan feminisasi bagian bunga jantan maupun bunga betina. Adam et al. (2005) melaporkan bahwa morfologi mantel terjadi karena transformasi stamen pada bunga jantan dan staminode (vestigial stamen) pada bunga betina menjadi struktur yang menyerupai karpel.
Menurut Adam et al. (2007a) pada bunga mantel jantan, transformasi stamen menjadi pseudokarpel berakibat pada sterilitas karena tidak terbentuk pollen. Sedangkan pada bunga mantel betina, fertilisasi pada kasus abnormal yang lebih ringan akan menghasilkan buah yang fertil. Namun pada kasus abnormal yang lebih berat akan menghasilkan buah partenokarpi atau perkembangan yang tertahan. Hartley (1988) menyatakan bahwa buah kelapa sawit yang mempunyai karpel tambahan disebut buah mantel.
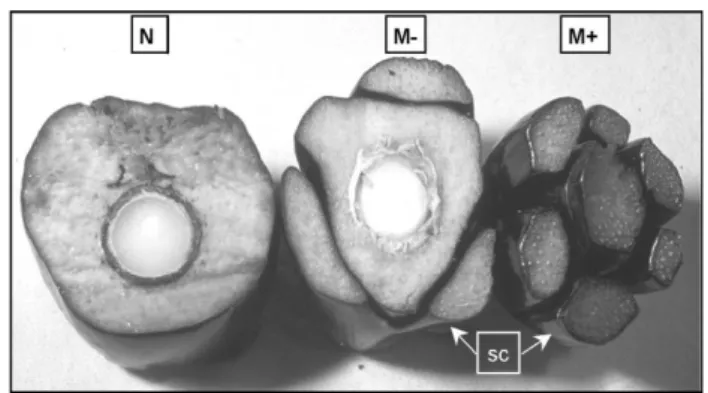
Menurut Hetharie (2008) karakterisasi tingkat abnormalitas pada buah mantel didasarkan atas batasan antara karpel tambahan dengan karpel utama, kondisi mesokarp serta keberadaan biji. Pada buah normal tidak ada karpel tambahan, mesokarp berdaging dan mempunyai biji. Pada buah abnormal ringan ada karpel tambahan namun batasannya dengan karpel utama hanya tampak di bagian apeks, mesokarp berdaging dan masih mempunyai biji. Pada buah abnormal berat batasan antar karpel tambahan dan utama terlihat sangat jelas dari bagian ujung hingga tengah buah, mesokarp masih berdaging dan mempunyai biji. Pada buah abnormal sangat berat karpel tambahan terpisah dari karpel utama dimulai dari ujung hingga ke sepertiga pangkal buah, mesokarp berserat bahkan berkayu serta tidak memiliki biji (Gambar 2).
Gambar 2 Potongan melintang pada buah kelapa sawit regeneran. N, buah normal; M-, buah mantel ringan; M, buah mantel berat; sc, supernumerary carpel (Sumber: Jaligot et al. 2000)
Timbulnya varian fenotip mantel ini hanya sekitar 5% dari kelapa sawit yang berasal dari kultur jaringan. Namun variasi yang dihasilkan bersifat sangat luas dan tidak dapat diprediksi, tergantung pada tanaman induk dan kondisi kultur in-vitro (Euwens et al. 2002). Tingkat abnormalitas sangat beragam, antara kelapa sawit yang berasal dari progeni klonal yang sama, antara infloresens yang tumbuh pada pohon yang sama, atau antara bunga yang menyusun infloresens yang sama. Suatu pembalikan yang spontan dan bertahap teramati di lapangan dan durasinya bergantung pada tingkat keparahan abnormalitas semula (Rizal & Parveez 2005). Hal ini mengindikasikan epigenetik (Tregear et al. 2002). Epigenetik adalah suatu fenomena yang berhubungan dengan perubahan ekspresi gen yang dapat kembali pulih tetapi bukan karena perubahan sekuens DNA (Kaeppler et al. 2000).
Pembalikan fenotip abnormal menuju normal memerlukan waktu bertahun-tahun. Periode non-produktif ini ditambahkan pula dengan masa 2 sampai 3 tahun perkembangan embrio somatik in-vitro serta 5 tahun kemudian sampai tanaman matang secara reproduktif. Abnormalitas pada organ reproduktif hanya dapat diketahui setelah tanaman berbunga atau berbuah. Akibatnya, abnormalitas mantel tidak hanya mengakibatkan penurunan dalam hal produksi minyak namun juga membuang waktu, area tanam dan infrastuktur bioteknologi (Jaligot et al. 2010).
Rival et al. (1997) mencoba menelusuri penyebab adanya varian somaklonal mantel ini melalui analisis aliran sitometri dan tidak menemukan adanya perubahan level ploidi pada fenotip abnormal kelapa sawit. Kubis et al. (2003)
7
menyatakan bahwa fenotip mantel tidak disebabkan oleh pengaturan kembali elemen transposon. Rival et al. (1998) juga tidak menemukan polimorfisme DNA yang dapat dihubungkan dengan fenotip mantel.
Fenotip mantel melibatkan transformasi homeotik organ bunga (Adam et al. 2005). Homeosis adalah penggantian sebagian atau keseluruhan dari salah satu organ dengan organ yang lain (Lehmann & Sattler 1992). Menurut Bowman et al. (1991) berdasarkan analisis genetik dari homeotik floral mutan, ditemukan bahwa sedikit perubahan dalam pola ekspresi gen MADS-box akan menentukan perubahan struktur bunga secara keseluruhan.
Faktor Transkripsi
Gen adalah unit hereditas, bagian kecil dari DNA yang membawa kode informasi untuk sekuens asam amino dari satu rantai polipeptida (pada umumnya) atau untuk menghasilkan molekul RNA non-coding. Suatu gen tidak berperan langsung dalam sintesis protein. Pada eukariot, DNA berada dalam nukleus sedangkan sintesis protein berlangsung di sitoplasma, sehingga keduanya tidak pernah bertemu. Suatu gen mengatur sintesis protein dengan cara mengirimkan kopi RNA yang mengandung kode informasi dari gen tersebut ke sitoplasma. Proses ini disebut transkripsi dan kopi RNA yang dihasilkan disebut messenger RNA (mRNA). Transkripsi adalah proses penting sebelum gen diekspresikan (Elliot & Elliot 2009).
Gen diinisiasi untuk menjalani transkripsi pada sekuens promoter dan RNA polimerase akan menempel di sekuens tersebut. Pada eukariot, RNA polimerase tidak berinisiatif untuk hadir begitu ada sekuens yang dikenalinya, melainkan diperintah oleh protein-protein (faktor transkripsi) yang terpasang terlebih dahulu pada sekuens elemen kontrol yang ada di daerah promoter (Gambar 3, Elliot & Elliot 2009).
Faktor transkripsi adalah protein yang menempel pada elemen kontrol dari gen eukariot dan bertanggung jawab untuk mengontrol tahap inisiasi transkripsi. Suatu faktor transkripsi didisain agar dapat menempel pada sekuens kontrol elemennya. Beberapa faktor transkripsi diaktifkan hanya apabila menerima sinyal yang tepat dari lingkungan, misalnya yang berasal dari hormon (Elliot & Elliot 2009).
Gambar 3 Domain faktor transkripsi (Sumber: Elliot & Elliot 2009)
Faktor Transkripsi
Elemen Kontrol DNA
Domain Pengikatan DNA Domain Aktivasi
Faktor transkripsi merupakan regulator penting dalam proses seluler dan kompleksitas organisme hidup memerlukan sejumlah besar faktor transkripsi. Faktor transkripsi memiliki peranan penting dalam gene networks, yaitu dengan berinteraksi dan mengatur gen lain (Riechmann & Ratcliffe 2000). Sebagian besar faktor transkripsi yang mengatur perkembangan pada tanaman mulai dari akar hingga bunga serta perkembangan buah, disandikan oleh gen MADS-box (Becker & Theissen 2003).
Gen MADS-box
Gen MADS-box dinamai dari singkatan huruf awal empat anggota pertamanya, yaitu Minichromosome maintenance 1 (MCM1; dari ragi),
AGAMOUS (AG; dari Arabidopsis thaliana), DEFICIENS (DEF; dari
Antirrhinum majus) dan Serum response factor (SRF; dari manusia) (Schwarz-Sommer et al. 1990; Shore & Sharrocks 1995). Menurut Alvarez-Buylla et al. (2000b) famili gen MADS-box dapat dibagi ke dalam dua kelas utama, yaitu Tipe I dan Tipe II. Kedua tipe gen ini ditemukan pada hewan, jamur dan tanaman. Namun fungsi gen MADS-box tipe I pada tanaman belum diketahui karena seluruh gen MADS-box tanaman yang sejauh ini telah berhasil dikarakterisasi dan berperan dalam perkembangan tanaman adalah termasuk dalam tipe II.
Faktor transkripsi dikelompokkan ke dalam suatu famili berdasarkan struktur dan interaksi faktor transkripsi tersebut dengan daerah spesifik DNA yang akan ditempelinya (Luscombe et al. 2000). Gen yang termasuk dalam MADS-box tipe II tanaman memperlihatkan kemiripan struktur domain yang disebut struktur domain MIKC, antara lain mencakup domain: MADS (M-), intervening (I-), keratin-like (K-) dan C-terminal (C-). Gen MADS-box tipe II tanaman selanjutnya dikenal dengan sebutan gen MIKC (Gambar 4) (Munster et al. 1997). Menurut Liu et al. (2001) faktor transkripsi tersusun atas empat daerah fungsional berdasarkan analisis struktur proteinnya, yaitu daerah pengikatan DNA, daerah regulasi transkripsi, daerah dimerisasi dan daerah lokalisasi inti. Dengan keempat daerah ini maka faktor transkripsi dapat melakukan fungsinya.
Gambar 4 Struktur domain MIKC pada gen MADS-box (Sumber: Munster et al. 1997)
Domain MADS (M-) disandikan oleh sekuens motif yang terkonservasi (Schwarz-Sommer et al. 1990) dengan panjang yang bervariasi. Namun panjang khasnya ada pada kisaran 168 hingga 180 bp. Hal ini berarti domain MADS menyandikan 56 hingga 60 asam amino (Lamb & Irish 2003). Domain MADS merupakan daerah yang paling konservatif dalam protein MADS-box (Riechmann et al. 1996). Sekuens motif dari domain MADS yang konservatif, selain
penempelan pada
DNA interaksi antar protein
mengandung karakteristik motif C-terminus
9
merupakan faktor utama yang menentukan penempelan pada DNA elemen kontrol, juga berperan dalam dimerisasi dan pengikatan faktor aksesori (Shore & Sharrocks 1995). Domain MADS (M-) berikatan dengan sequens DNA yang memiliki kesamaan yang tinggi terhadap motif CC(A/T)6
Domain-I yang relatif tidak konservatif menjadi penentu untuk pembentukan dimer selektif yang terlibat dalam pengikatan DNA. Domain-I yang terdapat pada seluruh protein MADS-box bervariasi dalam sekuens dan panjang (27 hingga 42 residu amino) (Riechmann & Meyerowitz 1997). Domain-K (keratin-like) ditunjukkan oleh suatu jarak reguler yang konservatif dari residu hidrofobik (Shore & Sharrocks 1995). Fungsinya adalah memediasi interaksi antar protein dan memulai dimerisasi melalui interaksi antar K-box yang ada pada protein-protein yang akan berdimer (Fan et al. 1997). Menurut Liu et al. (2001) daerah dimerisasi didefinisikan sebagai tempat bersatunya semua faktor transkripsi yang terlibat dalam regulasi gen tertentu.
GG yang disebut CarG-box (West et al. 1997).
Domain-C yang terletak pada daerah C-terminus protein MADS-box (yang kaya glutamin) merupakan daerah yang paling variabel baik dalam hal sekuens ataupun panjang. Domain ini terlibat dalam proses aktivasi transkripsi atau pembentukan kompleks multimerik faktor transkripsi (Cho et al. 1999).
Perluasan pada daerah C-terminus ini mengandung ≈ 30 asam amino (Shore & Sharrocks 1995). Menurut Liu et al. (2001) daerah regulasi transkripsi dibagi menjadi dua jenis, yaitu yang bersifat mengaktifkan dan yang menghambat transkripsi. Daerah yang mengaktifkan transkripsi biasanya tersusun oleh asam amino yang bersifat asam seperti prolin dan glutamin, sedangkan asam amino penyusun daerah penghambat transkripsi sampai sekarang belum jelas.
Protein MADS-box menempel pada DNA sebagai dimer. Struktur ini dapat berupa interaksi antara monomer dari protein yang sama membentuk homodimer atau antara monomer protein yang berbeda membentuk heterodimer. Sebagai contoh, protein SRF membentuk homodimer saat menempel secara in vitro pada DNA (Pellegrini et al. 1995), sedangkan protein MADS-box APETALA3 (AP3) dan PISTILLATA (PI) pada Arabidopsis merupakan heterodimer (Riechmann et al. 1996), namun beberapa protein MADS-box juga dapat membentuk kompleks yang lebih besar dalam mengatur perkembangan. Pada ragi, protein SQUA yang homodimer bergabung dengan DEF dan GLO yang merupakan heterodimer untuk menjalankan fungsi yang sama. Kompleks ini memperlihatkan afinitas penempelan yang lebih tinggi dibandingkan dengan saat masing-masingnya berdiri sendiri sebagai homodimer atau heterodimer (Egea-Cortines et al. 1999).
Anggota famili gen MADS-box terlibat dalam beberapa aspek perkembangan pada tanaman dan merupakan dasar untuk diversifikasi morfologi, terutama dalam organ reproduksi (Theissen et al. 2000). Gen MADS-box berperan penting dalam perkembangan bunga berdasarkan model kuartet atau model ABCDE (Theissen 2001). Model ini dijelaskan berdasarkan hipotesis bahwa pembentukan lingkaran konsentris (whorl) pada organ bunga diatur oleh kombinasi dari ekspresi satu set gen yang saling tumpang tindih (overlapping) (Zhao et al. 2010; Gambar 5).
Gen penentu identitas pembungaan dibagi ke dalam lima kelas yang berbeda, antara lain gen kelas A, B, C, D dan E yang berperan dalam lima fungsi homeotik yang berbeda, yaitu A menentukan sepal (daun kelopak), A + B + E
menentukan petal (mahkota), B + C + E menentukan stamen (organ bunga jantan), C + E menentukan karpel (organ bunga betina), dan C + D + E menentukan ovul (bakal biji) dalam karpel (Theiβen 2001, Gambar 5).
Gambar 5 Model penentuan identitas struktur bunga berdasarkan ekspresi gen MADS-box yang saling tumpang tindih. CAL, daun kelopak; COR, mahkota bunga; AN, organ bunga jantan; GY, organ bunga betina; OV, bakal biji (Sumber: Adam et al. 2007b) Sebagai konsekuensi dari fungsi tumpang-tindihnya, inaktivasi dari salah satu gen yang berperan dalam fungsi A, B atau C akan berakibat pada “overflowing” dari organ tetangganya (Zhao et al. 2010). Contoh tanaman yang paling sesuai untuk menggambarkan fenotip bunga mantel kelapa sawit adalah tanaman yang memiliki fungsi-B yang tidak sempurna, yaitu untuk fungsi petal dan stamen yang digantikan oleh sepal (fungsi A) dan karpel (fungsi C) (Coen & Meyerowitz 1991). Pada regeneran bunga mantel tanaman kelapa sawit, stamen atau staminode residual bunga jantan dan stamen pada bunga betina ditransformasi menjadi karpeloid dan karpel (Adam et al. 2005). Menurut Becker & Theissen (2003) gen-gen class A, B, C, D dan E yang telah diketahui terbagi ke dalam clade masing-masing berdasarkan kemiripan sekuens, antara lain SQUAMOSA (kelas A), DEFICIENS (kelas B), GLOBOSA (kelas B), AGAMOUS (kelas C dan D) dan AGL2-like (kelas E).
Pada infloresens bunga mantel kelapa sawit, level ekspresi seluruh gen MADS-box menunjukkan penurunan. Penurunan pada seluruh gen MADS-box ini disebabkan oleh regulasi yang terjadi antar gen MADS-box. Didasarkan pada fakta bahwa mereka menempeli situs yang sama, yaitu CarG-box (Adam et al.2007a).
Metilasi DNA dan Ekspresi Gen
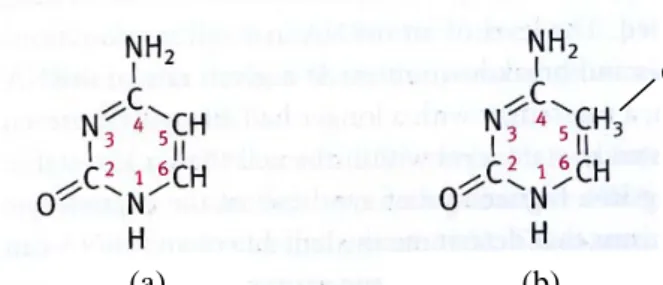
Metilasi adalah penambahan gugus metil ke posisi karbon kelima pada basa sitosin yang dikatalisis oleh enzim Metiltranferase (Lewin 2008, Gambar 6). Metilasi terjadi pada situs gen yang spesifik. Umumnya gugus metil ditemukan pada situs CG. Apabila residu Sitosin (C) pada sekuens pendek palindromik di kedua utas DNA mengalami metilasi, situs itu disebut fully methylated. Namun apabila hanya salah satu dari kedua utas DNA yang termetilasi, situs itu disebut hemimethylated (Lewin 2008).
11
Gambar 6 Metilasi pada basa sitosin. (a) Struktur Sitosin; (b) Struktur Metilsitosin (Sumber: Elliot & Elliot 2009)
Ada dua jenis enzim metiltransferase. Keduanya menggunakan S-adenosilmetionin (SAM) sebagai donor metil, yaitu de novo methyltrasferase dan maintenance methyltransferase. Saat replikasi DNA, enzim maintenance methyltransferase bekerja mengkopi pola metilasi utas DNA induk ke utas DNA anak yang mengandung pasangan GC komplementer. Dengan demikian dalam pembelahan sel, pola metilasi diturunkan. Namun pola metilasi ini tidak selalu diturunkan pada generasi selanjutnya, karena adanya proses demetilasi setelah fertilisasi.
Enzim de novo methyltransferase bekerja menghadirkan pola metilasi yang baru selama perkembangan organisme. Pola tersebut juga dapat berubah sewaktu-waktu. Hal ini menunjukkan bahwa dalam beberapa kasus pola metilasi “terkunci” dan terkadang diturunkan dengan “tidak stabil” oleh keturunannya. Kejadian ini disebut genetic imprinting, yang merupakan salah satu kasus pada kontrol epigenetik (Elliot & Elliot 2009).
Phillips et al. (1990) menyatakan bahwa keragaman somaklonal diduga berhubungan erat dengan perubahan pola metilasi DNA selama dalam kultur. Salah satu metode untuk memperkirakan status metilasi DNA adalah dengan menggunakan prinsip kromatografi untuk memfraksinasi nukleotida-nukleotida yang termetilasi dan pengukuran spektrofotometri untuk kuantifikasi. Metode ini memungkinkan untuk menentukan persentase nukleotida yang termetilasi (Mohan-Jain et al. 2002).
Metilasi DNA dapat ditentukan dengan cara analitis setelah sebelumnya DNA dihidrolisis terlebih dahulu secara enzimatik atau dengan asam. Nuklease adalah enzim yang memotong, memendekkan atau mendegradasi molekul asam nukleat. Nuklease mendegradasi molekul DNA dengan memecah ikatan fosfodiester yang menghubungkan satu nukleotida dengan nukleotida berikutnya pada utas DNA (Brown 1991).
Berdasarkan aktivitasnya terdapat dua kelas utama nuklease yaitu eksonuklease yang aktif pada bagian ujung molekul asam nukleat dan
endonuklease yang memotong rantai asam nukleat dari dalam.
Deoksiribonuklease memotong rantai DNA sedangkan ribonuklease memotong rantai RNA. Pemotongan tersebut menghasilkan torehan (titik) dalam sebuah molekul DNA berutas ganda serta menyebabkan hilangnya ikatan fosfodiester antara dua nukleotida yang berdekatan dalam satu utas (Rittie & Perbal 2008).
Status metilasi DNA dapat diperkirakan dengan menggunakan metode High Performance Liquid Chromatography (HPLC), namun jalannya proses analisis sering memakan waktu lama sehingga membatasi keluaran hasil dari sampel.
CH3
Perbaikan dalam waktu analisis diperoleh dengan Ultra Performance Liquid Chromatography (UPLC) (Swartz 2005).
UPLC adalah metode pemisahan analitik yang merupakan perluasan dari metode HPLC dalam hal kecepatan, sensitivitas dan resolusi. Teknologi ini memberi keuntungan maksimal dari prinsip kromatografi, karena proses pemisahan terjadi menggunakan kolom yang berisi partikel yang lebih kecil dan laju alir yang lebih tinggi untuk meningkatkan kecepatan dan mengurangi waktu running, serta menghasilkan resolusi dan sensitifitas yang lebih besar (Swartz 2005; Alexander 2008).
Metilasi DNA diimplikasikan pada pengaturan ekspresi gen (Elliot & Elliot 2009). Gardner et al. (1991) menyatakan bahwa ada korelasi antara level ekspresi gen dengan derajat metilasi, yaitu apabila metilasi rendah maka ekspresi gen tinggi atau sebaliknya.
Metilasi memiliki target, diantaranya promoter gen adalah target yang paling umum. Gen tidak aktif ketika promoter termetilasi, tetapi aktif apabila promoter tidak termetilasi (Lewin 2008). Metilasi pada promoter suatu gen juga dapat memblokir pengikatan faktor transkripsi pada daerah promoter tersebut (Elliot & Elliot 2009). Metilasi dan demetilasi sitosin pada daerah promoter merupakan mekanisme yang penting dalam meregulasi ekspresi gen pada sel dan jaringan spesifik (Renkens et al. 1992). Ehrlich & Ehrlich (1993) mengemukakan bahwa metilasi juga dapat menyebabkan terjadinya penarikan metil-DNA binding protein dan histon deasetilase yang akan mengubah struktur kromatin (chromatin remodelling) di sekitar daerah awal transkripsi pada gen sehingga menjadikan kromatin menutup kembali. Kedua mekanisme tersebut akan menghambat transkripsi dan menyebabkan gen inaktif.
Tingkat transkripsi dari gen dapat digunakan untuk mengetahui fungsi gen. Salah satu metode untuk mengidentifikasi perbedaan ekspresi gen adalah dengan membandingkan level transkrip mRNA (Mohan-Jain et al. 2002). Qualitative Real Time Polymerase Chain Reaction (qPCR) adalah suatu teknik yang paling sensitif untuk deteksi dan kuantifikasi mRNA yang ada pada saat ini (Kubista et al. 2006).
Dibandingkan dengan dua teknik kuantifikasi level mRNA lainnya, Northern blot analysis dan RNase protection assay, qPCR dapat digunakan untuk mengkuantifikasi sampel dalam jumlah yang lebih kecil. Teknik ini juga cukup sensitif untuk mengkuantifikasi RNA dari sel tunggal (Biederman et al. 2004). Real Time PCR adalah teknik paling sensitif dalam memonitor ekspresi gen, namun hanya memungkinkan untuk mendeteksi kopi tunggal transkrip mRNA. Tahap awal meliputi isolasi RNA total atau mRNA diikuti dengan sintesis cDNA sedangkan tahap kedua adalah amplifikasi cDNA menggunakan primer PCR khusus (Mohan-Jain et al. 2002).
Qualitative Real Time PCR (qPCR) berbeda dari PCR konvensional karena didasarkan atas deteksi dan kuantifikasi pancaran fluoresens oleh produk PCR selama proses amplifikasi (Kubista et al. 2006). Salah satu reporter fluoresens yang biasa digunakan dalam RT PCR, yaitu SYBR Green. SYBR Green akan berfluoresens hanya apabila menempel pada DNA utas ganda. Penggunaan SYBR Green relatif lebih mudah karena tidak membutuhkan desain probe (Dorak 2006).
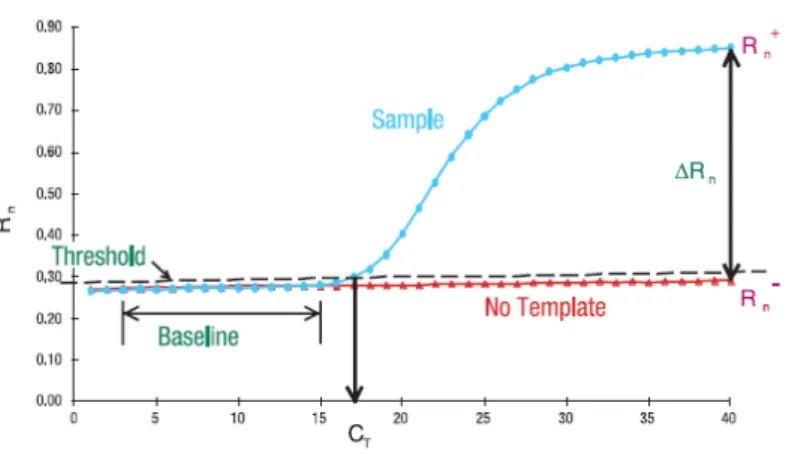
Reporter fluoresens akan meningkat sampai pada jumlah yang dapat terdeteksi oleh qPCR dan digambarkan dalam bentuk kurva amplifikasi. Kurva
13
amplifikasi mengandung beberapa informasi penting, yaitu (1) Threshold, adalah daerah deteksi pada saat jumlah reporter fluoresens pertama kali dapat dideteksi oleh mesin qPCR. Agar hasil perhitungan lebih akurat, threshold berada pada fase eksponensial, (2) Cycle Threshold (CT), yaitu siklus pada proses PCR saat jumlah reporter fluoresens berpotongan dengan garis threshold yang ditetapkan untuk perhitungan pada qPCR, (3) Rn, yaitu jumlah reporter fluoresens yang terdeteksi, dan (4) Cycle, yaitu jumlah siklus yang digunakan dalam proses PCR (Applied Biosystem 2007) (Gambar 7).
Gambar 7 Proses qPCR yang diamati dari peningkatan sinyal fluoresens (Sumber: Dorak 2006)
Kesalahan yang spesifik mudah sekali terjadi dalam reaksi qPCR dan mempengaruhi jumlah material awal di antara sampel. Hal ini khususnya berkaitan dengan sampel yang diperoleh dari individu yang berbeda dan akan menyebabkan mis-interpretasi dari profil ekspresi gen target yang dimaksud. Cara yang umum untuk meminimalisasi kesalahan ini serta untuk mengkoreksi variasi antar sampel adalah dengan mengamplifikasi suatu RNA seluler lain yang berperan sebagai pembanding internal, bersamaan dengan amplifikasi gen target yang diinginkan. Metode ini didasarkan pada asumsi bahwa level ekspresi dari gen normalisator tersebut tidak berubah pada setiap sampel (Dean et al.2002).
Suatu standar internal yang ideal haruslah terekspresi dalam level yang konstan di antara jaringan yang berbeda, bagian siklus sel ataupun pada tiap tahap perkembangan, serta tidak terpengaruh oleh perlakuan eksperimental apapun. Jenis gen seperti ini disebut housekeeping genes. Housekeeping genes mengkode protein yang umumnya berfungsi dalam metabolisme sel dasar (Blake et al. 2003). Aktin bersama-sama dengan gliseraldehid-3-fosfat dehidrogenase (GADPH), tubulin, siklofilin, elongation factor 1-α (ef-1α), ubikuitin, dan 18 Svedberg Units (S) rRNA (18S rRNA), terekspresi secara konstitutif dan terlibat dalam fungsi housekeeping dasar yang dibutuhkan dalam pemeliharaan sel. Oleh sebab itu, mereka umumnya digunakan sebagai kontrol endogenous internal untuk menormalisasi studi ekspresi gen (Sturzenbaum et al. 2001).
Elongation factor-1α (EF-1α) adalah sebuah protein yang mengikat aminoasil-transfer RNA ke ribosom selama proses sintesis protein. Protein ini telah ditetapkan sebagai kontrol invariant yang baik untuk menyesuaikan perbedaan akibat kesalahan pemipetan (tube-to-tube loading) atau degradasi (Dostal et al. 1994). Adam et al. (2007a) menggunakan gen EF1-α1 (nomor
aksesi AY550990) sebagai standar internal dari reaksi semikuantitatif RT-PCR pada penelitiannya terhadap abnormalitas kelapa sawit.
Terdapat dua macam perhitungan yang dapat dilakukan dengan Real Time PCR, yaitu (1) kuantitasi absolut yang digunakan untuk mengetahui jumlah molekul DNA yang ingin diketahui dengan menggunakan kurva standar yang sudah diketahui jumlah molekul DNA-nya, (2) kuantitasi relatif yang digunakan untuk mengetahui jumlah molekul DNA yang tidak diketahui dengan membandingkannya dengan sampel DNA yang lain yang digunakan sebagai pembanding (Applied Biosystem 2007).
Metode ΔΔCT Relative Quantification (Livak & Schmittgen 2001) melibatkan pembandingan nilai CT
dari gen target dengan gen pembanding, sehingga hasil akhir yang diperoleh hanya berupa rasio ekspresi gen target pada jaringan yang abnormal terhadap jaringan normal (kalibrator) serta relatif terhadap normalisator, yaitu gen pembanding. Hal ini disebabkan oleh ekspresi gen pembanding sebagai standar internal yang berada pada level konstan pada setiap sampel yang berbeda dan tidak terpengaruh oleh perlakuan eksperimental apapun. Dengan demikian rasio gen target terhadap gen referensi hanya akan bervariasi tergantung pada ekspresi gen target (Blake et al. 2003).
15
METODOLOGI
Waktu dan Tempat Penelitian
Penelitian ini dilaksanakan dari bulan Juni 2012 sampai April 2013 di Laboratorium Genomik dan Transkriptomik, Departemen Bioteknologi, PT. SMART Tbk., Bogor.
Bahan
Bahan yang digunakan dalam penelitian ini adalah buah muda normal dan buah muda mantel dengan tipe abnormalitas sangat berat. Buah abnormal dikelompokkan menjadi dua, yaitu buah abnormal yang diambil dari dalam tandan (Abn m) dan buah abnormal yang diambil dari luar tandan (Abn). Umur buah di bagian luar tandan lebih tua dibandingkan dengan umur buah di bagian dalam tandan. Buah berasal dari regeneran kelapa sawit Tenera berumur 15 tahun yang diambil dari perkebunan Socfindo. Primer gen MADS-box dan EF1-α1 diperoleh dari Kusnandar et al.(belum dipublikasikan).
Prosedur Kerja
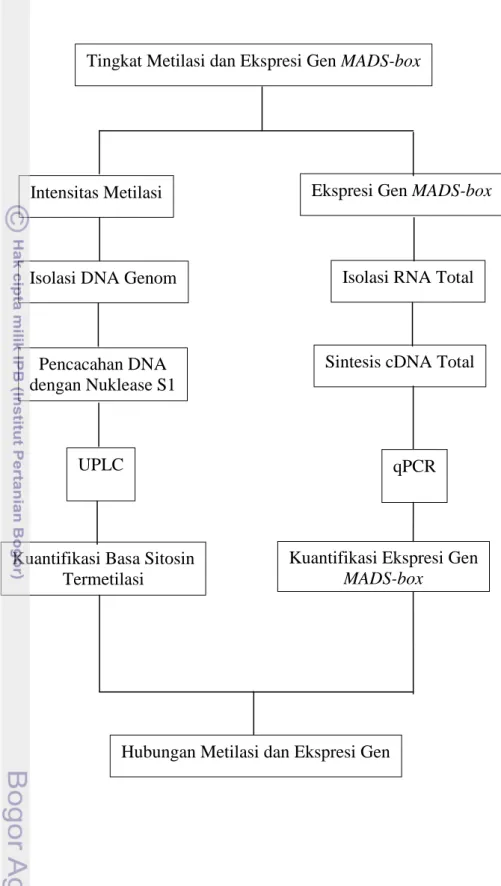
Penelitian ini terdiri dari dua eksperimen, yaitu (1) Analisis Ekspresi Gen MADS-box dan (2) Analisis Metilasi DNA. Alur penelitian disajikan pada Gambar 8.
1) Persiapan Sampel
Beberapa potongan tandan buah mantel dan buah normal yang masih muda diambil langsung dari kebun kemudian dimasukkan ke dalam kantong plastik dan diberi label.
Khusus buah untuk sampel RNA, diambil dari potongan tandan buah, dibersihkan, kemudian dipotong menjadi bagian kecil dan dimasukkan ke dalam Eppendorf yang telah diisi dengan larutan RNA later sebanyak ½ volume sampai sampel terendam. Sampel kemudian dibawa ke laboratorium di Bogor dan disimpan segera di dalam freezer bersuhu -20 0C sebelum diperlakukan lebih lanjut. Sampel dapat bertahan selama 7 hari dalam suhu ruang (selama pengiriman).
2) Analisis Ekspresi Gen MADS-box
Ekspresi gen MADS-box pada buah abnormal dan buah normal dihitung dengan menggunakan metode Quantitative Real-Time PCR (qPCR). DNA yang digunakan sebagai cetakan diperoleh dari proses transkripsi balik mRNA yang ada dalam RNA total menjadi cDNA. qPCR dilakukan dengan menggunakan EF1-α1 sebagai gen pembanding dan hasil qPCR divalidasi dengan kurva standar. Analisis hasil dilakukan dengan metode ΔΔ CT Relative Quantification (Livak & Schmittgen 2001).
Gambar 8 Alur Penelitian
Tingkat Metilasi dan Ekspresi Gen MADS-box
Intensitas Metilasi Ekspresi Gen MADS-box
Isolasi DNA Genom Isolasi RNA Total
Pencacahan DNA dengan Nuklease S1
Sintesis cDNA Total
UPLC qPCR
Kuantifikasi Basa Sitosin Termetilasi
Kuantifikasi Ekspresi Gen MADS-box
17
Ekstraksi RNA Total
RNA dari sampel buah untuk analisis ekspresi, diekstraksi menggunakan RNeasy® Plant Mini Kit (Cat. no 74904, Qiagen) sesuai dengan prosedur dari Qiagen. Sebanyak ± 450 µl bufer lisis (bufer RLC) yang telah terlebih dahulu diencerkan dengan β-Merkaptoetanol (10 µl β-ME per 1 ml bufer RLC) dipersiapkan dalam tabung Eppendorf 2 ml. Sampel digerus dalam Nitogen cair. Sebanyak ± 50 mg bubuk sampel yang telah halus dimasukkan segera ke dalam tabung Eppendorf yang telah dipersiapkan, kemudian divorteks.
Suspensi sel yang terbentuk dipindahkan ke dalam QIA shredder spin collumn (kolom insert ungu dalam tabung 2 ml) lalu disentifus pada 14000 rpm selama 2 menit. Supernatan dipipet dengan hati-hati kemudian ditransfer ke dalam tabung Eppendorf baru. Sebanyak 0.5 x volume etanol (96-100%) ditambahkan ke dalam supernatan kemudian diresuspensi secara perlahan-lahan.
Sebanyak ± 650 µl sampel tersebut kemudian dipindahkan ke dalam RNeasy spin column (kolom insert pink dalam tabung 2 ml) lalu disentrifus selama 15 detik pada kecepatan 10000 rpm dan cairan yang keluar dari kolom (flow trough) dibuang. Ke dalam kolom ditambahkan ± 750 µl bufer RW1 lalu disentrifus kembali selama 15 detik pada kecepatan 10000 rpm dan cairan yang keluar dari kolom dibuang. Selanjutnya ke dalam kolom ditambahkan ± 500 µl bufer RPE (yang telah diencerkan sebelumnya dengan etanol) lalu disentrifus selama 15 detik pada 10000 rpm untuk mencuci membran kolom, lalu cairan yang keluar dari kolom dibuang. Proses terakhir ini diulangi sekali lagi, namun sentrifugasi selanjutnya dilakukan selama 2 menit pada 10000 rpm untuk memastikan sisa etanol tidak terbawa di dalam kolom spin.
RNeasy spin column dipindahkan ke dalam tabung Eppendorf 2 ml baru kemudian disentrifus pada 16000 rpm selama 1 menit. RNeasy spin column dipindahkan lagi ke dalam tabung Eppendorf 1.5 ml baru, ditambahkan 30 µl Rnase free-water kemudian disentrifus pada 10000 rpm selama 1 menit untuk mengelusi RNA. RNeasy spin column lalu dibuang dan cairan yang merupakan hasil elusi RNA segera disimpan pada suhu -20 0
Keutuhan RNA total diuji menggunakan QIAxcel RNA Quality Control Kit (Qiagen) dan dijalankan pada alat QIAxcel analyzer system (Qiagen). Konsentrasi dan kemurnian RNA dihitung dengan mengukur absorbansinya menggunakan NanoDrop™ 2000c Specthrophotometer (Thermo Scientific) pada panjang gelombang 260, 280 dan 230 nm. Konsentrasi RNA dihitung berdasarkan asumsi bahwa satu satuan absorbansi pada panjang gelombang 260 nm setara dengan 40 μg/ml (Sambrook et al. 1989) sehingga konsentrasi RNA dapat dihitung dengan rumus sebagai berikut :
C.
Konsentrasi RNA = A 260 x 40 μg/ml x fp Keterangan :
A 260 : absorban yang terbaca pada λ 260 nm fp : faktor pengenceran
Kemurnian RNA terhadap protein dihitung berdasarkan rasio absorbansi pada panjang gelombang 260 dengan 280 nm, dan kemurnian RNA terhadap polisakarida dihitung dari rasio absorbansi pada panjang gelombang 260 dengan 230 nm. RNA yang murni mempunyai nilai ≥ 1,8 baik pada rasio absorbansi panjang gelombang 260/280 maupun 260/230 (Sambrook et al. 1989).
Sintesis cDNA Utas Tunggal
cDNA utas tunggal disintesis dari RNA total menggunakan QuantiTect® Reverse Transcription Kit (Cat. no. 205313, Qiagen) sesuai dengan prosedur dari Qiagen. Sebanyak 900 ng template RNA total dicampurkan dengan 2 μl gDNAwipeout buffer 7x dan dicukupkan dengan RNAse free water hingga total volume menjadi 14 μl. Campuran dihomogenkan kemudian diinkubasi pada 42 °C selama 2 menit untuk mengoptimalkan penghilangan DNA genomik pengkontaminan, setelah itu segera diletakkan di dalam es. Sebanyak 1 μl Quantiscript reverse transcriptase, 4 μl RT buffer 5x dan 1 μl RT primer mix ditambahkan ke dalam campuran hingga total volume seluruhnya menjadi 20 μl. Campuran dihomogenkan kemudian diinkubasi pada 42 °C selama 30 menit dan dilanjutkan pada 95 °C selama 3 menit untuk menginaktivasi Quantiscript reverse transcriptase.
Keberhasilan sintesis cDNA dan kemurnian cDNA dari DNA genom diverifikasi dengan PCR menggunakan primer forward ActF dan primer reverse ActR yang didisain spesifik untuk ekson 2 – ekson 3 dari gen Actin (EgACT1). Primer didisain menggunakan software Primer3Plus. Komposisi PCR yang digunakan adalah 1 µl cDNA, 1.5 µl Taq buffer 1x, 1.5 µl dNTP mix 4 mM, 1.5 µl Primer ActF 1 mM dan 1.5 µl Primer ActR 1 mM, 0.16 MgCl2
PCR dilakukan menggunakan alat Esco® PCR Cabinet dengan kondisi pra-PCR pada 95
40 mM, 0.12 µl enzim Taq DNA polimerase 0.5 U dan dicukupkan dengan Rnase-free water hingga volume akhir menjadi 15 µl.
0
C selama 5 menit, diikuti oleh denaturasi pada 95 0C selama 30 detik, annealing pada 60 0C selama 20 detik dan ekstensi pada 72 0C selama 30 detik yang dilakukan sebanyak 25 kali siklus, dan terakhir pasca PCR pada 72 0C selama 7 menit dilanjutkan dengan pendinginan pada 25 0
Amplifikasi dengan primer ActF dan ActR yang didisain mengapit ekson 2 – ekson 3 gen EgACT1, juga dilakukan terhadap DNA genom kelapa sawit sebagai sebagai kontrol terjadinya kontaminasi terhadap sintesis cDNA sekaligus kontrol keberhasilan sintesis cDNA. Keberhasilan sintesis cDNA diketahui dari terbentuknya satu pita berukuran ±162 bp. Apabila terjadi kontaminasi pada cDNA, maka akan diperoleh dua pita yang masing-masing berukuran ±162 bp (cDNA) dan ±242 bp (DNA genom) yang ±80 bp lebih panjang dari cDNA karena mengandung intron yang ada di antara ekson 2 – ekson 3 gen Actin (Shah et al. 1982).
C selama 1 menit.
Keutuhan DNA hasil amplifikasi dilihat pada gel agarose 1% (Invitrogen) dalam bufer TAE 1x (0.04 M Tris, 0.001 M EDTA-Na2.2H2O, 0.02 M Asam asetat pH 8.5). Untuk elektroforesis, 1 µl DNA dicampurkan dengan 1/6 volume loading dye (0.25% bromofenol blue, 0.25% silen sianol FF, 30% gliserol) dan dijalankan selama 30 menit pada tegangan 100 Volt. Visualisasi cDNA dilakukan di atas UV transluminator Gel-DocTM XR (BIORAD) setelah diwarnai dengan EtBr (0.5 µg/ml) selama 10 menit dan dibilas dengan dH2
O. Kuantifikasi Ekspresi Gen dengan qPCR
Level ekspresi gen MADS-box pada buah normal dan buah mantel dikuantifikasi menggunakan qPCR. Sebanyak 11 gen target MADS-box diamplifikasi menggunakan primer spesifik (Kusnandar et al. belum
19
dipublikasikan). Gen EgEF1-α1 juga diamplifikasi menggunakan primer spesifik sebagai gen referensi untuk kontrol ekspresi.
Level transkripsi gen target dan gen referensi untuk qPCR ditentukan dengan mesin qPCR Rotor-Gene®Q (Qiagen) menggunakan Quantifast SYBR Green PCR Kit (Cat. no. 204054, Qiagen) sesuai prosedur dari Qiagen. qPCR dilakukan dalam 384-well blocks dan dua ulangan per gen, per sampel cDNA.
Komposisi reaksi adalah 1 µl cDNA (900ng), 5 μLQuantifast SYBR Green RT-PCR Master Mix 2x, 2 μl primer mix gen MADS-box (50:50 campuran primer forward dan reverse, masing-masing 1 µM/ml)dan RNase-free water dalam total reaksi 10 μl. Kondisi reaksi adalah pra-PCR pada suhu 95 0C selama 10 menit, diikuti dengan 40 kali siklus dari denaturasi pada suhu 95 0C selama 30 detik, annealing pada suhu 60 0C selama 20 detik dan ekstensi pada suhu 72 0C selama 30 detik. Amplikon hasil dari RT-PCR dicek pada gel agarose untuk mendapatkan ekspresi secara semikuantitatif dan kualitas pita DNA.
Pembuatan Kurva Standar
Hasil amplifikasi masing-masing primer MADS-box terhadap utas pertama (first strand) cDNA digunakan untuk membuat kurva standar qPCR. Reaksi PCR yang digunakan adalah 1 µl cDNA utas tunggal, 1.5 µl Taq bufer 1x, 1.5 µl dNTP mix 4 mM, 3 µl Primer MADS-box 1 mM (Lampiran 1), 0.16 MgCl2, 0.12 µl enzim Taq DNA Polimerase 0.5 U dan dicukupkan dengan RNase-free water hingga volume akhir menjadi 15 µl. PCR dilakukan menggunakan alat Esco® PCR Cabinet dengan kondisi pra-PCR pada 950C selama 5 menit, diikuti dengan 25 kali siklus denaturasi pada 950C selama 30 detik, annealing pada 600C selama 20 detik dan ekstensi pada 720C selama 30 detik, dan terakhir pasca PCR pada 720C selama 7 menit diteruskan dengan pendinginan pada 250
Hasil PCR yang diperoleh selanjutnya dimurnikan menggunakan QIAquick PCR Purification Kit (Cat. no 28106, Qiagen) sesuai dengan prosedur dari Qiagen. Ke dalam sampel ditambahkan 5x volume bufer PB (yang sudah ditambahkan larutan pH indikator I) sambil dihomogenkan. Warna kuning menandakan campuran larutan berada pada pH optimal. Apabila larutan berwarna jingga atau ungu, ke dalam larutan ditambahkan 10 µl Sodium Asetat 3 M pH 5.0 sampai larutan berubah warna menjadi kuning.
C selama 1 menit.
QIAquick spin column disiapkan dalam tabung Eppendorf 2 ml yang sudah disediakan. Campuran larutan dipipet ke dalam kolom lalu disentrifus pada kecepatan 17900 x g (13000 rpm) selama 1 menit. Cairan yang keluar dari kolom dibuang dan kolom diletakkan kembali ke dalam tabung. Ke dalam larutan tersebut ditambahkan 0.75 ml bufer PE yang telah mengandung etanol kemudian disentrifus pada kecepatan 17900 x g (13000 rpm) selama 1 menit. Cairan yang keluar dari kolom dibuang dan kolom diletakkan kembali ke dalam tabung. Kolom disentrifus kembali pada kecepatan 17900 x g (13000 rpm) selama 1 menit untuk menghilangkan sisa etanol.
Kolom dipindahkan ke dalam tabung 1.5 ml yang baru. DNA kemudian di elusi dengan menambahkan 50 µl bufer EB (10 mM Tris-HCl, pH 8.5) ke bagian tengah kolom, diamkan selama 1 menit kemudian disentrifus pada kecepatan 17900 x g (13000 rpm) selama 1 menit.
Hasil purifikasi ini dianalisis dengan elektroforesis gel agarose 1% (Invitrogen) dalam bufer TAE 1x (0.04 M Tris, 0.001 M EDTA-Na2.2H2O, 0.02 M Asam asetat pH 8.5). Untuk elektroforesis, 1 µl DNA dicampurkan dengan 1/6 volume loading dye (0.25% bromofenol blue, 0.25% silen sianol FF, 30% gliserol) dan dijalankan selama 30 menit pada tegangan 100 Volt. Visualisasi DNA dilakukan di atas UV transluminator Gel-DocTM XR (BIORAD), setelah diwarnai dengan EtBr (0.5 µg/ml) selama 10 menit dan dibilas dengan dH2
Hasil purifikasi selanjutnya diencerkan ke dalam konsentrasi 0.1 ; 0.01 ; 0.001 dan 0.0001 ng/µl. Dari hasil pengenceran tersebut dibuat kurva standar dalam 2 ulangan menggunakan mesin Real-Time PCR Rotor-Gene®Q (Qiagen). Reaksi yang digunakan adalah 7 µl Rotor-Gene SYBR Green PCR Master Mix 2x, 2 µl primer MADS-box (50:50 campuran primer forward dan reverse masing-masingnya 1 µM/ml) dan 1 µl template hasil pengenceran.
O. Konsentrasi DNA ditetapkan dengan NanoDrop™2000c Specthrophotometer (Thermo Scientific) pada panjang gelombang 230, 260 dan 280 nm.
Kondisi reaksi adalah pra-PCR pada suhu 95 0C selama 10 menit, diikuti dengan 40 kali siklus dari denaturasi pada suhu 95 0C selama 30 detik, anealing pada suhu 60 0C selama 20 detik dan ekstensi pada suhu 72 0C selama 30 detik. Data kurva standar disimpan untuk analisis efisiensi amplifikasi.
Kuantifikasi Relatif
Hasil dari qPCR berupa kurva amplifikasi dari masing-masing reaksi dianalisis menggunakan program Relative Quantification dengan metode ΔΔ CT
berdasarkan metode Livak & Schmittgen (2001). Dari kurva amplifikasi diperoleh nilai CT dari masing-masing reaksi. Nilai Δ CT dari masing-masing perlakuan
diperoleh dari normalisasi gen target dengan gen pembanding, yaitu dengan cara mengurangi nilai CT gen target dengan nilai CT gen pembanding, sehingga rumus
untuk mendapatkan Δ CT adalah : Δ CT = CT MADS-box - CT EF1-α1. Sedangkan nilai
ΔΔ CT diperoleh dengan cara mengurangi nilai Δ CT perlakuan (buah abnormal)
dengan nilai Δ CT
Selanjutnya nilai ΔΔ C
kalibrator (buah normal).
T tersebut akan digunakan untuk mengetahui ekspresi
(RQ) gen MADS-box pada perlakuan relatif terhadap kalibrator dengan memasukkannya ke dalam rumus aritmatika 2 -ΔΔ CT. Analisis ekspresi gen dengan menggunakan qPCR ini diulang dua kali.
3) Analisis Metilasi DNA
Dalam tahapan ini dilakukan penghitungan persentase basa Sitosin termetilasi pada DNA buah normal dan buah abnormal hasil pencacahan enzim S1 Nuklease dan pemisahan dengan Ultra Performance Liquid Chromatography (UPLC).
Isolasi DNA Genomik
DNA genom dari sampel buah untuk analisis metilasi DNA diisolasi menggunakan Gen Elute™ Plant Genomic DNA Miniprep Kit (Cat. no. G2N350, Sigma-Aldrich) sesuai dengan prosedur dari Sigma Aldrich. Sampel dihaluskan dengan bantuan Nitrogen cair. Sebanyak ± 100 mg serbuk sampel dimasukkan ke
21
dalam tabung Eppendorf yang telah berisi 350 µl larutan lisis A dan 50 µl larutan lisis B kemudian divorteks untuk menghomogenkan.
Campuran diinkubasi selama 30 menit pada suhu 65 °C sambil sesekali divorteks untuk membantu proses lisis. Sebanyak 130 µl larutan presipitasi ditambahkan ke dalam sampel, lalu dibolak-balik sebentar untuk membantu menghomogenkan. Tabung Eppendorf kemudian disimpan di dalam es selama 5 menit untuk membantu proses presipitasi.
Tabung Eppendorf disentrifus pada kecepatan 16000 rpm selama 5 menit untuk mengendapkan debris seluler, protein dan polisakarida. Supernatan yang terbentuk disaring kembali melalui proses sentrifus pada kecepatan 16000 rpm selama 1 menit di dalam Gen Elute Filtration Column (kolom insert biru dalam tabung 2 ml). Kolom insert biru dibuang, kemudian ke dalam cairan yang keluar dari kolom (flow-trough) yang ada di dalam tabung ditambahi dengan 700 µl Binding Solution. Tabung lalu dibolak-balik sebentar untuk membantu menghomogenkan.
Sebanyak 500 µl larutan Column Preparation ditambahkan ke dalam Gen Elute Miniprep Binding Column (kolom insert merah dalam tabung 2 ml). Tabung kolom tersebut kemudian disentrifus pada 12000 rpm selama 1 menit dan cairan yang keluar dari kolom dibuang. Sebanyak 700 µl campuran larutan sampel dan Binding Solution dari proses sebelumnya dimasukkan ke dalam Gen Elute Miniprep Binding Column (kolom insert merah dalam tabung 2 ml) yang telah disiapkan. Tabung disentrifus pada 16000 rpm selama 1 menit, kemudian cairan yang keluar dari kolom dibuang. Proses ini diulang hingga sampel habis. Setelah selesai, binding column (kolom insert merah) dipindahkan ke dalam tabung 2 ml yang baru.
Ke dalam binding column yang ada pada tabung baru, ditambahkan ± 500 µl Wash Solution yang telah diencerkan terlebih dahulu dengan etanol. Tabung lalu disentrifus pada 16000 rpm selama 1 menit dan cairan yang keluar dari kolom dibuang. Proses ini diulangi sekali lagi, namun sentrifugasi selanjutnya dilakukan pada 16000 rpm selama 3 menit untuk mengeringkan kolom. Binding column lalu dipindahkan ke dalam tabung 2 ml yang baru, setelah itu ditambahkan ± 50 µl pre-warmed (65 °C) nuclease free water kemudian disentrifus pada 16000 rpm selama 1 menit.
Konsentrasi dan kemurnian DNA yang diperoleh, dihitung dengan mengukur absorbansinya menggunakan alat NanoDrop™ 2000c Specthrophotometer (Thermo Scientific) pada panjang gelombang 260, 280 dan 230 nm. Konsentrasi DNA dihitung berdasarkan asumsi bahwa satu satuan absorbansi pada panjang gelombang 260 nm setara dengan 50 μg/ml (Sambrook et al. 1989) sehingga konsentrasi DNA dapat dihitung dengan rumus sebagai berikut:
Konsentrasi DNA = A 260 x 50 μg/ml x fp Keterangan :
A 260 : absorban yang terbaca pada λ 260 nm fp : faktor pengenceran
Kemurnian DNA terhadap protein dihitung berdasarkan rasio absorbansi pada panjang gelombang 260 dengan 280 nm, dan kemurnian DNA terhadap polisakarida dihitung dari rasio absorbansi pada panjang gelombang 260 dengan
230 nm. DNA yang murni mempunyai nilai ≥ 1. 8 baik pada rasio absorbansi panjanggelombang 260/280 maupun 260/230 (Sambrook et al. 1989).
Keutuhan DNA dilihat pada gel agarose 1% (Invitrogen) dalam bufer TAE 1x (0.04 M Tris, 0.001 M EDTA-Na2.2H2O, 0.02 M Asam asetat pH 8.5). Untuk elektroforesis, 1 µl DNA dicampurkan dengan 1/6 volume loading dye (0.25% bromofenol blue, 0.25% silen sianol FF, 30% gliserol) dan dijalankan selama 30 menit pada tegangan 100 Volt. Visualisasi DNA dilakukan di atas UV transluminator Gel-DocTM XR (BIORAD) setelah diwarnai dengan 0.5 µg/ml Etidium Bromida (EtBr) selama 10 menit dan dibilas dengan dH2O. Sampel DNA yang diperoleh selanjutnya digunakan untuk analisis metilasi.
Pencacahan DNA Genomik dengan Enzim S1 Nuklease
Sebanyak 3.5 µg sampel DNA, 50 ng/µl un-methylated λ, 50 ppm larutan standar 5-Metil-2-Deoksisitidin (MDC) dan 2-Deoksisitidin (DC), dH2O serta blanko (no template control) ditambahkan dengan 10% S1 bufer nuklease10x (300 mM Na-asetat pH 4.6, 10 mM ZnSO4
Seluruh sampel lalu didenaturasi pada suhu 95 °C selama 10 menit dan segera didinginkan di dalam es. Sebanyak 3 U/µl (~ 1 µg DNA) enzim S1 nuklease (Thermo Scientific) yang sebelumnya telah diencerkan dengan S1 nuclease dillution buffer (20 mM Tris-HCl pH 7.5, 0.1 mM ZnSO
, 50% Gliserol).
4, 50 mM NaCl, 50% gliserol) kemudian ditambahkan ke dalam setiap sampel dengan volume akhir 70 µl. Sampel lalu diinkubasi pada suhu 37 °C selama 20 jam kemudian disentrifus pada 15000 g selama 5 menit. Keutuhan DNA sampel hasil digest kemudian dilihat dengan elektroforesis.
Kuantifikasi Metilasi DNA dengan UPLC
Sampel selanjutnya disuntikkan ke dalam suatu kolom berukuran 100 mm x 2.1 mm yang berisi partikel ACQUITY UPLC BEH C-18 berukuran 1.7 µm dan dipisahkan dengan alat Waters® ACQUITY UPLC™ System (Waters Corporation). Elusi dilakukan dalam fase gerak pada 97% buffer KH2PO4
Luas Area Sampel
dan 3% metanol pH 4.2 dengan kecepatan alir 1 ml/menit pada suhu ruang dan dideteksi pada panjang gelombang 254 nm. Efisiensi ditentukan berdasarkan retention time dan luas area puncak. Puncak nukleotida tunggal dari standar DC dan MDC selanjutnya digunakan untuk mengkuantifikasi kandungan sitidin (5’C) dan metilsitidin (5’mC) pada sampel, dengan rumus :
x Konsentrasi Standar
Luas Area Standar
Persentase kandungan metilsitidin (5mC) dari DNA genom kelapa sawit dihitung dengan rumus : (5’mC/ [5’mC + 5’C]) x 100% (Kubis et al. 2003).