KLASIFIKASI PROTEIN FAMILY MENGGUNAKAN
ALGORITME PROBABILISTIC NEURAL NETWORK (PNN)
RIZKY KURNIAWAN
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2014
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Klasifikasi Protein
Family Menggunakan Algoritme Probabilistic Neural Network (PNN) adalah
benar karya saya denganarahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, 2014
Rizky Kurniawan
ABSTRAK
RIZKY KURNIAWAN. Klasifikasi Protein Family Menggunakan Algoritme
Probabilistic Neural Network (PNN). Dibimbing oleh TOTO HARYANTO.
Protein terdiri dari rangkaian dan kombinasi dari 20 asam-amino
(amino-acid) . Protein yang satu memiliki susunan sekuens asam-amino yang berbeda
dengan protein lainnya. Klasifikasi protein sangat membantu sebagai petunjuk untuk mengetahui struktur, aktivitas dan peranan metabolismenya di dalam makhluk hidup. Karena banyaknya sekuens baru yang muncul diperlukan metode untuk mengklasifikasi protein ke dalam kelas yang sesuai. Tujuan dari penelitian ini adalah untuk menerapkan algoritme Probabilistic Neural Network (PNN) untuk melakukan klasifikasi protein family. Data protein family yang digunakan sebanyak tiga kelas, yaitu kelas 1-cysPrx_C, 4HBT dan ABC_Tran. Setiap kelas diambil sebanyak 100 data. Penelitian ini menggunakan ektraksi ciri rantai markov orde 1 dan 2. Nilai rata-rata akurasi terbaik Orde Satu sama dengan Nilai rata-rata akurasi terbaik Orde Dua sebesar 89.667%.
Kata kunci: klasifikasi,Probabilistic Neural Network, Ekstaksi Ciri Rantai Markov, Protein Family.
ABSTRACT
RIZKY KURNIAWAN. Protein Family Classification Based onProbabilistic Neural Network (PNN)Algorithm. Supervised by TOTO HARYANTO.
Proteins are composed of series and combination of 20 - amino acid. Each Protein has the composition of the amino - acid sequence which is different one from another. The classification of Protein is contributed as a guide to determine the structure, activity and role in the metabolism of organism. Since necessary to classifying proteins into appropriate classes, new sequences needed. Since the number of sequences that appear, new method of classifying proteins into appropriate classes. The purpose of this study is to apply the algorithm Probabilistic Neural Network ( PNN ) with four bias to classify the protein family. There are three classes of protein family data, namely class 1 - cysPrx_C , 4HBT and ABC_Tran. Each class is taken as 100 Data.
Keywords: classification, Probabilistic Neural Network (PNN), Feature Extraction
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Komputer
pada
Departemen Ilmu Komputer
KLASIFIKASI PROTEINFAMILY MENGGUNAKAN
ALGORITME PROBABILISTIC NEURAL NETWORK (PNN)
RIZKY KURNIAWAN
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2014
Penguji:
1. Dr. Wisnu Ananta Kusuma, S.T, M.T
Judul Skripsi : Klasifikasi Protein Family Menggunakan Algoritme Probabilistic
Neural Network (PNN)
Nama : Rizky Kurniawan NIM : G64096055
Disetujui oleh
Toto Haryanto, SKom MSi Pembimbing
Diketahui oleh
Dr Ir Agus Buono, MSi MKom Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga penulis dapat menyelesaikan skripsi yang berjudul “Klasifikasi Protein Family Menggunakan Algoritme Probabilistic Neural
Network (PNN)”. Shalawat serta salam penulis sampaikan kepada Nabi besar
Muhammad shallallahu ‘alaihi wasallam beserta keluarga, sahabat, serta umatnya hingga akhir zaman.
Terima kasih penulis ucapkan kepada seluruh pihak yang telah berperan membantu dalam penelitian ini, yaitu:
1. Ibu Suminah beserta Bapak Mudjijono atas kasih sayang, doa dan dorongan semangat yang telah diberikan kepada penulis sehingga penelitian ini dapat terselesaikan.
2. Kakak penulis Danu Aditya Yuda beserta keluarga yang telah memberikan dukungan dan semangat kepada penulis.
3. Nina Maria Priyatina, S. Kom beserta keluarga atas dukungan dan motivasi yang selama ini diberikan kepada penulis.
4. Bapak Toto Haryanto, S.Kom M.Si selaku pembimbing yang telah bersedia meluangkan waktunya untuk memberikan bantuan, arahan, serta kritik dan saran yang bermanfaat selama penelitian ini berlangsung.
5. Bapak Dr. Wisnu Ananta Kusuma, S.T, M.T dan Muhammad Asyhar Agmalaro, S.Si, M.Kom selaku dosen penguji yang bersedia meluangkan waktunya dalam seminar dan sidang.
6. Sony Muhammad rekan satu bimbingan penulis yang banyak bertukar fikiran dalam proses penyelesaian skripsi ini, serta Desta Sandya yang membantu dalam pembuatan aplikasi.
7. Teman-teman Ekstensi ILKOM angkatan 4 serta Rohis SMA N 4 Depok angkatan 2009, atas kerjasamanya selama penelitian.
Penulis menyadari bahwa masih terdapat banyak kekurangan dalam penulisan skripsi ini. Semoga karya ilmiah ini bermanfaat.
Bogor, 2014
DAFTAR ISI
DAFTAR TABEL vii
DAFTAR GAMBAR vii
DAFTAR LAMPIRAN viii
PENDAHULUAN 1 Latar Belakang 1 Tujuan 1 Manfaat Penelitian 2 Ruang Lingkup 2 TINJAUAN PUSTAKA 2 Protein 2 Sintesis Protein 3 Kode Genetik 4 ProteinFamily 6
Probabilistic Neural Network (PNN) 6
Ekstraksi Ciri Rantai Markov 7
K-Fold Cross Validation 9
METODE PENELITIAN 9
Kerangka Pemikiran 9
Pengambilan Data 10
Ekstraksi Ciri 10
Pembagian Data Latih dan Data Uji (K-Fold Cross Validation) 11
Analisa Hasil dan Model 11
Implementasi Aplikasi 12
HASIL DAN PEMBAHASAN 12
Pengambilan Data 12
Data Pelatihan dan Data Uji 13
Perhitungan Rata-rata Nilai Akurasi Orde 1 14
Perhitungan Rata-rata Nilai Akurasi Orde 2 16
SIMPULAN DAN SARAN 19
Saran 19
DAFTAR PUSTAKA 20
DAFTAR TABEL
1 Cofusion Matrix 11
2 Jenis protein yang digunakan dalam klasifikasi 13 3 Pembagian data uji dan data latih dengan 4-fold cross validation 14
4 Pengujian Orde 1 fold 1 14
5 Pengujian Orde 1 fold 2 14
6 Pengujian Orde 1 fold 3 15
7 Pengujian Orde 1 fold 4 15
8 Pengujian Orde 2 fold 1 16
9 Pengujian Orde 2 fold 2 17
10 Pengujian Orde 2 fold 3 17
11 Pengujian Orde 2 fold 4 18
DAFTAR GAMBAR
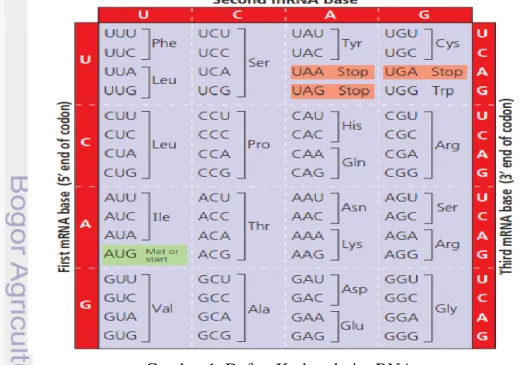
1 Daftar Kodon dari mRNA 5
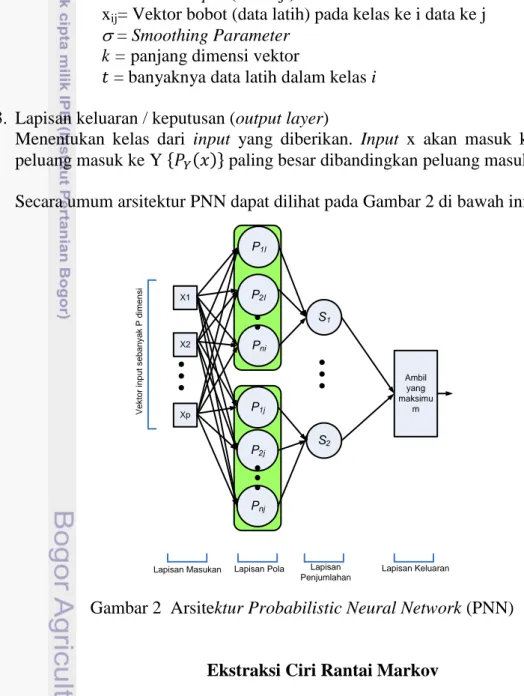
2 Arsitektur Probabilistic Neural Network (PNN) 7
3 Metode Penelitian 9
4 Contoh format FASTA 13
5 Hasil ekstraksi format FASTA 13
6 Grafik Nilai Rata-rata sensitivity dan specificity pada Orde 1 16 7 Grafik Perhitungan Nilai Rata-rata Akurasi pada Orde 1 16 8 Grafik Nilai Rata-rata sensitivity dan specificity pada Orde 2 16 9 Grafik Perhitungan Nilai Rata-rata Akurasi pada Orde 2 19
PENDAHULUAN
Latar Belakang
Protein adalah makromolekul yang paling banyak ditemukan di dalam sel makhluk hidup dan merupakan 50% atau lebih dari berat kering sel. Protein memiliki jumlah sangat bervariasi yang mulai dari struktur maupun fungsinya. Peranan protein diantaranya sebagai katalisator, pendukung, cadangan, sistem imun, alat gerak, sistem transport, dan respon kimiawi (Lehninger et al., 2004). Protein terdiri dari rangkaian dan kombinasi dari 20 asam-amino (amino-acid). Protein yang satu memiliki susunan sekuens asam-amino yang berbeda dengan protein lainnya. Klasifikasi protein sangat membantu sebagai petunjuk untuk mengetahui struktur, aktivitas dan peranan metabolismenya di dalam makhluk hidup. Terdapat beberapa keuntungan yang dapat diperoleh dari klasifikasi protein (Wu CH et.al 2003), diantaranya (1) memperbaiki hasil identifikasi protein yang sangat sulit apabila dilakukan dengan teknik pensejajaran pasangan asam amino (2) membantu dalam proses
maintenance yang ada dengan memberikan informasi berbasis family (3) membantu dalam
menemukembalikan informasi biologi yang relevan dari data sangat besar (4) membantu dalam melakukan analisis kekerabatan atau filogenetik.
Pangkalan data struktur primer protein yang berupa asam amino merupakan hasil translasi RNA dapat diklasifikasikan dengan beberapa kategori (Polanski dan Kimmel 2007) yaitu: (1) hierarchical family superfamilies/families pada tools PIR-PSD dan ProtoMap, (2) protein domain pada tools Pfam dan ProDom , (3) motif sekuens pada tools PROSITE dan PRINT, (4) struktur kelas protein pada tools SCOP dan CATH, dan (5) integrasi berbagai family pada tools iProClassn dan InterPro. Semua pangkalan data tersebut memiliki kegunaan yang spesifik.
Penelitian yang terkait dengan klasifikasi protein family pernah dilakukan oleh Muhammad (2013) menggunakan metode Rantai Markov. Data yang digunakan pada penelitian Muhammad sama dengan data yang digunakan pada penelitian ini, yaitu terbagi menjadi tiga kelas protein family yaitu kelas 1-cysPrx_C, 4HBT, dan ABC_Tran. Orde satu menghasilkan akurasi terbaik sebesar 96% dan orde dua menghasilkan akurasi terbaik sebesar 89.33%.
Penelitian terkait dilakukan oleh Fajar (2013). Fajar menggunakan metode ektraksi ciri Rantai Markov dengan orde satu dan orde dua serta metode klasifikasi Probabilistic
Neural Network (PNN) dengan data berupa sequence DNA. Penelitian ini menghasilkan
nilai sensitivity terbaik 0,7136 dan specificity terbaik 0,9284. Hasil ini diperoleh ketika mengunakan panjang sequence 1000 bp dan metode ekstraksi ciri Rantai Markov Orde Dua.
Berdasarkan latar belakang tersebut, maka pada penelitian ini ditujukan untuk mengimplementasikan algoritme Probabilistic Neural Network (PNN) dalam melakukan klasifikasi terhadap protein family.
Tujuan
Penelitian ini bertujuan untuk:
Menghasilkan model klasifikasi menggunakan metode ekstraksi ciri Rantai Markov Orde Satu dan Orde Dua dengan algoritme Probabilistic Neural Network (PNN)
2
Mengidentifikasi protein family terhadap tiga kelas protein family, yaitu kelas 1-cysPrx_C, 4HBT, dan ABC_Tran.
Manfaat Penelitian
Manfaat dari penelitian ini adalah dapat mengklasifikasikan protein family dengan menggunakan algoritme Probabilistic Neural Network (PNN) serta dapat menghasilkan sistem identifikasi protein family terhadap tiga kelas protein family yaitu kelas 1-cysPrx_C, 4HBT, dan ABC_Tran.
Ruang Lingkup
Ruang lingkup penelitian ini adalah:
Struktur protein yang diklasifikasikan hanya sampai pada tingkat struktur primer berupa deretan asam amino.
Data yang digunakan adalah data yang diambil dari pangkalan data Pfam dengan kategori protein domain. Data dapat di diunduh pada alamat ftp://ftp.sanger.ac.uk/pub/databases/pfam.
TINJAUAN PUSTAKA
Protein
Protein merupakan zat gizi yang sangat penting, karena yang paling erat hubungannya dengan proses-proses kehidupan. Nama protein berasal dari bahasa Yunani (Greek) proteus yang berarti “yang pertama” atau “yang terpenting”. Seorang ahli kimia Belanda yang bernama Mulder, mengisolasi susunan tubuh yang mengandung nitrogen dan menamakannya protein, terdiri dari satuan dasarnya yaitu asam amino (biasa disebut juga unit pembangun protein) (Suhardjo dan Clara, 1992).
Dalam proses pencernaan, protein akan dipecah menjadi satuan-satuan dasar kimia. Protein terbentuk dari unsur-unsur organik yang hampir sama dengan karbohidrat dan lemak yaitu terdiri dari unsur karbon (C), hidrogen (H), dan oksigen (O), akan tetapi ditambah dengan unsure lain yaitu nitrogen (N). Molekul protein mengandung pula fosfor, belerang, dan ada jenis protein yang mengandung unsur logam seperti besi dan tembaga. Molekul protein tersusun dari satuan-satuan dasar kimia yaitu asam amino. Dalam molekul protein, asam-asam amino ini saling berhubung-hubungan dengan suatu ikatan yang disebut ikatan peptida (CONH). Satu 4 molekul protein dapat terdiri dari 12 sampai 18 macam asam amino dan dapat mencapai jumlah ratusan asam amino (Suhardjo dan Clara, 1992).
Protein adalah molekul makro yang mempunyai berat molekul antara lima ribu hingga beberapa juta. Protein terdiri atas rantai-rantai asam amino, yang terikat satu sama lain dalam ikatan peptida. Asam amino yang terdiri atas unsur-unsur karbon, hidrogen, oksigen dan nitrogen, beberapa asam amino disamping itu mengandung unsur-unsur fosfor, besi, iodium, dan cobalt. Unsur nitrogen adalah unsur utama protein, karena terdapat di dalam semua protein akan tetapi tidak terdapat di dalam karbohidrat dan lemak. Unsur
3 nitrogen merupakan 16% dari berat protein. Molekul protein lebih kompleks daripada karbohidrat dan lemak dalam hal berat molekul dan keanekaragaman unit-unit asam amino yang membentuknya (Almatsier. S, 1989).
Berikut adalah 20 asam amino yang menyusun protein, dikelompokan menurut sifat dan struktur kimiawinya yaitu (1) Asam amino alifatik sederhana terdiri dari: Glisina (Gly, G), Alanina (Ala, A), Valina (Val, V), Leusina (Leu, L), Isoleusina (Ile, I), (2) Asam amino hidroksi-alifatik terdiri dari: Serina (Ser, S), Treonina (Thr, T), (3) Asam amino dikarboksilat (asam) terdiri dari: Asam aspartat (Asp, D), Asam glutamat (Glu, E), (4) Amida terdiri dari: Asparagina (Asn, N), Glutamina (Gln, Q), (5) Asam amino basa terdiri dari: Lisina (Lys, K), Arginina (Arg, R), Histidina (His, H) (memiliki gugus siklik), (6) Asam amino dengan sulfur terdiri dari: Sisteina (Cys, C), Metionina (Met, M), (7) Prolin: Prolina (Pro, P) (memiliki gugus siklik), (8) Asam amino aromatic terdiri dari: Fenilalanina (Phe, F), Tirosina (Tyr, Y), Triptofan (Trp, W).
Struktur protein dapat dilihat secara hierarki sebagai struktur primer, sekunder dan tersier (Polanski dan Kimmel 2007), yaitu : (1) Struktur Primer, yaitu sebuah protein yang tersusun dari serangkaian asam amino, (2) Struktur Sekunder, yaitu struktur primer yang berinteraksi dengan ikatan hidrogen sehingga menghasilkan struktur baru. Bentuk – bentuk struktur sekunder yang paling umum yaitu struktur alpha helix, beta sheets dan coil, (3) Struktur tersier, merupakan variasi dari berbagai bentuk struktur sekunder yang kemudian menghasilkan struktur yang baru, (4) Struktur kuartener, yaitu struktur yang dibangun dari sejumlah molekul protein.
Sintesis Protein
Proses sintesis atau pembentukan protein memerlukan adanya molekul RNA yang merupakan materi genetik di dalam kromosom, serta DNA sebagai pembawa sifat keturunan.Gen menspesifikasikan protein melalui transkripsi dan translasi (Campbell et al. 2011).
Gen menyediakan instruksi untuk membuat protein spesifik. Akan tetapi, gen tidak membangun protein secara langsung. Jembatan antara DNA dan sintesis protein adalah asam nukleat RNA. RNA mirip dengan DNA secara kimiawi, hanya saja RNA mengandung gula ribosa sebagai pengganti deoksiribosa dan mengandung basa bernitrogen urasil sebagai pengganti timin. Dengan demikian, setiap nukleotida di sepanjang untai DNA mengandung A, G, C, atau T sebagai basanya, sedangkan setiap nukleotida disepanjang untai RNA mengandung A, G, C, atau U sebagai basanya. Molekul RNA biasanya terdiri atas satu untai tunggal (Campbell et al. 2011).
Dalam RNA atau DNA, monomer adalah keempat tipe nukleotida, yang berbeda dalam kandungan basa benitrogen. Gen umumnya memiliki panjang yang mencapai ratusan atau ribuan nukleotida; masing-masing gen mengandung sekuens basa spesifik. Setiap polipeptida dari suatu protein juga mengandung monomer-monomer yang tertata dalam urutan linear tertentu ( struktur primer protein ), namun monomer-monomernya merupakan asam amino. Dengan demikian, asam nukleat dan protein mengandung informasi yang tertulis dalam dua bahasa kimiawi yang berbeda. Ini membutuhkan dua tahap utama dari DNA ke protein, yaitu transkripsi dan translasi (Campbell et al. 2011).
1. Transkripsi
Transkripsi adalah sintesis RNA dibawah arahan DNA. Kedua asam nukleat menggunakan bahasa yang sama, dan informasi hanya ditranskripsi, atau disalin, dari
4
satu molekul menjadi molekul lain. Selain menjadi cetakan untuk sintesis untai komplementer baru saat replikasi DNA, untai DNA juga bisa berperan sebagai cetakan untuk merakit sekuens nukleotida RNA komplementer. Untuk gen pengode protein, molekul RNA yang dihasilkan merupakan transkrip akurat dari instruksi pembangun protein yang dikandung oleh gen. molekul RNA transkrip bisa dikirimkan dalam banyak salinan. Tipe molekul RNA ini disebut RNA duta (messenger RNA, mRNA) karena mengandung pesan genetik dari DNA ke mekanisme penyintesis protein sel. 2. Translasi
Translasi adalah sintesis polipeptida yang terjadi dibawah arahan mRNA. Selama tahap ini terjadi perubahan bahasa. Sel harus menerjemahkan alias menstranslasikan sekuens basa molekul mRNA menjadi sekuens asam amino polipeptida. Tempat terjadinya translasi adalah ribosom, partikel-partikel kompleks yang memfasilitasi penautan teratur asam amino menjadi rantai polipetida. Translasi merupakan proses penerjemahan beberapa triplet atau kodon dari mRNA menjadi asam amino-asam amino yang akhirnya membentuk protein. Urutan basa nitrogen yang berbeda pada setiap triplet, akan diterjemahkan menjadi asam amino yang berbeda. Misalnya, asam amino fenilalanin diterjemahkan dari triplet UUU (terdiri dari 3 basa urasil), asam amino triptofan (UGG), asam amino glisin (GGC), dan asam amino serin UCA. Sebanyak 20 macam asam amino yang diperlukan untuk pembentukan protein merupakan hasil terjemahan triplet dari mRNA. Selanjutnya, dari beberapa asam amino (puluhan, ratusan, atau ribuan) tersebut dihasilkan rantai polipeptida spesifik dan akan membentuk protein spesifik pula.
Kode Genetik
Sintesis protein dikodekan dalam DNA, hanya terdapat empat macam basa nukleotida untuk menspesifikasikan 20 asam amino. Aliran informasi dari gen ke protein didasarkan padakode triplet (triplet code): instruksi-instruksi genetic untuk rantai polilpetida ditulis dalam DNA sebagai rangkaian kata tiga nukleotida yang tidak tumpang tindih. Misalnya, triplet basa AGT diposisi tertentu pada seuntai DNA menghasilkan penempatan asam amino serin pada posisi yang bersesuaian dari polipeptida yang sedang dibuat. Untuk setiap gen, hanya satu dari kedua untai DNA yang ditranskripsikan. Untaian ini disebut untai cetakan (template strand) karena menyediakan pola, atau cetakan, untuk sekuens nukleotida-nukleotida dalam transkrip RNA. Untai DNA tertentu merupakan untai cetakan untuk beberapa gen disepanjang molekul DNA, sedangkan untuk gen-gen lain untai komplementerlah yang berfungsi sebagai cetakan. Perlu diperhatikan untuk suatu gen, untai yang sama digunakan sebagai cetakan setiap kali gen tersebut ditranskripsi. Perpasangan serupa dengan yang terbentuk saat replikasi DNA, hanya saja U, pengganti untuk T pada RNA, berpasangan dengan A, dan nukleotida mRNA mengandung ribosa, bukan deoksiribosa. Seperti untai baru DNA, molekul RNA disintesis dengan arah yang anti pararel terhadap untai cetakan DNA. Misalnya triplet basa ACC pada DNA ( ditulis sebagai 3’-ACC-5’ ) menjadi cetakan untuk 5’-UGG-3’ pada molekul mRNA. Triplet basa mRNA disebut kodon ( codon ), dan biasanya ditulis dengan arah 5’ à 3’. Dalam contoh kita, UGG merupakan kodon untuk asam amino triptofan ( disingkat Trp ). Istilah kodon juga digunakan untuk triplet basa disepanjang untai bukan cetakan ( non template ). Kodon-kodon ini komplementer terhadap untai cetakan, dan dengan demikian bersekuens
5 identik dengan mRNA hanya saja mengandung T bukan U. (karena alasan ini, untai DNA bukan cetakan, terkadang disebut ‘untai pengode’).
Saat translasi, sekuens kodon di sepanjang molekul mRNA diterjemahkan atau ditranslasi, menjadi sekuens asam amino yang menyusun rantai polipeptida. Karena kodon merupakan triplet basa, jumlah nukleotida yang menyusun suatu pesan genetik pastilah tiga kali lebih banyak daripada jumlah asam amino dalam protein yang dihasilkan. Misalnya 300 nukleotida diperlukan disepanjang satu untai mRNA untuk menodekan asam-asam amino dalam polipeptida yang panjangnya 100 asam amino.
Kodon pertama kali dipecahkan pada tahun 1961 oleh Marshal Nirenberg dan Mathei. Mereka melakukan percobaan menggunakan E. coli dengan asam poli urasil. Menurut hasil percobaan tersebut, cetakan UUU yang dibawa oleh mRNA, artinya adalah asam amino fenilalanin. Dengan cara yang sama, triplet CCC diartikan sebagai prolin dan triplet AAA artinya asam amino lisin. Pada kamus kode genetik terdapat 64 kombinasi triplet untuk 20 asam amino. Jadi, terdapat asam amino tertentu melebihi triplet ( kodon). Setiap triplet disusun oleh 3 basa nitrogen. Rangkaian tiga basa nitrogen yang menyusun kode disebut triple atau trikodon atau kodon. Rangkaian tiga basa nitrogen yang ada pada DNA yang bertugas membuat kode-kode disebut kodogen ( agen pengkode ). Dari 64 triplet hanya 61 triplet yang mengodekan asam amino (lihat tabel kodon) . Ketiga kodon yang tidak mengodekan asam amino merupakan sinyal ‘stop’, atau kodon terminasi, yang menandai akhir translasi. Perhatikan bahwa kodon AUG berfungsi ganda: mengodekan asam amino metionin ( Met ) dan berfungsi sebagai sinyal ‘mulai’, atau kodon inisiasi. Pesan-pesan genetik dimulai dengan kodon mRNA, AUG, yang memberi sinyal pada mekanisme penyintesis protein untuk mulai mentraslasikan mRNA pada lokasi itu. ( karena AUG juga mengodekan metionin, rantai polipeptida diawali oleh Metionin saat disintesis. Akan tetapi, sejenis enzim mungkin akan segera menyingkirkan asam amino strarter dari rantai tersebut (Campbell et al. 2011).
6
ProteinFamily
Protein-protein yang dikelompokkan ke dalam kelas yang sama, pada dasarnya memiliki keterhubungan secara evolusi. Protein-protein yang dikelompokkan ke dalam kelas yang sama, memiliki nenek moyang yang sama (ancestor), dan secara umum memiliki kemiripan, baik dalam struktur tiga dimensinya, fungsi, maupun sekuens penyusunnya. Meskipun seringkali sulit untuk mengevaluasi seberapa signifikan baik tingkat kemiripan fungsi maupun struktur antara protein yang satu dengan yang lain, pada dasarnya protein yang tidak memiliki ancestor yang sama, memiliki perbedaan yang cukup signifikan pada sekuens penyusunnya. Hal ini menyebabkan banyak dikembangkannya metode untuk mengklasifikasikan protein family berdasarkan sekuens asam amino penyusunnya. Saat ini, telah ditemukan lebih dari 60.000 kelas family (Kunin et al. 2003) meskipun masih terdapat banyaknya ambiguitas mengenai definisi yang paling tepat mengenai protein family, menyebabkan banyaknya perbedaan hasil yang ditemukan antara peneliti yang satu dengan yang lain.
Probabilistic Neural Network (PNN)
PNN merupakan Artificial Neural Network (ANN) yang menggunakan teorema probabilitas klasik. PNN diperkenalkan oleh Donald Specht pada tahun 1990. PNN menggunakan pelatihan (training) supervised. Keuntungan utama menggunakan arsitektur PNN adalah Training data PNN mudah dan cepat (Wu et al. 2007).
Probabilistic Neural Network (PNN) merupakan Jaringan Saraf Tiruan (JST) yang
menggunakan teorema probabilitas klasik seperti pengklasifikasian Bayes dan penduga kepekatan Parzen. Proses yang dilakukan oleh PNN dapat berlangsung lebih cepat bila dibandingkan dengan JST Back Propagation. Hal ini disebabkan PNN hanya membutuhkan satu kali iterasi pelatihan bila dibandingkan dengan JST Back Propagation yang membutuhkan beberapa kali iterasi pelatihan (Specht 1990).
Struktur PNN terdiri atas empat lapisan, seperti yang terlihat pada Gambar 1. yaitu
input layer, pattern layer, summation layer, dan decision layer. Input layer merupakan
objek yang terdiri atas nilai ciri yang akan diklasifikasikan pada n kelas. Nilai-nilai k kemudian akan membentuk sebuah vektor masukan, vektor x. Proses-proses yang terjadi setelah fase input layer adalah:
1. Lapisan pola (pattern layer)
Pattern layer menggunakan satu node untuk setiap data pelatihan yang digunakan.
Setiap node pola merupakan selisih dari vektor masukan dengan vektor bobot xij, yaitu
rij = x - xij.rij kemudian dibagidengan faktor bias σ sehingga menghasilkan Pij. Dengan
demikian, persamaan yang digunakan adalah :
Ket: x= Vektor input (data uji)
xij= Vektor bobot (data latih) kelas ke i data ke j
7
2. Lapisan penjumlahan (summation layer)
Summation layer menerima masukan dari setiap node pattern layer yang terkait dengan
kelas yang ada. Hasil dari pattern layer setiap kelas diwakilkan oleh .Persamaan yang digunakan pada lapisan ini adalah:
Ket: x = Vektor input (data uji)
xij= Vektor bobot (data latih) pada kelas ke i data ke j
= Smoothing Parameter
k = panjang dimensi vektor
= banyaknya data latih dalam kelas i 3. Lapisan keluaran / keputusan (output layer)
Menentukan kelas dari input yang diberikan. Input x akan masuk kelas Y jika nilai peluang masuk ke Y paling besar dibandingkan peluang masuk ke kelas lainnya. Secara umum arsitektur PNN dapat dilihat pada Gambar 2 di bawah ini :
Ekstraksi Ciri Rantai Markov
Rantai Markov adalah pemograman dinamis stokastik untuk menangkap perilaku
user. Diperkenalkan oleh matematikawan Rusia bernama Prof Andrei A. Markov
(1856-1922) dan pertama kali diterbitkan hasilnya pada tahun 1906. Dengan menggunakan proses
X1 X2 Xp P1I Pni P1j P2j Pnj S1 S2 Ambil yang maksimu m V e k to r in p u t s e b a n y a k P d im e n s i
Lapisan Masukan Lapisan Pola Lapisan
Penjumlahan
Lapisan Keluaran P2I
8
Markov maka dimungkinkan untuk memodelkan fenomena stokastik dalam dunia nyata yang berkembang menurut waktu. Masalah dasar dari metode stokastik dengan proses Markov adalah menentukan deskripsi state yang sesuai, sehingga proses stokastik yang berpaduan akan benar-benar memiliki apa yang akan disebut sifat Markov (Markovian
property), yaitu pengetahuan terhadap state ini adalah cukup untuk memprediksi perilaku
stokastik yang akan datang (Ching dan Ng, 2006).
Suatu rantai Markov dikatakan diskret (discrete time Markov chain) jika ruang dari proses Markov tersebut adalah himpunan terbatas (finite) atau tercacah (countable), dengan himpunan indeks adalah T = (0, 1, 2, …). Jika nilai suatu state pada periode tertentu hanya bergantung pada satu periode sebelumnya, maka rantai tersebut disebut rantai Markov orde satu (first order Markov chain) dan jika nilai suatu state pada periode tertentu bergantung pada periode sebelumnya, maka rantai tersebut disebut rantai Markov orde m (m order
Markov chain). Rantai Markov orde satu secara matematika dirumuskan sebagai berikut.
Adapun rantai Markov orde m (m order Markov chain) secara matematika dirumuskan sebagai berikut.
Peluang bahwa Xn+1 berada pada state j jika Xn berada pada state i disebut sebagai
peluang transisi satu langkah (one step transition probability). Secara matematis dapat terlihat pada rumus di bawah ini:
Jika peluang ini bebas dari indeksnya, maka peluang ini disebut proses Markov dengan peluang transisi stasioner. Sehingga peluang transisi tersebut dirumuskan sebagai berikut:
Peluang transisi ini disusun dalam bentuk matriks, yang disebut peluang matriks transisi P, yang setiap unsurnya adalah Pi j yaitu P{Xn+1
= j | X
n= i} Matriks tersebut
terdapat di bawah ini:
P{X
n+1= j | X
n= i }
9 K-Fold Cross Validation
Cross validation merupakan metode untuk memperkirakan generalisasi galat
berdasarkan “resampling” K-fold cross validation membagi data menjadi k subset yang ukurannya hampir sama satu sama lain. Subset yang dihasilkanyaitu S1, S2, S3,..,Sk yang
digunakan sebagai data pelatihan dan data pengujian. Dalam metode ini dilakukan perulangan sebanyak k kali. Setiap kali perulangan, salah satu subset akan dijadikan data uji dan k-1 subset lainnya dijadikan sebagai data latih. Pada perulangan ke-i, subset digunakan sebagai data pengujian dan subset lainnya digunakan sebagai data pelatihan, dan seterusnya (Hastie et al. 2011).
METODE PENELITIAN
Kerangka Pemikiran
Penelitian ini dilakukan dengan beberapa tahapan proses untuk mengetahui tingkat akurasi yang diperoleh menggunakan algoritme Probabilstic Neural Network (PNN). Tahapan-tahapan mulai dari pengambilan data hingga implementasi akan dijelaskan pada ilustrasi sesuai Gambar 3.
10
Pengambilan Data
Data protein family diperoleh dari Pfam yang merupakan pusat database komprehensif yang meliputi berbagai macam koleksi data. Pfam memiliki koleksi kurang lebih 12.000 data protein family yang diperoleh dengan komputasional, eksperimen, penelitian dan penelian biologi mengenai spesifik protein, bentuk asal serta hasil evolusinya. Pfam juga telah dipergunakan secara luas oleh berbagai macam komunitas yang bergerak di bidang structural biologi untuk mengidentifikasi jenis protein baru.
Data yang digunakan terdiri dari tiga kelas protein family yaitu kelas 1-cysPrx_C, 4HBT dan ABC_Tran, yang masing-masing kelasnya mempunyai seratus data protein dengan format FASTA. Data dapat diunduh di http://www.pfam.sanger.co.uk.
Ekstraksi Ciri
Pada penelitian ini, matriks transisi akan dibangun berukuran 20 × 20 sesuai dengan banyaknya monomer atau jenis asam amino yang ada. Metode penyimpanan nilai sekuens ke dalam matriks transisi hanya menggunakan orde satu. Matriks transisi Rantai Markov Orde Satu dibentuk dari peluang-peluang munculnya komponen asam amino tertentu setelah sebelumnya merupakan tepat satukomponen asam amino tertentu. Dengan demikian matriks transisi Rantai Markov Orde Satu terdiri atas P(G|G), yaitu peluang munculnya komponen asam amino G setelah sebelumnya merupakan komponen asam amino G, P(G|A), P(G|V), P(G|I) dan seterusnya. Matrik transisi Rantai Markov Orde satu dapat dilihat dibawah ini:
Matriks transisi Rantai Markov Orde Dua dibentuk dari peluang-peluang munculnya komponen asam amino tertentu setelah sebelumnya merupakan tepat dua komponen asam amino tertentu. Dengan demikian matriks transisi Rantai Markov Orde Dua terdiri atas P(G|G), yaitu peluang munculnya komponen asam amino G setelah dua komponen sebelumnya merupakan komponen asam amino G, P(G|A), P(G|V), P(G|I) dan seterusnya.
Proses ekstraksi ciri dilakukan pada semua data, baik data latih maupun data uji. Jadi akan terdapat 300 matriks data hasil ektraksi ciri yang akan diproses ke tahap selanjutnya. Hasil ekstraksi ciri yang berupa matriks berukuran 20x20 lalu diubah menjadi sebuah vector berukuran 1x400 dengan cara meletakan baris kedua pada matrix tersebut setelah baris pertama, lalu baris ketiga setelah baris pertama dan kedua begitu seterusnya hingga terbentuk vector berukuran 1x400.
11
Pembagian Data Latih dan Data Uji (K-Fold Cross Validation)
Setelah dilakukan ekstraksi ciri kemudian dibagi menjadi data latih dan data uji meenggunakan metode k-fold cross validation dengan k = 4. Semua data akan dibagi menjadi 4 fold yaitu D1, D2, D3, D4 yang masing-masing fold memiliki data yang sama. Proses identifikasi akan dilakuakan 4 kali iterasi berdasarkan k-fold cross validation dan data latih serta data uji memiliki fold yang berbeda pada setiap iterasinya sehingga terciptanya model klasifikasi yang terbaik.
Probabilistic Neural Network (PNN)
Pada tahap ini dilakukan algoritme Probabilistic Neural Network (PNN) dengan data latih dan data uji yang telah dibagi pada proses k -fold cross validation. Pada setiap fold setiap data latih akan diproses ke semua data latih yang ada, dan hasilnya akan dijumlahkan sesuai dengan kelasnya masing-masing untuk menghitung besarnya peluang terhadap tiga kelas yang tersedia. Hasilnya akan ditentukan dengan melihat nilai peluang yang terbesar. Nilai bias yang digunakan pada proses ini adalah satu.
Analisa Hasil dan Model
Analisa hasil dan model dilakukan untuk memastikan bahwa model algoritme
Probabilistic Neural Network (PNN) yang dihasilkan adalah model yang terbaik. Dengan
model terbaik, akan menghasilkan akurasi yang paling tinggi dalam proses klasifikasi sehingga model tersebut yang nantinya akan digunakan pada saat implementasi.
Selain itu juga dilakukan analisis perhitungan nilai sensitivity dan specificity untuk setiap kelas yang ada. Dalam pencarian nilai sensitivity dan specificity dibutuhkan suatu matrix yang disebut confusion matrix.
Confusion matrix adalah sebuah tabel yang digunakan untuk memvisualisasikan
kinerja suatu algoritme. Setiap kolom dalam matriks merepresentasikan kelas yang diprediksikan, sedangkan setiap baris merepresentasikan kelas yang sebenarnya.
Contoh table of confusion perhitungan nilai sensitivity dan specificity untuk kelas
1-cysPrx_C dapat dilihat pada Tabel 1 di bawah ini. Tabel 1 Cofusion Matrix
Terdeteksi sebagai
sequence asam amino
kelas 1-cysPrx_C
Terdeteksi sebagai bukan sequence asam
amino kelas
1-cysPrx_C
Sequence asam amino uji
kelas 1-cysPrx_C tp fn
Bukan sequence asam
amino uji kelas
1-cysPrx_C
12
Keterangan:
tp : true positive (jumlah sequence DNA uji genus A yang tepat teridentifikasi
sebagai sequence DNA genus A)
tn : true negative (jumlah bukan sequence DNA uji genus A yang tepat
teridentifikasi sebagai bukan sequence DNA genus A)
fp : false positive (jumlah bukan sequence DNA uji genus A yang
teridentifikasi sebagai sequence DNA genus A)
fn : false negative (jumlah sequence DNA uji genus A yang teridentifikasi
sebagai bukan sequence DNA genus A)
Sensitivity mengukur proporsi positif yang diidentifikasi dengan benar, sedangkan specificity mengukur proporsi negatif yang diidentifikasi dengan benar.
Persamaan dari nilai Sensitivity adalah:
Persamaan dari nilai specificity adalah:
Dalam penelitian ini digunakan tiga kelas sehingga akan diperoleh tiga nilai sensitivity dan specificity. Ketiga nilai sensitivity dan specificity pada setiap fold tersebut kemudian dirata-ratakan sehingga diperoleh nilai sensitivity dan specificity yang merepresentasikan setiap fold.
Implementasi Aplikasi
Setelah mengetahui model yang terbaik kemudian diimplementasikan kedalam bentuk program berbasis website. Program ini akan membantu pengguna untuk mengklasifikasi protein family.
HASIL DAN PEMBAHASAN
Pengambilan Data
Data yang digunakan pada penelitian ini adalah kumpulan protein family yang terdapat pada pangkalan data bernama Pfam. Jenis-jenis protein yang di gunakan pada penelitian ini dapat dilihat pada Tabel 2.
13 Tabel 2 Jenis protein yang digunakan dalam klasifikasi
No Nama Keterangan
1 1-cysPrx_C C-terminal domain dari 1-Cys peroxiredoxin
(1-cysPrx), anggota dari superfamili peroxiredoxin yang melindungi sel terhadap oksidasi membrane.
2 4HBT Berfungsi untuk melakukan katalisasi dalam
proses biosintesis.
3 ABC_Tran Atau disebut juga ABC Transporter yang
menggunakan hidrolisis Adenosina trifosfat (ATP) untuk mentranslokasi berbagai senyawa melintasi membran biologis.
Data diambil secara acak. Setiap kelas masing-masing diambil seratus data. Sehingga terdapat tiga ratus data dalam penelitian ini. Pembagian data latih dan data uji dilakukan dengan menggunakan metode k-fold cross validation dengan k = 4, dimana terdiri dari 75% digunakan untuk data latih dan 25% digunakan untuk data uji. Data yang didapat adalah salah satu tipe data biologi yang disediakan yaitu FASTA. Contoh data dapat dilihat pada Gambar 4.
>B3ARY6_ECO57/154-186 PF10417.3;1-cysPrx_C; merupakan identifier pfam dengan 1-cysPrx_C merupakan keterangan kelas proteinfamily. Adapun deretan huruf dibawahnya merupakan sekuens asam amino atau protein. Setelah ini dilakukan ekstraksi data sehingga terjadi pemisahan antara identifier dengan sekuensnya. Lebih jelasnya dapat dilihat pada Gambar 5 berikut.
Data Pelatihan dan Data Uji
Data yang digunakan sebanyak tiga ratus data yang terbagi menjadi tiga kelas. Jadi masing-masing kelas terdiri dari seratus data. Setiap kelas terdiri dari 75 data latih dan 25 data uji. Pembagian data latih dan data uji menggunakan k-fold cross validation agar semua data pernah dijadikan data latih. Pembagian data menggunakan k-fold cross
validation dapat dilihat pada Tabel 3 berikut.
>B3ARY6_ECO57/154-186 PF10417.3;1-cysPrx_C; AAQYVASHPGEVCPAKWKEGEATLAPSLDLVGK
AAQYVASHPGEVCPAKWKEGEATLAPSLDLVGK Gambar 4 Contoh format FASTA
14
Tabel 3 Pembagian data uji dan data latih dengan
4-fold cross validation
fold Data Uji Data Latih
1 D4 D1, D2, D3
2 D3 D1, D2, D4
3 D2 D1, D3, D4
4 D1 D2, D3, D4
Perhitungan Rata-rata Nilai Akurasi Orde 1
Hasil perhitungan rata-rata nilai akurasi dengan menggunakan Ekstaksi Ciri Rantai Markov Orde 1 fold 1 menunjukkan akurasi setiap kelas dengan persentase seperti dilihat pada Tabel 4.
Tabel 4 Pengujian Orde 1 fold 1
1-cysPrx_C 4HBT ABC_Tran Akurasi (%)
1-cysPrx_C 22 0 3 88.00
4HBT 0 22 3 88.00
ABC_Tran 0 0 25 100.00
Rata-rata 92.00
Hasil akurasi pada Orde 1 fold 1 diperoleh rata-rata sebesar 92%. Kelas 1-cysPrx_C dapat terklasifikasi dengan benar sebanyak dua puluh dua, tiga sisanya terklasifikasi ke dalam kelas ABC_Tran. Kelas 4HBT dapat terklasifikasi dengan benar sebanyak dua puluh dua, tiga sisanya terklasifikasi ke dalam kelas ABC_Tran. Sedangkan Kelas ABC_Tran dapat terklasifikasi semua dengan benar.
Hasil perhitungan rata-rata nilai akurasi dengan menggunakan Ekstaksi Ciri Rantai Markov Orde 1 fold 2 menunjukkan akurasi setiap kelas dengan persentase seperti dilihat pada Tabel 5.
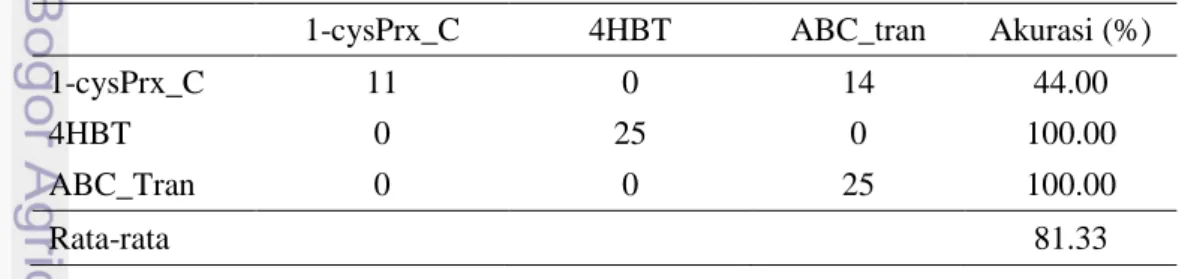
Tabel 5 Pengujian Orde 1 fold 2
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 11 0 14 44.00
4HBT 0 25 0 100.00
ABC_Tran 0 0 25 100.00
Rata-rata 81.33
Hasil akurasi pada Orde 1 fold 2 diperoleh rata-rata sebesar 81.33%. Kelas 1-cysPrx_C dapat terklasifikasi dengan benar sebanyak sebelas, empat belas sisanya terklasifikasi ke dalam kelas ABC_Tran. Sedangkan kelas 4HBT dan ABC_Tran dapat terklasifikasi semua dengan benar.
15 Hasil perhitungan rata-rata nilai akurasi dengan menggunakan Ekstaksi Ciri Rantai Markov Orde 1 fold 3 menunjukkan akurasi setiap kelas dengan persentase seperti dilihat pada Tabel 6.
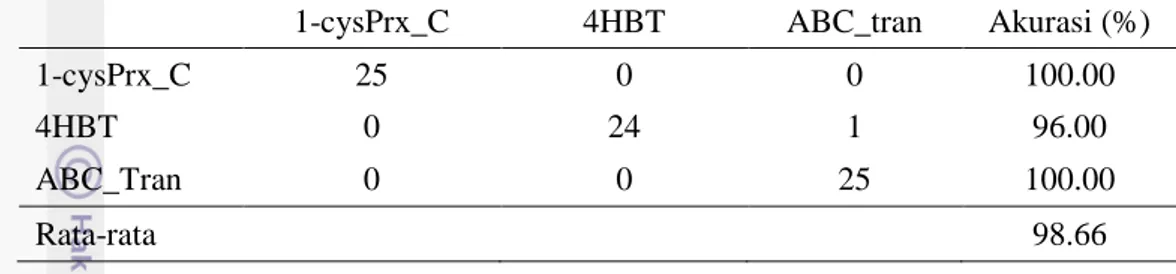
Tabel 6 Pengujian Orde 1 fold 3
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 25 0 0 100.00
4HBT 0 24 1 96.00
ABC_Tran 0 0 25 100.00
Rata-rata 98.66
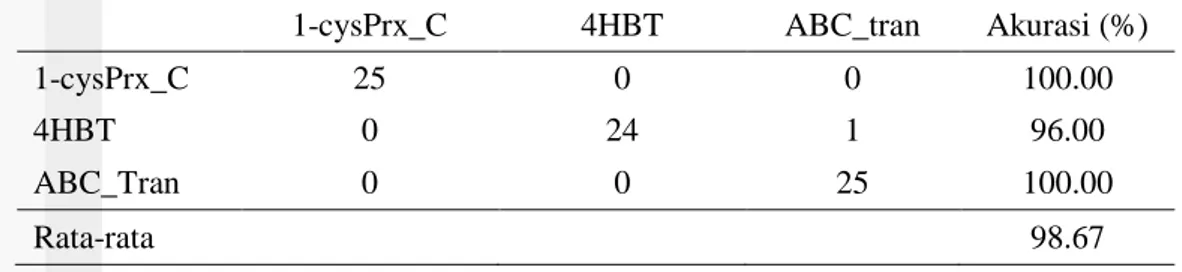
Hasil akurasi pada Orde 1 fold 3 diperoleh rata-rata sebesar 98.66%. Kelas 4HBT dapat terklasifikasi dengan benar sebanyak dua puluh empat, satu sisanya terklasifikasi ke dalam kelas ABC_Tran. Sedangkan kelas 1-cysPrx_C dan ABC_Tran dapat terklasifikasi semua dengan benar.
Hasil perhitungan rata-rata nilai akurasi dengan menggunakan Ekstaksi Ciri Rantai Markov Orde 1 fold 4 menunjukkan akurasi setiap kelas dengan persentase seperti dilihat pada Tabel 7.
Tabel 7 Pengujian Orde 1 fold 4
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 25 0 0 100.00
4HBT 0 11 14 44.00
ABC_Tran 0 0 25 100.00
Rata-rata 81.33
Hasil akurasi pada Orde 1 fold 4 diperoleh rata-rata sebesar 81.33%. Kelas 4HBT dapat terklasifikasi dengan benar sebanyak sebelas, empat belas sisanya terklasifikasi ke dalam kelas ABC_Tran. Sedangkan kelas 1-cysPrx_C dan ABC_Tran dapat terklasifikasi semua dengan benar.
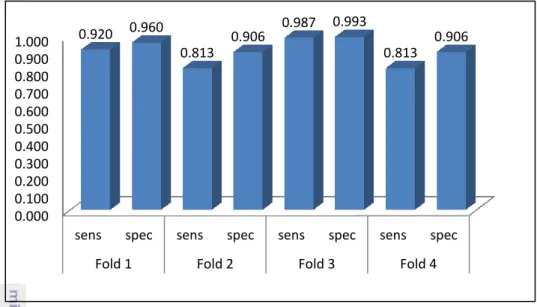
Setelah dilakukan Hasil perhitungan rata-rata nilai akurasi menggunakan Ekstaksi Ciri Rantai Markov Orde 1 dan klasifikasi menggunakan Probabilistic Neural Network (PNN), selanjutnya menghitung rata-rata nilai sensitivity dan specificity. Nilai sensitivity dan specificity yang dihasilkan oleh setiap fold pada Orde 1 dapat dilihat pada Gambar 6.
Pada Gambar 6 menunjukan bahwa nilai rata-rata sensitivity tertinggi pada Orde 1 terletak pada fold 3 dengan nilai 0.987 dan nilai rata-rata specificity tertinggi yang pada Orde 1 terletak pada fold 3 dengan nilai 0.993.
Berasarkan grafik pada Gambar 6 dan Gambar 7, dapat dilihat bahwa model terbaik yang dihasilkan dengan menggunakan Ekstraksi Ciri Rantai Markov Orde 1 dan klasifikasi Probabilistic Neural Network (PNN) adalah fold 3.
16
Perhitungan Rata-rata Nilai Akurasi Orde 2
Hasil perhitungan rata-rata nilai akurasi dengan menggunakan Ekstaksi Ciri Rantai Markov Orde 2 fold 1 menunjukkan akurasi setiap kelas dengan persentase seperti dilihat pada Tabel 8.
Tabel 8 Pengujian Orde 2 fold 1
1-cysPrx_C 4HBT ABC_Tran Akurasi (%)
1-cysPrx_C 24 0 1 96.00 4HBT 0 22 3 88.00 ABC_Tran 0 0 25 100.00 Rata-rata 94.67 0.000 0.100 0.200 0.300 0.400 0.500 0.600 0.700 0.800 0.900 1.000
sens spec sens spec sens spec sens spec
Fold 1 Fold 2 Fold 3 Fold 4
0.920 0.960 0.813 0.906 0.987 0.993 0.813 0.906 0 20 40 60 80 100 1 2 3 4 92 81.33 98.67 81.33 d al am % Fold
Gambar 6 Grafik Nilai Rata-rata sensitivity dan specificity pada Orde 1
17 Hasil akurasi pada Orde 2 fold 1 diperoleh rata-rata sebesar 94.67%. Kelas 1-cysPrx_C dapat terklasifikasi dengan benar sebanyak dua puluh empat, satu sisanya terklasifikasi ke dalam kelas ABC_Tran. Kelas 4HBT dapat terklasifikasi dengan benar sebanyak dua puluh dua, tiga sisanya terklasifikasi ke dalam kelas ABC_Tran. Sedangkan Kelas ABC_Tran dapat terklasifikasi semua dengan benar.
Hasil perhitungan rata-rata nilai akurasi dengan menggunakan Ekstaksi Ciri Rantai Markov Orde 2 fold 2 menunjukkan akurasi setiap kelas dengan persentase seperti dilihat pada Tabel 9.
Tabel 9 Pengujian Orde 2 fold 2
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 11 0 14 44.00
4HBT 0 24 1 96.00
ABC_Tran 0 0 25 100.00
Rata-rata 80.00
Hasil akurasi pada Orde 2 fold 2 diperoleh rata-rata sebesar 80%. Kelas 1-cysPrx_C dapat terklasifikasi dengan benar sebanyak sebelas, empat belas sisanya terklasifikasi ke dalam kelas ABC_Tran. Kelas 4HBT dapat terklasifikasi dengan benar sebanyak dua puluh empat, satu sisanya terklasifikasi ke dalam kelas ABC_Tran. Sedangkan Kelas ABC_Tran dapat terklasifikasi semua dengan benar.
Hasil perhitungan rata-rata nilai akurasi dengan menggunakan Ekstaksi Ciri Rantai Markov Orde 2 fold 3 menunjukkan akurasi setiap kelas dengan persentase seperti dilihat pada Tabel 10.
Tabel 10 Pengujian Orde 2 fold 3
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 25 0 0 100.00
4HBT 0 24 1 96.00
ABC_Tran 0 0 25 100.00
Rata-rata 98.67
Hasil akurasi pada Orde 2 fold 3 diperoleh rata-rata sebesar 98.67%. Kelas 4HBT dapat terklasifikasi dengan benar dua puluh empat, satu sisanya terklasifikasi ke dalam kelas ABC_Tran. Sedangkan kelas 1-cysPrx_C dan ABC_Tran dapat terklasifikasi semua dengan benar.
Hasil perhitungan rata-rata nilai akurasi dengan menggunakan Ekstaksi Ciri Rantai Markov Orde 2 fold 4 menunjukkan akurasi setiap kelas dengan persentase seperti dilihat pada Tabel 11.
18
Tabel 11 Pengujian Orde 2 fold 4
1-cysPrx_C 4HBT ABC_tran Akurasi (%)
1-cysPrx_C 25 0 0 100.00
4HBT 0 12 13 48.00
ABC_Tran 0 0 25 100.00
Rata-rata 82.67
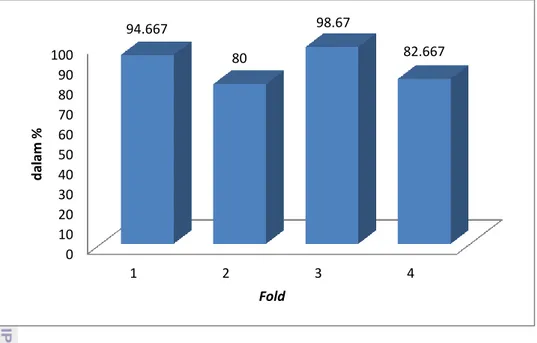
Hasil akurasi pada Orde 2 fold 4 diperoleh rata-rata sebesar 82.67%. Kelas 4HBT dapat terklasifikasi dengan benar dua belas, tiga belas sisanya terklasifikasi ke dalam kelas ABC_Tran. Sedangkan kelas 1-cysPrx_C dan ABC_Tran dapat terklasifikasi semua dengan benar.
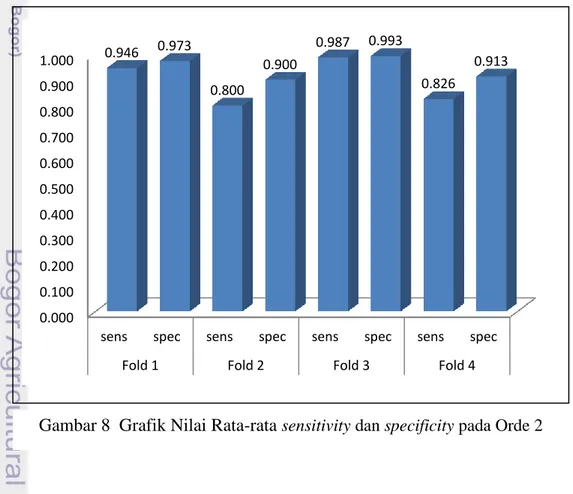
Setelah dilakukan Hasil perhitungan rata-rata nilai akurasi menggunakan Ekstaksi Ciri Rantai Markov Orde 2 dan klasifikasi menggunakan Probabilistic Neural Network (PNN), selanjutnya menghitung rata-rata nilai sensitivity dan specificity. Nilai sensitivity dan specificity yang dihasilkan oleh setiap fold pada Orde 2 dapat dilihat pada Gambar 8.
Pada Gambar 8 menunjukan bahwa nilai rata-rata sensitivity tertinggi pada Orde 2 terletak pada fold 3 dengan nilai 0.987 dan nilai rata-rata specificity tertinggi yang pada Orde 2 terletak pada fold 3 dengan nilai 0.993.
Berasarkan grafik pada Gambar 8 dan Gambar 9, dapat dilihat bahwa model terbaik yang dihasilkan dengan menggunakan Ekstraksi Ciri Rantai Markov Orde 2 dan klasifikasi Probabilistic Neural Network (PNN) adalah fold 3.
0.000 0.100 0.200 0.300 0.400 0.500 0.600 0.700 0.800 0.900 1.000
sens spec sens spec sens spec sens spec
Fold 1 Fold 2 Fold 3 Fold 4
0.946 0.973 0.800 0.900 0.987 0.993 0.826 0.913
19
Gambar 9 Grafik Perhitungan Nilai Rata-rata Akurasi pada Orde 2
SIMPULAN DAN SARAN
Simpulan
Berdasarkan penelitian yang telah dilakukan, dapat ditarik kesimpulan sebagai berikut.
Model dengan menggunakan Ekstraksi Ciri Rantai Markov Orde 1 dan 2 serta klasifikasi Probabilistic Neural Network (PNN) telah berhasil diimplementasikan.
Berdasarkan hasil penelitian, dapat diketahui bahwa model terbaik yang dihasilkan dengan menggunakan Ekstraksi Ciri Rantai Markov Orde 1 dan 2 serta klasifikasi Probabilistic Neural Network (PNN) adalah fold ketiga dimana nilai rata-rata akurasi
terbesar dan nilai rata-rata sensitivity dan specificity terbesar dari masing-masing Orde sama.
Saran
Saran yang dapat diberikan untuk mengembangkan penelitian ini agar lebih baik menjadi lebih baik lagi adalah:
Melibatkan kelas dan data yang lebih banyak lagi.
Menggunakan ektraksi ciri dan classifier yang berbeda.
0 10 20 30 40 50 60 70 80 90 100 1 2 3 4 94.667 80 98.67 82.667 d al am % Fold
20
DAFTAR PUSTAKA
Almatsier, S. 1989. Prinsip Dasar Ilmu Gizi. Jakarta : Penerbit Gramedia.
Cambell, Reece JB, Urry LA, Cain ML, Wasserman SA, Minorsky PV, Jackson RB. 2011.
Biology. San Francisco (US):Pearson Education.
Ching WK, Ng MK. 2006. Markov Chains Models, Algorithms and Applications. New York (US): Springer.
Fajar ML. 2013. Identifikasi DNA Bakteri Menggunakan Metode Ekstraksi Ciri Rantai Markov dengan Probabilistic Neural Network Sebagai Classifier [skripsi].Bogor (ID): Institut Pertanian Bogor.
Hastie T, Tibshirani R, Friedman J. 2011. The Elements of Statistical Learning: Data
Mining, Inference, and Prediction. New York (US): Springer.
Kunin V, Cases I, Enright A.J, De Lorenzo V, Ouzounis C.A. 2003. Myriads of Protein
Families, and Still Counting. Cambridge (GB): The European Bioinformatics
Institute.
Lehninger A L, Nelson D L, and Cox M M (2004). Lehninger Principles of Biochemistry, W. H. Freeman, New York.
Muhammad S. 2013. Klasifikasi Protein Family Menggunakan Metode Rantai Markov [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Polanski A, Kimmel M. 2007. Bioinformatics. Berlin (DE): Springer Science.
Specht, DF. 1990. Probabilistic Neural Netwok (PNN). NEURAL NETWORKS. 3: 109-118
Suhardjo dan Clara M.K. 1992. Prinsip-prinsip Ilmu Gizi. Yogyakarta: Kanisius.
Wu CH, Hongzhan H, Yeh LL, Barker WC. 2002. Protein Family Classificationand
Functional Annotation. Washington DC (US): Georgetown University Medical
21
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta, pada tanggal 29 Oktober 1988. Penulis merupakan anak kedua dari pasangan Mudjijono dan Suminah. Penulis lulus dari SMA Negeri 4 Depok pada tahun 2006. Pada tahun yang sama penulis diterima di Direktorat Diploma Institut Pertanian Bogor jurusan Teknik Komputer dan kemudian melanjutkan studi di ekstensi Departemen Ilmu Komputer IPB jurusan Ilmu Komputer.