PENGGUNAAN GEN
SITOKROM
β (Cyt β) DALAM IDENTIFIKASI
SPESIES ANJING, KUCING, DAN HARIMAU UNTUK MENJAMIN
KEASLIAN PRODUK PANGAN DAN OBAT
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2013
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Penggunaan Gen Sitokrom
β (Cyt β) dalam Identifikasi Spesies Anjing, Kucing, dan Harimau untuk Menjamin Keaslian Produk Pangan dan Obat adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dan karya tulis saya kepada Institut Pertanian Bogor.
Bogor, September 2013
Irine
ii
RINGKASAN
IRINE. Penggunaan Gen Sitokrom β (Cyt β) dalam Identifikasi Spesies Anjing, Kucing, dan Harimau untuk Menjamin Keaslian Produk Pangan dan Obat. Dibimbing oleh HENNY NURAINI dan CECE SUMANTRI.
Sampai saat ini, upaya pemalsuan produk pangan asal ternak dengan alasan ekonomi masih sering terjadi. Pengembangan metode identifikasi spesies pada produk pangan dan obat yang berasal dari hewan diharapkan dapat melindungi konsumen dari pemalsuan informasi. Kemajuan dibidang genetika molekuler memungkinkan proses pendeteksian dilakukan pada tingkat DNA sehingga hasil pengujian lebih akurat, walaupun telah mengalami proses pengolahan.
Penelitian tentang beberapa jenis daging telah dilakukan oleh beberapa peneliti dengan menggunakan DNA mitokondria (mtDNA). Penggunaan mtDNA didasarkan pada alasan jumlah kopi molekul mtDNA yang tinggi dalam sel dibandingkan dengan DNA inti, misalnya pada kasus dimana jumlah ekstraksi DNA sangat sedikit atau terdegradasi. Gen yang sering digunakan sebagai penanda spesies adalah gen sitokrom β (cyt β) karena mempunyai runutan sekuen basa nukleotida yang bervariasi. Penelitian ini bertujuan untuk pembuktian spesies anjing, kucing, dan harimau dalam menjamin keaslian produk asal ternak menggunakan marka spesifik berbasis gen sitokrom β (cyt β).
Jumlah sampel DNA yang digunakan untuk masing-masing hewan sebanyak tiga sampel, yang terdiri dari darah kucing, daging anjing, dan feses harimau, serta sebagai pembanding digunakan sampel darah dari berbagai jenis hewan antara lain kambing, ayam, sapi, domba, kuda, tikus serta sampel daging dari babi. Primer spesifik anjing, kucing, dan harimau dirancang menggunakan
software MEGA 5. Ekstraksi DNA dilakukan dengan metode fenol-kloroform, dan amplifikasi fragmen DNA menggunakan metode multipleks PCR. Multipleks PCR merupakan salah satu variasi dari PCR yang menggunakan banyak primer secara bersamaan untuk mengamplifikasi beberapa daerah target. Penelitian ini menunjukkan amplifikasi panjang fragmen yang berbeda pada spesies yang diuji (anjing, kucing, dan harimau) adalah 523, 568, dan 319 pb; dan pada spesies pembanding (kambing, ayam, sapi, domba, babi, kuda, dan tikus) adalah 157, 227, 274, 331, 398, 439, dan 603 pb. Kespesifikan spesies ditunjukkan dengan adanya persentase homologi primer reverse yang tinggi pada masing-masing spesies. Amplifikasi fragmen spesifik pada DNA total genom yang mempunyai panjang berdekatan harus dipisahkan untuk mencegah pita yang tumpang tindih diantara spesies. Hierarki taksonomi anjing, kucing, dan harimau sudah dapat dibedakan sampai pada tingkat spesies dengan menggunakan gensitokrom β (cyt β).
SUMMARY
IRINE. Using Cytochrome β(Cyt β) Gene for Species Identification of Dog, Cat, and Tiger to Ensure Food Authenticity and Drugs. Supervised by HENNY NURAINI and CECE SUMANTRI.
Adulteration food products from animal still happen until now. Species identification method development in food and medicine derived from animal was needed to prevent falsification information. Molecular genetic improvement allows the detection is carried out at the level of DNA, so the results more accurate, although processed.
Research on several meat have been conducted by several researchers using DNA mitochondrial DNA (mtDNA). Using mtDNA because it has high copy number in a cell, compared with nuclear DNA. In this case where the amount of extracted DNA is very small or degraded. Gene used as marker specific species is
cytochrome β (cyt β) because it has varied sequences. The objective of this research was to study species authentication (i.e. dog, cat, and tiger) to ensure animal origin in product using cytβ gene specific marker.
Total DNA samples for each species which identified are three samples were consisting blood (cat), cooked meat (dog), feces (tiger), and as a comparison used blood sample from several animal (i.e. goat, chicken, cattle, sheep, horse, rat) and pork. Reverse primer specific for dog, cat, and tiger was designed using MEGA 5 software. DNA extraction and fragment amplification was conducted using phenol-chloroform and multiplex PCR method, respectively. Multiplex PCR is a variant of PCR which many primers were used together for amplification of multiple region. This research showed that different length of amplification for the species tested (dog, cat, and tiger) were 523, 331, 319 bp, respectively; and for the comparison species (goat, chicken, cattle, sheep, pig, horse, and rat) were 157, 227, 274, 331, 398, 439, and 603 bp, respectively. Species specificity also indicated by high reverse primer homology percentage. Specific fragment amplification on genome pool with closed fragment length should be separate to avoid band overlapped between species. Taxonomy hierarchy (i.e. dog, cat, and tiger) could be distinguished to the species level.
iv
© Hak Cipta Milik IPB, Tahun 2013
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
PENGGUNAAN GEN
SITOKROM
β (Cyt β) DALAM IDENTIFIKASI
SPESIES ANJING, KUCING, DAN HARIMAU UNTUK MENJAMIN
KEASLIAN PRODUK PANGAN DAN OBAT
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2013
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Ilmu Produksi dan Teknologi Peternakan
Judul Tesis : Penggunaan Gen Sitokrom β (Cyt β) dalam Identifikasi Spesies Anjing, Kucing, dan Harimau untuk Menjamin Keaslian Produk Pangan dan Obat
Nama : Irine
NIM : D151110151
Disetujui oleh
Komisi Pembimbing
Diketahui oleh
Dr Ir Henny Nuraini, MSi Prof Dr Ir Cece Sumantri, MAgrSc
Ketua Anggota
Ketua Program Studi Dekan Sekolah Pascasarjana Ilmu Produksi dan Teknologi Peternakan
Prof Dr Ir Muladno, MSA Dr Ir Dahrul Syah, MscAgr
2
PRAKATA
Puji dan syukur penulis panjatkan kepada Tuhan Yang Maha Esa atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Oktober 2012 ini ialah pengembangan metode identifikasi spesies untuk melindungi konsumen dari pemalsuan informasi dengan judul: “Penggunaan Gen Sitokrom β (Cyt β) dalam Identifikasi Spesies Anjing, Kucing, dan Harimau untuk Menjamin Keaslian Produk Pangan dan Obat”.
Terima kasih penulis ucapkan kepada Dr Ir Henny Nuraini, Msi dan Prof Dr Ir Cece Sumantri, MAgrSc selaku pembimbing, serta Dr Jakaria, SPt, MSi yang telah banyak memberi saran. Di samping itu, penulis juga menyampaikan ucapan terima kasih kepada Prof Dr Ir Muladno, MSA selaku Kepala Laboratorium Genetika Molekuler Ternak, Fakultas Peternakan, Institut Pertanian Bogor dan kepada saudara Eryk Andreas, SPt, MSi atas bantuannya selama penelitian di Laboratorium Molekuler Ternak. Penghargaan juga disampaikan penulis kepada Prof Dr drh Dondin Sajuthi, MST dan Dr drh Ligaya I.T.A. Tumbelaka, SpMp, MSc atas perolehan sampel harimau. Ungkapan terima kasih juga disampaikan kepada orang tua, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, September 2013
3
DAFTAR ISI
DAFTAR TABEL ix
DAFTAR GAMBAR ix
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan 2
Manfaat 2
Hipotesis 2
2 TINJAUAN PUSTAKA 2
Deteksi Cemaran Pangan 2
Anjing (Canis lupus familiaris) dan Kucing (Felis catus) 3
Harimau (Panthera tigris) 4
DNA Mitokondria 5
Gen Sitokrom β (Cyt β ) 6
Polymerase Chain Reaction (PCR) 6
3 METODE 7
Waktu dan Tempat 7
Materi 8
Prosedur 9
Analisis Data 12
4 HASIL DAN PEMBAHASAN 13
Perancangan Primer Spesifik Anjing, Kucing, dan Harimau 13
Derajat Kesamaan Primer Spesifik 13
Kualitas DNA Total 14
Amplifikasi DNA Fragmen Spesifik Gen Cyt β pada Anjing, Kucing,
dan Harimau 19
Amplifikasi DNA Fragmen Spesifik Gen Cyt β pada Sepuluh Spesies
Hewan 20
Amplifikasi DNA Fragmen Spesifik Gen Cyt β pada DNA Total Genom 21 Amplifikasi DNA Fragmen Spesifik Gen Cyt β pada Obat Asal Hewan 22
5 SIMPULAN DAN SARAN 23
Simpulan 23
Saran 23
DAFTAR PUSTAKA 23
4
DAFTAR TABEL
1 Sampel DNA 8
2 Sekuen primer reverse gen cyt β 9
3 Sekuen primer reverse gen cyt βspesies anjing, kucing, dan harimau 13
4 Kriteria primer reverse anjing, kucing, dan harimau 13
5 Hasil uji homologi primer spesifik pada sepuluh jenis hewan 17
6 Hasil pengukuran konsentrasi DNA sepuluh spesies 18
7 Hasil pengukuran konsentrasi DNA obat 22
DAFTAR GAMBAR
1 Susunan genom mitokondria 5
2 Elektroforesis gel agarosa dari produk PCR enam daging 12
3 Fragmen DNA spesifik untuk setiap tipe hewan 12
4 Situs penempelan primer pada sekuen DNA mitokondria daerah
cyt βsepuluh jenis hewan 15
5 Fragmen DNA spesifik spesies anjing, kucing, dan harimau 19
6 Fragmen DNA spesifik untuk setiap jenis hewan 20
7 Amplifikasi fragmen spesifik dari DNA total 21
1
1 PENDAHULUAN
Latar Belakang
Indonesia mempunyai keragaman agama, budaya dan tradisi atau suatu kebiasaan yang bersifat turun temurun. Hal ini juga tercermin dari bervariasinya produk olahan daging maupun sumber bahan baku dagingnya. Sumber daging yang biasa digunakan untuk konsumsi berasal dari ternak: sapi, kerbau, kambing, domba, babi, kuda, dan unggas. Beberapa daerah di Indonesia yang mayoritas warganya mempunyai tradisi mengkonsumsi daging dari hewan yang tidak lazim (anjing) yaitu: Manado dan Minahasa yang mengenal masakan daging anjing dengan sebutan RW (rintek wuuk), budaya Batak dari Sumatera Utara
mengenalnya dengan kode “B1”, dan beberapa kota di Jawa, seperti Solo dan
Yogyakarta, sate dan tongseng yang memakai daging anjing disamarkan dengan
sebutan “jamu” dan “sengsu”. Daerah yang mengkonsumsi daging kucing sebagai
makanan khasnya adalah Tomohon yang terletak di provinsi Sulawesi Utara. Bagi agama tertentu, sumber daging tersebut dapat menjadi suatu larangan, namun tidak untuk agama atau budaya yang lain.
Penggunaan hewan yang tidak lazim, misalnya anjing dan kucing sebagai campuran bahan pangan perlu dideteksi untuk menjamin keaslian produk dan melindungi konsumen dari pemalsuan. Selain itu, penggunaan hewan yang dilindungi seperti harimau, sebagai bahan baku produk obat memerlukan pengawasan agar tidak terjadi kepunahan dari hewan asli Indonesia, karena digunakan sebagai bahan baku produk secara ilegal. Pemerintah sudah berusaha melindungi konsumen dengan adanya Undang-undang (UU No. 8 Tahun 1999 tentang Perlindungan Konsumen) dan Peraturan Pemerintah (Peraturan Pemerintah Nomor 28 Tahun 2004 tentang Keamanan, Mutu, dan Gizi Pangan). Namun, sampai saat ini, upaya pemalsuan produk pangan asal ternak dengan alasan ekonomi masih sering terjadi, seperti pemalsuan dan penggantian dengan spesies yang tidak diharapkan yang mempunyai nilai lebih rendah (Che Man et al.
2007; Rastogi et al. 2007). Oleh karena itu, untuk melindungi hak konsumen tersebut, maka diperlukan adanya label dari pangan tersebut yang menjelaskan spesies yang digunakan (Ballin 2010).
Kemajuan dibidang genetika molekuler memungkinkan proses deteksi dilakukan pada tingkat DNA sehingga hasil pengujian lebih akurat, walaupun telah mengalami proses pengolahan. Proses perbanyakan sekuen DNA melalui teknik multipleks Polymerase Chain Reaction (PCR) merupakan suatu teknik yang handal dalam menentukan adanya pemalsuan spesies dalam suatu produk. Matsunaga et al. (1999) menggunakan metode multipleks PCR untuk mengidentifikasi enam jenis daging (sapi, babi, ayam, domba, kambing, dan kuda) yang telah mengalami pemasakan. Variasi runutan basa nukleotida yang ada pada
2
Perumusan Masalah
Sampai saat ini, upaya pemalsuan produk pangan asal ternak dengan alasan ekonomi masih sering terjadi. Penggunaan hewan yang tidak lazim, misalnya anjing dan kucing sebagai campuran bahan pangan perlu dideteksi untuk menjamin keaslian produk. Selain itu, penggunaan hewan yang dilindungi seperti harimau, sebagai bahan baku produk obat memerlukan pengawasan, karena digunakan sebagai bahan baku produk secara ilegal. Kemajuan dibidang genetika molekuler memungkinkan proses pendeteksian dilakukan pada tingkat DNA sehingga hasil pengujian lebih akurat, walaupun telah mengalami proses pengolahan. Penelitian ini difokuskan pada teknik multipleks PCR dan penggunaan gen sitokrom β (cyt β) dalam mendeteksi dan mengidentifikasi spesies hewan secara cepat.
Tujuan
Penelitian ini bertujuan untuk pembuktian spesies anjing, kucing, dan harimau dalam menjamin keaslian produk asal ternak menggunakan marka spesifik berbasis gen sitokrom β (cyt β).
Manfaat
Pemanfaatan dan pengembangan penanda spesifik tersebut diharapkan dapat membantu dalam menyediakan teknologi yang aplikatif untuk mencegah terjadinya pemalsuan informasi khususnya produk pangan asal hewan, serta melindungi hewan asli Indonesia dari kepunahan.
Hipotesis
Penanda genetik seperti gen sitokrom β (cyt β) pada kambing (Capra hircus), ayam (Gallus gallus), sapi (Bos taurus), domba (Ovis aries), babi (Sus scrofa), kuda (Equus caballus), tikus (Rattus norvegicus), anjing (Canis lupus familiaris), kucing (Felis catus), dan harimau (Panthera tigris) memiliki fragmen unik yang mencirikan masing-masing jenis hewan tersebut.
2 TINJAUAN PUSTAKA
Deteksi Cemaran Pangan
3
spesies daging serta produk turunan hewan. Sejumlah metode analisis dikembangkan untuk identifikasi spesies yang berasal dari protein daging dan DNA. Metode yang didasarkan pada pemisahan fraksi protein termasuk elektroforesis, kromatografi, teknik immunologi (ELISA = enzyme-linked immunosorbent assay, CIE = counterimmunoelectrophoresis, RID = radial immunodiffusio) (Kesmen et al. 2007). Metode ini kurang sensitif untuk mengidentifikasi produk daging yang sudah mengalami pemasakan dengan suhu yang tinggi karena mungkin mengubah struktur dan kestabilan protein (Saez et al.
2004) serta merusak protein spesifik (Calvo et al. 2001). Metode analisis DNA merupakan salah satu strategi untuk identifikasi spesies daging, karena jika dibandingkan dengan protein, DNA lebih stabil terhadap perlakuan teknologi dan dianggap sebagai jaringan yang independen (Martinez dan Yman 1998). Metode analisis DNA yang didasarkan pada polymerase chain reaction (PCR) sangat potensial untuk mendeteksi spesies hewan yang digunakan, meskipun produk yang digunakan telah mengalami proses pemasakan (Kesmen et al. 2007).
Beberapa contoh pemalsuan yang terjadi di masyarakat diantaranya adalah 1) penggunaan lemak babi pada roti (Che Man et al. 2007) dan penggunaan usus babi sebagai pembungkus sosis (Nakyinsige et al. 2012), 2) penggunaan daging babi untuk mengganti daging sapi, ayam dan daging kambing, karena harganya yang lebih murah (Sahilah et al. 2011), 3) pemalsuan produk dendeng yak dengan dendeng sapi (Chen et al. 2010), 4) pemalsuan bakso sapi dengan bakso babi (Ali
et al. 2012). Selain itu, kemungkinan cemaran yang potensial terjadi di masyarakat adalah penggunaan hewan yang tidak lazim seperti anjing dan kucing (Abdel-Rahman et al. 2009). Penggunaan hewan yang dilindungi seperti harimau, sebagai bahan baku produk obat memerlukan pengawasan agar tidak terjadi kepunahan dari hewan asli Indonesia, karena digunakan sebagai bahan baku produk secara ilegal.
Anjing (Canis lupus familiaris) dan Kucing (Felis catus)
Belakangan ini, konsumen dikhawatirkan dengan berbagai isu, seperti pemalsuan daging. Keterangan komponen yang digunakan dalam proses atau campuran pangan tidak selalu tersedia, sehingga memungkinkan adanya pemalsuan dan penggantian produk dengan spesies yang tidak diharapkan dan bernilai lebih rendah. Identifikasi bahan atau komposisi campuran tidak selalu jelas dan diperlukan verifikasi bahwa komponen yang digunakan adalah asli dan berasal dari sumber yang dapat diterima konsumen. Oleh karena itu, untuk melindungi hak konsumen, peraturan dari setiap negara harus menggunakan label yang menjelaskan spesies yang digunakan dalam pengolahan (Abdel-Rahman et al. 2009).
Selain masalah pemalsuan daging, bukti forensik manusia yang dikumpulkan dari tempat kejadian perkara (TKP) sering tercampur dengan materi biologis anjing dan kucing. Amerika Serikat (US), 55% rumah tangga setidaknya memiliki satu anjing (68 juta anjing di US) dan/atau kucing (73 juta kucing di US). Pertukaran bukti biologis (rambut, feses, dan urin) hewan dengan tersangka kejahatan dan korban pada tempat kejadian sangat mungkin terjadi (Kanthaswamy
4
mitokondria cukup untuk menjelaskan bahwa anjing tidak berkontribusi sebagai bukti forensik (Angleby dan Savolainen 2005).
Harimau (Panthera tigris)
Harimau tersebar luas secara geografis, mendiami sebagian besar Asia, termasuk wilayah di antara Laut Caspian dan Laut Aral, Rusia Tenggara, dan Pulau Sunda (Hemmer 1987; Herrington 1987), dan diperkirakan besarnya populasi 100.000 pada tahun 1900 (Kitchener dan Dugmore 2000). Kehilangan habitat dan perburuan menyebabkan populasi harimau hanya sebagian dari ukuran sebelumnya, yang diperkirakan baru-baru ini hanya sekitar 3800-5000 harimau liar dengan titik tengah sekitar 4500 (Seidensticker et al. 2010). Sembilan subspesies dari harimau dikenali : Panthera tigris altica, P. t. amoyensis, P. t. tigris, P. t. corbetti, P. t. sumatrae, P. t. virgata, P. t. sondaica, P. t. balica dan P. t. “jacksoni”. Tiga subspesies diduga punah adalah P. t. virgata, P. t. sondaica,
dan P. t. balica, sementara P. t. amoyensis tidak ada lagi di alam liar dan hanya terdapat di penangkaran (Xu et al. 2007). Tersisa 5 subspesies yang terancam punah (Seidensticker et al. 2010), sehingga harus diketahui berapa banyak subspesies harimau yang dapat dikenali dengan pendekatan morfologi, biokimia, dan genetik molekuler (Mazak 2010; Luo et al. 2004).
Perburuan yang ekstrim dan perdagangan kulit harimau dan bagian tubuh untuk tujuan komersial merupakan faktor penting yang menyebabkan penurunan jumlah dari harimau liar. Hal ini terkait dengan peningkatan ketertarikan akan pengobatan tradisional serta didukung dengan perkembangan yang pesat dari ekonomi Asia sehingga meningkatkan permintaan secara pesat akhir-akhir ini (Wetton et al. 2004). Setiap bagian tubuh harimau dipercaya mempunyai efek terapi, seperti: kumis untuk menghilangkan sakit gigi, bola mata untuk menolong mengontrol epilepsi dan sup penis harimau membantu kejantanan. Tulang merupakan bagian yang sangat berharga untuk melawan arthritis dan untuk meningkatkan potensi pria (Mills dan Jackson 1994). Tulang harimau digunakan dalam obat tradisional Asia atau Traditional Chinese Medicine (TCM), dan biasanya dicampur dengan bahan yang berasal dari hewan lain atau bahan herbal untuk komponen resep (Wetton et al. 2004). Kulit harimau sangat dicari sebagai dekorasi rumah atau kostum pada beberapa negara, terutama Tibet (Wright 2010). Semua penggunaan dari bagian tubuh harimau menghasilkan keuntungan pasar yang tinggi di banyak negara (Kitpipit et al. 2012).
5
DNA Mitokondria
Mitokondria merupakan organel sel penghasil energi yang terdapat dalam sitoplasma. DNA mitokondria (mtDNA) adalah untai ganda yang mempunyai panjang 16.569 pb (pasang basa). Proporsi dari 4 basa nukleotida tidak seimbang, untai yang satu mengandung banyak basa guanine (G) dan adenosine (A) (heavy strand atau H-strand) dan yang lainnya kaya akan cytosine (C) dan thymine (T) yang disebut dengan light strand (L-strand) (Tully et al. 2001). Susunan genom mitokondria disajikan pada Gambar 1.
Gambar 1 Susunan genom mitokondria (Taylor dan Turnbull 2005)
Genom mitokondria hewan terdiri atas 13 protein yang disandikan oleh gen-gen, 2 gen penyandi untuk ribosomal RNA, 22 gen transfer RNA (tRNA) dan sebuah daerah non penyandi (Meyer 1994). Daerah non penyandi yang panjangnya sekitar 1100 pb sebagai daerah pengontrol yang disebut dengan
displacement loop (D-Loop). mtDNA hanya diwariskan dari ibu (Butler 2005). Sebuah mitokondria mengandung 2-10 kopi mtDNA dan terdapat sebanyak 1000 mitokondria per sel somatik (Budowle et al. 2003). Sementara untuk inti sel mengandung DNA inti sepanjang 3.2 x 109 pb dan terdapat 2 inti per sel (Butler 2005).
6
Gen Sitokrom β (Cyt β)
Gen cyt β menyandi pembentukan protein, terutama protein yang terlibat dalam transportasi elektron dalam mitokondria. Sekuen DNA mitokondria (mtDNA) mengandung gen cyt β (Irwin et al. 1991) yang digunakan untuk identifikasi spesies dan taksonomi filogenetik (Hsieh etal. 2001). Gen cyt β sering digunakan untuk membandingkan beberapa filogenetik spesies pada genus atau famili yang sama, keragaman gen cyt β digunakan untuk mendeteksi sumber susu yang berasal dari sapi (Bos), domba (Ovis), kambing (Capra), dan kerbau (Bubalus) (Lanzilao et al. 2005).
Sekuen gen cyt βyang berasal dari kucing (Felis catus) mempunyai panjang sekuen 1140 pb (Tamada et al. 2005), runutan gen cyt β anjing (Canis lupus familiaris) sepanjang 1140 pb (Kim et al. 1998), harimau (Panthera tigris) sepanjang 1140 pb (Cracraft et al. 1998), tikus (Rattus norvegicus) sepanjang 1143 pb (Naidu et al. 2010), kambing (Capra hircus)sepanjang 1140 pb (Liu et al. 2009), ayam (Gallus gallus) sepanjang 1143 pb (Shen dan Nakamura 2000), sapi (Bos taurus)sepanjang 1140 pb (Geng dan Chang 2008), domba (Ovis aries) sepanjang 1140 pb (Rezaei et al. 2009), babi (Sus scrofa) sepanjang 1140 pb (Han
et al. 2004b), dan kuda (Equus caballus) sepanjang 1139 pb (Han et al. 2004a). Matsunaga et al. (1999) mengembangkan metode multipleks PCR untuk mengidentifikasi enam jenis daging yaitu sapi, babi, ayam, domba, kambing dan kuda dengan menggunakan primer forward yang dirancang pada sekuen DNA gen cyt β “conserve”, sementara primer reverse pada sekuen DNA spesifik setiap spesies. Fragmen DNA spesifik diamplifikasi dari daging yang dimasak pada suhu 100-120 oC selama 30 menit dengan panjang fragmen DNA berturut-turut untuk kambing, ayam, sapi, domba, babi, kuda adalah 157, 227, 274, 331, 398, dan 439 pb. Sementara panjang fragmen DNA tikus hasil amplifikasi adalah 603 pb (Nuraini et al. 2012).
Polymerase Chain Reaction (PCR)
PCR adalah suatu teknik in vitro untuk menggandakan molekul DNA pada target tertentu dengan cara mensintesa molekul DNA baru yang berkomplemen dengan molekul DNA tersebut dengan menggunakan enzim polimerase dan oligonukleotida pendek sebagai primer. Metode ini berjalan secara enzimatik melalui mekanisme perubahan suhu (Buzdin dan Lukyanov 2007). Target PCR yang berupa DNA untai ganda, diekstraksi dari sel dan didenaturasi menjadi untai tunggal. Primer oligonukleotida spesifik untuk gen target tertentu, bersama dengan enzim (biasanya Taq polymerase, sebuah enzim yang thermostable dan
thermoactive yang berasal dari Thermus aquaticus) serta deoxynucleotide triphosphates (dNTPs), digunakan untuk mengamplifikasi gen target, menghasilkan untai ganda. Proses ini berlangsung secara otomatis, pada mesin
thermocycler, yang menyediakan kondisi thermal yang dibutuhkan untuk proses amplifikasi (Nollet dan Toldra 2011).
7
(annealing), ekstensi awal molekul DNA, dan tahap terakhir adalah ekstensi akhir. Setelah amplifikasi, produk PCR dielektroforesis menggunakan pewarna Ethidium Bromida (EtBr), dan divisualisasikan menggunakan sinar ultraviolet (Nollet dan Toldra 2011), dan dapat digunakan untuk analisis lebih lanjut (Weissensteiner et al. 2004). PCR seperti ini dinamakan PCR konvensional (Nollet dan Toldra 2011).
Multipleks PCR
Multipleks PCR merupakan salah satu variasi PCR yang menggunakan banyak primer secara bersama-sama untuk mengamplifikasi beberapa daerah target (Jain et al. 2007) dalam reaksi yang sama (Markoulatos et al. 2002). Multipleks PCR sangat potensial dalam menghemat waktu. Metode ini telah sukses mengamplifikasi banyak area dari DNA yang diujikan, termasuk analisis delesi gen (Chamberlain et al. 1988), mutasi dan analisis polimorfisme (Rithidech
et al. 1997), analisis kuantitatif (Zimmermann et al. 1996), dan transkripsi reverse
PCR (Crisan 1994). Optimasi komponen reaksi multipleks sangat tergantung pada konsentrasi primer, konsentrasi dNTPs dan MgCl2, konsentrasi buffer PCR, taq
DNA polymerase serta penggunaan adjuvant (DMSO, gliserol dan BSA) (Markoulatos et al. 2002). Meskipun dengan teknik unipleks (simpleks) PCR konvensional diperoleh hasil yang sama, pendekatan multipleks PCR memungkinkan untuk deteksi simultan yang cepat, praktis dan sederhana karena dilakukan sekaligus dalam satu reaksi (Kingombe et al. 2010). Ketepatan hasil dari produk PCR akan meningkat atau menurun sangat tergantung pada suhu
annealing (Henegariu et al. 1997).
Teknik yang handal untuk mengidentifikasi spesies asli dari komponen produk pangan yang berasal dari hewan diperlukan untuk tujuan pembuktian pangan. Multipleks PCR telah berhasil digunakan sebagai metode yang rutin, dengan sensitivitas yang tinggi, cepat, sederhana dan tidak mahal untuk membedakan jenis hewan (Jain et al. 2007).
3 METODE
Waktu dan Tempat
8
Materi
Sampel DNA
Sampel DNA yang digunakan dalam penelitian ini berasal dari isolasi darah kucing, feses harimau (koleksi sampel Taman Safari Bogor), dan survei di lapangan untuk daging anjing (gulai, rw (rica-rica), rebus), sebagai pembanding digunakan sampel darah dari berbagai jenis hewan antara lain kambing, ayam, sapi, domba, kuda, tikus serta sampel daging dari babi (Tabel 1). Sampel feses yang digunakan dalam penelitian ini berupa lendir mukosa feses basah. Lendir mukosa ini diharapkan mengandung jaringan epitel. Selanjutnya lendir mukosa disimpan pada larutan 1 x STE (sodium tris EDTA) (400 ml) untuk digunakan pada proses ekstraksi DNA. Selain itu, digunakan juga sampel obat harimau yang ada dipasaran.
Primer
Primer yang digunakan dalam amplifikasi fragmen DNA mengacu pada Matsunaga et al. (1999) dengan primer forward yang sama untuk semua jenis
hewan yaitu 5‟-GAC CTC CCA GCT CCA TCA AAC ATC TCA TCT TGA TGA AA-3‟. Primer reverse untuk mengamplifikasi fragmen spesifik kucing, anjing dan harimau dirancang dengan software MEGA 5. Sekuen primer reverse
pada hewan kambing, ayam, sapi, domba, babi, kuda, dan tikus dapat dilihat pada Tabel 2.
Tabel 1 Sampel DNA
Spesies Isolasi Sampel Jumlah
Anjing Daging 3
Kucing Darah 3
Harimau Sumatera Feses 3
Kambing Darah 3
Ayam Darah 3
Sapi Darah 3
Domba Darah 3
Babi Daging 3
Kuda Darah 3
9
Prosedur
Perancangan Primer Reverse Kucing, Anjing dan Harimau
Perancangan primer spesifik untuk kucing, anjing dan harimau berdasarkan runutan nukleotida gen cyt β pada kucing, anjing, dan harimau yang terdapat dalam GenBank (NCBI). Spesies kucing, anjing, dan harimau dalam hierarki taksonomi yaitu Felis catus (nomor akses GenBank AB194817), Canis lupus familiaris (nomor akses GenBank JF342903), dan Panthera tigris (nomor akses
GenBank EU184702). Sekuen gen tersebut kemudian dihomologikan dengan cyt β Capra hircus (nomor akses GenBank GU295658), Gallus gallus (nomor akses
GenBank GU261719), Bos taurus (nomor akses GenBank EU177824), Bos indicus (nomor akses GenBank JN817330), Ovis aries (nomor akses GenBank
HM236181), Sus scrofa (nomor akses GenBank DQ534707) dan Equus caballus
(nomor akses GenBank JF511459), Rattus norvegicus (nomor akses GenBank
JX105355). Setelah diketahui situs penempelan primer forward dan reverse pada masing-masing jenis hewan tersebut kemudian dilakukan perancangan primer
reverse kucing, anjing, dan harimau dengan melihat runutan basa yang hanya terdapat pada kucing, anjing, dan harimau tetapi tidak terdapat pada jenis hewan lain, sehingga diharapkan primer reverse tersebut dapat menjadi primer spesifik kucing, anjing, dan harimau.
Software yang digunakan untuk perancangan primer spesifik kucing, anjing dan harimau adalah MEGA 5. Tahapan penggunaan program yaitu (1) runutan nukleotida yang diperoleh dari GenBank dan primer dipastikan sudah dalam bentuk FASTA, (2) runutan nukleotida setiap jenis hewan dan primer tersebut dibuka pada software MEGA 5, (3) dilakukan multiple alignment dengan terlebih dahulu mencari situs penempelan primer forward untuk semua jenis hewan dan (4) ditentukan daerah spesifik untuk kucing, anjing, dan harimau yang tidak terdapat pada ketujuh jenis hewan yang digunakan.
Tabel 2 Sekuen primer reverse gen cyt β
Jenis Hewan Reverse (5‟-3‟) Hasil Amplifikasi Kambinga CTC GAC AAA TGT GAG TTA CAG AGG GA 157 pb Ayama AAG ATA CAG ATG AAG AAG AAT GAG GCG 227 pb Sapia CTA GAA AAG TGT AAG ACC CGT AAT ATA AG 274 pb Dombab CTA TGA ATG CTG TGG CTA TTG TCG CAA AT 331 pb Babia GCT GAT AGT AGA TTT GTG ATG ACC GTA 398 pb Kudaa CTC AGA TTC ACT CGA CGA GGG TAG TA 439 pb Tikusc GAA TGG GAT TTT GTC TGC GTT GGA GTT T 603 pb Keterangan: a Matsunaga et al. (1999)
b modifikasi Matsunaga et al. (1999)
c
10
Uji Homologi Primer Spesifik
Uji homologi primer spesifik terhadap sepuluh jenis hewan yang digunakan dalam penelitian ini dilakukan dengan genetic information processing software
GENETYX-WIN versi 4.0. Tahapan penggunaan program ini yaitu: (1) software
dibuka, (2) diklik „new sequence‟ pada „File’, (3) diberi nama primer pada ID
yang tersedia, (4) runutan primer diketik pada kotak, (5) diklik „search homology‟
pada „Nucleotide’, (6) sekuen cyt βyang sudah dalam bentuk FASTA dimasukkan
dengan cara diklik „add’lalu „ok’ dan hasil pengujian akan langsung ditampilkan.
Isolasi dan Ekstraksi DNA
Isolasi dan ekstraksi DNA dilakukan secara konvensional mengikuti metode (Sambrook dan Russel 2001) yang telah dimodifikasi dalam preparasi sampel dan degradasi protein.
Preparasi Sampel. Sampel darah dalam alkohol absolut diambil sebanyak 200 μl dan dipindahkan ke dalam tabung 1.5 ml. Sampel yang disimpan dalam alkohol terlebih dahulu dibersihkan dengan air distilasi sebanyak 1000 µl, kemudian disentrifugasi pada kecepatan 8000 rpm selama lima menit. Alkohol yang terbentuk di bagian atas tabung kemudian dibuang, langkah tersebut dilakukan sebanyak dua kali agar alkohol dalam sampel benar-benar hilang.
Degradasi Protein. Sampel darah yang telah dibersihkan dari alkohol, sampel daging (25 mg), dan sampel obat (25 mg) ditambahkan 1 x STE (sodium tris EDTA) dengan volume 300 µl, sementara sampel mukosa dalam larutan 1 x STE dari feses harimau diambil sebanyak 500 µl, selanjutnya ditambahkan 40 µl SDS (sodium dosesil sulfat) 10% dan 20 µl proteinase K 10 mg/ml. Tabung yang berisi campuran tersebut kemudian dihomogenkan dengan shaker dan diinkubasi pada suhu 55 oC selama 2 jam untuk mengoptimalkan pemecahan protein dalam sampel.
Degradasi Bahan Organik. Sampel yang telah diinkubasi ditambahkan fenol sebanyak 400 µl, CIAA (chlorofom : isoamyl alcohol dengan perbandingan 24 : 1) sebanyak 400 µl dan 40 µl NaCl 5M, kemudian dihomogenkan dengan menggunakan shaker pada suhu ruang selama 1 jam.
11
Pengujian DNA Total
Pengujian DNA hasil ekstraksi secara kualitatif dilakukan dengan elektroforesis pada gel 1%. Gel dibuat dari 0.3 gram agarosa dan 30 ml larutan
buffer (0.5 x TBE) yang dipanaskan pada microwave selama ± 5 menit. Larutan agarosa dibiarkan agak dingin sambil diaduk dengan magnetic stirrer, lalu ditambahkan 1.25 µl pewarna ethidium bromide. Hasil ekstraksi sebanyak 5 μl dicampur dengan 1 μl loading, kemudian dimasukkan pada masing-masing sisir yang terdapat pada gel agarosa 1%. Elektroforesis dilakukan selama 40 menit pada tegangan konstan 100 volt sampai campuran sampel dan loading dye tersebut mencapai garis ketiga pada bagian bawah cetakan gel.
Pengujian DNA hasil ekstraksi secara kuantitatif dilakukan dengan spektrofotometer GeneQuant 1300. Sampel DNA sebanyak 3 μl dimasukkan ke dalam tabung eppendorf 1.5 ml ditambah air distilasi sebanyak 597 μl. Larutan TE (Tris EDTA) 80% digunakan sebagai blanko dengan komposisi 3 μl larutan TE 80% ditambah air distilasi sebanyak 597 μl, kemudian dimasukkan dalam tabung
eppendorf 1.5 ml. Sampel dan blanko disentrifugasi menggunakan spin down
selama 0.5 menit, kemudian dilakukan pengujian dengan spektrofotometer.
DNA Total Genom
DNA dari sepuluh jenis hewan (kambing, ayam, sapi, harimau, domba, babi, kuda, anjing, kucing, tikus) dicampur dalam satu tabung dengan jumlah DNA masing-masing spesies 100 ng DNA. Selanjutnya, diambil DNA sebanyak 50 ng dari campuran tersebut dan didistribusikan ke dalam tiga tabung. Tabung pertama mengandung 50 ng sampel DNA dengan pereaksi PCR menggunakan sepuluh primer, tabung kedua mengandung 50 ng sampel DNA dengan lima primer (kambing, sapi, domba, kuda, kucing), tabung ketiga mengandung 50 ng sampel DNA dengan lima primer (ayam, harimau, babi, anjing, dan tikus).
Amplifikasi Fragmen DNA Spesifik
Amplifikasi ruas gen cyt β menggunakan teknik PCR (polymerase chain reaction) dengan mesin thermocycler. Komponen PCR yang digunakan terdiri dari 50 ng sampel DNA (termasuk DNA total genom), pereaksi PCR (air distilasi 9 μl, primer forward 1.667 pmol, primer reverse masing-masing 0.1667 pmol (untuk setiap spesies), 1 x buffer, dNTPs 0.267 mM, MgCl2 1.667 mM, dan enzim taq fermentas 1 unit) dengan volume akhir 15 μl. Sementara pereaksi PCR dengan lima primer terdiri dari air distilasi 9.5 μl, primer forward 0.833 pmol, primer
reverse masing-masing spesies 0.1667 pmol, 1 x buffer, dNTPs 0.267 mM, MgCl2
1.667 mM, dan enzim taq fermentas 1 unit. Amplifikasi in vitro dengan mesin
12
tersebut selesai, tabung diambil dan disimpan pada suhu 4 oC sampai akan dianalisis lebih lanjut.
Elektroforesis dan Visualisasi Produk PCR
Produk PCR divisualisasikan dengan teknik elektroforesis gel agarosa 1.5%. Gel dibuat dari 0.45 gram agarosa dan 30 ml larutan buffer (0.5 x TBE) yang dipanaskan pada microwave selama ± 5 menit. Larutan agarosa dibiarkan agak dingin sambil diaduk dengan magnetic stirrer, lalu ditambahkan 2.5 µl pewarna
ethidium bromide. Produk PCR sebanyak 5 μl dicampur dengan 1 μl loading dye, kemudian dimasukkan pada masing-masing sisir yang terdapat pada gel agarosa 1.5%. Elektroforesis dilakukan selama 40 menit pada tegangan konstan 100 volt sampai campuran sampel dan loading dye tersebut mencapai garis ketiga pada bagian bawah gel. Setelah elektroforesis selesai, gel diambil untuk dilakukan pemotretan menggunakan UV-Transilluminator.
Analisis Data
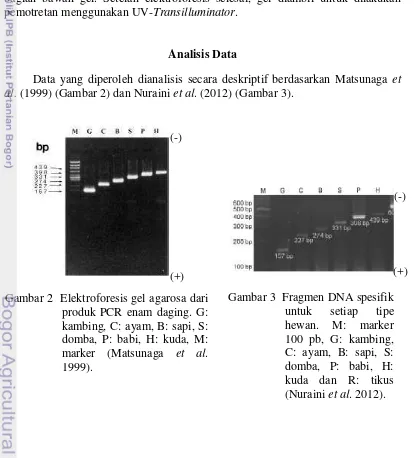
Data yang diperoleh dianalisis secara deskriptif berdasarkan Matsunaga et al. (1999) (Gambar 2) dan Nuraini et al. (2012) (Gambar 3).
Gambar 3 Fragmen DNA spesifik untuk setiap tipe hewan. M: marker 100 pb, G: kambing, C: ayam, B: sapi, S: domba, P: babi, H: kuda dan R: tikus (Nuraini et al. 2012). Gambar 2 Elektroforesis gel agarosa dari
produk PCR enam daging. G: kambing, C: ayam, B: sapi, S: domba, P: babi, H: kuda, M: marker (Matsunaga et al. 1999).
(-)
(+)
(-)
13
4
HASIL DAN PEMBAHASAN
Perancangan Primer Spesifik Anjing, Kucing, dan Harimau
Teknik multipleks PCR yang diaplikasikan dalam penelitian ini menggunakan satu primer forward dan sepuluh primer reverse secara bersamaan, untuk mengamplifikasi fragmen DNA sepuluh jenis hewan. Primer reverse anjing, kucing, dan harimau dirancang menggunakan program MEGA 5 (Tabel 3). Situs penempelan primer pada sekuen gen cyt β anjing, kucing dan harimau serta tujuh hewan lain disajikan pada Gambar 4.
Desain primer sangat penting pada teknik multipleks PCR, karena spesifisitas primer dan temperatur penempelan (Tm) sangat menentukan keberhasilan amplifikasi (Matsunaga et al. 1999). Primer yang digunakan dalam teknik multipleks PCR harus memenuhi beberapa syarat seperti: panjang primer 18-24 mer atau lebih, mengandung GC 35-60%, serta memiliki suhu annealing
55-58 oC atau lebih tinggi (Henegariu et al. 1997). Hasil rancangan primer reverse
anjing, kucing, dan harimau telah memenuhi syarat sebagai suatu rangkaian primer menurut Henegariu et al. (1997) dan ditampilkan pada Tabel 4.
Derajat Kesamaan Primer Spesifik
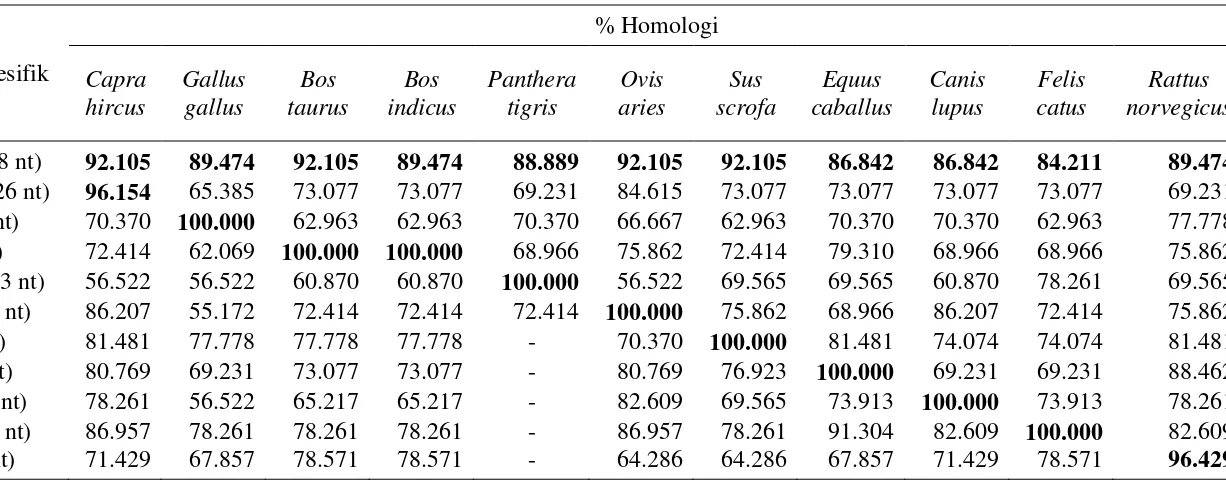
Uji homologi primer spesifik dilakukan menggunakan software GENETYX-WIN versi 4.0. Hasil uji homologi primer spesifik pada sekuen cyt β sepuluh jenis hewan disajikan pada Tabel 5. Hasil penelitian menunjukkan bahwa primer
forward memiliki persentase homologi yang relatif tinggi diantara spesies yaitu berkisar antara 84-92% (38 nukleotida), sehingga dapat digunakan sebagai primer umum. Runutan primer reverse memiliki persentase homologi yang tinggi pada
Tabel 3 Sekuen primer reverse gen cyt β spesies anjing, kucing, dan harimau
Jenis Hewan Reverse(5‟-3‟) Panjang Produk
PCR
Anjing TTG CTA GAG CTG CGA TGA TGA AA 523 pb
Kucing AGG GGT TGT TAG ATC CTG TTT CA 568 pb
Harimau TAG CCA TGA CCG TAA ACA ATA GC 319 pb
Tabel 4 Kriteria primer reverse anjing, kucing, dan harimau
Kriteria Primer anjing Primer kucing Primer harimau
Panjang primer (mer) 23 23 23
Kandungan basa GC (%) 43,48 43,48 43,48
Suhu annealing (oC) 60 60 60
14
satu jenis hewan dan lebih rendah pada hewan lainnya, sehingga dapat dikatakan bahwa primer reverse tersebut bersifat spesifik (Nuraini et al. 2012).
Matsunaga et al. (1999) menyatakan bahwa mismatch antara primer reverse
domba (5‟CTA TGA ATG CTG TGG CTA TTG TCG CA-3‟) dengan sekuen DNA kambing berbeda dua nukleotida, sementara pada penelitian ini hanya ditemukan perbedaan satu nukleotida, sehingga primer reverse domba pada penelitian ini dimodifikasi dengan menambahkan tiga basa pada ujung 3‟primer
reverse domba menjadi 5‟-CTA TGA ATG CTG TGG CTA TTG TCG CAA
AT-3‟. Mismatch (perbedaan) ujung 3‟ dari primer reverse domba terhadap sekuen DNA kambing sangat penting dalam mempengaruhi amplifikasi PCR, sehingga tidak dihasilkan pita domba pada sekuen DNA kambing (Matsunaga etal. 1999).
Penempelan primer reverse hanya pada sekuen spesifik jenis hewan tertentu disebabkan: 1) adanya mismatch ujung 3‟ pada setiap primer reverse (Matsunaga
et al. 1999), 2) mismatch diantara primer reverse terhadap masing-masing sekuen DNA menghasilkan suhu penempelan (Tm) yang berbeda (Viljoen et al. 2005).
Mismatch (perbedaan) pada penelitin ini berkisar antara 12-45%, setiap satu
mismatch menurunkan Tm 1.5 oC (Viljoen et al. 2005).
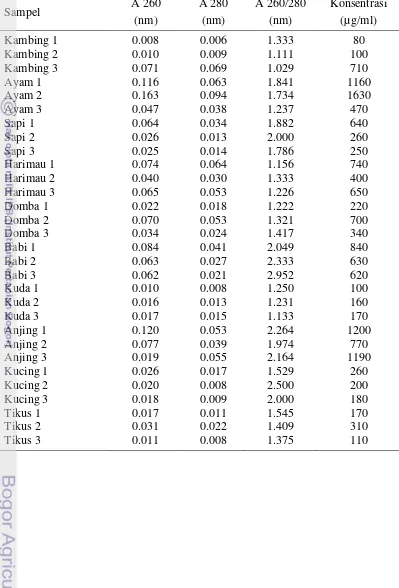
Kualitas DNA Total
Kemurnian DNA dari sampel (darah, feses, daging) hewan yang digunakan cukup baik dilihat dari rasio serapan A260/A280 berkisar antara 1.0 sampai 2.9 (Tabel 6). Rasio A260/A280 DNA murni adalah 1.8 (Sambrook dan Russel 2001; Clark dan Christopher 2001). Rasio A260/A280 yang kurang dari 1.8 menunjukkan bahwa DNA masih mengandung protein atau fenol (Sambrook dan Russel 2001; Clark dan Christopher 2001), rasio diatas 1.8 biasanya mengindikasikan kehadiran RNA. RNA biasanya didegradasi dengan menambahkan RNAse, sementara protein dirusak dengan denaturasi (Siun dan Beow 2009). Proses PCR akan terus berlangsung selama kontaminan tersebut tidak menghambat thermostable DNA atau degradasi DNA.
15
10 20 30 40 50 60 ....|....| ....|....| ....|....| ....|....| ....|....| ....|....|
Forward primer GACCTCCCAG CTCCATCAAA CATCTCATCT TGATGAAA
Capra_hircus ---A -C--- ---A ---CT TTGGATCCCT CCTAGGAATT
Rattus_norvegicus ---C- -C---T-- ---A ---CT TCGGTTCTCT ACTAGGAGTA
70 80 90 100 110 120 ....|....| ....|....| ....|....| ....|....| ....|....| ....|....|
Capra_hircus TGCCTAATCT TACAAATCCT GACAGGCCTA TTCCTAGCAA TACACTATAC ATCCGACACA
Gallus_gallus TGCCTCATGA CCCAAATCCT CACCGGCCTA CTACTAGCCA TGCACTACAC AGCAGACACA
Bos_taurus TGCCTAATCC TACAAATCCT CACAGGCCTA TTCCTAGCAA TACACTACAC ATCCGACACA
Bos_indicus TGCCTAATCC TACAAATCCT CACAGGCCTA TTCCTAGCAA TACACTACAC ATCCGACACA
Panthera_tigris TGCTTAATCT TACAAATCCT CACTGGCCTC TTTCTAGCCA TACACTACAC ATCAGACACA
Ovis_aries TGCTTAATTT TACAGATCCT AACAGGCCTA TTCCTAGCAA TACACTATAC ACCTGACACA
Sus_scrofa TGCCTAATCT TGCAAATCCT AACAGGCCTG TTCTTAGCAA TACATTACAC ATCAGACACA
Equus_caballus TGCCTAATCC TCCAAATCTT AACAGGCCTA TTCCTAGCCA TACACTACAC ATCAGACACG
Canis_lupus TGCTTGATTC TACAGATTCT AACAGGTTTA TTCTTAGCTA TGCACTATAC ATCGGACACA
Felis_catus TGCCTAATCT TACAAATCCT CACCGGCCTC TTTTTGGCCA TACACTACAC ATCAGACACA
Rattus_norvegicus TGCCTCATAG TACAAATCCT CACAGGCTTA TTCCTAGCAA TACACTACAC GTCTGATACC
130 140 150 160 170 180 ....|....| ....|....| ....|....| ....|....| ....|....| ....|....|
Capra_hircus ATAACAGCAT TTTCCTCTGT AACTCACATT TGTCGAGATG TAAATTATGG CTGAATCATC
Gallus_gallus TCCCTAGCCT TC---C-- -G-C----C- --C--GAACG TACAATACGG CTGACTCATC
Bos_taurus ACAACAGCAT TC--- T--C--T--C --C----ACG TGAACTACGG CTGAATCATC
Bos_indicus ACAACAGCAT TC--- T--C--T--C --C----ACG TGAACTACGG CTGAATCATC
Panthera_tigris ATAACCGCTT TC--A--A-- T--C--- --C--C-ACG TAAACTACGG TTGGATTATC
Ovis_aries ACAACAGCAT TC--- ---C--- --C----ACG TAAACTATGG CTGAATTATC
Sus_scrofa ACAACAGCTT TC--A--A-- T--A---C ---ACG TAAATTACGG ATGAGTTATT
Equus_caballus ACAACTGCCT TC--A--C-- C---C --C----ACG TTAACTACGG ATGAATTATT
Canis_lupus GCCACAGCTT T---A--A-- C--C---C --C----ACG TTAACTACGG CTGAATTATC
Felis_catus ATAACCGCCT T---A--A-- T--C---C ---C-ACG TTAATTATGG CTGAATCATC
Rattus_norvegicus ATAACAGCAT TC--A--A-- C--C---C --C----ACG TAAACTACGG CTGACTAATC
190 200 210 220 230 240 ....|....| ....|....| ....|....| ....|....| ....|....| ....|....|
Capra_hircus CGATACATAC ACGCAAACGG A--A---A-A ---T---- -CC-A--CAT ACATATCGGA
Gallus_gallus CGGAATCTCC ACGCAAACGG CGCCTCATTC TTCTTCATCT GTATCTTCCT TCACATCGGA
Bos_taurus CGATACATAC ACGCAAACGG A--T---A-G --T--T---- -CT-A-ATAT GCACGTAGGA
Bos_indicus CGATACATAC ACGCAAACGG A--T---A-G --T--T---- -CT-A-ATAT GCACGTAGGA
Panthera_tigris CGATATCTAC ATGCCAACGG A---CA-A ---T---- --C-A-ACAT GCACGTAGGA
Ovis_aries CGATACATAC ACGCAAACGG A--A---A-A --T--T---- -CC-A--TAT GCATGTAGGA
Sus_scrofa CGCTACCTAC ATGCAAACGG A--A--CA-G ---T--T- -CC-A--CAT CCACGTAGGC
Equus_caballus CGCTACCTCC ATGCCAACGG A--A---A-A --T--T---- -CC----CAT TCACGTAGGA
Canis_lupus CGCTATATGC ACGCAAATGG ---T--CA-A ---T---- -CC-A--CCT ACATGTAGGA
Felis_catus CGATATTTAC ACGCCAACGG A--T--TA-A ---T---- -CC-G-ACAT ACATGTAGGA
Rattus_norvegicus CGATACCTAC ACGCCAACGG ---A-A --T--- -CC-A--CCT CCATGTGGGA
250 260 270 280 290 300 ....|....| ....|....| ....|....| ....|....| ....|....| ....|....|
Capra_hircus CGAGGTC--- -C--T--A-- A--T--C--- ----AAACAT GAAACATTGG AGTAATC--C
Gallus_gallus CGAGG-C--- -C---C-- C---CTC-A- AAG-AAACCT GAAACACAGG AGTAATC--C
Bos_taurus CGAGGCTTAT ATTACGGGTC TTACACTTTT CTAGAAACAT GAAATATTGG AGTAATC--T
Bos_indicus CGAGGCTTAT ATTACGGGTC TTACACTTTT CTAGAAACAT GAAATATTGG AGTAATC--T
Panthera_tigris CGAGGAA--- -C---C-- C---C--C TC--AAACAT GAAATATCGG GATTGTGCTA
Ovis_aries CGAGG-C--- ----T--A-- A--T--C--C ----AAACAT GAAACATCGG AGTAATC--C
Sus_scrofa CGAGG-C--- -C---A-- C--T-TA--C ----AAACAT GAAACATTGG AGTAGTC---
Equus_caballus CGCGG-C-C- -C---C-- ---A--C ----AGACAT GAAACATTGG AATCATC---
Canis_lupus CGAGG-C--- ---A-- C--TGTA--C A---AAACAT GAAACATTGG AATTGTA---
Felis_catus CGGGGAA--- -C---C-- C---C--C TC--AGACAT GAAACATTGG AATCATA---
Rattus_norvegicus CGAGGAC--- -C--T--A-- C---C ----AAACCT GAAACATTGG GATCATC---
Primer_Panthera --- --- --- --- --- ---GCTA
310 320 330 340 350 360 ....|....| ....|....| ....|....| ....|....| ....|....| ....|....|
Capra_hircus CT-C-C*--* **---*-- --- -GCTATGTTT TACCATGAGG ACAAATATCA
Gallus_gallus CTCC-C--AC ----*--*-- C--C--TG-G -GCTATGTTC TCCCATGGGG CCAAATATCA
Bos_taurus CT-C-C--A- -*--*--*-- ---T--- -GATACGTCC TACCATGAGG ACAAATATCA
Bos_indicus CT-C-C--A- -*--*--*-- ---T--- -GATACGTCC TACCATGAGG ACAAATATCA
Panthera_tigris -T--- --- ---C--- -GATATGTCT TACCATGAGG ACAAATATCA
Ovis_aries CTATTTGCGA CAATAGCCAC AGCATTCATA GGCTATGTTT TACCATGAGG ACAAATATCA
Sus_scrofa CT*---C- -T--*--A-- ---C--- -GCTACGTCC TGCCCTGAGG ACAAATATCA
Equus_caballus CTT--C--A- -T--*--- ---G -GCTATGTCC TACCATGAGG CCAAATATCC
Canis_lupus -T*--C*-A* *---*--*-- ---G -GCTATGTAC TACCATGAGG ACAAATATCA
16
Rattus_norvegicus CT*---*-A- ----*--A-- T---G -GCTATGTAC TCCCATGAGG ACAAATATCA
Primer_Panthera TTGTTTACGG TCATGGCTA- --- --- --- ---
Sus_scrofa TTCTGAGGAG CTACGGTCAT CACAAATCTA CTATCAGCTA TCCCTTATAT CGGA--AGA-
Equus_caballus TTTTGAGGAG CA--A--- ---G--C--C ---AA TTCCCTACAT CGGTACTACC
Canis_lupus TTTTGAGGAG CA--T--A-- ---T---T --C--T--CA TCCCTTATAT CGGA---GA-
Felis_catus TTCTGAGGAG CA--C--A-- ---T--C--C --G---AA TTCCATACAT CGGG---GAA
Rattus_norvegicus TTCTGAGGAG C---A--A-- T---C--- T---TA TCCCTTACAT TGGG---
430 440 450 460 470 480 ....|....| ....|....| ....|....| ....|....| ....|....| ....|....|
Capra_hircus --A---A- ---G GGGATTCTCA GTAGACAAAG CCACTCTCAC CCGATTCTTC
Gallus_gallus --A--A---- --GC---G GGGATTTTCA GTCGACAACC CAACCCTTAC CCGATTCTTC
Bos_taurus T-A---A- ---G CGGATTCTCA GTAGACAAAG CAACCCTTAC CCGATTCTTC
Bos_indicus T-A---A- ---G CGGATTCTCA GTAGACAAAG CAACCCTTAC CCGATTCTTC
Panthera_tigris --- --- --- --- --- ---
Ovis_aries --A---A- ---G AGGATTCTCA GTAGACAAAG CTACCCTCAC CCGATTTTTC
Sus_scrofa ---A--A- ---G GGGCTTTTCC GTCGACAAAG CAACCCTCAC ACGATTCTTC
Equus_caballus CTCGTCGAGT GAATCTGAGG TGGATTCTCA GTAGACAAAG CCACCCTTAC CCGATTTTTT
Canis_lupus T-A--A--A- -G---G CGGCTTCTCA GTGGACAAAG CAACCCTAAC ACGATTCTTT
Felis_catus --A--A--A- -G---G GGGCTTCTCA GTAGACAAAG CCACCCTAAC ACGATTCTTC
Rattus_norvegicus --A---A- ---G AGGCTTCTCA GTAGACAAAG CAACCCTAAC ACGCTTCTTC
490 500 510 520 530 540 ....|....| ....|....| ....|....| ....|....| ....|....| ....|....|
Capra_hircus GCCTTCCACT TTATCCTCCC A--- A----C--C- -C-TAGTCCA CCTGCTTTTC
Gallus_gallus GCCTTACACT TCCTCCTCCC C--TGCA--- ----G-A-TA -T-TCATCCA CCTCACCTTC
Bos_taurus GCTTTCCATT TTATCCTTCC A--T--- AT---AA-T- -C-TAGTCCA CCTACTATTC
Bos_indicus GCTTTCCATT TTATCCTTCC A--T--- AT---AA-T- -C-TAGTCCA CCTACTATTC
Panthera_tigris --- --- --- --- --- ---
Ovis_aries GCCTTTCACT TTATTTTCCC A--- ---C--C- -C-TAGTTCA CCTACTCTTC
Sus_scrofa GCCTTGCACT TTATCCTGCC A---T A-C--C--C- --GCCGTACA TCTCCTATTC
Equus_caballus GCTTTCCACT TCATCCTACC C--- A----C--G- T-GTCGTACA TTTACTATTT
Canis_lupus GCATTCCATT TCATCCTCCC TTTCATCATC GCAGCTCTAG CAATAGTACA CCTCCTATTT
Felis_catus GCCTTCCACT TCATCCTTCC A---T--- T----CT--- --GCAGTACA CCTCTTATTC
Rattus_norvegicus GCATTCCACT TCATCCTCCC A---T--- --C--C--T- ---TTGTACA TCTTCTTTTC
550 560 570 580 590 600
Rattus_norvegicus CTCCAC---- ---A-- T---ACA GGATTAAACT CCGACGCAGA CAAAATCCCA
610
Rattus_norvegicus TTCCATCCAT
17
1
7
Tabel 5 Hasil uji homologi primer spesifik pada sepuluh jenis hewan
Primer Spesifik
% Homologi
Capra hircus
Gallus gallus
Bos taurus
Bos indicus
Panthera tigris
Ovis aries
Sus scrofa
Equus caballus
Canis lupus
Felis catus
Rattus norvegicus
Forward (38 nt) 92.105 89.474 92.105 89.474 88.889 92.105 92.105 86.842 86.842 84.211 89.474
Kambing (26 nt) 96.154 65.385 73.077 73.077 69.231 84.615 73.077 73.077 73.077 73.077 69.231
Ayam (27 nt) 70.370 100.000 62.963 62.963 70.370 66.667 62.963 70.370 70.370 62.963 77.778
Sapi (29 nt) 72.414 62.069 100.000 100.000 68.966 75.862 72.414 79.310 68.966 68.966 75.862
Harimau (23 nt) 56.522 56.522 60.870 60.870 100.000 56.522 69.565 69.565 60.870 78.261 69.565
Domba (29 nt) 86.207 55.172 72.414 72.414 72.414 100.000 75.862 68.966 86.207 72.414 75.862
Babi (27 nt) 81.481 77.778 77.778 77.778 - 70.370 100.000 81.481 74.074 74.074 81.481
Kuda (26 nt) 80.769 69.231 73.077 73.077 - 80.769 76.923 100.000 69.231 69.231 88.462
Anjing (23 nt) 78.261 56.522 65.217 65.217 - 82.609 69.565 73.913 100.000 73.913 78.261
Kucing (23 nt) 86.957 78.261 78.261 78.261 - 86.957 78.261 91.304 82.609 100.000 82.609
18
Tabel 6 Hasil pengukuran konsentrasi DNA sepuluh spesies
Sampel A 260
(nm)
A 280 (nm)
A 260/280 (nm)
Konsentrasi (µg/ml)
Kambing 1 0.008 0.006 1.333 80
Kambing 2 0.010 0.009 1.111 100
Kambing 3 0.071 0.069 1.029 710
Ayam 1 0.116 0.063 1.841 1160
Ayam 2 0.163 0.094 1.734 1630
Ayam 3 0.047 0.038 1.237 470
Sapi 1 0.064 0.034 1.882 640
Sapi 2 0.026 0.013 2.000 260
Sapi 3 0.025 0.014 1.786 250
Harimau 1 0.074 0.064 1.156 740
Harimau 2 0.040 0.030 1.333 400
Harimau 3 0.065 0.053 1.226 650
Domba 1 0.022 0.018 1.222 220
Domba 2 0.070 0.053 1.321 700
Domba 3 0.034 0.024 1.417 340
Babi 1 0.084 0.041 2.049 840
Babi 2 0.063 0.027 2.333 630
Babi 3 0.062 0.021 2.952 620
Kuda 1 0.010 0.008 1.250 100
Kuda 2 0.016 0.013 1.231 160
Kuda 3 0.017 0.015 1.133 170
Anjing 1 0.120 0.053 2.264 1200
Anjing 2 0.077 0.039 1.974 770
Anjing 3 0.019 0.055 2.164 1190
Kucing 1 0.026 0.017 1.529 260
Kucing 2 0.020 0.008 2.500 200
Kucing 3 0.018 0.009 2.000 180
Tikus 1 0.017 0.011 1.545 170
Tikus 2 0.031 0.022 1.409 310
19
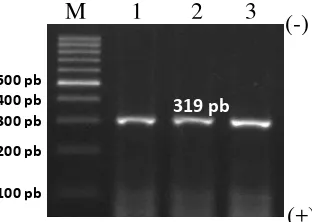
Amplifikasi DNA Fragmen Spesifik Gen Cyt βpada Anjing, Kucing, dan Harimau
Spesifitas primer diuji pada sampel olahan daging anjing, darah kucing, dan feses harimau. Produk olahan dari daging kucing masih jarang, namun fragmen DNA kucing tetap berhasil diamplifikasi dari darah. Begitu juga untuk sampel harimau, yang berhasil diamplifikasi dari feses segar. Penggunaan sumber sampel yang berbeda akan mempengaruhi proses ekstraksi DNA, sehingga memerlukan beberapa proses modifikasi (Nuraini et al. 2012). Berdasarkan konteks forensik, sampel yang diuji berpotensi untuk terdegradasi, DNA inti kemungkinan tidak menghasilkan hasil, oleh karena itu, DNA mitokondria mungkin digunakan sebagai alternatif untuk identifikasi spesies (Kitpipit et al. 2012).
Visualisasi hasil amplifikasi fragmen DNA masing-masing hewan pada gel agarosa 2% disajikan pada Gambar 5. Taksonomi harimau dan kucing mempunyai kesamaan sampai tingkat famili (Felidae) (Yu dan Zhang 2005; Weissengruber et al. 2002). Identifikasi spesies harimau dan kucing sudah dapat dibedakan sampai tingkat genus (harimau: Panthera, kucing: Felis) dengan penggunaan primer
reverse spesifik. Selain itu, primer reverse spesifik gen cyt β sudah dapat membedakan masing-masing hewan sampai pada tingkat spesies dalam hierarki taksonomi (harimau: Panthera tigris, kucing: Felis silvestris catus atau yang lebih dikenal dengan Felis catus, anjing: Canis lupus).
Gambar 5a Fragmen DNA spesifik spesies anjing. M: marker 100 pb, 1-3: DNA anjing (gulai, rica-rica, rebus).
20
Gambar 6 Fragmen DNA spesifik untuk setiap jenis hewan. M: marker 100 pb, G: kambing, C: ayam, B: sapi, T: harimau, S: domba, P: babi, H: kuda, D: anjing, F: kucing, R: tikus, Dw: kontrol negatif (-).
Amplifikasi DNA Fragmen Spesifik Gen Cyt βpada Sepuluh Spesies Hewan
Keberhasilan gen cyt β mengamplifikasi sepuluh jenis hewan dengan panjang fragmen yang berbeda menunjukkan spesifitas primer cyt β diantara spesies. Panjang fragmen amplifikasi dari kambing, ayam, sapi, domba, babi, kuda adalah 157, 227, 274, 331, 398, 439 pb (Matsunaga et al. 1999), tikus sepanjang 603 pb (Nuraini et al. 2012), sedangkan harimau, anjing dan kucing teramplifikasi sepanjang 319, 523, dan 568 pb. Visualisasi hasil amplifikasi fragmen DNA spesifik gen sitokrom β disajikan pada Gambar 6. Amplifikasi sekuen target DNA dari beberapa spesies dengan melibatkan banyak primer (menggunakan primer forward yang sama) pada reaksi yang sama merupakan salah satu variasi PCR yang disebut multipleks PCR (Matsunaga et al. 1999; Markoulatos et al. 2002; Jain et al. 2007). Penentuan spesies dengan PCR dipengaruhi oleh temperatur pemasakan, lama, dan ukuran fragmen DNA yang diamplifikasi (Martinez dan Yman 1998; Matsunaga et al. 1999; Arslan et al. Gambar 5c Fragmen DNA spesifik spesies harimau. M:
21
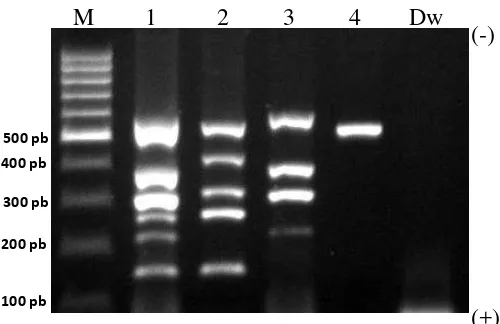
Amplifikasi DNA Fragmen Spesifik Gen Cyt β pada DNA Total Genom
Penelitian ini menunjukkan hanya enam pita (kambing, ayam, sapi, harimau, babi, dan kucing) yang teramplifikasi pada campuran DNA total sepuluh spesies hewan dengan menggunakan sepuluh primer (tabung pertama) (Gambar 7, nomor 1). Hal ini mungkin disebabkan adanya tumpang tindih pita diantara pita harimau dan domba; pita anjing, kucing, dan tikus. Molekul DNA untai ganda bermigrasi melewati matriks gel dengan laju yang berbanding terbalik dengan log 10 dari jumlah basa. Molekul yang lebih besar bermigrasi lebih lambat dari molekul kecil (Sambrook dan Russel 2001). Harimau (319 pb) dan domba (331 pb); anjing (523 pb), kucing (568 pb), and tikus (603 pb) mempunyai panjang fragmen yang berdekatan, sehingga laju migrasinya relatif sama dan menghasilkan pita yang tumpang tindih.
Oleh karena itu, pita yang tumpang tindih dan mempunyai panjang fragmen yang berdekatan dipisahkan. Tabung kedua yang mengandung DNA total genom dicampur dengan lima primer (kambing, sapi, domba, kuda, dan kucing), sementara tabung ketiga dicampur dengan primer ayam, harimau, babi, anjing, dan tikus). Hasil penelitian menunjukkan bahwa tabung kedua berhasil diamplifikasi dengan lima pita (kambing, sapi, domba, kuda, dan kucing), sementara tabung ketiga hanya berhasil diamplifikasi dengan empat pita (ayam, harimau, babi, dan tikus) tanpa pita anjing (Gambar 7, nomor 2 dan 3). Hasil yang sama juga diperoleh dari penelitian Matsunaga et al. (1999) yang menggunakan campuran primer dengan jumlah yang sama, tetapi tidak semua pita dari enam spesies (kambing, ayam, sapi, domba, babi, kuda) berhasil diamplifikasi. Umumnya, PCR kuantitatif sulit karena efisiensi amplifikasi yang tidak sama. Efisiensi amplifikasi dipengaruhi oleh perbedaan sekuen primer (Matsunaga et al. 1999). Primer yang lebih mudah menempel pada sekuen DNA akan mendominasi komponen pereaksi PCR.
Gambar 7 Amplifikasi fragmen spesifik dari DNA total. M: marker 100 pb, 1: kambing, ayam, sapi, harimau, babi, dan kucing (dari bawah ke atas); 2: kambing, sapi, domba, kuda, dan kucing (dari bawah ke atas); 3: ayam, harimau, babi, dan tikus (dari bawah ke atas); 4: anjing, Dw: kontrol negatif (-).
M 1 2 3 4 Dw
100 pb 200 pb 300 pb 400 pb 500 pb
(-)
22
Gambar 8 Amplifikasi fragmen spesifik obat harimau. M: marker 100 pb, G: obat + primer kambing, C: obat + primer ayam, B: obat + primer sapi, T: obat + primer harimau, S: obat + primer domba, P: obat + primer babi, H: obat + primer kuda, D: obat + primer anjing, F: obat + primer kucing, R: obat + primer tikus, Dw: kontrol negatif (-), A: kontrol positif feses harimau.
Amplifikasi DNA Fragmen Spesifik Gen Cyt β pada Obat Asal Hewan
Spesifitas primer diuji pada sampel obat harimau yang beredar dipasaran, dan didapatkan hasil pada Gambar 8. Sampel obat harimau mengandung DNA dengan konsentrasi 490 dan 750 µg/ml (Tabel 7). Hasil amplifikasi menunjukkan bahwa sampel obat tersebut tidak berasal dari harimau yang dibuktikan dengan tidak adanya pita harimau yang teramplifikasi. Kandungan DNA yang terdapat pada obat tersebut mungkin berasal dari tumbuh-tumbuhan atau hewan lain (selain kambing, ayam, sapi, harimau, domba, babi, kuda, anjing, kucing, dan tikus). Hal ini merupakan suatu bentuk pemalsuan informasi terhadap konsumen, seiring dengan permintaan pasar yang pesat dengan harga yang tinggi (Wetton et al. 2004), sementara populasi harimau yang hampir punah (Mazak 2010; Kitpipit et al. 2012).
Tabel 7 Hasil pengukuran konsentrasi DNA obat
Sampel A 260
(nm)
A 280 (nm)
A 260/280 (nm)
Konsentrasi (μg/ml)
Obat Harimau 1 0.075 0.062 1.210 750
Obat Harimau 2 0.049 0.040 1.225 490
M G C B T S P H D F R Dw A
319 pb
200 pb 300 pb 400 pb 500 pb
(-)
(+)
23
5 SIMPULAN DAN SARAN
Simpulan
Identifikasi spesies anjing, kucing dan harimau berhasil diamplifikasi dengan panjang fragmen yang berbeda-beda, yaitu 523, 568, dan 319 pb. Kespesifikan spesies anjing, kucing dan harimau juga ditunjukkan dengan adanya persentase homologi primer reverse yang tinggi pada masing-masing hewan. Metode multipleks PCR berhasil mengamplifikasi fragmen DNA dari sepuluh spesies, tetapi mempunyai batasan dalam mengamplifikasi total DNA dalam satu campuran. Penentuan spesies dengan multipleks PCR dipengaruhi oleh ukuran fragmen DNA yang diamplifikasi. Hierarki taksonomi anjing, kucing, dan harimau sudah dapat dibedakan sampai pada tingkat spesies dengan menggunakan gen sitokrom β (cyt β).
Saran
Amplifikasi fragmen spesifik pada DNA total genom yang mempunyai panjang berdekatan harus dipisahkan untuk mencegah pita yang tumpang tindih diantara spesies. Selain itu, pada kasus tertentu (misalnya pembuktian spesies), pita yang tumpang tindih dapat ditanggulangi dengan memodifikasi primer baru yang panjang fragmen amplifikasinya berjauhan.
DAFTAR PUSTAKA
Abdel-Rahman SM, El-Saadani MA, Ashry KM, Haggag AS. 2009. Detection of
adulteration and identification of cat‟s, dog‟s, donkey‟s and horse‟s meat using
species-specific PCR and PCR-RFLP Techniques. Aust J Basic Appl Sci 3: 1716-1719.
Achilli A, Olivieri A, Pellecchia M, Uboldi C, Colli L, Al-Zahery N, Accetturo M, Pala M, Kashani BH, Perego UA et al. 2008. Mitochondrial genomes of extinct aurochs survive in domestic cattle.
http://www.ncbi.nlm.nih.gov/nuccore/EU177824. [13 September 2012].
Ali ME, Hashim U, Mustafa S, Che Man YB, Dhahi TS, Kashif M, Uddin MK, Abd Hamid SB. 2012. Analysis of pork adulteration in commercial meatballs targeting porcine-specific mitochondrial cytochrome β gene by TaqMan probe real-time polymerase chain reaction. Meat Sci 91: 454-459. doi: 10.1016/j.meatsci.2012.02.031.
Angleby H, Savolainen P. 2005. Forensic informativity of domestic dog mtDNA control region sequences. Forensic Sci Int 154: 99-110. doi: 10.1016/j.forsciint.2004.09.132.