NINA RANTI HERMAN
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
tRNA Valin (tRNAVal) pada Reptilia. Dibimbing oleh ACHMAD FARAJALLAH dan BERRY JULIANDI.
Urutan nukleotida beberapa gen tRNAVal diekstrak dari genom lengkap mitokondria reptilia yang diambil dari GenBank serta dua genom parsial mitokondria milik Laboratorium Zoologi,
Institut Pertanian Bogor. Urutan nukleotida gen tRNAVal yang diperoleh kemudian dijajarkan berdasarkan struktur sekundernya. Gen ini terletak tepat di antara gen 12S rRNA dan 16S rRNA
tanpa adanya penataan ulang posisi maupun pseudogen. Komposisi nukleotida didominasi oleh
basa A dan T. Mutasi yang terjadi pada bagian batang kebanyakan adalah substitusi transisi. Gen
ini dapat digunakan dalam analisis filogenetik namun menghasilkan struktur pohon yang kurang
stabil. Selain urutan nukleotida pada bagian batang tRNA, nukleotida pada bagian lainnya (kecuali
bagian simpai D dan T) dapat diikutsertakan di dalam analisis. Pohon filogeni yang dihasilkan
mendukung formasi burung dan crocodilia dalam klad Archosauria, dan juga kecenderungan
kura-kura terhadap diapsida terutama Archosauria.
ABSTRACT
NINA RANTI HERMAN. Characterization of Mitochondrial DNA Sequence Encoding the tRNA
Valin (tRNAVal) Gene in Reptilia. Under the direction of ACHMAD FARAJALLAH dan BERRY JULIANDI.
We extracted tRNAVal gene sequences of some reptilians from complete mitochondrial genome sequences deposited in GenBank and from two partial mitochondrial genome sequences
property of Laboratory of Zoology, Bogor Agricultural University. These mitochondrial tRNAVal gene sequences were then aligned by virtue of their secondary structure. The gene itself was
located exactly between 12S rRNA and 16S rRNA genes without any rearrangements or
pseudogenes being observed. Nucleotide composition was dominated by A and T bases. Mutations
occurred in the stem region of the sequences were mostly transition kind of substitusion. This gene
was applicable in phylogenetic analysis but resulting less stabile trees. Besides the nucleotide
sequences of the stem region of tRNA, nucleotides in the other parts (except loops D and T) could
also be assigned in the analysis. The inferred trees supported the bird-crocodilian formation of
NINA RANTI HERMAN
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains pada
Departemen Biologi
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Nama
: Nina Ranti Herman
NRP
: G04400051
Menyetujui:
Pembimbing I
Pembimbing II
Dr. Ir. Achmad Farajallah, M.Si
NIP 131878947
Berry Juliandi, M.Si
NI 3010100093
Mengetahui:
Dekan Fakultas Matematika dan Ilmu Pengetahuan Alam
Institut Pertanian Bogor
Dr. drh. Hasim, DEA
NIP 131578806
sehingga karya ilmiah ini dapat terselesaikan. Tema yang dipilih dalam penelitian ini adalah
analisis urutan nukleotida, dengan judul Karakterisasi Urutan Nukleotida DNA Mitokondria
Penyandi tRNA Valin (tRNAVal) pada Reptilia
Penulis menyampaikan rasa terima kasih kepada bapak Dr. Ir. Achmad Farajallah, M.Si dan
bapak Berry Juliandi, M.Si atas kepercayaan dan kesabarannya dalam membimbing penulis. Di
samping itu, penghargaan setingginya penulis sampaikan kepada keluarga tercinta; mami dan papi
atas kasih sayang dan doa yang tak pernah putus, serta adik-adik terkasih Rika dan Rudy yang
selalu menghadirkan keceriaan. Ucapan terima kasih juga disampaikan kepada para sahabat
penulis: Tanty Indira atas kebersamaannya, Fitria Y. Bahri, S.Si atas motivasinya, D. Kurniawati,
S.Si dan Puji Rianti, S.Si atas inspirasinya, serta rekan-rekan Biopi 37 dan teman-teman TM3 atas
perhatian dan dorongan semangatnya. Penulis juga berterima kasih kepada keluarga besar
Laboratorium Zoologi atas dukungannya.
Karya ini tentu masih jauh dari sempurna. Namun penulis mengharapkan tulisan ini dapat
bermanfaat.
Bogor, November 2007
Karmela. Penulis merupakan anak pertama dari tiga bersaudara.
Setelah lulus dari SMU Islam Al-Azhar 1 Jakarta, penulis melanjutkan studinya di Institut
Pertanian Bogor, Fakultas Matematika dan Ilmu Pengetahuan Alam, Departemen Biologi pada
tahun 2000 melalui jalur Undangan Seleksi Masuk IPB (USMI). Penulis pernah berpartisipasi
sebagai anggota Tim Soal Lomba Cepat Tepat Biologi (LCTB) dalam rangkaian kegiatan Biologi
Interaktif (Bio-In) tahun 2001. Pada tahun 2003 penulis melaksanakan praktik lapang di
Laboratorium Mikrobiologi Rumah Sakit Pusat Pertamina (RSPP) Jakarta dengan judul Analisis
Halaman
DAFTAR GAMBAR ………. vii
DAFTAR LAMPIRAN ……….. vii
PENDAHULUAN Latar Belakang ………. 1
Tujuan Penelitian ……….. 2
Waktu dan Tempat ………... 2
BAHAN DAN METODE Data Urutan Nukleotida ……… 2
Penjajaran (Alignment) Urutan Nukleotida ……….. 2
Analisis Urutan Nukleotida dan Analisis Filogenetik ……….. 2
HASIL Analisis Urutan Nukleotida ……….. 2
Analisis filogenetik ………... 3
PEMBAHASAN ……… 3
KESIMPULAN ...………... 11
SARAN ……….. 11
DAFTAR PUSTAKA ……… 12
Halaman
1 Skema organisasi genom mitokondria vertebrata secara umum ………... 1
2 Struktur sekunder standar untuk tRNA (kecuali dua isoakseptor serin) ………. 2 3 Penjajaran urutan nukleotida gen tRNAVal mt menggunakan kriteria seperti yang
dipaparkan di dalam naskah …... 5
4 Urutan nukleotida gen tRNAVal mt reptilia setelah menghilangkan semua bagian simpai dan nukleotida pemisah, menyisakan 42 situs pada bagian batang ... 6
5 Pohon konsensus reptilia, burung, dan mamalia berdasarkan urutan nukleotida gen
tRNAVal pada bagian batang (42 basa) menggunakan metode Maximum Parsimony (MP) dengan bootstrap 100x ... 7
6 Pohon konsensus reptilia, burung, dan mamalia berdasarkan urutan nukleotida gen
tRNAVal pada bagian batang (42 basa) menggunakan metode Neighbor Joining (NJ) dengan kriteria Kimura 2-Parameter dan bootstrap 100x ... 8
7 Pohon konsensus reptilia, aves, dan mamalia berdasarkan urutan nukleotida gen tRNAVal tanpa bagian simpai D dan simpai T (57 basa) menggunakan metode Maximum
Parsimony (MP) dengan bootstrap 100x ... 9
8 Pohon konsensus reptilia, aves, dan mamalia berdasarkan urutan nukleotida gen tRNAVal tanpa bagian simpai D dan simpai T (57 basa) menggunakan metode Neighbor Joining
(NJ) dengan kriteria Kimura 2-Parameter dan bootstrap 100x ... 10
DAFTAR LAMPIRAN
Halaman
1 Daftar singkatan kode asam amino ………... 14
2 Genom mt hewan yang digunakan dalam analisis dan kode aksesnya pada Genbank ... 15
3 Komposisi basa pada bagian batang, jumlah nukleotida pada simpai D dan simpai T, serta
jumlah nukleotida total dari urutan nukleotida gen tRNAVal mt reptilia ………... 16 4 Rasio ts/tv dan jarak genetik reptilia berdasarkan urutan nukleotida gen tRNAVal mt pada
bagian batang (42 basa) ... 17
PENDAHULUAN
Latar Belakang
DNA mitokondria (mtDNA) vertebrata merupakan genom sirkular utas ganda. Genom ini menyandikan 13 protein subunit penyusun enzim-enzim fosforilasi oksidatif yang berfungsi dalam respirasi selular, dan 2 RNA ribosom (rRNA) serta 22 tRNA yang diperlukan dalam proses translasi protein mitokondria (mt). Terdapat pula ruas bukan penyandi (non-coding region) yang pada beberapa hewan berfungsi dalam pengaturan replikasi dan transkripsi (control region). Gen-gen mt tersebar pada kedua utas DNA, namun ada juga mtDNA yang seluruh gennya ditranskripsikan hanya dari satu utas DNA
Setiap utas DNA ditranskripsikan sebagai sebuah polisistron. Pada vertebrata, gen-gen tRNA pada mtDNA merupakan sinyal untuk pemrosesan polisistron pascatranskripsi (Ojala et al 1981). Gen-gen tRNA dalam mitokondria terletak menyebar di antara gen-gen penyandi protein. Urutan nukleotida (nt) gen-gen tRNA memungkinkannya untuk membentuk struktur sekunder yang melipat-lipat. Pada sebuah polisistron pascatranskripsi, enzim restriksi akan memotong tRNA dengan mengenali situs-situs lipatan struktur sekundernya, dan pada akhirnya memecah polisistron menjadi pesan-pesan yang spesifik gen.
Penelitian molekuler telah umum menggunakan mtDNA dalam analisis filogenetik. Beberapa karakteristik mtDNA yang menguntungkan di antaranya adalah ukuran yang relatif kecil (15-19 kb), susunan gen yang kompak hampir tanpa intron, dan sifat pewarisan maternal. Hasil penelitian Kumazawa dan Nishida (1993) menyimpulkan bahwa gen-gen tRNA pada mtDNA dapat digunakan untuk menentukan filogeni hewan pada percabangan dalam (deep-branch) yaitu percabangan yang terjadi 20-600 juta tahun yang lalu. Namun Springer et al (2001) mengemukakan bahwa ekson-ekson tertentu pada genom inti memiliki kinerja yang lebih baik untuk penentuan cabang dalam pada mamalia dibandingkan gen-gen pada mtDNA.
Berdasarkan klasifikasi tradisional, kelas Reptilia terdiri atas ordo Crocodilia (buaya), Squamata (kadal dan ular), Sphenodonta/Rhynchocephalia (tuatara), dan Testudinata (kura-kura). Kesemuanya termasuk ke dalam kelompok amniota
bersama kelas Aves dan Mamalia. Reptilia modern merupakan gabungan dua jalur (lineage) dari tiga jalur vertrebrata amniota; anapsida yang diwakili oleh kelompok kura-kura, dan diapsida yang diwakili oleh buaya, kadal, ular, dan tuatara. Namun kelas Reptilia mengecualikan burung, yang juga merupakan keturunan dari leluhur bersama diapsida. Oleh karena itu reptilia adalah kelompok yang parafiletik sehingga tidak memenuhi persyaratan kladistik dan tidak lagi dianggap sebagai takson yang valid dalam klasifikasi tradisional (Hickman et al 2000).
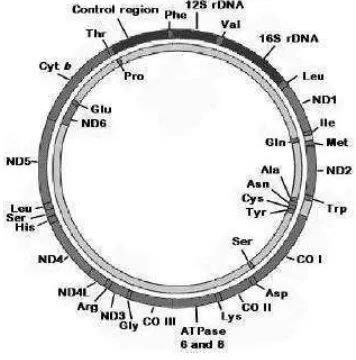
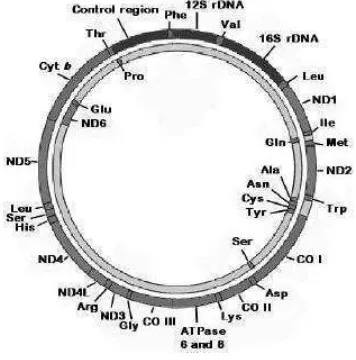
Gambar 1 Skema organisasi genom mitokondria vertebrata secara umum. Gen tRNA ditampilkan dengan singkatan 3 huruf untuk asam amino (lihat Lampiran 1). Gen penyandi protein ditampilkan sebagai berikut: Cyt b – sitokrom b; CO I-III – sitokrom oksidase subunit I-III; ND1-6 – NADH reduktase subunit 1-6 (gambar dari Pereira 2000).
berdasarkan gen-gen tRNA mt, dan oleh Janke dan Arnason (1997) yang melakukan analisis berdasarkan gen-gen penyandi protein mt.
Tujuan Penelitian
Penelitian ini dilakukan untuk mengetahui variasi basa nukleotida gen penyandi tRNA Valin (tRNAVal) dalam mtDNA. Selain itu, keefektifan gen tRNAVal dalam penentuan posisi filogenetik kelompok reptilia juga dieksplorasi.
Waktu dan Tempat
Penelitian ini dilaksanakan pada bulan Januari hingga Juli 2007, bertempat di Laboratorium Zoologi, Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor, Bogor.
BAHAN DAN METODE
Data Urutan Nukleotida
Data yang digunakan adalah urutan lengkap mtDNA dari beberapa spesies reptilia yang diambil dari GenBank (http://www.ncbi.nlm.nih.gov) serta dua data urutan mtDNA parsial milik Laboratorium Zoologi, Departemen Biologi, FMIPA IPB (lihat Lampiran 2 untuk rincian data). Dari data GenBank dipilih urutan nukleotida tRNAVal berdasarkan informasi yang
terdapat pada masing-masing entri. Data urutan nukleotida milik Laboratorium Zoologi FMIPA IPB dijajarkan dengan salah satu data urutan nukleotida gen tRNAVal mt yang telah diketahui kemudian ruas yang sejajar dipotong. Semua urutan nukleotida gen tRNAVal mt yang diperoleh selanjutnya digunakan dalam analisis.
Penjajaran (Alignment) Urutan
Nukleotida
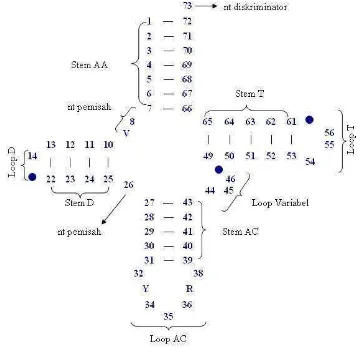
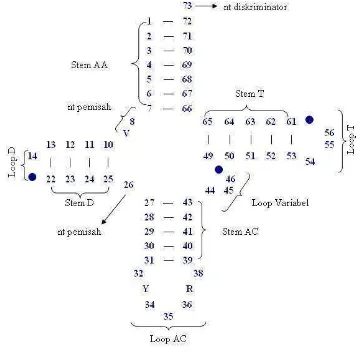
Semua data urutan nukleotida tRNAVal mt dijajarkan secara manual dengan mata berdasarkan kemampuannya membentuk struktur sekunder berupa lengan-lengan yang terdiri atas batang dan simpai (Gambar 2). Batang AA (asam amino) tidak memiliki simpai, berfungsi untuk membawa asam amino. Batang D (dihidrouridin) memiliki satu simpai. Batang AC (antikodon) memiliki satu simpai yang di dalamnya terdapat urutan nukleotida antikodon. Batang T (TψC) memiliki satu simpai. Di antara batang AA dan batang D serta antara batang D dan AC terdapat beberapa
nukleotida pemisah. Di antara batang AC dan batang T terdapat satu simpai tambahan (variabel). Nukleotida pada urutan paling akhir adalah satu nukleotida diskriminator.
Analisis Urutan Nukleotida dan Analisis Filogenetik
Seluruh analisis dilaksanakan menggunakan program MEGA 3.1 (Kumar et al 2004). Komposisi nukleotida ditentukan berdasarkan urutan nukleotida pada bagian batang gen tRNAVal mt. Perhitungan jarak genetik dilakukan berdasarkan urutan nukleotida pada bagian batang gen tRNAVal mt. Selain itu perhitungan jarak genetik juga dilakukan berdasarkan urutan nukleotida gen tRNAVal mt tanpa simpai D dan T. Konstruksi pohon filogenetik dilakukan menggunakan metode Maximum Parsimony (MP) dan Neighbor Joining (NJ) dengan kriteria Kimura-2-Parameter dan bootstrap 100x. Digunakan pula beberapa spesies burung dan mamalia sebagai pembanding (lihat Lampiran 2 untuk rincian data).
Gambar 2 Struktur sekunder standar untuk tRNA (kecuali dua isoakseptor serin). Nukleotida conserved ditunjukkan oleh R (purin), Y (pirimidin), dan V (nonuridin: A, G, atau C). Digambar kembali dari Kumazawa dan Nishida (1993). Sistem penomoran berdasarkan Sprinzl et al (1997).
HASIL
Analisis urutan nukleotida
diperoleh berkisar antara 15000 hingga 19000 bp. Data urutan nukleotida mtDNA parsial reptilia milik Laboratorium Zoologi, Departemen Biologi, FMIPA IPB berukuran 1330 dan 886 bp.
Semua gen tRNAVal mt yang teridentifikasi terletak di antara gen penyandi 12S rRNA dan 16S rRNA, seperti yang ditunjukkan pada Gambar 1. Tidak tercatat adanya penataan ulang posisi maupun kehadiran pseudogen tRNAVal mt.
Rataan komposisi nukleotida gen tRNAVal mt terdiri atas 57.7% A+T dan 42.3% G+C (Lampiran 3). Jumlah nukleotida penyusun tRNAVal mt berkisar antara 63 hingga 73 nt dengan rataan 67.97. Terdapat 7 bp pada batang AA, 4 bp pada batang D, masing-masing 5 bp pada batang AC dan T, 1 nt pemisah antara batang AA dan batang D, 2 nt pemisah antara batang D dan AC, dan 4 nt pada simpai variabel. Jumlah nt pada simpai D dan simpai T sangat bervariasi. Rataan jumlah nukleotida pada simpai D dan T adalah 6.0 dan 4.97 (Lampiran 3).
Analisis Filogenetik
Jarak genetik dihitung menggunakan pilihan Distance – Compute Pairwise pada program MEGA 3.1 (Lampiran 4). Berdasarkan urutan nukleotida gen tRNAVal mt pada bagian batang, rataan jarak genetik yang diperoleh adalah 0.37. Jarak genetik terkecil adalah 0.00 yang terjadi di antara tiga pasangan spesies yaitu D. subplana – P. sinensis, I. elongata – I. forstenii, dan G.gangeticus – C. niloticus, sedangkan jarak genetik terbesar adalah 1.97 yang terjadi di antara pasangan spesies L. dulcis – E. subglobosa. Berdasarkan urutan nukleotida gen tRNAVal mt tanpa simpai D dan T (lampiran 5), rataan jarak genetik yang diperoleh adalah 0.32. Jarak genetik terkecil adalah 0.00 yang terjadi di antara pasangan spesies I. elongata – I. forstenii, sedangkan jarak genetik terbesar adalah 1.01 yang terjadi di antara pasangan spesies B. constrictor – E. subglobosa.
Konstruksi pohon filogenetik dilakukan berdasarkan urutan nukleotida gen tRNAVal mt pada bagian batang (42 basa) dengan metode MP dan NJ (Gambar 5 dan 6) serta berdasarkan urutan nukleotida gen tRNAVal tanpa simpai D dan T (57 basa) dengan metode MP dan NJ (Gambar 7 dan 8). Semua pohon yang dihasilkan memiliki topologi yang mirip; spesies-spesies buaya, kura-kura dan burung membentuk satu
kelompok yang terpisah dari spesies-spesies kadal dan ular sedangkan spesies-spesies mamalia berada di luar sebagai outgroup.
PEMBAHASAN
Gen tRNAVal mt selalu ditemukan sebagai gen tunggal yang tidak membentuk kumpulan (cluster) dengan gen-gen tRNA lainnya. Kumpulan-kumpulan gen tRNA yang telah diketahui adalah IQM, WANCY, dan HSL (lihat Lampiran 1 untuk huruf singkatan asam amino). Posisi gen tRNAVal pada genom mitokondria adalah di antara gen 12S rRNA dan 16S rRNA, daerah yang memiliki tingkat keterpeliharaan (conservation rate) yang tinggi (Saccone et al 1999), dan tidak ditemukan adanya penataan ulang. Penataan ulang posisi gen dalam genom dapat menjadi karakter yang spesifik pada suatu takson. Beberapa penataan ulang posisi gen di antaranya adalah kumpulan gen tRNA HSL yang umum terdapat pada vertebrata mengalami penataan menjadi SHL pada Crocodilia, dan transposisi gen tRNA glutamin ke kumpulan gen tRNA lainnya (IQM-WANCY
→
IM-WQANCY) pada spesies ular blindsnake (Kumazawa & Nishida 1995). Selain itu telah ditemukan pula penataan letak gen pada Plastyternon megacephalum [Testudinata] berupa transposisi gen protein ND5 dan lima gen tRNA (H, S, L, T, P) yang menyisip di antara gen tRNA I dan Q pada kumpulan gen IQM (Parharn et al 2006).Ketiadaan penataan ulang gen tRNAVal kemungkinan disebabkan oleh posisinya di dalam genom mt. Gen tRNAVal terletak di antara gen 12S rRNA dan 16S rRNA. Hilangnya gen tRNAVal dari posisi tersebut karena mengalami penataan ulang ke posisi lain akan mengakibatkan rusaknya urutan nukleotida kedua gen rRNA sehingga proses translasi protein tidak dapat berjalan. Dengan kata lain sel akan mengalami kematian.
Tidak ditemukan pseudogen tRNAVal dalam mtDNA, baik yang berupa duplikasi gen utuh ataupun sebagian. Kumazawa et al (1996) menemukan adanya pseudogen urutan nukleotida control region tersisipkan ke dalam kumpulan gen IQM pada beberapa famili ular. Pseudogen lain yang telah diketahui di antaranya adalah pseudogen tRNA serin pada Mytilus californianus [Mollusca] (Beagly et al 1999).
pada reptilia sebesar 40.53% (± 2.22). Asimetri komposisi basa nukleotida ini terjadi pada semua genom mt Metazoa; komposisi G+C bernilai sangat rendah pada genom serangga dan nematoda, dan meningkat pada genom vertebrata hingga mencapai nilai tertinggi pada aves, mamalia (terutama primata) dan teleostei (Saccone et al 1999). Komposisi G+C berpengaruh terhadap kekuatan ikatan dan tingkat kestabilan DNA. Menurut Lynch (1997), bagian batang tRNA mitokondria hewan memiliki tingkat kestabilan dupleks yang rendah dibandingkan dengan tRNA inti, utamanya disebabkan oleh banyaknya insiden pasangan basa A-U yang melibatkan dua ikatan hidrogen daripada G-C yang melibatkan tiga ikatan hidrogen.
Semua gen tRNAVal mt reptilia dapat diidentifikasi berdasarkan struktur sekundernya yang berbentuk seperti daun semanggi (Kumazawa & Nishida 1993, Sprinzl et al 1997). Penjajaran urutan nukleotida ditampilkan pada Gambar 3. Urutan nukleotida gen tRNAVal mt melipat menjadi empat lengan (AA, D, AC, T) yang memiliki bentuk batang dan simpai. Keempat lengan tersebut terdapat secara utuh pada semua urutan nukleotida gen tRNAVal. Lengan AA berfungsi sebagai pembawa asam amino, lengan AC membawa urutan kodon pada bagian simpainya, lengan D dan T berperan dalam interaksi pembentukan struktur tersier yang menentukan fungsi tRNA. Wolstenholme et al (1987) mendapati seluruh tRNA pada Ascaris suum [Nematoda] tidak memiliki lengan T, namun dapat berfungsi normal karena adanya modifikasi pascatranskripsi (Sakurai et al 2005).
Variasi jumlah nukleotida penyusun gen tRNAVal mt terdapat pada simpai D dan T. Rataan jumlah nukleotida pada kedua situs tersebut adalah 6.00 dan 4.97. Lynch (1997) memperoleh nilai rataan jumlah nukleotida simpai D dan simpai T untuk reptilia dan burung sebesar 6.9 dan 7.0.
Beberapa situs tertentu pada simpai D dan T saling berinteraksi dalam pembentukan struktur tersier tRNA sehingga penjajaran pada kedua simpai tersebut harus mempertimbangan interaksi yang terjadi antara unsur-unsurnya. Hal ini, ditambah jumlah nukleotida yang bervariasi, membuat simpai D dan simpai T selalu memberikan hasil penjajaran yang ambigu (Kumazawa & Nishida 1993).
Jumlah nukleotida pada simpai D, simpai T, dan simpai variabel tRNA berbeda-beda. Situs-situs ini, bersama nukleotida-nukleotida pemisah dan diskriminator, biasanya tidak diikutsertakan dalam analisis (Kumazawa dan Nishida 1993). Pada penellitian ini, penghilangan nukleotida-nukleotida tersebut menghasilkan 42 basa pada bagian batang (Gambar 4). Namun spesies-spesies yang berkerabat dekat umumnya memiliki jumlah nukleotida yang sama pada simpai variabel, begitu juga pada nukleotida-nukleotida pemisah dan diskriminator, serta simpai AC. Oleh karena itu pada penelitian ini dilakukan analisis berdasarkan urutan nukleotida gen tRNAVal mt pada bagian batang (42 basa), dan berdasarkan urutan nukleotida gen tRNAVal mt tanpa simpai D dan T (57 basa).
Perhitungan jarak genetik berdasarkan 42 maupun 57 basa sama-sama menghasilkan jarak terkecil pada I. forstenii – I. Elongata. Berdasarkan 42 basa, jarak terkecil juga tejadi pada D. subplana – P. sinensis dan G. gangeticus – C. Niloticus (Lampiran 4). I. forstenii dan I. elongata merupakan anggota dari genus yang sama (Indotestudo). D. subplana dan P. sinensis bukan anggota dari genus yang sama namun kedua spesies ini tergabung dalam famili yang sama (Trionychidae). Begitu juga G. gangeticus – C. niloticus yang merupakan anggota dari famili yang sama (Crocodylidae). Perbedaan hasil yang diperoleh terutama disebabkan oleh pengaruh dari nukleotida-nukleotida pemisah dan diskriminator serta nukleotida-nukleotida pada simpai variabel yang diikutsertakan di dalam analisis.
Jarak genetik terbesar dari perhitungan berdasarkan 42 basa terjadi pada L. dulcis – E.subglobosa (Lampiran 4). E. subglobosa merupakan kura-kura famili Chelidae sedangkan L. dulcis [Leptotyphlopidae] (blindsnake) dianggap sebagai spesies ular yang paling awal berkembang dan memiliki laju evolusi yang lebih besar dari spesies-spesies ular primitif ataupun modern lainnya. Jarak genetik terbesar berdasarkan 57 basa terjadi pada B. constrictor – E. subglobosa (Lampiran 5) yang sebenarnya juga memiliki jarak genetik yang besar pada perhitungan berdasarkan 42 basa, namun nilainya sedikit di bawah L. dulcis – E. subglobosa.
AA D D AC AC T T AA ******* **** **** ***** ***** ***** ***** *******
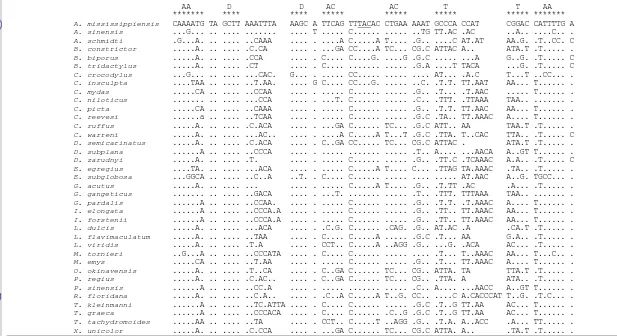
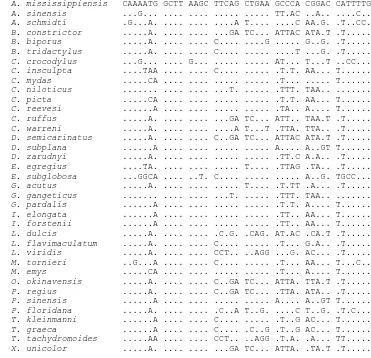
A. mississippiensis CAAAATG TA GCTT AAATTTA AAGC A TTCAG TTTACAC CTGAA AAAT GCCCA CCAT CGGAC CATTTTG A A. sinensis ...G... .. .... ... .... T ... C... ... ..TG TT.AC .AC ..A.. ....C.. . A. schmidti .G...A. .. .... ..CAAA .... . ....A C...A T.... .G.. ....C AT.AT AA.G. .T..CC. C B. constrictor ...A. .. .... .C.CA .... . ...GA CC....A TC... CG.C ATTAC A.. ATA.T .T... . B. biporus ...A. .. .... .CCA .... . C.... C....G. ....G .G.C ... ...A G..G. .T... C B. tridactylus ...A. .. .... .CT .... . C.... ... ... .G.A ....T TACA ...G. .T... C C. crocodylus ...G... .. .... ...CAC. G... . ... CC... ... .... AT... .A.C T...T ..CC... . C. insculpta ....TAA .. .... ..T.AA. .... G C.... CC...G. ... ..C. .T.T. TT.AAT AA... T... . C. mydas ...CA .. .... ..CCAA .... . ... C... ... .G.. .T... .T.AAC ... T... . C. niloticus ... .. .... ...CCA .... . ...T. C... ... .C.. .TTT. .TTAAA TAA.. ... . C. picta ...CA .. .... ..CAAA .... . ... C... ... .G.. .T.T. TT.AAC AA... T... . C. reevesi ...a .. .... ..TCAA .... . ... C... ... .G.C .TA.. TT.AAAC A.... T... . C. ruffus ...A. .. .... .C.ACA .... . ...GA C... TC... .G.C ATT.. AA TAA.T .T... . C. warreni ...A. .. .... ...AC.. .... . ....A C...A T...T .G.C .TTA. T..CAC TTA.. .T... C D. semicarinatus ...A. .. .... .C.ACA .... . C..GA CC... TC... CG.C ATTAC . ATA.T .T... . D. subplana ...A .. .... ..CCCA .... . ... ... ... .T.. A.... ...AACA A..GT T... . D. zarudnyi ...A. .. .... .T. .... . ... C... ... .G.. .TT.C .TCAAAC A.A.. .T... C E. egregius ....TA. .. .... ...ACA .... . ... C...A T.... C... .TTAG TA.AAAC .TA.. .T... . E. subglobosa ...GGCA .. .... ..C..A ..T. . C.... C... ... .... ... AT.AAC A..G. TGCC... . G. acutus ...A. .. .... ... .... . ... C...A T.... .G.. .T.TT .AC .A... .T... . G. gangeticus ... .. .... ..GACA .... . ...T. ... ... .T.. .TTT. TTTAAA TAA.. ... . G. pardalis ...A .. .... ..CCAA. .... . ... C... ... .G.. .T.T. .T.AAAC A.... T... . I. elongata ...A .. .... ..CCCA.A .... . ... C... ... .G.. .TT.. TT.AAAC AA... T... . I. forstenii ...A .. .... ..CCCA.A .... . ... C... ... .G.. .TT.. TT.AAAC AA... T... . L. dulcis ...A. .. .... ...ACA .... . .C.G. C... .CAG. .G.. AT.AC .A .CA.T .T... . L. flavimaculatum ...A. .. .... ..TAA .... . C.... C...A ... .G.C .T... AA G.A.. .T... . L. viridis ...A. .. .... .T.A .... . CCT.. C...A ..AGG .G.. ...G. .ACA AC... .T... . M. tornieri ..G...A .. .... ..CCCATA .... . C.... C... ... .... .T... T..AAAC AA... T...C.. . M. emys ...CA .. .... ..T.AA .... . ... C... ... .G.. .T... TT.AAAC A.... T... . O. okinavensis ...A. .. .... .T..CA .... . C..GA C... TC... CG.. ATTA. TA TTA.T .T... . P. regius ...A. .. .... .C.AC.. .... . C..GA C... TC... CG.. .TTA. A ATA.. .T... . P. sinensis ...A .. .... ..CC.A .... . ... ... ... .C.. A.... ...AACC A..GT T... . R. floridana ...A. .. .... ..C.A.. .... . .C..A C...A T..G. CC.. ....C A.CACCCAT T..G. .T.C... . T. kleinmanni ...A .. .... ..TC.ATTA .... . C.... C... ... .G.C .T..G TT.AA AC... T... . T. graeca ...A .. .... ..CCCACA .... . C.... C... .C..G .G.C .T..G TT.AA AC... T... . T. tachydromoides ...AA .. .... ..TA .... . CCT.. C...T ..AGG .G.. .T.A. A..ACC .A... TT... . X. unicolor ...A. .. .... .C.CCA .... . ...GA C... TC... CG.C ATTA. A.. .TA.T .T... .
Gambar 3 Hasil penjajaran urutan nukleotida gen tRNAVal mt reptilia secara manual dengan mata berdasarkan kemampuannya membentuk struktur sekunder. Nukleotida pada posisi
A. mississippiensis CAAAATG GCTT AAGC TTCAG CTGAA GCCCA CGGAC CATTTTG A. sinensis ...G... .... .... ... ... TT.AC ..A.. ....C.. A. schmidti .G...A. .... .... ....A T.... ....C AA.G. .T..CC. B. constrictor ...A. .... .... ...GA TC... ATTAC ATA.T .T... B. biporus ...A. .... .... C.... ....G ... G..G. .T... B. tridactylus ...A. .... .... C.... ... ....T ...G. .T... C. crocodylus ...G... .... G... ... ... AT... T...T ..CC... C. insculpta ....TAA .... .... C.... ... .T.T. AA... T... C. mydas ...CA .... .... ... ... .T... ... T... C. niloticus ... .... .... ...T. ... .TTT. TAA.. ... C. picta ...CA .... .... ... ... .T.T. AA... T... C. reevesi ...A .... .... ... ... .TA.. A.... T... C. ruffus ...A. .... .... ...GA TC... ATT.. TAA.T .T... C. warreni ...A. .... .... ....A T...T .TTA. TTA.. .T... D. semicarinatus ...A. .... .... C..GA TC... ATTAC ATA.T .T... D. subplana ...A .... .... ... ... A.... A..GT T... D. zarudnyi ...A. .... .... ... ... .TT.C A.A.. .T... E. egregius ....TA. .... .... ... T.... .TTAG .TA.. .T... E. subglobosa ...GGCA .... ..T. C.... ... ... A..G. TGCC... G. acutus ...A. .... .... ... T.... .T.TT .A... .T... G. gangeticus ... .... .... ...T. ... .TTT. TAA.. ... G. pardalis ...A .... .... ... ... .T.T. A.... T... I. elongata ...A .... .... ... ... .TT.. AA... T... I. forstenii ...A .... .... ... ... .TT.. AA... T... L. dulcis ...A. .... .... .C.G. .CAG. AT.AC .CA.T .T... L. flavimaculatum ...A. .... .... C.... ... .T... G.A.. .T... L. viridis ...A. .... .... CCT.. ..AGG ...G. AC... .T... M. tornieri ..G...A .... .... C.... ... .T... AA... T...C.. M. emys ...CA .... .... ... ... .T... A.... T... O. okinavensis ...A. .... .... C..GA TC... ATTA. TTA.T .T... P. regius ...A. .... .... C..GA TC... .TTA. ATA.. .T... P. sinensis ...A .... .... ... ... A.... A..GT T... P. floridana ...A. .... .... .C..A T..G. ....C T..G. .T.C... T. kleinmanni ...A .... .... C.... ... .T..G AC... T... T. graeca ...A .... .... C.... .C..G .T..G AC... T... T. tachydromoides ...AA .... .... CCT.. ..AGG .T.A. .A... TT... X. unicolor ...A. .... .... ...GA TC... ATTA. .TA.T .T...
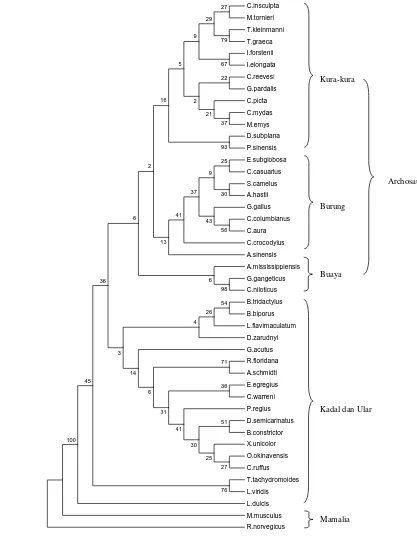
Gambar 5 Pohon konsensus reptilia, burung, dan mamalia berdasarkan urutan nukleotida gen tRNAVal pada bagian batang (42 basa) menggunakan metode Maximum Parsimony
(MP) dengan bootstrap 100x.
C.insculpta M.tornieri
T.kleinmanni
T.graeca
I.forstenii I.elongata
C.reevesi
G.pardalis
C.picta C.mydas
M.emys
D.subplana
P.sinensis
E.subglobosa C.casuarius
S.camelus
A.hastii G.gallus
C.columbianus
C.aura
C.crocodylus
A.sinensis A.mississippiensis
G.gangeticus
C.niloticus
B.tridactylus
B.biporus L.flavimaculatum
D.zarudnyi
G.acutus
R.floridana A.schmidti
E.egregius
C.warreni
P.regius
D.semicarinatus B.constrictor
X.unicolor
O.okinavensis
C.ruffus T.tachydromoides
L.viridis
L.dulcis
M.musculus R.norvegicus
98 93 79
76 71 67
56
54
51 43
30 25
9
37
41
27 25 30 41
37
36
31
27
29
26 22
21 9
2 5
16
6 14
13
6 2
6
4
3 36
45
100
Kura-kura
Buaya Burung
Kadal dan Ular
Archosauria
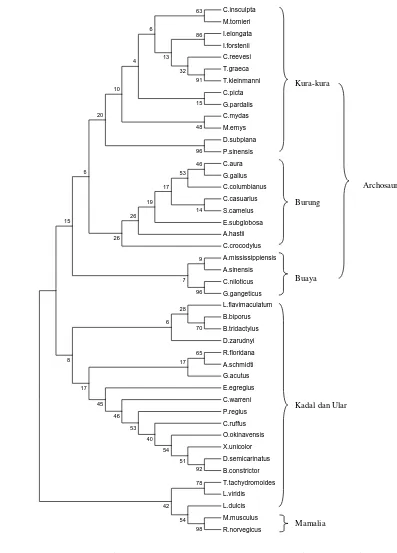
Gambar 6 Pohon konsensus reptilia, burung, dan mamalia berdasarkan urutan nukleotida gen tRNAVal pada bagian batang (42 basa) menggunakan metode Neighbor Joining (NJ) dengan kriteria Kimura 2-Parameter dan bootstrap 100x. Pohon NJ digambarkan secara rooted.
C.insculpta M.tornieri
T.kleinmanni
T.graeca
G.pardalis
C.picta C.mydas
M.emys
C.reevesi
I.forstenii I.elongata
D.subplana
P.sinensis
A.mississippiensis G.gangeticus
C.niloticus
A.sinensis
C.crocodylus E.subglobosa
C.casuarius
S.camelus A.hastii
C.columbianus
G.gallus
C.aura
R.floridana
A.schmidti B.tridactylus
B.biporus
G.acutus
L.flavimaculatum D.zarudnyi
E.egregius
C.warreni
P.regius C.ruffus
D.semicarinatus
B.constrictor
O.okinavensis X.unicolor
T.tachydromoides
L.viridis
L.dulcis
M.musculus R.norvegicus
95
95
90 85 80
70
65 50 30
20
5 30 50 60
35 35 35 35 50 45
40
40 35
30
25 10
5
15
35
35 30 20
10
5
30
15
15
5 10 40
40
100
Kura-kura
Buaya
Burung
Kadal dan Ular
Archosauria
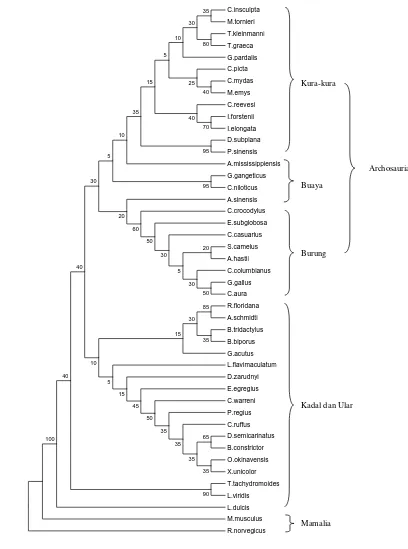
Gambar 7 Pohon konsensus reptilia, aves, dan mamalia berdasarkan urutan nukleotida gen tRNAVal tanpa bagian simpai D dan simpai T (57 basa) menggunakan metode Maximum Parsimony (MP) dengan bootstrap 100x.
C.mydas M.emys
C.picta
G.pardalis C.reevesi
T.kleinmanni
T.graeca
C.insculpta M.tornieri
I.forstenii
I.elongata D.subplana
P.sinensis
A.mississippiensis A.sinensis
C.niloticus
G.gangeticus C.crocodylus
A.hastii
S.camelus
E.subglobosa C.casuarius
G.gallus C.aura
C.columbianus
L.flavimaculatum
B.biporus B.tridactylus
D.zarudnyi
R.floridana A.schmidti
G.acutus
E.egregius C.warreni
C.ruffus
O.okinavensis
P.regius X.unicolor
D.semicarinatus
B.constrictor T.tachydromoides
L.viridis
L.dulcis M.musculus
R.norvegicus
98
92 90
73
70 64
64
63 56
27
18 24 50
49 43
40
36
24
9 18 22 26
26
24
22
19
15 6 4
2
6
14
14
12
6 2
7
6
3 26
38
100
Kura-kura
Buaya
Burung
Kadal dan Ular
Archosauria
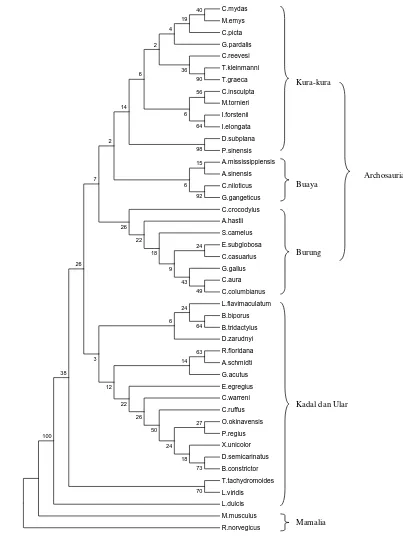
Gambar 8 Pohon konsensus reptilia, aves, dan mamalia berdasarkan urutan nukleotida gen tRNAVal tanpa bagian simpai D dan simpai T (57 basa) menggunakan metode Neighbor Joining (NJ) dengan kriteria Kimura 2-Parameter dan bootstrap 100x. Pohon NJ digambarkan secara rooted.
C.insculpta M.tornieri
I.elongata
I.forstenii
C.reevesi T.graeca
T.kleinmanni
C.picta G.pardalis
C.mydas
M.emys
D.subplana P.sinensis
C.aura
G.gallus C.columbianus
C.casuarius
S.camelus
E.subglobosa A.hastii
C.crocodylus
A.mississippiensis
A.sinensis C.niloticus
G.gangeticus
L.flavimaculatum B.biporus
B.tridactylus
D.zarudnyi R.floridana
A.schmidti
G.acutus
E.egregius C.warreni
P.regius
C.ruffus O.okinavensis
X.unicolor
D.semicarinatus
B.constrictor T.tachydromoides
L.viridis
L.dulcis
M.musculus R.norvegicus
96 96
92 91 86
78 70
65 63
51 54 40 53
46 53
48
46 45
32
28 14 17
19
26
26
15 13 6
4
10
20
17
17
9
7 6
15
6
8
42
54 98
Kura-kura
Buaya Burung
Kadal dan Ular
Mamalia
Semua pohon filogenetik dapat memisahkan kura-kura D. subplana dan P. sinensis yang tergabung dalam subordo Pleurodira (kura-kura cangkang lunak) dari spesies kura-kura lainnya yang merupakan anggota subordo Cryptodira (kura-kura cangkang keras). Namun beberapa spesies buaya (C.crocodylus dan A. sinensis) dan spesies kura-kura E. subglobosa cenderung bergabung dalam percabangan spesies-spesies burung. Ketidakstabilan juga terjadi dalam kelompok kadal. Spesies-spesies A. schmidti, B. biporus, B. tridactylus, D. zarudnyi, G. acutus, dan R. floridana dari superfamili Amphisbaenia cenderung bergabung, namun terpisah dari T. tachydromoides yang juga merupakan anggotanya.
Ketidakstabilan yang terjadi mungkin disebabkan oleh analisis yang hanya didasarkan pada satu gen saja. Cao et al (1994) telah menunjukkan bahwa set data yang besar, seperti serangkaian gen-gen yang berurutan, memberikan hasil yang lebih kuat dibandingkan dengan analisis yang didasarkan pada gen tunggal. Gen tRNAVal merupakan gen tunggal dengan urutan nt yang fungsional dan dapat digunakan dalam analisis filogeni, namun untuk memperoleh hasil yang lebih optimal masih diperlukan analisis bersama gen-gen lain.
Hal yang patut diperhatikan adalah posisi kura-kura di dalam pohon filogenetik yang dihasilkan. Kura-kura pada dasarnya adalah reptilia anapsida yang dicirikan oleh tidak adanya bukaan di belakang tulang orbit tengkorak, sedangkan buaya termasuk ke dalam reptilia diapsida yang memiliki dua bukaan di belakang tulang orbit tengkorak (Hickman et al 2000), begitu juga dengan burung. Topologi sister group kura-kura, buaya dan burung menyiratkan kecenderungan diapsida dari kura-kura dan afinitasnya terhadap kelompok diapsida archosauria. Penelitian-penelitian terdahulu juga sudah mengungkapkan afinitas kura-kura terhadap reptilia diapsida (Zardoya dan Meyer 1998), terutama Archosauria (Kumazawa dan Nishida 1999, Iwabe et al 2005).
Kecenderungan kura-kura kepada reptilia diapsida membuat pengelompokan lineage amniota (anapsida, diapsida, sinapsida) menjadi rancu. Hal ini disebabkan oleh reptilia yang tidak memiliki lubang di belakang orbit tengkorak (anapsida) dan reptilia yang memiliki dua lubang di belakang orbit tengkorak (diapsida) memiliki hubungan yang lebih dekat daripada spesies-spesies yang memiliki satu lubang di belakang orbit
tengkorak (sinapsida). Dengan demikian perlu dipertimbangkan kemungkinan bahwa karakter anapsida merupakan ciri sekunder dari diapsida. Selanjutnya perlu dikaji apakah pengelompokan anapsida, diapsida dan sinapsida masih relevan untuk digunakan.
KESIMPULAN
Gen tRNAVal mt reptilia yang terletak di antara gen 12S rRNA dan 16S rRNA tidak memiliki penataan ulang maupun pseudogen. Ukuran gen ini berkisar antara 63 hingga 73 nt dengan variasi pada jumlah nukleotida di posisi simpai D dan simpai T. Asimetri basa nukleotida terjadi dengan rataan komposisi A+T 57.7% dan G+C 42.3%.
Urutan nukleotida gen tRNAVal mt dapat digunakan dalam analisis filogenetik, namun perlu didukung oleh gen-gen lain untuk memperoleh hasil yang lebih baik. Analisis berdasarkan gen tRNAVal mt mendukung pandangan sistematik penggabungan burung dan buaya dalam klad Archosauria. Selain itu juga mendukung afinitas kura-kura terhadap diapsida, terutama Archosauria.
SARAN
DAFTAR PUSTAKA
Beagly CT, Okimoto R, Wolstenholme DR. 1999. Mytilus mitochondrial DNA contains a functional gene for a tRNASer(UCN) with a dihydrouridine
arm-replacement loop and a pseudo-tRNASer(UCN) gene. Genetics 152:641-652.
Cao Y, Adachi A, Janke A, Paabo S, Hasegawa M. 1994. Phylogenetic relationships among eutherian orders estimated from inferred protein sequnces of mitochondrial proteins: instability of a tree based on a single gene. J Mol Evol 39:519-527.
Hickman CP Jr, Roberts LS, Larson A. 2000. Integrated Principles of Zoology. Boston: McGraw-Hill.
Hedges SB. 1994. Molecular evidence for the origin of birds. Proc Natl Acad Sci USA 91:2621-2624.
Iwabe et al. 2005. Sister group relationship of turtles to the bird-crocodilian clade revealed by nuclear DNA-coded proteins. Mol Biol Evol 22(4):810-813.
Janke A, Arnason U. 1997. The complete mitochondrial genome of Alligator mississippiensis and the separation between recent Archosauria (birds and crocodiles). Mol Biol Evol 14:1266-1272. Kumar S, Tamura K, Nei M. 2004. MEGA 3:
Integrated software for molecular evolutionary genetics analysis and sequence alignment. Briefings in Bioinformatics 5:150-163.
Kumazawa Y, Nishida M. 1993. Sequence evolution of mitochondrial tRNA genes and deep-branch animal phylogenetics. J Mol Evol 37:380-389.
Kumazawa Y, Nishida M. 1995. Variations in mitochondrial tRNA gene organization of reptiles as phylogenetic markers. Mol Biol Evol 12(5):759-772.
Kumazawa Y. Nishida M. 1999. Complete mitochondrial DNA sequences of the green turtle and blue-tailed mole skink: statistical evidence for archosaurian affinity of turtles. Mol Biol Evol 16(6):784-792.
Kumazawa Y, Ota H, Nishida M, Ozawa T. 1996. Gene rearrangements in snake mitochondrial genomes: highly concerted evolution of control-region-like sequences duplicate and inserted into a tRNA gene cluster. Mol Biol Evol 13(9):1242-1254.
Lynch M. 1997. Mutation accumulation in nuclear, organelle, and prokaryotic
transfer RNA genes. Mol Biol Evol 14(9):914-925.
Ojala D, Montoya J, Attardi G. 1981. tRNA punctuation model of RNA processing in human mitochondria. Nature 290:470-474.
Parharn JF, Feldman CR, Boore JL. 2006. The complete mitochondrial genome of the enigmatic bigheaded turtle (Plastyternon): description of unusual genomic features and the reconciliation of phylogenetic hypotheses based on mitochondrial and nuclear DNA. BMC Evolutionary Biology 6(11). [terhubung berkala]. http://www.biomedcentral.com/ 147-2148/6/11 [4 Mei 2007].
Pereira SL. 2000. Mitochondrial genome organization and vertebrate phylogenetics. Genetics and Molecular Biology 23(4):742-752.
Puspa CS. 2005. Posisi filogenetik kura-kura Carettochelys insculpta (Reptilia: Testudinata) [Skripsi]. Bogor: Institut Pertanian Bogor.
Saccone C, De Giorgi C, Gissi C, Pesole G, Reyes A. 1999. Evolutionary genomics in Metazoa: the mitochondrial DNA as a model system. Gene 238:195-209. Sakurai M, Ohtsuki T. Watanabe K. 2005.
Modification at position 9 with 1-methyladenosine is crucial for structure and function of nematode mitochondrial tRNAs lacking the entire T-arm. Nucleic Acids Res 33:1653-1661.
Springer MS et al. 2001. Mitochondrial versus nuclear gene sequences in deep-level mammalian phylogeny rescontruction. Mol Biol Evol 18:132-143.
Sprinzl M, Horn C, Brown M, Ioudovitch A, Steinberg S. 1997. Compilation of tRNA sequences and sequences of tRNA genes. Nucleic Acids Res 26:148-153
Wolstenholme DR, MacFarlane JL, Okimoto R, Clary DO, Wahlethner JA. 1987. Bizarre tRNAs inferred from DNA sequences of mitochondrial genomes of nematode worms. Proc Natl Acad Sci USA 84:1324-1328.
Lampiran 1 Daftar singkatan kode asam amino
Singkatan satu huruf Singkatan tiga huruf Asam amino
A Ala Alanin
C Cys Sistein
D Asp Aspartat
E Glu Glutamat
F Phe Fenilalanin
G Gly Glisin
H His Histidin
I Ile Isoleusin
K Lys Lisin
L1 Leu(CUN) Leusin (CUN)
L2 Leu(UUR) Leusin (UUR)
M Met Metionin
N Asn Asparagin
P Pro Prolin
Q Gln Glutamin
R Arg Arginin
S1 Ser(AGN) Serin (AGN)
S2 Ser(UCN) Serin (UCN)
T Thr Treonin
V Val Valin
W Trp Triptofan
Y Tyr Tirosin
Lampiran 2 Genom mt hewan yang digunakan dalam analisis dan kode aksesnya pada Genbank
Kelompok Nama Spesies Kode Akses Genbank
Carettochelys insculpta (*) Chelonia mydas AB012104 Chrysemys picta AF069423 Chinemys reevesi AY676201 Dogania subplana AF366350 Emydura subglobosa (*) Geochelone pardalis DQ080041 Indotestudo elongata DQ656607 Indotestudo forstenii DQ080044 Malacochresus tornieri DQ080042
Manouria emys DQ080040
Pelodiscus sinensis AY962573 Testudo graeca DQ080050 Anapsida
Testudo kleinmanni DQ080048 Alligator mississippiensis AF069428 Alligator sinensis AF511507 Caiman crocodylus AJ404872 Crocodylus niloticus AJ810452 Archosauria
Gavialis gangeticus AB079596 Amphisbaena schmidti AY605475
Bipes biporus AY605480
Bipes tridactylus AY605477 Boa constrictor AB177354 Cordylus warreni AB079613 Cylindrophis ruffus AB179619 Dinodon semicarinatus AB008539 Diplometopon zarudnyi AY605474 Eumeces egregius AB016606 Geocalamus acutus AB162909 Lacerta viridis AM176577 Lepidophyma flavimaculatum AB162908 Leptotyphlops dulcis AB079242 Ovopis okinavensis AB175670
Phyton regius AB177878
Rhineura floridana AY605473 Takydromus tachydromoides AB080237
R
E
P
T
IL
IA
Lepidosauria
Xenopeltis unicolor AB179620 Cathartes aura AY463690 Cygnus columbianus DQ083161 Neognatha
Gallus gallus AB086102
Apteryx hastii AF338708 Casuarius casuarius AF338713
A
V
E
S
Paleognatha
Struthio camelus AF338715
Rattus norvegicus AJ428514
M
A
M
A
L
IA
Rodentia
Mus musculus domesticus AB042432
Lampiran 3 Komposisi basa pada bagian batang, jumlah nukleotida pada simpai D dan simpai T, serta jumlah nukleotida total dari urutan nukleotida gen tRNAVal mt reptilia
Komposisi basa (%) pada bagian batang
Jumlah nukleotida pada simpai Nama Spesies
T C A G D T
Jumlah nukleotida
total Carettochelys insculpta 28.6 19.0 35.7 16.7 7 6 70
Chelonia mydas 26.2 23.8 31.0 19.0 6 6 69
Chrysemys picta 28.6 19.0 35.7 16.7 6 6 69
Chinemys reevesi 28.6 16.7 35.7 19.0 6 7 70
Dogania subplana 28.6 19.0 33.3 19.0 6 7 70
Emydura subglobosa 19.0 31.0 23.8 26.2 6 6 69
Geochelone pardalis 31.0 16.7 33.3 19.0 7 7 71
Indotestudo elongata 31.0 16.7 35.7 16.7 8 7 72
Indotestudo forstenii 31.0 16.7 35.7 16.7 8 7 72
Malacochresus tornieri 23.8 23.8 33.3 19.0 8 7 72
Manouria emys 26.2 21.4 33.3 19.0 6 7 70
Pelodiscus sinensis 28.6 19.0 33.3 19.0 6 7 70
Testudo graeca 23.8 26.2 28.6 21.4 8 5 70
Testudo kleinmanni 26.2 23.8 31.0 19.0 9 5 71
Alligator mississippiensis Alligator sinensis Caiman crocodylus Crocodylus niloticus Gavialis gangeticus
23.8 26.2 26.2 35.7 35.7
26.2 26.2 23.8 16.7 16.7
28.6 28.6 26.2 31.0 31.0
21.4 19.0 23.8 16.7 16.7
7 5 7 6 6
4 3 4 6 6
68 65 68 69 69
Amphisbaena schmidti 21.4 28.6 28.6 21.4 6 5 68
Bipes biporus 28.0 21.4 31.6 19.0 4 4 65
Bipes tridactylus 21.4 26.2 23.8 28.6 3 4 64
Boa constrictor 33.3 16.7 35.7 14.3 5 3 65
Cordylus warreni 38.1 14.3 33.3 14.3 7 6 70
Cylindrophis ruffus 33.3 16.7 35.7 14.3 6 2 65
Dinodon semicarinatus 31.0 19.0 35.7 14.3 6 1 64
Diplometopon zarudnyi 28.6 21.4 31.0 19.0 3 7 67
Eumeces egregius 35.7 16.7 28.6 19.0 6 7 70
Geocalamus acutus 33.3 19.0 28.6 19.0 3 3 63
Lacerta viridis 21.4 26.2 28.6 23.8 4 4 65
Lepidophyma flavimaculatum 23.8 23.8 31.0 21.4 5 2 64
Leptotyphlops dulcis 23.8 28.6 31.0 16.7 6 2 65
Ovopis okinavensis 33.3 16.7 35.7 14.3 6 2 65
Phyton regius 28.6 19.0 35.7 16.7 7 1 65
Rhineura floridana 23.8 28.6 23.8 23.8 7 9 73
Takydromus tachydromoides 26.2 21.4 33.3 19.0 4 6 67
Xenopeltis unicolor 33.3 16.7 35.7 14.3 6 3 66
Lampiran 4 Rasio ts/tv dan jarak genetik reptilia berdasarkan urutan nukleotida gen tRNAVal mt pada bagian batang (42 basa)
SEQUENCE 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 1 A.mississippiensis 1.75 1.33 1 1.5 0.67 ∞ 6 ∞ 6 6 1.5 5.5 1.2 1.67 5 0.75 1 2 1.3 6 4 5 5 1.8 1 1.2 4 7 2.75 1.6 5 2.33 2.5 3.5 3.33 2.25 2 A.schmidti 0.33 1.8 9 5.5 8 3.5 3.67 2.5 2 3.33 2.5 5.5 1.4 6 3.3 ∞ 1.6 3.25 8 2 3.67 3.33 3.33 5 11 4 3.33 3.33 3 3.33 3.33 7 2.5 3 5 2.75 3 A.sinensis 0.19 0.46 1.17 1.6 1.75 2.33 1.75 2 1.25 1.75 1 2 1 1.8 2 0.6 0.67 2.4 1.5 1.25 1.25 1.75 1.75 2 0.5 1.83 1.5 1.75 2 1.33 2 2.75 1.2 1.6 3 1.6 4 B.biporus 0.16 0.31 0.41 4.33 0.5 3.33 4 2 2 4.5 2.33 13 1.75 4 3.5 7 2 2.67 3.5 2 4 4.5 4.5 3 ∞ 3 3.5 4.5 2.67 5 3.5 3.5 2.33 2.33 4.5 4 5 B.constrictor 0.51 0.45 0.41 0.62 4 1.67 2.2 1.67 0.71 2 1.5 0 1.33 ∞ 2 3 1.67 2.83 2.67 0.71 2 4.5 4.5 7 3 17 2 2.6 0.5 3 2 3.67 3 3 5.33 0 6 B.tridactylus 0.13 0.27 0.33 0.08 0.55 3.33 1.5 1.67 2 1.75 1 13 2 3.67 1.25 6 1.5 1.2 ∞ 2 1.5 1.75 1.75 6 1.5 1.5 1.25 1.75 3.67 2 1.25 ∞ 0.8 1.2 5 3.67 7 C.crocodylus 0.24 0.77 0.3 0.44 0.57 0.44 11 ∞ 10 11 4 5.5 2 1.8 8 2 2.2 2.2 3.33 10 9 10 10 2.2 2.67 2.6 9 12 2.75 2.6 8 3.67 5 6 5.33 2.75 8 C.insculpta 0.2 0.49 0.33 0.3 0.58 0.29 0.41 4 2.5 ∞ 3 4 1.17 2 ∞ 2.33 1.17 10 1 2.5 ∞ ∞ ∞ 1.83 3 2 ∞ ∞ 2 2 ∞ 3 2 4 1.25 2.2 9 C.mydas 0.11 0.48 0.26 0.26 0.57 0.22 0.32 0.13 8 2 0.5 6 1.4 1.83 5 1 1.2 4 1.67 8 2 3 3 2 1.33 4.5 0 5 3 1.8 5 3.33 1.5 2.5 2.33 2.5 10 C.niloticus 0.2 0.52 0.26 0.38 0.36 0.38 0.36 0.19 0.27 2.5 1.67 2 0.33 0.86 5 0.4 0.5 5 1 ∞ 2.5 2 2 1.22 1 1.5 3.5 4 1.2 0.67 5 2.75 2.33 3 2.75 1.2 11 C.picta 0.2 0.44 0.33 0.35 0.52 0.33 0.41 0.05 0.08 0.19 3 3.67 1 2.2 ∞ 2 1 10 0.75 2.5 ∞ ∞ ∞ 1.67 3.5 2.25 ∞ ∞ 2.2 2.25 ∞ 2.75 4 6 1.5 2 12 C.reevesi 0.13 0.48 0.29 0.3 0.51 0.29 0.3 0.1 0.08 0.22 0.1 2.5 0.71 1.67 4 0.75 0.71 4.5 1 1.67 1 1 1 1.43 1.67 1.8 1 4 1.67 1.6 4 1.8 1 2 1.4 1.5 13 C.ruffus 0.45 0.45 0.52 0.53 0.1 0.53 0.45 0.55 0.51 0.26 0.49 0.48 1.33 0.25 3.67 3.5 2.33 4.5 9 2 4 3 3 2.33 9 5 3.67 4.33 0.5 1 3.67 12 3 3 17 0.5 14 C.warreni 0.33 0.37 0.36 0.33 0.19 0.38 0.52 0.41 0.37 0.22 0.36 0.36 0.19 1.67 1.67 0.4 1.5 1.86 1 0.33 1 0.83 0.83 6 1 6 1 1.5 5 1.5 1.67 1.75 1.8 2 6 5 15 D.semicarinatus 0.57 0.51 0.46 0.55 0.02 0.49 0.63 0.52 0.63 0.4 0.58 0.57 0.13 0.22 2.2 3.5 2 2.67 3 0.86 2.2 2 2 8 2.67 16 2.2 2.4 0 2 2.2 4 2.75 2.75 5 0.5 16 D.subplana 0.16 0.44 0.38 0.26 0.52 0.26 0.27 0.2 0.16 0.4 0.2 0.13 0.49 0.57 0.58 2.67 1.67 9 2.25 5 ∞ ∞ ∞ 1.67 4.5 2.75 ∞ ∞ 2.2 3.25 ∞ 2.25 6 8 2.75 2 17 D.zarudnyi 0.19 0.32 0.22 0.23 0.23 0.2 0.38 0.3 0.22 0.19 0.26 0.19 0.26 0.19 0.26 0.34 0.5 2.75 6 0.4 1.67 1.33 1.33 2.67 3 3 1.33 2.67 1.75 1.67 2.67 9 1.25 1.75 3.67 1.5 18 E.egregius 0.29 0.41 0.29 0.38 0.22 0.29 0.58 0.41 0.33 0.25 0.36 0.36 0.3 0.13 0.26 0.57 0.16 1.71 0.5 0.5 1 0.83 0.83 5 1 6.5 1 1.5 8 2.5 1.67 2.25 1.4 1.8 6 6 19 E.subglobosa 0.4 0.68 0.65 0.34 1.61 0.33 0.45 0.36 0.3 0.81 0.36 0.35 1.58 0.87 1.26 0.31 0.54 0.77 2.4 5 10 11 11 2.43 3.33 2.4 8 11 2.67 2.8 9 2 4.5 5.5 2.4 2.83 20 G.acutus 0.19 0.27 0.29 0.26 0.34 0.17 0.44 0.22 0.22 0.22 0.19 0.29 0.31 0.22 0.39 0.42 0.2 0.16 0.65 1 1 1.25 1.25 5 2.5 2 1.25 1.75 3 1.5 2.25 ∞ 1 1.4 4.5 2.33 21 G.gangeticus 0.2 0.52 0.26 0.38 0.36 0.38 0.36 0.19 0.27 0 0.19 0.22 0.26 0.22 0.4 0.4 0.19 0.25 0.81 0.22 2.5 2 2 1.33 1 1.5 3.5 4 1.2 0.67 5 2.75 2.33 3 2.75 1.2 22 G.pardalis 0.13 0.49 0.26 0.3 0.52 0.29 0.31 0.05 0.08 0.19 0.05 0.05 0.55 0.36 0.58 0.14 0.22 0.36 0.36 0.22 0.19 ∞ ∞ 1.67 3 2.25 ∞ ∞ 2.2 2.25 ∞ 2.5 3 5 1.75 2 23 I.elongata 0.16 0.44 0.33 0.35 0.46 0.33 0.36 0.08 0.1 0.16 0.08 0.05 0.39 0.32 0.52 0.17 0.19 0.32 0.41 0.26 0.16 0.08 ∞ 1.83 3.5 2.5 ∞ ∞ 2 2 ∞ 2.75 3 5 1.75 1.8 24 I.forstenii 0.16 0.44 0.33 0.35 0.46 0.33 0.36 0.08 0.1 0.16 0.08 0.05 0.39 0.32 0.52 0.17 0.19 0.32 0.41 0.26 0.16 0.08 0 1.83 3.5 2.5 ∞ ∞ 2 2 ∞ 2.75 3 5 1.75 1.8 25 L.dulcis 0.46 0.81 0.38 0.6 0.23 0.51 0.58 0.63 0.52 0.46 0.57 0.62 0.3 0.51 0.27 0.57 0.34 0.4 1.97 0.4 0.46 0.57 0.63 0.63 2 5 1.67 2.17 9 5 1.17 6 2.2 2.2 5 7 26 L.flavimaculatum 0.16 0.41 0.25 0.11 0.39 0.13 0.34 0.23 0.19 0.22 0.26 0.22 0.31 0.22 0.34 0.35 0.1 0.22 0.44 0.19 0.22 0.23 0.26 0.26 0.38 4 2.5 3.5 2.33 3 4.5 4.5 1.67 2.33 4.5 2.67 27 L.viridis 0.33 0.55 0.63 0.23 0.91 0.29 0.74 0.38 0.46 0.51 0.42 0.46 0.81 0.51 0.78 0.54 0.39 0.57 0.65 0.38 0.51 0.42 0.48 0.48 0.4 0.3 2.25 2.5 16 ∞ 2.75 2 3 3 2 17 28 M.emys 0.13 0.44 0.29 0.26 0.52 0.26 0.31 0.11 0.02 0.26 0.05 0.05 0.49 0.36 0.58 0.14 0.19 0.36 0.27 0.26 0.26 0.05 0.08 0.08 0.57 0.19 0.42 ∞ 2.2 2.25 ∞ 2.25 3 5 1.75 2 29 M.tornieri 0.23 0.44 0.33 0.35 0.74 0.33 0.46 0.08 0.16 0.3 0.14 0.13 0.62 0.51 0.65 0.24 0.34 0.51 0.41 0.33 0.3 0.14 0.11 0.11 0.8 0.26 0.48 0.14 2.4 2.5 ∞ 3.25 3 5 1.75 2.6 30 O.okinavensis 0.54 0.6 0.52 0.49 0.08 0.49 0.54 0.52 0.6 0.33 0.58 0.57 0.08 0.16 0.05 0.58 0.33 0.27 1.26 0.39 0.33 0.58 0.52 0.52 0.31 0.3 0.78 0.58 0.65 2 2.2 4 3 3 16 ∞
31 P.regius 0.41 0.44 0.46 0.4 0.1 0.38 0.74 0.38 0.46 0.29 0.42 0.41 0.16 0.13 0.08 0.68 0.22 0.19 0.84 0.29 0.29 0.42 0.38 0.38 0.4 0.23 0.55 0.42 0.48 0.08 3.25 2.5 3.33 3.33 6.5 4 32 P.sinensis 0.16 0.44 0.38 0.26 0.52 0.26 0.27 0.2 0.16 0.4 0.2 0.13 0.49 0.57 0.58 0 0.34 0.57 0.31 0.42 0.4 0.14 0.17 0.17 0.57 0.35 0.54 0.14 0.24 0.58 0.68 2.25 6 8 2.75 2 33 R.floridana 0.3 0.23 0.54 0.26 0.49 0.24 0.49 0.6 0.44 0.54 0.54 0.46 0.46 0.33 0.55 0.42 0.31 0.42 0.52 0.32 0.54 0.48 0.54 0.54 0.51 0.35 0.38 0.42 0.68 0.55 0.48 0.42 2 2.4 6.5 4 34 T.kleinmanni 0.19 0.48 0.33 0.3 0.6 0.25 0.4 0.08 0.13 0.3 0.13 0.1 0.6 0.46 0.54 0.2 0.26 0.37 0.35 0.29 0.3 0.1 0.1 0.1 0.58 0.22 0.39 0.1 0.1 0.6 0.44 0.2 0.52 ∞ 1.2 3.25 35 T.graeca 0.26 0.6 0.41 0.3 0.6 0.33 0.51 0.13 0.19 0.39 0.2 0.16 0.6 0.52 0.54 0.27 0.33 0.46 0.45 0.37 0.39 0.16 0.16 0.16 0.58 0.3 0.39 0.16 0.16 0.6 0.44 0.27 0.65 0.05 1.2 3.25 36 T.tachydromoides 0.44 0.81 0.6 0.35 0.93 0.4 0.93 0.26 0.3 0.54 0.29 0.37 0.91 0.51 0.81 0.54 0.49 0.51 0.65 0.35 0.54 0.33 0.33 0.33 0.4 0.35 0.16 0.33 0.33 0.78 0.57 0.54 0.57 0.33 0.33 16 37 X.unicolor 0.42 0.54 0.41 0.55 0.05 0.49 0.54 0.58 0.48 0.33 0.52 0.51 0.08 0.16 0.08 0.52 0.29 0.2 1.61 0.3 0.33 0.52 0.46 0.46 0.23 0.34 0.91 0.52 0.74 0.05 0.1 0.52 0.55 0.68 0.68 0.78
Lampiran 5 Rasio ts/tv dan jarak genetik reptilia berdasarkan urutan nukleotida gen tRNAVal mt tanpa simpai D dan T (57 basa)
SEQUENCE 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 1 A.mississippiensis 1.5 0.8 1.8 1.6 0.6 ∞ 2.5 ∞ 3.5 8 3 7 1.3 2 2.5 1 0.9 5.5 1.5 3 6 7 7 2.2 1.5 1.3 6 8 2.6 1.7 2.5 1.3 4 5 4.3 2.4 2 A.schmidti 0.33 1 5.5 3.3 3 2.7 2.5 1.7 1.3 2 1.8 3 1.6 2.8 1.8 10 1.3 2.3 4 1.4 2.2 2 2 3 6 3 2 2.2 1.7 1.7 1.8 1.8 1.8 2.2 3 1.7 3 A.sinensis 0.22 0.48 0.9 1 1.4 1.3 1.3 1.2 0.6 1.1 0.8 1.4 0.6 1.2 1.1 0.4 0.4 1.5 0.9 0.8 0.9 1.1 1.1 1.3 0.4 1.2 1 1 1.2 0.9 1.1 1.1 0.9 1.1 2 1 4 B.biporus 0.23 0.29 0.45 2.5 1 3.5 3 2 1.7 3.7 2 7 1.6 2.8 2.5 9 1.6 2.8 2.3 1.8 3.3 3.7 3.7 2.8 2.5 2 3 4 2.6 3 2.5 1.5 2 2 4 2.6 5 B.constrictor 0.54 0.41 0.48 0.56 2 1.5 1.9 1.5 0.7 1.7 1.3 0.2 1 1 1.6 1.6 2.7 2.5 2.5 0.8 1.7 1.6 1.6 3 2.5 9.5 1.7 2.3 1 2.5 1.6 3.3 2.2 2.2 3.6 0.3 6 B.tridactylus 0.16 0.26 0.39 0.11 0.55 2.6 1.6 1.2 1.3 1.3 0.9 4.7 1.5 2.2 0.7 3.5 1 1.1 2.3 1.1 1.2 1.3 1.3 3.3 0.8 1 1 1.5 2 1.3 0.7 1.8 0.7 1 3 2 7 C.crocodylus 0.22 0.61 0.3 0.45 0.49 0.44 3.3 ∞ 5.5 13 5.5 7 1.9 1.9 5 2 1.7 6 3 6 11 12 12 2.6 2.8 2.5 11 13 2.6 2.5 5 2 6.5 7.5 6.3 2.8 8 C.insculpta 0.31 0.56 0.42 0.38 0.63 0.43 0.41 1.8 1.3 1.7 2 4.3 1.3 2 2.8 2.2 1.4 2.8 1.3 1.5 2 2.3 2.3 2.1 2.8 2 2.3 2 2 2 2.8 1.9 1.8 2.3 2 2.3 9 C.mydas 0.12 0.36 0.28 0.25 0.49 0.23 0.27 0.23 4 2 1 6.5 1.1 1.9 3 0.8 1 4.5 1.3 4.5 2 3 3 2 1.3 1.5 0 6 2.4 1.5 3 1.7 2 3 2.7 2.2 10 C.niloticus 0.18 0.42 0.27 0.36 0.38 0.36 0.29 0.3 0.21 1.7 1.5 1.8 0.3 0.9 6 0.3 0.3 3.8 0.7 ∞ 1.7 1.3 1.3 1.1 0.8 1.1 2.3 2.7 0.9 0.5 5.5 1.8 2 2.5 2.4 1 11 C.picta 0.19 0.34 0.33 0.32 0.46 0.3 0.33 0.16 0.05 0.16 4 4 0.9 2.2 8 1.5 0.9 11 0.6 2 ∞ ∞ ∞ 1.7 2.7 1.8 ∞ ∞ 1.8 1.8 8 1.6 5 7 1.8 1.8 12 C.reevesi 0.16 0.4 0.3 0.25 0.42 0.27 0.29 0.25 0.07 0.2 0.09 2.5 0.6 1.6 3 0.8 0.8 5.5 1 1.8 2 2 2 1.6 1.3 1.7 2 6 1.6 1.5 3 1.3 1 2 1.8 1.3 13 C.ruffus 0.39 0.38 0.46 0.39 0.13 0.42 0.39 0.59 0.36 0.23 0.35 0.31 0.8 0.4 3.3 2.7 1.8 5 5 2 4.3 3.3 3.3 2.7 4.5 4 4 5 0.7 1 3.3 3.3 3 3 19 0.3 14 C.warreni 0.36 0.28 0.41 0.28 0.2 0.33 0.5 0.53 0.33 0.25 0.33 0.3 0.18 1 1.3 0.5 1.3 1.7 1 0.4 0.9 0.8 0.8 3.3 0.8 4.3 0.9 1.4 1.5 0.8 1.3 1.1 1.3 1.4 3.3 1.3 15 D.semicarinatus 0.55 0.48 0.49 0.48 0.04 0.47 0.5 0.55 0.5 0.38 0.47 0.43 0.13 0.25 2 2.3 2.3 2.7 2.2 1 2.2 2 2 5 1.8 6 2.2 2.5 1 4 2 2.8 2.4 2.4 4.5 1 16 D.subplana 0.14 0.4 0.39 0.31 0.54 0.25 0.26 0.34 0.16 0.32 0.19 0.16 0.41 0.53 0.55 1.8 1.2 5 1.7 5 6 7 7 1.6 2.8 2 6 9 1.7 2.3 ∞ 1.8 4 5 2.6 1.7 17 D.zarudnyi 0.2 0.24 0.27 0.21 0.28 0.18 0.34 0.37 0.18 0.18 0.2 0.18 0.23 0.18 0.28 0.31 0.3 2.4 2 0.4 1.3 1 1 2 1.3 1.8 1 2.3 1.2 1 1.8 1.8 1.2 1.6 3 1.2 18 E.egregius 0.27 0.36 0.32 0.43 0.23 0.36 0.46 0.39 0.3 0.25 0.33 0.35 0.31 0.18 0.28 0.49 0.2 1.3 0.6 0.4 0.9 0.8 0.8 2.8 1.2 4.7 0.9 1.1 4.5 2 1.2 1.8 1.3 1.6 3.3 4 19 E.subglobosa 0.29 0.51 0.49 0.34 1.01 0.33 0.32 0.34 0.23 0.49 0.27 0.29 0.78 0.67 0.85 0.26 0.4 0.53 2.2 4 11 12 12 2.6 3 2.2 9 11 2.4 2.5 5 1.3 5.5 6.5 2.8 2.7 20 G.acutus 0.2 0.21 0.33 0.28 0.31 0.2 0.38 0.3 0.18 0.2 0.16 0.25 0.26 0.2 0.37 0.36 0.18 0.16 0.47 0.8 0.8 1 1 3.3 3 2 1 1.6 1.8 1 1.7 5 1 1.3 3 1.6 21 G.gangeticus 0.16 0.46 0.3 0.4 0.41 0.33 0.32 0.33 0.23 0.04 0.18 0.23 0.25 0.27 0.42 0.26 0.2 0.27 0.54 0.22 2 1.7 1.7 1.3 1 1.3 2.7 3 1 0.6 5.5 2.2 2.3 2.8 2.6 1.1 22 G.pardalis 0.14 0.37 0.27 0.29 0.46 0.28 0.27 0.18 0.05 0.16 0.04 0.05 0.38 0.33 0.47 0.14 0.18 0.33 0.27 0.18 0.18 ∞ ∞ 1.7 2.3 1.8 ∞ ∞ 1.8 1.8 6 1.4 4 6 2 1.8 23 I.elongata 0.16 0.34 0.33 0.32 0.43 0.3 0.3 0.2 0.07 0.14 0.06 0.05 0.29 0.3 0.43 0.16 0.16 0.3 0.3 0.2 0.16 0.06 ∞ 1.8 2.7 2 ∞ ∞ 1.7 1.6 7 1.6 4 6 2 1.7 24 I.forstenii 0.16 0.34 0.33 0.32 0.43 0.3 0.3 0.2 0.07 0.14 0.06 0.05 0.29 0.3 0.43 0.16 0.16 0.3 0.3 0.2 0.16 0.06 0 1.8 2.7 2 ∞ ∞ 1.7 1.6 7 1.6 4 6 2 1.7 25 L.dulcis 0.37 0.52 0.36 0.48 0.26 0.41 0.44 0.59 0.34 0.33 0.36 0.43 0.23 0.41 0.26 0.43 0.25 0.34 0.77 0.29 0.36 0.36 0.4 0.4 1.8 3.3 1.7 2.3 4.5 3.3 1.6 2.4 2.4 2.4 5.5 4 26 L.flavimaculatum 0.2 0.32 0.3 0.14 0.31 0.18 0.34 0.34 0.18 0.22 0.23 0.18 0.23 0.18 0.31 0.34 0.14 0.23 0.38 0.16 0.25 0.2 0.23 0.23 0.31 4.5 2 3 1.6 1.8 2.8 2.5 1.3 1.8 3.3 1.6 27 L.viridis 0.3 0.38 0.56 0.25 0.62 0.3 0.56 0.43 0.33 0.39 0.31 0.36 0.54 0.38 0.6 0.43 0.31 0.42 0.47 0.25 0.42 0.31 0.34 0.34 0.29 0.23 1.8 2.2 5.3 7 2 1.3 2.5 2.5 1.3 6 28 M.emys 0.14 0.34 0.3 0.26 0.46 0.25 0.27 0.2 0.02 0.2 0.04 0.05 0.35 0.33 0.47 0.14 0.16 0.33 0.21 0.2 0.23 0.04 0.06 0.06 0.36 0.18 0.31 ∞ 1.8 1.8 6 1.3 4 6 2 1.8 29 M.tornieri 0.19 0.37 0.3 0.35 0.65 0.33 0.33 0.18 0.14 0.23 0.12 0.14 0.46 0.46 0.56 0.21 0.28 0.39 0.27 0.28 0.26 0.12 0.1 0.1 0.51 0.26 0.37 0.12 2.2 2.2 9 1.9 5 7 2.3 2.5 30 O.okinavensis 0.44 0.46 0.49 0.44 0.11 0.43 0.44 0.55 0.4 0.27 0.4 0.43 0.09 0.2 0.07 0.46 0.28 0.23 0.71 0.31 0.3 0.4 0.36 0.36 0.23 0.28 0.5 0.4 0.47 2 1.7 2.4 2.6 2.6 8.5 ∞
31 P.regius 0.36 0.36 0.45 0.38 0.14 0.36 0.56 0.43 0.33 0.25 0.31 0.33 0.16 0.18 0.09 0.51 0.2 0.18 0.56 0.25 0.27 0.31 0.28 0.28 0.29 0.23 0.39 0.31 0.37 0.05 2.3 1.7 2.8 2.8 4.7 4 32 P.sinensis 0.14 0.4 0.39 0.31 0.54 0.25 0.26 0.34 0.16 0.29 0.19 0.16 0.41 0.53 0.55 0.02 0.31 0.49 0.26 0.36 0.29 0.14 0.16 0.16 0.43 0.34 0.43 0.14 0.21 0.46 0.51 1.7 4 5 2.6 1.7 33 R.floridana 0.3 0.23 0.52 0.33 0.41 0.31 0.43 0.63 0.36 0.4 0.43 0.42 0.41 0.33 0.48 0.4 0.31 0.31 0.42 0.26 0.47 0.39 0.43 0.43 0.4 0.31 0.3 0.36 0.5 0.4 0.36 0.36 1.4 1.6 2.6 2.6 34 T.kleinmanni 0.21 0.4 0.33 0.25 0.47 0.25 0.36 0.23 0.11 0.25 0.12 0.07 0.38 0.36 0.4 0.21 0.23 0.36 0.29 0.25 0.28 0.09 0.09 0.09 0.4 0.18 0.31 0.09 0.12 0.44 0.34 0.21 0.46 ∞ 1.6 2.6 35 T.graeca 0.26 0.47 0.39 0.25 0.47 0.3 0.43 0.28 0.16 0.31 0.16 0.11 0.38 0.39 0.4 0.26 0.28 0.43 0.36 0.3 0.34 0.14 0.14 0.14 0.4 0.23 0.31 0.14 0.16 0.44 0.34 0.26 0.54 0.04 1.6 2.6 36 T.tachydromoides 0.38 0.52 0.55 0.35 0.68 0.38 0.66 0.34 0.23 0.4 0.23 0.31 0.59 0.41 0.64 0.44 0.38 0.41 0.48 0.26 0.44 0.25 0.25 0.25 0.29 0.29 0.14 0.25 0.28 0.52 0.42 0.44 0.44 0.28 0.28 9 37 X.unicolor 0.4 0.46 0.42 0.44 0.07 0.43 0.48 0.65 0.37 0.3 0.4 0.36 0.07 0.18 0.07 0.46 0.28 0.21 0.85 0.28 0.33 0.4 0.36 0.36 0.21 0.28 0.6 0.4 0.56 0.06 0.09 0.46 0.44 0.44 0.44 0.57
NINA RANTI HERMAN
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
tRNA Valin (tRNAVal) pada Reptilia. Dibimbing oleh ACHMAD FARAJALLAH dan BERRY JULIANDI.
Urutan nukleotida beberapa gen tRNAVal diekstrak dari genom lengkap mitokondria reptilia yang diambil dari GenBank serta dua genom parsial mitokondria milik Laboratorium Zoologi,
Institut Pertanian Bogor. Urutan nukleotida gen tRNAVal yang diperoleh kemudian dijajarkan berdasarkan struktur sekundernya. Gen ini terletak tepat di antara gen 12S rRNA dan 16S rRNA
tanpa adanya penataan ulang posisi maupun pseudogen. Komposisi nukleotida didominasi oleh
basa A dan T. Mutasi yang terjadi pada bagian batang kebanyakan adalah substitusi transisi. Gen
ini dapat digunakan dalam analisis filogenetik namun menghasilkan struktur pohon yang kurang
stabil. Selain urutan nukleotida pada bagian batang tRNA, nukleotida pada bagian lainnya (kecuali
bagian simpai D dan T) dapat diikutsertakan di dalam analisis. Pohon filogeni yang dihasilkan
mendukung formasi burung dan crocodilia dalam klad Archosauria, dan juga kecenderungan
kura-kura terhadap diapsida terutama Archosauria.
ABSTRACT
NINA RANTI HERMAN. Characterization of Mitochondrial DNA Sequence Encoding the tRNA
Valin (tRNAVal) Gene in Reptilia. Under the direction of ACHMAD FARAJALLAH dan BERRY JULIANDI.
We extracted tRNAVal gene sequences of some reptilians from complete mitochondrial genome sequences deposited in GenBank and from two partial mitochondrial genome sequences
property of Laboratory of Zoology, Bogor Agricultural University. These mitochondrial tRNAVal gene sequences were then aligned by virtue of their secondary structure. The gene itself was
located exactly between 12S rRNA and 16S rRNA genes without any rearrangements or
pseudogenes being observed. Nucleotide composition was dominated by A and T bases. Mutations
occurred in the stem region of the sequences were mostly transition kind of substitusion. This gene
was applicable in phylogenetic analysis but resulting less stabile trees. Besides the nucleotide
sequences of the stem region of tRNA, nucleotides in the other parts (except loops D and T) could
also be assigned in the analysis. The inferred trees supported the bird-crocodilian formation of
PENDAHULUAN
Latar Belakang
DNA mitokondria (mtDNA) vertebrata merupakan genom sirkular utas ganda. Genom ini menyandikan 13 protein subunit penyusun enzim-enzim fosforilasi oksidatif yang berfungsi dalam respirasi selular, dan 2 RNA ribosom (rRNA) serta 22 tRNA yang diperlukan dalam proses translasi protein mitokondria (mt). Terdapat pula ruas bukan penyandi (non-coding region) yang pada beberapa hewan berfungsi dalam pengaturan replikasi dan transkripsi (control region). Gen-gen mt tersebar pada kedua utas DNA, namun ada juga mtDNA yang seluruh gennya ditranskripsikan hanya dari satu utas DNA
Setiap utas DNA ditranskripsikan sebagai sebuah polisistron. Pada vertebrata, gen-gen tRNA pada mtDNA merupakan sinyal untuk pemrosesan polisistron pascatranskripsi (Ojala et al 1981). Gen-gen tRNA dalam mitokondria terletak menyebar di antara gen-gen penyandi protein. Urutan nukleotida (nt) gen-gen tRNA memungkinkannya untuk membentuk struktur sekunder yang melipat-lipat. Pada sebuah polisistron pascatranskripsi, enzim restriksi akan memotong tRNA dengan mengenali situs-situs lipatan struktur sekundernya, dan pada akhirnya memecah polisistron menjadi pesan-pesan yang spesifik gen.
Penelitian molekuler telah umum menggunakan mtDNA dalam analisis filogenetik. Beberapa karakteristik mtDNA yang menguntungkan di antaranya adalah ukuran yang relatif kecil (15-19 kb), susunan gen yang kompak hampir tanpa intron, dan sifat pewarisan maternal. Hasil penelitian Kumazawa dan Nishida (1993) menyimpulkan bahwa gen-gen tRNA pada mtDNA dapat digunakan untuk menentukan filogeni hewan pada percabangan dalam (deep-branch) yaitu percabangan yang terjadi 20-600 juta tahun yang lalu. Namun Springer et al (2001) mengemukakan bahwa ekson-ekson tertentu pada genom inti memiliki kinerja yang lebih baik untuk penentuan cabang dalam pada mamalia dibandingkan gen-gen pada mtDNA.
Berdasarkan klasifikasi tradisional, kelas Reptilia terdiri atas ordo Crocodilia (buaya), Squamata (kadal dan ular), Sphenodonta/Rhynchocephalia (tuatara), dan Testudinata (kura-kura). Kesemuanya termasuk ke dalam kelompok amniota
bersama kelas Aves dan Mamalia. Reptilia modern merupakan gabungan dua jalur (lineage) dari tiga jalur vertrebrata amniota; anapsida yang diwakili oleh kelompok kura-kura, dan diapsida yang diwakili oleh buaya, kadal, ular, dan tuatara. Namun kelas Reptilia mengecualikan burung, yang juga merupakan keturunan dari leluhur bersama diapsida. Oleh karena itu reptilia adalah kelompok yang parafiletik sehingga tidak memenuhi persyaratan kladistik dan tidak lagi dianggap sebagai takson yang valid dalam klasifikasi tradisional (Hickman et al 2000).
Gambar 1 Skema organisasi genom mitokondria vertebrata secara umum. Gen tRNA ditampilkan dengan singkatan 3 huruf untuk asam amino (lihat Lampiran 1). Gen penyandi protein ditampilkan sebagai berikut: Cyt b – sitokrom b; CO I-III – sitokrom oksidase subunit I-III; ND1-6 – NADH reduktase subunit 1-6 (gambar dari Pereira 2000).
berdasarkan gen-gen tRNA mt, dan oleh Janke dan Arnason (1997) yang melakukan analisis berdasarkan gen-gen penyandi protein mt.
Tujuan Penelitian
Penelitian ini dilakukan untuk mengetahui variasi basa nukleotida gen penyandi tRNA Valin (tRNAVal) dalam mtDNA. Selain itu, keefektifan gen tRNAVal dalam penentuan posisi filogenetik kelompok reptilia juga dieksplorasi.
Waktu dan Tempat
Penelitian ini dilaksanakan pada bulan Januari hingga Juli 2007, bertempat di Laboratorium Zoologi, Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor, Bogor.
BAHAN DAN METODE
Data Urutan Nukleotida
Data yang digunakan adalah urutan lengkap mtDNA dari beberapa spesies reptilia yang diambil dari GenBank (http://www.ncbi.nlm.nih.gov) serta dua data urutan mtDNA parsial milik Laboratorium Zoologi, Departemen Biologi, FMIPA IPB (lihat Lampiran 2 untuk rincian data). Dari data GenBank dipilih urutan nukleotida tRNAVal berdasarkan informasi yang
terdapat pada masing-masing entri. Data urutan nukleotida milik Laboratorium Zoologi FMIPA IPB dijajarkan dengan salah satu data urutan nukleotida gen tRNAVal mt yang telah diketahui kemudian ruas yang sejajar dipotong. Semua urutan nukleotida gen tRNAVal mt yang diperoleh selanjutnya digunakan dalam analisis.
Penjajaran (Alignment) Urutan
Nukleotida
Semua data urutan nukleotida tRNAVal mt dijajarkan secara manual dengan mata berdasarkan kemampuannya membentuk struktur sekunder berupa lengan-lengan yang terdiri atas batang dan simpai (Gambar 2). Batang AA (asam amino) tidak memiliki simpai, berfungsi untuk membawa asam amino. Batang D (dihidrouridin) memiliki satu simpai. Batang AC (antikodon) memiliki satu simpai yang di dalamnya terdapat urutan nukleotida antikodon. Batang T (TψC) memiliki satu simpai. Di antara batang AA dan batang D serta antara batang D dan AC terdapat beberapa
nukleotida pemisah. Di antara batang AC dan batang T terdapat satu simpai tambahan (variabel). Nukleotida pada urutan paling akhir adalah satu nukleotida diskriminator.
Analisis Urutan Nukleotida dan Analisis Filogenetik
Seluruh analisis dilaksanakan menggunakan program MEGA 3.1 (Kumar et al 2004). Komposisi nukleotida ditentukan berdasarkan urutan nukleotida pada bagian batang gen tRNAVal mt. Perhitungan jarak genetik dilakukan berdasarkan urutan nukleotida pada bagian batang gen tRNAVal mt. Selain itu perhitungan jarak genetik juga dilakukan berdasarkan urutan nukleotida gen tRNAVal mt tanpa simpai D dan T. Konstruksi pohon filogenetik dilakukan menggunakan metode Maximum Parsimony (MP) dan Neighbor Joining (NJ) dengan kriteria Kimura-2-Parameter dan bootstrap 100x. Digunakan pula beberapa spesies burung dan mamalia sebagai pembanding (lihat Lampiran 2 untuk rincian data).
Gambar 2 Struktur sekunder standar untuk tRNA (kecuali dua isoakseptor serin). Nukleotida conserved ditunjukkan oleh R (purin), Y (pirimidin), dan V (nonuridin: A, G, atau C). Digambar kembali dari Kumazawa dan Nishida (1993). Sistem penomoran berdasarkan Sprinzl et al (1997).
HASIL
Analisis urutan nukleotida
[image:30.612.324.506.352.528.2]berdasarkan gen-gen tRNA mt, dan oleh Janke dan Arnason (1997) yang melakukan analisis berdasarkan gen-gen penyandi protein mt.
Tujuan Penelitian
Penelitian ini dilakukan untuk mengetahui variasi basa nukleotida gen penyandi tRNA Valin (tRNAVal) dalam mtDNA. Selain itu, keefektifan gen tRNAVal dalam penentuan posisi filogenetik kelompok reptilia juga dieksplorasi.
Waktu dan Tempat
Penelitian ini dilaksanakan pada bulan Januari hingga Juli 2007, bertempat di Laboratorium Zoologi, Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor, Bogor.
BAHAN DAN METODE
Data Urutan Nukleotida
Data yang digunakan adalah urutan lengkap mtDNA dari beberapa spesies reptilia yang diambil dari GenBank (http://www.ncbi.nlm.nih.gov) serta dua data urutan mtDNA parsial milik Laboratorium Zoologi, Departemen Biologi, FMIPA IPB (lihat Lampiran 2 untuk rincian data). Dari data GenBank dipilih urutan nukleotida tRNAVal berdasarkan informasi yang
terdapat pada masing-masing entri. Data urutan nukleotida milik Laboratorium Zoologi FMIPA IPB dijajarkan dengan salah satu data urutan nukleotida gen tRNAVal mt yang telah diketahui kemudian ruas yang sejajar dipotong. Semua urutan nukleotida gen tRNAVal mt yang diperoleh selanjutnya digunakan dalam analisis.
Penjajaran (Alignment) Urutan