KEMIRIPAN GENETIK DAN POLA PENYEBARAN SERBUK
SARI POPULASI KELAPA KOPYOR PATI BERDASARKAN
ANALISIS MARKA SSR DAN SNAP
RINI ISMAYANTI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Kemiripan Genetik dan Pola Penyebaran Serbuk Sari Populasi Kelapa Kopyor Pati Berdasarkan Analisis Marka SSR dan SNAP adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Oktober 2013
Rini Ismayanti NIM A253100031
RINGKASAN
RINI ISMAYANTI. Kemiripan Genetik dan Pola Penyebaran Serbuk Sari Populasi Kelapa Kopyor Pati Berdasarkan Analisis Marka SSR dan SNAP. Dibimbing oleh DEWI SUKMA dan SUDARSONO.
Bunga betina buah kelapa kopyor yang diserbuki oleh kelapa berbuah normal diduga menyebabkan penurunan produksi buah kopyor, sehingga perlu diketahui pola penyebaran serbuk sari pada populasi campuran antara kelapa berbuah kopyor dengan kelapa berbuah normal. Selain itu, evaluasi kemiripan genetik pada populasi tertentu dan progeninya juga perlu dilakukan. Tujuan penelitian ini adalah (1) mempelajari kemiripan genetik populasi kelapa berbuah kopyor di Kabupaten Pati, Jawa Tengah dan progeninya, (2) mempelajari pola penyebaran serbuk sari populasi kelapa campuran antara kelapa berbuah kopyor dengan kelapa berbuah normal, dan (3) mengevaluasi keterkaitan antara penyebaran serbuk sari dengan produksi buah kopyor. Populasi tanaman dewasa yang dievaluasi sebagai kandidat tetua sebanyak 95 individu dan populasi progeni sebanyak 84 bibit yang dipanen dari 15 pohon induk betina terpilih. Induk betina dipilih berdasarkan data produksi buah yang tinggi diperoleh dari pengamatan di lapangan. Semua populasi digunakan untuk menganalisis kemiripan dan struktur genetik serta penyebaran serbuk sari. Setiap pohon dewasa yang dievaluasi diberi nomor dengan spidol dan dicatat posisi koordinatnya menggunakan GPS. Penelitian ini terdiri atas dua tahapan yaitu tahapan analisis kemiripan genetik antar individu pada populasi tanaman dewasa serta populasi progeninya serta tahapan analisis pola penyebaran serbuk sari menggunakan marka molekuler SSR dan SNAP.
Penelitian kedua bertujuan untuk mengevaluasi pola penyebaran serbuk sari pada seluruh provenan menggunakan marka SSR dan SNAP. Marka SSR sebanyak empat dan marka SNAP satu digunakan untuk identifikasi genotipe 95 provenan dan 84 progeni yang dipanen dari 15 induk betina terpilih. Analisis parental menggunakan Perangkat lunak CERVUS berdasarkan data genotipe. Jarak antar pohon tetua jantan dan betina dihitung menggunakan data GPS setelah tetua jantan teridentifikasi. Jarak penyebaran serbuk sari terdekat adalah 0 m atau terjadi penyerbukan sendiri, sedangkan jarak terjauh adalah 61.8 m. Penyerbukan paling banyak terjadi pada jarak 0-10 m dengan persentase 33.3%.
Arah yang tidak beraturan antara pohon pendonor serbuk sari dengan penerima serbuk sari mengindikasikan bahwa penyerbukan dibantu oleh serangga polinator. Persentase penyerbukan silang induk betina kelapa Dalam kopyor, kelapa Genjah kopyor dan kelapa Hibrida kopyor berurutan adalah 100%, 72.73% dan 82.14%. Penyerbukan sendiri pada induk betina kelapa Dalam kopyor, kelapa Genjah kopyor dan kelapa Hibrida kopyor berurutan adalah 0%, 27.27% dan 17.86%. Pohon normal mendonorkan serbuk sari ke kelapa kopyor sebanyak 8.33%. Kehadiran pohon kelapa yang berbuah normal yang berada di sekitar pohon kelapa berbuah kopyor memiliki peluang untuk mengurangi produksi buah kopyor.
SUMMARY
RINI ISMAYANTI. Genetic Similarity and Pollen Dispersal Analysis on Pati Kopyor Coconut Population Based on SSR and SNAP Marker Analysis. Supervised by DEWI SUKMA and SUDARSONO.
Pollination of female flower of kopyor coconut by male pollen of normal coconut could result in reduced kopyor fruit yield. Therefore, investigating pattern of pollen dispersal in a mix population of kopyor and normal coconut provenances would be beneficial. Moreover, evaluating genetic similarity among the studied population and their progenies would also beneficial for predicting the quality of produced seed nuts. The objectives of this experiment were : (1) to evaluated genetic similarity of the kopyor coconut parents population and their progenies, (2) to evaluate pattern of pollen dispersal among mix population of kopyor and normal fruit producing coconut in Pati, Central Java, (3) evaluated relevancy of pollen dispersal among mix population with their kopyor fruit yield. There were 95 adult palms evaluated as male parent candidate and 84 seeds harvested from 15 selected female parents as progeny population. The female parents were selected based on their high productivity from field evaluation. All population was used for genetic structure and similarity analysis and also pollen dispersal. Every adult palms were numbered using a marker and noted for the coordinate position using GPS. This research consisted of two stages, the first analyzed of genetic similarity among individual of adult plants and their progenies, and the second analyzed the pollen dispersal using molecular markers SSR and SNAP.
The analyses of the genetic similarities were initiated with primer screening. Among 36 SSR loci tested, 32 loci resulted in a (+) product when they were used to amplify coconut DNA and only CnCir_B12, CnCir_86, CnCir_87 and CnCir_56 resulted in polymorphic SSR markers. Moreover, SNAP locus corresponding to position 14 of SNP of coconut sucrose synthase gene were also used. These loci were used to genotype candidate parents and progenies population. Results of the experiment indicated the average of allele per locus and average of PIC were 4.5 and 0.46, respectively. Cluster analysis using DICE coefficient based on molecular data resulted in an estimate of intra population of adult palms similarity and intra population of progenies were at least 18% and 43%, respectively. Intra population of progenies were more closely related than intra population of adult palms. Results of STRUCTURE analysis grouped most of the individuals into either Tall, Dwarf or Hybrid types, as they were suspected based on the morphology of the palms. However, a number of individuals grouped as Tall, and some as Dwarf based on their morphology, were actually identified as Hybrids based on alleles constitutions of the evaluated markers.
CERVUS software. The distances between male and female parents trees were calculated based on the GPS data after male parents identification. Results of the experiment indicated self-pollinations occurred in a number o sampled female parents since the distance of pollen dispersal was 0 m. Meanwhile, the furthest distance of pollen dispersal was 61.8 m. On the other hand the majority of pollination occurs at a distance of 0-10 m (33.3%).
In term of pollen dispersal, the position of donor pollen relative to the recipient one was random. This indicated that the coconut pollen dispersal in the regions was not transferred by wind but probably was by insect pollinators help. Outcrossing rate in Tall, Dwarf and Hybrid type of coconuts were calculated as 100%, 72.7% and 82.1%, respectively. Therefore, levels of self-pollination in Tall, Dwarf and Hybrid coconut were 0%, 27.3% and 17.9%. There was a normal coconut in the region donating its pollen to kopyor female parents at the level of 8.3%. The existence of normal coconut palms around the kopyor coconut one might possibly reduce the kopyor fruit yield.
.
© Hak Cipta Milik IPB, Tahun 2013
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Pemuliaan dan Bioteknologi Tanaman
KEMIRIPAN GENETIK DAN POLA PENYEBARAN SERBUK
SARI POPULASI KELAPA KOPYOR BERDASARKAN
ANALISIS MARKA SSR DAN SNAP
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2013
Judul Tesis : Kemiripan Genetik dan Pola Penyebaran Serbuk Sari Populasi Kelapa Kopyor Pati Berdasarkan Analisis Marka SSR dan SNAP Nama : Rini Ismayanti
NIM : A253100031
Disetujui oleh Komisi Pembimbing
Dr. Dewi Sukma, SP, M.Si Ketua
Prof. Dr. Ir. Sudarsono, M.Sc Anggota
Diketahui oleh
Ketua Program Studi
Pemuliaan dan Bioteknologi Tanaman
Dr. Ir. Trikoesoemaningtyas, M.Sc
Dekan Sekolah Pascasarjana
Dr. Ir. Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia, kekuatan, kemudahan serta rahmat-Nya sehingga penelitian dan penulisan tesis ini berhasil diselesaikan dengan baik. Tesis yang berjudul Kemiripan Genetik dan Pola Penyebaran Serbuk Sari Populasi Kelapa Kopyor Berdasarkan Analisis Marka SSR dan SNAP ini merupakan tugas akhir penulis untuk memperoleh gelar Magister Sains dari Program Studi Pemuliaan dan Bioteknologi Tanaman, Sekolah Pascasarjana Institut Pertanian Bogor.
Terima kasih penulis ucapkan kepada Dr. Dewi Sukma, SP., M.Si dan Prof. Dr. Ir. Sudarsono, M.Sc selaku pembimbing atas bimbingan, motivasi dan arahannya selama perencanaan, pelaksanaan serta penulisan tesis ini. Terima kasih kepada proyek penelitian KKP3T 2011 yang berjudul “Peningkatan Persentase Buah Kopyor (75%) Melalui Pemuliaan Tanaman dan Deteksi Dini Bibit Kelapa Kopyor dengan Marka Molekuler” di bawah koordinasi Prof. Dr. Ir. Sudarsono, M.Sc atas pendanaan dan bantuan fasilitas dalam pelaksanaan penelitian ini.
Terima kasih juga diucapkan kepada Bapak Ir. Ismail Maskromo, M.S atas bantuan, masukan, dan sharingnya selama penelitian. Terima kasih pada teman-teman Pascasarjana program studi Pemuliaan dan Bioteknologi Tanaman angkatan 2010 atas kekompakan, kerjasama serta dukungan dan semangatnya. Kepada rekan-rekan dan teknisi di Plant Molecular Biology Laboratorium (PMB Lab) yang banyak membantu dalam kegiatan penelitian ini juga diucapkan terima kasih. Tak lupa penulis ucapkan terima kasih setinggi-tingginya kepada kedua orang tua Bapak Dr. Mappaganggang Sodding Pabbage, MS dan Ibu dr.Isdiana Kaelan, Sp.Rad adik-adik tercinta Akhmad Setiadi dan Cynthia Balqis, serta keluarga besar atas semangat dan doanya sehingga pendidikan ini dapat diselesaikan dengan baik.
Mohon maaf atas segala kekurangan dan kesalahan dalam penulisan karya ilmiah ini. Semoga karya ilmiah ini bermanfaat bagi pengembangan ilmu pengetahuan. Khususnya bidang pemuliaan tanaman dan pertanian pada umumnya.
Bogor, Oktober 2013
DAFTAR ISI
DAFTAR TABEL xii
DAFTAR GAMBAR xii
DAFTAR LAMPIRAN xiii
1 PENDAHULUAN 1
Tujuan Penelitian 2
Manfaat Penelitian 2
2 TINJAUAN PUSTAKA 4
3 ANALISIS KEMIRIPAN GENETIK POPULASI KELAPA KOPYOR
KOPYOR PATI MENGGUNAKAN MARKA MOLEKULER
Abstrak 11
Pendahuluan 12
Bahan dan Metode 13
Hasil dan Pembahasan 17
Simpulan 28
4 ANALISIS PENYEBARAN SERBUK SARI KELAPA KOPYOR PATI MENGGUNAKAN MARKA SSR DAN SNAP
Abstrak 31
Pendahuluan 32
Bahan dan Metode 32
Hasil dan Pembahasan 35
Simpulan 44
5 PEMBAHASAN UMUM 47
6 SIMPULAN DAN SARAN
Simpulan 50
Saran 50
DAFTAR PUSTAKA 51
LAMPIRAN 59
DAFTAR TABEL
2.1 Perbandingan tampilan fisik dari beberapa sampel kelapa 6 3.1 Contoh skoring data biner dan data genotipe berdasarkan hasil
amplifikasi lokus CnCir_56 pada Gambar 3.1 16
3.2 Profil primer menggunakan marka SSR dan SNAP 19 3.3 Perbedaan data morfologi dan data molekuler terhadap tipe kelapa 27 4.1 Persentase penyerbukan sendiri dan penyerbukan silang kelapa
berbuah kopyor berdasarkan jenis induk betinanya 41 4.2 Rata-rata produktifitas buah kelapa kopyor yang digunakan sebagai
tetua betina di Pati pada bulan Juni 2011 43
4.3 Kontribusi serbuk sari dan persentase produksi buah kelapa kopyor 43
DAFTAR GAMBAR
1.1 Bagan alir kegiatan penelitian analisis penyebaran serbuk sari kelapa
kopyor Pati 3
2.1 Perbedaan fenotipik endosperma antara buah kelapa kopyor dan buah
kelapa normal 5
3.1 Perbedaan pola pita polimorfik pada lokus CnCir_56 dan pita
monomorfik pada lokus CnCir_K8 16
3.2 Pola pita DNA menggunakan primer SSR CnCir_56 (230–180 bp) 17 3.3 Pola pita DNA menggunakan primer SSR CnCir_86 (188-163bp) 17
3.4 Pola pita DNA menggunakan primer SSR CnCir_87 (158-170bp) 18
3.5 Pola pita DNA menggunakan primer SSR CnCir_B12 (151- 198 bp) 18
3.6 Visualisasi DNA hasil amplifikasi primer SNP pada posisi 14 gen
SUS1 18
3.7 Dendrogram kemiripan genetik pada 95 individu tanaman kelapa
dewasa pada populasi kelapa kopyor Pati 22
3.8 Dendrogram kemiripan genetik pada 84 bibit pada populasi progeni dari sejumlah induk betina kelapa berbuah kopyor 23
3.9 Analisis struktur populasi tanaman kelapa dewasa menggunakan
STRUCTURE dengan K=2 25
3.10 Analisis struktur populasi tanaman kelapa dewasa dan progeninya
menggunakan STRUCTURE dengan K=2 26
4.1 Peta sebaran pohon induk betina dan pohon kelapa dewasa lainnya
sebagai kandidat tetua jantan 36
4.2 Visualisasi primer CnCir_56 marka SSR pada induk betina dan
progeninya 37
4.3 Visualisasi primer situs SNP #14 dari fragmen gen SUS1 marka SNAP 37
4.4 Representasi pola penyerbukan pohon induk 67 39
4.5 Persentase penyerbukan yang terjadi berdasarkan jarak antara pohon
induk jantan dengan pohon induk betina 40
4.6 Tipe bibit kelapa berdasarkan jenis tetua jantan dan betina yang
DAFTAR LAMPIRAN
1 Pembuatan Larutan Stok untuk analisis molekuler 59
2 Daftar 36 primer SSR yang digunakan 61
3 Representasi induk jantan sebagai donor serbuk sari untuk dua progeni yang dianalisis pada pohon induk betina no.37 63 4 Representasi induk jantan sebagai donor serbuk sari untuk lima
progeni yang dianalisis pada pohon induk betina no.39 64 5 Representasi induk jantan sebagai donor serbuk sari untuk dua progeni
yang dianalisis pada pohon induk betina no.44 65 6 Representasi induk jantan sebagai donor serbuk sari untuk enam
progeni yang dianalisis pada pohon induk betina no. 51 66 7 Representasi induk jantan sebagai donor serbuk sari untuk dua progeni
yang dianalisis pada pohon induk betina no. 53 67 8 Representasi induk jantan sebagai donor serbuk sari untuk dua progeni
yang dianalisis pada pohon induk betina no. 58 68 9 Representasi induk jantan sebagai donor serbuk sari untuk tujuh
progeni yang dianalisis pada pohon induk betina no. 59 69 10 Representasi induk jantan sebagai donor serbuk sari untuk sembilan
progeni yang dianalisis pada pohon induk betina no.68 70 11 Representasi induk jantan sebagai donor serbuk sari untuk enam
progeni yang dianalisis pada pohon induk betina no.69 71 12 Representasi induk jantan sebagai donor serbuk sari untuk delapan
progeni yang dianalisis pada pohon induk betina no.84 72 13 Representasi induk jantan sebagai donor serbuk sari untuk enam
progeni yang dianalisis pada pohon induk betina no.85 73 14 Representasi induk jantan sebagai donor serbuk sari untuk enam
progeni yang dianalisis pada pohon induk betina no.88 74 15 Representasi induk jantan sebagai donor serbuk sari untuk tujuh
progeni yang dianalisis pada pohon induk betina no.89 75 16 Representasi induk jantan sebagai donor serbuk sari untuk dua progeni
1
PENDAHULUAN
Kelapa kopyor adalah kelapa mutan yang merupakan salah satu komoditas perkebunan Indonesia bernilai ekonomi tinggi. Ciri dari kelapa kopyor adalah memiliki endosperma bertekstur lunak dan tidak melekat pada tempurungnya (Santoso 1996). Sifat tersebut menyebabkan timbulnya suara khas apabila kelapa kopyor diguncang.
Kabupaten Pati, Jawa Tengah, merupakan sentra produksi kelapa kopyor terbaik di Indonesia karena di daerah tersebut telah tumbuh dan berkembang kelapa Genjah berbuah kopyor. Kementrian Pertanian Republik Indonesia pada tahun 2010 telah melepas tiga varietas unggul lokal kelapa Genjah kopyor Pati. Varietas tersebut adalah kelapa kopyor Genjah Hijau Pati, kopyor Genjah Kuning Pati dan kopyor Genjah Cokelat Pati (Maskromo et al. 2011a).
Permasalahan dalam pengembangan kelapa kopyor adalah penyediaan bibit kelapa kopyor yang berkualitas dan terjamin keasliannya dalam jumlah banyak. Hal ini terkait dengan belum tersedianya teknologi untuk membedakan bibit kelapa berbuah kopyor atau yang berbuah normal. Hasil observasi menunjukkan bahwa tidak ada perbedaan secara morfologi antara pohon kelapa berbuah normal dan pohon kelapa berbuah kopyor (Maskromo 2005).
Permasalahan lain adalah rendahnya kuantitas hasil buah kopyor yang dipanen. Akibatnya produksi buah kelapa kopyor masih belum dapat memenuhi kebutuhan konsumen. Pertanaman kelapa berbuah kopyor di Indonesia umumnya masih ditanam bersama dengan kelapa normal dari jenis kelapa Dalam, Genjah, dan Hibrida. Adanya kelapa berbuah normal di antara pertanaman kelapa berbuah kopyor diduga berpotensi mempengaruhi produktivitas buah kopyor yang dipanen (Sudarsono et al. 2012). Keberadaan pohon dewasa kelapa Dalam berbuah normal yang cenderung menyerbuk silang (Pandin 2009), diduga dapat berpengaruh negatif terhadap produksi buah kopyor. Pohon tersebut dapat menyebarkan serbuk sari pembawa sifat buah normal ke bunga betina pada pohon kelapa berbuah kopyor. Akibat dari penyerbukan seperti itu adalah diperolehnya buah kelapa normal (Sudarsono et al. 2012).
Evaluasi penyebaran serbuk sari (pollen dispersal) pada pertanaman kelapa kopyor perlu dilakukan berdasarkan berbagai hal tersebut. Arah penyebaran serbuk sari dapat memberikan informasi vektor apa yang membantu penyerbukan pada pohon kelapa kopyor. Jarak antara pohon tetua jantan dengan tetua betina juga dapat mengindikasikan vektor yang berperan dalam penyerbukan.
Penyebaran serbuk sari dapat dipelajari dengan metode pewarnaan serbuk sari (Blair dan Williamson 2010) atau menggunakan marka molekuler (Austerlitz
et al. 2004). Marka molekuler yang telah digunakan dalam analisis penyebaran serbuk sari adalah marka RAPD pada Ilex paraguariensis (Cansian et al. 2010) dan marka SSR (Single Sequence Repeat) pada tanaman Hymenaea courbaril
(Carneiro et al. 2011), tanaman pinus (Feng et al. 2010) dan kelapa (Pandin et al.
yang dapat menggandakan DNA target (Semagn et al. 2006). SNAP merupakan marka yang berbasis perbedaan basa nukleotida tunggal dari sekuens DNA pada umumnya. Marka SNAP memiliki keuggulan dibanding dengan marka SSR yaitu dapat mendeteksi variasi dalam sekuens DNA yang berkorelasi dengan perbedaan fenotipe tanaman (Manju dan Arunachalam 2011). Marka SNAP telah digunakan sebagai penanda genetik pada padi (Lestari 2013), jagung (Mammadov 2010) dan
Beta vulgaris (Mohring et al. 2004).
Tujuan Penelitian
Tujuan yang ingin dicapai dalam penelitian ini adalah :
1) Mendapatkan informasi mengenai kemiripan genetik individu-individu tanaman kelapa dalam satu populasi.
2) Memperoleh pola penyebaran serbuk sari pada kelapa berbuah kopyor dan kelapa berbuah normal dalam satu areal pertanaman berdasarkan data marka SSR dan SNAP.
3) Mengevaluasi keterkaitan antara penyebaran serbuk sari dengan produktivitas buah kopyor.
Manfaat Penelitian
TINJAUAN PUSTAKA
Morfologi Kelapa
Kelapa anggota familia Palmaceae atau Arecaceae, memiliki banyak arti penting dalam kehidupan manusia karena semua organ tanaman ini dapat dimanfaatkan (Novarianto 2008). Ciri-ciri pohon kelapa menurut Chan dan Elevitch (2006) adalah memiliki batang tunggal dan beruas dengan tinggi mencapai 30 m dan diameter kanopi 8-9 m. Akar berbentuk serabut, tebal, berkayu dan adaptif pada lahan berpasir pantai. Daun tersusun secara majemuk dan menyirip sejajar tunggal, pelepah terletak pada ibu tangkai daun, duduk pada batang (roset batang). Warna pada tangkai daun (petiole) mengindikasikan warna buah pada kelapa.Bunga kelapa merupakan bunga majemuk yang dilindungi oleh
spatha. Bunga jantan dan betina terdapat pada satu tangkai utama yang disebut
spadix, setiap spadix terdiri atas 40-60 cabang (spikelet) dengan ribuan bunga jantan. Letak bunga bunga betina terletak di pangkal, sedangkan bunga jantan di bagian atas bunga betina hingga ujung spikelet. Buah kelapa memiliki tiga lapisan, yaitu eksokarp (kulit tipis terluar yang memiliki lapisan lilin) berwarna kuning, hijau, jingga atau coklat, mesokarp berupa lapisan serat yang lebih tebal atau sering disebut sabut (husk), dan endokarp yang keras disebut batok (shell), yang melindungi biji. Endokarp dan biji hanya dipisahkan oleh membran yang melekat pada sisi dalam dari endokarp. Biji kelapa memiliki tiga mikrofil (micropyle) dan hanya satu yang mengindikasikan keberadaan embrio. Embrio kelapa berukuran kecil dan akan membesar ketika buah siap untuk berkecambah. Endosperma biji kelapa terdiri atas endosperma cair yang mengandung banyak enzim dan endosperma fase padat yang mengendap pada dinding endokarp ketika buah menua (kernel).
Tanaman kelapa menurut Maskromo et al (2007b) dibagi dalam dua tipe yaitu kelapa Dalam dan kelapa Genjah. Masing-masing tipe memiliki karakteristik yang berbeda. Kelapa Dalam memiliki ciri khas batang besar, mempunyai bol (pembengkakan) pada pangkal batang, mulai berbunga pada umur 5-7 tahun, buah berukuran cukup besar tetapi jumlahnya sedikit. Kelapa Genjah memiliki batang lebih kecil dibanding kelapa Dalam, tidak memiliki bol, mulai berbunga pada umur 3-4 tahun, buah berukuran lebih kecil dan banyak. Karakteristik kelapa Dalam dan kelapa Genjah juga dimiliki oleh kelapa kopyor.
Kelapa Kopyor
Hasil penelitian Maskromo (2005) menyatakan bahwa kelapa berbuah kopyor dari segi morfologi sama dengan tanaman kelapa lainnya, yang membedakan adalah bagian endospermanya (Gambar 2.1). Maskromo et al.
kopyor akan menghasilkan bunyi yang kurang nyaring dibanding kelapa normal, karena sebagian atau seluruh endosperma fase padatnya sudah lepas dari tempurungnya. Buah kopyor juga dapat diidentifikasi dengan ketukan, tetapi memerlukan keterampilan khusus untuk dapat melakukannya. Tukang ketuk kelapa yang sudah ahli dalam identifikasi buah kopyor disebut “tukang totok”. Tingkat akurasi penentuan buah kopyornya dapat mencapai 99% (Sudarsono et al.
2012). Buah dengan sifat kopyor dihasilkan dari pohon kelapa tertentu yang sebagian besar buahnya mempunyai endosperma normal dan sebagian kecil abnormal (kopyor) (Wahyuni 2000). Pohon kelapa kopyor hanya mempunyai buah kelapa kopyor dengan frekuensi antara 3-4 buah kopyor per tandan.
Abnormalitas endosperma kelapa kopyor bersifat genetik dan dikendalikan oleh gen mutan resesif (Santos 1999). Sifat kopyor dibawa oleh pasangan alel resesif, yaitu 50% dari induk betina dan 50% dari induk jantan. Buah kopyor akan terbentuk jika terjadi penyerbukan antara polen dan stigma yang masing-masing membawa alel resesif penentu sifat kopyor. Sifat kopyor secara genetik ditentukan oleh kontribusi genetik endosperma (induk betina) dan serbuk sari (induk jantan) (Tahardi 1997).
Gambar 2.1 Perbedaan fenotipik endosperma antara (a) buah kelapa kopyor dan (b) buah kelapa normal. Ea=endosperma abnormal pada buah kelapa kopyor. En=endosperma normal pada buah kelapa normal.
A
B
Tabel 2.1. Perbandingan tampilan fisik dari beberapa sampel kelapa
Perbandingan berat buah, diameter buah, volume air dan ketebalan endosperma pada buah kelapa normal dengan buah kelapa kopyor dapat dilihat pada Tabel 2.1. Perbedaan tidak hanya terjadi pada fenotipe endosperma antara kelapa kopyor dengan kelapa normal, tetapi juga pada senyawa-senyawa kimia yang dikandungnya. Air kelapa kopyor memiliki kandungan sukrosa yang tinggi (60.8%), sedangkan pada air kelapa muda normal hanya memiliki glukosa dan fruktosa sebagai gula utamanya. Vitamin B dan vitamin C pada endosperma kelapa kopyor lebih tinggi dibanding pada endosperma kelapa normal yang tua, tetapi lebih rendah dibanding endosperma kelapa normal yang masih muda. Asam amino total pada air kelapa kopyor lebih tinggi dibanding dengan kelapa normal (Santoso et al. 1996).
Sistem Penyerbukan Kelapa
Penyerbukan atau polinasi adalah jatuhnya serbuk sari dari kotak sari (antera) ke kepala putik (stigma) dalam satu bunga atau bunga yang berbeda. Penyerbukan tumbuhan dapat terjadi secara biotik dan abiotik. Penyerbukan biotik terjadi dengan bantuan hewan, sedangkan penyerbukan abiotik terjadi dengan bantuan angin, air dan gravitasi (Liverdi 2008). Jarak penyebaran serbuk sari pada tanaman yang menyerbuk sendiri (autogamy) lebih rendah dibandingkan dengan tanaman menyerbuk silang (Boer 2007).
Kelapa Dalam pada umumnya merupakan tanaman menyerbuk silang, oleh karena itu tampilannya sangat beragam (Pandin 2009a). Kelapa Dalam memiliki bunga jantan yang matang lebih dulu dibanding bunga betina. Bunga betina siap diserbuki ketika bunga jantan umumnya sudah rontok sehinga terjadi penyerbukan silang (Heliyanto, 2010). Kelapa Genjah pada umumnya memiliki pola penyerbukan sendiri meskipun masih memungkinkan terjadinya penyerbukan silang sehingga menyebabkan tingginya tingkat kemiripan genetik pada kelapa Genjah (Hannum et al. 2003). Bunga betina dan bunga jantan pada kelapa Genjah masak secara bersamaan sehingga peluang untuk menyerbuk sendiri sangat besar (Heliyanto, 2010).
Penelitian Ramirez et al. (2004) menyatakan sebanyak 59% penyerbukan kelapa dibantu oleh serangga lebah madu. Lebah membantu proses penyerbukan silang, sehingga meningkatkan produktivitas tanaman budidaya. Potensi ini dimanfaatkan dengan cara meletakkan koloni lebah pada areal tanaman budidaya yang daya serbuknya rendah. Perpindahan lebah dari satu bunga ke bunga yang lain mempercepat proses polinasi karena serbuk sari banyak menempel pada kaki dan perut dari lebah (Liferdi 2008).
Penanda Genetik
Penanda (marka) genetik dikenal ada tiga macam, yaitu marka morfologi, marka biokimia (isozim), dan marka DNA (molekuler) (Liu dan Wu 1998). Penanda biokimia (isozim) pada kelapa telah digunakan untuk studi keragaman (Novarianto dan Hartana 1995). Keterbatasan dari marka-marka biokimia dan morfologi terbatas dalam jumlah dan dipengaruhi oleh faktor-faktor lingkungan atau fase perkembangan dari tanaman (Iriani 2011).
Penanda molekuler pertama tanpa aplikasi PCR mulai diperkenalkan sejak tahun 1980-an, yaitu RFLP (Restriction Fragment Length Polymorphism) (Schulman 2007). Setelah itu muncul penanda yang menggunakan aplikasi PCR, yaitu RAPD (Random Amplified Polymorphic DNA). Marka RAPD memiliki kekurangan yaitu tidak dapat membedakan lokus heterozigot dengan homozigot. Setelah itu berkembang mikrosatelit atau SSR (Simple Sequence Repeat) pada tahun 1990 (Semagn, 2006). Sebuah penanda yang ideal seharusnya memiliki tingkat polimorfisme yang tinggi sehingga dapat digunakan untuk mengukur keragaman genetik dan tidak dipengaruhi oleh faktor lingkungan; marka tersebar merata di seluruh genom; marka dapat mendeteksi perbedaan nukleotida dan dapat diwariskan dari tetua ke progeninya (Lowe et al. 2005).
Semagn (2006) menyatakan bahwa penanda molekuler secara garis besar dibagi menjadi tiga kelompok berdasarkan metode deteksinya, yaitu (i) Marka berbasis hibridisasi seperti RFLP, (ii) Marka berbasis PCR, seperti RAPD, AFLP, ISSR, SSR, dan (iii) Marka berbasis sekuens DNA seperti SNP.
2006). Identifikasi perbedaan individu tanaman dapat menggunakan marka molekuler untuk perlindungan kultivar tanaman (Pabendon 2007).
Simple sequence repeats (Mikrosatelit)
Pengulangan ruas basa DNA diklasifikasikan berdasarkan panjang dan jumlah ruas berulang di dalam genom yang dapat berupa : (1) DNA Satelit, adalah DNA yang memiliki pengulangan sangat tinggi biasanya antara 1000–100.000 kopi, sering berada pada bagian heterokromatin; (2) Minisatelit, memiliki pengulangan yang lebih sedikit yaitu 10–60 pasang basa; (3) Mikrosatelit (SSR), disebut juga fragmen berulang sederhana atau fragmen berulang sederhana, memiliki pengulangan lebih pendek pada 1-6 pasang basa, terdistribusi lebih banyak pada lokus genom; (4) Midisatelit, memiliki ruas berulang yang merupakan kombinasi dari satelit dan minisatelit (Pandin 2009b).
Simple sequence repeats juga dikenal dengan mikrosatelit terdiri atas pengulangan beberapa basa nukleotida, berupa dinukleotida, trinukleotida, atau tetranukleotida, yang tersebar disepanjang genom kebanyakan spesies eukariotik (Powell 1996). Jumlah pengulangan nukleotida berkisar antara 5-40 kali (Selkoe dan Toonen 2006) atau kurang dari 100 kali (Karp et al. 1997). Panjang pengulangan ini bervariasi tergantung individu/varietas dan diwariskan kepada generasi berikutnya. Motif pengulangan nukleotida yang paling banyak ditemukan pada manusia adalah AC atau TC, sedangkan pada tanaman adalah AT, AG, dan TC (Powell 1996).
Kelebihan marka mikrosatelit adalah jumlahnya yang banyak di dalam genom tanaman, bersifat polimorfik, mudah dideteksi dengan PCR, waktu deteksi yang dibutuhkan singkat, bersifat co-dominan, dan membutuhkan DNA dalam jumlah sedikit. Marka mikrosatelit mampu membedakan individu yang heterozigot maupun dan homozigot (Wright dan Benzen 1994).
Ekstraksi DNA merupakan langkah awal dalam melakukan analisis marka molekuler. Tahap kegiatan berikutnya dilanjutkan dengan amplifikasi PCR. Amplifikasi PCR pada SSR menggunakan primer forward dan reverse khusus yang akan menempel pada suhu annealing tertentu pada template DNA. Fragmen hasil PCR kemudian dapat dipisahkan dengan teknik elektroforesis menggunakan gel poliakrilamid. Deteksi DNA dilakukan dengan pewarnaan silver atau dengan menggunakan sistem deteksi fluorescent. Gel poliakrilamid memiliki resolusi yang lebih tinggi dibandingkan dengan gel agarose (Semagn 2006).
Single nucleotide amplified polymorphism (SNAP)
SNP yang melimpah membuat marka SNAP lebih menarik dibanding marka lainnya, termasuk dalam mengembangkan penanda bagi gen target tertentu (Lestari dan Koh 2013).
Deteksi marka SNAP yang bersifat ko-dominan, berdasarkan pada amplifikasi PCR dengan primer yang berbasis pada informasi sekuen untuk gen spesifik. Keunggulan teknik SNAP adalah lebih mudah dan lebih hemat waktu dibandingkan dengan teknik SSR (Yang et al. 2011). Marka SNAP juga memiliki tingkat kesalahan yang lebih rendah dibandingkan dengan marka SSR. Marka SNAP saat ini telah digunakan sebagai penanda genetik untuk berbagai fungsi pemuliaan tanaman, misalnya analisis keragaman genetik, pembuatan linkage map, dan Marker Assisted Selection (Chen et al. 2011). Kelemahan dari teknik SNAP adalah memerlukan informasi keragaman sekuen untuk suatu gen yang menjadi target analisis (Mammadov et al. 2012).
Makapuno adalah salah satu kelapa unik yang juga memiliki endosperma yang abnormal. Endosperma makapuno lebih tebal dan lembut serta fase cairnya sangat kental sepeti oli. Hal tersebut disebabkan oleh terdegradasinya galaktomanan oleh α-D-galaktosidase (Samonthe 1988). Sintesis sukrosa sejalan dengan proses degradasi galaktomanan menjadi manosa dan galaktosa, sehinga enzim sucrose synthase (SUS) diduga mempengaruhi morfologi endosperma pada kelapa kopyor. Keragaman DNA dari gen SUS dapat digunakan untuk menghasilkan marka SNAP dan dievaluasi untuk menduga keragaman genetik pada kelapa kopyor (Sukendah 2007).
Penyebaran Serbuk Sari
Aliran gen atau gene flow adalah proses transfer informasi genetik melalui penyebaran serbuk sari (penyebaran gamet jantan) dan melalui penyebaran benih (migrasi) (Mallet 2001). Aliran gen merupakan proses yang alami yang terjadi pada tanaman yang menyebabkan gen-gen dalam tanaman berpindah. Proses aliran gen dapat terjadi pada tanaman yang memiliki keserasian secara seksual antara tanaman domestik maupun kerabat liarnya (Pandin 2009). Analisis aliran gen melalui serbuk sari dalam suatu populasi dapat digunakan untuk menduga apakah terjadi perkawinan antara tanaman yang berbeda (outcrossing) atau dengan tanaman yang sama (selfing) (Boer, 2007).
Hamrick dan Trapnell (2011) mengatakan bahwa pola penyebaran biji dapat dianalisis menggunakan dua metode, yaitu :
a. Metode tak langsung meliputi analisis struktur genetik populasi menggunakan marka genetik yang diwariskan secara maternal misalnya menggunakan cpDNA (DNA kloroplas) dan mtDNA (DNA mitokondria) dalam satu populasi.
Sistem perkawinan pada tanaman dapat diketahui melalui analisis pola penyebaran serbuk sari. Penelitian Carneiro et al. (2011) menyatakan bahwa tanaman Hymenaea coubaril melakukan penyerbukan sendiri. Hal tersebut bertentangan dengan penelitian sebelumnya oleh Dunphy et al. (2004) yang menyatakan bahwa H. coubaril memiliki ketidaksesuaian secara seksual (self incompability). Penebangan pohon H. coubaril secara bebas dalam areal perhutanan dapat mengakibatkan berkurangnya pohon yang reproduktif. Kondisi tersebut mengakibatkan tanaman terisolasi, sehingga persentase penyerbukan sendiri dapat meningkat (Carneiro et al. 2011).
11
ANALISIS KEMIRIPAN GENETIK POPULASI KELAPA
KOPYOR PATI MENGGUNAKAN MARKA MOLEKULER
Abstract
Kopyor coconuts is an exotic coconut originated from Indonesia. Pati, Central Java is one of the kopyor coconut producing areas in Indonesia. This research was conducted to evaluate genetic similarity among accessions of kopyor and normal coconut provenances existed in the region. Four SSR loci and one SNAP locus (SNP #14 of SUS1) were selected to access genetic variability among provenances after evaluating 36 SSR and 4 SNP loci. The materials evaluated include 95 accessions of a mixture of normal and kopyor coconut producing adult palms, consisted of Tall, Dwarf and Hybrid types and their 84 progenies. It harvested from 15 randomly selected female parents from the same gardens. Results of the experiment indicated the average of allele per locus and PIC number was 4.5 and 0.46, respectively. Cluster analysis using UPGMA based on molecular data resulted in an estimate of female similarity intra adult population and intra progenies population were at least 18% and 43%, respectively. That intrapopulation of progenies were more closely related than intrapopulation of adult palms. Results of STRUCTURE analysis were grouped most of the individuals into either Tall, Dwarf or Hybrid types, as they were suspected based on the morphology of the palms. However, a number of individuals grouped as Tall, and some as Dwarf based on their morphology, were actually identified as Hybrids based on alleles constitutions of the evaluated markers.
Pendahuluan
Kelapa di Indonesia memiliki beberapa keunikan yang menjadi daya tarik tersendiri. Kelapa eksotik yang ada di Indonesia antara lain adalah kelapa lilin yang endospermanya agak kenyal, kelapa kenari yang endospermanya renyah dan manis, kelapa hijau yang sabutnya berwarna merah muda, serta kelapa kopyor yang memiliki endosperma yang terlepas dari tempurungnya (Maskromo 2013).
Jumlah tanaman kelapa kopyor sangat terbatas karena merupakan hasil mutasi alami (Maskromo 2005). Keterbatasan jumlah serta tingginya permintaan masyarakat meningkatkan harga jual buah kelapa kopyor. Tanaman kelapa kopyor dilaporkan ditemukan di daerah tertentu seperti Pati, Sumenep, Jember, Banyuwangi (Sukendah 2009), Tangerang, Ciomas, dan Kecamatan Kalianda Lampung Selatan (Maskromo 2005).
Buah kelapa mutan dengan fenotipik endosperma abnormal juga dilaporkan di beberapa negara. Di Filipina dikenal kelapa mutan dengan nama macapuno dan di Thailand dengan nama dikiri (Maskromo 2005). Kelapa tersebut memiliki endosperma bertekstur menyerupai kelapa lilin dan rasanya kurang manis sehingga kurang diminati oleh konsumen. Kelapa kopyor memiliki perbedaan pada endospermanya yang lembut serta rasa yang manis, sehingga kelapa kopyor lebih disukai. Kelapa kopyor menjadi buah eksklusif dengan harga jual di pedagang pengumpul berkisar antara Rp.20.000–Rp.30.000/buah sedangkan di pasar swalayan bisa mencapai Rp.50.000/buah.
Kelapa dibagi menjadi tiga tipe, yaitu kelapa Dalam, kelapa Genjah dan kelapa Hibrida. Ciri kelapa Dalam adalah memiliki tinggi di atas 15 m dan bagian pangkal batang membesar (bole). Kelapa tipe Genjah pada umumnya memiliki batang pendek berkisar 12 meter dan agak kecil, tidak memiliki bole (Pandin 2009). Kelapa Hibrida merupakan gabungan sifat antara kelapa Genjah dengan kelapa Dalam.
Jenis kelapa Genjah maupun kelapa Dalam berpeluang untuk menghasilkan buah kopyor (Maskromo et al. 2007). Populasi kelapa penghasil buah kopyor di Pati sangat beragam jenisnya mulai dari kelapa Genjah berbuah kopyor, kelapa Dalam berbuah kopyor, bahkan terdapat kelapa Hibrida alami yang berbuah kopyor (Sudarsono et al. 2012). Kelapa penghasil buah kopyor yang dikembangkan di beberapa daerah umumnya berjenis kelapa Dalam yang memiliki persentase produksi buah kopyor yang rendah 2-10% (Maskromo dan Novarianto2007). Kelapa Genjah penghasil buah kopyor telah berkembang sejak puluhan tahun yang lalu di Kabupaten Pati (Maskromo et al. 2011a).
Marka molekuler adalah suatu penanda berbasis DNA yang bermanfaat dalam mengidentifikasi perbedaan tanaman secara individu melalui profil unik secara alelik. Profil dan similaritas genetik setiap genotipe dapat dilakukan langsung melalui analisis DNA (Azrai 2006). Marka SSR menarik dikembangkan khususnya pada spesies yang menunjukkan variasi genetik rendah, pada populasi
inbred dan populasi yang diperoleh dari daerah-daerah berdekatan sehingga sulit dipilah-pilah dengan pendekatan lain (Saptahadi et al. 2011).
penelitian ini adalah (1) Menganalisis kemiripan genetik pada populasi kelapa berbuah kopyor di Pati, Jawa Tengah serta populasi progeninya menggunakan marka SSR dan SNAP. (2) Menganalisis struktur genetik kelapa tipe Dalam, kelapa tipe Genjah, dan kelapa tipe Hibrida.
Bahan dan Metode
Waktu dan Tempat Penelitian
Penelitian dilaksanakan mulai bulan Oktober 2011 hingga Januari 2013. Sampel tanaman diambil pada bulan Juli 2011di Desa Sambiroto, Kabupaten Pati, Jawa Tengah. Kegiatan penelitian dilakukan di Laboratorium Biologi Molekuler Tanaman (PMB Lab) Departemen Agronomi dan Hortikultura Fakultas Pertanian Institut Pertanian Bogor.
Bahan Tanaman
Materi genetik penelitian ini menggunakan dua populasi, yaitu populasi kandidat tetua dan populasi progeni. Populasi kandidat tetua sebanyak 95 aksesi terdiri atas 66 pohon kelapa tipe Genjah, 18 pohon kelapa tipe Dalam dan 11 pohon kelapa tipe Hibrida. Populasi progeni sebanyak 84 individu yang merupakan hasil panen buah kelapa kopyor dan non kopyor dari 15 pohon induk betina terpilih di lokasi yang sama.
Anak daun pada setiap pohon diambil sebanyak dua helai kemudian dipotong-potong kurang lebih sepanjang 10 cm. Potongan daun lalu dibungkus dengan aluminium foil dan diberi label sesuai dengan nomor pohon asal daun tersebut. Daun yang telah dibungkus disimpan di dalam kotak plastik pada tempat yang lembab selama tiga hari. Sampel daun disimpan di dalam freezer pada suhu -20 °C setelah berada di laboratorium dan siap dilakukan isolasi DNA.
Buah kelapa normal yang dipanen dikecambahkan di lapang untuk ekstraksi DNA. Daun muda diambil dari bibit berumur 3 bulan setelah perkecambahan. Isolasi DNA pada buah kelapa kopyor diambil dari embrionya.
Isolasi DNA
DNA diisolasi menggunakan CTAB mengikuti metode Rohde et al. (1995) dengan beberapa modifikasi. Daun kelapa muda dipotong kurang lebih seberat 0.3-0.4 g kemudian digerus dengan buffer lisis sebanyak 2 ml, PVP 0.007 g dan 2-mercaptoetanol sebanyak 10 µl. Hasil gerusan daun kemudian diinkubasi dalam
Supernatan dipindah ke tube eppendorf baru kemudian ditambahkan isopropanol dingin sebanyak 0.8 volume dari supernatan dan natrium asetat sebanyak 0.1 volume supernatant kemudian diinkubasi dalam freezer semalaman. Suspensi kemudian disentrifugasi hingga diperoleh pellet DNA. Pellet DNA dicuci dengan menggunakan ethanol 70% dingin 500 μl, lalu disentrifugasi dan dikeringkan. Pellet DNA diencerkan menggunakan aquabidest sebagai suspensi DNA.
Purifikasi DNA dilakukan untuk menghilangkan kontaminan RNA. RNase sebanyak 3 μl ditambahkan ke dalam suspensi DNA kemudian diinkubasi selama 1 jam pada suhu 37 0C. Suspensi DNA ditambahkan dengan satu volume fenol. dan campuran kloroform:isoamilalkohol (24:1). Suspensi DNA dipresipitasi dengan 0.8 volume isopropanol dan 0.1 volume natrium asetat lalu dibilas dengan alkohol 70% dingin. Pelet DNA kering diberi 300 µl aquabidest dan disimpan dapat diestimasi. Gel agarose diwarnai dengan ethidium bromide selanjutnya diamati dengan UV transiluminator Vilber Lourmat dan dokumentasi dengan kamera digital.
Amplifikasi PCR (Polymerase Chain Reaction)
Amplifikasi menggunakan mesin PCR Perkin Elmer GeneAmp PCR System 2400 dan PCR kit KAPA 2G FAST dengan total reaksi 25 µl. Mix PCR merupakan resep campuran untuk satu reaksi yaitu PCR buffer 5X 5 µl, MgCl2 25 mM 0.5 µl, dNTP 10 mM 0.5 µl, satu unit Taq polymerase 0.01 µl, dan
aquabidest 13.5 µl. Primer forward dan reverse untuk satu reaksi sebanyak 1.5 µl ditambahkan ke dalam mix PCR. DNA working solution sebanyak 4 µl disiapkan. Tahapan PCR dimulai dengan denaturasi awal 95 ºC selama 3 menit, tahap denaturasi 95 ºC selama 15 detik, tahap annealing 51-55 ºC selama 15 detik (suhu yang berbeda untuk tiap primer), tahap elongasi 72 ºC selama 1 detik, dan dilakukan pengulangan siklus-siklus tersebut sebanyak 35 kali. Tahap elongasi terakhir pada suhu 72 ºC selama 10 menit. Hasil PCR bisa disimpan pada suhu 4 ºC atau -21 °C untuk pemakaian dalam jangka waktu yang lama. Produk PCR dikonfirmasi dengan gel agarose 1% dalam buffer TBE 0.5X pada tegangan 80 V selama 30 menit yang dielektroforesis dengan Cole Parmer®.
PAGE (Polyacrilamid Gel Electroforesis)
Elektroforesis vertikal menggunakan alat Cole-Parmer® Dedicated Height Sequencers.
Setiap produk PCR dicampur dengan loading dye kemudian didenaturasi selama 10 menit kemudian diletakkan dalam es yang telah dihancurkan. Pre run
dilakukan pada 100 watt selama 30 menit. Elektroforesis dilakukan pada 60 watt selama 90 menit dan sebagai pembanding digunakan ladder 100 bp.
Deteksi fragmen DNA menggunakan silver staining
Pewarnaan silver mengikuti metode Creste et al. (2001) dengan modifikasi. Proses pewarnaan memiliki lima tahapan. Pertama adalah tahap fiksasi gel selama 10 menit. Plate dibilas dengan aquadest selama 1 menit. Kedua adalah tahap nitrit acid selama 3 menit, kemudian dibilas dengan aquadest selama 1 menit. Ketiga adalah tahap pewarnaan perak nitrat selama 20 menit, ketika proses staining akan berakhir, larutan developing ditambahkan 1.5 ml formaldehid dan 200 µl sodium thiosulfate. Plate dicuci dengan aquadest secara cepat, sekitar 5-10 detik. Keempat adalah tahap developer selama 5-7 menit hingga pita pada kaca muncul. Kelima adalah tahap reaction stop selama 5 menit, kemudian dicuci selama 5 menit di dalam 1 L aquadest. Plate dikeringkan pada suhu ruang dengan posisi tegak semalaman hingga benar-benar kering. Visualisasi akhir dan pemberian skor dilakukan di atas light table.
Seleksi primer dan identifikasi genotipe tetua dan progeni
Seleksi primer dilakukan untuk memperoleh primer-primer yang polimorfik pada kelapa. Contoh perbedaan pola pita monomorfik dan polimorfik dapat dilihat pada Gambar 3.1. Primer SSR yang polimorfik diseleksi dari 36 primer (Lebrun et al. 2001) pada DNA dari 6 genotipe yang dipilih acak. Dua genotipe mewakili kelapa Genjah, dua genotipe mewakili kelapa Dalam dan dua genotipe mewakili kelapa Hibrida. Primer-primer yang terpilih digunakan untuk mengidentifikasi genotipe pada populasi kandidat tetua sejumlah 95 genotipe serta populasi progeni berjumlah 84 genotipe. Marka SNAP untuk situs SNP #14 dari fragmen gen SUS1
(sucrose synthase).
Analisis data
Gambar 3.1 Perbedaan pola pita polimorfik pada lokus CnCir_56 yang memiliki empat alel (a) dan pita monomorfik pada lokus CnCir_K8 yang
Data biner digunakan untuk menganalisis kemiripan genetik menggunakan perangkat lunak komputer Numerical Taxonomy and Multivariate System
NTSYSpc 2.02 (Rohlf 1995). Output yang dihasilkan adalah dendogram pengelompokan atau clustering kemiripan genetik berdasarkan koefisien DICE yaitu F
,
dimana F adalah nilai kemiripan antara individu a dan b; Nab adalah jumlah pita yang sama posisinya antara individu a dan b; Na dan Nb adalah jumlah pita pada masing-masing individu a dan b.Data genotipe digunakan dalam analisis perangkat lunak komputer CERVUS 2.0, POPGENE versi 1.31, dan STRUCTURE. Nilai PIC (Polymorphic Information Content), Ho (observed heterozigosity) dan He (expectedheterozigosity) diperoleh dari analisis perangkat lunak komputer CERVUS 2.0 (Marshal et al. 1998). Nilai Na (number of allele) dan Ne (effective of allele) diperoleh dari analisis perangkat lunak POPGENE versi 1.31 (Yeh et al 1999). Perangkat lunak STRUCTURE (Pritchard et al. 2000) digunakan untuk mengamati struktur genetik tiap individu pada kedua populasi. Running STRUCTURE menggunakan 10 000 Length of burn periode, 100 000 MCMC (Monte Carlo Montev Chain) dan 20 iterations. Penentuan nilai K (pengelompokan) terbaik mengikuti metode Evano et al. (2005). Penentuan nilai
K terbaik juga bisa dilakukan secara online pada situs taylor0.biology.ucla.edu/structureHarvester/.
Hasil dan Pembahasan
Seleksi Primer dan Identifikasi Genotipe
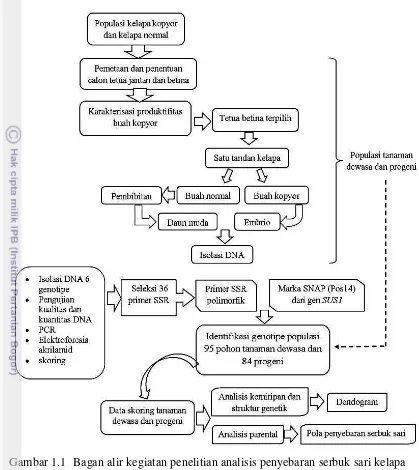
Kegiatan seleksi primer dilakukan untuk memperoleh primer-primer yang dapat mengamplifikasi DNA dan polimorfik terhadap populasi dan progeni yang digunakan. Hasil penelitian menunjukkan dari 36 primer SSR yang digunakan (Lampiran 2) pada tahap seleksi primer menggunakan enam genotipe hanya ada 32 primer yang dapat mengamplifikasi dan 4 primer diantaranya yang polimorfik.
Gambar 3.2 Pola pita DNA menggunakan primer SSR CnCir_56 (230–180 bp). M=marka Ladder 100 bp (100, 200)
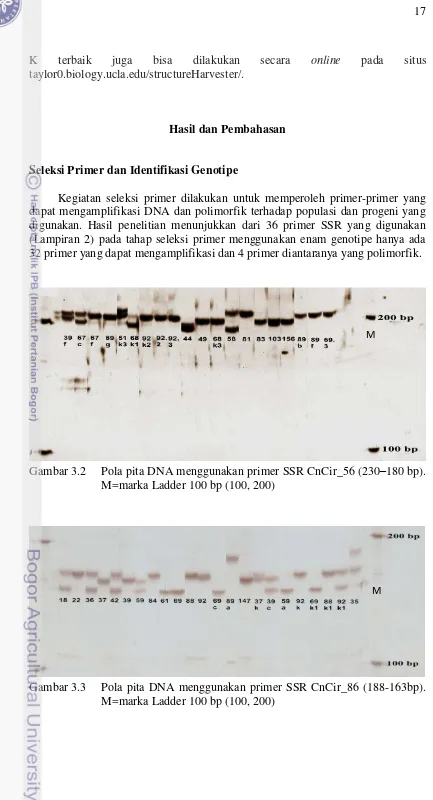
Gambar 3.3 Pola pita DNA menggunakan primer SSR CnCir_86 (188-163bp). M=marka Ladder 100 bp (100, 200)
M
Gambar 3.4 Pola pita DNA menggunakan primer SSR CnCir_87 (158-170bp). M=marka Ladder 100 bp (100, 200)
Gambar 3.5 Pola pita DNA menggunakan primer SSR CnCir_B12 (151- 198bp). M=marka Ladder 100 bp (100, 200)
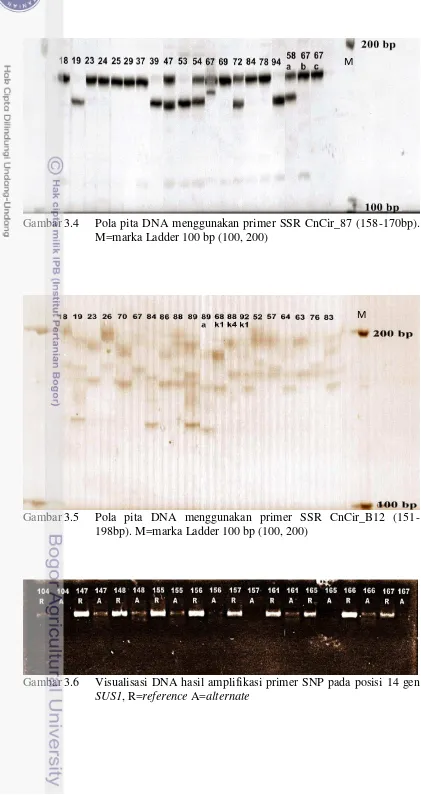
Gambar 3.6 Visualisasi DNA hasil amplifikasi primer SNP pada posisi 14 gen
SUS1, R=reference A=alternate
M
Tabel 3.2 Profil primer menggunakan marka SSR dan SNAP
Keterangan : PIC = Polymorphic Information Content , Ta = Temperature annealing
Ho = Observed heterozigosity, He = Expected heterozigosity, Na =
number of alleles, Ne = effective of alleles, Y = G/C
Profil keempat lokus SSR yang polimorfik serta marka SNAP dapat dilihat pada Tabel 3.2. Penelitian ini menghasilkan empat primer yang polimorfik meskipun menggunakan primer-primer yang telah digunakan sebelumnya (Lebrun
et al. 2001). Misalnya pada lokus CnCir_H4 yang digunakan pada penelitian Noel
et al. (2011) memperoleh 6 alel, penelitian Martinez et al. (2009) dan Ribeiro et al. (2010) memperoleh 3 alel, tetapi untuk populasi kelapa kopyor di Pati lokus tersebut menunjukkan pita monomorfik. Lokus CnCir_B12 dalam kasus ini memiliki jumlah alel yang lebih sedikit dibanding penelitian Martinez et al.
(2009) yang memperoleh 8 alel, Ribeiro et al. (2010) dan Shalini et al. (2007) memperoleh 9 alel, Rajesh et al. (2008) memperoleh 13 alel, sedangkan pada penelitian ini hanya diperoleh 6 alel. Rata-rata jumlah alel per lokus untuk marka SSR adalah 4.5 dengan kisaran jumlah alel 4-6. Jumlah rata-rata alel per lokus hampir sama dengan yang diperoleh oleh Konan et al. (2007) yaitu 4.83 alel per lokus yang menggunakan 21 aksesi kelapa dan 13 lokus SSR.
Hal ini disebabkan karena populasi yang digunakan pada penelitian sebelumnya menggunakan wild tipe hasil eksplorasi dari berbagai wilayah dan menggunakan populasi kelapa Dalam. Populasi yang digunakan dalam penelitian ini adalah populasi yang memiliki hubungan kekerabatan, sehingga sulit untuk menemukan primer yang polimorfik atau dapat membedakan antar genotipe. Primer yang polimorfis dibutuhkan untuk dapat menganalisis keragaman genetik populasi tanaman dengan memperlihatkan keragaman pola pita yang terbentuk (Chakravarthi dan Naravaneni 2006). Pola pita polimorfis dapat terjadi sebagai akibat dari perbedaan pengulangan basa dari tiap individu (Hildebrand et al.
1992).
dalam analisisnya (Sajib et al. 2012). Sardou et al. (2011) mengatakan bahwa PIC yang tinggi mengindikasikan kemampuan membedakan individu juga tinggi. Primer CnCir_B12 memiliki kemampuan yang tinggi untuk membedakan setiap genotipe dalam populasi, sedangkan primer CnCir_87 kemampuan membedakan genotipenya pada populasi ini kurang. Nilai PIC berkisar antara 0 hingga 1. Nilai 0 artinya pada lokus tersebut memiliki hanya satu alel (monomorfik) sedangkan nilai PIC 1 artinya lokus tersebut memiliki alel yang tidak terbatas (Hildebrand et al. 1992).
Lokus CnCir_B12 memiliki nilai He tertinggi (0.63) dan CnCir_87 terendah (0.36). Nilai He yang tinggi mengindikasikan bahwa pada lokus tersebut memiliki tingkat keragaman yang tinggi (Boer 2007). Rata-rata nilai He adalah 0.52. Nilai He yang tinggi pada CnCir_B12 disebabkan karena banyak genotipe pada lokus tersebut yang bersifat heterozigot, sedangkan primer CnCir_87 kebanyakan menghasilkan satu alel per genotipe (homozigot). Nilai He selalu lebih tinggi dibandingkan nilai PIC.
Nilai rata-rata PIC dan He tergolong sedang untuk populasi kelapa kopyor di Pati menggunakan analisis lima lokus menunjukkan bahwa kurangnya keragaman genetik pada populasi tersebut. Hal tersebut didukung dengan sulitnya memperoleh primer yang polimorfik. Semakin rendah tingkat polimorfis, maka tingkat keragamannya juga semakin rendah. Hal ini sejalan dengan informasi dari petani yang menanam buah (progeni) di lokasi yang berdekatan dengan pohon induknya, sehingga terbentuk suatu struktur famili dalam populasi. Bibit kelapa yang ditanam merupakan campuran bibit kelapa berbuah kopyor dan bibit kelapa berbuah normal, karena masih sulit membedakan bibit kelapa normal dengan kelapa kopyor.
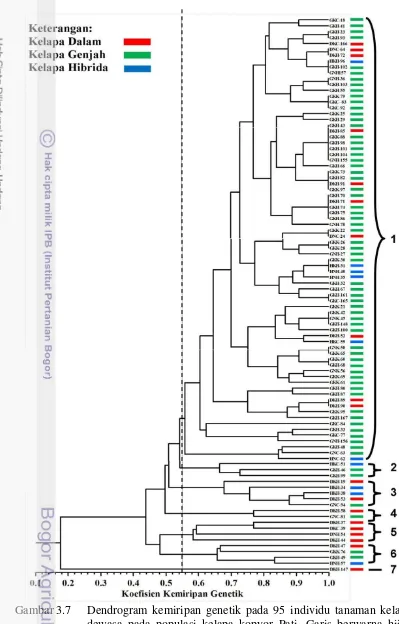
Kemiripan Genetik
Analisis kemiripan genetik pada populasi tanaman kelapa dewasa menggunakan koefisien DICE mengelompok menjadi tujuh berdasarkan kemiripan 55% atau mengelompok menjadi tiga berdasarkan kemiripan 45% (Gambar 3.7). Kelompok satu dan enam terdiri dari kelapa Genjah, kelapa Dalam dan kelapa Hibrida, tetapi kelapa Genjah lebih dominan. Kelompok dua terdiri atas kelapa Genjah dan kelapa Hibrida. Kelompok tiga terdiri dari kelapa Genjah, kelapa Hibrida dan kelapa Dalam. Kelompok empat terdiri atas kelapa Dalam dan kelapa Hibrida. Kelompok lima dan tujuh hanya terdiri dari pohon kelapa Dalam. Seluruh individu tanaman dalam populasi tetua menjadi satu kelompok dengan kemiripan 18%.
heterozigot maupun homozigot yang diperoleh dari pohon yang berbuah kopyor, sehingga menyebabkan turunannya memiliki kekerabatan yang dekat.
Hasil yang sama juga dilaporkan dalam penelitian Maskromo (2005) yang mengatakan bahwa pohon kelapa berbuah kopyor dari lokasi yang sama membentuk kelompok dengan kemiripan antara 61% hingga 100%. Misalnya kemiripan genetik pada populasi kelapa berbuah kopyor di Lampung memiliki kisaran 62% hingga 100%, sedangkan di daerah Pati memiliki kisaran 61% hingga 100%, dan di daerah Sumenep memiliki kisaran 55% hingga 100%. Kemiripan genetik di daerah Pati pada penelitian ini lebih rendah dibanding penelitian Maskromo (2005). Hal itu disebabkan karena perbedaan jumlah tanaman yang digunakan, yaitu hanya 30 tanaman kelapa dewasa, sedangkan pada penelitian ini menggunakan 95 tanaman kelapa dewasa.
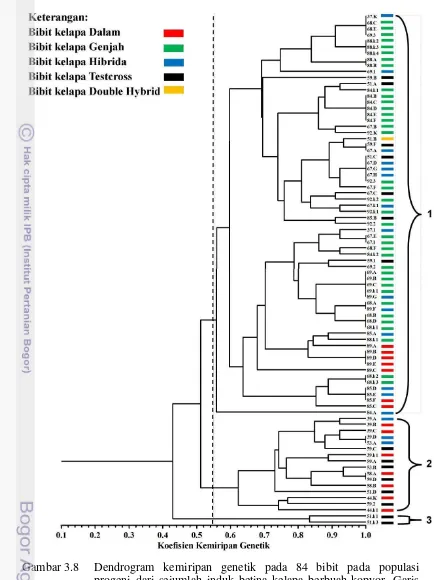
Analisis kemiripan genetik pada populasi progeni menggunakan data molekuler mengelompok menjadi tiga bagian berdasarkan kemiripan 55% (Gambar 3.8). Kelompok satu terdiri atas bibit kelapa tipe Dalam, Genjah, Hibrida, testcross dan double hybrid tetapi didominasi oleh bibit kelapa tipe Genjah. Kelompok dua terdiri dari bibit kelapa tipe Dalam, Hibrida, dan testcross
tetapi didominasi oleh bibit kelapa tipe Dalam dan testcross. Kelompok tiga hanya terdiri oleh bibit kelapa tipe testcross. Bibit kelapa tipe testcross dibedakan menjadi dua yaitu testcross Dalam dan testcross Genjah. Bibit kelapa testcross
Dalam adalah bibit kelapa yang terbentuk dari induk kelapa Hibrida dengan induk kelapa Dalam. Bibit kelapa testcross Genjah adalah bibit kelapa yang terbentuk dari induk kelapa Hibrida dengan induk kelapa Genjah.
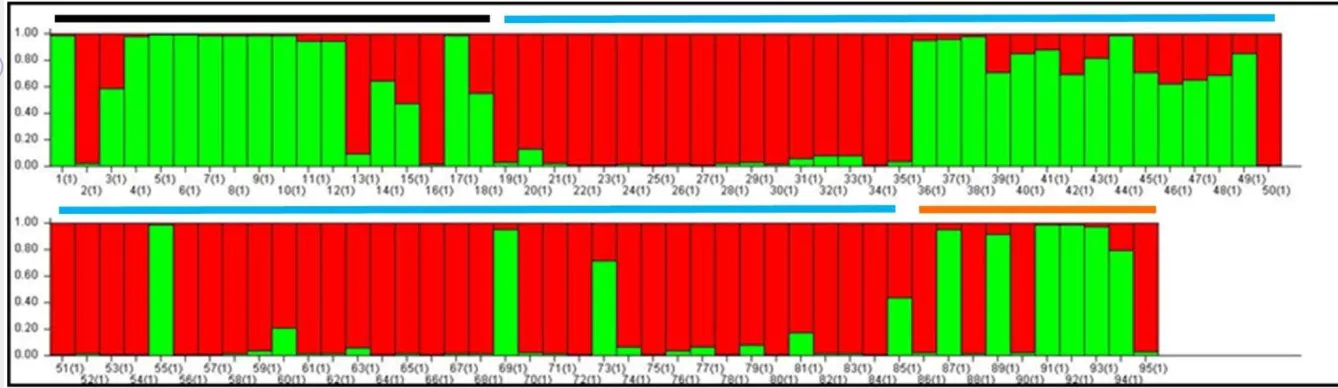
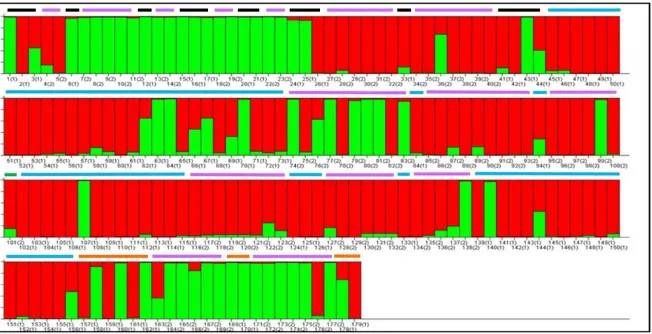
Susunan struktur genetik antar populasi dapat dilihat menggunakan program komputer STRUCTURE (Pritchard et al. 2000). Variasi genetik dalam populasi tanaman kelapa dewasa (Gambar 3.10) serta populasi tanaman dewasa dengan progeninya (Gambar 3.9) memiliki nilai K = 2, artinya bahwa dalam populasi tersebut mengelompok menjadi dua subgroup yang dilambangkan dengan warna merah dan hijau. Setiap individu ditampilkan dalam garis vertikal. Warna yang sama pada individu yang berbeda menunjukkan bahwa mereka adalah satu kelompok atau memiliki genetik yang mirip. Warna yang berbeda dalam satu individu mengindikasikan persentase genetik yang diperoleh dari masing-masing kelompok. Genotipe progeni disusun disamping induk betinanya diberi label (2) sedangkan populasi kelapa dewasa diberi label (1) pada Gambar 3.10.
Analisis struktur genetik dapat mengelompokkan kelapa berdasarkan jenisnya, yaitu kelapa Dalam dan kelapa Genjah. Kelapa Dalam dikelompokkan dengan warna hijau sedangkan kelapa Genjah dikelompokkan dengan warna merah. Kelapa hibrida adalah perpaduan antara kelapa Genjah dan kelapa Dalam, sehingga warna pada individu kelapa hibrida adalah merah dan hijau. Jika kelapa hibrida memiliki warna hijau yang lebih dominan maka sifat kelapa Hibrida tersebut lebih mirip dengan kelapa Dalam. Jika kelapa Hibrida memiliki warna merah yang lebih dominan maka sifat kelapa Hibrida tersebut lebih mirip dengan kelapa Genjah.
25
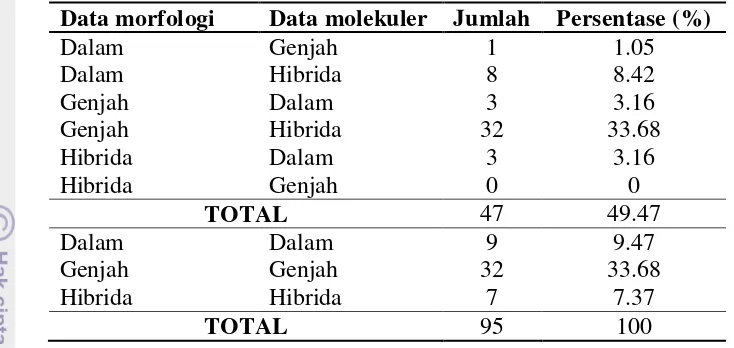
Tabel 3.3 Perbedaan data morfologi dan data molekuler terhadap tipe kelapa Data morfologi Data molekuler Jumlah Persentase (%)
Dalam Genjah 1 1.05
Penentuan tipe kelapa berdasarkan data morfologi memiliki perbedaan dengan data molekuler. Penentuan tipe kelapa berdasarkan data molekuler didasarkan pada analisis perangkat lunak komputer STRUCTURE. Perubahan identifikasi tersebut dapat dilihat pada Tabel 3.3. Perubahan yang paling banyak terjadi adalah kelapa Genjah berdasarkan data morfologi berubah menjadi kelapa Hibrida berdasarkan data molekuler. Selain itu, terdapat pula perubahan dari kelapa Genjah ke kelapa Dalam, maupun sebaliknya. Tipe kelapa yang berubah secara keseluruhan adalah 47 pohon (49.47%).
Hasil analisis struktur genetik menggunakan perangkat lunak STRUCTURE juga memperlihatkan adanya kecenderungan terjadi pengelompokan antara kelapa Genjah dengan kelapa Dalam (Gambar 3.9). Struktur genetik pada progeni sebagian besar mengikuti induk betinanya, tetapi juga ada yang mengikuti induk jantannya (Gambar 3.10). Jika progeni berwarna hijau, artinya sebagian besar genetik pada progeni tersebut bersifat kelapa Dalam, begitupun sebaliknya. Kelapa Hibrida merupakan kombinasi warna merah dan hijau. artinya kelapa Hibrida memiliki komposisi genetik yang merupakan kombinasi kelapa Genjah dengan kelapa Hibrida. Alel-alel yang dominan muncul pada kelapa Dalam ditampilkan dalam warna hijau. Alel-alel yang dominan muncul pada kelapa Genjah ditampilkan dalam warna merah.
Simpulan
Kisaran nilai PIC dan nilai He menunjukkan bahwa kemiripan genetik pada populasi kelapa kopyor Pati tergolong moderat. Tipe kelapa yang ditentukan melalui observasi morfologi di lapangan memiliki perbedaan pada tingkat molekuler dengan tingkat kemiripan genetik tergolong moderat.
Daftar Pustaka
Azrai M. 2006. Sinergi teknologi marka molekuler dalam pemuliaan tanaman jagung. Jurnal Litbang Pertanian. 25:81-89.
Boer D. 2007. Keragaman dan Struktur Genetik Populasi Jati Sulawesi Tenggara Berdasarkan Marka Mikrosatelit. [Disertasi]. Indonesia (ID) : Institut Pertanian Bogor.
Brody JR & Kern SE. 2004. Sodium boric acid: a Tris-free, cooler conductive medium for DNA electrophoresis. Bio Tech. 36:214-6.
Creste S, Neto AT, Figueira A. 2001. Detection of single sequence repeat polymorphisms in denaturing polyacrylamide sequencing gels by silver staining. Plant Mol Biol Rep. 19:299–306.
Chakravarthi BK and Naravaneni R. 2006. SSR marker based DNA fingerprinting and diversity study in rice (Oryza sativa L.). African J Biotech. 5:684-688.
Evanno G, Regnaut S, Goudet J. 2005. Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study. Mol Ecol. 14:2611-2620.
Heliyanto B dan Tenda ET. 2010. Varietas kelapa Dalam unggul spesifik Gorontalo. Bul Palma. 38:73-89.
Hildebrand CE, Torney DC, Wagner RP. 1992. Informativeness of polymorphic DNA markers. Los Alamos Sci. 20:100-102.
Konan KJN, Koffi KE, Konan JL, Lebrun P, Dery SK, Sangare A. 2007. Microsatellite gene diversity in coconut (Cocos nucifera L.) accessions resistants to lethal yellowing disease. African J Biotech. 6:341-347.
Margarita MH, Alan WM, Lalith P, Joanne R, Raymond JS. 2010. Ambiguous genetic relationships among coconut (Cocos nucifera L.) cultivars: the effects of outcrossing, sample source and size, and method of analysis.
Genet Resour Crop Evol. 57:203-217.
Martinez RT, Luc B, Angelique B, and Michael D. 2009. Characterization of the genetic diversity of the Tall coconut (Cocos nucifera L.) in the Dominican Republic using microsatellite (SSR) markers. Tree Genets Genomes. 6:73-81.
Maskromo I. 2005. Kemiripan Genetik Populasi Kelapa Berbuah Kopyor Berdasarkan Karakter Morfologi dan Penanda DNA SSRs (Simple Sequence Repeats) [Tesis]. Indonesia (ID) : Institut Pertanian Bogor. Maskromo I, Mashud N, Hutapea R, Novarianto H. 2007. Keragaman tipe kelapa
kopyor di Indonesia. Di dalam: Elsje TT, Miftahorrachman, Meldy LAH, Arie AL, Abener L, Donata SP, Nurhaini M, editor. Prosiding Konferensi Nasional Kelapa VI Buku II; 2006 Mei 16-18; Gorontalo, Indonesia. Bogor (ID): Pusat Penelitian dan Pengembangan Perkebunan. Hlm 294-299.
Noel KKJ, Edmond KK, Konan KJL, Konan KE. 2011. Microsatellite gene diversity within Philippines dwarf coconut palm (Cocos nucifera L.) resources at Port-Bouet, Cote d’lvoire. Sci Res Essay. 6:5986-5992.
Pritchard JK, Stephens M, Donnelly P. 2000. Inference of population structure using multilocus genotype data. Genetics. 155:945-959.
Rajesh MK, Nakarajan P, Jerard BA, Arunachalam V, Dhanapal R. 2008. Microsatellite variability of coconut accessions (Cocos nucifera L.) from Andaman and Nicobar Island. Curr Sci. 94:1627-1631.
Ribeiro FE, Baudouin L, Lebrun P, Chaves LJ, Brondani C, Zucchi MI, Vencovsky R. 2010. Population structure of Brazilian tall coconut (Cocos nucifera L.) by microsatellite markers. Genet Mol Bio. 33:696-702. Rohde W, Kullaya A, Rodriguez J, Ritter E. 1995. Genome analysis of Cocos
nucifera L by PCR Amplification of spacer sequences separating a subset of copies-like EcoR1 repetitive elements. J Genet Breed. 49:170-186.
Rohlf FJ. 1995. NTSYS-pc Numerical Taxonomy and Multivariate Analysys System Version 2.0. New York (US): Exeter Software.
Sajib AM, M.M. Hossain, A.T.M.J. Mosnaz, H. Hossain, M.M. Islam, M.S. Ali, SH. Prodhan. 2012. SSR marker-based molecular characterization and genetic diversity analysis of aromatic landreces of rice (Oryza sativa L.).
Samonthe LJ, Mendoza EMT, Ilag LL, De La Cruz ND, Ramirez DA. 1989. Galactomannan degrading enzym in maturing normal and makapuno and germinating normal coconut endosperm. Phytochem. 28:2269–2273. Saptahadi D, Hartati RRS, Setiawan A, Heliyanto B, Sudarsono. 2011.
Pengembangan marka simple sequence repeat untuk Jatropha spp. Jurnal Litri. 17:140-149.
Sardou MA, Baghizadeh A, Tavasoli A, and Babaei S. 2011. The use of microsatellite markers for genetic diversity assessment of genus
Hordeum L. in Kerman province (Iran). African J Biotech. 10:1516-1521. Shalini KV, Manjunatha S, Lebrun P, Berger A, Baudouin L, Pirany N, Rangananth RM, Prasad DT. 2007. Identification of molecular markers associated with mite resistance in coconut (Cocos nucifera L.). Genome.
50:35-42.
Sudarsono, Sudrajat, Novarianto H, Hosang MLA, Dinarti D, Rahayu MR, Maskromo I. 2012. Produksi bibit kopyot true to type dengan persilangan terkontrol dan peningkatan produksi buah kopyor dengan polinator lebah madu. Laporan Akhir Program Hi Link. Bogor (ID): Institut Pertanian Bogor.
Sukendah. 2009. Pembiakan Kopyor In Vitro dan Analisis Molekuler Kelapa Kopyor [Disertasi]. Indonesia (ID) : Institut Pertanian Bogor.
ANALISIS PENYEBARAN SERBUK SARI KELAPA KOPYOR
PATI MENGGUNAKAN MARKA SSR DAN SNAP
Abstract
Pollen and seed dispersal could be used to estimate gene flow among plants in a population. In kopyor coconut, pollination of normal pollen (pollen produced by normal coconut) reduced kopyor coconut yield. Therefore, it was necessary to evaluate the effect of pollen dispersal on kopyor fruit yield the mix garden containing normal and kopyor coconut provenances. The objectives of this experiment were to evaluate pollen dispersal among coconut provenances using SSR and SNAP markers and its effects on kopyor fruit yield. Fifteen coconut provenances were selected as female parents among a total of 95 adult palms. The coordinate positions of the adult palm were determined using GPS. Four SSR markers and one SNAP marker were used to genotype all provenances and 84 progenies harvested from 15 female parents. Parent-offspring relationships were determined based on genotype data using CERVUS software. The result of analysis indicated the farthest distance of pollen dispersal recorded in this evaluation was 61.8 m. The yield of kopyor fruit harvested was 2-3 fruits per bunch. Moreover, a number of progenies obtained pollens from normal fruit producing palms. The existence of normal palms in the garden affected negatively on kopyor fruit production.
Pendahuluan
Kelapa kopyor memiliki potensi dan peluang sebagai komoditi ekspor, tetapi rendahnya produksi buah kopyor yang diperoleh petani dari pohon kelapa berbuah kopyor masih menjadi kendala dalam pengembangannya (Maskromo et al. 2102). Pertanaman kelapa kopyor di Pati umumnya berada dalam pekarangan penduduk. Pohon kelapa berbuah kopyor ditanam dalam populasi campuran dengan kelapa normal Dalam, Genjah, maupun Hibrida.
Kelapa Dalam umumnya menyerbuk silang sedangkan kelapa Genjah umumnya menyerbuk sendiri (Pandin 2009). Kelapa normal yang hadir dalam areal pertanaman kelapa kopyor, baik itu tipe Dalam maupun tipe Genjah diduga mempengaruhi jumlah produksi buah kelapa kopyor. Hal ini disebabkan karena pohon kelapa berbuah normal dengan genotipe KK dapat mengkontribusikan gamet K ke pohon kelapa lain disekitarnya sehingga mengakibatkan buah kopyor tidak terbentuk. Syarat terbentuknya buah kopyor adalah induk betina harus bersifat kopyor heterozigot atau kopyor homozigot dengan genotipe Kk atau kk
dan mendapatkan gamet k dari induk jantan (Maskromo et al. 2011b).
Kelapa normal yang menyerbuki kelapa kopyor dapat dibuktikan melalui analisis penyebaran serbuk sari atau analisis induk jantan. Serbuk sari pada populasi kelapa kopyor di Pati dapat dibawa oleh vektor serangga maupun angin. Salah satu cara untuk mempelajari analisis penyebaran serbuk sari adalah menggunakan penanda molekuler genetik (Hamrick dan Trapnell 2011).
Marka yang sering digunakan dalam analisis penyebaran serbuk sari adalah marka SSR. Marka SSR telah digunakan dalam analisis penyebaran serbuk sari pada tanaman jati (Prabha et al. 2011), Prunus mahaleb (Garcia et al. 2005),
Pinus koraiensis (Feng et al. 2010), Hymenaea courbaril L. (Carneiro et al. 2011) dan analisis induk jantan pada tanaman kelapa kelapa (Pandin et al. 2008). Marka SNAP memiliki keuggulan dibanding dengan marka SSR yaitu dapat mendeteksi variasi dalam sekuens DNA yang menyebabkan perbedaan fenotipe (Manju dan Arunachalam 2011). Tujuan penelitian ini adalah (1) mempelajari penyebaran serbuk sari kelapa kopyor di Pati Jawa Tengah. (2) Mengamati produktivitas pohon kelapa berbuah kopyor dan mempelajari hubungannya dengan pola penyebaran serbuk sari.
Bahan dan Metode
Waktu dan Tempat Penelitian