SELEKSI CIRI MORFOLOGI UNTUK MEMBANGUN KUNCI
IDENTIFIKASI ASTERINACEAE BERDASARKAN
PRINCIPAL COMPONENT ANALYSIS
KURNIA NURAENI
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
SELEKSI CIRI MORFOLOGI UNTUK MEMBANGUN KUNCI
IDENTIFIKASI ASTERINACEAE BERDASARKAN
PRINCIPAL COMPONENT ANALYSIS
KURNIA NURAENI
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2012
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar
Sarjana Komputer pada
ABSTRACT
KURNIA NURAENI. Selection of Morphological Characters for Setting Up Asterinaceae
Identification Key Using Principal Component Analysis. Supervised by SRI NURDIATI and GAYUH
RAHAYU.
Characters of each fungal species may show similarities to others, such that they are related to
each other. In this case, the characters of 90 species of Asterina, 2 species of Asterolibertia, 7 species
of Lembosia, and 2 species of Parasterinella (Asterinaceae) were studied. These species are delimited
based on 116 morphological characters with three different character states (71 nominal, 33 ordinal,
and 12 numerical). Recognizing each species using all those characters is rather difficult. Therefore,
selection of representative characters is needed. Selection was done by reducing the number of
features to the minimum without losing the essential information using Principal Component Analysis
(PCA) approach. Eventhough PCA is usually applied for one type of data, in this research PCA was
experimented for mixed data types. The purpose was to select the most representative morphological
traits. Selected traits were then used as parameters in the construction of interactive Asterinaceae
identification system using the principal component scores. The result of this research shows that PCA
can be used to reduce the traits with different data types. Its 23 characters out of 116 characters were
selected. The accuracy of the identification system based on those 23 traits was 100%, as all
Asterinaceae species can be identified.
Judul Skripsi
: Seleksi ciri morfologi untuk membangun kunci identifikasi Asterinaceae
berdasarkan Principal Component Analysis
Nama
: Kurnia Nuraeni
NRP
: G64080065
Menyetujui:
Pembimbing I
Pembimbing II
Dr. Ir. Sri Nurdiati, M. Sc.
Dr. Ir. Gayuh Rahayu
NIP. 19601126 198601 2 001
NIP. 19580105 198303 2 002
Mengetahui:
Ketua Departemen Ilmu Komputer
Dr. Ir. Agus Buono, M.Si, M.Kom
NIP. 19660702 199302 1 001
KATA PENGANTAR
Segala puji bagi Allah
subhanahu wata’ala
atas segala limpahan rahmat serta karunia-Nya
sehingga penulis mampu menyelesaikan penelitian ini dengan baik.
Shalawat dan salam penulis
sampaikan kepada Nabi Muhammad
shallallahu ‘alaihi wasallam serta kepada keluarganya,
sahabatnya, serta para pengikutnya hingga akhir zaman. Penulis juga menyampaikan terima kasih
kepada seluruh pihak yang telah berperan dalam penelitian ini, yaitu:
1
Almarhum Ayahanda Kinang, Ibunda Eri, serta Adik Muhammad Isya Ramadhan dan
Muhammad Ali Abdurrahman atas doa, kasih sayang, dukungan, serta motivasi kepada penulis
untuk penyelesaian penelitian ini.
2
Ibu Dr. Ir. Sri Nurdiati, M. Sc dan Dr. Ir. Gayuh Rahayu selaku dosen pembimbing yang telah
memberi banyak ide, saran, bantuan, serta dukungan sampai selesainya penelitian ini.
3
Bapak Mushthofa, S. Kom, M.Sc selaku dosen penguji yang telah memberi masukan dan saran
pada penelitian ini.
4
Bapak Hendra Rahmawan, S. Kom, M.T selaku pembimbing akademik yang telah memberikan
bimbingan kepada penulis selama penulis menyelesaikan masa belajar di jurusan Ilmu
Komputer, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
5
Seluruh staf pengajar dan karyawan Departemen Ilmu Komputer, FMIPA IPB yang telah
membantu memfasilitasi segala keperluan kuliah dan birokrasi yang harus diselesaikan penulis.
6
Darina Putri Siswantoro, Linda Dwi Roswitasari, Zola Mukhda, Lizza Amini Gumilar, Latifah
Hanum P., Miftahurrohman, Girisa Hartiwi dan semua teman-teman Pondok Nuansa Sakinah
atas segala kebersamaan suka duka selama hampir 3 tahun tinggal bersama dengan penulis.
7
Alif Kurniawan atas dukungan, motivasi, dan segala bentuk bantuannya dalam penyelesaian
penelitian ini.
8
Vininta Ayudiana Fitriani dan rekan-rekan seperjuangan di Ilmu Komputer IPB angkatan 45
atas segala kebersamaan, bantuan, dukungan, serta kenangan bagi penulis selama menjalani
masa studi. Semoga kita bisa berjumpa kembali kelak sebagai orang-orang sukses.
9
Rekan-rekan satu bimbingan, Muhammad Rifkiaansyah, Indra Lesmana, Cipta Wiraswasta,
Neri Petri Anti, Yuli Susanti, dan Rizka Paramitha semoga lancar dalam melanjutkan
penelitiannya.
10
Adik-adik panti asuhan Raksa Putra dan para anak jalanan yang selalu meringankan beban
pikiran, memberikan motivasi dan pelajaran hidup bagi penulis dengan keceriaan, cerita, dan
kisah hidup kalian sehingga penulis tetap semangat dalam menyelesaikan penelitian.
Penulis berharap penelitian ini dapat memberikan manfaat, khususnya bagi peneliti Ilmu
Komputer dan Institut Pertanian Bogor serta Departemen Pertanian pada umumnya.
Bogor, Juli 2012
RIWAYAT HIDUP
Penulis dilahirkan di Bekasi pada tanggal 22 Mei 1990. Penulis merupakan anak pertama dari
pasangan almarhum Kinang dan Eri. Pada tahun 2008, penulis menamatkan pendidikan di SMA
Negeri 2 Cikarang Utara, Kota Bekasi. Penulis lulus seleksi masuk Institut Pertanian Bogor (IPB)
pada tahun yang sama melalui jalur Undangan Seleksi Masuk IPB dan diterima sebagai mahasiswa di
Departemen Ilmu Komputer, Fakultas Matematika dan Ilmu Pengetahuan Alam.
DAFTAR ISI
Halaman
DAFTAR TABEL ... vi
DAFTAR GAMBAR ... vi
DAFTAR LAMPIRAN ... vi
PENDAHULUAN
Latar Belakang ... 1
Tujuan ... 1
Ruang Lingkup ... 1
METODE PENELITIAN
Gambaran Umum Penelitian ... 1
Studi Literatur ... 1
Analisis Jenis Data ... 2
Analisis Faktor ... 2
Uji Matriks Korelasi ... 2
Principal Component Analysis ... 3
Pembuatan Sistem Identifikasi ... 5
Pengujian dan Analisis ... 5
HASIL DAN PEMBAHASAN
Analisis Jenis Data ... 6
Analisis Faktor ... 6
Uji Matriks Korelasi ... 6
Uji Kaiser Meyer Olkin (KMO), Uji Barlett, dan Uji Measures of Sampling
Adequacy (MSA)... 6
Principal Component Analysis ... 6
Uji Communalities ... 6
Total Variance Explained ... 6
Component Matrix dan Rotated Component Matrix ... 7
Component Score Coefficient Matrix ... 8
Pembuatan Sistem Identifikasi ... 8
Pengujian dan Analisis ... 9
SIMPULAN DAN SARAN
Simpulan... 10
Saran ... 10
DAFTAR PUSTAKA ... 10
vi
DAFTAR TABEL
Halaman
1 Ciri-ciri terpilih, nilai loading factor, dan nilai summated scales ... 7
2 Penamaan ciri, nilai factor loading, dan component score coefficient matrix ... 7
DAFTAR GAMBAR
Halaman
1 Gambaran umum proses penelitian. ... 2
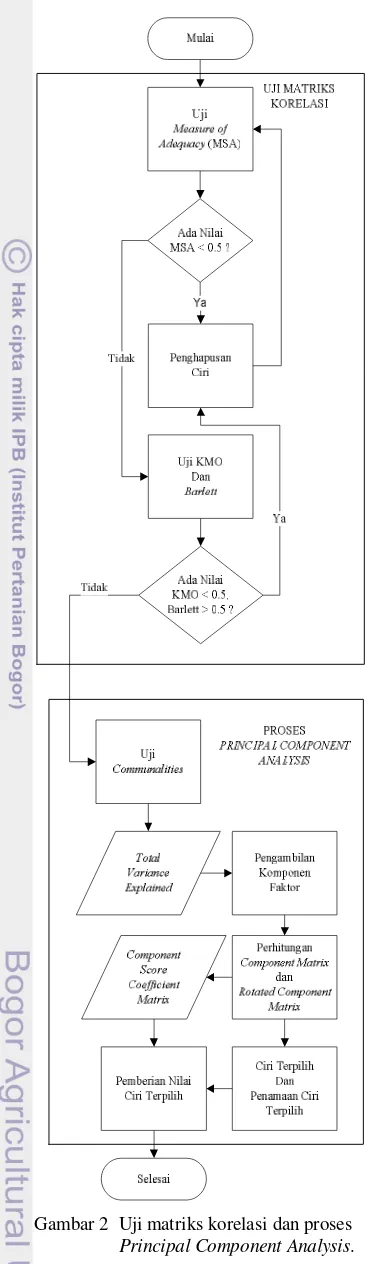
2 Uji matriks korelasi dan proses Principal Component Analysis. ... 4
3 Antarmuka awal sistem identifikasi. ... 9
4 Antarmuka tentang sistem. ... 9
5 Antarmuka identifikasi. ... 9
6 Antarmuka peringatan. ... 9
7 Antarmuka hasil identifikasi (1). ... 9
8 Antarmuka hasil identifikasi (2). ... 9
DAFTAR LAMPIRAN
Halaman
1 Data asli ... 12
2 Matriks korelasi ... 19
3 Uji KMO dan uji Barlett ... 24
4 Anti-image correlation ... 25
5 Communalities ... 30
6 Total variance explained ... 31
7 Component matrix dan rotated component matrix ... 32
PENDAHULUAN
Latar Belakang
Cendawan merupakan suatu kelompok
mikroorganisme yang sangat besar dan dapat
ditemukan pada semua relung ekologi. Menurut
Hawksworth (1991), diperkirakan 1.500.000
spesies cendawan terdapat di dunia dan 69.000
spesies telah dideskripsikan. Pengelompokan
spesies
cendawan
tersebut
dilakukan
berdasarkan morfologi.
Menurut Gandjar
et al. (2006), satu jenis
cendawan dapat dibedakan dengan jenis
cendawan
lain
berdasarkan
bentuk
morfologinya. Asterinaceae merupakan salah
satu cendawan yang hidup di permukaan daun.
Di seluruh dunia telah dikenal sebanyak 336
spesies
Asterina (Kirk
et al. 2008). Rahayu
(1992) telah mendeskripsikan 90 spesies
Asterina asal Australia. Spesies-spesies ini
dibangun berdasarkan 116 ciri morfologi.
Berdasarkan ciri-ciri ini, Rahayu (1992) juga
telah mengenal 2 spesies
Asterolibertia, 7
spesies Lembosia, dan 2 spesies
Parasterinella.
Ciri-ciri yang terdapat pada setiap spesies
tersebut terkadang memiliki kemiripan yang
menyebabkan beberapa spesies tersebut saling
terkait satu sama lain.
Pada saat ini, pengenalan spesies-spesies
berdasarkan 116 ciri tersebut masih sulit. Oleh
karena itu, seleksi ciri pembeda perlu dilakukan
dengan mereduksi jumlah ciri sampai tingkat
minimum tanpa kehilangan informasi awalnya.
Cara tersebut diharapkan dapat mempermudah
pengenalan spesies-spesies cendawan. Metode
Principal
Component
Analysis
(PCA)
merupakan salah satu analisis statistik yang
dapat diterapkan untuk mereduksi jumlah ciri
(Jolliffe 2002).
Pada umumnya, PCA digunakan untuk
mereduksi satu jenis data. Penelitian yang
dilakukan oleh Triyoga (2011) dan Kurniawati
(2008) menggunakan metode PCA pada jenis
data numerik. Ahamad (1967) menggunakan
data numerik untuk menganalisis faktor-faktor
yang
memengaruhi
tingginya
tingkat
kriminalitas. Roswitasari (2012) menggunakan
data ordinal untuk menganalisis faktor-faktor
yang memengaruhi perilaku konsumen dalam
keputusan pembelian susu cair Ultra Milk.
Dalam penelitian ini PCA diujicobakan
untuk jenis data campuran. Data tersebut
merupakan data hasil pengelompokan spesies
Asterinaceae
yang
telah
dispesifikasikan
menurut morfologi. Melalui data tersebut,
penelitian ini berusaha menyeleksi 116 ciri
morfologi menggunakan metode PCA dengan
cara mereduksi dan mencari variabel ciri
penting yang dapat mewakili 116 ciri tersebut.
Ciri terpilih hasil seleksi digunakan sebagai
parameter dalam pembuatan sistem identifikasi
Asterinaceae
sederhana dengan memanfaatkan
nilai principal component-nya.
Tujuan
Tujuan dari penelitian ini yaitu:
mereduksi ciri dan mencari ciri penting yang
dapat mewakili 116 ciri lain dengan
menggunakan metode
Principal Component
Analysis (PCA) pada jenis data ciri
Asterinaceae yang berbeda, dan
membuat sistem identifikasi Asterinaceae
sederhana
dengan
memanfaatkan nilai
principal component.
Ruang Lingkup
Data yang digunakan untuk penelitian ini
berasal dari hasil penelitian Departemen
Biologi,
Fakultas
Matematika
dan
Ilmu
Pengetahuan Alam, Institut Pertanian Bogor
yang
berjudul
Australian
hyphopodiate
Asterinaceae: a taxonomic revision. Data yang
digunakan ialah nama-nama jenis spesies
Asterinaceae beserta ciri-cirinya yang telah
diklasifikasikan berdasarkan morfologi. Data
tersebut berjumlah 101 spesies Asterinaceae
beserta 116 ciri.
Sistem identifikasi Asterinaceae hanya
mampu
mendeteksi
spesies
berdasarkan
masukan yang sesuai dengan sumber data yang
telah ditentukan.
METODE PENELITIAN
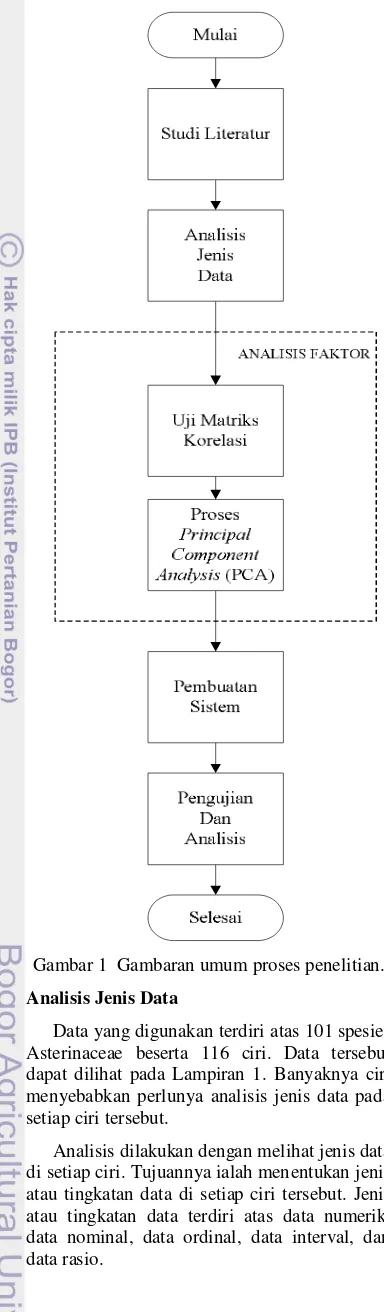
Gambaran Umum Penelitian
Gambaran umum tahapan proses pada
penelitian ini dapat dilihat pada Gambar 1.
Selanjutnya, analisis faktor terbagi menjadi 2
proses, yaitu pengujian matriks korelasi dan
proses perhitungan PCA yang dapat dilihat pada
Gambar 2.
Studi Literatur
Gambar 1 Gambaran umum proses penelitian.
Analisis Jenis Data
Data yang digunakan terdiri atas 101 spesies
Asterinaceae beserta 116 ciri. Data tersebut
dapat dilihat pada Lampiran 1. Banyaknya ciri
menyebabkan perlunya analisis jenis data pada
setiap ciri tersebut.
Analisis dilakukan dengan melihat jenis data
di setiap ciri. Tujuannya ialah menentukan jenis
atau tingkatan data di setiap ciri tersebut. Jenis
atau tingkatan data terdiri atas data numerik,
data nominal, data ordinal, data interval, dan
data rasio.
Analisis Faktor
Menurut Santoso dan Tjiptono (2001),
analisis faktor pada prinsipnya digunakan untuk
mereduksi data, yaitu proses untuk meringkas
sejumlah variabel menjadi jumlah yang lebih
sedikit dan menamakannya sebagai faktor. Jadi,
10 atribut tersebut dapat diringkas menjadi
hanya 3 faktor utama. Proses analisis faktor
mencoba menemukan hubungan antarsejumlah
variabel yang saling independen satu dengan
yang lain sehingga dapat dibuat satu atau
beberapa kumpulan variabel yang lebih sedikit
dari jumlah variabel awal (Santoso 2002).
Dalam analisis faktor, ada beberapa tahapan
yang harus dilakukan, yaitu pengujian matriks
korelasi dan pencarian faktor dengan
Principal
Component Analysis (PCA). Proses analisis ini
dilakukan
dengan
bantuan
software
PASWStatistics18.
Uji Matriks Korelasi
Menurut
Simamora
(2005),
multikolinearitas adalah korelasi antarvariabel.
Multikolinearitas cenderung dihindari dalam
regresi linear berganda, namun diinginkan
dalam analisis faktor. Bahkan, analisis faktor
tidak dapat dilakukan jika tidak terdapat
multikolinearitas. Pada tahap ini, 116 ciri yang
telah dianalisis jenis data tersebut akan
dilakukan uji matriks korelasi dengan 3 macam
pengujian, yaitu:
1
Uji Measures of Sampling Adequacy (MSA)
Pengujian
ini
bertujuan
mengetahui
koefisien korelasi parsial antarvariabel. Korelasi
parsial
merupakan
korelasi
yang
tidak
dipengaruhi oleh variabel lain. Menurut
Walpole (1983), perhitungan matematis untuk
mendapatkan koefisien korelasi parsial ialah:
r
y2.1=
r
y2-
r
y1r
12(1
-
r
y21)
(1
-
r
12 2
)
dengan
r
y2.1= koefisien korelasi parsial antara Y dan X
2sementara X
1dibuat tetap
r
y1= koefisien korelasi biasa antara Y dan X
1r
y2= koefisien korelasi biasa antara Y dan X
2r
12= koefisien korelasi biasa antara X
1dan X
2r
2y1= kuadrat koefisien korelasi biasa antara
Y
dan X
1r
212= kuadrat koefisien korelasi biasa antara X
1Angka MSA berkisar dari 0 sampai 1
dengan kriteria:
MSA = 1, variabel tersebut dapat diprediksi
tanpa kesalahan oleh variabel lain.
MSA
≥ 0.5, variabel masih bisa diprediksi
dan bisa dianalisis lebih lanjut.
MSA < 0.5, variabel tidak bisa diprediksi
dan tidak bisa dianalisis lebih lanjut, atau
dikeluarkan dari variabel lainnya.
2
Uji Kaiser Meyer Olkin (KMO)
Pengujian
ini
bertujuan
mengetahui
kelayakan analisis faktor sebuah data. Formula
untuk menghitung KMO sebagai berikut:
KMO=
∑ ∑rij 2∑ ∑rij2+ ∑ ∑aij2
untuk i
≠
j
dengan
r
ij=
koefisien korelasi sederhana antara variabel
i dan variabel j
a
ij=
koefisien korelasi parsial antara variabel
i
dan variabel j.
Nilai KMO yang kecil mengindikasikan
bahwa penggunaan analisis faktor harus
dipertimbangkan kembali. Hal ini disebabkan
korelasi antarvariabel tidak dapat diterangkan
oleh variabel lain.
Kaiser (1974) menetapkan karakteristik nilai
KMO yang terdiri atas:
jika 0.90 < nilai KMO
≤ 1.00 berarti data
sangat baik untuk analisis faktor,
jika 0.80 < nilai KMO
≤ 0.90 berarti
data
baik untuk analisis faktor,
jika 0.70 < nilai KMO
≤ 0.80 berarti data
agak baik untuk analisis faktor,
jika 0.60 < nilai KMO
≤ 0.70 berarti data
lebih dari cukup untuk analisis faktor,
jika 0.50
≤ nilai KMO ≤ 0.60 berarti d
ata
cukup untuk analisis faktor, dan
jika nilai KMO < 0.50 berarti data tidak
layak untuk analisis faktor.
3
Uji Barlett
Menurut Widarjono (2010), jika sebagian
besar nilai koefisien korelasi kurang dari 0.5,
dilakukan uji
Barlett. Uji
Barlett bertujuan
mengetahui
matriks
korelasi
tersebut
merupakan matriks identitas atau bukan.
Menurut Morrison (2005), variabel X
1,
X
2,…, X
Pdikatakan bersifat saling bebas
(independent)
jika
matriks
korelasi
antarvariabel membentuk matriks identitas.
Urutan pengujiannya yaitu:
Hipotesis
H
0: matriks korelasi merupakan matriks
identitas.
H
1: matriks korelasi bukan merupakan
matriks identitas.
Statistik Uji
x
obs2=
(
N-1
)
-
(
2
P+5
)
6
ln
|
R
|
dengan
N = jumlah observasi
p = jumlah variabel
|
R
|
= determinan matriks korelasi.
Keputusan
Pengujian Barlett akan menolak H
0jika nilai
x
obs2> x
tabel.
Selain itu, melalui
output dari
software
PASWStatistics18, kriteria uji
Barlett juga
dapat dilakukan dengan melihat nilai p-value
(signifikansi), yaitu terima H
0jika Sig. > 0.05
atau tolak H
0jika Sig. < 0.05.
Jika H
0diterima, analisis faktor tidak layak
dilakukan. Sebaliknya, jika tolak H
0, berarti
analisis faktor layak dilakukan. Hal ini
disebabkan oleh adanya hubungan antarvariabel
karena matriks korelasi tersebut berbeda secara
signifikan dengan matriks identitas atau bukan
merupakan matriks identitas.
Principal Component Analysis
Principal
Component
Analysis
(PCA)
merupakan salah satu metode dasar analisis
faktor. Menurut Jolliffe (2002), salah satu
pendekatan untuk mengurangi peubah-peubah
yang mungkin tidak memiliki kaitan dengan
peubah yang ingin diteliti ialah dengan
mengurangi peubah-peubah yang memberikan
kontribusi informasi yang sedikit pada variasi
data. PCA merupakan salah satu metode untuk
menentukan
peubah-peubah
yang
dapat
Gambar 2 Uji matriks korelasi dan proses
Principal Component Analysis.
Menurut
Hair
et
al.
(2010),
PCA
menggunakan total varian dalam analisisnya.
PCA dapat dilakukan jika nilai
common
variance lebih besar dari
specific dan
error
variance. Hal ini dilakukan karena PCA
bertujuan mengetahui jumlah faktor minimal
yang dapat diekstrak.
Common variance adalah
variance suatu
variabel yang juga dimiliki variabel-variabel
lain.
Specific variance adalah
variance yang
dimiliki hanya oleh sebuah variabel.
Error
variance adalah variance yang disebabkan oleh
kesalahan pengukuran, kesalahan alat ukur, atau
kesalahan pemilihan sampel.
Menurut
Widarjono
(2010),
PCA
merupakan teknik analisis statistik untuk
mentransformasi variabel-variabel asli yang
masih saling berkorelasi menjadi satu set
variabel baru yang tidak berkorelasi lagi.
Variabel-variabel baru tersebut disebut sebagai
komponen utama yang merupakan kombinasi
linear dari variabel-variabel asli.
Keragaman total dapat didefinisikan sebagai
Var
=
λ
1+
λ
2+…+
λ
pdengan
λ
1+
λ
2+…+
λ
padalah
akar ciri komponen utama. Besarnya proporsi
dari varian total populasi yang dapat dijelaskan
oleh komponen utama ke-j yaitu:
Proporsi
i=
λ
λ
j1
+
λ
2+…+
λ
p×100%
dengan j = 1, 2, …, p.
Pada tahap ini, data 116 ciri yang telah diuji
matriks
korelasi
akan
dilakukan
proses
Principal Component Analysis (PCA) dengan
beberapa tahapan. Tahapan tersebut terdiri atas:
1
Uji Communalities
Communalities
bertujuan
menunjukan
banyaknya varian yang dapat dijelaskan oleh
faktor yang diekstrak.
2
Total Variance Explained
Setelah dilakukan uji
communalities, akan
dihasilkan
total variance explained. Melalui
total variance explained ini dapat dilakukan
pengambilan komponen faktor.
3
Pengambilan Komponen Faktor
Menurut Widarjono (2010), pemilihan
komponen utama yang digunakan ialah jika
nilai akar cirinya lebih dari 1 (
λ
1>1).
4
Perhitungan Component Matrix dan Rotated
Component Matrix
Pada
komponen
faktor
utama
yang
dihasilkan pada total variance explained, akan
dilakukan perhitungan
component matrix. Hal
ini bertujuan mengetahui ada tidaknya korelasi
antara setiap faktor dan variabel-variabel
analisis yang telah diekstrak. Setelah dihasilkan
component matrix, matriks tersebut dirotasi
yang akan menghasilkan
rotated component
matrix. Rotasi ini dilakukan dengan tujuan
meyakinkan bahwa di antara faktor-faktor yang
diekstrak tidak terdapat korelasi.
5
Ciri Terpilih dan Penamaan Ciri Terpilih
Melalui nilai
rotated component matrix
dapat ditentukan ciri terpilih yang terdapat pada
setiap komponen faktor. Hal tersebut dilakukan
dengan
membandingkan
nilai
rotated
component matrix masing-masing ciri di setiap
komponen faktor berbeda, dan diambil nilai
rotated component matrix yang besar. Menurut
Gorsuch (1983), nilai rotasi variabel yang
paling besar pada salah satu faktor menandakan
bahwa variabel tersebut berada pada faktor
tersebut.
Dari beberapa ciri terpilih tersebut akan
ditentukan beberapa ciri yang akan mewakili
setiap komponen faktor. Menurut Santoso
(2002), terdapat 3 metode
yang dapat
digunakan, yaitu:
menggunakan satu variabel awal yang
memiliki faktor
loading
terbesar (disebut
surrogate variable) untuk mewakili setiap
faktor,
menggunakan
factor scores untuk membuat
satu atau beberapa variabel yang lebih
sedikit dan berfungsi untuk menggantikan
variabel asli yang sudah ada, atau
menggunakan
summated scales yaitu nilai
setiap faktor adalah rata-rata nilai semua
variabel yang tergabung dalam faktor
tersebut.
Penelitian ini menggunakan summated scale
untuk mendapatkan ciri terpilih. Alasan
pemilihan
summated
scale
dibandingkan
metode lain karena beberapa kelebihan yang
dimiliki
summated scale dibandingkan dengan
metode lain (factor scores dan
surrogate
variable).
Hair
et
al.
(2010)
mengemukakan
kelebihan-kelebihan yang dimiliki
summated
scale, antara lain
summated scale merupakan
kompromi antara
surrogate variable dan
factor
scores sehingga mengurangi
meassurement
error. Cara ini dulu hanya bisa dilakukan
dengan
pengolahan
structural
equation
modeling (SEM). Namun, dengan menggunakan
summated scale,
error dapat juga dikurangi
pada metode pengolahan yang lebih sederhana
dibandingkan SEM (misalkan regresi linear
berganda). Kelebihan lain dari summated scale
ialah mewakili aspek dari beberapa konsep yang
ada dan mudah untuk direplikasi pada penelitian
lainnya.
6
Component Score Coefficient Matrix
Selain digunakan untuk pemilihan ciri dan
penamaan ciri terpilih, nilai
rotated component
matrix
juga
digunakan
untuk
mencari
component
scrore
coefficient
matrix.
Component score coefficient matrix bertujuan
memberikan nilai pada ciri terpilih.
Pembuatan Sistem Identifikasi
Sistem identifikasi ini dibuat dengan
menggunakan
bahasa
pemrograman
Java.
Sistem ini memanfaatkan nilai component score
coefficient matrix dalam mendeteksi 101 spesies
Asterinaceae. Antarmuka sistem berupa aplikasi
berbasis
desktop. Masukan yang digunakan
pada sistem ini ialah kumpulan komponen
faktor yang telah diekstrak dan telah dilakukan
penamaan ciri terpilih. Keluaran dari sistem ini
ialah salah satu nama dari 101 spesies
Asterinaceae yang sesuai dengan data.
Spesifikasi perangkat keras dan perangkat
lunak yang digunakan dalam pembuatan sistem
sebagai berikut:
prosesor Intel Core Duo 2.0 GHz,
memori 2 GB,
hard disk 500 GB,
sistem operasi Windows 7 Ultimate
bahasa pemrograman Java, dan
lingkungan pengembangan (IDE) NetBeans
versi 7.1.1.
Pengujian dan Analisis
Pada tahap ini, pengujian akan dilakukan
pada sistem yang telah dibuat dengan
memasukkan beberapa ciri (komponen faktor
yang telah dilakukan penamaan ciri terpilih).
Setelah itu, dilihat keluaran dari sistem.
Pengujian ini dilakukan sebanyak 101 kali.
HASIL DAN PEMBAHASAN
Analisis Jenis Data
Melalui analisis ini dapat diketahui bahwa
116 ciri tersebut menghasilkan 3 jenis data ciri
yang berbeda. Tiga jenis data tersebut terdiri
atas 71 ciri dengan jenis data nominal, 33 ciri
dengan jenis data ordinal, dan 12 ciri dengan
jenis data numerik. Selain itu, setelah dilakukan
analisis secara manual terhadap 116 ciri, ada
beberapa variabel yang perlu dihapuskan, di
antaranya ciri ke-36 dan ke-102 karena kedua
ciri tersebut memiliki nilai yang seragam.
Analisis Faktor
Analisis faktor tidak dapat dilakukan jika
ketiga jenis data yang berbeda pada setiap ciri
tersebut digabung sehingga perlu dilakukan uji
matriks korelasi terlebih dahulu pada 3 jenis
data tersebut secara terpisah. Hal ini dilakukan
untuk memastikan nilai signifikansi
Barlett
≤
0.05 dan nilai Measures of Sampling Adequacy
dan KMO
pada masing-masing jenis data
tersebut
≥ 0.5.
Tujuannya untuk mengetahui
ketiga jenis data tersebut dapat dilakukan
analisis faktor atau tidak. Setelah dilakukan uji
matriks korelasi, maka ketiga jenis data tersebut
dapat digabung untuk dianalisis faktor. Tujuan
akhirnya ialah mendapatkan komponen faktor
utama hasil penggabungan ketiga jenis data
tersebut.
Uji Matriks Korelasi
Matriks korelasi tiap jenis data setelah
dilakukan penghapusan data dan penggabungan
jenis data dapat dilihat pada Lampiran 2. Pada
setiap matriks korelasi tersebut dapat dilihat
bahwa koefisien korelasi banyak sekali yang
nilainya kurang dari 0.5 sehingga dapat
dilakukan pengujian lebih lanjut.
Uji Kaiser Meyer Olkin (KMO), Uji Barlett,
dan Uji Measures of Sampling Adequacy
(MSA)
Matriks
korelasi
71
ciri
nominal
menghasilkan nilai KMO sebesar 0.746 dan
nilai Barlett dengan Sig. sebesar 0.000. Nilai ini
didapatkan setelah penghapusan ciri nominal
sebanyak 46 ciri. Selanjutnya, matriks korelasi
33 ciri ordinal menghasilkan nilai KMO sebesar
0.668 dan nilai
Barlett dengan Sig. sebesar
0.000. Nilai ini didapatkan setelah penghapusan
ciri ordinal sebanyak 22 ciri, sedangkan 12 ciri
numerik menghasilkan nilai KMO sebesar
0.722 dan nilai
Barlett dengan Sig. sebesar
0.000. Nilai ini didapatkan setelah penghapusan
ciri numerik sebanyak 4 ciri.
Adanya penghapusan beberapa ciri tersebut
dilakukan karena saat pengujian
Measures of
Sampling Adequacy
(MSA)
pada
masing-masing ciri, ada beberapa ciri yang nilai
Measures of Sampling Adequacy (MSA) kurang
dari 0.5. Menurut Hair
et al. (2010),
penghapusan dilakukan dengan tujuan agar pada
variabel-variabel tersebut dapat dilakukan
analisis faktor. Setelah dilakukan penghapusan
ciri, pada masing-masing ciri tersebut diperoleh
nilai
Barlett dengan Sig. sebesar 0.000, KMO
dan Measures of Sampling Adequacy (MSA)
≥
0.05.
Dengan demikian, total ciri yang digunakan
setelah penghapusan berjumlah 44 ciri dari 116.
Data-data tersebut terdiri atas 25 ciri dari 71 ciri
nominal, 11 ciri dari 33 ciri ordinal, dan 8 ciri
dari 12 ciri numerik.
Selanjutnya, dilakukan uji KMO kembali
terhadap 44 ciri tersebut. Penggabungan ketiga
jenis data ciri menghasilkan nilai KMO sebesar
0.707 dan nilai
Barlett dengan Sig. sebesar
0.000. Nilai ini didapatkan setelah penghapusan
ciri sebanyak 3 ciri. Dengan demikian, total data
yang akan digunakan untuk tahap selanjutnya
ialah 41 ciri.
Nilai pengujian KMO dan
Barlett dapat
dilihat pada Lampiran 3, sedangkan nilai
Measures of Sampling Adequacy
(MSA)
yang
didapat dari
Anti-image correlation dapat
dilihat pada pada Lampiran 4.
Principal Component Analysis
Ada beberapa tahapan proses PCA yang
dilakukan pada 41 ciri yang telah diuji matriks
korelasi. Tahapan-tahapan tersebut dijelaskan
sebagai berikut.
Uji Communalities
Pada tahap ini, dapat diketahui bahwa ciri
X
1mempunyai nilai
communalities sebesar
0.646. Hal ini berarti sekitar 64.6% ciri X
1dapat
dijelaskan
oleh
faktor
yang
terbentuk.
Sementara itu, pada ciri X
2, nilai communalities
yang didapatkan sebesar 0.657. Hal ini berarti
sekitar 65.7% ciri X
2dapat dijelaskan oleh
faktor yang terbentuk. Selanjutnya, hasil untuk
beberapa ciri lainnya dapat dilihat pada
Lampiran 5.
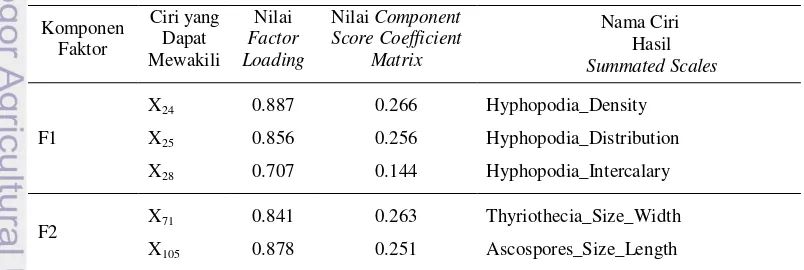
Total Variance Explained
Tabel 1 Ciri-ciri terpilih, nilai loading factor,
dan nilai summated scales
Komponen
Faktor
Ciri
Terpilih
Nilai
Loading
Factor
Nilai
Sumated
Scales
F1
X
6X
13X
19X
24X
25X
28X
43X
540.408
0.530
0.435
0.887
0.856
0.707
0.386
0.545
0.594
F2
X
33X
52X
71X
105X
1060.377
0.566
0.841
0.878
0.815
0.695
F3
X
111X
112X
113X
1160.787
0.763
0.792
0.796
0.785
F4
X
85X
86X
87X
920.799
0.820
0.767
0.876
0.816
F5
X
16X
170.885
0.898
0.892
F6
X
1X
53X
1030.544
0.689
0.711
0.648
F7
X
2X
18X
20X
60X
74X
760.608
0.352
0.508
0.730
0.429
0.522
0.525
F8
X
3X
4X
270.803
0.835
0.499
0.712
F9
X
50.751
0.751
F10
X
59X
62X
72X
840.555
0.532
0.569
0.441
0.524
F11
X
70.643
0.643
Component Matrix dan Rotated Component
Matrix
Melalui nilai
rotated component matrix
(loading factor) yang seluruhnya dapat dilihat
pada Lampiran 7, terdapat ciri-ciri terpilih yang
dapat mewakili 11 komponen faktor yang
terbentuk. Ciri-ciri beserta nilai
loading factor
dan summated scales tersebut dapat dilihat pada
Tabel 1.
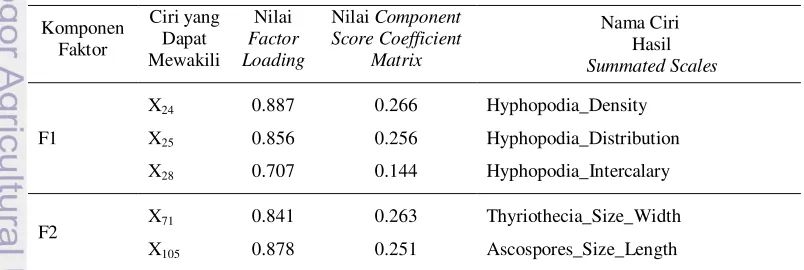
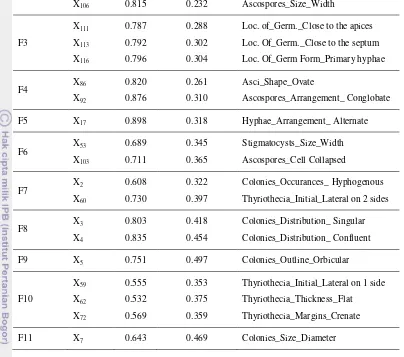
Tabel 2 Penamaan ciri, nilai factor loading, dan component score coefficient matrix
Komponen
Faktor
Ciri yang
Dapat
Mewakili
Nilai
Factor
Loading
Nilai Component
Score Coefficient
Matrix
Nama Ciri
Hasil
Summated Scales
F1
X
24X
25X
280.887
0.856
0.707
0.266
0.256
0.144
Hyphopodia_Density
Hyphopodia_Distribution
Hyphopodia_Intercalary
F2
X
71X
1060.815
0.232
Ascospores_Size_Width
F3
X
111X
113X
1160.787
0.792
0.796
0.288
0.302
0.304
Loc. of_Germ._Close to the apices
Loc. Of_Germ._Close to the septum
Loc. Of_Germ Form_Primary hyphae
F4
X
86X
920.820
0.876
0.261
0.310
Asci_Shape_Ovate
Ascospores_Arrangement_ Conglobate
F5
X
170.898
0.318
Hyphae_Arrangement_ Alternate
F6
X
53X
1030.689
0.711
0.345
0.365
Stigmatocysts_Size_Width
Ascospores_Cell Collapsed
F7
X
2X
600.608
0.730
0.322
0.397
Colonies_Occurances_ Hyphogenous
Thyriothecia_Initial_Lateral on 2 sides
F8
X
3X
40.803
0.835
0.418
0.454
Colonies_Distribution_ Singular
Colonies_Distribution_ Confluent
F9
X
50.751
0.497
Colonies_Outline_Orbicular
F10
X
59X
62X
720.555
0.532
0.569
0.353
0.375
0.359
Thyriothecia_Initial_Lateral on 1 side
Thyriothecia_Thickness_Flat
Thyriothecia_Margins_Crenate
F11
X
70.643
0.469
Colonies_Size_Diameter
Setelah ciri terpilih didapat, dicari ciri-ciri
yang mewakili 11 komponen faktor dari
kumpulan ciri terpilih. Hal ini dilakukan dengan
membandingkan nilai rotated component matrix
dengan nilai
summated scales pada setiap
faktor. Ciri yang diambil adalah ciri yang nilai
rotated component matrix lebih besar atau sama
dengan nilai
summated scales pada setiap
faktornya. Penamaan ciri, nilai factor loading,
dan
component score coefficient matrix dapat
dilihat pada Tabel 2.
Dengan demikian,
faktor
ke-1 dapat
diwakilkan dengan ciri X
24, X
25,
dan
X
28, faktor
ke-2 dapat diwakilkan dengan ciri X
71, X
105, dan
X
106, sedangkan faktor ke-3 oleh ciri X
111, X
113,
dan X
116, selanjutnya untuk faktor ke-4 sampai
dengan ke-11 dapat dilihat pada Tabel 2.
Component Score Coefficient Matrix
Komponen faktor yang telah dilakukan
penamaan ciri akan diberi nilai. Nilai ini
digunakan sebagai parameter untuk pembuatan
sistem. Nilai-nilai tersebut dapat dilihat pada
Tabel
2,
sedangkan
nilai
keseluruhan
component score coefficient matrix dapat dilihat
pada Lampiran 8.
Pembuatan Sistem Identifikasi
Gambar 3 Antarmuka awal sistem identifikasi.
Jika pengguna memilih tombol Tentang
Sistem, akan muncul antarmuka seperti yang
dapat dilihat pada Gambar 4.
Gambar 4 Antarmuka tentang sistem.
Antarmuka Tentang Sistem memiliki 1
tombol Keluar. Tombol ini digunakan pengguna
jika ingin keluar dari antarmuka Tentang Sistem
dan kembali ke antarmuka awal sistem
identifikasi.
Ketika
pengguna
memilih
tombol
Identifikasi, pengguna akan diberikan beberapa
pertanyaan berupa 23 ciri seperti yang dapat
dilihat pada Gambar 5. Pengguna juga dapat
memilih tombol
back jika ingin kembali ke
antarmuka awal sistem atau tombol Identifikasi
untuk melanjutkan proses pencarian.
Gambar 5 Antarmuka identifikasi.
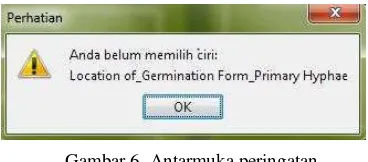
Jika pengguna tidak memasukkan semua
atau salah satu informasi dari 23 ciri, akan
muncul peringatan seperti yang dapat dilihat
pada Gambar 6.
Gambar 6 Antarmuka peringatan.
Selanjutnya, setelah pengguna mengisi
secara lengkap 23 ciri dan memilih tombol
Identifikasi, akan muncul hasil pencarian
spesies Asterinaceae. Jika data masukan sesuai
dengan sumber data, nama salah satu spesies
Asterinaceae dari berbagai varian spesies
Asterinaceae akan muncul seperti yang dapat
dilihat pada Gambar 7.
Gambar 7 Antarmuka hasil identifikasi (1).
Namun, jika masukan pengguna tidak sesuai
dengan sumber data, sistem tidak dapat
menemukan jenis spesies Asterinaceae yang
dicari, seperti yang dapat dilihat pada Gambar
8.
Gambar 8 Antarmuka hasil identifikasi (2).
Pengujian dan Analisis
Pengujian dilakukan sebanyak 101 kali
dengan memasukkan 23 ciri yang sesuai dengan
sumber
data.
Setelah
dianalisis
dengan
membandingkan
keluaran
sistem
dengan
SIMPULAN DAN SARAN
Simpulan
Simpulan dari penelitian ini antara lain:
melalui jenis data Asterinaceae yang
berbeda-beda, reduksi menggunakan metode
Principal Component Analysis (PCA) dapat
dilakukan dan telah menghasilkan 23 ciri
penting yang dapat mewakili 116 ciri yang
lain, dan
dengan
memanfaatkan
nilai
principal
component,
telah
dihasilkan
sistem
identifikasi Asterinaceae sederhana yang
mampu mengenali 101 spesies Asterinaceae.
Saran
Saran untuk penelitian selanjutnya yaitu:
1
Memerhatikan kelemahan dari penggunaan
floating point yang memengaruhi presisi
proses identifikasi yang menyebabkan hasil
identifikasi tidak tepat.
2
Membuat sistem identifikasi Asterinaceae
yang lebih kompleks dengan memanfaatkan
hasil PCA yang ada.
3
Menguji metode untuk kasus data lain,
meskipun
metode
yang
digunakan
memperoleh hasil dengan akurasi yang
sangat tinggi.
DAFTAR PUSTAKA
Ahamad B. 1967. An analysis of crimes by the
method of principal components. The Royal
Statistical Society 16: 17-35.
Gandjar, Indrawati, Sjamsuridzal W, Oetari A.
2006.
Mikologi: Dasar dan Terapan.
Jakarta: Yayasan Obor Indonesia.
Gorsuch RL. 1983.
Factor Analysis. Ed ke-2.
Hillsdale: Lawrence Erlbaum.
Hair JR, Black WC, Babin BJ, Anderson RE.
2010. Multivariate Data Analysis. Ed ke-7.
New Jersey: Prentice-Hall.
Hawksworth DL. 1991. The fungal dimension
biodiversity: magnitude, significance, and
conservation.
Mycological Research 95(6):
641-655.
Jolliffe
IT.
2002.
Principal
Component
Analysis.
Ed ke-2. New York:
Springer-Verlag.
Kaiser HF. 1974. An index of factorial
simplicity. Psyscho 39: 31-36.
Kirk PM, Cannon PF, Minter DW, Stalpers JA.
2008.
Dictionary of The Fungi. Ed ke-10.
United Kingdom: CABI Europe.
Kurniawati A. 2008. Ukuran dan bentuk tubuh
ayam arab, ayam kampung, dan ayam
pelung berdasarkan analisis komponen
utama
[skripsi].
Bogor:
Fakultas
Peternakan, Institut Pertanian Bogor.
Morrison DF. 2005.
Multivariate Statistical
Methods. Ed ke-4. Belmont: Brooks/Cole
Thomson Learning.
Rahayu G. 1992. Australian hyphopodiate
Asterinaceae:
a
taxonomic
revision
[disertasi]. Armidale: Departement of
Botany, The University of New England.
Roswitasari LD. 2012. Analisis faktor-faktor
yang mempengaruhi perilaku konsumen
dalam keputusan pembelian susu cair Ultra
Milk (studi kasus mahasiswa S1 Institut
Pertanian Bogor) [skripsi]. Bogor: Fakultas
Ekonomi
dan
Manajemen,
Institut
Pertanian Bogor.
Santoso S, Tjiptono F. 2001. Riset Pemasaran:
Konsep dan Aplikasi dengan SPSS. Jakarta:
Elex Media Komputindo.
Santoso S. 2002.
Buku Latihan SPSS Statistik
Multivariate.
Jakarta:
Elex
Media
Komputindo.
Simamora B. 2005.
Analisis Multivariate
Pemasaran. Jakarta: Gramedia.
Triyoga
E.
2011.
Analisis
faktor-faktor
dominan yang memengaruhi pendapatan
pelabuhan sunda kelapa tahun 2004-2011
[skripsi]. Bogor: Fakultas Ekonomi dan
Manajemen, Institut Pertanian Bogor.
Walpole ER. 1982.
Pengantar Statistika. Ed
ke-3. Sumantri B, penerjemah; Jakarta:
Gramedia. Terjemahan dari: Introduction to
Statistics.
Lampiran 1 Data asli
a)
Australian taxa studied
No. FungiName No. FungiName
1 A. cordyline 61 A. piperina
2 A. libertiae 62 A. orites
3 A. tuberculata 63 A. clematidis
4 A. capparidicola 64 A. trichilliae
5 A. bottomleyae 65 A. correicola var. correicola
6 A. formosana 66 A. correicola var. euodiae
7 A. hypsophilae 67 A. correicola var. queenslandica
8 A. perrottetiae var. perrottetiae 68 A. decora
9 A. perrottetiae var. ampulliformia 69 A. eudiae
10 A. malandaensis 70 A. loranthi-rhododendricoli
11 A. stipitipodia var. stipitipodia 71 A. decipiens
12 A. stipitipodia var. dilleniae 72 A. knysnae var. gracilipodia
13 A. carbonacea var. acanthopoda 73 A. argophylli
14 A. carbonacea var. oppositipodia 74 A. cuttsiae
15 A. styracina 75 A. xumenensis
16 A. alchorneae-javensis var. alchorneae-javensis 76 A. citriobati
17 A. alchorneae-javensis var. raripoda 77 A. recisa
18 A. excoecariae 78 A. polyloba
19 A. fraseriana 79 A. fieldiae
20 A. radio-fissilis 80 A. diplopoda
21 A. hoveifolia 81 A. reclinata
22 A. nova-anglica 82 A. phaleriae
23 A. oxylobii 83 A. strebli
24 A. platystoma 84 A. celtidicola
25 A. nothofagi 85 A. sponiae
26 A. puellaris 86 A. pouzolziae
27 A. peraffinis 87 A. homaliicola
28 A. loganicearum 88 A. scolopiarum
29 A. randiae-benthamianae 89 A. adeniae
30 Asterina sp. aff. pusilla 90 A. undulate
31 A. aemula var. endiandrae 91 Asterolibertia diplosporae
32 A. cinnamomi-virentis 92 Asterolibertia schroteri var. licaniae
33 A. litseae 93 L. pothoricola
34 A. xanthogloea 94 L. lobatipodia
35 A. daphnandrae 95 L. bramstonensis
36 A. doryphoricola 96 L. endriandrae
37 A. palmeriae 97 Lembosia sp. A
38 Asterina sp. aff. artabotrydis 98 L. capnoides
39 Asterina sp. A 99 L. graphioides
40 A. melanomera 100 Parasterinella compacta
41 A. uvariicola var. australiensis 101 Parasterinella drymidis
42 A. eupomatiae
43 A. pycnanthi
44 A. australiensis
45 A. drimidicola
46 A. oppositipodia
47 A. queenslandica
48 A. alpine
49 A. elaeocarpi var. elaeocarpi
50 A. elaeocarpi var. longipodia
51 A. elaeocarpicola var. elaeocarpicola
52 A. elaeocarpicola var. brevipodia
53 A. elaeocarpicola var. parvuli
54 A. sloaneae
55 A. triumfetticola
56 A. decumana
57 A. pemphidioides
58 A. quarta
59 A. syzygiicola
Lanjutan
b)
List of characters, character states and coding
List of Characters (X) Character States and Coding
Colonies Occurances 1 epigenous present (1), absent (0), miss. val. (-1) 2 hyphogenous present (1), absent (0), miss. val. (-1)
Distribution 3 singular present (1), absent (0), miss. val. (-1) 4 confluent present (1), absent (0), miss. val. (-1)
Outline 5 orbicular present (1), absent (0), miss. val. (-1) 6 irregular present (1), absent (0), miss. val. (-1)
Size 7 diameter quantitative continuous (mm), miss. val. (-1)
Hyphae Colour 8 light brown present (1), absent (0), miss. val. (-1) 9 brown present (1), absent (0), miss. val. (-1) 10 dark brown present (1), absent (0), miss. val. (-1)
Flowing 11 straight present (1), absent (0), miss. val. (-1) 12 flexuous present (1), absent (0), miss. val. (-1)
Width 13 quantitative continuous (µm) quantitative continuous (µm), miss. val. (-1)
Septation Branching 14 distinct, indistinct distinct (0), indistinct (1), miss. val. (-1)
Density 15 density numerous (2), rare (1), miss. val. (-1)
Arrangement 16 unilateral more (4), moderate (3), less (2), no (1), miss. val. (-1) 17 alternate more (4), moderate (3), less (2), no (1), miss. val. (-1) 18 opposite to another branch more (4), moderate (3), less (2), no (1), miss. val. (-1) 19 opposite to hyphopodia more (4), moderate (3), less (2), no (1), miss. val. (-1)
Angles 20 wide present (1), absent (0), miss. val. (-1) 21 acute present (1), absent (0), miss. val. (-1)
Network 22 loose present (1), absent (0), miss. val. (-1) 23 close present (1), absent (0), miss. val. (-1)
Hyphopodia Density 24 density numerous (4), moderate (3), few (2), no (1), miss. val. (-1)
Distribution 25 distribution regular (1), irregular (0), miss. val. (-1)
Location 26 middle present (1), absent (0), miss. val. (-1) 27 distal present (1), absent (0), miss. val. (-1) 28 intercalary present (1), absent (0), miss. val. (-1)
Colour 29 colour darker than hyphae (1), similar to hyphae (0), miss. val. (-1)
Arrangement 30 cluster more (4), moderate (3), less (2), no (1), miss. val. (-1) 31 unilateral more (4), moderate (3), less (2), no (1), miss. val. (-1) 32 alternate more (4), moderate (3), less (2), no (1), miss. val. (-1) 33 opposite more (4), moderate (3), less (2), no (1), miss. val. (-1)
Septation 34 no septate more (4), moderate (3), less (2), no (1), miss. val. (-1) 35 1-septate more (4), moderate (3), less (2), no (1), miss. val. (-1) 36 2-septate more (4), moderate (3), less (2), no (1), miss. val. (-1)
Stigmatopodia Shape 37 shape cylindrical (1), not cylindrical (0), miss. val. (-1)
Size 38 length quantitative continuous (µm), miss. val. (-1) 39 width quantitative continuous (µm), miss. val. (-1)
Stigmatocysts Gross Shape 40 uniform, versiform uniform (1), versiform (0), miss. val. (-1)
41 hemispherical more (4), moderate (3), less (2), no (1), miss. val. (-1) 42 ovate more (4), moderate (3), less (2), no (1), miss. val. (-1) 43 cylindrical more (4), moderate (3), less (2), no (1), miss. val. (-1) 44 vermiform more (4), moderate (3), less (2), no (1), miss. val. (-1) 45 ampulliform more (4), moderate (3), less (2), no (1), miss. val. (-1) 46 conical more (4), moderate (3), less (2), no (1), miss. val. (-1) 47 bifid or trifid more (4), moderate (3), less (2), no (1), miss. val. (-1)
Outlines 48 entire more (4), moderate (3), less (2), no (1), miss. val. (-1) 49 sinuous more (4), moderate (3), less (2), no (1), miss. val. (-1) 50 lobate more (4), moderate (3), less (2), no (1), miss. val. (-1) 51 deeply lobate more (4), moderate (3), less (2), no (1), miss. val. (-1)
Size 52 length quantitative continuous (µm), miss. val. (-1) 53 width quantitative continuous (µm), miss. val. (-1)
Lanjutan
List of Characters Character States and Coding
Thyriothecia Initial 59 lateral on 1 side present (1), absent (0), miss. val. (-1) 60 lateral on 2 sides present (1), absent (0), miss. val. (-1) 61 terminal on stalk present (1), absent (0), miss. val. (-1)
Thickness 62 flat present (1), absent (0), miss. val. (-1) 63 slightly convex present (1), absent (0), miss. val. (-1) 64 moderately convex present (1), absent (0), miss. val. (-1) 65 strongly convex present (1), absent (0), miss. val. (-1)
Outlines 66 orbicular present (1), absent (0), miss. val. (-1) 67 Ellipsoid present (1), absent (0), miss. val. (-1) 68 Linear present (1), absent (0), miss. val. (-1) 69 X or Y present (1), absent (0), miss. val. (-1)
Size 70 Length quantitative continuous (µm), miss. val. (-1) 71 Width quantitative continuous (µ,m), miss. val. (-1)
Margins 72 Crenate present (1), absent (0). miss. val. (-1) 73 fimbriate, short, loose present (1), absent (0), miss. val. (-1) 74 fimbriate, short, close present (1), absent (0), miss. val. (-1) 75 fimbriate, long, loose present (1), absent (0), miss. val. (-1) 76 fimbriate, long, close present (1), absent (0), miss. val. (-1)
Cover Wall 77 straight present (1), absent (0), miss. val. (-1) 78 flexuous present (1), absent (0), miss. val. (-1)
Cell Wall 79 isodiametrirc present (1), absent (0), miss. val. (-1) 80 rectangular present (1), absent (0), miss. val. (-1)
Opening 81 stellate cracks present (1), absent (0), miss. val. (-1) 82 longitudinal slit present (1), absent (0), miss. val. (-1) 83 cell disintegration present (1), absent (0), miss. val. (-1)
Basal Wall 84 radiate present (1), absent (0), miss. val. (-1)
Asci Number 85 number numerous (2), few (1), miss. val. (-1)
Shape 86 ovate present (1), absent (0), miss. val. (-1) 87 clavate present (1), absent (0), miss. val. (-1)
Size 88 length quantitative continuous (µm), miss. val. (-1) 89 width quantitative continuous (µm), miss. val. (-1)
Number of Ascospores 90 ascospores 8-spored only (1), 8-spored and less (0), miss. val. (-1)
Hamathecia 91 hamathecia present (1), absent (0), miss. val. (-1)
Ascospores Arrangement 92 conglobate present (1), absent (0), miss. val. (-1) 93 seriate present (1), absent (0), miss. val. (-1)
Colour 94 pale brown present (1), absent (0), miss. val. (-1) 95 brown present (1), absent (0), miss. val. (-1) 96 dark brown present (1), absent (0), miss. val. (-1)
Constriction 97 slightly yes (1), no (0), miss. val. (-1) 98 moderately yes (1), no (0), miss. val. (-1) 99 strongly yes (1), no (0), miss. val. (-1)
Cell Apices 100 round on both apices (3), one apex (2), no (1), miss. val. (-1) 101 gradually attenuated on both apices (3), one apex (2), no (1), miss. val. (-1) 102 bent on both apices (3), one apex (2), no (1), miss. val. (-1)
Cell Collapsed 103 cell collapsed present (1), absent (0), miss. val. (-1)
Cell Equality 104 cell equality subequal (1), unequal (0), miss. val. (-1)
Size 105 length quantitative continuous (µm), miss. val. (-1) 106 width quantitative continuous (µm), miss. val. (-1)
Surfaces 107 smooth present (1), absent (0), miss. val. (-1) 108 granulose present (1), absent (0), miss. val. (-1) 109 verrucose present (1), absent (0), miss. val. (-1) 110 spinulose present (1), absent (0), miss. val. (-1)
Location of Germination 111 close to the apices yes (1), no (0), miss. val. (-1) 112 middle yes (1), no (0), miss. val. (-1) 113 close to the septum yes (1), no (0), miss. val. (-1)
Lanjutan
c)
Data matrix of taxa studied
Taxa Number Coding of Each Character State (1-116)
1 1 1 1 1 0 1 9 0 0 1 1 0 4 1 2 4 4 1 1 1 0 1 0 2 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 0 4 4 1 1 1 1 1 4 3 2 1 11 9 4 1 1 2 1 1 1 0 1 1 0 0 1 1 0 0 225 225 0 1 0 0 0 1 0 0 1 1 0 0 0 -1 1 1 50 39 1 1 1 0 0 0 1 1 0 0 2 1 1 0 1 29.5 13.5 1 0 0 1 1 1 0 0 0 1
2 1 1 1 1 0 1 1 0 1 0 0 1 5 1 2 4 4 1 1 1 0 0 1 2 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 0 4 4 1 1 1 1 1 4 3 2 1 6 9.5 4 1 1 2 1 1 1 0 1 1 0 0 1 1 0 0 200 200 1 0 0 0 0 1 0 0 1 1 0 0 0 -1 1 1 38.5 29 1 0 1 0 0 0 1 1 0 0 3 1 1 0 1 26.5 11 1 0 0 1 1 1 0 0 1 1
3 1 1 1 1 1 1 5 0 0 1 1 1 5 1 1 1 1 4 4 1 0 0 1 3 1 1 0 1 0 1 4 4 1 4 1 1 0 0 0 1 4 4 2 1 1 1 1 4 2 1 1 11.5 8.5 4 2 1 2 1 1 1 0 0 1 0 0 1 0 0 0 180 180 1 0 0 1 0 0 1 1 0 1 0 1 0 1 1 1 41 29 1 0 1 0 0 1 1 0 0 1 2 2 1 0 1 30 13 0 0 0 1 1 0 0 0 1 0
4 1 1 0 1 1 0 4 1 0 1 0 1 4 0 2 4 4 2 2 1 0 0 1 3 1 1 1 1 0 1 4 4 2 1 4 1 1 6 5 1 1 4 1 1 1 1 1 4 3 2 1 8 6.5 4 1 1 3 2 0 1 0 0 0 1 0 1 0 0 0 224 224 1 1 0 1 0 1 1 1 0 1 0 0 0 2 1 0 40 32 1 1 1 0 0 1 0 1 0 0 3 1 1 0 1 21.5 10.5 0 0 1 0 1 0 0 0 1 1
5
1 0 -1 -1 0 1 5 1 0 1 0 1 6.5 0 -1 2 2 4 1 1 0 1 0 3 1 1 1 1 0 1 2 2 4 4 1 1 0 0 0 1 1 1 4 1 1 2 1 4 1 1 1 11.5 6 4 1 1 3 1 0 1 0 0 0 1 0 1 0 0 0 240 240 0 0 0 0 1 1 0 1 0 1 0 0 0 2 0 1 56 43 1 0 1 0 0 0 1 0 1 0 3 1 1 0 1 32 16.5 0 0 1 0 1 0 0 0 0 1
6
1 0 0 1 0 1 4 0 1 0 1 0 5.5 0 -1 1 1 4 4 1 0 0 1 3 1 1 0 1 0 1 2 2 4 4 1 1 0 0 0 1 1 1 4 1 1 1 1 4 1 1 1 8.5 9 4 2 1 2 1 0 1 0 0 0 1 0 1 0 0 0 370 370 0 0 1 0 0 1 0 0 1 1 0 0 -1 2 0 1 56 43 1 0 1 0 0 0 1 0 1 0 3 1 1 0 1 32.5 15.5 1 0 0 0 -1 -1 -1 -1 -1 -1
7 0 1 0 1 0 1 6 0 0 1 0 1 4 0 -1 4 4 1 1 1 0 0 1 3 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 1 4 1 1 1 1 1 1 4 2 1 1 13 5.5 4 2 1 2 2 0 1 0 0 0 1 0 1 0 0 0 224 224 0 0 0 0 1 1 0 1 0 1 0 0 0 2 1 1 50 41 1 0 1 0 0 0 1 1 0 0 3 2 1 0 1 34 15 0 0 1 0 1 0 0 0 1 1
8 1 0 0 1 0 1 2 0 0 1 1 0 4 0 -1 4 4 2 1 1 0 0 1 3 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 1 1 1 4 1 1 1 1 4 4 1 1 14 4.5 1 1 1 1 4 0 1 0 0 0 1 0 1 0 0 0 152 152 1 1 0 0 0 1 0 1 0 1 0 0 0 2 1 0 30 30 1 0 1 0 1 0 0 1 0 0 3 1 1 0 1 22 10 0 0 1 0 1 0 0 0 1 0
9 1 1 0 1 0 1 3 0 0 1 0 1 4 0 -1 4 4 2 2 1 0 0 1 3 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 1 1 1 4 1 1 3 1 4 3 1 1 10 4 4 1 1 3 3 0 1 0 0 0 1 0 1 0 0 0 144 144 0 1 0 0 0 1 0 0 1 1 0 0 0 -1 1 0 36 36 1 0 1 0 1 0 0 1 0 0 3 1 1 0 1 23 9 1 0 1 0 1 0 0 0 1 0
10 1 0 0 1 0 1 5 0 1 0 0 1 4 1 1 4 4 2 2 1 0 1 0 3 1 0 1 1 0 1 4 4 1 4 1 1 0 0 0 0 1 1 4 1 1 1 1 1 1 4 3 10 7.5 4 1 1 3 1 0 1 0 0 0 1 0 1 0 0 0 192 192 0 0 0 1 0 0 1 1 0 1 0 0 -1 2 1 0 -1 -1 -1 0 -1 0 0 1 0 1 1 0 3 1 1 0 1 30 11 0 0 1 0 1 0 0 1 0 0
11 1 0 1 1 0 1 4 0 1 1 1 1 4 1 2 4 4 2 2 1 0 1 0 3 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 0 4 4 4 1 1 1 1 2 4 4 1 9 8 4 1 1 1 1 0 1 0 0 0 1 0 1 0 0 0 320 320 1 0 0 0 0 0 1 1 0 1 0 0 0 2 1 1 34 24 1 1 1 0 0 1 1 0 0 1 3 1 1 0 1 19 8 0 0 1 0 1 0 0 1 0 1
12 1 0 1 1 0 1 4 0 1 1 1 1 4 1 2 4 4 2 2 1 0 1 0 3 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 0 4 4 1 1 1 1 1 2 4 4 1 9 8 4 1 1 1 1 0 1 0 0 0 1 0 1 0 0 0 320 320 1 1 0 0 0 0 1 1 0 1 0 0 0 1 1 0 28 25 1 0 1 0 0 1 1 0 0 1 3 1 1 0 1 20.5 9 1 0 0 0 1 0 0 1 0 1
13 1 1 1 1 0 1 8 0 0 1 1 1 5 0 2 1 1 1 4 1 0 1 0 3 1 0 1 1 0 1 4 4 2 4 1 1 0 0 0 1 1 1 4 1 1 3 1 4 1 1 1 11 6.5 4 1 1 4 1 0 1 0 0 0 1 0 1 0 0 0 228 228 0 0 1 0 1 1 0 0 1 1 0 0 0 2 0 1 81 58.5 1 0 1 0 0 0 1 0 0 1 3 1 1 0 1 37.5 15 1 0 0 0 1 0 0 0 1 0
14 1 0 1 0 0 1 4 0 0 1 0 1 8 0 2 1 1 1 4 1 0 1 0 3 1 0 1 1 0 1 2 2 4 4 1 1 0 0 0 1 1 1 4 3 1 2 1 4 1 1 1 13 7 4 1 1 2 1 0 1 0 0 0 1 0 1 0 0 0 320 320 0 0 1 0 0 1 0 0 1 1 0 0 -1 2 1 0 -1 -1 1 -1 1 0 0 0 1 0 0 1 3 1 1 0 1 35 14 1 0 0 0 1 0 0 0 1 0
15 1 0 1 0 0 1 8 0 0 1 1 1 4.5 0 2 1 1 1 4 1 0 1 0 3 1 0 1 1 0 1 4 4 2 4 1 1 0 0 0 1 4 4 1 1 1 1 1 4 1 1 1 8 6.5 2 1 1 4 1 0 1 0 0 0 1 0 1 0 0 0 256 256 0 0 1 0 1 1 0 0 1 1 0 0 0 2 1 0 -1 -1 1 0 1 0 0 0 1 0 0 1 3 1 1 0 1 33 13.5 1 0 0 0 1 0 0 0 1 0
16 1 1 1 1 0 1 4 0 1 1 0 1 5 1 1 -1 2 2 4 1 0 0 1 3 1 1 1 1 0 1 4 4 2 1 4 1 1 5.5 5 0 1 1 1 1 1 1 1 2 2 4 1 9 7 1 1 1 1 4 0 1 0 0 0 1 0 1 0 0 0 200 200 1 0 0 1 0 1 1 1 1 1 0 0 0 2 1 0 31 31 1 1 1 0 0 1 1 1 0 0 3 1 1 0 1 23.5 10 0 0 1 0 1 0 0 1 0 1
17
0 1 0 1 0 1 10 0 0 1 0 1 3.5 0 2 1 4 4 1 1 0 1 0 1 0 1 0 1 0 1 4 4 1 1 4 1 1 5 4.5 0 1 1 1 1 1 1 1 1 4 4 1 9 10 4 2 1 2 4 0 1 0 0 0 1 0 1 0 0 0 160 160 0 0 0 1 0 0 1 0 1 1 0 0 0 1 1 1 1 1 1 1 1 0 0 0 1 1 0 1 3 1 1 0 1 24.5 10.5 0 0 0 1 1 -1 --1 --1 --1 --1
18
1 1 0 1 0 1 2 1 0 0 0 1 3 0 -1 4 4 3 3 0 1 1 0 3 1 0 1 1 0 1 4 4 1 4 1 1 0 0 0 0 1 4 4 1 1 1 1 1 4 4 1 7 7 4 1 1 1 1 1 1 0 0 0 1 0 1 0 0 0 145 145 1 1 0 1 0 0 1 1 0 1 0 0 0 1 1 1 1 1 1 1 1 0 0 0 1 1 0 1 3 1 1 0 1 24.5 10.5 0 0 0 1 1 1 1 -1 --1 --1
19
1 1 1 1 0 1 6 0 0 1 0 1 5 0 -1 4 4 3 3 1 0 0 1 3 1 1 1 1 0 1 4 4 2 1 4 1 1 5.5 5 0 1 4 4 1 1 1 1 4 3 1 1 7.5 8.5 1 4 4 1 3 0 1 0 0 0 1 0 1 0 0 0 220 220 1 1 0 0 0 1 1 1 1 1 0 0 0 2 1 0 42.5 34 1 0 1 0 0 0 1 1 0 0 3 1 1 0 1 22 11 0 0 0 1 1 0 0 1 0 0
20
1 1 1 1 0 0 4 0 1 0 1 0 4 0 -1 4 4 3 1 1 0 0 1 3 1 1 1 1 0 1 4 4 1 1 4 1 1 7.5 3.5 1 1 1 1 1 1 1 1 2 2 4 1 8 7.5 1 4 1 4 4 0 1 0 0 0 1 0 1 0 0 0 136 136 0 0 0 0 0 1 0 0 1 1 0 0 0 2 1 0 27 23 1 1 1 0 1 0 0 1 0 0 3 1 1 0 1 17.5 8.5 0 0 1 0 1 1 1 1 0 0
21
1 0 0 1 1 1 4 0 0 1 1 1 4.5 0 -1 4 4 3 3 1 0 0 1 3 1 0 1 1 0 1 4 4 1 4 1 1 0 0 0 1 4 4 4 1 1 1 1 4 1 1 1 8.5 8.5 4 2 1 2 1 1 0 0 0 0 0 1 1 0 0 0 240 240 1 0 0 1 0 1 0 1 0 1 0 0 0 2 1 1 43 29 1 1 1 0 1 0 0 1 0 0 3 1 1 1 1 25.5 11 0 1 0 0 1 0 0 0 1 0
22 1 1 0 1 0 0 -1 0 0 1 1 0 4.5 0 -1 1 1 4 4 1 0 0 1 3 1 0 1 1 0 1 4 4 1 4 1 1 0 0 0 1 4 4 1 1 1 1 1 4 1 1 1 7 7 4 1 1 1 1 1 1 0 0 0 1 1 1 0 0 0 160 160 1 0 0 1 0 1 0 1 0 0 0 1 -1 2 1 0 24 21 0 0 1 0 1 0 0 1 0 0 3 1 1 1 1 20 8.5 1 0 0 0 1 1 0 0 1 1
23 1 1 -1 -1 1 1 5 0 1 0 1 0 5 0 -1 4 4 3 1 1 0 0 1 3 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 1 4 4 4 1 1 1 1 4 4 1 1 8 7 4 3 1 3 1 1 1 0 1 0 0 1 1 0 0 0 200 200 1 0 1 0 0 1 1 1 1 1 0 0 0 2 1 0 42 34 1 0 1 0 0 0 1 1 0 0 3 2 1 0 1 24.5 11.5 0 1 0 0 1 1 0 0 1 0
24
1 0 1 1 0 1 8 0 0 1 1 1 4 0 -1 2 2 4 4 1 0 0 1 3 1 1 0 1 0 1 4 4 1 4 1 1 0 0 0 1 3 3 3 1 1 1 1 4 1 1 1 6.5 5 4 2 2 1 1 1 1 0 0 0 0 1 1 1 0 0 288 156 1 1 0 0 0 -1 -1 0 0 0 1 1 -1 1 0 1 44 24 1 1 1 0 1 0 0 1 0 0 3 1 1 0 1 18.5 16.5 0 1 0 0 0 1 0 0 0 1
25
1 1 1 1 0 1 10 0 0 1 1 0 5 0 -1 2 2 4 4 1 0 1 0 3 1 0 1 1 0 1 3 3 3 4 1 1 0 0 0 1 1 4 4 1 1 3 1 4 3 1 1 7.5 6.5 4 2 1 2 1 0 1 1 0 0 1 0 1 0 0 0 210 210 1 1 0 1 0 1 0 0 1 1 0 0 0 2 1 0 45 45 1 0 1 0 0 0 1 0 0 1 3 1 1 0 1 29.5 13 1 0 1 0 1 1 0 0 1 0
26 1 1 1 1 1 1 4 0 0 1 1 0 3.5 1 1 2 2 4 4 1 0 1 0 2 1 1 0 1 0 1 4 4 1 4 1 1 0 0 0 1 4 2 2 1 1 1 1 4 1 1 1 6.5 7 4 2 1 2 1 0 1 0 1 0 0 0 1 0 0 0 132 132 0 1 0 0 0 1 0 1 0 1 0 0 0 2 1 0 35 30 1 1 -1 -1 1 0 0 0 1 0 1 3 1 0 0 17.5 7 1 0 0 0 1 1 0 0 0 1
27 1 1 1 1 0 1 2 0 1 0 1 1 4 0 2 2 2 4 4 1 0 1 0 2 1 1 1 1 0 1 4 4 2 1 4 1 0 3.5 5 1 1 2 1 1 1 1 1 2 3 4 1 7 6.5 1 1 4 4 3 0 1 0 0 0 1 0 1 0 0 0 160 160 0 1 0 1 0 0 1 1 0 1 0 0 1 2 1 0 28 25 1 1 1 0 1 0 1 1 0 0 3 1 1 0 0 18.5 8 1 0 1 0 1 0 0 1 0 1
28
Lanjutan
Taxa Number Coding of Each Character State (1-116)
29
1 1 1 1 0 1 5 0 0 1 1 0 7 1 -1 4 4 2 2 1 0 1 0 3 1 0 1 1 0 1 4 4 3 4 1 1 0 0 0 1 1 1 4 1 1 3 1 4 3 1 1 15 9.5 4 1 3 3 1 0 1 1 0 0 0 1 1 0 0 0 320 320 0 0 0 1 0 -1 -1 -1 -1 1 0 0 0 2 0 1 66 44 1 1 1 0 0 0 1 0 0 1 3 1 1 0 1 31 14 1 0 0 0 1 0 0 1 0 0
30 1 1 1 0 1 0 3 0 0 1 1 0 4.5 0 -1 2 2 4 4 0 1 0 1 3 1 0 1 1 0 1 4 4 1 4 1 1 0 0 0 0 1 1 1 1 1 1 1 1 1 4 4 9 10 4 1 2 1 1 0 0 1 0 0 1 1 1 0 0 0 120 120 0 0 1 0 0 1 0 0 1 1 0 0 0 2 1 0 31 31 0 0 1 0 0 0 1 0 0 1 3 1 1 0 1 15 10 1 0 0 0 1 0 0 1 0 1
31
1 0 0 1 0 1 5 1 0 0 1 0 5.5 0,-1 2 2 4 1 1 0 1 0 3 1 0 1 1 0 1 4 4 3 4 1 1 0 0 0 1 1 1 4 1 1 3 1 4 1 1 1 13 7 4 2 1 2 3 0 0 1 1 0 0 0 1 0 0 0 320 320 0 0 0 0 1 0 1 0 1 1 0 0 0 -1 -1 -1 -1 -1 -1 -1 -1 -1 0 0 1 1 0 0 2 2 1 0 1 36 11 1 0 0 0 1 0 0 0 1 0
32
0 1 0 1 0 1 8 1 0 0 1 0 3 1 -1 4 4 2 2 1 0 1 0 3 1 1 0 1 0 1 4 4 1 4 1 1 0 0 0 0 1 1 4 1 1 1 1 4 4 4 1 11 8.5 3 1 1 4 1 0 1 0 0 0 1 0 1 0 0 0 176 176 0 0 0 1 1 1 0 0 1 1 0 0 0 2 1 1 38 30 1 1 1 0 0 0 1 0 0 1 3 2 1 0 1 23.5 10.5 0 0 0 1 1 1 1 -1 --1 --1
33 1 1 1 1 0 1 3 0 0 1 1 0 3 1 1 2 2 4 1 1 0 1 1 3 1 0 1 1 0 1 4 4 2 4 1 1 0 0 0 1 1 1 4 1 3 3 1 4 1 1 1 9.5 4.5 4 1 1 1 1 1 1 0 0 0 1 0 1 0 0 0 160 160 1 0 1 0 1 1 1 0 1 1 0 0 0 2 1 1 28 24 1 0 1 0 0 1 0 1 0 0 3 2 1 0 1 16.5 6.5 1 0 1 0 1 1 1 0 0 1
34
1 1 0 1 0 1 8 1 0 0 0 1 4 1 -1 4 4 3 1 1 0 1 0 3 1 1 0 1 0 1 4 4 1 2 4 1 1 0 0 0 4 3 1 1 1 1 1 4 3 1 1 10 7 4 2 1 2 1 1 1 0 0 0 1 0 1 0 0 0 80 80 1 0 0 0 0 0 1 1 0 1 0 0 1 1 1 0 22.5 22.5 1 0 1 0 0 1 0 0 1 0 3 1 1 0 1 14.5 7 0 0 1 0 1 1 1 1 -1 --1
35 1 1 1 0 0 1 3 0 1 0 1 1 3.5 0 -1 4 4 2 2 1 0 1 1 3 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 1 1 1 4 1 1 1 1 4 3 1 1 12 6 4 2 2 2 2 1 1 0 0 0 1 0 1 0 1 0 255 255 1 1 1 1 1 0 1 0 1 1 0 0 0 2 1 0 48 44 1 0 1 1 1 0 1 1 0 0 3 2 1 0 1 29 11.5 1 0 0 1 0 1 0 0 1 1
36
1 1 1 1 1 1 7 0 1 0 1 1 3.5 0 -1 3 3 4 4 1 0 0 1 3 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 1 4 1 1 1 1 2 1 4 1 1 1 10.5 11.5 4 2 1 2 1 0 0 1 1 1 0 0 1 1 0 0 330 330 1 1 0 0 0 1 0 0 0 1 0 1 0 2 1 1 72 36 1 1 1 0 0 0 1 1 0 0 3 2 1 1 1 29 14 1 0 0 0 1 1 1 1 0 1
37
1 0 0 1 -1 -1 5 0 0 1 0 1 5 1 -1 4 4 2 2 1 0 1 0 3 1 1 0 1 0 1 4 4 1 1 4 1 1 4.5 5 1 1 4 1 1 1 1 1 4 2 1 1 6 6 4 2 1 2 1 1 1 0 0 0 1 0 1 0 0 0 144 144 1 1 0 1 0 1 0 1 0 1 0 0 0 1 1 0 42.5 36 1 0 1 0 0 1 0 1 0 0 3 1 1 0 1 21.5 10 0 0 1 0 1 0 0 0 0 1
38 1 0 0 1 -1 -1 5 0 0 1 0 1 5 1 -1 4 4 2 2 1 0 1 0 3 1 1 0 1 0 1 4 4 1 1 4 1 1 4.5 5 1 1 1 4 1 1 2 1 4 2 1 1 8.5 6.5 1 1 1 4 3 1 0 0 0 0 1 0 1 0 0 0 152 152 1 0 1 0 0 1 0 0 1 1 0 0 0 1 1 0 33 30 1 0 1 0 0 0 1 0 0 1 3 1 1 0 1 23 9 0 0 0 1 1 1 0 1 0 1
39 1 0 0 1 0 1 4 0 0 1 0 1 4 1 1 4 4 2 2 1 0 1 0 3 1 1 1 1 0 1 4 4 1 1 4 1 1 5.5 5 0 1 1 4 1 1 1 1 1 1 4 1 7 11 4 1 1 2 1 0 1 0 0 0 1 0 1 0 0 0 192 192 1 1 0 0 0 0 1 0 1 1 0 0 0 1 1 0 33 30 1 0 1 0 0 0 1 0 0 1 3 1 1 0 1 23 9 0 0 0 1 1 1 0 1 0 1
40
1 0 0 1 0 1 10 1 0 0 1 1 6.5 1 -1 4 4 2 2 1 0 1 0 3 1 1 0 1 0 1 4 4 1 4 1 1 0 0 0 1 4 1 1 1 1 1 1 4 1 1 1 7.5 9.5 4 1 1 1 1 1 1 0 0 0 1 0 1 0 0 0 200 200 0 0 0 1 0 -1 -1 -1 -1 1 0 0 0 3 1 0 41 38 1 0 1 0 0 1 0 1 0 0 3 1 1 0 1 24.5 12 1 0 0 0 1 0 0 1 0 1
41 1 0 0 1 0 1 6 1 0 0 1 0 3 1 1 2 2 4 1 1 0 1 0 3 1 1 0 1 0 1 4 4 1 4 3 1 0 6 4.5 0 4 1 3 1 1 1 1 4 3 1 1 6 4.5 4 1 1 1 1 0 1 0 0 0 1 0 1 0 0 0 106 106 0 1 0 1 0 0 1 1 0 1 0 0 0 1 1 0 30 26 1 0 1 0 1 0 0 0 1 0 3 2 1 0 1 18 8.5 1 0 0 0 1 0 0 0 0 1
42 1 1 1 1 0 1 7 1 1 1 1 1 4.5 0 2 4 4 2 2 1 0 1 0 3 1 1 1 1 0 1 4 4 2 1 4 1 1 4.5 5 0 1 1 4 1 1 1 1 4 2 2 1 9 7.5 2 1 4 4 2 1 1 0 0 0 1 0 1 0 0 0 190 190 1 1 0 1 0 0 1 0 1 1 0 0 0 2 1 0 31 31 1 1 1 0 0 0 1 1 0 0 3 2 1 0 1 21 11 0 0 0 1 1 1 0 1 0 1
43
1 1 1 1 1 1 -1 0 1 0 1 1 5 0,-1 2 2 4 1 1 0 1 0 2 0 0 1 1 0 1 4 4 1 4 1 1 0 0 0 1 1 4 1 1 1 1 1 4 2 1 1 11 9.5 4 2 1 2 1 0 1 0 1 1 0 0 1 0 0 0 240 240 1 0 0 0 0 1 0 0 1 1 0 0 -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 0 0 1 0 0 1 3 2 1 0 1 37 17 0 0 0 1 1 0 0 0 1 0
44
0 1 1 1 0 1 12 0 0 1 1 1 4.5 1 -1 4 4 2 2 1 0 1 0 3 1 1 1 1 0 1 4 4 2 4 1 1 0 0 0 0 4 1 4 1 1 2 1 4 2 2 1 10 8.5 4 1 1 1 1 0 1 0 1 0 1 0 1 0 0 0 280 280 1 0 1 0 0 0 1 0 1 1 0 0 0 2 1 0 39 39 1 -1 1 0 0 0 1 0 0 1 3 1 1 0 1 29.5 14 0 0 0 1 1 0 0 1 0 0
45 0 1 1 1 0 1 8 0 0 1 0 1 4 1 1 4 4 2 1 1 0 1 0 1 0 1 1 1 0 1 4 4 2 4 1 1 0 0 0 0 1 1 1 1 1 1 1 1 4 4 1 9 8.5 4 1 2 1 1 0 1 0 1 0 1 0 1 0 0 0 260 260 1 0 0 0 0 0 1 0 1 1 0 0 0 2 1 0 35 35 1 -1 1 0 0 0 1 0 0 1 3 1 1 0 1 23.5 11.5 0 0 0 1 1 1 0 1 0 1
46
1 0 1 1 0 1 8 0 0 1 1 1 5.5 0 -1 2 2 4 1 1 0 0 1 3 1 1 1 1 0 1 2 2 4 4 1 1 0 0 0 1 1 4 1 1 1 2 1 4 1 1 1 8.5 7.5 4 2 1 2 1 0 1 0 1 1 0 0 1 0 0 0 300 300 1 1 0 0 0 1 0 0 1 1 0 0 0 2 1 0 42 41 1 1 1 0 0 0 1 0 0 1 3 1 1 0 1 26 14.5 0 0 1 0 1 1 1 1 1 0
47
1 0 0 1 0 1 6 0 0 1 0 1 4.5 0 -1 2 2 4 4 1 0 1 0 3 1 1 1 1 0 1 4 4 1 4 1 1 0 0 0 1 1 1 4 1 1 1 1 4 2 1 1 11.5 5 4 2 1 2 2 0 1 0 0 0 1 0 1 0 0 0 160 160 1 0 0 0 0 1 0 0 1 1 0 0 0 1 1 0 30 26 1 1 1 0 0 0 1 0 0 1 3 1 1 0 1 31.5 10 0 0 0 1 1 0 0 1 0 0
48
0 1 0 1 0 1 3 0 0 1 1 0 5.