DINAMIKA DAN INTERAKSI SOLITON DNA
MODEL PEYRARD-BISHOP-DAUXOIS
SWITENIA WANA PUTRI
DEPARTEMEN FISIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Switenia Wana Putri. Dinamika dan Interaksi Soliton DNA
Model
Peyrard-Bishop-Dauxois.
Dibimbing oleh
: Dr. Husin Alatas.
Abstrak
Soliton DNA model PBD menggambarkan dinamika peregangan (denaturasi) pada ikatan hidrogen antar nukleotida dalam rantai DNA yang berbeda yang direpresentasikan oleh potensial Morse. Dengan menggunakan metode beda hingga (finite-difference) dibantu dengan interpolasi Lagrange, maka diperoleh solusi numerik dari dinamika dan interaksi soliton DNA model PBD dengan memberikan gangguan pada solusi stabilnya. Hasil yang diperoleh menunjukkan bahwa solusi numerik dari dinamika dan interaksi soliton DNA model PBD yang diberikan efek gangguan akan menunjukkan hasil yang berbeda dengan solusi eksak tanpa gangguan. Ada tiga gangguan yang diberikan pada solusi stabil ini. Gangguan pertama dan kedua berupa gangguan pada amplitudo menunjukkan bahwa soliton DNA yang pada awalnya stabil mengalami peristiwa undulasi, dimana terjadi penyempitan soliton dan kenaikan amplitudo. Pada gangguan ketiga diberikan gangguan untuk tiga beda fase yang berbeda, dimana akan terbentuk dua soliton yang mengalami superposisi dengan interaksi antar soliton yang berbeda-beda pada setiap beda fasenya. Untuk beda fase θ=0, kedua soliton akan saling tarik menarik, lalu kedua soliton akan saling tolak menolak dengan perubahan asimetrik pada θ=�
2, dan saling tolak menolak dengan perubahan simetrik pada θ=�.
Kata kunci: soliton DNA, PBD, denaturasi, metode beda hingga, interpolasi Lagrange.
Judul : Dinamika dan Interaksi Soliton DNAModel Peyrard-Bishop-Dauxois
Nama : Switenia Wana Putri
NRP : G74070026
Menyetujui,
Pembimbing
Dr. Husin Alatas
NIP : 19710604 199802 1 001
Mengetahui,
Ketua Departemen Fisika FMIPA IPB
Dr. Akhiruddin Maddu
NIP : 19660907 199802 1 000
DINAMIKA DAN INTERAKSI SOLITON DNA
MODEL PEYRARD-BISHOP-DAUXOIS
Switenia Wana Putri G74070026
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains pada Fakultas Matematika dan Ilmu Pengetahuan Alam
Institut Pertanian Bogor
DEPARTEMEN FISIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
i
KATA PENGANTAR
Puji syukur penulis panjatkan ke hadirat Allah swt. karena berkat rahmat, kasih sayang, dan karunia yang tak terhingga dari-Nya penulis dapat menyelesaikan penulisan laporan tugas akhir yang berjudul “Dinamika dan Interaksi Soliton DNAModel Peyrard-Bishop-Dauxois”. Laporan ini disusun sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains pada Fakultas Matematika dan Ilmu Pengetahuan Alam Institut Pertanian Bogor. Dalam skripsi ini tertuang penjelasan tentang hasil penelitian tugas akhir yang telah dilakukan oleh penulis dalam beberapa bulan ke belakang.
Penulis juga ingin menyampaikan ucapan terima kasih kepada pihak-pihak yang telah membantu dalam penyelesaian penulisan usulan penelitian ini, yaitu kepada : 1. Bapak Dr. Husin Alatas selaku dosen pembimbing yang telah memberikan
bimbingan, masukan, dan semangat kepada penulis.
2. Ibu Mersi Kurniati, M.Si dan Bapak Dr. Ir. Irmansyah selaku dosen penguji serta staf dosen Departemen Fisika FMIPA IPB lainnya yang telah memberikan bimbingan, masukan, dan ilmu-ilmu yang bermanfaat selama penulis menempuh pendidikan S1 ini.
3. Mama dan papa selaku orang tua, adik Erin dan Melly, serta keluarga besar di Palembang yang selalu memberi dorongan baik secara materi maupun spiritual. 4. Izzatu Yazidah dan Dede Hermanudin sebagai partners satu topik penelitian ini,
terima kasih untuk bantuan dan dukungan yang luar biasa dari kalian.
5. Keluarga di rumah kedua “Arsida 4”, Erna Piantari, Mery Purnamasarie, Hesti Paramita Sari, dan Rithoh Yahya. Thanks for being so lovely and take care of me here.
6. Teman-teman seperjuangan di Fisika 44. 7. Keluarga besar Departemen Fisika FMIPA IPB.
8. Teman-teman Ikamusi IPB, teman-teman dari TK sampai SMA, dan teman-teman IPB lainnya yang tidak dapat disebutkan satu per satu, terima kasih untuk doa dan semangatnya.
Penulis menyadari bahwa skripsi ini masih jauh dari sempurna. Oleh karena itu, saran dan kritik yang bersifat membangun sangat diperlukan bagi penulis. Semoga tulisan ini bermanfaat bagi semuanya.
Bogor, Juli 2012
ii
DAFTAR RIWAYAT HIDUP
Penulis dilahirkan di Palembang pada tanggal 16 Desember 1989 dari
pasangan Isman Prabujaya dan Ilya Rosdiana. Penulis menyelesaikan
pendidikan Taman Kanak-Kanak hingga Sekolah Menengah Atas di
Sumatera Selatan, yaitu TK Tri Daqa Kartika II Sriwijaya Karang
Endah, SDN 1 Karang Endah, SMP Negeri 2 Gelumbang, dan SMA
Negeri 1 Prabumulih. Pada tahun 2007, penulis diterima menjadi
mahasiswa IPB di Departemen Fisika Fakultas Matematika dan Ilmu Pengetahuan Alam
melalui jalur Undangan Seleksi Masuk IPB (USMI). Selain aktif mengikuti kegiatan
akademik, penulis juga aktif di beberapa organisasi kemahasiswaan di IPB seperti di
Ikatan Keluarga Mahasiswa Bumi Sriwijaya, Himpunan Mahasiswa Fisika, Komunitas
Seni Budaya Masyarakat Roempoet, dan dalam kepanitiaan pada beberapa acara yang
diselenggarakan oleh mahasiswa IPB. Penulis juga berkesempatan menjadi asisten
praktikum mata kuliah Fisika Dasar TPB IPB dan mata kuliah Eksperimen Fisika II di
iii
DAFTAR ISI
Halaman
DAFTAR GAMBAR ... iv
DAFTAR LAMPIRAN ... vi
BAB 1 PENDAHULUAN ... 1
1.1 Latar Belakang ... 1
1.2 Tujuan Penelitian ... 1
1.3 Perumusan Masalah ... 2
1.4 Hipotesis ... 2
BAB 2 TINJAUAN PUSTAKA ... 2
2.1 Deoxyribose Nucleic Acid (DNA) ... 2
2.2 Solusi Soliton DNA Model PBD Teraproksimasi ... 3
BAB 3 METODE PENELITIAN ... 7
3.1 Tempat dan Waktu Penelitian ... 7
3.2 Peralatan ... 7
3.3 Metode Penelitian ... 7
3.3.1 Studi Pustaka ... 8
3.3.2 Pembuatan Program Simulasi ... 8
3.3.2.1 Metode Beda Hingga (Finite-Difference) ... 8
3.3.2.2 Interpolasi Lagrange ... 9
3.3.3 Skala Ulang Parameter ... 11
BAB 4 HASIL DAN PEMBAHASAN ... 12
4.1 Simulasi Perambatan Soliton akibat Gangguan pada Amplitudo ... 13
4.2 Simulasi Interaksi Dua Buah Soliton ... 15
BAB 5 KESIMPULAN ... 18
SARAN – SARAN ... 19
DAFTAR PUSTAKA ... 19
iv
DAFTAR GAMBAR
Halaman
Gambar 1. (a) Struktur untai komplementer DNA yang membentuk DNA
beruntai ganda ... 2
(b) Struktur molekul DNA. Atom karbon berwarna hitam, oksigen merah, nitrogen biru, fosfor hijau, dan hidrogen putih ... 2
Gambar 2. Proses replikasi DNA ... 3
Gambar 3 Solusi soliton pada DNA model PBD... 4
Gambar 4. Representasi grafis model pegas sederhana untuk rantai DN... 4
Gambar 5. Kurva suatu fungsi f(x) yang dibagi sama besar berjarak h ... 8
Gambar 6. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 stabil ... 12
(a) profil soliton DNA dalam tiga dimensi ... 12
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik berwarna merah menunjukkan grafik pada saat Tawal, dan grafik biru menunjukkan grafik pada saat Takhir ... 12
Gambar 7. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 Gangguan I ... 13
(a) profil soliton DNA dalam tiga dimensi ... 13
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik berwarna merah menunjukkan grafik pada saat Tawal, grafik biru menunjukkan grafik pada saat Takhir, dan grafik hijau menunjukkan saat terjadinya undulasi ... 13
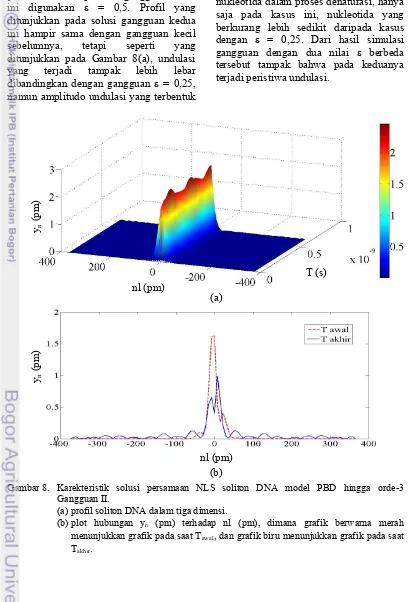
Gambar 8. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 Gangguan II ... 14
(a) profil soliton DNA dalam tiga dimensi ... 14
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik berwarna merah menunjukkan grafik pada saat Tawal, dan grafik biru menunjukkan grafik pada saat Takhir ... 14
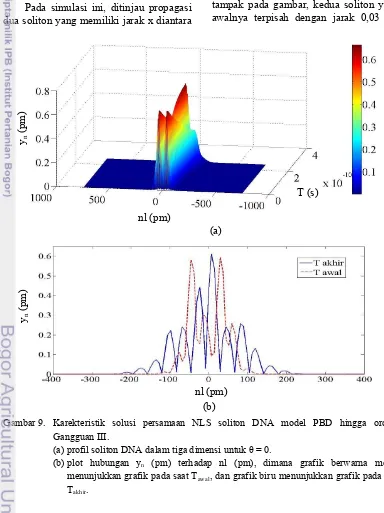
Gambar 9. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 Gangguan III ... 15
v
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik
berwarna merah menunjukkan grafik pada saat Tawal, dan
grafik biru menunjukkan grafik pada saat Takhir ... 15
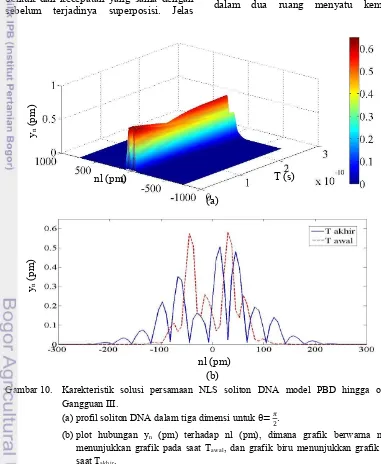
Gambar 10. Karekteristik solusi persamaan NLS soliton DNA model PBD
hingga orde-3 Gangguan III ... 16
(a) profil soliton DNA dalam tiga dimensi untuk θ = �
2 ... 16
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik
berwarna merah menunjukkan grafik pada saat Tawal, dan
grafik biru menunjukkan grafik pada saat Takhir ... 16
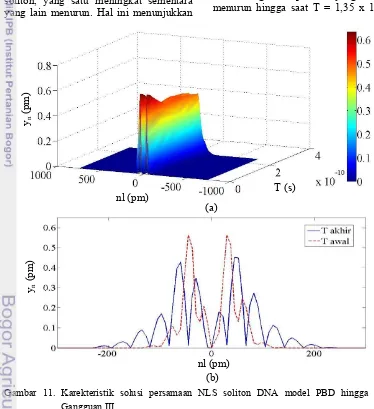
Gambar 11. Karekteristik solusi persamaan NLS soliton DNA model PBD
hingga orde-3 Gangguan III ... 17
(a) profil soliton DNA dalam tiga dimensi untuk θ = π ... 17 (b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik
berwarna merah menunjukkan grafik pada saat Tawal, dan
vi
DAFTAR LAMPIRAN
Halaman
Lampiran A. Diagram Alir Penelitian ... 22
Lampiran B. SkalaUlang Parameter Persamaan NLS Tahap 1 ... 23
Lampiran C. Skala Ulang Parameter Persamaan NLS Tahap 2 ... 23
BAB 1
PENDAHULUAN
1.1 Latar Belakang
Dinamika transkripsi Deoxyribo Nucleic Acid (DNA) adalah salah satu gejala fisis yang paling menarik dalam biofisika modern karena terkait dengan dasar kehidupan.1 Pada tahun-tahun terakhir gagasan bahwa eksitasi nonlinier memainkan peran dalam dinamika DNA telah menjadi semakin populer, sehingga selanjutnya semakin banyak penelitian-penelitian terkait dengan dinamika soliton DNA ini sendiri, seperti penelitian Englander et al2, Yomosa3, Takeno dan Homma4, Peyrard-Bishop1, Peyrard-Bishop-Dauxois5, Zhang6 dan lain-lain. Ini merupakan suatu hal yang sangat menarik, menelisik bagaimana “cetak biru” ini dipandang dari sudut pandang seorang fisikawan, tidak hanya di bidang Biologi atau Biokimia.
Banyak penelitian Fisika berkaitan dengan gerak internal amplitudo besar DNA sampai pada kesimpulan bahwa molekul dapat ditinjau sebagai sistem dinamika nonlinier yang terkait dengan gejala soliton.1-6 Struktur heliks ganda DNA mengalami dinamika yang sangat kompleks. Pengetahuan tentang dinamika tersebut dapat memberikan wawasan yang dalam mengenai berbagai fenomena biologis terkait seperti transkripsi, translasi dan mutasi. Diyakini dengan pemahaman akan mekanisme organisme elementer (DNA), kelak diharapkan fenomena makroskopis organisme hidup bisa dijelaskan dan diprediksi dengan akurat dan mudah. Permasalahan utama dalam biofisika sendiri adalah bagaimana menghubungkan sifat fungsional DNA dengan struktur dan karakteristik fisik dinamik-nya. Dari sudut pandang fisikawan, molekul DNA tak lain adalah sistem yang terdiri dari banyak atom berinteraksi, tersusun dengan cara tertentu dalam ruang.7 Sebuah hirarki dari model yang paling penting bagi dinamika nonlinier DNA disajikan oleh Yakushevich.8
Salah satu model dinamika DNA telah diajukan oleh Peyrard dan Bishop1 yang kemudian dikembangkan oleh Dauxois.9,10 Selanjutnya dalam laporan tugas akhir ini, model tersebut disingkat menjadi model PBD. Model PBD terutama ditujukan untuk menggambarkan proses peregangan pasangan basa DNA (denaturasi). Berbeda dengan dua paper Peyrard-Bishop11 sebelumnya, dalam model PBD juga diperhitungkan efek helikoidal yang menggambarkan interaksi antara dua rantai DNA akibat struktur heliks.
Soliton DNA model PBD menggambarkan dinamika peregangan pada ikatan hidrogen antar nukleotida dalam rantai yang berbeda yang direpresentasikan oleh potensial Morse, yang terlokalisasi dan bersifat stabil. Dalam model pendekatan potensial Morse hingga orde 4 dikendalikan oleh persamaan Nonlinear Schrodinger (NLS).12 Profil sebaran rapat energinya menyerupai gundukan yang terpusat dalam rentang ruang berhingga. Setiap soliton dicirikan oleh sifat tidak berubahnya topologi yang menunjukkan sifat kestabilannya. Pada penelitian ini, dilakukan analisis numerik yaitu membuat simulasi model menggunakan pemrograman MATLAB dari solusi analitik persamaan NLS yang telah diperoleh pada penelitian Hermanudin12 sebelumnya. Untuk menentukan solusi numerik dari persamaan NLS ini, metode yang digunakan adalah metode beda hingga (finite- difference) dibantu dengan interpolasi Lagrange.
1.2 Tujuan Penelitian
Gambar 1.13 (a) Struktur untai komplementer DNA yang membentuk DNA beruntai ganda.
(b) Struktur molekul DNA. Atom karbon berwarna hitam, oksigen merah, nitrogen biru, fosfor hijau, dan hidrogen putih.
1.3
Perumusan Masalah
Pada penelitian sebelumnya, telah diperoleh solusi analitik untuk dinamika DNA model PBD hingga orde ke-4.12 Lalu ada penelitian ini akan ditinjau pemecahan solusi numerik persamaan soliton DNA model PBD dengan memberikan efek gangguan menggunakan metode beda hingga untuk dinamika DNA model PBD hingga orde ke-3.
1.4
Hipotesis
Solusi numerik yang akan diperoleh dari dinamika dan interaksi DNA model PBD yang diberikan efek gangguan akan menunjukkan hasil yang berbeda dengan solusi eksak tanpa gangguan.
BAB 2
TINJAUAN PUSTAKA
2.1
Deoxyribose Nucleic Acid
(DNA)
DNA atau Deoxyribo Nucleic Acid merupakan substansi penurunan sifat yang erat kaitannya dengan hampir semua aktivitas biologi. DNA adalah asam nukleotida, biasanya dalam bentuk heliks ganda yang mengandung instruksi genetik yang menentukan perkembangan biologis dari seluruh bentuk kehidupan sel. DNA berbentuk polimer panjang nukleotida, mengkode barisan residu asam amino
dalam protein dengan menggunakan kode genetik, sebuah kode nukleotida triplet.13 DNA pada pengertian sempit adalah suatu cetakan untuk membuat protein.14 DNA membawa informasi genetik sel dan terdiri dari ribuan gen. Setiap gen berfungsi sebagai cetakan untuk membangun sebuah molekul protein. Protein melakukan tugas-tugas penting untuk fungsi sel atau berfungsi sebagai blok bangunan. Aliran informasi dari gen ini menentukan komposisi protein dan fungsi dari sel itu sendiri.15
DNA tersusun sebagai untai komplementer dengan ikatan hidrogen di antara mereka. Masing-masing untai DNA
adalah rantai kimia „batu bata penyusun‟,
yakni nukleotida, yang terdiri dari empat tipe: Adenine (A), Cytosine (C), Guanine (G) dan Thymine (T).13 DNA mengandung informasi genetika yang diwariskan oleh keturunan dari suatu organisme; informasi ini ditentukan oleh barisan pasangan basa. Sebuah untai DNA mengandung gen,
sebagai „cetak biru‟ organisme. Rantai DNA memiliki lebar 22-24 Å, sementara panjang satu unit nukleotida 3,3 Å.13 Struktur komplementer dan molekul DNA ditunjukkan oleh Gambar 1.
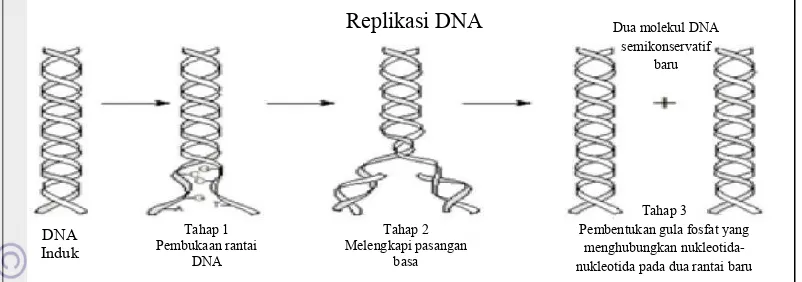
Replikasi DNA adalah proses penggandaan rantai ganda DNA. Proses replikasi DNA ini ditunjukkan oleh Gambar 2.
Dua molekul DNA semikonservatif
baru
3
Gambar 2. Proses Replikasi DNA.16
Satu pengamatan penting yang menuntun Watson dan Crick ke penemuan terkenal dari struktur heliks ganda DNA adalah basa cenderung berpasangan melalui ikatan hidrogen dan pasangan yang terbentuk oleh purin dan pirimidin memiliki ukuran yang hampir sama sehingga pasangan-pasangan demikian membentuk struktur dua rantai nukleotida berikatan hidrogen yang sangat teratur. Barisan pasangan basa dalam DNA mengkode informasi untuk mensintesa protein, adalah polimer yang terdiri dari 20 asam amino berbeda. Ikatan hidrogen pada pasangan basa dalam DNA ini pada suatu saat dapat mengalami pemutusan yang disebut dengan proses denaturasi. Selama replikasi DNA, yang terjadi selama pembelahan sel, misalnya molekul disalin dengan cara membukanya seperti ritsleting.Transkripsi DNA adalah pembacaan gen tunggal untuk mensintesa protein.5
Pada sel, replikasi DNA terjadi sebelum pembelahan sel. Sel prokariot terus-menerus melakukan replikasi DNA, sedangkan sel eukariot waktu terjadinya replikasi DNA sangat teratur, yaitu pada fase S daur sel, sebelum mitosis atau meiosis I. Penggandaan tersebut memanfaatkan enzim DNA polimerase yang membantu pembentukan ikatan antara nukleotida-nukleotida penyusun polimer DNA. Proses replikasi DNA dapat pula dilakukan in vitro dalam proses yang disebut reaksi berantai polimerase (PCR). Replikasi DNA dikatakan semikonservatif
jika masing-masing strand DNA berlaku sebagai template untuk sintesa rantai baru, dua molekul DNA baru akan dihasilkan, masing-masing dengan satu rantai baru dan satu rantai lama. Proses ini disebut replikasi semikonservatif. 17
2.2 Solusi Soliton DNA Model PBD
Teraproksimasi
Untuk mendeskripsikan gerak internal DNA dimulai dengan memilih model aproksimasi yang bersesuaian. Biasanya pilihan model bergantung pada masalah yang ditinjau dan akurasi deskripsi yang diperlukan.18 Dalam hal ini untuk gejala denaturasi DNA digunakan model PBD. Sedang untuk mempelajari dinamika model nonlinier DNA model PBD tersebut, diperlukan suatu algoritma yang mencakup beberapa unsur, yaitu memilih gerakan yang dominan, mengkonstruksikan persamaan diferensial parsial terkait dan mencari solusi untuk persamaan, serta menginterpretasikan solusi yang diperoleh.8
Gejala denaturasi pada dinamika DNA ditunjukkan oleh adanya solusi soliton. Representasi grafis dari solusi persamaan gerak yang memiliki solusi soliton ditunjukkan oleh Gambar 3 yang ditafsirkan sebagai keadaan peregangan untai ganda DNA. Dalam dinamika replikasi DNA, DNA diasumsikan mengalami tiga gerakan, yaitu gerak longitudinal (searah sumbu x), gerak bending (searah sumbu y), dan gerak memutar (torsional). Tahap 1 Pembukaan rantai DNA DNA Induk Tahap 2 Melengkapi pasangan basa Tahap 3 Pembentukan gula fosfat yang
menghubungkan nukleotida-nukleotida pada dua rantai baru
2 2 � − 2 −1 − 2 (6)
= +1+ −1−2 −
+ℎ+ −ℎ + 2
4
4
Gambar 3. Solusi soliton pada DNA model PBD
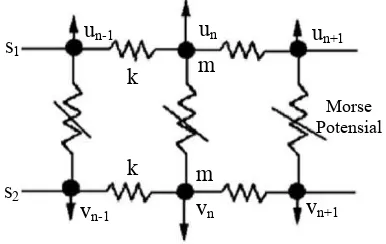
Bentuk B-DNA dalam model Watson-Crick merupakan heliks ganda, terdiri atas dua alur s1 dan s2 (Gambar 4)
yang dihubungkan oleh interaksi secara harmonik dengan tetangga terdekat di sepanjang rantai. Untai dipasangkan satu sama lain oleh suatu ikatan hidrogen, yang bersesuaian untuk perpindahan transversal nukleotida.5
Berdasarkan Gambar 4, diasumsikan bahwa massa nukleotida pada DNA sebagai m dan kopling k konstan di sepanjang untai yang sama.5 Struktur helikoid dari untai DNA menunjukkan bahwa nukleotida dari untai yang berbeda menjadi cukup dekat, sehingga nukleotida-nukleotida tersebut dapat berinteraksi melalui filamen air, artinya bahwa nukleotida n salah satu rantai berinteraksi dengan (n+h) dan (n-h) nukleotida lainya. Nukleotida selanjutnya mengalami perpindahan gerak transversal un dan vn dari posisi kesetimbangannya di
sepanjang arah ikatan hidrogen, Hamiltonian untuk rantai DNA menjadi 9,10
�=
2
2+ 2 +
2 −
−1 2+ − −1 2 +
2 − +ℎ
2+ −
−ℎ 2 + � − − −1 2
... (1)
dimana k adalah konstanta harmonik helicoid untuk untai yang sama; K adalah konstanta harmonik helicoid untuk untai yang berbeda; H adalah Hamiltonian potensial morse yang mendekati potensial ikatan hidrogen. D adalah kedalaman potensial morse; dan a adalah jarak antar nukleotida pada rantai yang berbeda.
Gambar 4. Representasi grafis model pegas sederhana untuk rantai DNA.5
Dengan membuat transformasi ke koordinat pusat massa yang mewakili gerak tranversal ke dalam dan keluar, maka akan lebih mudah menggambarkan gerakan dua alur, yaitu
= +
2 , =
−
2 ... (2)
Bentuk Persamaan (1) setelah di transformasi ke koordinat pusat massa Persamaan (2) :
� =
2 2+
2 2 − −1 2
+
2 2 − + +ℎ 2
+
2 − + −ℎ 2 ... (3)
� =
2 2+
2 2 − −1 2
+
2 + +ℎ
2+ +
−ℎ 2 +
� − 2 −1 2 ... (4)
dari hubungan Persamaan (3) dan (4) akan diperoleh persamaan dinamika DNA yang menggambarkan gelombang linear dan non linear
= +1+ −1−2 +
+ℎ+ −ℎ −2 ... (5) dan
Seperti dijelaskan dengan asumsi DNA bergerak dengan amplitudo sangat kecil dalam beberapa artikel dapat menerapkan transformasi
= Φn atau Φ =
�; ( ≪1)... (7)
s1
s2
un-1 un un+1
vn
m m k
k
vn-1 vn+1
Morse Potensial
nl (pm)
yn
�2 = � 2
2
cos 2 −1 −2 cos 2 ℎ +1+4 2 �1 2
5
5
Substitusi Persamaan (6) ke Persamaan (7) (yang artinya diasumsikan bahwa osilasi nukleotida cukup besar untuk anharmonik, tetapi masih cukup kecil sehingga partikel berosilasi di sekitar bagian bawah potensial Morse) menjadi
Φ = Φ +1+Φ −1−2Φ −
Φ +ℎ +Φ −ℎ + 2Φ −
�2(Φ + Φ 2+ 2 Φ 3)... (8)
dimana: �2=4
2�
, =−3
2, = 7 2
3 (9)
agar Persamaan (8) dapat dipecahkan, harus diterapkan pendekatan semi-diskrit yang artinya bahwa mencari solusi dalam bentuk gelombang soliton DNA model PBD.
Φ =�1 , � +
�0 ,
+�2 , 2� 3�
+ . + 3 ... (10)
dengan � ≡ � = − , ... (11) dimana l adalah jarak antara dua nukleotida tetangga pada untai yang sama;
ω adalah frekuensi optik dari getaran
pendekatan linier; q adalah bilangan gelombang soliton DNA; dan c.c adalah istilah conjugate-complex dari fungsi F0.5
Untuk memecahkan Persamaan (10), fungsi Fi akan ubah ke bentuk kontinu.
dengan mengambil batas → dan menerapkan transformasi
= , = ... (12)
sehingga menghasilkan pendekatan kontinu berikut
� ±ℎ → � , ±� , ℎ+
1
2� ( , )
2 2ℎ2 ... (13) dimana FZ dan FZZ berarti turunan
terhadap Z. Hal ini memungkinkan untuk mendapatkan ekspresi baru untuk fungsi
Φ ( ) :
Φ =�1 , � + �0 , +
�2 , 2� + .
=�1 � + �0+�2 2� +
�1∗ − � + �2∗ −2� ... (14)
Dari Persamaan (10) - (14) diperoleh keadaan untuk semua persamaan dalam Persamaan (8). Kasus kontinu dari Persamaan (8) adalah menyamakan koefisien untuk berbagai keadaan. Sebagai contoh, dengan menyamakan koefisien untuk � kemudian diperoleh hubungan dispersi
2=2 1−cos +2 cos ℎ +
1+ �2 ... (15) Selanjutnya, dengan menurunkan Persamaan (15) secara implisit terhadap q ( ) maka akan diperoleh kecepatan grup
�� = sin( )− sin( ℎ ) ... (16)
Dengan cara yang sama yaitu menyamakan koefisien untuk 0= 1
yang artinya cos = cos ℎ = 1, diperoleh
�0= − 2
1+4
�
2
�1 2 ... (17)
dengan �=−2 1 + 4 �
2
−1
maka diperoleh hubungan
�0=� �1 2 ... (18) Asumsikan 2� untuk koefisien �2
... (19)
dengan
= �
2
2
cos 2 −1 −2 cos 2 ℎ +1+4 2 hubungannya menjadi
�2= �12 ... (20) Fungsi F0 dan F2 kemudian dapat
dinyatakan ke dalam bentuk fungsi F1
dengan menggunakan transformasi koordinat yang baru lagi
= − �� atau =�� + , �=
...(21)
6
2�
1 −2 �1 − 2�1 = {2�1 cos −1 + 2 �1 sin +
2 2�1 cos } � − {2�1 cos ℎ + 1 + 2 ℎ�1 sin ℎ + 2 2ℎ2�
1 cos( ℎ )} � −
�2 �1+ 2 2 �+ + 3 �1 1�1 � ... (22)
Berdasarkan Persamaan (22) maka persamaan NLS dapat ditulis
�1�+ 1 2
2
cos − ℎ2cos( ℎ ) −
��2�1 − �22 2 �+ +3 �11�1=0 ... (23)
dengan koefisien dispersi
= 1
2 2
cos − ℎ2cos( ℎ ) −
��2 ... (24)
=− �
2
2 2 �+ + 3 ... (25) Maka persamaan NLS pada Persamaan (23) dapat ditulis menjadi5
�1�+ �1 + �12�1= 0 ... (26) Untuk menyelesaikan Persamaan (26) harus menggunakan ansatz (tebakan), sehingga dapat diperoleh solusi eksak soliton DNA. Persamaan ansatz dari persamaan NLS tersebut diberikan
�1 ,� = ( ) �� ... (27)
dengan σ merupakan frekuensi gelombang
soliton DNA yang berperan sebagai variabel bebas dan ( ) merupakan fungsi real. Kemudian Persamaan (27) disubstitusikan ke Persamaan (26)
−� + 22+ 3= 0 ... (28) Selanjutnya Persamaan (28) dikali dengan
, diperoleh
−� + 22 + 3 = 0 ... (29) Persamaan (29) dapat dituliskan kembali dalam bentuk
1
2 −�
2+ 2+
2
4 = 0 ... (30)
yang mengindikasikan bahwa
−� 2+ 2+
2
4 = ... (31)
dimana c merupakan sebuah konstanta. Selanjutnya kembali akan diberikan batasan pada solusi yang memiliki kondisi
→ 0 dan u → 0 pada S → ± ∞ dan mengimplikasikan c = 0.
Dari hasil ini dapat ditulis kembali Persamaan (31) menjadi
2 =1 � 2−
2
4 ... (32a)
atau
2�− 2
=
2 / ... (32b)
Misalkan = 2� dan
= 2� , maka ruas kanan Persamaan (32b) dalam variable ψ dapat ditulis menjadi
2�
− 2 → 2� ... (33) Integrasikan ruas kanan Persamaan (33) terhadap variable ψ, diperoleh
2� =
1
2�
1
− ... (34)
Ruas kanan Persamaan (34) dalam variabel u dapat dinyatakan
1
2�
1
− = 1
2�
2� 1− 1− 2
2�
Sedangkan integrasi ruas kanan untuk Persamaan (32b) diperoleh
2 / = 2 ... (36)
Dengan demikian pesamaan yang harus dipecahkan adalah
2� 1− 1− 2
2�
= � (37)
Dari Persamaan (37) diperoleh persamaan untuk u
=
2 2� �
1+ 2 �
... (38)
Bentuk solusi Persamaan (38) dapat diubah menjadi bentuk trigonometri berikut
= 2�s ℎ � ... (39) Substitusi Persamaan (39) ke Persamaan (27) maka diperoleh
�1 ,� = 2�
s ℎ � �� (40)
Solusi eksak untuk Φ ( ) dalam ekspresi F1 berdasarkan Persamaan (14), (18) dan
(20) adalah
Φ =�1 � + � �1 2+
�12 2� + . ... (41) Substitusi Persamaan (38) ke Persamaan (39)
Φ = 2�s ℎ � ��+� +
2� � s ℎ �
2
+
2� s ℎ �
2
2 (��+� )+
. ... (42a)
Φ = 2�s ℎ � 2cos ��+
� +2� s ℎ �
2
dengan F = 2�s ℎ � ,
maka s
ubstitusi Persamaan (42b) ke Persamaan (7) diperoleh12= � 2cos ��+� +
2�2 �+ 2 cos 2(��+� )
(43)
Penurunan untuk solusi analitik ini diperoleh dan dapat dilihat secara lengkap pada penelitian Hermanudin12. Dalam perhitungan pada penelitian ini juga, digunakan nilai variabel-variabel karakteristik DNA, yaitu sebagai berikut k = 3K = 24 N/m,
l = 3,4 x 10−10 m, m = 5,1 x 10−25 kg,
a = 2 x 1010 m−1, D = 0,1 eV.
Massa yang digunakan dalam hal ini merupakan massa rata-rata untuk empat buah nukleotida (h = 4).9,10
BAB 3
METODE PENELITIAN
3.1 Tempat dan Waktu Penelitian
Penelitian ini dilakukan di Laboratorium Fisika Teori dan Komputasi, Departemen Fisika, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor dari bulan Februari 2011 sampai dengan Januari 2012.
3.2 Peralatan
Peralatan yang digunakan adalah perangkat komputer dengan software yang digunakan MATLAB R2010b dan Microsoft Office 2007. Sebagai pendukung penulis menggunakan sumber literatur, yaitu jurnal-jurnal ilmiah, buku-buku, dan sumber-sumber lain yang terkait.
3.3 Metode Penelitian
Metode penelitian ini adalah mencari solusi numerik dari dinamika gangguan soliton DNA model Peyrard-Bishop-Dauxois (PBD) orde ke-3 dengan menggunakan metode finite-difference
2� s ℎ �
2
8
dibantu dengan interpolasi Lagrange. Langkah-langkah yang dilakukan secara garis besar adalah studi pustaka dan pembuatan program simulasi.
3.3.1
Studi Pustaka
Studi pustaka dilakukan dengan membaca dan memahami jurnal-jurnal dan buku-buku yang berkaitan dengan penelitian ini. Studi pustaka juga membantu dalam menganalisis hasil simulasi yang dilakukan
.
3.3.2
Pembuatan Program Simulasi
Membuat sintak simulasi dengan bantuan software MATLAB menggunakan interpolasi Lagrange dan metode finite-difference. Termasuk dengan mengubah persamaan dinamika DNA model PBD ke dalam persamaan finite-difference dan interpolasi Lagrange.
3.3.2.1 Metode Beda Hingga (Finite Difference)
Sebuah aplikasi penting dari persamaan diferensial adalah dalam analisis numerik, terutama dalam persamaan diferensial numerik, tujuannya untuk menentukan solusi numerik dari persamaan diferensial biasa dan parsial. Idenya adalah dengan menggantikan turunan yang muncul dalam persamaan diferensial dengan persamaan finite-difference yang teraproksimasi. Metode yang dihasilkan kemudian disebut metode beda hingga.19 Atau dalam kata lain, metode beda hingga (finite-difference) diterapkan untuk persamaan diferensial dengan melibatkan pergantian semua turunan dengan formula perbedaan (difference).20 Metode ini digunakan untuk membantu dalam menentukan solusi numerik dari Persamaan NLS (26) sebelumnya [halaman 11]. Persamaan tersebut kemudian dapat diubah ke dalam bentuk persamaan differensial biasa, menjadi
�+ 2�2+ � 2� = 0 ... (44)
Maka persamaan tersebut dapat diselesaikan dengan melakukan pendekatan numerik beda hingga untuk masing-masing turunan parsial.
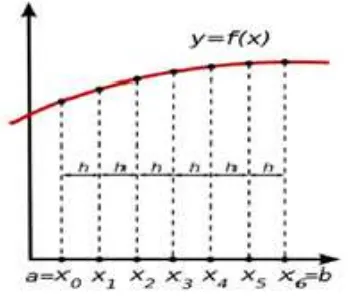
Untuk melakukannya pertama kali pilih angka integer sembarang yaitu N dimana N > 0 dan membagi interval [a, b] dengan (N + 1) sebagaimana diilustrasikan pada Gambar 5, sedemikan rupa sehingga
ℎ= −
+1 ... (45)
Dengan demikian maka titik-titik x yang merupakan sub-interval antara a dan b dapat dinyatakan sebagai21
= + ℎ, = 0,1,…, + 1 ... (46) Karena Persamaan (44) dibangun oleh dua parameter dan , maka selanjutnya parameter juga dapat dituliskan sebagai
= + ℎ, = 0,1,…, + 1 .... (47) dimana ℎ dapat pula dideskripsikan
sebagai Δ dan Δ (ℎ=Δ =Δ ) dengan menyesuaikan parameter yang dimaksud.
Selanjutnya Persamaan (44) akan diubah ke dalam bentuk formula metode beda hingga. Pencarian solusi persamaan diferensial melalui pendekatan numerik dilakukan dengan memanfaatkan polinomial Taylor yang dapat dituliskan
� +∆ =� +∆ � + ∆ 2 2 2� 2 + ∆ 3 3! 3�
3 +⋯
... (48)
(55a) 9
sedangkan polinomial Taylor untuk � +1 dan � −1 dapat dinyatakan
� +1 =� +∆
�
+∆ 2
2 2� 2 + ∆ 3 3! 3�
3 +⋯ ... (49)
� −1=� − ∆ � +∆
2 2 2� 2 + ∆ 3 3! 3�
3 +⋯ ... (50) Jika Persamaan (49) dikurangi dengan Persamaan (50) maka
� +1− � −1 = 2∆ � +
2(∆ )
3
3!
3�
3 +⋯ ... (51) Turunan � terhadap adalah
�
= � +1−� −1
2∆ −
(∆ )3 3!
3�
3 +⋯
... (52a)
atau
�
− � +1−�−1
2∆ ≈
(∆ )3 3!
3�
3 +⋯
... (52b)
Selanjutnya ruas kanan pada Persamaan (52b) dapat dikatakan sebagai error dari Persamaan (51) yang artinya nilainya dapat diabaikan dengan memberikan
notasi O. Sehingga Persamaan (51) dapat
dituliskan kembali menjadi
� = � +1−�−1
2∆ + (∆ ) ... (53)
Kemudian jika Persamaan (49) dijumlahkan dengan Persamaan (50), maka
� +1+� −1= 2� + (∆ )2
2�
2 +
2(∆ )4
4!
4�
4 +⋯ ... (54) Turunan kedua dari � terhadap adalah
2�
2 =
�+1−2� +�−1
∆ 2 +
(∆ )2 12 4� 4 atau 2� 2 =
�+1−2� +�−1
∆ 2 + (∆
2) ... (55b)
Selanjutnya dilakukan aproksimasi turunan waktu ( �) pada Persamaan (44) berdasarkan Persamaan (53). Dimana dapat dituliskan persamaannya
�
+1,
= �
+1+�
2∆ (∆ ) ... (56) Sementara aproksimasi untuk turunan
kedua terhadap jarak ( 2�2) pada Persamaan (44) berdasarkan Persamaan (55b) dalam iterasi waktu ke- dapat dituliskan persamaannya
2�
2 =
� −1+2� −� −1
∆ 2 + (∆
2) ... (57)
Kemudian Persamaan (56) dan (57) disubstitusikan ke Persamaan (44) maka
� , +1 −�( , −1)
2Δt +
� +1, − 2� , + �( −1, )
Δx2 + �
2� = 0
� , +1 − � , −1 = 2 Δt
Δx2 (� +1, −2� , +� −1, )
+ �( , )2�( , ) � , +1 = 2 �
� 2 (� +1, −2� , +� −1, ) +
�( , )2�( , ) +� ,
−1 ... (58)
Persamaan (58) inilah yang selanjutnya diubah ke dalam bahasa pemrograman MATLAB.
3.3.2.2 Interpolasi Lagrange
0 1 2 3 4
ℎ ℎ ℎ ℎ ℎ
10
10
interval [a, b]. Sebelum menentukan nilai a dan b, harus diketahui terlebih dahulu nilai titik sebelum a dan nilai titik-titik setelah b, agar mempermudah dalam menentukan nilai a dan b yang dimaksud. Untuk itu digunakanlah interpolasi Lagrange dalam menentukan titik-titik tersebut.
Interpolasi Lagrange diterapkan untuk mendapatkan fungsi polinomial P(x) berderajat tertentu yang melewati sejumlah titik data. Misalnya, untuk mendapatkan fungsi polinomial berderajat satu yang melewati dua buah titik yaitu (x0, y0) dan (x1, y1).21 Langkah pertama
yang dilakukan adalah mendefinisikan fungsi berikut
0 = − 1
0− 1 ... (59a) dan
1 = − 0
1− 0 ... (59b) kemudian definisikan fungsi polinomial sebagai berikut
= 0 0+ 1( ) 1... (60) Substitusi Persamaan (59a) dan (59b) ke Persamaan (60), maka akan didapat
= − 1
0− 1 0
+ −
1− 0 1
... (61)
dan ketika = 0
0 = 0− 1
0− 1 0
+ 0−
1− 0 1
= 0... (62a)
dan pada saat = 1
1 = 1− 1
0− 1 0
+ 1−
1− 0 1
= 1 ...(62b)
Dari persamaan tersebut dapat disimpulkan bahwa Persamaan (61) benar-benar melewati titik ( , ) dan
( 1, 1).22
Persamaan (61) dinamakan interpolasi Lagrange derajat 1. Nama interpolasi ini diambil dari nama penemunya, yaitu Joseph Louis Lagrange yang berkebangsaan Perancis. Bentuk
umum interpolasi Lagrange derajat ≤ n
untuk (n+1) titik berbeda adalah
=
=0
= 0 0 + 1 1 +⋯+ … … … …. (63)
dengan i = 0, 1, 2, ...., n dan
= −
− =
− 0 − 1 … − −1 − +1 … −
( − ) − … − −1 − +1 …( − )
=0
≠
… …(64)
Mudah dibuktikan bahwa:
= 1 , = 0 , ≠
dan polinom interpolasi P(x) melalui setiap titik data.23
Jika terdapat N data yang terdiri dari titik-titik 0, 1, 2, 3, dan seterusnya,
dan jarak antara titik satu dengan lainnya adalah h, maka Persamaan (63) dapat ditulis untuk tiga titik terdekat ( 0, 1, 2, 3)
= − 1 ( − 2)
0− 1 ( 0− 2) 0
+ − 0 ( − 2)
1− 0 ( 1− 2) 1
+ − 0 ( − 1)
2− 0 ( 2− 1) 2
= −2ℎ (−3ℎ) −ℎ (−2ℎ) 0+
−ℎ (−3ℎ)
ℎ(−ℎ) 1+
−ℎ (−2ℎ) 2ℎ (ℎ) 2
1 +
2Δt �1 2�1+
� , ... (67)
� , +1 = 2�2 Δt
Δx2 (� +1, −
2� , + � −1, ) +
11
Persamaan (65) kemudian dibuat dalam bentuk algoritma dengan menggunakan bahasa MATLAB.
3.3.3 Skala Ulang Parameter
Dalam pengerjaannya, nilai variabel-variabel yang digunakan pada persamaan NLS ini masih mengalami masalah dalam perhitungan numeriknya, dimana nilai yang digunakan masih terlalu besar untuk cakupan numerik pada software MATLAB sehingga error yang dihasilkan juga menjadi cukup besar. Untuk mengatasi masalah ini, maka dilakukanlah skala ulang terhadap nilai parameter pendukung. Skala ulang parameter ini tidak mengubah persamaan, tetapi dilakukan dengan cara memasukkan variabel pengali lain yang nilainya kecil yang dapat ditentukan sendiri, ke dalam parameter t, x, dan F, sehingga nilai ketiga parameter tersebut menjadi lebih kecil (penurunan lengkap dapat dilihat pada Lampiran B dan C)
→� ; → �
� ; dan � → �
�
... (66)
dimana � dan merupakan variabel skala ulang yang dimaksud. Nilai kedua variabel ini harus disesuaikan dengan parameter t, x, dan F agar diperoleh hasil yang tepat. Dalam hal ini nilai variabel � dan berturut-turut digunakan nilai 0,0005 dan 0,03 untuk gangguan pertama dan kedua, serta 0,001 dan 0,03 untuk gangguan ketiga. Nilai ini diperoleh dari hasil trial and error yang dilakukan selama penelitian.
Parameter t, x, dan F yang telah diskala ulang, kemudian disubstitusi ke Persamaan (58), sehingga persamaannya menjadi:
Selain persamaan utama, persamaan anzats yang digunakan juga mengalami perubahan. Dengan cara yang sama variabel t, x, dan F yang telah diskala ulang dan disubstitusi ke Persamaan (40) [halaman 7], sehingga diperoleh hasil
�= 2sech
� ... (68)
Dalam penelitian ini, solusi numerik dari persamaan NLS stabil diberikan tiga kasus gangguan. Hal ini dengan cara memberikan variabel tambahan pada persamaan ansatz, sehingga dapat diamati perubahan dari masing-masing keadaan setelah diberi gangguan. Ketiga persamaan tersebut adalah
� , = 2 ℎ
� 1 + ... (69a)
� , = 2 ℎ 1 +
�
... (69b)
� , = 2( ℎ + 0
� + ℎ − 0
� �)... (69c)
Persamaan (69a), (69b), dan (69c) selanjutnya digunakan untuk menggantikan Persamaan ansatz (68) solusi stabil. Sama seperti Persamaan (68) persamaan-persamaan tersebut digunakan untuk menentukan solusi baru dari persamaan NLS soliton DNA yang digunakan. Dari ketiga persamaan ini akan dihasilkan solusi baru yang berbeda dengan solusi stabilnya.
BAB 4
HASIL DAN PEMBAHASAN
Melalui penerapan metode beda-hingga dengan interpolasi Lagrange sebagai syarat batas terkait, maka solusi numerik dari dinamika dan interaksi soliton DNA model PBD dapat dicari dan disajikan dalam bentuk grafik tiga dimensi. Hasil perhitungan dibagi menjadi empat keadaan, yaitu keadaan tanpa gangguan kecil, dengan gangguan kecil, serta interaksi antara dua buah solusi soliton. Untuk solusi numerik pada kondisi stabil (tanpa gangguan), dihasilkan solusi soliton yang memiliki
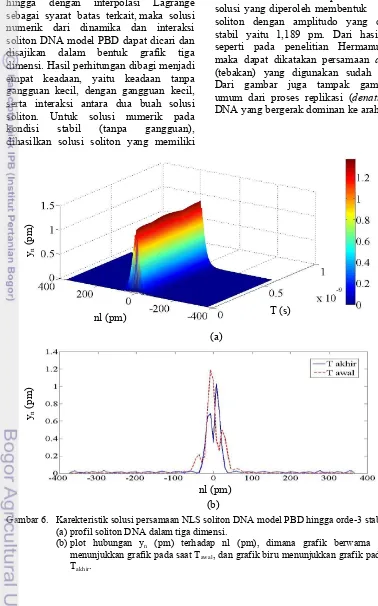
karakteristik stabil sejak waktu awal (T = 0) hingga waktu akhirnya. Hasil solusi numerik pada kondisi stabil ini ditunjukkan oleh Gambar 6.
Dari Gambar 6 tampak bahwa dari solusi yang diperoleh membentuk profil soliton dengan amplitudo yang cukup stabil yaitu 1,189 pm. Dari hasil ini, seperti pada penelitian Hermanudin13, maka dapat dikatakan persamaan ansatz (tebakan) yang digunakan sudah tepat. Dari gambar juga tampak gambaran umum dari proses replikasi (denaturasi) DNA yang bergerak dominan ke arah un.
Gambar 6. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 stabil. (a) profil soliton DNA dalam tiga dimensi.
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik berwarna merah menunjukkan grafik pada saat Tawal, dan grafik biru menunjukkan grafik pada saat Takhir.
yn
(p
m)
(a)
yn
(p
m)
nl (pm) (b)
13
4.1. Simulasi Perambatan Soliton
Akibat
Gangguan
pada
Amplitudo
Setelah diperoleh solusi untuk persamaan NLS kubik soliton DNA model PBD yang stabil, selanjutnya, solusi stabil tersebut diberikan gangguan kecil terhadap amplitudo yaitu dengan cara mengalikan Persamaan (68) yang stabil dengan suatu nilai 1 + [lihat Persamaan (69a) pada halaman 11].
Dalam hal ini digunakan nilai ε = 0,25. Pada kasus pertama ini, tampak terjadi perubahan dari solusi stabil (Gambar 6) sebelumnya setelah diberikan gangguan. Soliton DNA yang terbentuk mengalami undulasi seperti yang ditunjukkan pada Gambar 7(a). Pada saat
undulasi terjadi penyempitan yang diiringi dengan kenaikan amplitudo, yang pada waktu awal amplitudo sebesar 1,495 pm, sedangkan pada puncak undulasinya amplitudo mencapai 2,87 pm. Pada Gambar 7(b) juga ditunjukkan bahwa amplitudo untuk solusi gangguan pertama ini lebih tinggi daripada solusi stabilnya. Hal ini menunjukkan bahwa gangguan yang diberikan pada ansatz akan mempengaruhi amplitudo dari soliton. Dengan memberikan gangguan pada solusi stabilnya, artinya akan mengubah profil soliton sendiri. Selain mengalami perubahan amplitudo, tampak pula pada Gambar 7(b), soliton mengalami dispersi yang lebih besar daripada solusi stabilnya (Gambar 6(b)).
Gambar 7. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 Gangguan I.
(a) profil soliton DNA dalam tiga dimensi.
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik berwarna merah menunjukkan grafik pada saat Tawal, grafik biru menunjukkan grafik pada saat Takhir, dan grafik hijau menunjukkan saat terjadinya undulasi.
yn
(p
m)
yn
(p
m)
nl (pm)
nl (pm) T (s)
(a)
14
Artinya, gangguan yang diberikan juga mempengaruhi hubungan dispersi pada persamaan Hamiltoniannya.
Selanjutnya, gangguan kecil kembali diberikan pada persamaan ansatz untuk solusi stabil dengan cara mengubah Persamaan (68) dengan Persamaan (69b) [lihat halaman 11]. Untuk gangguan kedua
ini digunakan ε = 0,5. Profil yang ditunjukkan pada solusi gangguan kedua ini hampir sama dengan gangguan kecil sebelumnya, tetapi seperti yang ditunjukkan pada Gambar 8(a), undulasi yang terjadi tampak lebih lebar dibandingkan dengan gangguan ε = 0,25, namun amplitudo undulasi yang terbentuk
lebih kecil, yaitu 2,437 pm. Dari Gambar 8(b) juga terlihat bahwa amplitudo soliton ini lebih tinggi daripada solusi stabilnya. Dispersi yang terjadi pada kasus dengan
gangguan ε = 0,5 lebih besar dan sama
seperti kasus ε = 0,25 dimana undulasi
pada kasus dengan ε = 0,5 tersebut juga mengakibatkan pengurangan jumlah nukleotida dalam proses denaturasi, hanya saja pada kasus ini, nukleotida yang berkurang lebih sedikit daripada kasus
dengan ε = 0,25. Dari hasil simulasi
gangguan dengan dua nilai ε berbeda
tersebut tampak bahwa pada keduanya terjadi peristiwa undulasi.
Gambar 8. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 Gangguan II.
(a) profil soliton DNA dalam tiga dimensi.
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik berwarna merah menunjukkan grafik pada saat Tawal, dan grafik biru menunjukkan grafik pada saat Takhir.
yn
(p
m)
yn
(p
m)
nl (pm)
nl (pm)
T (s)
(a)
15
Ketika terjadi penyempitan pada soliton atau dalam kata lain terjadi undulasi, efek nonlinier mengalami ketidakstabilan yang lebih dominan daripada efek dispersinya. Setiap terjadi undulasi menunjukkan terjadinya pengurangan jumlah eksitasi nukleotida yang terlibat dalam proses denaturasi, dimana nukleotida yang pada awalnya dapat meregang terhalangi oleh efek nonlinier ini.
4.2. Simulasi Interaksi Dua Buah
Soliton
Pada simulasi ini, ditinjau propagasi dua soliton yang memiliki jarak x diantara
keduanya. Persamaan modifikasi yang digunakan adalah Persamaan (69c), [lihat halaman 11] dengan membuat variasi pada
nilai θ, yaitu θ = 0, θ=�
2, dan θ = π. Hasil
simulasi untuk θ = 0 ditunjukkan oleh
Gambar 9. Gambar 9(a) menunjukkan bahwa untuk kasus dengan θ = 0 ini, akan terbentuk dua buah soliton yang kemudian mengalami superposisi dengan memberikan kondisi x = 0,03 pm dan
θ = 0. Artinya diberikan jarak antara soliton satu dengan yang lain adalah 0,03 pm dengan beda fase 0. Pada gangguan ini tampak pada gambar, kedua soliton yang awalnya terpisah dengan jarak 0,03 pm
Gambar 9. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 Gangguan III.
(a) profil soliton DNA dalam tiga dimensi untuk θ = 0.
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik berwarna merah menunjukkan grafik pada saat Tawal, dan grafik biru menunjukkan grafik pada saat Takhir.
yn
(p
m)
yn
(p
m)
nl (pm)
nl (pm)
T (s)
(a)
16
menjalar dengan bentuk dan kecepatan yang sama, namun pada T = 1,37 x 10-10 s kedua soliton semakin mendekat dan keduanya berangsur-angsur bergabung menjadi satu soliton (mengalami superposisi). Sebelum terjadi superposisi, masing-masing soliton memiliki amplitudo sebesar 0,6594 pm. Amplitudo terus menurun hingga 0.44 pm pada T = 1,37 x 10-10 s, kemudian seiring bertambahnya waktu terjadi superposisi dan amplitudo berangsur-angsur kembali meningkat seperti amplitudo awalnya. Jika iterasi diteruskan (T diperpanjang) kedua gelombang terpisah kembali dengan bentuk dan kecepatan yang sama dengan sebelum terjadinya superposisi. Jelas
bahwa kondisi ini menggambarkan keadaan tarik-menarik antar kedua soliton.
Perubahan ini tampak jelas pada Gambar 9(b), dimana pada grafik berwarna merah yang menunjukkan kondisi pada T = 0, masih terdapat dua buah profil soliton yang terpisah, tetapi kemudian pada grafik biru yang menunjukkan kondisi pada T akhir, hanya terdapat satu profil soliton. Dua soliton yang terbentuk menunjukkan bahwa nukleotida yang terlibat dalam proses denaturasi terlokalisasi dalam dua ruang. Artinya, ketika terjadi superposisi, nukleotida-nukleotida yang terlokalisasi dalam dua ruang menyatu kembali.
Gambar 10. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 Gangguan III.
(a) profil soliton DNA dalam tiga dimensi untuk θ=�
2.
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik berwarna merah menunjukkan grafik pada saat Tawal, dan grafik biru menunjukkan grafik pada saat Takhir.
yn
(p
m)
yn
(p
m)
nl (pm)
nl (pm)
T (s)
(a)
17
Sementara itu untuk kondisi θ=�
2, hasil simulasi ditunjukkan oleh Gambar 10. Pada Gambar 10(a) tampak bahwa, pada awalnya terbentuk dua buah soliton yang terpisah dengan jarak 0,03 pm dengan amplitudo dan kecepatan yang sama, namun kemudian amplitudo berangsur-angsur mengecil seiring pertambahan waktu dari keadaan awal amplitudo
0,6237 pm, kemudian pada saat T = 1,35 x 10-10 s hingga waktu akhir
amplitudo salah satu soliton kembali meningkat, namun pada soliton yang lain, amplitudo mengalami penurunan. Artinya, terjadi perbedaan amplitudo dari kedua soliton, yang satu meningkat sementara yang lain menurun. Hal ini menunjukkan
kedua soliton saling tolak menolak dan mengalami perubahan yang asimetrik.
Dapat terlihat jelas juga dari grafik hubungan yn (pm) dan nl (pm) pada
Gambar 10(b), awalnya profil amplitudo dua soliton sama (grafik merah), namun kemudian di T akhir, kedua soliton memiliki amplitudo yang berbeda dari sebelumnya, selain itu terjadi pula dispersi yang cukup signifikan dari kondisi awalnya (grafik berwarna biru). Untuk
θ = π, profil soliton yang terbentuk
ditunjukkan oleh Gambar 11. Dari Gambar 11(a), tampak dua soliton yang terbentuk awalnya memiliki profil yang sama dengan amplitudo yang semakin menurun hingga saat T = 1,35 x 10-10 s.
Gambar 11. Karekteristik solusi persamaan NLS soliton DNA model PBD hingga orde-3 Gangguan III.
(a) profil soliton DNA dalam tiga dimensi untuk θ = π.
(b) plot hubungan yn (pm) terhadap nl (pm), dimana grafik berwarna merah menunjukkan grafik pada saat Tawal, dan grafik biru menunjukkan grafik pada saat Takhir.
yn
(p
m)
yn
(p
m)
nl (pm)
nl (pm) (a)
T (s)
Kemudian amplitudo soliton kembali meningkat (meskipun tidak signifikan) seiring dengan pertambahan waktu hingga iterasi akhir. Jarak antara kedua soliton tersebut juga semakin melebar dari kondisi awalnya (Gambar 11(b)). Pada waktu awal, amplitudo masing-masing soliton adalah 0,5642 pm, sedangkan di akhir amplitudo menurun menjadi 0,4524 pm. Berbeda dengan kondisi sebelumnya
(θ = �2), masing-masing soliton mengalami perubahan yang sama, atau dapat dikatakan kedua soliton saling tolak menolak tetapi dengan perubahan yang simetrik.
Dari ketiga kasus interaksi dua soliton yang diamati, tampak bahwa dinamika soliton DNA model PBD bergantung pada fase awal yang cukup memberikan dampak yang signifikan pada dinamikanya. Terlihat bahwa soliton mengalami undulasi, kenaikan dan penurunan amplitudo, serta dispersi yang semakin meningkat.
BAB 5
KESIMPULAN
Pada penelitian sebelumnya, telah diperoleh solusi analitik untuk persamaan NLS kubik DNA model PBD.12 Selanjutya, berdasarkan solusi analitik yang telah diperoleh, dikembangkan kembali penelitian untuk mencari solusi numerik persamaan NLS kubik DNA model PBD dengan menggunakan metode beda hingga dan interpolasi Lagrange. Dari hasil yang diperoleh menunjukkan bahwa solusi antara solusi analitik dan solusi numerik sudah sesuai. DNA model PBD yang digunakan pada penelitian ini menunjukkan perilaku dinamika DNA pada saat proses denaturasi replikasi DNA. Hasil dari solusi numerik yang dilakukan diperolehlah profil soliton dinamika terkait. DNA mengalami dinamika yang cukup stabil dari proses awal hingga akhirnya. Hasil ini juga menunjukkan bahwa persamaan ansatz yang diperoleh dari solusi analitik sudah tepat.
Dalam penelitian ini, persamaan ansatz yang diperoleh dari solusi analtik persamaan NLS kubik DNA model PBD, diberikan gangguan. Dilakukan sebanyak tiga gangguan pada solusi stabil persamaan NLS DNA model PBD ini. Masing-masing hasil solusi numerik dari beberapa kasus gangguan yang ditinjau menunjukkan hasil yang berbeda-beda. Untuk gangguan yang relatif kecil diperoleh hasil bahwa terjadi peristiwa undulasi pada soliton yang awalnya stabil. Undulasi ini mengakibatkan soliton semakin menyempit dan mengalami kenaikan amplitudo. Hasil ini menunjukkan bahwa pada saat undulasi, terjadi pengurangan jumlah nukleotida yang terlibat dalam proses denaturasi pada rantai DNA. Selain itu, gangguan ini juga menyebabkan efek pada koefisien dispersinya, sehingga tampak terjadi dispersi pada solusi numeriknya. Untuk gangguan yang lebih besar, diperoleh hasil yang hampir sama dengan kasus gangguan sebelumnya. Perbedaannya, pada gangguan yang lebih besar undulasi yang terjadi lebih lebar dan amplitudo puncak undulasinya lebih rendah jika dibandingkan dengan gangguan yang lebih kecil. Artinya, pengurangan jumlah nukleotida yang terlibat dalam proses denaturasi pada rantai DNA lebih sedikit. Selain itu, dispersi yang terjadi tampak lebih besar untuk gangguan yang lebih besar. Peristiwa undulasi yang terjadi pada kedua gangguan tersebut ini menunjukkan bahwa efek nonlinier mengalami ketidakstabilan yang lebih besar daripada efek dispersinya, yang menyebabkan soliton mengalami penyempitan. Efek dispersi lebih dominan ketidakstabilannya pada saat soliton tidak mengalami undulasi (keadaan sebaliknya).
Berbeda dengan kasus gangguan kecil, pada interaksi dua soliton dengan jarak x, pada proses denaturasinya, nukleotida terlokalisasi dalam dua ruang. Telah ditinjau kasus tiga kondisi fase awal yaitu dengan membuat variasi pada beda fase antara kedua soliton, pada saat θ = 0,
�
soliton bergerak dengan amplitudo yang sama yang nilainya terus menurun, kemudian saat T = 1,37 x 10-10 s, kedua soliton mengalami superposisi, sehingga terbentuklah satu soliton. Soliton ini kemudian bergerak dengan mengalami peningkatan amplitudo hingga waktu akhir. Dinamika ini akan berlanjut, dimana soliton akan kembali terpisah pada T tertentu kemudian mengalami superposisi kembali, begitu pula seterusnya. Untuk beda fase θ = �
2, kedua soliton saling tolak menolak dan mengalami perubahan yang asimetrik. Pada selang waktu T = 0 s hingga T = 1,35 x 10-10 s soliton bergerak dengan amplitudo yang sama yang nilainya terus
menurun. Kemudian pada T > 1,35 x 10-10 s hingga akhir iterasi
waktu, jarak antar kedua soliton semakin melebar, lalu salah satu soliton bergerak dengan mengalami peningkatan amplitudo, sedangkan soliton yang lain terus bergerak dengan amplitudo yang semakin menurun dari kondisi awalnya. Dan untuk beda fase θ = π, kedua soliton tolak menolak tetapi dengan perubahan yang simetrik. Pada selang waktu T = 0 s hingga T = 1,35 x 10-10 s dua soliton bergerak dengan amplitudo yang sama yang nilainya terus menurun, selanjutnya pada saat T > 1,35 x 10-10 s hingga akhir iterasi waktu, jarak antar kedua soliton semakin melebar, kemudian amplitudo kedua soliton kembali meningkat (walaupun tidak terlalu signifikan) dan terus bergerak stabil hingga T akhir. Dari hasil yang diperpoleh ini, dapat disimpulkan pula bahwa hasil solusi numerik yang diperoleh dari dinamika DNA model PBD yang diberikan efek gangguan akan menunjukkan hasil yang berbeda dengan solusi eksak tanpa gangguan.
SARAN
–
SARAN
Penelitian tentang dinamika permodelan DNA merupakan salah satu hal yang menarik untuk dilakukan. Untuk pengembangan selanjutnya, diharapkan dapat melakukan beberapa hal,
diantaranya dengan menggunakan metode numerik yang memiliki tingkat akurasi yang lebih baik lagi, sehingga dapat dilakukan perhitungan dengan lebih cepat dan tepat. Selain itu, dapat pula dilakukan (baik numerik maupun analitik) ekspansi potensial morse dengan orde yang lebih tinggi. Dengan mengambil ekspansi hingga orde yang lebih tinggi diharapkan akan diperoleh hasil yang lebih akurat seperti keadaan sebenarnya. Pada penelitian ini, permodelan yang digunakan masih bersifat ideal, dalam artian mengabaikan pengaruh-pengaruh luar yang bersifat mengganggu. Selanjutnya mungkin dapat dilakukan penelitian dengan memperhitungkan pengaruh-pengaruh luar tersebut. Dalam permodelan DNA sendiri ada banyak sekali model yang terus dikembangkan, yang mungkin dapat diteliti selain daripada model PBD ini.
DAFTAR PUSTAKA
1. Peyrard, M. dan Bishop, A.R. (1989). Statistical mechanics of a nonlinear model for DNA denaturation. Phys. Rev. Lett. 62, 2755-2758.
2. Englander, S.W. et al. (1980). Nature of the open state in long polynucleotide double helices: possibility of soliton excitations. Proc. Natl. Acad. Sci. USA
77, 7222-7226.
3. Yomosa, S. (1984). Solitary excitations in deoxyribonucleic acid (DNA) double helices. Phys. Rev. A 30, 474-480.
4. Homma, S. dan Takeno, S. (1984). A coupled base-rotator model for structure and dynamics of DNA, Prog. Theor. Phys. 72, 679-693.
5. Zdravković, S., Tuszyński, J.A. dan
Satarić, M.V. (2005). Peyrard-Bishop-Dauxois model of DNA dynamics and impact of viscosity. Journal of
Computational and Theoretical
Nanoscience Vol.2: 1–9.
11. Zdravković, S. dan Satarić, M.V. (2001). Impact of viscosity on DNA dynamics. Phys. Scripta 64, 612-615.
20
absorption in DNA, Phys. Rev. A-40, 2148-2153.
7. Hadi, M. dan Nurlita, I. “Soliton dan
DNA”. Fisikanet. 2006. Web. 17
Februari 2011.<http://fisikanet.lipi.go. id/utamacgi?artikel&1151902055&33>
8. Yakushevich, L.V. (1998). Nonlinear Physics of DNA. Wiley Series in Nonlinear Science, John Wiley, Chichester, 181.
9. Dauxois, T. (1991). Dynamics of breathers modes in a nonlinear
“helicoidal” model of DNA. Phys. Lett. A-159, 390-395.
10. Dauxois, T. dan Peyrard, M. (1991). Dynamics of breather modes in a nonlinear helicoidal model of DNA.
Lecture Notes in Physics 393,
Dijon,p.79.
12. Hermanudin, D. (2011). Efek osilasi anharmonik pada soliton Deoxyribo Nucleic Acid Peyrard-Bishop-Dauxois. Skripsi. Departemen Fisika-FMIPA IPB.
13. Anonim. “DNA”. Wikipedia. 2011. Web. 02 Maret 2011. <http://en.wikipedia.org/DNA>
14. Hamdani, S. “DNA sebagai Kode
Genetik”. 2012. Web. 03 Mei 2012.
<http://catatankimia.com/catatan/dna-sebagai-kode-genetik.html>
15. Anonim. “DNA-RNA-Protein”. 2012. Web. 03 Mei 2012. <http://www.nobelprize.org/educatio nal/medicine/dna/>
16. Anonim. “DNA vs. RNA and replication”.2011. Web. 18 Februari
2011.<http://apbio2-chs-09.wikispaces.com/DNA+vs.+RNA+ and+replication>
17. Anonim. “Replikasi DNA”. Wikipedia. 2010. Web. 18 Februari 2011.<http://id.wikipedia.org/Replika si DNA>
18. Ludmila, V. dan Yakushevich. (2001). Is DNA a nonlinear dynamical system where solitary conformational waves are possible?. J. Biosci., Vol.26, No.3: 305-313.
19. Anonim. “Finite Difference”. Wikipedia. 2011. Web. 17 Februari 2011.<http://en.wikipedia.org/wiki/Fi nite_difference>
20. Recktenwald, G.W. “ Finite-Difference Approximations to The
Heat Equation”. 2004. Web. 17
Februari 2011.
<www.f.kth.se/~jjalap/numme/FDhea t.pdf>
21. Suparno, S. (2008). Komputasi untuk
Sains dan Teknik –Menggunakan
Matlab-. Departemen Fisika FMIPA, Univeristas Indonesia,181-182.
22. Supriyanto. (2007). Komputasi untuk
Sains dan Teknik. Universitas
Indonesia. Jakarta.
22
Lampiran A.
Diagram Alir Penelitian
Mulai
Penelusuran literatur
Membuat sintak simulasi MATLAB dengan menggunakan metode beda hingga (finite-difference)
Menganalisis hasil solusi numerik
23
Lampiran B.
Skala Ulang Parameter Persamaan NLS Tahap 1
Persamaan NLS awal
�+ 2�2+ �1 2�
1 = 0 ... (26)
dengan persamaan ansatz (tebakan)
� = 2�s ℎ � �� ... (40)
misalkan:
� = ; =
� ; � =� � ... (B.1)
Substitusi permisalan pada persamaan (64) ke (26)
� �+ �
2�
2+ �
2�= 0... (B.2)
� � +� 2�2+ � 2�= 0 ... (B.3)
� � � +� � 2� 2 + � � � 2� = 0 ... (B.4)
� +
2�
2+ �
2
� = 0 ... (B.5)
Substitusi permisalan pada persamaan (B.1) ke (40)
� =�
� ... (B.1)
� =
�
2�
sech �
� ... (B.6)
� = 2 sech ... (B.7)
Lampiran C.
Skala Ulang Parameter Persamaan NLS Tahap 2
Skala ulang tahap 2 dilakukan, karena pada skala ulang tahap 1, skala yang diperoleh
masih terlalu besar, dan belum compatible dengan software MATLAB yang digunakan.
24
Misalkan:
=
� ; � = � ... (C.1)
Substitusi persamaan (C.1) ke (B.5), sehingga
�+� 2�
2+
3 � 2�= 0 ... (C.2)
�+� 2�
2 +
2 � 2�= 0 ... (C.3) Substitusi persamaan (C.1) ke (B.7)
�= �
�= 2sech
� ... (C.4)
Jika dilakukan substitusi persamaan (C.1) ke persamaan (B.1), maka variabel t, x, dan F
dapat disederhanakan:
� = ; → �
� ; � → � �
25
Lampiran D.
Source Code
Program dengan
software
Matlab
clear allclc
% >> ..:Definisi Kondisi Awal dan Inkremen:.. <<
M = input ('M = '); N = input ('N = ');
% x01 = input ('x01 = '); % teta = input ('teta = ');
% x01 dan teta digunakan pada saat menjalankan simulasi gangguan ketiga;
epsilon = input ('epsilon = ');
% gunakan epsilon=0.25 untuk gangguan kedua; % gunakan epsilon=0.5 untuk gangguan ketiga;
dx = 0.01;
dt = 2.5000e-005; x0 = -N/2*dx;
% >> ..:Definisi Nilai Parameter Pembangun:.. <<
D = 0.1*1.6e5; mass=5.1e-13; a=3e-2;
k=24; K=8; l=340; lamda=10*l; q=2*pi/lamda; h=4;
sigma=1e10; tau=0; e2=0.001;
% >> ..:Definisi hubungan dispersi:.. <<
wg =2*a*sqrt(D/mass);
w=sqrt(wg+(2*k*(1-cos(q*l))+2*K*(cos(q*l*h)+1))/mass);
% >> ..:Definisi kecepatan group:.. <<
Vg=(l*(k*sin(l*q)-K*h*sin(l*q*h))/(mass*w));
% >> ..:Definisi koefisien orde-1 sampai orde-3:.. <<
alfa =(-3*a)/sqrt(2); beta =7*a^2/3;
% >> ..:Definisi hubungan F1 dengan F0 dan F2:.. <<
miu=-2*alfa/(1+4*K/(mass*wg.^2));
delta=(wg.^2)*alfa/(4*w^2+2*k*(cos(2*q*l-1)/mass-2*K*(cos(2*h*q*l)+1)...
/mass-wg^2));
% >> ..:Definisi koefisien dispersi(P) dan koefisien nonlinear(Q):.. <<
P=(((l^2)*(k*cos(l*q)-K*(h^2)*cos(l*q*h))/mass)-Vg^2)/(2*w); Q=(-wg.^2*(2*alfa*(miu+delta)+3*beta)/(2*w));
% >> ..:Parameter rescale:.. <<
26
psi = 0.03;
% >> ..:Matriks F dan Y:.. <<
F=zeros(N,M); Y=zeros(N,M);
for n=1:N
x(n,1)=x0+(n-1)*dx;
F(n,1)=((sqrt(2)/psi)*sech(x(n,1)/sqrt(phi));
%.: untuk gangguan pertama :.
% F(n,1)=((sqrt(2)/psi)*sech(x(n,1/sqrt(phi)))*(1+epsilon);
%.: untuk gangguan kedua :.
% F(n,1)=((sqrt(2)/psi)*sech((1+epsilon)*(x(n,1)/sqrt(phi))*
% (1+epsilon);
%.: untuk gangguan ketiga :.
% F(n,1)=(sqrt(2)/psi)*((sech(((x(n,1)+x01)/sqrt(phi))))+...
% ((sech((x(n,1)-x01)/sqrt(phi)))*(exp(1i*teta)));
end
% >> ..:Running Program:.. <<
for m=1:M
for n=2:N-1
% >> ..:Interpolasi Lagrange:.. <<
if n==2
F(n-1,m)=3*F(n,m)-3*F(n+1,m)+F(n+2,m); end
if n==N-1
F(n+1,m)=3*F(n,m)-3*F(n-1,m)+F(n-2,m); end
% >> ..:Metode Finite Difference:.. <<
if m==1
F(n,m+1)=1i*((phi^2*r*(F(n+1,m)-2*(F(n,m))+F(n-1,m))+...
(psi^2*dt*(conj(F(n,m))*(F(n,m)^2))+F(n,m); else
F(n,m+1)=2*1i*((phi^2*r*(F(n+1,m)-2*(F(n,m))+F(n- 1,m)))+...
(psi^2*dt*(conj(F(n,m))*(F(n,m)^2)))+F(n,m-1); end
t(m)=(m-1)*dt;
theta(n,m)=(n*q*l)-(w*t(m)/e2); Y(n,m)=
((e2*(psi*F(n,m)/(sqrt(Q/sigma))))*(2*(cos((sigma*tau)+...
theta(n,m)))))+((e2^2*((psi*F(n,m)/(sqrt(Q/sigma)))^2))...
*(miu+(2*delta*(cos(2*((sigma*tau)+theta(n,m))))))); %Y adalah persamaan gelombang untuk soliton DNA%
end
m
end
% >> ..:Grafik 3 dimensi soliton DNA:.. <<
figure
surf(t/sigma,x*sqrt(P/sigma)/sqrt(phi),abs(Y(:,1:M))); view(0,90);
colorbar
[image:36.595.92.493.1.818.2]27
xlabel ('T'); ylabel ('x'); zlabel ('Y');
% >> ..:Grafik Hubungan Y terhadap x:.. <<
figure
plot((x*sqrt(P/sigma)),abs(Y(:,0))); xlabel ('x(pm)');
ylabel ('Y(pm)');
figure
plot((x*sqrt(P/sigma)),abs(Y(:,M))); xlabel ('x(pm)');
[image:37.595.96.516.47.811.2]