PERANCANGAN PRIMER UNTUK PENGEMBANGAN
SISTEM DETEKSI BERBASIS PCR (
Polymerase Chain Reaction
)
PADA
Ganoderma
spp.
NURUL RAHAYU
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Perancangan Primer untuk Pengembangan Sistem Deteksi Berbasis PCR (Polymerase Chain Reaction) pada Ganoderma spp. adalah benar karya bersama saya dengan komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, September 2013 Nurul Rahayu NIM G34080068
ABSTRAK
NURUL RAHAYU. Perancangan Primer untuk Pengembangan Sistem Deteksi Berbasis PCR (Polymerase Chain Reaction) pada Ganoderma spp. Dibimbing oleh UTUT WIDYASTUTI dan NAMPIAH SUKARNO.
Ganoderma spp diketahui merupakan salah satu penyebab penyakit pada kelapa sawit. Penelitian ini bertujuan merancang dan menguji primer spesifik terhadap Ganoderma spp. untuk pengembangan sistem deteksi berbasis PCR. Penelitian dilakukan dengan mengumpulkan data sekuen dari daerah β-tubulin dan ITS dari data base nukleotida yang tersedia. Selanjutnya dianalisis untuk merancang primer menggunakan sofware primer 3. Primer yang dirancang selanjutnya digunakan untuk mendeteksi DNA yang berasal dari miselia Ganoderma spp. yang berasal dari IPBCC kode 10.658 (GIPB) dan tiga tubuh buah Ganoderma sp. dari Lombok (GLom). Penelitian ini berhasil merancang dua pasang primer yang berdasarkan daerah ITS yaitu Gan-ITS12 dan β-tubulin yaitu primer β-Gan. Hasil PCR menunjukkan bahwa primer Gan-ITS12 mampu mengamplifikasi Ganoderma spp. pada tingkat spesies, sedangkan primer β-Gan mampu mengamplifikasi pada tingkat genus. Hasil uji spesifitas primer pada DNA yang berasal dari kelompok cendawan Basidiomycota tidak berhasil mengamplifikasi DNA, sedangkan pada kelompok selain Basidiomycota berhasil mengamplifikasi DNA walaupun dengan pita yang terbentuk lebih dari satu. Hal ini mengindikasikan bahwa primer yang dirancang bekerja spesifik pada Ganoderma spp.
Kata kunci: Ganoderma sp., ITS, PCR, β-tubulin
ABSTRACT
NURUL RAHAYU. Development of Dectection System base on PCR Through Design Primer for Ganoderma spp.. Supervised by UTUT WIDYASTUTI and NAMPIAH SUKARNO.
Ganoderma spp. has been known a pathogenic fungus for palm oil. The aim of this research was to design specific primers and test it to Ganoderma spp. in order to obtain new detection system base on PCR method. Two pairs of primers were designed successfully; Gan-ITS12 primer derived from ITS region and β -Gan primer derivied from β-tubulin region. This two pairs primers were used to identified DNA from mycellia Ganoderma spp. from two different sample sources i.e IPBCC with code 10.658 (GIPB) and three different of Ganoderma sp. from Lombok (Glom1, Glom2, Glom3). The result indicates both Gan-ITS
primer and β-Gan primer can amplify Ganoderma spp. on species and genus level respectively. Specifity of primers were tested to DNA from Basidiomycota group and non-Basidiomycota group. The results showed this two pairs primer can not amplification DNA from Basidiomycota group. But, this primers can amplified non-Basidiomycota groups even with more than one band formed.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Biologi
PERANCANGAN PRIMER UNTUK PENGEMBANGAN
SISTEM DETEKSI BERBASIS PCR (
Polymerase Chain Reaction
)
PADA
Ganoderma
spp.
NURUL RAHAYU
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
Judul Skripsi : Perancangan Primer untuk Pengembangan Sistem Deteksi Berbasis PCR (Polymerase Chain Reaction) pada Ganoderma spp.
Nama : Nurul Rahayu NIM : G34080068
Disetujui oleh
Dr Ir Utut Widyastuti, MSi Pembimbing I
Dr Ir Nampiah Sukarno Pembimbing II
Diketahui oleh
Dr Ir Iman Rusmana, MSi Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan dan sebagai syarat untuk memperoleh gelar Sarjana Sains Departemen Biologi FMIPA IPB. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Juli 2012 ini ialah sistem deteksi penanda molekular secara dini, dengan judul Perancangan Primer untuk Pengembangan Sistem Deteksi Berbasis PCR (Polymerase Chain Reaction) pada Ganoderma spp.. Terima kasih penulis ucapkan kepada Proyek Hibah Kerjasama Internasional DIKTI Tahun 2010 yang telah memberikan dana kepada Dr Ir Utut Widyastuti, MSi dan terimaksih juga kepada Laboratorium BIORIN PPSHB-IPB atas fasilitas penelitian yang diberikan.
Penghargaan dan terima kasih diberikan kepada Ibu Dr Ir Utut Widyastuti, MSi dan Ibu Dr Ir Nampiah Sukarno selaku dosen pembimbing yang telah sepenuh hati memberikan bimbingan, motivasi, dan saran kepada penulis selama penyelesaian karya ilmiah ini. Ucapan terima kasih juga penulis sampaikan kepada Ibu Dr Rita Megia, DEA selaku dosen penguji yang telah banyak memberikan saran dan masukan untuk perbaikan penulisan karya ilmiah ini.
Ucapan terima kasih juga ingin disampaikan kepada Ibu Winarni Widayati, Bapak Basuki dan seluruh keluarga yang selalu bermurah hati untuk mendoakan dalam menempuh pendidikan dan mencari ilmu pengetahuan, Dini Ayu Lestari dan Anniza Dian Pertiwi atas doa, perhatian, dan segala dukungan. Terima kasih penulis sampaikan kepada Gina, Salma, dan Rani teman seperjuangan dalam penelitian ini, teman-teman Tumbang tersayang, Asrama Putri Darmaga, seluruh staf Departemen Biologi, Biorin Crew, dan teman-teman Biologi 45 yang telah membantu selama berlangsungnya kegiatan karya ilmiah.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL ix
DAFTAR GAMBAR ix
DAFTAR LAMPIRAN ix
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
METODE 2
Waktu dan Tempat 2
Bahan 2
Alat 3
Prosedur Penelitian 3
1Perancangan Primer 4
2Pengujian Primer 4
2.1 Pengujian primer pada Ganoderma spp. 4
2.1.1 Penyiapan kultur cendawan 4
2.1.2 Isolasi DNA genomik cendawan dari miselia 5 2.1.3 Amplifikasi DNA Ganoderma spp. dengan primer yang
dirancang 5
2.1.4 Sekuensing hasil PCR dan analisis bioinformatika 6
2.2 Pengujian kespesifikan primer 6
2.2.1 Isolasi DNA genomik cendawan dari tubuh buah 6 2.2.2 Amplifikasi DNA uji dengan primer yang dirancang 6
HASIL 6
Perancangan Primer 6
Pengujian Primer 8
1 Pengujian primer pada Ganoderma spp. 8
2 Pengujian kespesifikan primer 10
PEMBAHASAN 11
SIMPULAN 13
SARAN 13
DAFTAR PUSTAKA 14
LAMPIRAN 16
DAFTAR TABEL
1 Perancangan desain primer 7
2 Analisis Bioinformatika keempat sampel Ganoderma spp.
menggunakan primer Beta-Gan 9
DAFTAR GAMBAR
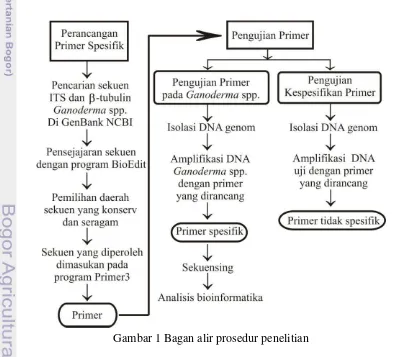
1 Bagan alir prosedur penelitian 3
2 Posisi amplifikasi kedua primer yang diharapkan akan terbentuk 7 3 Hasil isolasi DNA keempat sampel Ganoderma spp. 8 4 Amplifikasi PCR pada rDNA sampel GIPB menggunakan kedua
pasang primer. 8
5 Amplifikasi PCR kedua pasang primer menggunakan tiga sampel
DNA Ganoderma sp. Lombok (GLom1, GLom2, GLom3) 9 6 Peta DNA yang terbentuk hasil amplifikasi primer Beta-Gan pada
sampel GIPB dan GLom 10
7 Hasil uji kespesifikan primer (Beta-Gan:A, ITS12:B) menggunakan
DNA cendawan kelompok Basidiomycota. 10
8 Hasil uji kespesifikan primer (Beta-Gan:A, ITS12:B) menggunakan
DNA cendawan bukan kelompok Basidiomycota. 11
DAFTAR LAMPIRAN
1 Siklus hidup Ganoderma sp. menurut Esser dan Kuenen 1967 16
2 Hasil runutan DNA daerah β-Tubulin cendawan GIPB 17
3 Hasil runutan DNA daerah β-Tubulin cendawan GLom 19
4 Hasil isolasi tubuh buah cendawan Basidiomycota 21 5 Runutan konserv DNA daerah ITS1,5.8,ITS2 dan hasil perancangan
primer 22
PENDAHULUAN
Latar Belakang
Indonesia merupakan penghasil minyak sawit terbesar kedua setelah Malaysia. Kelapa sawit adalah tanaman komoditas perkebunan yang penting dan masih memiliki prospek pengembangan yang bagus. Komoditas kelapa sawit, baik berupa bahan mentah maupun hasil olahannya, menduduki peringkat ketiga penyumbang devisa nonmigas tebesar bagi Indonesia setelah karet dan kopi (Sastrosayono 2003). Oleh karena itu, komoditi ini perlu mendapat perhatian. Usaha peningkatan produksi perkebunan kelapa sawit secara rutin terkendala dengan masalah penyakit serius yang disebabkan oleh cendawan patogen Ganoderma spp.. Menurut Menteri Pertanian Suswono kerugian akibat serangan ganoderma pada kelapa sawit sangat besar. Secara nasional, dengan luas perkebunan sawit 83 juta hektar (ha), tingkat serangan Ganoderma sebesar 1% bisa menyebabkan kerugian lebih dari Rp 2 triliun per tahun. Sedangkan, tingkat serangan bisa mencapai lebih dari 2%, terutama pada kebun yang sudah peremajaan (replanting) beberapa kali.
Ganoderma spp. ialah anggota cendawan divisi Basidiomycota, kelas Agaricomycetes, ordo Polyporales, famili Ganodermataceae, ordo Ganoderma. Miselia cendawan ini bereproduksi secara seksual dengan cara menghasilkan tubuh buah yang disebut basidiokarpus. Basidiokarpus menghasilkan banyak basidiospora yang merupakan spora seksual. Reproduksi aseksual pada cendawan ini lebih jarang terjadi. Miselia cendawan ini mampu tumbuh sangat cepat, misalnya pada media serbuk batang kelapa sawit atau potongan akar kelapa sawit, dan mulai membentuk bakal basidiokarpus setelah 30 hari inokulasi, dan tumbuh sempurna setelah 90 hari. Siklus hidup cendawan Ganoderma spp. menurut Esser dan Kuenen (1967) bisa dilihat pada Lampiran 1. Jika media tempat hidup cendawan ini adalah batang kelapa sawit maka dibutuhkan waktu bertahun-tahun hingga tumbuh tubuh buah basidiokarpus sempurna (Semangun 1988). Cendawan Ganoderma spp. menyebabkan penyakit busuk pangkal batang (BPB) pada tanaman kelapa sawit. Penyakit ini menyerang sistem perakaran dan gejala penyakit yang ditimbulkannya hanya terlihat sangat lambat pada akhir infeksi sehingga tanaman yang sakit tidak dapat diselamatkan. Kerugian serius terjadi pada generasi tanaman lanjut setelah mengalami masa panen sebanyak tiga kali atau tanaman generasi ketiga dan serangan penyakit ini dapat mencapai 60% populasi tanaman (Singh 1991). Oleh karena itu pengembangan sistem deteksi dini yang cepat dan akurat sangat dibutuhkan untuk mengurangi dampak yang akan terjadi akibat serangan cendawan tersebut.
2
alternatif teknik untuk mendeteksi dan mengawasi keberadaan cendawan (Suanthie et al. 2009) seperti Ganoderma spp.. Perbedaan profil fragmen DNA hasil amplifikasi dengan PCR dapat digunakan sebagai alat untuk membedakan mikroba pada tingkat genus, spesies bahkan genotipe spesifik dari patogen (Edel 1998). Penggabungan metode PCR dan perancangan primer untuk mengamplifikasi berbagai daerah yang terdapat pada DNA cendawan sangat mendukung upaya pengembangan perangkat deteksi cendawan patogen tanaman. Perbedaan penelitian yang dilakukan ini dengan penelitian-penelitian sebelumnya adalah penggunaan primer dan tahapan siklus hidup pada cendawan Ganoderma sp.. Pada penelitian sebelumnya diperlukan sampel berupa tubuh buah cendawannya, selanjutnya digunakan primer universal (Purnamasari et al. 2012). Namun pada penelitian ini menggunakan sampel miselia yang keberadaannya lebih awal, dan primer yang spesifik untuk cendawan target.
Pada cendawan sistem penanda molekular digunakan berada pada daerah 400-600 dari subunit ribosom, daerah cistron Internal Transcribe Spacer (ITS),
daerah β-tubulin, A (BenA), dan 1-α (EF-1α), serta gen CO1 (Seifert et al. 2007). Penelitian yang akan dilakukan menggunakan dua dari daerah tersebut yaitu
daerah ITS dan β-tubulin. Daerah ITS biasanya mengalami perubahan atau mutasi sehingga dapat berbeda atau bervariasi di antara spesies. Menurut Schoch et al. (2012) daerah ITS secara formal telah diusulkan sebagai barkode marker utama pada Fungi, sedangkan DNA pada daerah β-tubulin penting dalam studi rekonstruksi filogenetik di antara cendawan disemua tingkatan (Thon & Royse 1999).
Tujuan Penelitian
Penelitian ini bertujuan untuk merancang dan menguji primer dengan PCR untuk mendeteksi keberadaan cendawan Ganoderma spp secara dini.
METODE
Waktu dan Tempat
Penelitian ini dilaksanakan dari bulan Juli 2012 sampai bulan Februari 2013 di laboratorium BIORIN Pusat Penelitian Sumberdaya Hayati dan Bioteknologi (PPSHB), Institut Pertanian Bogor.
Bahan
3 bukan kelompok Basidiomycota yaitu: Mortierella sp., Hypoxylon sp., Xilaria sp., dan Penicillium sp.. Selain itu, digunakan media Potato Dextrose Agar (PDA), media Potato Dextrose Broth (PBD), nitrogen cair, CTAB 2x, PVP, fenol, CI, PCI, NaOAC, ethanol 100%, ethanol 70%, RNAse, enzim taq polymerase dan buffer-nya, dNTPs, primer reverse dan primer forward, ddH2O, gel agarose, TAE
1x, loading dye, dan Ethidium Bromida.
Alat
Alat yang digunakan pada penelitian ini adalah laptop atau komputer yang terhubung ke internet, laminar, shaker, mortar, water bath, mesin sentrifugasi, mesin vakum, mesin spektrofotometri, freezer, mesin PCR, mesin elektroforesis, UV transluminator, dan alat-alat laboratorium lainnya .
Prosedur Penelitian
Tahapan prosedur yang digunakan pada penelitian ini terdiri dari dua bagian utama yaitu perancangan primer dan pengujiannya. Alur prosedur penelitian disajikan pada Gambar 1.
4
1 Perancangan Primer
Analisis primer forward dan reverse menggunakan sekuen daerah ITS dan
β-tubulin RNA Ganoderma spp. yang diperoleh dari http://www.ncbi. nlm.nih.gov. Sekuen DNA Ganoderma spp. yang digunakan sebagai acuan desain primer pada penelitian ini terdiri dari dua daerah konservasi yaitu daerah ITS dan
β-tubulin RNA. Masing-masing sekuen lengkap dari setiap daerah konservasi, disejajarkan untuk mendapatkan sekuen yang paling seragam sebagai acuan desain primer. Setelah itu, primer didesain berdasarkan daerah konservasinya menggunakan Primer3 yang terdapat di http://frodo.wi.mit.edu/. Hasil desain primer yang dipilih adalah primer yang memiliki presentasi GC sebesar 50-55%, angka dimer dan hairpin yang kecil, dan suhu leleh 60-62 oC. Presentasi GC ditentukan dengan cara menghitung banyaknya basa G dan C yang ada pada oligonukleotida primer, dikalikan dengan panjang basa total oligonukleotida, dibagi seratus. Dimer merupakan terbentuknya ikatan antara sesama primer forward, sesama primer reverse, ataupun primer reverse dan primer forward. Hairpin merupakan terbentuknya ikatan antara satu basa, dengan basa lainnya dalam satu sekuen primer. Suhu leleh secara kasar dapat dihitung dengan rumus Tm = 2(A+T) + 4(G+C). Primer hasil rancangan ini selanjutnya disintesis nukletidanya (1st Base, Singapura).
2 Pengujian Primer
Pengujian primer hasil rancangan dilakukan menggunakan DNA cendawan kelompok Ganoderma spp.. Sedangkan, untuk mengetahui kespesifikannya digunakan sampel DNA cendawan kelompok Basidiomycota, dan kelompok bukan Basidiomycota.
2.1 Pengujian primer pada Ganoderma spp.
2.1.1 Penyiapan kultur cendawan
5 PDB, diremajakan di atas shaker pada suhu ruang, masing-masing tahap peremajaan membutuhkan waktu selama 7-8 hari.
2.1.2 Isolasi DNA genomik cendawan dari miselia
Isolasi DNA genomik cendawan dilakukan dengan metode Bluhm et al. (2002) yang telah dimodifikasi. Keempat isolat Ganoderma spp. diisolasi dari media PDB, miselia diambil secukupnya, disaring mengunakan kertas saring steril, dikeringkan menggunakan vakum, kemudian digerus menggunakan N2 cair dalam
mortar. Hasil penggerusan dimasukkan ke dalam tabung eppendorf yang telah diisi dengan 600 L CTAB 2% yang sudah diinkubasi pada suhu 65 ⁰C dalam waterbath. Tabung berisi hasil gerusan ini dibolak-balik dan diinkubasi dalam waterbath pada suhu 65 ⁰C. Setelah 30 menit, diangkat dan disimpan dalam es. Setelah kurang lebih lima menit, ditambahkan ± 600 L CI dengan perbandingan volume 24:1, dibolak-balik, dan disentrifus pada suhu 4 ⁰C dengan kecepatan 10.000 rpm. Setelah 10 menit, sebanyak 600 µL supernatan diambil, ditambahkan ± 600 µL PCI dengan perbandingan volume 25:24:1, dibolak-balik, dan disentrifus pada suhu 4 ⁰C dengan kecepatan 10.000 rpm. Setelah lima menit, supernatan diambil secukupnya, ditambah 2 M NaOAC pH 5,2 sebanyak 0,1 kali volume supernatan yang diambil, ditambah etanol 100% sebanyak 2 kali volume supernatan yang diambil, dan disimpan dalam freezeer selama ± 24 jam. Setelah 24 jam, disentrifugasi pada suhu 4 ⁰C dengan kecepatan 10.000 rpm. Setelah 30 menit, supernatan dibuang dan ditambahkan 500 µL ETOH 70%, disentrifugasi dengan suhu 4 ⁰C pada 10.000 rpm. Setelah 10 menit supernatan dibuang dan dikeringkan dalam vakum. Setelah 30 menit ditambahkan RNAse (1 mg/mL) sebanyak 0,2 kali volume, disimpan dalam oven bersuhu 37 ⁰C selama 10 menit, dan diinaktifkan pada suhu 70 ⁰C selama 10 menit. Elektroforesis dilakukan untuk mengetahui keberhasilan proses isolasi DNA sampel. Hasil isolasi DNA yang diperoleh selanjutnya siap digunakan atau disimpan dalam freezer.
2.1.3 Amplifikasi DNA Ganoderma spp. dengan primer yang dirancang Amplifikasi potongan daerah ITS dan β-tubulin terhadap isolat sampel dilakukan dengan mesin PCR. Sebanyak 1 µL DNA sampel konsentrasi 100 ng/µ L dimasukkan ke dalam 9 µL mix PCR yang terdiri dari 0,1 µL enzim taq polymerase 5 U/µL; 0,25 µL primer forward 10 pm; 0,25 µL primer reverse 10 pm; 1 µL dNTPs 2 mM; 1 µL buffer; dan 6,4 µL ddH2O. Volume campuran reaksi
sebanyak 10 µL dimasukkan dalam mesin PCR dengan program denaturasi awal 95 ⁰C selama 5 menit, denaturasi 94 ⁰C selama 30 detik, penempelan 52 ⁰C selama 30 detik, dan ekstensi 72 ⁰C selama 30 detik yang dijalankan sebanyak 35 siklus. Setelah itu, dilanjutkan dengan pasca ekstensi 72 ⁰C selama 7 menit dan pendinginan 15 ⁰C selama 20 menit.
6
2.1.4 Sekuensing hasil PCR dan analisis bioinformatika
Sekuensing dilakukan pada hasil amplifikasi keempat sampel Ganoderma spp.. Hasil amplifikasi PCR disiapkan hingga volume campurannya mencapai 60 µL menggunakan pasangan primer dengan program yang sama. Sebagian hasil PCR ini, sebanyak 5 µL digunakan untuk cek elektroforesis. Sisanya dikirim ke perusahaan analisa DNA untuk sekuensing. Hasil sekuensing kemudian dibandingkan dengan sekuen nukleotida dari GenBank menggunakan megaBLAST yang dilakukan di http://www.ncbi.nlm.nih.gov. (Lampiran 2 dan 3) untuk mengidentifikasi DNA cendawan yang mampu mengamplifikasikan pita. 2.2 Pengujian kespesifikan primer
Pengujian primer dilakukan terhadap DNA cendawan dari kelompok Basidiomycota dan yang bukan dari kelompok Basidiomycota. DNA cendawan kelompok Basidiomycota yang digunakan adalah DNA cendawan Enoki (Flammulina velutipes), Buna Shimeji (Hypsizygus tessellatus), Champignon (A. bisporus), Jamur Tiram (Pleurotus ostreatus), dan Jamur Merang (Volvariella volvacea). Cendawan kelompok bukan Basidiomycota yang digunakan yaitu sampel DNA dari Penicillium sp., Hypoxilon sp., Mortierella sp., dan Xilaria sp.. 2.2.1 Isolasi DNA genomik cendawan dari tubuh buah
Isolasi DNA genomik cendawan dari tubuh buah menggunakan metode yang sama dengan yang berasal dari miselia, dengan modifikasi yang sedikit berbeda. Kelima tubuh buah dari cendawan yang bukan dari kelompok Basidiomycota diambil sekitar 0,5 gram, kemudian digerus menggunakan N2 cair
dalam mortar. Hasil penggerusan dimasukkan ke dalam tabung eppendorf yang telah diisi dengan 600 L CTAB 2% dan PVP yang sudah diinkubasi pada suhu 65 ⁰C dalam waterbath. Tabung berisi hasil gerusan ini dibolak- balik dan diinkubasi dalam waterbath pada suhu 65 ⁰C. Setelah 30 menit, diangkat dan disimpan dalam es. Setelah kurang lebih lima menit, ditambahkan ± 600 L fenol dan dihomogenkan. Langkah berikutnya sama dengan tahapan ektraksi DNA dari miselia.
2.2.2 Amplifikasi DNA uji dengan primer yang dirancang
Proses amplifikasi pada tahap ini sama dengan proses pada amplifikasi DNA Ganoderma spp. dengan primer yang dirancang.
HASIL
Perancangan Primer
7 Berdasarkan hasil pensejajaran kumpulan sekuen daerah β-tubulin DNA Ganoderma spp. diperoleh dua sekuen oligonukleotida pasangan primer forward dan reverse dengan kode A. Primer forward yaitu Beta-Gan F, primer reverse yaitu Beta-Gan R. Berdasarkan hasil pensejajaran kumpulan sekuen daerah ITS rDNA Ganoderma spp., diperoleh dua sekuen oligonukleotida yang merupakan pasangan primer forward dan reverse dengan kode B. Primer forward yaitu Gan-ITS12 F, primer reverse yaitu Gan-Gan-ITS12 R. Runutan masing-masing dari daerah konserv dapat dilihat pada Lampiran 5 dan 6. Panjang masing-masing oligonukleotida adalah 20 basa. Kedua pasang primer tersebut memiliki nilai %GC dan Tm yang sama yaitu 50% dan 58,4 ⁰C. Ukuran amplikon yang diharapkan pada pasangan primer Beta-Gan sekitar 305 bp, sedangkan pada pasangan primer Gan-ITS12 sekitar 308 bp (Tabel 1). Posisi penempelan primer hasil amplifikasi yang diharapkan pada kedua daerah ITS dan β-tubulin penempelan primer Gan-ITS12 308 bp±
Sekuen DNA spp.
8
Pengujian Primer 1 Pengujian primer pada Ganoderma spp.
1.1 Amplifikasi hasil isolasi DNA genom Ganoderma spp.
DNA genom berhasil diisolasi dari keempat sampel Ganoderma spp. yaitu GIPB, GLom1, GLom2, GLom3. Keberhasilan isolasi dapat terlihat dengan melakukan elektroforesis kemudian divisualisasikan pada UV. Hasil dokumentasi
menunjukkan adanya pita yang terbentuk sejajar dengan DNA marker yang
berarti DNA berhasil diisolasi (Gambar 3).
1.2 Amplifikasi DNA Ganoderma spp. dengan primer yang dirancang
Tahap awal pengujian hasil desain primer dapat bekerja pada DNA Ganoderma spp. adalah dengan melakukan metode PCR menggunakan sampel DNA Ganoderma sp. koleksi IPBCC (GIPB). Suhu annealing kedua primer sebelumnya telah ditentukan untuk mendapatkan hasil amplifikasi pita yang jelas dengan melakukan percobaan PCR beberapa kali pada penggunaan suhu annealing 50, 52, 54, 56, 58, dan 60 ⁰C. Kedua pasang primer yang menunjukkan terbentuknya pita yang jelas dan terang dengan ukuran amplikon ± 300 bp pada suhu annealing 52 ⁰C (Gambar 4).
Gambar 3 Hasil isolasi DNA keempat sampel Ganoderma spp.
Gambar 4 Amplifikasi PCR pada DNA sampel GIPB menggunakan kedua pasang primer
Keterangan:
GIPB: sampel Ganoderma sp. koleksi IPBCC kode 10.658
GLom1: sampel Ganoderma sp. pertama dari Lombok
GLom2: sampel Ganoderma sp. kedua dari Lombok
GLom3: sampel Ganoderma sp. ketiga dari Lombok
Keterangan:
9 Pengujian primer selanjutnya dilakukan menggunakan tiga sampel DNA Ganoderma sp. dari Lombok (GLom1, GLom2, GLom3) hasilnya menunjukkan terbentuknya pita dengan ukuran ± 200 bp (Gambar 5). Hasil pengujian primer Gan-ITS12 tidak dapat teramplifikasi pada sampel DNA Ganoderma sp. Lombok (GLom1, GLom2, GLom3), meskipun telah dicoba pada suhu annealing yang berbeda pada proses PCR. Hal tersebut menunjukkan bahwa primer tersebut tidak mampu menempel pada Ganoderma sp. Lombok.
1.3 Sekuensing hasil PCR dan analisis bioinformatika
Amplifikasi PCR yang dapat terbentuk kemudian dilakukan metode PCR Scale-up untuk pasangan primer Beta-Gan pada keempat sampel Ganoderma spp.. Metode PCR Scale-up adalah memperbanyak volume pada reaksi PCR normal sebanyak enam kali lipatnya. Hasil PCR Scale-up kemudian dikirim ke perusahaan analisa DNA untuk dilakukan sekuensing. Hasil sekuensing kemudian diolah menggunakan software BioEdit. Hasil pensejajaran DNA sekuensing selanjutnya di Blast pada program NCBI dengan laptop yang terhubung ke internet (Lampiran 2 dan 3).
Hasil sekuensing menunjukkan bahwa pita DNA yang teramplifikasi menggunakan primer Beta-Gan adalah benar adanya DNA Ganoderma spp. (Tabel 2). Sampel DNA GIPB hasil PCR yang telah dianalisis teridentifikasi merupakan genus Ganoderma applanatum dengan nilai query coverage 99%, nilai E-value 9e-124, dan nilai max ident atau kemiripan 95%, sedangkan sampel DNA GLom1, GLom2 dan GLom 3 menunjukkan jenis yang sama yaitu Gambar 5 Amplifikasi PCR kedua pasang primer menggunakan tiga sampel DNA
Ganoderma sp. Lombok (GLom1, GLom2, GLom3)
Tabel 2 Analisis Bioinformatika keempat sampel Ganoderma spp. menggunakan primer Beta-Gan
GIPB JQ675614.1 Ganoderma applanatum strain
ATCC 44053 beta-tubulin gene 99% 9e-124 95% GLom1 JQ675670.1 Ganoderma tornatum strain
CBS 109679 beta-tubulin gene 16% 3.6 89% GLom2 JQ675670.1 Ganoderma tornatum strain
CBS 109679 beta-tubulin gene 16% 3.7 89% GLom3 JQ675670.1 Ganoderma tornatum strain
10
Ganoderma tornatum dengan nilai query coverage 16%, nilai E-value 3.6, dan nilai kemiripan 89%.
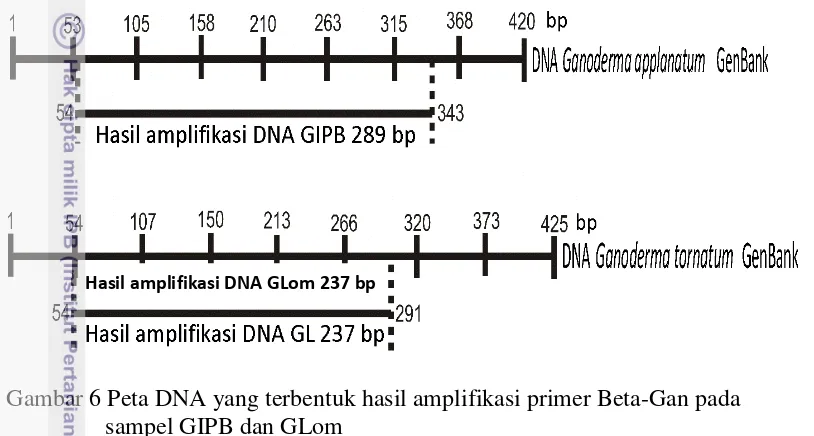
Ukuran pita amplifikasi yang berbeda antara GIPB dan Glomb (Gambar 5) menunjukkan adanya perbedaan posisi penempelan primer Beta-Gan (Gambar 6). Primer forward menempel pada posisi yang sama yaitu posisi ke 54 pada dua sekuen cendawan Ganoderma yang berbeda spesies, sedangkan posisi akhir penempelan primer reverse terletak berbeda pada GIPB dan GLom.
2 Pengujian kespesifikan primer
Primer yang telah dirancang diuji kespesifikannya terhadap sesama cendawan yang berada pada satu kelompok Basidiomycota, karena Ganoderma termasuk dalam kelompok ini. Cendawan tersebut sama-sama mampu membentuk tubuh buah berumur panjang. Cendawan tersebut yaitu jamur Enoki (Flammulina velutipes), jamur Buna Shimeji (Hypsizygus tessellatus), jamur Champignon (A. bisporus), jamur Tiram (Pleurotus ostreatus), dan jamur Merang (Volvariella volvacea). Cendawan-cendawan tersebut sebelumnya telah diisolasi DNAnya (Lampiran 4) Hasil amplifikasi menunjukkan bahwa kedua pasang primer tidak mampu mengamplifikasi DNA pada semua sampel cendawan uji (Gambar 7). Gambar 6 Peta DNA yang terbentuk hasil amplifikasi primer Beta-Gan pada
sampel GIPB dan GLom
Hasil amplifikasi DNA GLom 237 bp
Gambar 7 Hasil uji kespesifikan primer (Beta-Gan:A, ITS12:B) menggunakan DNA cendawan kelompok Basidiomycota
Keterangan:
Kolom: M marker 100bp, 1 Enoki (Flammulina velutipes),
2 Buna Shimeji (Hypsizygus tessellatus), 3 Champignon (A. bisporus),
11 Uji kespesifikkan primer juga dilakukan terhadap beberapa sampel DNA cendawan yang bukan kelompok Basidiomycota, yaitu Penicillium sp. (cendawan bermitospora), Hypoxilon sp. (Ascomycota), Mortierella sp. (Zygomycota), dan Xilaria sp. (Ascomycota). Cendawan tersebut merupakan spesies yang mewakili kelompok cendawan sejati. Hasil amplifikasi menunjukkan bahwa primer yang digunakan menghasilkan amplifikasi DNA dengan jumlah pita yang lebih dari satu pita (Gambar 8).
PEMBAHASAN
Ukuran amplikon primer yang di harapkan akan terbentuk pada kedua primer berukuran 305 bp untuk BGan dan 308 bp untuk GanITS. Kriteria penting yang harus dipenuhi dalam merancang primer antara lain panjang primer, komposisi basa, dan suhu leleh. Panjang primer yang baik dan ideal berkisar 18-30 nukleotida dengan komposisi nukleotida berkisar 40-60%. Suhu leleh (Tm) yang baik berkisar 52-58 ⁰C dan sebaiknya basa nitrogen pasa ujung 3’ adalah G atau C, karena ikatannya lebih kuat dan dibutuhkan ATP lebih banyak untuk melepaskan ikatannya bila dibandingkan ikatan A dengan T dan untuk menghindari kesalahan penempelan sehingga tidak terbentuk pita ganda, yaitu terbentuknya dua pita yang dapat terlihat dari hasil visulaisasi elektroforesis pada UV, karena primer menempel pada daerah lain di lokasi DNA template yang menandakan primer tidak spesifik (Abd-Elsalam 2003). Primer yang telah dirancang tersebut akan menentukan keberhasilan ketika proses PCR, sehingga untuk mengetahui keberhasilannya perlu dilakukan pengujian primer terhadap DNA target yang digunakan sebagai penentu keberhasilan perancangan primer dan DNA lain untuk mengetahui kespesifikannya.
Berdasarkan siklus hidupnya keberadaan miselia pada cendawan lebih awal dan cepat dari pada keberadaan tubuh buah pada divisi Basidiomycota, sehingga penelitian ini dapat diterapkan lebih awal untuk mengetahui keberadaan cendawan Ganoderma sp. yang menginfeksi tanaman kelapa sawit. Penelitian ini mampu mendeteksi keberadaan cendawan Ganoderma sp. lebih dini yaitu pada tahap perkembangan miselia, sehingga diharapkan dapat digunakan untuk mendeteksi sebelum lahan yang akan menjadi perkebunan kelapa sawit ditanami kelapa sawit. Pengujian kedua pasang primer pada sampel GIPB menunjukkan hasil amplifikasi pita DNA yang jelas dan terang pada ukuran ± 300 bp. Hal tersebut menunjukkan bahwa primer dapat bekerja pada sampel Ganoderma. Suhu
Gambar 8 Hasil uji kespesifikan primer menggunakan DNA cendawan bukan kelompok Basidiomycota
Keterangan:
A primer Beta-Gan, B primer Gan- ITS12 Kolom: M marker 1 kb
12
annealing kedua pasang primer sebelumnya telah ditentukan untuk mendapatkan hasil amplifikasi pita yang jelas dengan melakukan percobaan PCR beberapa kali pada penggunaan suhu annealing 50, 52, 54, 56, 58, dan 60 ⁰C, hingga ditentukan suhu yang tepat adalah 52 ⁰C. Uji selanjutnya pada sampel GLom1, 2, dan 3 dengan pasangan primer Beta-Gan menunjukkan perbedaan ukuran amplifikasi dengan DNA GIPB. Hal tersebut mungkin disebabkan karena perbedaan spesies dan keragaman urutan basa pada DNA masing-masing sampel. Namun, pada pasangan primer Gan-ITS12 tidak teramplifikasi pada sampel GLom1, 2, dan 3. Dari hasil analisis sekuensing dan analisis bioinformatik diketahui bahwa sampel GIPB dan GLom adalah dua spesies berbeda.
Analisis bioinformatik NCBI terdapat nilai query coverage yang menunjukkan ukuran panjang basa yang sama antara nukleotida yang dimiliki dengan nukleotida yang terdapat pada GenBank, semakin panjang nukleotida yang sama pada sampel uji dengan nukleotida yang terdapat pada GenBank maka nilainya akan semakin besar dan sampel uji akan semakin homolog dengan nukleotida yang ada pada GenBank. Nilai E-value menunjukkan tingkat kesalahan yang terjadi ketika proses pensejajaran antara urutan nukleotida sampel uji dengan urutan nukleotida yang terdapat dalam GenBank. Nilai max ident menunjukkan nilai kemiripan, semakin besar nilai maka semakin mirip nukleotida sampel uji dengan nukleotida data GenBank. Sampel uji GIPB menunjukkan ketiga nilai analisis yang cukup baik, sedangkan sampel uji GLom1, 2, dan 3 ketiga nilainya kurang baik. Hasil pensejajaran hasil sekuensing menggunakan BioEdit pada sampel GLom masih menunjukkan adanya banyak variasi urutan basa dengan DNA G. applanatum GenBank, sehingga nilai query coverage, E-value, dan max ident hasil analisis rendah. Nilai query coverage yang tidak sampai 20% menunjukkan ukuran nukleotida yang terbentuk lebih pendek dan adanya variasi yang tinggi dari seluruh DNA hasil Blast yang ada. Nilai tersebut mempengaruhi nilai E-value yang hasilnya mencapai 3,7. Penyebab lain dari kecilnya ketiga nilai tersebut berdasarkan Purnamasari et al. (2012) hal ini mungkin dapat dijelaskan oleh sifat tubuh buah yang merupakan hasil perkawinan heterokarion antar hifa yang berbeda. Untuk menghasilkan tubuh buah, Ganoderma harus melakukan reproduksi seksual dengan hifa yang berbeda, dan rekombinasi DNA yang terjadi waktu reproduksi seksual berkontribusi terhadap keragaman genetika yang tinggi. Hasil yang diperoleh menunjukkan bahwa primer Gan-ITS12 mampu menjadi penanda deteksi dini pada tingkat spesies yaitu Ganoderma applanatum, sedangkan primer Beta-Gan mampu menjadi penanda deteksi dini pada tingkat genus Ganoderma dengan ukuran amplifikasi yang mungkin bervariasi ketika dilakukan PCR dan elektroforesis.
13
SIMPULAN
Penelitian ini berhasil merancang 2 pasang primer yaitu primer Beta-Gan dan ITS12. Primer Beta-Gan mampu digunakan sebagai penanda molekular dini untuk mendeteksi keberadaan Ganoderma spp.. Primer Gan-ITS12 mampu untuk mendeteksi keberadaan Ganoderma applanatum yang merupakan salah satu cendawan Ganoderma penyebab penyakit pada tanaman kelapa sawit.
SARAN
14
DAFTAR PUSTAKA
Abd-Elsalam KA.2003. Bioinformatic tools and guideline for PCR primer design. Afr J Biotechnol. [Internet]. [diunduh 22 Feb 2013]; 2(5):91-95. Tersedia pada: http://www.academicjournals.org/AJB.
Bluhm BH, Flaherty JE, Cousin MA, dan Woloshuk. 2002. Multiplex Polymerase Chain Reaction Assay fo the Differential Detection of Trichothecene and Fumonisin-Producing Species of Fusarium in Cornmeal. Journal of Food Protection .65:1955-1961.
Chong KP. 2010. The Role of Phenolic In The Interaction Between Oil Palm and Ganoderma boninense The Causal Agen of Basal Stem Rot. [Thesis]. [Internet]. [diunduh 1Sept 2013]; Nottingham (GB): The University of Nottingham. Tersedia pada: http://etheses.nottingham.ac.uk/2817/1/537652.pdf.
Darmono TW, Suharyanto, Darussamin A. 1993. Polyclonal antibody against washing filtrate of mycelium culture of Ganoderma sp.. Menara Perkebunan. 61:67-72.
Darmono TW, Suharyanto. 1995. Recognition of field materials of Ganoderma sp. associated with basal stem rot in oil pal with polyclonal antibody. Menara Perkebunan. 65:15-22.
Edel V. 1998. Polymerase chain reaction in mycology: an overview. Bridge PD et al., editor. Applications of PCR in mycology. United Kingdom: CAB International. [Internet]. [diunduh 13 Juli 2013]; P. 1-20. Tersedia pada: www.bnski.com/File/Attachments/634954096433764218.pdf
Esser K, Kuenen R. 1967. Genetics of Fungi. New York (US): Springer Verlag. Gunawan AW, Rahayu G, Dharmaputra OS, Sudirman LI, Sukarno N, Listiyowati
S. 2011. Cendawan Dalam Praktik Laboratorium. Edisi ke-4. Bogor (ID): IPB Press.
Purnamasari MI, Prihatna C, Gunawan AW, dan Suwanto A. 2012. Isolasi dan Identifikasi Secara Molekular Ganoderma spp. yang Berasosiasi dengan Penyakit Busuk Pangkal Batang di Kelapa Sawit. Jurnal Fitopatologi Indonesia 8(1): 9-15.
Sastrosayono S. 2003. Budi daya Kelapa Sawit. Agromedia Pustaka. Jakarta. [Internet]. [diunduh 13 Juli 2013]. Tersedia pada: http://books.gooGLome. co.id/books?id=Ezi93yp_fucC&source=gbs_navlinks_s
Scoch CL, Seifert KA, Huhndorf S, Robert V, Spouge JL, Levesque CA, Chen W, Fungal Barcoding Consortium. 2012. Nuclear ribosomal internal trascribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences, USA. [Internet]. [diunduh 22 Agustus 2013]; 109(16):6241-6246. Tersedia pada: http://www.pnas.org/content/109/16/6241.full.pdf
Seifert KA, Samson RA, deWaard JR, Houbraken J, levesque CA, Moncalvo JM, Seize GLOM, Hebert PDN. 2007. Prospect for fungus identification using CO1 DNA barcodes, with Penicillium as a test case. Proceedings of the National Academy of Sciences, USA. [Internet]. [diunduh 13 Juli 2013]; 104(10): 3901-3906. Tersedia pada: http://www.pnas.org/cgi/content/full/0611691104/DC1. Semangun H. 1988. Penyakit-penyakit Tanaman Perkebunan di Indonesia.
15 Singh G. 1991. Ganoderma the scourge of oli palm in the coastal areas. [abstrak]. Di dalam: Darus A,Sukaimi J, editor. Proceedings of Ganoderma workshop; 11 September 1990; Bangi Selangor Malaysia. [Internet]. [diunduh 25 Mei 2013]; hlm 7-35. Tersedia pada: http://www.cabdirect.org/abstracts/19932328524 Suanthie Y, Cousin MA, Woloshuk CP. 2009. Multiplex real-time PCR for
Detection and Qualification of Mycotoxigenic Aspergillus, Penicillium and Fusarium. [abstrak]. Journal of Stored Products Research. [Internet]. [diunduh 25 Mei 2013]; 45:139-145. Tersedia pada: http://www.scopus.com /record/ display.
Suharyanto, Darmono TW. 1998. Karakterisasi parsial eksudat dan filtrat kultur Ganoderma sp. untuk membantu dalam pengembangan perangkat identifikasi antibodi monoklonal. Menara Perkebunan. [Internet]. [diunduh 1 Sept 2013]; 66(2):96-103. Tersedia pada: http://www.pustaka.litbang.deptan.go.id/bptpi /lengkap/IPTANA/fullteks/MP/1998/mp11.pdf.
16
Lampiran 1 Siklus hidup Ganoderma sp. menurut Esser dan Kuenen 1967
Keterangan gambar:
1. Basidiospora dihasilkan oleh basidium
2. Basidiospora yang menyebar jika sampai pada lingkungan yang sesuai akan berkecambah menjadi miselia. Dua miselia yang cocok akan bertemu.
3. Selanjutnya miselia hasil perkawinan akan tumbuh lebih cepat dibandingkan dengan miselia induknya.
4. Setelah 30 hari pembentukan miselia, mulai terbentuk basidiokarpus dan akan sempurna setelah 90 hari (Pada media yang diteliti).
5. Permukaan Basidiokarpus dilapisi sel-sel terminal yang disebut basidia. 6. Tahap 6. Ke 7. Pada satu sel basidia terjadi penyatuan intisel.
7. Intisel menyatu disebut basidium.
17 Lampiran 2 Hasil runutan DNA daerah β-Tubulin cendawan GIPB
Urutan nukleotida hasil sekuensing cendawan GIPB
TGATCGTATGATGGCCACGTTCTCCGTTGTCCCCTCGCCAAAGGTGTCTGACACC
Hasil analisis bioinformatika menggunakan megaBLAST dari NCBI Ganoderma applanatum strain ATCC 44053 beta-tubulin gene, partial cds GenBank: JQ675614.1
LOCUS JQ675614 419 bp DNA linear PLN 15-AUG-2012 DEFINITION Ganoderma applanatum strain ATCC 44053 beta-tubulin gene, partial
cds.
ACCESSION JQ675614
VERSION JQ675614.1 GI:400201353 KEYWORDS .
SOURCE Ganoderma applanatum ORGANISM Ganoderma applanatum
Eukaryota; Fungi; Dikarya; Basidiomycota; Agaricomycotina;
TITLE Genetic diversity analysis of Ganoderma species and development of a specific marker for identification of medicinal mushroom
Ganoderma lucidum
JOURNAL Afr J Microbiol Res 6 (25), 5417-5425 (2012) REFERENCE 2 (bases 1 to 419)
AUTHORS Park,Y.J., Kwon,O.C., Son,E.S., Yoon,D.E., Han,W., Yoo,Y.B.and Lee,C.S.
JOURNAL Submitted (16-FEB-2012) Dept. of Applied Biochemistry, Konkuk University, 322 Danwoldong, Chung-Ju, Chungbuk 380-701, Rep. of Korea
18
Lanjutan Lampiran 2
Sequencing Technology :: Sanger dideoxy sequencing ##Assembly-Data-END##
FEATURES Location/Qualifiers source 1..419
/organism="Ganoderma applanatum" /mol_type="genomic DNA"
/strain="ATCC 44053"
/culture_collection="ATCC:44053" /db_xref="taxon:29884"
mRNA join(<1..345,401..>419) /product="beta-tubulin" CDS join(<1..345,401..>419) /codon_start=3
/product="beta-tubulin" /protein_id="AFP73547.1" /db_xref="GI:400201354"
/translation="GAGMGTLLISKIREEYPDRMMATFSVVPSPKVSD TVVEPYNATLSVHQLVENSDQTFCIDNEALYDICFRTLKLTTP TYGDLNHLVSIVMSGITTCLRFPGQLNSDLRKLAVNMVPFPR L"
ORIGIN
19 Lampiran 3 Hasil runutan DNA daerah β-Tubulin cendawan GLom
Urutan nukleotida hasil sekuensing cendawan GLom
TGATCGTATGATGGCCACGTTCATATGGTAGTCCCCTAAGAACAATATGCTGATA GTTCGACGGCAGCATCATGGTGGATTGGCGCTAGGCAACTTAGCCTGTTTTGGTG AGTAGATTCTGGACGCTGGCGATTGGCCAAAGAAACTTAACAGCCATCGGGAATC AGCAGAGGATGGCCTTAATGTAGTAGTGCAGGAAGTTGGCTGTCAACAA
Hasil analisis bioinformatika menggunakan megaBLAST dari NCBI Ganoderma tornatum strain CBS 109679 beta-tubulin gene, partial cds GenBank: JQ675670.1
LOCUS JQ675670 419 bp DNA linear PLN 15-AUG-2012 DEFINITION Ganoderma tornatum strain CBS 109679 beta-tubulin gene, partial cds.
ACCESSION JQ675670
VERSION JQ675670.1 GI:400201465 KEYWORDS .
SOURCE Ganoderma tornatum ORGANISM Ganoderma tornatum
Eukaryota; Fungi; Dikarya; Basidiomycota; Agaricomycotina; Agaricomycetes; Polyporales; Ganodermataceae; Ganoderma. REFERENCE 1 (bases 1 to 419)
AUTHORS Park,Y.-J., Kwon,O.-C., Son,E.-S., Yoon,D.-E., Han,W., Nam,J.-Y.,
Yoo,Y.-B. and Lee,C.-S.
TITLE Genetic diversity analysis of Ganoderma species and development of a specific marker for identification of medicinal mushroom
Ganoderma lucidum
JOURNAL Afr J Microbiol Res 6 (25), 5417-5425 (2012) REFERENCE 2 (bases 1 to 419)
AUTHORS Park,Y.J., Kwon,O.C., Son,E.S., Yoon,D.E., Han,W., Yoo,Y.B. and
Lee,C.S.
TITLE Taxonomy of Ganoderma lucidum from Korea Based on rDNA and Partial
JOURNAL Submitted (16-FEB-2012) Dept. of Applied Biochemistry, Konkuk University, 322 Danwoldong, Chung-Ju, Chungbuk 380-701, Rep. of Korea
COMMENT ##Assembly-Data-START##
20
Lanjutan Lampiran 3
FEATURES Location/Qualifiers source 1..419
/organism="Ganoderma tornatum" /mol_type="genomic DNA" /strain="CBS 109679"
/culture_collection="CBS:109679" /db_xref="taxon:101942"
mRNA join(<1..345,401..>419) /product="beta-tubulin" CDS join(<1..345,401..>419) /codon_start=3
/product="beta-tubulin" /protein_id="AFP73603.1" /db_xref="GI:400201466"
/translation="GAGMGTLLISKIREEYPDRMMATFSVVPSPKVSD TVVEPYNATLHQLVENSDQTFCIDNEALYDICFRTLKLTTPTY GDLNHLVSIVMSGITTCLRFPGQLNSDLRKLAVNMVPFPRL" ORIGIN
1 ccggtgcagg catgggtacc ctcctgatct cgaagattag ggaagaatac cccgatcgta
21
Lampiran 4 Hasil isolasi tubuh buah cendawan Basidiomycota
Keterangan:
Kolom M adalah DNA marker lambda ( )
Kolom 1 adalah DNA jamur Enoki (Flammulina velutipes)
Kolom 2 adalah DNA jamur Buna Shimeji (Hypsizygus tessellatus) Kolom 3 adalah DNA jamur Champignon (A. bisporus)
22
Lampiran 5 Runutan konserv DNA daerah ITS1,5.8,ITS2 dan hasil perancangan primer
OLIGO start len tm gc% any 3' LEFT PRIMER 144 20 59.87 50.00 3.00 1.00 TCTGTGCCTGCGTTTATCAC
RIGHT PRIMER 451 20 59.95 50.00 2.00 0.00 TAAGAGGAGCCGACCAAGAA
SEQUENCE SIZE: 587
INCLUDED REGION SIZE: 587
PRODUCT SIZE: 308, PAIR ANY COMPL: 3.00, PAIR 3' COMPL: 0.00
GAAGGATCATTATCGAGTCTTGACCGGGTTGTAGCTGGCCTTCCGAGGCATGTGCACGCC CTGCTCATCCACTCTACACCTGTGCACTTACTGTGGGCTTCAGATCGTAAAGCAGGGCCC TTCACCGGGCTTTGCAGGACGTGTCTGTGCCTGCGTTTATCACAAACTCTATAAAGTATC
23 Lampiran 6 Runutan konserv DNA daerah β-tubulin dan hasil perancangan primer
start len tm gc% any 3' LEFT PRIMER 56 20 60.75 50.00 6.00 2.00 GATCGTATGATGGCCACGTT
RIGHT PRIMER 360 20 59.87 50.00 7.00 3.00 TGTTGACAGCCAACTTCCTG
SEQUENCE SIZE: 447
INCLUDED REGION SIZE: 447
PRODUCT SIZE: 305, PAIR ANY COMPL: 4.00, PAIR 3' COMPL: 0.00
1 CGGTGCAGGCATGGGTACCCTCCTGATCTCGAA---GATCAGGGAAGAATACCCCGATCG
61 TATGATGGCCACGTTC-TCCGTT---GTCCCCTCACCGAAGGTATCCGACACCGT
121 CGTYGAGC-CGTACAACGCTACGCTCTCCGTTCACCAGCTCGTTGAGAACTCGGACCAGA 181 CCTTCTGTATCGATAACGAGGCGCTCTA-CGACATTTGCTTCAGGACGCTCAAGCTTACG
241 ACACCGACAT--ATGGTGACCTCAACCACCTCGTC---TCCATCGTCATGTCGGGTATCA 301 CGACTTGCTTGCGCTTCCCTGGTCAGCTCAACTCGGACCTCAGGAAGTTGGCTGTCAACA
24
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta pada tanggal 13 April 1990 dari ayah Basuki dan ibu Winarni Widayati. Penulis merupakan anak tunggal. Penulis menyelesaikan pendidikannya di TK Islam Soraya pada tahun 1996, SDN Jatiasih 4 pada tahun 2002, MDA Al-Ikhlas pada tahun 2002, SMPN 9 Bekasi pada tahun 2005, SMAN 6 Bekasi pada tahun 2008. Pada tahun yang sama penulis lulus seleksi masuk Institut Pertanian Bogor (IPB) melalui jalur Undangan Seleksi Masuk IPB (USMI) di Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam.
Selama mengikuti perkuliahan, penulis pernah menjadi asisten praktikum mata kuliah Genetika Molekuler pada tahun ajaran 2012/2013. Penulis aktif di beberapa organisasi, diantaranya sebagai Staf Rohis Kelas Serum-G FMIPA IPB pada tahun 2009-2010, Staf Sosial Kemasyarakatan LDK Al Hurriyyah IPB pada tahun 2008-2010, Staf Kepengurusan Asrama Putri Darmaga pada tahun 2009-2011. Menjadi panitia di berbagai kegiatan kampus dan asrama. Pada tahun 2012 penulis mengikuti Program Kreativitas Mahasiswa dalam bidang Kewirausahaan dengan judul program “GUITEC Rice: Alternatif Diversifikasi Pangan Pertama Di Indonesia dengan Memanfaatkan Bruguiera gymnorrhiza yang Dibalut Daun Tectona grandis” dan berhasil didanai DIKTI.
Pada tahun 2010, penulis melakukan Studi Lapang di Taman Wisata Alam Pangandaran Ciamis, Jawa Barat dengan judul laporan “Pengembangan Produk Kerajinan Berbahan Dasar Keanekaragaman Hayati dan Aspek Pelestariannya di Taman Wisata dan Cagar Alam Pangandaran”. Pada tahun 2011, penulis melakukan Praktik Kerja Lapang di PT. Tirta Investama/ Aqua Plant Citeureup
dari bulan Juli sampai bulan Agustus dengan judul laporan “Analisis Mikrobiologi dari Bahan Baku sampai Produk Jadi Di PT Tirta Investama/ Aqua Plant Citeureup ”.
Sebagai salah satu syarat kelulusan studi di Fakultas Matematika dan Ilmu