i Pa
g
e
i
UKURAN KEMIRIPAN
SWEETSPOT SIMILARITY
PADA
TEMU KEMBALI INFORMASI ANOTASI
GENE ONTOLOGY
BERBASIS SOLR
DIRMAN HAFIZ
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
iii Pa
g
e
iii
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Ukuran Kemiripan Sweetspot Similarity Pada Temu Kembali Informasi Anotasi Gene Ontology Berbasis SOLR adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
DIRMAN HAFIZ. Ukuran Kemiripan Sweetspot Similarity Pada Temu Kembali Informasi Anotasi Gene Ontology Berbasis SOLR. Dibimbing oleh YENI HERDIYENI dan JULIO ADISANTOSO.
Biodiversitas Informatik adalah sebuah upaya membuat sumber informasi keanekaragaman hayati dalam format digital. Biodiversitas informatik menggunakan struktur ontologi dalam memetakan pengetahuannya. Gene Ontology (GO) adalah sebuah ontologi di bidang biologi molekular yang dikembangkan oleh Gene Ontology Consortium. Gene Ontology memiliki 3 komponen, yaitu molecular function, biological process, dan cellular component. Dibutuhkan mesin pencari yang relevan serta mampu mengolah data biodiversitas yang besar. Apache SOLR merupakan mesin pencari yang dibangun pada Apache Lucene. Uji kemiripan pada SOLR Sweetspot similarity dapat mengantisipasi masalah perbedaan panjang anotasi GO dengan melakukan normalisasi panjang dokumen. Normalisasi panjang dokumen ditentukan berdasarkan perbandingan dari beberapa parameter dan didapatkan nilai min 51, max 100, dan steepness 0.4 sebagai acuan yang terbaik. Hasil dari penelitian ini menunjukkan nilai Mean Average Precision dan R-Precision sweetspot similarity lebih baik dibandingkan dengan classic similarity. Kata kunci: Apache SOLR, Biodiversitas Informatic, Gene Ontology, Mesin
pencari, Sweetspot Similarity
ABSTRACT
DIRMAN HAFIZ. Similarity Measure of Gene Ontology Annotation Information Retrieval Based on Sweetspot Similarity using SOLR. Supervised by YENI HERDIYENI and JULIO ADISANTOSO.
Biodiversity Informatics is an effort to make the source of biological diversity information into digital format. Biodiversity Informatics uses ontology structure for knowledge mapping. Gene Ontology (GO) is a field of ontology in molecular biology developed by Gene Ontology Consortium. Gene Ontology has three components, which are the molecular function, biological process, and cellular component. A relevant search engine that can process big data of biodiversity is required. Apache SOLR is a search engine that is built on Apache Lucene. Similarity test in SOLR sweetspot similarity could anticipate GO annotation length differences problems with the normalized length of the document. Normalization length of the document is determined based on a comparison of some parameters and the values of min 51, max 100, and the steepness 0.4 are found to be the best reference. The results of this research show that the MAP and R-Precision value of sweetspot similarity is better than classic similarity.
iii Pa
g
e
iii
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer
pada
Departemen Ilmu Komputer
UKURAN KEMIRIPAN
SWEETSPOT SIMILARITY
PADA
TEMU KEMBALI INFORMASI ANOTASI
GENE ONTOLOGY
BERBASIS SOLR
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2016
v Pa
g
e
v
Judul Skripsi : Ukuran Kemiripan Sweetspot Similarity Pada Temu Kembali Informasi Anotasi Gene Ontology Berbasis SOLR
Nama : Dirman Hafiz NIM : G64120035
Disetujui oleh
Dr Yeni Herdiyeni, SSi MKom Pembimbing I
Ir Julio Adisantoso, MKom Pembimbing II
Diketahui oleh
Dr Ir Agus Buono, MSi MKom Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Januari 2016 ini ialah information retrieval, dengan judul Ukuran kemiripan Sweetspot Similarity Pada Temu Kembali Informasi Anotasi Gene Ontology Berbasis SOLR.
Penulisan skripsi ini merupakan salah satu syarat memperoleh gelar Sarjana Komputer pada Program Studi Ilmu Komputer Institut Pertanian Bogor. Tugas akhir ini tidak mungkin dapat diselesaikan tanpa adanya bantuan dari berbagai pihak. Oleh karena itu, penulis mengucapkan terima kasih dan penghargaan kepada: 1 Ibu penulis Nur Umi Legiatri, Ayah penulis Sulistyono, Kakak penulis Agus Adi Saputro dan Jamilludin Noer, Adik penulis Hendra Satria, dan juga keluarga lainnya yang telah memberikan dukungan, doa serta motivasi untuk keberhasilan studi.
2 Ibu Dr Yeni Herdiyeni, SSi MKom dan Bapak Ir Julio Adisantoso, MKom selaku dosen pembimbing yang telah memberikan bimbingan, saran, arahan dan bantuan selama penyusunan skripsi.
3 Bapak Dr Ir Agus Buono, MSi MKom selaku Ketua Program Studi Ilmu Komputer IPB.
4 Seluruh dosen dan staf pegawai tata usaha Departemen Ilmu Komputer IPB yang telah banyak membantu selama masa perkuliahan hingga penelitian.
5 Teman satu kelompok bimbingan yang senantiasa membantu dan memotivasi dalam penyelesaian penelitian ini.
6 Seluruh teman-teman Program S1 Ilmu Komputer angkatan 49 atas kebersamaan dan persaudaraan selama 3 tahun ini.
7 Sahabat penulis yaitu Amelia Lindani, Basyiru Rahman, Desi Rosdiana, Fide Kristopan, Galih Puspitasari, Isabella Sianturi, Ridwan Agung, Ruth Meliani Hutapea, Umdatul Qori selaku sahabat yang telah memberikan motivasi serta dukungan selama ini.
8 Teman TPB QO8 dan Teman kontrakan Supernova yang telah memberikan memberikan kenangan serta menemani penulis selama kuliah di IPB.
Semoga segala bantuan, bimbingan, motivasi, dan dukungan yang telah diberikan kepada penulis senantiasa dibalas oleh Allah subhanahu wa ta’ala. Semoga karya ilmiah ini bermanfaat dan menambah wawasan bagi pembaca.
v
ii Pa
g
e
vii
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 3
Ruang Lingkup Penelitian 3
TINJAUAN PUSTAKA 3
Web Semantik 3
Ontologi 4
Gene Ontology 4
SOLR 5
Indexing 6
Classic Similarity 6
Sweetspot Similarity 7
Evaluasi 9
METODE 10
Data Penelitian 10
Tahapan Penelitian 10
Pengumpulan Dokumen 11
Kueri 12
Praproses 12
Sweetspot Similarity 14
Evaluasi 15
HASIL DAN PEMBAHASAN 17
Sweetspot Similarity 17
Evaluasi 18
SIMPULAN DAN SARAN 25
Simpulan 25
Saran 25
DAFTAR PUSTAKA 26
LAMPIRAN 27
RIWAYAT HIDUP 51
3 Panjang anotasi Gene Ontology 14
4 Nilai Min Max metode Sweetspot Similarity 15 5 Hasil score anotasi retrieval kueri Pericycle 17 6 Hasil anotasi dari kueri pedicel metode Sweetspot Similarity 18
7 Golden list dari kueri 19
8 Perbandingan nilai MAP dan R-Precision pada parameter min max 20 9 Perbandingan nilai MAP dan R-Precision untuk steepness pada min 51
dan max 100 21
10 Perbandingan nilai MAP dan R-Precision untuk Classic Similarity dan
Sweetspot Similarity 21
11 Hasil anotasi dari kueri Parenchyma metode Classic Similarity 22 12 Hasil anotasi dari kueri Parenchyma metode Sweetspot Similarity 23 13 Hasil anotasi dari kueri Lignin metode Sweetspot Similarity 24
DAFTAR GAMBAR
1 Visualisasi hubungan class, property, dan individual 4
2 Graf dalam GO 5
3 Normalisasi LengthNorm Sweetspot Similarity 8
4 Tahapan penelitian 10
parameter min max terbaik 20
11 Grafik interpolasi 11 titik recall precision nilai steepness 0.4 pada min
51 dan max 100 21
12 Grafik perbandingan interpolasi 11 titik recall precision Sweetspot
Similarity dan Classic Similarity 22
DAFTAR LAMPIRAN
4 Precision Recall Metode Sweetspot Similarity ( Min =101 Max =150
5 Precision Recall Metode Sweetspot Similarity ( Min =151 Max =200
Steepness = 0.5) 31
6 Precision Recall Metode Sweetspot Similarity ( Min =201 Max =250
Steepness = 0.5) 32
7 Precision Recall Metode Sweetspot Similarity ( Min =1 Max =100
Steepness = 0.5) 33
8 Precision Recall Metode Sweetspot Similarity ( Min =1 Max =150
Steepness = 0.5) 34
9 Precision Recall Metode Sweetspot Similarity ( Min =1 Max =200
Steepness = 0.5) 35
10 Precision Recall Metode Sweetspot Similarity ( Min =1 Max =250
Steepness = 0.5) 36
11 Precision Recall Metode Sweetspot Similarity ( Min =51 Max =150
Steepness = 0.5) 37
12 Precision Recall Metode Sweetspot Similarity ( Min =51 Max =200
Steepness = 0.5) 38
13 Precision Recall Metode Sweetspot Similarity ( Min =51 Max =250
Steepness = 0.5) 39
14 Precision Recall Metode Sweetspot Similarity ( Min =101 Max =200
Steepness = 0.5) 40
15 Precision Recall Metode Sweetspot Similarity ( Min =101 Max =250
Steepness = 0.5) 41
16 Precision Recall Metode Sweetspot Similarity ( Min =151 Max =250
Steepness = 0.5) 42
17 Precision Recall Metode Sweetspot Similarity ( Min =51 Max =100
Steepness = 0.2) 43
18 Precision Recall Metode Sweetspot Similarity ( Min =51 Max =100
Steepness = 0.4) 44
19 Precision Recall Metode Sweetspot Similarity ( Min =51 Max =100
Steepness = 0.6) 45
20 Precision Recall Metode Sweetspot Similarity ( Min =51 Max =100
Steepness = 0.8) 46
21 Precision Recall Metode Sweetspot Similarity ( Min =51 Max =100
Steepness = 1) 47
22 Precision Recall Metode Classic Similarity 48
PENDAHULUAN
Latar Belakang
Indonesia adalah negara kepulauan yang memiliki cakupan yang sangat luas dimana didalamnya hidup flora dan fauna yang sangat beragaram. Tingginya keanekaragaman hayati atau yang biasa dikenal dengan biodiversitas dan tingkat endemisme menempatkan Indonesia sebagai laboratorium alam yang sangat unik untuk tumbuhan tropik dengan berbagai fenomenanya (Walujo 2011). Web merupakan salah satu dari sekian banyak cara untuk memperoleh informasi tentang biodiversitas. Jumlah data yang sangat banyak dengan format yang berbeda-beda menimbulkan kesulitan bagi pengguna untuk mengakses informasi (Amanqui et al. 2014).
Kesulitan dalam mengakses informasi biodiversitas dan konsep pemahaman makna dapat diatasi dengan menggunakan konsep semantic web. Semantic web merupakan sebuah generasi baru dari web yang mencoba untuk merepresentasikan informasi yang dapat digunakan oleh mesin, tidak hanya untuk menampilkan, tetapi juga untuk automasi, integrasi dan penggunaan kembali oleh berbagai aplikasi untuk mempermudah pencarian bagi pengguna dan mesin (Amanqui et al. 2014). Semantic web memungkinkan mesin untuk memproses informasi yang tersedia dan akan bertindak layaknya seorang manusia (Mukhopadhyay et al. 2011). Salah satu penerapan konsep semantic web adalah ontologi. Sebagian pengembangan biodiversitas sudah mengimplementasikan semantic web yang menggunakan terminologi ontologi dalam merepresentasikan pengetahuan. Pada sistem Entrez di National Center for Biotechnology (NCBI) menyediakan akses informasi medikal dan informasi tersebut dapat diambil dengan menggunakan ontologi (Sarkar dan Indra 2007).
Ontologi merupakan teknik merepresentasikan suatu pengetahuan yang dapat berupa fakta ataupun ide. Pengetahuan tersebut didefinisikan ke dalam hubungan dan klasifikasi dari suatu konsep pada domain yang spesifik (Jepsen 2009). Saat ini ontologi sudah banyak diterapkan dalam berbagai domain pengetahuan, salah satunya biologi. Gene Ontology adalah sebuah ontologi di bidang biologi molekular yang dikembangkan oleh Gene Ontology Consortium. Dokumen ontologi disimpan dalam format RDF dan OWL yang berbasis representasi pengetahuan pada web, dan mengizinkan pengguna untuk mendefinisikan term, hubungan antar term dan menetapkan batasan-batasan pada data yang terstruktur dengan baik (Mukhopadhyay et al. 2007). RDF merupakan model data dasar untuk penulisan statement sederhana tentang objek web (resource). Model data RDF tidak bersandar pada Extensible Markup Language (XML) namun RDF memiliki sintaks berbasis XML (Antoniou dan Hermalen 2008).
2
2
(platform) pencarian open-source yang dibangun pada Apache Lucene menggunakan bahasa pemrograman Java. Kemampuan SOLR adalah mampu mengolah data dalam jumlah yang besar dan mengindeks secara otomatis. Fitur utama dari SOLR yaitu server yang dapat berkomunikasi melalui HTTP dengan menggunakan XML dan JSON data format, konfigurasi file (Smiley dan Pugh 2011). Sehingga SOLR dapat menginput dokumen ontologi dengan model data RDF dengan melakukan beberapa tahapan perubahan RDF menjadi XML.
Untuk meningkatkan kinerja mesin pencari diperlukan sebuah similarity yang menghasilkan output lebih relevan. Ukuran kemiripan tidak hanya menghitung banyaknya nilai term frekuensi dalam dokumen namun diperlukan normalisasi panjang dokumen. Normalisasi diperlukan karena dokumen yang lebih panjang mengandung nilai term frekuensi (tf) yang lebih besar dibandingkan dengan dokumen yang lebih pendek. sedangkan nilai tf yang lebih besar belum tentu mencirikan dokumen tersebut relevan (Manning dan Raghavan 2008). SOLR memiliki beberapa similarity yang dapat digunakan dalam package similarity, adalah classic similarity dan sweetspot similarity. Classic Similarity merupakan gabungan dari Boolean similarity, TF-IDF dan cosine similarity. Sedangkan Sweetspot similarity merupakan pengembangan dari classic similarity yang berfokus pada normalisasi panjang dokumen. Pada penelitian ini dokumen ontologi yang digunakan memiliki panjang dokumen yang beragam. Normalisasi panjang dokumen dilakukan dengan menggunakan metode sweetspot similarity. Normalisasi panjang dokumen dalam sweetspot similarity memungkinkan panjang dokumen yang lebih besar memiliki nilai normalisasi yang lebih besar dibandingkan dengan panjang dokumen yang lebih sedikit. Hal tersebut agar menyetarakan nilai normalisasi pada panjang dokumen berdasarkan tingkat kepentingan dari panjang dokumen. Pada sweetspot similarity normalisasi panjang dokumen diubah menjadi sebuah nilai dengan interval nol sampai satu. Penelitian yang dilakukan Cohen dan Amiatay (2007) membandingkan beberapa normalisasi panjang dokumen serta melakukan beberapa modifikasi dalam average tf. Nilai evaluasi dalam penelitian tersebut menghasilkan metode sweetspot similarity lebih baik dibandingkan dengan beberapa metode normalisasi panjang dokumen lainnya.
Perumusan Masalah
Rumusan permasalahan dalam penelitian ini, yaitu:
1 Bagaimana menggunakan struktur ontologi untuk pencarian informasi sistem pada temu kembali informasi ?
2 Bagaimana menerapkan sweetspot similarity mengukur kemiripan anotasi pada Gene Ontolology menggunakan SOLR?
3 Bagaimana kinerja sweetspot similarity dibandingkan dengan classic similarity pada SOLR ?
Tujuan Penelitian
Tujuan dari penelitian ini, yaitu:
1 Mengukur kemiripan anotasi Gene Ontology menggunakan metode sweetspot similarity
3
Penelitian ini diharapkan dapat mempercepat proses pencarian pada sistem temu kembali informasi sehingga pengguna bisa mendapatkan dokumen yang lebih relevan, dengan mengetahui metode pengukuran kemiripan yang baik dan efisien.
Ruang Lingkup Penelitian
Ruang lingkup penelitian ini antara lain:
1 Kueri yang digunakan diambil dari buku Anatomy of Flowering Plants pada bagian batang (Rudal 2007).
2 Relasi anotasi Gene Ontology tidak diperhitungkan dalam ukuran kemiripan 3 Penggunaan relasi anotasi Gene Ontology hanya is_a dan berada pada tingkat
level pertama
TINJAUAN PUSTAKA
Semantic Web
Semantic web diperkenalkan oleh Berners-Lee, Hendler dan Lassila pada tahun 2001. Semantik web adalah sebuah teknologi yang bertujuan untuk membuat dokumen web dapat dimengerti oleh komputer (machine-readable data) (Davies et. al 2006). Semantic web merupakan representasi dari data di dalam World Wide Web, berdasar pada Resource Description Framework (RDF) yang mengintegrasikan berbagai aplikasi menggunakan XML sebagai sintaks dan URI sebagai penamaan (Antoniou dan Hermalen 2008). Sebuah semantic search engine meyimpan informasi semantic tentang web resources dan mampu memecahkan suatu kueri yang kompleks. Semantic search membantu agar kueri yang diberikan oleh pengguna dapat dimengerti dengan baik oleh mesin pencari, karena semantik berarti suatu pembelajaran tentang arti yang terkandung dari suatu bahasa, kode, atau jenis representasi lain, sehingga mesin pencari dapat menampilkan hasil yang paling relevan sesuai dengan maksud dari kueri
RDF (Resource Description Framework)
eXtensible Markup Language (XML) adalah suatu bahasa yang direpresentasikan dengan metadata. XML merepresentasikan informasi agar dapat dengan mudah diakses oleh mesin. XML tidak menyediakan tag-tag tertentu, tetapi penggunanya dapat mendefiniskan tag secara mandiri. RDF mengadopsi sintaks-sintaks yang dimiliki oleh XML. Menurut Antoniou dan Hermalen (2008), RDF memiliki konsep dasar RDF yaitu resource, properties, dan statement. Resource adalah objek atau tentang sesuatu yang ingin diungkapkan. Beberapa contoh resource seperti judul buku, pengarang, penerbit, orang dan sebagainya. Setiap resource memiliki Universal Resource Identifier (URI). Properties adalah deskripsi hubungan antar resource, misalnya properti yang menghubungkan antara buku
4
4
Flower
Color Ontologi
Ontologi merupakan teknik merepresentasikan suatu pengetahuan yang dapat berupa fakta ataupun ide. Pengetahuan tersebut didefinisikan ke dalam hubungan dan klasifikasi dari suatu konsep pada domain yang spesifik (Jepsen 2009). Pengetahuan yang didefinisikan ke dalam hubungan dimaksudkan agar mesin dapat membaca dan mengerti konsep dari data yang bersifat abstrak atau dikenal dengan machine readable, adapun pengetahuan tersebut dapat direpresentasikan dalam sebuah model (Bermejo 2007).
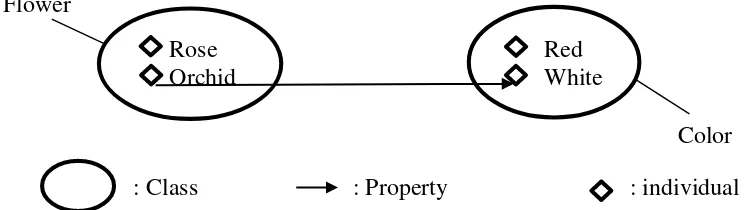
Ontologi memiliki tiga komponen dasar, yaitu class, property, dan individual (Liu dan Ozsu 2009). Class merepresentasikan konsep dari entitas dalam suatu domain. Class merupakan himpunan abstrak dari sebuah objek. Di dalam suatu class dapat terkandung suatu individual atau class lain. Sebuah class dapat dijelakan menggunakan property, misalnya pada class flower memiliki property yaitu HasColor. Selain itu, suatu class dapat mengandung beberapa individual, misalnya class Color memiliki beberapa individual yaitu red dan white. Visualisasi hubungan class, property, dan individual dapat dilihat pada Gambar 1.
Rose Red
Orchid White
: Class : Property : individual Gambar 1 Visualisasi hubungan class, property, dan individual
Gene Ontology
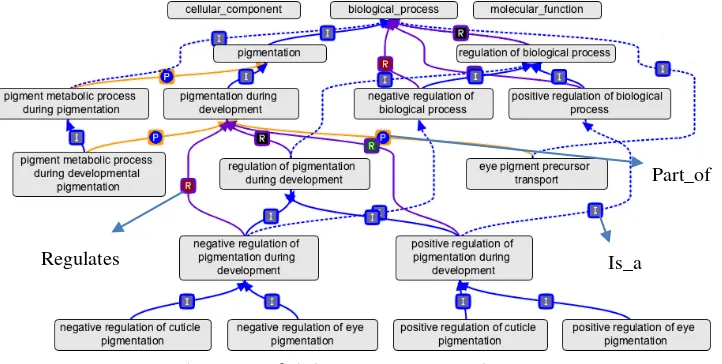
Gene Ontology (GO) merupakan sebuah ontologi yang menjelaskan tentang biologi molekular yang dikembangkan oleh Gene Ontology Consortium dan dapat diunduh pada situs geneontology.org. Gene Ontology yang mengandung sekitar 30.000 term ini memiliki 3 komponen, yaitu molecular function, biological process, dan cellular component (Consortium 2004). Cellular components (CC), merupakan sebuah komponen sel dan penjelesan struktur subselular dan makromolekuler yang lebih kompleks. Molecular Function (MF), menggambarkan aktivitas-aktivitas dari sel namun tidak menentukan dimana, kapan, atau dalam konteks apa aktitivitas tersebut terjadi. Biological process (BP), menjelaskan tujuan biologis yang dicapai oleh satu atau lebih molecular function. Perbedaan antara biological process dan molecular function adalah bahwa secara umum sebuah proses harus memiliki lebih dari satu langkah berbeda.
5
5
misalnya pada pigmentation merupakan bagian dari biological process. Relasi part_of (P) GO digunakannya untuk representasi bagian dari substan dan proses dan dari fungsi atau aktivitas, misalnya pada pigment metabolic process during development merupakan representasi dari bagian pigmentation pigment during development. Regulates (R) menjelaskan bahwa suatu proses secara langsung mempengaruhi manifestasi dari proses lain, misalnya pigmentation during development dipengaruhi oleh manifestasi dari negative regulation of pigmentation during development dan positive regulation of pigmentation during development. Kosa-kata dalam GO tersusun sebagai sebuah rooted Directed Acyclic Graph (rDAG) atau sebuah graf berarah yang mempunyai node akar sebagai parent dan tidak memiliki cycle. Anotasi dalam gene ontologi memiliki beberapa kategori diantaranya label, definisi, sinonim, dan relasi hubungan konsep seperti yang dijelaskan dalam Gambar 2. Label merupakan sebuah judul dari anotasi. Definisi merupakan penjelasan secara spesifik dari anotasi. Sedangkan sinonim merupakan makna lain dari sebuah anotasi yang memiliki kemiripan makna. Visualisasi graf GO terdapat dalam gambar 2.
Gambar 2 Graf dalam GO (genontology.org)
SOLR
SOLR merupakan aplikasi mesin pencari yang dibangun di atas Lucene sebagai mesin indexing dan berbasis enterprise. SOLR dapat menangani pencarian pada data dokumen yang besar secara cepat, dan akurat, karena sintaks untuk mencari dokumen yang digunakan adalah NoSQL (Shahi 2015). Apache SOLR memiliki fitur utama seperti mencari teks dengan cepat, fected search, pengelompokan secara dinamis, integrasi database, menangani beragam dokumen input seperti PDF, Word dan lain-lain. Kelebihan utama SOLR dibanding Lucene yaitu server yang dapat berkomunikasi melalui HTTP dengan menggunakan format data XML dan JSON, konfigurasi file, cache untuk mempercepat respon pencarian, administrative interface berbasis web, faceting hasil pencarian, dan geospatial search (Smiley dan Pugh 2011).
SOLR terdiri atas sistem admin dan rancangan contoh user interface untuk melakukan pencarian. Sistem admin digunakan untuk membuat core yang berisi dokumen korpus, menginput dokumen, menganalisa dokumen yang diinput,
Part_of
6
6
menganalisa hasil pencarian dan lain sebagainya. Sementara untuk melakukan pencarian dapat langsung menggunakan browse. BioSolr merupakan salah satu pengembangan SOLR untuk penelitian biodiversitas dengan struktur ontologi. BioSolr sudah diimplementasikan ke bentuk web yaitu pada http://www.ebi.ac.uk/
Indexing
Inverted Index merupakan proses untuk mengurutkan kata yang berada dalam dokumen setelah melalui proses tokenisasi, case folding, dan pembuangan stopwords berdasarkan lokasi dokumen dimana kata tersebut berada (Mahapatra dan Biswas 2011). SOLR menggunakan inverted index dalam menyimpan hasil indexing. Hal ini dikarenakan inverted index dapat memfasilitasi pencarian yang dilakukan dengan sangat cepat dan mampu menangani kueri masukan meskipun jumlahnya banyak (McCandless et al. 2010). Proses indexing dalam penelitian ini dilakukan oleh Apache SOLR setelah dokumen dimasukan ke dalam tool document input Apache SOLR.
Classic Similarity
Scoring merupakan bagian yang digunakan untuk memberikan bobot terhadap dokumen yang akan ditampilkan saat user memberikan kueri masukan ke dalam mesin pencari. Scoring menentukan urutan dokumen yang ditampilkan berdasarkan nilai pembobotan yang dihasilkan (Graigger dan Potter 2014). Ukuran kemiripan default pada SOLR yaitu Classic Similarity. Classic Similarity merupakan gabungan dari Boolean similarity, TF-IDF dan cosine similarity. Boolean melakukan filter pada dokumen yang mengandung kata pada kueri masukan, TF-IDF melakukan pembobotan terhadap kata dalam dokumen dan kumpulan dokumen sedangkan cosine similarity melakukan uji kemiripan dokumen dengan kueri menggunakan ukuran kemiripan vektor, dimana setiap kueri masukan dan term di dalam dokumen dibandingkan. Pengukuran dilakukan dengan membandingkan sudut yang dibentuk antara kueri dan dokumen yang ada. Setiap kata dalam dokumen direpresentasikan ke dalam vektor sehingga dapat ditentukan nilai jarak yang menggambarkan hubungan antara kueri dan dokumen (Manning dan Raghavan. 2008)
Formula scoring classic similarity dalam Lucene pada Persamaan 1.
7 ukuran untuk seberapa banyak dokumen yang relevan dengan faktor banyaknya term kueri. Persamaan 4 menunjukkan implementasi dari coord factor, dengan max overlaps merupakan jumlah kata pada kueri dan overlaps merupakan jumlah dokumen yang mengandung kata pada kueri. Pada penelitian ini tidak menggunakan formula coord factor dikarenakan panjang kueri tidak mempengaruhi metode yang digunakan.
=√ . � 2 × ∑1 × .� 2 [5]
qN(q) atau queryNorm merupakan nilai normalisasi pada kueri, bertujuan untuk melakukan normalisasi terhadap nilai scoring antara kueri yang ada dengan seluruh dokumen. q.getboost menyatakan boosting yang diberikan pada sebuah kueri dan t.Bst adalah nilai boost pada term (t) untuk kueri (q) pada saat pencarian yang diminta oleh user. Apabila tidak terdapat boost pada kueri maka secara otomatis nilai queryNorm bernilai satu
, = ℎ . ∏ . [6]
LengthNorm = 1
√L [7]
Fungsi N(t,d) merupakan Normalisasi term dalam dokumen. Dalam normalisasi tersebut terdapat enkapsulasi beberapa boost pada field pada saat
indexing dan faktor normalisasi panjang dokumen. Fungsi . menyatakan
boost yang dilakukan terhadap dokumen atau field saat proses indexing dan L merupakan panjang dari suatu dokumen. Pada Persamaan 7 fungsi LengthNorm merupakan normalisasi panjang dokumen. Dokumen yang memiliki kata paling pendek dalam sekumpulan dokumen akan mendapatkan bobot lebih besar karena memuat kepentingan kueri pada dokumen tersebut lebih besar dari yang lain. Namun, hasil dari nilai norm akan dilakukan proses pengubahan float menjadi single byte sebelum disimpan. Pada saat Pencarian, nilai byte norm dibaca berdasarkan index directory dan kemudian di ubah kembali menjadi nilai float. Pengubahan float menjadi single byte akan mengurangi ukuran index. Kompresi nilai norm akan menghemat memori saat pencarian, dikarenakan saat pencarian ke masing-masing field, normalisasi ke semua dokumen dilakukan di dalam memori.
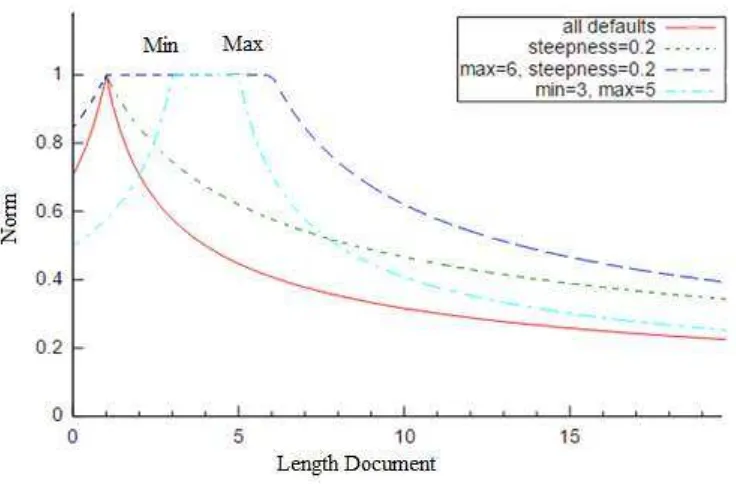
Sweetspot Similarity
8
8
lebih besar dibandingkan dengan panjang dokumen yang lebih sedikit. Hal tersebut agar menyetarakan nilai normalisasi pada panjang dokumen berdasarkan tingkat kepentingan dari panjang dokumen. Pada sweetspot similarity normalisasi panjang dokumen diubah menjadi sebuah nilai dengan interval nol sampai satu.
LengthNorm = 1
√ � × |L− i | + |L+ ax|− ax− i +1 [8]
Gambar 3 Normalisasi LengthNorm Sweetspot Similarity
9
Evaluasi merupakan cara yang digunakan untuk mengukur kinerja sistem temu kembali informasi. Metode yang umum digunakan dalam evaluasi adalah precision dan recall. Precision menyatakan rasio dokumen yang relevan dari seluruh dokumen yang ditampilkan. Sedangkan recall menyatakan rasio dokumen yang ditampilkan dari seluruh dokumen yang relevan. (Manning dan Raghavan 2008). Formula untuk precision dan recall dapat dilihat pada Persamaan 9 dan Persamaan 10.
Tabel 1 Confusion Matriks
Relevant Nonrelevant
Retrieved True positives (tp) False positives (fp)
Not retrieved False negatives (fn) True negatives (tn)
� � = / + [9]
= / + [10]
Pada Tabel 1, True positve merupakan jumlah dokumen relevan yang dapat ditampilkan oleh sistem. False positve merupakan jumlah dokumen yang tidak relevan yang ditampilkan oleh sistem. True negative merupakan jumlah dokumen yang tidak relevan dan tidak ditampilkan oleh sistem. False negative merupakan jumlah dokumen relevan dan tidak ditampilkan oleh sistem. Ukuran efisiensi dari sebuah mesin pencari dapat dilihat dengan grafik interpolasi yang digambarkan Pada kurva precision and recall dengan melihat plot pada 11 titik recall (Manning dan Raghavan 2008). Pengukuran efisiensi dari sebuah mesin pencari dilihat dari nilai MAP. MAP (Mean Average Precision) merupakan standarisasi pengukuran kualitas hasil pencari yang telah disepakati oleh TREC (Text Retrieval Community). MAP dapat menggambarkan perbedaan yang baik diantara dua jenis hasil mesin pencari, MAP menghitung nilai rataan precision dari sejumlah kueri yang diujikan, Persamaan MAP dapat dilihat pada formula berikut:
= 1
10
10
METODE
Data Penelitian
Data yang digunakan pada penelitian ini sebagai dokumen input adalah dokumen Gene Ontology yang diperoleh dari website geneontology.org. Dokumen yang dimaksud adalah anotasi yang ada di dalam GO.
Tahapan Penelitian
Tahapan yang dilakukan pada penelitian ini dapat dilihat pada Gambar 4. Penelitian dimulai dengan mengumpulkan dokumen ontologi dan kueri, kemudian melakukan praposes dalam SOLR, melakukan pengukuran kemiripan dengan metode Sweetspot Similarity dan evaluasi dengan menghitung nilai precision, recall, MAP, dan R-Precision.
11
Dokumen didapatkan dengan mengunduh dokumen Gene Ontology dari situs geneontology.org yang berjumlah 44698 anotasi. Format GO yang diunduh adalah go.owl (RDF-XML) yang memiliki hubungan hierarki dasar seperti is_a dan part_of dapat dilihat pada Gambar 5.
<rdf:RDF>
<go:term rdf:about="http://www.geneontology.org/go#GO:0000001"> <go:accession>GO:0000001</go:accession>
<go:name>mitochondrion inheritance</go:name> <go:synonym>mitochondrial inheritance</go:synonym> <go:definition>The distribution of mitochondria
<go:is_a rdf:resource="http://www.geneontology.org/go#GO:0048308" /> <go:is_a rdf:resource="http://www.geneontology.org/go#GO:0048311" />
Gambar 5 Metadata RDF GeneOntology
Mengubah format RDF-XML menjadi XML dilakukan dengan melakukan transformasi dengan menggunakan perintah berikut setelah meng-install package xsltproc pada sistem komputer:
$ xsltproc –novalid go2solr.xsl [nama_file] > output.xml
Lampiran 23 menjelaskan proses transformasi yang dilakukan program xsltproc. Field yang terdapat dalam anotasi semuanya diubah kedalam sintaks XML. Namun pada hubungan relasi is_a dan part_of penunjukkan value berdasarkan dari
rdf:resource . Output yang dihasilkan dari transformasi tersebut adalah anotasi-
anotasi dalam GO yang ditandai oleh tag doc. Contoh anotasi GO metadata XML dapat dilihat pada Gambar 6.
<doc>
<field name="id">GO:0000001</field>
<field name="go_name">mitochondrion inheritance</field> <field name="namespace">biological_process</field>
<field name="go_definition">The distribution of mitochondria <field name="go_synonym">mitochondrial inheritance</field> <field name="is_a">GO:0048308</field>
<field name="is_a">GO:0048311</field> </doc>
Gambar 6 Metadata XML GeneOntology
Selanjutnya dokumen XML hasil transformasi dimasukkan ke dalam server SOLR. Konfigurasi yang harus dilakukan agar SOLR dapat mengenali serta mengetahui apa saja yang akan diambil dari dokumen XML tersebut. Mengubah schema.xml pada konfigurasi SOLR dengan mendaftarkan field mana saja dalam dokumen ontologi yang akan diindeks dan menentukan tipe serta bagaimana field tersebut akan di-parsing.
<field name=”go_name” type=”text_general” indexed=”true” stored=”true” multiValued=”false”/>
<field name=”go_synonym” type=”text_general” indexed=”true”
stored=”true” multiValued=”true”/>
12
12
<field name=”id” type=”string” indexed=”true” required=”true” stored=”true” multiValued=”false”/>
Atribut type berfungsi untuk mengetahui tahap praproses apa yang harus dilakukan SOLR terhadap dokumen. Atribut stored berfungsi menyimpan setiap value yang ada pada dokumen sehingga dapat ditampilkan. Atribut multivalued berfungsi agar sistem dapat menyimpan single atau multivalue. Dan atribut indexed berfungsi melakukan indexing dan perhitungan pada setiap field. Field dengan indexed =
true memiliki makna bahwa field tersebut akan ikut diindeks dalam SOLR. Field
dengan required = true memiliki makna bahwa field tersebut wajib ada dalam
setiap anotasi pada GO, contohnya ID. Sedangkan field dengan multivalued =
true berarti bahwa field tersebut dapat memiliki nilai lebih dari satu, seperti
go_synonym.
Kueri
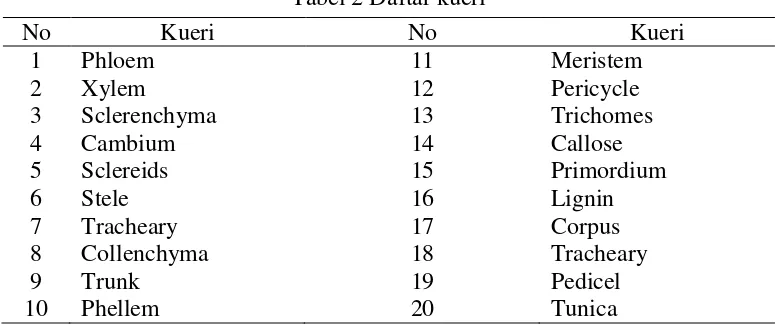
Kueri diperoleh dari buku Anatomy of Flowering Plants (Rudall 2007) sebanyak 20 term pada bagian tentang batang. Daftar kueri yang digunakan dapat dilihat pada Tabel 2. Definisi dari masing-masing daftar kueri dapat dilihat pada Tabel Lampiran 1.
Tabel 2 Daftar kueri
No Kueri No Kueri
1 Phloem 11 Meristem
2 Xylem 12 Pericycle
3 Sclerenchyma 13 Trichomes
4 Cambium 14 Callose
Pada tahap ini dilakukan pengolahan dokumen yang merupakan bahan mentah menjadi kata-kata yang siap dihitung bobotnya. Tahap ini dilakukan secara otomatis dalam SOLR. Tahapan yang dilakukan pada praproses dapat dilihat pada Gambar 7.
Gambar 7 Tahapan praproses 1 Tokenisasi
Tahap ini dilakukan dengan memotong string masukan berdasarkan tiap kata yang dimasukan dalam sebuah array. Pada umumnya setiap kata teridentifikasi dan terpisahkan dengan kata lain oleh karakter spasi. Proses tokenisasi dilakukan oleh Apache SOLR dengan memotong setiap kalimat yang terdapat dalam dokumen menjadi potongan-potongan kata.
Dokumen
13
Pada tahap ini dilakukan pengubahan semua huruf dalam dokumen menjadi huruf kecil. Karakter selain huruf dihilangkan dan dianggap sebagai delimiter (pembatas).
3 Filtering
Stopwords merupakan kata umum yang sering muncul dalam suatu dokumen dengan jumlah besar namun seringkali tidak memiliki makna, sehingga dapat diabaikan di dalam pengolahan. Stopword dibuang untuk meringankan komputasi dan mempercepat waktu pemrosesan pencarian. Sebelumnya daftar kata yang digunakan sebagai stopword dikumpulkan dalam suatu file. Kemudian sistem membaca file tersebut untuk dilakukan penghapusan ketika kata dalam dokumen identik dengan kata yang berada dalam file stopwords.
Tahapan praproses dilakukan secara otomatis dalam SOLR, terbagi atas empat tahap sesuai pada Gambar 7. Perlu dilakukan konfigurasi praproses pada skema SOLR dapat dilihat pada Gambar 8.
<fieldType name="text_general" class="solr.TextField" positionIncrementGap="100">
<analyzer type="index">
<tokenizer class="solr.StandardTokenizerFactory"/> <filter class="solr.StopFilterFactory" ignoreCase="true
words="stopwords.txt" />
<filter class="solr.LowerCaseFilterFactory"/> </analyzer>
Gambar 8 Konfigurasi praproses pada skema SOLR
Proses tokenisasi dilakukan setelah dokumen diinput kedalam SOLR. Tokenisasi dilakukan pada tahap awal indexing dengan cara memisahkan setiap kata yang ada dalam dokumen, salah satunya dengan mengidentifikasi karakter spasi. Dalam proses ini juga dilakukan penghapusan pada tag-tag XML pada dokumen awal. Pada skema dalam SOLR tokenisasi ditandai dengan tag tokenizer dan merupakan proses yang pertama kali dilakukan di bagian analyzer.
Proses case folding dilakukan setelah dokumen atau anotasi dalam GO sudah menjadi token-token. Pada skema SOLR proses case folding ditandai dengan tag filter pada kelas LowerCaseFilterFactory untuk mengubah semua huruf menjadi huruf kecil.
14
14
Sweetspot Similarity
Tahap pengukuran kemiripan dilakukan setelah praproses. Penelitian ini menggunakan metode Sweetspot Similarity, sehingga metode similarity default yang ada di SOLR harus diubah terlebih dahulu dengan cara menambah pengaturan berikut pada skema SOLR.
<similarity similarity class="solr.SchemaSimilarityFactory" /similarity>
kemudian pada Fieldtype tambahkan pengaturan sweetspot similarity dan paramater min, max, dan steepness. Masukkan nilai min, max pada Tabel 4, dan nilai steepness 0.5.
<similarity class="solr.SweetSpotSimilarityFactory"> <int name="lengthNormMin">1</int>
<int name="lengthNormMax">1</int>
<float name="lengthNormSteepness">0.5</float> </similarity>
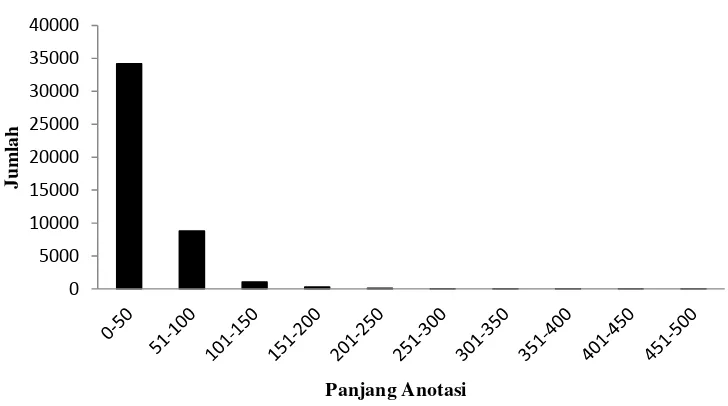
Tahap dilakukan penentuan nilai min max sesuai dengan karakteristik panjang dokumen anotasi Gene Ontology yang dapat dilihat pada Tabel 3.
15
15
Gambar 9 Diagram pesebaran jumlah panjang anotasi GO
Berdasarkan Tabel 3 dan Gambar 9 ditentukan 15 kombinasi min max yaitu dengan jumlah dokumen yang lebih dari 100. Kombinasi min max dapat dilihat pada Tabel 4.
Tabel 4 Nilai Min Max metode Sweetspot Similarity
No. Min Max No. Min Max kemiripannya dengan kueri. Setelah menemukan parameter min dan max dengan nilai evaluasi terbaik maka tahap selanjutnya adalah menentukan nilai evaluasi untuk parameter steepness yang terbaik. Parameter steepness yang diuji adalah 0.2; 0.4; 0.6; 0.8; 1.0.
Evaluasi
Pada tahap ini dilakukan evaluasi untuk mengukur kinerja dari masing-masing kombinasi paramater min, max, dan steepnes sweetspot similarity dan membandingkan dengan Classic Similarity, yang merupakan metode pengukuran kemiripan default pada SOLR. Evaluasi dilakukan dengan membandingkan nilai precision,recall, MAP, dan R-Precision pada masing-masing metode.
16
16
Lingkungan Pengembangan
Penelitian ini dilakukan dengan menggunakan perangkat keras dan perangkat lunak sebagai berikut:
Perangkat keras berupa komputer personal dengan spesifikasi sebagai berikut:
• Processor Intel Core i3-2100
• RAM 4 GB
• 14.0” WXGA LED
• 500 GB HDD
Perangkat lunak yaitu sebagai berikut:
• Sistem operasi Windows 8
• Bahasa pemrograman Python 2.7
• Notepad++ dan Sublime Text 3.0 sebagai text editor
17
Kueri berdasarkan pada Tabel 2 diukur kemiripannya dengan hasil indexing yang dilakukan oleh SOLR menggunakan sweetspot similarity. Terdapat dua contoh yang akan dibahas adalah kueri “pericycle” dan “pedicel” .Pericycle merupakan selapis sel parenkima yang terletak antara endodermis dan silinder pusat. Pada hasil pencarian menggunakan SOLR, SOLR mengembalikan beberapa dokumen yang dianggap relevan berikut informasi mengenai jumlah dokumen yang dikembalikan dan Penjelasan singkat mengenai perhitungan score. Pada Tabel 5 dapat dilihat hasil anotasi pada kueri “pericycle” dengan menggunakan nilai min 51, max 100, dan steepness 0.4 sebagai parameter sweetspot similarity
Tabel 5 Hasil score anotasi retrieval kueri Pericycle
GO_ID Tf idf queryNorm FieldNorm LengDoc Score
GO:0010311 1 106.531 1 1 60 106.531
GO:0048527 1 106.531 1 1 54 106.531
GO:0010102 1 106.531 1 0.5 48 53.265
Pada tabel 5 dijelaskan bahwa pada kueri “pericycle” dihasilkan nilai tf yang merupakan akar dari jumlah term yang mengandung kata “pericycle” pada setiap anotasi. Nilai idf didapat dari hasil Persamaan 3 dengan nilai numdocs adalah 44698 dan docfreq bernilai tiga. Nilai queryNorm secara otomotis bernilai satu dikarenakan pada metode penelitian ini tidak menggunakan boosting untuk kueri. Nilai fieldNorm merupakan nilai yang didapat normalisasi panjang dokumen namun pada metode sweetspot similarity terdapat parameter min, max dimana panjang dokumen akan dinormalisasikan ke menjadi nilai interval nol sampai satu. Panjang dokumen yang berada diantara nilai min dan nilai max akan bernilai satu. Sedangkan range panjang dokumen diluar nilai max dan min mengalami normalisasi panjang dokumen sesuai dengan Persamaan 7. Pada kueri “pericycle” untuk GO:0010311 dan GO:0048527 fieldNorm bernilai satu dikarenakan panjang dokumen kedua anotasi tersebut berada pada range panjang dokumen nilai min dan max. Sedangkan untuk nilai fieldNorm dengan GO:0010102 bernilai 0.5 dikarenakan panjang anotasi GO:0010102 berada di luar range panjang dokumen nilai min dan nilai max. Urutan berdasarkan dengan besarnya nilai score dan apabila nilai score sama seperti pada GO:0010311 dan GO:0048527 maka urutan berdasarkan letak anotasi dalam kumpulan dokumen.
Contoh kasus kelemahan metode sweetspot similarity
18
18
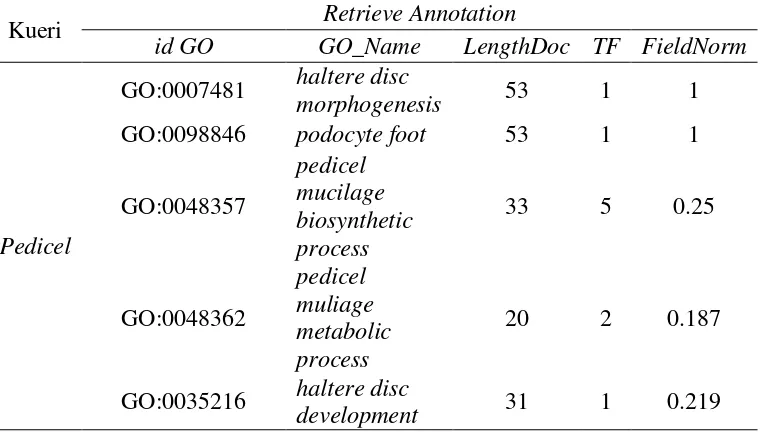
memiliki arti penting mengenai pedicel namun keluaran hasil anotasi menempatkan pada urutan tearatas. Sedangkan GO:0048357 yang berlabelkan pedicel mucilage biosynthetic process termasuk dalam kategori biology process memiliki TF paling besar, kata “pedicel” terdapat empat pada field synonym dan satu pada field label. Isi anotasi ini merupakan proses kimia dari pedicel mucilage. GO_id ini berada pada urutan ketiga dikarenakan panjang dokumen berada diluar range panjang anotasi min dan max.
Tabel 6 Hasil anotasi dari kueri pedicel metode Sweetspot Similarity
Kueri Retrieve Annotation
Tahap evaluasi dimulai dengan mengumpulkan kueri beserta dokumen-dokumen yang dianggap relevan terhadap kueri tersebut. Dokumen yang diambil dari situs Ontobee ontobee.org dengan menggunakan Sparql untuk suatu kueri diasumsikan relevan dengan kueri tersebut. Berikut kode Sparql yang dimasukkan: PREFIX obo-term: <http://purl.obolibrary.org/obo/>
SELECT ?s ?label ?definition
FROM <http://purl.obolibrary.org/obo/merged/GO> { ?s a owl:Class .
?s rdfs:label ?label .
?s obo-term:IAO_0000115 ?definition . FILTER regex(?definition, "<term-query>") .}
Pada Baris satu dari SPARQL yang digunakan menyatakan pendefinisian PREFIX rdfs untuk http://purl.obolibrary.org/obo/. Baris dua menyatakan penggunaan fungsi SELECT untuk memilih isi dari variabel subjek label dan definition. Tanda “?” pada SPARQL tersebut menyatakan suatu variabel. Baris tiga menyatakan fungsi penggunakan FROM untuk merujuk pada http://purl.obolibrary.org/obo/merged/GO. Baris empat menjelaskan bahwa variabel subjek berasal dari PREFIX owl:Class. Baris lima menjelaskan dimana
19
19
fungsi FILTER untuk menyaring hasil pemilihan bersyarat berupa definition yang mengandung dari input “term-query”
Tabel 7 Golden list dari kueri Kueri Jumlah relevan
Phloem 11
Xylem 15
Sclerenchyma 3
Cambium 3
Sclereids 1
Stele 1
Parenchyma 7
Collenchyma 1
Trunk 36
Phellem 1
Meristem 57
Pericycle 3
Trichomes 2
Callose 25
Primordium 29
Lignin 35
Corpus 13
Tracheary 1
Pedicel 5
Tunica 2
Evaluasi percobaan nilai min dan max
20
20
Gambar 10 Grafik perbandingan interpolasi 11 titik recall precision 3 nilai parameter min max terbaik
Tabel 8 Perbandingan nilai MAP dan R-Precision pada parameter min max
No. Min Max MAP R-Precision
Pada gambar 10 dapat dilihat bahwa nilai mn 51 max 100 relatif lebih baik dibanding kedua kombinasi min max lainnya. Untuk nilai min 1 max 50 dan min 1 max 100 pada saat recall 0.2 sampai recall 0.9 memiliki nilai precision lebih kecil. Hal tersebut disebabkan oleh beberapa kueri seperti “xylem”, “phloem”, “callose”,
“parenchyma”, “primordium”, “trunk” memiliki nilai precision semakin menurun seiring interpolasi nilai recall. Nilai MAP dan R-Precision yang dihasilkan pada Tabel 8 menunjukkan nilai yang relatif sama. Namun pada min 51 dan max 100 nilai MAP dan R-Precision menunjukkan nilai yang terbaik
Evaluasi percobaan nilai steepness
Pada Tabel Lampiran 17 sampai 21 menunjukkan nilai percobaan steepness memiliki nilai 11 titik recall relatif sama. Pada tabel 9 menunjukkan nilai MAP yang sama dan R-Precision menunjukkan nilai steepness 0.4 terbaik diantara lainnya termasuk nilai steepness default. Hal tersebut menunjukkan nilai steepness
21
21
juga mempengaruhi hasil retrieve yang dikeluarkan. Interpolasi 11 titik recall precision untuk percobaan nilai steepness dapat dilihat pada Gambar 11.
Tabel 9 Perbandingan nilai MAP dan R-Precision untuk paramater steepness pada minimal 51 dan maximal 100
Min 51 Max
100
Steepnesss MAP R-Precision
0.2 0.898 0.799
Gambar 11 Grafik interpolasi 11 titik recall precision nilai steepness 0.4 pada min 51 dan max 100
Perbandingan Classic Similarity dan Sweetspot Similarity
Setelah ditentukan parameter sweetspot yang terbaik yaitu nilai min 51, max 100, dan steepness 0.4 kemudian dibandingkan dengan classic similarity dengan menggunakan evaluasi MAP yang dapat dilihat pada Tabel 10 dan interpolasi 11 titik recall precision pada gambar 12.
Tabel 10 Perbandingan nilai MAP dan R-Precision untuk Classic Similarity dan Sweetspot Similarity
Ukuran Kemiripan MAP R-Precision
Classic Similarity 0.865 0.797
Sweetspot Similarity 0.898 0.802
Metode ukuran kemiripan default yang ada pada SOLR, yaitu classic similarity juga menggunakan kueri dan golden list yang sama. Pada Tabel 10 menunjukkan metode classic similarity menghasilkan nilai MAP lebih kecil dibandingkan dengan metode Sweetspot Similarity dapat dilihat pada Tabel 11. Hal ini bearti urutan anotasi yang relevan yang dikeluarkan metode sweetspot similarity lebih baik dibandingkan dengan classic similarity
22
22
Gambar 12 Grafik perbandingan interpolasi 11 titik recall precision Sweetspot Similarity dan Classic Similarity
Pada Gambar 12 dapat dilihat interpolasi 11 titik recall precision sweetspot similarity lebih baik dibandingkan dengan classic similarity. Nilai 11 titik recall precision sweetspot similarity pada tabel Lampiran 3 menunjukkan saat nilai recall 0 - 0.1, nilai precision menunjukkan sebesar 0.9 sedangkan nilai 11 titik recall precision classic similarity pada tabel Lampiran 22 menunjukkan saat nilai recall 0-0.1, nilai precision menunjukkan di titik 0.882 . Hal tersebut bermakna bahwa dari 10% anotasi yang ditampilkan sweetspot similarity menghasilkan anotasi yang lebih relevan. Ketika recall 0.9 - 1, kedua metode memiliki nilai precision sama yaitu 0.541 . Hal tersebut bermakna dari semua anotasi yang ditampilkan sekitar 89% anotasi relevan yang ditampilkan oleh kedua metode tersebut.
Contoh kasus kueri yang menyebabkan perbedaan nilai evaluasi
Tabel 11 Hasil anotasi dari kueri Parenchyma metode Classic Similarity
23
23
Tabel 12 Hasil anotasi dari kueri Parenchyma metode Sweetspot Similarity
Kueri Retrieve Annotation
Pada Tabel 11 dan 12 terlihat perbedaan urutan pada hasil keluaran anotasi. Perbedaan urutan dikarenakan penggunaan normalisasi panjang dokumen berbeda yang mempengaruhi nilai pada fieldNorm. FieldNorm pada metode classic similarity dihasilkan dari nilai normalisasi panjang dokumen sesuai dengan Persamaan 7 . Sedangkan pada metode sweetspot similarity normalisasi panjang dokumen yang berada pada range min 51 sampai max 100 bernilai satu. Apabila panjang dokumen anotasi berada diluar range maka nilai fieldNorm akan bernilai kurang dari satu. Berdasarkan pada Gambar 9, tingkat kepentingan kueri terhadap dokumen berpengaruh terhadap pesebaran jumlah panjang anotasi. Namun, dalam sweetspot similarity tingkat kepentingan kueri terhadap dokumen tertinggi bukan berada pada jumlah panjang dokumen yang terkecil. Jumlah panjang anotasi pada range 51 – 100 memiliki tingkat kepentingan terhadap kueri lebih besar dibandingkan dengan panjang anotasi pada range 1 – 50 ataupun range panjang anotasi lainnya sesuai pada tabel 4.
Pada Golden list kueri anotasi “parenchyma” yang relevan berjumlah tujuh sedangkan yang dikeluarkan keduanya metode tersebut adalah enam anotasi yang dikeluarkan. Terdapat satu anotasi yang dianggap tidak termasuk dalam daftar golden list yaitu GO:1903866. Id tersebut dianggap tidak relevan karena term parenchyma berada pada field synonym. Golden list yang didapat menggunakan sparql ontobee hanya mencari term pada bagian label dan definisi dalam setiap anotasi. Sehingga banyak term yang terdapat pada field synonym dinyatakan tidak relevan oleh golden list. Pada classic similarity id yang tidak relevan tersebut berada pada urutan pertama sedangkan pada sweetspot similairity berada pada urutan terakhir.
24
24
tidak relevan pada kedua metode. Nilai R-Precision yang didapatkan pada kedua metode adalah 0.714. Hal tersebut dikarenakan jumlah anotasi pada golden list untuk kueri “parenchyma” yang dapat dilihat pada tabel 7 berjumlah tujuh. Sedangkan nilai dokumen yang relevan untuk urutan sampai ke tujuh adalah lima anotasi yang relevan.
Contoh Hasil anotasi panjang dokumen yang beragam
Tabel 13 Hasil anotasi dari kueri Lignin metode Sweetspot Similarity Retrieve Annotation
Pada kueri “lignin” jumlah anotasi yang dikeluarkan sistem adalah 31. Dari 31 jumlah anotasi yang dikeluarkan terdapat beragam panjang anotasi. Normalisasi dilakukan berdasarkan dari penetapan paramater sweetspot similarity yaitu nilai min 51, max 100, dan steepness 0.4. Keberagaman panjang anotasi akan dinormalisasi menjadi nilai 1 apabila panjang anotasi berada pada diantara min dan max seperti pada GO:0048224. Tabel 13 menunjukkan dalam sweetspot similarity tingkat kepentingan kueri terhadap dokumen pada range 51 – 100 memiliki nilai tertingi dibandingkan dengan panjang anotasi pada range 1 - 51 dan 101 – 150. Hal tersebut menunjukkan penyetaraan normalisasi yang dilakukan sweetspot similarity melihat dari sudut karakteristik panjang dokumen yang ditunjukkan pada Gambar 9 terhadap kepentingan kueri.
Contoh kasus nilai precision dan recall terendah
Pada hasil 11 titik recall precision yang terdapat pada tabel Lampiran 2 sampai tabel Lampiran 22 terdapat kueri yang menunjukkan nilai precision dan nilai recall nol. Selain disebabkan oleh hasil term yang dikeluarkan sistem berada pada field synonym, term kueri yang di cari oleh sistem berupa exact match sehingga term yang memiliki kemiripan tidak teretrieve oleh sistem, contohnya pada kueri
25
25
SIMPULAN DAN SARAN
Simpulan
Penelitian ini menggunakan anotasi Gene Ontology sebagai dokumen input dan metode Sweetspot Similarity sebagai metode ukuran kemiripan yang digunakan. Metode Sweetspot Similarity melakukan normalisasi panjang dokumen dengan parameter nilai min, max, dan steepness. Apabila panjang dokumen berada pada range nilai min dan nilai max maka fieldNorm akan bernilai satu. Pada penelitian ini nilai min, max, dan steepness ditentukan berdasarkan evaluasi MAP , R-Precision, dan gambaran interpolasi 11 titik recall precision. Parameter nilai min 51 dan max 100 menunjukkan nilai MAP, R-Precision, dan interpolasi 11 titik recall precision terbaik. Sedangkan untuk nilai steepness yang menunjukkan nilai MAP dan R-Precision terbaik adalah 0.4. Namun pada evaluasi interpolasi 11 titik recall precision didapatkan hasil yang relatif sama diantara semua percobaan nilai steepness.
Perbandingan antara metode classic similarity dan sweetspot similarity evaluasi yang dilakukan dalam segi MAP dan R-Precision menunjukkan metode sweetspot similarity lebih baik. Sedangkan pada gambaran 11 titik recall precision pada titik recall awal sampai titik recall 0.9 sweetspot relatif lebih baik. Namun pada titik recall satu , titik nilai precision kedua metode adalah sama yaitu 0.541. Beberapa dokumen yang dianggap tidak relevan oleh golden list disebabkan oleh letak term pada anotasi berada pada synonym. Sedangkan golden list yang ada letak term pada anotasi yang berada pada label dan definisi. Selain itu term kueri yang di cari oleh sistem berupa exact match sehingga term yang memiliki kemiripan tidak teretrieve oleh sistem.
Saran
Saran untuk penelitian selanjutnya agar menghasilkan pengembangan yang lebih baik yaitu sebagai berikut:
1 Pada Golden list dilakukan pencarian dokumen yang relevan berdasarkan field synonym.
2 Menarik hierarki lebih banyak dari ontologi, tidak hanya is_a.
26
26
DAFTAR PUSTAKA
Amanqui F K, Serique K J, Cardoso S D, Santos J L D, Albuquerque A, Moreira A. 2014. Improving Biodiversity Data Retrieval Through Semantic Search and Ontologies. WIAT. Doi:10.1109/WI-IAT.2014.44
Antoniou, G and F Hermalen (2008). A Semantic Web Primer, Second edition. Cambridge (GB): The MIT
Ashburner, M. 2000. Gene Ontology Tool for the Unification of Biology. California(US): Stanford University School of Medicine
Bermejo J. 2007. A Simplified Guide to Create an Ontology. Madrid (ES): ASLab. Cohen D, Amiatay E, Camel D. 2005. 1 Million Queries Track. Di Dalam: Lucene and Juru at Trec 2007; 31 September 2005; Haifa,Israel. Haifa(IL): IBM Haifa Research Lab. hlm 3-7.
Consortium GO. 2004. The gene ontology (GO) database and informatics resource. Nucleic Acids Research. 32(90001):258D-261. doi:10.1093/nar/gkh036. Davies J,Studer R,Warren P.2006.Semantic web Teknologies Trends and Research
in Ontology-based Systems. John Wiley & Sons, Chichester.
Graigger T, Potter T.2014. Solr In Action. Shelter Island (US): Manning
Mahapatra AK, Biswas S.2011. Inverted indexes: types and techniques. IJCSI (International Journal of Computer Science Issues). 8(4):384-392.
Manning CD, Raghavan P, Schütze H. 2008. An Introduction to Information Retrieval. Cambridge (UK): Cambridge Univ Pr.
McCandless M, Hatcher E, Gospodnetić O.2010. Lucene in Action. Ed ke-2. Greenwich (US): Manning.
Mukhopadhyay D, Banik A, Mukherjee S, Bhattacharya J, Kim YC. 2007. A domain specific ontology based semantic web search engine. Di dalam: The 7th International Workshop MSPT 2007 Proceedings. 2007 Feb 5. Republic of Korea (KR): Youngil Publication. hlm pp.81-89.
Rudall P.2007. Anatomy of Flowering Plants an Introduction to Structure an Development. Ed ke-3. Leiden(NL):Cambridge University Press.
Shahi D. 2015. Apache Solr a Practical Approach to Enterprise Search. Moodie M, editor. New York (US): Apress.
Smiley D, Pugh E. 2011. Apache Solr 3 Enterprise Search Server. Birmingham: Packt Publishing Ltd.
Sarkar, Indra N.2007. Biodiversity informatics: organizing and linking information across the spectrum of life dalam: Briefings in Bioinformatics 8 (5), pp. 347–357.
27
27
Lampiran 1 Definisi dari term kueri
No Kueri Definisi
1 Corpus bagian pusat titik tumbuh yang memiliki kemampuan membelah
ke segala arah
2 meristem jaringan pada tumbuhan berwujud sekumpulan sel-sel punca yang
aktif melakukan pembelahan sel
3 Tunica lapisan terluar dari titik tumbuh akar adan batang, yang
selanjutnya berkembang menjadi jaringan primer
4 sclereids bagian dari sclerenchyma dimana terjadi dalam berbagai bentu
yang secara luas didistribusikan ke seluruh tanaman
5 Phloem jaringan pengangkut yang berfungsi menyalurkan zat-zat makanan
hasil fotosintesis dari daun ke seluruh bagian tumbuhan
6 Xylem sebagai tempat pengangkutan air dan zat-zat mineral dari akar ke
bagian daun
7 pericycle selapis sel parenkima yang terletak antara endodermis dan silinder
pusat pada akar tumbuhan
8 trichomes tonjolan epidermis yang terdiri dari satu atau lebih sel
9 stele bagian sentral dari akar atau batang yang berisi jaringan yang
berasal dari prokambium tersebut
10 Pedicel batang yang menempel satu bunga ke perbungaan.
11 tracheary bagian xylem yang berfungsi sebagai penghantar air, terdiri dari
trakedi dan pembuluh.
12 sclerenchyma untuk memberikan dukungan mekanik, terutama pada
bagian-bagian tanaman yang tidak lagi memanjangkan.
13 Cambium
lapisan jaringan meristematik pada tumbuhan yang sel-selnya aktif membelah dan bertanggung jawab atas pertumbuhan sekunder tumbuhan
14 collenchyma hidup hanya selama jatuh tempo dan memiliki dua dinding (primer
dan sekunder)
15 callose Pori-pori yang diperkuat oleh trombosit dari polisakarida
16 Parenchyma
untuk membentuk korteks, atau pengisi utama, batang, lapisan dalam sel dalam daun, bahan endosperma yang memberi makan benih tumbuh, dan pulpa buah
17 Primordium Bakal daun tumbuh disisi meristem apikal
18 trunk menyediakan struktur columnar yang kuat dari manacabang yang
melekat
19 phellem produk phellogen yang terbentuk ke arah luar, dalam
penyusunannya biasa disebut sel – sel gabus
20 lignin zat yang bersama-sama dengan selulosa adalah salah satu sel yang
28
28
Lampiran 2 Precision Recall Metode Sweetspot Similarity ( Min =1 Max =50 Steepness = 0.5)
Kueri Titik Recall
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 0,9 1
Corpus 1 1 1 1 1 1 0,9091 0,9091 0 0 0
meristem 1 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0
Tunica 0 0 0 0 0 0 0 0 0 0 0
sclereids 1 1 1 1 1 1 1 1 1 1 1
Phloem 1 1 1 1 0,9231 0,9231 0,9231 0 0 0 0
Xylem 1 1 0,8824 0,8824 0,8824 0,8824 0,8824 0,8824 0,8824 0,8824 0,8824
pericycle 1 1 1 1 1 1 1 1 1 1 1
trichomes 1 1 1 1 1 1 1 1 1 1 1
stele 1 1 1 1 1 1 1 1 1 1 1
Pedicel 1 1 1 1 1 1 1 1 1 1 1
tracheary 0 0 0 0 0 0 0 0 0 0 0
sclerenchyma 1 1 1 1 1 1 1 1 1 1 1
Cambium 1 1 1 1 0 0 0 0 0 0 0
collenchyma 1 1 1 1 1 1 1 1 1 1 1
callose 1 1 0,9615 0,9615 0,9615 0,9615 0,9615 0,9615 0,9615 0,9615 0,9615 Parenchyma 1 1 1 0,8333 0,8333 0,8333 0,8333 0,8333 0 0 0
Primordium 1 1 1 1 1 1 1 1 1 1 0,9667
trunk 1 1 1 1 1 1 0,9722 0,9722 0,9722 0,9722 0
phellem 1 1 1 1 1 1 1 1 1 1 1
lignin 1 1 1 1 1 1 0,9677 0,9677 0,9677 0 0
29
29
Lampiran 3 Precision Recall Metode Sweetspot Similarity ( Min =51 Max =100 Steepness = 0.5)
Kueri Titik Recall
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 0,9 1
Corpus 1 1 1 1 0,9091 0,9091 0,9091 0,9091 0 0 0
meristem 1 1 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0
Tunica 0 0 0 0 0 0 0 0 0 0 0
sclereids 1 1 1 1 1 1 1 1 1 1 1
Phloem 1 1 1 1 1 1 0,9231 0 0 0 0
Xylem 1 1 1 1 1 1 1 1 1 0,9333 0,8824
pericycle 1 1 1 1 1 1 1 1 1 1 1
trichomes 1 1 1 1 1 1 1 1 1 1 1
stele 1 1 1 1 1 1 1 1 1 1 1
Pedicel 1 1 1 1 1 1 1 1 1 1 1
tracheary 0 0 0 0 0 0 0 0 0 0 0
sclerenchyma 1 1 1 1 1 1 1 1 1 1 1
Cambium 1 1 1 1 0 0 0 0 0 0 0
collenchyma 1 1 1 1 1 1 1 1 1 1 1
callose 1 1 1 1 1 1 1 1 1 1 0,9615
Parenchyma 1 1 1 1 1 1 1 1 0 0 0
Primordium 1 1 1 1 1 1 1 1 1 1 1
trunk 1 1 1 1 1 1 1 1 1 1 0
phellem 1 1 1 1 1 1 1 1 1 1 1
lignin 1 1 1 1 1 1 1 0,9677 0,9677 0 0
30
30
Lampiran 4 Precision Recall Metode Sweetspot Similarity ( Min =101 Max =150 Steepness = 0.5)
Kueri Titik Recall
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 0,9 1
Corpus 1 1 1 1 0,9091 0,9091 0,9091 0,9091 0 0 0
meristem 1 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0
Tunica 0 0 0 0 0 0 0 0 0 0 0
sclereids 1 1 1 1 1 1 1 1 1 1 1
Phloem 1 1 1 1 1 0,9231 0,9231 0 0 0 0
Xylem 1 1 1 1 1 1 1 1 0,9333 0,9333 0,8824
pericycle 1 1 1 1 1 1 1 1 1 1 1
trichomes 1 1 1 1 1 1 1 1 1 1 1
stele 1 1 1 1 1 1 1 1 1 1 1
Pedicel 1 1 1 1 1 1 1 1 1 1 1
tracheary 0 0 0 0 0 0 0 0 0 0 0
sclerenchyma 1 1 1 1 1 1 1 1 1 1 1
Cambium 1 1 1 1 0 0 0 0 0 0 0
collenchyma 1 1 1 1 1 1 1 1 1 1 1
callose 1 1 0,96 0,96 0,96 0,96 0,96 0,96 0,96 0,96 0,9259
Parenchyma 1 1 1 1 1 1 1 1 0 0 0
Primordium 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667
trunk 1 1 1 1 1 1 1 1 1 1 0
phellem 1 1 1 1 1 1 1 1 1 1 1
lignin 1 1 1 1 1 1 1 1 0,9677 0 0
31
31
Lampiran 5 Precision Recall Metode Sweetspot Similarity ( Min =151 Max =200 Steepness = 0.5)
Kueri Titik Recall
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 0,9 1
Corpus 1 1 1 1 0,9091 0,9091 0,9091 0,9091 0 0 0
meristem 1 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0
Tunica 0 0 0 0 0 0 0 0 0 0 0
sclereids 1 1 1 1 1 1 1 1 1 1 1
Phloem 1 1 1 1 0,9231 0,9231 0,9231 0 0 0 0
Xylem 1 1 1 1 1 1 0,9333 0,9333 0,9333 0,9333 0,8824
pericycle 1 1 1 1 1 1 1 1 1 1 1
trichomes 1 1 1 1 1 1 1 1 1 1 1
stele 1 1 1 1 1 1 1 1 1 1 1
Pedicel 1 1 1 1 1 1 1 1 1 1 1
tracheary 0 0 0 0 0 0 0 0 0 0 0
sclerenchyma 1 1 1 1 1 1 1 1 1 1 1
Cambium 1 1 1 1 0 0 0 0 0 0 0
collenchyma 1 1 1 1 1 1 1 1 1 1 1
callose 1 1 1 0,96 0,96 0,96 0,96 0,96 0,96 0,96 0,9259
Parenchyma 1 1 1 1 1 1 1 1 0 0 0
Primordium 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667
trunk 1 1 1 1 1 1 1 1 1 1 0
phellem 1 1 1 1 1 1 1 1 1 1 1
lignin 1 1 1 1 1 1 1 1 0,9677 0 0
32
32
Lampiran 6 Precision Recall Metode Sweetspot Similarity ( Min =201 Max =250 Steepness = 0.5)
Kueri Titik Recall
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 0,9 1
Corpus 1 1 1 1 0,9091 0,9091 0,9091 0,9091 0 0 0
meristem 1 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0
Tunica 0 0 0 0 0 0 0 0 0 0 0
sclereids 1 1 1 1 1 1 1 1 1 1 1
Phloem 1 1 1 1 0,9231 0,9231 0,9231 0 0 0 0
Xylem 1 1 1 1 1 1 0,9333 0,9333 0,9333 0,9333 0,8824
pericycle 1 1 1 1 1 1 1 1 1 1 1
trichomes 1 1 1 1 1 1 1 1 1 1 1
stele 1 1 1 1 1 1 1 1 1 1 1
Pedicel 1 1 1 1 1 1 1 1 1 1 1
tracheary 0 0 0 0 0 0 0 0 0 0 0
sclerenchyma 1 1 1 1 1 1 1 1 1 1 1
Cambium 1 1 1 1 0 0 0 0 0 0 0
collenchyma 1 1 1 1 1 1 1 1 1 1 1
callose 1 1 1 1 0,96 0,96 0,96 0,96 0,96 0,96 0,9259
Parenchyma 1 1 1 1 1 1 1 1 0 0 0
Primordium 1 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667 0,9667
trunk 1 1 1 1 1 1 1 1 1 1 0
phellem 1 1 1 1 1 1 1 1 1 1 1
lignin 1 1 1 1 1 1 1 1 0,9677 0 0
33
33
Lampiran 7 Precision Recall Metode Sweetspot Similarity ( Min =1 Max =100 Steepness = 0.5)
Kueri Titik Recall
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 0,9 1
Corpus 1 1 1 1 0,9091 0,9091 0,9091 0,9091 0 0 0
meristem 1 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0,9623 0
Tunica 0 0 0 0 0 0 0 0 0 0 0
sclereids 1 1 1 1 1 1 1 1 1 1 1
Phloem 1 1 1 1 0,9231 0,9231 0,9231 0 0 0 0
Xylem 1 1 1 0,9091 0,9091 0,9091 0,9091 0,8824 0,8824 0,8824 0,8824
pericycle 1 1 1 1 1 1 1 1 1 1 1
trichomes 1 1 1 1 1 1 1 1 1 1 1
stele 1 1 1 1 1 1 1 1 1 1 1
Pedicel 1 1 1 1 1 1 1 1 1 1 1
tracheary 0 0 0 0 0 0 0 0 0 0 0
sclerenchyma 1 1 1 1 1 1 1 1 1 1 1
Cambium 1 1 1 1 0 0 0 0 0 0 0
collenchyma 1 1 1 1 1 1 1 1 1 1 1
callose 1 1 1 1 1 1 1 0,9615 0,9615 0,9615 0,9615
Parenchyma 1 1 1 1 1 1 0,8333 0,8333 0 0 0
Primordium 1 1 1 1 1 1 1 1 1 1 0,9667
trunk 1 1 1 1 1 1 1 1 0,9722 0,9722 0
phellem 1 1 1 1 1 1 1 1 1 1 1
lignin 1 1 1 1 1 1 0,9677 0,9677 0,9677 0 0