DETEKSI IKAN TENGGIRI (
Scomberomorus
sp.) DAN PRODUK
OLAHANNYA MENGGUNAKAN DNA
BARCODING
DEDEN YUSMAN MAULID
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis yang berjudul “Deteksi ikan tenggiri (Scomberomorus sp.) dan produk olahannya menggunakan DNA
barcoding” adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Juni 2016
Deden Yusman Maulid
RINGKASAN
DEDEN YUSMAN MAULID. Deteksi Ikan Tenggiri (Scomberomorus sp.) dan Produk Olahannya Menggunakan DNA Barcoding. Dibimbing oleh NURJANAH, MALA NURILMALA, dan HAWIS MADDUPPA.
Ikan tenggiri merupakan salah satu jenis ikan yang sering digunakan sebagai bahan baku produk perikanan seperti pempek, bakso, otak-otak, kerupuk, dan lain-lain. Ketersediaan bahan baku yang terbatas serta keuntungan secara ekonomi sering dijadikan alasan terjadinya pemalsuan produk (mislabeling). Deteksi pemalsuan produk dapat dilakukan dengan penelusuran informasi secara morfologi, protein, dan DNA. Identifikasi secara morfologi dan analisis protein dapat dilakukan untuk ikan segar sedangkan untuk produk olahan perikanan tidak dapat dilakukan karena telah terjadi perubahan bentuk, ukuran, serta denaturasi protein selama proses produksi. DNA barcoding merupakan teknik identifikasi spesies yang dapat digunakan baik untuk sampel ikan segar maupun sampel produk olahan. Tujuan dari penelitian ini adalah menentukan keaslian produk olahan ikan tenggiri (Scomberomorus sp.) melalui pendekatan DNA barcoding
menggunakan dua gen mitokondria yaitu cytochrome oxidase sub unit I (COI) dan
cytochrome b (cyt b).
Penelitian ini dibagi menjadi dua tahap. Tahap pertama meliputi pengukuran fisik (morfometrik dan rendemen) serta pengukuran kimia (proksimat). Penelitian tahap dua yaitu tahap molekuler (DNA barcoding) dimulai dari isolasi DNA, desain primer, uji kuantitatif DNA, amplifikasi DNA, elektroforesis, dan konstruksi pohon filogenetik. Sampel terdiri dari ikan segar (6 sampel), tenggiri giling (3), pempek (4), bakso (6), otak-otak (5), dan kerupuk (6). Sampel dikoleksi dari beberapa tempat yaitu Jakarta, Bogor, Banten, Bandung, Makasar, dan Palembang.
Ciri morfometrik menunjukkan bahwa sampel ikan segar (6 sampel) terbagi menjadi dua kelompok spesies yaitu tenggiri papan (Scomberomorus commerson) dan tenggiri totol (Scomberomorus guttatus). Ciri khas ikan tenggiri totol adalah terdapat corak bulat yang tersebar tidak teratur di atas linea lateralis
(LL) sedangkan tenggiri papan memiliki corak garis vertikal. Ikan tenggiri papan dan tenggiri totol memiliki bagian daging putih yang lebih besar dibandingkan daging merah yaitu 46,67% dan 48,38%. Hasil SDS-PAGE menunjukkan bahwa pada ikan segar dan sampel ikan giling terdapat pita protein yang jelas, sedangkan pada sampel produk olahan pita proteinnya tidak tergambarkan secara jelas.
Hasil analisis DNA barcoding menunjukkan terdapat 9 dari 30 (30%) sampel tidak mengandung ikan tenggiri seperti yang tercantum pada labelnya yaitu pempek (2 sampel), bakso (2), dan kerupuk (5). Nilai homologi spesies yang teridentifikasi sebagai tenggiri papan (S. commerson) dan tenggiri totol (S. guttatus) berkisar antara 97%-99%.
SUMMARY
DEDEN YUSMAN MAULID. Detection of Mackerel Fish (Scomberomorus sp.) and its Processed Products Using DNA Barcoding. Supervised by NURJANAH, MALA NURILMALA, and HAWIS MADDUPPA
Mackerel is often used as a raw material of fish products such as pempek, meatball, otak-otak, and crackers. Both limited availability of raw materials and economic benefits mostly become the reason for the counterfeiting of the products (mislabeling). Adulteration of raw material could be detected by any identification methods such as morphological identification, protein analysis, and DNA analysis. Both morphological identification and proteins analysis could be performed for whole fish, but not for processed fish products due to shape and size changes and protein denaturation during production process. DNA barcoding is species identification technique which can be used either for whole fish samples or processed products. The aim of this study was to authenticate mackerel products (Scomberomorus sp.) by DNA barcoding method using two mitochondrial genes: cytochrome b (cyt b) and cytochrome c subunit I (COI).
This study was divided into two stages. First, physical (morphometric and yield) and chemical (proximate) measurement. The second was molecular stage (DNA barcoding): DNA isolation, primer design, purification and concentration of DNA, DNA amplification, electrophoresis, and phylogenetic tree construction. Sample consists of whole fish (6 samples), minced fish (3), pempek (4), meat ball (6), otak-otak (5), and snack (6). Samples was collected from Jakarta, Bogor, Bandung, Banten, Palembang, and Makassar.
Morphometric characteristics showed that whole fish samples (6 samples) were identified into two groups of species, king mackerel (Scomberomorus commerson) and korean mackerel (Scomberomorus guttatus). Main characteristic of king mackerel is irregular pattern over the linea lateralis while king mackerel has a pattern of vertical lines. King mackerel and korean mackerel have 46.67% and 48.38% greater white meat than the red meat. SDS-PAGE results showed that whole and milled fish have a clear protein bands while processed products were not clearly ilustrated.
DNA barcoding analysis indicated that 9 of 30 (30%) samples did not contain mackerel as listed on the label. They are pempek (2 samples), meatball (2), and snack (5). Homology of identified species (king and korean mackerel)
ranged between 97% -99%.
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Teknologi Hasil Perairan
DETEKSI IKAN TENGGIRI (
Scomberomorus
sp.) DAN PRODUK
OLAHANNYA MENGGUNAKAN DNA
BARCODING
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2016
Deteksi Ikan Tenggiri (Scomberomorus sp.) dan Produk Olahannya Menggunakan DNA Barcoding
Deden Yusman Maulid C351130011
Disetujui oleh Komisi Pembimbing
Prof Dr Ir Nurjanah, MS Ketua
Dr Mala Nurilmala, SPi, MSi Anggota
Dr Hawis Madduppa, SPi, MSi Anggota
Diketahui oleh
Ketua Program Studi Teknologi Hasil Perairan
Dr Ir Wini Trilaksani, MSc
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji syukur kepada Allah SWT, karena atas rahmat dan hidayah-Nya Penulis dapat menyelesaikan penelitian yang berjudul “Deteksi Ikan Tenggiri (Scomberomorus sp.) dan Produk Olahannya Menggunakan DNA Barcoding”.
Penelitian ini didanai oleh Kementrian Riset, Teknologi, dan Pendidikkan Tinggi dalam program hibah kompetensi atas nama Dr Mala Nurilmala, SPi, MSi.
Penulis mengucapkan terimakasih yang sebesar-besarnya kepada:
1. Prof Dr Ir Nurjanah, MS, Dr Mala Nurilmala, SPi, MSi, Dr Hawis Madduppa, SPi, MSi selaku dosen pembimbing yang telah memberikan bimbingan, arahan, dukungan, semangat kepada penulis sehingga penulis dapat menyelesaikan tesis ini.
2. Prof Dr Ir Linawati Hardjito, MS selaku dosen penguji luar komisi yang telah memberikan bimbingan dan arahan sehingga penulis mampu menyelesaikan tesis ini.
3. Ketua program studi, Dr Ir Wini Trilaksani, MSc dan perwakilan tim gugus kendali mutu, Dr Asadatun Abdullah, SPi, MSi atas masukan kepada penulis. 4. Orang tua beserta istri yang telah memberikan dukungan baik materil maupun
spiritual kepada penulis.
5. Teman-teman satu tim penelitian DNA barcoding yang saya banggakan. Laboran yang telah membantu penelitian saya (Ibu Ema, Mas Ipul, Mbak Dini, Faqih, Mbak Nuring, Lita, Yustin, Fajrin, dan Andhra)
6. Keluarga besar mahasiswa sekolah pascasarjana Teknologi Hasil Perairan, yang telah memberikan dorongan semangat baik selama penelitian maupun saat penyusunan tesis ini.
7. Direktorat Jendral Pendidikkan Tinggi (Dikti) yang telah membiayai penulis selama masa perkuliahan melalui Beasiswa Pendidikan Perguruan Tinggi Dalam Negeri (BPPDN).
8. Kementrian Riset Teknologi dan Pendidikkan Tinggi yang telah mendanai penelitian ini pada program hibah kompetensi atas nama Dr Mala Nurilmala SPi, MSi.
9. Terima kasih penulis sampaikan kepada semua pihak yang telah membantu penyelesaian tesis ini.
Penulis menyadari bahwa masih terdapat kekurangan dalam penyusunan tesis ini. Penulis sangat mengharapkan kritik dan saran yang membangun sehingga bermanfaat untuk pembaca dan masyarakat secara umum. Semoga karya ilmiah ini bermanfaat.
Bogor, Juni 2016
DAFTAR ISI
DAFTAR TABEL vii
DAFTAR GAMBAR vii
DAFTAR LAMPIRAN vii
1 PENDAHULUAN
Latar Belakang 1
Rumusan Masalah 2
Tujuan Penelitian 3
Manfaat Penelitian 3
2 METODE
Waktu dan Tempat 3
Bahan dan Alat 3
Prosedur Kerja 4
Morfometrik 5
Rendemen 5
Analisis Proksimat (AOAC 2005) 5
SDS-PAGE (Sodium dedosil sulphate-polyacrylamid gel elechtrophoresis) 6
Analisis Molekuler 7
Isolasi DNA 7
Konsentrasi dan Kemurnian DNA 7
Desain Primer 7
Amplifikasi DNA dan Elektroforesis 8
Identifikasi Spesies dan Sekuensing 8
Pohon Filogenetik (Tamura et al. 2011) 8
3 HASIL DAN PEMBAHASAN
Morfometrik 9
Rendemen 10
Komposisi Kimia 10
SDS-PAGE 11
Isolasi DNA 14
Desain Primer 12
Amplifikasi DNA dan Elektroforesis 14
Identifikasi Spesies dan Sekuensing 17
Pohon Filogenetik 18
4 SIMPULAN DAN SARAN
Simpulan 20
Saran 20
DAFTAR PUSTAKA 21
DAFTAR TABEL
1Proses PCR (Polymerase Chain Reaction) ... 8
2Panjang dan berat ikan tenggiri segar ... 9
3Rendemen ikan tenggiri ... 10
4Kandungan kimia ikan tenggiri segar dan produk olahannya (%) ... 10
5Konsentrasi dan kemurnian DNA sampel ... 13
6Hasil identifikasi spesies berdasarkan analisis BLAST (COI dan Cyt b) ... 17
DAFTAR GAMBAR
1Alur penelitian deteksi ikan tenggiri dan olahannya ... 42Hasil SDS PAGE ikan tenggiri dan olahannya. ... 11
3Hasil elektroforesis (COI) ikan tenggiri segar dan produk olahannya. ... 15
4Hasil elektroforesis (cytb) ikan tenggiri segar dan produk olahannya. ... 16
5Pohon filogenetik sampel dengan primer COI ... 18
6Pohon filogenetik dengan menggunakan primer cyt b ... 19
DAFTAR LAMPIRAN
1Sampel dan lokasi pengambilan ... 242Alat dan bahan isolasi DNA, amplifikasi DNA, dan elektroforesis ... 25
3Tabel protein berdasarkan berat molekul sebagai marka SDS-PAGE ... 25
4Hasil elektroforegram sampel menggunakan primer spesifik COI dan Cytochrome b ... 26
5Nomor accession sample dari genbank untuk marka cytochrome b ... 27
1
1
PENDAHULUAN
Latar Belakang
Ikan tenggiri merupakan salah satu jenis ikan keluarga scombridae yang terdiri dari 15 genus dan 51 spesies (Orrell et al. 2006). Ikan tenggiri tersebar dari tepi samudera sampai perairan lepas pantai yang dangkal. Habitat yang disukai oleh ikan tenggiri memiliki ciri fisik di antaranya salinitas yang rendah dengan kekeruhan tinggi (Mcpherson 1985). Ikan tenggiri merupakan salah satu komoditas penting bagi Indonesia. Indonesia menjadi negara dengan hasil tangkapan ikan tenggiri terbesar di dunia. Hasil penangkapan global ikan tenggiri pada tahun 2013 mengalami peningkatan sebesar 2,8% dari tahun sebelumnya yaitu sebesar 936.004 ton (FAO 2013).
Ikan tenggiri telah dimanfaatkan dalam berbagai produk olahan seperti pempek, bakso, otak-otak, dan kerupuk. Produk olahan ikan dalam kemasan memiliki daya tarik tersendiri bagi konsumen. Berbagai keuntungan yang diperoleh adalah awet dan instan. Semakin bervariasinya produk perikanan memunculkan kemungkinan adanya pemalsuan produk oleh produsen dalam rangka meningkatkan keuntungan (Jacquet dan Pauly 2008). Bentuk pemalsuan dapat berupa penggunaan ikan substitusi, yaitu jenis ikan yang berbeda dari jenis ikan yang ada pada label atau mencampurnya dengan ikan jenis lain (Mackie et al. 1999). Substitusi merupakan cara pemalsuan paling mudah dilakukan pada ikan olahan karena sulitnya mengidentifikasi produk, sehingga peluang terjadinya
mislabeling menjadi meningkat (Haye et al. 2012).
Pemalsuan produk perikanan menyebabkan kerugian karena biaya yang dikeluarkan lebih tinggi untuk produk yang telah dicampur atau diganti dengan bahan baku yang lebih murah. Pemalsuan produk perikanan juga dapat menyebabkan gangguan kesehatan (Leeuwen et al. 2009), seperti alergi pada jenis ikan tertentu, atau keracunan karena penggunaan ikan beracun seperti ikan buntal (Huang et al. 2014). Adanya pemalsuan ini juga mengganggu hak konsumen
dalam memperoleh “enforcement of labelling regulation” (Mackie et al. 1999). Pemalsuan ini harus mendapat perhatian khusus dalam rangka penjaminan keamanan pangan dan perlindungan hak konsumen. EU Council Regulation No. 1379/2013 sudah menetapkan aturan tentang pelabelan menggunakan nama komersial dan nama ilmiah untuk produk perikanan di Eropa (Eur-lex 2013). Di Indonesia pelabelan produk pangan sudah diatur dalam peraturan pemerintah (PP) no 69 tahun 1999, selain itu hak-hak konsumen dilindungi oleh undang-undang nomor 8 tahun 1999. Pemalsuan produk perikanan masih saja terjadi seperti pemalsuan pada filet tuna dan filet dori (Dwiyitno 2010). Upaya yang dapat dilakukan adalah dengan menerapkan peraturan dan undang-undang secara taat azas. Dalam penerapan peraturan tersebut diperlukan pengujian keaslian produk, termasuk produk ikan dan seafood, atau dikenal dengan istilah autentikasi.
2
2013). Identifikasi dengan pendekatan protein yang pernah berkembang juga belum mampu mengidentifikasi produk olahan dengan baik. Upaya yang dapat digunakan untuk autentikasi produk ini adalah dengan pendekatan DNA, yakni dengan perbanyakan untai DNA menggunakan mesin PCR. DNA masih dapat diidentifikasi, bahkan dalam jumlah sampel yang sedikit, rusak dan tidak utuh (Tsai et al. 2007).
Beberapa metode PCR telah dikembangkan di antaranya adalah single strand conformation polymorphism (SSCP), restriction fragment length polymorphism (RFLP), real time PCR, multiplex PCR, microsatellite, dan DNA
barcoding. SSCP digunakan untuk melihat laju mutasi yang terjadi pada suatu spesies, RFLP digunakan untuk melihat keragaman genetik antar individu dalam satu spesies, real time PCR digunakan untuk mendeteksi keberadaaan suatu species sekaligus dapat menghitung secara kuantitatif produk PCR yang dihasilkan. Multiplex PCR digunakan untuk mengetahui keberadaan suatu spesies dengan sekuens lebih dari satu, microsatellite digunakan untuk mendeteksi kekerabatan individu dalam satu species yang sama, sedangkan DNA barcoding
digunakan untuk mendeteksi suatu spesies yang belum diketahui menggunakan marka molekuler dari DNA mitokondria. Analisis spesies menggunakan DNA mitokondria memiliki kelebihan di antaranya jumlahnya lebih banyak, ukurannya lebih kecil, sekuens dari beberapa organisme akuatik dengan pendekatan mitokondria tersedia lengkap, rentang intron (non-coding) tidak ada (Mackie et al.
1999), dan mitokondria DNA lebih sensitif terhadap mutasi. DNA barcoding
merupakan metode yang paling banyak diaplikasikan dalam forensik taksonomi (Dawnay et al. 2007) karena efektif dalam mengidentifikasi berbagai kondisi sampel uji, baik sampel utuh maupun sudah rusak, dan tidak menghasilkan data yang ambigu (Wong 2011).
Pendekatan yang digunakan untuk analisis DNA adalah mengamplifikasi segmen DNA dari cytochrome b dan menentukan sekuensnya (Mackie et al. 1999) atau amplifikasi cytochrome c oxydase I (COI). Roe dan Sperling (2007) menyatakan COI memiliki tingkat keragaman yang tinggi sehingga dapat mengidentifikasi pada tingkat spesies. Hold et al. (2001) menyatakan bahwa produk perikanan yang mengalami proses pembekuan, pemanasan, penambahan garam, dan pengolahan lainnya dapat menggunakan cytochrome b (cyt b). Espiñeira et al. (2009) menggunakan cyt b untuk mengetahui pemalsuan produk yang berbahan baku dari ikan Scombridae.
Rumusan Masalah
Bahan baku yang terbatas dan mendapatkan keuntungan yang sebesar-besarnya sering dijadikan alasan oleh pihak yang tidak bertanggung jawab untuk memalsukan produk perikanan. Identifikasi bahan baku pada produk olahan perikanan sulit dilakukan secara morfologi karena sudah terjadi perubahan bentuk dan ukuran selama proses pengolahan menyebabkan perlunya cara alternatif untuk mengetahui spesies yang digunakan sebagai bahan baku. DNA barcoding
3
Tujuan Penelitian
Tujuan dari penelitian ini adalah menentukan keaslian produk olahan ikan tenggiri (Scomberomorus sp.) melalui pendekatan DNA barcoding menggunakan gen target pada mitokondria yaitu cytochrome oxidase sub unit I (COI) dan
cytochrome b (cyt b).
Manfaat Penelitian
Manfaat penelitian ini adalah teknik DNA barcoding dapat diaplikasikan sebagai salah satu metode identifikasi spesies untuk ikan tenggiri dan olahannya sehingga dapat mencegah terjadinya pemalsuan produk perikanan secara cepat dan akurat, serta meningkatkan keamanan pangan dan kenyamanan konsumen.
2
METODE
Waktu dan Tempat
Penelitian dilaksanakan pada bulan Desember 2014 sampai dengan Agustus 2015 di Laboratorium Karakteristik Bahan Baku, Departemen Teknologi Hasil Perairan, Laboratorium Biodiversitas dan Biosistematika Departemen Teknologi Ilmu Kelautan Fakultas Perikanan dan Ilmu Kelautan IPB.
Bahan dan Alat
Alat yang digunakan dalam penelitian ini adalah freezer, pipet tips (Axygen Scientific, California-USA), mikro pipet (Thermo scientific, vantaa-finland), tabung mikro (Qiagen, Venlow-Netherland), sentrifuse perfectspin 24 plus (peqlab biotechnologie GMvH, Erlanger-Jerman), mesin PCR Termocycler Biometra T1 (Biometra GMbH, Gṏttingen-Jerman), Inkubator Digital Block Heater HX-1 (Peqlab Biotechnologie GmbH, Erlangen-Jerman), elektroforesis Mupid-Exu, (Advance, Tokyo-Japan), timbangan digital Adam vw 254 (Adam equipment co. Milton Keynes-England), spindown perfectspin mini (Peqlab Biotechnologie GmbH, Erlangen-Jerman), vortex peqTwist vortex mixer (Peqlab Biotechnologie GmbH, Erlangen-Jerman), microwave Panasonic NN-SM320M, alat sinar Ultraviolet Viewer (UV-1) (Extragene Inc, Taichung City-Taiwan), nanodrop nanofotometer p360 (implant GmbH eschtzbogeen-Jerman), electroforesis vertical TU100YK (Scie-plus Ltd, Cambridge-England).
Bahan yang digunakan dalam penelitian ini adalah ikan tenggiri segar (tenggiri papan atau Scomberomorus commerson 3 sampel dan tenggiri totol atau
4
Vaenlow-Netherland), DNA dneasy mericoon food kit (Qiagen Vaenlow-Netherland), larutan mix Kapa Taq Extra Hotstart ReadyMix (kappa biosystem,Massachusetts-USA), etanol 96%, kloroform, agarose (Vivantis Invitrogen, Massachussetts-USA), Etidium Bromida, gel loading bufer (Invitrogen, Massachussetts-USA ), dan DNA ladder (Invitrogen, Massachussetts-US).
Prosedur Kerja
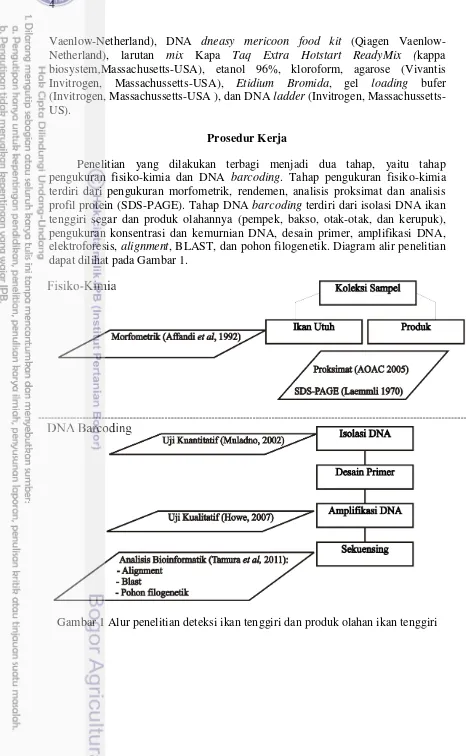
Penelitian yang dilakukan terbagi menjadi dua tahap, yaitu tahap pengukuran fisiko-kimia dan DNA barcoding. Tahap pengukuran fisiko-kimia terdiri dari pengukuran morfometrik, rendemen, analisis proksimat dan analisis profil protein (SDS-PAGE). Tahap DNA barcoding terdiri dari isolasi DNA ikan tenggiri segar dan produk olahannya (pempek, bakso, otak-otak, dan kerupuk), pengukuran konsentrasi dan kemurnian DNA, desain primer, amplifikasi DNA, elektroforesis, alignment, BLAST, dan pohon filogenetik. Diagram alir penelitian dapat dilihat pada Gambar 1.
5
Morfometrik
Morfometrik adalah pengukuran bagian tubuh ikan seperti panjang total dan panjang baku. Pengukuran panjang total ikan segar dilakukan dari ujung kepala sampai ujung ekor dan pengukuran panjang baku dilakukan dari ujung kepala sampai pangkal ekor. Pengamatan ciri fisik ikan tenggiri mengacu pada Collette et al. (1983).
Rendemen
Pengukuran rendemen dilakukan dengan mengukur berat tiap bagian tubuh yang terdiri dari kulit, tulang, daging putih, jeroan, daging merah, dan kepala kemudian dibandingkan dengan bobot awal (utuh) (Hoar dan Randall, 1970). Rendemen dihitung berdasarkan rumus berikut:
Rendemen (%) = Bobot bagian tubuh ikan tenggiri
Bobot total x 100%
Analisis Proksimat (AOAC 2005)
Analisis proksimat terdiri dari penentuan kadar air, lemak, abu, protein dan karbohidrat. Analisis proksimat mengacu pada AOAC (2005). Perhitungan karbohidrat dilakukan secara by difference.
Kadar air
Kadar air ditentukan berdasarkan perbedaan berat contoh sebelum dan sesudah dikeringkan. Mula-mula cawan kosong dikeringkan dalam oven selama 30 menit dengan suhu 105oC kemudian disimpan dalam desikator selama 15 menit, kemudian ditimbang. Sampel sebanyak 3-5 g dimasukkan ke dalam cawan kemudian dikeringkan dalam oven 105oC selama 6 jam. Cawan disimpan dalam
desikator selama 30 menit, kemudian ditimbang. Kadar air ditentukan dengan rumus :
Kadar air (%) = B-C
B-A
x 100%
Ket: A= berat cawan kosong (gram); B= berat cawan dengan sampel sebelum dikeringkan (gram); C= berat cawan dengan sampel setelah dikeringkan (gram)
Kadar lemak
6
Kadar lemak (%) = B-A
S x 100%
Ket: S= berat contoh sampel (gram); A= berat labu lemak tanpa lemak (gram); B= berat labu lemak dengan lemak (gram)
Kadar abu
Sampel ditimbang 3-5 g kemudian dimasukkan ke dalam cawan, dibakar di atas bunsen sampai tidak berasap. Sampel dimasukkan ke dalam tanur pengabuan, dibakar hingga diperoleh abu berwarna abu-abu. Pengabuan dilakukan dalam dua tahap, yaitu pengabuan pertama pada suhu 400oC kemudian pengabuan kedua menggunakkan suhu 550oC. Cawan disimpan dalam desikator setelah beratnya tetap (konstan) cawan kemudian ditimbang. Kadar abu ditentukan dengan rumus:
Kadar abu (%) = A
B
x 100%
Ket : A = berat abu (gram), B = berat contoh (gram) Kadar protein
Bahan ditimbang 0,5-1 g. Bahan dimasukkan ke dalam labu kjeldahl, kemudian ditambahkan 0,5 g selenium dan 7 mL H2SO4 pekat. Sampel kemudian
didestruksi sampai larutan berwarna jernih kehijauan dan uap SO2 hilang. Hasil
destruksi ditambah akuades dan dimasukkan ke dalam labu destilasi. NaOH 33% ditambahkan ke dalam labu destilasi dan kemudian dilakukan destilasi, destilat ditampung dalam 20 mL larutan asam borat 3% kemudian dititrasi dengan HCl standar (indikator metil merah).
Kadar karbohidrat (by difference)
Kadar karbohidrat dihitung dengan menghitung sisa (by difference) dari selisih berat utuh (100%) yaitu dengan rumus sebagai berikut:
Kadar karbohidrat (%)= 100%-(%air+%abu+%lemak+%protein)
(Sodium dedosyl sulphate-polyacrylamid gel elechtrophoresis) SDS-PAGE
Sodium dedosyl sulphate-polyacrylamid gel elechtrophoresis (SDS-PAGE) bertujuan untuk memisahkan protein dan melihat pola protein pada suatu sampel yang diuji. Pengujian SDS-PAGE yang dilakukan berdasarkan metode Laemmli (1970). Elektroforesis dijalankan secara konstan pada arus 15 mA dan voltase 150 volt selama 3 jam. Bahan SDS-PAGE dalam penelitian ini menggunakan 3%
7
Isolasi DNA
Isolasi DNA dilakukan menggunakan Dneasy blood and tissue kit yang didapatkan dari PT Genecraft Labs. Sampel ikan segar dipotong kecil kemudian ditimbang 10-25 mg selanjutnya disimpan dalam tabung mikrosentrifuse 1,5 mL, kemudian ditambahkan 180 µL bufer lisis yang mengandung detergen sodium dedosyl sulphate (SDS) dan 2 µL proteinase k, divortex selama 15 detik, inkubasi dilakukan pada suhu 56°C selama 1-2 jam sampai lisis. Selama inkubasi divortex setiap 15 menit. Sebanyak 200 µL larutan bufer yang mengandung guanidium chloride dan lisin ditambahkan untuk merusak membran sel, divortex beberapa detik. Larutan tersebut dimasukkan ke dalam spin column 2 mL, disentrifugasi selama 1 menit pada kecepatan 8000 rpm. Cairan yang terdapat di bawah dibuang, kemudian saringan dipindahkan ke dalam spin column yang baru. Sebanyak 500 µL larutan bufer yang mengandung guanidium chloride yang lebih pekat dibandingkan bufer sebelumnya ditambahkan dan disentrifugasi 8000 rpm, selama satu menit. Cairan pada lapisan bawah dibuang dan dimasukkan tube pada spin column yang baru. Sebanyak 500 µL larutan bufer yang mengandung sodium azide dimasukkan dan disentrifugasi pada 14000 rpm, selama 1 menit. Column
diambil, kemudian spin column dipindahkan pada tabung sentrifugasi yang baru 1,5 mL. Elusi DNA dilakukan dengan menambahkan 200 µL bufer yang mengandung (tris-EDTA) pada bagian tengah membran.
Konsentrasi dan Kemurnian DNA
Alat yang digunakan dalam pengukuran konsentrasi dan kemurnian DNA yaitu spektrofotometer UV-Vis. Sampel diambil sebanyak 1 µL, kemudian diteteskan pada piringan kecil yang terletak di dalam nanodrop.yang telah dibersihkan dengan akuades. Program nanodrop diatur untuk sampel kurang dari 20 µL. Proses pengukuran dimulai dengan cara menekan tombol running pada display nanodrop. Hasil pengukuran ditampilkan pada layar monitor nanodrop.
Desain Primer
8
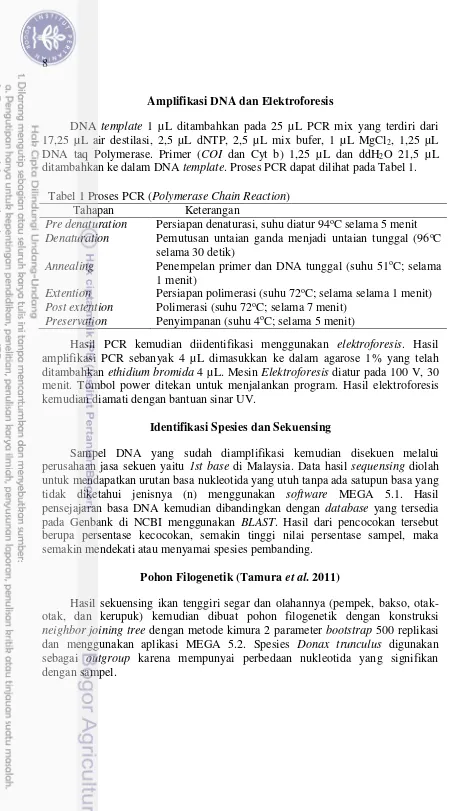
Amplifikasi DNA dan Elektroforesis
DNA template 1 µL ditambahkan pada 25 µL PCR mix yang terdiri dari 17,25 µL air destilasi, 2,5 µL dNTP, 2,5 µL mix bufer, 1 µL MgCl2, 1,25 µL
DNA taq Polymerase. Primer (COI dan Cyt b) 1,25 µL dan ddH2O 21,5 µL
ditambahkan ke dalam DNA template. Proses PCR dapat dilihat pada Tabel 1.
Hasil PCR kemudian diidentifikasi menggunakan elektroforesis. Hasil amplifikasi PCR sebanyak 4 µL dimasukkan ke dalam agarose 1% yang telah ditambahkan ethidium bromida 4 µL. Mesin Elektroforesis diatur pada 100 V, 30 menit. Tombol power ditekan untuk menjalankan program. Hasil elektroforesis kemudian diamati dengan bantuan sinar UV.
Identifikasi Spesies dan Sekuensing
Sampel DNA yang sudah diamplifikasi kemudian disekuen melalui perusahaan jasa sekuen yaitu 1st base di Malaysia. Data hasil sequensing diolah untuk mendapatkan urutan basa nukleotida yang utuh tanpa ada satupun basa yang tidak diketahui jenisnya (n) menggunakan software MEGA 5.1. Hasil pensejajaran basa DNA kemudian dibandingkan dengan database yang tersedia pada Genbank di NCBI menggunakan BLAST. Hasil dari pencocokan tersebut berupa persentase kecocokan, semakin tinggi nilai persentase sampel, maka semakin mendekati atau menyamai spesies pembanding.
Pohon Filogenetik (Tamura et al. 2011)
Hasil sekuensing ikan tenggiri segar dan olahannya (pempek, bakso, otak-otak, dan kerupuk) kemudian dibuat pohon filogenetik dengan konstruksi
neighbor joining tree dengan metode kimura 2 parameter bootstrap 500 replikasi dan menggunakan aplikasi MEGA 5.2. Spesies Donax trunculus digunakan sebagai outgroup karena mempunyai perbedaan nukleotida yang signifikan dengan sampel.
Tabel 1 Proses PCR (Polymerase Chain Reaction)
Tahapan Keterangan
Pre denaturation Persiapan denaturasi, suhu diatur 94oC selama 5 menit
Denaturation Pemutusan untaian ganda menjadi untaian tunggal (96oC selama 30 detik)
Annealing Penempelan primer dan DNA tunggal (suhu 51oC; selama 1 menit)
Extention Persiapan polimerasi (suhu 72oC; selama selama 1 menit)
Post extention Polimerasi (suhu 72oC; selama 7 menit)
9
3
HASIL DAN PEMBAHASAN
Sampel yang dikumpulkan berjumlah 30 sampel yang terdiri dari: 3 tenggiri papan segar, 3 tenggiri totol segar, 3 tenggiri giling, 4 jenis pempek, 6 jenis bakso, 5 jenis otak-otak, dan 6 jenis kerupuk. Sampel diperoleh dari berbagai kota yaitu Jakarta, Bogor, Bandung, dan Makasar. Sampel ikan segar yaitu ikan tenggiri papan dan ikan tenggiri totol seperti terlihat pada Lampiran 1.
Morfometrik
Ukuran ikan diamati berdasarkan panjang dan beratnya. Panjang ikan yang diukur terdiri dari panjang total dan panjang baku. Panjang total dihitung dari bagian paling depan kepala sampai bagian paling belakang ekor. Panjang baku dihitung dari bagian paling ujung kepala sampai pangkal ekor. Ciri fisik yang diamati terdiri dari kepala, corak tubuh, dan jumlah sirip. Panjang total ikan tenggiri papan 41±1 cm dan ikan tenggiri totol 36,83±0,29 cm. Pengukuran panjang dan berat ikan tenggiri secara lengkap ditampilkan pada Tabel 2.
Ikan tenggiri papan yang dikoleksi memiliki panjang dan berat yang lebih besar dibandingkan ikan tenggiri totol. Affandi et al. (1992) menyatakan bahwa pertumbuhan ikan dipengaruhi faktor internal maupun faktor eksternal. Faktor internal meliputi genetik, jenis kelamin, usia, dan hormon. Faktor eksternal ketersediaan makanan dan kondisi fisik lingkungan perairan. Ikan tenggiri papan dan tenggiri totol memiliki kemiripan bentuk dan warna rahang yaitu bentuk rahang memanjang dan lancip pada arah mulut dengan warna putih keperakan. Ciri fisik khas tenggiri papan adalah yaitu pada tubuhnya memiliki pola garis vertikal dari sisi dorsal sampai anal hampir sepanjang tubuhnya dan berubah menjadi pola bintik hitam pada bagian ekor (kaudal), sirip punggung terdiri dari dua bagian (depan dan belakang). Ikan tenggiri totol memiliki pola bintik-bintik kehitaman sepanjang linea lateralis tersebar acak, sirip punggung terdiri dari dua bagian (depan dan belakang).
Collette et al. (1983) menyatakan bahwa ciri spesies Scomberomorus commerson di antaranya adalah sirip dorsal terdiri dari dua bagian (depan dan belakang) terdiri dari sekitar 14-16 buah. Corak tubuh berupa garis vertikal yang membungkuk pada bagian punggung dengan jumlah sekitar 50 buah pada ikan dewasa dan 20 buah pada ikan kecil biasanya terputus pada bagian perut. Spesies
Scomberomorus guttatus memiliki ciri sirip dorsal terdiri dari dua bagian, corak tubuh membentuk bintik hitam yang tersebar acak sepanjang linea lateralis bagian atas, sisik berwarna putih keperakan. Berdasarkan ciri fisik tersebut, ikan tenggiri papan memiliki kemiripan dengan spesies Scomberomorus commerson dan ikan tenggiri totol memiliki kemiripan dengan spesies Scomberomorus guttatus.
Tabel 2 Panjang dan berat ikan tenggiri segar
Sampel Panjang (cm) Berat (gram)
Total Baku
10
Tabel 4 Kandungan kimia ikan tenggiri segar dan produk olahannya (%)
Sampel Kadar Air Abu Lemak Protein Karbohidrat Tenggiri papan 77,27±1,41 1,42±0,25 0,54±0,18 18,56±0,32 2,21±1,43 Tenggiri totol 76,58±0,59 1,73±0,32 0,49±0,06 19,91±0,17 1,29±0,72 Pempek 52,44±0,50 1,91±0,01 0,55±0,01 1,96±0,09 43,41±0,6 Bakso 19,79±0,13 4,52±0,11 10,56±0,15 9,00±0,06 56,13±0,23 Kerupuk 4,77±0,02 2,89±0,13 2,54±0,18 2,58±0,18 87,22±0,05 Mackerel* 75,67±0,12 1,42±0,01 0,89±0,01 9,47±0,16 0,70±0,01
*)Sutarshiny and Sivhashantini 2011
Rendemen
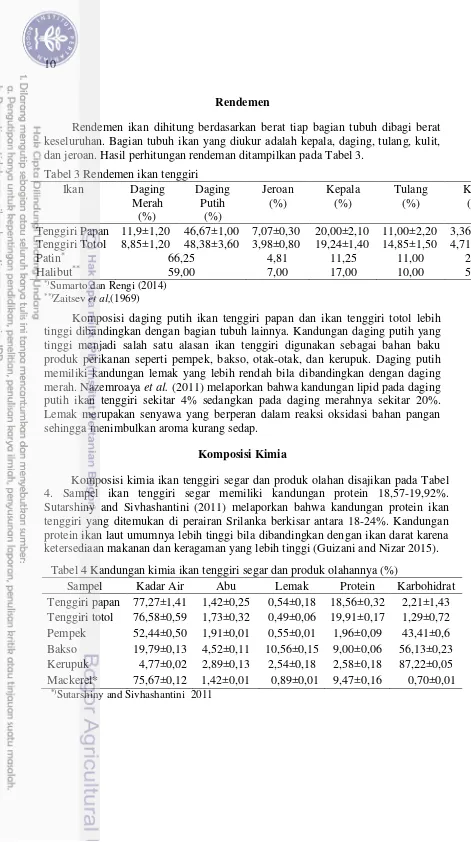
Rendemen ikan dihitung berdasarkan berat tiap bagian tubuh dibagi berat keseluruhan. Bagian tubuh ikan yang diukur adalah kepala, daging, tulang, kulit, dan jeroan. Hasil perhitungan rendeman ditampilkan pada Tabel 3.
Tabel 3 Rendemen ikan tenggiri
Ikan Daging
Merah (%) Daging Putih (%) Jeroan (%) Kepala (%) Tulang (%) Kulit (%) Tenggiri Papan 11,9±1,20 46,67±1,00 7,07±0,30 20,00±2,10 11,00±2,20 3,36±0,10 Tenggiri Totol 8,85±1,20 48,38±3,60 3,98±0,80 19,24±1,40 14,85±1,50 4,71±0,40
Patin* 66,25 4,81 11,25 11,00 2,62
Halibut** 59,00 7,00 17,00 10,00 5,00
*)Sumarto dan Rengi (2014) **)Zaitsev et al,(1969)
Komposisi daging putih ikan tenggiri papan dan ikan tenggiri totol lebih tinggi dibandingkan dengan bagian tubuh lainnya. Kandungan daging putih yang tinggi menjadi salah satu alasan ikan tenggiri digunakan sebagai bahan baku produk perikanan seperti pempek, bakso, otak-otak, dan kerupuk. Daging putih memiliki kandungan lemak yang lebih rendah bila dibandingkan dengan daging merah. Nazemroaya et al. (2011) melaporkan bahwa kandungan lipid pada daging putih ikan tenggiri sekitar 4% sedangkan pada daging merahnya sekitar 20%. Lemak merupakan senyawa yang berperan dalam reaksi oksidasi bahan pangan sehingga menimbulkan aroma kurang sedap.
Komposisi Kimia
11
Kadar air dan kandungan protein ikan segar lebih tinggi bila dibandingkan dengan produk olahan, sedangkan kadar abu, lemak, dan karbohidrat lebih rendah dari produk olahan. Pengurangan kadar air dan kandungan protein pada produk olahan disebabkan oleh pemanasan dan penambahan bahan lain selama proses produksi. Peningkatan kandungan abu, lemak dan karbohidrat disebabkan oleh penambahan garam-garam mineral, minyak, dan bahan lain yang mengandung karbohidrat tinggi seperti tepung beras dan tepung singkong (Shaltout 1993; Chang and Chen. 2013; Sengun et al. 2014; Turp et al. 2016)
SDS-PAGE
SDS-PAGE digunakan untuk melihat dugaan awal kandungan protein dan pola penyebaran pita protein suatu sampel. Sampel yang diuji terdiri dari dua jenis yaitu ikan segar dan produk olahannya (pempek, bakso, otak-otak, dan kerupuk). Pita profil protein dibandingkan dengan marka. Hasil SDS-PAGE diperlihatkan pada Gambar 2.
Hasil SDS-PAGE pada sampel ikan segar dan giling menunjukkan pita protein yang jelas sedangkan sampel produk (no 5-9) tidak menggambarkan pola pita protein yang jelas, hal ini disebabkan karena pada sampel ikan segar dan giling rata-rata belum mengalami denaturasi protein. Produk olahan telah mengalami denaturasi selama proses pengolahan.
Denaturasi protein adalah perubahan struktur protein baik struktur sekunder, tersier maupun kuartener karena faktor peubah, baik fisik, maupun kimia. Bettelheim et al (2010) menyebutkan bahwa protein dapat terdenaturasi oleh beberapa faktor di antaranya suhu, asam, basa, alkohol, dan ion logam. Suhu dapat mengganggu ikatan hidrogen pada subunit rantai kuartener pada suatu protein. Perubahan tersebut mengakibatkan berubahnya sifat fisik maupun fungsi secara keseluruhan.
Gambar 2 Hasil SDS PAGE ikan tenggiri dan olahannya.
(M= Marka; 1= tenggiri papan utuh; 2= tenggiri totol utuh; 3= tenggiri giling 1; 4= tenggiri giling 2; 5= otak-otak 1; 6= Otak-otak 2; 7= kerupuk 1; 8= kerupuk 2; 9= bakso 1).
12
pengolahan. Sampel kerupuk dan bakso (7 dan 9) menunjukkan tidak terdapat satupun pita protein. Hal ini menunjukkan bahwa semua protein yang terdapat pada sampel tersebut telah terpotong menjadi peptida yang berukuran lebih kecil dari 8,8 kDa sehingga peptida tersebut tidak terdeteksi. Syahrudin (2013) melaporkan bahwa denaturasi pada daging ikan yang disebabkan oleh penggaraman dapat memotong protein menjadi ukuran lebih kecil sehingga dapat menyebabkan tidak terdeteksinya protein pada saat pengujian SDS-PAGE. Sutanto (2010) menyatakan terdapat fraksi protein yang hilang akibat denaturasi oleh panas selama pengolahan pada bakso yang diduga terbuat dari daging babi. Panas menyebabkan energi kinetik pada molekul protein meningkat dan mengganggu ikatan hidrogen. Perlakuan panas yang tinggi dan berlangsung dalam waktu yang lama dapat menyebabkan protein terdegradasi ke dalam ukuran yang lebih kecil (Winarno 1997). Proses yang terjadi selama pemasakan menyebakan rusaknya struktur protein sehingga tidak dapat digambarkan melalui pita SDS-PAGE. Identifikasi bahan baku (spesies) pada produk tidak dapat dilakukan dengan SDS-PAGE, oleh karena itu perlu adanya analisis lanjut yakni dengan DNA barcoding.
Isolasi DNA
Isolasi DNA yang dilakukan merupakan modifikasi dari metode cetyl trimethylammonium bromide (CTAB). Prinsip isolasi DNA yaitu pelisisan sel, ekstraksi, pengendapan DNA, dan pemurnian DNA (Nishiguchi et al. 2002). Proses pemecahan membran sel (lisis) bertujuan untuk mengeluarkan isi sel dilakukan secara kimiawi dengan menambahkan bufer ATL yang mengandung detergen SDS. Detergen SDS dapat melarutkan lipid pada membran sel sehingga terjadi distabilisasi membran sel dan dapat mengurangi aktifitas enzim nuklease yang dapat mendegradasi DNA (Surzycki 2000). Penambahan protease K dapat mempercepat proses lisis karena dapat mendegradasi protein globular dan merusak rantai polipeptida dalam komponen sel (Ahmed et al. 2014). Isolasi DNA menggunakan kit isolasi yaitu Dneasy blood and tissue yang didapatkan dari PT Genecraft Labs. Proses ekstraksi yaitu pemisahan molekul DNA dari bahan lain yang terdapat pada sel dilakukan dengan menambahkan bufer AL yang mengandung lisin dan guanidium chloride. Lisin dan garam guanidium chloride
dapat merusak membran organel sel seperti mitokondria dan nucleus. Pemisahan DNA dilakukan berdasarkan perbedaan berat molekul dengan bantuan sentrifugasi. Proses pengendapan DNA dilakukan dengan penambahan bufer AW 1 dan AW 2 yang mengandung isopropanol dan guanidium chloride.
13
nilai konsentrasi di atas 2,0 menunjukkan telah terkontaminasi oleh RNA. Hasil pengukuran konsentrasi dan kemurnian DNA ditampilkan pada Tabel 5.
Semua sampel memiliki DNA dengan konsentrasi yang berbeda. Konsentrasi DNA yang tertinggi terdapat pada sampel ikan utuh tenggiri papan yaitu 185,5 ng/µL. Konsentrasi DNA yang terendah terdapat pada sampel kerupuk 5 yaitu 0,55 ng/µL. Konsentrasi DNA semua ikan segar (69,5-185,5 ng/µL) lebih besar dibandingkan dengan produk olahan (0,55-29,5 ng/µL). Pencampuran dengan bahan lain dan proses selama pengolahan menyebabkan DNA pada olahan tenggiri berkurang. Sebagian besar sampel (21 dari 30 sampel) memiliki nilai kemurnian DNA di luar kisaran nilai ideal (1,8-2,0), namun sampel masih dapat diamplifikasi dengan mesin PCR. Ahmed et al (2014) menyatakan bahwa kemurnian DNA lebih kecil dari 1,8 untuk sampel bakteri Methycylin resistant Staphylococcus aureus (MRSA) masih dapat diamplifikasi oleh mesin PCR dan memberikan pita DNA yang jelas ketika dielektroforesis. Suhu annealing yang tepat dan waktu penempelan primer pada DNA template yang cukup memperbesar peluang keberhasilan amplifikasi DNA.
Tabel 5 Konsentrasi dan kemurnian DNA sampel
No Sampel Konsentrasi (ng/µL) Kemurnian (260/280)
1 Tenggiri Papan 1 69,60 1,70
2 Tenggiri Papan 2 185,50 2,09
3 Tenggiri Papan 3 125,20 2,08
4 Tenggiri totol 1 87,00 2,07
5 Tenggiri totol 2 102,70 1,99
6 Tenggiri totol 3 70,00 2,09
7 Tenggiri giling 1 43,90 2,03
8 Tenggiri giling 2 56,40 1,80
9 Tenggiri giling 3 32,70 1,80
10 Pempek 1 5,90 1,50
11 Pempek 2 4,00 1,70
12 Pempek 3 3,40 1,70
13 Pempek 4 29,50 1,90
14 Bakso 1 4,20 1,50
15 Bakso 2 2,20 1,70
16 Bakso 3 3,10 1,40
17 Bakso 4 24,95 1,90
18 Bakso 5 20,50 1,40
19 Bakso 6 10,50 2,10
20 Otak-otak 1 19,40 1,70
21 Otak-otak 2 9,30 1,80
22 Otak-otak 3 15,50 2,00
23 Otak-otak 4 18,50 1,80
24 Otak-otak 5 11,00 1,70
25 Kerupuk 1 6,10 2,20
26 Kerupuk 2 6,80 1,70
27 Kerupuk 3 7,60 1,50
28 Kerupuk 4 3,75 2,00
29 Kerupuk 5 0,55 1,22
14
Desain Primer
Primer yang didesain berdasarkan DNA mitokondria yaitu gen penyandi COI dan cyt b. Gen penyandi COI dan cyt b sering digunakan untuk mengidentifikasi spesies. Primer didapatkan dari spesies yang memiliki kekerabatan yang sama dan yang berdekatan dengan sampel yang digunakan yaitu dari family Scombridae. Primer terdiri dari dua bagian yaitu forward dan reverse. Panjang gen target COI yaitu 560 basa dan cyt b 780 basa. Panjang primer yang ideal adalah sekitar 18-24 basa. Primer yang pendek berpengaruh terhadap rendahnya keakuratan pada saat penempelan terhadap cetakan DNA gen target yang diinginkan sedangkan primer yang terlalu panjang akan menyebabkan tingginya suhu leleh. Peluang terjadinya struktur sekunder kedua jenis primer tersebut adalah tidak ada berdasarkan pengujian oligoevaluator. apabila ada struktur sekunder maka kemungkinan primer tersebut menempel pada bagian primer itu sendiri sehingga proses amplifikasi tidak terjadi. Struktur sekunder harus dihindari supaya proses amplifikasi berjalan. Primer bagian forward
menempel pada salah satu utas DNA template dan primer bagian reverse
menempel pada utas yang lainnya. Jarak antara ujung primer forward dan ujung primer reverse disebut panjang gen target. Panjang gen target cyt b 780 pasang basa dan COI 560 pasang basa.
Amplifikasi DNA dan Elektroforesis
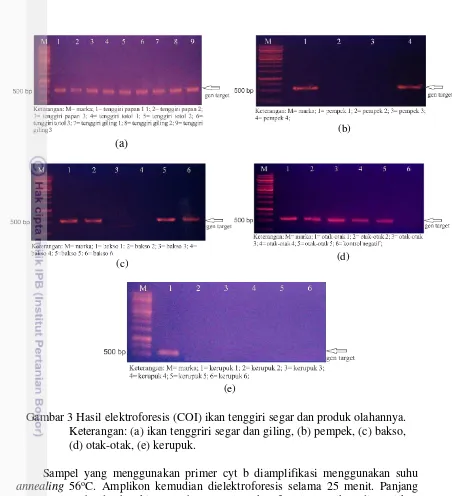
Sampel dengan primer COI diamplifikasi menggunakan suhu annealing
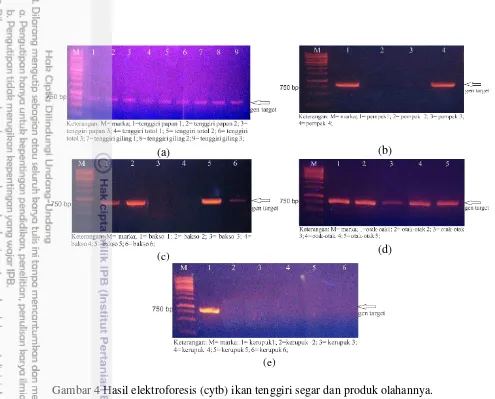
51oC. Amplikon kemudian dielektroforesis selama 25 menit. Panjang gen target COI adalah sekitar 560 basa. Hasil elektroforesis amplikon ditampilkan pada Gambar 3 (COI).
DNA bermuatan negatif, sehingga molekul DNA akan bergerak dari kutub negatif ke kutub positif. DNA yang bermuatan negatif akan ditarik oleh muatan listrik positif, sehingga molekul DNA akan terpisah sesuai dengan ukuran berat molekulnya (Howe 2007). Molekul DNA yang terpisah kemudian divisualisasi menggunakan sinar UV dan akan berpendar karena pewarna yang ditambahkan seperti ethidium bromida.
15
(a)
(b)
(c) (d)
[image:31.595.94.546.73.569.2](e)
Gambar 3 Hasil elektroforesis (COI) ikan tenggiri segar dan produk olahannya. Keterangan: (a) ikan tenggriri segar dan giling, (b) pempek, (c) bakso, (d) otak-otak, (e) kerupuk.
Sampel yang menggunakan primer cyt b diamplifikasi menggunakan suhu
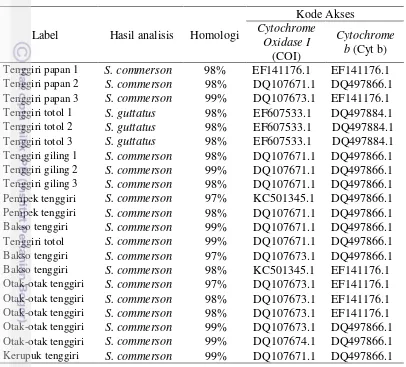
annealing 56oC. Amplikon kemudian dielektroforesis selama 25 menit. Panjang gen target cyt b adalah sekitar 780 basa. Hasil elektroforesis amplikon ditampilkan pada Gambar 4.
16
(a) (b)
(c) (d)
[image:32.595.29.524.76.475.2](e)
Gambar 4 Hasil elektroforesis (cytb) ikan tenggiri segar dan produk olahannya. Keterangan: (a) ikan tenggriri segar dan giling, (b) pempek, (c) bakso, (d) otak-otak, (e) kerupuk.
17
Identifikasi Spesies dan Sekuensing
[image:33.595.110.516.187.554.2]Hasil sekuensing kemudian disejajarkan menggunakan software Mega 5.2. Hasil pensejajaran kemudian dianalisis secara online melalui BLAST. Hasil analisis BLAST dapat dilihat pada Tabel 6 (COI dan Cyt b).
Tabel 6 Hasil identifikasi spesies berdasarkan analisis BLAST (COI dan Cyt b)
Label Hasil analisis Homologi
Kode Akses
Cytochrome Oxidase I
(COI)
Cytochrome b (Cyt b)
Tenggiri papan 1 S. commerson 98% EF141176.1 EF141176.1
Tenggiri papan 2 S. commerson 98% DQ107671.1 DQ497866.1
Tenggiri papan 3 S. commerson 99% DQ107673.1 EF141176.1
Tenggiri totol 1 S. guttatus 98% EF607533.1 DQ497884.1
Tenggiri totol 2 S. guttatus 98% EF607533.1 DQ497884.1
Tenggiri totol 3 S. guttatus 98% EF607533.1 DQ497884.1
Tenggiri giling 1 S. commerson 98% DQ107671.1 DQ497866.1
Tenggiri giling 2 S. commerson 99% DQ107671.1 DQ497866.1
Tenggiri giling 3 S. commerson 98% DQ107671.1 DQ497866.1
Pempek tenggiri S. commerson 97% KC501345.1 DQ497866.1
Pempek tenggiri S. commerson 98% DQ107671.1 DQ497866.1
Bakso tenggiri S. commerson 99% DQ107671.1 DQ497866.1
Tenggiri totol S. commerson 99% DQ107671.1 DQ497866.1
Bakso tenggiri S. commerson 97% DQ107673.1 DQ497866.1
Bakso tenggiri S. commerson 98% KC501345.1 EF141176.1
Otak-otak tenggiri S. commerson 97% DQ107673.1 EF141176.1
Otak-otak tenggiri S. commerson 98% DQ107673.1 EF141176.1
Otak-otak tenggiri S. commerson 98% DQ107673.1 EF141176.1
Otak-otak tenggiri S. commerson 99% DQ107673.1 DQ497866.1
Otak-otak tenggiri S. commerson 99% DQ107674.1 DQ497866.1
Kerupuk tenggiri S. commerson 99% DQ107671.1 DQ497866.1
Berdasarkan Tabel 6, dua puluh satu sampel menunjukkan kesesuaian antara bahan baku yang tercantum pada kemasan dengan hasil analisis BLAST. Hasil analisis BLAST dilihat berdasarkan persentase nilai identity. Nilai persentase
18
Pohon Filogenetik
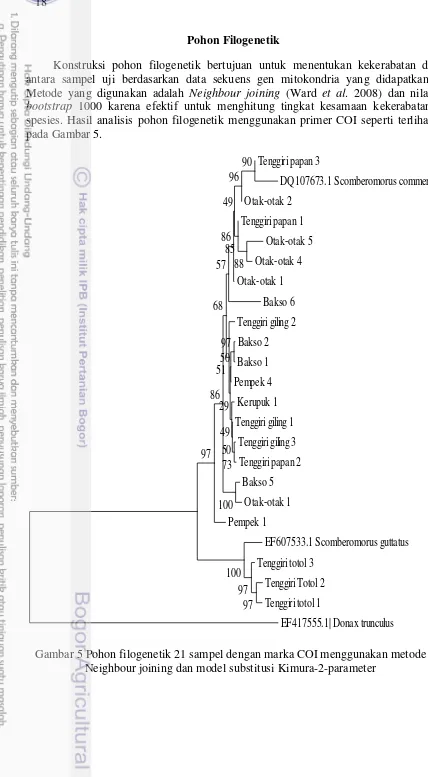
Konstruksi pohon filogenetik bertujuan untuk menentukan kekerabatan di antara sampel uji berdasarkan data sekuens gen mitokondria yang didapatkan. Metode yang digunakan adalah Neighbour joining (Ward et al. 2008) dan nilai
[image:34.595.50.478.62.839.2]bootstrap 1000 karena efektif untuk menghitung tingkat kesamaan kekerabatan spesies. Hasil analisis pohon filogenetik menggunakan primer COI seperti terlihat pada Gambar 5.
Gambar 5 Pohon filogenetik 21 sampel dengan marka COI menggunakan metode Neighbour joining dan model substitusi Kimura-2-parameter
Tenggiri papan 3
DQ107673.1 Scomberomorus commerson
Otak-otak 2
Tenggiri papan 1
Otak-otak 5
Otak-otak 4
Otak-otak 1
Bakso 6
Tenggiri giling 2
Bakso 2
Bakso 1
Pempek 4
Kerupuk 1
Tenggiri giling 1
Tenggiri giling 3
Tenggiri papan 2
Bakso 5
Otak-otak 1
Pempek 1
EF607533.1 Scomberomorus guttatus
Tenggiri totol 3
Tenggiri Totol 2
Tenggiri totol 1
19
Spesies Scomberomorus commerson dan Scomberomorus guttatus diperoleh dari genebank dengan accession number DQ107673.1 (Scomberomorus commerson) dan EF607533.1 (Scomberomorus guttatus). Donax trunculus
digunakan sebagai pembanding outgrup dengan accession number EF417555.1. Hasil analisis pohon filogenetik menggunakan primer cyt b seperti terlihat pada Gambar 6. Spesies Scomberomorus commerson dan Scomberomorus guttatus diperoleh dari genebank dengan accession number DQ497866.1 (Scomberomorus commerson), DQ497868.1 (Scomberomorus guttatus). Donax trunculus digunakan sebagai pembanding outgrup dengan accession number EF417554.1.
Gambar 6 Pohon filogenetik 21 sampel dengan marka cytochrome b menggunakan metode Neighbour Joining dan model substitusi Kimura-2-parameter
[image:35.595.82.530.153.790.2]Tenggiri papan 1 Tenggiri papan 2 Otak-otak 1 Bakso 5 Otak-otak 5 Kerupuk 1 Bakso 2 Pempek 4 Otak-otak 3 Giling 1 Bakso 6 Otak-otak 4
DQ497866.1| Scomberomorus commerson Tenggiri papan 3
Giling 2 Giling 3 Bakso 1 Otak-otak 2
Pempek 1
DQ497868.1| Scomberomorus guttatus Tenggiri totol 1
Tenggiri totol 2 Tenggiri totol 3
20
Pengelompokkan ikan berdasarkan pohon filogenetik untuk primer COI dan cyt b memberikan hasil yang sama. Sampel ikan segar terbagi menjadi dua kelompok yaitu tenggiri papan satu kelompok dengan Scomberomorus commerson dan tenggiri totol satu kelompok dengan Scomberomorus guttatus. Semua sampel produk olahan masuk ke dalam kelompok Scomberomorus commerson. Hasil ini sama dengan hasil analisis BLAST yang menyatakan bahwa semua sampel produk terdeteksi sebagai spesies Scomberomorus commerson. Tingkat akurasi dan ketetapan posisi dari percabangan yang terbentuk ditunjukkan oleh nilai persentasi bootstrap yang berkisar antara 1-100%. Nilai bootstrap
mendekati 100% dan atau sama dengan 100% menunjukkan percabangan sangat kuat (Prehadi et al. 2015). Konstruksi pohon filogenetik dengan marka molekuler COI memiliki nilai bootstrap 97% untuk cabang kelompok Scomberomorus commerson dan 100% untuk cabang kelompok Scomberomorus guttatus. Nilai
bootstrap pada pohon filogenetik dengan marka molekuler cyt b adalah 100% untuk cabang kelompok Scomberomorus commerson dan 70% untuk cabang kelompok Scomberomorus guttatus. Nilai bootstrap 100% atau mendekati 100% pada setiap percabangan kelompok baik pada marka molekuler COI maupun cyt b menunjukkan bahwa percabangan kelompok yang terbentuk merupakan cabang yang kuat.
4
SIMPULAN DAN SARAN
Simpulan
DNA barcoding dapat digunakan untuk identifikasi spesies pada produk olahan ikan secara akurat. Sembilan sampel dari tiga puluh sampel yang dikoleksi (30%), tidak mengandung ikan tenggiri seperti tercantum pada kemasannya.
Saran
21
DAFTAR PUSTAKA
[AOAC] Association of Official Analytical and Chemistry. 2005. Official Methods of Analysis of The Association of official Analytical of Chemist.
Arlington, Virginia, (USA): The Association of Official Analytical of Chemist, Inc.
Affandi R, Sjafei DS, Raharjo MF, Sulistiono. 1992. Ikhtiologi. Bogor (ID). Pedoman Kerja Laboratorium. Departemen Pendidikan dan kebudayaan. Direktorat Jendral Pendidikan tinggi. Pusat Antar Universitas Ilmu Hayat. Institut Pertanian Bogor.
Ahmed OB, Ashgar AH, Elhassan MM. 2014. Comparison of three dna extraction methods for PCR analysis of bacterioal genomic DNA. Academic Journal. Vol 8(6): 598-602.
Bettelheim FA, Brown WH, Campbell MK, Farrell SO. 2010. Introduction to Organic and Biochemistry. Seventh edition. London (UK). ISBN-10:0495391964.p337.
Cawthorn DM, Steinman HA, Witthuhn RC. 2012. DNA barcoding reveals a high incidence of fish species misrepresentation and substitution on the South African market. Food Control 46:30-40.
Chang HC, Chen HH. 2013. Association between textural profiles and surface electromyographic (sMEG) behaviors of microwavable cassava cuttlefish crackers with various expansion ratios. Food Research International
53(3):334-341.
Collette BB, Nauen CE. 1983. FAO species catalogue. Vol 2. Scombrids of The World: An Annotated and Illustrated Catalogue of Tunas, Mackerel, Bonitos and Related Spesies Known to Date. Rome (ITA): FAO.
Dawnay N, Ogden R, McEwing R, Carvalho GR, Thorpe RS. 2007. Validation of the barcoding gene coi for use in forensic genetic species identification.
Forensic Science International. 173:1-6.
Dwiyitno. 2010. Deteksi pemalsuan ikan menggunakan analisis DNA. Dikutip dari laman (www.ristek.go.id) diakses pada bulan maret 2016.
Espiñeira M, Gonzalez-Lavín N, Vieites JM, Santaclara FJ. 2009. Development of a method for the identification of scombroid and common substitute species in seafood products by FINS. Food Chemistry 117: 698-704. Eur-Lex (European legal text). 2013. Regulation (EU) no 1379/2013 of the
European Parliament and of the Council. Available from www.eur-lex.europa.eu/legal-content.
FAO (Food and Agriculture Organization of the United Nations). 2013. Fishery statistical collection. Global capture production. Available from www.fao.org/fishery/statistics/global-capture production.
Filonzi L, Stefania C, Marina V, Francesco NM. 2010. Molecular barcoding reveals mislabeling of commercial fish products in Italy. Food Research International Vol 43 (5):1383-1388.
22
Haye PA, Segovia NI, Vera R, Gallardo MA, Gallardo-Escárate C. 2012. Authentication of commercialized crab-meat in Chile using DNA Barcoding. Food Control 25: 239-244.
Hoar WS, Randall DJ. 1970. Fish Physiology. Forth Volume. London (UK). Academic press.
Hold GL, Russell VJ, Pryde SE, Rehbein H, Quinteiro J, Rey-Mendez m, Sotelo CG, Pérez-Martin R, Santos AT, Rosa C. 2001. Validation of a PCR of a PCR-RFLD based method for the identification of salmon species in food products. Eur Food Res Technol 212: 385-389.
Howe C. 2007. Gen Cloning and Manipulation. Second Edition. Cambridge University press. New York (USA).
Huang YR, Yin MC, Hsieh YL, Yeh YH, Yang YC, Chung YL, Hsieh CHE. 2014. Authentication of consumer fraud in Taiwanese fish production by molecular trace evidence and forensically informative nuclear sequencing.
Food Research International 55: 294-302.
Jacquet JL, Pauly D. 2008. Trade secrets: renaming and mislabeling of seafood.
Marine Policy 32(3): 309-318.
Jérôme M, Lemaire C, Verrez-Bagnis V, Etienne M. 2003. Direct sewuencing method for species identification of canned sardine and sardine-type products. Journal Agriculture Food Chemistry 51: 7326-7332.
Laemmli UK. 1970. Cleavage of structural protein during the assembly of the heat of bacteriophag T4. Nature. 227(10):680-685.
Leeuwen SP, van Velzen MJ, Swart CP, van der Veen I, Traag WA, de Boer J. 2009. Halogenated contaminants in farmed salmon, trout, tilapia, pangasius, and shrimp. Environmental Science and Technology 43:4009-4015.
Mackie IM, Pryde SE, Gonzales-Sotelo C, Medina I, Peréz-Martin R, Quinteiro J, Rey-Mendez M, Rehbein H. 1999. Challenges in the identification of species of vanned fish. Trend and Food Science and Technology 10: 9-14. Marko PB, Lee SC, Rice AM, Gramling JM, Fitzhenry TM, McAlister JS. 2004.
Mislabeling of a depleted reef fish. Nature 430:309-310.
Mcpherson GR. 1985. Northern line fishery for mackerel still important. Aust fish. 44(8):12-14.
Muladno, 2002. Seputar Teknologi Rekayasa Genetika. Bogor (ID): Pustaka Wirausaha Muda.
Nazemroaya S, Sahari MA, Rezae M. 2011 Identification of fatty acid in mackerel (Scomberomorus commerson) and shark (Charcharhinus dussumieri) fillets and their changes during six month of frozen storage at -18 C.
J.Agr.Sci. Tech Vol.13:553-566.
Nishiguchi MK, Daukakis P, Egan M. 2002. DNA Isolation Procedures. Bassel (SWZ). Berkauser.
Orrell TM, Collette BB, Jhonson GD. 2006. Molecular data support separate scombroid and xiphioid clades. Bulletin of marine science. 79(3):505-519. Pearson WR. 2013. An introduction to sequences similiarity (homology)
searching. Bioinformatics Vol 3(1):1-3.
23
species landed in muncar fisheries landing site in comparison with southern java fishing port. Biodiversitas Volume 16(1):55-61.
Roe AD, Sperling FAH. 2007. Patterns of evolution of mytochodria cytochrome COI and II DNA and implications for DNA barcoding. Molecular Phylogenetic and Evolution 44:325-345.
Sambrook J, Russell DW. 2001. Molecular Cloning : a Laboratory Manual 3rd Edition. New York (USA) : Cold Spring Harbor.
Sengun IY, Turp GY, Icier F, Kindirci P, Kor G. 2014. Effects of ohmic heating for pre-cooking or meatball on some quality and safety attributes. Food Science and Technology 55(4):232-239.
Shaltout OE. 1993. Chip-like cod based crackers: acceptability and chemical composition. Food Tech 26(6):558-562.
Sumarto, Rengi P. 2014. Pengembangan penerapan produksi bersih hasil pengolahan perikanan berbasis ikan patin. Pusat penelitian lingkungan hidup. Universitas Riau.
Sutanto E. 2010. Penggunaan sds-page untuk karakterisasi fraksi protein sebagai alternatif metode identifikasi pencampuran daging babi ke dalam bakso. Jurnal Ternak 1(1):1-7
Sutarshiny S, Sivhashantini K. 2011. Proximate composition of three species of scombreroides fish from sri lankan water. Asian Journal of Clinical Nutrition 3 (3):103-111.
Surzycki S. 2000. Basic techniques in Molecular Biology. Heidelberg New York (USA). Springer-verlag.
Syahrudin H. 2013. Pengaruh penggaraman terhadap protein ikan layang. Jurnal Ilmiah Mahasiswa Universitas Surabaya 2(1):1-6.
Tamura K, Peterson D, Stecher G, Nei M, Kumar S. 2011. Mega 5: moleculer evolutionary genetics analysis using maximum likelihood distance, and maximum parsimony methods. Mol biol evol 10: 2731-9.
Tsai LC, Huang MT, Hsiao CT. 2007. Species identification of animal specimens by cytochrome b gene. Forensic Science Journal 6(1):63-65.
Turp GY, Icier F, Kor G. 2016. Influence of infrared final cooking on color, texture and cooking charachteristics of ohmically pre-cooking meatball.
Meat science 114(1):46-53.
Ward RD, Holmes BH, White WT, Last PR, 2008. DNA barcoding australasian chondrichtylan: result and possible uses in conservation. Freswater Res. 59(1): 57-71.
Winarno FG. 1997. Kimia pangan dan gizi. Jakarta (INA). Gramedia.
Wong LL. 2011. DNA barcoding and related molecular markers for fish species authentication. Phylogenetic Assessment and Population Studies. Auburn University. Auburn. Alabama. 118 p.
Zaitsev V, Kizevetter I, Laqunov L, Makarava T, Minder L, Podesvalov. 1969.
Fish Curing and Processing. MIR Publ., Moscow, 722p.
24
.Lampiran 1 Sampel dan lokasi pengambilan
Sampel yang digunakan dalam penelitian ini sebanyak 30 sampel yang terdiri dari 6 ikan segar dan 24 produk yang terbuat dari ikan tenggiri berdasarkan keterangan yang tercantum pada kemasaannya. Berikut adalah contoh sampel yang dikoleksi:
No Jenis
[image:40.595.33.502.141.758.2]Sampel
Gambar Jumlah Lokasi
1 Ikan segar 6 Jakarta
2 Tenggiri Giling
3 Bogor
3 Pempek 4 Bogor
4 Bakso 6 Bandung,
Bogor
5 Otak-otak 5 Makassar,
Pandeglang, Bogor
6 kerupuk 6 Bogor,
25
Lampiran 2 Alat dan bahan isolasi DNA, amplifikasi DNA, dan elektroforesis
DNA kit isolasi Qiagen Kappa Master mix Agarose
Mesin PCR Mesin Elektroforesis
Lampiran 3 Tabel protein berdasarkan berat molekul sebagai marka SDS-PAGE Berat Molekul (kDa) Protein
192 Myosin
112 β-galaktosidase
85 Bovin serum albumin
60 Glutamat dehidrogenase
47 Ovalbumin
35 Carbonic anhydrase
28 Myoglobin
21 Lysozime
26
Lampiran 4 Hasil elektroforegram sampel menggunakan primer spesifik COI dan
Cytochrome b
No Sampel Keterangan
1 Tenggiri papan 1 +
2 Tenggiri papan 2 +
3 Tenggiri papan 3 +
4 Tenggiri totol 1 +
5 Tenggiri totol 2 +
6 Tenggiri totol 3 +
7 Tenggiri giling 1 +
8 Tenggiri giling 2 +
9 Tenggiri giling 3 +
10 Pempek 1 +
11 Pempek 2 -
12 Pempek 3 -
13 Pempek 4 +
14 Bakso 1 +
15 Bakso 2 +
16 Bakso 3 -
17 Bakso 4 -
18 Bakso 5 +
19 Bakso 6 +
20 Otak-otak 1 +
21 Otak-otak 2 +
22 Otak-otak 3 +
23 Otak-otak 4 +
24 Otak-otak 5 +
25 Kerupuk 1 +
26 Kerupuk 2 -
27 Kerupuk 3 -
28 Kerupuk 4 -
29 Kerupuk 5 -
27
Lampiran 5 Nomor accession sample dari genbank marka cytochrome b
Sample Nama Ilmiah Submission ID Accession number
Tenggiri papan 1 Scomberomorus commerson 1907017 KX002075 Tenggiri papan 2 Scomberomorus commerson 1907017 KX002076 Tenggiri papan 3 Scomberomorus commerson 1907017 KX002077 Tenggiri totol 1 Scomberomorus guttatus 1907017 KX002078 Tenggiri totol 2 Scomberomorus guttatus 1907017 KX002079 Tenggiri totol 3 Scomberomorus guttatus 1907017 KX002080 Tenggiri giling 1 Scomberomorus commerson 1907017 KX002081 Tenggiri giling 2 Scomberomorus commerson 1907017 KX002082 Tenggiri giling 3 Scomberomorus commerson 1907017 KX002083
Pempek 2 Scomberomorus commerson 1907022 KX002084
Bakso 1 Scomberomorus commerson 1907022 KX002085
Bakso 2 Scomberomorus commerson 1907022 KX002086
Bakso 5 Scomberomorus commerson 1907022 KX002087
Bakso 6 Scomberomorus commerson 1907022 KX002088
Otak-otak 1 Scomberomorus commerson 1907022 KX002089 Otak-otak 2 Scomberomorus commerson 1907022 KX002090 Otak-otak 3 Scomberomorus commerson 1907022 KX002091 Otak-otak 4 Scomberomorus commerson 1907022 KX002092
28
Lampiran 6 Contoh sampel submit ke genbank
LOCUS Seq1 736 bp DNA linear VRT 30-MAR-2016 DEFINITION Scomberomorus commerson isolate DYM SC1 cytochrome b (cytb) gene, partial CDS.
ACCESSION Seq1 VERSION
KEYWORDS .
SOURCE mitochondrion Scomberomorus commerson (narrow-barred Spanish mackerel)
ORGANISM Scomberomorus commerson
Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Actinopterygii; Neopterygii; Teleostei; Neoteleostei;
Acanthomorphata; Pelagiaria; Scombriformes; Scombridae; Scomberomorus.
REFERENCE 1 (bases 1 to 736)
AUTHORS Maulid,D.Y., Nurilmala,M., Nurjanah,--., Madduppa,H., Widyastuti,U. and Kusuma,W.A.
TITLE Detection of Mackerel Fish (Scomberomorus sp.) and its Processed Products Using DNA Barcoding
JOURNAL Unpublished
REFERENCE 2 (bases 1 to 736)
AUTHORS Maulid,D.Y., Nurilmala,M., Nurjanah,--., Madduppa,H., Widyastuti,U. and Kusuma,W.A.
TITLE Direct Submission
JOURNAL Submitted (30-MAR-2016) Aquatic Products Technology, Bogor Agricultural University, Lingkar akademis Rd, Bogor, West Java 16680, Indonesia
COMMENT Bankit Comment: ALT EMAIL:[email protected]. Bankit Comment: TOTAL # OF SEQS:10.
Bankit Comment: TOTAL # OF SETS:10.
##Assembly-Data-START##
Sequencing Technology :: Sanger dideoxy sequencing ##Assembly-Data-END##
FEATURES Location/Qualifiers source 1..736
/organism="Scomberomorus commerson" /organelle="mitochondrion" /mol_type="genomic DNA" /db_xref="taxon:218798" /country="Indonesia" /collection_date="20-nov-2014"
/collected_by="personal: Deden Yusman Maulid" gene <1..>736
/gene="cytochrome b" CDS <1..>736
/gene="cytochrome b" /note="Mitochondrial gene" /codon_start=1 /transl_table=2 /product="cytochrome b" /translation="LYYGSYLYKETWNIGVVLLLLVMMTAFVGYVLPWGQMSFWGATV ITNLLSAVPYVGTMLVEWIWGGFSVDNATLTRFFAFHFLFPFVILAMTILHLLFLHET GSNNPMGLNSNADKISFHPYFSYKDLLGFAVLLIALVSLALFSPNLLGDPDNFTPANP MVTPPHIKPEWYFLFAYAILRSIPNKLGGVLALLASILVLMVVPFLHTSKQRALTFRP ASQFLFWALIADVAILTWIGGMPAEQP"
BASE COUNT 178 a 253 c 110 g 195 t ORIGIN
29
RIWAYAT HIDUP
Penulis dilahirkan di Kabupaten Garut, Jawa Barat pada 31 Oktober 1987 dari pasangan Endang Dahya (Alm) dan Rosyati. Penulis merupakan anak ke-9 dari 9 bersaudara. Penulis memulai jenjang pendidikan formal di SDN Banjarwangi II (tahun 1994-2000), kemudian melanjutkan pendidikan di SLTPN 2 Bandung (tahun 2000-2003). Pendidikan menengah atas ditempuh di SMAN 4 Bandung dan lulus pada tahun 2006. Penulis menyelesaikan gelar sarjana di Fakultas Perikanan dan Ilmu Kelautan, Universitas Padjadjaran pada tahun 2010.