ISOLASI DNA DARI SAMPEL DAGING DAN SIRIP DELAPAN
SPESIES IKAN LAUT UNTUK PCR-RFLP
PUTRI EKANDINI
FAKULTAS KEDOKTERAN HEWAN INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN SUMBER
INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Isolasi DNA dari Sampel Daging dan Sirip Delapan Spesies Ikan Laut untuk PCR-RFLP adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, September 2014
Putri Ekandini
ABSTRAK
PUTRI EKANDINI. Isolasi DNA dari Sampel Daging dan Sirip Delapan Spesies Ikan Laut untuk PCR-RFLP. Dibimbing oleh KUSDIANTORO MOHAMAD dan WAHONO ESTHI PRASETYANINGTYAS.
Penelitian ini bertujuan untuk membandingkan konsentrasi, tingkat kemurnian, visualisasi elektroforesis, dan penggunaan DNA hasil isolasi untuk
polymerase chain reaction-restriction fragment length polymorphism (PCR-RFLP) dari delapan spesies ikan laut. Sampel DNA yang diperoleh dari daging dan sirip ikan baronang (Siganus javus), kerapu (Epinephelus sp), ekor kuning (Caesionidae sp), kakap merah (Lutjanus campechanus), kurisi (Nemipterus nematophorus), kue (Caranx melampygus), bawal hitam (Parastromateus niger), dan selar (Selaroides leptolepis) diisolasi menggunakan metode presipitasi amonium asetat. Primer universal sitokrom b digunakan untuk mengamplifikasi mtDNA menggunakan PCR. Amplikon dipotong menggunakan enzim restriksi
Hinf I. Hasil menunjukkan bahwa konsentrasi dan kemurnian DNA sampel daging dan sirip tidak berbeda nyata (P>0.05) yaitu berturut-turut 22.01±20.29 dan 1.44±0.27 untuk konsentrasi dan kemurnian DNA sampel daging, serta 52.86±41.37 dan 1.54±0.23 untuk konsentrasi dan kemurnian DNA sampel sirip. Sampel DNA ikan laut dapat diisolasi dari seluruh sampel daging dan sirip tetapi hanya satu sampel yaitu dari sirip ikan selar yang memiliki konsentrasi yang tinggi (150.50 ng/μl) dan kemurnian yang baik (rasio A260/280=1.91) serta menunjukkan hasil positif setelah PCR-RFLP dengan enzim Hinf I dan menghasilkan dua pita dengan ukuran 332 bp dan 27 bp.
ABSTRACT
PUTRI EKANDINI. DNA Isolation from Meat and Fin of Eight Marine Fish Specieses for PCR-RFLP. Supervised by KUSDIANTORO MOHAMAD and WAHONO ESTHI PRASETYANINGYAS.
The aim of this research is to compare DNA concentration, purity, electrophoresis visualization, and capability of DNA for polymerase chain reaction-restriction fragment length polymorphism (PCR-RFLP) analysis from eight marine fishes. The DNA samples were isolated from meat and fin tissues of streaked spinefoot (Siganus javus), grouper (Epinephelus sp), fusiliers (Caesionidae sp), red snapper (Lutjanus campechanus), double whip thread fin bream (Nemipterus nematophorus), trevally (Caranx melampygus), black pomfret (Parastromateus niger), and yellow stripe scad (Selaroides leptolepis) fishes using ammonium acetate precipitation method. Universal primer of cytochrome b was used to amplify mtDNA using PCR and the amplicon was digested using restriction enzyme Hinf I. The result showed that the average of concentration and purity value of fish DNA were no different (P>0.05) between meat and fin samples (22.29±20.29 and 1.44±0.27 for DNA concentration and purity value of meat samples, respectively; and 52.86±41.37 and 1.54±0.23 for DNA concentration and purity value of fin samples, respectively). Marine fish DNA could be isolated from all samples but only one sample from fin of yellow striped scad fish showed high concentration (150.50 ng/μl) with good purity (A260/280=1.91) and this DNA was succesfully separated into two bands with 332 bp and 27 bp after PCR–RFLP using enzyme Hinf I.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Kedokteran Hewan pada
Fakultas Kedokteran Hewan
ISOLASI DNA DARI SAMPEL DAGING DAN SIRIP DELAPAN
SPESIES IKAN LAUT UNTUK PCR-RFLP
PUTRI EKANDINI
FAKULTAS KEDOKTERAN HEWAN INSTITUT PERTANIAN BOGOR
Judul Skripsi : Isolasi DNA dari Sampel Daging dan Sirip Delapan Spesies Ikan Laut untuk PCR-RFLP
Nama : Putri Ekandini
NIM : B04100015
Disetujui oleh
Drh Kusdiantoro Mohamad, MSi PAVet Pembimbing I
Drh Wahono Esthi Prasetyaningtyas, MSi PAVet Pembimbing II
Diketahui oleh
Drh Agus Setiyono, MS PhD APVet Wakil Dekan
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah Subhanahu Wa Ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Judul yang dipilih dalam penelitian yang dilaksanakan pada bulan Januari sampai Juli 2014 ini adalah Isolasi DNA dari Sampel Daging dan Sirip Delapan Spesies Ikan Laut untuk PCR-RFLP.
Terima kasih penulis ucapkan kepada Bapak Drh Kusdiantoro Mohamad, MSi PAVet dan Ibu Drh Wahono Esthi Prasetyaningtyas, MSi PAVet selaku pembimbing, Dr Drh Ita Djuwita, Mphil PAVet (K) (alm) yang telah merancang, membimbing, dan memberikan bantuan dana penelitian, serta Dr Drh Ni Luh Putu Ika Mayasari yang telah memberikan bantuan bahan penelitian. Di samping itu, penghargaan penulis sampaikan kepada Wahyudin, Amd selaku staf Laboratorium Embriologi Fakultas Kedokteran Hewan Institut Pertanian Bogor dan teman-teman sesama penelitian (Dwi Budiono, Juita Siregar, Fitri Susana, Siti Khadijah, Deny Putra Romadhon, dan Fatimah). Penulis menyadari bahwa penelitian ini masih banyak kekurangan karena keterbatasan yang dimiliki. Oleh karena itu, penulis sangat mengharapkan saran yang membangun untuk memperlancar dan memperoleh hasil penelitian selanjutnya yang lebih baik.
Semoga karya ilmiah ini bermanfaat.
Bogor, September 2014
DAFTAR ISI
Analisis Kuantitatif dan Kualitatif DNA 4
METODE 5
Visualisasi Hasil Isolasi Dengan Elektroforesis 7
Penghitungan Ukuran Fragmen Restriksi 7
Pengukuran Konsentrasi dan Kemurnian DNA 8
HASIL DAN PEMBAHASAN 8
Visualisasi DNA Hasil Isolasi 8
Konsentrasi dan Kemurnian DNA Hasil Isolasi 10
DAFTAR TABEL
1 Taksonomi delapan jenis ikan laut 2
2 Kualitas visualisasi hasil isolasi sampel daging dan sirip ikan laut 9 3 Konsentrasi dan kemurnian DNA yang dikoleksi dari berbagai jenis
ikan laut 10
DAFTAR GAMBAR
1 Visualisasi DNA hasil isolasi sampel daging dan sirip ikan laut pada gel
agarosa 0.8% dan 1% 8
2 Visualisasi amplikon DNA sirip ikan selar setelah PCR dengan primer
cyt b dan setelah PCR dilanjutkan dengan pemotongan dengan enzim
Hinf I 11
DAFTAR LAMPIRAN
1 Analisis ragam jumlah rata-rata konsentrasi sampel DNA daging dan
sirip ikan laut 16
2 Analisis ragam jumlah rata-rata tingkat kemurnian sampel DNA dan
sirip ikan laut 17
PENDAHULUAN
Latar Belakang
Identifikasi spesies ikan sangat penting terutama untuk menjaga keberagaman jenis hewan, proses karantina, diagnostik klinik, dan kontrol sampel makanan. Identifikasi spesies ikan dapat dilakukan menggunakan morfologi luar seperti bentuk tubuh, pola dan warna, jumlah dan ukuran sisik, jumlah dan posisi sirip, serta jumlah dan tipe sirip renang. Namun dengan keberagaman morfologi yang ada akan menjadi sulit untuk mengidentifikasi dengan menggunakan perbedaan morfologi saja. Terdapat keterbatasan untuk identifikasi spesies bahkan dengan spesimen utuh, karena beberapa jenis ikan memiliki variasi atau perbedaan yang kecil antar spesies. Selain itu, apabila salah satu ciri khas morfologi hilang karena suatu proses (misal canning atau filetting), identifikasi akan semakin sulit dilakukan (Teletchea 2009).
Indonesia memiliki kekayaan jenis ikan yang beragam. Terbukti bahwa seperempat jenis ikan laut dan ikan air tawar dari seluruh dunia terdapat di Indonesia, namun informasi mengenai variasi genetik jenis ikan di Indonesia belum banyak dipelajari. Salah satu teknik untuk mengetahui data molekuler spesies hewan adalah dengan menggunakan metode polymerase chain reaction-restriction fragment length polymorphism (PCR-RFLP), karena menurut Minarovic et al. (2010) penggunaan PCR pada DNA mitokondria merupakan metode modern untuk mengidentifikasi spesies hewan.
Isolasi DNA merupakan tahap awal dari rangkaian berbagai teknologi analisis DNA. Proses pemecahan membran sel dan membran inti dibutuhkan untuk mengisolasi DNA kemudian dilanjutkan dengan pemisahan DNA dari berbagai komponen sel lain. Pada saat melakukannya, DNA harus dijaga agar tidak rusak dan dalam bentuk rantai panjang (Fatchiyah et al. 2011).
Isolasi DNA digunakan dalam berbagai macam aplikasi sains, medis, dan forensik. Isolat DNA asal hewan atau tumbuhan dapat dimanfaatkan untuk analisis single nucleotide polymorphism (SNP), analisis metilasi DNA, copy number variation (CNV) atau comparative genomic hybridization (CGH), PCR, dan lain-lain. Banyak penelitian telah dilakukan dengan berbagai metode isolasi DNA dengan kualitas dan kuantitas DNA yang berbeda-beda.
Berbagai macam sampel jaringan dapat digunakan untuk mengisolasi DNA dari ikan antara lain daging (Saputra 2009), sirip dan sisik (Wasko et al. 2003), mukus (Vin et al. 2011), serta darah (Turtinen dan Juran 1998). Metode pengambilan sampel untuk isolasi DNA terbagi menjadi metode invasif dan non invasif. Metode non invasif dalam pengambilan sampel DNA akan menghindari kerusakan komoditas. Jenis sampel yang termasuk non invasif pada ikan yaitu sisik, sirip, dan mukus. Pada mamalia dan unggas, sampel yang umum digunakan dengan metode non invasif yaitu folikel rambut, buccal swab, dan urin.
2
barcoding (Teletchea 2009), serta PCR–RFLP untuk identifikasi spesies (Wolf et al. 1999).
Tujuan Penelitian
Tujuan dari penelitian ini adalah membandingkan konsentrasi, tingkat kemurnian, visualisasi elektroforesis, dan penggunaan DNA hasil isolasi untuk PCR-RFLP dari delapan spesies ikan laut menggunakan sampel daging dan sirip.
Manfaat Penelitian
Teknik isolasi DNA ikan laut ini dapat digunakan untuk berbagai keperluan analisis molekuler seperti PCR-RFLP, sequencing, mikrosatelit, hibridisasi, dan lain-lain. Penelitian ini diharapkan dapat memberikan referensi mengenai jenis sampel yang tepat untuk isolasi DNA pada ikan laut sehingga diperoleh sampel DNA dengan konsentrasi dan kemurnian yang lebih tinggi.
TINJAUAN PUSTAKA
Ikan Laut
Lautan Indonesia merupakan wilayah marine mega biodiversity terbesar di dunia dengan kekayaan 8500 spesies ikan, 555 spesies rumput laut, dan 950 spesies biota terumbu karang (Noegroho 2013). Menurut kamus besar bahasa indonesia (KBBI 2008), ikan merupakan binatang bertulang belakang yang hidup dalam air, berdarah dingin, umumnya bernafas dengan insang, biasanya tubuhnya bersisik, bergerak dan menjaga keseimbangan badannya menggunakan sirip.
Ikan terbagi menjadi dua golongan berdasar habitatnya yaitu ikan laut dan ikan air tawar. Pada penelitian ini, jenis ikan yang akan diteliti adalah ikan air laut yang biasa dikonsumsi, terdiri dari baronang, kerapu, ekor kuning, kakap merah, kurisi, kue, bawal hitam, dan selar. Kedelapan jenis ikan tersebut termasuk dalam filum chordata, kelas actinopterygii, dan ordo perciformes dengan famili, genus, dan spesies dapat dilihat pada Tabel 1.
Tabel 1 Taksonomi delapan jenis ikan laut yang digunakan dalam penelitian
Jenis Famili Genus Spesies
Baronang Siganidae Siganus S. javus
Kerapu Serranidae Epinephelus Ephinephelus sp.
Ekor Kuning Caesionidae Caesionida Caesionidae sp
Kakap Merah Lutjanidae Lutjanus L. camphenatus
Kurisi Nemipteridae Nemipterus N. nematophorus
Kue Carangidae Caranx C. melampygus
Bawal Hitam Carangidae Parastromateus P. niger
Selar Carangidae Selaroides S. letolepis
3 Isolasi DNA
Isolasi DNA merupakan tahap pertama dari berbagai teknologi analisis DNA yang diperoleh dari kromosom inti maupun dari organel, yaitu mitokondria dan kloroplas. Langkah-langkah yang diperlukan terdiri dari pemecahan membran sel dan membran inti yang dilanjutkan dengan pemisahan DNA dari berbagai komponen sel lain. Isolat DNA harus dijaga agar tidak rusak dan didapatkan dalam bentuk rantai yang panjang (Fatchiyah et al. 2011). Hasil isolasi DNA dapat berupa DNA genom, yaitu keseluruhan DNA yang terdapat dalam suatu sel atau DNA fragmen, keduanya dapat digunakan untuk analisis molekuler lanjutan. Tujuan dari isolasi yaitu menghasilkan DNA yang berkualitas serta dapat diukur konsentrasi dan tingkat kemurniannya. DNA yang berkualitas memiliki kontaminan yang rendah dan dapat tervisualisasi pada saat elektroforesis (Putri 2010).
Sebagai bahan dasar untuk analisis lanjutan seperti PCR, RFLP, kloning atau sequencing, maka DNA yang digunakan harus bersih dari kontaminan (mempunyai kemurnian yang tinggi) dan memiliki berat molekul yang tinggi (rantai yang panjang). Selama proses ekstrasi DNA, beberapa kerusakan yang dapat terjadi antara lain fragmentasi selama proses isolasi, degradasi oleh enzim nuklease, kontaminasi oleh polisakarida, dan metabolit sekunder saat isolasi (Fatchiyah et al. 2011)
Terdapat beberapa jenis prosedur yang berbeda untuk isolasi DNA di antaranya metode presipitasi amonium asetat (Sambrook et al.1982), metode CTAB (cationic hexadecyl trimethyl ammonium bromide) (Rogers dan Bendich 1994), metode menggunakan thermal lysis (Buwono dan Rosidah 2010), dan metode isolasi dengan menggunakan berbagai jenis kit komersial.
PCR-RFLP
Polymerase chain reaction (PCR) merupakan teknik yang sensitif untuk mengamplifikasi segmen spesifik pada DNA dengan cepat. Teknik ini dapat membentuk milyaran salinan fragmen DNA spesifik atau gen untuk mendeteksi dan mengidentifikasi sekuens gen melalui tahapan uji selanjutnya. Setiap uji PCR membutuhkan DNA template, primer nukleotida, dan DNA polimerase. Enzim DNA polimerase penting karena menghubungkan nukleotida yang terpisah menjadi satu kesatuan untuk membentuk produk PCR.
Proses PCR terdiri dari tiga tahapan yang berulang. Pertama, larutan reaksi akan dipanaskan di atas titik didih dari dua untai DNA komplemen sehingga DNA target yang akan membuat untaian tersebut menjadi terpisah, proses ini disebut denaturasi. Kemudian suhu diturunkan sehingga primer yang spesifik akan berikatan dengan DNA target, proses ini disebut hibridisasi atau annealing. Tahap terakhir, suhu akan dinaikkan kembali, pada saat ini DNA polimerase dapat memanjangkan primer dengan cara menambahkan nukleotida pada untai DNA. Setiap pengulangan dari tiga tahapan ini akan menambah jumlah salinan molekul DNA menjadi dua kali lipat (Garibyan dan Avashia 2013).
4
spesifik dengan enzim endonuklease. Pemotongan ini akan menghasilkan fragmen yang lebih kecil dengan ukuran berbeda-beda tergantung pada letak titik pemotongan. Fragmen yang berbeda ini akan dipisahkan melalui elektroforesis agarosa (Teletchea 2009). Enzim endonuklease atau enzim restriksi merupakan kunci penting dalam pemotongan fragmen DNA. Enzim bakterial ini akan berikatan pada daerah spesifik pada DNA kemudian memotong rantai pada kedua untai. Setiap enzim restriksi memiliki target sekuens yang berbeda pada setiap spesies (Becker et al 2000) oleh karena itu, setiap spesies akan memiliki pola daerah pemotongan yang berbeda (Clark dan Pazdernik 2011). Apabila DNA dari organisme yang berbeda dipotong oleh enzim restriksi yang sama, maka dapat dihasilkan pola fragmen yang berbeda. Teknik ini juga dapat diaplikasikan pada DNA dari dua individu dari spesies yang sama. Apabila perbedaan sekuens jatuh pada daerah enzim restriksi yang dikenali, maka akan terlihat polimorfologi panjang fragmen restriksi (Clark dan Pazdernik 2011).
Menurut Fatchiyah dan Arumingtyas (2011), aplikasi teknik PCR-RFLP biasa digunakan untuk mendeteksi diversitas genetik, hubungan kekerabatan, sejarah domestikasi, asal dan evolusi suatu spesies, aliran gen (genetic drift) dan seleksi, pemetaan keseluruhan genom, pengamanan gen-gen target yang diekspresikan (tagging gene), isolasi gen-gen yang berguna dari spesies liar, serta mengonstruksi pustaka DNA. Pascoal et al. (2004) melaporkan bahwa metode PCR-RFLP adalah metode yang cepat dan mudah untuk dilakukan dalam menganalisis dan mengidentifikasi daging, baik dari daging yang matang maupun mentah yang mengandung satu atau lebih spesies yang berbeda.
Kelebihan dari PCR-RFLP di antaranya adalah akurasi yang tinggi dan mudah ditransfer antar laboratorium. Selain itu, RFLP dapat mendeteksi adanya heterozigositas meskipun tanpa informasi sekuens target. PCR-RFLP sering direkomendasikan untuk analisis filogenetik antar spesies yang berkerabat karena didasarkan homolog sekuens. Sedangkan kekurangan dari teknik ini adalah dibutuhkan DNA dengan kemurnian tinggi dalam jumlah banyak, beberapa spesies mempunyai level polimorfisme yang rendah sehingga sedikit lokus yang terdeteksi diperlukan pustaka probe yang sesuai, dibutuhkan waktu yang banyak dan biaya yang relatif besar. Selain itu, kerugian dari metode ini yaitu pemrosesan yang tidak lengkap dapat terjadi dan variasi yang spesifik dapat terhapus atau bahkan tercipta tambahan daerah pemotongan (Lockley dan Bardsley 2000).
Analisis Kuantitatif dan Kualitatif DNA
Analisis kuantitatif dilakukan untuk mengetahui kualitas hasil isolasi DNA meliputi konsentrasi dan tingkat kemurnian DNA genom. Analisis ini dapat dilakukan dengan menggunakan alat spektrofotometer. Analisis kualitatif DNA genom dapat dilakukan dengan elektroforesis pada gel agarosa.
5 kemurnian DNA berkorelasi dengan kualitasnya. DNA dikatakan berkualitas jika memiliki tingkat kemurnian yang tinggi, karena telah termurnikan dari kontaminan seperti RNA, protein, dan lipid. Tingkat kemurnian DNA diukur dengan membandingkan absorbansi asam nukleat pada panjang gelombang A260 nm dan protein pada A280 nm. Kontaminasi DNA dapat dilihat dengan semakin meningkatnya nilai absorbansi protein.
Analisis kualitatif DNA genom menggunakan elektroforesis pada gel agarosa merupakan metode standar yang digunakan untuk identifikasi, pemisahan, dan purifikasi fragmen DNA. Pewarna ethidium bromida digunakan sebagai alat identifikasi dan mengukur semikualitatif fragmen DNA yang terpisah di dalam gel. Ethidium bromida akan terikat di antara dua untai ganda DNA sehingga pita DNA dalam gel agarosa akan berpendar karena pewarna ini mengandung zat fluoresen. Ikatan DNA dengan ethidium bromida ini akan terekspos pada sinar UV tingkat medium, sekitar panjang gelombang () 300 nm (Fatchiyah 2011). Intensitas fluoresen dapat diukur dengan menggunakan DNA penanda standar sehingga kuantitas DNA dapat diperkirakan, misalnya antara 0.5020 µg/ml.
METODE
Tempat dan Waktu Penelitian
Penelitian ini dilaksanakan di Laboratorium Pendidikan dan Layanan Fakultas Kedokteran Hewan, Institut Pertanian Bogor dalam kurun waktu Januari–Juli 2014.
Bahan Penelitian
6
Alat Penelitian
Alat yang digunakan dalam penelitian ini adalah alat diseksi, timbangan analitik, pipet mikro (ukuran 1–10 μl, 10–100 μl, dan 100–1000 μl), tip mikro, tabung sampel, tabung mikro 1.5 ml, erlenmeyer, gelas piala, parafilm, vortex, waterbath, microwave, elektroforesis gel agarosa, sentrifus (Kokusan H1500DR®), spektrofotometer (NanoDrop 2000 Thermo Scientific®), mesin PCR (AB Gene Amp® PCR System 9700).
Prosedur Penelitian
Pengumpulan Sampel
Sampel delapan ikan laut konsumsi yaitu ikan baronang (Siganus javus), kerapu (Epinephelus sp), ekor kuning (Caesionidae sp), kakap merah (Lutjanus campechanus), kurisi (Nemipterus nematophorus), kue (Caranx melampygus), bawal hitam (Parastromateus niger), dan selar (Selaroides leptolepis) diperoleh dari pasar sekitar Bogor, kemudian sampel tersebut disimpan di dalam freezer di suhu -20 °C sampai proses selanjutnya.
Isolasi DNA
Metode isolasi yang digunakan mengacu pada Sambrook et al. (1982). Sebanyak 100 mg masing-masing sampel dihaluskan di dalam tabung mikro 1.5 ml menggunakan gunting. Sampel yang telah halus ditambah larutan lysis buffer
sebanyak 500 μl, kemudian diinkubasi pada suhu 55 °C selama 1 jam dan digoyang sekali-sekali. Kemudian 6 μl proteinase K (10 mg/ml) ditambahkan ke dalam larutan tersebut dan diinkubasi kembali di suhu 55 °C selama 3 jam. Sebanyak 3 μl (20 mg/ml) RNAse ditambahkan ke dalam tabung kemudian tabung dibolak-balik sebanyak 25 kali, lalu diinkubasi pada suhu 37 °C selama 1530 menit. Setelah itu tabung yang berisi larutan tersebut disimpan di dalam pecahan es selama 30 menit dan dilanjutkan dengan sentrifugasi pada kecepatan 9000 g selama 30 menit.
Supernatan yang diperoleh dimasukkan ke dalam tabung baru dan ditambahkan larutan amonium asetat 5 M sebanyak 500 μl, divortex selama 20 detik pada kecepatan maksimum, kemudian didiamkan selama 10 menit pada suhu ruang. Selanjutnya tabung disentrifugasi pada kecepatan 4300 g pada suhu 4 °C selama 15 menit, supernatan dipindahkan ke dalam tabung baru dan disentrifugasi kembali pada kecepatan 4300 g pada suhu 4 °C selama 15 menit. Supernatan hasil sentrifugasi dipindahkan ke dalam tabung baru yang sudah diisi isopropanol absolut sebanyak 600 μl, sampel dicampur dengan cara dibolak-balik, kemudian didiamkan selama 1 malam pada suhu ruang sampai terbentuk endapan Setelah itu dilakukan sentrifugasi pada kecepatan 4300 g pada suhu 4 °C selama 5 menit sehingga DNA akan terlihat sebagai endapan putih kecil di dasar tabung.
7 dikeringkan dengan cara didiamkan pada suhu ruang semalam atau diinkubasi pada suhu 75 °C selama 5 menit. MilliQ steril ditambahkan ke dalam tabung mikro yang berisi endapan DNA sebanyak 3050 μl lalu diinkubasi pada suhu 60 °C selama 3 menit. Tabung yang berisi DNA diberi label dan disegel menggunakan parafilm kemudian disimpan di dalam freezer (-20 °C).
Amplifikasi Fragmen DNA Mitokondria (mtDNA)
Proses amplifikasi fragmen mtDNA dilakukan dengan menggunakan metode PCR. Reaksi PCR menggunakan primer universal sitokrom b. Larutan reaksi terdiri dari 3 μl DNA template, primer sitokrom b1 (10 μM) dan primer
sitokrom b2 (10 μM) masing-masing 1 μl, Invitrogen® PCR Reagen Supermix 45 μl, sehingga diperoleh total volume dalam 1× reaksi PCR adalah 50 μl.
Tabung PCR yang sudah berisi larutan tersebut dimasukkan ke dalam mesin PCR yang sudah diprogram dengan siklus yang digunakan dimulai dengan 1 siklus denaturasi awal pada suhu 95 °C selama 5 menit, dilanjutkan 35 siklus yang terdiri dari denaturasi pada suhu 95 °C selama 45 detik, annealing primer pada suhu 55 °C selama 45 detik, extention pada suhu 72 °C selama 1 menit, dan 1 siklus extention akhir pada suhu 72 °C selama 3 menit.
Pemotongan Amplikon DNA
Proses pemotongan amplikon DNA sirip ikan selar dalam penelitian ini dilakukan menggunakan enzim Hinf I (10 U/μl). Reagen yang digunakan dalam pemotongan DNA hasil PCR dengan Hinf I secara berurutan adalah ddH2O sebanyak
12 μl, buffersebanyak 2.5 μl, enzim Hinf I sebanyak 0.5 μl, DNA hasil PCR sebanyak
10 μl. Kemudian DNA hasil PCR diinkubasi pada suhu 37 °C selama 2 jam.
Visualisasi Hasil Ektraksi dengan Elektroforesis
Hasil isolasi DNA dan PCR-RFLP dari sirip dan daging delapan spesies ikan air laut konsumsi divisualisasikan menggunakan gel elektroforesis yang kemudian diamati dengan ultraviolet (UV) transluminator. Proses visualisasi DNA menggunakan gel agarosa 0.8% dengan komposisi 0.4 gram agarosa dicampur dengan 50 ml TAE atau agarosa 1% dengan komposisi 0.5 gram agarosa dicampur dengan 50 ml TAE. Hasil PCR divisualisasi menggunakan elektroforesis gel agarosa 1%, sedangkan hasil pemotongan amplikon divisualisasi menggunakan elektroforesis gel agarosa 1.5% dengan komposisi 0.375 gram agarosa dicampur dengan 25 ml TAE. Larutan gel agarosa dipanaskan dalam microwave kemudian didinginkan di dalam air, lalu ditambahkan 2.5 μl ethidium bromida (5mg/ml), setelah itu dibuat sumur untuk memasukkan DNA dengan cara memasukkan sisir pembentuk pada cetakan. Setelah gel agarosa dituangkan ke dalam cetakan, gel agarosa didiamkan hingga menjadi padat. Masing-masing sampel DNA dimasukkan ke dalam sumur setelah ditambahkan
loading dye dengan perbandingan 1:1 yaitu 3 μl DNA dan 3 μl loading dye. Proses elektroforesis dilakukan dengan voltase 100 V selama 45 menit.
Penghitungan Ukuran Fragmen Restriksi
8
x untuk jarak migrasi DNA (cm). Langkah kedua migrasi DNA ladder diukur pada foto atau gambar kemudian diplot pada kertas Semi-Log sehingga terbentuk garis lurus atau garis miring. Langkah berikutnya adalah pengukuran migrasi DNA setiap fragmen restriksi dan diplot pada kertas Semi-Log sama seperti sebelumnya sehingga dapat diketahui berat molekul DNA setiap fragmen restriksi (Sambrook et al. 1982).
Pengukuran Konsentrasi dan Kemurnian DNA
Pengukuran konsentrasi dan kemurnian 16 sampel DNA yang telah diisolasi dilakukan di laboratorium Institut Pertanian Bogor Culture Collection (IPBCC) dengan menggunakan spektrofotometer. Sebanyak 1 μl dari masing-masing sampel DNA diletakkan di atas pedestal bagian bawah, kemudian ditutup menggunakan pedestal bagian atas. Kabel fiber optik akan berkontak dengan sampel dan pengukuran spektro dilakukan menggunakan software pada komputer. Pengukuran dilakukan pada panjang gelombang A260 dan A280 dengan pengulangan dilakukan sebanyak dua kali.
Data-data konsentrasi dan kemurnian dari sampel DNA yang diperoleh dianalisa menggunakan program statistical package for the social sciences (SPSS) v.22 dengan metode analysis of variance (ANOVA) satu arah.
HASIL DAN PEMBAHASAN
Visualisasi DNA Hasil Isolasi
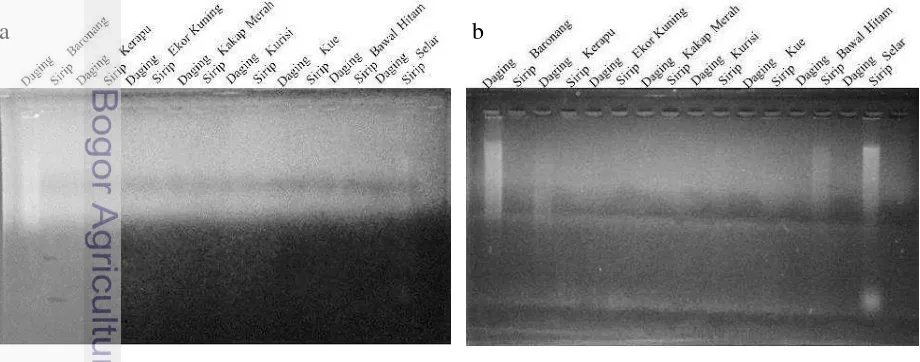
Hasil elektroforesis DNA hasil isolasi yang berasal dari delapan spesies ikan laut konsumsi menunjukkan hanya sampel DNA daging ikan baronang dan sirip selar yang terlihat jelas pada gel agarosa 0.8%. Setelah menggunakan gel agarosa 1%, maka sampel DNA daging ikan baronang, daging kerapu, sirip bawal hitam, dan sirip selar terlihat jelas pada hasil elektroforesis (Gambar 1, Tabel 2).
Gambar 1 Visualisasi DNA hasil isolasi sampel daging dan sirip ikan laut pada gel agarosa 0.8% (a) dan 1% (b)
9 Sampel DNA daging ikan kerapu dan sirip ikan bawal hitam tidak terlihat setelah dielektroforesis pada gel agarosa 0.8% tetapi terlihat pada gel agarosa 1%. Sementara itu jenis sampel daging dan sirip dari jenis ikan lain tidak terlihat baik pada gel agarosa 0.8% dan 1%. Hal ini dipengaruhi beberapa faktor di antaranya ukuran molekul DNA dan konsentrasi gel agarosa. Molekul yang lebih besar akan bergerak lebih lambat dibanding molekul yang lebih kecil dan DNA linier bergerak lebih cepat dibanding DNA sirkuler. Konsentrasi gel agarosa yang lebih rendah memiliki pori yang lebih renggang dibanding gel agarosa dengan konsentrasi tinggi memungkinkan molekul DNA akan lebih cepat bergerak melewati gel agarosa.
Menurut Fatchiyah (2010), laju migrasi elektroforesis DNA dipengaruhi beberapa faktor yaitu ukuran molekul DNA, konsentrasi gel agarosa, konformasi DNA, penggunaan voltase dan arah bidang elektris, komposisi basa DNA atau temperatur, keberadaan pewarna DNA, serta komposisi buffer elektroforesis.
Tabel 2 Kualitas visualisasi DNA hasil isolasi sampel daging dan sirip ikan laut
Jenis Ikan Jenis Sampel Hasil Elektroforesis
Gel agarosa 0.8% Gel agarosa 1%
- : Tidak terlihat, +: Terlihat cukup jelas, ++ : Terlihat jelas, +++ : Terlihat sangat jelas
Menurut Muladno (2002), isolasi DNA merupakan langkah penting dalam memurnikan DNA dimana DNA dipisahkan dari komponen seluler lain seperti protein, RNA, dan lipid. Terdapat banyak jenis prosedur yang berbeda untuk isolasi DNA, dimulai dengan melisiskan sel, diikuti deproteinasi dan terakhir memulihkan DNA. Perbedaan dari berbagai macam prosedur tersebut terletak pada proses deproteinasi dan hasil berat molekul DNA yang diperoleh.
10
yang diinginkan sesuai kemudahan pelaksanaan, ketersediaan sumber daya manusia, fasilitas, dan dana (Ardiana 2009).
Metode standar yang digunakan untuk memisahkan, mengidentifikasi, dan memurnikan fragmen DNA adalah elektroforesis gel agarosa. Teknik ini sederhana, cepat, dan mampu memisahkan campuran potongan DNA sesuai dengan ukurannya secara akurat. Pewarna yang mengandung fluoresen seperti ethidium bromida digunakan untuk mendeteksi potongan-potongan DNA berupa pita DNA pada gel agarosa (Fatchiyah 2010).
Jenis sampel yang pernah digunakan untuk isolasi DNA dari ikan antara lain daging (Saputra 2009), sirip dan sisik (Wasko et al. 2003), mukus (Vin et al.
2011), serta darah (Turtinen dan Juran 1998) dengan menggunakan metode yang berbeda-beda. Dalam penelitian ini, jenis sampel sirip lebih disukai karena bersifat non invasif dan dapat dilakukan ketika hewan masih hidup. Sedangkan jenis sampel daging bersifat invasif dan dapat merusak ikan sebagai komoditas makanan. Oleh karena itu, sampel sirip dapat digunakan sebagai alternatif untuk menggantikan sampel daging.
Konsentrasi dan Kemurnian DNA Hasil Isolasi
Hasil spektrofotometer untuk mengukur konsentrasi dan tingkat kemurnian menunjukkan sampel DNA yang berasal dari sirip ikan memiliki konsentrasi dan kemurnian yang tidak berbeda (P>0.05) dibandingkan dengan sampel DNA yang berasal dari daging ikan (Tabel 3).
Tabel 3 Konsentrasi dan kemurnian DNA yang dikoleksi dari berbagai jenis ikan laut
Rata-rata ±SD 22.01±20.29a 52.86±41.37a 1.54±0.23b 1.44±0.27b Superscript yang sama diperbandingkan dan menunjukan hasil yang tidak berbeda nyata (P>0.05) SD = simpangan baku
11 Pengukuran spektrofotometer biasa dilakukan dengan cara membandingkan nilai absorbansi A260 nm untuk DNA dan nilai absorbansi A280 nm untuk protein. Nilai perbandingan (rasio) A260/280 normal untuk DNA adalah 1.82.0. Jika nilai rasio lebih rendah dari 1.8 maka terdapat kontaminan protein atau fenol, tetapi jika nilai rasio lebih tinggi dari 2.0 maka terdapat kontaminan berupa RNA (Tataurov et al. 2008, Fatchiyah 2011).
Sebanyak tujuh dari delapan sampel daging dan sirip menghasilkan DNA dengan rasio A260/280 yang lebih rendah dari 1.8, mengindikasikan adanya kontaminasi dari protein atau fenol. DNA yang murni harus bebas dari protein dan oligopeptida. Kontaminasi oleh protein dapat berasal dari komponen sel yang tidak lisis selama proses isolasi atau berasal dari fenol sebagai bahan yang ditambahkan pada proses isolasi untuk mempresipitasi DNA (Bellard et al. 1973). Pada penelitian ini kontaminasi yang mungkin terjadi adalah kontaminan fenol sebagai akibat proses penghilangan fenol saat isolasi tidak sempurna. Pemurnian DNA yang terkontaminasi dapat dilakukan dengan menggunakan berbagai jenis
kit komersial.
PCR-RFLP DNA Hasil Isolasi
Setelah dilakukan proses PCR pada seluruh sampel DNA hasil isolasi, hanya diperoleh satu sampel DNA yaitu dari sirip ikan selar yang menunjukkan hasil positif pita pada 359 bp setelah dielektroforesis (Gambar 2a). Kocher dan White (1989) menyatakan bahwa penggunaan primer universal sitokrom b akan menghasilkan amplikon dengan panjang 359 bp. Kemudian amplikon dipotong dengan menggunakan enzim restriksi Hinf I dan terpotong menjadi dua fragmen (Gambar 2b). Setelah dilakukan pengukuran secara manual, maka diketahui bahwa panjang fragmen amplikon tersebut adalah 332 bp dan 27 bp (Lampiran 3).
Gambar 2 Visualisasi amplikon DNA sirip ikan selar setelah PCR dengan primer
cyt b (a) dan setelah PCR dilanjutkan dengan pemotongan dengan enzim Hinf I (b)
12
Tabel 3). Konsentrasi template DNA 1 ng merupakan konsentrasi minimal yang dapat digunakan dalam proses PCR (Grunewald 2003). Amplifikasi yang tidak berhasil disebabkan karena DNA tidak bebas dari kontaminan seperti protein atau bahan-bahan sisa proses isolasi seperti alkohol dan fenol dan ditunjukkan oleh rasio A260/280 di bawah 1.8 atau di atas 2.0.
Hasil amplifikasi PCR dapat dipotong dengan menggunakan enzim restriksi
Hinf I karena memiliki titik restriksi yang sesuai untuk enzim tersebut. Enzim restriksi tertentu akan memotong pada urutan pengenal dan tidak memotong daerah urutan lainnya. Enzim Hinf I dapat mengenali urutan pentanukleotida dengan urutan basa GANTC dan memotong pada titik di antara basa G dan A (Fatchiyah dan Aruminingtyas 2011). Selain pada penelitian ini, enzim Hinf I
telah dilaporkan sebelumnya dapat membedakan spesies ikan Scomber scombrus
dan Ruvettus pretiosus menggunakan primer universal cyt b (Nebola et al. 2010). Teknik PCR-RFLP memiliki banyak kegunaan dan aplikasi di bidang biologi dan keanekaragaman hayati, di antaranya ialah identifikasi spesies. Selain itu teknik ini lebih mudah dalam pelaksanaannya dibandingkan dengan teknik molekuler yang lain seperti sequencing, mikrosatelit, dan lain-lain. Berbagai jenis spesies ikan air tawar (Saputra 2009), satwa liar (Wolf 1999), dan hewan domestik (Bravi et al. 2004) telah berhasil diidentifikasi menggunakan teknik ini berdasarkan perbedaan potongan DNA setelah dipotong dengan enzim restriksi tertentu.
Uji DNA diperlukan di Indonesia untuk mengidentifikasi jenis spesies ikan baru (UNDIP 2013), identifikasi stok ikan (Syahailatua 2010), membedakan ikan dari kekerabatan yang dekat (species identification) (Ariyanti 2012), kasus pencurian (illegal fishing), serta keperluan perdagangan (terutama untuk ikan-ikan yang mahal). Hal ini menunjukkan bahwa teknik ini berguna untuk negara seperti Indonesia yang memiliki keanekaragaman hayati yang tinggi.
SIMPULAN DAN SARAN
SIMPULAN
Isolasi DNA delapan ikan laut menunjukkan konsentrasi dan kemurnian yang tidak berbeda antara sampel daging dan sampel sirip, yaitu 22.01±20.29 dan 1.44±0.27 untuk konsentrasi dan kemurnian DNA sampel daging, serta 52.86±41.37 dan 1.54±0.23 berturut-turut untuk konsentrasi dan kemurnian DNA sampel sirip. Sampel DNA ikan laut dapat diisolasi dari daging dan sirip namun hanya satu sampel dari sirip ikan selar yang memiliki konsentrasi yang tinggi (150.50 ng/μl) dan kemurnian yang baik (rasio A260/280=1.91) serta menunjukkan hasil positif setelah PCR-RFLP dengan enzim Hinf I dan menghasilkan dua pita dengan ukuran 332 bp dan 27 bp.
SARAN
13 untuk isolasi DNA dari ikan lebih disarankan karena bersifat non invasif dan menghasilkan nilai konsentrasi dan tingkat kemurnian yang tidak berbeda nyata dibanding sampel daging yang bersifat invasif. Konsentrasi dan tingkat kemurnian DNA perlu diperhatikan agar proses amplifikasi PCR dapat berlangsung secara optimal.
DAFTAR PUSTAKA
Ardiana DW. 2009. Teknik isolasi DNA genom tanaman pepaya dan jeruk dengan menggunakan modifikasi buffer CTAB. Bul Tek Pertan. 14 (1):12-16. Ariyanti Y. 2012. Aplikasi DNA barcode pada penentuan spesies ikan danau laut
tawar, Nanggroe Aceh Darussalam [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Becker WM, Kleinsmith LJ, Hardin J. 2000. The World of The Cell. Ed ke-4. New York (US): The Benjamin Publishing Company.
Bellard MG, Outdet P, Chambon P. 1973. Isolation of high molecular weight DNA from mammalian cells. Eur J Biochem. 36:32-38.
Bravi CM, Liron JP, Mirol PM, Ripoli MV, Garcia PP, Giovambattista G. 2004. A simple method for domestic animal identification in Argentina using PCR-RFLP analysis of cytochrome b gene. Leg Med. 6:246-251.
Buwono ID, Rosidah. 2010. Uji Sensitifitas Metode One Step dan Nested PCR terhadap Deteksi Penyakit KHV pada Ikan Mas (Cyprinus carpio). Lembaga Penelitian dan Pengembangan Masyarakat Universitas Padjajaran.
Clark DP, Pazdernik NJ. 2011. Biotechnology: Academic Cell. Massachusetts (US): Academic Press.
Fatchiyah. 2010. DNA qualitative and quantitative analysis [Internet]. [diacu 2014 Agustus 13]. Tersedia dari: http://fatchiyah.lecture.ub.ac.id/teaching-res ponsibility/general/dna-qualitative-quantitative-analysis/.
Fatchiyah, Rahayu S, Aruminingtyas EL. 2011. Isolasi DNA. Di dalam : Fatchiyah, Arumingtyas EL, Widyarti S, Rahayu S. Biologi Molekular-Prinsip Dasar Analisis. Jakarta (ID): Erlangga. hlm 21-32.
Fatchiyah. 2011. Uji Kuantitatif dan Kualitatif. Di dalam: Fatchiyah, Arumingtyas EL, Widyarti S, Rahayu S. Biologi Molekular-Prinsip Dasar Analisis. Jakarta (ID): Erlangga. hlm 34-37.
Fatchiyah, Aruminingtyas EL. 2011. Restriction Fragment Length Polymorphism (RFLP). Di dalam: Fatchiyah, Arumingtyas EL, Widyarti S, Rahayu S.
Biologi Molekular-Prinsip Dasar Analisis. Jakarta (ID): Erlangga. hlm 42-45.
Froese R, Pauly D. 2014. Fish base [Internet]. [diacu 2014 September 9]. Tersedia dari: http://www.fishbase.org.
Garibyan L, Avashia N. 2013. Polymerase chain reaction. J Invest dermatol. 133 (6):1-4.
14
[KBBI] Kamus Besar Bahasa Indonesia. 2008. Ikan [Internet]. [diacu 2014 September 6]. Tersedia dari: bahasa.kemdiknas.go.id/kbbi/index.php. Kocher TD, White TJ. 1989. Evolutionary analysis via PCR. Di dalam: Erlich H
A, editor. PCR Technology: Principles and Aplication for DNA amplification. New York (US): Stockton Press. hlm 137-147.
Lockley A, Bardsley R. 2000. Novel method for discrimination of tuna (Thunnus thynnus) and bonito (Sarda sarda) DNA. J Agric Food Chem. 48:4463-4468.
Minarovic T, Trakovicka A, Rafayova A, Lieskovska Z. 2010. Animal species identification by PCR-RFLP of cytochrome b. Anim Sci Biotechnol. 43 (1):296-299.
Muladno. 2002. Seputar Teknologi Rekayasa Genetika. Bogor (ID): Pustaka Wirausaha Muda.
Nebola M, Borilova G, Kasalova J. 2010. PCR-RFLP analysis of DNA for the differentiation of fish species in seafood samples. Bull Vet Inst Pulawy. 54: 49-53.
Noegroho A. 2013. Keanekaragaman hayati laut Indonesia terbesar di dunia [Internet]. [diacu 2014 Agustus 27]. Tersedia dari: http://kkp.go.id/ index.php/arsip/c/9822/keanekaragaman-hayati-laut-indonesia-terbesar-di-berbasis PCR-sequencing dan perbandingan metode ekstraksi DNA [skripsi]. Bogor (ID): Institut Pertanian Bogor. manual. Ed ke-1. New York (US): Cold Spring Harbor Laboratory.
Sambrook J, Fritsch FF, Maniastis T. 1989. Molecular Cloning. A laboratory manual. Ed ke-2. New York (US): Cold Spring Harbor Laboratory.
Saputra D. 2009. Gambaran restriction fragment length polymorphism (RFLP)
gen sitokrom b DNA mitokondria dari sembilan spesies ikan air tawar konsumsi [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Syahailatua A. 2010. Identifikasi stok ikan, prinsip dan kegunaannya. Oseana.
18(2):55-63.
Tataurov AV, You Y, Owczarzy R. 2008. Predicting ultraviolet spectrum of single stranded and double stranded deoxyribonucleic acids. Biophys Chem. 133 (1-3):66-70.
Teletchea F. 2009. Molecular dentification methods of fish species: reassesment and possible applications. Rev Fish Biol Fish. 19:265-293.
15 [UNDIP] Universitas Diponegoro (ID). 2013. Uji DNA untuk identifikasi ikan spesies baru dari Kaimana Papua [Internet]. [diacu 2014 Agustus 25]. Tersedia dari: http://msdp.undip.ac.id/s3/berita-143-uji-dna-untuk-identifi kasi-ikan-spesies-baru-dari-kaimana-papua.html.
Vin ALL, Adam A, Tedder A, Arnold KA, Mable BK. 2011. Validation of swabs as non destructive and relatively non-invasive DNA sampling method in fish. Mol Ecol Resour. 11 (1):107-109.
Wasko AP, Martins C, Oliveira C, Foresti F. 2003. Non-destructive genetic sampling in fish. An improved method for DNA isolation from fish fins and scales. Her. 138:161-165.
16
17 Lampiran 2 Analisis ragam jumlah rata-rata tingkat kemurnian sampel DNA
18
Lampiran 3 Perhitungan manual fragmen restriksi Hinf I sirip ikan selar
Keterangan : DNA Ladder : 100 bp : 3.1 cm
200 bp : 2.6 cm
300 bp : 2.4 cm
400 bp : 2.1 cm
Fragmen restriksi sirip selar : 2.3 cm (332 bp)
B
era
t m
olekul (
bp)
19 RIWAYAT HIDUP
Penulis dilahirkan di Sukabumi pada 13 Agustus 1993. Penulis merupakan anak pertama dari tiga bersaudara dari pasangan Yudhi Rhahadian dan Leni Nuraeni. Penulis bersekolah dari sekolah dasar (SD) sampai dengan sekolah menengah atas (SMA) di Kota Sukabumi. SD Negeri Brawijaya Kota Sukabumi ditempuh penulis selama enam tahun dan lulus pada tahun 2005. Jenjang pendidikan selanjutnya adalah SMP Negeri 1 Kota Sukabumi yang ditempuh selama dua tahun dan lulus pada tahun 2007. Setelah lulus SMP, penulis melanjutkan bersekolah di SMA Negeri 1 Kota Sukabumi dan lulus pada tahun 2010. Penulis diterima di IPB melalui jalur undangan seleksi masuk IPB (USMI) pada tahun 2010.