PENGARUH VARIASI TEMPERATUR UNFOLDING
TERHADAP TRAYEKTORI REFOLDING PROTEIN 1GB1
RIZKY AMELIA
DEPARTEMEN FISIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Pengaruh Variasi Temperatur Unfolding terhadap Trayektori Refolding Protein 1GB1 adalah benar karya saya dengan arahan dari dosen pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
RIZKY AMELIA. Pengaruh Variasi Temperatur Unfolding terhadap Trayektori Refolding Protein 1GB1. Dibimbing oleh TONY SUMARYADA dan SETYANTO TRI WAHYUDI.
Protein 1GB1 adalah jenis protein immunoglobulin dari Streptococcus kelompok G yang berfungsi membantu organisme menghindari pertahanan inang. Proses unfolding dan refolding protein penting untuk dipelajari. Penelitian ini bertujuan mempelajari dan membandingkan trayektori proses unfolding dan refolding protein 1GB1 akibat pengaruh suhu. Proses unfolding dilakukan pada suhu 500 K dan 525 K selama 2 ns. Koordinat dari unfolding dijadikan masukan awal simulasi refolding selama 20 ns pada suhu 200 K. Metode simulasi dinamika molekul dimulai dengan preparasi, minimisasi, pemanasan atau pendinginan, ekuilibrasi dan production run. Protein 1GB1 dapat terdenaturasi dengan cepat pada suhu yang lebih tinggi yaitu 525 K dalam waktu 162 ps. Protein native yang telah terdenaturasi dapat kembali ke struktur awalnya melalui proses refolding, menghasilkan bentuk konformasi dan kesesuaian urutan residu antara struktur protein native dengan hasil simulasi yang berbeda, untuk suhu 500 K memiliki kesesuaian sebesar 85.71 %, sedangkan untuk suhu 525 K sebesar 73.21 %. Proses unfolding dan refolding protein 1GB1 memiliki trayektoriyang berbeda. Kata kunci : simulasi dinamika molekul, protein 1GB1, refolding, unfolding
ABSTRACT
RIZKY AMELIA, Effect of Unfolding Temperature Variation on Refolding Trajectory of Protein 1GB1. Supervised by TONY SUMARYADA and SETYANTO TRI WAHYUDI.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Fisika
PENGARUH VARIASI TEMPERATUR UNFOLDING
TERHADAP TRAYEKTORI REFOLDING PROTEIN 1GB1
RIZKY AMELIA
DEPARTEMEN FISIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PRAKATA
Syukur Alhamdulillah penulis panjatkan ke Hadirat Allah subhanahu wa
ta’ala yang telah melimpahkan rahmat dan karunia-Nya, sehingga penulis dapat menyelesaikan karya ilmiah ini dengan judul “Pengaruh Variasi Temperatur Unfolding terhadap Trayektori Refolding Protein 1GB1”. Karya ilmiah ini disusun sebagai salah satu syarat kelulusan program sarjana di Departemen Fisika, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Ucapan terima kasih penulis sampaikan kepada :
1. Direktorat Jenderal Pendidikan Tinggi (DIKTI) Kementerian Pendidikan dan Kebudayaan Republik Indonesia melalui program beasiswa Bidikmisinya yang telah diamanahkan kepada penulis.
2. Kedua orang tua dan kakak-kakak yang senantiasa selalu memberikan kasih sayang, perhatian, dukungan dan do’a kepada penulis dalam semua pencapaian ini.
3. Bapak Dr Tony Ibnu Sumaryada selaku dosen pembimbing yang telah membantu dan memberikan saran serta motivasi kepada penulis selama perkuliahan di Departemen Fisika sampai penyusunan tugas akhir dan mencapai kelulusan.
4. Bapak Dr Setyanto Tri Wahyudi selaku dosen pembimbing kedua atas bimbingan dan saran yang diberikan kepada penulis selama penelitian. 5. Bapak Heriyanto Syafutra selaku dosen penguji yang telah memberikan
masukan dan saran kepada penulis.
6. Mochammad Alief Diego dan keluarga atas kasih sayang, perhatian, dukungan dan doa yang diberikan kepada penulis.
7. Seluruh dosen pengajar dan staf Departemen Fisika IPB atas bimbingan dan bantuan yang diberikan kepada penulis.
8. Uni Della dan Ka Nia yang telah membantu penulis dalam penelitian serta rekan-rekan mahasiswa/i fisika yang senantiasa memberikan do’a, motivasi dan saran.
Semoga karya ilmiah ini bermanfaat.
Bogor, Mei 2015
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 1
Manfaat Penelitian 2
Hipotesis 2
TINJAUAN PUSTAKA 2
Protein 1GB1 2
Proses Unfolding-Refolding Protein 3
METODE 4
Waktu dan Tempat 4
Alat 4
Prosedur Penelitian 4
HASIL DAN PEMBAHASAN 5
Struktur Sekunder Protein 5
Energi Konformasi 8
Jari-jari Girasi 9
RMSD 9
SASA 10
SIMPULAN DAN SARAN 11
Simpulan 11
Saran 11
DAFTAR PUSTAKA 12
LAMPIRAN 13
DAFTAR TABEL
1 Residu penyusun struktur sekunder protein 1GB1 2
DAFTAR GAMBAR
1 Struktur Sekunder Protein 1GB1 (a) unfolding 500 K selama 2 ns, (b) refolding 200 K selama 20 ns (dari simulasi unfolding pada suhu 500 K), (c) unfolding 525 K selama 2 ns dan (d) refolding 200 K selama 20 ns (dari simulasi unfolding pada suhu 525 K) 6 2 Perubahan konformasi struktur sekunder selama simulasi unfolding 500
K dan refolding 200 K 7
3 Perubahan konformasi struktur sekunder selama simulasi unfolding 525
K dan refolding 200 K 7
4 Energi konformasi selama simulasi (a) unfolding 500K dan refolding 200 K dari simulasi unfolding suhu 500 K, (b) unfolding 525 K dan refolding 200 K dari simulasi unfolding suhu 525 K 8 5 Jari- jari girasi selama simulasi (a) unfolding 500K dan refolding 200
K dari simulasi unfolding suhu 500 K, (b) unfolding 525 K dan refolding 200 K dari simulasi unfolding suhu 525 K 9 6 RMSD selama simulasi (a) unfolding 500K dan refolding 200 K dari
simulasi unfolding suhu 500 K, (b) unfolding 525 K dan refolding 200
K dari simulasi unfolding suhu 525 K 10
7 SASA selama simulasi (a) unfolding 500K dan refolding 200 K dari simulasi unfolding suhu 500 K, (b) unfolding 525 K dan refolding 200
K dari simulasi unfolding suhu 525 K 10
DAFTAR LAMPIRAN
1 Diagram alir penelitian 14
2 Persentase kesesuaian urutan residu penyusun antara struktur protein
PENDAHULUAN
LATAR BELAKANG
Protein adalah makromolekul yang tersusun dari bahan dasar asam amino yang menyusun protein sebanyak 20 macam. Protein terdapat dalam sistem hidup semua organisme pada tingkat rendah maupun tinggi.1 Protein G domain B1 (1GB1) adalah jenis protein immunoglobulin dari Streptococcus kelompok G yang berfungsi untuk membantu organisme menghindari pertahanan inang yaitu manusia.2 Protein 1GB1 dapat dipelajari sebagai model protein dalam menjelaskan proses folding (pelipatan) dan unfolding (pembukaan lipatan) karena strukturnya sederhana, termasuk protein kecil (memiliki 56 rantai samping) dan sifat kestabilan termalnya tinggi.3
Perkembangan teknologi mengenai metode penentuan protein terkait keberadaan dan keadaan protein mempunyai arti penting dalam kehiduan sehari-hari karena terkait dengan kondisi kesehatan manusia.4 Pengkajian bagaimana protein mengalami folding dan unfolding masih dikembangkan baik secara eksperimental maupun secara komputasional.5 Seiring dengan meningkatnya kemampuan komputer, proses simulasi dapat mendeskripsikan secara lebih terperinci mengenai dinamika protein.6
Metode dinamika molekul (MD) merupakan salah satu metode komputasi fisika yang popular untuk mensimulasikan gerak atom dan molekul. Dengan metode MD gerak atom-atom bahan jika mengalami pengaruh dari luar seperti akibat pemanasan, dapat diamati dari waktu ke waktu. Secara ringkas metode MD memerlukan informasi koordinat awal atom, kondisi simulasi (temperatur, tekanan, rapat partikel, dan lain-lain), fungsi potensial interaksi antar atom untuk obyek yang akan disimulasikan dan spesifikasi obyek yang disimulasikan (massa, muatan, jumlah atom, dan lain-lain). Pada dasarnya dinamika molekul memerlukan informasi yang akurat untuk fungsi potensial interaksi tersebut. Semakin akurat fungsi potensial yang menggambarkan interaksi antar partikel, atom dan molekul maka semakin akurat hasil simulasi yang kita dapatkan.7 Oleh karena itu penelitian ini mensimulasikan keseluruhan protein G doimain B1 di berbagai variasi suhu menggunakan program not just another molecular dynamics program (NAMD).
Perumusan Masalah
1. Bagaimana pengaruh suhu terhadap dinamika dan kestabilan protein 1GB1 selama proses unfolding dan refolding?
2. Apakah trayektori dari proses unfolding protein 1GB1 sama dengan proses refolding protein 1GB1?
Tujuan Penelitian
2
Manfaat Penelitian
Penelitian ini diharapkan dapat memberikan pemahaman terhadap mekanisme unfolding dan refolding protein 1GB1 akibat pengaruh suhu.
Hipotesis
Adanya perbedaan trayektoriproses unfolding protein 1GB1 pada suhu 500 K dan 525 K dengan trayektoriproses refolding protein 1GB1 pada suhu 200 K.
TINJAUAN PUSTAKA
Protein 1GB1
Protein 1GB1 adalah protein dari kelompok G Streptococcus dengan domain B1. Permukaan multidomain selnya besar dan berfungsi untuk membantu organisme menghindari pertahanan inang melalui sifat pengikatan protein. Protein 1GB1 terdiri dari 56 residu yang membentuk 2 pasang beta-hairpin, 4 coil, 2 turn dan 1 alpha-helix.2 Struktur protein terdiri dari struktur primer, sekunder, tersier, dan kuartener. Struktur sekunder merupakan struktur tiga dimensi dari rangkaian asam amino pada protein yang distabilkan oleh ikatan hidrogen. Struktur sekunder ini terdiri dari alpha helix, beta sheet, turn, dan coil.8
Tabel 1. Residu penyusun struktur sekunder protein 1GB13 No. Struktur Sekunder Residu Index Kode Residu
1 β
-sheet β-sheet 1 2 s/d 8 T, Y, K, L, I, L, N β-sheet 2 13 s/d 19 K, G, E, T, T, T, E β-sheet 3 43 s/d 46 E, W, T, Y, D β-sheet 4 51 s/d 55 T, F, T, V, T 2 α-helix 23 s/d 36 A, A, T, A, E, K, V, F, K, Q, Y, A, N, D, N
3 turn 9 s/d 12 G, K, T, L
47 s/d 50 D, A, T, K
4 coil
1 M
20 s/d 22 A, V, D 37 s/d 42 G, V, D, G
3 Proses Unfolding dan Refolding Protein
Sebagai fungsi biologisnya, protein akan berada pada keadaan folding (pelipatan). Pada penelitian terkait, proses simulasi diterapkan pada suhu tinggi yang bertujuan untuk mempercepat laju reaksi unfolding dalam rentang yang terjangkau karena jika dilakukan di suhu rendah maka akan membutuhkan waktu simulasi yang sangat lama. Proses unfolding dan refolding dapat dirangkum dalam 4 tahap yaitu :
F adalah frayed, keadaan folded atau keadaan native; H adalah keadaan tanpa struktur sekunder tapi inti masih terbungkus, S adalah keadaan inti sebagian tersolvasi; dan U adalah keadaan unfolded lengkap. Refolding merupakan kebalikan dari proses unfolding dimana penurunan suhu diterapkan pada protein dalam keadaan unfolded.9 Proses unfolding dan refolding protein dapat dijelaskan melalui beberapa parameter, diantaranya adalah :
1. Energi
Dalam simulasi, energi didefinisikan sebagai energi potensial molekul penyusunnya. Energi potensial dapat dipengaruhi energy akibat interaksi internal yang meliputi energi ikatan, sudut ikatan dan sudut dihedral dan interaksi eksternal yang meliputi interaksi kovalen dan ikatan. Jenis interaksi non-ikatan adalah interaksi elektrostatik (potential Coulomb) dan interaksi van der Waals (potential Lennard-Jones).10
2. Jari-jari Girasi
Jari-jari girasi adalah parameter yang menjelaskan konformasi seimbang dari sistem total dan kekompakan protein.10 Peningkatan nilai jari-jari girasi suatu simulasi menunjukkan volume struktur protein yang semakin membesar secara geometri. Volume yang meningkat menggambarkan semakin berkurangnya kerapatan dan kekompakan struktur protein.2
3. Root Mean Square Deviation (RMSD)
RMSD adalah jarak rata-rata antara konformasi dan struktur referensi dari dua buah atom.11 Besar kecilnya nilai RMSD bergantung pada atom yang digunakan serta dapat menjelaskan prosedur pelipatan protein dan mengetahui waktu saat perubahan konformasi.12
4. Solvent Accessible Surface Area (SASA)
4
METODE
Waktu dan Tempat Penelitian
Penelitian ini dilaksanakan pada bulan September 2014 sampai bulan April 2015. Tempat penelitian dilakukan di Laboratorium Fisika Teori dan Komputasi, Departemen Fisika, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Bahan
Bahan yang digunakan dalam simulasi dinamika molekuler teknik in silico ini berupa data koordinat hasil NMR protein G domain B1, yang dapat diunduh di Protein Data Bank (http://www.rscb.org/pdb/) dengan indeks 1GB1.
Alat
Alat yang digunakan berupa perangkat keras dan perangkat lunak. Perangkat keras terdiri atas komputer dengan spesifikasi RAM 8 GB, Quad Core Processor (Intel Corei7), Graphic Card NVIDIA Ge Force GTS 9400, dan sistem operasi LINUX Ubuntu versi 12.04. Perangkat lunak berupa NAMD (Not Just Another Molecular Dynamics Program) versi 2.9 untuk simulasi, VMD (Visual Molecular Dynamics Program) versi 1.9.1 untuk preparasi dan analisis, CatDCD versi 4.0 untuk pengolahan data, VBA Ms.Excel 2010 untuk smoothing grafik, Gneplot 4.6.4, dan Gimp versi 2.6 untuk konversi format gambar.
Prosedur Penelitian
Studi pustaka
Studi pustaka dilakukan agar dapat mempelajari konsep protein G domain B1 (1GB1), simulasi dinamika molekul, proses unfolding dan refolding dan analisis dinamika molekul.
Preparasi molekul
5 harus dinetralkan dengan menambahkan penetral sistem NaCl dengan konsentrasi 0.15 mol/L, tahap ini dilakukan agar simulasi yang dilakukan tidak terpengaruh oleh faktor lain kecuali suhu.
Simulasi dinamika molekul
Tahap simulasi dilakukan dengan menggunakan program NAMD. Simulasi yang dilakukan adalah unfolding dan refolding yang masing-masing simulasi di awali dengan proses minimisasi dan dilakukan pada temperatur transisi 500 K dan 525 K. Pada minimisasi, data masukannya adalah data yang dihasilkan dari proses preparasi. Minimisasi bertujuan untuk meminimalkan energi pada molekul agar berada pada keadaan stabil. Proses unfolding, diawali dengan proses pemanasan pada suhu awal 0 K sampai 500 K dan 525 K. Selanjutnya, sistem diekuilibrasi dengan protokol Langevin yang bertujuan agar suhu tetap berada di suhu akhir. Tahap terakhir adalah production run dengan menahan suhu pada 500 K sampai 500 K dan 525 K sampai 525 K selama 2 ns.
Proses refolding yaitu simulasi dinamika molekul yang berkebalikan dari proses unfolding, artinya pada refolding protein kembali melipat. Untuk proses refolding data koordinat awal yang digunakan adalah data koordinat protein saat unfolding. Data masukan awal refolding terlebih dahulu di preparasi dan minimisasi seperti proses unfolding. Selanjutnya dilakukan ekuilibrasi, pemanasan dan production run pada suhu dasar 200 K selama 20 ns.
Analisis Data
Data yang diperoleh dari tahap simulasi dinamika molekul masih mengandung air, kemudian air dihilangkan terlebih dahulu. Dengan menggunakan program VMD, data tersebut diolah berdasarkan parameter struktur sekunder, jari-jari girasi, RMSD dan SASA. Hasil pengolahan data menghasilkan grafik yang dibutuhkan untuk menganalisa kestabilan protein 1GB1 dan sebagai parameter pembanding antara trayektoridari proses unfolding dan refolding protein 1GB.
HASIL DAN PEMBAHASAN
Struktur Sekunder
6
(a) (b)
(b) (d)
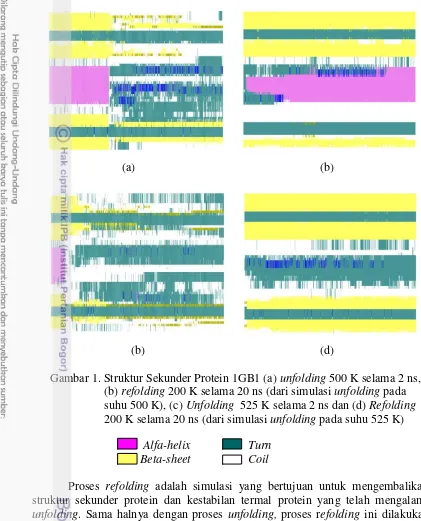
Gambar 1. Struktur Sekunder Protein 1GB1 (a) unfolding 500 K selama 2 ns, (b) refolding 200 K selama 20 ns (dari simulasi unfolding pada suhu 500 K), (c) Unfolding 525 K selama 2 ns dan (d) Refolding 200 K selama 20 ns (dari simulasi unfolding pada suhu 525 K)
Alfa-helix Turn Beta-sheet Coil
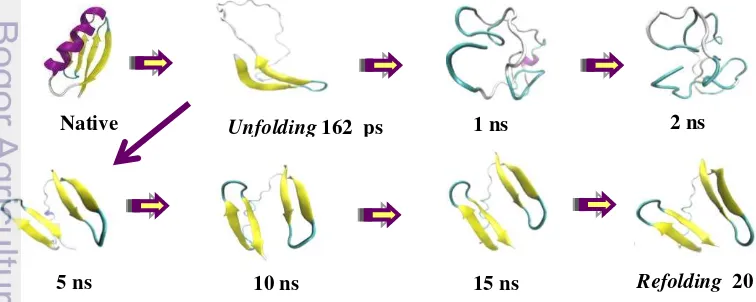
7 Pada gambar 2 dan 3 adalah inti dari proses simulasi unfolding dan refolding protein 1GB1, yang direpresentasikan melalui skema pembentukan struktur sekunder dari protein selama simulasi. Gambar 2 terlihat protein 1GB1 dalam keadaan native masih memiliki struktur lengkap yaitu 1 alfa-helix, 2 beta-sheet, 2 coil dan 2 turn. Protein native mengalami unfolding pada suhu 500 K di waktu 688 ps, terlihat perubahan struktur protein yaitu alfa-helix dan beta-sheet berubah menjadi coil dan turn. Kemudian pada koordinat ini dijadikan masukan awal proses refolding, terlihat pada simulasi 5 ns terbentuk kembali 1 alfa-helix yang belum sempurna dan 3 lembar beta-shteet. Selama 20 ns simulasi refolding, struktur protein yang telah unfolding menunjukan perubahan struktur mendekati protein native yaitu 1 alfa-helix sempurna, dan 3 lembar beta-sheet.
Gambar 2. Perubahan konformasi struktur sekunder selama simulasi unfolding 500 K dan refolding 200 K
Gambar 3 merepresentasikan struktur protein native mengalami unfolding pada waktu simulasi 162 ps, strukturnya mengalami perubahan yaitu alfa-helix yang telah hancur serta 2 lembar beta-sheet yang telah berubah menjadi coil dan turn. Sedangkan konformasi akhir hasil simulasi selama 2 ns sudah hancur secara keseluruhan. Dan koordinat akhir ini tidak digunakan sebagai masukan awal proses refolding karena akan sangat sulit dan membutuhkan waktu yang lebih lama agar struktur kembali ke keadaan native.
Gambar 3. Perubahan konformasi struktur sekunder selama simulasi unfolding 525 K dan refolding 200 K
Native Unfolding 688 ps 1 ns
5 ns 10 ns 15 ns Refolding 20 ns
2 ns
Native Unfolding 162 ps 1 ns
5 ns 10 ns 15 ns Refolding 20
8
Proses refolding pada suhu 525 K membutuhkan waktu yang lebih lama dalam proses pembentukan struktur sekunder dibandingkan simulasi pada suhu 500 K, karena dengan memberikan waktu simulasi yang sama yaitu 20 ns, urutan struktur yang terbentuk tidak sama. Terlihat hanya 4 lembar beta-sheet yang terbentuk selama 20 ns dan alfa-helix tidak terbentuk sama sekali (Gambar 3). Perubahan setiap 5 ns ini memperlihatkan proses penyempurnaan dari beta-sheet. Transisi temperatur yang diberikan, semakin memperjelas bahwa semakin tinggi suhu yang diberikan semakin cepat protein native mengalami unfolding. Karena pada simulasi suhu 525 K struktur sekunder lebih hancur, maka semakin lama waktu yang dibutuhkan oleh protein untuk refolding (native state).
Energi Konformasi
(a) (b)
Gambar 4. Energi konformasi selama simulasi (a) unfolding 500K dan refolding 200 K dari simulasi unfolding pada suhu 500 K, (b) unfolding 525 K
dan refolding 200 K dari simulasi unfolding pada suhu 525 K Energi konformasi dibutuhkan oleh protein untuk mengubah bentuk
konformasi. Artinya, semakin besar energi konformasi yang dibutuhkan maka perubahan konformasi yang terjadi pada protein juga semakin besar atau keadaan stabilitas protein menurun. Grafik pada Gambar 4(a) dan 4(b) telah diperhalus dengan moving average untuk setiap 50 frame. Gambar 4(a) menunjukkan bahwa energi konformasi yang dibutuhkan oleh protein untuk terdenaturasi (unfolding) pada 688 ps adalah 1343.70 kcal/mol. Sedangkan pada keadaan protein refolding, energi konformasi mengalami penurunan yang signifikan yaitu 1303.10 kcal/mol.
9 Jari – jari Girasi
(a) (b)
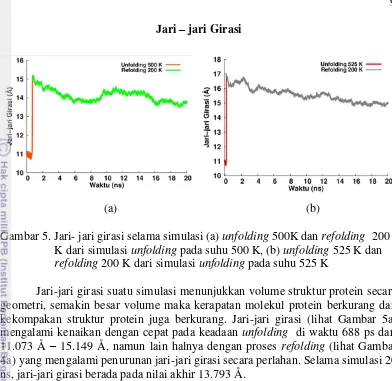
Gambar 5. Jari- jari girasi selama simulasi (a) unfolding 500K dan refolding 200 K dari simulasi unfolding pada suhu 500 K, (b) unfolding 525 K dan
refolding 200 K dari simulasi unfolding pada suhu 525 K Jari-jari girasi suatu simulasi menunjukkan volume struktur protein secara
geometri, semakin besar volume maka kerapatan molekul protein berkurang dan kekompakan struktur protein juga berkurang. Jari-jari girasi (lihat Gambar 5a) mengalami kenaikan dengan cepat pada keadaan unfolding di waktu 688 ps dari 11.073 Å – 15.149 Å, namun lain halnya dengan proses refolding (lihat Gambar 4a) yang mengalami penurunan jari-jari girasi secara perlahan. Selama simulasi 20 ns, jari-jari girasi berada pada nilai akhir 13.793 Å.
Gambar 5(b) merupakan perubahan jari-jari girasi pada simulasi dengan suhu yang lebih tinggi yaitu 525 K, terlihat jari-jari girasi mengalami peningkatan yang sangat cepat yaitu 11.087 Å – 16.993 Å pada waktu 162 ps. Hal ini menunjukan peningkatan volume struktur protein yang besar artinya kerapatan molekul dan kekompakan struktur protein pada simulasi dengan pemberian suhu yang lebih tinggi mengalami penurunan yang juga besar. Begitu juga dengan proses refolding mengalami penurunan yang sangat lambat sampai nilai akhir jari-jari girasi sebesar 15.076 Å. Dalam hal ini struktur protein pada simulasi unfolding dengan suhu yang lebih rendah memiliki kekompakan molekul protein yang lebih tinggi dibanding suhu yang lebih tinggi.
RMSD
10
(a) (b)
(b)
Gambar 6. RMSD selama simulasi (a) unfolding 500K dan refolding 200 K dari simulasi unfolding pada suhu 500 K, (b) unfolding 525 K dan
refolding 200 K dari simulasi unfolding pada suhu 525 K
Artinya protein telah mengalami unfolding parsial, dan pada keadaan ini koordinat protein dijadikan masukan awal untuk proses refolding. Refolding dilakukan terhadap struktur protein yang telah mengalami unfolding, dengan simulasi yang lebih lama yaitu 20 ns pada suhu 200 K, nilai RMSD mengalami penurunan yang signifikan yaitu 3.229 Å. Grafik pada Gambar (b) merepresentasikan bahwa protein ter-unfolding pada suhu 525 K dengan nilai RMSD 7.784 Å dan saat protein refolding nilai RMSD mengalami penurunan yang signifikan yaitu 1.870 Å artinya protein telah berada pada kedaan native namun belum sempurna. Perbedaan suhu yang diberikan, mempengaruhi proses simulasi baik lamanya waktu simulasi ataupun nilai RMSD yang diperoleh. Simulasi unfolding pada suhu 525 K lebih cepat dibandingkan pada suhu 500 K.
SASA
(a) (b)
Gambar 5. SASA selama simulasi (a) unfolding 500K dan refolding 200 K dari simulasi unfolding pada suhu 500 K, (b) unfolding 525 K dan
refolding 200 K dari simulasi unfolding pada suhu 525 K
11 Analisis SASA pada simulasi unfolding dan refolding protein digunakan untuk mengetahui seberapa besar luas area permukaan protein yang dapat diakses oleh molekul pelarut (dalam hal ini air). Pola grafik yang terbentuk hampir sama dengan pola grafik jari-jari girasi. Luas area permukaan protein yang diakses oleh air pada proses unfolding pada suhu 500 K (lihat Gambar a) adalah 6734.324 Å dan hal ini menunjukan seberapa besar inti hidropobik dari molekul protein yang telah dirusak. Namun lain halnya dengan proses refolding yang merepresentasikan luas area permukaan protein yang diakses oleh air mengalami penurunan yaitu 6298.987 Å selama 20 ns.
Proses refolding ini bertujuan untuk mengembalikan inti hidropobik molekul protein yang telah dirusak secara perlahan. Begitu juga dengan simulasi pada suhu 525 K, yang mengalami peningkatan nilai SASA secara signifikan yaitu 6986.511 Å, dalam hal ini terlihat bahwa peningkatan suhu menyebabkan luas area permuakaan protein yang diakses oleh air semakin besar, sehingga inti hidropobik molekul protein juga mengalami kerusakan yang lebih besar dibanding simulasi pada suhu 500 K. Grafik pada Gambar b menunjukkan bahwa luas area permuakaan protein yang diakses air mengalami penurunan hingga 6414.389 Å.
SIMPULAN DAN SARAN
Simpulan
Telah dilakukan simulasi unfolding pada suhu 500 K dan 525 K, serta simulasi refolding pada suhu 200 K. Diperoleh bahwa hasil simulasi dengan suhu yang lebih tinggi akan mempercepat proes unfolding protein. Hasil simulasi refolding dengan struktur awal dari simulasi unfolding pada suhu 500 K menghasilkan bentuk struktur akhir yang lebih mendekati struktur asli (native state). Sedangkan simulasi refolding dari struktur hasil simulasi unfolding pada suhu 525 K, menghasilkan struktur akhir yang masih jauh dari struktur asli protein 1GB1, ditandai dengan tidak terbentuknya struktur alfa-helix.
Saran
12
DAFTAR PUSTAKA
1. Katili, AS. Struktur dan fungsi protein kolagen. Jurnal Pelangi Ilmu. Vol. 02, No. 5, Mei 2009.
2. Gronenborn AM, Clore GM. Structural studies of immunoglobulin binding domains of Streptococcal protein G. Immunomethods. Maryland: Academic Press Inc. 1993.
3. Sawitri, Kania. Pengaruh mutasi terhadap kestabilan termal protein 1gb1 [tesis]. Bogor : Institut Pertanian Bogor. 2014
4. Sari, Mayang. Identifikasi protein menggunakan fourier transform infrared. [skripsi]. Depok : Universitas Indonesia. 2011.
5. Shen T, Cao Y, Zuang S, Li H .Engineered Bi-Histidine metal chelation sites map the structure of the mechanical unfolding transition state of an elastomeric protein domain GB1. Biophysical Journal. 2012; 103: 807-816. 6. Granata D, Camilloni C, Vendruscolo M, Laio A. Characterization of the
free-energy landscapes of proteins by NMR-guided metadynamics. PNAS. 2013; 110(17): 66817-6822.
7. Widiasih, et.al. Penerapan Metode Dinamika Molekul untuk Pembelajaran : Konsep Tittk Leleh dan Perubahan Wujud. Jurnal Teori dan Aplikasi Fisika. 2013; Vol. 01, No. 02.
8. Hati, Jellyta. Analisis kestabilan protein 1GB1 menggunakan simulasi dinamika molekul. [skripsi]. Bogor : Institut Pertanian Bogor. 2014.
9. Pande, V. S., Rokhsar, D. S. 1999. Molecular dynamics simulations of unfolding and refolding of beta-hairpin fragment of protein G. Biophysics Journal. Vol. 96, August 1999, pp. 9062-9067. USA: Proc. Natl. Acad. Sci. 10.Randy, Ahmad. Desain peningkatan termostabilitas lipase b candida antartica
dengan rekayasa penambahan ikatan disulfida pada enzim. [tesis]. Depok: Universitas Indonesia.
11.Micaelo, Nuno. Analysis of molecular simulation experiment [bahan presentasi] . Portugal: Universidade do Minho. 2010.
12.Aksimentiev, Alek, et.al. 2013. Using VMD. www.ks.uiuc.edu. (diakses 18 Oktober 2014).
13.JA, Marsh, SA Teichmann. Relative solvent accessible surface area predicts protein conformational changes upon binding. 2010. UK: MRC Laboratory of Molecular Biology, Hills Road, Cambridge.
14.Sharma RD, Lynn AM, Sharma PK, Rajnee, Jawaid S. High temperature unfolding of Bacillus anthracis amidase-03 by molecular dynamics simulations.
13 Lampiran 1. Diagram alir penelitian
Mulai
Persiapan Alat dan Bahan
Refolding
Pendinginan
Ekuilibrasi
Production run Koordinat unfolded 1GB1
Minimisasi Preparasi molekul
protein
Analisis Data
Selesai
Output : Data koordinat 1GB1
hasil refolding
Penyusunan laporan Unfolding
Pemanasan
Ekuilibrasi
Production run Minimisasi Koordinat 1GB1.pdb
Preparasi molekul protein
Output : Data koordinat 1GB1
14
Lampiran 2. Persentase kesesuaian urutan residu penyusun antara struktur protein native denganhasilsimulasi refolding
15 RIWAYAT HIDUP