KESTABILAN TERMAL ASAM AMINO ENZIM LIPASE
BACILLUS SUBTILIS
MENGGUNAKAN
SIMULASI DINAMIKA MOLEKUL
NYA DANIATY MALAU
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Kestabilan Termal Asam Amino Enzim Lipase Bacillus subtilis Menggunakan Simulasi Dinamika Molekul adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
NYA DANIATY MALAU. Kestabilan Termal Asam Amino Enzim Lipase Bacillus subtilis Menggunakan Simulasi Dinamika Molekul. Dibimbing oleh TONY IBNU SUMARYADA dan LAKSMI AMBARSARI.
Lipase A Bacillus subtilis (BsL) secara ekstensif dipelajari dalam produksi biodiesel, deterjen, surfaktan, pangan, produk farmasi, kosmetik, industri kertas, dan nutrisi. Salah satu kekurangan penggunaan BsL adalah suhu optimumnya yang relatif rendah pada 37 oC (310 K). Tujuan penelitian ini adalah untuk merancang mutan BsL termostabil dibanding BsL wild type melalui rekayasa mutasi residu pengunci kestabilan BsL. Simulasi dinamika molekul dilakukan dengan mengamati proses unfolding atau denaturasi termal BsL yang dipercepat dengan melakukan simulasi pada suhu tinggi. Simulasi dinamika molekul BsL dilakukan menggunakan perangkat lunak NAMD (Not Just Another Molecular Dynamic) pada suhu 400-500 K. Pemilihan residu yang dimutasi, didasarkan pada hasil analisis Interaksi Elektrostatis, Ikatan disulfida dan Interaksi Hidrofobik. Berdasarkan hasil analisis tersebut dirancang tiga mutan enzim BsL yaitu Mutan-1 (Iso151Val), Mutan-2 (Glu65Asp), dan Mutan-3 (Ile135Cys/Hsd145Cys). Parameter yang digunakan untuk membandingkan termostabilitas enzim mutan dengan wild type adalah RMSD (Root Mean Square Deviation), SASA (Solvent Accessible Surface Area), Rg (Radius of Gyration), RMSF (Root Mean Square Fluctuation) dan Struktur Sekunder. Simulasi dinamika molekul yang dilakukan pada ketiga mutan tersebut menunjukkan bahwa Mutan-1 (Iso151Val) dengan pemberian temperatur 525 K selama 20 ns (nano second) memiliki termostabilitas yang lebih baik dibanding wild type. Sehingga dapat disimpulkan bahwa interaksi hidrofobik penting dalam peningkatan stabilitas termal enzim lipase Bacillus subtilis. Pengaruh mutasi dari hasil analisa Interaksi hidrofobik pada peningkatan kestabilan termal untuk mutan-1 (Iso151Val) dengan pemberian temperatur 500 K mampu meningkatkan stabilitas termal yang dibuktikan oleh penurunan nilai parameter RMSD, Rg, RMSF, SASA dan Struktur Sekunder yaitu RMSD mampu diturunkan sebesar 7.7 Å, nilai Rg mampu diturunkan sebesar 3.41 Å dan nilai SASA mampu diturunkan sebesar 1504 Å2. Untuk struktur
beta-sheet, mampu mempertahankan sekitar 16% dibandingkan wild-type yang sama sekali tidak memiliki struktur beta-sheet. Untuk struktur alpa-helix, mutan-1 tidak begitu mempengaruhi komposisi alpa-helix nya. Diharapkan hasil penelitian ini dapat memberikan saran rancangan mutasi yang dapat diimplementasikan pada laboratorium basah (wet experiment).
SUMMARY
NYA DANIATY MALAU. Termal Stability of the Amino Acid Enzyme Lipase Bacillus subtilis Using Molecular Dynamics Simulation. Supervised by TONY IBNU SUMARYADA and LAKSMI AMBARSARI.
Bacillus subtilis lipase A (BsL) is extensively studied in enzymatic production of biodiesel, production of detergents, surfactants, food, pharmaceutical products, cosmetics, paper industry and nutrition. One drawback of using BsL is its relatively low optimum temperature at 37 oC (310 K). The objective of this research is to design BsL mutant more thermostable than the wild-type BsL by engineering mutations in residue stabilizer BsL. Molecular dynamic simulation was conducted to get better insight on the process of thermal denaturation or unfolding in BsL. Thermal denaturation of BsL was accelerated by conducting simulation at high temperature. Molecular dynamic simulation of BsL was performed with NAMD software package at 400-500 K. Selection of mutated residues was based on electrostatic interaction, disulfida bond, hidrofobic interaction analysis of BsL. From those analyses, three mutants were designed, which are Mutant-1 (Iso155Val), Mutant-2 (Ile135Cys/Hsd156Cys), and Mutant-3 (Glu65Asp). Parameters that were used to compare the thermostability of mutant with wild type enzyme were RMSD (Root Mean Square Deviation), SASA (Solvent Accessible Surface Area), Rg (Radius of Gyration), RMSF (Root Mean Square Fluctuation) and Secondary Structure. Molecular dynamics simulations were performed on all three mutants showed that the mutant-1 (Iso155Val) by giving the temperature of 525 K for 20 ns has a better thermostability compared to the wild type BSL. It concluded that hydrophobic interaction is important in improving the thermal stability of the enzyme lipase B. subtilis. Effect of mutation of hydrophobic interaction analysis results in an increase in the thermal stability of mutant-1 (Iso151Val) by giving a temperature of 500 K for 50 ns can improve thermal stability as evidenced by a decrease in the value of the parameter RMSD, Rg, RMSF, SASA and Secondary Structure is RMSD able lowered by 7.7 Å, the value of Rg able lowered by 3.41 Å and value SASA able lowered by 1504 Å2. For the beta-sheet structure, is able to retain about 16% compared to wild-type who did not have the beta-sheet structure. For the alpha-helix structure, mutan-1 did not influence alpa-helix composition. The resulted mutant design will be used as a suggestion to engineer BsL mutant in wet experiment.
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Biofisika
KESTABILAN TERMAL ASAM AMINO ENZIM LIPASE
BACILLUS SUBTILIS
MENGGUNAKAN
SIMULASI DINAMIKA MOLEKUL
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
Judul Tesis : Kestabilan Termal Asam Amino Enzim Lipase Bacillus subtilis Menggunakan Simulasi Dinamika Molekul
Nama : Nya Daniaty Malau
NIM : G751130031
Disetujui oleh Komisi Pembimbing
Dr. Tony Ibnu Sumaryada
Ketua Dr. Laksmi Ambarsari Anggota
Diketahui oleh
Ketua Program Studi Biofisika Dekan Sekolah Pascasarjana IPB
Dr Mersi Kurniati, M.Si Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah Yang Maha Kuasa atas segala kasih dan karunia-Nya yang tidak berkesudahan sehingga karya ilmiah yang berjudul Kestabilan Termal Asam Amino Enzim Lipase Bacillus subtilis Menggunakan Simulasi Dinamika Molekul dapat diselesaikan.
Terima kasih penulis ucapkan kepada Bapak Dr. Tony Ibnu Sumaryada dan Ibu Dr. Laksmi Ambarsari selaku pembimbing. Dr. Setyanto Tri Wahyudi selaku penguji yang telah banyak memberi saran dan masukan. Disamping itu, penghargaan penulis sampaikan kepada DIKTI selaku pemberi beasiswa BPPDN yang telah membantu pendanaan selama kuliah dan penelitian di Biofisika IPB. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, seluruh keluarga serta teman-teman, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 3
Manfaat Penelitian 3
Ruang Lingkup Penelitian 3
2 TINJAUAN PUSTAKA 3
Lipase A Bacillus subtilis Ekstraseluler (BsL) 3
Stabilitas Termal Protein 4
Simulasi Dinamika Molekul 5
3 METODE 6
Bahan 6
Alat 6
Prosedur Analisis Data 6
4 HASIL DAN PEMBAHASAN 10
Validasi Metode Simulasi Dinamika Molekul 10
Analisis Kestabilan Termal Wild-type Bacillus subtilis 11
Pemilihan Mutan Protein Bacillus subtilis 15
Analisis Kestabilan Termal Mutan Bacillus subtilis 22
5 SIMPULAN DAN SARAN 27
Simpulan 27
Saran 27
DAFTAR PUSTAKA 28
LAMPIRAN 32
DAFTAR TABEL
1 Nilai parameter enzim pada 50 ns 12
2 Perubahan komposisi struktur sekunder pada temperatur 400 K, 450 K,
475 K dan 500 K masing-masing pada 50 ns 13
3 Pemilihan Mutan dan Nilai ∆∆Gsolv 19
4 Nilai akhir parameter enzim pada suhu 500 K saat 50 ns 25 5 Perubahan komposisi struktur sekunder pada temperatur 500 K saat 50
ns 26
DAFTAR GAMBAR
1 Representasi struktur sekunder enzim lipase Bacillus subtilis serta letak
daerah sisi aktifnya. 4
2 Perbandingan nilai faktor-β kristal 1ISP (Biru) dengan faktor-β simulasi
(Orange) pada temperatur 300 K 11
3 Perubahan analisis parameter enzim pada temperatur 400 K, 450 K, 475 K dan 500 K masing-masing selama 50 ns (a). RMSD (b). Rg (c).SASA
(d). RMSF 12
4 (a) Persentase perubahan komposisi struktur sekunder selama simulasi
di 500 K selama 50 ns ; α-helix (Ungu), β-sheet (Kuning), Turn (Biru) dan Coil (Hitam) (b) Perubahan struktur pada simulasi 500 K selama 50 ns ; α-helix(Ungu), β-sheet (Kuning) 3-10helix (Biru), Turn (Hijau) dan
Coil (Putih) 14
5 (a) Jarak ikatan interaksi elektrostatistik antara Asp64 – Lys35 (Merah) dan Glu65 – Lys35 (Hitam) selama proses simulasi pada 500 K (b) Gaya elektrostatik antara Asp64 – Lys35 (Merah) dan Glu65 – Lys35
(Hitam) selama proses simulasi pada 500 K 15
6 Target mutasi pada enzim wild type dan mutan-mutan yang dihasilkan 16 7 Perubahan energi bebas solvasi mutasi (a). Asp64Asn (Biru), Asp64Glu
(Hitam), Glu65Gln (Hijau), Glu65Asp (Merah) (b). Ala146Cys (Ungu), Glu155Cys (Biru), Gly158Cys (Merah), Hsd156Cys (Hijau), Ile135Cys (Hitam) (c). Ile73Gln (Biru Muda), Ile75Leu (Biru), Ile151Ser (Hijau), Ile151Val (Merah), Val59Ile (ungu), Val59Thr (Hitam) 18 8 Posisi calon mutan berdasarkan interaksi elektrostatik (a) Glu64
(Hitam)-Lys35 (Merah) (b) Asp65 (Hitam)-Lys35(Merah) 19 9 Posisi calon mutan berdasarkan ikatan disulfida (a) Ile135
(Hjau)-Hsd146 (Orange) (b) Ile145 (Hijau)-(Hjau)-Hsd146(Orange) (c) Gly155
(Hijau)-Gly158 (Orange) 20
10 Posisi calon mutan berdasarkan interaksi hidrofobik : Val59 (Pink) ,
Ile73 (Abu-Abu) dan Ile 151 (Hijau) 19
11 Perubahan analisis parameter enzim pada mutan-1, mutan-2, mutan-3 dan wild-type dengan pemberian temperatur 525 K selama 20 ns
(a). RMSD (b). Rg (c). SASA (d). RMSF 22
12 Perubahan komposisi struktur sekunder pada 1, 2, mutan-3 dan wild-type dengan pemberian temperatur 525 K selama 20 ns (a).
13 Representasi struktur sekunder lipase Bacillus subtilis (a) Native (b)
Mutan-1 23
14 Perbandingan nilai faktor-β Mutan-1 (Biru), faktor-β wild-type (Orange) dan faktor-β eksperimen pada temperatur 300 K 24 15 Perubahan analisis parameter enzim pada mutan-1 dan wild-type
dengan pemberian temperatur 500 K selama 50 ns (a). RMSD (b). Rg
(c). SASA (d). RMSF 25
16 Perubahan komposisi struktur sekunder pada temperatur 500 K selama
50 ns. 26
17 Representasi struktur sekunder enzim lipase Bacillus subtilis setelah simulasi selama 50 ns pada temperatur 500 K (a) Wild-type (b) Mutan-1
DAFTAR LAMPIRAN
1 Residu penyusun struktur sekunder enzim lipase 1ISP 32
2 Diagram alir penelitian 33
3 Tabel pasangan jembatan garam yang muncul selama simulasi 50 ns
pada temperatur 500 K 34
4 Prediksi mutasi hasil keluaran perangkat lunak “Disulfide by DesignTM” 34 5 Pasangan mutasi hasil keluaran perangkat lunak “Disulfide by DesignTM”
yang berada pada β–sheet maupun α-helix 35
1
PENDAHULUAN
Latar Belakang
Enzim adalah biokatalisator yang dapat mengurangi dampak pencemaran dan pemborosan energi karena reaksinya tidak membutuhkan energi tinggi, bersifat spesifik, dan tidak beracun (Aunstrup et al. 1979). Lipase (Acylhydrolases triasilgliserol, E.C.3.1.1.3) merupakan enzim yang menghidrolisis trigliserida dan mengubahnya menjadi gliserol dan asam lemak (Prasad 2015). Lipase banyak diaplikasikan pada pembuatan obat-obatan, industri pengolahan makanan, deterjen dan pengolahan air limbah (Corzo dan Revah 1999). Mikroorganisme penghasil lipase yaitu bakteri (Kulkarni dan Gadre 2002), jamur (Fodiloglu dan Erkmen 1999), ragi (Corzo dan Revah 1999) dan actinomycetes (Sommer et al. 1997).
Beberapa lipase yang dihasilkan oleh bakteri spesies bacillus seperti B. subtilis, B. pumilis, B. licheniformis, B. thermoleovorans, B. stearothermophilus, dan B. Sphaericuspossess, banyak dimanfaatkan untuk bidang bioteknologi (Nthangeni et al. 2001; Rahman et al. 2003; Ruiz et al. 2003). Enzim lipase yang dihasilkan dua jenis bakteri mesofilik B. subtilis dan B. pumilus berbeda dari lipase Bacillus lainnya. Kedua lipase ini memiliki massa molekul yang relatif kecil yaitu sebesar 19.6 kDa sedangkan B. themocatenulatus, B. thermoleovorans, dan B. stearothermophilus merupakan lipase yang relatif besar dengan massa molekul sekitar 43 kDa (Shah dan Bhatt 2011).
Lipase B. subtilis adalah lipase yang tepat untuk mempelajari struktur dan fungsi enzim karena ukurannya yang kecil (~19.6 kDa) dan spesifisitas substratnya yang luas (Dartois et al. 1992; Eggert et al. 2000). Lipase B. subtilis menghasilkan dua jenis lipase yaitu Lipase A (LipA) dan Lipase B (LipB) yang dihasilkan pada medium kultur dan bergantung pada komposisi media pertumbuhan. LipA adalah lipase yang diproduksi dalam medium yang kaya komposisi media pertumbuhan dan pada medium yang minimal komposisi media pertumbuhan, sedangkan LipB hanya diproduksi dalam medium yang kaya komposisi media pertumbuhan (Eggert et al. 2001).
Lipase A B. subtilis (BsL) adalah lipase yang berasal dari bakteri mesofilik B. subtilis yang dihasilkan secara ekstraseluler dan bekerja pada temperatur optimum yaitu 370C, sehingga memiliki kestabilan termal yang rendah (Gertie et al. 2001). Beberapa aplikasi lipase dalam bidang industri memerlukan lipase yang termostabil. Hal tersebut diperlukan karena penggunaan suhu tinggi dalam proses industri akan meningkatkan kecepatan reaksi sehingga mengurangi jumlah enzim yang dibutuhkan. Kemungkinan adanya kontaminasi mikrobial oleh mikroba mesofilik yang mengganggu dapat dikurangi, kelarutan substrat dan senyawa - senyawa kimia lainnya meningkat (Haki dan Rakshit 2003). Sehingga dilakukan berbagai teknik untuk meningkatkan stabilitas termal enzim tersebut. Salah satu cara yang dapat dilakukan untuk meningkatkan termostabilitas suatu enzim adalah dengan teknik rekayasa protein.
2
proses terjadinya unfolding (Pikkemaat et al. 2002; Han et al. 2009; Liu et al. 2003; Kim et al. 2010). Sejauh ini hasil penelitian dengan berbagai metode baik eksperimen, statistik maupun komputasi telah menemukan bahwa sifat stabilitas termal dipengaruhi oleh adanya interaksi hidrofobik, ikatan disulfida, ikatan hidrogen, interaksi elektrostatik dan lain-lain (Jaenicke dan Zavodszky 1990; Jaenicke dan Bohm 1998; kumar et al. 2000b; Daniel dan Cowan 2000; Das dan Gerstein 2000; Gianese et al. 2000).
Dari hasil analisis sifat stabilitas termal tersebut maka dapat ditentukan residu-residu yang memiliki pengaruh yang signifikan terhadap kestabilan enzim. Mencari residu yang menjadi pengunci kestabilan enzim dan menggantinya menjadi residu yang lebih stabil akan meningkatkan kestabilan termal enzim tersebut (Liu et al. 2003).
Mutasi protein tersebut dapat dilakukan dengan mempelajari struktur protein melalui pendekatan simulasi dinamika molekul (Molecular Dynamic Simulation) atau pemodelan komputer (Han et al. 2009; Santarossa et al. 2005). Strategi simulasi yang dilakukan untuk mempelajari perubahan struktur protein selama proses denaturasi/ unfolding adalah dengan melakukan simulasi pada temperatur yang sangat tinggi (Li dan Dagget 1994; Day et al. 2002; Becker et al. 2001; Liu dan wang 2003; Purmonen et al. 2007). Penggunaan temperatur tinggi menyebabkan simulasi pada proses perubahan struktur dapat terjadi lebih cepat sehingga penggunaan sumber daya komputasi dapat dikurangi.
Analisis hasil simulasi tersebut dapat dimanfaatkan oleh peneliti secara in-vitro sebagai bahan pertimbangan untuk menentukan residu asam amino yang akan dimutasi pada rekayasa genetik. Untuk meningkatkan kestabilan termal wild-type, pada suhu 500 K, Ashutosh et al. (2014) telah menganalisis wild-type Bacillus subtilis ekstraseluler (BsL) yang telah di mutasi secara in-vitro dengan memutasi 12 residu dan diperoleh penurunan RMSD sebesar 3.567 Å.
Penelitian ini bertujuan untuk menganalisis kestabilan termal enzim lipase A B. subtilis ekstraseluler (BsL) wild type (Kode PDB : 1ISP) dengan mengamati peristiwa unfolding yang terjadi untuk mencari residu-residu penstabil. Selanjutnya dirancang mutan yang termostabil dengan cara mengganti residu pengunci kestabilan enzim dengan residu yang diduga lebih stabil. Selanjutnya membandingkan hasil analisis kestabilan termal enzim wild-type dan mutan yang telah dirancang dengan membandingkan parameter Root Mean Squared Deviation (RMSD), Radius of Gyration (Rg), Solvent Accessible Surface Area (SASA), Root Mean Squared Fluctuation (RMSF) dan Struktur Sekunder.
Perumusan Masalah
3
Tujuan Penelitian
Penelitian ini bertujuan untuk menganalisis kestabilan termal enzim lipase A B. subtilis (BsL) wild type dengan mengamati peristiwa unfolding yang terjadi untuk mencari residu-residu penstabil. Selanjutnya dirancang mutan yang termostabil dengan cara mengganti residu pengunci kestabilan enzim dengan residu yang diduga lebih stabil. Selanjutnya membandingkan hasil analisis kestabilan termal enzim wild-type dan mutan yang telah dirancang dengan membandingkan parameter Root Mean Squared Deviation (RMSD), Radius of Gyration (Rg), Solvent Accessible Surface Area (SASA), Root Mean Squared Fluctuation (RMSF) dan Struktur Sekunder.
Manfaat Penelitian
Manfaat dari penelitian ini adalah dapat memberikan saran rancangan mutasi enzim lipase B. subtilis termostabil yang dapat diimplementasikan secara in-vitro (wet experiment) untuk melakukan rekayasa genetik pada enzim lipase B. subtilis.
Ruang Lingkup Penelitian
Penelitian ini berupa simulasi dinamika molekuler pada temperatur yang sangat tinggi untuk menganalisis sifat termal dari enzim lipase B. subtilis. Simulasi dinamika molekuler untuk mengamati fenomena unfolding dilakukan pada temperatur 300 K selama 2 ns untuk validasi metode, temperatur 400 K, 450 K, 475 K dan 500 K selama 50 ns untuk simulasi pada wild-type. Sedangkan untuk mutan dilakukan dengan temperatur 525 K selama 20 ns dan 500 K selama 50 ns.
2
TINJAUAN PUSTAKA
Lipase A Bacillus subtilis Ekstraseluler (BsL)
Bacillus subtilis adalah bakteri gram-positif yang ditemukan di tanah, air dan dalam tanaman. Organisme ini sangat menarik untuk dipelajari karena memiliki efisiensi yang tinggi dalam mensekresikan protein seperti lipase, amilase dan protease (Harwood 1992).
4
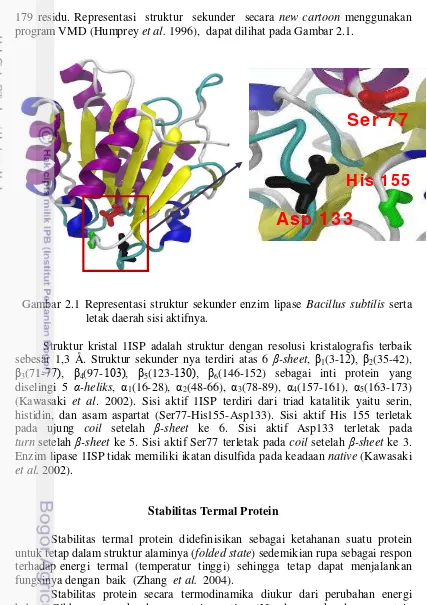
179 residu. Representasi struktur sekunder secara new cartoon menggunakan program VMD (Humprey et al. 1996), dapat dilihat pada Gambar 2.1.
Se r 7 7
H is 1 5 5
Asp 1 3 3
Gambar 2.1 Representasi struktur sekunder enzim lipase Bacillus subtilis serta letak daerah sisi aktifnya.
Struktur kristal 1ISP adalah struktur dengan resolusi kristalografis terbaik sebesar 1,3 Å. Struktur sekunder nya terdiri atas 6 β-sheet, β1(3-12), β2(35-42),
β3(71-77), β4(97-103), β5(123-130), β6(146-152) sebagai inti protein yang
diselingi 5 α-heliks, α1(16-28), α2(48-66), α3(78-89), α4(157-161), α5(163-173)
(Kawasaki et al. 2002). Sisi aktif 1ISP terdiri dari triad katalitik yaitu serin, histidin, dan asam aspartat (Ser77-His155-Asp133). Sisi aktif His 155 terletak pada ujung coil setelah β-sheet ke 6. Sisi aktif Asp133 terletak pada turn setelah β-sheet ke 5. Sisi aktif Ser77 terletak pada coil setelah β-sheet ke 3. Enzim lipase 1ISP tidak memiliki ikatan disulfida pada keadaan native (Kawasaki et al. 2002).
Stabilitas Termal Protein
Stabilitas termal protein didefinisikan sebagai ketahanan suatu protein untuk tetap dalam struktur alaminya (folded state) sedemikian rupa sebagai respon terhadap energi termal (temperatur tinggi) sehingga tetap dapat menjalankan fungsinya dengan baik (Zhang et al. 2004).
Stabilitas protein secara termodinamika diukur dari perubahan energi bebas Gibbs antara keadaan protein native (N) dengan keadaaan protein terdenaturasi/ unfolded (U) pada kesetimbangan dinamik (N ⇌U) yang juga disebut sebagai perubahan energi bebas unfolding protein (Kumar dan Nussinov 2001) :
5
Dimana ∆H adalah perubahan entalpi dan ∆S adalah entropi antara protein dalam keadaan native dengan keadaan unfolded. Penggunaan model sederhana untuk kesetimbangan ini dilakukan dengan model dua keadaan (two state unfolding), dimana protein hanya berada pada dua keadaan diskret, yaitu N dan U. Keadaan N adalah keadaan dimana kekompakan struktur tercapai dan protein melipat secara sendiri. Struktur kristal yang ditentukan secara eksperimental dapat dianggap mewakili keadaan N, walaupun sebenarnya keadaan N tidak terdiri dari konformasi tunggal melainkan berupa sekumpulan struktur dengan konformasi pada tingkatan yang sama (Buechner dan Kiefhaber 2005).
Entalpi dari reaksi unfolding protein bernilai positif, sehingga secara entalpi, keadaan native lebih disukai dan lebih stabil dibanding keadaan unfolded. Interaksi-interaksi internal seperti ikatan hidrogen dan interaksi van der Waals intramolekular memberi efek menstabilkan secara entalpik, sedangkan ekspos interior protein terhadap protein pada keadaan unfolding bersifat destabilisasi secara entalpik (Buechner dan Kiefhaber 2005).
Proses unfolding protein disertai oleh peningkatan entropi sistem, sehingga secara keadaan unfolding lebih disukai secara entropik. Faktor pertama yang menyebabkan peningkatan entropi adalah interaksi gugus-gugus yang terekspos kepada pelarut pada keadaan unfolded. Faktor lainnya adalah bertambahnya jumlah konformasi protein yang dapat diakses oleh protein keadaan unfolded dibanding konformasi yang mungkin diakses keadaan native. Secara keseluruhan, kestabilan struktur dicapai secara entalpik yang diimbangi oleh destabilisasi entropik. Kesetimbangan ke arah keadaan native tercapai ketika nilai entalpik cenderung lebih besar dibanding entropi destabilisasi (Buchner dan Kiefhaber 2005).
Simulasi Dinamika Molekul
Biomolekul seperti protein dan asam nukleat merupakan sistem yang dinamis. Simulasi dinamika molekul merupakan salah satu bagian dari pendekatan komputasi dimana atom dan molekul dibiarkan saling berinteraksi selama periode waktu tertentu sehingga tingkah laku sistem dapat teramati.
Metode simulasi dinamika molekul pertama kali diperkenalkan oleh Alder dan Wainwright pada akhir tahun 1950. Pada awalnya digunakan untuk mempelajari permukaan yang keras (hard spheres). Sedangkan simulasi dinamika molekul terhadap sistem nyata baru dilakukan pada tahun 1974. Tahun 1977 teknik ini pertama kali digunakan untuk kajian biomolekul dengan menggunakan bovine pancreatic trypsin inhibitor (BPTI) (McCammon et al. 1997).
6
Gaya Intramolekul
Dalam simulasi dinamika molekul, fungsi energi potensial diberikan oleh medan gaya, yaitu fungsi yang mendefinisikan gaya-gaya yang bekerja pada suatu atom individual pada keadaan energi rendah (kesetimbangan termal) (Ponder dan Case 2003). Ada dua kelompok interaksi yang dapat memberikan pengaruh terhadap energi potensial, yaitu interaksi internal dan eksternal. Interaksi internal didefinisikan sebagai interaksi kovalen antar atom yang disebut sebagai interaksi ikatan (bonded interaction), meliputi uluran ikatan (bonded streching) yaitu interaksi kovalen antara dua atom pada jarak kesetimbangan tertentu, sudut ikatan dan sudut dihedral. Pada simulasi dinamika molekul, ikatan kovalen didefinisikan sebagai pegas dengan pergerakan harmonis (Becker 2001). Sedangkan interaksi eksternal mengekspresikan interaksi non-ikatan. Interaksi non-ikatan mempresentasikan interaksi fleksibel diantara pasangan atom atau partikel. Dua jenis interaksi non-ikatan paling umum yang dapat mengakibatkan perubahan energi potensial adalah interaksi elektrostatik (Potential Coulomb) dan interaksi van der Waals (Potential Lennard Jones) (Becker et al. 2001).
( ) = ∑ ( − , ) + ∑ ( − , ) + ∑ ( 1 +
cos ( − ) ) + ∑ ∑ 4 − +
(2)
3
METODE
Bahan
Bahan yang digunakan dalam penelitian ini adalah data eksperimen hasil X-Ray Diffraction berupa data koordinat lipase B. subtilis (Kawasaki et al. 2002) yang dapat diunduh di Protein Data Bank (http://www.rscb.org/pdb/) (Berman et al. 2000) dengan kode PDB 1ISP.
Alat
7 perangkat lunak yang digunakan untuk prediksi mutasi sepasang residu menjadi sistein adalah “Disulfide by DesignTM V 1.2” (Dombkowski 2003) untuk
Struktur kristal Lipase A Bacillus subtilis (Kode PDB : 1ISP) (Kawasaki et al. 2002) yang digunakan pada simulasi diperoleh dari bank data Protein Data Bank (PDB), (http://www.rscb.org/pdb/) (Berman et al. 2000). Penentuan struktur kristal 1ISP dilakukan dengan metode sinar X yang memiliki resolusi 1.3 Å dengan jumlah residu penyusunnya 179. Pada berkas tersebut terdapat 1 frame sebagai model konformasi. Sebelum digunakan sebagai data awal dalam penelitian, data koordinat terlebih dahulu dipreparasi karena data tersebut masih terdapat molekul air dan ligan yaitu Gliserol. Selanjutnya di atur pH sesuai dengan pH alami enzim tersebut yaitu 10 dengan menggunakan web server H++ (Gordon et al. 2005). Program psfgen digunakan untuk menerapkan medan gaya ke dalam sistem molekul. Medan gaya yang digunakan untuk mendefinisikan energi potensial struktur kristal adalah CHARMM22 (Brooks et al. 1983). Solvasi model molekul menggunakan TIP3P sebagai molekul pelarut (Jorgensen et al. 1983) dengan dimensi kotak sebesar 70 Å x 70 Å x 70 Å. Secara total, jumlah molekul air yang ditambahkan pada kotak adalah 30404 molekul air. Penambahan molekul air secara eksplisit dilakukan sebagai pendekatan terhadap keadaan molekul dalam pelarut, dimana interaksi dengan molekul pelarut ikut mempengaruhi konformasi molekul. Untuk menetralkan total muatan sistem, empat ion Cl- ditambahkan untuk menetralkan empat ion Na+.
Kondisi Simulasi
Keseluruhan simulasi menggunakan parameter integrasi waktu (time step) setiap 2 femto detik (fs). Simulasi dilakukan dengan menggunakan metode periodic boundary condition (PBC) untuk menghilangkan efek tegangan permukaan dan untuk mencapai kondisi dengan kerapatan dan tekanan yang lebih seragam (Allen dan Tildesley 1989). Algoritma SHAKE dengan toleransi 10-5 diterapkan kedalam sistem untuk mengekang seluruh ikatan yang
8
Adapun proses pemanasan molekul yang telah diminimisasi menggunakan perangkat lunak NAMD dilakukan secara bertahap dari suhu 0 K sampai 300 K, 400 K, 450 K, 475 K, 500 K untuk wild-type, 500 K dan 525 K untuk mutan. Dilakukan selama 20 ps dengan kenaikan setiap 25 K. Selanjutnya dilakukan ekuilibrasi yang bertjuan untuk menahan protein stabil di dalam sistem simulasi. Ekuilibrasi dilakukan dengan protokol Langevin selama 40 ps.
Production Run
Setelah molekul enzim terekuilibrasi dengan baik maka tahap selanjutnya adalah tahap produksi (Production Run). Konstrain yang diterapkan pada proses ekuilibrasi kemudian dihilangkan pada proses production run sehingga molekul protein bebas bergerak. Production run dilakukan selama 2 ns dengan temperatur 300 K untuk validasi metode, selama 50 ns dengan temperatur 400 K, 450 K, 475 K, dan 500 K untuk wild-type dan selama 20 ns dengan temperatur 525 K untuk uji mutan serta selama 50 ns dengan 500 K untuk mutan-1.
Analisis Simulasi Dinamika Molekul
Untuk menganalisis kestabilan dan fleksibilitas enzim wild-type dan mutan serta mengevaluasi proses simulasi yang telah dilakukan, sejumlah parameter dianalisis terhadap hasil simulasi meliputi Root Mean Square Deviation
(RMSD), Jari-Jari Girasi (Rg), Root-mean-square fluctuation (RMSF), Solvent Accessible Surface Area(SASA) dan analisis struktur sekunder
Root-mean-square deviation (RMSD) adalah akar kuadrat rata-rata penyimpangan koordinat atom dari posisi referensi. RMSD merupakan ukuran perbedaan struktur protein selama proses simulasi terhadap struktur awal protein. Analisis ini difokuskan pada perubahan struktur atom C-α asam amino penyusun protein yang didefinisikan sebagai berikut :
RMSD = ∑ ( ( ) ( ) ) (3) dengan N adalah jumlah total atom C-α yang terdapat pada protein sedangkan ri(t2) dan ri(t1) adalah koordinat atom C-α pada waktu t2 dan pada
waktu t1. Untuk mendapatkan gambaran profil kestabilan sistem yang
disimulasikan serta tahap-tahap transisi pada proses simulasi termal protein, maka hasil analisis RMSD tersebut akan di plot dalam bentuk grafik antara RMSD terhadap waktu simulasi (Becker et al. 2001; Coutsias et al. 2004).
9 protein. Nilai jari-jari girasi dihitung menggunakan fungsi posisi atom terhadap pusat massa protein. Richards 1971). Nilai SASA memberikan gambaran terhadap struktur tersier protein. Pada umumnya, protein mengemas struktur tersiernya sedemikian rupa sehingga gugus-gugus yang bersifat hidrofobik akan berada di bagian dalam sedangkan gugus-gugus asam amino yang bersifat hidrofil akan terdapat di bagian luar. Ketika proses simulasi termal berlangsung, kestabilan struktur tersier protein dan derajat keeksposuran dapat diamati dengan melakukan perhitungan SASA.
Struktur sekunder protein tersusun atas interaksi lokal inter-residu yang pada umumnya dimediasi oleh ikatan hidrogen. Struktur sekunder yang paling umum adalah α-helix dan β-sheet. Analisis struktur sekunder dilakukan untuk melihat kestabilan struktur sekunder selama proses simulasi berlangsung yang dikarakterisasi dengan perubahan komposisi α-helix, β-sheet, turn dan coil. Perhitungan struktur sekunder dilakukan dengan menggunakan plug-in yang terdapat pada perangkat lunak VMD yang didasarkan atas algoritma STRIDE (Structural identification) (Frishman dan Argos 1995).
Prediksi Pasangan Residu yang akan Dimutasi Menjadi Sistein
Pasangan residu yang akan dapat dimutasi menjadi sistein pada Bacillus subtilis diprediksi menggunakan perangkat lunak “Disulfide by DesignTM” (Dombkowski 2003). Masukan data yang diperlukan adalah struktur kristal Bacillus subtilis (Kode PDB 1ISP). Kriteria yang digunakan adalah Optimum Chi3-angle = +100o/-80o, tolerance ± 30o; dan Ca-Cb-S-angle=114.60, tolerance± 10o. Keluaran dari perangkat lunak ini adalah beberapa pasangan residu yang dapat dimutasi menjadi sistein dan dapat membentuk ikatan disulfida berdasarkan kriteria geometrik (jarak dan sudut). Pembuatan struktur enzim mutan juga dilakukan dengan perangkat lunak ini dengan memilihi mutasi yang diinginkan. Keluaran struktur mutan didapat dalam bentuk file pdb (*.pdb).
Penentuan Mutan dengan Teknik simulasi FEP (Free Energy Perturbation) Untuk menganalisis efek mutasi terhadap stabilitas termal enzim secara kuantitatif, dilakukan perhitungan nilai perubahan energi bebas (∆∆Gsolv ) melalui pendekatan free energy perturbation (FEP). Jika perhitungan nilai
10
bahwa mutan lebih stabil dibandingkan wild-type nya (Kollman 1993; Ghoufi et al. 2004).
Simulasi FEP dilakukan dengan dua kondisi yaitu sistem tanpa air (vacuo) dan sistem dengan air (aqua). Simulasi vacuo dilakukan dengan kenaikan λ sebesar 0.05 selama 10 ps dan diambil nilai energi bebas selama 100 ps untuk setiap λ sedangkan simulasi aqua dilakukan dengan kenaikan λ sebesar 0.05 yang diekulibrasi selama 100 fs dan diambil nilai energi bebas selama 500 fs setiap nilai λ. Kedua simulasi dilakukan tanpa menggunakan soft-core potential untuk menghilangkan interaksi topologi target dengan lingkungan pada saat nilai λ mendekati 0 atau 1. Struktur asli dan mutan tidak saling berinteraksi selama simulasi.
Simulasi Dinamika Molekul Mutan
Molekul BsL yang telah dimutasi (mutan) kemudian dianalisis kestabilannya pada temperatur tertentu dengan metode simulasi dinamika molekul. Simulasi dilakukan pada temperatur 525 K uji mutan dan 500 K untuk mutan-1. Tahapan simulasi dinamika molekular mutan sama seperti tahapan simulasi wild type, yaitu preparasi, minimisasi, pemanasan dan ekuilibrasi, serta production run. Parameter-parameter kondisi dan sistem simulasi yang digunakan juga sama seperti simulasi wild type. Kemudian dilakukan analisis terhadap trajektori simulasi dinamika molekul yang dihasilkan. Termostabilitas mutan dan wild type dibandingkan dengan melakukan analisis terhadap parameter struktur protein seperti RMSD, Rg, RMSF, SASA dan Struktur Sekunder.
4
HASIL DAN PEMBAHASAN
Penelitian pada enzim atau protein termostabil dapat memberikan pengetahuan baru tentang faktor-faktor yang menentukan stabilitas termal enzim, maupun aplikasi praktis pada bidang industri. Pemahaman tentang faktor penentu stabilitas termal suatu protein atau enzim dapat dijadikan sebagai acuan untuk merekayasa protein serta menghindari kesalahan target mutasi yang dapat berakibat fatal terhadap kestabilan maupun aktivitas enzim.
Penelitian yang telah dilakukan terbagi atas tiga kelompok, meliputi simulasi kestabilan termal enzim wild-type, simulasi FEP untuk menganalisis calon mutan dan simulasi kestabilan termal mutan.
Validasi Metode Simulasi Dinamika Molekul
Validasi teknik percobaan penting dilakukan agar percobaan yang dilakukan dapat mewakili fenomena yang akan dipelajari. Begitu juga dengan metode simulasi dinamika molekul dapat divalidasi dengan membandingkan data simulasi faktor-βdengan data eksperimennya (Van gunsteren 1998).
11 pada file bank data (PDB) menggambarkan koordinat kesetimbangan atau rata-rata, dan bukanlah koordinat pasti (rigid) dari molekul protein dikarenakan adanya perbedaan struktur antar molekul dalam kisi kristal. Pergeseran atom dari posisi kesetimbangan dapat diamati dari pergeseran kerapatan elektron yang terdapat pada bank data suatu kristal protein.
Nilai faktor-β simulasi, diperoleh dari data RMSF (root mean-square fluctuation) hasil simulasi. Dari RMSF dapat dapat dihitung faktor-β dengan persamaan berikut (Kuzmanic dan Zagrovic 2010) :
= (5) Nilai RMSF yang digunakan adalah RMSF atom-atom C-α dari simulasi dinamika molekul pada temperatur 300 K yang dihitung dengan waktu simulasi 2 ns dengan struktur pembanding kristal 1ISP dari PDB. Hasil perbandingan menunjukkan bahwa faktor-β simulasi dinamika molekul dan faktor-β
kristalografi tidak berbeda secara signifikan (Gambar 4.1). Secara kesuluruhan keduanya memiliki pola yang sama.
(2)
(13)
(120)
(44)
Gambar 4.1 Perbandingan nilai faktor-β kristal 1ISP (Biru) dengan faktor-β
simulasi (Orange) pada temperatur 300 K
Residu-residu yang memiliki perbedaan nilai faktor-β yang cukup besar adalah pada residu 2, 13, 44, 120. Pada daerah-daerah tersebut faktor-β simulasi lebih besar dibanding faktor-β kristalografi. Hal ini disebabkan adanya perbedaan kondisi protein pada simulasi dan kristalografi. Pada sistem simulasi, protein berada dalam kondisi terlarut dalam air sehingga residu penyusunnya menjadi lebih fleksibel dibandingkan dengan residu dalam kondisi kristal yang rigid.
Analisis Kestabilan Termal Wild-type Bacillus subtilis
12
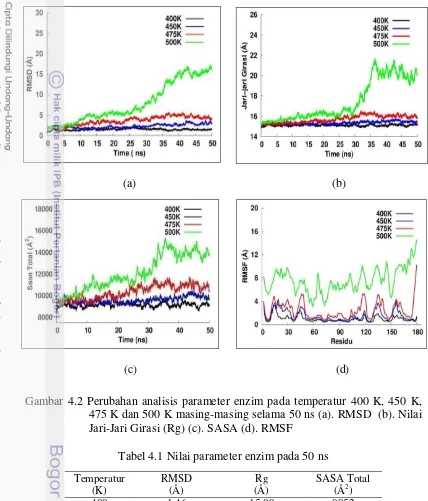
Sekunder. Gambar 4.2 menunjukkan fluktuasi nilai RMSD, Rg, dan SASA pada variasi temperatur 400 K, 450 K, 475 K dan 500 K selama 50 ns.
(a) (b)
(c) (d)
Gambar 4.2 Perubahan analisis parameter enzim pada temperatur 400 K, 450 K, 475 K dan 500 K masing-masing selama 50 ns (a). RMSD (b). Nilai Jari-Jari Girasi (Rg) (c). SASA (d). RMSF
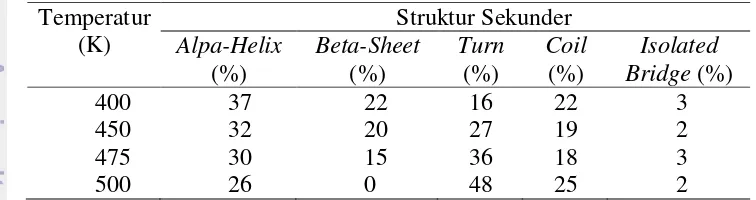
Tabel 4.1 Nilai parameter enzim pada 50 ns
Nilai RMSD, Rg dan SASA pada temperatur 400 K, 450 K dan 475 K cukup konstan dan tidak mengalami peningkatan nilai yang signifikan. Adanya kenaikan temperatur pada sistem tersebut belum mempengaruhi kestabilan protein. Berbeda halnya dengan suhu 500 K, pada temperatur ini nilai RMSD, Rg
Temperatur
(K) RMSD (Å) (Å) Rg SASA Total (Å2)
400 1.46 15.09 8852
450 2.76 15.29 9615
475 3.87 15.76 10401
13 dan SASA cukup konstan sampai periode waktu 25 ns. Kemudian terjadi peningkatan nilai RMSD, Rg dan SASA yang cukup tajam. Adanya kenaikan nilai RMSD, Rg dan SASA yang cukup signifikan pada rentang 28 – 42 ns mengindikasikan telah terjadi proses unfolding.
Nilai tertinggi parameter RMSD, Rg dan SASA selama 50 ns dapat dilihat pada Tabel 4.1. Nilai RMSD, Rg maupun SASA pada 500 K meningkat sangat tinggi jika dibandingkan dengan suhu 475 K, 450 K dan 400 K. Pada suhu 400 K, 450 K dan 475 K kenaikan nilai RMSD hanya sekitar 1.5 sampai 2 kali semula sedangkan untuk suhu 500 K, RMSD meningkat 5 kali lipat. Untuk Rg pada suhu 400 K, 450 K dan 475 K kenaikan Rg hanya sekitar 1.3% sampai 3% sedangkan untuk suhu 500 K Rg meningkat cukup signifikan yaitu sebesar 28%. Untuk SASA pada suhu 400 K, 450 K dan 475 K kenaikan SASA hanya sekitar 8% sedangkan untuk suhu 500 K SASA meningkat pesat sebesar 35% .
Untuk parameter RMSF dari gambar 4.2.d terlihat bahwa simulasi pada temperatur 400 K, 450 K dan 475 K nilai RMSF tidak mengalami peningkatan yang signifikan. Pola yang diperlihatkan juga cenderung sama pada ketiga variasi suhu. Ini menandakan bahwa protein masih cenderung stabil, walaupun ada beberapa puncak yang muncul yang menggambarkan residu yang fleksibel, tetapi jumlahnya masih tergolong sedikit. Sedangkan pada temperatur 500 K nilai RMSF meningkat cukup signifikan dan pola nilainya juga sangat berbeda dibandingkan variasi suhu lainnya. Seiring meningkatnya temperatur, residu-residu yang semula rigid menjadi fleksibel, yang ditandai oleh banyaknya puncak yang muncul. Hal ini mengindikasikan bahwa telah terjadi proses unfolding pada protein.
Analisis perubahan struktur sekunder merupakan analisis yang dapat digunakan sebagai penanda telah terjadinya unfolding protein, terutama hilang/ rusaknya struktur heliks dan sheet (Day et al. 2002; Li et al. 1994). Analisis struktur sekunder juga dapat melengkapi pemahaman akan perubahan struktur selama simulasi yang sebelumnya dilakukan lewat analisis RMSD, Rg dan SASA.
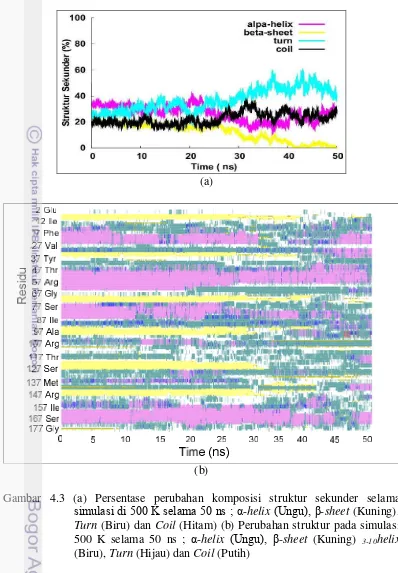
Komposisi struktur sekunder terutama alpa-helix maupun beta-sheet pada temperatur 400 K, 450 K dan 475 K selama 50 ns tidak mengalami perubahan yang signifikan. Sedangkan pada temperatur 500 K komposisi alpa-helix maupun beta-sheet menurun sangat drastis (Tabel 4.2).
Tabel 4.2 Perubahan komposisi struktur sekunder pada temperatur 400 K, 450 K, 475 K dan 500 K masing-masing pada 50 ns
14
(a)
(b)
Gambar 4.3 (a) Persentase perubahan komposisi struktur sekunder selama
simulasi di 500 K selama 50 ns ; α-helix (Ungu), β-sheet (Kuning), Turn (Biru) dan Coil (Hitam) (b) Perubahan struktur pada simulasi 500 K selama 50 ns ; α-helix (Ungu), β-sheet (Kuning) 3-10helix
(Biru), Turn (Hijau) dan Coil (Putih)
15 Pada gambar 4.3.a memperlihatkan bahwa komposisi alpa-helix dan beta-sheet pada suhu 500 K menurun drastis sampai nol pada rentang waktu 28 ns hingga 42 ns sedangkan komposisi coil dan turn meningkat. Hal ini menandakan pada temperatur 500 K, protein telah mengalami unfolding sama seperti hasil analisa RMSD, Rg dan SASA. Rusaknya struktur sekunder dari enzim Bacillus subtilis pertama sekali terjadi pada α-helix 3 pada rentang waktu 22 ns dan kemudian disusul oleh α-helix 1 pada waktu 28 ns. Sedangkan α-helix 2 dan ke 4
hanya mengalami kerusakan yang relatif sedikit jika dibandingkan α-helix lainnya. Untuk struktur β-sheet, yang pertama kali mengalami kerusakan adalah β -sheet 2, 4, 5 dan 6 pada rentang waktu 28 ns. Kemudiaan pada 30 ns kerusakan dialami β-sheet ke 1 dan 3.
Pemilihan Mutan Protein Bacillus subtilis
Pemilihan residu-residu yang akan dimutasi untuk meningkatkan kestabilan enzim lipase B.subtilis didasarkan pada analisis interaksi elektrostatik, ikatan disulfida dan interaksi hidrofobik. Analisa tersebut hanya dilakukan pada suhu 500 K karena analisis ini dilakukan untuk mencari calon mutan yang akan dimutasi. Pada suhu ini protein sudah unfolding, sehingga mudah untuk mencari residu-residu penstabil protein.
Interaksi Elektrostatik/ Jembatan Garam
Interaksi elektrostatik yang sering disebut sebagai interaksi pasangan ion (ion pairs interaction) (Vieille dan Zeikus 2001) atau jembatan garam (salt bridge) (Tomazic dan Klibanov 1988) merupakan interaksi antara residu-residu asam amino bermuatan. Penelitian mengenai pentingnya interaksi elektrostatik pertama kali dilaporkan oleh Perutz (1978) yang menyatakan bahwa interaksi ini memiliki kontribusi signifikan untuk menstabilkan protein.
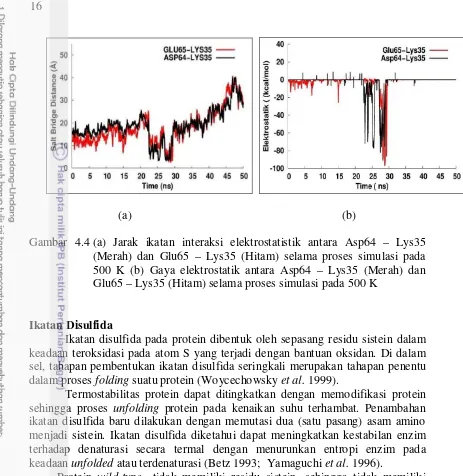
Terdapat 57 pasangan jembatan garam pada temperatur 500 K selama 50 ns. Selanjutnya jembatan garam diplot antara jarak (Å) dengan waktu (ns). Ada dua kurva jembatan garam yang polanya sama dengan pola kurva RMSD dan SASA yang meningkat pada selang waktu 28 ns, yaitu jembatan garam Asp64-Lys35 dan Glu65-Lys35 (Gambar 4.4.a). Kenaikan tajam nilai RMSD dan SASA tersebut disebabkan putusnya kedua jembatan garam tersebut. Sehingga ketika dilakukan mutasi secara in-silico calon mutan yang akan dimutasi adalah residu-residu asam amino yang diduga berperan dalam interaksi elektrostatik dan menggantinya dengan residu asam amino yang lain atas dasar kemiripan struktur.
Selain itu, jika diamati dari energi elektrostatiknya (Gambar 4.4.b) kedua pasang jembatan garam ini akan kehilangan energinya pada selang waktu 28 ns, waktu ini sama dengan waktu unfolding nya. Ini menandakan bahwa kedua pasang jembatan garam ini merupakan pengunci kestabilan termal enzim Bacillus subtilis.
16
(a) (b)
Gambar 4.4 (a) Jarak ikatan interaksi elektrostatistik antara Asp64 – Lys35 (Merah) dan Glu65 – Lys35 (Hitam) selama proses simulasi pada 500 K (b) Gaya elektrostatik antara Asp64 – Lys35 (Merah) dan Glu65 – Lys35 (Hitam) selama proses simulasi pada 500 K
Ikatan Disulfida
Ikatan disulfida pada protein dibentuk oleh sepasang residu sistein dalam keadaan teroksidasi pada atom S yang terjadi dengan bantuan oksidan. Di dalam sel, tahapan pembentukan ikatan disulfida seringkali merupakan tahapan penentu dalam proses folding suatu protein (Woycechowsky et al. 1999).
Termostabilitas protein dapat ditingkatkan dengan memodifikasi protein sehingga proses unfolding protein pada kenaikan suhu terhambat. Penambahan ikatan disulfida baru dilakukan dengan memutasi dua (satu pasang) asam amino menjadi sistein. Ikatan disulfida diketahui dapat meningkatkan kestabilan enzim terhadap denaturasi secara termal dengan menurunkan entropi enzim pada keadaan unfolded atau terdenaturasi (Betz 1993; Yamaguchi et al. 1996).
Protein wild-type tidak memiliki residu sistein, sehingga tidak memiliki ikatan disulfida. Untuk meningkatkan kestabilan termal protein perlu ditambahkan ikatan disulfida dengan memutasi sepasang residu menjadi sistein. Perangkat lunak yang digunakan untuk prediksi mutasi sepasang residu menjadi sistein adalah “Disulfide by DesignTM” (Dombkowski 2003).
Hasil prediksi pasangan residu keluaran adalah 29 pasang residu yang mungkin untuk dimutasi. Beberapa residu yang terlibat dalam pembentukan struktur heliks atau sheet tidak dipilih sebagai target mutasi karena alasan sterik (Pikkemaat et al. 2002). Dengan mengamati letak pada struktur helix atau pada sheet (Lampiran 5) terpilihlah 10 pasang residu yang diprediksi dapat membentuk ikatan disulfida.
17
Gambar 4.5 Target mutasi pada enzim wild type dan mutan-mutan yang dihasilkan
Interaksi Hidrofobik
Di antara sekian banyak faktor penentu stabilitas termal protein, interaksi hidrofobik juga dipertimbangkan sebagai faktor dominan penentu kestabilan protein (Mathews dan van Holde 1996). Hal ini didasari atas fenomena bahwa ketika protein melipat (folded) sebagian besar residu non polar (~80%) akan terkubur di dalam interior protein, terlindungi dari pelarut sehingga menyebabkan suatu protein dapat mempertahankan integritasnya (Klapper 1971). Fenomena tersebut dikenal sebagai pengaruh hidrofobik (hydrophobic effect).
Dari hasil analisa RMSF diperoleh tiga residu yang rigid dan merupakan residu yang hidrofobik, yaitu Ile73, Ile151 dan Val59. Sehingga diduga residu ini adalah residu hidrofobik penstabil pada protein tersebut. Ketiga residu tersebut kemudian dimutasi dan diperoleh 6 variasi mutan yaitu Ile73Gln, Ile151Ser dan Val59Thr yang berfungsi menghilangkan efek hidrofobik. Kemudian variasi mutan Ile73Leu, Ile151Val dan Val59Ile untuk mempertahankan interaksi hidrofobiknya.
Untuk menganalisis efek mutasi terhadap stabilitas termal enzim secara kuantitatif, dilakukan perhitungan nilai perubahan energi bebas (∆∆Gsolv )
melalui pendekatan free energy perturbation (FEP). Jika perhitungan nilai
∆∆Gsolv positif mengindikasikan bahwa mutan lebih tidak stabil dibandingkan
wild-type nya dan sebaliknya jika perhitungan ∆∆Gsolv negatif mengindikasikan
bahwa mutan lebih stabil dibandingkan wild-type nya (Kollman 1993; Ghoufi et al.2004).
Analisis perubahan energi bebas solvasi
Keluaran simulasi FEP adalah nilai ΔG (Kcal/mol) untuk setiap nilai. Gambar 4.6 adalah gambar grafik untuk nilai ΔΔGsolv pada calon mutan. Dari gambar dapat kita amati bahwa terdapat mutan yang memiliki nilai ΔΔGsolv
positif dan nilai ΔΔGsolv negatif. Nilai ΔΔGsolv tersebut diperoleh dari selisih
ΔG dari simulasi tanpa pelarut (vacuo) dan simulasi dengan pelarut (aqua)
didefinisikan sebagai ΔΔGsolv (Kcal/mol) atau perubahan energi bebas
solvasi. Nilai tersebut menyatakan jumlah energi yang dibutuhkan melakukan mutasi. Mutan yang memiliki nilai ΔΔGsolv yang negatif yaitu Glu65Asp, Ile135Cys/Hsd156Cys, Ile151Val dan Val59Thr. Hal ini mengindikasikan bahwa keempat mutan tersebut lebih stabil dibandingkan wild-typenya.
Dari Tabel 4.3 juga memperlihatkan nilai ΔΔGsolv pada semua calon mutan dan diperoleh 4 mutan yang diduga lebih stabil dari wild-type nya yaitu Glu65Asp, Ile135Cys/Hsd156Cys, Ile151Val dan Val59Thr. Glu65Asp adalah mutan yang diperoleh berdasarkan analisis interaksi elektrostatik. Glu65Asp lebih stabil dibandingkan dengan Asp64Glu walaupun keduanya memiliki kesamaan yaitu dirancang untuk tetap mempertahankan interaksi elektrostatik. Hal ini
18
dikarenakan Asp memiliki rantai samping (R = -CH2-COOH) yang lebih pendek
dibandingkan Glu (R = -CH2-CH2-COOH) sehingga mampu mengurangi kontak
yang tidak sesuai (unfavorable) dengan pelarut (Vieille dan Zeikus 2001). Nilai pKa pada rantai samping kedua Asp dan Glu juga mempengaruhi. Untuk Asp nilai pKa rantai kedua adalah 3.9 sedangkan untuk Glu adalah 4.2 (Nurbaiti 2009). Hal ini menyebabkan Asp lebih mudah mempertahankan muatan negatif nya daripada Glu (Mathews dan van Holde 1996). Sehingga interaksi elektrostatik antara Asp dengan Lys menjadi lebih kuat dibandingkan Glu. Sehingga Asp lebih mampu menstabilkan enzim.
(a) (b)
(c)
19
Tabel 4.3 Pemilihan Mutan dan Nilai ∆∆Gsolv Analisa Pemilihan
Mutan Residu yang akan dimutasi Mutan (Kkal/mol) ∆∆Gsolv
Interaksi Elektrostatik Asp 64 Asn 15.5813
Glu 6.9903
Glu65 Asp -6.207
Gln 21.7704
Ikatan Disulfida Ile 135 Cys -7.48769
Leu 145 Cys Unstable
Gly 158 Cys 1.50212
Ala 146 Cys 2.20751
Gly 155 Cys 5.70778
Hsd 156 Cys 10.597
Interaksi Hidrofobik Ile 73 Leu 3.9217
Gln 5.8102
Ile 151 Ser 1.805775
Val -1.9624
Val 59 Ile 1.36309
Thr -1.5060661
Glu 6 4
Lys 3 5
(a)
Asp 6 5
Lys 3 5
(b)
20
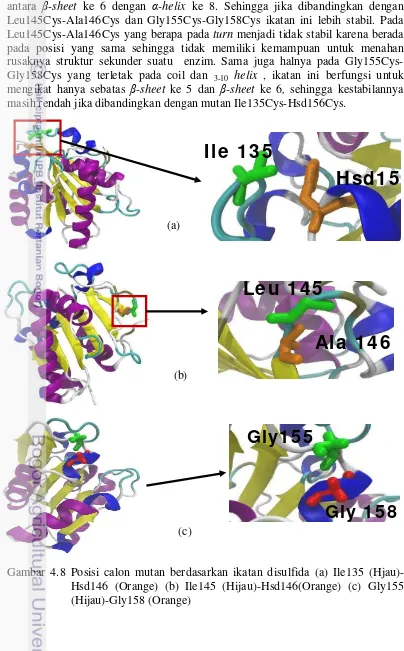
Untuk mutan Ile135Cys-Hsd156Cys adalah mutan yang diperoleh berdasarkan analisis ikatan disulfida. Residu Ile135 berada pada turn dan residu Hsd156 berada pada coil. Ikatan ini berfungsi untuk meningkatkan kestabilan antara β-sheet ke 6 dengan α-helix ke 8. Sehingga jika dibandingkan dengan Leu145Cys-Ala146Cys dan Gly155Cys-Gly158Cys ikatan ini lebih stabil. Pada Leu145Cys-Ala146Cys yang berapa pada turn menjadi tidak stabil karena berada pada posisi yang sama sehingga tidak memiliki kemampuan untuk menahan rusaknya struktur sekunder suatu enzim. Sama juga halnya pada Gly155Cys-Gly158Cys yang terletak pada coil dan 3-10 helix , ikatan ini berfungsi untuk
mengikat hanya sebatas β-sheet ke 5 dan β-sheet ke 6, sehingga kestabilannya masih rendah jika dibandingkan dengan mutan Ile135Cys-Hsd156Cys.
I le 1 3 5
H sd1 5
(a)
Le u 1 4 5
Ala 1 4 6
(b)
Gly1 5 5
Gly 1 5 8
(c)Gambar 4.8 Posisi calon mutan berdasarkan ikatan disulfida (a) Ile135
21 Untuk mutan Ile151Val adalah mutan yang diperoleh berdasarkan analisis interaksi hidrofobik. Residu Ile 151 ini terletak pada β-sheet ke 6, sehingga melalui preferensi konformasi (Biological magnetic resonance data bank) yang terlampir (Lampiran 6) residu ini memiliki karakteristik Strong former dalam membentuk konformasi β-sheet dengan kemampuan sebesar 1.60, sedangkan residu 151Val memiliki kemampuan sebagai Strong former β-sheet sebesar 1.65 sehingga residu Valin lebih stabil jika dibandingkan dengan residu Isoleusin. Sedangkan pada mutan lainnya yaitu Ile151Ser akan menurunkan kestabilan karena residu Isoleusin yang sifatnya Strong former β-sheet akan berubah menjadi Weak former β-sheet . Untuk mutan Ile73Leu (berada pada β -sheet ke 3) akan merubah sifat residu Isoleusin yang tadinya Strong former β -sheet menjadi Breaker former β-sheet sehingga kestabilan menjadi rendah. Untuk mutan Ile73Gln (berada pada β-sheet ke 3) akan merubah sifat residu Isoleusin yang tadinya Strong former β-sheet menjadi Former β-sheet sehingga kestabilannya masih dibawah mutan Ile151Val. Untuk mutan Val59Ile (berada pada α-helix ke 2) akan merubah sifat residu Valin yang tadinya Former α-helix menjadi Weak former α-helix sehingga kestabilan menjadi rendah. Untuk mutan Val59Thr (berada pada α-helix ke 2) akan merubah sifat residu Valin yang tadinya Former α-helix menjadi Indifferent α-helix sehingga kestabilan menjadi rendah juga. Walaupun nilai ΔΔGsolv negatif tetapi nilainya masih lebih rendah dibandingkan nilai ΔΔGsolv pada Ile151Val, sehingga Val59Thr tidak dipilih untuk diuji dengan simulasi MD.
V a l 5 9
I le 7 3
I le 1 5 1
Gambar 4.9 Posisi calon mutan berdasarkan interaksi hidrofobik : Val59 (Pink) , Ile73 (Abu-Abu) dan Ile 151 (Hijau)
22
Analisis Kestabilan Termal Mutan Bacillus subtilis
Untuk mengetahui parameter yang paling mempengaruhi kestabilan pada enzim B.subtilis maka dilakukan simulasi dinamika molekul untuk mutan yang telah diuji dengan simulasi FEP. Pada enzim Bacillus subtilis ada 3 variasi yaitu mutan-1, mutan-2 dan mutan-3. Mutan-1 adalah hasil analisa dari interaksi hidrofobik yaitu mengubah Isoleusin pada residu 151 menjadi Valin, mutan-2 adalah hasil analisa dari interaksi elektrostatik yaitu mengubah Glutamat pada residu 65 menjadi Aspartat dan mutan-3 adalah hasil analisa dari ikatan disulfida yaitu mengubah Isoleusin pada residu 135 dan histidin pada residu 156 menjadi sistein.
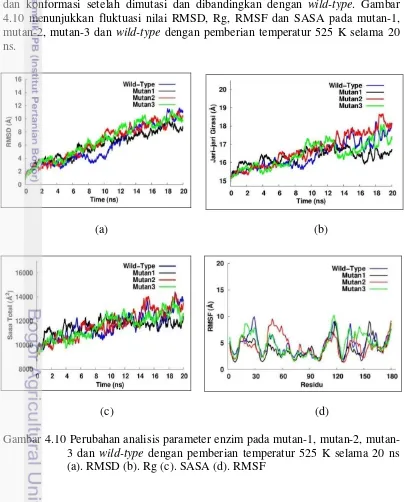
Selanjutnya diamati proses unfolding dengan menganalisis sejumlah parameter yaitu RMSD, Jari-jari Girasi (Rg), SASA, RMSF, Struktur Sekunder dan konformasi setelah dimutasi dan dibandingkan dengan wild-type. Gambar 4.10 menunjukkan fluktuasi nilai RMSD, Rg, RMSF dan SASA pada mutan-1, mutan-2, mutan-3 dan wild-type dengan pemberian temperatur 525 K selama 20 ns.
(a) (b)
(c) (d)
23 Analisis struktur sekunder juga menunjukkan bahwa mutan-1 lebih stabil dibandingkan dengan wild-type mutan-3, maupun mutan-2. Pada Gambar 4.11 terlihat bahwa mutan-1 hingga konformasi akhir memiliki komposisi beta sheet dan alpa-helix yang lebih banyak dibandingkan wild-type, mutan-2 dan mutan-3, sehingga struktur sekunder yang dimilikinya lebih stabil. Mutan-3 memiliki komposisi alpa-helix yang lebih besar tetapi komposisi beta-sheet sama dengan kondisi wild-type dan mutan-2 memiliki komposisi beta-sheet lebih besar tetapi komposisi alpa-helix rendah.
(a) (b) Gambar 4.11 Perubahan komposisi struktur sekunder pada mutan-1, mutan-2,
mutan-3 dan wild-type dengan pemberian temperatur 525 K selama 20 ns (a). Alpa-helix (b). Beta-sheet
Dari hasil analisis simulasi diatas dapat disimpulkan bahwa interaksi hidrofobik adalah interaksi yang memiliki pengaruh yang signifikan pada kestabilan termal enzim lipase Bacillus subtilis.
Untuk mengamati seberapa kuat pengaruh ikatan hidrofobik pada kestabilan termal enzim B.subtilis saat terjadi unfolding, maka pada mutan-1 yang merupakan mutan terbaik dianalisa dengan melakukan simulasi dinamika molekuler. Simulasi dinamika molekuler dilakukan pada suhu normal yaitu sekitar 300 K selama 2 ns untuk dua kondisi yakni wild-type dan mutan.
(a) (b)
24
Analisa faktor-β dilakukan untuk melihat perubahan pada residu penyusun enzim setelah dimutasi apakah memiliki perubahan dibandingkan kondisi wild-type nya. Pada gambar 4.13 terlihat nilai faktor-β dari enzim lipase Bacillus subtilis mutan-1, wild-type dan eksperimen. Pada residu 16 nilainya diturunkan dari 75 Å menjadi 25 Å. Begitu juga dengan residu 38 dirubah dari 24 Å menjadi 8 Å. Walaupun terdapat juga peningkatan pada residu 120 dari 38 Å menjadi 62 Å dan pada residu 130 nilainya berubah dari 8 Å menjadi 64 Å. Ini terjadi karena fleksibilitas residu didalam air, tetapi pola yang terbentuk masih sama dan hal ini menandakan kondisi enzim setelah dimutasi masih memiliki sifat yang sama dengan tanpa dimutasi. Hanya ada perbedaan sifat pada residu 16, 38, 120, 130 dan 156.
(16)
(130)
(120)
(38) (156)
Gambar 4.13 Perbandingan nilai faktor-β Mutan-1 (Biru), faktor-β wild-type (Orange) dan faktor-β eksperimen pada temperatur 300 K
Selanjutnya dilakukan simulasi dinamika molekuler pada temperatur unfolding yaitu 500 K selama 50 ns untuk melihat seberapa jauh pengaruh mutasi yang dilakukan terhadap kestabilan termal enzim lipase B. subilis mampu melalui analisa parameter RMSD, Rg, RMSF, SASA dan struktur sekunder.
Nilai RMSD, Rg, RMSF dan SASA pada gambar 4.14 menunjukkan bahwa mutan-1 lebih stabil dibandingkan wild-type. Hal ini ditunjukkan oleh nilai konformasi akhir dari RMSD, Rg, RMSF dan SASA pada mutan-1 lebih kecil dibandingkan nilai wild-type.
Nilai RMSD mutan-1 mampu diturunkan sebesar 7.7 Å, nilai Rg mutan-1 mampu diturunkan sebesar 3.41 Å dan nilai SASA mutan-1 mampu diturunkan sebesar 1 504 Å2 (Tabel 4.4). Penurunan yang signifikan pada nilai RMSD, Rg dan SASA pada mutan-1 dibandingkan wild-type menunjukkan adanya peningkatan kestabilan struktur protein.
25
(a) (b)
(c) (d)
Gambar 4.14 Perubahan analisis parameter enzim pada mutan-1 dan wild-type dengan pemberian temperatur 500 K selama 50 ns (a). RMSD (b). Rg (c). SASA (d). RMSF
Tabel 4.4 Nilai parameter enzim pada suhu 500 K saat 50 ns
Untuk parameter struktur sekunder, yaitu struktur alpa-helix, mutan-1 hanya mampu mempertahankan struktur ini sekitar 23% dibandingkan wild-type yang mampu mempertahankan sekitar 26% struktur. Perbedaannya yang tidak terlalu jauh ini menandakan bahwa untuk alpa-helix mutasi yang dilakukan tidak berpengaruh secara signifikan pada struktur sekunder alpa-helix nya. Sedangkan untuk struktur beta-sheet mutan-1 mampu mempertahankan strukturnya sekitar 16% dibandingkan wild-type yang sama sekali tidak memiliki struktur beta-sheet (Tabel 4.5). Sehingga mutan-1 mampu mengatasi kerusakan struktur beta-sheet sehingga kemungkinan enzim masih tetap bisa berfungsi sebagaimana mestinya.
Temperatur
(K) RMSD (Å) (Å) Rg SASA Total (Å2)
Wild-type 16.45 20.24 14 029
26
Sehingga jika dibandingkan dengan kondisi native enzim, yang memiliki alpa-helix sebesar 37% dan beta sheet sebesar 19% maka mutan-1 mampu mempertahankan kestabilan enzim lebih baik daripada wild-type ketika diganggu dengan pemberian temperatur yang cukup tinggi.
(a)
(b
Gambar 4.15 Perubahan Komposisi struktur sekunder pada mutan-1 dan wild-type dengan pemberian temperatur 500 K selama 50 ns (a). Alpa-helix (b). Beta-sheet.
Tabel 4.5 Perubahan komposisi struktur sekunder pada temperatur 500 K saat 50 ns
Temperatur
(K) Alpa-Helix Struktur Sekunder (%)
Beta-Sheet (%)
Turn (%)
Coil (%)
Beta-Bridge (%) Native
Wild-type
39
26 19 0 15 52 25 29 2 2
Mutan-1 23 16 37 22 2
(a) (b)
27
SIMPULAN DAN SARAN
Simpulan
Enzim lipase Bacillus subtilis mengalami unfolding pada temperatur 500 K saat 28 ns. Residu-residu yang bertanggung jawab terhadap kestabilan termal enzim Bacillus subtilis berdasarkan analisa interaksi elektrostatik yaitu Glutamat pada residu 65, berdasarkan ikatan hidrofobik yaitu Isoleusin pada residu 151, berdasarkan ikatan disulfida yaitu Isoleusin pada residu 135 dan Histidin pada residu 156.
Tiga mutan berhasil dirancang yaitu mutan-1 adalah Iso151Val dari hasil analisa ikatan hidrofobik, mutan-2 adalah Glu65Asp dari hasil analisa interaksi elektrostatik dan mutan-3 adalah Ile135Cys dan Hsd145Cys dari hasil analisa ikatan disulfida.Dari hasil analisis parameter RMSD, Jari-jari Girasi (Rg), SASA, RMSF, Struktur Sekunder dan konformasi tersebut disimpulkan bahwa Mutan-1 yaitu Iso155Val adalah mutan yang paling stabil diantara ketiga mutan tersebut. Sehingga Interaksi Hidrofobik memiliki pengaruh yang sangat signifikan pada kestabilan termal Enzim Lipase Bacillus subtilis.
Pengaruh mutasi dari hasil analisa Interaksi hidrofobik yaitu pada mutan-1 (Iso151Val) pada suhu 500 K mampu meningkatkan stabilitas termal yang dibuktikan oleh penurunan nilai parameter RMSD, Rg, RMSF, SASA dan Struktur Sekunder yaitu RMSD mampu diturunkan sebesar 7.7 Å, nilai Rg mampu diturunkan sebesar 3.41 Å dan nilai SASA mampu diturunkan sebesar 1504 Å2. Untuk struktur beta-sheet, mampu dipertahankan sekitar 16% dibandingkan wild-type yang sama sekali tidak memiliki struktur beta-sheet. Untuk struktur alpa-helix, mutan-1 tidak begitu mempengaruhi komposisi alpa-helix nya.
Saran
28
DAFTAR
PUSTAKA
Allen MP, Tildesley DJ. 1989. Computer Simulation of Liquids, Oxford University Press, USA.
Aunstrup KO, Andressen, Falch EA, Nielsen. 1979. Production of Microbial Enzymes. Microbial Technology. Vol. 1. Academic Press Inc. New York Becker OM, Mackerell ADJr, Roux B, Watanabe M. 2001. Computational
Biochemistry and Biophysics. New York: Marcel Dekker.
Berman HM, Westbrook J, Feng Z, Gilliland G, Bhat TN, Weissig H, Shindyalov IN, Bourne PE. 2000. The Protein Data Bank. Nucleic Acids Res. 28: 235-242. Betz SF. 1993. Disulfide bonds and the stability of globular proteins. Protein Science. 2: 1551-1558.
Brooks BR, Bruccoleri RE, Olafson BD, States D J, Swaminathan S, Karplus M. 1983. CHARMM: A Program for Macromolecular 138 Energy, Minimization, dan Dynamics Calculations. J. Comput. Chem. 4(2):187-217.
Buchner J, Kiefhaber T. 2005. Protein folding handbook. Part I. Weinheim: WILEY-VCH Verlag GmbH & Co. KGaA.
Bui JM, Gsponer J, Vendruscolo M, Dobson CM. 2009. Analysis of sub- and supra- motions in protein Gβ1 using molecular dynamics simulations. Biophyics Journal. 97: 2513-2520
Bussi G, Donadio D, Parrinello M. 2007. Canonical sampling through velocity rescaling. J. Chem. Phys. 126: 014101
Corzo G, Revah S. 1999. Production and characteristics of the lipase from Yarrowia lipolytica 681. Bioresource Technology. 70: 173–180.
Coutsias EA, Seok C, dan Dill KA. 2004. Using quaternions to calculate RMSD, J. Comput. Chem. 25(15) : 1849-1857.
Daniel RM, Cowan DA. 2000. Biomolecular stability dan life at high temperatures, Cell. Mol. Life Sci. 57: 250-264.
Darden T, York D, Pedersen L. 1993. Particle mesh Ewald: An N. log(N) method for Ewald sums in large systems. J. Chem. Phys. 98(12) : 10089-10092.
Dartois V, Baulard A, Schanck K, Colson C. 1992. Cloning, nucleotide sequence and expression in Escherichia coli of a lipase gene from Bacillus subtilis 168. Biochim Biophys Acta. 3: 253–260.
Das R, Gerstein M. 2000. The stability of thermophilic protein : a study based on comprehensive genome comparison. Funct. Integr. Genom. 1: 76-88. Day R, Bennion BJ, Ham S, Daggett V. 2002. Increasing temperature
accelerates protein unfolding without changing the pathway of unfolding.J. Mol. Biol. 322: 189-203
Dombkowski AA. 2003. Disulfide by DesignTM : a computational method for the rational design of disulfide bonds in proteins. Bioinformatics. 19: 1852 1853
29 Eggert T, Pouderoyen G, Dijkstra B, Jaeger K. 2001. Lipolytic enzymes LipA and
LipB from Bacillus subtilis differ in regulation of gene expression,
Feller SE, Zhang Y, Pastor RW, Brooks BR. 1995. Constant pressure molecular dynamics simulation: The Langevin piston method. J. Chem. Phys. 103 (11): 4613-4621.
Fodiloglu S, Erkmen O. 1999. Lipase production by Rhizopus oryzae growing on different carbon and nitrogen sources. Journal of the Science of Food and Agriculture. 79 : 1936–1938.
Frishman D, Argos P. 1995. Knowledge-based protein secondary structure assignment.Proteins: Structure, Function, and Genetics. 23: 566-579. Gertie van P, Thorsten E , Karl-Erich J.,BaukeW, Dijkstra. 2001. The Crystal
Structure of Bacillus subtilis Lipase : A Minimal α/β Hydrolase Fold
Enzyme. J. Mol. Biol. 309: 215 – 226.
Ghoufi A, Bonal C, Morel JP, Morel-Desrosiers N, Malfreyt P. 2004. Gibbs Free Energy Perturbation Calculations, An Application to the Binding of Alkylammonium Cations by Water-Soluble Calixarene. J. Phys. Chem. B. 108 : 11744-11752.
Gianese G, Bossa F, Pascarella S. 2002. Comparative structural analysis of psychoprilic dan meso- dan thermophilic enzymes. Proteins. 2: 236-249.
Gordon JC, Myers JB, Folta T, Shoja V, Heath LS, Onufriev A. 2005. H++: a server for estimating pKas and adding missing hydrogens to macromolecules. Nucleic Acids Research. 33 : 368 – 371.
Haki GD, Rakshit SK. 2003. Developments in industrially important thermostable enzymes: a review. Bioresource Technology. 89 : 17-34. Han Z, Han S, Zheng S, Lin Y. 2009. Enhancing thermostability of a
Rhizomucor mieheilipase by engineering a disulfide bond and displaying on the yeast cell surface. Appl. Microbiol. Biotechnol. 85: 117-126.
Harwood, CR. 1992. Bacillus subtilis and its relatives: molucular biological and industrial work hourse. Trends Biotechnol. 10 : 247-256.
Humphrey W, Dalke A, Schulten K. 1996. VMD: visual molecular dynamics. J. Mol. Graph, 14(1) : 27 – 38.
Jaenicke R, Böhm G. 1998. The stability of proteins in extreme environments. Curr. Opin. Struct. Biol. 6: 738-748.
Jaenicke R, Závodszky P. 1990. Proteins under extreme physical conditions. FEBS Lett. 2: 344-349.