ANALISIS EKSPRESI GEN PADA EMBRIOGENESIS
SOMATIK DI TANAMAN KELAPA SAWIT
NURYANTI SYARIYANTO
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa Tesis yang berjudul Analisis Ekspresi Gen pada Embriogenesis Somatik di Tanaman Kelapa Sawit adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor dan PT SMART Tbk.
Bogor, Mei 2016
Nuryanti Syariyanto
RINGKASAN
NURYANTI SYARIYANTO. Analisis Ekspresi Gen Pada Embriogenesis Somatik di Tanaman Kelapa Sawit. Dibimbing oleh UTUT WIDYASTUTI dan NURITA TORUAN-MATHIUS.
Teknik kultur jaringan merupakan salah satu teknik perbanyakan tanaman kelapa sawit untuk skala produksi (mass production) karena dapat menghasilkan tanaman yang memiliki sifat yang sama dengan induknya (true to type). Namun, teknik ini masih kurang efisien karena keberhasilan pembentukan embrio somatik-nya yang masih rendah (3-6%), oleh karena itu perlu dilakukan identifikasi gen-gen yang terlibat dalam embriogenesis pada tanaman kelapa sawit.
Pada penelitian sebelumnya dengan teknik microarray, diperoleh beberapa gen yang potensial dan terlibat dalam pembentukan embrio somatik di tanaman kelapa sawit, namun ekspresi gen dari kandidat gen-gen tersebut belum divalidasi, oleh karena itu penelitian ini bertujuan: (1) seleksi gen-gen terkait embriogenesis somatik dari hasil
microarray berdasarkan pendekatan Bioinformatik dan (2) menganalisis ekspresi gen-gen tersebut menggunakan qPCR. Penelitian ini diharapkan diperoleh kandidat gen-gen-gen-gen terkait embriogenesis somatik yang dapat digunakan sebagai marka untuk deteksi dini kalus-kalus yang memiliki kemampuan embriogenik pada tanaman kelapa sawit.
Penelitian secara garis besar dibagi menjadi dua bagian, yaitu: (1) seleksi gen-gen terkait embriogenesis somatik dan (2) menganalisis ekspresi gen-gen tersebut menggunakan qPCR. Pada tahap seleksi gen dengan Bioinformatik, gen-gen hasil dari analisis dengan microarray diseleksi berdasarkan nilai ekspresinya (fold change), fungsi gen, dan faktor-faktor yang mempengaruhi embriogenesis.
Analisis ekpsresi gen dilakukan dengan menggunakan sampel kalus (nodular callus) dan embrio somatik (coleoptilar stage) yang merupakan hasil kultur jaringan dari tiga progeni tanaman kelapa sawit (Tenera). RNA dari dua jenis sampel diekstraksi, kemudian diubah menjadi cDNA utas tunggal. Selanjutnya dilakukan analisis ekspresi gen menggunakan teknik qPCR. Ekspresi gen dikuantifikasi dengan metoda Relative Quantification Standart Curve. Pada analsis tersebut kalus digunakan sebagai kalibrator sampel (kontrol) dan gen 40S ribosomal protein (40s) digunakan gen pembanding (reference gene).
Hasil seleksi gen-gen yang terlibat dalam embriogenesis somatik melalui pendekatan Bioinformatik diperoleh sebanyak sepuluh kandidat gen yang terdiri atas: tujuh gen yang up-regulated dan tiga gen down-regulated. Kesepuluh gen tersebut kemudian dilakukan analisis melt curve sehingga diperoleh empat gen yang primernya terbukti spesifik dan dapat digunakan dalam analisis qPCR, yaitu: IAA-amino acid hydrolase ILR1-like1 (ilr1), late embryogenesis abundant (lea2), 26S proteasome non-ATPase (26sp), dan trehalose phosphate synthase (tps6). Hasil analisis ekspresi gen dengan menggunakan qPCR menunjukkan bahwa gen ilr1 dan lea2 terekspresi lebih tinggi pada embrio somatik (coleoptilar stage) dibandingkan dengan sampel kalus (kontrol). Meskipun tingginya ekspresi kedua gen tersebut berbeda pada setiap progeni. Kedua gen ini berpotensi digunakan sebagai penanda untuk mendeteksi adanya embrio somatik (coleoptilar stage).
SUMMARY
NURYANTI SYARIYANTO. Gene Expression Analysis of Somatic Embryogenesis in Oil Palm. Supervised by UTUT WIDYASTUTI and NURITA TORUAN-MATHIUS.
Tissue culture is one of techniques to multiply elite palms for production true to type generation. However, this technique is still susceptible by low embryogenesis rate (3-6%). Therefore, identification of genes related to embryogenesis in oil palm is needed to improve the efficiency in oil palm tissue culture.
Previous research with microarray technique showed some potential candidate genes related to oil palm somatic embryogenesis, but the expression of those genes has not yet been validated. Thus, the aim of this study is to: (1) select genes related in somatic embryogenesis from microarray result based on Bioinformatic and (2) validate the expression level of those genes using quantitative real-time PCR (qPCR). In the future, the candidate genes might be used in developing molecular marker for early detection to differentiate the embryogenic and non-embryogenic cultures.
This research consist of two parts: (1) selection of genes related in somatic embryogenesis based on Bioinformatic and (2) analysis expression of genes related in somatic embryogenesis used qPCR. In genes selection, embryogenesis related genes were choosen based on fold change, function of the genes and factors of embryogenesis.
In gene expression analysis, nodular callus and somatic embryo of E. guineensis
Tenera at coleoptilar stage were collected from three palms for RNA extraction. The first strand cDNA was synthesized from RNA and used for gene expression analysis. The expression of embryogenesis-related genes were analyzed using Relative Quantification Standard Curve method. Callus was used as calibrator sample and 40S ribosomal protein S27-2 (40s) gene as reference gene.
Ten genes related to embryogenesis were selected based on Bioinformatic, which consist of seven up-regulated genes and three down-regulated genes. Melt curve analysis of those ten primer of target genes showed that only four primers was specific and can be used for qPCR analysis, such as: IAA-amino acid hydrolase ILR1-like1 (ilr1), late embryogenesis abundant (lea2), 26S proteasome non-ATPase (26sp), and trehalose phosphate synthase (tps6). qPCR result showed that ilr1 and lea2 genes were highly expressed on somatic embryo (coleoptilar stage) in compared to callus, but expression of those genes was different in each progeny. ilr1 and lea2 genes may potentially be involved in oil palm somatic embryogenesis.
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian,
penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB dan PT SMART Tbk.
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Bioteknologi
ANALISIS EKSPRESI GEN PADA EMBRIOGENESIS
SOMATIK DI TANAMAN KELAPA SAWIT
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2016
ii
iii Judul Tesis : Analisis Ekspresi Gen Pada Embriogenesis Somatik di Tanaman
iv
PRAKATA
Puji dan syukur penulis panjatkan kepada Tuhan Yang Maha Esa atas segala karunia-Nya sehingga penelitian ini berhasil diselesaikan. Penelitian yang berjudul Analisis Ekspresi Gen Pada Embriogenesis Somatik di Tanaman Kelapa Sawit, ini ditujukan sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada Program Studi Bioteknologi Sekolah Pascasarjana IPB Bogor.
Penulis mengucapkan terima kasih kepada semua pihak yang telah membantu dalam penyusunan penelitian ini baik secara langsung maupun tidak langsung. Ucapan terima kasih penulis sampaikan kepada Dr Ir Utut Widyastuti, MSi, sebagai ketua komisi pembimbing dan Dr Nurita Toruan-Mathius, MS, sebagai anggota komisi pembimbing atas arahan, bimbingan, serta motivasi yang diberikan kepada penulis dari awal penelitian hingga penyusunan tesis ini. Kepada Prof Ir Sudarsono, MSc PhD atas saran, masukan serta kesediaannya menjadi penguji luar komisi, dan kepada Dr. dr Sri Budiarti atas kesediaannya sebagai moderator dalam ujian tesis. Terima kasih juga penulis sampaikan kepada Prof Dr Ir Suharsono, DEA selaku Ketua Program Studi Bioteknologi IPB yang telah memberi arahan dan bimbingan selama penulis menempuh studi S2 Bioteknologi. Kepada Prof Ir Antonius Suwanto, MSc PhD atas perhatian, motivasi dan izin sit in di mata kuliah Ekspresi Gen dan Pengendaliannya.
Penulis juga menyampaikan terima kasih kepada Bapak Jo Daud Dharsono selaku Head of Upstream PT SMART Tbk yang telah memberi izin dan kesempatan kepada penulis untuk melanjutkan studi S2, Bapak Dr Tony Liwang selaku Division Head dan Ibu Lisa Muliani, MSc selaku Dept. Head Tissue Culture yang telah memberikan kesempatan, motivasi dan dukungan selama studi. Kepada Bapak Dr Condro Utomo, Dr Roberdi, Reno Tryono, Zulfikar, Hadi, Ibu Angeli Karmorahardjo, dan Widyah Budinarta yang telah berperan dalam mengkaji serta memberikan saran dalam rangka penyusunan publikasi. Kepada Maharani Anischan, Andree, Fajri, Analekta, Wulan A, dan Sigit atas sharing pengetahuan mengenai qPCR, kepada Chris Darmawan dan Bu Widyartini atas sharing
pengetahuan dalam perancangan primer, kepada Yogo AN dan Dwi Yono atas pelatihan analisa data statistik, juga kepada Intana dan Lutfi atas pelatihan pengunaan alat di laboratorium Bioteknologi.
Ungkapan terima kasih juga disampaikan kepada kedua orang tua, Agus Syariyanto dan Wong Wan Jin, kedua kakak serta adik atas doa dan dukungannya selama studi. Terima kasih juga kepada Tami, Fitria, Bu Sussi, Pak Hadi, Yeni, Qurrota A’yun, teman-teman di Sekolah Pascasarjana Bioteknologi angkatan 2012 serta rekan-rekan dari PT SMART Tbk atas dukungan dan bantuannya selama penelitian studi S2 ini.
Penulis menyadari bahwa penelitian dan penyusunan tesis ini masih jauh dari sempurna namun semoga hasil penelitian ini dapat bermanfaat.
Bogor, Mei 2016
v
Analisis Hasil Microarray dengan Bioinformatik 8
Perancangan Primer 10
Analisis Ekspresi Gen dengan qPCR 11
3 METODE 14
Bahan 14
Prosedur Kerja 15
Seleksi Gen menggunakan Pendekatan Bioinformatik 15
Isolasi RNA dan Sintesis cDNA Sampel 16
Perancangan, Optimasi, dan Validasi Primer 16
Pembuatan Standar qPCR 17
Seleksi Gen Pembanding 17
Analisis Ekspresi Gen dengan qPCR 17
4 HASIL DAN PEMBAHASAN 19
Seleksi Gen Embriogenesis dengan Pendekatan Bioinfromatik 19
Isolasi RNA dan Sintesis cDNA Sampel 21
Perancangan, Optimasi, dan Validasi Primer 24
Seleksi Gen Pembanding 29
Analisis Ekspresi Gen dengan qPCR 30
vi
DAFTAR TABEL
1 Gen-gen yang up-regulated di embrio somatik kelapa sawit
(Budinarta et al. 2012) 9
2 Informasi asal tanaman dari sampel kalus dan embrio somatik 14
3 Kandidat gen embriogenesis somatik 19
4 Fungsi dari kandidat gen embriogenesis somatik 19
5 Konsentrasi, kemurnian, serta nilai intergritas RNA dari sampel kalus dan embrio somatik pada ketiga tanaman kelapa sawit 22
6 Sekuen nukleotida dari primer gen pembanding (primer I) 24
7 Sekuen nukleotida dan hasil optimasi primer gen target (primer I) 24
8 Hasil BLASTN gen target dan pembanding primer I 25
9 Sekuen nukleotida dan hasil optimasi primer gen target (primer II) 26
10 Sekuen nukleotida primer gen pembanding (primer II) 27
11 Hasil BLASTN gen target dan pembanding primer set II 28
12 Hasil BLASTN dan BLASTX pada primer set II 28
13 Persamaan, nilai R2, dan persentase efisiensi dari masing-masing kurva standar gen target dan gen pembanding 31
14 Nilai Cq pada LOD dan NTC dari masing-masing gen target dan
gen pembanding 32
DAFTAR GAMBAR
1 Alur proses kultur jaringan tanaman kelapa sawit 5
2 Diagram skematik jalur pembentukan embrio somatik 6
3 Fase perkembangan embrio somatik dari kultur kelapa sawit 7
4 Gen-gen yang terlibat dalam proses embriogenesis somatik 8
5 Kurva amplifikasi pada qPCR 12
6 Efisiensi Amplifikasi PCR 12
7 Sampel kalus dan embrio somatik 14
8 Diagram alir kegiatan penelitian 15
9 Heat map dari 1.083 gen hasil seleksi microarray 20
10 Interaksi antar gen up-regulated dan down-regulated hasil analisis
dengan Cytoscape 21
11 RNA hasil isolasi dari kalus (C) dan embrio somatik (E) tanaman
kelapa sawit 22
12 Hasil analisis integritas RNA kalus 23
13 Hasil analisis integritas RNA embrio somatik 23
14 Hasil sintesis cDNA dari sampel kalus dan embrio somatik 24
15 Hasil optimasi primer set II yang tidak spesifik (terdapat dimer) 27
16 Hasil analisis Melt curve pada gen target dan gen pembanding 28
vii
18 Perbandingan nilai Cq dari ketiga gen (act3, 40s, dan sodm) pembanding pada sampel kalus (Cal) dan embrio somatik (Emb) 30
19 Kurva standar dari masing-masing gen target 31
20 Pita hasil amplifikasi qPCR dari keempat gen target (palm 1) 32
21 Hasil analisis ekspresi gen sampel kalus dan embrio somatik 33
22 Jalur biosintesis auksin (Tryptophan-independent pathways) 34
23 Sistem ekspresi gen auksin dan gen 26S Proteasome (SCFTIR1) 34
24 Transport auksin pada tahap awal pembentukan embrio
Arabidopsis. 35
25 ABA-mediated abiotic stress response 36
26 Trehalose pathway in plants 37
DAFTAR LAMPIRAN
1 Data hasil pensejajaran sekuen cDNA hasil sekuensing dengan sekuen cDNA masing-masing gen hasil design primer I 44
2 Data hasil pensejajaran sekuen cDNA hasil sekuensing dengan sekuen cDNA masing-masing gen hasil design primer II 50
3 Hasil analsis qPCR dari keempat kandidat gen embriogenesis 52
4 Hasil analisis Statistik: One-way Anova dan Dunnett t-tests dari gen
E. guineensis IAA-amino acid hydrolase ILR1-like 1 (ilr1) 53
5 Hasil analisis Statistik: One-way Anova dan Dunnett t-tests dari gen
1
PENDAHULUAN
Latar Belakang
Kelapa sawit merupakan tanaman yang paling produktif dalam menghasilkan minyak nabati (±4 ton ha-1 tahun-1) dibandingkan dengan tanaman lainnya seperti kanola, bunga matahari, dan kedelai (<1 ton ha-1 tahun-1) (Oil World 2013). Kebutuhan akan minyak kelapa sawit ini terus meningkat setiap tahunnya.
Perbanyakan tanaman kelapa sawit dengan teknik kultur jaringan diperlukan dalam rangka meningkatkan jumlah bibit dan produktivitas tanaman kelapa sawit. Menurut Soh (2012), teknik kultur jaringan mampu meningkatkan oil yield sekitar 12-15% dengan mengkulturkan sebanyak 5% dari ortet terpilih serta dapat meningkatkan sekitar 30% dengan mengkulturkan kembali (recloning) hasil dari klon-klon yang unggul. Teknik kultur jaringan ini masih memiliki kendala, yaitu rendahnya persentase pembentukan kalus (19%) (Corley & Tinker 2003) dan pembentukan embrio somatik (3-6%) (Wooi 1995).
Beberapa faktor yang dapat mempengaruhi keberhasilan embriogenesis pada kelapa sawit antara lain genotipe (Sanputawong & Te-chato 2008), media kultur, zat pengatur tumbuh, sumber karbon (Kramut & Te-Chato 2010), kompetensi sel, dan kondisi lingkungan fisik kultur. Banyak penelitian yang telah dilakukan untuk mengatasi kendala tersebut namun belum ada yang dapat secara efisien dan cepat dalam meningkatkan keberhasilan embriogenesis.
Pendekatan molekuler dengan menggunakan marka merupakan pendekatan yang paling efektif dan cepat dalam membedakan kalus embriogenik dari kalus yang non-embriogenik. Pembuatan marka ini diperlukan pemahaman kultur jaringan di tingkat molekuler. Gen-gen yang terkait embriogenesis pada tanaman kelapa sawit telah berhasil diidentifikasi dengan menggunakan beberapa teknik analisis molekuler, antara lain: DNA Chip Technology/microarray (Low et al. 2006), Expressed Sequence Tags (ESTs) (Lin et al. 2009) dan cDNA Amplified Fragment Length Polymorphism (cDNA-AFLP) (Pattarapimol et al. 2015).
Shariff et al. (2008) melaporkan gen-gen terkait embriogenesis pada kelapa sawit antara lain: lipid transfer protein homolog (wbp1a), somatic embryogenesis receptor kinase 1 (serk1), dan defensin (egad1). Pada tanaman lain (wortel dan
Arabidopsis), gen-gen embriogenesis terdiri atas Late Embryogenic Abundant (lea),
Somatic Embryogenesis Receptor Kinase (serk), Agamous-like 15 (agl15), Baby Boom (BBM), Leafy Cotyledon 1 (lec1), Fusca3 (fus3), dan Leafy Cotyledon 2
(lec2) (Ikeda et al. 2006). Gen-gen yang terkait dalam embriogenesis somatik juga banyak dibahas dalam ulasan Chugh & Khurana (2002) dan Elhiti et al. (2013).
2
Perumusan Masalah
Perbanyakan kelapa sawit dengan teknik kultur jaringan masih terkendala karena rendahnya persentase pembentukan embrio somatik (sekitar 3-6%), oleh karena itu perlu dilakukan analisis terhadap gen-gen yang terlibat pada embriogenesis somatik. Pada penelitian sebelumnya telah dilakukan analisis ekspresi gen pada embriogenesis somatik tanaman kelapa sawit dengan teknik
microarray, namun belum dilakukan validasi untuk gen yang terlibat pada
coleoptilar stage. Sehubungan dengan hal tersebut, maka dilakukan penelitian untuk mendapatkan gen yang dapat digunakan sebagai penanda ekspresi gen-gen yang terkait pada embriogen-genesis somatik.
Tujuan Penelitian
Penelitian ini bertujuan untuk mendapatkan gen-gen terkait embriogenesis somatic (coleoptilar stage) dari hasil microarray berdasarkan pendekatan Bioinformatik dan berdasarkan analisis ekspresi gen menggunakan qPCR.
Manfaat Penelitian
Manfaat dari penelitian ini antara lain adalah diperoleh kandidat gen-gen terkait embriogenesis yang dapat digunakan sebagai marka pembentukan embrio somatik pada tahap kalus. Pengunaan gen sebagai marka juga bertujuan agar dapat dilakukan seleksi dini terhadap kalus embriogenik dan kalus nonembriogenik, sehingga dapat meningkatkan persentase pembentukan somatik embriogenesis. Pada akhirnya teknik perbanyakan tanaman kelapa sawit dapat menjadi lebih cepat dan efisien.
Hipotesis
3
2
TINJAUAN PUSTAKA
Tanaman Kelapa Sawit
Tanaman Kelapa sawit (E. guineensis Jacq.) berasal dari Benua Afrika. Pemberian nama E. guineensis Jacq. ini diberikan oleh seorang botanist yang bernama Jacquin pada 1763. Elaeis berasal dari Bahasa Yunani, elaion yang berarti minyak dan guineensis yang menunjukkan tempat asalnya, yaitu pantai Guinea (Corley & Tinker 2003). Kelapa sawit pertama kali diperkenalkan di Indonesia oleh pemerintah kolonial Belanda pada tahun 1848. Perkebunan kelapa sawit pertama di Indonesia berlokasi di Pantai Timur Sumatera (Deli) dan Aceh (Fauzi et al. 2002).
Tanaman kelapa sawit secara klasifikasi termasuk dalam divisi
Spermatophyte, subdivisi Angiospermae, kelas Monocotyledonae, ordo Palmales, famili Palmaceae, dan Genus Elaeis. Kelapa sawit memiliki empat spesies, yaitu
E. guineensis, E. oleifera, E. odora, dan E. madagascariensis Becc. (Corley & Tinker 2003).
Bagian tanaman kelapa sawit yang bernilai ekonomi tinggi adalah buahnya yang tersusun dalam sebuah tandan yang biasa disebut dengan TBS (tandan buah segar). Buah sawit terdiri atas daging buah/mesocarp menghasilkan minyak sawit kasar (crude palm oil atau CPO) sebanyak 20-24% dan bagian inti sawit menghasilkan minyak inti sawit (palm kernel oil atau PKO) 3-4%. Minyak sawit dan minyak inti sawit umumnya digunakan untuk pangan dan nonpangan (Sunarko 2007).
Pada bidang pangan, minyak sawit atau minyak inti sawit digunakan sebagai bahan untuk membuat minyak goreng, lemak pangan, margarin, lemak khusus (substitusi cacao butter), kue, biskuit, atau es krim. Produk pangan ini umumnya dihasilkan melalui proses fraksinasi, rafinasi, dan hidrogenasi. Pada nonpangan, minyak sawit atau minyak inti sawit digunakan sebagai bahan untuk membuat sabun, deterjen, surfaktan, pelunak (plasticizer), pelapis (surface coating), pelumas, sabut metalik, bahan bakar mesin diesel, atau kosmetik. Produk nonpangan ini dihasilkan melalui proses hidrolisis (splitting) (Sunarko 2007).
Tanaman kelapa sawit berdasarkan ketebalan cangkang dan daging buahnya dapat dibedakan menjadi: Dura, Pisifera, Tenera, dan Macro carya. Dura memiliki cangkang yang tebal (2-8 mm), pada bagian luar cangkang tidak terdapat lingkaran serabut, daging buah relatif tipis (35-50%) terhadap buah, kernel (daging buah) besar dengan kandungan minyak yang rendah, dan dalam persilangan dipakai sebagai pohon induk betina (Fauzi et al. 2002).
Pisifera memiliki ciri sebagai berikut: ketebalan cangkang sangat tipis bahkan hampir tidak ada, daging buah tebal (lebih tebal dari daging buah Dura), daging biji sangat tipis, tidak dapat diperbanyak tanpa menyilangkan dengan jenis lain, dan dipakai sebagai pohon induk jantan.Tenera adalah hasil dari persilangan Dura dan Pisifera (Fauzi et al. 2002).
4
Daun kelapa sawit membentuk susunan daun majemuk. Daun terdiri atas kumpulan anak daun (leaflets) yang menyerupai helaian (lamina) dan tulang anak daun (midrib). Daun kelapa sawit juga terdiri atas rachis yang merupakan tempat anak daun melekat, dan tangkai daun (petiole) yang merupakan bagian antara daun dan batang. Terakhir, daun terdiri atas seludang daun (sheath) yang berfungsi sebagai pelindung kuncup dan memberi kekuatan pada batang.
Daun kelapa sawit dihasilkan dalam urut-urutan yang teratur menurut deret
Fibonacci (1, 2, 3, 5, 8, 13, dst.) yang merupakan hasil penjumlahan dari dua angka sebelumnya. Umumnya daun kelapa sawit memiliki filotaksis (susunan daun) yang merupakan kelipatan angka 8. Daun termuda yang sudah mengembang sempurna secara konvensional dinamakan daun nomor 1, sedangkan daun yang masih terbungkus selundang (pupus daun atau spear leaf) dinamakan daun nomor negatif (-1, -2, dan seterusnya) (Pahan 2008). Pada penelitian ini kalus yang dihasilkan dari hasil kultur jaringan berasal dari eksplan daun yang masih terbungkus selundang (pupus daun atau spear leaf).
Kultur Jaringan Tanaman
Kultur jaringan adalah suatu upaya untuk mengisolasi bagian-bagian tanaman (protoplas, sel, jaringan, dan organ), kemudian mengkulturkannya pada nutrisi buatan yang steril di bawah kondisi lingkungan terkendali sehingga bagian-bagian tanaman tersebut dapat beregenerasi menjadi tanaman lengkap (Zulkarnain 2009).
Keberhasilan teknik kultur jaringan sebagai sarana perbanyakan tanaman secara vegetatif pertama kali dilaporkan oleh White 1934 pada kultur akar tanaman tomat (Zulkarnain 2009). Perbanyakan tanaman dengan teknik kultur jaringan ini dapat menghasilkan tanaman baru dalam jumlah yang banyak dan dalam waktu yang singkat serta memiliki sifat yang sama dengan induknya.
Teknik kultur jaringan bermanfaat dalam beberapa hal diantaranya perbanyakan klon secara cepat, menghasilkan tanaman yang secara genetik seragam serta bebas patogen, dapat mempermudah dalam seleksi tanaman, membuat stok tanaman mikro, pelestarian plasma nutfah, produksi tanaman sepanjang tahun, dan memperbanyak tanaman yang sulit diperbanyak secara vegetatif konvensional (Zulkarnain 2009).
Gamborg dan Shyluk (1981) mengelompokkan teknik kultur jaringan tanaman berdasarkan jenis eksplannya menjadi kultur organ (biji, meristem, nodus tunggal, potongan daun, akar, serta tunas), kultur kalus, kultur sel, dan kultur protoplas. Hartman et al. (1990) menggunakan istilah yang lebih spesifik, yaitu mikropropagasi. Teknik mikropropagasi ini adalah pemanfaatan teknik kultur jaringan dalam upaya perbanyakan tanaman, dimulai dari pengkulturan bagian tanaman secara aseptik di dalam tabung kultur atau wadah lain yang serupa. Hartman et al. (1990) juga mengemukakan lima tipe dasar dari mikropropagasi yaitu kultur meristem, proliferasi tunas aksilar, induksi pucuk adventif, organogenesis, dan embriogenesis somatik.
5 teknik embriogenesis somatik tidak langsung (Wong et al. 1997). Tahapan dan durasi setiap proses perbanyakan tanaman kelapa sawit melalui teknik kultur jaringan (somatik embriogenesis tidak langsung) dapat terlihat pada Gambar 1.
Gambar 1 Alur proses kultur jaringan tanaman kelapa sawit (PT SMART Tbk 2012)
Embriogenesis somatik
Embriogenesis somatik adalah proses perkembangan embrio lengkap dari sel-sel vegetatif atau sel-sel-sel-sel somatik yang diperoleh dari berbagai sumber eksplan yang ditumbuhkan pada sistem kultur jaringan (Hartman et al. 1990). Embriogenesis somatik adalah proses sel somatik (non gametic) mengalami diferensiasi membentuk struktur bipolar yang terdiri atas akar dan tunas (Smith 2000).
Embriogenesis somatik ini dapat dibedakan menjadi dua yaitu direct embryogenesis dan indirect embryogenesis. Pada direct embryogenesis
(embriogenesis langsung), sel-sel jaringan eksplan langsung membentuk embrio somatik, sedangkan pada indirect embryogenesis (embriogenesis tidak langsung), eksplan yang dikulturkan akan mengalami dediferensiasi membentuk kalus yang selanjutnya berdiferensiasi membentuk embrio somatik. Kalus yang mampu berdiferensiasi menjadi embrio somatik disebut dengan kalus embriogenik.
Beberapa faktor yang mempengaruhi pembentukan embrio somatik adalah jenis eksplan, gula, sumber nitrogen, serta zat pengatur tumbuh. Penggunaan eksplan yang bersifat meristematik umumnya memberikan keberhasilan pembentukan embrio somatik yang lebih tinggi. Eksplan yang digunakan dapat berupa aksis embrio zigotik muda dan dewasa, kotiledon, mata tunas, epikotil maupun hipokotil. Gula berfungsi di samping sebagai sumber karbon, juga berguna untuk mempertahankan tekanan osmotik media (Purnamaningsih 2002).
6
amino seperti glutamin dan kasein hidrolisat, sangat penting untuk inisiasi dan perkembangan embrio somatik.
Embrio somatik dapat diinduksi dengan memberikan perlakuan stress pada kultur. Perlakuan stress tersebut dapat berupa stress osmotik, heavy metal ion, dan stress dehydration. Stress osmotik dapat dilakukan dengan memberikan konsentrasi tertentu dari berbagai jenis gula, seperti sorbitol, manitol, sukrosa, selain itu stress osmotik juga dapat dilakukan dengan pemberian NaCl, sedangkan contoh stress
heavy metal ion, yaitu dengan pemberian CdCl pada medium. Stress lainnya untuk menginduksi terbentuknya embrio somatik, yaitu drought stress dan cold stress
(Ikeda-Iwai et al. 2003).
Pembentukan embrio dari sel somatik terjadi melalui beberapa tahapan, yaitu: sel somatik yang bersifat meristematik maupun non-meristematik berde-diferensiasi menjadi sel-sel yang belum terberde-diferensiasi (sel-sel kalus), selanjutnya sel-sel ini diinduksi untuk menjadi sel-sel embrionik (mengekspresikan sifat totipotensi). Sel embrionik selanjutnya berkomitmen untuk menjadi sel yang meristematik kemudian sel tersebut berkembang menjadi embrio (Elhiti et al. 2013) (Gambar 2).
Gambar 2 Diagram skematik jalur pembentukan embrio somatik (Elhiti et al. 2013) Fase perkembangan embrio somatik pada dikotil berbeda dengan monokotil. Pada embrio somatik pada tanaman dikotil, terdiri atas: fase globular, hati, torpedo, dan kotiledon (cotyledonary), sedangkan pada tanaman monokotil seperti tanaman kelapa sawit, tahapan perkembangan embrio somatik meliputi fase globular,
7
Gambar 3 Fase perkembangan embrio somatik dari kultur kelapa sawit. (a).
nodular aggregate callus. (b) kalus embriogenik dan embrio fase globular. (c). kalus embriogenik. (d-e) embrio globular. (f) scutellar. (g) early coleoptilar. (h-i) late coleoptilar. (j) embrio yang menuju tahap germinasi. (a-j) sampel didokumentasikan dengan Stereo microscope Nikon SMZ800. (garis pada a & h = 5 mm, garis pada b-g, i-j = 2 mm) (Syariyanto et al. 2014)
Gen-Gen Terkait Embriogenesis
Gen-gen yang terlibat dalam embriogenesis pada tanaman kelapa sawit maupun tanaman lain sudah banyak diteliti antara lain: ACC oxidase merupakan gen yang terekspresi pada protodermis-like layer ini berhubungan pada tahap awal embriogenesis kelapa sawit (See 2002) dan serine/threonine kinase diekspresikan meningkat pada kalus embriogenik dari kelapa sawit (Ooi 2003). Gen-gen yang terlibat selama proses somatik embriogenesis juga telah dikelompokkan menjadi beberapa kelompok gen oleh Chugh & Khurana (2002) (Gambar 4).
8
Gambar 4 Gen-gen yang terlibat dalam proses embriogenesis somatik (Chugh & Khurana 2002)
Analisis gen terkait embriogenesis kelapa sawit juga dilakukan oleh Low et al. (2008) yang menyatakan bahwa lipid transfer protein sangat terekspresi pada kalus embriogenik, sedangkan glutathione S-transferase terekspresi kuat pada kalus non embriogenik.
Gen-gen terkait embriogenesis pada tanaman lain (wortel dan Arabidopsis) antara lain Late Embryogenic Abundant (lea), Somatic Embryogenesis Receptor Kinase (serk) Agamous-like 15 (agl15), Baby Boom (bbm), Leafy Cotyledon 1
(lec1), Fusca3 (fus3) and Leafy Cotyledon 2 (lec2) (Ikeda et al. 2006).
Analisis Hasil Microarray dengan Bioinformatik
Analisis ekspresi gen terkait embriogenesis telah dilakukan terhadap kalus embriogenik dan non embriogenik kelapa sawit menggunakan Microarray oleh Budinarta et al. (2012). Hasil microarray yang telah dinormalisasi selanjutnya dianalisis menggunakan Program Bioinformatik ArrayStar® 5.0. Kegunaan program ini antara lain untuk analisis ekspresi gen hasil dari analisis microarray, analisis SNP, analisis Transcription Factor Binding Sites, dan identifikasi Micro -RNAs.
Program ArrayStar® 5.0 dilengkapi analisis data secara statistik. Data hasil
9 Kandidat-kandidat gen kemudian dianotasi menggunakan BLASTX
algorithm (Altschul et al. 1990) dengan menggunakan e-value 10-6 dengan
databaseSwissprot. BLASTX bertujuan membandingkan sekuen nukleotida yang ditranslasi menjadi protein dengan protein dalam database.
Anotasi gene ontology (GO) dapat dilakukan menggunakan Program
Blast2Go (Conesa et al. 2005). Anotasi gen bertujuan untuk mengetahui lokasi sel/jaringan/organ di mana gen tersebut terekspresi, fungsi gen, serta peranan gen dalam biological pathway.
Interaksi antar gen dapat terlihat dengan menggunakan Program Cytoscape. Kandidat gen terkait dipilih berdasarkan nilai ekspresi (fold change), fungsi gen, serta faktor-faktor yang mempengaruhi embriogenesis. Masing-masing kandidat gen kemudian dilakukan perancangan primer (Design Primer).
Pada penelitian sebelumnya, Budinarta et al. (2012) telah menganalisis ekspresi gen terkait embriogenesis somatik dengan menggunakan teknik
microarray. Pada analisis tersebut dibandingkan sampel kalus dengan sampel embrio somatik. Sampel embrio yang digunakan pada analisis ini merupakan gabungan embrio somatik dari berbagai tahap perkembangan embrio (globular,
scutellar dan coleoptilar). Hasil dari penelitian tersebut diperoleh gen-gen yang up-regulated pada embrio somatik (Tabel 1).
Tabel 1 Gen-gen yang up-regulated di embrio somatik kelapa sawit (Budinarta et al. 2012)
No Info gen Fungsi Folding change
1 Serine carboxypeptidase-like 42 Serine-type carboxypeptidase activity 170.5
2 Metallothionein Metal ion binding 155.5
3 Uncharacterized protein At5g22580 - * 267.3
4 ADT1 - * 49.5
5 Homeobox-leucine zipper protein roc2 sequence-specific DNA binding, unfolded protein binding, sequence-specific DNA binding
transcription factor activity 64.0
6 Ribosome-recycling factor translation factor activity, nucleic acid binding 64.5
7 RFA2 protein binding, nucleic acid binding, 158.3
8 Phototropin-2
protein serine/threonine kinase activity, blue light photoreceptor activity, structural molecule activity, cysteine-type endopeptidase inhibitor activity involved in apoptotic process, FMN binding, identical protein binding, ATP binding, two-component sensor activity
124.8
9 Iaa-amino acid hydrolase ilr1-like 1 IAA-amino acid conjugate hydrolase activity, metallopeptidase activity 142.6
10 GTR1 monosaccharide transmembrane transporter activity, sugar:hydrogen symporter activity,
10
No Info gen Fungsi Folding change
11 Dihydroxy-2-naphthoyl- peroxisomal 3-hydroxypropionyl-CoA dehydratase activity, bicarbonate binding,
1,4-dihydroxy-2-naphthoyl-CoA synthase activity 186.1
12 BRM
ATP-dependent DNA helicase activity,
transcription factor binding, nucleotide binding, methylated histone residue binding, chromatin binding, nucleosome-dependent ATPase activity,
53.5
13 14 kda proline-rich protein lipid binding 350.7
14 ZNRF3 ubiquitin conjugating enzyme binding; ubiquitin-protein ligase activity 55.9
15 Dna-directed rna polymerase iii subunit rpc4 - * 108.5
16 SYT2 threonine-tRNA ligase activity, nucleotide binding, protein homodimerization activity 51.3
17 Inv1 sucrose 1F-fructosyltransferase activity, sucrose alpha-glucosidase activity, fructan
beta-fructosidase activity 436.7
18 5-oxoprolinase ame nucleotide binding 76.8
19 SEC16 - * 225.2
20 40s ribosomal protein s20-1 RNA binding, structural constituent of ribosome 214.6
21 SIL1 unfolded protein binding 106.5
* Keterangan: belum diketahui fungsinya
Perancangan Primer
Primer dapat dirancang dengan menggunakan program yang dapat diakses secara online seperti Primer3 atau Primer Blast yang terdapat pada NCBI. Primer
qPCR for SYBR® Green detection dirancang sesuai Wang & Seed (2006) : 1. Panjang primer berkisar 18-24 nukleotida
2. Panjang produk amplifikasi <250 bp (panjang yang ideal <150 bp) 3. Perbedaan Tm (melting temperature) antar primer forward dan primer
reverse sebaiknya ≤2 °C
4. Primer memiliki Tm berkisar 50-60 °C (ideal: 55-59 °C)
5. Primer tidak memiliki pengulangan basa nukleotida (runs) yang sama lebih dari enam ulangan.
6. Primer tidak memiliki pengulangan basa nukleotida Guanin (G) lebih dari tiga pengulangan secara berturut-turut
7. Primer memiliki persentase GC sekitar 50%
8. Primer tidak memiliki GC clamp pada ujung utas 3’
11
Analisis Ekspresi Gen dengan qPCR
Teknik kuantitatif real-time PCR (qPCR) pertama kali dikembangkan oleh Higuchi pada tahun 1993. Teknik ini sangat sensitif sehingga memungkinkan amplifikasi dan kuantifikasi urutan asam nukleat tertentu melalui deteksi produk PCR secara langsung selama proses PCR berlangsung. qPCR menggubah dari sinyal fluorescent dari setiap reaksi ke dalam nilai numerik untuk setiap sampel (Shipley 2006). Teknik qPCR ini telah diaplikasikan dalam analisis ekspresi gen
putative serine/threonine kinase pada kultur jaringan kelapa sawit (Ooi et al. 2008). Kuantifikasi DNA target dapat dengan mudah dilakukan dengan menentukan jumlah siklus PCR dan kuantifikasi dimulai sejak produk PCR pertama terdeteksi. Hal ini berbeda dengan PCR konvensional yang mendeteksi pada akhir proses PCR (end point detection).
Pewarna fluorescentSYBR Green I akan mengikat semua molekul DNA utas ganda dan akan tereksitasi memancarkan sinyal fluorescent apabila dilewati oleh DNA polymerase. Eksitasi dan emisi maksimal dari SYBR Green berada di 494 nm dan 521 nm. Sinyal fluorescent ini terdeteksi pada saat PCR berada pada tahapan
extension. Kekuatan sinyal meningkat dengan meningkatnya jumlah siklus karena akumulasi PCR produk. Penggunaan SYBR Green memungkinkan analisis dari banyak target yang berbeda tanpa harus mensintesis probe berlabel untuk target tertentu.
Intensitas sinyal fluoresen akan meningkat hingga pada jumlah yang dapat terdeteksi oleh alat qPCR dan digambarkan dalam bentuk kurva amplifikasi (Gambar 5). Kurva amplifikasi mengandung beberapa informasi penting, yaitu:
1. Baseline, adalah level sinyal yang dideteksi oleh mesin qPCR selama proses awal qPCR. Baseline dapat disebut juga background atau noise. Umumnya
baseline ini terbentuk pada siklus ke-3 sampai 15. Penetapan baseline akan menentukan akurasi penetapan CT, di mana garis threshold harus berada di atas baseline.
2. Threshold, adalah level sinyal yang mencerminkan mulainya peningkatan yang signifikan pada fase eksponensial.
3. Cycle, adalah jumlah siklus yang digunakan dalam proses PCR. 4. Rn, adalah jumlah sinyal reporter fluoresen yang terdeteksi.
5. Cycle Threshold (CT) atau cycle quantification (Cq), adalah siklus di mana intensitas sinyal fluoresen pada reaksi PCR berpotongan dengan garis
12
Gambar 5 Kurva amplifikasi pada qPCR (Qiagen 2010)
Analisis ekspresi gen dengan qPCR didahului dengan melakukan analisis efisiensi PCR dari masing-masing primer gen yang digunakan. Analisis efisiensi PCR tersebut dilakukan dengan cara membuat kurva standar dari masing-masing primer yang digunakan. Hanya gen-gen yang kemiringan plot kurva standarnya yang memenuhi syarat sebanding/bernilai 1 (Gambar 6). Gen target yang memiliki kurva standar yang sejajar dengan kemiringan kurva standar gen pembanding saja yang dapat dilanjutkan pada tahapan penghitungan ΔΔCT menurut metode penghitungan Livak & Schmittgen (2001).
Efisiensi amplifikasi antara dua gen dapat berbeda karena perbedaan pada situs penempelan primer, sekuen produk, dan ukuran produk PCR. Apabila diasumsikan efisiensi amplifikasi adalah sebanding, maka amplifikasi dari produk yang lebih panjang akan menghasilkan sinyal yang lebih banyak dibandingkan dengan produk yang lebih pendek.
Efisiensi PCR dapat dihitung dengan menganalisis serangkaian pengenceran
template, memplot nilai Cq terhadap jumlah log template, dan menentukan kemiringan hasil dari kurva standar. Nilai efisiensi PCR dapat dihitung berdasarkan nilai slope (S) dengan menggunakan rumus berikut:
Efisiensi PCR (%) = (10(-1/S) – 1) x 100
S : slope dari kurva standar
13
Kemiringan kurva standar memberikan indikasi efisiensi real-time PCR. Sebuah kemiringan -3.322 berarti bahwa PCR memiliki nilai efisiensi 1 atau 100%, dan jumlah produk PCR dua kali lipat selama setiap siklus. Sebuah kemiringan kurang dari -3.322 (misalnya -3.8) merupakan indikasi dari efisiensi PCR <1. Secara umum, sebagian besar reaksi amplifikasi tidak mencapai 100% efisiensi karena keterbatasan eksperimental. Sebuah kemiringan lebih besar dari -3.322 (misalnya, -3.0) menunjukkan efisiensi PCR yang lebih besar dari 100%. Hal ini dapat terjadi ketika nilai-nilai diukur dalam fase nonlinier reaksi atau dapat menunjukkan adanya inhibitor dalam reaksi.
Nilai Relative Quantification (RQ) dapat dihitung berdasarkan berberapa rumus/metode antara lain metode comparativeΔΔCT (sekarang disebut ΔΔCq) dan metode Relative Quantification Standart Curve. Pada metode comparative ΔΔCq berdasarkan metode Livak dan Schmittgen (2001), nilai Cq dari masing-masing reaksi diperoleh dari kurva amplifikasi. Nilai ΔCq dari masing-masing perlakuan didapatkan dari normalisasi gen target dengan gen pembanding yaitu dengan cara mengurangi nilai Cq gen target dengan nilai Cq gen pembanding. Level ekspresi gen dianalisis berdasarkan nilai Quantification Cycle (Cq) dengan formula sebagai berikut :
Δ Cq control = Cq control – Cq internal kontrol Δ Cq treatment = Cqtreatment– Cqinternal kontrol ΔΔ Cq = Cqcontrol– Cqtreatment
Respective gene expression level = 2- ΔΔCT
Penghitungan dengan RQ berdasarkan metode Relative Quantification Standart Curve (Applied Biosystems 2008):
� � = � �
� � � = � �
14
3
METODE
Penelitian ini dilaksanakan pada bulan Agustus 2014 - Oktober 2015 di
General Laboratory dan Tissue Culture Research Laboratory, Plant Production and Biotechnology Division, PT SMART Tbk, Sentul.
Bahan
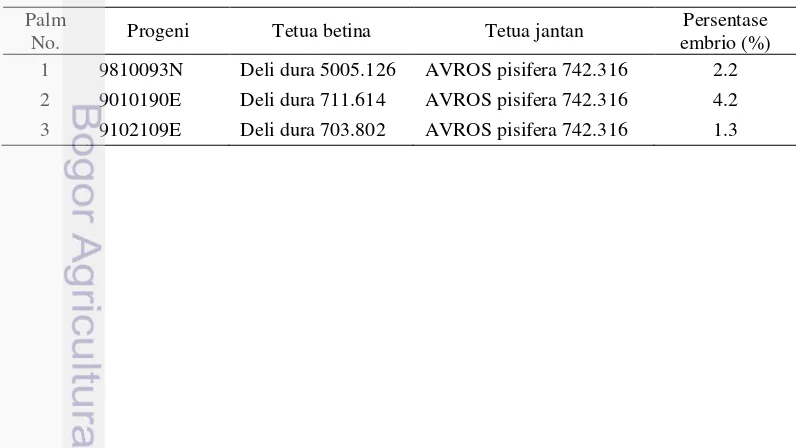
Bahan yang digunakan dalam penelitian ini adalah kalus (nodular callus) dan embrio somatik (coleoptilar stage) hasil kultur jaringan tanaman kelapa sawit Tenera, yaitu persilangan dari Dura (Deli) dan Pisifera (AVROS) yang diperoleh dari Laboratorium Kultur Jaringan Riset PT SMART Tbk (Gambar 7). Sampel tersebut merupakan hasil dari inokulasi eksplan daun muda yang masih terbungkus seludang (pupus daun) melalui teknik somatik embriogenesis tidak langsung. Sampel kalus and embrio somatik dari tiga tanaman kelapa sawit yang berbeda progeni digunakan dalam ektraksi RNA (Tabel 2).
Gambar 7 Sampel kalus dan embrio somatik. (a). Sampel kalus (nodular callus) dan (b) embrio somatik (coleoptilar stage)
Tabel 2 Informasi asal tanaman dari sampel kalus dan embrio somatik Palm
No. Progeni Tetua betina Tetua jantan
Persentase embrio (%)
1 9810093N Deli dura 5005.126 AVROS pisifera 742.316 2.2
2 9010190E Deli dura 711.614 AVROS pisifera 742.316 4.2
15
Prosedur Kerja
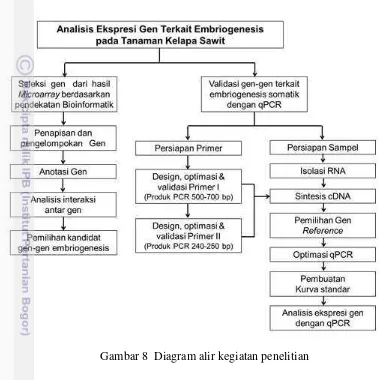
Penelitian ini terdiri atas dua kegiatan, yaitu (1) Seleksi gen-gen terkait embriogenesis menggunakan pendekatan Bioinformatik dan (2) Analisis ekspresi gen terkait embriogenesis pada kelapa sawit menggunakan qPCR (Gambar 8).
Gambar 8 Diagram alir kegiatan penelitian
Seleksi Gen menggunakan Pendekatan Bioinformatik
Kandidat gen terkait embriogenesis diseleksi berdasarkan hasil analisis
microarray dari sampel kalus dan embrio somatik tanaman kelapa sawit yang dilakukan pada penelitian sebelumnya oleh Budinarta 2012. Sebanyak 66.545 sekuen dari hasil microarray diseleksi dengan menggunakan uji statistik Moderated t-test, FDR (Benjamini Hochberg), 8 Fold change pada tingkat kepercayaan 99% yang terdapat pada program Bioinformatik ArrayStar® 5.0. Kandidat gen hasil seleksi kemudian dikelompokkan secara Hierrachical sehingga diperoleh Heat map
yang menunjukkan gen-gen yang terekspresi tinggi (up-regulated)dan terekspresi rendah (down-regulated) pada kedua sampel.
16
terekspresinya gen, serta peranannya (gene ontology) dengan menggunakan program Blast2Go (Conesa et al. 2005). Interaksi antar gennya dianalisis dengan menggunakan program Cytoscape. Sebanyak sepuluh kandidat gen terkait embriogenesis dipilih berdasarkan hasil analisis microarray dan literatur yang selanjutnya dilakukan perancangan primer untuk analisis ekspresi gen.
Isolasi RNA dan Sintesis cDNA Sampel
RNA diisolasi dari sampel kalus dan embrio somatik dengan menggunakan kit NucleoSpin® RNA (Macherey-Nagel 2014) diikuti dengan perlakuan DNAse. Kualitas dan kuantitas (konsentrasi) RNA diukur dengan menggunakan
NanoDropTM 2000c Spectrophotometer (Thermo Scientific, USA) pada panjang gelombang 230, 260, dan 280 nm. Sebanyak 10 µl sampel RNA dielektroforesis pada denaturing Formaldehyde gels dengan buffer MOPS 1X (Mansour dan Pestov 2013) pada 100 volt selama 30 menit, diwarnai dengan EtBr dan didokumentasikan dengan UV trans-iluminator Gel-DocTM XR (BIORAD). Analisis kemurniaan dan integritas RNA dilakukan dengan menggunakan 200 ng RNA pada alat elektroforesis kapiler sesuai protokol QIAxcel RNA QC Kit (Qiagen 2013). Intergritas RNA dinyatakan dalam RNA Integritas Score (RIS). RNA hasil isolasi disimpan pada suhu -80 °C.
Hasil isolasi RNA yang berupa mRNA diubah menjadi cDNA dengan menggunakan QuantiTect® Reverse Transcription Kit (Qiagen 2009). Keberhasilan sintesis cDNA dideteksi dengan cara cDNA sampel amplifikasi menggunakan primer 40s (primer I) yang dirancang diantara kedua ekson (mengapit intron). Ukuran pita hasil amplifikasi cDNA dengan primer gen 40s (primer I), yaitu 456 bp dan apabila terjadi kontaminasi DNA maka akan terdapat pita dengan ukuran ±1000 bp. cDNA disimpan pada suhu -40 °C.
Perancangan, Optimasi, dan Validasi Primer
Perancangan primer dilakukan pada kesepuluh gen target terkait embriogenesis dan kelima gen pembanding. Primer dirancang melalui dua tahap, yaitu: primer I untuk menghasilkan produk PCR dengan ukuran ±500-700 bp. Hasil sekuen dari primer I selanjutnya digunakan sebagai acuan dalam perancangan primer qPCR (Primer II, ±240-250 bp). Primer I dan II dirancang menggunakan program Primer-BLAST sesuai dengan persyaratan perancangan primer qPCR menurut Wang dan Seed (2006).
17 Validasi primer dilakukan dengan cara: pita cDNA hasil dari amplifikasi masing-masing primer yang terdapat pada gel agarosa, dipotong dan diipurifikasi dengan menggunakan QiAquick® Gel Extraction Kit (Qiagen 2008) sesuai dengan protokol produsen kemudian disekuensing dan susunan nukleotida hasil sekuensing dibandingkan (BLASTN) dengan sekuen cDNA dari masing-masing gen pada database NCBI.
Pembuatan Standar qPCR
Kurva standar dibuat dengan mengamplifikasi cDNA dengan masing-masing primer dengan mix PCR yang terdiri atas: 1 µl cDNA (1,5 µg µl-1), 14 µl primer mix (10 µM), 10,5 µl 10X DreamTaq Buffer, 10,5 µl dNTP Mix, 4,2 µl MgCl2, 0,84 µl Dream Taq DNA Polymerase (Thermo Scientific), dan nuclease-free water
hingga volume 100 µL. Reaksi PCR yang digunakan, yaitu: predenaturasi pada 94 °C selama 5 menit, proses amplifikasi dilakukan sebanyak 35 siklus yang terdiri atas: denaturasi pada 94 ºC selama 1 menit, anealing pada suhu 62 °C atau 64 °C selama 30 detik, dan ekstensi pada 72 °C selama 1 menit. Proses amplifikasi diakhiri dengan pasca ekstensi pada 72 °C selama 7 menit.
Hasil dari amplifikasi cDNA tersebut dielektroforesis pada gel agarosa 2% (b/v) pada 100V selama 90 menit, pita cDNA pada gel dipotong, dan dipurifikasi dengan menggunakan QiAquick® Gel Extraction Kit (Qiagen 2008) sesuai dengan
protokol produsen. cDNA hasil purifikasi tersebut lalu dibuat pengenceran bertingkat dari konsentrasi 10-1 ng/µL sampai dengan 10-7 ng/µL. Analisis qPCR dilakukan berdasarkan hubungan linear antara nilai Cq dengan nilai log dari konsentrasi cDNA (R2>0.98), sehingga dapat dihitung konsentrasi sampel berdasarkan nilai Cq dari masing-masing sampel.
Seleksi Gen Pembanding
Pemilihan gen pembanding dilakukan dengan cara masing-masing primer gen pembanding dianalisis kespesifikkannya melalui melt curve analisis. Primer yang spesifik hanya akan menghasilkan satu puncak pada melt curve. Primer yang spesifik selanjutnya dilakukan analisis absolut quantitation pada kedua jenis sampel yaitu kalus dan embrio somatik. Gen pembanding yang dipilih untuk analisis qPCR adalah gen yang stabil pada kedua jenis sampel yang diuji, yaitu gen yang menghasilkan nilai Cq yang relatif sama pada kedua jenis sampel tersebut.
Analisis Ekspresi Gen dengan qPCR
Ekspresi dari kandidat gen embriogenesis dikuantifikasi menggunakan qPCR dengan metoda Relative Quantification Standart Curve (Larionov et al. 2005). Kalus digunakan sebagai kalibrator sampel. Reaksi qPCR dilakukan pada
MicroAmp Fast Optical 96-well reaction plate 0.1 mL (Applied Biosystems) dengan menggunakan QuantiFast®SYBR® Green PCR Kit (Qiagen 2011) dan alat Applied
18
Publication of Quantitative Real Time PCR Experiments (MIQE) Guidelines (Bustin et al. 2009).
Mix qPCR (@ 10 µL/reaksi) terdiri atas: 200 ng/µL sampel cDNA utas tunggal, 0,2 µM mix primer, 5 µL QuantiFast SYBR Green PCR Master Mix (2x), dan RNase-Free Water hingga total volume 10 µL dengan kondisi reaksi PCR yang diawali dengan proses predenaturasi pada 95 ºC selama 5 menit, dilanjutkan dengan amplifikasi sebanyak 40 siklus yang terdiri atas: 95 ºC selama 10 detik (denaturasi), 62 ºC selama 30 detik (annealing dan ekstensi), dan dilanjutkan dengan analisis
melt curve yang reaksinya terdiri atas: 95 ºC selama 15 detik, 60 ºC selama 1 menit, 95 ºC selama 15 detik, dan 60 ºC selama 15 detik.
Analisis ekspresi gen dilakukan dengan menggunakan qPCR secara manual pipetting pada sampel (kalus dan embrio somatik) dari ketiga tanaman kelapa sawit yang masing-masing reaksi qPCR dilakukan sebanyak tiga ulangan (triplo). Nilai Cq dan quantity dari sampel diperoleh secara automatis dari hasil analisis qPCR dengan software 7500 v2.0.6, sedangkan nilai Relative Quantiation (RQ) dihitung secara manual dengan Excel sesuai dengan rumus dari Real Time Quantitative PCR Guide from Applied Biosystems (Applied Biosystems 2008):
� � = � �
� � � = �
�
� � =� � � � �
Nilai RQ selanjutnya dianalisis dengan One-way Anova dan Dunnett t-tests
19
4
HASIL DAN PEMBAHASAN
Seleksi Gen Embriogenesis dengan Pendekatan Bioinfromatik
Sebanyak 66.545 sekuen dari hasil analisis microarray terhadap sampel kalus dan embrio somatik kultur kelapa sawit (Budinarta et al. 2012), setelah diseleksi dengan Moderated t-test, FDR (Benjamini Hochberg), 8 Fold change pada tingkat kepercayaan 99% yang terdapat pada program Bioinformatik ArrayStar® 5.0 diperoleh sebanyak 1.087 gen (573 up-regulated genes dan 514 down-regulated genes). Sebanyak sepuluh gen terkait embryogenesis dipilih berdasarkan nilai ekspresinya (fold change) dari hasil analisis microarray, fungsi gen, dan faktor-faktor yang mempengaruhi embryogenesis. Kesepuluh kandidat gen terkait embriogenesis somatik berserta fungsinya dapat terlihat pada Tabel 3 dan Tabel 4. Tabel 3 Kandidat gen embriogenesis somatik
No Gen Description Fold change
1 26sp 26s proteasome non-atpase regulatory subunit 13 15.13 Down
2 ilr1 Auxin amidohydrolase/iaa-amino acid hydrolase
ilr1-like 1 142.63 Up
3 inv1 Invertase/beta-fructofuranosidase 1 436.68 Up
4 lea Late embryogenesis abundant protein 1.93 Down
5 nltp Non-specific lipid-transfer protein akcs9 16.99 Up
6 qltq3 Qltg3-1/ proline-rich protein 350.75 Up
7 phot2 Serine threonine protein/ phototropserine Threonine
protein or phototropin-2in-2 124.80 Up
8 serk Somatic embryogenesis receptor kinase 5.79 Up
9 tps6 Trehalose-6-phosphate Synthase 11.44 Down
10 hsp Heat shock protein 405.89 Up
Tabel 4 Fungsi dari kandidat gen embriogenesis somatik
Gen Functional Process
26sp inositol or phosphatidylinositol kinase activity
ilr1 calcium ion binding, hydrolase activity GO:0016787, protein binding, protein binding, metallopeptidase activit, IAA-Ala conjugate hydrolase activity
inv1 beta-fructofuranosidase activity, sucrose alpha-glucosidase activity,
6G-fructosyltransferase activity, 1,2-beta-fructan 1F-fructosyltransferase activity
lea2 nucleic acid binding, response to stres, response to abiotic stimulus
nltp lipid binding
20
Gen Functional Process
phot2 phosphorelay sensor kinase activity, protein serine/threonine kinase activity; structural molecule activity, ATP binding, blue light photoreceptor activity,
FMN binding, identical protein binding
serk2
peptide receptor activity, transmembrane receptor protein serine/threonine kinase activity, ATP binding, ubiquitin protein ligase binding, receptor serine/threonine kinase binding, identical protein binding, protein heterodimerization activity
tps6
alpha,alpha-trehalose-phosphate synthase (UDP-forming) activity ,
glucosylglycerol-phosphate synthase activity, trehalose-phosphatase activity
hsp response to heat, hydrogen peroxide, ethanol, arsenic-containing substance, cadmium ion, and response to copper ion
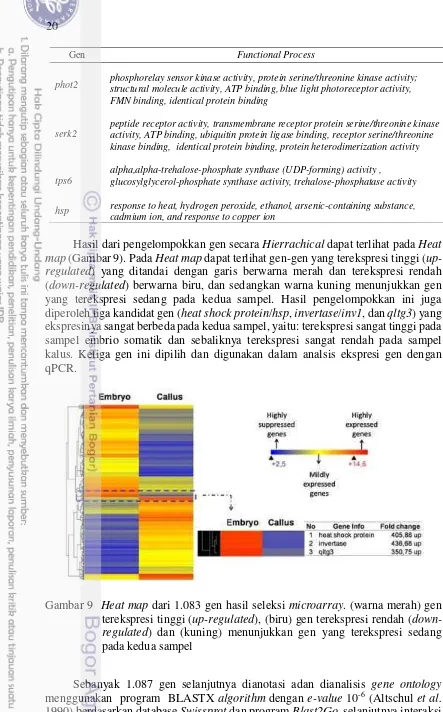
Hasil dari pengelompokkan gen secara Hierrachical dapat terlihat pada Heat map (Gambar 9). Pada Heat map dapat terlihat gen-gen yang terekspresi tinggi ( up-regulated) yang ditandai dengan garis berwarna merah dan terekspresi rendah (down-regulated) berwarna biru, dan sedangkan warna kuning menunjukkan gen yang terekspresi sedang pada kedua sampel. Hasil pengelompokkan ini juga diperoleh tiga kandidat gen (heat shock protein/hsp, invertase/inv1, dan qltg3) yang ekspresinya sangat berbeda pada kedua sampel, yaitu: terekspresi sangat tinggi pada sampel embrio somatik dan sebaliknya terekspresi sangat rendah pada sampel kalus. Ketiga gen ini dipilih dan digunakan dalam analsis ekspresi gen dengan qPCR.
Gambar 9 Heat map dari 1.083 gen hasil seleksi microarray. (warna merah) gen terekspresi tinggi (up-regulated), (biru) gen terekspresi rendah ( down-regulated) dan (kuning) menunjukkan gen yang terekspresi sedang pada kedua sampel
Sebanyak 1.087 gen selanjutnya dianotasi adan dianalisis gene ontology
menggunakan program BLASTX algorithm dengan e-value 10-6 (Altschul et al.
21 antar gennya dianalisis dengan menggunakan program Cytoscape versi 2.8 dan database BioGRID 3.2 (Gambar 10). Hasil analisis interaksi gen ini dapat terlihat terdapat beberapa tiga gen yang interaksi gen-nya paling banyak/tinggi (berwarna merah), yaitu: pat1, hht1, dan tps1. Gen pat1 merupakan gras family transcription
factor, hht1 merupakan hydroxycinnamoyl shikimate quinate
hydroxycinnamoyltransferase-like protein, dan tps1 adalah trehalose-6-phosphate synthase. Salah satu gen yang memiliki tingkat interaksi tertinggi yaitu tps1 atau
tps6 ini dipilih dalam analisis ekspresi gen dengan qPCR. Gen tps1 ini merupakan gen yang berperan dalam pembentukan gula osmotik (trehalose) yaitu sejenis gula/sumber karbon yang dibentuk dalam kondisi drought stress.
Gambar 10 Interaksi antar gen up-regulated dan down-regulated hasil analisis dengan Cytoscape. Lingkaran besar (merah) gen yang memiliki interaksi gen paling tinggi dengan gen lain, (oranye) interaksi gen sedang, (kuning) interaksi gen rendah, dan lingkaran paling kecil (hijau) gen yang interaksinya paling rendah
Isolasi RNA dan Sintesis cDNA Sampel
22
Gambar 11 RNA hasil isolasi dari kalus (C) dan embrio somatik (E) tanaman kelapa sawit
Tabel 5 Konsentrasi, kemurnian, serta nilai intergritas RNA dari sampel kalus dan embrio somatik pada ketiga tanaman kelapa sawit
Sampel
Sambrook et al. (1989) menyatakan bahwa RNA yang berkualitas baik adalah RNA yang memiliki nilai OD260/280 dengan rasio 1.8-2.0. Perbandingan nilai absorbansi pada panjang gelombang 260 nm dan 280 nm (A260/280) menyatakan nilai kemurnian RNA dari kontaminasi protein, sedangkan A260/230 menunjukkan kemurnian RNA dari kontaminasi bahan kimia lainnya seperti EDTA yang biasa digunakan dalam proses isolasi RNA. Nilai integritas RNA biasanya dinyatakan dalam RIN/RQI, namun karena pada penelitian ini menggunakan alat Qiaxcel (Qiagen, German) maka nilai tersebut dinyatakan dalam RNA Integritas Score
23 nilainya berarti kualitas RNA semakin baik (RNA tidak terdegradasi). Hasil analisis integritas RNA dari masing-masing sampel kalus dan embrio somatik dapat terlihat pada Gambar 12 dan Gambar 13.
Gambar 12 Hasil analisis integritas RNA kalus
24
RNA yang telah berhasil diisolasi kemudian diubah menjadi cDNA utas tunggal dengan menggunakan QuantiTect® Reverse Transcription Kit (Qiagen 2009). Keberhasilan sintesis cDNA utas tunggal ini dapat diketahui dari ukuran pita yang terbentuk dari hasil amplifikasi cDNA dengan primer 40s (primer I) yang telah dirancang di antara dua ekson. Primer ini menghasilkan pita berukuran ±456 bp pada cDNA, sedangkan bila terkontaminasi DNA maka pita akan berukuran ±1.000 bp. cDNA dari sampel RNA kalus dan somatik embrio telah berhasil disintesis tanpa terkontaminasi DNA dapat terlihat pada Gambar 14.
Gambar 14 Hasil sintesis cDNA dari sampel kalus dan embrio somatik. (a) kalus dan (b) embrio somatik
Perancangan, Optimasi, dan Validasi Primer
Perancangan primer dilakukan sebanyak dua tahap, yaitu pada tahap pertama (primer I) dirancang primer yang menghasilkan produk PCR dengan ukuran agak panjang (500-700 bp). Pembuatan primer I ini bertujuan untuk verifikasi susunan nukleotida hasil sekuensing dari masing-masing gen dengan database cDNA gen kelapa sawit yang telah dimiliki pada penelitian sebelumnya. Hasil optimasi primer gen pembanding dan gen target (primer I) dapat terlihat pada Tabel 6 dan Tabel 7. Tabel 6 Sekuen nukleotida dari primer gen pembanding (primer I)
Gen Primer pair 5' → 3' Ukuran
Amplikon (bp) Ta (°C)
act3 F GAGATTCAGGTGTCCAGAGGT 589 60
R CATAGATAAGGAGAGAGCGTGC
ef2 F GAAGGAGATGGAAGGTGGAAG 528 64
R GGTTGAAGGTGAGGAAGCC
40s F GCTCCTTCCCTTCCTCTCAA 456 60
R CCTGGACTAACAAAATCACCGAT
sodm F CATTCAGGGAAAGGTCCATCT 621 62
R ACATCACAAACTACAACAAGGC
tubβ1 F AGGTGGTTCAAGTCTCCAAAG 700 58
R TTCTCTCCGCCGTCAAAATG
25
Gen Primer pair 5' → 3' Ukuran
Amplikon (bp) Ta (°C)
26sp F TCATCTTTAGCCGACCTTCG 555 66
R ATTCACCAGCCCGCCTTTT
ilr1 F TCGCAGGTAAGAAAGGATGTT 640 62
R GGGAGCACAAAAGCAAAGTC
inv1 F ACATACGGACACAAGCAACA 561 64
R CGACTACGGCAAGTTCTACG
lea2 F TACAAACCCCAATCCCATCC 559 60
R GTCCCAAAGAGCAGAACCAA
nltp F GCATCCTTCCTCTTCCTATCC 448 66
R GACAAGCCCTCCTCACAAA
qltg3 F GCTTCCACATCCCTCCTCCT 500 56
R GACCAAACAACGACACCACA
phot2 F CAAAGGTTGGAGGAAGGTGA 513 64
R TGGGTTGTGGGGATACTGG
serk2 F CTCCACTTGCTGCCATTCCT 605 66
R GCCTCCATCTGAACCACCAC
tps6 F ATTCCTGTTGCTGACTGTGG 528 62
R GCTTCTTACTGCTTGTTCTGAG
hsp F CCCACAAAACACACCAACCC 536 58
R CCAAGCACACCAAGATTCCG
Sekuen nukleotida dari hasil primer I ini kemudian digunakan sebagai acuan dalam merancang primer qPCR yang menghasilkan produk PCR yang berukuran 240-250 bp. Hasil BLASTN dari primer I menujukkan primer gen target dan pembanding telah berhasil dirancang spesifik sesuai gen yang ditargetkan (Tabel 8).
Tabel 8 Hasil BLASTN gen target dan pembanding primer I
Primer Description E-value Ident
26sp PREDICTED: Elaeis guineensis 26S proteasome non-ATPase
regulatory subunit 13 homolog B 0.0 99%
ilr1 PREDICTED: Elaeis guineensis IAA-amino acid hydrolase
ILR1-like 1 0.0 99%
inv1 PREDICTED: Elaeis guineensis beta-fructofuranosidase 1 0.0 99%
lea2 PREDICTED: Elaeis guineensis uncharacterized LOC105049817 0.0 99%
nltp PREDICTED: Phoenix dactylifera non-specific lipid-transfer protein
2-like 0.0 83%
qltg3 PREDICTED: Elaeis guineensis 14 kDa proline-rich protein
DC2.15-like 0.0 98%
phot2 PREDICTED: Elaeis guineensis phototropin-2-like 0.0 100%
serk2 PREDICTED: Elaeis guineensis somatic embryogenesis receptor
kinase 2-like 0.0 99%
tps6 PREDICTED: Elaeis guineensis alpha,alpha-trehalose-phosphate
synthase [UDP-forming] 6-like 0.0 99%
26
Primer Description E-value Ident
protein-like
act3 PREDICTED: Elaeis guineensis actin-3-like 0.0 99%
ef2 PREDICTED: Elaeis guineensis elongation factor 2-like 0.0 99%
40s PREDICTED: Elaeis guineensis 40S ribosomal protein S27-2 0.0 96%
Sodm PREDICTED: Elaeis guineensis superoxide dismutase [Mn],
mitochondrial 0.0 99%
tubβ1 PREDICTED: Elaeis guineensis tubulin beta-1 chain 0.0 99%
Hasil optimasi primer II baik gen target maupun gen pembanding ternyata banyak terdapat pita yang tidak spesifik yaitu dengan adanya dimer (Tabel 9 dan Tabel 10), hal ini juga terlihat pada gel elektroforesis pada Gambar 15. Primer yang dapat digunakan dalam analisis dengan qPCR harus primer yang spesifik, yaitu primer yang hanya membentuk satu pita tunggal tanpa dimer pada gel elektroforesis, selain itu primer juga hanya membentuk satu puncak pada hasil analisis melt curve (Bustin et al. 2009).
Tabel 9 Sekuen nukleotida dan hasil optimasi primer gen target (primer II)
Gen Primer pair 5' → 3'
26sp F AGCCTCTCGGTTCATCTTATTG 250 60 Spesifik
R GTTGTTTTGGTCTCCTTCTGC
ilr1 F CTCCTCAATTCGTTGCTTCAGTT 250 58 Spesifik
R CGGAAAGGGAGGTCATGGT
inv1 F GCCAACACGAGCAATCCAAA 240 66 ns, dimer
R TGACGAGGTGGAGAGCCTAA
lea2 F ACAAACCCCAATCCCATCC 243 64 Spesifik
R AATCGGCACATCAACAATGAG
nltp F CCTGAACGGGTCTCCACCAA 250 64 ns, dimer
R GCTAACACTGCCAACACTCTG
qltg3 F TGCCCACCACCTCCTTCTAA 248 64 ns, dimer
R ACAGGGACGTTGAGCTTGAT
phot2 F AAGTCGGTCAATACCACATGC 240 64 ns, dimer
R CACTTCCTTTCAGACTCCAACAC
serk2 F AGTCCTTTCACCCAGTCAAGC 250 64 ns, dimer
R AAGCAGTTGTTGGAGACTTCGG
tps6 F CAGGAGAGGGGCTTATCACC 240 58 Spesifik
R GCTTCTTACTGCTTGTTCTGAG
hsp F CGCCGCAATCTCATCGGTTA 246 66 ns, dimer
27 Tabel 10 Sekuen nukleotida primer gen pembanding (primer II)
Gen Primer pair 5' → 3'
Ukuran Amplikon
(bp)
Ta (°C) Spesifisitas Amplikon
act3 F ATATCAGGAAGGACTTGTATGG 240 60 Spesifik
R CAGGACCACATTCATCATACTCT
ef2 F ACTTCTTGGTTGTTGGGTCA 250 62 ns, dimer
R GCCTATCAGACTTTTCTTCGTG
40s F GCTCCTTCCCTTCCTCTCAA 246 60 Spesifik
R TACCACACACAACAACAGTCT
sodm F TATCCAGAGCAAGCCACACC 242 60 Spesifik
R AGACTCCTCCAAGGTCGTC
tubβ1 F TCGCAGTTCTCAGCCTCTTT 247 62 ns, dimer
R GCGTCAATGTCTACTACAACGA ns = primer tidak spesifik
Gambar 15 Hasil optimasi primer set II yang tidak spesifik (terdapat dimer). Marker (1 Kb+), kalus (C), dan embrio somatic (E)
28
Gambar 16 Hasil analisis Melt curve pada gen target dan gen pembanding
Tabel 11 Hasil BLASTN gen target dan pembanding primer set II
Gen Definisi/Keterangan gen E-value Ident (%)
ilr1 PREDICTED: Elaeis guineensis IAA-amino acid hydrolase
ILR1-like 1 (LOC105047633) 8.0E-82 99
lea2 PREDICTED: Elaeis guineensis uncharacterized
(LOC105049817) 7.0E-93 100
26sp PREDICTED: Elaeis guineensis 26S proteasome
non-ATPase regulatory subunit 13 homolog B (LOC105060648) 4.0E-90 100
tps6 PREDICTED: Elaeis guineensis alpha-trehalose-phosphate
synthase [UDP-forming] 6-like (LOC105048485) 4.0E-85 100
40s PREDICTED: Elaeis guineensis 40S ribosomal protein S27-2
(LOC105035175) 2.0E-98 100
Tabel 12 Hasil BLASTN dan BLASTX pada primer set II
Gen Accession number Location of amplicon Chromosome Conserved Domains
ilr1 XM_010926645.1
(2258580..2296247) Unplaced Scaffold -
tps6 XM_010927796.1
NC_025999.1 (12213872..12217392, complement)
7 HAD_like superfamily
40s XM_010910623.1 NW_011551536.1
(241058..242434) Unplaced Scaffold
29
Seleksi Gen Pembanding
Pemilihan gen pembanding dilakukan dengan cara masing-masing primer gen pembanding dianalisis kespesifikkannya melalui melt curve analisis. Primer yang spesifik hanya akan menghasilkan satu puncak pada hasil analisis melt curve. Hasil analisis melt curve menggunakan alat qPCR menunjukkan bahwa dari kelima gen
reference hanya tiga gen yang spesifik, antara lain: gen act1, 40s, dan tubβ1 (Gambar 17).
Ketiga primer yang spesifik selanjutnya dilakukan analisis absolut quantitation pada kedua jenis sampel, yaitu kalus dan embrio somatik. Gen
reference yang digunakan untuk analisis qPCR adalah gen yang stabil pada kedua jenis sampel yang diuji, yaitu gen yang menghasilkan nilai Cq yang relatif sama pada kedua jenis sampel tersebut. Gen 40s dipilih sebagai gen pembanding karena terbukti lebih stabil pada kedua jenis sampel dibandingkan gen pembanding lainnya (Gambar 18). Gen pembanding 40s juga merupakan gen yang direkomendasikan untuk digunakan dalam analisis ekspresi gen untuk sampel kultur jaringan kelapa sawit menurut penelitian Chan et al. (2014).
30
Gambar 18 Perbandingan nilai Cq dari ketiga gen (act3, 40s, dan sodm) pembanding pada sampel kalus (Cal) dan embrio somatik (Emb)
Analisis Ekspresi Gen dengan qPCR
Analisis ekspresi gen terkait embryogenesis dilakukan pada sampel kalus dan embrio somatik menggunakan metode Relative Quantification Standart Curve. Metode ini merupakan metode terpercaya serta merupakan metode alternatif yang lebih mudah dibandingkan perhitungan berdasarkan efisiensi PCR pada relative real time PCR (Larionov et al. 2005).
Kurva standar qPCR dibuat dari amplifikasi cDNA dengan masing-masing primer gen target dan gen pembanding. Pada penelitian ini kalus digunakan sebagai
calibrator sampel (kontrol), maka kurva standar dibuat dengan menggunakan sampel cDNA kalus. Hasil amplifikasi dari cDNA kemudian dilarikan pada gel agarose yang selanjutnya dilakukan purifikasi dengan cara gel extraction
menggunakan QiAquick® Gel Extraction Kit (Qiagen 2008). Hasil purifikasi
tersebut lalu diukur dengan Nanodrop dan dibuat pengenceran bertingkat dari konsentrasi 10-1 ng/µL sampai dengan 10-7 ng/µL.