IDENTIFIKASI GENOTIPE YANG MEMBERIKAN
KONTRIBUSI TERHADAP INTERAKSI
GENOTIPE × LINGKUNGAN PADA MODEL AMMI
RUSIDA YULIYANTI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN SUMBER
INFORMASI
Dengan ini saya menyatakan bahwa tesis Identifikasi Genotipe yang Memberikan Kontribusi terhadap Interaksi Genotipe × Lingkungan pada Model AMMI adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau yang dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam daftar pustaka di bagian akhir tesis ini.
Bogor, Februari 2009
ABSTRACT
RUSIDA YULIYANTI. Identification of The Contributing Genotypes for Genotipe × Environment Interaction on AMMI Model. Under direction of AHMAD ANSORI MATTJIK and TOTONG MARTONO.
Additive Main Effect and Multiplicative Interactions (AMMI) Models have been utilized in analysis of the effects of the Genotype × Environment Interaction (GEI) on multienvironmental trials. However, the models could not be used for post-hoc tests of genotype contribution in the interaction effects. The tests are called tests of subhypothesis. By conducting this test, one could identify the contributing genotypes in GEI. Critical point in this test is unknown, so that it’s value must be approximated by bootstrap method. This research used maize data from PT.Kreasidharma and Bioseed Inc. In this data, there are 12 genotypes which are tested in 16 different locations. As the result, the identified contributing genotypes in GEI are BIO 9900, BIO 1169 dan BIO9899.
RINGKASAN
RUSIDA YULIYANTI. Identifikasi Genotipe yang Memberikan Kontribusi terhadap Interaksi Genotipe × Lingkungan pada Model AMMI . Di bawah bimbingan AHMAD ANSORI MATTJIK sebagai ketua dan TOTONG MARTONO sebagai anggota.
Model AMMI telah mampu menerangkan pengaruh interaksi genotipe × lingkungan, dan sebenarnya bisa dilakukan pengujian mengenai kontribusi yang diberikan oleh genotipe dan lingkungan terhadap pengaruh interaksi tersebut dengan pengujian subhipotesis yang disampaikan oleh Marasinghe (1980).
Pengujian subhipotesis memerlukan nilai statistik Λ dan nilai kriteria uji (qφ)
untuk menarik kesimpulan hasil pengujian. Karena distribusi Λ belum diketahui
sehingga didekati dengan distribusi empirik guna mengaproksimasi nilai kriteria
uji. Nilai kriteria uji bagi sebaran Λ ditentukan dengan metode Bootstrap.
Penelitian ini bertujuan mendeskripsikan metode pengujian subhipotesis pada model AMMI dan mengimplementasikan metode pada tujuan pertama terhadap data percobaan jagung dalam upaya mengidentifikasi genotipe yang berkontribusi terhadap interaksi genotipe × lingkungan. Data penelitian ini merupakan data sekunder hasil percobaan tanaman jagung hibrida yang dilakukan oleh Bioseed Genetic International, INC bekerjasama dengan PT. Mitra Kreasidharma. Percobaan dilakukan di 16 lokasi dengan 12 genotipe.
Metode subhipotesis Marasinghe dapat digunakan untuk menguji pengaruh genotipe terhadap interaksi genotipe × lingkungan pada model AMMI.
Matriks kontras H pada hipotesis nol H0 merepresentasikan tujuan pengujian
tersebut. Pengujian subhipotesis terhadap data percobaan jagung menunjukkan bahwa genotipe yang memberikan kontribusi terhadap interaksi genotipe × lingkungan adalah genotipe A (BIO 9900), C (BIO 1169) dan I (BIO9899).
©Hak cipta milik IPB, Tahun 2009
Hak cipta dilindungi Undang-undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan
atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan,
penelitian, penulisan karya ilmiah, penyusunan laporan, penyusunan kritik atau
tinjauan suatu masalah; dan pengutipan tidak merugikan kepentingan yang wajar
IPB.
Dilarang mengumumkan atau memperbanyak sebagian atau seluruh karya tulis
IDENTIFIKASI GENOTIPE YANG MEMBERIKAN
KONTRIBUSI TERHADAP INTERAKSI
GENOTIPE × LINGKUNGAN PADA MODEL AMMI
RUSIDA YULIYANTI
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Sekolah Pascasarjana Institut Pertanian Bogor
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Judul Tesis : Identifikasi Genotipe yang Memberikan Kontribusi terhadap Interaksi Genotipe × Lingkungan pada Model AMMI
Nama : Rusida Yuliyanti
NRP : G151050151
Program Studi : Statistika
Disetujui Komisi Pembimbing
Prof. Dr. Ir. A. A. Mattjik, M.Sc Dr. Totong Martono Ketua Anggota
Diketahui
Ketua Program Studi Statistika Dekan Sekolah Pascasarjana IPB
Dr. Ir. Aji Hamim Wigena, M.Sc Prof.Dr. Ir. Khairil A. Notodiputro, M.S
PRAKATA
Puji syukur penulis panjatkan kepada Allah SWT atas berkat Rahmat dan
Karunia-Nya sehingga penulis dapat menyelesaikan tesis yang berjudul ”Identifikasi Genotipe yang Memberikan Kontribusi terhadap Interaksi Genotipe
× Lingkungan pada Model AMMI”.
Pada kesempatan ini, dengan penuh rasa hormat penulis mengucapkan terima kasih kepada:
1. Prof. Dr. Ir. A. A. Mattjik, M.Sc, sebagai ketua komisi pembimbing dan
Dr. Totong Martono sebagai anggota komisi pembimbing yang telah memberikan bimbingan, masukan dan saran yang sangat berarti dalam penyusunan tesis ini.
2. Dr. Ir. Asep Saefuddin, M.Sc sebagai penguji Luar Komisi pada Ujian Tesis.
3. Direktorat Jendral Pendidikan Tinggi yang telah mendanai penelitian ini yang
merupakan bagian Hibah Penelitian Tim Pascasarjana dengan Nomor : 266/13.11/PL/2008 tanggal 2 April 2008.
4. Keluarga dan teman-teman mahasiswa pascasarjana Statistika yang telah
membantu penulis dan memberi dukungan selama penyusunan tesis ini.
5. Semua pihak yang telah membantu penulis baik secara fisik, ilmu maupun
dukungan moral dalam penyusunan usulan penelitian ini.
Akhirnya penulis mohon maaf jika tesis ini masih jauh dari kesempurnaan. Semoga tulisan ini bermanfaat bagi pihak yang membutuhkannya.
Bogor, Februari 2009 Penulis
RIWAYAT HIDUP
x
DAFTAR ISI
Halaman
DAFTAR TABEL ... xi
DAFTAR GAMBAR ... xi
DAFTAR LAMPIRAN ... xii
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan Penelitian ... 2
TINJAUAN PUSTAKA ... 3
Model Interaksi Multiplikatif pada Rancangan Faktorial Dua Faktor ... 3
Model AMMI (Additive Main Effect and Multiplicative Interactions) ... 5
Perhitungan Jumlah Kuadrat AMMI ………... 6
Penentuan Banyaknya Komponen AMMI ... 6
BAHAN DAN METODE ... 8
Bahan ... 8
Metode Analisis ... 8
HASIL DAN PEMBAHASAN ……… . 11
Pengelompokan Genotipe ……….. .. 11
Pengujian Pengaruh Interaksi Kelompok Genotipe × Lingkungan ... 13
Model AMMI ………... 14
Pengujian Subhipotesis pada Model AMMI 2 ... 15
SIMPULAN DAN SARAN………... .. 20
Simpulan ... 20
Saran ………. 20
xi
DAFTAR TABEL
Halaman
1 Tabel Analisis Ragam AMMI ……… 7
2 Korelasi Komponen Hasil Penen dengan Hasil Panen ……….. 12
3 Analisis Ragam dari Hasil Panen ... 14
4 Persentase Keragaman Interaksi Kelompok Genotipe × Lingkungan ... 15
5 Matriks Kontras Pengujian Subhipotesis ... 15
6 Hasil Aproksimasi Nilai Kriteria Uji ………. 16
7 Hasil Pengujian Subhipotesis pada AMMI 2 ………... 18
DAFTAR GAMBAR
Halaman 1 Proses Resampling untuk Aproksimasi Nilai Kriteria Uji ... 102 Biplot melalui AMMI 2 untuk Hasil Panen ………. 11
3 Dendrogram Genotipe Berdasarkan Karakteristik Kadar Air Panen, Umur Berbunga Betina, Berat Tongkol dan Hasil Panen ... 13
xii
DAFTAR LAMPIRAN
Halaman
1 Deskripsi Lokasi Penelitian ... 22
2 Kode genotipe ... 23
3 Persentase Keragaman Interaksi Genotipe × Lokasi ... 24
4 Analisis Box Cox Transformation ... 25
5 Uji Asumsi dalam Normalitas Galat dan Kehomogenan Ragam pada Analisis Ragam untuk Data Hasil Panen .….……… 26
1
PENDAHULUAN
Latar Belakang
Rancangan percobaan faktorial dengan klasifikasi dua faktor telah banyak diterapkan dalam berbagai bidang, salah satunya pada percobaan agronomi yang melibatkan faktor genotipe dan faktor lingkungan. Model linier percobaan ini biasanya terdiri dari pengaruh faktor utama aditif dan pengaruh interaksi dari kedua faktor tersebut. Hal menarik yang ingin dikaji dalam percobaan ini adalah pengaruh interaksi kedua faktor tersebut, yang digunakan untuk mendeteksi genotipe-genotipe yang mempunyai daya adaptasi yang tinggi di berbagai kondisi lingkungan dan bisa dimodelkan dengan satu atau lebih pola interaksi multiplikatif.
Salah satu model interaksi multiplikatif yang telah banyak digunakan untuk menjelaskan pengaruh interaksi (dalam hal ini interaksi antara genotipe dan lingkungan) dan juga biasa digunakan untuk analisis kestabilan terhadap hasil
percobaan multilokasi adalah model AMMI (Additive Main Effect and
Multiplicative Interactions). AMMI, yang sebenarnya telah dikembangkan oleh Mandel pada tahun 1961 (Husein, 2000), juga mampu menjelaskan rata-rata pengaruh genotipe dan interaksi genotipe × lingkungan dengan menggunakan pendekatan analisis komponen utama (AKU). Gauch (1990) mengemukakan bahwa model AMMI merupakan suatu model gabungan dari pengaruh aditif pada analisis ragam dan pengaruh multiplikatif pada analisis komponen utama.
2 keterangan di atas, apabila genotipe dan lingkungan disetarakan dengan faktor baris dan faktor kolom, dan interaksi genotipe × lingkungan dimodelkan dengan AMMI, yang merupakan model interaksi multiplikatif, maka kita bisa mengaplikasikan metode Marasinghe untuk menguji sumbangan faktor genotipe pada pengaruh interaksi genotipe × lingkungan. Bila suatu genotipe tertentu nyata berkontribusi terhadap interaksi genotipe × lingkungan maka genotipe tersebut berinteraksi dengan lingkungan atau bisa dikatakan daya adaptasinya di berbagai lingkungan kurang atau cenderung tidak stabil. Sebaliknya, jika genotipe tersebut tidak nyata berkontribusi terhadap interaksi genotipe × lingkungan maka genotipe tersebut tidak berinteraksi dengan lingkungan atau bisa dikatakan daya adaptasinya di berbagai lingkungan cukup tinggi atau cenderung stabil.
Pengujian subhipotesis ini membutuhkan informasi nilai kriteria uji untuk
mengetahui nilai batasan tolak H0 dan menarik kesimpulan. Karena nilai kriteria
uji tersebut belum diketahui maka salah satu cara guna mengetahui nilai tersebut
adalah dengan mengaproksimasi. Proses aproksimasi yang dilakukan
menggunakan metode Bootstrap yaitu me-resampling data dengan pengembalian.
Tujuan Penelitian
Penelitian ini bertujuan :
1. mendeskripsikan metode pengujian subhipotesis pada model AMMI,
2. mengimplementasikan metode pada tujuan pertama terhadap data percobaan
3
TINJAUAN PUSTAKA
Model Interaksi Multiplikatif pada Rancangan Faktorial Dua Faktor
Perhatikan rancangan percobaan faktorial dua faktor dengan interaksi yang
terdiri atas a faktor baris dan b faktor kolom. Misalkan yij merupakan respon dari
faktor baris ke-i pada faktor kolom ke-j, µ adalah nilai rata-rata umum, τi adalah
pengaruh faktor baris ke-i, βj adalah pengaruh faktor kolom ke-j, γij merupakan
pengaruh interaksi antara faktor baris ke-i dan faktor kolom ke-j, dan εij adalah
pengaruh acak dari faktor baris ke-i pada faktor kolom ke-j yang menyebar
Normal (0,σ2). Model rancangan tersebut ialah (Marasinghe, 1980)
ij ij j i ij
y =µ+τ +β +γ +ε
(1)
dengan asumsi τ'1a =0, 1 0
' =
b
β , 1'aΓ=0' dan Γ1b =0 jika Γ=[γij]a×b .
Marasinghe (1980) mendeskripsikan parameter interaksi γij pada model
(1) dalam bentuk bilinier berikut
∑
= = k r jr ir r ij l 1 θ α
γ , dan k ≤min(b−1,a−1)
dengan unsur-unsur dari vektor α'r =
[
α1r α2r ... αar]
, r = 1, 2,…, k,merupakan parameter interaksi faktor baris; sedangkan unsur-unsur vektor
[
r r br]
r θ θ θ
θ' = 1 2 ... , r = 1, 2,…, k, merupakan parameter interaksi faktor
kolom. Dalam ungkapan bilinier tersebut diasumsikan :
k
l l
l1 ≥ 2 ≥...≥ dan A'A= B'B = Ik
dengan A=
[
α1 α2 ... αr ... αk]
dan B =[
θ1 θ2 ... θr ... θk]
.Dengan demikian model interaksi multiplikatif dapat ditulis dalam bentuk
ij k r jr ir r j i ij l
y =µ+τ +β +
∑
α θ +ε=1 (2) atau dapat pula ditulis dalam notasi matriks seperti berikut
( )
l B E ADY =µ1a1b' +τ1b' +1aβ' + k ' +
(3)
dengan matriks data Y berordo a×b dan D(lk) adalah matriks diagonal berordo k
yang unsur-unsur diagonal utamanya ialah l1, l2, ... , lk, sedangkan E matriks
4 Dalam hal ini yang menjadi perhatian pada model (3) ialah pengujian subhipotesis terhadap parameter interaksi pengaruh faktor baris yang dapat diuji dengan menyususn hipotesis berbentuk
0 :
0 HΑ=
H dan
0 :
1 HΑ≠
H , yang artinya ada sekurang-kurangnya satu ungkapan
berbentuk H1:Hαr ≠0, 1≤r≤k
dengan H merupakan matriks kontras dan berordo s×a.
Hal ini disebabkan karena hipotesis
( )
b s k B l D H H × = Α 0 : '
0 identik dengan
a s H H × = Α 0 : 0 .
Sebagai ilustrasi, misalnya untuk menguji α1r =α2r =α3r =α4r =0
untuk r = 1, 2,…, k, maka matriks H dapat berbentuk
− × − − − × = 0 ... 0 0 ... 0 0 ... 0 ) 4 ( 3 | | | 1 1 0 0 0 1 1 0 0 0 1 1 4 3 3 a k HDengan anggapan k sudah ditentukan dan memisalkan Z=[zij] sebagai
matriks interaksi berordo a×b dengan zij = yij − yi..−y.j+ y.., maka menurut
Marasinghe (1980) hipotesis di atas dapat diuji dengan menggunakan statistik
∑
∑
∑
∑
∑∑
= = = = = =−
−
=
Λ
a i k r r b j ij k r r a i b j ijz
z
1 1 * 1 2 1 1 1 2λ
λ
(4)dengan λr merupakan akar ciri terbesar ke-r dari matriks Z’Z dan λr* merupakan
akar ciri terbesar ke-r dari matriks
(
I−H−H)
ZZ', sedangkan H− adalah matrikskebalikan Moore-Penrose dari matriks H. Hipotesis nol H0 ditolak jika Λ<qφ
dengan
(
Λ
<
φ)
=
φ
0
q
P
H . Simulasi Monte Carlo atau Bootstrap dapat digunakan5
Model AMMI (Additive Main Effect and Multiplicative Interactions)
AMMI merupakan suatu teknik analisis data percobaan dua faktor yaitu faktor genotipe dan lingkungan dengan pengaruh utama perlakuan bersifat aditif sedangkan pengaruh interaksi yang bersifat multiplikatif dimodelkan dengan model bilinier. Model AMMI merepresentasikan observasi ke dalam komponen
sistematik yang terdiri atas pengaruh utama (main effect) dan pengaruh interaksi
melalui suku-suku multiplikatif (multiplicative interactions), di samping
komponen acak sisaan atau galat. Komponen acak pada model ini diasumsikan menyebar Normal dengan ragam konstan. Berarti model percobaan faktorial dua faktor yang akan dimodelkan dengan AMMI sama seperti pada model (1), dengan genotipe merupakan faktor baris, sedangkan lingkungan sebagai faktor kolom.
Pada dasarnya analisis AMMI menggabungkan analisis ragam aditif bagi pengaruh utama perlakuan dengan analisis komponen utama dengan pemodelan bilinier bagi pengaruh interaksi yang memanfaatkan penguraian nilai singular (SVD) pada matriks interaksi, sehingga model percobaan faktorial dua faktor menjadi
( )
B EAD
Y = a b + b + a + t +
' '
' '
1 1 1
1 τ β λ
µ
(5)
dengan D
( )
λt adalah matriks diagonal berordo t yang unsur-unsur diagonalutamanya ialah λ1, λ2,..., λt, λt merupakan nilai singular untuk
komponen bilinier ke-t (λt merupakan akar ciri terbesar ke-t dari matriks ZZ’ dan
t
λ λ
λ1 ≥ 2 ≥...≥ ), dan δij adalah simpangan dari pemodelan bilinier (Crossa
dalam Mattjik dan Sumertajaya, 2002).
Asumsi-asumsi pada model AMMI identik dengan asumsi pada model
interaksi multiplikatif yang diungkapkan oleh Marasinghe (1980) dalam menyusun metode pengujian subhipotesis untuk melakukan identifikasi faktor baris (genotipe dalam model AMMI) yang memberikan kontribusi terhadap interaksi baris × kolom (genotipe × lingkungan dalam model AMMI). Oleh karena itu pengujian subhipotesis pada model AMMI dapat dilakukan dengan metode yang diusulkan oleh Marasinghe (1980). Hipotesis nol
6
dengan H merupakan matriks kontras dan berordo s×a; A=
[
α1 α2 ... αt]
pada model AMMI-t dapat diuji dengan statistik Λ pada persamaan (4) untuk k = t
dengan kriteria yang sama dalam menolak hipotesis H0. Nilai kriteria uji qφ dapat
diperoleh dengan proses Bootstrap.
Perhitungan Jumlah Kuadrat AMMI
Perhitungan pengaruh aditif genotipe dan lingkungan serta jumlah kuadrat dan kuadrat tengah pada model AMMI dilakukan seperti analisis ragam pada umumnya, tetapi berdasarkan pada data rataan per genotipe × lingkungan. Pengaruh ganda genotipe dan lingkungan pada interaksi diduga dengan
... . . ...
. y y y
y
zij = ij − i − j + sehingga jumlah kuadrat interaksi dapat diturunkan
sebagai berikut :
(
)
) ( )
( . .. .. ... 2
. 2 Z Z teras r y y y y r z r GL
JK ij i j
j i ij ′ = + − − = =
∑
∑
(6)Berdasarkan teorema pada aljabar matriks bahwa teras dari suatu matriks
sama dengan jumlah seluruh akar ciri matriks tersebut,
(
)
∑
= = k r r a aZZ tr 1
' λ , maka
jumlah kuadrat untuk pengaruh interaksi komponen ke-r adalah akar ciri ke-r
pada pemodelan bilinier tersebut
( )
λr , jika analisis ragam dilakukan terhadaprataan per genotipe × lingkungan. Jika analisis ragam dilakukan terhadap data
sebenarnya maka jumlah kuadratnya adalah banyak ulangan kali akar ciri ke-r.
Pengujian masing-masing komponen ini dilakukan dengan membandingkannya
terhadap kuadrat tengah galat gabungan (Gauch dalamMattjik, 2000).
Penentuan Banyaknya Komponen AMMI
Salah satu metode yang digunakan untuk menentukan banyaknya Komponen Utama Interaksi (KUI) yang dipertahankan dalam model AMMI
(Gauch dalam Mattjik, 2000) yaitu Metode Keberhasilan Total (postdictive
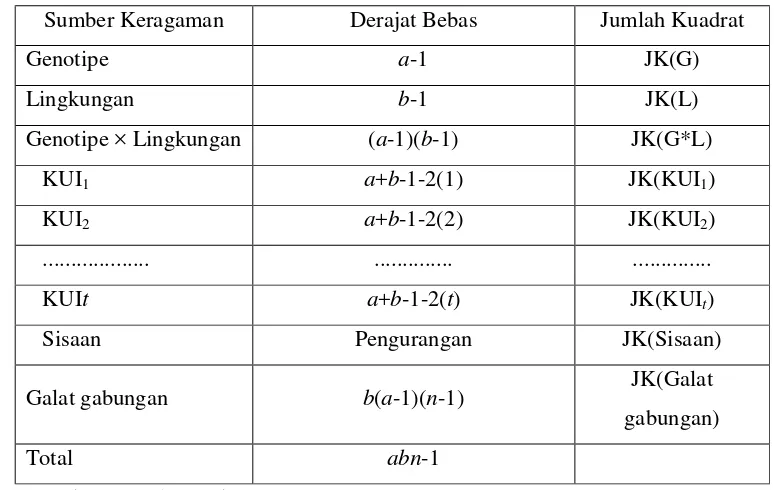
7 Sedangkan banyaknya komponen AMMI sesuai dengan banyaknya KUI yang nyata pada uji-F analisis ragam. Untuk KUI yang tidak nyata digabungkan dengan sisaan. Metode ini diusulkan oleh Gollob yang selanjutnya direkomendasikan oleh Gauch (dalam Mattjik, 2000). Tabel analisis AMMI (Tabel 1) merupakan perluasan dari tabel penguraian jumlah kuadrat interaksi menjadi beberapa jumlah kuadrat KUI.
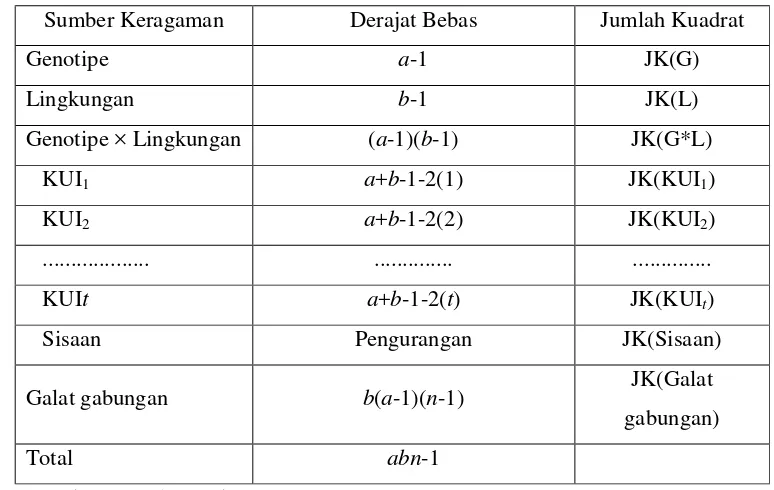
Tabel 1 Tabel Analisis Ragam AMMI
Sumber Keragaman Derajat Bebas Jumlah Kuadrat
Genotipe a-1 JK(G)
Lingkungan b-1 JK(L)
Genotipe × Lingkungan (a-1)(b-1) JK(G*L)
KUI1 a+b-1-2(1) JK(KUI1)
KUI2 a+b-1-2(2) JK(KUI2)
... ... ...
KUIt a+b-1-2(t) JK(KUIt)
Sisaan Pengurangan JK(Sisaan)
Galat gabungan b(a-1)(n-1) JK(Galat
gabungan)
Total abn-1
8
BAHAN DAN METODE
Bahan
Bahan yang akan digunakan dalam penelitian ini merupakan data sekunder hasil percobaan multilokasi tanaman jagung hibrida yang dilakukan oleh Bioseed Genetic International, INC bekerjasama dengan PT. Mitra Kreasidharma pada bulan Juli 2006 sampai dengan April 2007. Percobaan dilakukan di 16 lokasi
(sebagai faktor lingkungan, b = 16) dengan a sebanyak 12 genotipe seperti
tercantum pada Lampiran 1 dan Lampiran 2, serta di setiap kombinasi genotipe
dan lokasi diulang sebanyak tiga kali (n = 3). Sedangkan data mentah penelitian
ini telah terlampir di Jaya (2009).
Peubah respon yang diamati adalah hasil panen (ton/ha) yang diukur dari hasil kering jagung dengan kadar air maksimum 15%, sedangkan karakteristik genotipe (peubah amatan lain) selain hasil yang juga digunakan dalam pengelompokan genotipe adalah
a. Kadar air saat panen yaitu kadar air dari hasil panen jagung dalam persentase
yang diukur saat panen.
b. Umur berbunga betina yaitu jumlah hari dari tanam sampai 50% tanaman
telah keluar rambut tongkol.
c. Berat tongkol panen yaitu rataan berat tongkol pada saat panen dalam satuan
ton/ha.
Metode Analisis
Pengujian subhipotesis memerlukan nilai statistik uji (Λ) dan nilai kriteria
uji (qφ) untuk menarik kesimpulan hasil pengujian. Karena distribusi Λ belum
diketahui sehingga didekati dengan distribusi empirik guna mengaproksimasi nilai
kriteria uji. Nilai kriteria uji bagi sebaran Λ ditentukan dengan metode Bootstrap.
Hasil metode ini akan baik jika n > 4 agar proses Bootstrap, resampling data
9
1. Acuan kemiripan karakteristik interaksi genotipe × lingkungan : gunakan
biplot melalui AMMI-2 terhadap data.
2. Acuan korelasi peubah respon dengan peubah amatan lainnya : gunakan
analisis gerombol hierarki dengan jarak Mahalanobis.
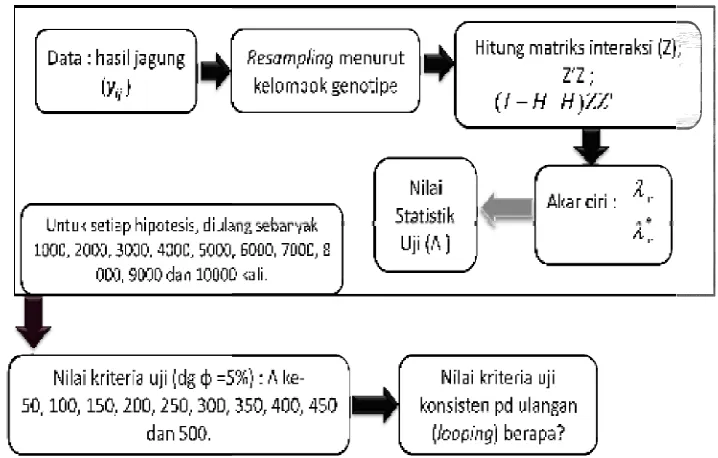
Tahapan yang dilakukan dalam pengujian subhipotesis adalah
1. Persiapan data untuk proses Bootstrap.
Selama n < 4 lakukan pengelompokan genotipe dengan salah satu metode yang telah dijelaskan di atas.
2. Pemodelan peubah respon dengan model AMMI.
Jika langkah (1) tidak dikerjakan, maka peubah respon pada model AMMI berupa data semula. Dalam hal lainnya peubah respon tersebut berupa hasil dari pengelompokan pada langkah (1). Misalkan hasilnya berupa model
AMMI-t.
3. Menentukan statistik Λ untuk pengujian subhipotesis H0:HΑ=0, dengan
langkah berikut :
Rumuskan matriks kontras H.
Hitung matriks Z.
Hitung t akar ciri terbesar dari matriks Z’Z, katakan λ1, λ2, ..., λt.
Hitung t akar ciri terbesar dari matriks
(
I−H−H)
ZZ', katakanλ1*, λ2*,…, λt*.
Hitung statistik Λ.
4. Penentuan nilai kriteria uji qφ dengan metode Bootstrap.
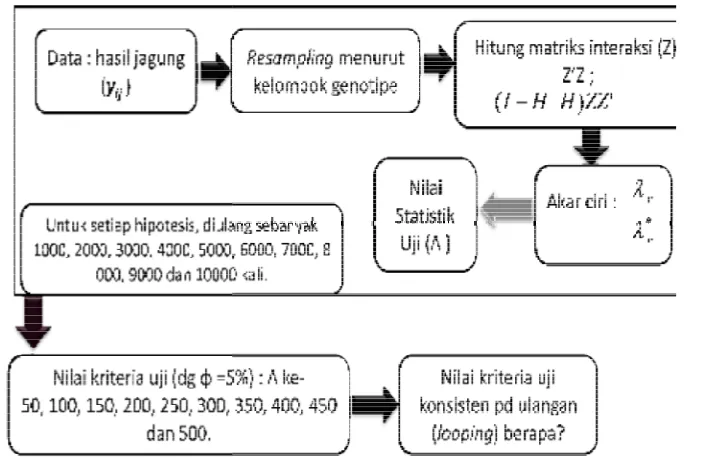
Proses resampling dilakukan dengan pengembalian diulang sebanyak
1000, 2000, 3000, 4000, 5000, 6000, 7000, 8000, 9000 dan 10000 kali
untuk melihat kekonsistenan qφ. Proses resampling terlihat pada
Gambar 1.
Menentukan nilai qφ yang konsisten untuk setiap pengujian, berdasarkan
Gambar 1 Proses Resamp
5. Pengujian subhipotesis pa
Mengambil keputusan
Tolak H0:HΑ =0
Menarik kesimpulan b
Penelitian ini mengg pemodelan AMMI, MINITA genotipe dan R versi 2.8.0 dip data percobaan serta proses a
pling untuk Aproksimasi Nilai Kriteria Uji
pada langkah (3).
an dengan kriteria berikut
jika Λ<qφ dan ( ) 5%
0 Λ<qφ =
PH
n berdasarkan hasil pengujian tersebut.
ggunakan perangkat lunak GenStat versi 8 un
AB versi 14 dan SAS versi 9.1 untuk pengelompok
dipakai dalam proses perhitungan nilai statiatik Λ
aproksimasi nilai kriteria uji.
11
HASIL DAN PEMBAHASAN
Pengelompokan Genotipe
Data percobaan yang digunakan dalam penelitian ini mempunyai ulangan hanya tiga, oleh karena itu dilakukan pengelompokan genotipe guna
memperbanyak ulangan agar variasi data yang tersedia dalam proses resampling
memadai. Pengelompokan genotipe dilakukan dengan dua cara yaitu
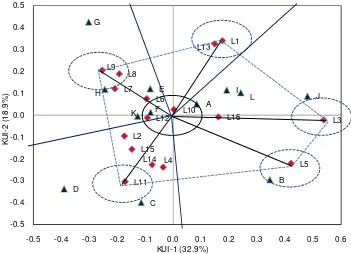
1. Pengelompokan genotipe menggunakan biplot melalui AMMI-2. Berdasarkan
hasil biplot pada Gambar 2, kedua belas genotipe dikelompokan menjadi lima kelompok yaitu
a. Kelompok I (KI) : genotipe A (BIO 9900), F (BC 41399) dan K (P-12).
b. Kelompok II (KII) yaitu genotipe B (BIO 1263).
c. Kelompok III (KIII) yaitu genotipe C (BIO 1169) dan D (BC 42521).
d. Kelompok IV (KIV) yaitu genotipe E (BC 42683), G ( BC 2630) dan H (C
42882-A).
e. Kelompok V (KV) yaitu genotipe I (BIO 9899), J (BISI-2) dan L (C-7 ).
Gambar 2 Biplot melalui AMMI 2 untuk Hasil Panen A B C D E F G
H I J
K L L1 L2 L5 L4 L3 L6 L7 L8 L9 L10 L11 L12 L13 L14 L15 L16 -0.5 -0.4 -0.3 -0.2 -0.1 0.0 0.1 0.2 0.3 0.4 0.5
-0.5 -0.4 -0.3 -0.2 -0.1 0.0 0.1 0.2 0.3 0.4 0.5 0.6
12
2. Pengelompokan genotipe menggunakan analisis gerombol hierarki dengan
jarak Mahalanobis, dengan acuan korelasi peubah respon hasil panen dengan peubah amatan lainnya, dalam hal ini adalah komponen hasil panen. Menurut Nur et al, (2007), komponen hasil panen yang dapat dijadikan indikator hasil panen adalah jumlah tanaman panen, umur berbunga betina, berat tongkol, dan kadar air panen. Berdasarkan nilai korelasi pada Tabel 2, komponen hasil panen yang nyata berkorelasi dengan hasil panen adalah peubah kadar air saat panen, umur berbunga betina dan berat tongkol. Oleh karena itu pengelompokan genotipe dilakukan menggunakan kriteria kemiripan kadar air saat panen, umur berbunga betina, berat tongkol dan hasil panen.
Tabel 2 Korelasi Komponen Hasil Penen dengan Hasil Panen Kadar
Air Panen
Jumlah Tanaman
Panen
Jumlah Tongkol
Panen
Umur Berbunga
Betina
Berat Tongkol
Nilai
Korelasi 0.189 -0.021 0.058 0.259 0.624
P-value 0.000 0.613 0.166 0.000 0.000
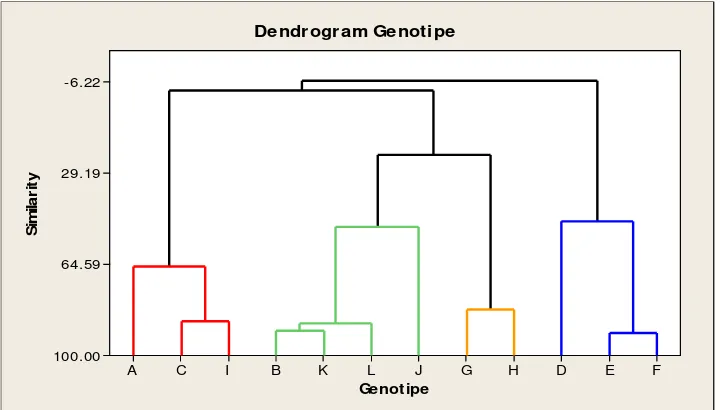
Berdasarkan dendrogram pada Gambar 3, keduabelas genotipe
diklasifikasikan ke dalam empat kelompok yaitu :
a. Kelompok 1 (K1) terdiri atas genotipe A (BIO 9900), C (BIO 1169) dan I
(BIO9899).
b. Kelompok 2 (K2) terdiri atas genotipe B (BIO 1263), J (BISI-2), K (P-12)
dan L (C-7).
c. Kelompok 3 (K3) terdiri atas genotipe G (BC 2630) dan H (C 42882-A).
d. Kelompok 4 (K4) terdiri atas genotipe D (BC 42521), E (BC 42683) dan F
13 Gambar 3 Dendrogram Genotipe Berdasarkan Karakteristik Kadar Air Panen,
Umur Berbunga Betina, Berat Tongkol dan Hasil Panen
Pengelompokan genotipe dengan biplot melalui AMMI-2 menghasilkan kelompok yang beranggotakan satu genotipe, berarti masih ada kelompok yang mempunyai ulangan tiga (n < 4). Oleh karena itu hasil pengelompokan yang digunakan dalam analisis selanjutnya adalah pengelompokan berdasarkan kriteria kemiripan kadar air saat panen, umur berbunga betina, berat tongkol dan hasil panen.
Pengujian Pengaruh Interaksi Kelompok Genotipe × Lingkungan
Sebelum mengkaji pengaruh interaksi kelompok genotipe × lingkungan. kita harus menguji pengaruh interaksi tersebut nyata atau tidak dengan melakukan analisis ragam dari respon hasil penen. Analisis ragam yang dilakukan harus memenuhi asumsi normalitas galat dan kehomogenan ragam. Oleh karena itu
dilakukan transformasi akar kuadrat sesuai dengan hasil analisis Box Cox
Transformation pada Lampiran 4 dengan nilai lamda optimal sebesar 0.50. Setelah dilakukan transformasi akar kuadrat, asumsi normalitas galat dan kehomogenan ragam telah terpenuhi seperti tercantum pada Lampiran 5. Hasil analisis ragam dari respon hasil panen pada Tabel 3 menunjukkan bahwa pengaruh interaksi kelompok genotipe × lingkungan nyata pada taraf 5% berarti
Genot ipe
S
im
ila
ri
ty
F E D H G J L K B I C A -6.22
29.19
64.59
100.00
14 kelompok genotipe memberikan respon hasil panen yang tidak sama di lingkungan yang berbeda. Selain itu juga terlihat bahwa faktor kelompok genotipe
dan lingkungan nyata pada taraf 5% (nilai-p yang kurang dari 5%) berarti ada
perbedaan rata-rata hasil panen antara kelompok genotipe dan rata-rata hasil panen untuk setiap lingkungan. Hal tersebut menunjukkan bahwa jenis genotipe dan kondisi lingkungan tempat tumbuh sangat berpengaruh terhadap hasil panen jagung.
Tabel 3 Analisis Ragam dari Hasil Panen
Sumber Keragaman db Jumlah
Kuadrat
Kuadrat
Tengah F Nilai-p
Kelompok Genotipe 3 16.402 5.467 15.29 0.000
Lingkungan 15 796.533 53.102 148.49 0.000
Kelompok
Genotipe*Lingkungan 45 31.380 0.697 1.95 0.002
Sisaan 128 45.774 0.358
Total 191 890.088
Model AMMI (Additive Main Effect and Multiplicative Interactions)
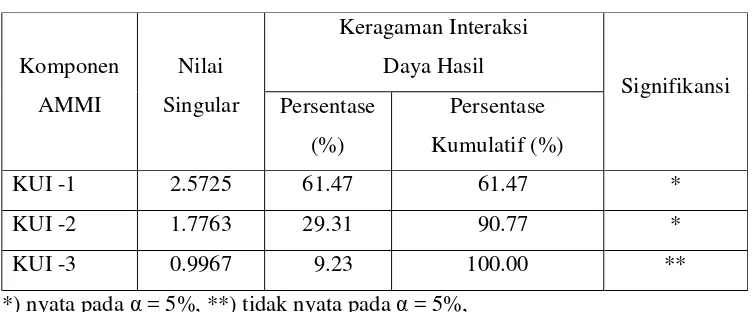
Pengaruh interaksi yang nyata pada percobaan ini biasanya dimodelkan dengan pola interaksi multiplikatif yaitu dengan model AMMI untuk mengetahui
struktur interaksi kelompok genotipe × lingkungan. Berdasarkan Tabel 4di bawah
ini, penguraian pengaruh interaksi kelompok genotipe × lingkungan untuk respon hasil panen nyata sampai KUI-2 dengan 90.77% keragaman yang mampu dijelaskan sehingga model AMMI yang digunakan cukup sampai AMMI-2 dengan model berikut :
ij r
ij jr ir r j
i ij
y
=
µ
+
τ
+
β
+
∑
λ
α
θ
+
δ
+
ε
=2
1
ˆ
ˆ
ˆ
ˆ
15 Tabel 4 Persentase Keragaman Interaksi Kelompok Genotipe × Lingkungan
Komponen AMMI
Nilai Singular
Keragaman Interaksi Daya Hasil
Signifikansi Persentase
(%)
Persentase Kumulatif (%)
KUI -1 2.5725 61.47 61.47 *
KUI -2 1.7763 29.31 90.77 *
KUI -3 0.9967 9.23 100.00 **
*) nyata pada α = 5%, **) tidak nyata pada α = 5%,
KUI adalah Komponen Utama Interaksi
Pengujian Subhipotesis pada Model AMMI 2
Hasil analisis ragam di atas menyatakan bahwa pengaruh interaksi kelompok genotipe × lingkungan nyata dan pola interaksinya signifikan pada model AMMI-2. Karena pengaruh interaksi kelompok genotipe × lingkungan nyata maka untuk mengetahui kelompok genotipe yang berkontribusi terhadap pengaruh interaksi tersebut dilakukan pengujian subhipotesis antar kelompok
genotipe, dengan matriks kontras H yang diuji tertera pada Tabel 5.
Tabel 5 Matriks Kontras Pengujian Subhipotesis
Kelompok Genotipe yang Dibandingkan Matriks Kontras (H)
K1K2 [ 1 -1 0 0 ]
K1K3 [ 1 0 -1 0 ]
K1K4 [ 1 0 0 -1 ]
K2K3 [ 0 1 -1 0 ]
K2K4 [ 0 1 0 -1 ]
K3K4 [ 0 0 1 -1 ]
K1K2K3 [ 2 -1 -1 0 ]
K1K2K4 [ 2 -1 0 -1 ]
K1K3K4 [ 2 0 -1 -1 ]
16 Pengujian tersebut memerlukan nilai kriteria uji untuk mengetahui nilai
batasan tolak H0 dan menarik kesimpulan. Karena nilai kriteria uji tersebut belum
diketahui maka salah satu cara guna mengetahui nilai tersebut adalah dengan mengaproksimasi. Proses aproksimasi yang dilakukan menggunakan metode
Bootstrap yaitu me-resampling data dengan pengembalian. Nilai kriteria uji yang
dihasilkan pada proses resampling yang diulangan sebanyak c kali merupakan
nilai statistik Λ yang ke- (5% × c) dari hasil resampling yang telah diurutkan.
Berarti untuk proses resampling yang diulang sebanyak 1000 kali diperoleh 1000
nilai Λ dan nilai kriteria ujinya adalah nilai Λ ke-50 dari 1000 nilai Λ yang telah
diurutkan. Sedangkan untuk proses resampling yang diulang sebanyak 2000,
3000, 4000, 5000, 6000, 7000, 8000, 9000 dan 10000 kali, nilai kriteria ujinya
secara berurutan adalah nilai Λ yang 100, 150, 200, 250, 300,
ke-350, ke-400, ke-450, dan ke-500.
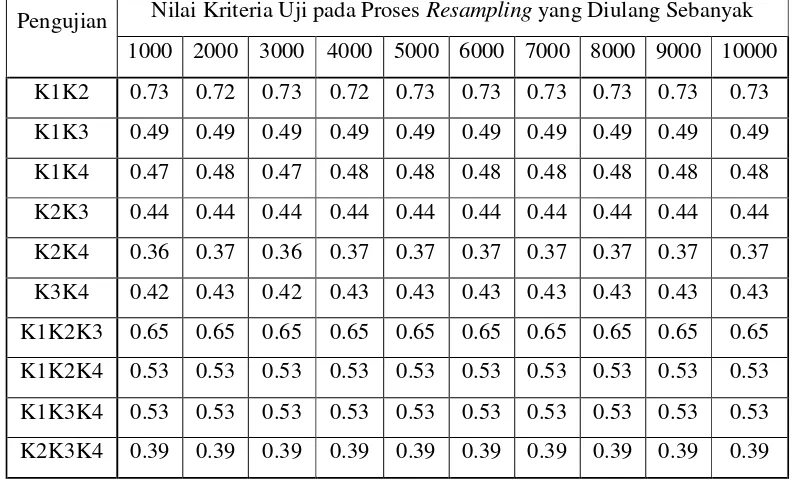
Tabel 6 Hasil Aproksimasi Nilai Kriteria Uji
Pengujian Nilai Kriteria Uji pada Proses Resampling yang Diulang Sebanyak
1000 2000 3000 4000 5000 6000 7000 8000 9000 10000
K1K2 0.73 0.72 0.73 0.72 0.73 0.73 0.73 0.73 0.73 0.73
K1K3 0.49 0.49 0.49 0.49 0.49 0.49 0.49 0.49 0.49 0.49
K1K4 0.47 0.48 0.47 0.48 0.48 0.48 0.48 0.48 0.48 0.48
K2K3 0.44 0.44 0.44 0.44 0.44 0.44 0.44 0.44 0.44 0.44
K2K4 0.36 0.37 0.36 0.37 0.37 0.37 0.37 0.37 0.37 0.37
K3K4 0.42 0.43 0.42 0.43 0.43 0.43 0.43 0.43 0.43 0.43
K1K2K3 0.65 0.65 0.65 0.65 0.65 0.65 0.65 0.65 0.65 0.65
K1K2K4 0.53 0.53 0.53 0.53 0.53 0.53 0.53 0.53 0.53 0.53
K1K3K4 0.53 0.53 0.53 0.53 0.53 0.53 0.53 0.53 0.53 0.53
K2K3K4 0.39 0.39 0.39 0.39 0.39 0.39 0.39 0.39 0.39 0.39
Penentuan nilai kriteria uji yang digunakan dalam pengambilan keputusan adalah dengan memilih nilai kriteria uji hasil aproksimasi yang nilainya telah
konsisten (sama) pada proses resampling yang diulang berapapun. Kekonsistenan
17 untuk pengujian K1K3, K2K3, K1K2K3, K1K2K4, K1K3K4 dan K2K3K4 telah
konsisten pada proses resampling yang diulang 1000 kali. Sedangkan nilai kriteria
uji pada pengujian K1K2 konsisten pada proses resampling yang diulang 5000
kali, kemudian untuk pengujian K1K4, K2K4, dan K3K4 nilai kriteria ujinya
konsisten pada proses resampling yang diulang 4000 kali.
Gambar 4 Kekonsistenan Nilai Kriteria Uji
Setelah mendapatkan nilai kriteria uji di setiap pengujian tersebut. pengujian subhipotesis yang dilakukan antar kelompok genotipe telah bisa
diambil keputusan apakah terima atau tolak Ho dengan cara membandingkan nilai
statistik uji (Λ) terhadap nilai kriteria uji yang diperoleh dari proses aproksimasi
untuk setiap pengujian, seperti tercantum pada Tabel 7.
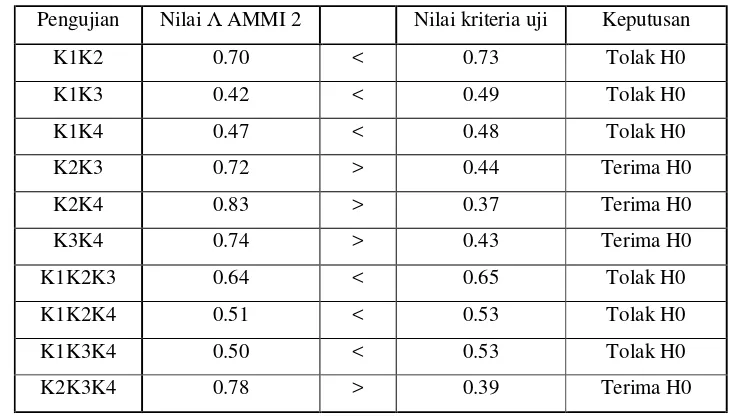
Pengujian K1K2, K1K3, K1K4, K1K2K3, K1K2K4 dan K1K3K4
mempunyai nilai Λ yang lebih kecil daripada nilai kriteria ujinya sehingga tolak
Ho, sedangkan nilai Λ pengujian K2K3, K2K4, K3K4 dan K2K3K4 lebih besar
daripada nilai kriteria ujinya sehingga terima Ho pada taraf nyata 5%. Berdasarkan
hasil pengujian ini berarti :
a. Hanya K1 saja yang nyata berkontribusi terhadap interaksi kelompok genotipe
× lingkungan berarti genotipe-genotipe yang termasuk dalam kelompok 0.3
0.35 0.4 0.45 0.5 0.55 0.6 0.65 0.7 0.75
0 2000 4000 6000 8000 10000
K1K2
K1K3
K1K4
K2K3
K2K4
K3K4
K1K2K3
K1K2K4
K1K3K4
18 genotipe 1 yaitu genotipe A (BIO 9900), C (BIO 1169) dan I (BIO9899) berinteraksi dengan lingkungan sehingga relatif kurang mampu beradaptasi di semua lingkungan yang dicobakan.
b. K2, K3 dan K4 tidak nyata memberikan kontribusi terhadap interaksi
[image:30.612.135.502.312.520.2]kelompok genotipe × lingkungan atau hanya menyumbangkan pengaruh aditif (utama) saja sehingga genotipe-genotipe yang termasuk dalam ketiga kelompok tersebut yaitu genotipe B (BIO 1263), J (BISI-2), K (P-12), L (C-7), G (BC 2630), H (C 42882-A), D (BC 42521), E (BC 42683) dan F (BC 41399) tidak berinteraksi dengan lingkungan dan relatif bisa ditanam dan beradaptasi dengan baik di semua lingkungan yang dicobakan.
Tabel 7 Hasil Pengujian Subhipotesis pada AMMI 2
Pengujian Nilai Λ AMMI 2 Nilai kriteria uji Keputusan
K1K2 0.70 < 0.73 Tolak H0
K1K3 0.42 < 0.49 Tolak H0
K1K4 0.47 < 0.48 Tolak H0
K2K3 0.72 > 0.44 Terima H0
K2K4 0.83 > 0.37 Terima H0
K3K4 0.74 > 0.43 Terima H0
K1K2K3 0.64 < 0.65 Tolak H0
K1K2K4 0.51 < 0.53 Tolak H0
K1K3K4 0.50 < 0.53 Tolak H0
K2K3K4 0.78 > 0.39 Terima H0
Pengujian subhipotesis pada model AMMI tersebut menghasilkan bahwa hanya kelompok genotipe pertama saja yang berbeda, atau terbentuk dua kelompok baru yaitu
a. Kelompok baru pertama (KB1) yang terdiri atas genotipe pada K1, yaitu
genotipe A (BIO 9900), C (BIO 1169) dan I (BIO9899).
b. Kelompok baru kedua (KB2) yang terdiri atas genotipe pada K2, K3 dan K4,
19 Sedangkan hasil pengelompokan genotipe dengan menggunakan analisis gerombol (terlihat pada Gambar 3), jika dipotong menjadi dua kelompok berarti kelompok genotipe ke-4 (K4) saja yang berbeda, dimana kedua kelompok tersebut beranggotakan sebagai berikut
a. Kelompok satu (KS), terdiri atas genotipe pada K1, K2 dan K3, yaitu genotipe
A (BIO 9900), C (BIO 1169), I (BIO9899), B (BIO 1263), J (BISI-2), K (P-12), L (C-7), G (BC 2630) dan H (C 42882-A).
b. Kelompok dua (KD), terdiri atas genotipe pada K4, yaitu genotipe D (BC
42521), E (BC 42683) dan F (BC 41399.
Berdasarkan hasil pengujian subhipotesis dan pengelompokan dengan analisis gerombol, terjadi perbedaan hasil pengelompokan genotipe. Salah satu penyebab perbedaan tersebut adalah pengelompokan genotipe dengan analisis gerombol mengacu pada peubah respon hasil panen dan peubah komponen hasil panen yang berkorelasi dengannya, sedangkan pada pengujian subhipotesis yang diuji hanya peubah respon hasil panen saja. Berarti hasil pengujian subhipotesis sensitif terhadap hasil pengelompokan.
20
SIMPULAN DAN SARAN
Simpulan
1. Metode subhipotesis Marasinghe dapat digunakan untuk menguji pengaruh
genotipe terhadap interaksi genotipe × lingkungan pada model AMMI.
Matriks kontras H pada hipotesis nol H0 merepresentasikan tujuan pengujian
tersebut.
2. Pengujian subhipotesis terhadap data percobaan jagung menunjukkan bahwa
genotipe yang memberikan kontribusi terhadap interaksi genotipe × lingkungan adalah genotipe A (BIO 9900), C (BIO 1169) dan I (BIO9899).
Saran
Perlu dicari metode lain dalam aproksimasi sebaran Λ, yang tidak
21
DAFTAR PUSTAKA
Gauch, J.R. 1990. Full and Reduced Models for Yield Trials. Theoritical and Applied Genetics. 80: 153-160.
Hussein, M.A., Bjornstad, and Aastveit. 2000. SASG × ESTAB: A SAS
program for computing genotype × environment stability statistics. Agron.
J. 92:454-459.
Jaya, I.G.D.N.M.2009. Analisis Interaksi Genotipe × Lingkungan Menggunakan Model Persamaan Struktural [tesis]. Bogor : Sekolah Pascasarjana, Institut Pertanian Bogor.
Marasinghe, M.G. 1980. Testing Subhypothesis In The Multiplicative Interaction Model [dissertation]. Kansas : Department of Statistics. Kansas State University.
Mattjik, A.A. 2000. Pendugaan Data Hilang dengan Algoritma EM-AMMI pada
Percobaan Lokasi Ganda. Forum Statistika dan Komputasi. Vol. 5 No. 1.
Mattjik, A.A., dan Sumertajaya, I.M. 2002. Perancangan Percobaan dengan Aplikasi SAS dan Minitab. Bogor : IPB Press.
Nur, A., Isnaeni, M., Iriany, R.N., dan Takdir, A. 2007. Stabilitas Komponen Hasil sebagai Indikator Stabilitas Hasil Genotipe Jagung Hibrida.
Penelitian Pertanian Tanaman Pangan Vol. 26 No. 2 : 106-107.
22 Lampiran 1 Deskripsi Lokasi Penelitian
Propinsi Kabupaten Kecamatan Desa Musim 2006/2007
Kemarau Hujan
Jawa Tengah Boyolali Banyodono Ketaon L1
Sulawesi
Selatan Barru Barru Kemiri L2
Sulawesi
Selatan Maros Moncongloe
Moncongloe
Bulu L3
Lampung Metro Metro Timur Yoso Mulyo L14 L4
Lampung Central
Lampung Ratu Nuban Sido waras L5
Jawa Timur Jombang Kedung
Mulyo Brodot L6
Jawa Timur Malang Tumpang Wringinsongo L7
Sumatera Utara
Deli
Serdang Namo Rambe Kuta Tengah L12 L8
Sumatera Utara
Serdang
Bedagai Sei Rampah
Cempedak
Lobang L9
Jawa Barat Kota Bogor Bogor Barat Pabuaran L10
Jawa Tengah Klaten Gemblengan Kalikotes L11
Sumatera
Utara Langkat Binjai Sambirejo L13
Jawa Timur Jember Ambulu Pontang L15
23 Lampiran 2 Kode genotipe
Kode Genotipe Asal
A BIO 9900 Bioseed
B BIO 1263 Bioseed
C BIO 1169 Bioseed
D BC 42521 Bioseed
E BC 42683 Bioseed
F BC 41399 Bioseed
G BC 2630 Bioseed
H C 42882 –A Bioseed
I BIO 9899 Bioseed
J BISI – 2 PT. BISI
K P – 12 PT. Dupont
24 Lampiran 3 Persentase Keragaman Interaksi Genotipe × Lokasi
Komponen AMMI
Nilai Singular
Keragaman Interaksi Daya Hasil
Signifikansi Persentase
(%)
Persentase Kumulatif (%)
KUI 1 0.7965 32.88 32.88 *
KUI 2 0.6034 18.87 51.75 *
KUI 3 0.5285 14.48 66.23 *
KUI 4 0.4664 11.27 77.50 *
KUI 5 0.3977 8.20 85.70 *
KUI 6 0.3239 5.44 91.13 **
KUI 7 0.2457 3.13 94.26 **
KUI 8 0.2203 2.51 96.78 **
KUI 9 0.1768 1.62 98.40 **
KUI 10 0.1464 1.11 99.51 **
KUI 11 0.0974 0.49 100.00 **
KUI 12 0.0000 0.00 100.00 **
*) nyata pada α = 5%, **) tidak nyata pada α = 5%, KUI adalah Komponen
25
Lampiran 4 Analisis Box Cox Transformation
Analisis Box Cox Transformation untuk hasil panen diperoleh nilai lamda
optimum adalah 0.50 sehingga data hasil panen ditransformasi akar kuadrat untuk memenuhi asumsi normalitas galat dan kehomogenan ragam pada analisis ragam.
Lambda
S
tD
e
v
5.0 2.5
0.0 -2.5
-5.0 12
10
8
6
4
2
0
Low er CL Upp er CL
Limit
Lambda
0.50 ( using 95.0% confidence) Estimate 0.54 Low er CL 0.34 Upper CL 0.74 Round ed Valu e
Lampiran 5 Uji Asumsi dalam Normalitas Galat dan Kehomogenan Ragam pada Analisis Ragam untuk Data Hasil Panen
L O K A S I _ 1
95% Bonferroni Confidence I ntervals for StDevs
16 15 14 13 12 11 10 9 8 7 6 5 4 3 2 1
0.25 0.20
0.15 0.10
0.05 0.00
Bartlet t's Test
0.237 Test Stat ist ic 20.01 P-Value 0.076
Levene's Test Test Stat ist ic 1.15 P-Value
27 Lampiran 6 Program R untuk pengujian subhipotesis pada model AMMI 2
{
awal<-data_interaksi baru<-matrix(awal,4,16) data<-t(baru)
rataancol <- NULL for (i in 1:4) {
rataancol <- c(rataancol.mean(data[,i])) }
colom <- matrix(rataancol,16,4,T) rataanrow <- NULL
for (i in 1:16) {
rataanrow <- c(rataanrow.mean(data[I,])) }
row <- matrix(rataanrow,16,4) rataanrowcol <- mean(data) zt<-data-colom-row+rataanrowcol z<-t(zt)
ztz<-zt%*%z aciri1<-eigen(ztz) ac1<-aciri1[[1]] tr1<-sum(ac1) ac11<-sum(ac1[1]) ac12<-sum(ac1[1:2]) iden4<-diag(rep(1,4))
kontrasK1K2 <- c( 1, -1, 0, 0 ) hK1K2<- matrix(kontrasK1K2,1,4) hminK1K2<-c(0.5,-0.5,0,0)
28 hinvhK1K2<-hinvK1K2%*%hK1K2
selisihK1K2<-iden4-hinvhK1K2 ztselisihK1K2<-zt%*%selisihK1K2 ztselisihzK1K2<-ztselisihK1K2%*%z aciriK1K2<-eigen(ztselisihzK1K2) acK1K2<-aciriK1K2[[1]]
trK1K2<-sum(acK1K2) ac1K1K2<-sum(acK1K2[1]) ac2K1K2<-sum(acK1K2[1:2])
lamda1K1K2<-(tr1-ac11)/(tr1-ac1K1K2) lamdaK1K2<-(tr1-ac12)/(tr1-ac2K1K2)
kontrasK1K3 <- c( 1, 0, -1, 0 ) hK1K3<- matrix(kontrasK1K3,1,4) hminK1K3<-c(0.5,0,-0.5, 0)
hinvK1K3<- matrix(hminK1K3,4,1) hinvhK1K3<-hinvK1K3%*%hK1K3 selisihK1K3<-iden4-hinvhK1K3 ztselisihK1K3<-zt%*%selisihK1K3 ztselisihzK1K3<-ztselisihK1K3%*%z aciriK1K3<-eigen(ztselisihzK1K3) acK1K3<-aciriK1K3[[1]]
trK1K3<-sum(acK1K3) ac1K1K3<-sum(acK1K3[1]) ac2K1K3<-sum(acK1K3[1:2])
lamda1K1K3<-(tr1-ac11)/(tr1-ac1K1K3) lamdaK1K3<-(tr1-ac12)/(tr1-ac2K1K3)
kontrasK1K4 <- c( 1, 0,0, -1 ) hK1K4<- matrix(kontrasK1K4,1,4) hminK1K4<-c(0.5,0,0,-0.5)
29 hinvhK1K4<-hinvK1K4%*%hK1K4
selisihK1K4<-iden4-hinvhK1K4 ztselisihK1K4<-zt%*%selisihK1K4 ztselisihzK1K4<-ztselisihK1K4%*%z aciriK1K4<-eigen(ztselisihzK1K4) acK1K4<-aciriK1K4[[1]]
trK1K4<-sum(acK1K4) ac1K1K4<-sum(acK1K4[1]) ac2K1K4<-sum(acK1K4[1:2])
lamda1K1K4<-(tr1-ac11)/(tr1-ac1K1K4) lamdaK1K4<-(tr1-ac12)/(tr1-ac2K1K4)
kontrasK2K3 <- c( 0, 1, -1, 0 ) hK2K3<- matrix(kontrasK2K3,1,4) hminK2K3<-c(0, 0.5,-0.5, 0)
hinvK2K3<- matrix(hminK2K3,4,1) hinvhK2K3<-hinvK2K3%*%hK2K3 selisihK2K3<-iden4-hinvhK2K3 ztselisihK2K3<-zt%*%selisihK2K3 ztselisihzK2K3<-ztselisihK2K3%*%z aciriK2K3<-eigen(ztselisihzK2K3) acK2K3<-aciriK2K3[[1]]
trK2K3<-sum(acK2K3) ac1K2K3<-sum(acK2K3[1])
lamda1K2K3<-(tr1-ac11)/(tr1-ac1K2K3) ac2K2K3<-sum(acK2K3[1:2])
lamdaK2K3<-(tr1-ac12)/(tr1-ac2K2K3)
30 hinvhK2K4<-hinvK2K4%*%hK2K4
selisihK2K4<-iden4-hinvhK2K4 ztselisihK2K4<-zt%*%selisihK2K4 ztselisihzK2K4<-ztselisihK2K4%*%z aciriK2K4<-eigen(ztselisihzK2K4) acK2K4<-aciriK2K4[[1]]
trK2K4<-sum(acK2K4) ac1K2K4<-sum(acK2K4[1])
lamda1K2K4<-(tr1-ac11)/(tr1-ac1K2K4) ac2K2K4<-sum(acK2K4[1:2])
lamdaK2K4<-(tr1-ac12)/(tr1-ac2K2K4)
kontrasK3K4 <- c( 0, 0, 1, -1 ) hK3K4<- matrix(kontrasK3K4,1,4) hminK3K4<-c(0, 0, 0.5, -0.5) hinvK3K4<- matrix(hminK3K4,4,1) hinvhK3K4<-hinvK3K4%*%hK3K4 selisihK3K4<-iden4-hinvhK3K4 ztselisihK3K4<-zt%*%selisihK3K4 ztselisihzK3K4<-ztselisihK3K4%*%z aciriK3K4<-eigen(ztselisihzK3K4) acK3K4<-aciriK3K4[[1]]
trK3K4<-sum(acK3K4) ac1K3K4<-sum(acK3K4[1])
lamda1K3K4<-(tr1-ac11)/(tr1-ac1K3K4) ac2K3K4<-sum(acK3K4[1:2])
lamdaK3K4<-(tr1-ac12)/(tr1-ac2K3K4)
k1k2k3<-matriksH-H1
31 ztselisihzK1K2K3<-ztselisihK1K2K3%*%z
aciriK1K2K3<-eigen(ztselisihzK1K2K3) acK1K2K3<-aciriK1K2K3[[1]]
trK1K2K3<-sum(acK1K2K3) ac1K1K2K3<-sum(acK1K2K3[1])
lamda1K1K2K3<-(tr1-ac11)/(tr1-ac1K1K2K3) ac2K1K2K3<-sum(acK1K2K3[1:2])
lamdaK1K2K3<-(tr1-ac12)/(tr1-ac2K1K2K3)
k1k2k4<- matriksH-H2
hinvhK1K2K4<- matrix(k1k2k4,4,4) selisihK1K2K4<-iden4-hinvhK1K2K4 ztselisihK1K2K4<-zt%*%selisihK1K2K4 ztselisihzK1K2K4<-ztselisihK1K2K4%*%z aciriK1K2K4<-eigen(ztselisihzK1K2K4) acK1K2K4<-aciriK1K2K4[[1]]
trK1K2K4<-sum(acK1K2K4) ac1K1K2K4<-sum(acK1K2K4[1])
lamda1K1K2K4<-(tr1-ac11)/(tr1-ac1K1K2K4) ac2K1K2K4<-sum(acK1K2K4[1:2])
lamdaK1K2K4<-(tr1-ac12)/(tr1-ac2K1K2K4)
k1k3k4<- matriksH-H3
hinvhK1K3K4<- matrix(k1k3k4,4,4) selisihK1K3K4<-iden4-hinvhK1K3K4 ztselisihK1K3K4<-zt%*%selisihK1K3K4 ztselisihzK1K3K4<-ztselisihK1K3K4%*%z aciriK1K3K4<-eigen(ztselisihzK1K3K4) acK1K3K4<-aciriK1K3K4[[1]]
trK1K3K4<-sum(acK1K3K4) ac1K1K3K4<-sum(acK1K3K4[1])
32 ac2K1K3K4<-sum(acK1K3K4[1:2])
lamdaK1K3K4<-(tr1-ac12)/(tr1-ac2K1K3K4)
k2k3k4<- matriksH-H4
hinvhK2K3K4<- matrix(k2k3k4,4,4) selisihK2K3K4<-iden4-hinvhK2K3K4 ztselisihK2K3K4<-zt%*%selisihK2K3K4 ztselisihzK2K3K4<-ztselisihK2K3K4%*%z aciriK2K3K4<-eigen(ztselisihzK2K3K4) acK2K3K4<-aciriK2K3K4[[1]]
trK2K3K4<-sum(acK2K3K4) ac1K2K3K4<-sum(acK2K3K4[1])
lamda1K2K3K4<-(tr1-ac11)/(tr1-ac1K2K3K4) ac2K2K3K4<-sum(acK2K3K4[1:2])
ABSTRACT
RUSIDA YULIYANTI. Identification of The Contributing Genotypes for Genotipe × Environment Interaction on AMMI Model. Under direction of AHMAD ANSORI MATTJIK and TOTONG MARTONO.
Additive Main Effect and Multiplicative Interactions (AMMI) Models have been utilized in analysis of the effects of the Genotype × Environment Interaction (GEI) on multienvironmental trials. However, the models could not be used for post-hoc tests of genotype contribution in the interaction effects. The tests are called tests of subhypothesis. By conducting this test, one could identify the contributing genotypes in GEI. Critical point in this test is unknown, so that it’s value must be approximated by bootstrap method. This research used maize data from PT.Kreasidharma and Bioseed Inc. In this data, there are 12 genotypes which are tested in 16 different locations. As the result, the identified contributing genotypes in GEI are BIO 9900, BIO 1169 dan BIO9899.
RINGKASAN
RUSIDA YULIYANTI. Identifikasi Genotipe yang Memberikan Kontribusi terhadap Interaksi Genotipe × Lingkungan pada Model AMMI . Di bawah bimbingan AHMAD ANSORI MATTJIK sebagai ketua dan TOTONG MARTONO sebagai anggota.
Model AMMI telah mampu menerangkan pengaruh interaksi genotipe × lingkungan, dan sebenarnya bisa dilakukan pengujian mengenai kontribusi yang diberikan oleh genotipe dan lingkungan terhadap pengaruh interaksi tersebut dengan pengujian subhipotesis yang disampaikan oleh Marasinghe (1980).
Pengujian subhipotesis memerlukan nilai statistik Λ dan nilai kriteria uji (qφ)
untuk menarik kesimpulan hasil pengujian. Karena distribusi Λ belum diketahui
sehingga didekati dengan distribusi empirik guna mengaproksimasi nilai kriteria
uji. Nilai kriteria uji bagi sebaran Λ ditentukan dengan metode Bootstrap.
Penelitian ini bertujuan mendeskripsikan metode pengujian subhipotesis pada model AMMI dan mengimplementasikan metode pada tujuan pertama terhadap data percobaan jagung dalam upaya mengidentifikasi genotipe yang berkontribusi terhadap interaksi genotipe × lingkungan. Data penelitian ini merupakan data sekunder hasil percobaan tanaman jagung hibrida yang dilakukan oleh Bioseed Genetic International, INC bekerjasama dengan PT. Mitra Kreasidharma. Percobaan dilakukan di 16 lokasi dengan 12 genotipe.
Metode subhipotesis Marasinghe dapat digunakan untuk menguji pengaruh genotipe terhadap interaksi genotipe × lingkungan pada model AMMI.
Matriks kontras H pada hipotesis nol H0 merepresentasikan tujuan pengujian
tersebut. Pengujian subhipotesis terhadap data percobaan jagung menunjukkan bahwa genotipe yang memberikan kontribusi terhadap interaksi genotipe × lingkungan adalah genotipe A (BIO 9900), C (BIO 1169) dan I (BIO9899).
1
PENDAHULUAN
Latar Belakang
Rancangan percobaan faktorial dengan klasifikasi dua faktor telah banyak diterapkan dalam berbagai bidang, salah satunya pada percobaan agronomi yang melibatkan faktor genotipe dan faktor lingkungan. Model linier percobaan ini biasanya terdiri dari pengaruh faktor utama aditif dan pengaruh interaksi dari kedua faktor tersebut. Hal menarik yang ingin dikaji dalam percobaan ini adalah pengaruh interaksi kedua faktor tersebut, yang digunakan untuk mendeteksi genotipe-genotipe yang mempunyai daya adaptasi yang tinggi di berbagai kondisi lingkungan dan bisa dimodelkan dengan satu atau lebih pola interaksi multiplikatif.
Salah satu model interaksi multiplikatif yang telah banyak digunakan untuk menjelaskan pengaruh interaksi (dalam hal ini interaksi antara genotipe dan lingkungan) dan juga biasa digunakan untuk analisis kestabilan terhadap hasil
percobaan multilokasi adalah model AMMI (Additive Main Effect and
Multiplicative Interactions). AMMI, yang sebenarnya telah dikembangkan oleh Mandel pada tahun 1961 (Husein, 2000), juga mampu menjelaskan rata-rata pengaruh genotipe dan interaksi genotipe × lingkungan dengan menggunakan pendekatan analisis komponen utama (AKU). Gauch (1990) mengemukakan bahwa model AMMI merupakan suatu model gabungan dari pengaruh aditif pada analisis ragam dan pengaruh multiplikatif pada analisis komponen utama.
2 keterangan di atas, apabila genotipe dan lingkungan disetarakan dengan faktor baris dan faktor kolom, dan interaksi genotipe × lingkungan dimodelkan dengan AMMI, yang merupakan model interaksi multiplikatif, maka kita bisa mengaplikasikan metode Marasinghe untuk menguji sumbangan faktor genotipe pada pengaruh interaksi genotipe × lingkungan. Bila suatu genotipe tertentu nyata berkontribusi terhadap interaksi genotipe × lingkungan maka genotipe tersebut berinteraksi dengan lingkungan atau bisa dikatakan daya adaptasinya di berbagai lingkungan kurang atau cenderung tidak stabil. Sebaliknya, jika genotipe tersebut tidak nyata berkontribusi terhadap interaksi genotipe × lingkungan maka genotipe tersebut tidak berinteraksi dengan lingkungan atau bisa dikatakan daya adaptasinya di berbagai lingkungan cukup tinggi atau cenderung stabil.
Pengujian subhipotesis ini membutuhkan informasi nilai kriteria uji untuk
mengetahui nilai batasan tolak H0 dan menarik kesimpulan. Karena nilai kriteria
uji tersebut belum diketahui maka salah satu cara guna mengetahui nilai tersebut
adalah dengan mengaproksimasi. Proses aproksimasi yang dilakukan
menggunakan metode Bootstrap yaitu me-resampling data dengan pengembalian.
Tujuan Penelitian
Penelitian ini bertujuan :
1. mendeskripsikan metode pengujian subhipotesis pada model AMMI,
2. mengimplementasikan metode pada tujuan pertama terhadap data percobaan
3
TINJAUAN PUSTAKA
Model Interaksi Multiplikatif pada Rancangan Faktorial Dua Faktor
Perhatikan rancangan percobaan faktorial dua faktor dengan interaksi yang
terdiri atas a faktor baris dan b faktor kolom. Misalkan yij merupakan respon dari
faktor baris ke-i pada faktor kolom ke-j, µ adalah nilai rata-rata umum, τi adalah
pengaruh faktor baris ke-i, βj adalah pengaruh faktor kolom ke-j, γij merupakan
pengaruh interaksi antara faktor baris ke-i dan faktor kolom ke-j, dan εij adalah
pengaruh acak dari faktor baris ke-i pada faktor kolom ke-j yang menyebar
Normal (0,σ2). Model rancangan tersebut ialah (Marasinghe, 1980)
ij ij j i ij
y =µ+τ +β +γ +ε
(1)
dengan asumsi τ'1a =0, 1 0
' =
b
β , 1'aΓ=0' dan Γ1b =0 jika Γ=[γij]a×b .
Marasinghe (1980) mendeskripsikan parameter interaksi γij pada model
(1) dalam bentuk bilinier berikut
∑
= = k r jr ir r ij l 1 θ α
γ , dan k ≤min(b−1,a−1)
dengan unsur-unsur dari vektor α'r =
[
α1r α2r ... αar]
, r = 1, 2,…, k,merupakan parameter interaksi faktor baris; sedangkan unsur-unsur vektor
[
r r br]
r θ θ θ
θ' = 1 2 ... , r = 1, 2,…, k, merupakan parameter interaksi faktor
kolom. Dalam ungkapan bilinier tersebut diasumsikan :
k
l l
l1 ≥ 2 ≥...≥ dan A'A= B'B = Ik
dengan A=
[
α1 α2 ... αr ... αk]
dan B =[
θ1 θ2 ... θr ... θk]
.Dengan demikian model interaksi multiplikatif dapat ditulis dalam bentuk
ij k r jr ir r j i ij l
y =µ+τ +β +
∑
α θ +ε=1 (2) atau dapat pula ditulis dalam notasi matriks seperti berikut
( )
l B E ADY =µ1a1b' +τ1b' +1aβ' + k ' +
(3)
dengan matriks data Y berordo a×b dan D(lk) adalah matriks diagonal berordo k
yang unsur-unsur diagonal utamanya ialah l1, l2, ... , lk, sedangkan E matriks
4 Dalam hal ini yang menjadi perhatian pada model (3) ialah pengujian subhipotesis terhadap parameter interaksi pengaruh faktor baris yang dapat diuji dengan menyususn hipotesis berbentuk
0 :
0 HΑ=
H dan
0 :
1 HΑ≠
H , yang artinya ada sekurang-kurangnya satu ungkapan
berbentuk H1:Hαr ≠0, 1≤r≤k
dengan H merupakan matriks kontras dan berordo s×a.
Hal ini disebabkan karena hipotesis
( )
b s k B l D H H × = Α 0 : '
0 identik dengan
a s H H × = Α 0 : 0 .
Sebagai ilustrasi, misalnya untuk menguji α1r =α2r =α3r =α4r =0
untuk r = 1, 2,…, k, maka matriks H dapat berbentuk
− × − − − × = 0 ... 0 0 ... 0 0 ... 0 ) 4 ( 3 | | | 1 1 0 0 0 1 1 0 0 0 1 1 4 3 3 a k HDengan anggapan k sudah ditentukan dan memisalkan Z=[zij] sebagai
matriks interaksi berordo a×b dengan zij = yij − yi..−y.j+ y.., maka menurut
Marasinghe (1980) hipotesis di atas dapat diuji dengan menggunakan statistik
∑
∑
∑
∑
∑∑
= = = = = =−
−
=
Λ
a i k r r b j ij k r r a i b j ijz
z
1 1 * 1 2 1 1 1 2λ
λ
(4)dengan λr merupakan akar ciri terbesar ke-r dari matriks Z’Z dan λr* merupakan
akar ciri terbesar ke-r dari matriks
(
I−H−H)
ZZ', sedangkan H− adalah matrikskebalikan Moore-Penrose dari matriks H. Hipotesis nol H0 ditolak jika Λ<qφ
dengan
(
Λ
<
φ)
=
φ
0
q
P
H . Simulasi Monte Carlo atau Bootstrap dapat digunakan5
Model AMMI (Additive Main Effect and Multiplicative Interactions)
AMMI merupakan suatu teknik analisis data percobaan dua faktor yaitu faktor genotipe dan lingkungan dengan pengaruh utama perlakuan bersifat aditif sedangkan pengaruh interaksi yang bersifat multiplikatif dimodelkan dengan model bilinier. Model AMMI merepresentasikan observasi ke dalam komponen
sistematik yang terdiri atas pengaruh utama (main effect) dan pengaruh interaksi
melalui suku-suku multiplikatif (multiplicative interactions), di samping
komponen acak sisaan atau galat. Komponen acak pada model ini diasumsikan menyebar Normal dengan ragam konstan. Berarti model percobaan faktorial dua faktor yang akan dimodelkan dengan AMMI sama seperti pada model (1), dengan genotipe merupakan faktor baris, sedangkan lingkungan sebagai faktor kolom.
Pada dasarnya analisis AMMI menggabungkan analisis ragam aditif bagi pengaruh utama perlakuan dengan analisis komponen utama dengan pemodelan bilinier bagi pengaruh interaksi yang memanfaatkan penguraian nilai singular (SVD) pada matriks interaksi, sehingga model percobaan faktorial dua faktor menjadi
( )
B EAD
Y = a b + b + a + t +
' '
' '
1 1 1
1 τ β λ
µ
(5)
dengan D
( )
λt adalah matriks diagonal berordo t yang unsur-unsur diagonalutamanya ialah λ1, λ2,..., λt, λt merupakan nilai singular untuk
komponen bilinier ke-t (λt merupakan akar ciri terbesar ke-t dari matriks ZZ’ dan
t
λ λ
λ1 ≥ 2 ≥...≥ ), dan δij adalah simpangan dari pemodelan bilinier (Crossa
dalam Mattjik dan Sumertajaya, 2002).
Asumsi-asumsi pada model AMMI identik dengan asumsi pada model
interaksi multiplikatif yang diungkapkan oleh Marasinghe (1980) dalam menyusun metode pengujian subhipotesis untuk melakukan identifikasi faktor baris (genotipe dalam model AMMI) yang memberikan kontribusi terhadap interaksi baris × kolom (genotipe × lingkungan dalam model AMMI). Oleh karena itu pengujian subhipotesis pada model AMMI dapat dilakukan dengan metode yang diusulkan oleh Marasinghe (1980). Hipotesis nol
6
dengan H merupakan matriks kontras dan berordo s×a; A=
[
α1 α2 ... αt]
pada model AMMI-t dapat diuji dengan statistik Λ pada persamaan (4) untuk k = t
dengan kriteria yang sama dalam menolak hipotesis H0. Nilai kriteria uji qφ dapat
diperoleh dengan proses Bootstrap.
Perhitungan Jumlah Kuadrat AMMI
Perhitungan pengaruh aditif genotipe dan lingkungan serta jumlah kuadrat dan kuadrat tengah pada model AMMI dilakukan seperti analisis ragam pada umumnya, tetapi berdasarkan pada data rataan per genotipe × lingkungan. Pengaruh ganda genotipe dan lingkungan pada interaksi diduga dengan
... . . ...
. y y y
y
zij = ij − i − j + sehingga jumlah kuadrat interaksi dapat diturunkan
sebagai berikut :
(
)
) ( )
( . .. .. ... 2
. 2 Z Z teras r y y y y r z r GL
JK ij i j
j i ij ′ = + − − = =
∑
∑
(6)Berdasarkan teorema pada aljabar matriks bahwa teras dari suatu matriks
sama dengan jumlah seluruh akar ciri matriks tersebut,
(
)
∑
= = k r r a aZZ tr 1
' λ , maka
jumlah kuadrat untuk pengaruh interaksi komponen ke-r adalah akar ciri ke-r
pada pemodelan bilinier tersebut
( )
λr , jika analisis ragam dilakukan terhadaprataan per genotipe × lingkungan. Jika analisis ragam dilakukan terhadap data
sebenarnya maka jumlah kuadratnya adalah banyak ulangan kali akar ciri ke-r.
Pengujian masing-masing komponen ini dilakukan dengan membandingkannya
terhadap kuadrat tengah galat gabungan (Gauch dalamMattjik, 2000).
Penentuan Banyaknya Komponen AMMI
Salah satu metode yang digunakan untuk menentukan banyaknya Komponen Utama Interaksi (KUI) yang dipertahankan dalam model AMMI
(Gauch dalam Mattjik, 2000) yaitu Metode Keberhasilan Total (postdictive
7 Sedangkan banyaknya komponen AMMI sesuai dengan banyaknya KUI yang nyata pada uji-F analisis ragam. Untuk KUI yang tidak nyata digabungkan dengan sisaan. Metode ini diusulkan oleh Gollob yang selanjutnya direkomendasikan oleh Gauch (dalam Mattjik, 2000). Tabel analisis AMMI (Tabel 1) merupakan perluasan dari tabel penguraian jumlah kuadrat interaksi menjadi beberapa jumlah kuadrat KUI.
Tabel 1 Tabel Analisis Ragam AMMI
Sumber Keragaman Derajat Bebas Jumlah Kuadrat
Genotipe a-1 JK(G)
Lingkungan b-1 JK(L)
Genotipe × Lingkungan (a-1)(b-1) JK(G*L)
KUI1 a+b-1-2(1) JK(KUI1)
KUI2 a+b-1-2(2) JK(KUI2)
... ... ...
KUIt a+b-1-2(t) JK(KUIt)
Sisaan Pengurangan JK(Sisaan)
Galat gabungan b(a-1)(n-1) JK(Galat
gabungan)
Total abn-1