ORGANISASI GENOM MITOKONDRIA CRUSTACEA
MARIA ULFA
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Organisasi Genom Mitokondria (mtDNA) Crustacea adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Agustus 2014
Maria Ulfa
ABSTRAK
MARIA ULFA. Organisasi Genom Mitokondria Crustacea. Dibimbing oleh ACHMAD FARAJALLAH dan DYAH PERWITASARI.
Kandungan gen dalam genom mitokondria sangat stabil antar taksa hewan. Mulai dari invertebrata sampai vertebrata.Tetapi organisasi gennya sangat beragam antar taksa.Penelitain ini bertujuan untuk mempelajari organisasi gen pada mtDNA Crustacea, terutama pada kejadian translokasi rRNA.Data yang digunakan pada penelitian ini adalah data organisme Crustacea yang diunduh dari situs http://ncbi.nlm.nih.gov. Skema organisasi gen dibuat menggunakan Corel Draw dan Microsoft Paint. Jumlah data yang diperoleh 292 entri, yang terdiri dari 6 Ordo, yaitu Malacostraca 70 spesies, Maxillopoda 30 spesies, Branchiopoda 6 spesies, Ostracoda, Remipedia, dan Cephalocarida, masing-masing 1 spesies. Secara keseluruhan diperoleh 63 tipe organisasi gen. Berdasarkan tipe gen yang paling banyak dari setiap Ordo, ditemukan banyak translokasi gen di sekitar klaster rRNA, yaitu NAD1, valin (V), leusin (L), dan control region (CR).
Kata kunci: filogeni, invertebrata, Malacostraca, rRNA, translokasi
ABSTRACT
1 M
MARIA ULFA. The Rearrangements of Crustaceans Mitochondrial DNA. Supervised by ACHMAD FARAJALLAH and DYAH PERWITASARI.
The content of genes in the mitochondrial genome (mtDNA) in very stable between animal taxonomical, from invertebrates to vertebrates. But the rearrangements are very diverse between taxa group. The aim of this research is to learn the rearrangement of Crustaceans mtDNA, especially the rRNA translocation. The data used in this research is Crustaceans which download from http:// ncbi.nlm.nih.gov. The gene rearrangements were created using CorelDraw and Microsoft Paint. The amount of data obtained 292 entries, which consisted of 6 Orders, 70 species of Malacostraca, 30 species of Maxillopoda, 6 species of Branchiopoda, and one for each Ostracods, Remipeds, and Cephalocarids. According to the majority type of each Order, the result showed many of translocation gene in rRNA cluster, they were NAD1, valine (V), leucine (L), and control region (CR).
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Biologi
pada
Departemen Biologi
ORGANISASI GENOM MITOKONDRIA CRUSTACEA
MARIA ULFA
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Judul Skripsi : Organisasi Genom Mitokondria Crustacea Nama : Maria Ulfa
NIM : G34070077
Disetujui oleh
Dr Achmad Farajallah Pembimbing I
Dr RR Dyah Perwitasari Pembimbing II
Diketahui oleh
Dr Iman Rusmana Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikandalam memenuhi salah satu syarat mendapatkan gelar Sarjana Sains di Departemen Biologi FMIPA IPB. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Oktober 2012 ini adalah analisis genom DNA mikondria, dengan judul Organisasi Genom Mitokondria Crustacea.
Terima kasih penulis sampaikan kepada Ibu dan Ayah beserta seluruh keluarga, yang senantiasa memberi dukungan, doa dan kasih sayang. Terima kasih penulis ucapkan kepada Bapak Dr.Ir. Achmad Farajallah, MSi.dan Ibu Dr.Ir. Dyah Perwitasari, MSc. selaku pembimbing yang senantiasa membimbing dan memberi arahan. Terima kasih kepada seluruh staf Departemen Biologi IPB atas bimbingannya selama penulis menjalani studi di IPB.Di samping itu, penghargaan penulis sampaikan kepada Bapak Dr. Iman Rusmana selaku Ketua Departemen Biologi dan juga penguji luar komisi, Ibu Dr. Tridiati selaku komdik Departemen Biologi, Ibu Dr. Utut Widyastuti selaku sekertaris FMIPA, Ibu NinaRatna Djuwita, MSi. selaku Pembimbing Akademik yang telah membantu penulis selama menjalani studi.
Penulis juga sampaikan terima kasih kepada teman-teman Biopat, Tia, Mami, Mala, Joe, Eko, Om, Fahmi, Mini, Ivan, Komal, dan lainnya.Kepada adik-adik 45, Tami, Esa, Andri, dan Mae.Kepada adik-adik-adik-adik 46, Ziah, Okta, Heca, Abduh, Ayoy dan Pipina. Kepada anggota Zoo Corner, Bu Ana, Kak Andi, Kak Wildan, Kak Silvi, Kak Bulan, Kak Sars, Kak Nunus, Kak Dini, Pak Bambang, Pak Kiranadi, Mba Puji, Mba Kanti, Ulil, Via, Ega. Kepada teman-teman 348, Yuyun, Ica, dan Nengnong. Kepada keluarga Puri Fikriyah, Niki, Gama, Eka, Meta, Rina, Bu Wiwin, dan Pak Maman.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2014
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Tujuan Penelitian 1
METODE 2
Bahan 2
Analisis Data 2
HASIL DAN PEMBAHASAN 3
Hasil 3
Pembahasan 7
SIMPULAN 9
DAFTAR PUSTAKA 10
LAMPIRAN 12
DAFTAR TABEL
1 Nama gen dan alternatifnya di situs GenBank 2
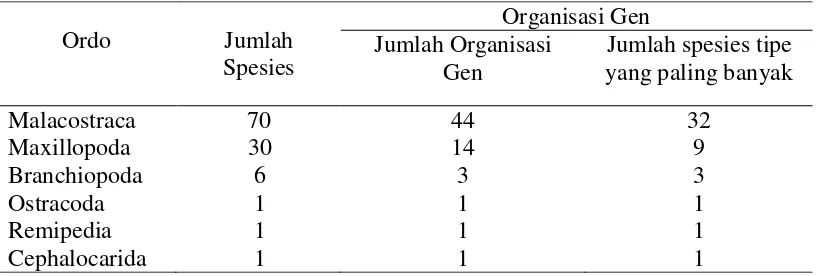
2 Data jumlah spesies dan jumlah tipe organisasi gen pada tiap Ordo 4
DAFTAR GAMBAR
1 Organisasi mtDNA Malacostraca 5
2 Organisasi mtDNA Maxillopoda 6
3 Organisasi mtDNA Branchiopoda, Ostracoda, Remipedia, dan
Cephalocarida 6
4 Tipe organisasi gen yang paling banyak pada tiap Ordo 7 5 Contoh organisasi gen mikondria pada vertebrta 7
6 Translokasi rRNA Crustacea 9
7 Perbandingan translokasi gen organisme Insekta (D. yakuba) dan Crustacea (P. notialis, P. longivcarpus, dan D. pulex) 9
DAFTAR LAMPIRAN
1 Nomor akses data mtDNA Crustacea di situs GenBank 12 2 Contoh data runutan lengkap nukleotida mtDNA anggota Crustacea
1
PENDAHULUAN
Sejak runutan lengkap genom mitokondria (mtDNA) manusia berhasil dipetakan (Anderson et al. 1981), kemudian runutan lengkap mtDNA pada beberapa spesies juga terus dilakukan. Dalam rentang waktu tahun 1981 sampai dengan tahun 2011, runutan lengkap mtDNA sudah berhasil dipetakan untuk 2458 spesies hewan (http://ncbi.nlm.nih.gov). Kecepatan pemetaan mtDNA tersebut tidak bisa dilepaskan dari popularitasnya sebagai penanda genetik dalam berbagai studi makro dan mikroevolusi pada beragam taksa hewan. Popularitas tersebut didukung oleh beberapa keunggulan mtDNA dibanding genom inti, yaitu jumlah kopi genomnya sangat banyak per sel, laju evolusinya relatif cepat, tidak mengalami rekombinasi dan diwariskan secara maternal atau uniparental.Selain itu, kandungan gen dalam mtDNA relatif sedikit dan sangat stabil antar taksa hewan sehingga konsep homologi relatif mudah tercapai untuk keperluan menelusuri hubungan kekerabatan antar taksa hewan (Boore 1999).
Bentuk mtDNA hewan berupa molekul sirkuler dengan ukuran berkisar antara 14,000 dan 39,000 pasangan basa (pb). Pada hewan, mtDNA umumnya menyandikan 37 gen, yaitu yang menyandikan rRNA ada 2 gen, tRNA ada 22 gen, menyandikan protein 13 gen, dan satu ruas noncoding yang berukuran besar yang dikenal sebagai control region (Boore 1999). Gen-gen yang saling berdekatan banyak ditemukan saling tumpang tindih tanpa adanya intron. Kodon stop beberapa gennya tidak sempurna yang kemudian mengalami modifikasi paska transkripsi (Kilpert dan Podsiadlowski 2006). Selain itu, ruas mtDNA yang menyandikan suatu gen bisa ada di utas heavy (utas positif) maupun di utas light
(utas negatif) (Boore et al. 1995).
Kelas Crustacea merupakan anggota Filum Arthropoda bersama-sama dengan Insekta, Ararachanida, Millipeda, Centipeda, Symphyla, dan Nymphonidae, sehingga saat ini hubungan filogenetik Crustacea di dalam filum Arthropoda masih menjadi perdebatan. Berdasarkan 13 gen penyandi protein mtDNA, Insecta merupakan nenek moyang dari Crustacea (Hickerson dan Cunningham 2000). Kemudian, berdasarkan beberapa karakter diagnostik yang ada di kepala (antena, mandibula dan maksila) kelompok Crustacea ini bersifat monofiletik (Koenamann 2009). Peneliti lain mengemukakan bahwa karakter yang digunakan untuk menyebutkan sifat monomorfik Crustacea bersifat pleseiomorf, terutama pada Ordo Mandibulata (Ritcher et al. 2007). Hal yang sama juga untuk Ordo-ordo yang lain dari Crustacea, sifat monofiletiknya tidak pernah disepakati, baik yang diturunkan dari data molekular (DNA dan protein), karakter morfologi pada hewan yang hidup maupun yang sudah menjadi fosil (Giribet et al. 2001, Ritcher 2002, Brusca dan Brusca 2000, Elfoson et al. 1990, Ruppert et al. 2004).
Tujuan Penelitian
2
METODE
Bahan
Bahan yang digunakan adalah runutan nukleotida lengkap mtDNA taksa Crustacea yang diunduh dari basis data nukleotida National Centre for Biotechnology Information (NCBI).
Analisis Data
Pengunduhan Data
Data runutan nukleotida lengkap mtDNA diunduh dari portal data
http://ncbi.nlm.gov yang menyatukan data mtDNA dari European Molecular Biology Laboratorium (EMBL), DNA Database of Japan (DDBJ) dan GenBank.
Kata kunci pencarian yang digunakan adalah “complete mitochondri*” pada opsi
pencarian pull-down menu nucleotida. Tanda wildcard (*) digunakan untuk meluaskan pilihan ke kata mitochondria, mitochondrion, ataupun mithocondrial. Data yang diperoleh diseleksi berdasarkan taksa dengan cara mengganti opsi pencarian pull-down menu tree menjadi taxonomic group. Semua data dalam taksa Crustacea diunduh dalam format lengkap GenBank.
Penyortiran Data
Kata kunci “complete genome” tidak spesifik digunakan untuk whole genome. Oleh karena itu, data yang telah diunduh disortir berdasarkan ukurannya, yaitu yang berukuran antara 14.000 - 25.000 pb (Boore, 1999). Setelah itu, data yang diperoleh kemudian dicermati secara lebih detail berkenaan dengan beragam
feature yang mengarah ke mitochondrial complete genome, antara lain bentuk
molekul sirkular, kode akses yang dimulai dengan “NC_”, asal organismenya “Crustacea”, dan jenis organelnya “mitochondrion”. Kode akses “NC_” adalah
kode yang dibuat oleh staf GenBank setelah data runutan nukleotida yang diunggah ke situs GenBank diverifikasi.
Pensejajaran Organisasi Gen
Nama-nama gen diseragamkan mengikuti Ogoh dan Ohmiya (2004) (Tabel 1). Semua titik awal runutan nukleotida dari data dipindahkan ke ujung 5’ gen CO1. Notasi untuk gen mengikuti features genome yang dibuat oleh setiap penulis. Nama-nama gen tersebut kemudian diurutkan menjadi organisasi gen menggunakan CorelDraw dan Microsoft Paint. Untuk memudahkan analisis, semua data dipilah-pilah berdasarkan tingkatan taksa Ordo.
Tabel 1 Nama gen dan alternatifnya di situs GenBank (http://ncbi.nlm.nih.gov)
Nama Gen Kepanjangan Alternatif
CO1 Cytochrome c oxidase subunit 1 COX1, COI, cox1, coI CO2 Cytochrome c oxidase subunit 2 COX2, COII, cox2, coII CO3 Cytochrome c oxidase subunit 2 COX3, COIII, cox3, coIII
3 ATP6 ATP synthase F0 subunit 6 ATPase subunit 8
ATP8 ATP synthase F0 subunit 8 ATPase subunit 8 ND1 NADH dehydrogenase subunit 1 NAD1, nad1 ND2 NADH dehydrogenase subunit 2 NAD2, nad2 ND3 NADH dehydrogenase subunit 3 NAD3, nad3 ND4 NADH dehydrogenase subunit 4 NAD4, nad4 ND4L NADH dehydrogenase subunit 4L NAD4L, nd4l ND5 NADH dehydrogenase subunit 5 NAD1, nd5 ND6 NADH dehydrogenase subunit 6 NAD1, nd6
12S rRNA- small subunit 12S s-RNA, rrnS, 12S rRNA, 12S ribosomal RNA 16S rRNA- small subunit 16S l-RNA, rrnL, 16S rRNA,
16S ribosomal RNA
Ala tRNA- Alanine trnA, tRNA-Ala
Cys tRNA- Cysteine trnC, tRNA-Cys
Asp tRNA- Aspartic Acid trnD, tRNA-Asp
Glu tRNA- Glutamic Acid trnE, tRNA-Glu
Phe tRNA- Phenylalanine trnF, tRNA-Phe
Gly tRNA- Glycine trnG, tRNA-Gly
His tRNA- Histidine trnH, tRNA-His
Ile tRNA- Isoleucine trnI, tRNA-Ile
Lys tRNA- Lysine trnK, tRNA-Lys
Leu tRNA- Leucine trnL, tRNA-Leu
Met tRNA- Methionine trnM, tRNA-Met
Asn tRNA- Asparagine trnN, tRNA-Asn
Pro tRNA-Proline trnP, RNA-Pro
Gln tRNA- Glutamine trnQ, tRNA-Gln
Arg tRNA- Arginine trnR, tRNA-Arg
Ser tRNA- Serine trnS, tRNA-Ser
Thr tRNA- Threonine trnT, tRNA-Thr
Val tRNA-Valine trnV, tRNA-Val
Trp tRNA- Tryptophan trnW, tRNA-Trp
Tyr tRNA- Tyrosine trnY, tRNA-Tyr
CR Control region D-loop, repeat_region
NC Noncoding region misc_feature
HASIL DAN PEMBAHASAN
Hasil
4
full disajikan dalam Lampiran 2. Semua data (109 data) mempunyai organisasi mtDNA yang saling berbeda. Data tersebut terdiri dari Malacostraca sebanyak 70 spesies, Maxillopoda 30 spesies, Branchiopoda 6 spesies, Ordo Ostracoda, Remipedia, dan Cephalocarida masing-masing 1 spesies (Tabel 2).
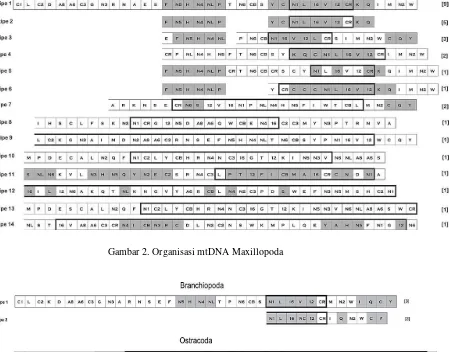
Tipe yang paling banyak diwakili oleh Ordo Malacostraca, yaitu sebanyak 44 tipe. Tipe yang paling banyak (tipe 1), yaitu 23 spesies (Gambar 1). Pada Ordo Maxillopoda, ditemukan 30 tipe organisasi gen, yang paling banyak yaitu sebanyak 9 spesies (Gambar 2). Pada Ordo Branchiopoda, ditemukan 2 tipe organisasi gen. Kedua tipe memiliki jumlah spesies yang sama, yaitu masing-masing 3 spesies.(Gambar 3). Sedangkan pada Ordo Ostracoda, Remipedia, dan Cephalocarida yang hanya diwakili oleh masing-masing 1 spesies (Gambar 3).
Berdasarkan data yang telah dipetakan, secara keseluruhan ditemukan gen komplemen (gen dengan arah penyandian yang berlawanan) sebanyak 34% dan nonkomplemen (gen dengan arah penyandian yang sama) sebanyak 66%. Skema organisasi gen dibuat berdasarkan translokasi yang ada pada tiap spesies. Tipe organisasi gen yang paling banyak jumlah spesiesnya dijadikan pembanding bagi tipe lainnya. Kecuali Ordo pada Branchiopoda, karena kedua tipe jumlah spesiesnya sama. Pada skema organisasi gen, warna abu-abu merupakan gen komplemen. Spasi kosong menunjukkan bahwa susunan gennya sama dengan organisasi gen tipe 1. Garis tebal pada tepi kotak menunjukkan translokasi rRNA yang dibatasi oleh NAD 1, valin (V), leusin (L), dan control region (CR). Sedangkan tanda kurung siku ([ ]) menunjukkan jumlah spesies (Gambar 1, 2, 3, 4, 5, dan 6).
Berdasarkan tipe yang paling banyak dari tiap Ordo, ditemukan 6 variasi translokasi rRNA (Gambar 4). Translokasi rRNA tipe A adalah yang paling banyak ditemukan pada kelompok Crustacea, yaitu 50 spesies. Pada translokasi rRNA Crustacea, ditemukan banyak delesi, insersi, komplemen, dan subtitusi (Gambar 6).
Tabel 2 Data jumlah spesies dan jumlah tipe organisasi gen pada tiap Ordo
5
6
Gambar 2. Organisasi mtDNA Maxillopoda
7
Gambar 4.Tipe organisasi gen yang paling banyak pada setiap Ordo
Gambar 5. Translokasi rRNA Crustacea dari tipe yang paling banyak pada setiap Ordo
Pembahasan
8
42,000 pb. Hal ini disebabkan oleh delesi dan insersi gen-gen mtDNA dalam jumlah besar. Selain itu, ada juga fragmen-fragmen yang diduplikasi secara berulang (Raymond et al. 1999)
Karakter translokasi gen mitokondria bisa digunakan untuk menganalisis hubungan filogenetik suatu taksa (Boore et al. 1998). Secara keseluruhan, translokasi gen pada mtDNA Crustacea sangat bervariasi. Oleh, karena itu analisis penelitian ini difokuskan pada translokasi rRNA, karena klasternya lebih kecil dibandingkan tRNA dan gen penyandi protein. Sebagai pembanding, pada kelompok vertebrata, seperti amphibi, aves, mammalia, reptil, dan pisces, translokasi gennya relatif sama dan jarang ditemukan gen kompelemen (Haring et al. 2001, Parham et al. 2006, Gilkeson et al. 2007, Garvin et al. 2010, Ryu et al.
2011). Gen-gen yang umum ditemukan di sekitar klaster tRNA adalah NAD1, valin (V), leusin (L), Fenilalanin (F) dan control region. Gen-gen tersebut juga relatif sama seperti yang ditemukan pada Crustacea, kecuali gen fenilalanin (F) (Gambar 6). Gen-gen rRNA pada organisme Arthropoda biasanya ditemukan pada posisi yang berdekatan, dan dibatasi oleh control region (CR) (Shao dan Barker 2003).
Dalam laporan Hickerson dan Cunningham (2000) disebutkan bahwa kelompok Insekta merupakan nenek moyang dari kelopmpok Crustacea. Dalam laporannya, disebutkan ada delapan translokasi gen pada kepiting Pagurus longicarpus yang identik dengan Drosopila yakuba dan Daphnia pulex, yaitu tRNA leusin (L), glisin (G), alanin (A), isoleusin (I), metionin (M), triptofan (Y), prolin (P), valin (V), glutamin (Q), AT-rich region, dan gen penyandi protein NAD2 dan NAD3 (Gambar 7). Hasil yang sama pada karakter translokasi gen penyandi protein NAD2 dan NAD3 juga dilaporkan oleh Black dan Roehrdanz (1998) dan Campbell dan Barker (1999). Hanya tRNA valin (V) saja terlibat dalam inversi transkripsi (Gambar 7). Kedelapan translokasi gen tersebut juga ditemukan pada famili Decapoda, termasuk juga gen-gen yang tidak ditemukan pada Drosopila yakuba dan Daphnia pulex, juga tidak ditemukan pada Penaeus notialis. Keidentikan karakter translokasi tersebut, diduga muncul akibat homoplasi, yaitu suatu sifat yang pada yang ditemukan pada suatu individu yang berevolusi secara independen dan tidak diturunkan dari nenek moyang.
9
Gambar 6. Contoh organisasi gen mikondria pada vertebrta
Gambar 7. Perbandingan translokasi gen organisme Insekta (D. yakuba) dan Crustacea (P. notialis,
P. longivcarpus, dan D. pulex)
SIMPULAN
10
DAFTAR PUSTAKA
Anderson S, Bankier AT, Barrel BG, de Buji MH, Coulson AR, Droujin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG. 1981. Sequence and organization of the human mithocondrial genome. Nature 290: 457-464.
Black WCI, Roehrdanz RL. 1998. Mitochondrial gene order is not conserved in Arthropods: prostriate and metastriate tick mitochondrial genomes. Mol Biol Evol 15: 1772-1785.
Boore JL, Collins TM, Stanton D, Daehler LL, Brown WM. 1995. Deducing the pattern of Arthropod phylogeny from mitochondrial DNA rearrangements.
Nature 376: 163-165.
Boore JL, Lavrov DV, Brown WM. 1998. Gene translocation links Insects and Crustaceans. Nature392: 667-668.
Boore JL. 1999. Animal mitochondial genomes. Nucleid Acids Research. 27: 1767-1780.
Brusca RC, Brusca GJ. 2004. Invertebrates. Systematic Biology: 53 (4): 664-666. Campbell NJH, Barker SC. 1998.An unprecedented major rearrangement in an
Arthropod mitochondrial genome.Mol Biol Evol 15: 1786-1787.
Elofsson R, Hessler RR. 1990. Central nervous system of Hutchinsoniella macracantha (Cephalocarida). Journal of Crustacean Biology 10: 423-439.
Finnila S. 1999. Phylogenetic analysis of mitochondrial DNA: detection of mutations in patients with occipital stroke. [disertasi]. Oulu: University of Oulu.
Garvin MR, Saitoh K, Churikov DY, Brykov VA, Gharrett AJ. 2010. Single nucleotide polymorphisms in chum salmon (Oncorhynchus keta) mitochondrial DNA derived from restriction site haplotype information.
Genome: 53(7): 501-507.
Gilkeson G, Tomlinson S, Holers VM, Rohrer B. 2007. Targeting complement factor h for treatment of diseases. [disertasi]. Colorado: University of Colorado.
Giribet G, Carranza S, Baguna J, Riurot M, Ribera C. First molecular evidance for existance of a Tardigra Arthropoda clade. 1999. Mol Biol Evol 13 (1): 76-84.
Giribet G. Edgecombe GD, Wheeler WC. 2001. Arthropod phylogeny based on eight molecular loci and morphology. Nature 413: 157-161.
Haring E, Kruckenhauser L, Gamauf A, Riesing MJ, Pinsker W. 2001. The complete sequence of the mitochondrial genome of Buteo buteo (Aves, Accipitridae) indicates an early split in the phylogeny of raptors. Mol Biol Evol 18(10): 1892-1904.
Hickerson MJ, Cunningham CW. 2000. Dramatic mitochondrial gene rearrangements in the hermit crab Pagurus longicarpus (Crustacea, Anomura).Mol Biol Evol17(4): 639-644.
11 Koenemann S, Olesen J, Alwes F, Iliffe TM, Hoenemann M, Ungerer P, Wolff C,Scholtz G. 2009. The post-embryonic development Remipedia (Crustacea): additional results and new insights. Development Genes and.Evolution219: 131-145.
Ogoh K, Ohmiya,Y. 2004. Complete mitochondrial DNA sequence of the sea-firefly, Vargula hilgendorfii (Crustacea, Ostracoda) with duplicate control regions. Gene 327 (1): 131-139.
Parham JF, Macey JR, Papenfuss TJ, Feldman CR, Turkozan O, Polymeni, Boore J. 2006. The phylogeny of Mediterranean tortoises and their close relatives based on complete mitochondrial genome sequences from museum specimens. Mol Phylogenet Evol 38 (1): 50-64.
Raymond R, Marcade I, Bouchon D, Rigaud T, Bossy JP, Grosset CS. 1999. Organization of the Large Mitochondrial Genome in the Isopod
Armadillidium vulgare. Genetics 151: 203-210.
Richter S. 2002. The Tetraconata concept:hexapod-Crustacean relationships and the phylogeny of Crustacea. Diversity & Evolution 2: 217-237.
Richter S, Olesen J, Wheeler WC. 2007. Phylogeny of Branchiopoda (Crustacea) based on a combined analysis of morphological data and six molecular loci.
Cladistics 23: 301-336.
Ruppert EE. Fox RS, Barnes RD. 2004. Invertebrate zoology: a functional evolutionary approach. Belmont: Brooks Cole Thomson.
Ryu SH, Hwang UW. 2011. Complete mitochondrial genome of the Seoul frog
Rana chosenica (Amphibia, Ranidae): comparison of Rana chosenica and
Rana plancyi.Mitochondrial DNA: 22 (3): 53-54.
Shao R, Barker SC. 2003. The highly rearranged mitochondrial genome of the plague thrips, thrips imaginis (Insecta: Thysanoptera): convergence of two novel gene boundaries and an extraordinary arrangement of rRNA Genes.
Mol Bio Evol 20(3): 362-370.
12
LAMPIRAN
Lampiran 1 Nomor akses data mtDNA Crustacea di situs GenBank
Kode Akses Ordo Kode Akses Ordo Kode Akses Ordo
NC_002184 Malacostraca NC_014854 Malacostraca NC_020311 Malacostraca NC_004251 Malacostraca NC_014883 Malacostraca NC_020312 Malacostraca NC_005037 Malacostraca NC_015073 Malacostraca NC_020313 Malacostraca NC_006081 Malacostraca NC_015607 Malacostraca NC_020314 Malacostraca NC_006281 Malacostraca NC_016015 Malacostraca NC_020351 Malacostraca NC_006880 Malacostraca NC_016184 Malacostraca NC_021458 Malacostraca NC_006891 Malacostraca NC_016192 Malacostraca NC_021753 Malacostraca NC_006916 Malacostraca NC_016925 Malacostraca NC_021754 Malacostraca NC_006992 Malacostraca NC_016926 Malacostraca NC_021971 Malacostraca NC_007010 Malacostraca NC_017600 Malacostraca NC_003979 Maxillopoda NC_007379 Malacostraca NC_017760 Malacostraca NC_005934 Maxillopoda NC_007442 Malacostraca NC_017868 Malacostraca NC_005935 Maxillopoda NC_007443 Malacostraca NC_018097 Malacostraca NC_005936 Maxillopoda NC_007444 Malacostraca NC_018778 Malacostraca NC_006293 Maxillopoda NC_008412 Malacostraca NC_019606 Malacostraca NC_007215 Maxillopoda NC_008413 Malacostraca NC_019607 Malacostraca NC_008742 Maxillopoda NC_009626 Malacostraca NC_019608 Malacostraca NC_008831 Maxillopoda NC_009679 Malacostraca NC_019609 Malacostraca NC_008974 Maxillopoda NC_011013 Malacostraca NC_019610 Malacostraca NC_012455 Maxillopoda NC_011243 Malacostraca NC_019653 Malacostraca NC_019627 Maxillopoda NC_011597 Malacostraca NC_019654 Malacostraca NC_000844 Branchiopoda NC_011598 Malacostraca NC_019655 Malacostraca NC_001620 Branchiopoda NC_012060 Malacostraca NC_019656 Malacostraca NC_004465 Branchiopoda NC_012217 Malacostraca NC_019657 Malacostraca NC_006079 Branchiopoda NC_012565 Malacostraca NC_019658 Malacostraca NC_021382 Branchiopoda NC_012566 Malacostraca NC_019659 Malacostraca NC_021383 Branchiopoda NC_012567 Malacostraca NC_019660 Malacostraca NC_005937 Cephalocarida NC_012569 Malacostraca NC_019661 Malacostraca NC_005306 Ostracoda NC_012572 Malacostraca NC_019662 Malacostraca NC_005938 Remipedia NC_012738 Malacostraca NC_020020 Malacostraca
NC_013032 Malacostraca NC_020021 Malacostraca
NC_013246 Malacostraca NC_020022 Malacostraca
NC_013480 Malacostraca NC_020023 Malacostraca
NC_013713 Malacostraca NC_020024 Malacostraca
NC_013819 Malacostraca NC_020025 Malacostraca
NC_013976 Malacostraca NC_020026 Malacostraca
NC_014339 Malacostraca NC_020027 Malacostraca
NC_014342 Malacostraca NC_020029 Malacostraca
NC_014492 Malacostraca NC_020040 Malacostraca
NC_014687 Malacostraca NC_020310 Malacostraca
13
Lampiran 2 Contoh data runutan lengkap nukleotida mtDNA anggota Crustacea serta featurenya.
LOCUS NC_013032 14113 bp DNA circular INV 15-AUG-2010 DEFINITION Metacrangonyx longipes mitochondrion, complete genome.
ACCESSION NC_013032
VERSION NC_013032.1 GI:254798853 DBLINK Project: 39687
BioProject: PRJNA39687 KEYWORDS RefSeq.
SOURCE mitochondrion Metacrangonyx longipes (subterranean amphipod) ORGANISM Metacrangonyx longipes
Eukaryota; Metazoa; Ecdysozoa; Arthropoda; Crustacea; Malacostraca; Eumalacostraca; Peracarida; Amphipoda; Gammaridea; Hadzioidea; Metacrangonyctidae; Metacrangonyx.
REFERENCE 1 (bases 1 to 14113)
AUTHORS Bauza-Ribot,M.M., Jaume,D., Juan,C. and Pons,J.
TITLE The complete mitochondrial genome of the subterranean crustacean Metacrangonyx longipes (Amphipoda): A unique gene order and
JOURNAL Submitted (24-JUL-2009) National Center for Biotechnology Information, NIH, Bethesda, MD 20894, USA
REFERENCE 3 (bases 1 to 14113) AUTHORS Pons,J.
TITLE Direct Submission
JOURNAL Submitted (03-MAR-2008) Pons J., Biodiversity and Conservation, IMEDEA (CSIC-UIB), Mallorca, Spain, Miquel Marques, 21, Esporles, Balearic Islands, 07190, SPAIN
16
tRNA complement(12176..12235) /product="tRNA-Leu"
/anticodon=(pos:complement(12204..12206),aa:Leu,seq:tag) /db_xref="GeneID:8220994"
rRNA complement(12206..13342) /product="l-rRNA"
/note="16S ribosomal RNA" /db_xref="GeneID:8221012" tRNA complement(13299..13361) /product="tRNA-Val"
/anticodon=(pos:complement(13331..13333),aa:Val,seq:tac) /db_xref="GeneID:8220987"
rRNA complement(13343..14037) /product="s-rRNA"
/note="12S ribosomal RNA" /db_xref="GeneID:8221021" misc_feature 14038..14113
/note="control region" ORIGIN
1 ttataaatta attatttttt ctcaagagta attaattaaa ggtcttataa tataataacc 61 gaaatataaa actcttagag tttgcccggt aaaaatataa ggaggctcta caggggatat
--- runutan nukleotida dihapus ---
13981 agaacaaaag ctttaaatat ttaagttttt aataaaaata atcaaacaac aattaagttt 14041 tttttttttt ttaaaaaaaa aaaaaaactt ctagacttag tctaattagt attttcaaaa 14101 tactgttaag aag
17
RIWAYAT HIDUP
Penulis lahir pada tanggal 9 Mei 1989 di Jakarta, dan merupakan anak pertama dari pasangan Sutrisno dan Lie Kui Lian.
Pada usia 5 tahun mulai memsauki bangku sekolah taman kanak-kanak di TK Tunas Harapan Bangsa (1994), SDN 06 Pagi Jakarta (1995-2001), SMPN 23 Petang Jakarta (2001-2004), SMAN 40 Jakarta (2004-2007). Pada tahun 2007 diterima di Institut Pertanian Bogor (USMI) dengan Program Biologi.
Selama masa studi di IPB, penulis aktif dalam organisasi Himpunan Mahasiswa Biologi (HIMABIO).Penulis pernah mengikuti Studi Lapangan di di Wana Wisata Cangkuang, Sukabumi Jawa Barat dengan judul laporan “Lumut
Sejati di Cangkuang”. Pada bulan Juli-Agustus 2010 penulis melakukan kegiatan Praktik Lapangan di Pusat Pendidikan dan Latihan Serikat Petani Indonesia
(Pusdiklat SPI) dengan judul “Penyilangan Kelinci di Pusat Pendidikan dan