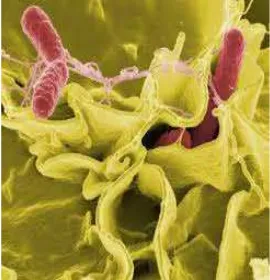
OPTIMASI POLYMERASE CHAIN REACTION (PCR)
UNTUK DETEKSI Salmonella spp. PADA UDANG SEGAR
ULFAH AMALIA
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Optimasi Polymerase Chain Reaction (PCR) untuk Deteksi Salmonella spp. pada Udang Segar adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Desember 2013
RINGKASAN
ULFAH AMALIA. Optimasi Polymerase Chain Reaction (PCR) untuk Deteksi
Salmonella spp. pada Udang Segar. Dibimbing oleh RATIH DEWANTI-HARIYADI dan ACHMAD POERNOMO.
Udang merupakan salah satu hasil perikanan sumber protein dan sumber devisa non migas penting di Indonesia. Di samping peningkatan volume produksi baik untuk pemenuhan ekspor dan kebutuhan dalam negeri, Indonesia juga tidak lepas dari masalah penolakan ekspor udang oleh negara pengimpor. Penolakan ini disebabkan oleh berbagai alasan terutama mutu dan sanitasi, residu antibiotik, dan isu lingkungan. Keberadaan Salmonella spp. pada udang telah seringkali dilaporkan di Indonesia dan di dunia. USFDA dalam waktu 5 tahun terakhir mencatat kasus penolakan impor produk perikanan dari Indonesia karena
Salmonella spp. sebesar 65%. Standar Nasional Indonesia No. 01-2728.1-2006 menetapkan bahwa jumlah Salmonella spp. pada udang segar harus negatif dalam 25 g, karena bakteri tersebut bersifat patogen dan dapat menimbulkan gangguan kesehatan. Pengujian Salmonella spp. pada seafood umumnya dilakukan dengan menggunakan metode konvensional yang membutuhkan waktu 5 hari. Berbagai upaya dilakukan untuk mempercepat metode deteksi Salmonella spp., diantaranya dengan metode PCR (Polymerase Chain Reaction). Variasi batas deteksi dan akurasi untuk mendeteksi Salmonella spp. telah diamati dengan penggunaan primer yang berbeda, diantaranya primer penyandi gen oriC yang menargetkan replikasi Salmonella spp., primer penyandi gen invAdan invE yang menargetkan virulensi, primer penyandi gen ompC yang menyandikan outer membrane protein
dan primer hns yang menyandikan DNA pengikat protein. Penelitian ini bertujuan untuk mengembangkan prosedur PCR untuk mendeteksi Salmonella spp. secara langsung dari udang, dan membandingkannya dengan metode konvensional.
Penelitian dilakukan dalam 3 tahap yaitu: (a) optimasi metode PCR dengan kondisi suhu annealing yang berbeda (b) uji sensitivitas dan spesifisitas metode PCR dan (c) deteksi Salmonella spp. dari udang dengan menggunakan metode PCR terpilih dan metode konvensional. Hasil penelitian ini menunjukkan bahwa suhu annealing 64 0C optimum untuk mendeteksi fragmen gen invA Salmonella
spp. yang berukuran 284 bp. Metode PCR terpilih memiliki limit deteksi sebesar
27.81 µg/mL untuk pengenceran ekstrak DNA dan 1 cfu/mL untuk pengenceran
kultur Salmonella dengan spesifitas 100%. Metode PCR tersebut mampu
mendeteksi 6 serovar Salmonella yang berbeda (S. Enteritidis, S. Hadar, S.
Heidelberg, S. Kentucky, S. Paratyphi dan S. Typhimurium), dan tidak mengenali bakteri non Salmonella spp. Aplikasi metode PCR terpilih pada Salmonella spp. yang diisolasi dari udang segar di pasar basah, setelah tahap pengayaan selektif
menunjukkan bahwa ke-16 sampel positif mengandung Salmonella spp.
Sementara itu, metode konvensional hanya mampu mendeteksi 3 sampel sebagai
positif mengandung Salmonella spp. Penggunaan primer yang menargetkan gen
invA potensial untuk mendeteksi Salmonella spp. pada udang.
SUMMARY
ULFAH AMALIA. Polymerase Chain Reaction Optimization for Salmonella
Detection on Shrimp. Supervised by RATIH DEWANTI-HARIYADI and ACHMAD POERNOMO.
Shrimp is an important protein source and non-oil commodity for foreign trade in Indonesia. Increase in the production volume for export and domestic demand is frequently challenged with rejection of shrimp exports to the importing country. This rejection is caused by many reasons, especially quality and sanitation, antibiotic residue and environmental issues. Presence of Salmonella
spp. in shrimp has been frequently reported in Indonesia and worldwide. Within the last 5 years, USFDA recorded that 65% of the refusal of fishery products from Indonesia was due to Salmonella spp. The Indonesian National Standard 01-2728.1-2006 requires that Salmonella spp. on fresh shrimp should be negative in 25 g, because the bacteria are pathogenic and can cause health problems. Testing of Salmonella spp. in seafood is generally done using conventional methods that take 5 days. Various attempts were made to speed up the detection time of
Salmonella spp., including by PCR. Variation of detection limits and accuracy for the detection of Salmonella spp. has been observed with different primers i.e. primers oriC targeting the replication gene of Salmonella spp., invA and invE (virulence genes), ompC which encodes outer membrane proteins and hns a DNA binding protein gene. This study aims to develop a PCR procedure for the detection of Salmonella spp. in shrimp, and compare it with conventional methods.
The research was conducted in the following three phases: (a) optimization of PCR method with different annealing temperature (b) sensitivity and specificity assays of the PCR method and (c) detection of Salmonella spp. from shrimp using the PCR method selected and a conventional method. The results show that an annealing temperature of 640C is optimum to detect the 284 bp fragment of
Salmonella invA gene. The PCR based detection method has a limit detection of 27.81 µg/mL and 1 cfu/mL of viable salmonellae with 100% specificity. The PCR based assay could detect 6 different Salmonella serovars (S. Enteritidis, S. Hadar,
S. Heidelberg, S. Kentucky, S. Paratyphi and S. Typhimurium) and none of the non salmonellae isolates. Application of the PCR assay to detect Salmonella in shrimp after selective enrichment step suggested that all 16 samples are positive for Salmonella. Meanwhile the conventional method only detected 3 samples as positive for Salmonella. Use of a primer targeting invA gene is potential for detection of salmonellae in shrimp.
© Hak Cipta Milik IPB, Tahun 2013
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Teknologi Ilmu Pangan
OPTIMASI POLYMERASE CHAIN REACTION (PCR)
UNTUK DETEKSI Salmonella spp. PADA UDANG SEGAR
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2013
Judul Tesis : Optimasi Polymerase Chain Reaction (PCR) untuk Deteksi
Salmonella spp. pada Udang Segar Nama : Ulfah Amalia
NRP : F251110131
Disetujui oleh
Komisi Pembimbing
Ketua
Prof Dr Ir Ratih Dewanti-Hariyadi, MSc
Anggota
Dr Ir Achmad Poernomo, MAppSc
Diketahui oleh
Ketua Program Studi Ilmu Pangan
Prof Dr Ir Ratih Dewanti-Hariyadi, MSc
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
Tanggal Ujian: 6 November 2013
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Desember 2012 ini ialah
Salmonella spp., dengan judul Optimasi Polymerase Chain Reaction (PCR) untuk Deteksi Salmonella spp. pada Udang Segar.
Terima kasih penulis ucapkan kepada Ibu Prof Dr Ir Ratih Dewanti-Hariyadi, MSc dan Bapak Dr Ir Achmad Poernomo, MAppSc selaku pembimbing, serta Ibu Dr Ir Harsi Dewantari Kusumaningrum yang telah banyak memberikan saran. Di samping itu, penghargaan penulis sampaikan kepada Bapak Prof Hari Eko Irianto, Ibu Yusma Yennie, SPi, MSi dari Balai Besar Penelitian dan Pengembangan Pengolahan Produk dan Bioteknologi Kelautan dan Perikanan (BBP4-BKP) beserta staf, yang telah memberikan izin untuk melakukan penelitian serta membantu selama pengumpulan data. Ungkapan terima kasih juga disampaikan kepada suami, anak, ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, Desember 2013
DAFTAR ISI
DAFTAR TABEL vii
DAFTAR GAMBAR viii
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 3
Hipotesis Penelitian 3
2 TINJAUAN PUSTAKA 3
Karakteristik Salmonella spp 3 Sumber Kontaminasi dan Penyebaran Salmonella spp 5 Prevalensi Salmonella spp. pada Udang 5 Metode Deteksi Salmonella spp 8
3 METODE 10
Tempat dan Waktu 10
Bakteri Uji 11
Bahan dan Media 11
Alat 12
Prosedur 13
I. Optimasi Suhu Annealing untuk Pengembangan Metode PCR 13 II. Uji Sensitivitas dan Spesifisitas metode PCR 15 III. Deteksi Salmonella spp. pada Udang Segar dengan Metode
Konvensional dan Metode PCR Terpilih
16
4 HASIL DAN PEMBAHASAN 17
I. Suhu Annealing Optimum untuk Pengembangan Metode PCR 17 II.Sensitivitas dan Spesifisitas Metode PCR 19 III.Deteksi Salmonella spp. pada Udang Segar dengan Metode
Konvensional dan Metode PCR Terpilih
22
a. Deteksi Salmonella spp. dengan Metode Konvensional 22 b. Deteksi Salmonella spp. pada Udang Segar dengan Metode
PCR
26
5 SIMPULAN DAN SARAN 28
DAFTAR PUSTAKA 29
LAMPIRAN 37
DAFTAR TABEL
DAFTAR GAMBAR
1 Volume ekspor komoditi perikanan menurut komoditas utama 1
2 Nomenklatur Salmonella 4
3 Insiden Salmonella pada beberapa produk udang di Asia 6
4 Dominasi serotype Salmonella terkait penyakit pada manusia, seafood,
dan lingkungan perairan
7
5 Pasangan primer oligonukleotida yang digunakan untuk amplifikasi gen pada Salmonella spp.
12
6 Hasil uji Salmonella spp. pada TSIA dan LIA 16
7 Persentase koloni yang diduga Salmonella setelah uji konfirmasi pada media TSIA dan LIA
25
8 Hasil identifikasi 13 jenis bakteri bukan Salmonella spp. pada sampel udang segar
26
1 Sel Salmonella spp 3
2 Peta amplifikasi gen invA Salmonella spp. pada 284 bp dengan menggunakan pasangan primer F139 dan R141
12
5 Visualisasi DNA hasil amplifikasi gen invA S. Typhimurium pada berbagai suhu annealing, lajur 1-6 secara berurutan : 59; 59.5; 60.8; 62; 64; 64.5 0C, 7 : S. aureus sebagai kontrol negatif, lajur M merupakan
marker DNA ladder 100 bp
18
6 Visualisasi DNA hasil amplifikasi gen invA Salmonella pada gel agarosa 1.5% (TBE). Batasan PCR dalam mendeteksi gen invA Salmonella, (a). Pengenceran DNA, lajur 1-10 : DNA S. Typhimurium mulai dari 100 – 10-9; lajur 11 : DNA S. aureus sebagai kontrol negatif; lajur M : marker
DNA ladder 100 bp. (b). Pengenceran kultur, lajur 1-10 : DNA S.
Typhimurium mulai dari 109 – 1 cfu/mL; lajur 11 : DNA S. aureus
sebagai kontrol negatif; lajur M : marker DNA ladder 100 bp
20
7 Spesifisitas PCR dievaluasi melalui 6 isolat Salmonella dengan serovar berbeda dan 5 isolat bakteri non-Salmonella
21
9 Hasil positif pada media MKTTn dan RV 23
10 Koloni tipikal dan koloni atipikal Salmonella pada media BSA 23
11 Hasil positif media TSIA dan LIA pada penelitian 24
12 Hasil identifikasi Salmonella dengan Microgen GN-ID System 26
13 Visualisasi DNA hasil amplifikasi gen invA Salmonella pada gel
agarosa 1.5% (TBE). Kemampuan PCR dalam mendeteksi gen invA
Salmonella, (a). Lajur 1: S. Typhimurium sebagai kontrol positif; lajur 2-9 : sampel udang segar ke-1 sampai ke-8; lajur 10 : S. aureus sebagai kontrol negatif; lajur M : marker DNA ladder 100 bp. (b). Lajur 1: S.
Typhimuriumsebagai kontrol positif; lajur 2-9 : sampel udang segar ke-9 sampai ke-16; lajur 10 : S. aureus sebagai kontrol negatif; lajur M :
marker DNA ladder 100 bp
PENDAHULUAN
Latar Belakang
Udang merupakan salah satu hasil perikanan yang telah banyak dimanfaatkan baik sebagai sumber protein hewani maupun sebagai komoditas ekspor non-migas. Berdasarkan data Pusat Data Statistikdan Informasi, KKP (2012), tentang ekspor hasil perikanan, terlihat udang menduduki urutan pertama (Tabel 1) dalam hal volume.
Tabel 1 Volume ekspor komoditi perikanan menurut komoditas utama
Komoditi Volume per tahun (ton)
2007 2008 2009 2010 2011 Pusat Data Statistik dan Informasi, Kementerian Kelautan Perikanan(2012)
Tabel 1 menunjukkan adanya kenaikan volume ekspor pada tahun 2008 dan tahun 2011, dan penurunan volume ekspor udang pada tahun 2009 dan 2010. Penurunan volume ekspor ini diduga disebabkan oleh adanya penolakan ekspor udang dari Indonesia oleh negara importir, antara lain seperti standar mutu dan sanitasi, residu antibiotik, animal welfare, dan isu lingkungan. Cemaran yang ditemukan pada udang segar meliputi Salmonella, Vibrio parahaemolyticus dan
Vibrio cholera. Keberadaan Salmonella spp. pada ikan dan produk perikanan dari negara-negara Asia Tenggara telah dilaporkan, seperti Sri Lanka, Vietnam, Thailand, Thaiwan dan Indonesia (Phan et al., 2005). Dewanti-Hariyadi et al.
(2005) melaporkan bahwa 2 dari 32 sampel udang yang ditangkap di laut terkontaminasi Salmonella spp.; sedangkan pada sampel udang yang dipanen ditemukan 4 sampel terkontaminasi dari 32 sampel yang ada. Pada tahun 2011, USFDA mencatat 2 kasus cemaran Salmonella spp. pada produk udang dari 2 perusahaan pengolahan udang di Indonesia. Sementara itu, data lain USFDA menyebutkan pada periode Januari-Desember 2010, kasus penolakan impor tertinggi produk perikanan dari Indonesia disebabkan oleh cemaran bakteri patogen Salmonella spp. yaitu sebesar 65 %.
Salmonella spp. dapat mencemari udang dari lingkungan perairan asal udang, maupun dari penanganan pasca panen. AdanyaSalmonella dalam udang untuk ekspor mungkin disebabkan oleh penjaminan mutu yang belum memadai. Untuk memverifikasi sistem penjaminan mutu umumnya dilakukan pengujian pada produk akhir agar memenuhi standar yang ada. Meskipun tidak dapat menjamin keamanan, pengujian produk dapat meningkatkan keyakinan akan diperolehnya produk yang memenuhi standar. Umumnya pengujian Salmonella
2
Perumusan Masalah
Pengujian Salmonella spp. pada seafood umumnya dilakukan dengan menggunakan metode konvensional yang membutuhkan waktu setidaknya 5 hari untuk melengkapi analisa (Kumar et al.,2008b). Devananda (2011) menyatakan bahwa meskipun metode konvensional efektif dan telah digunakan untuk mengisolasi dan mengidentifikasi Salmonella spp. dalam makanan, tetapi prosedur yang digunakan lama dan membutuhkan banyak tenaga. Metode ini memberikan sensitivitas satu sel Salmonella spp. per 25 gram makanan, tetapi deteksinya dapat dihambat oleh adanya mikroba lain yang berkompetisi dengan
Salmonella spp. selama kultur pengayaan. Metode kuantitatif Polymerase Chain Reaction (PCR) dapat digunakan sebagai sarana yang dapat mempersingkat waktu reaksi serta meningkatkan deteksi kuantitatif kontaminasi Salmonella (Anderson
et al., 2011; Postollec et al., 2011). Penelitian oleh Kumar et al. (2008a) menyatakan bahwa sensitivitas deteksi Salmonella pada ikan, udang, mussel,
kepiting, oyster dan kerang dari pasar ikan lokal di Cochin (India), paling tinggi ditunjukkan oleh metode PCR sebesar 31.6%, diikuti dengan metode ELISA sebesar 23.7% dan metode kultur sebesar 21.3%.
Ragam batas deteksi dan akurasi telah diamati dengan penggunaan primer yang berbeda (Malorny et al., 2003). Beberapa primer dijadikan acuan untuk mengamplifikasi dan mendeteksi Salmonella spp. berdasarkan keberadaan gen yang dimilikinya, seperti primer yang mengamplifikasi 429 bp fragmen kromosom DNA (Aabo et al.,1993), primer oriC yang menargetkan replikasi
Salmonella spp. (Mc Carthy et al., 2009), invA dan invE (gen virulensi) (Shanmugasamy et al., 2011; Deekshit et al., 2013) dan ompC yang menyandikan
outer membrane protein (Devananda, 2011). PCR dapat menargetkan sekuen genetik seperti gen virulensi bakteri, dan sangat berpotensi untuk pegujian spesifik. Akan tetapi, metode cepat ini masih memerlukan tahap pengayaan. (Hyeon et al., 2010; Kobayashi et al., 2009). Pada penelitian ini dilakukan deteksi
Salmonella spp. pada udang dengan metode PCR yang diawali dengan tahap pengayaan, dengan menggunakan pasangan primer 139 F danR 141 (Rahn et al., 1992; Garrido et al., 2013) yang menyandikan gen invA. Primer ini digunakan karena gen invA terpelihara (conserved), artinya terdapat kemiripan sekuen basa oligonukleotida pada hampir semua serotipe Salmonella spp. (Galan et al., 1991).
Tujuan Penelitian
Penelitian ini bertujuan untuk :
a. mengoptimalkan prosedur PCR untuk mendeteksiSalmonellaspp.dari udang
b. mengetahui sensitivitas dan spesifisitas dari prosedur PCR
Manfaat Penelitian
Hasil dari penelitian ini diharapkan dapat memberikan informasi bagi pihak-pihak yang terlibat dalam kegiatan pengujian rutin Salmonella spp. pada udang, dan di masa yang akan datang diharapkan prosedur yang dikembangkan ini dapat diaplikasikan ke berbagai jenis pangan lainnya.
Hipotesis Penelitian
Hipotesis dalam penelitian ini adalah :
Optimasi metode PCR dengan penggunaan primer gen penyandi invA dapat dikembangkan untuk mendeteksi Salmonella spp yang diisolasi dari udang segar di Indonesia.
TINJAUAN PUSTAKA
Karakteristik Salmonella spp.
Salmonella merupakan bakteri Gram negatif berbentuk batang yang tidak membentuk spora. Bakteri ini bersifat patogen yang berbahaya bagi manusia dan hewan lainnya. Habitat utama Salmonella adalah saluran usus hewan (burung, reptil, hama tanaman) dan manusia. Salmonella juga terdapat di bagian tubuh yang lain serta di udara terutama udara yang tercemar. Dalam studi di rumah pemotongan babi, Kampelmacher menemukan Salmonella di limpa, hati, empedu, sendi, dan feses babi (Jay et al., 2005). Salmonella pada makanan ditemukan pada kacang-kacangan, salad dressing, mayonnaise, susu, dan makanan lainnya (Jay et al., 2005). Centers for Disease Control and Prevention (CDC) melaporkan kejadian luar biasa (KLB) di Amerika Serikat dimana 127 orang dari 15 negara bagian, 261 orang dari 24 negara bagian dan 84 orang dari 18 negara bagian, secara berurutan, terinfeksi akibat mengonsumsi buah mangga, melon dan ketimun yang terkontaminasi Salmonella (CDC, 2012; CDC, 2013). Selai kacang yang mengandung Salmonella juga dilaporkan menjadi penyebab KLB yang mengakibatkan 42 orang dari 20 negara bagian dirawat di rumah sakit.
4
Salmonella berukuran relatif kecil, yaitu sekitar 0.7 – 1.5 x 2.0 – 5.0 μm (Bell dan Kyriakides, 2003). Beberapa galur Salmonella bersifat dapat memfermentasi laktosa diantaranya yaitu Salmonella Heidelberg, Salmonella
Anatum, Salmonella Sendai, Salmonella Typhimurium dan Salmonella
Newwington. Berdasarkan sistem CDC, genus Salmonella spp. terdiri dari dua spesies, yang secara terperinci tersaji pada Tabel 2.
Tabel 2Nomenklatur Salmonella spp.
Posisi taksonomi Nomenklatur Genus Salmonella
Spesies dan sub spesies • enterica, terbagi menjadi 6 sub spesies : - S. enterica subsp. Enterica (I) - S. enterica subsp. Salamae (II) - S. enterica subsp. Arizonae (IIIa) - S. enterica subsp. Diarizonae (IIIb) - S. enterica subsp. Houtenae (IV) - S. enterica subsp. Indica (VI)
• bongori (V) Sumber : Brenner et al. (2000)
Salmonella hidup secara anaerobik fakultatif. Bakteri ini tidak dapat berkompetisi secara baik dengan mikroba-mikroba umum yang terdapat di dalam makanan. Oleh karena itu, pertumbuhannya sangat terhambat dengan adanya bakteri-bakteri lain, misalnya bakteri pembusuk, bakteri genus Escherichiae dan bakteri asam laktat. Salmonella biasanya bersifat motil dan mempunyai flagella peritrikus, kecuali S. Gallinarum dan S. Pullorum, karena tidak mempunyai flagella. Selain karena tidak memiliki flagella, jenis Salmonella yang bersifat tidak motil disebabkan karena kesalahan pemasangan subunit flagella atau kekurangan fungsi motorik pada anggota selnya (D’Aoust, 2000).
Salmonella umumnya dapat tumbuh pada media yang memiliki Aw di atas 0.94 dan pH 4.1-9.0 dengan pH optimum 7-7.5. Nilai pH minimum bervariasi tergantung pada serotipe, suhu inkubasi, komposisi media, aw, dan jumlah sel. Pada pH di bawah 4.1, Salmonella akan mati secara perlahan. Selain itu
Salmonella dapat tumbuh pada suhu 5.2-43°C, dengan suhu optimum 35-43°C. Berbeda dengan Staphylococcus, Salmonella tidak tahan terhadap kadar garam tinggi. Salmonella akan mati jika berada pada media dengan kadar garam di atas 9% (Food Safety Authority of Ireland, 2011).
Infeksi akibat Salmonella tergantung pada sistem kekebalan tubuh inang dan faktor virulensi Salmonella, yang disebut Salmonella Pathogenicity Islands
(SPIs), ragam bakteri dan interaksinya dengan lingkungan (Huehn et al., 2010). Berbagai SPIs yang mengkode sistem sekresi tipe III yang mentransfer protein bakteri ke sitosol sel inang. Gen SPI juga berperan dalam perkembangbiakan
Salmonella dalam sel inang dan menyebabkan infeksi sistemik (Schmidt dan Hensel, 2004; Courtneyet al., 2006). Gen SPI juga menyandikan operon mgtBC
yang diperlukan untuk kelangsungan hidup intra-makrophage. Selain itu, serovar
Sumber Kontaminasi dan Penyebaran Salmonella spp.
Salmonella secara luas tersebar pada hampir semua habitat ekologi, isolasi dari tanah, air, makanan dan saluran pencernaan dari manusia dan hewan. Kecenderungan terhadap semua Salmonella yang dapat menyebabkan infeksi sistemik maupun enterik terhadap manusia dan hewan menjadikan Salmonella
sebagai patogen yang sangat penting. Meskipun banyak perkembangan higienis pada produksi makanan, tetapi Salmonella tetap menjadi sangat penting sebagai bakteri yang menyebabkan penyakit bawaan pangan (Hui et al., 2001). Salmonella
memiliki potensi menjadi salah satu bakteri patogen penyebab penyakit pada pencernaan manusia dan hewan di dunia (D'Aoust andMaurer, 2007). Umumnya, pangan yang berkaitan dengan salmonellosis pada manusia meliputi ternak, daging sapi, babi, telur, susu dan seafood.
Salmonella banyak tersebar di alam terutama pada udara yang tercemar. Namun habitat utamanya adalah saluran usus binatang dan manusia. Bakteri ini dapat diisolasi dari sampel feses, makanan, dan sampel dari lingkungan.
Salmonella pada makanan terdapat pada kacang-kacangan, salad, mayonaise, susu (Jay, 2000). Salmonella merupakan salah satu masalah penting bagi kesehatan manusia dan hewan karena dapat menyebabkan gangguan kesehatan dan kematian. Infeksi Salmonella terutama disebabkan oleh penanganan yang buruk dan konsumsi pangan mentah ataupun kurang matang. Salmonellosis sangat mudah ditularkan dari hewan ke manusia baik secara langsung ataupun melalui perantara seperti produk makanan yang berasal dari hewan, tumbuhan dan lingkungan. Salmonella enterica Serovar Typhimurium dan Enteridis merupakan salah satu penyebab utama gastroenteritis (Jay dan Davey, 1989).
Bakteri Salmonella di laut dan perairan air tawar berasal dari limbah organik domestik, industri dan dari tempat-tempat rekreasi (sumber mata air, danau dan pantai). Keberadaan bakteri Salmonella dalam suatu perairan laut dapat diindikasikan dengan keberadaan bakteri indikator yaitu Escherichia coli karena bakteri ini sangat erat hubungannya dengan bakteri Salmonella. Semakin tinggi kandungan bakteri E. coli maka semakin positif peluang bakteri Salmonella akan ditemukan dalam suatu perairan. Bakteri Salmonella sebenarnya adalah bakteri dari air tawar, kehadirannya di laut disebabkan terbawa oleh aliran sungai atau air buangan. Keberadaannya di laut dapat menyebabkan banyak hasil laut seperti ikan, udang, kerang-kerangan dan lainnya terkontaminasi oleh bakteri Salmonella
dan Shigella.
Prevalensi Salmonella spp. pada Udang
Air laut umumnya bebas Salmonella, tetapi perairan pantai, estuaria dan karang dapat terkontaminasi dari air buangan manusia dan pertanian, sehingga
Salmonella kadang ditemukan pada ikan mentah dan kerang. Kondisi lingkungan dan insiden Salmonellosismemang berkorelasi. Hasil investigasi Martinez-Urtaza
et al. (2003) menunjukkan bahwa 2.9% dari 381 sampel kerang yang diambil dari lokasi panen positif terinfeksi Salmonella dan hanya 1.6% dari 2599 sampel kerang yang telah didepurasi yang positif terinfeksi Salmonella.
6
selama pemanenan dan pengepakan dapat menjadi penyebab tingginya kontaminasi Salmonella pada produk ikan dan kerang dari Asia. Asia merupakan penghasil separuh dari produk akuakultur dunia, dan studi isolat Salmonella
menunjukkan salah satu potensi pembawa Salmonella pada manusia di Asia berasal dari ikan dan kerang (D’Aoust 2000). Di Thailand hasil analisis terhadap air mendapatkan 984 isolat Salmonella dengan serovar yang banyak ditemukan adalah S. Weltreveden (14.5%), S. Anatum (11.5%), S. Rissen (9.5%), dan S. Derby (7.2%). Bangtrakulnonth et al. (2003) menyebutkan hasil analisis pada
seafood selama tahun 1993-2000, dari 1007 isolat yang didapat serovar S. Weltreveden juga merupakan serovar yang sering ditemukan dengan prevalensi sebesar 26%.
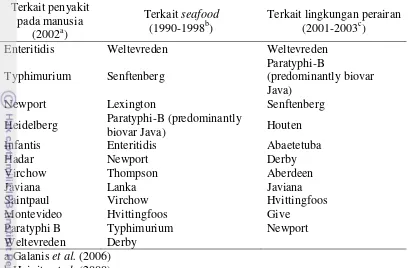
Hasil penelitian Susanto (1998) terhadap udang segar di DKI dan Jawa Barat menunjukkan bahwa udang segar yang didapat dari Kandang Haur dan Cilincing terkontaminasi oleh S. Paratyphi A, dan diduga kontaminasi berasal dari lingkungan habitat udang yang tercemar oleh kotoran manusia dan hewan. Hasil penelitian lain yang dilakukan oleh Slamet (2000) menunjukkan bahwa udang segar dari Tanjung Kait dan Gebang telah terkontaminasi oleh Salmonella dan diduga mikroorganisme tersebut adalah S. Paratyphi A. D’Aoust (2000) melaporkan beberapa insiden Salmonella pada udang yang didapat dari beberapa negara Asia hingga tahun 1995 (Tabel 3). Prevalensi Salmonella terbesar sebanyak 30% ditemukan pada udang segar yang berasal dari India pada tahun 1990.
Tabel 3 Insiden Salmonella pada beberapa produk udang di Asia
Negara Asal Produk Jumlah Sampel
yang Diuji Jumlah Positif (%) 1. India
Tabel 4 Dominasi serotype Salmonella terkait penyakit pada manusia, seafood,
Enteritidis Weltevreden Weltevreden
Typhimurium Senftenberg
Paratyphi-B
(predominantly biovar Java)
Newport Lexington Senftenberg
Heidelberg Paratyphi-B (predominantly
biovar Java) Houten Infantis Enteritidis Abaetetuba
Hadar Newport Derby
Virchow Thompson Aberdeen
Javiana Lanka Javiana
Saintpaul Virchow Hvittingfoos Montevideo Hvittingfoos Give
Paratyphi B Typhimurium Newport Weltevreden Derby
a Galanis et al. (2006) b Heinitz et al. (2000)
c Hatha et al.(1997); Koonse et al. (2005); Kumar et al. (2009a); Norhana et al.(2010)
Meratanya kontaminasi Salmonella pada seafood berasal dari perairan Asia-Pasifik dan Afrika dilaporkan karena perairan tersebut memiliki tingkat
Salmonella yang tinggi dibandingkan dengan bagian lain di dunia (Heinitz et al., 2000). Keragaman serovar terkait ikan dan produk perikanan tertinggi terdapat di Asia Tenggara diikuti Amerika Selatan (FAO, 2010).
Hatha et al. (1997) menyebutkan bahwa terdapat Salmonella pada udang segar dan beku yang dikumpulkan dari tempat penampungan, retail, penjualan, impor, tempat pengolahan dan lain-lain. Salmonella ditemukan pada udang yang diolah, peralatan dan lantai serta air yang digunakan selama proses pengolahan. Kejadian tersebut paling banyak pada produk udang segar (10–14%), diikuti dengan sampel swab lantai (4%), peralatan (2%) dan air untuk pengolahan (1%).
8
Metode Deteksi Salmonella spp.
1. Metode Konvensional
Deteksi Salmonella spp. dalam makanan dengan metode konvensional termasuk pra pengayaanselama 16 – 20 jam untuk mengetahui resusitasi dan multiplikasi dari sel sub lethal yang rusak, pengayaan selama 18 – 48 jam untuk meningkatkan rasio Salmonella spp. dibandingkan mikroba lain dan pengayaan selektif selama 24 – 48 jam untuk menunjukkan koloni tipikal Salmonella spp. Selanjutnya adalah identifikasi secara biokimia dari koloni yang diduga adalah
Salmonella spp. selama 4 – 48 jam, yang diikuti dengan serotyping (ISO 6579:2002).
Pra Pengayaan
Lactose broth (LB) dan buffered peptone water (BPW) merupakan media yang umum digunakan pada tahapan pra pengayaan Salmonella spp. BPW umum digunakan di dalam pekerjaan rutin (van Leusden et al.,1982). Pra pengayaan umum direkomendasikan untuk segala sampel yang terkontaminasi mikroba
Enterobactericeae (Varnam and Evans, 1991).
Pengayaan Selektif
Inhibitor utama yang digunakan pada pengayaan selektif Salmonella spp. adalah bile salt, tetrathionate, selenite dan dyes termasuk brilliant green, malachite green. Media yang umum digunakan adalah tetrathionate broth
(Kauffman, 1935), selenite cystine broth (North and Bartram, 1953), brilliant
green–MacConkey broth (King and Metzger, 1968) dan
Rappaport-Vassiliadis(RV) broth (RVB) (Harvey and Price, 1982).
Untuk menggunakan media RV, penting dilakukan pengecekan rasio sampel terhadap broth yang digunakan. Untuk SCB dan TTB, rasio inokulasi 1:10 umum digunakan sedangkan untuk RV, rasio inokulasi lebih kecil yaitu 1 : 100 (Varnam and Evans, 1991). SCB dan TTB diinkubasi pada suhu 37°C, sedangkan RV pada suhu 43 °C selama 24 jam. SCB dan TTB umumnya digunakan pada tahapan pengayaan selektif untuk mengisolasi Salmonella berdasarkan metode yang dikeluarkan oleh Bacteriological Analitical Manual (BAM), sedangkan pada metode ISO 6579:2002, media untuk pengayaan selektif menggunakan Muller-Kauffmann Tetrathionate-Novobiocin Broth (MKTTn).
Pemupukan Selektif
Media agar selektif untuk Salmonella spp. mengandung selective agent
Uji Konfirmasi Biokimia dan Serologi
Media yang umum digunakan untuk mengidentifikasi Salmonella spp. pada pengayaan campuran adalah triple sugar iron agar (TSI) dan lysine iron agar (LIA), sedangkan urea broth digunakan untuk identifikasi Salmonella spp. dalam pengayaan murni (Moats 1981, USFDA 2003). Uji serologi digunakan untuk mengkonfirmasi isolat-isolat secara biokimia. Uji serologi polyvalen flagellar (H), uji serological somatic (O) dan uji serologi Spicer-Edwards
direkomendasikan oleh USFDA (2003).
Metode konvensional telah lama digunakan dan efektif untuk mengisolasi dan mengidentifikasi mikroba patogen dalam makanan. Namun, tahapan-tahapan pengayaan dan karakterisasi biokimia membutuhkan waktu yang cukup lama hingga beberapa hari (Devananda, 2011)
2. Polymerase Chain Reaction (PCR)
PCR merupakan suatu teknik perbanyakan molekul DNA dengan ukuran tertentu secara enzimatik melalui mekanisme perubahan suhu (Sambrook et al.,1989). Secara ringkas, prinsip PCR dapat dijelaskan sebagai berikut : pada suhu 94-950C, DNA mengalami denaturasi yang menyebabkan pemisahan untai ganda menjadi untai tunggal. Waktu yang diperlukan untuk proses ini sekitar 30 detik pada suhu 950C atau 15 detik pada suhu 970C. Apabila DNA target mengandung banyak nukleotida Guanin/Citosin (G/C), suhu denaturasi dapat ditingkatkan. Denaturasi yang tidak lengkap akan menyebabkan renaturasi secara cepat, sedangkan waktu denaturasi yang terlalu lama dapat mempengaruhi kerja enzim Taq polymerase. Hal ini sangat berpengaruh terhadap keberhasilan proses PCR. Umumnya sebelum proses siklus PCR dimulai sering sekali dilakukan pre denaturasi selama 3-5 menit, untuk meyakinkan bahwa molekul DNA target yang ingin dilipatgandakan jumlahnya benar-benar terdenaturasi (Promega, 2001).
Suhu annealing dalam suatu tahapan PCR merupakan hal yang sangat penting untuk menghindari kesalahan penempelan primer. Primer yang baik berukuran 18-25 basa, mengandung 50-60% G + C dan memiliki suhu leleh (Tm) yang sama. Suhu annealing yang digunakan umumnya 50C di bawah Tm, dimana formula untuk menghitung Tm = 4 (G+C) + 2 (A+T). Penelitian Deekshit et al.
(2013) menghasilkan suhu 55 0C sebagai suhu annealing paling optimum untuk mendeteksi Salmonella spp. pada seafood dengan menggunakan 3 pasangan primer yang berbeda antara lain primer penyandi gen invA, gen ssaT dan gen
sseF. Muladno (2010) menyatakan bahwa kisaran suhu annealing antara 360C sampai 720C. Semakin panjang ukuran primer, semakin tinggi temperaturnya.
10
penyelesaian DNA. Akurasi dan reliabilitas tergantung pada uji templat kuantitas, kualitas dan desain optimal. PCR tidak digunakan sebagai pengganti teknik mikrobiologi konvensional, tetapi dapat dipertimbangkan karena memerlukan sedikit waktu untuk melengkapi analisa. Teknis mikrobiologi konvensional sering dianggap kurang menuntut dibanding PCR. Namun, PCR dapat mendeteksi bakteri yang tidak dapat tumbuh di pengayaan. PCR melengkapi dan meningkatkan metode mikrobiologi tradisional dengan meningkatkan kecepatan, sensitivitas, dan spesifisitas untuk mendeteksi patogen pada ikan. PCR dapat dilakukan dengan cepat di lapangan dan membatasi jumlah culture dan isolasi untuk beberapa sampel teridentifikasi positif oleh PCR untuk mengurangi beban kerja, tenaga kerja, sumber daya, dan biaya (Mufty, 2008).
Beberapa penelitian terkait deteksi Salmonella spp. dengan metode PCR telah dilaporkan. Sabarinath et al.(2007) menggunakan 3 set primer yang masing-masing menyandikan gen antara lain invA, hns dan invEuntuk melihat keragaman genetik Salmonella spp. yang diisolasi di seafood dari pantai Barat Daya India.
Hasilnya menunjukkan bahwa primer yang menyandikan gen hns
memilikikemampuan deteksi yang signifikan (63%) dari 100 sampel.Kumar et al.
(2008a) yang melaporkan adanya perbedaan nilai sensitivitas antara metode pengayaan, ELISA, dan PCR dalam mendeteksi keberadaan Salmonella spp. pada
seafood, dimana berdasarkan penelitian tersebut, PCR merupakan metode dengan deteksi yang paling sensitif dibandingkan dengan ELISA dan pengayaan. Heint et al. (2006) dan Cheng et al. (2008) menggunakan primer penyandi gen invA dengan didahului tahap pengayaan untuk mendeteksi Salmonella spp. pada sampel udang, salmon, daging ayam dan sapi serta bubuk cabe. Primer penyandi gen oriC digunakan oleh McCarthy et al. (2009) untuk mendeteksi Salmonella spp. pada keju dan daging. Selanjutnya Upadhyay et al. (2010) juga melaporkan bahwa perlakuan pengayaan sebelum PCR berpotensi untuk diaplikasikan dalam deteksi
Salmonella spp. yang secara alamiah mengkontaminasi makanan, lingkungan dan sampel klinis.
METODE
Tempat dan Waktu
Penelitian ini dilakukan selama 4 bulan mulai Desember 2012 hingga April 2013 di Laboratorium Mikrobiologi dan Laboratorium Bioteknologi, Balai Besar Penelitian dan Pengembangan Pengolahan Produk dan Bioteknologi Kelautan dan Perikanan (BBP4-BKP), Kementerian Kelautan dan Perikanan, Slipi, Jakarta Pusat.
Bakteri Uji
Bakteri yang digunakan dalam penelitian ini adalah isolat bakteri
Salmonella Typhymurium (S. Typhimurium), Eschericia coli (E. coli), Listeria monocytogenes (L. monocytogenes), Proteus mirabilis (P. mirabilis),
Staphylococcus aureus (S. aureus) dan Vibrio parahaemolyticus (V.
parahaemolyticus) yang diperoleh di Laboratorium Mikrobiologi Balai Besar Penelitian dan Pengembangan Pengolahan Produk dan Bioteknologi Kelautan dan Perikanan (BBP4-BKP), Slipi Jakarta Pusat, serta 5 isolat Salmonella dengan serovar yang berbeda (S. Enteritidis, S. Hadar, S. Heidelberg, S. Kentucky, dan S.
Paratyphi) yang diperoleh dari koleksi SEAFAST CENTER.
Bahan dan Media
Sebanyak 16 sampel udang segar diperoleh dari 5 pasar domestik (basah) yang ada di Jakarta. Media untuk pengayaan : Buffered Peptone Water (BPW),
Rappaport-Vassiliadis (RV) broth (RVB), Muller-KauffmannTetrathionate-Novobiocin Broth (MKTTn), media pemupukan selektif: Brilliance Salmonella Agar (BSA) dan Xylose Lysine Deoxycholate Agar (XLDA) (Oxoid, England), uji biokimiawi Salmonella spp. presumtif dari isolat udang antara lain:Triple Sugar Iron Agar (TSIA), Lysine Iron Agar(LIA), Microgen GN-ID Test Kit, dan alkohol 70%.
Bahan yang digunakan untuk isolasi DNA bakteri antara lain media pertumbuhan bakteri yaitu TSB (Tryptone Soya Broth) (CM 0129 Oxoid, UK); bahan-bahan untuk ekstraksi DNA menggunakan PureLinkTMGenomic DNA Mini Kit (invitrogenTM) yang terdiri dari Lysis/Binding Buffer; Digestion Buffer; Wash Buffer 1; Wash Buffer 2; Elution Buffer (10 mM Tris-HCl, pH 9.0, 0.1 mM EDTA), RNase A (20 mg/ml) dalam 50 mM Tris HCl, pH 8.0, 10 mM EDTA; Proteinase K (20 mg/ml); Spin Columns; Collection Tubes (2.0 ml); isopropanol (Merck, Darmstadt, Germany), etanol 100% dan larutan lysozyme (Promega, USA).
Bahan-bahan yang digunakan untuk analisis gel elektroforesis DNA antara lain bufer TBE (Tris-Asetat-EDTA), agarosa (Fermentas, US), pewarna SYBR® Safe DNA Gel Stain (invitrogenTM), DNA ladder 100 bp dan akuabides steril.
12
polymerase, bufer PCR, MgCl2, dan dNTP (masing-masing 0.4 mM dATP, dCTP,
dGTP dan dTTP), akuabides steril, DNA cetakan (template) dan primer. Primer untuk mengamplifikasi gen penyandi invA pada Salmonella spp. berdasarkan penelitian Rahn et al. (1992) (Tabel 5). Primer dipesan dari AIT-Biotech (Singapore).
Tabel 5 Pasangan primer oligonukleotida yang digunakan untuk amplifikasi gen
Salmonella spp.
Primer Gen
target Sekuen Oligonukleotida
Lokasi gen invA
Ukuran
amplikon (bp) Referensi
F139
invA
GTGAAATTATCGCCACGTTCGGGCAA 287 - 312
284 Rahn et
al.(1992)
R141 TCATCGCACCGTCAAAGGAACC 571- 550
Alat
Alat-alat yang digunakan antara lain microtube steril, tabung spin column, waterbath shaker, termometer, inkubator, pipet volumetrik berukuran 10 ml dan 25 ml, pipet mikro beserta tip berukuran 1 ml, 0.1 ml dan 2-20 μl, batang gelas pengaduk, sudip, jarum ose, labu takar berukuran 50 ml dan 1000 ml, erlenmeyer berukuran 100 ml, 125 ml, 250 ml, dan 500 ml, botol Schott Duran berukuran 100 ml, 250 ml, 500 ml dan 1000 ml, tabung reaksi bertutup, gelas ukur, cawan petri, plastik steril, pH meter, penangas air (hot plate), microcentrifuge dengan kekuatan 18000 rpm, pengaduk magnet (magnetic stirer), oven, vortex, bunsen, timbangan elektrik, aluminium foil, perangkat elektroforesis (Bio-Rad), perangkat PCR
Applied Biosystem 2720 Thermal Cycler (Foster City, California), Geldoc XR (Bio-Rad) dan Spektrofotometer UV-Vis (Shimadzu).
F139 (287 – 312)
R141 (571 – 550)
284 bp
Prosedur
Secara garis besar penelitian ini dilakukan melalui tahapan-tahapan sebagai berikut : (I) optimasi metode PCR dengan menggunakan DNA bakteri
S.Typhimurium biakan murni (II) uji sensitivitas dan spesifisitas metode PCR, (III) deteksi Salmonella spp. pada udang segar dengan menggunakan metode konvensional dan metode PCR yang terpilih. Diagram alir penelitian diperlihatkan pada Gambar 3.
Gambar 3 Diagram alir penelitian
I. Optimasi Suhu Annealing untuk Pengembangan Metode PCR
Isolasi DNA Genom Bakteri (PureLinkTMGenomic DNA Mini Kit, invitrogenTM) Enam biakan murni Salmonella antara lain :S. Enteritidis, S. Hadar, S.
Heidelberg, S. Kentucky, S.Paratyphi dan S. Typhimurium serta lima biakan murni non Salmonella antara lain : E. coli, L. monocytogenes, P. mirabilis, S. aureus dan V. parahaemolyticus ditumbuhkan pada media TSB selama 24 jam±2 jam pada waterbath shaker. Sel bakteri yang tumbuh dipanen dengan cara melakukan sentrifugasi kultur biakan bakteri (1.5 ml) pada 4 000 rpm selama 10 menit. Pelet yang diperoleh diresuspensi dalam 190 μl TE Bufer dan 10 μl
(I) Optimasi suhu annealing untuk pengembangan metode PCR, dengan protokol sebagai berikut :
1. Pra denaturasi selama 1 menit suhu 950C 2. Denaturasi selama 30 detik suhu 950C
3. Penempelan primer (annealing) selama 30 detik dengan variasi suhu yang berbeda antara lain : 59; 59.5; 60.8; 62; 64; dan 64.50C
4. Pemanjangan primer selama 30 detik suhu 720C 5. Final extension selama 4 menit suhu 720C
Tahap 2, 3 dan 4 mengalami pengulangan siklus masing-masing sebanyak 35 kali
(modifikasi Rahn et al., 1992)
(III) Deteksi Salmonella spp. pada udang segar dengan metode konvensional dan metode PCR terpilih a. Metode konvensional menggunakan metode ISO
6579: 2002
b. Metode PCR dimulai dari pengayaan selektif pada media MKTTn
14
Lysozyme (untuk Gram negatif) atau 100 μl TE Bufer dan 100 μl Lysozyme (untuk Gram positif), kemudian diinkubasi pada suhu 30oC selama 10 menit untuk Gram negatif dan 30 menit untuk Gram positif. Selanjutnya sel disentrifugasi pada 4000 rpm selama 5 menit pada suhu kamar, pelet yang diperoleh diresuspensi dalam 400 μl DNA Lysis Bufer T, ditambahkan 20 μl Proteinase K, 15 μl RNaseA, divorteks selama 10 detik dan diinkubasi pada shaker waterbath suhu 700C selama 30 menit. Setelah 30 menit, ditambahkan 200 μl DNA Binding Buffer dan dicampur hingga merata dengan menggunakan pipetor.
Campuran larutan di atas dipindahkan ke dalam spin column berikut tabung pasangannya lalu disentrifugasi pada 10000 rpm selama 1 menit untuk memisahkan fase campuran. Fase padatan (berada di dalamspin column) yang turun ke tabung dibuang dan spin column dipindahkan ke tabung pasangan yang baru, lalu ditambahkan dengan 650 μl DNA Wash Buffer yang sudah mengandung 100% ethanol, kemudian disentrifugasi pada 10000 rpm selama 10 menit hingga diperoleh kembali fase terpisah. Fase padatan dipindahkan ke tabung pasangan yang baru, kemudian ditambahkan lagi dengan 650 μl DNA Wash Buffer yang sudah mengandung 100% ethanol, kemudian disentrifugasi pada 10000 rpm selama 2 menit. Selanjutnya, spin column ditempatkandi tabung mikro steril 1.5 ml yang baru, dan ditambahkan dengan 50 μl DNA Elution Buffer dan dibiarkan selama 3 menit pada suhu kamar. Presipitasi DNA dilakukan dengan sentrifugasi pada 6000 rpm selama 1 menit. DNA akan terelusi dari spin column, dan langkah ini diulangi sebanyak 1 kali. Untuk penyimpanan jangka panjang, larutan DNA disimpan pada suhu -20oC. Verifikasi DNA dilakukan dengan elektroforesis menggunakan gel agarosa 1.5% pada 120 V 1x bufer TBE. Gel lalu diwarnai dengan SYBr SAFE dan divisualisasi pada Geldoc XR (Bio-Rad).
Elektroforesis Gel Agarosa (Brown, 1992)
DNA genom bakteri yang berukuran besar (0,1-20 kb) dapat dipisahkan dengan elektroforesis gel agarosa. DNA merupakan molekul bermuatan negatif sehingga bila diletakkan di medan listrik maka DNA akan bermigrasi dari kutub negatif ke kutub positif. Kecepatan pergerakan DNA tergantung pada ukuran molekul DNA, kerapatan gel dan arus listrik yang diberikan untuk memigrasikan molekul DNA. Sebelum dilakukan elektroforesis, suspensi DNA dicampur dengan penyangga muatan berwarna (loading dye) yang berfungsi untuk menambah densitas sehingga DNA berada di bagian bawah sumur. Pewarna (loading dye) ditambahkan untuk memudahkan peletakan sampel DNA ke dalam sumur.
Loading dye juga berfungsi agar DNA dapat bergerak ke arah anoda dengan laju yang diperkirakan sehingga dapat sebagai tanda migrasi DNA (Brown, 1992).
Visualisasi DNA (Brown, 1992)
dilihat dengan menyinari gel dengan sinar UV pada UV transiluminator (Brown 1992).
Kuantifikasi DNA (Johnson and Tyler, 1993)
Untuk tujuan PCR, maka DNA dikuantifikasi terlebih dahulu dengan spektrofotometer UV-Vis. Konsentrasi dan kemurnian isolat DNA ditentukan dengan mengukur absorbansi pada panjang gelombang 260 nm dan 280 nm. Isolat DNA diencerkan 1 : 100 menggunakan ddH2O dan diukur absorbansinya (A260
dan A280). Konsentrasi DNA (µg/ml) dapat dilihat dengan rumus A260 x 50 x
faktor pengenceran (1 OD A260 = 50 µg/ml dsDNA). Kemurnian DNA dapat
ditentukan dengan mengukur rasio A260/A280. Kemurnian DNA yang baik
memiliki nilai rasio antara 1.9 sampai 2.0 (Johnson and Tyler, 1993).
Amplifikasi dan Visualisasi GeninvA (modifikasi Rahn et al., 1992)
Proses amplifikasi dengan PCR dilakukan menggunakan 15 μl campuran
reaktan yang masing-masing mengandung 1.5 μl primer forward dan reverse (20pmol/μl), 7.5 μl PCR Master Mix (Fermentas) yang terdiri dari 0.05 U/μl Taq
DNA polimerase, bufer PCR, MgCl2 dan dNTP (masing-masing 0.4 mM dATP,
dCTP, dGTP dan dTTP); dan akuabides steril. Volume DNA cetakan bervariasi tergantung pada konsentrasi DNA yang diperoleh.
Protokol PCR yang digunakan terdiri dari 6 kondisi suhu annealing yang berbeda yaitu :
1. Denaturasi (950C, 30 detik), annealing (590C, 30detik), elongasi atau pemanjangan primer (720C, 30 detik) dengan siklus sebanyak 35kali. 2. Denaturasi (950C, 30 detik), annealing (59.50C, 30 detik), elongasi atau
pemanjangan primer (720C, 30 detik) dengan siklus sebanyak 35 kali. 3. Denaturasi (950C, 30 detik), annealing (60.80C, 30 detik), elongasi atau
pemanjangan primer (720C, 30 detik) dengan siklus sebanyak 35 kali. 4. Denaturasi (950C, 30 detik), annealing (620C, 30 detik), elongasi atau
pemanjangan primer (720C, 30 detik) dengan siklus sebanyak 35 kali. 5. Denaturasi (950C, 30 detik), annealing (640C, 30 detik), elongasi atau
pemanjangan primer (720C, 30 detik) dengan siklus sebanyak 35 kali. 6. Denaturasi (950C, 30 detik), annealing (64.50C, 30 detik), elongasi atau
pemanjangan primer (720C, 30 detik) dengan siklus sebanyak 35 kali. Masing-masing kondisi tersebut diawali dengan pra denaturasi (950C, 1 menit) dan diakhiri dengan final extension (720C, 4 menit).
Selanjutnya, sebanyak 2 μl hasil PCR dielektroforesis pada gel agarosa 1.5% (w/v), dengan menggunakan bufer TBE 1x pada voltase 100.
II. Uji Sensitivitas dan Spesifisitas Metode PCR (Upadhyay et al., 2010; Deekshit et al., 2013)
Sensitivitas PCR dievaluasi dengan membuat variasi konsentrasi DNA
terekstrak dari S. Typhimurium. Ekstrak DNA diencerkan hingga 10 kali
pengenceran dengan deionized water. Disamping itu, juga dibuat variasi kultur
S.Typhimuriumyang telah ditumbuhkan semalam dan diencerkan hingga 10 kali
16
berurutan digunakan sebagai kontrol positif dan kontrol negatif di setiap uji PCR. Hasil PCR dielektroforesis dan dilakukan pencatatan terhadap DNA dengan konsentrasi terendah yang memberikan hasil positif.
Spesifisitas PCR dievaluasi dengan menguji 6 isolat serovar Salmonella
yang berbeda (S. Enteritidis, S. Hadar, S. Heidelberg, S. Kentucky, S. Paratyphi, dan S. Typhimurium) dengan menggunakan kontrol negatif S. aureus, kemudian diuji silang dengan 5 isolat non Salmonella yang sering mengkontaminasi makanan khususnya seafood (Eschericia coli, Listeria monocytogenes, Proteus mirabilis, Staphylococcus aureus, dan Vibrio parahaemolyticus), dengan menggunakan kontrol positif S. Typhimurium.
III. Deteksi Salmonella spp. pada Udang Segar dengan Metode Konvensional dan metode PCR terpilih
a. Metode konvensional (ISO 6579: 2002)
Sampel udang segar diambil dari 5 pasar basah di Jakarta. Pra pengayaan
sampel dilakukan dengan memasukkan 25 g sampel ke dalam 225 ml Buffered
Peptone Water (BPW) (Oxoid,England) dan diinkubasi pada suhu 37ºC selama 24 jam, dan kemudian dilakukan pengayaan selektif pada media MKTTn (Oxoid,
England) selama 24 jam pada suhu 370C. Metode konvensional dilakukan
berdasarkan ISO 6579:2002. Dengan metode ini pengayaan selektif dilakukan
pada media Rappaport-Vassiliadis Broth (RV) (Oxoid, England) dan MKTTn serta diinkubasi selama 24 jam pada suhu 420C dan 370C, secara berurutan, diikuti dengan isolasi selektif pada Xylose Lysine Deoxycholate Agar (XLDA) (Oxoid, England) dan Brilliance Salmonella Agar (BSA) (Oxoid, England) dan diinkubasi selama 24 jam pada suhu 37ºC. Koloni tipikal digores pada agar miring Triple Sugar Iron Agar (TSIA) (Oxoid, England) dan Lysine Iron Agar (LIA) (Oxoid, England), dan diinkubasi selama 24 jam pada suhu 37ºC.
Uji biokimia digunakan untuk mengkonfirmasi koloni positif pada agar selektif. TSIA dan LIA merupakan media yang umum digunakan untuk identifikasi Salmonella pada kultur campuran, sedangkan urea broth digunakan untuk mengidentifikasi Salmonella pada kultur murni (Moats 1981, USFDA, 2003). Koloni yang mencirikan Salmonella spp. pada TSIA dan LIA diamati dengan reaksi seperti yang tercantum pada Tabel 6.
Tabel 6 Hasil uji Salmonella spp. pada TSIA dan LIA
Media Bagian Miring
Uji biokimia lanjutan dilakukan dengan sistem identifikasi untuk semua
lubang-lubang yang terdapat dalam strip.Selanjutnya ditambahkan 3-4 tetes mineral oil ke dalam lubang nomor 1, 2, 3 dan 9. Menginkubasi selama 18-24 jam pada suhu 35-370C.Prosedur pembacaan hasil uji biokimia lanjutan tersaji pada Lampiran 1.
b. Metode PCR
Secara garis besar, 16 sampel udang segar diambil dari 5 pasar basah. 25 g sampel diperkaya dalam 225 ml Buffered Peptone Water (BPW) (Oxoid,England) dan diinkubasi pada suhu 37ºC selama 24 jam, dan kemudian dilakukan pengayaan selektif pada media MKTTn (Oxoid, England) selama 24 jam pada suhu 370C. Bakteri yang tumbuh pada media MKTTn disentrifugasi pada 20000 rpm selama 5 menit dan DNA diekstrak untuk mendapatkan sel pelet.Metode ekstraksi DNA dilakukan seperti yang telah tersebut pada subsubbab “Isolasi DNA Genom Bakteri”, kemudian dilakukan uji PCR.
HASIL DAN PEMBAHASAN
I. Suhu Annealing Optimum untuk Pengembangan Metode PCR
Ekstrak DNA genom bakteri
Visualisasi hasil isolasi DNA S. Typhimurium disajikan pada gambar 4.
Gambar 4 Visualisasi DNA (lajur M :marker DNA ladder 100 bp, lajur 1 : S.
Typhimurium, lajur2 : E. coli, lajur 3 : L. monocytogenes, lajur 4 : P. mirabilis, lajur 5 : S. aureus, lajur 6 : V. parahaemolyticus, lajur 7 : air)
Visualisasi DNA dalam penelitian ini menggunakan pewarna SYBr SAFE untuk mewarnai DNA pada gel, sedangkan pada penelitian-penelitian sebelumnya menggunakan EtBr (ethidium bromide).Pewarna SYBr SAFE dipilih karena memiliki beberapa kelebihan dibandingkan EtBr, antara lain dapat menekan biaya 14% lebih murah (berdasarkan studi yang dilakukan oleh Forsyth Institute, Boston), lebih aman untuk lingkungan dan yang terpenting tidak beracun, sedangkan EtBr bersifat karsinogenik (Muladno, 2010).
Berdasarkan pengukuran dengan spektrofotometer pada panjang gelombang (optic density/OD) 260 nm dan 280 nm diperoleh konsentrasi DNA genom dari S. Typhimurium tersebut sebesar2.486 μg/ml. Menurut Wilkerson et
18
al. (1993), konsentrasi DNA yang baik untuk PCR berkisar antara 0.5 sampai
6.5μg/mL. Pada panjang gelombang 260 nm yang terdeteksi adalah material genetik DNA; sedangkan pada panjang gelombang 280 nm yang terdeteksi adalah protein (Sambrook et al., 1989).
Kemurnian DNA diukur dengan melihat rasio absorbansi pada OD 260 dengan OD 280. Kemurnian DNA genom hasil ekstraksi baik karena masuk dalam cakupan nilai 1.8 – 2.0, yaitu sebesar 1.88. Nilai perbandingan yang kurang dari 1.8 atau lebih dari 2.0 menunjukkan bahwa preparasi DNA terkontaminasi oleh fenol atau protein lainnya (Brown, 1992).
Amplifikasi Gen invA
Gambar 5 menunjukkan visualisasi produk PCRhasil amplifikasi gen invA dari isolat S. Typhimurium dengan 6 suhu annealing yang berbeda antara lain : 59; 59.5; 60.8; 62; 64 dan 64.50C.
Gambar 5 Visualisasi DNAhasil amplifikasi gen invA S. Typhimuriumpada berbagai suhu annealing, lajur 1-6 secara berurutan : 59; 59.5; 60.8;
62; 64; 64.5 0C, 7 :S. aureus sebagai kontrol negatif, lajur M
merupakan marker DNA ladder 100 bp
Pengembangan yang dilakukan dalam penelitian ini terletak pada protokol PCR yang menerapkan 6 kondisi suhu annealing, yang berada pada kisaran 59oC hingga 64.5oC. Gradien suhu annealing pada penelitian ini berdasarkan suhu leleh (Tm) yang dimiliki pasangan primer penyandi gen invA, dimana Tm F139 sebesar 780C dan Tm R141 sebesar 680C. Suhu annealing yang digunakan umumnya 50C dibawah Tm primer, dan berkisar antara 360C hingga 720C (Muladno, 2010). Oleh karena itu, pada penelitian ini dilakukan optimasi suhu annealing dengan kisaran suhu seperti tersebut di atas. Kary B. Mullis pada tahun 1985 menyatakan bahwa tahap annealing merupakan tahap yang paling penting dalam running PCR, karena apabila suhu yang digunakan kurang optimum, maka akan terjadi
mispriming yaitu penempelan primer pada tempat yang salah.
Gambar 5memperlihatkan visualisasi DNA S. Typhimuriumpada kisaran suhu 590C hingga 64.50C tampak sangat jelas dan memiliki ukuran produk PCR (amplikon) yang sama dengan ketebalan pita yang sama tampak sangat jelas, namun tidak untuk S. aureus sebagai kontrol negatif. Ketebalan yang hampir sama
M 1 2 3 4 5 6 7
ini menunjukkan bahwa kisaran suhu yang digunakan dalam penelitian ini spesifik. Yuwono (2006) menyatakan bahwa pada suhu annealing yang lebih tinggi dari 370C, yaitu antara 55 hingga 650C, maka spesifisitas reaksi amplifikasi akan meningkat. Deekshit et al. (2013) menggunakan berbagai macam primer salah satunya primer penyandi gen invA untuk menentukan suhu annealing paling optimum yaitu pada 550C selama 1.5 menit. Secara keseluruhan, amplifikasi spesifik tergantung pada primer yang menempel ke target selama siklus amplifikasi. Design primer dan komponen bufer yang buruk, serta suhu annealing
merupakan beberapa faktor yang dapat mengurangi optimasi tahap annealing.
Demikian juga pada tahap extension, dimana target primer spesifik dipengaruhi oleh aktivitas enzim, MgCl2, ketersediaan komponen deoxyribonucleoside
triphosphates (dNTPs), dan kemurnian DNA target. Oleh karena itulah mayoritas optimasi metode untuk meningkatkan fungsi PCR diarahkan pada faktor-faktor yang mempengaruhi annealing/extension (Devananda, 2011).
Berdasarkan ketebalan pita yang tampak, dapat disimpulkan bahwa suhu
annealing 640C menghasilkan pita yang paling tebal sehingga untuk tahap penelitian selanjutnya digunakan suhu 640C sebagai suhu annealing. Penentuan suhu 640C sebagai suhu annealing dalam tahapan PCR pada penelitian ini serupa dengan penelitian yang dilakukan oleh Upadhyay et al. (2010) dan Zou et al.
(2011).
II. Sensitivitas dan Spesifisitas Metode PCR
Sensitivitas PCR dalam penelitian ini bertujuan untuk mengetahui limit deteksi PCR dengan menggunakan pasangan primer F-139 dan R-141 yang menargetkan gen invA Salmonella. Hasilnya ditunjukkan pada Gambar 6.
Gambar 6 menunjukkan bahwa pita spesifik dari gen invA sebesar 284 bp
dapat dideteksi pada pengenceran DNA hingga 10-5, dengan konsentrasi DNA
sebesar 27.81 µg/mL. Ketika DNA diekstrak dari kultur S. Typhimuriumyang
diencerkan, gen invA dapat dideteksi hingga pada pengenceran kultur tertinggi
yaitu pada 1 cfu/ml. DNA dari kontrol negatif, S. aureus tidak memberikan
amplikon. Hasil yang diperoleh pada penelitian ini memiliki limit deteksi yang lebih tinggi dibandingkan penelitian-penelitian sebelumnya. Kimura et al. (1999)
melaporkan bahwa uji PCR mampu mendeteksi keberadaan Salmonella pada
sampel feses hingga 3 cfu per tabung PCR Salmonella spp.dalam kultur biakan (120 cfu/mLkultur TSB). Cheng et al. (2008) melaporkan bahwa dengan
menggunakan Real Time PCR dan primer penyandi gen invA, yang diawali
dengan pengayaan selektif, Salmonella spp. pada udang masih dapat terdeteksi hingga 0.04 cfu/g. Deteksi gen invA pada Salmonella spp. dengan PCR yang
dilakukan Uphadhyay et al. (2010) hanya mendeteksi hingga 104 cfu/mL.
Penelitian Deekshit et al. (2013) melaporkan bahwa dengan pengayaan dalam
lactose broth (LB) selama 4 – 20 jam, kultur Salmonella Newport dapat terdeteksi hingga pengenceran 10-8, yang setara dengan 9.1 cfu/mL. Selanjutnya Garrido et
al. (2013) dengan menggunakan qPCR berhasil mendeteksi keberadaan
20
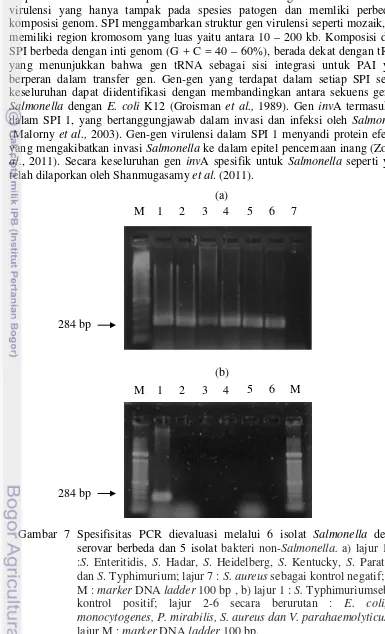
Spesifisitas PCR dalam penelitian ini mampu menghasilkan pita tunggal
pada 284 bp keseluruhan 6 isolat Salmonella dengan serovar yang berbeda
(Gambar 7). Gambar 7 (b) menunjukkan bahwa tidak adanya amplifikasi pada 5 isolat non – Salmonella, mengindikasikan bahwa spesifisitas PCR pada penelitian ini sebesar 100%. Disamping itu, gen invA yang terkandung dalam primer F-139 dan R-141 spesifik untuk mendeteksi serovar Salmonella. Gen invA Salmonella
ini merupakan gen virulensi dimana ketersediaan gen invA ini menunjukkan tingkat patogenitas yang mempengaruhi infeksi Salmonella.
Data terbaru dari Karunasagar et al. (2012) menyebutkan bahwa
Salmonella memiliki tingkat patogenitas yang disebut dengan Salmonella Patogenicity Island (SPI), dan sudah teridentifikasi sebanyak 17 SPI. Patogenitas ini mempengaruhi karakteristik virulensi Salmonella, dimana SPI 1 dan SPI 2
284 bp
Gambar 6 Batasan PCR dalam mendeteksi gen invA Salmonella, (a).
Pengenceran DNA, lajur 1-10 : DNA S. Typhimurium mulai
dari 100 – 10-9; lajur 11 : DNA S. aureus sebagai kontrol negatif; lajur M : marker DNA ladder 100 bp. (b). Pengenceran kultur, lajur 1-10 :DNA S. Typhimurium mulai dari 109 – 1 cfu/mL; lajur 11 : DNA S. aureus sebagai kontrol negatif; lajur M :
marker DNA ladder 100 bp.
(a)
(b)
M 1 2 3 4 5 6 7 8 9 10 11 M
M 1 2 3 4 5 6 7 8 9 10 11 M
berperan dalam invasi dan pertumbuhan sel dalam inang. SPI ini memiliki gen virulensi yang hanya tampak pada spesies patogen dan memliki perbedaan komposisi genom. SPI menggambarkan struktur gen virulensi seperti mozaik, dan memiliki region kromosom yang luas yaitu antara 10 – 200 kb. Komposisi dasar SPI berbeda dengan inti genom (G + C = 40 – 60%), berada dekat dengan tRNA yang menunjukkan bahwa gen tRNA sebagai sisi integrasi untuk PAI yang berperan dalam transfer gen. Gen-gen yang terdapat dalam setiap SPI secara keseluruhan dapat diidentifikasi dengan membandingkan antara sekuens genom
Salmonella dengan E. coli K12 (Groisman et al., 1989). Gen invA termasuk ke dalam SPI 1, yang bertanggungjawab dalam invasi dan infeksi oleh Salmonella
(Malorny et al., 2003). Gen-gen virulensi dalam SPI 1 menyandi protein efektor yang mengakibatkan invasi Salmonella ke dalam epitel pencernaan inang (Zou et al., 2011). Secara keseluruhan gen invA spesifik untuk Salmonella seperti yang telah dilaporkan oleh Shanmugasamy et al. (2011).
III. DeteksiSalmonella spp. pada udang segar dengan metode konvensional dan metode PCR terpilih
M 1 2 3 4 5 6 7
Gambar 7 Spesifisitas PCR dievaluasi melalui 6 isolat Salmonella dengan serovar berbeda dan 5 isolat bakteri non-Salmonella. a) lajur 1 – 6 :S. Enteritidis, S. Hadar, S. Heidelberg, S. Kentucky, S. Paratyphi, dan S. Typhimurium; lajur 7 : S. aureus sebagai kontrol negatif; lajur M : marker DNA ladder 100 bp , b) lajur 1 : S. Typhimuriumsebagai
kontrol positif; lajur 2-6 secara berurutan : E. coli, L.
monocytogenes, P. mirabilis, S. aureus dan V. parahaemolyticus dan lajur M : marker DNA ladder 100 bp.
(a)
(b)
M 1 2 3 4 5 6 M 284 bp
22
Sampel udang segar yang digunakan pada penelitian ini berasal dari 5 pasar tradisional dengan total sampel sebanyak 16 sampel. Rata-rata ke-16 sampel tersebut dijual dalam kondisi tanpa pendinginan dan dalam keadaan terbuka.
a. Deteksi Salmonella spp. dengan Metode Konvensional
Hasil menunjukkan bahwa dari 16 sampel udang segar yang ditumbuhkan pada media TSB, seluruhnya menunjukkan kekeruhan (positif).
Tahap selanjutnya adalah pengayaan selektif dengan menggunakan dua jenis media yaitu Muller-Kauffmann Tetrathionate-Novobiocin Broth (ISO)
(MKTTn) dan Rappaport Vassiliadis (RV). Kedua media tersebut secara selektif memperkaya jumlah Salmonella yang terdapat pada sampel. Pada media MKTTn, senyawa selektif berupa garam empedu menghambat pertumbuhan bakteri gram positif. Novobiocin Supplement yang terkandung dalam media dapat meningkatkan pertumbuhan Salmonella. Selain itu terdapat senyawa selektif seperti natrium tiosulfat dan tetrationat untuk menghambat pertumbuhan bakteri koliform. Tetrationat terbentuk di dalam media akibat penambahan iodin dan kalium iodida (I2-KI). Pada media MKTTn, Salmonella dapat tumbuh karena
memiliki enzim tetrationat reduktase (Oxoid Manual,2009). Adanya enzim tetrationat reduktase pada Salmonella menyebabkan Salmonella tahan terhadap efek toksik dari tetrationat (S4O62-) selama pengayaan. Pada media RV senyawa
selektif seperti malachite green dan magnesium klorida yang dikombinasikan dengan pH rendah (5,2 ±2) menghambat pertumbuhan mikroba alami yang berasal dari saluran pencernaan selain Salmonella (D’Aoust, 1989). Selain itu, pertumbuhan Salmonella didukung juga dengan adanya soy peptone pada media.
Soy peptone yang terdapat pada media RV berfungsi sebagai sumber nitrogen, karbon, dan asam amino bagi Salmonella (Oxoid Manual, 1995). Pada kedua media hasil menunjukkan positif yaitu terjadi kekeruhan pada media seperti pada Gambar 9. Hasil menunjukkan bahwa dari 16 sampel isolat dari TSB yang diinokulasikan ke dalam media MKTTn dan RV, keseluruhannya menunjukkan hasil positif, yang berupa kekeruhan dan pengendapan pada media MKTTn serta kekeruhan pada media RV.
Selanjutnya dilakukan isolasi Salmonella dengan menggunakan dua media spesifik yaitu Xylose Deoxycholate Agar (XLDA), dan Brilliance Salmonella Agar (BSA). Koloni tipikal pada media XLDA berwarna merah muda dengan atau tanpa warna hitam di tengahnya, beberapa akan tampak sebagai koloni yang besar, berwarna hitam mengkilap di tengahnya atau tampak sebagai koloni yang semuanya berwarna hitam (BAM, 2007). Koloni tipikal pada BSA berwarna ungu. Sekeliling koloni biasanya akan berwarna biru pada awalnya dan akan menjadi hitam dengan bertambahnya waktuinkubasi, yang dinamakan halo effect. Koloni tipikal dan atipikal pada media BSA dapatdilihat pada Gambar 10.
Koloni tipikal maupun tidak tipikal Salmonella yang diisolasi dari media XLDA dan BSA selanjutnya diinokulasikan pada media agar miring Triple Sugar Iron Agar (TSIA) dan Lysine Iron Agar (LIA) untuk konfirmasi biokimia dengan cara gores dan tusuk, kemudian diamati pertumbuhannya setelah diinkubasi pada Gambar 10 Koloni tipikal (kanan) dan koloni atipikal (kiri) Salmonella pada media BSA
Gambar 9 Hasil positif pada media MKTTn dan RV MKTTn
24
suhu 35 ± 2°C selama 24 jam. Konfirmasi biokimia pada TSIA ditandai dengan terbentuknya warna merah di bagian permukaan dan warna kuning (menghasilkan asam) di bagian dasar tabung, dengan atau tanpa terbentuknya gas pada agar. Warna merah terjadi karena Salmonella dapat memfermentasi glukosa yang jumlahnya terbatas dalam media, sehingga jika glukosa habis bakteri ini menggunakan pepton sebagai sumber energi yang terjadi di permukaan agar dan menghasilkan produk sampingan berupa basa (merah). Terbentuknya H2S ditandai
dengan warna hitam karena kandungan natrium tiosulfat pada agar direduksi oleh H2S yang kemudian bereaksi dengan garam besi menghasilkan warna hitam.
Konfirmasi biokimia pada LIA ditandai dengan terbentuknya warna ungu di bagian permukaan dan berwarna hitam di bagian dasar tabung (menghasilkan H2S). Warna ungu terjadi karena Salmonella dapat mendekarboksilasi lisin
menghasilkan amin kadaverin yang ditunjukkan dengan berubahnya indikator pH bromkresol ungu menjadi warna ungu. Reaksi biokimia yang menunjukkan hasil positif dapat dilihat pada Gambar 11.
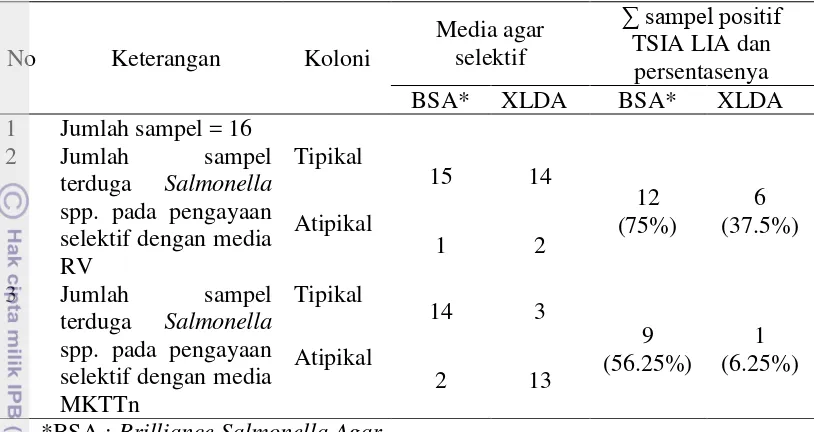
Persentase koloni yang diduga Salmonella setelah uji konfirmasi biokimia pada media TSIA dan LIA miring disajikan pada Tabel 7. Tabel 7 memperlihatkan hasil bahwa media yang paling banyak menghasilkan uji positif konfirmasi biokimia adalah koloni tipikal maupun atipikal dari BSA baik dari media pengaya selektif MKTTn dan RV, dimana 9 dari 16 sampel (56.25%) yang berasal dari MKTTn dan 1 dari 16 sampel (6.25%) yang berasal dari RV diduga
Salmonella. Hasil analisa dari media XLDA menunjukkan bahwa 12 dari 16 sampel (75%) yang berasal dari MKTTn dan 6 dari 16 sampel (37.5%) yang berasal dari RV diduga sebagai Salmonella.
Tabel 7 Persentase koloni yang diduga Salmonella setelah uji konfirmasi pada media TSIA dan LIA
No Keterangan Koloni
Media agar
*BSA : Brilliance Salmonella Agar
Berdasarkan hasil konfirmasi tersebut terlihat bahwa kemungkinan tertinggi mendapatkan koloni yang diduga Salmonella adalah dengan mengisolasi koloni dari media BSA, baik dari media pengaya RV maupun MKTTn dimana 72.41% (21 dari 29 total isolat terpilih) dari koloni tipikal diduga Salmonella pada media BSA teridentifikasi sebagai Salmonella setelah uji biokimia pada TSIA dan LIA sementara koloni tipikal pada media XLDA hanya 41.18% (7 dari 17 total isolat terpilih) yang terkonfirmasi sebagai Salmonella dengan uji di atas.
BSA merupakan media agar selektif yang fleksibel digunakan dengan berbagai macam prosedur dalam mendeteksi Salmonella. Adapun senyawa selektif yang terdapat dalam BSA adalah adanya dua kromogen yang menargetkan enzim spesifik yaitu caprylate esterase dan ß-glucosidase. Kerja kedua enzim tersebut menghasilkan warna menonjol dari koloni. Warna yang dihasilkan tergantung enzim yang dimiliki oleh organisme. Kerja caprylate esterase, tampak di semua Salmonella, menghasilkan koloni berwarna ungu. Beberapa spesies
Enterobacteriaceaelain juga memproduksi caprylate esterase, tapi
penghambatannya berbeda dengan Salmonella karena keberadaan substrat
ß-glucosidase. Hal ini mengakibatkan munculnya koloni berwarna biru, sehingga memudahkan untuk membedakan dengan koloni Salmonella yang berwarna ungu. Tidak semua Salmonella akan tumbuh sama baiknya pada semua media agar cawan selektif untuk menekan tumbuhnya kontaminan nonSalmonella sehingga proses perbaikan dari jenis Salmonella kemungkinan besar memerlukan dua atau lebih media agar cawan selektif. Kesalahan dalam deteksi ketika melihat koloni pada media agar cawan juga dapat terjadi karena tidak ada media selektif yang secara penuh bersifat selektif (Oxoid Manual,1995).
Hasil positif pada media TSIA dan LIA selanjutnya dikonfirmasi dengan perangkat Microgen GN-ID System dengan microwell test strips GNA untuk memastikannya sebagai Salmonella. Perangkat Microgen GN-ID System