PENGARUH PERBANDINGAN VOLUME DARAH DAN
LISIS BUFFER SERTA KECEPATAN SENTRIFUGASI
TERHADAP KUALITAS PRODUK DNA PADA
SAPI FRIESIAN HOLSTEIN (FH)
SKRIPSI
KOKOM KOMALASARI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
KOKOM KOMALASARI. D14051702. 2009. Pengaruh Perbandingan Volume Darah dan Lisis Buffer serta Kecepatan Sentrifugasi Terhadap Kualitas Produk DNA pada Sapi Friesian Holstein (FH). Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir. Ronny Rachman Noor, MRur.Sc Pembimbing Anggota : Dr. Ir. Endang Tri Margawati, M. Agr.Sc
Deoxyribonucleic Acid (DNA) adalah molekul terkecil dari suatu sel berbentuk untai panjang yang membawa informasi genetik dan mengontrol semua fungsi seluler pada semua bentuk kehidupan. DNA diperoleh dengan cara ekstraksi dari jaringan atau darah segar hewan. Terdapat berbagai metode ekstraksi DNA, salah satunya adalah phenol-choroform-isoamyl alcohol (PCI) yang digunakan dalam penelitian ini. Tujuan penelitian ini yaitu mencari perbandingan yang optimal antara volume darah segar dan lisis buffer dengan kecepatan sentrifugasi untuk memperoleh produk DNA yang berkualitas tinggi.
Materi penelitian berupa darah segar Friesian Holstein (FH) yang diperoleh dengan cara penyedotan dengan jarum suntik (G18) dari pembuluh darah ekor sapi yang dialirkan ke dalam tabung vacum berisi 15% EDTA, sebanyak ±5 ml per ekor. Darah diperoleh dari 4 ekor sapi sesuai dengan jumlah ulangan (4). Rancangan percobaan yang digunakan adalah rancangan acak lengkap faktorial dengan dua factor (3x2). Faktor pertama yaitu kombinasi volume darah dan lisis buffer (µl darah : µl lisis buffer), terdapat 3 kombinasi, yaitu 100 : 800, 200 : 700, dan 300 : 600. Faktor kedua yaitu kecepatan sentrifugasi (10.000 rpm dan 12.000 rpm). Dengan demikian terdapat 6 kombinasi perlakuan. Masing-masing perlakuan diulang 4 kali. Data yang dikoleksi berupa konsentrasi DNA dan kemurnian DNA pada rasio OD 260/280 yang diukur dengan GeneQuant DNA calculator. Sementara kualitas DNA dicek melalui gel agarose (1%) yang dialiri listrik 100 voltage selama 1 jam. Data dianalisis dengan sidik ragam (ANOVA). Perbedaan antara perlakuan diuji dengan Tukey pada taraf perbedaan 5% (p<0,05).
Hasil penelitian menunjukkan bahwa kombinasi volume darah dan lisis buffer dengan dua kecepatan sentrifugasi yang berbeda tidak berpengaruh terhadap konsentrasi DNA. Konsentrasi DNA relatif lebih tinggi diperoleh pada perlakuan perbandingan volume darah : lisis buffer (300µl : 600µl), konsentrasi DNA relatif meningkat dengan meningkatnya volume darah. Tingkat kemurnian DNA yang diperoleh rata-rata 1,3 (260/280), artinya DNA yang diperoleh masih terbawa bahan kimia lain. Tingkat kemurnian yang optimal yaitu 1,8. Kualitas DNA pada hasil elektroforesis menunjukan hasil berbanding lurus dengan konsentrasi DNA, dan menunjukkan pita tunggal.
ABSTRACT
THE EFFECT OF BLOOD AND BUFFER LYSIS COMPOSITION AND SPINNING SPEED ON DNA YIELDS OF Friesian Holstein (FH)
Komalasari, K., R. R. Noor and E. T. Margawati
Deoxyribonucleic Acid (DNA) is smallest part (molecule) of a cell in the form of a long chain that carries genetic information and controlling all cellular functional in all life form. The DNA can be obtained by extracting tissue or fresh whole blood of animal. There are several methods of DNA extraction, one of which is phenol-choroform-isoamyl alcohol (PCI) that used in this research. The aim of this research was to determine the best combination of fresh blood and buffer lysis with two differences of spinning speed on DNA quantity and quality. The fresh whole blood of Friesian Holstein (FH) dairy cattle was collected using a vacuum tube (vacutainer) containing 15% EDTA with a 18G needle to suck the blood of tail vena with the amount of ±5 ml per head. A 3x2 factorial completely random design was used. First factor was three combinations of fresh whole blood and buffer lysis volume (µl), i.e., 100 : 800, 200 : 700, and 300 : 600. Second factor was spinning speeds, i.e., 10.000 rpm and 12.000 rpm. There were 6 combination treatments used in this research which replicated 4 times of each treatment. Data of DNA concentration was collected by a GeneQuant DNA calculator at the 260 nm and 280 nm wave length of optical density (OD). The quality of DNA was obtained by running the DNA through electrophoresis with 1% gel agarose and electricity flow of 100 voltage for an hour. The DNA purity was examined at OD 260/280 ratios. DNA concentration and purity were analyzed by using ANOVA, the differences between theatments were tested by tukey test at the level of 5% (p<0.05). The result showed that there was no significant effect of combination of fresh blood and buffer lysis at both spinning speeds (p>0.05). The highest DNA concentration was obtained from the treatment of ratio of 300 µl blood and 600 µl buffer lysis. The DNA concentration tended to increase with the increase of blood volume. The DNA purity was obtained at the ratio of 1.3 (260/280) meaning that the DNA was a slighty contaminated by the chemical used. A ratio of 1.8 is the optimal purity of DNA. The DNA quality linearly correlated to DNA concentration and showed a single band. Keywords: blood, DNA extraction, phenol-choroform-isoamyl alcohol (PCI),
PENGARUH PERBANDINGAN VOLUME DARAH DAN
LISIS BUFFER SERTA KECEPATAN SENTRIFUGASI
TERHADAP KUALITAS PRODUK DNA PADA
SAPI FRIESIAN HOLSTEIN (FH)
KOKOM KOMALASARI
D14051702
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
PENGARUH PERBANDINGAN VOLUME DARAH DAN
LISIS BUFFER SERTA KECEPATAN SENTRIFUGASI
TERHADAP KUALITAS PRODUK DNA PADA
SAPI FRIESIAN HOLSTEIN (FH)
Oleh
KOKOM KOMALASARI
D14051702
Skripsi ini telah disetujui dan disidangkan di hadapan Komisi Ujian Lisan pada tanggal 18 Juni 2009
Pembimbing Utama
Prof. Dr. Ir. Ronny R. Noor, MRur.Sc.
Pembimbing Anggota
Dr. Ir. Endang T. Margawati, M.Agr.Sc.
Dekan
Fakultas Peternakan Institut Pertanian Bogor
Dr. Ir. Luki Abdullah, M.Sc.Agr.
Ketua Departemen
Ilmu Produksi dan Teknologi Peternakan Institut Pertanian Bogor
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 8 Desember 1986 di Pangkalan Boros, Sumedang. Penulis anak kedua dari dua bersaudara dari pasangan Bapak Wiharya dan Ibu Uka. Pendidikan dasar diselesaikan pada tahun 1999 di SD Negeri Boros, Sumedang. Pendidikan lanjutan menengah pertama diselesaikan pada tahun 2002 di SLTP Negeri II Buahdua, Sumedang dan pendidikan menengah atas diselesaikan pada tahun 2005 di SMU Negeri I Cimalaka, Sumedang. Penulis diterima sebagai mahasiswa Program Studi Ilmu Produksi dan Teknologi Peternakan, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB (USMI) pada tahun 2005.
Penulis aktif di berbagai organisasi meliputi Staf ahli divisi Unggas, Himpunan Mahasiswa Ilmu Produksi dan Teknologi Peternakan (HIMAPROTER), Fakultas Peternakan (2006-2007), sekertaris umum Famm Al An’Am, Fakultas Peternakan Institut Pertanian Bogor (2007-2008), dan aktif pada berbagai kegiatan kampus (2005-2008) serta pernah menjadi asisten mata kuliah Tingkah Laku dan Kesejahteraan Ternak (2009).
KATA PENGANTAR
Bismillahirrahmaniirahim,
Alhamdulillah setelah disiapkan dalam waktu yang cukup singkat akhirnya skripsi ini telah diselesaikan. Puji dan syukur penulis panjatkan ke hadirat Allah SWT yang telah memberikan rahmat, karunia, rizki, dan nikmat iman dan islam yang telah diberikan sehingga penulis memperoleh kemudahan dalam menyusun dan menyelesaikan skripsi ini yang berjudul “Pengaruh Perbandingan Volume Darah dan Lisis Buffer serta Kecepatan Sentrifugasi terhadap Kualitas Produk DNA
pada Sapi Friensian Holstein (FH)”. Shalawat dan salam semoga tetap tercurahkan ke junjungan kita Nabi Muhammad SAW.
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan di Fakultas Peternakan, Institut Pertanian Bogor. Di samping itu penulisan skripsi ini bertujuan untuk memberikan satu sumbangan untuk kemajuan di dunia peternakan, khususnya bioteknologi molekuler hewan.
Penelitian dilaksanakan di Laboraturium Biologi Molekuler Hewan, Pusat Penelitian Bioteknologi-LIPI Cibinong pada bulan Februari-April 2009. Sampel darah diambil dari sapi FH dengan jarum G-18 pada pembuluh halus di ekor kemudian diekstraksi dengan metode phenol-choroform-isoamyl alcohol (PCI) dengan modifikasi volume darah dan lisis buffer dengan kecepatan sentrifugasi yang berbeda. Penelitian ini diharapkan dapat memperoleh dan mengetahui kombinasi optimal antara volume darah dan lisis buffer dengan kecepatan sentrifugasi dalam mengoptimalkan produk DNA yang dihasilkan.
Akhirnya tiada gading yang tak retak, penulis menyadari bahwa karya kecil ini masih belum sempurna. Penulis mengharapkan semoga skripsi ini dapat bermanfaat bagi pembaca dan menjadi salah satu sumber ilmu pengetahuan.
DAFTAR ISI
Sejarah dan Perkembangan Teknologi DNA ... 5
Perkembangan Penelitian DNA pada Hewan ... 6
DNA (Deoxyribonucleic Acid) ... 7
Ekstraksi DNA ... 9
Phenol-Choroform-Isoamyl alkohol (PCl)... 10
Rasio ... 17
Kualitas DNA ... 19
KESIMPULAN DAN SARAN ... 21
Kesimpulan ... 21
Saran... 21
UCAPAN TERIMA KASIH ... 22
DAFTAR PUSTAKA ... 23
DAFTAR TABEL
Nomor Halaman
1. Pengaruh Perbandingan Volume Darah dan Lisis Buffer dengan Kecepatan Sentrifugasi terhadap Konsentrasi DNA (Rataan ± SE) .. 16 2. Pengaruh Perbandingan Volume Darah dan Lisis Buffer dengan
PENGARUH PERBANDINGAN VOLUME DARAH DAN
LISIS BUFFER SERTA KECEPATAN SENTRIFUGASI
TERHADAP KUALITAS PRODUK DNA PADA
SAPI FRIESIAN HOLSTEIN (FH)
SKRIPSI
KOKOM KOMALASARI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
KOKOM KOMALASARI. D14051702. 2009. Pengaruh Perbandingan Volume Darah dan Lisis Buffer serta Kecepatan Sentrifugasi Terhadap Kualitas Produk DNA pada Sapi Friesian Holstein (FH). Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir. Ronny Rachman Noor, MRur.Sc Pembimbing Anggota : Dr. Ir. Endang Tri Margawati, M. Agr.Sc
Deoxyribonucleic Acid (DNA) adalah molekul terkecil dari suatu sel berbentuk untai panjang yang membawa informasi genetik dan mengontrol semua fungsi seluler pada semua bentuk kehidupan. DNA diperoleh dengan cara ekstraksi dari jaringan atau darah segar hewan. Terdapat berbagai metode ekstraksi DNA, salah satunya adalah phenol-choroform-isoamyl alcohol (PCI) yang digunakan dalam penelitian ini. Tujuan penelitian ini yaitu mencari perbandingan yang optimal antara volume darah segar dan lisis buffer dengan kecepatan sentrifugasi untuk memperoleh produk DNA yang berkualitas tinggi.
Materi penelitian berupa darah segar Friesian Holstein (FH) yang diperoleh dengan cara penyedotan dengan jarum suntik (G18) dari pembuluh darah ekor sapi yang dialirkan ke dalam tabung vacum berisi 15% EDTA, sebanyak ±5 ml per ekor. Darah diperoleh dari 4 ekor sapi sesuai dengan jumlah ulangan (4). Rancangan percobaan yang digunakan adalah rancangan acak lengkap faktorial dengan dua factor (3x2). Faktor pertama yaitu kombinasi volume darah dan lisis buffer (µl darah : µl lisis buffer), terdapat 3 kombinasi, yaitu 100 : 800, 200 : 700, dan 300 : 600. Faktor kedua yaitu kecepatan sentrifugasi (10.000 rpm dan 12.000 rpm). Dengan demikian terdapat 6 kombinasi perlakuan. Masing-masing perlakuan diulang 4 kali. Data yang dikoleksi berupa konsentrasi DNA dan kemurnian DNA pada rasio OD 260/280 yang diukur dengan GeneQuant DNA calculator. Sementara kualitas DNA dicek melalui gel agarose (1%) yang dialiri listrik 100 voltage selama 1 jam. Data dianalisis dengan sidik ragam (ANOVA). Perbedaan antara perlakuan diuji dengan Tukey pada taraf perbedaan 5% (p<0,05).
Hasil penelitian menunjukkan bahwa kombinasi volume darah dan lisis buffer dengan dua kecepatan sentrifugasi yang berbeda tidak berpengaruh terhadap konsentrasi DNA. Konsentrasi DNA relatif lebih tinggi diperoleh pada perlakuan perbandingan volume darah : lisis buffer (300µl : 600µl), konsentrasi DNA relatif meningkat dengan meningkatnya volume darah. Tingkat kemurnian DNA yang diperoleh rata-rata 1,3 (260/280), artinya DNA yang diperoleh masih terbawa bahan kimia lain. Tingkat kemurnian yang optimal yaitu 1,8. Kualitas DNA pada hasil elektroforesis menunjukan hasil berbanding lurus dengan konsentrasi DNA, dan menunjukkan pita tunggal.
ABSTRACT
THE EFFECT OF BLOOD AND BUFFER LYSIS COMPOSITION AND SPINNING SPEED ON DNA YIELDS OF Friesian Holstein (FH)
Komalasari, K., R. R. Noor and E. T. Margawati
Deoxyribonucleic Acid (DNA) is smallest part (molecule) of a cell in the form of a long chain that carries genetic information and controlling all cellular functional in all life form. The DNA can be obtained by extracting tissue or fresh whole blood of animal. There are several methods of DNA extraction, one of which is phenol-choroform-isoamyl alcohol (PCI) that used in this research. The aim of this research was to determine the best combination of fresh blood and buffer lysis with two differences of spinning speed on DNA quantity and quality. The fresh whole blood of Friesian Holstein (FH) dairy cattle was collected using a vacuum tube (vacutainer) containing 15% EDTA with a 18G needle to suck the blood of tail vena with the amount of ±5 ml per head. A 3x2 factorial completely random design was used. First factor was three combinations of fresh whole blood and buffer lysis volume (µl), i.e., 100 : 800, 200 : 700, and 300 : 600. Second factor was spinning speeds, i.e., 10.000 rpm and 12.000 rpm. There were 6 combination treatments used in this research which replicated 4 times of each treatment. Data of DNA concentration was collected by a GeneQuant DNA calculator at the 260 nm and 280 nm wave length of optical density (OD). The quality of DNA was obtained by running the DNA through electrophoresis with 1% gel agarose and electricity flow of 100 voltage for an hour. The DNA purity was examined at OD 260/280 ratios. DNA concentration and purity were analyzed by using ANOVA, the differences between theatments were tested by tukey test at the level of 5% (p<0.05). The result showed that there was no significant effect of combination of fresh blood and buffer lysis at both spinning speeds (p>0.05). The highest DNA concentration was obtained from the treatment of ratio of 300 µl blood and 600 µl buffer lysis. The DNA concentration tended to increase with the increase of blood volume. The DNA purity was obtained at the ratio of 1.3 (260/280) meaning that the DNA was a slighty contaminated by the chemical used. A ratio of 1.8 is the optimal purity of DNA. The DNA quality linearly correlated to DNA concentration and showed a single band. Keywords: blood, DNA extraction, phenol-choroform-isoamyl alcohol (PCI),
PENGARUH PERBANDINGAN VOLUME DARAH DAN
LISIS BUFFER SERTA KECEPATAN SENTRIFUGASI
TERHADAP KUALITAS PRODUK DNA PADA
SAPI FRIESIAN HOLSTEIN (FH)
KOKOM KOMALASARI
D14051702
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
PENGARUH PERBANDINGAN VOLUME DARAH DAN
LISIS BUFFER SERTA KECEPATAN SENTRIFUGASI
TERHADAP KUALITAS PRODUK DNA PADA
SAPI FRIESIAN HOLSTEIN (FH)
Oleh
KOKOM KOMALASARI
D14051702
Skripsi ini telah disetujui dan disidangkan di hadapan Komisi Ujian Lisan pada tanggal 18 Juni 2009
Pembimbing Utama
Prof. Dr. Ir. Ronny R. Noor, MRur.Sc.
Pembimbing Anggota
Dr. Ir. Endang T. Margawati, M.Agr.Sc.
Dekan
Fakultas Peternakan Institut Pertanian Bogor
Dr. Ir. Luki Abdullah, M.Sc.Agr.
Ketua Departemen
Ilmu Produksi dan Teknologi Peternakan Institut Pertanian Bogor
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 8 Desember 1986 di Pangkalan Boros, Sumedang. Penulis anak kedua dari dua bersaudara dari pasangan Bapak Wiharya dan Ibu Uka. Pendidikan dasar diselesaikan pada tahun 1999 di SD Negeri Boros, Sumedang. Pendidikan lanjutan menengah pertama diselesaikan pada tahun 2002 di SLTP Negeri II Buahdua, Sumedang dan pendidikan menengah atas diselesaikan pada tahun 2005 di SMU Negeri I Cimalaka, Sumedang. Penulis diterima sebagai mahasiswa Program Studi Ilmu Produksi dan Teknologi Peternakan, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB (USMI) pada tahun 2005.
Penulis aktif di berbagai organisasi meliputi Staf ahli divisi Unggas, Himpunan Mahasiswa Ilmu Produksi dan Teknologi Peternakan (HIMAPROTER), Fakultas Peternakan (2006-2007), sekertaris umum Famm Al An’Am, Fakultas Peternakan Institut Pertanian Bogor (2007-2008), dan aktif pada berbagai kegiatan kampus (2005-2008) serta pernah menjadi asisten mata kuliah Tingkah Laku dan Kesejahteraan Ternak (2009).
KATA PENGANTAR
Bismillahirrahmaniirahim,
Alhamdulillah setelah disiapkan dalam waktu yang cukup singkat akhirnya skripsi ini telah diselesaikan. Puji dan syukur penulis panjatkan ke hadirat Allah SWT yang telah memberikan rahmat, karunia, rizki, dan nikmat iman dan islam yang telah diberikan sehingga penulis memperoleh kemudahan dalam menyusun dan menyelesaikan skripsi ini yang berjudul “Pengaruh Perbandingan Volume Darah dan Lisis Buffer serta Kecepatan Sentrifugasi terhadap Kualitas Produk DNA
pada Sapi Friensian Holstein (FH)”. Shalawat dan salam semoga tetap tercurahkan ke junjungan kita Nabi Muhammad SAW.
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan di Fakultas Peternakan, Institut Pertanian Bogor. Di samping itu penulisan skripsi ini bertujuan untuk memberikan satu sumbangan untuk kemajuan di dunia peternakan, khususnya bioteknologi molekuler hewan.
Penelitian dilaksanakan di Laboraturium Biologi Molekuler Hewan, Pusat Penelitian Bioteknologi-LIPI Cibinong pada bulan Februari-April 2009. Sampel darah diambil dari sapi FH dengan jarum G-18 pada pembuluh halus di ekor kemudian diekstraksi dengan metode phenol-choroform-isoamyl alcohol (PCI) dengan modifikasi volume darah dan lisis buffer dengan kecepatan sentrifugasi yang berbeda. Penelitian ini diharapkan dapat memperoleh dan mengetahui kombinasi optimal antara volume darah dan lisis buffer dengan kecepatan sentrifugasi dalam mengoptimalkan produk DNA yang dihasilkan.
Akhirnya tiada gading yang tak retak, penulis menyadari bahwa karya kecil ini masih belum sempurna. Penulis mengharapkan semoga skripsi ini dapat bermanfaat bagi pembaca dan menjadi salah satu sumber ilmu pengetahuan.
DAFTAR ISI
Sejarah dan Perkembangan Teknologi DNA ... 5
Perkembangan Penelitian DNA pada Hewan ... 6
DNA (Deoxyribonucleic Acid) ... 7
Ekstraksi DNA ... 9
Phenol-Choroform-Isoamyl alkohol (PCl)... 10
Rasio ... 17
Kualitas DNA ... 19
KESIMPULAN DAN SARAN ... 21
Kesimpulan ... 21
Saran... 21
UCAPAN TERIMA KASIH ... 22
DAFTAR PUSTAKA ... 23
DAFTAR TABEL
Nomor Halaman
1. Pengaruh Perbandingan Volume Darah dan Lisis Buffer dengan Kecepatan Sentrifugasi terhadap Konsentrasi DNA (Rataan ± SE) .. 16 2. Pengaruh Perbandingan Volume Darah dan Lisis Buffer dengan
DAFTAR GAMBAR
Nomor Halaman
1. Sel-sel dalam Darah Mamalia ... 4 2. Struktur Adenin, Guanin, Sitosin, Urasil, dan Timin ... 8 3. Hasil Pemotretan Elektroforesis dari DNA Marker (1 Kb dan Enam
DAFTAR LAMPIRAN
Nomor Halaman
1. Diagram Alir Prosedur Ekstraksi DNA Metode Sambrook et al.,1989 ... 27 2. Reagent-reagent yang Digunakan untuk Ekstraksi DNA Metode
Phenol-Choroform-Isoamyl alkohol (PCI) ... 29 3. Data Kuantifikasi DNA (Kosentrasi DNA, Rasio OD 260/280, 260
nm, dan 280 nm) ... 31 4. Analisis Ragam Kosentrasi DNA terhadap Pengaruh Perbandingan
Volume Darah dan Lisis Buffer dengan Kecepatan Sentrifugasi yang Berbeda ... 32 5. Analisis Ragam Rasio 260/280 DNA terhadap Pengaruh
PENDAHULUAN
Latar Belakang
Kesenjangan yang cukup besar antara persediaan susu dan permintaan susu menjadi masalah yang penting dalam penyediaan protein hewani bagi masyarakat luas. Sampai saat ini permintaan susu semakin terus meningkat dengan bertambahnya jumlah penduduk pada setiap tahunnya. Peternakan sapi merupakan salah satu komponen subsektor peternakan yang mampu meningkatkan kesejahteraan sebagian masyarakat pedesaan dan memberikan perbaikan gizi bagi generasi mendatang. Namun, rendahnya produktivitas sapi perah dibandingkan laju kebutuhan, rentannya terhadap penyakit dan keterbatasan kemampuan peternakan untuk memanajemen perusahaan menjadi suatu kendala untuk memenuhi kebutuhan tersebut. Rataan produksi susu sapi Friesian Holstein (FH) di Indonesia sekitar 10 liter per hari per ekor (Sudono et al., 2003). Rendahnya produktivitas ini disebabkan beberapa faktor diantaranya faktor lingkungan, faktor genetik dan faktor interaksi antara lingkungan dan genetik. Usaha-usaha perlu dilakukan untuk meningkatkan produktivitas sapi perah domestik.
Perbaikan mutu genetik sapi Friesian Holstein yang banyak dilakukan pada saat ini adalah secara konvensional, yaitu berdasarkan seleksi morfologi. Namun, cara ini belum memberikan hasil yang optimal karena disamping membutuhkan waktu yang lama juga mengeluarkan biaya yang cukup besar. Menghadapi masalah itu, perlu adanya terobosan baru yang dapat dilakukan untuk mempercepat proses peningkatan mutu genetik adalah melalui teknologi DNA. Teknologi tersebut dapat memberikan solusi lebih tepat melalui identifikasi gen-gen berasosiasi dengan sifat-sifat bernilai ekonomi tinggi seperti tingginya produksi susu dapat diidentifikasi dan ditelusuri pada sapi FH.
berkualitas baik dapat diperoleh dengan waktu dan biaya yang minimal merupakan satu hal yang menarik dalam penelitian genetika molekuler.
Berdasarkan keperluan dan jumlah DNA yang diperlukan, maka banyak prosedur-prosedur ekstraksi yang telah dilakukan dan dikembangkan dalam menghasilkan DNA yang berkualitas tinggi. Setiap prosedur memiliki keunggulan dan kelemahan tersendiri. DNA dapat diperoleh dari berbagai sumber umumnya diperoleh dari daging, darah, dan organ-organ tubuh lainnya (Sambrook et al., 1989). Darah merupakan sumber DNA yang sering dilakukan karena kemudahan dalam koleksi sampel.
Ekstraksi DNA yang dilakukan pada studi ini berasal dari darah segar sapi FH dengan menggunakan metode phenol-choroform-isoamyl alcohol (PCI) dengan modifikasi komposisi volume darah dan lisis buffer dengan kecepatan sentrifugasi yang berbeda. Penelitian ini diharapkan dapat memperoleh dan mengetahui efisiensi kombinasi antara volume darah dan lisis buffer dengan kecepatan sentrifugasi dalam mengoptimalkan produk DNA yang lebih baik untuk diaplikasikan pada penelitian berikutnya.
Tujuan
TINJAUAN PUSTAKA
Sapi Friesian Holstein
Sapi perah Friesian Holstein (FH) berasal dari Netherland, propinsi Friesland, Belanda. Sapi Friesian Holstein (FH) merupakan bangsa sapi perah tertua kedua setelah Brown Swiss. Di Indonesia sapi FH sudah banyak dikembangkan karena telah banyak disilangkan dengan sapi lokal. Hasil persilangan dikenal dengan nama sapi Grati, sapi perah jenis baru yang sesuai dengan kondisi di Indonesia. Produksi susu sapi FH di Indonesia mencapai rata-rata 3.660 liter per laktasi dengan bobot sapi dewasa mencapai ± 700 kg (Eko et al., 2009). Sedangkan menurut Sudono et al. (2003) produksi rata-rata sapi FH 10 liter per ekor per hari atau ± 3.050 kg per laktasi.
Ciri-ciri yang paling menonjol pada sapi perah Friesian Holstein (FH) yaitu warna tubuhnya memiliki dua warna, hitam dan putih atau merah dan putih. Sapi perah Friesian Holstein (FH) menghasilkan produksi susu yang paling besar diantara sapi perah yang lainnya (Tyler and Ensminger, 2006). Sedangkan menurut Santoso (2008), ciri-ciri sapi Friesian Holstein (FH) yang baik adalah tubuh luas ke belakang seperti gergaji, sistem dan bentuk perambingan baik dan puting simetris, efesiensi pakan yang dialihkan untuk produksi susu tinggi, dan sifatnya baik dan bijak.
Kendala-kendala pengembangan sapi Friesian Holstein (FH) ada dua faktor diantaranya, faktor teknis dan faktor pemerintah. Faktor teknis meliputi pemuliaan, tata laksana pemberian pakan dan tata laksana pemeliharaan. Sedangkan faktor pemerintah adalah dalam memperhatikan kegiatan persusuan, memelihara kebijakan yang kondusif dengan memberikan fasilitas dan dana untuk mengembangkan peternakan (Prahasta et al., 2008).
Darah
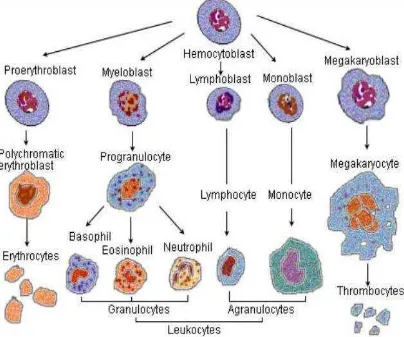
terdiri dari eritrosit, leukosit dan trombosit digambarkan seperti Gambar 1 (Wardhani, 2008).
Gambar 1. Sel-sel dalam Darah Mamalia
Sejarah dan Perkembangan Teknologi DNA
Teknologi DNA rekombinan merupakan hasil dari sejumlah pemotongan DNA dan penyambungan kembali potongan-potongan DNA tersebut. Masalah yang berhubungan dengan bioteknologi dan perbaikan mutu genetik ternak yaitu pertama mengidentifikasi gen-gen yang bermanfaat dan mengintroduksikannya ke dalam genome ternak sehingga dapat diwariskan secara mantap dari generasi ke generasi (Martojo, 1992). Menurut Susanti dan Ariani (2004) teknologi DNA rekombinan atau rekayasa genetika merupakan suatu metode yang dilakukan untuk memanipulasi DNA suatu makhluk hidup tertentu guna memperoleh sifat-sifat tertentu dalam organisme tersebut.
Tahun 1926 Muller, seorang Pelopor ilmu biologi molekuler mulai mengenalkan peran gen. Dalam bukunya yang berjudul “The Gene as the Basis of
Life”, Muller menyatakan bahwa gen dapat dipandang sebagai sebuah atom biologis
yang bertanggung jawab sepenuhnya untuk ciri-ciri fisiologis dan morfologis dari bentuk-bentuk kehidupan (Hardjosubroto, 1999).
Perkembangan teknologi DNA rekombinan dimulai dari berbagai penemuan dan penelitian oleh para ahli genetika melokuler. Penelitian Griffith yang menjadi titik awal bagi penelitian 14 tahun untuk mencari identitas substansi pentransformasi yang dilakukan oleh Avery. Avery memurnikan berbagai macam zat kimia dari berbagai bakteri-bakteri patogenik yang telah dimatikan dengan panas, kemudian mencoba mentransformasikan bakteri nonpatogenik hidup dengan setiap zat kimia, hanya DNA yang mampu melakukan transformasi tersebut. Oleh karena itu tahun 1944, Avery dan koleganya mengumumkan bahwa agen pentransformasi tersebut adalah DNA. berikutnya pada tahun 1952, Alfred Hershey dan Martha Chase menemukan bahwa DNA merupakan materi genetik dari suatu fase yang dikenal T2. Bukti lain oleh ahli biokimia Erwin Chargaff menganalisis komposisi basa DNA dari sejumlah organisme yang berbeda diperoleh bahwa komposisi DNA dari setiap spesies berbeda-beda dan menemukan keteraturan yang khas dalam rasio dari basa-basa nukleotida (Campbell et al., 2002)
memotong fragmen DNA, pada tahun 1962 adanya DNA ligase, enzim yang dapat menyambung fragmen DNA yang telah dipotong. Tahun 1967 telah diletakkan dasar bagi terbentuknya DNA rekombinan (Alberts et al., 1994). Selanjutnya, teknologi rekombinan DNA semakin sempurna dengan ditemukan teknik perunutan DNA (Sequencing) pertengahan 1970 yang dapat menentukan urutan fragmen DNA secara cepat. Teknologi DNA berfungsi dalam studi ekspresi gen yang menguntungkan seperti hormon, enzim pembentukan organisme transgenik dengan sifat unggul yang lainnya (Nicholl, 1996).
Perkembangan teknologi DNA rekombinan berfungsi dalam studi ekspresi gen, produksi protein yang sangat menguntungkan seperti hormon dan enzim, serta pembentukan organisme transgenik (tumbuhan, hewan dan manusia). DNA rekombinan juga bermanfaat untuk teknologi, kesehatan dan aplikasi yang lainnya yang biasa dimanfaatkan (Nicholl, 1996).
Perkembangan Penelitian DNA pada Hewan
Tahun 1972, ketika Cohen, Berg, dan Boyer yang melakukan penelitian bersama di Universitas Stanford berhasil menggabungkan gen dari seekor katak pada bakteri (Djojosoebagio, 1996). Perkembangan selanjutnya dari DNA adalah pembentukan hewan trasngenik, yaitu hewan yang telah mengalami rekayasa dalam susunan DNA-nya melalui introduksi gen asing. Tahun 1981 Palmiter dan Brinster berhasil memproduksi tikus transgenik, kemudian diikuti oleh Spradling dan Rublin yang menghasilkan lalat buah transgenik. Pembentukan hewan transgenik pada ternak domestik berhasil dilakukan pada penghujung tahun 1980 dan pada awal 1990 (Boyd dan Samid, 1993).
Keberhasilan manusia merekayasa ternak untuk digunakan sebagai ‘pabrik’
pengganda protein yang dibutuhkan oleh manusia, misalnya protein laktoperin yang terdapat pada air susu ibu (ASI) telah berhasil diproduksi oleh sapi yang telah direkayasa. Keberhasilan yang sangat memuaskan juga terjadi pada tikus sebagai hewan transgenik pada tahun 1986 sebagai model untuk merekayasa ternak-ternak lainnya. Contoh lain, kambing transgenik mampu memproduksi Tissue Plasminogen Aktivator (TPA) secara luas untuk pengobatan penyakit jantung (Muladno, 2002).
mengisolasi molekul DNA manusia yang member kode genetika untuk pembentuan suatu protein. Kedua, teknologi untuk menyusun molekul DNA tersebut ke dalam embrio ternak.
Bioteknologi Peternakan di Indonesia telah dirumuskan dalam lokakarya Nasional I Bioleknologi peternakan tahun 1995, bahwa pengertian bioteknologi peternakan dapat diartikan memanfaatkan proses biologi melalui rekayasa genetik dan rekayasa proses untuk menghasilkan ternak dan produk ternak yang berkualitas. Bioteknologi peternakan di Indonesia ruang lingkupnya meliputi: inseminasi buatan, transfer embrio, dan rekayasa genetik (Saefuddin, 1996). Sedangkan Direktorat Jendral Peternakan mendefinisikan bioteknologi peternakan sebagai pemanfaatan proses biolgis melalui rakayasa genetika dan proses untuk menghasilkan ternak dan produk peternakan yang berkualitas (Hardjosubroto, 1999).
DNA (Deoxyribonucleic Acid)
Deoxyribonucleic Acid (DNA) adalah bagian terkecil molekul dari suatu sel
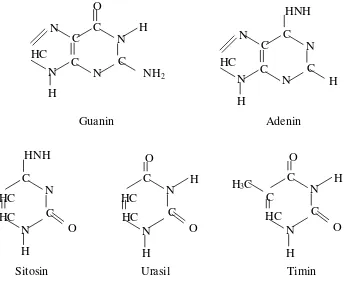
berbentuk untai panjang yang membawa informasi genetik dan mengontrol semua fungsi seluler pada semua bentuk kehidupan (Zaid et al., 1999). Gen disusun oleh suatu substansi yang disebut dengan DNA (Deoxyribonucleic Acid). Substansi DNA terdiri dari dua untaian panjang terpilin yang membentuk double helix (seperti tangga pilin). Setiap dua untaian DNA disusun oleh ribuan unit nukleotida. Setiap nukleotida disusun oleh basa nitrogen, gula deoksiribosa (deoxyribose) dan asam fosfat. Basa nitrogen dalam DNA ada empat macam yaitu adenine (A), guanine (G), sitosin (C), dan timin (T). Sitosin selalu berpasangan dengan guanin dan adenin selalu berpasang dengan timin (Noor, 2008). Struktur basa nitrogen yaitu adenin, guanin, sitosin, urasil, dan timin Gambar 2 (Jusuf, 2001).
Muladno (2002) menjelaskan bahwa DNA terdapat pada semua makhluk hidup mulai dari mikroorganisme sampai organisme tingkat tinggi seperti manusia, hewan dan tumbuhan. DNA terdapat di dalam sel dan di dalam inti sel. DNA yang terdapat di dalam sel dapat berupa DNA mitokondria, DNA kloroplast atau DNA penyusun kromosom, sedangkan DNA yang terdapat dalam inti sel disebut juga sebagai DNA inti. Sedangkan menurut Maclean (1987) DNA (Deoxyribonucleic Acid) merupakan materi genetik yang ada di dalam semua organisme yang hidup.
dalam sel berbeda-beda tiap organisme. Rentang jumlah DNA yaitu dari 4 x 106 pasang di dalam E. coli sampai kira-kira 109 pasang di dalam manusia.
Gambar 2. Struktur Adenin, Guanin, Sitosin, Urasil, dan Timin
Basa nitrogen menempel pada posisi karbon 1’ dari pentose, sedangkan gugus phospat pada posisi karbon 3’ atau karbon 5’ dari pentose. Satu nukleotida dan nukleotida lainnya dapat dibedakan pada basa nitrogennya. Serangkaian nukleotida
dapat terbentuk dengan mengikatkan gugus hidroksi pada karbon 3’ dari satu pentose dan gugus phosphat pada gugus 5’ dari pentose sebelahnya. Struktur molekul DNA terdiri atas dua rangkaian nukleotida yang tersusun secara linier. Kedua rangkaian yang saling berikatan itu terbentuk seperti tali terpilin. Oleh karenanya molekul DNA dikatakan double heliks atau heliks ganda (Alberts et al., 1994).
DNA dapat terdenaturasi oleh temperatur dan pH yang ekstrim. Denaturasi ini dapat melepaskan ikatan double heliks seperti diantara ikatan hidrogen yang saling melengkapi dapat terganggu. Denaturasi DNA dapat menyebabkan melting dan dapat memisahkan untaian DNA (Sheeler dan Bianchi, 1987). Sifat kimia yang dimiliki DNA heliks ganda yaitu kelenturan dan kestabilan. Kelenturan terdapat dalam perpasangan kedua utasan yang pasangan basanya diikat oleh ikatan hidrogen.
Ikatan hidrogen merupakan ikatan yang lemah sehingga mudah lepas dan mudah terbentuk kembali. Sehingga pasangan utas ganda DNA mudah terurai dan mudah terbentuk kembali tanpa merusak ikatan polipeptidanya. Sifat ini sangat penting karena DNA akan mengalami replikasi atau transkripsi. Sedangkan sifat kimia yang stabil dilihat dari adanya pilinan utas ganda yang menempatkan pasangan basa pada bagian dalam dan ikatan fosfat pada bagian luar. Molekul fosfat merupakan melekul yang bersifat hidrofolik dan basa-basa merupakan molekul yang bersifat hidrofobik. Sifat ini cukup memelihara kesetabilan DNA (Jusuf, 2001).
Ekstraksi DNA
Ekstraksi DNA merupakan suatu langkah awal dalam teknologi DNA. Molekul DNA ini harus diekstraksi dari asal usul tempatnya. Banyak molekuler termasuk DNA bersifat labil dan mudah kehilangan aktifitas biologinya. Oleh karena itu ekstraksi harus dilakukan dalam kondisi ringan yaitu dengan menggunakan larutan encer dan menghindari kondisi pH, tekanan osmotik yang ekstrim dan suhu tinggi (Murray, 1995). Proses ekstraksi DNA sel hewan biasanya diawali dengan merusak dinding sel menggunakan proteinase-K, EDTA dan SDS, yang diikuti ekstraksi menggunakan phenol (Sambrook et al., 1989). Senyawa-senyawa tersebut akan merusak integrasi dinding sel secara kimiawi.
Cara ekstraksi DNA dari berbagai sumber berbeda namun pada prinsipnya sama. Hanya ada beberapa modifikasi tertentu yang biasa dilakukan untuk menghancurkan inhibitor yang ada di dalam masing-masing sumber. Isolasi DNA dari organisme eukaryote biasanya dilakukan melalui penghancuran sel, pemisahan protein dan RNA, dan pemurnian DNA. Secara kimiawi penghancuran sel dilakukan dengan memanfaatkan senyawa kimia seperti lisozim, EDTA (etilendiamin tetraasetat) dan SDS (sodium dodesil sulfat). Fungsi dari EDTA adalah sebagai
perusak sel dengan cara mengikat ion magnesium sementara SDS berfungsi untuk merusak membram sel (Muladno, 2002). Metode yang sering digunakan untuk pengendapan DNA adalah etanol (Brown, 1996).
Metode-metode ekstraksi DNA yang biasa dilakukan diantaranya phenol-choroform-isoamyl alcohol (PCI), hight salt method dan choroform iso amyl alcohol
menggunakan NaCl konsentrasi tinggi juga mengunakan fenol dan iso amyl alcohol (CIAA). Keuntungannya hight salt method tidak menggunakan bahan beracun atau non-toksik ( Prasetyo, 2005).
Phenol-Choroform-Isoamyl alkohol (PCI)
Phenol-Choroform-Isoamyl alkohol (PCI) merupakan senyawa kimia yang
digunakan untuk mengekstraksi DNA yaitu untuk memisahkan protein dari asam nukleat. Metode ini menguntungkan karena dapat mengdeproteinnasi secara efisien karena penggunaan dua macam larutan yaitu fenol dan klorofrom yang lebih efektif dalam mengendapkan protein dan menghambat aktifitas RNAase dibandingkan dengan menggunakan satu larutan (Sambrook et al., 1989). Keunggulan lainnya yaitu tidak membutuhkan waktu yang lama untuk pemurnian DNA. Penambahan Phenol-Choroform-isoamyl Alkohol (PCI) diharapkan akan menghasilkan DNA yang murni
dan tidak terkontaminasi oleh protein dan akan dihasilkan rasio Optimal Density (OD) 260/280 yang berada diantara 1,8-2,0. Hasil yang terbaik untuk memperoleh DNA, yaitu melakukan pencucian dua kali dengan menggunakan Phenol-Choroform-isoamyl Alkohol (PCI) ( Khosravinia et al., 2007).
Penentuan Kuantitas DNA
Konsentrasi DNA diukur secara Sprektrofotometri dengan menggunakan GeneQuant DNA calculator. Dasar teknik ini adalah DNA yang dapat menyerap
sinar ultraviolet dengan kuat pada panjang gelombang 260 nm, sedangkan 280 nm merupakan daerah serapan protein. Panjang gelombang dengan nilai serapan 1,0 (A260=1,0 ) setara dengan 50 mg DNA utas ganda per milliliter larutan. Oleh karena itu, didapatkan bahwa konsentrasi DNA (µ g/ml) diperoleh dari perkalian antara faktor pengencer, faktor konversi (50 µg/ml) dan Absorbansi (A260). Tingkat kemurnian DNA baik jika nilai rasio yang diperoleh adalah antara 1.8-2.0 (Sambrook et al., 1989).
sampel. Rasio 260 nm dan 280 nm yang kurang dari 1.8 kemungkinan terkontaminasi protein atau terkontaminasi oleh phenol. Rasio 260/280 nilainya 1.8 mengindikasikan murni untuk DNA dan 2.0 murni untuk RNA (Nicholl, 1996). Rasio optical density (OD) menurut Montgomery and Sise (1990) apabila 260 nm dan 280 nm berada di antara 1.8-2.0 menunjukkan bahwa proses deproteinasi pada metode tersebut baik.
Penentuan Kualitas DNA
Elektroforesis adalah sutu teknik pemisahan molekul selular berdasarkan atas ukurannya. Elektroforesis menggunakan medan listrik yang dialirkan pada suatu medium yang mengandung sempel yang akan dipisahkan. Teknik ini dapat digunakan dengan memanfaatkan muatan listrik yang ada pada makromolekul, misalnya DNA yang bermuatan negatif (Triwibowo, 2008). Menurut Winarno dan Agustinah (2007) elektroforesis adalah suatu cara pemisahan campuran dan beberapa senyawa dengan melakukan suspensi ke dalam air dan kemudian diberikan aliran listrik. Gel yang ditempatkan ke dalam sumur elektroforesis yang mengandung larutan buffer dan dialiri listrik, molekul DNA yang bermuatan negatif pada pH netral akan bergerak kearah positif. DNA bergerak melalui gel pada kecepatan yang berbeda tergantung ukurannya.
METODE
Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboraturium Biologi Molekuler Hewan, Pusat Penelitian Bioteknologi-LIPI Cibinong. Penelitian dilaksanakan selama dua bulan dari bulan Februari-April 2009.
Materi
Sampel dan Bahan
Sampel yang digunakan adalah darah segar sapi FH dari empat ekor sapi yang berasal dari Laboratorium Lapang Hewan Pusat Bioteknologi-LIPI Cibinong. Darah diambil dengan jarum G-18 pada pembuluh darah (vena) halus di ekor. Darah dikoleksi langsung ke dalam tabung 10 ml mengandung 15 % EDTA (Etilendiamin tetraaserat) sebagai antikoagulasi.
Bahan-bahan yang digunakan untuk ekstraksi DNA yaitu, 5 ml darah segar sapi FH, 16,8 ml lisis buffer (1M Tris HCl pH 8, 5M NaCl, 5M EDTA dan 1 % SDS), Proteinase-K (Progen) 0,48 ml, phenol-choroform-isoamyl alkohol (PCI) (25:24:1) 14,4 ml, 3M NaOAc (Natrium Acetat) pH 5,2 sebanyak 0,72 ml, 12 ml etanol 70 %, 14,4 ml etanol 96 % dan buffer TE pH 8 (1M Tris dan 0,5M EDTA) 1,2 ml.
Bahan-bahan yang digunakan untuk kuantifikasi DNA yaitu, DNA 5 µl tiap sempel, TE buffer, H2O steril dan etanol 70 %.
Bahan-bahan yang digunakan untuk elektroforesis yaitu, DNA 35 µl, gel agarose 1 % (Invitrogen ultra pure +M Agarose), TBE 1x, ethidium bromida, 14 µl loading dye (1 ml G190A 24267202), 10,5µl marker DNA 1 kb (Biolabs ladder
N32325), alkohol, H2O dan film polaroid.
Peralatan
refrigerator, elektroforesis (Biorad), ultraviolet light, dan timbangan analitik (Precisa XT 120 A).
Rancangan Statistik
Penelitian ini menggunakan Rancangan Acak Lengkap Faktorial (3x2) dengan dua faktor. Faktor pertama yaitu tiga kombinasi volume darah dan lisis buffer (µl darah : µl lisis buffer), yaitu 100 : 800, 200 : 700, dan 300 : 600. Faktor kedua yaitu kecepatan sentrifugasi, 10.000 rpm dan 12.000 rpm. Dengan demikian penelitian ini melibatkan enam perlakuan (3x2), tiap perlakuan diulang empat kali. Sebagai ulangan adalah sapi, sehingga penelitian ini menggunakan empat ekor sapi dan jumlah sempel sebanyak 24. Model rancangan tersebut menurut Steel dan Torrie (1991) adalah sebagai berikut:
Yijk = μ + Ai + Bj + ABij +
ε
ijkKeterangan :
Yijk : Variabel respon akibat pengaruh komposisi lysis buffer ke-i dan taraf kecepatan sentrifugasi ke-j pada ulangan ke-k
µ : Nilai tengah umum
Ai : Pengaruh komposisi lysis buffer level ke-i Bj : Pengaruh kecepatan sentrifugasi levek ke-j
ABij : Pengaruh interaksi antara komposisi lsis buffer ke-i dengan kecepatan sentrifugasi ke-j
Εij : Galat percobaan pada unit percobaan ke-k dalam kombinasi perlakuan ke-ij
Analisis Data
Data berupa kuantitas dan kemurnian DNA yang diperoleh dari setiap perlakuan akan dianalisis menggunakan sidik ragam (ANOVA). Sebelum dianalisis data diuji kehomogenan, kenormalan dan kebebasan galat terlebih dahulu, apabila data memenuhi uji asumsi maka data langsung dianalisis. Apabila data tidak memenuhi asumsi maka data di transformasi. Perbedaan antar perlakuan akan diuji dengan menggunakan Tukey pada taraf perbedaan 5 % (p<0,05).
DNA yang digunakan adalah 1kb Biolabs DNA Ladder N32325, hanya sebagai kontrol ukuran berat molekul.
Prosedur
Metode ekstraksi DNA yang digunakan adalah modifikasi phenol-choroform-isoamyl alcohol (PCI) menurut Sambrook et al. (1989) adalah sebagai berikut:
Koleksi Darah Segar
Koleksi darah diperoleh sebanyak 5-6 ml dalam tabung 10 ml yang berisi EDTA sebagai antikoagulasi.
Koleksi Darah Putih
Koleksi darah putih dilakukan dengan menggunakan lysis buffer, komposisi yang digunakan adalah perbandingan volume darah segar dengan lisis buffer yaitu 100 µl : 800 µl, 200 µl : 700 µl, dan 300 µl : 600µl. Komposisi darah dan lisis buffer yang telah dicampurkan kemudian dihomogenkan. Selanjutnya dilakukan presipitasi protein.
Penghilangan Protein
Darah yang telah homogen ditambahkan 20 µl proteinase-K, diinkubasi pada suhu 56oC selama 1 jam setiap 20 menit tabung digoyangkan. Ke dalam larutan tersebut ditambahkan 600 µl phenol-choroform-isoamyl alkohol (PCI) dengan perbandingan (25:24:1), inkubasi kembali pada suhu -20 oC selama 30 menit. Setelah itu, disentrifugasi sesuai perlakuan (10.000 rpm dan 12.000 rpm) selama 10 menit pada suhu 10 0C.
Presipitasi dan Koleksi DNA
Kuantifikasi DNA
DNA terkoleksi dikalkulasi dengan mesin GeneQuant DNA calculator. Dasar perhitungan kemurnian DNA tersebut dibandingkan pada panjang gelombang 260 nm dengan 280 nm. Mesin GeneQuant DNA calculator dinyalakan tunggu sinyal sampai keluar instrument ready, tombol DNA ditekan dan dimasukan TE buffer sebagai blanko, ditekan tombol set ref ditunggu sampai muncul pada layar niai 0,000 pada semua peubah. Sampel DNA yang telah diterapkan dalam kuvet sebanyak 5 µl, kemudian tekan enter tunggu sampai nilai keluar. Nilai yang tertera pada layar yang dicatat yaitu nilai konsentrasi (ng/µl), nilai absorbansi 260 nm, 280 nm dan nilai rasio 260/280. Kuantifikasi dilanjutkan pada sampel berikutnya sebanyak 24 sampel.
Kualifikasi DNA
Gel agarosa 1% dilarutkan dalam TBE 1X (20 gram agarosa dan 200 ml TBE 1X) didihkan sampai larut, kemudian dibiarkan beberapa menit hingga suhunya turun 50-60 0C. Sisir elektroforesis dipasang di salah satu ujung baki, kemudian gel agarosa dituangkan ke dalam baki cetakan, tunggu hingga gel mengeras, dimasukkan ke dalam tanki elektroforesis yang telah diisi dengan larutan buffer TBE 1X. Masing-masing 5 µl sampel dicampurkan dengan 2 µl loading dye. Sedangkan untuk DNA Ladder digunakan ladder 1 kb sebanyak 1.5 µ l yang dicampurkan dengan 2 µl loading dye. Selanjutnya dimasukkan ke dalam sumur, dicatat nomor sumur dan
sampel DNA yang dimasukkan. Elektroforesis dijalankan dengan alir listrik 100 voltage, selama 60 menit, tekan tombol run. Setelah itu, gel direndam dalam larutan ethidium bromide selama 30 menit. Cuci gel yang telah direndam ethidium bromide
HASIL DAN PEMBAHASAN
Keperluan dasar penelitian di bidang biologi molekuler adalah adanya DNA yang cukup. Oleh karenanya banyak metode ekstraksi DNA yang dilakukan untuk memperoleh cara yang lebih efektif dalam menghasilkan DNA. Sampel yang digunakan diperoleh dari darah segar sapi Friesian Holstein (FH) yang dikoleksi dari Laboratorium Lapang Hewan Pusat Bioteknologi-LIPI Cibinong. Sampel tersebut diekstraksi menggunakan metode Sambrook et al., (1989) yang telah dimodifikasi. Indikasi keberhasilan ekstraksi DNA perlu diketahui kuantitas dan kualitas DNA yang dihasilkan. Secara kuantitas bisa dilihat dari nilai konsentrasi DNA dan nilai OD 260/280. Sedangkan secara kualitas bisa dilihat dari hasil elektroforesis dan kemurnian DNA.
Kuantitas DNA
Konsentrasi DNA
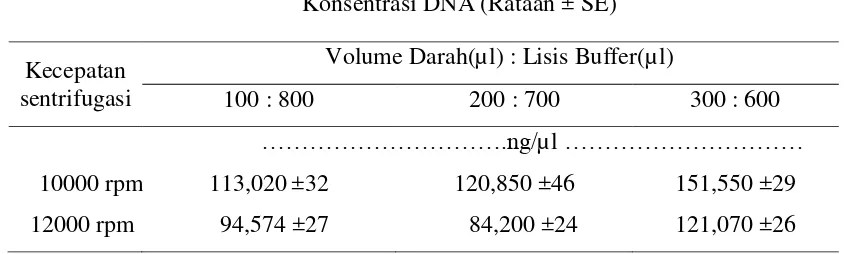
Hasil Perhitungan konsentrasi DNA dengan GeneQuant DNA calculator dari 24 sampel tergantung dari volume darah yang digunakan. Semakin banyak volume darah yang digunakan maka relatif semakin meningkat konsentrasi DNA yang dihasilkan. Konsentrasi DNA yang diperoleh berkisar antara 84,200 ± 24 ng/µl-151,550 ± 29 ng/µl. Volume darah 300 µl menghasilkan konsentrasi DNA yang relatif lebih tinggi (151,550 ± 29 ng/µl) pada kecepatan sentrifugasi 10.000 rpm. Demikian juga pada kecepatan sentrifugasi 12.000 rpm menghasilkan DNA yang relatif tinggi pada volume darah 300 µl yaitu rata-rata 121,070 ± 26 ng/ul dibandingkan dengan volume darah 100 µl dan 200 µl . Konsentrasi DNA darah sapi segar Friesian Holstein FH secara lengkap disajikan pada Tabel 1.
Tabel. 1 Pengaruh Perbandingan Volume Darah dan Lisis Buffer dengan Kecepatan Sentrifugasi terhadap Konsentrasi DNA (Rataan ± SE)
Secara statistik konsentrasi DNA yang dihasilkan tidak berbeda nyata (p>0,05) pada masing-masing kecepatan sentrifugasi. Hal ini menunjukkan nilai konsentrasi DNA yang dihasilkan dari setiap perlakuan pada setiap kecepatan sentrifugasi hampir sama. Hasil statistik tidak ada interaksi antara perbandingan volume darah dan lisis buffer dengan kecepatan sentrifugasi yang berbeda terhadap konsentrasi DNA yang dihasilkan. Namun, peningkatan konsentrasi DNA pada perbandingan volume darah : lisis buffer (300 µl : 600 µl) pada kecepatan sentrifugasi yang berbeda menunjukkan bahwa konsentrasi DNA yang dihasilkan relatif meningkat berbanding terbalik dengan volume lisis buffer yang digunakan.
DNA terdapat di dalam inti sel, pada darah mamalia dewasa inti sel berada di dalam sel darah putih (leukosit). Dellman dan Brown (1989) menyebutkan bahwa eritrosit pada mamalia dewasa tidak berinti dan leukosit memiliki inti sel. Volume darah yang digunakan lebih banyak menghasilkan leukosit yang lebih banyak dengan demikian menghasilkan konsentrasi DNA yang relatif lebih tinggi. Kecepatan sentrifugasi 10.000 rpm menghasilkan rata-rata konsentrasi DNA yang relatif lebih tinggi dibandingkan pada kecepatan sentrifugasi 12.000 rpm. Sentrifugasi dilakukan untuk memisahkan DNA dan komponen-komponen lain, tetapi kemungkinan DNA dapat tercampur dengan komponen yang lain seperti protein, RNA, lipid, dan polisakarida pada putaran sentrifugasi yang lebih cepat. Berdasarkan hasil ini menunjukkan kecepatan dan lama sentrifugasi harus benar-benar diperhatikan.
Rasio DNA
Tingkat kemurnian DNA dapat ditentukan dengan cara menghitung rasio antara nilai 260 nm dan 280 nm pada sampel DNA yang diukur melalui GeneQuant DNA calculato. Nilai 260 merupakan nilai maksimal DNA dapat menyerap cahaya.
DNA masih rendah. Rasio kurang dari 1,8 mengindikasikan adanya kontaminasi protein dan phenol (Brown, 1996). Selain kontaminasi protein dan phenol, rasio DNA yang kurang dari 1,7 disebabkan adanya kontaminasi bahan kimia lainnya seperti Tris, EDTA, etanol, sodium asetat yang akan menyebabkan kontaminasi pada sempel (Khosravinia et al., 2007). Hasil kemurnian DNA pada rasio 260/280 disajikan pada Tabel 2.
Tabel 2. Pengaruh Perbandingan Volume Darah dan Lisis Buffer dan Kecepatan Sentrifugasi terhadap Rasio OD 260/280 (Rataan ± SE)
Rasio OD 260/280 (Rataan ± SE) Kecepatan
Secara statistik tidak ada interaksi antara perbandingan volume darah dan lisis buffer dengan kecepatan sentrifugasi terhadap kemurnian DNA (p>0,05). Kemurnian DNA juga tidak dipengaruhi oleh perbandingan volume darah dan lisis buffer. Sedangkan kecepatan sentrifugasi dapat mempengaruhi tingkat kemurnian DNA, dengan kecepatan 10.000 rpm telah mampu mengendapkan lebih banyak dan mampu memisahkan molekul DNA dengan molekul yang lainnya. Sedangkan pada kecepatan sentrifugasi 12.000 rpm diperkirakan putaran terlalu cepat sehingga DNA kemungkinan dapat tercampur lagi dengan komponen yang lain. Kecepatan sentrifugasi 10.000 rpm menghasilkan kemurnian DNA rata-rata (1,368) sedangkan pada putaran 12.000 rpm menghasilkan kemurnian DNA rata-rata (1,187).
tinggi. Lisis buffer pada penelitian ini berfungsi untuk melisis sel. Komposisi lisis buffer diantaranya EDTA dan SDS, EDTA untuk merusak dinding sel dengan cara mengikat ion magnesium. Ion ini berfungsi untuk mempertahankan integritas sel maupun mempertahankan aktivitas enzim nuklease yang merusak asam nukleat. Sedangkan SDS untuk merusak membran sel (Muladno, 2002). Penambahan lisis buffer yang lebih besar (800 µl) diduga lebih efektif melisis sel darah merah pada volume darah yang lebih sedikit (100 µl). Kemurnian DNA relatif menurun dari perbandingan volume darah dan lisis buffer 100 µl : 800 µl, 200 µl : 700 µl, dan 300 µl : 600 µl.
Banyak faktor yang menyebabkan kemurnian pada penelitian ini masih sedikit rendah. Selain terkontaminasi bahan kimia, sampel juga masih terdapat RNA dan protein, sehingga hasilnya bukan DNA murni. Dalam meningkatkan kemurnian DNA yang diperoleh masih harus dilakukan tahap pemurnian dengan cara pencucian dengan beberapa kali dan penambahan PCI dua kali (Khosravinia et al., 2007). Selain itu, ketelitian dalam bekerja merupakan salah satu hal yang harus diperhatikan karena dapat mempengaruhi kemurnian DNA yang dihasilkan juga.
Kualitas DNA
Kualitas DNA hasil ekstraksi bisa dilihat pada gel agarose 1% setelah dielektroforesis pada 100 voltage selama 1 jam. Pengukuran panjang molekul DNA diperlukan pengukur DNA yang disebut molecule weight dna marker (Muladno, 2002). Marker yang digunakan untuk membandingkan ukuran molekul DNA dalam penelitian ini adalah berukuran 1 kb (kilobasa) DNA Ladder N32325. Ukuran DNA sampel dapat diperkirakan dengan cara melihat posisi DNA terhadap DNA pengukur yang memang sudah diketahui panjangnya. Namun, pengukuran seperti ini masih bersifat perkiraan. Elektroforesis dalam penelitian ini tujuannya untuk melihat ekspresi DNA dan melihat keberhasilan ekstraksi DNA yang dilakukan.
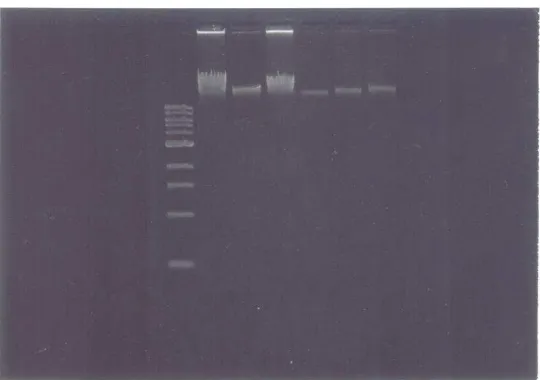
Perkiraan ukuran DNA pada keenam perlakuan (Gambar 3) lebih dari 1.000 pasang basa (pb) atau 1 kilobasa (kb). Besar kecilnya konsentrasi DNA dapat ditunjukkan dengan tipis dan tebalnya pita DNA. Perbandingan darah dan lisis buffer (100 µl : 800 µl) dan (300µl : 600 µl) dengan kecepatan sentrifugasi 10.000 rpm menunjukkan ukuran molekul besar. Pita-pita pada perlakuan tersebut terlihat smear memperlihatkan molekul DNA tidak turun ke bawah karena konsentrasi yang tinggi. Perbandingan darah dan lisis buffer 100 µl : 800 µl adalah 108.7 ng/ul dan 300 µl : 600 µl adalah 200 ng/ul. Pita yang paling tipis ditunjukkan pada perbandingan darah dan lisis buffer 100 µl : 800 µl pada kecepatan sentrifugasi 12.000 rpm dengan konsentrasi DNA yang paling rendah (74,5 ng/µl).
M T1 T2 T3 T4 T5 T6
Gambar 3. Hasil Pemotretan Elektroforesis dari DNA Marker (M) I Kb dan Enam Sempel DNA (T1-T6)
KESIMPULAN DAN SARAN
Kesimpulan
1. Kombinasi perbandingan volume darah dan lisis buffer dengan kecepatan sentrifugasi tidak mempengaruhi konsentrasi DNA
2. Konsentrasi DNA relatif tinggi diperoleh pada perlakuan yang menggunakan darah paling banyak (300 ul). Konsentrasi DNA relatif tinggi sebanding dengan banyaknya volume darah yang digunakan
3. Penambahan lisis buffer yang lebih banyak (800 µl) memberikan kemurnian yang relatif lebih tinggi
4. Nilai kemurnian DNA rata-rata 1,3 masih sedikit terkontaminasi bahan-bahan kimia lain
5. Kecepatan sentrifugasi 10.000 rpm dapat meningkatkan kemurnian DNA dan menghasilkan konsentrasi DNA yang relatif lebih banyak
6. Berdasarkan elektroforesis ketebalan pita DNA berbanding lurus dengan konsentrasi DNA
Saran
1. Perlu dilakukan penelitian lebih lanjut menggunakan perbandingan darah dan lisis buffer yang berbeda dengan penambahan PCI dan pencucian lebih dari satu kali
2. Perlu dicoba Ekstraksi DNA dengan materi yang berbeda, seperti semen, bulu, feses dan jaringan
UCAPAN TERIMA KASIH
Alhamdulillah, dengan rasa syukur penulis panjatkan kehadirat Allah SWT yang telah melimpahkan nikmat, rahmat dan hidayah-Nya sehingga penulis dapat menyelesaikan studi ini. Shalawat serta salam semoga senantiasa tercurahkan kepada Nabi Muhammad sebagai suri teladan kita.
Ayahanda, ibunda, dan teteh tercinta, terima kasih yang tak terhingga yang senantiasa melimpahkan doa, nasehat, kasih sayang, motivasi dan kekuatan kepada penulis untuk menjalani kehidupan hingga saat ini dan masa yang akan datang. Dede lia dan dede engguh keponakanku yang selalu memberikan keceriaan.
Penulis mengucapkan terima kasih yang sebesar-besarnya dan penghargaan yang setinggi-tingginya kepada Prof. Dr. Ir. Ronny R. Noor, MRur.Sc dan Dr. Ir. Endang T. Margawati, M. Agr.Sc selaku dosen pembimbing atas semua bimbingan, masukan dan arahannya selama penulis melakukan penelitian dan penyusunan skripsi ini hingga tahap akhir. Dr. Ir. Cece Sumantri M. Agr.Sc dan Dr. Ir. Nahrowi, MSc penulis haturkan terima kasih selaku dosen penguji. Penulis juga mengucapkan terima kasih kepada Dr. Ir. Sri Supraptini Mansjoer selaku dosen akademik, Ir. Niken Ulupi, MS dan Dr. Ir. Henny Nuraini, MSi yang telah memberikan pengalaman hidup selama perkuliahan, nasihat, masukan, arahan dan saran-sarannya akan selalu penulis ingat.
Ucapan terima kasih, spesial penulis sampaikan kepada semua rekan-rekan 42, Sahabat-sahabatku Ayu (seperjuangan dalam penelitian), Heni, Uny, Oel, Tray, Neng Ia, Hida, Ala, Ninu, Pipit, dan temen kos Bateng 23’ atas suasana kekeluargaan dan kebersamaan yang telah diberikan sehingga selalu memberikan keceriaan. Terima kasih kepada Prayogo Hadi dan keluarganya yang selalu memberikan kasih sayang, motivasi, dan yang selalu ada di hati penulis.
Terakhir penulis ucapkan terima kasih kepada seluruh civitas akademik Fakultas Peternakan, Institut Pertanian Bogor. Semoga skripsi ini bermanfaat bagi penulis khususnya dan pembaca pada umumnya.
Bogor, Juli 2009
DAFTAR PUSTAKA
Alberts, B., A. Johnson., J. Lewis., M. Raff., K. Roberts dan P. Walter. 2002. Molecular Biology of The Cell. Fourt Edition. Garland Science, a member of the taylor and Francis Group 29 west 35th, New York.
Alberts, B., D. Bray., J. Lewis., Raff., K. Robert dan J. D. Watson. 1994. Biologi Molekuler Sel. Mengenal Sel. Edisi ke-2, Gramedia, Jakarta.
Boyd A. L. dan D. Samid. 1993. Molekuler biology of transgenic animals. Journal of Animal Science. Vol. 71. Suppl. 3; 9-1.
Brown. 1996. Gene Cloning. An Introduction. third edition. Chapman dan Hall, Boundrary Raw, London.
Campbell, N. A., J. B. Reece dan L. G. Mitchell. 2002. Biologi. Edisi ke 5, Jilid 1. Terjemahan: Rahayu. Erlangga, Jakarta.
Dellman, H. D dan E. M. Brown. 1989. Buku Teks Histologi Veteriner I. Terjemahan: R. Hartanto. Universitas Indonesia Press, Jakarta.
Djojosoebagio, S. 1996. Peningkatan Produktivitas melalui penerapan bioteknologi. Seminar Nasional Peternakan dan Veteriner. Puslitbangnak, Bogor.
Eko, T. S., M. E. Sawitri dan Murharlien. 2009. Budi Daya 22 Ternak Potensi. Penebar Swadaya, Jakarta.
Frandson, R. D. 199. Anatomi dan Fisiologi Ternak. Terjemahan: Srigandono dan Koen Praseno. Fakultas Peternakan Universitas Diponegoro. Gadjah Mada University Press, Yogyakarta.
Hardjosubroto, W. 1999. Pengantar Genetika Hewan. Fakultas Peternakan Gadjah Mada Yogyakarta. CV. Makmur, Yogyakarta.
Isnaeni, W. 2006. Fisiologi Hewan. Kanisius, Yogyakarta.
Jusuf, M. 2001. Genetika 1 Struktur dan Ekspresi Gen. Institut Pertanian Bogor, Bogor.
Khosravinia, H dan K. P. Ramesha. 2006. Influence of EDTA and magnesium on DNA extraction from blood samples and specificity of polimerase chain reaction. African Journal of Biotechnolody, Vol. 6(3), pp. 184-187.
Khosravinia, H., N. N. Murthy., D. T Parasad dan N. Pirany. 2007. Optimazing factors influencing DNA extraction from fresh whole avian blood. African Journal of Biotechnolody, Vol. 6(4), pp. 481-486.
Maclean, N. 1987. Macmillan Dictionary of Genetics and Cell Biology. The Macmillan Press Ltd, London and Basingstoke.
Mortojo, H. 1992. Peningkatan Mutu Genetik Ternak. Departemen Pendidikan dan Kebudayaan, Direktorat Jenderal Pendidikan Tinggi, Pusat Antar Universitas Bioteknologi, Institut Pertanian Bogor, Bogor.
Muladno. 2002. Teknologi Rekayasa Genetika. Pustaka Wirausaha Muda dan UCC Faundation, Bogor.
Murray, R.K. 1995. Biokimia Harper. Edisi ke-22. EGC penerbit Buku Kedokteran, Jakarta.
Nicholl, D. S. T. 1996. An Introduction to Genetic Engineering. Cambridge University Press.
Noor, R. R. 2008. Genetik Ternak. Penebar Swadaya, Jakarta.
Old, R.W dan S.B. Primrose. 1989. Prinsip-prinsip Manipulasi Gen. Blackwell Scientific Publications, London.
Prahasta, A., H. Masturi., T. Dhalika. 2008. Budidaya Usaha Pengolahan Agrobisnis Ternak Sapi. CV Pustaka Grafika, Bandung.
Prasetyo, A. 2005. Metode ekstraksi DNA identifikasi gen kappa kasein (k- kasein) pada sapi fresian holstein (FH) di peternakan rakyat. Skripsi. Departemen Ilmu Produksi Ternak. Fakultas Peternakan. Institut Pertanian Bogor, Bogor.
Saefudin. 1996. Ternak transgenik dan pandangan genetika kuantitatif. Kumpulan Makalah Ilmiah Hasiil Penelitian Bioteknologi. Pusat antar Universitas Bioteknologi, Institut Pertanian Bogor, Bogor.
Sheeler, P dan D. E. Bianchi. 1987. Cell and Molecular Biology. California State University, Northridge.
Sambrook, J., E. F. Fritsch, dan T. Maniatis. 1989. Moleculer Cloning. A Laboratory Manual. Cold Spring Harbour Lab. CSH, New York.
Santoso. 2008. Mengelola Peternakan Sapi Secara Profesional. Penebar Swadaya, Jakarta.
Steel, R. G. D. dan J. H. Torrie. 1991. Prinsip dan Prosedur Statistik. Terjemahan: B. Sumantri. Gramedia, Jakarta.
Sudono., F. Rosdiana dan B. S. Setiawan. 2003. Beternak Sapi secara Intensif. Agro Media Pustaka, Jakarta.
Susanti, E. VH dan S. R. D. Ariani. 2004. Kloning gen penisilin v asilase dari Bacillus sp bac4 melalui pembuatan pustaka genom. Biodiservitas. Vol (5). Hal 1-6.
Triwibowo, Y. 2008. Biologi Molekuler. Erlangga, Jakarta.
Tyler, H. D dan M. E. Ensminger. 2006. Dairy Cattle Science. Pearson Prentice Hall, Upper saddle River, New Jersey Columbus, Ohio.
Winarno dan W. Agustinah. 2007. Pengantar Bioteknologi. M-Brio Biotekindo Press, Bogor.
Lampiran 1. Diagram Alir Prosedur Ekstraksi DNA Metode Sambrook et al.,1989
a. Lisis sel darah merah dan penghilangan protein
Tambahkan 20 µl proteinase K
Sentrifugasi sesuai perlakuan selama 10 menit pada suhu 10 0C.
Inkubasi pada suhu -20 oC selama 30 menit
Tambahkan 600 µl PCI (25:24:2) Inkubasi pada suhu 56oC selama 1 jam,
b. Ekstraksi DNA
Lapisan yang paling atas pindahkan ke tabung baru
Inkubasi pada suhu -20 oC selama 30 menit
Tambahkan 30 µl 3M NaOAc dan 600 µl etanol 96% dihomogenkan
Pelet dikeringkan dan tambahkan 50 µl TE buffer
DNA disimpan pada suhu -20oC sebagai stok untuk kuantifikasi dan elektroforesis
Supernatan dibuang, pelet dicuci dengan 500 µl etanol 70%
Sentrifugasi sesuai perlakuan selama 10 menit pada suhu 10 0C.
Lampiran 2. Reagent-reagent yang digunakan untuk ekstraksi DNA metode Phenol Choroform-Isoamyl alkohol (PCI)
1. Lisis Buffer 50 ml
1M Tris HCl : 2,5 ml
5M NaCl : 10 ml
0,5 EDTA pH 8 : 2 µl
10 % SDS : 1 ml
Up to H2O : 50 ml
2. Proteinase K 20 mg/ml untuk 24 sempel tiap sempel 20 µl
Proteinase K : 13,4 mg
H2O destilasi : 666,6 µl
3. Phenol-Choroform-Isoamyl alkohol (PCI) 20 ml Phenol : Choroform : Isoamyl alcohol
25 : 24 : 1
Phenol : 10 ml
Choroform : 9,6 ml
Isoamyl alcohol : 0,4 ml
4. 3M NaOAC 5 ml
3M NaOAC : 1,23045 gr
Up to H2O : 5 ml
5. TE Buffer pH 8 300 ml
1M Tris : 2,5 ml
0.5 EDTA : 50 ml
Mili Q : 247,5 ml
6. 1M Tris 50 ml
1M Tris : 6,057 gr
Up to H2O : 50 ml
7. TBE 1X 100 ml
Tris : 5,4 gr
Boric Acid : 2,75 gr
0.5 M EDTA : 2 ml
Up to H2O : 100 ml
8. Gel agarose 1 %
Agarose : 2 gr
Lampiran 3. Data Kuantifikasi DNA (Konsentrasi DNA, Rasio OD 260/280, 260nm, dan 280 nm)
No Perlakuan Konsentrasi ng/ml Rasio 260/280 260 nm 280 nm
1 T1S1 61,8 1,168 0,618 0,528
2 T1S2 203 1,133 2,03 1,792
3 T1S3 78,6 1,572 0,786 0,5
4 T1S4 108,7 1,704 1,087 0,638
5 T2S1 128,5 1,428 1,285 0,9
6 T2S2 109,7 1,154 1,097 0,951
7 T2S3 57,7 1,468 0,577 0,393
8 T2S4 187,5 1,433 1,875 1,308
9 T3S1 89,6 1,12 0,896 0,8
10 T3S2 59,6 1,469 0,598 0,407
11 T3S3 200 1,464 2 1,366
12 T3S4 257 1,308 2,57 1,965
13 T4S1 42,2 1,431 0,422 0,295
14 T4S2 74,5 1,061 0,745 0,702
15 T4S3 152,1 1,056 1,521 1,441
16 T4S4 109,5 1,484 1,095 0,738
17 T5S1 45,1 1,229 0,451 0,367
18 T5S2 27,1 1,168 0,271 0,232
19 T5S3 111,1 1,148 1,111 0,968
20 T5S4 153,5 1,067 1,535 1,439
21 T6S1 78,7 1,151 0,787 0,684
22 T6S2 170,1 1,112 1,701 1,53
23 T6S3 161,7 1,051 1,617 1,539
Lampiran 4. Analisis Ragam Konsentrasi DNA terhadap Pengaruh Perbandingan Volume Darah dan Lisis Buffer dengan Kecepatan Sentrifugasi yang berbeda
Sumber Keragaman db JK KT F P lisis buffer 2 5867 2934 0,74 0,491 sentrifugasi 1 4882 4882 1,23 0,282 lisis buffer*sentrifugasi 2 343 171 0,04 0,958 Error 18 71374 3965
Total 23 82466
S = 62,9699 R-Sq = 13,45% R-Sq(adj) =0,00%
Lampiran 5. Analisis Ragam Rasio OD 260/280 DNA terhadap Pengaruh Perbandingan Volume Darah Lisis Buffer dengan Kecepatan Sentrifugasi yang berbeda
Sumber Keragaman db JK KT F P Lisis buffer 2 0,000854 0,000427 0,36 0,700 sentrifugasi 1 0,00693 0,006936 5,91 0,026 Lisis buffer*sentrifugasi 2 0,000283 0,000142 0,12 0,887 Error 18 0,021118 0,001173
Total 23 0.029191