KLASIFIKASI FRAGMEN
METAGENOME
MENGGUNAKAN
KNN DAN PNN DENGAN EKSTRAKSI FITUR
GRAY LEVEL
CO-OCCURRENCE MATRIX
(GLCM) PADA VARIASI
PANJANG FRAGMEN
MUHAMMAD DHIRA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Klasifikasi Fragmen Metagenome Menggunakan KNN dan PNN dengan Ekstraksi Fitur Gray Level Co-occurrence Matrix (GLCM) pada Variasi Panjang Fragmen adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
MUHAMMAD DHIRA. Klasifikasi Fragmen Metagenome Menggunakan KNN dan PNN dengan Ekstraksi Fitur Gray Level Co-occurrence Matrix (GLCM) pada Variasi Panjang Fragmen. Dibimbing oleh AZIZ KUSTIYO dan WISNU ANANTA KUSUMA
Bioinformatika adalah kajian ilmu yang berkembang sangat pesat di Indonesia. Kajian yang menjadi konsentrasi dalam penelitian ini adalah klasifikasi fragmen metagenom ke dalam beberapa taksonomi menggunakan pendekatan tekstur Gray Level Co-occurence Matrix (GLCM) yang memiliki 13 fitur. Proses klasifikasi menggunakan 50 organisme dengan 5-fold cross validation. Kombinasi sekuens DNA yang terdapat di dalam suatu fragmen dipandang sebagai citra 1xN dengan N adalah panjang fragmen yang dibentuk dalam matrix dua dimensi. Matrix yang telah terbentuk akan diformulasikan ke dalam 13 fitur GLCM. Hasil dari fitur tersebut akan diklasifikasian menggunakan teknik PNN dan KNN. Pada akhir penelitian dilakukan uji klasifikasi menggunakan 4 variasi panjang fragmen, yaitu 200 bp, 1 Kbp, 3 Kbp, 10 Kbp, dan didapatkan nilai akurasi setiap panjang fragmen 100%. Dapat disimpulkan bahwa variasi panjang fragmen tidak memengaruhi akurasi. Selain itu, dapat disimpulkan bahwa metode ekstraksi fitur GLCM memiliki prospek yang baik untuk klasifikasi fragmen metagenome. Kata kunci: Fragmen, GLCM, KNN, PNN
ABSTRACT
MUHAMMAD DHIRA. Metagenome Fragment Binning Using KNN and PNN With Gray Level Co-Occurrence Matrix (GLCM) on the Variation of the Length of Fragments. Supervised with AZIZ KUSTIYO and WISNU ANANTA KUSUMA
Bioinformatics is a field of study which is developing rapidly in Indonesia. The main focus of this study is to classify metagenome fragment into some taxonomies using GLCM that has 13 features. The training data used in the classification process are 50 organisms with 5-fold cross validation. The DNA combination sequences inside a fragment can be seen as a 1xN image, where N is the fragment’s length in two-dimension’s matrix. The result from those features will be classified using PNN and KNN. The research shows that the accuracy percentage with all lengths variety, including 200 bp, 1 Kbp, 3 Kbp, and 10 Kbp, are 100%. It can be stated that the variety fragment’s length does not affect the accuracy. In addition, it can be concluded that GLCM feature extraction method can be prospectively implemented for classifying metagenome fragment.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Komputer
pada
Departemen Ilmu Komputer
KLASIFIKASI FRAGMEN
METAGENOME
MENGGUNAKAN
KNN DAN PNN DENGAN EKSTRAKSI FITUR
GRAY LEVEL
CO-OCCURRENCE MATRIX
(GLCM) PADA VARIASI
PANJANG FRAGMEN
MUHAMMAD DHIRA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
Judul Skripsi : Klasifikasi Fragmen Metagenome Menggunakan KNN dan PNN dengan Ekstraksi Fitur Gray Level Co-occurrence Matrix (GLCM) pada Variasi Panjang Fragmen
Nama : Muhammad Dhira NIM : G64100068
Disetujui oleh
Aziz Kustiyo, SSi, MKom Pembimbing I
Dr Eng Wisnu Ananta Kusuma, ST, MT Pembimbing II
Diketahui oleh
Dr Ir Agus Buono, MSi, MKom Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Desember 2013 ini ialah klasifikasi fragmen metagenome, dengan judul Klasifikasi Fragmen Metagenome Menggunakan KNN dan PNN dengan Ekstraksi Fitur Gray Level Co-occurrence Matrix (GLCM) pada Variasi Panjang Fragmen. Penulis juga menyampaikan terima kasih kepada seluruh pihak yang telah berperan dalam penelitian ini, yaitu: 1 Kedua orangtua, kakak, adik, dan keluarga atas doa, motivasi, dan kasih
sayangnya untuk menyelesaikan penelitian ini.
2 Bapak Aziz Kustiyo, SSi, MKom dan Bapak Dr Eng Wisnu Ananta Kusuma selaku dosen pembimbing yang telah memberi ide, saran, dan bantuan hingga penelitian ini selesai.
3 Bapak Toto Haryanto, SKom, MSi selaku dosen penguji yang telah memberi saran dalam penelitian ini.
4 Keluarga besar MAX!! yang membuat saya berkembang secara moral dan membantu music terus berkembang selama saya kuliah di IPB.
5 Rekan satu bimbingan, yaitu Machmum Aliefiya atas kerjasamanya selama ini.
6 Kresna Harimurti, Dimas Napitupulu, Disqa dewintami dan rekan-rekan PIXELS 47 atas segala kebersamaan, bantuan, dan dukungan selama menjalani masa studi.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
METODE 3
Pengambilan Data 3
Praproses Data 4
Ekstraksi Ciri 4
K-fold cross validation 7
Probabilictic Neural Network (PNN) 7
K-Nearest Neighbor (KNN) 8
Analisis dan Evaluasi 8
HASIL DAN PEMBAHASAN 9
Pengambilan Data 9
Praproses Data 9
Ekstraksi Ciri 10
Model klasifikasi Data 11
SIMPULAN DAN SARAN 12
Simpulan 12
Saran 12
DAFTAR PUSTAKA 13
DAFTAR GAMBAR
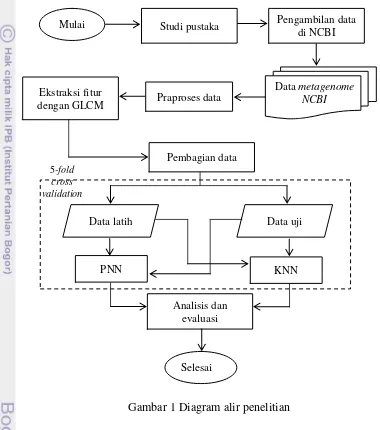
1 Diagram alir penelitian 3
2 Tahap mengubah sekuens DNA menjadi matrix 4
3 Potongan fragmen dengan panjang 200 bp 10
4 Potongan hasil ekstraksi fitur 11
5 Boxplot perbandingan variasi panjang dari fitur ASM 11 DAFTAR LAMPIRAN
1 Daftar nama organisme 14
2 Daftar nama genus 15
3 Daftar nama data latih dan data uji 16
1
PENDAHULUAN
Latar Belakang
Teknologi yang berhubungan dengan bioinformatika mulai berkembang di Indonesia. Banyak ahli yang ingin menggali lebih dalam potensi yang dapat dihasilkan dari bidang yang tergolong baru ini. Bioinformatika adalah salah satu cabang ilmu biologi yang merupakan perpaduan antara biologi dan teknik informasi. Bioinformatika juga dapat disebut ilmu biologi yang analisis datanya disimpan dalam database.
Studi yang menjadi fokus utama dalam penelitian ini adalah metagenomics. Berbeda dengan studi tentang genomics, metagenomics tidak memerlukan pure clonal cultures dari individu tertentu. DNA yang berasal dari suatu komunitas mikrob dapat diperoleh melalui sequencing secara langsung (Mchardy 2007).
Sequencing secara langsung dari komunitas mikrob dapat menyebabkan kesalahan rekayasa atau perakitan fragmen dalam suatu kelompok mikroorganisme yang menyebabkan dihasilkannya cymerics contigs. Solusi yang dapat diterapkan untuk meminimalkan adanya cymerics contigs adalah melakukan sequence assembly dan binning. Dalam hal ini, binning yang dimaksud adalah melakukan pengelompokan dengan supervised atau unsupervised learning sampai ke tahap genus, mengingat penglasifikasian sampai tahap spesies sulit dilakukan.
Metode yang telah digunakan dalam penelitian sebelumnya ialah multiclass
support vector machine (SVM) dengan frekuensi k-mers sebagai fiturnya (McHardy
et al. 2007 dalam Ariny 2013). Klasifikasi fragmen metagenome menghasilkan
akurasi yang cukup tinggi yaitu 60% sampai 90% dengan panjang ≥ 5 Kbp. Akan tetapi akurasi menurun drastis hingga 30% apabila diuji dengan panjang ≤ 3 Kbp. Selain itu, metode ini mengeluarkan biaya yang cukup mahal karena waktu penghitungan kernel yang cukup lama dan pemodelan SVM yang cukup kompleks yaitu dengan 5-mers. Oleh karena itu, penelitian ini akan dicoba melalui sudut pandang yang berbeda yaitu menggunakan pendekatan tekstur dengan hanya 2-mers.
2
Setelah ketiga belas ciri dihitung akan digunakan kalsifikasi KNN dan PNN untuk menentukan pengelompokkan antar genus. PNN digunakan karena hanya mengalami satu kali iterasi dalam prosesnya. Hasil yang didapat akan dihitung peluang terbesarnya untuk menentukan apakah suatu organisme telah diklasifikasikan secara tepat. Sementara itu KNN digunakan karena prosesnya sangat sederhana, hanya menentukan jarak ketetanggaan dengan k yang telah ditentukan. Kedua tenik klasifikasi ini digunakan untuk dibandingkan hasilnya. Setelah dibandingkan akan ditentukan teknik klasifikasi yang tebaik diantara keduanya.
Perumusan Masalah
Pemetaan Fragmen metagenome ke dalam suatu matrix menjadi sangat krusial karena akan dihitung 13 ciri fitur dari matrix tersebut dengan orientasi sudut 0º. Pertanyaan yang muncul pada penelitian ini sebagai berikut:
1 Bagimana menerapkan metode GLCM terhadap sekuens DNA dari fragmen metagenom?
2 Berapa akurasi yang diperoleh dengan menggunakan metode KNN dan PNN?
3 Bagaimana pengaruh panjang fragmen terhadap akurasi? 4 Bagaimana pengaruh nilai k terhadap kinerja KNN?
Tujuan Penelitian Tujuan dari penelitian ini, yaitu:
1 Menerapkan metode GLCM pada sekuens DNA.
2 Menerapkan PNN dan KNN pada klasifikasi sekuens DNA berdasarkan fitur dari GLCM.
3 Mengetahui berapa akurasi PNN terhadap sekuens DNA. 4 Mengetahui akurasi seluruh k pada KNN.
Manfaat Penelitian
Penelitian ini diharapkan dapat menjadi sarana pengembangan klasifikasi fragmen metagenome menggunakan ekstraksi ciri citra dan dapat digunakan untuk pengklasifikasian mikroorganisme jenis baru.
.
Ruang Lingkup Penelitian Ruang lingkup dari penelitian ini meliputi:
1 Orientasi sudut pada fitur GLCM terbatas pada 0°.
2 Data bioinformatika berupa DNA yang terbatas hanya 5 genus yaitu Burkholderia, Clostridium, Mycobacterium, Staphylococcus dan Streptococcus.
3
METODE
Penelitian dimulai dengan mengambil data, praproses data, ekstraksi fitur dengan GLCM, pembagian data latih dan data uji, klasifikasi PNN dan KNN, dan analisis evaluasi. Ilustrasi metode penelitian dapat dilihat pada Gambar 1
Pengambilan Data
Data yang diambil adalah data metagenome yang diambil dari National Centre for Biotechnology Information (NCBI). NCBI merupakan institusi yang terkait dengan biologi molekuler dan menjadi pusat informasi tentang perkembangan. Data yang diambil berupa sekuens DNA dengan format FastA. Alamat untuk mengunduh data ini yaitu ftp://ftp.ncbi.nih.gov/ genomes/Bacteria/.
Gambar 1 Diagram alir penelitian 5-fold
cross validation
Mulai Pengambilan data
di NCBI Studi pustaka
Data metagenome NCBI
Ekstraksi fitur dengan GLCM
Pembagian data
Data latih Data uji
Analisis dan evaluasi
KNN PNN
4
Praproses Data
Tekstur adalah sifat-sifat atau karakteristik yang dimiliki suatu daerah (dalam hal ini isi dari matrix-nya) yang cukup signifikan dan daerah tersebut secara alami mempunyai sesuatu secara berulang. Dalam hal ini pengertian tekstur yang dimaksud adalah keteraturan dari sekuens DNA dari suatu mikroorganisme yang membentuk pola-pola tertentu. Sekuens DNA dikatakan mempunyai informasi tekstur jika sekuens DNA antar satu mikroorganisme dan lainnya mempunyai kemiripan dari segi jarak dan orientasi sudut. Dalam penelitian ini jarak dan orientasi sudut yang digunakan adalah 2-mers dan 0°.
Pada tahap praproses sekuens DNA setiap fragmen dipetakan ke dalam matrix 4x4 sesuai banyaknya fragmen dengan jarak 1 (bersebelahan). Setelah itu semua matrix yang telah dihitung ditotal menjadi satu matrix. Suatu nilai dari setiap panjang yang telah dihitung akan dilakukan normalisasi, yaitu membagi suatu elemen dengan jumlah dari seluruh elemen. Secara umum, analisis tesktur dapat dilakukan dengan dua pendekatan yaitu pendekatan struktural dan statistikal. GLCM merupakan salah satu metode yang paling umum untuk menganalisis tekstur.
GLCM dapat dibentuk dengan cara berikut (Lihat Gambar 2) :
1 Tentukan elemen yang terkandung dalam sekuens DNA yaitu A, T, G dan C.
2 Bentuk matrix A dengan ukuran 4x4 yaitu sesuai dengan banyaknya elemen yang terkandung dalam DNA dimana elemen menyatakan jumlah kemunculan dari DNA yang bertetangga dengan interval 1 untuk setiap satu fragmen metagenom dengan orientasi sudut 0°.Fragmen yang digunakan adalah dengan panjang 200 bp, 1 Kbp, 3 Kbp, dan 10kbp dengan banyaknya fragmen menyesuaikan.
3 Bentuk matrix co-occurrence dengan cara membagi elemen dengan jumlah (sekuens-1) atau total dari semua elemen . Dengan demikian dapat dikatakan bahwa matrix A telah dinormalisasi.
Gambar 2 Tahap mengubah sekuens DNA menjadi matrix
Ekstraksi Ciri
5 suatu sekuens ke dalam sekelompok nilai ciri yang sesuai. Ekstraksi ciri pada sekuens DNA dapat dilakukan jika dan hanya jika matrix co-ocurrence telah dinormalisasi. Haralick et al. (1973) mengusulkan berbagai fitur ciri tekstural yang dapat diekstraksi dari matrix co-occurence. Haralick mengungkapkan bahwa beberapa dari ekstraksi ciri citra merupakan perhitungan untuk pengenalan karakteristik citra meliputi homogenitas, kontras dan keberadaan tekstur dalam suatu citra. Dalam hal ini, kemiripan sekuens satu DNA dengan yang lainnya.
Akan tetapi, sulit untuk menentukan apakah suatu organisme dikatakan sebagai penciri walaupun fitur telah digunakan untuk menentukan karakteristik dari organisme tersebut. Dalam penelitian ini dilakukan 13 fitur yang diusulkan oleh Haralick, yang akan ditentukan sebagai penciri dan bukan penciri.
Keempat belas fitur tersebut adalah: 1 ASM (Angular Second Moment)
Homogenitas dari Sekuens DNA
ASM = ∑ ∑
Dimana p(i,j) menyatakan nilai dari baris i dan kolom j dalam matrix co-occurrence yang telah dinormalisasi.
2 Contrast
Menunjukkan ukuran penyebaran (momen inersia) elemen-elemen matrix. Jika letaknya jauh dari diagonal utama, nilai kekontrasan besar. Secara visual, nilai kekontrasan adalah ukuran variasi antar kombinasi dengan interval tertentu pada DNA.
Con = ∑ |-|2 3 Correlation
Ukuran ketergantungan linear pada sekuens DNA sehingga dapat dilihat ciri tektstural dari sekuens tersebut.
Cor = ∑ ∑ (- )(- ) dengan:
µi = nilai rata-rata baris ke-i matrix p
µj = nilai rata-rata kolom ke-j matrix p
= standar deviasi baris ke-i matrix p = standar deviasi kolom ke-j matrix p
4 Variance
6
Var = ∑ ∑
5 Inverse Different Moment
Menunjukkan kehomogenan sekuens DNA dalam kombinasi yang sejenis. Sekuens DNA yang sejenis akan memilik IDM yang besar
IDM = ∑ 1
|-|
6 Entropy
Menunjukkan ukuran ketidakteraturan bentuk. Harga ENT besar untuk sekuens DNA kombinasi yang sejenis merata dan bernilai kecil jika sekuens DNA tersebut kombinasinya bervariasi.
ENT = - ∑ log
7 Sum Entropy
SENT = ∑
8 Sum Average
AVER = ∑ 9 Sum Variance
SVAR = ∑ 10 Difference Variance
DVAR = variance of 11 Difference Entropy
DENT = ∑ 12 Information Measures of Correlation1
IMC1=
13 Information Measures of Correlation 2
IMC2 = ⁄
7 HXY1 = ∑ ∑
HXY2 = ∑ ∑
14 Maximal Correlation Coefficient(tidak digunakan)
MCC = ⁄ dimana
Q(i,j) = ∑
K-fold cross validation
K-fold cross validation adalah metode pembagian sebuah kelompok data yang akan dibagi ke dalam data latih dan data uji . Pembuatan partisi dilakukan dengan cara melakukan pengolahan data sebanyak k kali dengan menggunakan k-1 data latih dan sisanya data uji. Akurasi didapat dari rata-rata seluruh k percobaan (Zhang dan Wu 2011) dalam Amalia (2013). Penelitian ini menggunakan k sebesar 5 dan data berupa 50 organisme (Lihat Lampiran 1) yang terbagi ke dalam 5 genus (Lihat Lampiran 2) dengan masing- masing genus terdapat 10 mikroorganisme. Data tersebut akan dibagi ke dalam 4/5 data latih dan 1/5 data uji yaitu 10 data uji dan 40 data latih. Oleh karena itu akan terbentuk matrix citra latih berukuran 40x5 dan citra uji 10x5. Pembagian data yang dilatih dan diuji dapat dilihat pada Lampiran 3.
Probabilictic Neural Network (PNN)
PNN adalah teknik klasifikasi yang mengadaptasi bayesian network dan analisis algoritme statistik yaitu kernel fisher discriminant analysis. Terdapat empat tahap pengoperasian PNN yang terangkum dalam empat layer. Layer pertama adalah input hasil dari ketiga belas ekstraksi ciri pada matrix co-occurrence.
Layer kedua menghitung jarak antara vektor masukan pada data uji dan data latih yang akan dibagi ke dalam fakor penghalus. Faktor penghalus yang digunakan adalah 0.1. Faktor penghalus digunakan untuk menghaluskan fakor kernel, dalam hal ini faktor kernel yang dimaksud adalah fungsi Gauss. Setelah itu hasil perhitungannya dibagi kedalam fungsi parzen untuk mengukur Probabilistic density function. Layer ketiga menghitung kontribusi dari setiap input-an dan menghitung keluaran berupa peluang dari vektor.
p p | 1 2 2
∑ exp
2 2 1
dengan:
8
p(x|A) = peluang bersyarat x jika masuk ke dalam kelas A
xAi = vektor data latih kelas A urutan ke-i
d = dimensi vektor masukan
N = jumlah pola pelatihan seluruh kelas NA = jumlah pola pelatihan pada kelas A
= faktor penghalus
Layer keempat menganalisa masukan data uji yang akan diklasifikasikan ke dalam suatu kelas berdasarkan nilai peluang tertinggi. Metode PNN lebih banyak digunakan dalam klasifikasi dibandingkan dengan multilayer perceptron. PNN hanya mengalami satu kali iterasi (Specht 1990) dalam Faturohman (2009) dan dapat menghasilkan output yang lebih akurat, yaitu berupa nilai peluang yang merepresentasikan suatu data uji masuk dalam klasifikasi kelas tertentu.
K-Nearest Neighbor (KNN)
Algoritme K-Nearest Neighbor (KNN) adalah sebuah metode yang menggunakan klasifikasi berdasarkan data pembelajaran yang memiliki jarak terdekat dari suatu objek. Tujuan dari algoritme ini adalah mengklasifikasikan obyek baru berdasarkan training sample. Classifier hanya berdasarkan pada memori dengan menggunakan klasifikasi ketetanggaan sebagai nilai prediksi dari query instance yang baru.
Jarak yang digunakan untuk menentukan ketetanggaan dari suatu titik digunakan jarak eucledian. Jarak eucledian berfungsi untuk mengukur kedekatan antar obyek yang berdekatan sebagai sebuah interpretasi (Han et al. 2011). Kedekatan antara dua obyek ditentukan oleh sebuah kelas yang paling banyak ditemui pada k buah tetangga dari suatu obyek tersebut. Pada penelitian kali ini digunakan k=10.
Nilai k yang terbaik untuk algoritme KNN tergantung pada data. Nilai k yang tinggi dapat mengurangi efek noise pada klasifikasi dengan akibat antar kelas kabur atau sulit dibedakan. Langkah untuk menghitung menggunakan metode KNN sangat sederhana. Pertama menentukan jumlah k terdekat (k=10), kemudian menghitung kuadrat euclid masing-masing obyek yang diberikan. Lalu mengurutkan objek-objek tersebut kedalam kelompok yang mempunyai Euclid terkecil. Terakhir mengumpulkan klasifikasi nearest neighbor menggunakan kategori nearest neighbor yang dapat memprediksi nilai query instance (data uji) yang telah dihitung.
Analisis dan Evaluasi
Setelah seluruh output dari KNN dan PNN telah ditentukan, akan dihitung akurasi tiap kelasnya dengan cara:
akurasi ∑data uji benar
9 Lingkungan Pengembangan
Spesifikasi perangkat keras yang digunakan untuk penelitian ini sebagai berikut:
Processor Intel® CoreTM i7 CPU Memori RAM 4 GB
Harddisk 640 GB
Spesifikasi perangkat lunak yang digunakan untuk penelitian ini sebagai berikut:
Sistem operasi : Windows 7 Simulasi sequencer : MetaSim Compiler : MATLAB
HASIL DAN PEMBAHASAN
Pengambilan Data
Data yang diambil dari NCBI diambil sebanyak 50 organisme dari 5 genus berupa sekuens DNA.
Praproses Data
Data DNA yang terkumpul dalam bentuk sekuens DNA akan diekstraksi dengan panjang 200 bp, 1 Kbp, 3 Kbp,dan 10 Kbp dengan masing-masing 50 organisme menggunakan metasim. Terdapat aturan untuk menentukan berapa banyak fragmen yang diperlukan untuk panjang setiap fragmen yang digunakan. Untuk menentukan perbandingan panjang dan banyaknya fragmen digunakan rumus berikut:
n= banyak fragmen yang dibutuhkan l= panjang fragmen yang dibutuhkan
L= total rata-rata dari seluruh panjang mikroorganisme
10
Panjang fragmen
Banyak fragmen
200 bp 45000
1 Kbp 36000
3 Kbp 12000
10 Kbp 4000
Tabel 1 Perbandingan panjang dan banyak fragmen
Dapat dilihat dari pola tersebut bahwa semakin panjang fragmen maka semakin sedikit jumlah fragmen yang dibutuhkan. Contoh output dari metasim dengan panjang fragmen 200 dapat dilihat pada Gambar 3.
Gambar 3 Potongan fragmen dengan panjang 200 bp Ekstraksi Ciri
Setelah semua sekuens DNA dipotong ke dalam beberapa variasi panjang, sekuens tersebut diekstraksi cirinya menggunakan GLCM. Matrix co-occurrence dihitung berdasarkan sudut dan jarak yang telah ditentukan. Dalam kasus ini jaraknya adalah satu dengan sudut 0º. Baris DNA tersebut akan dihitung pasangan antar elemennya dengan jarak 1. Banyaknya pasangan tersebut akan dimasukkan kedalam matrix 4x4 sesuai dengan banyaknya elemen dari DNA yaitu A, T, G dan C. Setelah itu matrix dinormalisasi dan dihitung untuk 13 fitur Haralick.
11
Gambar 4 Potongan hasil ekstraksi fitur
Model klasifikasi Data
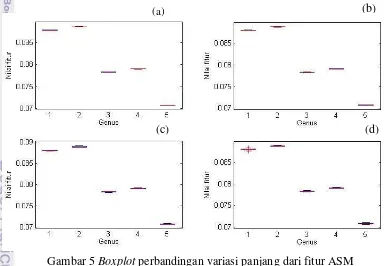
Pembagian terhadap data latih dan uji menggunakan k-fold cross validation. Dalam penelitian ini digunakan dengan 5 fold. Karena total sekuens setiap panjang yang berbeda terdapat 50 data dari 50 organisme, maka data latih sebanyak 40 dan data uji sebanyak 10 dengan 5 jenis fold. Klasifikasi menggunakan PNN yang nantinya akan dilihat apakah model klasifikasi yang digunakan cocok. Hasil menunjukkan bahwa semua jenis panjang fragmen memiliki akurasi bernilai 100%. Dapat dilihat pada Gambar 5 yaitu beberapa boxplot dari fitur ASM dengan panjang 200 bp (a), 1 Kbp (b), 3 Kbp (c) dan 10 Kbp (d) terhadap 5 genus tersebut. Gambar tersebut menghasilkan perbedaan yang sangat signifikan dari setiap genus.
Gambar 5 Boxplot perbandingan variasi panjang dari fitur ASM
Sebagai contoh fitur ASM pada keempat variasi panjang tesebut menunjukkan tingkat homogenitas dari sebuah data. Semakin kecil rentang nilainya, maka semakin tinggi homogenitasnya. Gambar 5 memiliki rentang rata-rata antara 0.07-0.095 yang berarti kemiripan antar genus tersebut tinggi. Gambar
(c) (a)
12
5 memperlihatkan bahwa hasil pengelompokkan antar genusnya dapat dibedakan walaupun hasil dari ekstraksi ciri tiap panjangnya berbeda. Disimpulkan bahwa variasi panjang fragmen yang dilakukan pada penelitian ini tidak menimbulkan perbedaan hasil klasifikasi antar genusnya. Pada Lampiran 4 akan diperlihatkan boxplot dari ketiga belas ciri dengan panjang 200 bp.
Pada klasifikasi KNN, percobaan dilakukan dengan panjang 200 bp. Hasil menunjukkan tidak ada perbedaan dengan klasifikasi PNN. Tidak ada perbedaan hasil pada percobaan berikutnya dengan panjang 1 Kbp, 3 Kbp, dan 10 Kbp. Percobaan juga dilakukan dengan mengganti nilai k yaitu dengan k=1,..,10. Hasilnya menunjukkan hal yang sama dengan percobaan sebelumnya yaitu 100%. Semua fitur dari metode GLCM memperlihatkan hasil yang sama seperti gambar diatas yaitu terdapat berbedaan yang signifikan antar genusnya. Sehingga dapat diketahui bahwa PNN dan KNN secara sempurna dapat mengklasifikasikan antar genus karena perbedaan yang dapat dilihat dari boxplot untuk setiap fiturnya.
SIMPULAN DAN SARAN
Simpulan
Model identifikasi terhadap fragmen metagenome menggunakan PNN dan KNN berhasil dilakukan. Akurasi tertinggi untuk PNN didapat dengan nilai 100%. Percobaan menggunakan panjang yang beragam dengan coverage yang sama tidak memberikan dampak yang signifikan terhadap hasil akurasi. Fragmen dengan panjang 200 bp, 1 Kbp, 3 Kbp dan 10 Kbp adalah sama, yaitu 100%. Dapat dikatakan bahwa seluruh data dapat diklasifikasikan berdasarkan genusnya secara sempurna. Klasifikasi KNN didapat dengan nilai yang sama yaitu 100% untuk semua k. Dalam hal ini diambil k yang paling dekat yaitu k=1 karena mengambil jarak tetangga yang paling dekat.
Seluruh percobaan yang telah dilakukan dapat disimpulkan bahwa hasil yang sempurna disebabkan oleh data yang sedikit dan sempit jangakuannya, yaitu hanya terdiri 50 organisme dengan variasi dari 5 genus yang berbeda. Jarak yang digunakan hanya kombinasi dari sebelah sekuens DNA-nya dengan jarak 1 dengan orientasi sudut 0°.
Saran
Beberapa saran untuk penelitian selanjutnya:
1 Menggunakan pendekatan GLCM memakai seluruh sudut yaitu 0°, 45°, 90°, 135°.
2 Menggunakan data dengan tingkatan takson yang mirip (satu genus satu ordo). 3 Menggunakan data lebih banyak.
13
DAFTAR PUSTAKA
Amalia, RH. 2013. Identifikasi citra hama tanaman menggunakan gray level co-occurence matrix dan klasifikasi probabilistic neural network [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Ariny. 2013. Klasifikasi fragmen metagenome menggunakan metode support vector machine (SVM) [skripsi]. Bogor (ID): Institut Pertanian Bogor. Fathurohman Z. 2009. Pengembangan probabilistic neural network untuk
penentuan kematangan belimbing manis [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Han J, Kamber, Pei J. 2011. Data Mining Concepts and Techniques. Edition. San francisco (US): Elsevier.
Haralick MR, Shanmugan K, Dinstein I. 1973. Textural features for image classification. IEEE Transactions on Systems, Man, and Cybernetics. 3(6):610-621. doi: 10.1109/tsmc.1973.4309314
McHardy AC, Martín HG, Tsirigos A, Hugenholtz P, Rigoutsos I. 2007. Accurate phylogonetic classification of variabel-length DNA fragments. Nature Methods. 4(1):63–72. doi: 10.1038/nmeth976.
McHardy AC, Rigoutsos I. 007. What’s in the mi : phylogenetic classification of metagenome sequence samples. Current Opinion in Microbiology. 10(5):499–503. doi: 10.1016/j.mib.2007.08.004.
Specht DF. 1990. Probabilistic neural networks and the polynomial adalines as complementary techniques for classification. IEEE Transaction on Neural Networks, 1(1), hal. 111-121.
14
Lampiran 1 Daftar nama organisme
No Nama organisme No Nama organisme
1 Burkholderia ambifaria AMMD chromosome chromosome 1
26 Mycobacterium gilvum PYR-GCK chromosome
2 Burkholderia ambifaria MC40-6 chromosome chromosome 1
27 Mycobacterium leprae TN chromosome
3 Burkholderia cenocepacia AU 1054 chromosome 3
28 Mycobacterium marinum M 4 Burkholderia cenocepacia HI2424
chromosome chromosome 1
29 Mycobacterium smegmatis str. MC2 155
5 Burkholderia cenocepacia J2315 chromosome chromosome 1
30 Mycobacterium sp. JLS chromosome 6 Burkholderia cenocepacia MC0-3
chromosome chromosome 1
31 Staphylococcus aureus RF122 7 Burkholderia mallei ATCC 23344
chromosome chromosome 1
32 Staphylococcus aureus subsp. aureus COL chromosome
8 Burkholderia mallei NCTC 10229 chromosome I
33 Staphylococcus aureus subsp. aureus JH1
9 Burkholderia mallei NCTC 10247 chromosome I
34 Staphylococcus aureus subsp. aureus JH9
10 Burkholderia mallei SAVP1 chromosome I
35 Staphylococcus aureus subsp. aureus MRSA252 chromosome
11 Clostridium acetobutylicum ATCC 824 36 Staphylococcus aureus subsp. aureus MSSA476 chromosome
12 Clostridium beijerinckii NCIMB 8052 chromosome
37 Staphylococcus aureus subsp. aureus Mu3
13 Clostridium botulinum A3 str. Loch Maree
38 Staphylococcus aureus subsp. aureus Mu50
14 Clostridium botulinum A str. ATCC 19397
39 Staphylococcus aureus subsp. aureus MW2
15 Clostridium botulinum A str. ATCC 3502
40 Staphylococcus aureus subsp. aureus N315
16 Clostridium botulinum A str. Hall 41 Streptococcus agalactiae 2603V/R 17 Clostridium botulinum B1 str. Okra 42 Streptococcus agalactiae A909 18 Clostridium botulinum B str. Eklund
17B
43 Streptococcus agalactiae NEM316 19 Clostridium botulinum E3 str. Alaska
E43
44 Streptococcus equi subsp. zooepidemicus MGCS10565
20 Clostridium botulinum F str. Langeland 45 Streptococcus gordonii str. Challis substr. CH1
21 Mycobacterium abscessus chromosome 46 Streptococcus mutans UA159 chromosome
22 Mycobacterium avium 104 47 Streptococcus pneumoniae CGSP14 23 Mycobacterium avium subsp.
paratuberculosis K-10
48 Streptococcus pneumoniae D39 24 Mycobacterium bovis AF2122/97 49 Streptococcus pneumoniae G54 25 Mycobacterium bovis BCG str. Pasteur
1173P2
15 Lampiran 2 Daftar nama genus
No Genus
16
Lampiran 3 Daftar nama data latih dan data uji Percobaan 1
Data latih
No Nama organisme No Nama organisme
1 Burkholderia cenocepacia AU 1054 chromosome 3
21 Mycobacterium leprae TN chromosome
2 Burkholderia cenocepacia HI2424 chromosome chromosome 1
22 Mycobacterium marinum M 3 Burkholderia cenocepacia J2315
chromosome chromosome 1
23 Mycobacterium smegmatis str. MC2 155
4 Burkholderia cenocepacia MC0-3 chromosome chromosome 1
24 Mycobacterium sp. JLS chromosome
5 Burkholderia mallei ATCC 23344 chromosome chromosome 1
25 Staphylococcus aureus subsp. aureus JH1
6 Burkholderia mallei NCTC 10229 chromosome I
26 Staphylococcus aureus subsp. aureus JH9
7 Burkholderia mallei NCTC 10247 chromosome I
27 Staphylococcus aureus subsp. aureus MRSA252 chromosome 8 Burkholderia mallei SAVP1
chromosome I
28 Staphylococcus aureus subsp. aureus MSSA476 chromosome 9 Clostridium botulinum A3 str. Loch
Maree
29 Staphylococcus aureus subsp. aureus Mu3
10 Clostridium botulinum A str. ATCC 19397
30 Staphylococcus aureus subsp. aureus Mu50
11 Clostridium botulinum A str. ATCC 3502
31 Staphylococcus aureus subsp. aureus MW2
12 Clostridium botulinum A str. Hall 32 Staphylococcus aureus subsp. aureus N315
13 Clostridium botulinum B1 str. Okra 33 Streptococcus agalactiae NEM316 14 Clostridium botulinum B str. Eklund
17B
34 Streptococcus equi subsp. zooepidemicus MGCS10565
15 Clostridium botulinum E3 str. Alaska E43
35 Streptococcus gordonii str. Challis substr. CH1
16 Clostridium botulinum F str. Langeland 36 Streptococcus mutans UA159 chromosome
17 Mycobacterium avium subsp. paratuberculosis K-10
37 Streptococcus pneumoniae CGSP14
18 Mycobacterium bovis AF2122/97 38 Streptococcus pneumoniae D39 19 Mycobacterium bovis BCG str. Pasteur
1173P2
39 Streptococcus pneumoniae G54 20 Mycobacterium gilvum PYR-GCK
chromosome
40 Streptococcus pneumoniae Hungary19A-6
Data Uji
No Nama organisme No Nama organisme
1 Burkholderia ambifaria AMMD chromosome chromosome 1
17 Lanjutan
No Nama organisme No Nama organisme
2 Burkholderia ambifaria MC40-6 chromosome chromosome 1
7 Staphylococcus aureus RF122
3 Clostridium acetobutylicum ATCC 824 8 Staphylococcus aureus subsp. aureus COL chromosome
4 Clostridium beijerinckii NCIMB 8052 chromosome
9 Streptococcus agalactiae 2603V/R 5 Mycobacterium abscessus chromosome 10 Streptococcus agalactiae A909
Percobaan 2 Data latih
No Nama organisme No Nama organisme
1 Burkholderia ambifaria AMMD chromosome chromosome 1
21 Mycobacterium leprae TN chromosome
2 Burkholderia ambifaria MC40-6 chromosome chromosome 1
22 Mycobacterium marinum M 3 Burkholderia cenocepacia J2315
chromosome chromosome 1
23 Mycobacterium smegmatis str. MC2 155
4 Burkholderia cenocepacia MC0-3 chromosome chromosome 1
24 Mycobacterium sp. JLS chromosome 5 Burkholderia mallei ATCC 23344
chromosome chromosome 1
25 Staphylococcus aureus RF122 6 Burkholderia mallei NCTC 10229
chromosome I
26 Staphylococcus aureus subsp. aureus COL chromosome
7 Burkholderia mallei NCTC 10247 chromosome I
27 Staphylococcus aureus subsp. aureus MRSA252 chromosome
8 Burkholderia mallei SAVP1 chromosome I
28 Staphylococcus aureus subsp. aureus MSSA476 chromosome
9 Clostridium acetobutylicum ATCC 824 29 Staphylococcus aureus subsp. aureus Mu3
10 Clostridium beijerinckii NCIMB 8052 chromosome
30 Staphylococcus aureus subsp. aureus Mu50
11 Clostridium botulinum A str. ATCC 3502
31 Staphylococcus aureus subsp. aureus MW2
12 Clostridium botulinum A str. Hall 32 Staphylococcus aureus subsp. aureus N315
13 Clostridium botulinum B1 str. Okra 33 Streptococcus agalactiae 2603V/R
14 Clostridium botulinum B str. Eklund 17B
34 Streptococcus agalactiae A909 15 Clostridium botulinum E3 str. Alaska
E43
35 Streptococcus gordonii str. Challis substr. CH1
16 Clostridium botulinum F str. Langeland 36 Streptococcus mutans UA159 chromosome
18 Lanjutan
No Nama organisme No Nama organisme
19 Mycobacterium bovis BCG str. Pasteur 1173P2
39 Streptococcus pneumoniae G54 20 Mycobacterium gilvum PYR-GCK
chromosome
40 Streptococcus pneumoniae Hungary19A-6
Data uji
No Nama organisme No Nama organisme
1 Burkholderia cenocepacia AU 1054 chromosome 3
6 Mycobacterium bovis AF2122/97 2 Burkholderia cenocepacia HI2424
chromosome chromosome 1
7 Staphylococcus aureus subsp. aureus JH1
3 Clostridium botulinum A3 str. Loch Maree
8 Staphylococcus aureus subsp. aureus JH9
4 Clostridium botulinum A str. ATCC 19397
9 Streptococcus agalactiae NEM316 5 Mycobacterium avium subsp.
paratuberculosis K-10
10 Streptococcus equi subsp. zooepidemicus MGCS10565
Percobaan 3 Data latih
No Nama organisme No Nama organisme
1 Burkholderia ambifaria AMMD chromosome chromosome 1
21 Mycobacterium leprae TN chromosome
2 Burkholderia ambifaria MC40-6 chromosome chromosome 1
22 Mycobacterium marinum M 3 Burkholderia cenocepacia AU 1054
chromosome 3
23 Mycobacterium smegmatis str. MC2 155
4 Burkholderia cenocepacia HI2424 chromosome chromosome 1
24 Mycobacterium sp. JLS chromosome
5 Burkholderia mallei ATCC 23344 chromosome chromosome 1
25 Staphylococcus aureus RF122 6 Burkholderia mallei NCTC 10229
chromosome I
26 Staphylococcus aureus subsp. aureus COL chromosome
7 Burkholderia mallei NCTC 10247 chromosome I
27 Staphylococcus aureus subsp. aureus JH1
8 Burkholderia mallei SAVP1 chromosome I
28 Staphylococcus aureus subsp. aureus JH9
9 Clostridium acetobutylicum ATCC 824 29 Staphylococcus aureus subsp. aureus Mu3
10 Clostridium beijerinckii NCIMB 8052 chromosome
30 Staphylococcus aureus subsp. aureus Mu50
11 Clostridium botulinum A3 str. Loch Maree
31 Staphylococcus aureus subsp. aureus MW2
12 Clostridium botulinum A str. ATCC 19397
19 Lanjutan
No Nama organisme No Nama organisme
13 Clostridium botulinum B1 str. Okra 33 Streptococcus agalactiae 2603V/R 14 Clostridium botulinum B str. Eklund
17B
34 Streptococcus agalactiae A909 15 Clostridium botulinum E3 str. Alaska
E43
35 Streptococcus agalactiae NEM316 16 Clostridium botulinum F str. Langeland 36 Streptococcus equi subsp.
zooepidemicus MGCS10565
17 Mycobacterium abscessus chromosome 37 Streptococcus pneumoniae CGSP14 18 Mycobacterium avium 104 38 Streptococcus pneumoniae D39 19 Mycobacterium avium subsp.
paratuberculosis K-10
39 Streptococcus pneumoniae G54
20 Mycobacterium bovis AF2122/97 40 Streptococcus pneumoniae Hungary19A-6
Data uji
No Nama organisme No Nama organisme
1 Burkholderia cenocepacia J2315 chromosome chromosome 1
6 Mycobacterium gilvum PYR-GCK chromosome
2 Burkholderia cenocepacia MC0-3 chromosome chromosome 1
7 Staphylococcus aureus subsp. aureus MRSA252 chromosome
3 Clostridium botulinum A str. ATCC 3502
8 Staphylococcus aureus subsp. aureus MSSA476 chromosome
4 Clostridium botulinum A str. Hall 9 Streptococcus gordonii str. Challis substr. CH1
5 Mycobacterium bovis BCG str. Pasteur 1173P2
10 Streptococcus mutans UA159 chromosome
Percobaan 4 Data latih
No Nama organisme No Nama organisme
1 Burkholderia ambifaria AMMD chromosome chromosome 1
21 Mycobacterium bovis BCG str. Pasteur 1173P2
2 Burkholderia ambifaria MC40-6 chromosome chromosome 1
22 Mycobacterium gilvum PYR-GCK chromosome
3 Burkholderia cenocepacia AU 1054 chromosome 3
23 Mycobacterium smegmatis str. MC2 155
4 Burkholderia cenocepacia HI2424 chromosome chromosome 1
24 Mycobacterium sp. JLS chromosome 5 Burkholderia cenocepacia J2315
chromosome chromosome 1
25 Staphylococcus aureus RF122 6 Burkholderia cenocepacia MC0-3
chromosome chromosome 1
26 Staphylococcus aureus subsp. aureus COL chromosome
7 Burkholderia mallei NCTC 10247 chromosome I
20 Lanjutan
No Nama organisme No Nama organisme
8 Burkholderia mallei SAVP1 chromosome I
28 Staphylococcus aureus subsp. aureus JH9
9 Clostridium acetobutylicum ATCC 824 29 Staphylococcus aureus subsp. aureus MRSA252 chromosome 10 Clostridium beijerinckii NCIMB 8052
chromosome
30 Staphylococcus aureus subsp. aureus MSSA476 chromosome 11 Clostridium botulinum A3 str. Loch
Maree
31 Staphylococcus aureus subsp. aureus MW2
12 Clostridium botulinum A str. ATCC 19397
32 Staphylococcus aureus subsp. aureus N315
13 Clostridium botulinum A str. ATCC 3502
33 Streptococcus agalactiae 2603V/R 14 Clostridium botulinum A str. Hall 34 Streptococcus agalactiae A909 15 Clostridium botulinum E3 str. Alaska
E43
35 Streptococcus agalactiae NEM316 16 Clostridium botulinum F str. Langeland 36 Streptococcus equi subsp.
zooepidemicus MGCS10565
17 Mycobacterium abscessus chromosome 37 Streptococcus gordonii str. Challis substr. CH1
18 Mycobacterium avium 104 38 Streptococcus mutans UA159 chromosome
19 Mycobacterium avium subsp. paratuberculosis K-10
39 Streptococcus pneumoniae G54 20 Mycobacterium bovis AF2122/97 40 Streptococcus pneumoniae
Hungary19A-6 Data uji
No Nama organisme No Nama organisme
1 Burkholderia mallei ATCC 23344 chromosome chromosome 1
6 Mycobacterium marinum M 2 Burkholderia mallei NCTC 10229
chromosome I
7 Staphylococcus aureus subsp. aureus Mu3
3 Clostridium botulinum B1 str. Okra 8 Staphylococcus aureus subsp. aureus Mu50
4 Clostridium botulinum B str. Eklund 17B
9 Streptococcus pneumoniae CGSP14
5 Mycobacterium leprae TN chromosome 10 Streptococcus pneumoniae D39 Percobaan 5
Data latih
No Nama organisme No Nama organisme
1 Burkholderia ambifaria AMMD chromosome chromosome 1
21 Mycobacterium bovis BCG str. Pasteur 1173P2
2 Burkholderia ambifaria MC40-6 chromosome chromosome 1
21 Lanjutan
No Nama organisme No Nama organisme
3 Burkholderia cenocepacia AU 1054 chromosome 3
23 Mycobacterium leprae TN chromosome
4 Burkholderia cenocepacia HI2424 chromosome chromosome 1
24 Mycobacterium marinum M 5 Burkholderia cenocepacia J2315
chromosome chromosome 1
25 Staphylococcus aureus RF122 6 Burkholderia cenocepacia MC0-3
chromosome chromosome 1
26 Staphylococcus aureus subsp. aureus COL chromosome
7 Burkholderia mallei ATCC 23344 chromosome chromosome 1
27 Staphylococcus aureus subsp. aureus JH1
8 Burkholderia mallei NCTC 10229 chromosome I
28 Staphylococcus aureus subsp. aureus JH9
9 Clostridium acetobutylicum ATCC 824 29 Staphylococcus aureus subsp. aureus MRSA252 chromosome
10 Clostridium beijerinckii NCIMB 8052 chromosome
30 Staphylococcus aureus subsp. aureus MSSA476 chromosome
11 Clostridium botulinum A3 str. Loch Maree
31 Staphylococcus aureus subsp. aureus Mu3
12 Clostridium botulinum A str. ATCC 19397
32 Staphylococcus aureus subsp. aureus Mu50
13 Clostridium botulinum A str. ATCC 3502
33 Streptococcus agalactiae 2603V/R 14 Clostridium botulinum A str. Hall 34 Streptococcus agalactiae A909 15 Clostridium botulinum B1 str. Okra 35 Streptococcus agalactiae NEM316 16 Clostridium botulinum B str. Eklund
17B
36 Streptococcus equi subsp. zooepidemicus MGCS10565
17 Mycobacterium abscessus chromosome 37 Streptococcus gordonii str. Challis substr. CH1
18 Mycobacterium avium 104 38 Streptococcus mutans UA159 chromosome
19 Mycobacterium avium subsp. paratuberculosis K-10
39 Streptococcus pneumoniae CGSP14 20 Mycobacterium bovis AF2122/97 40 Streptococcus pneumoniae D39
Data uji
No Nama organisme No Nama organisme
1 Burkholderia mallei NCTC 10247 chromosome I
6 Mycobacterium sp. JLS chromosome 2 Burkholderia mallei SAVP1
chromosome I
7 Staphylococcus aureus subsp. aureus MW2
3 Clostridium botulinum E3 str. Alaska E43
8 Staphylococcus aureus subsp. aureus N315
4 Clostridium botulinum F str. Langeland 9 Streptococcus pneumoniae G54 5 Mycobacterium smegmatis str. MC2
155
22
Lampiran 4 Daftar boxplot semua fitur dengan panjang 200 bp
Boxplot fitur Contrast
Boxplot fitur Correlation
23 Lanjutan
Boxplot fitur Inverse Different Moment
Boxplot fitur Entropy
24
Lanjutan
Boxplot fitur Sum Variance
Boxplot fitur Difference Variance
25 Lanjutan
Boxplot fitur Inverse Measures of Correlation 1
Boxplot fitur Inverse Measures of Correlation 2
26
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta pada tanggal 29 April 1992. Penulis merupakan anak kedua dari tiga bersaudara pasangan Ir. Indrayana LS dan (alm) Ir. Alda Djumeralda. Penulis mengenyam pendidikan dasar di SD Negeri 08 Bengkulu (1998-2002) yang kemudian pindah ke SD Islam Al-Azhar 23 Bekasi (2002-2004). Penulis melanjutkan pendidikan menengah pertama di SMP Labschool Jakarta (2004-2007). Kemudian, penulis melanjutkan pendidikan menengah atas di SMA Labschool Jakarta (2007-2010). Penulis berkesempatan melanjutkan studi di Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB (USMI) di Departemen Ilmu Komputer, Fakultas Matematika dan Ilmu Pengetahuan Alam.