Analisis Cemaran Daging Babi
Pada Produk Bakso Sapi
Yang Beredar Di Wilayah Ciputat Menggunakan
Real-Time Polymerase Chain Reaction
(PCR) dengan Metode
Hydrolysis
Probe
SKRIPSI
Diajukan sebagai salah satu syarat untuk memperoleh gelar Sarjana Farmasi
SULAIMAN RASYID
NIM : 108102000040
FAKULTAS KEDOKTERAN DAN ILMU KESEHATAN
PROGRAM STUDI FARMASI
v
Nama : Sulaiman Rasyid
Program Studi : Farmasi
Judul : Analisis Cemaran Daging Babi pada Produk Bakso Sapi yang Beredar di Wilayah Ciputat Menggunakan Real-Time Polymerase Chain Reaction (PCR) dengan Metode Hydrolysis Probe.
Harga daging sapi semakin meningkat dari waktu ke waktu. Faktor tersebut mendorong produsen yang tidak bertanggungjawab untuk mencampur daging sapi
dengan daging babi demi mendapat keuntungan yang besar. Padahal Islam melarang umatnya untuk mengkonsumsi babi walau hanya sedikit. Metode yang
handal, efisien dan terpercaya diperlukan untuk dapat mendeteksi cemaran daging babi pada produk daging olahan. Penelitian ini menganalisis cemaran babi menggunakan Real-Time PCR dengan Metode Hydrolysis Probe yang memiliki keunggulan dari segi spesifisitas DNA yang di amplifikasi. Isolat DNA dari 2 kontrol positif, 2 kontrol negatif dan 8 sampel didapatkan menggunakan kit komersial dengan hasil konsentrasi dan kemurnian yang baik. Amplifikasi dilakukan menggunakan primer spesifik sapi dan babi yang didisain pada daerah DNA mitokondria sitokrom b dengan amplikon 120 bp dan 131 bp. Suhu
annealing yang ditetapkan pada sapi adalah 61oC dan babi 60oC. Kurva amplifikasi menggunakan primer spesifik babi menunjukkan bahwa kontrol positif teramplifikasi dengan nilai CP 16,74 dan 30,37 sedangkan kontrol negatif dan semua sampel tidak terdeteksi.
vi
Name : Sulaiman Rasyid
Major : Pharmacy
Title : Analysis of Pork Contamination in Beef Meat Ball which are available throughout Ciputat area Using Real Time Polymerase Chain Reaction (PCR) with Hydrolysis Probe Method.
The price of beef is increasing from time to time. Because of this factor, The
producer who doesn’t have responsibility is unfairly mixing beef with pork to get huge profit. Though in religion of Islam forbid muslims to consume pork even
only slightly. The reliable, efficient and trusted method are required to detect pork contamination in processed meat products. This study was to analyzed the pork contamination using Real Time PCR with Hydrolysis Probe method. The advantage of this method is DNA amplification specificity. DNA was isolated from Two positive control, Two negative control and 8 sample. The isolate of DNA was obtained using a commercial kit with a good result of concentration and purity. Amplification was performed using cattle and pork specific primers. The primers was designed in the area of Cytochrome b mithocondrial DNA with amplicon of 120 bp and 131 bp. The annealing temperature specified in cattle and pork consecutively is 61oC and 60oC. The amplification curve using specific pork primers showed the positive control amplified with yield of CP is 16,74 and 30,37 while the negative control and all of the samples were not detected.
vii
KATA PENGANTAR
Segala puji dan syukur kehadirat Allah SWT yang senantiasa meberikan jalan keluar bagi hamba Nya yang meminta jalan keluar. Atas rahmat Nya skripsi yang berjudul “Analisis Cemaran Daging Babi pada Produk Bakso Sapi yang
Beredar di Wilayah Ciputat Menggunakan Real-Time Polymerase Chain Reaction (PCR) dengan metode Hydrolysis Probe” ini berhasil penulis selesaikan. Sholawat beriring salam semoga selalu tercurah limpah kepada baginda Rasulullah SAW, manusia terbaik sepanjang zaman, teladan umat manusia menjalani kehidupan.
Skripsi ini disusun untuk memenuhi salah satu syarat dalam ujian akhir guna
mendapatkan gelar Sarjana Farmasi (S.Far) pada Program Studi Farmasi, Fakultas Kedokteran dan Ilmu Kesehatan, Universitas Islam Negeri (UIN) Syarif
Hidayatullah Jakarta. Rampungnya penelitian dan penulisan skripsi ini pastilah atas bantuan berbagai pihak, dalam kesempatan ini penulis ingin menyampaikan ucapan terimakasih yang sedalam-dalamnya kepada :
1. Bapak Dr. H. Arif Sumantri, M.Kes. selaku Dekan Fakultas Kedokteran dan Ilmu Kesehatan (FKIK) UIN Syarif Hidayatullah Jakarta.
viii
menempuh pendidikan farmasi di FKIK UIN Syarif Hidayatullah Jakarta. 6. Teman-teman angkatan Betalaktam yang telah menjadi memori berkesan
selama penulis menuntut ilmu.
7. Kak Rahmadi, Kak Rani, Kak Tiwi, Kak Eris, Kak Lisna, Kak Lilis dan Kak Ai yang telah membantu penulis dalam melakukan penelitian di laboratorium. 8. Teman-teman seperjuangan tim PCR dari angkatan ke angkatan terutama
Afifah dan Yanti juga Putri Rahmawati, Dienar Fitri dan adik-adikku Rian Hidayat dan Fathiya, Semoga bekal yang didapat bermanfaat untuk kehidupan kita di masa depan.
9. Tim Roche Indonesia: Pak Deka, Pak Yos, Ibu Helen yang membantu penulis dalam penyelesaian terkait RT-PCR.
10.Iyus, Rian, Suparman, Dendi dan Ali serta saudara-saudaraku dalam lingkaran surga, semoga kebersamaan kita tetap di eratkan hingga kaki kita benar-benar
telah berada di surga Nya.
11.Sahabat-sahabat seperjuangan di jalan dakwah yang senantiasa saling mendoakan, siapapun dan dimanapun kalian berada, semoga kita istiqomah dan tetap dalam keyakinan menjadi umat terbaik di bumi ini.
Semoga semua bantuan yang telah diberikan mendapat ganjaran terbaik di sisi Allah SWT. Sekali lagi mohon maaf jikalau penulis hanya bisa memberikan ucapan terimakasih yang sedalam-dalamnya. Semoga skripsi ini bisa bermanfaat bagi pengembangan ilmu pengetahuan dan menjadi amal jariah bagi penulisnya. Aamiin.
Jakarta, 24 Juni 2015
ix
Sebagai sivitas akademika Universitas Islam Negeri (UIN) Syarif Hidayatullah Jakarta, saya yang bertanda tangan di bawah ini :
Nama : Sulaiman Rasyid
NIM : 108102000040
Program Studi : Strata-1 Farmasi
Fakultas : Kedokteran dan Ilmu Kesehatan Jenis Karya : Skripsi
Demi pengembangan ilmu pengetahuan, saya menyetujui skripsi/karya ilmiah
saya dengan judul :
Analisis Cemaran Daging Babi pada Produk Bakso Sapi yang Beredar di Wilayah Ciputat Menggunakan Real-Time Polymerase Chain Reaction (PCR)
dengan Metode Hydrolysis Probe
untuk dipublikasikan atau ditampilkan di internet atau media lain yaitu Digital Library Perpustakaan Universitas Islam Negeri (UIN) Syarif Hidayatullah Jakarta untuk kepentingan akademik sebatas sesuai dengan Undang-Undang Hak Cipta. Demikian pernyataan publikasi karya ilmiah ini saya buat dengan sebenarnya.
Dibuat di : Jakarta Pada tanggal : 24 Juni 2015
Yang Menyatakan,
x
2.2 Babi dan Keharamannya ... 5
2.3 Sel ... 6
2.4 Asam Nukleat dan Protein ... 8
2.5 DNA ... 9
2.5.1 Pengertian DNA ... 9
2.5.2 DNA Mitokondria ... 12
xi
2.6.2 Tahapan PCR... 20
2.7 Real-Time PCR... 22
2.7.1 Prinsip Analisis ... 22
2.7.2 Analisis menggunakan metode Hydrolysis Probe ... 24
BAB 3 METODOLOGI PENELITIAN ... 27
3.1 Tempat dan Waktu Penelitian ... 27
3.1.1 Tempat ... 27
3.4.2.2 Proses Melisiskan Sampel dan Mengikat DNA ... 29
3.4.2.3 Proses Pemurnian dan Elusi DNA ... 30
3.4.3 Analisis Hasil Isolasi DNA dengan Spektrofotometri UV untuk DNA ... 31
3.4.4 Uji Spesifisitas Primer dan Probe dengan metode BLAST menggunakan database NCBI ... 32
3.4.5 Amplifikasi DNA menggunakan Real-Time PCR dengan metode Hydrolysis probe ... 32
BAB 4 HASIL DAN PEMBAHASAN ... 34
4.1 Hasil Analisis Isolat DNA dengan Spektrofotometer UV ... 34
xii
4.3.2 Hasil Amplifikasi DNA menggunakan Real-Time PCR dengan
metode Hydrolysis Probe menggunakan Primer Sapi... 39
4.3.2.1 Hasil Analisis Menggunakan Metode Analisis Second Derivative Maximum ... 41
4.3.2.2 Hasil Analisis Menggunakan Metode Analisis Fit Point ... 42
4.3.2.3 Perbandingan antara Analisis Second Derivative Maximum dan Fit Point pada Primer Sapi ... 44
4.3.3 Hasil Amplifikasi DNA menggunakan Real-Time PCR dengan metode Hydrolysis Probe menggunakan Primer Babi ... 45
4.3.3.1 Hasil Analisis Menggunakan Metode Analisis Second Derivative Maximum ... 46
4.3.3.2 Hasil Analisis Menggunakan Metode Analisis Fit Point ... 47
4.3.3.3 Perbandingan antara Analisis Second Derivative Maximum dan Fit Point pada primer sapi... 48
4.3.4 Perbandingan antara metode analisis baik Second Derivative Maximum maupun Fit Point pada sampel dengan primer sapi dan primer babi ... 49
BAB 5 KESIMPULAN DAN SARAN ... 50
5.1 Kesimpulan ... 50
5.2 Saran ... 50
DAFTAR PUSTAKA ... 51
xiii
Gambar 2.1 Sel Prokariotik dan Eukariotik ... 6
Gambar 2.2 Asam Nukleat: DNA dan RNA ... 8
Gambar 2.3 Struktur Nukleotida ... 9
Gambar 2.4 Struktur Basa Nitrogen Purin dan Pirimidin ... 10
Gambar 2.5 Struktur double helix DNA ... 11
Gambar 2.6 Susunan dan Replikasi DNA ... 12
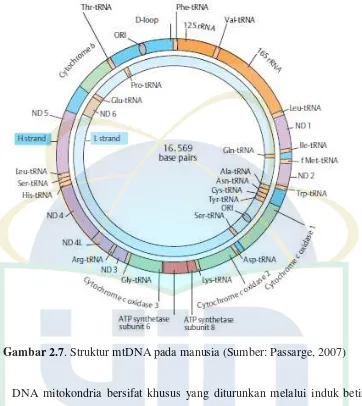
Gambar 2.7 Struktur mtDNA pada Manusia ... 14
Gambar 2.8 Simulasi Proses PCR ... 17
Gambar 2.9 Siklus PCR ... 20
Gambar 2.10 Bentuk Kurva pada Real-Time PCR ... 23
Gambar 4.1 Hasil Uji spesifisitas primer dan probe sapi dengan program BLAST pada laman NCBI ... 37
Gambar 4.2 Hasil Uji spesifisitas primer dan probe babi dengan program BLAST pada laman NCBI ... 38
Gambar 4.3 Kurva Amplifikasi RT-PCR menggunakan Primer Sapi ... 39
xiv
Tabel 1. Perbandingan sel eukariotik dan prokariotik ... 7 Tabel 2. Tahapan Proses Fluoresensi Hydrolysis Probe ... 25 Tabel 3. Susunan basa primer dan probe untuk DNA sapi dan babi (Tanabe
et a.l, 2007) ... 28 Tabel 4. Program amplifikasi Real-Time PCR (Rochec, 2008) ... 33 Tabel 5. Konsentrasi dan kemurnian DNA hasil isolasi ... 34 Tabel 6. Nilai CP Menggunakan Metode analisis Second Derivative
Maximum pada sampel dengan primer sapi ... 41 Tabel 7. Nilai CP Menggunakan Metode analisis Fit Point dengan variasi
nilai treshold pada sampel dengan primer sapi ... 42 Tabel 8. Perbandingan Nilai CP Menggunakan Metode analisis Second
Derivative Maximum dan Fit Point pada treshold 0,75 dengan primer sapi ... 43
Tabel 9. Nilai CP Menggunakan Metode analisis Second Derivative Maximum pada sampel dengan primer babi ... 45 Tabel 10. Nilai CP Menggunakan Metode analisis Fit Point dengan variasi
nilai treshold pada sampel dengan primer sapi ... 46 Tabel 11a. Perbandingan Nilai CP Menggunakan Metode analisis Second
Derivative Maximum dan Fit Point pada treshold 0,914 dengan primer babi ... 47 Tabel 11b. Perbandingan Nilai CP Menggunakan Metode analisis Second
Derivative Maximum dan Fit Point pada treshold 1,733 dengan primer babi ... 47 Tabel 12. Perbandingan Nilai CP Menggunakan Metode analisis Second
Derivative Maximum pada primer Sapi dan Babi ... 48 Tabel 13a. Perbandingan Nilai CP Menggunakan Metode analisis Fit Point
dengan treshold 0,9 pada primer Sapi dan Babi ... 48 Tabel 13b. Perbandingan Nilai CP Menggunakan Metode analisis Fit Point
xv
Lampiran 1. Alur Penelitian ... 53
Lampiran 2. Spesifikasi Kit isolasi DNA High Pure PCR Template Preparation ... 54
Lampiran 3. Alur kerja Isolasi DNA menggunakan kit High Pure PCR Template Preparation ... 55
Lampiran 4a. Membuat larutan induk primer dan probe ... 56
Lampiran 4b. Campuran reaksi master mix untuk amplifikasi DNA... 57
Lampiran 5. Perhitungan Tm (Melting Temperature) primer ... 57
Lampiran 6. Hasil Optimasi Suhu Annealing Primer Tanabe et al. dengan Metode Gradien PCR (Rahmawati, 2012) dengan modifikasi ... 58
Lampiran 7. Hasil Kurva Amplifikasi Primer Sapi setelah modifikasi treshold dalam perhitungan nilai CP dengan nilai treshold 0,5617 ... 59
Lampiran 8. Hasil Kurva Amplifikasi Primer Sapi setelah modifikasi treshold dalam perhitungan nilai CP dengan nilai treshold 0,7500 ... 60
Lampiran 9. Hasil Kurva Amplifikasi Primer Sapi setelah modifikasi treshold dalam perhitungan nilai CP dengan nilai treshold 0,9000 ... 61
Lampiran 10. Hasil Kurva Amplifikasi Primer Sapi setelah modifikasi treshold dalam perhitungan nilai CP dengan nilai treshold 1,3300 ... 62
Lampiran 11. Hasil Kurva Amplifikasi Primer Babi setelah modifikasi treshold dalam perhitungan nilai CP dengan nilai treshold 0,9000 ... 63
Lampiran 12. Hasil Kurva Amplifikasi Primer Babi setelah modifikasi treshold dalam perhitungan nilai CP dengan nilai treshold 0,9400 ... 64
Lampiran 13. Hasil Kurva Amplifikasi Primer Babi setelah modifikasi treshold dalam perhitungan nilai CP dengan nilai treshold 1,3300 ... 65
Lampiran 14. Hasil Kurva Amplifikasi Primer Babi setelah modifikasi treshold dalam perhitungan nilai CP dengan nilai treshold 1,7330 ... 66
xvi BHQ-1 : Black Hole Quencher-1
BLAST : Basic Logical Aligment Search Tool
bp : Base pair
CP : Crossing Point
cyt b : Cytochrome b
dATP : Deoxyadenosine Triphosphate
dCTP : Deoxycytidine Triphosphate
dGTP : Deoxyguanosine Triphosphate
dNTP : Deoxyribonuleaside Triphosphate
dTTP : Deoxythymidine Triphosphate
DNA : Deoxyribonucleic Acid
FAM : Fluorescein Amidite
mtDNA : mitochondrial DNA
NCBI : National Center for Biotechnology Information
NTC : No Template Control
PCR : Polymerase Chain Reaction
qPCR : Quantitative Polymerase Chain Reaction
RE : Retikulum Endoplasma RNA : Ribonucleic Acid
Tm : Temperature Melting
xvii
Maximum
BLAST : Basic Logical Aligment Search Tool merupakan program untuk menganalisis kesejajaran urutan basa query (DNA atau protein) dengan urutan basa DNA atau protein dari
database NCBI
Blastn : Nucleotide Blast atau biasa disebut blastn merupakan salah satu fasilitas dari program BLAST untuk menganalisis kesejajaran nukleotida yang dimasukkan pada query dengan nukleotida pada database NCBI
CP : Crossing Point merupakan angka siklus yang menunjukkan
awal permulaan akumulasi amplikon telah memasuki peningkatan log-linear
Fit Point : Metode penentuan CP melalui pertemuan antara garis
treshold dengan kurva amplifikasi
Garis Treshold : Garis horizontal penanda siklus awal dari reaksi PCR yaitu saat sinyal fluorescent berada pada titik terendah
NCBI : National Center for Biotechnology Information merupakan suatu institusi yang dimiliki United States National Library of Medicine yang berperan sebagai sumber informasi perkembangan biologi molekuler. Dari situs NCBI dapat diakses database bioteknologi meliputi genebank, urutan basa DNA atau protein juga publikasi-publikasi ilmiah.
Second Derivative : Metode penentuan CP dalam instrumen LightCycler 480 dimana CP diperoleh berdasarkan saat kurva amplifikasi mengalami kenaikan yang tajam
Query : Urutan basa yang dimasukkan ke dalam program BLAST untuk diketahui kesejajarannya dengan data yang tersedia Tm : Temperature Melting atau suhu lebur adalah suhu saat
1 UIN Syarif Hidayatullah Jakarta
1.1 Latar Belakang
Kehalalan suatu makanan yang akan dikonsumsi adalah suatu syarat ketetapan Agama Islam yang wajib dijalankan oleh umatnya. Suatu makanan dapat dikatakan halal apabila tidak dilarang oleh nash-nash agama. Makanan yang halal bisa menjadi haram apabila makanan tersebut tidak baik untuk dikonsumsi. Babi merupakan hewan yang secara keseluruhan diharamkan
untuk dikonsumsi oleh umat Islam (Q.S Al-Baqarah : 173, Al-Ma’idah : 3, Al-An’am : 145 dan An-Nahl : 115). Babi diketahui sebagai inang dari banyak macam parasit dan penyakit berbahaya. Sistem biokimia babi mengeluarkan hanya 2% dari seluruh kandungan asam uratnya, sedangkan 98% sisanya tersimpan dalam tubuhnya (Wijaya, 2009). Wajib bagi pemerintah Indonesia yang mayoritas masyarakatnya beragama Islam untuk memperhatikan kehalalan makanannya dari campuran daging babi.
Dalam era perdagangan global, dimungkinkan terjadinya impor bahan makanan dalam bentuk olahan atau mentah dari negara lain ke Indonesia tanpa melalui pengujian yang mendalam. MUI (Majelis Ulama Indonesia) selaku perkumpulan ahli ilmu agama Islam tertinggi di negeri ini melalui LPPOM (Lembaga Pusat Pengkajian Obat dan Makanan) telah melakukan sertifikasi halal terhadap produk-produk yang ber edar termasuk produk pangan daging. Namun masih ditemukan beberapa kasus pencampuran daging babi pada produk daging sapi olahan. Tujuan pencampuran tersebut untuk menghasilkan produk akhir dengan harga yang relatif lebih murah dibandingkan jika menggunakan bahan aslinya, mengingat harga daging sapi terus meningkat (Margawati dan Ridwan, 2010).
UIN Syarif Hidayatullah Jakarta
Pada akhir tahun 2007 di kota Jambi ditemukan kandungan daging babi dalam bakso sapi, pada Mei 2010 kasus yang sama juga kembali terulang (jambi-independent.co.id). Selain beredar di Kota Jambi, produk bakso berlabelkan bakso sapi yang mengandung babi juga membuat gegar kota Palembang pada bulan maret di tahun yang sama (okezone.com). Akibat adanya kasus bakso babi di tahun 2007 menyebabkan 80% pedagang bakso eceran bangkrut. Kejadian tersebut belum membuat pelanggar hukum jera. Semakin dekat, kasus kembali terjadi pada bulan Desember 2012 di Pasar Cipete, Jakarta Selatan. Ditemukan sebuah kios penggilingan yang menjual bakso yang di campur daging babi (detik.com). Bulan April tahun lalu masyarakat Jakarta kembali dikejutkan dengan terbongkarnya bakso oplosan babi di Tambora, Jakarta Barat (sindonews.com). Yang cukup hangat pada 12 Februari kasus yang sama terjadi di buah batu, Bandung (Jpnn.com) dan
paling terbaru akhir Maret lalu kota Sukabumi mengalami kejadian serupa (antaranews.com). Hal tersebut tentunya sangat meresahkan penduduk daerah
tersebut dan juga penduduk Indonesia yang sebagian besar adalah muslim. Hingga hari ini teknologi biologi molekuler terus mengalami perkembangan dan kemajuan yang pesat. Teknologi tersebut telah dapat diaplikasikan dan mempermudah pengujian akan adanya kontaminasi bahan lain diluar bahan aslinya. Pengujian cemaran daging babi dalam berbagai produk daging olahan seperti daging bakso dapat dideteksi melalui amplifikasi PCR. Margawati dan Ridwan (2010) telah melakukan pengujian pencemaran campuran daging babi pada produk bakso. Sebelumnya Calvo et al, (2001) melakukan identifikasi daging babi pada produk makanan olahan dan mentah melalui amplifikasi PCR. Pada tahun 2008 Alaraidh juga berhasil melakukan isolasi DNA dan amplifikasi PCR dari sampel daging yang terkontaminasi daging babi di pasar Arab Saudi. Sistem TaqMan Real-Time PCR dengan probe Minor Groove Binding (MGB) juga telah digunakan pada pendeteksian kuantifikasi DNA sapi, babi, domba, ayam, kalkun dan burung onta pada sampel yang kompleks (Lopez-Andreo et al,2005).
UIN Syarif Hidayatullah Jakarta
yang identik. DNA merupakan molekul yang stabil dalam proses ekstraksi, dan analisis DNA sangat mungkin dikerjakan dari beberapa tipe sampel yang berbeda (Jain, 2004). Dengan demikian upaya mendeteksi adanya campuran daging babi dalam produk daging sapi olahan dapat dilakukan.
Tujuan dari penelitian ini adalah untuk menganalisis ada atau tidaknya kandungan daging babi pada produk daging sapi olahan. Sampel yang digunakan pada penelitian ini adalah bakso sapi. Pengujian dilakukan melalui amplifikasi DNA menggunakan Real-Time PCR. Real-Time PCR merupakan metode terkini untuk amplifikasi PCR. Pada Real-Time PCR jumlah DNA yang diamplifikasi bisa langsung diamati secara real-time sehingga tidak memerlukan analisis dengan elektroforesis gel untuk mengetahui produk PCR. Real-Time PCR lebih dikenal sebagai quantitative PCR karena kemampuan analisisnya yang sensitif dan spesifik sehingga mengurangi
kesalahan pada hasil (Burns et al, 2005).
1.2 Rumusan Masalah
1. Bagaimana kondisi optimal amplifikasi DNA pada bakso menggunakan primer spesifik babi dan sapi dengan Real-Time PCR dengan metode
Hydrolysis Probe?
2. Apakah bakso sapi yang beredar di wilayah Ciputat tercemar daging babi?
1.3 Hipotesis
1. Real-Time PCR dapat mengamplifikasi DNA menggunakan primer spesifik babi dan sapi dengan metode Hydrolysis Probe.
UIN Syarif Hidayatullah Jakarta
1.4 Tujuan Penelitian
Mendeteksi keberadaan kandungan babi dalam bakso sapi yang dijual di
wilayah Ciputat melalui amplifikasi DNA menggunakan Real-Time PCR.
1.5 Manfaat Penelitian
Manfaat dari penelitian ini adalah memberikan informasi kepada masyarakat tentang keamanan dan kehalalan produk bakso sapi yang beredar di wilayah Ciputat. Hal ini dilakukan sebagai dharma UIN terhadap masyarakat sekitar, sehingga masyarakat lebih berhati-hati dan bijaksana
UIN Syarif Hidayatullah Jakarta
BAB II
TINJAUAN PUSTAKA
2.1 Bakso
Bakso adalah makanan terbuat dari daging, udang, ikan yang dicincang dan dilumatkan bersama tepung kanji, biasanya bulat-bulat (KBBI, 2008) Bakso daging menurut SNI No: 01-3818-1995 adalah produk makanan berbentuk bulatan atau lain yang diperoleh dari campuran daging ternak (kadar daging tidak kurang dari 50 persen) dan pati atau serealia dengan atau
tanpa bumbu BTP (bahan tambahan pangan) yang diizinkan. Pembuatan bakso biasanya menggunakan daging yang segar. Daging segar (pre-rigor) adalah daging yang diperoleh setelah pemotongan hewan tanpa mengalami proses pendinginan terlebih dahulu. Fase pre-rigor berlangsung selama 5 sampai 8 jam setelah postmortem. Bakso dapat dikelompokkan menurut jenis daging yang digunakan dan berdasarkan perbandingan jumlah tepung pati yang digunakan. Berdasarkan jenis daging sebagai bahan baku untuk membuat bakso, maka dikenal bakso sapi, bakso ayam, bakso ikan, bakso kerbau, dan bakso kelinci (Gaffar, 1998 dalam Saddam 2013).
2.2 Babi dan Keharamannya
UIN Syarif Hidayatullah Jakarta
suis type 2 (group R), Swine vesicular disease, Taenia solum, Trichinella spiralis, Yersinia enterocolitica, Yersinia pseudotuberculosis (Wijaya, 2009).
Sebagai muslim yang taat dan menjadikan kitab suci Al-Qur’an sebagai pedoman hidup, dilarang menganiaya diri sendiri termasuk mengkonsumsi sesuatu yang membahayakan bagi kita. Apalagi terkait pengharaman daging babi secara jelas difirmankan oleh Allah kepada Rasulullah “Katakanlah: "Tidak kudapati di dalam apa yang diwahyukan kepadaku, sesuatu yang diharamkan memakannya bagi yang ingin memakannya, kecuali daging hewan yang mati (bangkai), darah yang mengalir, daging babi - karena semua itu kotor - atau hewan yang disembelih bukan atas (nama) Allah. Tetapi barangsiapa terpaksa bukan karena menginginkan dan tidak melebihi (batas darurat) maka sungguh, Tuhan-mu Maha Pengampun, Maha Penyayang." (QS.Al-An’am:145)
2.3 Sel
Sel adalah komponen dasar dan unit terkecil dari kehidupan. Organisme pertama yang ada semenjak 1 milyar tahun yang lalu terdiri dari sel tunggal. Organisme sederhana yang ada terdiri dari hanya 1 sel. Satu karakteristik kunci organisme yang hidup adalah dapat mereplikasi atau mereproduksi dirinya. Banyak organisme sel tunggal yang bereproduksi dengan membelah
diri menjadi 2 salinan baru yang identik dari dirinya. Sebaliknya, organisme multiseluler bereproduksi dengan cara yang bervariasi. (Cain, 2002)
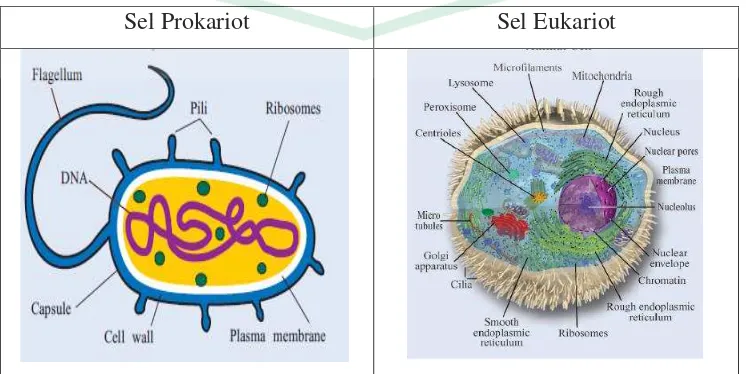
Gambar 2.1. Sel Prokariotik dan Eukariotik (Sumber: Brooks, 2003)
UIN Syarif Hidayatullah Jakarta
Ketika organisme menghasilkan benih, bertelur, atau membelah diri sekalipun, semua bereproduksi menggunakan molekul yang dikenal dengan sebutan DNA (deoxyribonucleic Acid). (Cain, 2002)
Organisme yang hidup saat ini dibagi dalam dua kelompok besar, yaitu
prokariot dan eukariot. Berikut adalah perbandingan antara prokariot dan
eukariot menurut Koolman et al, (2005) dalam bukunya yang berjudul ”Atlas Berwarna dan Teks Biokimia”
Tabel 1. Perbandingan sel eukariotik dan prokariotik (Koolman et al, 2005)
Prokariotik Eukariotik
Bersel satu Bersel satu atau banyak
Organel, sitoskelet, alat pembelahan sel :
Tidak Ada Ada, rumit dan terspesialisasi
DNA Kecil, sirkular, tidak ada intron,
terdiri dari plasmid-plasmid Besar, dalam inti sel, banyak intron
RNA: Sintesis dan Pematangan
Sederhana, didalam sitoplasma Rumit, didalam inti sel
Protein: Sintesis dan Pematangan
mampu menyesuaikan diri Kebanyakan aerobik
Endositosis dan Eksositosis
UIN Syarif Hidayatullah Jakarta
2.4 Asam Nukleat dan Protein
Asam nukleat dan protein merupakan senyawa polimer utama yang terdapat
pada sel. Asam nukleat berfungsi menyimpan dan mentransmisikan informasi
genetik dalam sel (Gaffar, 2007). Protein seperti asam nukleat, juga berupa rantai dari unit kecil polimer. Protein terdiri dari rantai asam amino, terdapat 20 jenis asam amino yang berbeda pada protein. Asam amino tergabung bersama di dalam protein lewat ikatan peptida sehingga disebut polipeptida. Protein terdiri dari satu atau lebih polipeptida. Kebanyakan protein berfungsi untuk menjaga dan memberikan bentuk pada sel. Selain itu protein juga berperan sebagai hormon yang mengirimkan sinyal antar sel. Protein bertindak sebagai enzim yang menjadi katalis ratusan reaksi yang diperlukan
dalam kehidupan (Weaver, 2001)
Sel mempunyai dua jenis molekul asam nukleat yaitu asam
deoksiribonukleat (DNA) yang berfungsi sebagai penyimpan informasi. Dan asam ribonukleat (RNA) berperan sebagai ekspresi gen dan bio-sintesis protein. Semua asam nukleat dibentuk dari komponen-komponen nukleotida
yang terdiri atas satu basa, satu gula dan satu residu fosfat. DNA dan RNA dapat dibedakan dari jenis gulanya dan pada salah satu dari basanya (Koolman et al, 2005).
UIN Syarif Hidayatullah Jakarta
2.5 DNA
2.5.1 Pengertian DNA
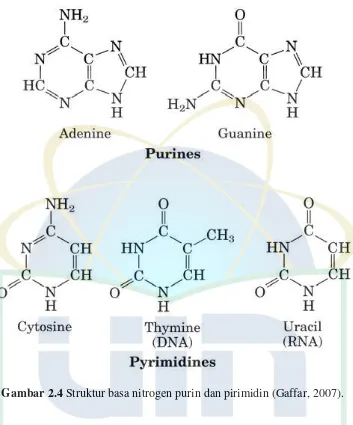
DNA dan RNA merupakan polimer linier (polinukleotida) yang tersusun dari subunit atau monomer nukleotida. Pada awal 1930-an, Levene, W. Jacobs mengemukakan bahwa RNA terdiri dari gula ribosa dan empat basa nitrogen sedangkan DNA terdiri gula yang berbeda yaitu deoksiribosa dan empat buah basa (Weaver, 2001). Komponen penyusun nukleotida terdiri dari tiga jenis molekul, yaitu gula pentosa (deoksiribosa pada DNA atau ribosa pada RNA), basa nitrogen, dan gugus fosfat (Gambar 2.3). Basa yang
ditemukan pada nukleotida adalah basa purin (adenin = A, guanin = G) dan basa pirimidin (cytosin = C, tymin = T, urasil = U) (Gambar 2.4). Monomer nukleotida mempunyai gugus hidroksil pada posisi karbon 3’, gugus fosfat pada posisi karbon 5’ dan basa pada posisi karbon 1’ molekul gula. Nukleotida satu dengan yang lainnya berikatan melalui ikatan fosfodiester antara gugus 5’fosfat dengan gugus 3’hidroksil. (Gaffar, 2007).
UIN Syarif Hidayatullah Jakarta
Gambar 2.4 Struktur basa nitrogen purin dan pirimidin (Gaffar, 2007).
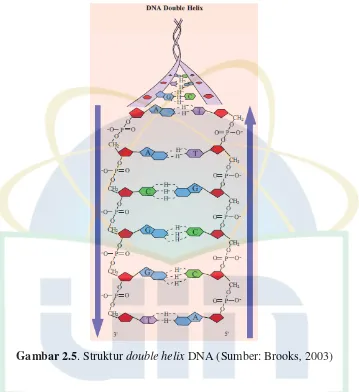
Pada tahun 1953, Watson dan Crick mengemukakan bahwa struktur
molekul DNA merupakan rantai heliks ganda yang memutar kekanan (Gaffar, 2007). Struktur molekul DNA terdiri atas dua rangkaian nukleotida yang tersusun secara linier. Kedua rangkaian yang saling berikatan itu terbentuk seperti tali berpilin, sehingga molekul DNA dikatakan sebagai double helix
UIN Syarif Hidayatullah Jakarta
pasangan G dan C terbentuk dengan tiga ikatan. Oleh karena itu, pasangan G dan C lebih stabil daripada pasanagan A dan T (Muladno, 2010).
Gambar 2.5. Struktur double helix DNA (Sumber: Brooks, 2003)
UIN Syarif Hidayatullah Jakarta
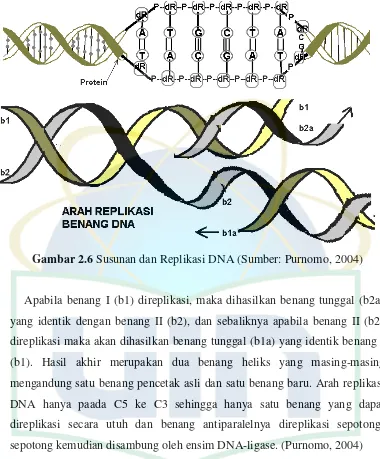
Gambar 2.6 Susunan dan Replikasi DNA (Sumber: Purnomo, 2004)
Apabila benang I (b1) direplikasi, maka dihasilkan benang tunggal (b2a) yang identik dengan benang II (b2), dan sebaliknya apabila benang II (b2) direplikasi maka akan dihasilkan benang tunggal (b1a) yang identik benang I (b1). Hasil akhir merupakan dua benang heliks yang masing-masing mengandung satu benang pencetak asli dan satu benang baru. Arah replikasi DNA hanya paada C5 ke C3 sehingga hanya satu benang yang dapat direplikasi secara utuh dan benang antiparalelnya direplikasi
sepotong-sepotong kemudian disambung oleh ensim DNA-ligase. (Purnomo, 2004) Sifat Fisika DNA adalah DNA akan terpisah dari rantai komplemennya
(denaturasi) pada suhu mendekati titik didih dan pH ekstrim (pH < 3 atau pH > 10) dan bisa bergabung kembali (renaturasi) pada suhu ± 600C (Watson et al,1988). DNA menyerap sinar UV dengan panjang gelombang 260 nm (Sambrook et al, 1989).
2.5.2 DNA Mitokondria
UIN Syarif Hidayatullah Jakarta
demikian mitokondria disebut ”pembangkit tenaga” bagi sel (Koolman et al, 2005).
Keseluruhan mitokondria dalam satu sel mencapai hingga 25% volume sel. Mitokondria dikelilingi oleh dua membran yaitu membran dalam dan membran luar. Ruang antara membran dalam dan luar disebut ruang antar membran. Membran bagian dalam membentuk lipatan-lipatan yang disebut kristae dimana terdapat enzim-enzim oksidase Membran dalam juga memiliki permukaan yang besar yang mengelilingi ruang matriks. Matriks ini mengandung DNA, RNA, ribosom dan berbagai enzim yang berperan dalam oksidasi zat-zat makanan. (Koolman et al, 1994).
Mitokondria memiliki perangkat genetik sendiri yaitu DNA mitokondria atau sering disingkat mtDNA. Dengan bantuan DNAnya, mitokondria mempunyai kemampuan untuk mensintesis sendiri beberapa proteinnya.
Namun bagian terbesar protein mitokondria disandi di dalam inti sel, kemudian disintesis pada ribosom yang bebas dalam sitoplasma dan diimpor
ke dalam mitokondria (Koolman et al, 1994).
Ukuran genom mitokondria minimum untuk berfungsinya mitokondria hewan multiseluler adalah 14.000 pasang basa dari total ukuran yang berkisar hingga 39.000 pasang basa. DNA mitokondria merupakan DNA rantai ganda yang berbentuk sirkuler. Ukuran DNA mitokondria relatif sangat kecil dibandingkan dengan ukuran genom intinya. (Solihin, 1994).
DNA mitokondria hewan secara umum memiliki jumlah dan jenis gen yang sama yaitu 13 daerah yang mengkode protein (URF1, URF2, URF3, URF4, URF5, URF6, URFA6L, URF4L, Cytochrome Oxidase unit I,
UIN Syarif Hidayatullah Jakarta
Gambar 2.7. Struktur mtDNA pada manusia (Sumber: Passarge, 2007)
DNA mitokondria bersifat khusus yang diturunkan melalui induk betina tanpa mengalami rekombinasi. Adanya sifat tersebut dapat digunakan untuk suatu rekonstitusi historik dari genealogi matrilinier suatu spesies maupun antar populasi yang ada. Beberapa hal yang mendukung penggunaan mtDNA sebagai penanda dalam studi keragaman genetik dan studi biologi populasi pada hewan yaitu (Solihin, 1994):
1. DNA mitokondria terdapat dalam jumlah kopi yang tinggi. Jumlah kopi yang tinggi ini menjadikannya mudah diisolasi dan dipurifikasi untuk berbagai keperluan analisis genom.
2. Ukuran DNA mitokondria relatif kecil (14-39 kb) sehingga dapat dipelajari sebagai satu kesatuan yang utuh.
3. Bagian-bagian dari genom mitokondria berevolusi dengan kecepatan yang
UIN Syarif Hidayatullah Jakarta
4. DNA mitokondria hewan tidak memiliki intron ataupun spacer yang berukuran besar antar gennya.
5. DNA mitokondria bersifat khusus karena diturunkan melalui induk betinanya tanpa mengalami rekombinasi (strict matertralinheritance). 6. DNA mitokondria sangat polimorf, baik untuk intrapopulasi maupun untuk
interspesies. 2.5.3 Isolasi DNA
Semua organisme disusun oleh sel yang mengandung elemen genetik yang sama yaitu DNA yang terdapat dalam kromosom. Kromosom eukariot berbentuk linier sedangkan kromosom prokariot berbentuk sirkular. Selain itu prokariot juga mengandung satu atau lebih plasmid. Plasmid merupakan molekul DNA sirkular dengan ukuran yang jauh lebih kecil dibanding kromosom (Gaffar, 2007).
Prinsip isolasi DNA adalah memisahkan DNA dari komponen-komponen sel lain. Isolasi DNA dari organisme eukariot dilakukan melalui proses penghancuran membran sel (lisis), pemusnahan protein dan RNA, dan pemanenan DNA (Muladno, 2010). Membran sel dilisis dengan menambahkan buffer yang mengandung satu atau lebih deterjen, contohnya SDS (B), NP-40, atau Triton X-100 untuk membebaskan isinya. Kotoran sel yang ditimbulkan akibat pengrusakan oleh detergen tersebut dibersihkan
dengan cara sentrifugasi Kemudian pada ekstrak sel tersebut ditambahkan protenase yang berfungsi untuk mendegragasi protein dan RNAse yang
berfungsi untuk mendegragasi RNA, sehingga tertinggal hanyalah DNA. Selanjutnya ekstrak tersebut dipanaskan sampai suhu 900C untuk menginaktivasi enzim yang mendegradasi DNA (DNAse). Larutan DNA kemudian dipresipitasi dengan etanol dan bisa dilarutkan lagi dengan air (Gaffar, 2007).
UIN Syarif Hidayatullah Jakarta
termasuk darah, daging, kultur sel, sampel klinis (sputum, feses), jaringan hewan, ekor tikus, ragi dan banyak lagi (Roched,2012)
2.6 Polymerase Chain Reaction (PCR)
Reaksi berantai polymerase (Polymerase Chain Reaction, PCR) adalah suatu metode enzimatis untuk amplifikasi DNA dengan cara in vitro. PCR ini pertama kali dikembangkan pada tahun 1985 oleh Kary B. Mullis. (Yusuf, 2010). DNA polymerase ini biasanya disintesis dari mikroorganisme yang hidup pada suhu panas, seperti Thermophilus aquaticus, sehingga enzim yang berasal dari mikroorganisme tersebut disebut Taq polymerase (Passarge, 2007). Dengan teknik ini sejumlah fragmen kecil DNA yang diinginkan akan diamplifikasi secara eksponensial sampai jutaan kali dalam beberapa jam (Sulistyaningsih, 2007).
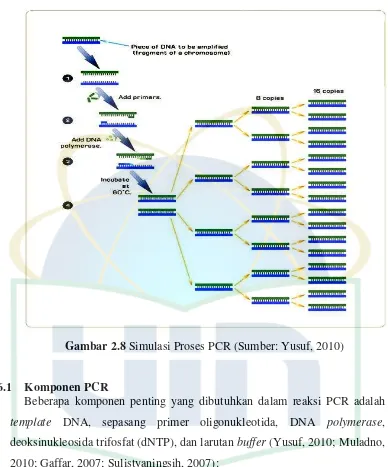
PCR digunakan untuk menggadakan jumlah molekul DNA pada target tertentu dengan cara mensintesis molekul DNA baru yang berkomplemen dengan molekul DNA target tersebut melalui bantuan enzim dan oligonukleotida sebagai primer dalam suatu thermocycle (Muladno, 2010). Amplifikas DNA pada PCR dapat dicapai bila menggunakan primer oligonukleotida yang disebut amplimers. Primer DNA adalah suatu sekuens oligonukleotida pendek yang berfungsi mengawali sintesis rantai DNA
(Yusuf, 2010). PCR memungkinkan dilakukannya pelipatgandaan suatu fragmen DNA Primer yang berada sebelum daerah target disebut primer
UIN Syarif Hidayatullah Jakarta
Gambar 2.8 Simulasi Proses PCR (Sumber: Yusuf, 2010)
2.6.1 Komponen PCR
Beberapa komponen penting yang dibutuhkan dalam reaksi PCR adalah
template DNA, sepasang primer oligonukleotida, DNA polymerase, deoksinukleosida trifosfat (dNTP), dan larutan buffer (Yusuf, 2010; Muladno, 2010; Gaffar, 2007; Sulistyaningsih, 2007):
1. Template DNA
Template DNA adalah molekul DNA untai ganda yang mengandung
sequen target yang akan diamplifikasi. Ukuran DNA bukan merupakan faktor utama keberhasilan PCR, berapapun panjangnya jika tidak mengandung sequen yang diinginkan maka tidak akan berhasil proses suatu PCR, namun sebaliknya jika ukuran DNA tidak terlalu panjang tapi mengandung sequen yang diinginkan maka PCR akan berhasil (Sulistyaningsih, 2007).
UIN Syarif Hidayatullah Jakarta
kuantitas atau konsentrasi (Yusuf, 2010). Jika konsentrasinya terlalu rendah maka primer mungkin tidak dapat menemukan target dan jika konsentrasi terlalu tinggi akan meningkatkan kemungkinan mispriming. Disamping itu perlu diperhatikan kemurnian template karena akan mempengaruhi hasil reaksi (Sulistyaningsih, 2007).
2. Primer
Susunan primer merupakan salah satu kunci keberhasilan PCR. Pasangan primer terdiri dari 2 oligonukleotida yang mengandung 18-28 nukleotida dan mempunyai 45-60% GC content yang digunakan untuk mengawali sintesis rantai DNA (Yusuf, 2010). Sequen primer yang lebih pendek akan memicu amplifikasi produk PCR non spesifik. Ujung 3’ primer penting dalam menentukan spesifisitas dan sensitivitas PCR. Ujung ini tidak boleh mempunyai 3 atau lebih basa G atau C, karena
dapat menstabilisasi annealing primer non spesifik. Disamping itu ujung 3’ kedua primer tidak boleh komplementer satu dengan yang lain, karena hal ini akan mengakibatkan pembentukan primer-dimer yang akan menurunkan hasil produk yang diinginkan. Ujung 5’ primer tidak terlalu penting untuk annealing primer, sehingga memungkinkan untuk menambahkan sequen tertentu misalnya sisi restriksi enzim, start codon
ATG atau sequen promoter (Sulistyaningsih, 2007). Untuk merancang urutan primer, perlu diketahui urutan nukleotida pada awal dan akhir DNA target. Primer oligonukleotida di sintesis menggunakan suatu alat yang disebut DNA synthesizer (Gaffar, 2007)
Spesifisitas PCR sangat tergantung pada suhu melting (Tm) primer, yaitu suhu dimana separuh jumlah primer annealing pada template. Tm
UIN Syarif Hidayatullah Jakarta
3. DNA polymerase
DNA polymerase adalah enzim yang mengkatalisis polimerisasi DNA. Dalam sejarahnya, PCR dilakukan dengan menggunakan Klenow fragment DNA Polimerase I selama reaksi polimerisasinya. Enzime ini ternyata tidak aktif secara termal selama proses denaturasi, sehingga peneliti harus menambahkan enzim di setiap siklusnya. Dalam perkembangannya, kini banyak digunakan enzim Taq DNA polymerase
yang memiliki keaktifan pada suhu tinggi sehingga penambahan enzim tidak perlu dilakukan disetiap siklus dan proses PCR dapat dilakukan dalam satu mesin (Gaffar, 2007). Enzim ini diperoleh dari Eubacterium
yang disebut Thermus aquaticus, spesies ini diisolasi dari taman
Yellowstone pada tahun 1969. Enzim polimerase Taq tahan terhadap pemanasan berulang-ulang karena akan membantu melepaskan ikatan
primer yang tidak tepat dan meluruskan wilayah yang mempunyai struktur sekunder (Yusuf, 2010)
Enzim Taq DNA polymerase terdiri atas dua macam yaitu enzim alami yang diisolasi dari sel bakteri Thermus aquaticus dan enzim rekombinan yang disintesis didalam sel bakteri Escherichia coli
(Muladno, 2010).
Enzim ini masih mempunyai aktivitas eksonuklease dari 5’ ke 3’ tetapi tidak mempunyai aktivitas eksonuklease dari 3’ ke 5’. Konsentrasi enzim yang dibutuhkan untuk PCR biasanya 0,5-2,5 unit. Kelebihan jumlah enzim mengakibatkan akumulasi produk non spesifik, sedangkan jika terlalu rendah maka dihasilkan sedikit produk yang diinginkan (Sulistyaningsih, 2007).
4. Deoxynucleotide Triphosphate (dNTP)
UIN Syarif Hidayatullah Jakarta
ketepatan PCR. Konsentrasi masing-masing dNTP harus seimbang untuk meminimalkan kesalahan penggabungan. Deoxynucleotide Triphosphate
akan menurunkan Mg2+ bebas sehingga mempengaruhi aktivitas
polymerase dan menurunkan annealing primer. Konsentrasi dNTP yang rendah akan meminimalkan mispriming pada daerah non target dan menurunkan kemungkinan perpanjangan nukleotida yang salah. Oleh karena itu spesifisitas dan ketepatan PCR meningkat pada konsentrasi dNTP yang lebih rendah (Sulistyaningsih, 2007).
5. Larutan buffer
Larutan buffer yang biasa digunakan untuk reaksi PCR umumnya mengandung 10 – 50mM Tris-HCl pH 8,3-8,8 (suhu 20o C); 50 mM KCl; 0,1% gelatin atau BSA (Bovine Serum Albumin); Tween 20 sebanyak 0,01% atau dapat diganti dengan Triton X-100 sebanyak 0,1%;
disamping itu perlu ditambahkan 1,5 mM MgCl2 (Yusuf, 2010).
Optimalisasi konsentrasi ion Mg2+ merupakan hal yang penting.
Konsentrasi ion ini mempengaruhi beberapa hal yaitu annealing primer, suhu pemisahan untai template dan produk PCR, spesifisitas produk, pembentukan primer-dimer serta aktivitas dan ketepatan enzim Taq Polymerase. PCR harus mengandung 0,5-2,5 µM Mg2+ dari total konsentrasi dNTP. Konsentrasi yang lebih tinggi akan meningkatkan produk PCR tetapi menurunkan spesifisitasnya. Konsentrasi ion ini tergantung pada konsentrasi bahan-bahan yang mengikatnya seperti dNTP, EDTA dan fosfat (Sulistyaningsih, 2007).
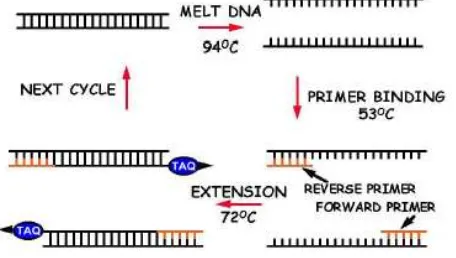
2.6.2 Tahapan PCR
UIN Syarif Hidayatullah Jakarta
Berikut ini merupakan tahapan yang terjadi pada proses PCR (Yusuf, 2010: Muladno, 2010; Gaffar, 2007; Sulistyaningsih, 2007):
1. Denaturasi
Selama proses denaturasi, DNA untai ganda akan membuka menjadi dua untai tunggal. Hal ini disebabkan karena suhu denaturasi yang tinggi menyebabkan putusnya ikatan hidrogen diantara basa-basa yang komplemen. Pada tahap ini, seluruh reaksi enzim tidak berjalan (Gaffar, 2007). Denaturasi biasanya dilakukan antara suhu 90-950 C selama 3 menit untuk meyakinkan bahwa molekul DNA yang ditargetkan ingin dilipatgandakan jumlahnya benar-benar telah terdenaturasi menjadi untai tunggal. (Muladno, 2010). Denaturasi yang tidak lengkap mengakibatkan DNA mengalami renaturasi (membentuk DNA untai ganda lagi) secara cepat, dan ini mengakibatkan gagalnya proses PCR. (Yusuf, 2010). Untuk denaturasi berikutnya, waktu
yang diperlukan hanya 30 detik pada suhu 950 C atau 15 detik pada suhu 970 C (Muladno, 2010).
Suhu denaturasi dipengaruhi oleh sequen target. Jika sequen target kaya akan G-C maka diperlukan suhu yang lebih tinggi. Suhu denaturasi yang terlalu tinggi dan waktu denaturasi yang terlalu lama mengakibatkan hilangnya atau berkurangnya aktivitas enzim Taq polymerase. (Muladno, 2010; Sulistyaningsih, 2007).
2. Penempelan primer
Pada tahap penempelan primer (annealing), primer akan menuju daerah yang spesifik yang komplemen dengan urutan primer. Kriteria yang umum digunakan untuk merancang primer yang baik adalah bahwa primer sebaiknya berukuran 18 – 25 basa, mengandung 50 – 60 % G+C dan untuk kedua primer tersebut sebaiknya sama. Sekuens DNA dalam masing-masing primer itu sendiri juga sebaiknya tidak saling berkomplemen, karena hal ini akan mengakibatkan terbentuknya struktur sekunder pada primer tersebut dan mengurangi efisiensi PCR (Yusuf, 2010). Pada proses annealing ini, ikatan hidrogen akan terbentuk antara primer dengan urutan komplemen pada
UIN Syarif Hidayatullah Jakarta
menjadi sangat kuat dan tidak akan putus kembali apabila dilakukan reaksi polimerisasi selanjutnya (Gaffar, 2007).
Suhu dan lamaya waktu yang dibutuhkan untuk annealing primer tergantung pada komposisi basa, panjang, dan konsentrasi primer (Sulistyaningsih, 2007). Waktu annealing yang biasa digunakan dalam PCR adalah 30 – 45 detik. Semakin panjang ukuran primer, semakin tinggi temperaturnya. Kisaran temperatur penempelan yang digunakan adalah antara 36oC sampai dengan 72oC, namun suhu yang biasa dilakukan itu adalah antara 50 – 60oC. (Yusuf, 2010)
3. Reaksi polimerisasi
Umumnya reaksi polimerisasi (extension) atau perpanjangan rantai, terjadi pada suhu 720 C karena merupakan suhu optimum Taq polymerase. Primer yang telah menempel tadi akan mengalami perpanjangan pada sisi 3’nya dengan penambahan dNTP yang komplemen dengan template oleh DNA
polymerase (Gaffar 2007).
Kecepatan penyusunan nukleotida oleh enzim tersebut pada suhu 720 C diperkirakan antara 35 sampai 100 nukleotida per detik, bergantung pada buffer, pH, konsentrasi garam, dan molekul DNA target. Dengan demikian, untuk produk PCR sepanjang 2000 pasang basa, waktu 1 menit sudah lebih dari cukup untuk tahap pemanjangan primer ini. Biasanya di akhir siklus PCR, waktu yang digunakan untuk tahap ini diperpanjang sampai 5 menit, sehingga seluruh produk PCR diharapkan berbentuk DNA untai ganda (Muladno, 2010).
2.7 Real-time PCR 2.7.1 Prinsip Analisis
Real-Time PCR merupakan teknologi terkini untuk amplifikasi DNA. Pada
Real-Time PCR jumlah DNA yang diamplifikasi bisa langsung diamati secara seketika sehingga tidak memerlukan analisis dengan elektroforesis gel untuk mengetahui produk PCR. Real-Time PCR lebih dikenal sebagai quantitative
PCR karena kemampuan analisisnya yang sensitif, spesifik dan reproducibility
UIN Syarif Hidayatullah Jakarta
Instrumen Real-Time PCR mendeteksi amplikon dengan mengukur peningkatan pewarna (dye) fluorescent yang berpendar ketika terikat dengan
double-stranded DNA. Karena sifat inilah maka pertumbuhan fragment DNA hasil amplifikasi dapat diikuti secara seketika, semakin banyak DNA yang terbentuk semakin tinggi pula intensitas fluorescent yang dihasilkan.
Quantitative PCR dimungkinkan dapat mendeteksi secara akurat konsentrasi DNA hingga hitungan pikogram atau setara dengan sel tunggal karena sensitifitas dye yang sangat tinggi. Hasil peningkatan fluorescent digambarkan melalui kurva amplifikasi yang menunjukkan tiga fasa yaitu fasa awal, fasa eksponensial atau puncak dan fasa plateau atau stabil (Vaerman, 2004).
Gambar 2.10 Bentuk Kurva pada Real-Time PCR (Sumber: BioRad, 2006)
Instrument Real-Time PCR memiliki tiga komponen utama yaitu thermal block cycler sebagai akurasi data, optical system sebagai deteksi data, dan
software sebagai analisis data. Real-Time PCR juga dapat menganalisis banyak sampel dalam waktu bersamaan menggunakan multiwell plates
UIN Syarif Hidayatullah Jakarta
2.7.2 Analisis menggunakan Metode Hydrolysis Probe
Instrumen Real-Time PCR menggunakan pewarnaan flurosensi secara Online dan Real Time, baik untuk memonitor Hasil dari produk PCR
selama siklus berlangsung maupun setelah siklus pada proses melting hasil produk PCR untuk menganalisis Melting Curve. (Rochea, 2008)
Ada beberapa analisis pewarnaan yang dapat dilakukan antara lain: (Rochea, 2008)
1. Uji Deteksi Sequence Independent
Mengandalkan fluorophores yang mengikat semua DNA molekul untai ganda (dsDNA) terlepas dari urutan basanya; misalnya SYBR Green I.
2. Uji Sequence-Specific Probe Binding
Mengandalkan fluorophores yang berpasangan ke probe oligonukleotida dengan sequence-specific yang berhibridisasi dengan urutan komplementernya dalam target produk PCR yaitu Metode Simpel Probe, Hybridization Probe (Hyb Probe) dan Hydrolysis Probe
Hydrolysis probe menggunakan oligo nukleotida spesifik berkomplemen dengan DNA target disebut probe. Probe dilabeli oleh dua molekul, yaitu reporter pada ujung 5’ probe yang merupakan pewarna flurosensi dan quencher pada ujung 3’ probe yang merupakan molekul
UIN Syarif Hidayatullah Jakarta
Tabel 2. Tahapan Proses Fluoresensi Hydrolysis Probe
No Gambar Keterangan
Hydrolysis Probe membawa dua
fluorescent dye berdekatan dengan
quencher dye menekan sinyal
fluorescent reporter. Ujung 3’ dari
probe terfosforilasi sehingga tidak dapat memanjang selama PCR. Di tahap annealing, primer dan probe sama-sama menempel ke urutan basa pada target spesifik
Ketika DNA polimerase memanjangkan primer dan bertemu dengan probe. Kemudian memecah probe pada ujung 5’ sehingga menggantikan posisinya dan terus memanjang sampai membentuk amplikon baru.
Saat probe terpecah, reporter tidak lagi berdekatan dengan quencher
sehingga dapat mengeimisikan cahaya fluorescent yang dibaca oleh detektor. Semakin tinggi flurosens yang dipancarkan dari reporter secara langsung berkorelasi dengan akumulasi pelepasan molekul
reporter dye (sekaligus menandakan jumlah produk PCR yang
UIN Syarif Hidayatullah Jakarta
Hydrolysis probe biasa digunakan dalam multipleks quantitative Real-Time PCR yang menggunakan DNA target dan pasangan lebih dari satu dalam satu reaksi karena probe akan berikatan secara spesifik dengan beberapa DNA target yang berbeda (BioRad, 2006). Metode Hydrolysis probe memiliki keunggulan dibandingkan dengan SYBR Green karena dapat mengamplifikasi DNA lebih spesifik (Izzah, 2014).
Pewarna fluorescent yang digunakan dalam Hydroysis probe bermacam ragamnya yang disesuaikan dengan penggunaannya. Metode Hydrolysis
probe dapat digunakan secara terpisah atau dengan kombinasi pewarna. Dalam penggunaannya dengan format deteksi monocolor biasa digunakan reporter FAM dengan nilai eksitasi dan emisi berturut-turut 483 dan 533, pewarna lainnya yang tersedia untuk deteksi multicolor antara lain Cyan 500, Hex, Red 610 dan Cy 5 dengan nilai eksitasi dan emisi dalam keadaan normal
berturut turut 450 dan 500, 523 dan 568, 558 dan 610 serta 615 dan 670. Sebagai quencher terutama untuk deteksi multicolor digunakan quencher dye
27 UIN Syarif Hidayatullah Jakarta
3.1 Tempat dan Waktu Penelitian 3.1.1 Tempat
Penelitian ini dilaksanakan di Laboratorium Analisa Obat dan Pangan Halal dan Laboratorium Penelitian II Fakultas Kedokteran dan Ilmu Kesehatan Universitas Islam Negeri Syarif Hidayatullah, Jakarta.
3.1.2 Waktu
Waktu pelaksanaan penelitian dilakukan dari bulan Maret 2014 hingga Desember 2014.
3.2 Alat dan Bahan 3.2.1 Alat
Real-Time PCR (LightCycler® 480 - Roche), Multiwell plate 96 (Roche), Spektrofotometer UV DNA (BioDrop DUO), Refrigerated Microcentrifuge (5417R – Eppendorf), Timbangan Analitik. Mikropipet 0,5-10 μl; 2-20 μl; 20-200 μl (Biorad), Mikrotips volume 10 μl; 200 μl; 1000 μl (Genfollower), PCR tube dan microcentrifuge tube volume 1,5 μl (Biogenix), Microcentrifuge (Wiggenhauser), Vortex (Wiggenhauser),
Filter tube dan Collection tube (Kit High Pure PCR Template Preparation
- Roche), Digital waterbath (Eyela), Pisau steril, dan Alat gelas yang digunakan adalah Gelas ukur 100 ml, Gelas beker 500 ml (Pyrex), Kaca arloji, Spatula, dan Batang pengaduk.
3.2.2 Bahan
Daging sapi segar, daging babi segar, 7 produk bakso sapi yang beredar di sekitar kampus UIN Jakarta; 1 produk bakso sapi yang berada di pusat perbelanjaan di sekitar kampus UIN Jakarta, satu set reagen isolasi DNA yang terdiri dari Tissue Lysis Buffer; Binding Buffer; Proteinase K; Inhibitor Removal Buffer; Washing Buffer; dan Elution Bufffer (Kit High Pure PCR Template Preparation), ddH2O (Roche), Aqua
bidest (IKA pharmindo), Isopropanol dan Etanol absolut (Merck), Probe
UIN Syarif Hidayatullah Jakarta
Tabel 3. Susunan basa primer dan probe untuk DNA sapi dan babi (Tanabe et a.l, 2007)
Babi Forward 5’-CTTGCAAATCCTAACAGGCCTG-3’
Reverse 5’-CGTTTGCATGTAGATAGCGAATAAC-3’
Probe 5’-(FAM)-ACAGCTTTCTCATCAGTTAC-(BHQ1)-3’ Sapi Forward 5’-CCCGATTCTTCGCTTTCCAT-3’
Reverse 5’-CTACGTCTGAGGAAATTCCTGTTG-3’
Probe 5’-(FAM)-CATCATAGCAATTGCC-(BHQ1)-3’
3.3 Tahapan Penelitian 1. Pengumpulan sampel
2. Isolasi DNA sampel dan kontrol 3. Analisis Isolat DNA
4. Amplifikasi DNA menggunakan Real-Time PCR
3.4 Prosedur Kerja 3.4.1 Pengumpulan sampel
Pengumpulan sampel dilakukan terhadap 8 sampel bakso sapi dengan produsen berbeda yang beredar di wilayah Ciputat. 6 sampel dipilih secara random dari produsen tradisional yang menjajakan produknya dengan menetap di kios permanen di sekitar kampus UIN Syarif Hidayatullah. 2 sampel lainnya diperoleh dari produsen yang produknya telah memiliki merek dagang (branded), telah luas dikenal masyarakat dan memiliki banyak cabang selain di sekitar Ciputat. Pengumpulan sampel dilakukan pada tanggal 10 November 2014 dengan cara membeli secara tunai. Sampel dari produsen tradisional didapatkan dalam keadaan belum di hidangkan sedangkan sampel branded diperoleh dalam keadaan siap saji lengkap dengan bumbu-bumbu penyedap, rempah-rempah maupun kuah kaldu panas yang biasa menyertainya.
3.4.2 Isolasi DNA
UIN Syarif Hidayatullah Jakarta
3.4.2.1Preparasi Sampel a. Daging Segar
Masing-masing daging sapi segar maupun daging babi segar dihaluskan dengan cara dicincang menggunakan pisau steril. Daging
yang telah dihaluskan ditimbang sebanyak 50 mg dan kemudian
Simulasi bakso dibuat mengacu kepada resep tradisional dengan perlakuan yang sama baik pada pembuatan bakso sapi maupun babi. 50 g daging yang telah di haluskan di campur dengan 100 g tepung terigu dan 100 g tepung kanji, tambahkan 100 ml air sedikit demi sedikit sambil di aduk merata perlahan-lahan. Untuk penyedap ditambahkan 3 siung bawang putih yang telah di haluskan, aduk sampai terbentuk adonan. Adonan kemudian dibentuk menyerupai bola, seperti bakso lalu direbus dengan air mendidih hingga mengambang. Semua proses dilakukan menggunakan alat yang terpisah serta tempat pengerjaan yang berbeda untuk menghindari kontaminasi.
Setelah menjadi bakso, potong menjadi 2 bagian sama besar. Pada bagian dalam di posisi tengah bakso yang telah terpotong, sayat
secukupnya daging bakso tersebut. Sayatan bakso kemudian dicincang hingga halus dan ditimbang sebanyak 50 mg, masukkan ke dalam
microsentrifuge tube dan sampel siap dilanjutkan ke proses selanjutnya. Proses ini juga berlaku bagi sampel bakso yang dikumpulkan dari produsen dengan terlebih dahulu mencuci bakso yang didapatkan menggunakan air mengalir sebelum di lakukan preparasi.
3.4.2.2Proses Melisiskan Sampel dan Mengikat DNA (Rocheb,2012)
UIN Syarif Hidayatullah Jakarta
Larutan yang terbentuk ditambah dengan 200 µl Binding Buffer, vortex hingga homogen dan inkubasi kembali pada suhu 70oC selama 10 menit. Lanjutkan dengan penambahan isopropanol 100 µl, vortex hingga homogen. Untuk menghilangkan partikel-partikel tidak larut bisa dilakukan dengan mempipet larutan dengan micro pipet 1ml. Ketika cairan dikeluarkan maka partikel-partikel tidak larut akan tertinggal di tips.
Masukkan cairan yang telah bebas dari partikel tidak larut ke dalam
filter tube yang telah dipasangkan dengan collection tube. Sentrifugasi menggunakan refrigerated centrifuge selama 1 menit dengan putaran 8700 rpm. DNA akan tertinggal pada kapas fiberglas di filter tube. Siap untuk proses pemurnian dan elusi.
3.4.2.3Proses Pemurnian dan Elusi DNA (Rocheb,2012)
Buang collection tube beserta cairan yang tertampung didalamnya.
Filter tube yang mengandung DNA dipasangkan kembali dengan
collection tube baru, kemudian tambahkan 500 µl inhibitor removal buffer
ke dalam filter tube. Sentrifugasi kembali dengan putaran 8700 rpm selama 1 menit.
Lepaskan filter tube dan kembali pasangkan dengan collection tube
baru. Buang collection tube beserta cairan yang ditampungnya. Tambahkan 500 µl wash buffer ke dalam filter tube, lakukan sentrifugasi dengan putaran 8700 rpm selama 1 menit.
Proses sebelumnya diulangi untuk benar-benar didapatkan DNA yang murni. Lepaskan filter tube dan kembali pasangkan dengan collection tube
baru. Buang collection tube beserta cairan yang ditampungnya. Tambahkan 500 µl wash buffer ke dalam filter tube, lakukan sentrifugasi dengan putaran 8700 rpm selama 1 menit kembali. Tahap akhir pemurnian setelah cairan di dalam collection tube dibuang, pasangkan kembali filter tube dan collection tube tersebut. Kemudian sentrifugasi dengan kecepatan maksimum yakni 15000 rpm selama 10 detik.
UIN Syarif Hidayatullah Jakarta
kecepatan 8700 rpm selama 1 menit. Larutan dalam microsentrifuge tube
yang keluar melewati filter tube kini telah mengandung DNA.
3.4.3 Analisis Hasil Isolasi DNA dengan Spektrofotometer UV untuk DNA (BioDrop, 2015)
Instrumen BioDrop DUO yang digunakan dalam analisis ini menggunakan sistem layar sentuh. Setelah dihidupkan dan proses kalibrasi selesai maka akan tampil 6 menu, pilih “Life Science”, kemudian antara “Nucleic Acid” dan “Protein” pilih “Nucleic Acid”, kemudian “DNA”.
Mode pengukuran konsentrasi dan kemurnian DNA pun berhasil
ditampilkan.
Sebelum dilakukan pengukuran, lakukan beberapa penyesuaian antara lain: Pada menu “pathlength” pilih µ Lite 0.5mm. Untuk mendapatkan hasil pengukuran dalam nanogram, pada menu “unit” ubah “µg/ml” menjadi “ng/µl”. Setelah pengaturan selesai, tekan “next” yang dilambangkan dengan simbol anak panah ke kanan, pengukuran telah dapat dilakukan.
Sebelum mengukur sampel terlebih dahulu dilakukan analisis blangko, blangko dalam hal ini adalah elution buffer. Bersihkan sampel port dengan tisu, 2 µl elution buffer dimasukkan ke dalam sampel port kemudian pilih “Blangko” dengan simbol kuvet tanpa isi. Setelah itu di layar akan menampilkan nilai konsentrasi 0,000 ng/µl dan pada nama sampel akan tertera reference. Pengukuran sampel dilakukan sama seperti perlakuan pada blangko, masukan sampel satu persatu dengan sebelumnya tidak lupa selalu membersihkan sampel port dengan tisu sebelum sampel baru dimasukkan. Selanjutnya pilih “Measure”dengan simbol kuvet berisi pada layar. Akan ditampilkan nilai konsentrasi beserta kemurnian DNA.
3.4.4 Uji Spesifisitas Primer dan Probe dengan metode BLAST menggunakan Database NCBI
Dari laman website NCBI, uji spesifisitas dilakukan dengan terlebih dahulu menuju ke laman BLAST. Pada laman BLAST, klik menu “Nucleotide Blast”. Setelah itu masukkan urutan basa yang akan diujikan di kolom “Enter Query Sequence”. Pisahkan antar urutan basa dengan
UIN Syarif Hidayatullah Jakarta
Muncul ditampilan berupa data yang berisikan daftar spesies yang memiliki kemiripan dengan data yang diujikan.
3.4.5 Amplifikasi DNA menggunakan Real-Time PCR dengan metode Hydrolysis Probe
Langkah pertama pembuat larutan induk primer dan probe dibuat dengan konsentrasi 100 µM sesuai dengan instruksi yang terdapat dalam dokumen dari produsen, dari larutan induk tersebut diencerkan konsentrasinya menjadi 10 µM. Pengenceran dibuat sesuai perhitungan (lampiran 4a) dengan mencampurkan 10 µl larutan induk dan 90 µl
aquadest di dalam microcentifuge tube, kemudian dihomogenkan dengan menaikturunkan pegas micropipet.
Pembuatan master mix disesuaikan protokol yang ada (lampiran 4b) mengacu pada penelitian sebelumnya dimana konsentrasi primer ditetapkan sebesar 0,8 µM dan probe 0,2 µM (Izzah, 2014). Sebelum melakukan pencampuran dilakukan perhitungan (lampiran 4a), kemudian setiap reaksi master mix dibuat dengan mencampurkan secara berurutan 1,6 µl primer forward, 1,6 µl primer reverse, 1,4 µl aquadest, 0,4 µl probe dan 10 µl probe master. Setelah memasukkan probe dan probe master, campuran tidak boleh terlalu banyak di homogenkan untuk menghindari kerusakan. Ketika proses pencampuran sampai pada saat pemasukkan ke dalam instrumen, larutan diwajibkan tidak terpapar cahaya. Penambahan DNA template sebesar 5 µl menjadikan total keseluruhan campuran menjadi 20 µl.
Sebelum running, dilakukan pengaturan subset dan sample editor serta program amplifikasi dengan LightCycler 480® Software 1,5 seperti pada tabel berikut:
Tabel 4. Program amplifikasi Real-Time PCR (Rochec, 2008)
Setup
Detection Format Block Type Reaction Volume Mono Color
Hydrolysis Probe 96 20 µ L
UIN Syarif Hidayatullah Jakarta
Amplification 40 Quantification
Cooling 1 None
*Keterangan: Suhu 61oC untuk primer sapi & Suhu 60oC untuk primer babi (Lampiran 6)
Setelah campuran master mix dan program amplifikasi siap, terlebih dahulu masukkan 5 µl sampel atau DNA template pada multiwell plate
sesuai dengan pengaturan yang telah dibuat pada subset editor. Kemudian masukkan 15 µl master mix ke setiap sampel secara perlahan dan dihomogenkan juga dengan sangat perlahan. Kemudian multiwell plate
34 UIN Syarif Hidayatullah Jakarta
BAB 4
HASIL DAN PEMBAHASAN
Penelitian ini menganalisis cemaran daging babi pada produk bakso sapi menggunakan Real-Time PCR dengan metode Hydrolysis Probe. Cemaran daging babi dapat diketahui melalui amplifikasi DNA ditunjukkan dengan naiknya kurva amplifikasi apabila terjadi kontaminasi daging babi dalam produk bakso sapi yang diuji.
4.1Hasil Analisis Isolat DNA dengan Spektrofotometer UV
Berdasarkan hasil analisis, proses ekstraksi dan isolasi DNA menggunakan kit komersial High Pure PCR Template preparation pada penelitian ini menghasilkan isolat DNA yang cukup baik. Isolat DNA didapatkan dengan menggunakan prinsip memisahkan DNA yang terikat pada filter dari pengotor lalu mengelusinya, menyesuaikan dengan protokol dan fungsi dari reagen dalam kit tersebut (Lampiran 2 & 3). Lisis sel dilakukan dengan inkubasi menggunakan Proteinase K dimana keberadaan garam kaotropik menyebabkan inaktifasi seketika semua nuklease. Sentrifugasi membuat terjadinya ikatan yang selektif antara asam nukleat dengan kapas fiberglas pada filter. Kemudian terjadi ikatan asam nukleat kembali selama proses wash and spin yang merupakan tahapan pembersihan molekul-molekul kecil. Tahap terakhir digunakan dapar rendah garam untuk melepaskan asam nukleat yang diperoleh dari kapas fiberglas. (Rocheb, 2012)
Isolat DNA yang diperoleh dianalisis dengan Spektrofotometer UV khusus analisis asam nukleat dengan volume mikro. Dari analisis ini didapat konsentrasi dan kemurnian Isolat DNA. Hasil analisis dapat dilihat pada tabel berikut:
Tabel 5. Konsentrasi dan kemurnian DNA hasil isolasi
No. Isolat DNA Konsentrasi
UIN Syarif Hidayatullah Jakarta
Nilai konsentrasi dan kemurnian DNA diperoleh melalui pengukuran menggunakan Spektrofotometer UV khusus analisis volume mikro dengan limit deteksi 1 ng/µl dan akurasi panjang gelombang ± 2 nm. (BioDrop, 2015).
Analisis dilakukan pada panjang gelombang 260 nm dan 280 nm. Dari perbandingan panjang gelombang 260 nm dan 280 nm di dapat rasio angka
yang mencerminkan tingkat kemurnian DNA terhadap protein.
Konsentrasi DNA pada Daging Sapi (DS) adalah 77,52 ng/µl dan Daging
UIN Syarif Hidayatullah Jakarta
konsentrasi pada daging segar. Konsentrasi yang diperoleh dari semua sampel memenuhi syarat untuk diujikan menggunakan Real-Time PCR.
Kemurnian dari Isolat DNA yang diperoleh akan mempengaruhi akurasi percobaan. Sampel dengan tingkat kemurnian rendah/ tidak murni menunjukkan adanya kontaminasi yang akan menyebabkan hasil amplifikasi tidak spesifik. Kontaminasi dengan protein yang terjadi pada isolat DNA dikarenakan proses ekstraksi yang kurang sempurna. DNA dikatakan murni dari campuran protein apabila nilai perbandingan A260/A280 berkisar antara 1,8 sampai 2,0 (Sambrook et al., 1989).
Kemurnian atau nilai perbandingan A260/A280 pada sampel daging sapi dan daging babi masing-masing 1,913 dan 1,824. Angka tersebut telah masuk dalam kisaran kemurnian yang baik. Berbeda dengan daging segar, hasil yang bervariasi ditunjukkan pada sampel bakso. Angka kemurnian Simulasi Bakso
Sapi; Simulasi Bakso Babi; Bakso A; Bakso F; Bakso G; Bakso I; Bakso Ki; Bakso Ko; Bakso Mr.B dan Bakso SR masing-masing adalah 1,638; 1,897;
1,716; 1,747; 1,629; 1,707; 1,773; 1,629; 1,880 dan 1,642. Hanya Simulasi Bakso Babi dan Bakso Mr.B saja yang tergolong memiliki kemurnian ideal yakni dengan angka 1,897 dan 1,880. Sisanya mendapat angka tidak terpaut jauh dibawah 1,8. Rasio ~ 1,8 umumnya dapat diterima untuk DNA dikatakan murni (NanoDrop, 2007).
Konsentrasi dan kemurnian Isolat DNA merupakan faktor penting dalam analisis DNA. Konsentrasi yang baik tanpa di iringi kemurnian yang baik pula sangat memungkinkan terjadinya kesalahan analisis disebabkan DNA yang tidak spesifik. Sebaliknya, kemurnian yang baik namun tidak dibarengi pula dengan konsentrasi yang baik menyebabkan tidak teramplifikasinya DNA sampel jika konsentarsinya rendah atau justru menghasilkan amplifikasi yang berlebihan jika konsentrasinya tinggi sehingga berakibat terjadinya kesulitan dalam menganalisis sampel yang di uji karena kurva amplifikasinya terlalu cepat mencapai fase plateau.
UIN Syarif Hidayatullah Jakarta
mengetahui tingkat spesifisitas primer dan probe yang digunakan apakah hanya mengamplifikasi satu jenis spesies saja.
Uji spesifisitas primer dilakukan dengan memasukan urutan basa dari primer forward, probe dan pasangan dari primer reverse untuk kemudian di cari kemiripannya dengan urutan basa yang ada dalam database NCBI. Pengunjung dapat memilih sumber database sebagai acuan pencarian, dalam hal ini database yang digunakan adalah nucleotida collection (nr/nt). Dari pencarian tersebut ditampilkan spesies-spesies yang memiliki kemiripan urutan basanya dengan urutan basa dari data yang dimasukkan, maka didapatlah spesies yang paling identik. (NCBI, 2015)
Gambar 4.1 Hasil Uji spesifisitas primer dan probe sapi dengan program BLAST pada laman NCBI
*Keterangan: = primer forward ; = probe = RT primer reverse