ISOLASI DAN KARAKTERISASI FRAGMEN GEN
LEAFY
DARI BANTALAN BUNGA KAKAO
RATNA DEWI ESKUNDARI
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2010
ABSTRAK
RATNA DEWI ESKUNDARI. Isolasi dan Karakterisasi Fragmen Gen
LEAFY
dari
Bantalan
Bunga
Kakao.
Dibimbing
oleh
EDY
DJAUHARI
PURWAKUSUMAH dan TETTY CHAIDAMSARI.
Produktivitas tanaman kakao di Indonesia masih tergolong
rendah
.Informasi
dan pengetahuan pada tingkat molekuler tentang proses perkembangan organ
regeneratif kakao dapat menjadi pemecahan efektif untuk masalah regeneratif
yang berhubungan dengan rendahnya produktivitas tanaman kakao. Gen
LEAFY
merupakan gen kunci dalam transisi meristem tunas menjadi meristem bunga.
Penelitian ini bertujuan untuk mengisolasi gen
LEAFY
dari bantalan bunga kakao
dan menganalisis ekspresi gen
LEAFY
pada beberapa jaringan tanaman kakao.
Isolasi gen
LEAFY
dengan teknik
Reverse Transcriptase Polymerase Chain
Reaction
(RT-PCR) menunjukkan ketiga pasangan primer spesifik yang dirancang
berdasarkan daerah terkonservasi ternyata dapat mengamplifikasi fragmen gen
target. Selanjutnya fragmen ini disisipkan ke dalam vector pGEM-T
Easy
dan
selanjutnya ditransformasikan ke dalam kompeten
Escherichia coli
XL-1
Blue
.
Hasil analisis bioinformatika terhadap hasil sekuensing menunjukkan bahwa
fragmen gen target mempunyai tingkat homologi yang sangat tinggi terhadap gen
LEAFY
pada variatas tanaman lain di basis data bank gen. Karakterisasi gen target
diawali dengan isolasi RNA beberapa jaringan tanaman kakao kemudian
diamplifikasi dengan teknik RT-PCR. Amplifikasi dengan suhu annealing 62 ºC
serta perbanyakan sebanyak 33 siklus berhasil menunjukkan keberadaan gen
ABSTRACT
RATNA DEWI ESKUNDARI. Isolation and Characterization
LEAFY
Gene
Fragment from Cacao’s Flower Cushion. Under the direction of EDY
DJAUHARI PURWAKUSUMAH and TETTY CHAIDAMSARI.
Cocoa plant productivity in Indonesia is still low. Information and knowledge
at the molecular level about the process of development of regenerative organs of
cocoa can be an effective solution to problems associated with the regenerative
low productivity of cocoa plants.
LEAFY
gen is key gene in the shoot meristem
transition to flower one. The purposes of the research were to isolate
LEAFY
gene
from cacao’s flower cushion and analyse its expresion in several cacao’s tissues.
Isolation of
LEAFY
gene using
Reverse Transcriptase Polymerase Chain Reaction
(RT-PCR) technique showed that three spesific primer pairs designed based
conserved area can amplify the target gene. Futhermore this fragment were
inserted into pGEM-T
Easy
vector then transformed into competent cell
E. Coli
XL-1
Blue
.
The result of bioinformatic analysis showed that the fragment of target
gene has a high homology to
LEAFY
gene of other plants in gene bank’s database.
Characterization of target gene was initiated with RNA isolation phase from
several cacao’s tissues then those were amplified with RT-PCR technique.
Amplification at 62 ºC of annealing temperature for 33 cycles was successful to
ISOLASI DAN KARAKTERISASI FRAGMEN GEN
LEAFY
DARI BANTALAN BUNGA KAKAO
RATNA DEWI ESKUNDARI
Skripsi
sebagai salah satu syarat untuk memperoleh
Sarjana Sains pada
Program Studi Biokimia
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2010
Judul Skripsi : Isolasi dan Karakterisasi Fragmen Gen
LEAFY
dari Bantalan
Bunga Kakao
Nama : Ratna Dewi Eskundari
NRP
: G84051829
Disetujui
Komisi Pembimbing
Drs. Edy Djauhari Purwakusumah, M.Si Dr. Tetty Chaidamsari, M.Si
Ketua Anggota
Diketahui
Dr. I Made Artika, M. App. Sc
Ketua Departemen Biokimia
PRAKATA
Puji dan syukur penulis panjatkan kepada Alloh SWT karena berkat rahmat
dan karunia-Nya penulis dapat menyelesaikan penelitian ini. Penelitian ini
berjudul Isolasi dan Karakterisasi Fragmen Gen
LEAFY
dari Bantalan Bunga
Kakao (
Theobroma cacao
L.). Kegiatan penelitian ini dilakukan di Laboratorium
Biologi Molekuler dan Rekayasa Genetika, Balai Penelitian Bioteknologi
Perkebunan Indonesia (BPBPI) dari bulan Februari hingga Juli 2009.
Penulis mengucapkan terima kasih kepada semua pihak yang telah membantu
dalam penyelesaian penelitian ini, antara lain kepada Drs. Edy Djauhari
Purwakusumah, M.Si. selaku pembimbing utama, Dr. Tetty Chaidamsari, M.Si.
selaku pembimbing kedua, Dr. Darmono Taniwiryono, M.Sc. sebagai Kepala
BPBPI. Tak lupa penulis menyampaikan terima kasih kepada orang tua dan adik
penulis untuk semua doa, dukungan, dan bimbingan yang sangat berarti bagi
penulis. Ucapan terima kasih juga penulis sampaikan kepada Alm. Mardadi
Danusaputro (eyang kakung) yang selalu memberikan semangat untuk terus
belajar kepada penulis. Penulis juga berterima kasih kepada Mbak Nina, Mbak
Riana, Mbak Herti, Mbak Alin, Bu Rita, Bu Nova, seluruh staf BPBPI,
teman-teman Biokimia ‘42(khususnya Mbak Saeli, Mbak Izzah, dan Mbak Ellen), Mbak
Oca, dan juga teman-teman di kos Ananda. Ungkapan terima kasih juga
disampaikan kepada Hariyanto yang telah memberikan semangat untuk tidak
pernah menyerah dalam menghadapi ujian hidup.
Penulis menyadari masih banyak kekurangan dan kesalahan yang harus
diperbaiki, namun harapan penulis semoga penelitian ini dapat memberikan
manfaat bagi semua orang yang membacanya.
Bogor, Februari 2010
RIWAYAT HIDUP
Penulis dilahirkan di Wonogiri pada tanggal 23 Agustus 1987 dari pasangan
Drs. Tri Kuncoro, M. Pd dan Esti Rahayu, S. Pd. Penulis merupakan anak pertama
dari dua bersaudara.
Tahun 2005 penulis lulus dari SMA Negeri 1 Wonogiri dan melanjutkan
pendidikannya di IPB melalui jalur Undangan Seleksi Masuk IPB. Penulis
memilih jurusan Biokimia, Departemen Biokimia, Fakultas Matematika dan Ilmu
Pengetahuan Alam sebagai pilihan studi di IPB. Selama mengikuti perkuliahan di
IPB, penulis pernah menjadi asisten praktikum Keteknikan Asam Nukleat (KAN)
pada tahun 2008/2009 Penulis melakukan Praktik Lapang di Laboratorium
Mikrobiologi, Balai Besar Pengembangan Teknologi Tepat Guna Lembaga Ilmu
Pengetahuan Indonesia (B2PTTG-LIPI) Subang dengan judul Uji Antibakteri
DAFTAR ISI
Halaman
DAFTAR GAMBAR ... ix
DAFTAR TABEL ... ix
DAFTAR LAMPIRAN ... x
PENDAHULUAN ... 1
TINJAUAN PUSTAKA
Kakao ... 1
Gen Pembungaan ... 2
Isolasi dan Karakterisasi Gen ... 3
Polymerase Chain Reaction
(PCR) ... 3
Reverse Transcriptation Polimerase Chain Reaction
(RT-PCR) ... 5
BAHAN DAN METODE
Bahan dan Alat ... 5
Metode ... 6
HASIL DAN PEMBAHASAN
Isolasi RNA Total Bantalan Bunga Kakao ... 8
Sintesis Utas Pertama cDNA dan Amplifikasi Fragmen gen
LEAFY
.... 8
Kloning Fragmen Gen
LEAFY
... 9
Analisis Urutan Basa Fragmen Gen Terklon ... 10
Karakterisasi gen
LEAFY
pada Berbagai Jaringan ... 11
SIMPULAN DAN SARAN ... 12
DAFTAR PUSTAKA ... 13
DAFTAR GAMBAR
Halaman
1 Bantalan bunga kakao, bagian bunga kakao, dan diagram bunga ... 2
2 Model ABC pada perkembangan bunga ... 2
3 Peta plasmid pGEM-T
Easy
... 3
4 Tahap amplifikasi DNA dengan PCR ... 4
5 Tahap yang tejadi pada RT-PCR ... 5
6 Hasil elektroforesis RNA total bantalan bunga kakao ... 8
7 Hasil PCR gen
LEAFY
pada elektroforesis gel agarosa 1% ... 9
8 Koloni biru putih hasil kloning fragmen gen
LEAFY
... 9
9 Hasil elektroforesis PCR koloni primer LEAFY-F1 ... 10
10 Hasil elektroforesis PCR koloni primer dgLFY-F1 ... 10
11 Hasil elektroforesis RNA berbagai jaringan tanaman kakao ... 12
12 Amplifikasi berbagai jaringan tanaman kakao siklus 30, 33, dan 35 ... 12
DAFTAR TABEL
Halaman
1 Hasil analisis BLASTX dengan primer LEAFY-F1 + TcLFY R971 ... ..11
2 Hasil analisis BLASTX gen
LEAFY
lengkap ... ..11
DAFTAR LAMPIRAN
Halaman
1 Tahap penelitian ... 16
2 Primer -primer spesifik fragmen gen
LEAFY
... 17
3 Program PCR untuk amplifikasi fragmen gen
LEAFY
... 18
4 Hasil analisis BLASTX dengan primer LEAFY-F1 + TcLFY R971 ... 19
5 Hasil analisis BLASTX fragmen gen
LEAFY
lengkap ... 20
6 Hasil analisis kuantitatif RNA beberapa jaringan tanaman kakao ... 21
7 Primer -primer spesifik fragmen gen
LEAFY
... 21
8 Elektroforegram gen
LEAFY
dengan primer LEAFY-F1 + TcLFY R
971... 23
9 Elektroforegram gen
LEAFY
dengan primer LEAFY-F1 + TcLFY R
260full.... 24
PENDAHULUAN
Kakao (Theobroma cacao L.) merupakan salah satu tanaman perkebunan yang mempunyai nilai ekonomi tinggi sebagai komoditi ekspor. Indonesia termasuk dalam tiga besar produsen kakao dunia bersama Ghana dan Pantai Gading. Indonesia menghasilkan 456.500 ton buah kakao dari sekitar luas ares 0,6 juta hektar. Lebih dari 70% produksi kakao Indonesia diekspor ke Eropa dan Amerika Serikat dengan nilai US$ 503 juta (Direktorat Jendral Bina Produksi Perkebunan 2001). Selain ke Eropa dan Amerika Serikat, kakao Indonesia juga diekspor ke Singapura dan Rusia. Tingginya permintaan terhadap kakao serta harga yang bagus dan stabil dari kakao di pasar dunia mendorong usaha untuk meningkatkan produktivitas tanaman kakao.
Kebutuhan kakao yang setiap tahun meningkat menyebabkan berbagai provinsi di Indonesia melakukan ekstensifikasi areal penanaman kakao guna meningkatkan produksi nasional. Salah satu provinsi yang sedang melakukan ekstensifikasi adalah Sumatera Barat dan direncanakan menjadi sentra kakao nasional pada masa yang akan datang. Adanya program ekstensifikasi areal penanaman kakao, diharapkan dapat meningkatkan produksi kakao nasional sehingga dapat bersaing dengan negara produsen lainnya. Indonesia harus menyediakan tanaman kakao yang mempunyai produktivitas tinggi dan kualitas biji yang baik untuk mempertahankan sebagai negara produsen kakao (Wardojo 1992).
Produktivitas tanaman kakao di Indonesia masih tergolong rendah. Dua masalah besar yang dapat menurunkan produksi adalah serangan penggerek buah kakao (PBK) dan pembungaan yang tidak konsisten dengan layu pentil. Usaha untuk meningkatkan produktivitas tanaman kakao mendorong studi molekuler gen-gen pembungaan pada tanaman kakao. Informasi dan pengetahuan pada tingkat molekuler tentang proses perkembangan organ regeneratif kakao dapat menjadi pemecahan efektif untuk masalah regeneratif yang berhubungan dengan rendahnya produktivitas tanaman kakao.
Isolasi dan karakterisasi dari lokus yang berperan dalam pembentukan bunga dapat menunjukkan gen-gen regulator bunga berinteraksi dalam perkembangan bunga.
Gen-gen pembungaan yang penting pada identitas meristem selama fase reproduktif di antaranya LEAFY (LFY), APETALA 1 (AP1),
APETALA 2 (AP2), dan CAULIFLOWER
(CAL). Mutasi pada gen-gen tersebut dapat mengakibatkan penghambatan pada proses pembungaan atau perubahan bentuk pada identitas bunga (Sartika 2006).
Salah satu gen yang berperan utama dalam pembentukkan bunga adalah gen
LEAFY. Berdasarkan penelitian yang
dilakukan oleh tim peneliti dari Universitas Salk di Amerika Serikat, dapat diketahui bahwa tanaman akan cepat berbunga dan tunas lateral akan menjadi bunga tunggal apabila gen LEAFY dibuat terekspresi (“on” position). Gen LEAFY ini pertama kali dianalisis pada tanaman rumput liar Arabidopsis thaliana (Henahan 1995). Di dalam Arabidopsis, perubahan dari fase vegetatif ke reproduktif dikontrol secara ketat oleh banyak faktor. Gen LEAFY mempunyai posisi sentral dan merupakan gen kunci yang diperlukan untuk pembentukan bunga normal.
Pada penelitian ini dipelajari karakterisasi gen LEAFY dari bantalan bunga kakao menggunakan primer-primer yang telah dirancang berdasarkan daerah terkonservasi. Penelitian ini bertujuan mengisolasi dan mengkarakterisasi gen LEAFY dari bantalan bunga kakao. Hipotesis pada penelitian ini adalah gen LEAFY dapat diisolasi dari bantalan bunga kakao. Hasil penelitian ini diharapkan dapat memberikan informasi mengenai gen LEAFY pada kakao sehingga produksi kakao dapat meningkat.
TINJAUAN PUSTAKA
Kakao
Tanaman kakao (Theobroma cacao L.) merupakan salah satu tanaman perkebunan yang dapat tumbuh baik di Indonesia, terutama di dataran rendah. Klasifikasi lengkap tanaman kakao adalah divisi Spermatophyta, subdivisi Angiospermae, kelas Dicotyledone, ordo Malvales, famili Sterculiaceae, genus Theobroma, dan spesies Theobroma cacao L. Tanaman kakao dikenal di Indonesia sejak tahun 1560, namun baru menjadi komoditi yang penting sejak tahun 1951 (Putri 2004). Kakao merupakan tumbuhan tahunan (perennial) berbentuk pohon, di alam dapat mencapai ketinggian 10 m. Meskipun demikian, dalam
pembudidayaan tingginya dibuat tidak lebih dari 5 m dengan tajuk menyamping yang meluas. Hal ini dilakukan untuk memperbanyak cabang produktif.
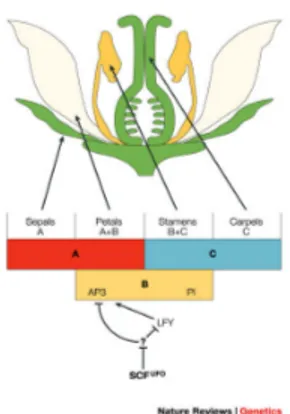
Pohon kakao yang telah mencapai umur di atas 3 tahun akan menghasilkan banyak bunga pada musim tertentu. Bunga muncul dari bantalan bunga yaitu suatu jaringan meristem pada batang dan cabang kayu sehingga disebut bunga yang bersifat cauliflorous atau truncate (Gambar 1a). Seperti ditunjukkan Gambar 1b, bunga kakao mempunyai beberapa bagian bunga yaitu sepal, petal, kepala sari, tangkai sari, putik, dan ovarium. Bunganya berukuran kecil (diameternya sekitar 15 mm) dan berbentuk tangkai yang memanjang (Chaidamsari et al. 2006). Bunga kakao bersifat hermaprodit dan mempunyai susunan K5 C5 A5 + 5G(5) yaitu 5 sepal, 5 petal, 10 stamen, dan 5 karpel (Wood 1975). Kakao secara umum adalah tumbuhan menyerbuk silang dan memiliki sistem inkompatibilitas-sendiri. Walaupun demikian, beberapa varietas kakao mampu melakukan penyerbukan sendiri dan menghasilkan jenis komoditi dengan nilai jual yang lebih tinggi (Wood 1975). Penyerbukan juga dapat dibantu serangga kecil tetapi hanya sekitar 1-5% bunga yang sukses diserbuki dan berubah menjadi buah (Chaidamsari et al. 2006).
Gambar 1 Bantalan bunga kakao (a); bagian-bagian bunga kakao (b): Se, sepal; Pe, petal; Sta, kepala sari; Std, tangkai sari; Pi, putik; Ov, ovarium (Chaidamsari et al. 2006).
Gen Pembungaan
Gen-gen yang terlibat dalam pengaturan proses pembungaan biasanya tergolong pada kelompok gen MADS-box yang mempunyai peran kunci dalam spesifikasi identitas organ-organ bunga (Soltis et al. 2002). Model ABC dari perkembangan bunga angiospermae ditemukan oleh E. Coen dan Elliot Meyerowitz pada tahun 1991. Gen
pembungaan berfungsi dari fase awal pembentukan identitas pembungaan hingga pembentukan bunga.
Organ pembungaan pada tanaman tingkat tinggi diatur dalam empat kelompok yang terdiri atas sepal, petal, stamen, dan karpel (Gambar 2). Ekspresi gen kelas A spesifik membentuk sepal pada lingkaran pertama. Kombinasi gen kelas A dan B spesifik untuk pembentukan petal pada lingkaran kedua. Kombinasi gen-gen B dan C spesifik dalam pembentukan stamen pada lingkaran ketiga. Sedangkan ekspresi gen kelas C akan spesifik dalam pembentukan karpel pada lingkaran keempat. Penelitian mengenai proses dan gen-gen yang terlibat dalam pembungaan telah banyak dilakukan pada tanaman model seperti Arabidopsis thaliana dan Antirrhinum majus, namun pada tanaman berkayu penelitian ini masih jarang dilakukan.
Karakteristik awal dari dua jenis gen kakao yang mengkode faktor transkripsi dari kelas MADS-box diperkirakan mempengaruhi regulasi waktu pembungaan dan pembentukan bunga. Berdasarkan hasil penelitian yang dilakukan oleh Chaidamsari
et al. 2006 dapat diketahui bahwa
APETALA1 (TcAP1) Kakao gen homolog dari Arabidopsisberpengaruh baik terhadap koregulasi pada inisiasi pembungaan dan pada determinasi identitas dua kelopak bagian luar organ pembungaan seperti sepal dan petal. Kakao homolog dari AGAMOUS Arabidopsis gene (TcAG) berpengaruh pada pembentukan kelopak bagian dalam dari organ pembungaan yaitu tangkai sari, benang sari, dan ovarium. TcAG juga terekspresi pada dinding buah kakao pada berbagai tingkat perkembangannya.
Gambar 2 Model ABC pada perkembangan bunga (Sullivan 2003 ).
Isolasi dan Karakterisasi Gen
Serangkaian studi genetik yang dikombinasikan dengan studi kimia, yang dilaksanakan oleh banyak peneliti dari berbagai lembaga dengan waktu yang cukup lama, telah membawa kepada kesimpulan bahwa materi genetik disusun oleh asam nukleat yaitu asam deoksiribonukleat (DNA) dan asam ribonukleat (RNA). Materi genetik sering diperlukan sebagai sumber gen untuk diklon (Jusuf 2001). Untuk keperluan ini, proses isolasi mutlak dilakukan sebab isolasi materi genetik merupakan proses untuk mengekstrak (memisahkan) materi genetik dari sel yang mengandung berbagai komponen seperti plasma sel, organel sel, dinding sel, dan membran sel. Proses isolasi RNA secara garis besar terdiri atas tiga tahap yaitu pemecahan sel dan solubilisasi membran, isolasi dan pemurnian RNA, dan pemekatan RNA.
Tahap pemecahan sel dan solubilisasi membran sebaiknya dilakukan secara cepat dan tuntas. Bufer lisis hendaknya secepat mungkin dapat mencapai isi sel untuk menginaktifkan kerja ribonuklease. Tahap isolasi dan pemurnian RNA membutuhkan larutan untuk menghilangkan kontaminan seperti protein dan DNA. Ekstraksi fenol dan kloroform dapat menghilangkan kontaminan protein sedangkan penghilangan kontaminan DNA dapat dilakukan dengan presipitasi litium klorida (LiCl). Pemekatan RNA merupakan tahap akhir dalam proses pemurnian RNA. Cara yang paling efektif untuk memekatkan RNA adalah melalui presipitasi menggunakan berbagai kombinasi garam dan alkohol. Asam nukleat akan membentuk kompleks dengan garam sehingga membantu pengendapan oleh etanol (Liu et al. 1998).
Setelah molekul DNA rekombinan berhasil dikonstruksi, tahap berikutnya adalah pengklonan gen. Tahap ini mencakup ligasi, transformasi, dan PCR koloni. Vektor kloning merupakan molekul DNA yang dapat bereplikasi secara mandiri dan dapat digunakan sebagai pembawa molekul materi genetik lain yang tidak mempunyai kemampuan untuk bereplikasi sendiri di dalam sel (Glick & Pasternak 1994). Vektor kloning (plasmid) umumnya membawa satu atau sejumlah gen yang dapat berupa gen pembawa sifat resisten terhadap antibiotik dan penyandi enzim restriksi. Sebagian besar percobaan pengklonan menggunakan bakteri Escherichia coli (E.coli) sebagai inang sehingga vektor yang tersedia untuk
bakteri ini jumlah dan ragamnya paling tinggi serta dapat digunakan untuk mengekspresikan gen asing dalam rangka memproduksi protein tertentu yang bernilai komersial dalam skala besar.
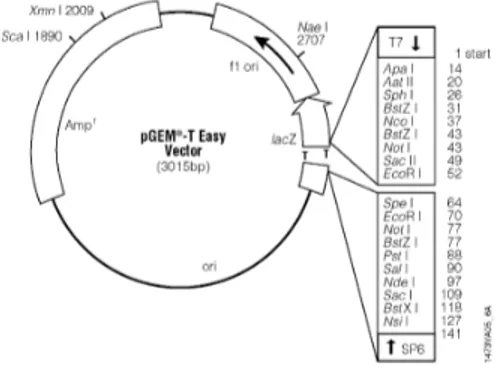
Salah satu contoh plasmid untuk bakteri E.coli adalah pGEM-T Easy (Gambar 3). Plasmid pGEM-T Easy berukuran 3015 bp dan resisten terhadap Ampisilin. Selain itu, plasmid pGEM-T Easy mempunyai banyak situs restriksi seperti situs restriksi untuk enzim Apa I, Sac I, BstX I, dan Nsi I.
Setelah tahapan kloning selesai, maka plasmid diisolasi dan dimurnikan. Pemurnian plasmid dilakukan supaya didapatkan plasmid DNA yang membawa gen target dalam keadaan murni terbebas dari kontaminan komponen sel inang E.coli. Gen target yang terdapat pada plasmid selanjutnya disekuensing dan hasil sekuensingnya dianalisis kemudian dibandingkan dengan gen referensi yang terdapat dalam basis data bank gen. Berdasarkan berbagai informasi analisis molekuler gen-gen pembungaan dan perkembangan data-data berupa urutan DNA dan protein, maka penggunaan ilmu komputer dan teknologi informasi sangat penting untuk menganalisis data, khususnya gen-gen yang berupa informasi molekuler (urutan DNA atau protein) sehingga dapat menghasilkan suatu informasi baru (Sartika 2006).
Gambar 3 Peta plasmid pGEM-T Easy (Promega 2009).
Polymerase Chain Reaction (PCR)
PCR merupakan teknik yang digunakan untuk mengamplifikasi sekuen asam nukleat menggunakan polimerisasi berulang dari sekuen DNA. Teknik ini disusun dan dipraktikkan oleh Kary B. Mullis pada
pertengahan tahun 1985. Kesederhanaan dan tingginya tingkat kesuksesan amplifikasi sekuen DNA yang diperoleh menyebabkan teknik ini semakin luas penggunaannya (Saiki et al. 1988).
Teknik PCR yang ditemukan Kary B. Mullis menggunakan fragmen Klenow DNA Polimerase I E. coli untuk mensintesis sekuen DNA yang baru. Namun, enzim ini mempunyai keterbatasan, yaitu terdenaturasi pada suhu tinggi (suhu denaturasi utas ganda DNA) pada tahap pertama proses PCR, sehingga keaktifan enzim tersebut hilang saat proses pemanjangan (elongation). Oleh karena itu, enzim harus ditambahkan setiap tahap, setelah suhu untuk proses ekstensi diturunkan sampai 37 °C. Hal ini membuat PCR berjalan lambat dan cenderung membuat kesalahan (Saiki et al. 1988). Tetapi pada saat ini telah digunakan Taq polimerase yang diisolasi dari bakteri Thermus aquaticus yang tergolong Archea termofilik. Enzim ini bersifat tahan panas mengingat proses PCR yang melibatkan suhu tinggi sehingga aplikasi PCR pada saat ini menjadi lebih luas dan proses kerjanya menjadi lebih cepat.
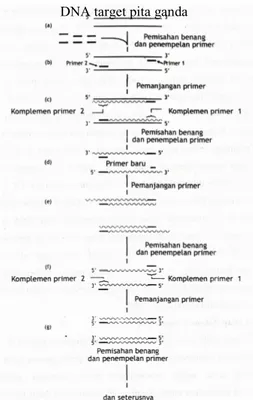
Proses PCR memerlukan dua macam oligonukleotida (primer) yang masing-masing berhibridisasi dengan salah satu utas DNA yang akan diamplifikasi pada sisi yang berbeda dan keempat deoksinukleosida trifosfat (dNTP) dalam jumlah yang cukup, serta suatu DNA polimerase khusus yang tahan panas (Koolman & Rohmn 2005). Reaksi PCR merupakan sebuah siklus yang berlangsung dalam tiga tahapan. Siklus diawali dengan tahap denaturasi pada suhu tinggi (90-95°C) yang mengakibatkan untai ganda DNA mengalami pemisahan menjadi untai tunggal. Tahap selanjutnya adalah penempelan primer (annealing) biasanya terjadi pada suhu 45-55°C, primer akan melekat pada DNA cetakan sesuai dengan komplementasi basa nukleotidanya. Tahap yang terakhir yaitu pemanjangan nukleotida (elongation), pembentukan molekul DNA menggunakan molekul-molekul dNTP yang merupakan komponen dari cetakan pada suhu 70-75°C (Innis et al. 1991).
Tahap pertama, yaitu denaturasi utas ganda DNA menjadi dua utas tunggal DNA dilakukan melalui pemanasan pada temperatur tertentu atau melalui pemberian bahan kimia. Temperatur yang biasa digunakan untuk tahapan denaturasi antara 93-96°C atau hingga 98°C jika polimerase ekstrim termostabil yang digunakan.
Temperatur denaturasi tersebut dijaga selama 1-9 menit. Penjagaan temperatur dilakukan untuk meyakinkan bahwa DNA cetakan dan primer benar-benar terdenaturasi (ikatan hidrogen antara pasangan basa dari utas ganda DNA terputus sehingga DNA menjadi dua utas tunggal yang terpisah), DNA polimerase berperan dalam memasangkan setiap dNTP yang komplemen terhadap basa pada utas tunggal DNA setelah primer menempel pada utas tunggal DNA tersebut.
Tahap denaturasi kemudian dilanjutkan dengan tahap penempelan primer. Temperatur reaksi pada tahap ini diturunkan sehingga primer dapat menempel pada utas tunggal DNA cetakan. Gerak Brown menyebabkan primer bergerak di sekeliling campuran reaksi dan ikatan-ikatan hidrogen DNA-DNA dibentuk serta dirusak secara konstan antara primer dan cetakan. Ikatan yang stabil hanya dibentuk pada saat sekuen primer komplemen dengan sekuen cetakan dan basa yang komplemen menempel dengan bantuan Taq polimerase sehingga sintesis DNA baru dimulai. Langkah amplifikasi DNA dapat diamati pada Gambar 4.
DNA target pita ganda
Gambar 4 Tahap amplifikasi DNA dengan PCR (Reece 2004).
Polimerasasi nukleotida yang komplementer dengan urutan nukleotida DNA cetakan terjadi pada tahap selanjutnya, yaitu tahap pemanjangan nukleotida. Reaksi polimerisasi dimulai dari ujung 5’-fosfat dan berakhir pada ujung 3’ gugus hidroksil (OH) (Newton 1995). Tahap pemanjangan nukleotida umumnya terjadi pada temperatur 72°C karena pada temperatur tersebut Taq polimerase bekerja optimal untuk sintesis DNA. Tahap ini biasanya dipertahankan selama 2 menit untuk memberi kesempatan terjadinya sintesis DNA (Innis et al. 1991). Tahap elongasi akhir selama 5-15 menit setelah siklus terakhir selesai diperlukan untuk meyakinkan bahwa proses pemanjangan nukleotida telah berlangsung secara utuh.
Reverse Transcriptase PCR (RT-PCR)
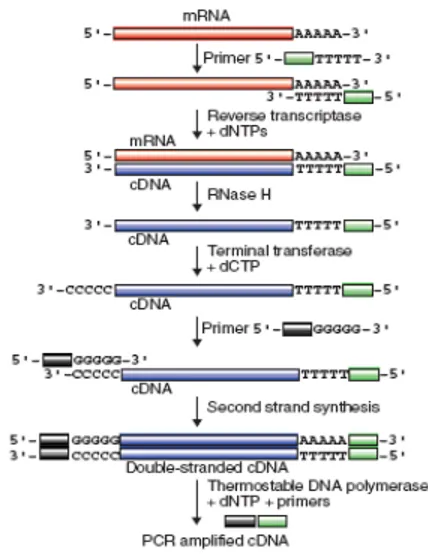
RT-PCR merupakan metode amplifikasi cDNA (complementary DNA), yaitu DNA hasil proses transkripsi balik menggunakan RNA sebagai cetakan. Teknik ini tergolong sensitif dan antara lain dapat digunakan untuk pembuatan pustaka cDNA, mengidentifikasi mutasi maupun polimorfisme pada sekuen hasil transkripsi, menentukan tingkat ekspresi gen, dan mendeteksi keberadaan molekul RNA dalam suatu sampel. Teknik RT-PCR yang dikembangkan sangat spesifik sehingga dapat digunakan walaupun jumlah RNA yang akan dianalisis sedikit (O’Connell 2002).
Transkripsi balik (reverse transcription) terhadap molekul mRNA dilakukan terlebih dahulu pada proses RT-PCR ini sehingga diperoleh molekul cDNA (complementary DNA). Molekul cDNA tersebut selanjutnya digunakan sebagai cetakan untuk proses PCR selanjutnya (Sambrook et al. 2001). Prinsip dasar dari teknik ini adalah menggunakan suatu enzim reverse transcriptase (RT) yang akan mensintesis suatu cDNA dari mRNA yang digunakan sebagai cetakan. Beberapa enzim RT yang digunakan secara komersial diisolasi dari sel yang terinfeksi oleh Moloney murine
leukimia virus (MMLV) dan Avian
myleoblastosis virus (AMV). Proses sintesis cDNA utas ganda terdapat pada Gambar 5.
Saat proses transkripsi balik, primer oligo dT akan menempel pada bagian poli-A mRNA. Enzim RT mengkatalisis pembentukkan first strand (utas pertama) cDNA dengan memasangkan basa (dNTP) yang komplementer terhadap basa pada
mRNA. RNase H akan memisahkan mRNA dari molekul hibrid yang terbentuk. Enzim terminal transferase kemudian mensintesis sekuen C berulang (ekor poli-C) pada ujung 3’ cDNA sebagai situs untuk penempelan primer oligo-dG. Selanjutnya terjadi pembentukkan utas cDNA kedua sehingga terbentuk cDNA utas ganda (Reece 2004).
Gambar 5 Proses-proses yang terjadi pada tahap RT-PCR (Reece 2004).
BAHAN DAN METODE
Bahan dan Alat
Bahan-bahan yang digunakan pada penelitian ini adalah sampel bantalan bunga kakao dari Balai Penelitian dan Bioteknologi Perkebunan Indonesia (BPBPI), polivinilpoli-pirolidon (PVPP), dietilpirokarbonat (DEPC), bufer (tris HCl 1 M pH 8.0, NaCl 5 M, EDTA 0.5 M pH 8.0, N-cetyl-N,N,N-trimetilamonium bromida (CTAB), dan β-merkaptoetanol), etanol
absolut, etanol 70%,
kloroform:isoamilalkohol (24:1), ddH2O,
fenol:kloroform:isoamilalkohol (25:24:1), fenol, natrium asetat 3 M pH 5.2, LiCl 10 M, N2 cair, Transcriptor First Strand cDNA
Synthesis kit (Invitrogen), cetakan utas pertama bantalan bunga kakao, primer LEAFY-F1, primer dgLFY-F1, primer TcLFY R971, primer TcLFY R380full, primer
TcLFY R260full, primer TcLFY-09F1, primer
TcLFY-GW Rev4, buffer RT-PCR, MgCl2,
dNTP, Taq polimerase, air molekuler (MW), Extraction and Purification DNA kit (Invitrogen), vektor pGEM-T Easy (Promega), T4 DNA ligase, bufer ligasi, sel
kompeten E.coli XL-1 Blue, media SOC, media LA, Ampisilin (100ppm), X-Gal (40ppm), IPTG 0.1mM, agarosa (Roche), buffer TBE 0.5X, etidium bromida 1% (w/v), larutan pemberat (brom fenol biru 2.5%, sukrosa 40%), marker 1 kb plus DNA ladder (Invitrogen), High Pure Plasmid Isolation Kit (Roche).
Alat-alat yang digunakan pada penelitian ini adalah mortar, lemari asam, sentrifus Beckman Allegra 64R, sentrifus Eppendorf 5417R, penangas air, tabung sentrifus 30 mL, tabung mikro 2 mL dan 1.5 mL, pipet Mohr, pipet Eppendorf, Decby freezer -40°C, Sansio freezer -20°C, spektrofotometer UV-VIS Beckman Coulter-DU 530, sisir dan cetakan agar, bak elektroforesis, adaptor 100 Volt, UV T2201 (Sigma), mesin PCR (Biometra T-Personal), DNA speed vacuum 110 savant, inkubator bergoyang, lemari laminar, otoklaf, pH meter, cawan petri, gelas piala, labu Erlenmeyer, dan gelas ukur.
Metode
Isolasi RNA Total Bantalan Bunga Kakao
RNA total diisolasi dari bantalan bunga kakao dengan menggunakan prosedur Chaidamsari et al. (2005). Sebanyak 1.5 gram sampel dihaluskan dengan nitrogen cair beserta satu sudip PVPP. Setelah itu ditambahkan 15 mL bufer ekstraksi dan diinkubasi dalam penangas air pada suhu 65°C selama 1 jam. Selanjutnya sampel diekstraksi dengan kloroform:isoamilalkohol (24:1) dengan volume yang sama sebelum penambahan. Campuran ekstraksi tersebut kemudian disentrifus dengan kecepatan 19.621 g selama 15 menit pada suhu ruang, supernatan diambil lalu dilakukan ekstraksi ulang menggunakan fenol:kloroform: isoamilalkohol (25:24:1), dan disentrifugasi pada kondisi yang sama. Supernatan diekstraksi kembali dengan kloroform:isoamilalkohol (24:1) lalu disentrifugasi pada kondisi yang sama sebanyak dua kali pengulangan. Supernatan selanjutnya ditambahkan LiCl hingga konsentrasi akhir 2M kemudian diendapkan pada 4°C selama semalam.
Endapan RNA hasil pengendapan semalam lalu disentrifugasi dengan kecepatan 19.621 g selama 30 menit pada 4°C, pelet yang dihasilkan dilarutkan dengan 750 µL ddH2O DEPC dan dipindahkan ke
dalam tabung mikro 2 mL, kemudian larutan tersebut diekstraksi dengan fenol sebanyak 1
volume (750 µL) lalu disentrifugasi dengan kecepatan 13.362 g selama 15 menit pada 4°C. Selanjutnya supernatan yang didapatkan dipindahkan ke tabung mikro 2 mL yang baru dan diekstrak kembali dengan campuran fenol: kloroform : isoamilalkohol (25:24:1) sebanyak volume supernatan yang didapatkan lalu disentrifugasi dengan kecepatan 13.362 g selama 15 menit pada 4°C. Supernatan yang dihasilkan diekstraksi kembali dengan kloroform:isoamilalkohol (24:1) lalu disentrifugasi pada kondisi yang sama. Supernatan yang didapatkan dipindahkan ke dalam tabung mikro 1.5 mL lalu ditambahkan Na-asetat 3M pH 5.8 sebanyak 0.1 kali volume supernatan dan etanol absolut 3 kali volume supernatan yang diperoleh. Campuran kemudian diendapkan selama satu malam pada suhu -70°C. Setelah satu malam, campuran disentrifugasi dengan kecepatan 13.362 g selama 30 menit pada 4°C, pelet yang diperoleh selanjutnya dicuci dengan 100µL etanol 70%, kemudian disentrifugasi lagi pada 13.362 g pada suhu 4ºC selama 2 menit. Pelet kemudian dikeringkan, disentrifus pada 13.362 g pada suhu 4ºC selama 2 menit, dan terakhir dilarutkan dengan 30 µL ddH2O DEPC.
Uji Kuantitatif dan Kualitatif RNA Total
Larutan RNA hasil isolasi dianalisis kemurnian dan konsentrasinya. Kemurnian RNA hasil isolasi ditentukan dengan dua cara. Cara pertama adalah membandingkan absorbansi RNA sampel tersebut pada panjang gelombang 260 nm dengan 280 nm dan juga dengan panjang gelombang 230 nm. Kemurnian RNA dilihat dari perbandingan A260/280 dan A260/230 yang berkisar 1.8-2.0 (Sambrook et al. 1989). Cara kedua adalah dengan dielektroforesis dalam gel agarosa 1% pada tegangan listrik 25 Volt selama 2 jam dan diamati jumlah pita yang terbentuk. Konsentrasi RNA dihitung dengan perbandingan bahwa nilai pada serapan 260 nm merupakan serapan RNA dengan konsentrasi 40 µg/mL.
Sintesis utas pertama cDNA
Sintesis utas pertama dilakukan untuk mendapatkan cDNA dari RNA total hasil isolasi. Ke dalam tabung mikro 200 µL dimasukkan 1 µL dNTP mix, RNA total dengan konsentrasi 2 µg/mL, 1 µL oligo dT, dan ddH2O DEPC sehingga total larutan
menjadi 10 µL. Campuran tersebut diinkubasi pada suhu 65°C selama 5 menit
pada mesin PCR. Setelah 5 menit, campuran diangkat dan dimasukkan ke dalam es. Selanjutnya ke dalam tabung mikro tadi dimasukkan 2 µL transcriptor RT reaction buffer 5X, 4 µL MgCl2, 2 µL DTT, dan 1 µL
RNase out, kemudian campuran tersebut diinkubasi kembali pada suhu 42°C selama 2 menit. Setelah itu ditambahkan 1 µL transcriptor reverse transcriptase dan diinkubasi kembali pada suhu 42°C selama 50 menit serta 70ºC selama 15 menit. Sebanyak 1 µL RNase H selanjutnya dimasukkan ke campuran tersebut, diinkubasi lagi pada suhu 37°C selama 20 menit, dan terakhir disimpan di dalam lemari es dengan suhu -20°C.
Isolasi dan Amplifikasi Fragmen Gen LEAFY dengan Metode RT-PCR
Gen LEAFY diisolasi dan diamplifikasi menggunakan utas pertama bantalan bunga kakao. Proses ini menggunakan kit dari Invitrogen. Ke dalam tabung mikro 200 µL kosong dimasukkan 3 µL MW, 1 µL cetakan, 1 µL primer F, dan 1 µL primer R kemudian dicampur. Selanjutnya dimasukkan sebanyak 14.5 µL MW, 2.5 µL bufer, 0.75 µL MgCl2, 1 µL dNTPs, dan
0.25 µL Taq polimerase ke tabung mikro tersebut sehingga volume total larutan menjadi 25 µL. Larutan tersebut kemudian dimasukkan ke dalam mesin PCR yang telah diatur programnya sebanyak 35 siklus.
Proses amplifikasi dilakukan dengan program PCR seperti yang tertera pada Lampiran 3. Setelah itu disimpan di lemari es pada suhu -20 °C. Hasil PCR kemudian dielektroforesis menggunakan elektroforesis gel agarosa 1% (Sambrook et al. 1989) dengan tegangan listrik 75 Volt selama 1-1.5 jam.
Ekstraksi dan Purifikasi DNA hasil PCR
Ekstraksi dan purifikasi DNA hasil PCR dilakukan dengan menggunakan kit dari Invitrogen. Bagian gen yang memiliki fragmen gen dipotong menggunakan pisau scalpel dan disimpan pada suhu 4°C. Selanjutnya potongan gel dipindahkan ke dalam tabung mikro 2 mL, ditimbang, dan dilarutkan dengan GS1 sebanyak 3x volume gel. Suspensi selanjutnya diinkubasi pada suhu 50°C selama 15 menit. Larutan selanjutnya dipindahkan ke dalam tabung baru yang telah ditambahkan kolom dan disentrifus pada 13.362 g selama 2 menit. Selanjutnya pelet yang didapatkan ditambahkan 500 µL GS1, diinkubasi pada
suhu ruang selama 1 menit, dan disentrifugasi lagi pada kondisi yang sama. Pelet yang didapatkan kemudian ditambahkan 700 µL W9, diinkubasi pada suhu ruang selama 5 menit, dan disentrifugasi lagi pada kondisi yang sama. Selanjutnya kolom dipindahkan ke tabung mikro 1.5 mL, dielusi tepat di bagian tengah dengan 30 µL bufer TE hangat (65-70°C), diinkubasi pada suhu ruang selama 1 menit, disentrifugasi pada kondisi yang sama, dan terakhir dielektroforesis pada gel agarosa 1%.
Kloning Fragmen Gen dengan pGEM-T Easy
Fragmen gen yang sudah dipurifikasi kemudian diligasi ke dalam pGEM-T Easy (Promega) dengan cara sebanyak 0.5 µL vektor pGEM-T Easy, 5 µL bufer ligasi, 3.5 µL insert, serta 1 µL T4 ligase dimasukkan ke dalam tabung mikro 200 µL dan diinkubasi selama semalam pada suhu 4 °C. Hasil ligasi sebanyak 10µL selanjutnya ditransformasikan ke dalam 200 µL sel kompeten E.coli XL-1 Blue secara perlahan dan disimpan di dalam es selama 30 menit. Setelah itu diberi kejut panas 42 °C selama 50 detik dan dimasukkan ke dalam es lagi selama 10 menit. Selanjutnya ditambahkan 800 µL medium LB + glukosa 20 mM tanpa antibiotik dan diinkubasi pada suhu 37 ºC selama 90 menit. Setelah 90 menit, suspensi sel transforman ditransfer pada media seleksi (medium LA + Ampisilin + IPTG + X-Gal) di cawan petri dan disebar. Medium kemudian diinkubasi pada 37°C selama semalam hingga terbentuk koloni putih biru. Seleksi transforman dilakukan dengan pengamatan terhadap koloni yang terbentuk. Koloni putih yang terbentuk selanjutnya diambil untuk PCR koloni dan duplikat koloni.
Konfirmasi Koloni Transforman yang Membawa Fragmen Sisipan
Konfirmasi koloni transforman dilakukan dengan teknik PCR koloni. PCR koloni dilakukan menggunakan primer universal M13F dan R serta koloni bakteri sebagai cetakan. Hasil PCR koloni diverifikasi pada gel agarosa. Koloni terpilih kemudian dikulturkan dalam medium LB cair yang telah ditambahkan Ampisilin dan diinkubasi pada inkubator bergoyang pada suhu 37°C 150 rpm.
Kultur bakteri yang tumbuh selanjutnya diisolasi DNA plasmidnya menggunakan
High Pure Plasmid Isolation Kit (Roche). Sebanyak 0.5-4.0 mL kultur bakteri dimasukkan ke dalam tabung mikro 2 mL dan disentrifugasi pada 5.939 g selama 1 menit. Pelet yang didapatkan selanjutnya diresuspensikan dengan 250 µL bufer suspensi, 250 µL bufer lisis, dan 350 µL bufer pengikat, diinkubasi di es selama 5 menit, dan disentrifugasi pada 15.682 g selama 10 menit. Supernatan yang didapatkan kemudian dipindahkan ke dalam filter dan disentrifugasi pada 15.682 g selama 1 menit. Pelet yang didapatkan selanjutnya ditambahkan 500 µL bufer pencuci I dan disentrifugasi pada 15.682 g selama 1 menit. Selanjutnya pelet yang didapatkan ditambahkan 700 µL bufer pencuci II dan disentrifugasi lagi pada 15.682 g selama 1 menit. Terakhir pelet yang didapatkan ditambahkan 30 µL bufer dan selanjutnya diverifikasi dengan gel agarosa 1%.
Sekuensing Gen Terklon
Pengurutan basa (sekuensing) fragmen gen terklon dilakukan di Charoen Phokphand, Jakarta. Primer yang digunakan untuk sekuensing adalah primer universal M13F dan M13R. Hasil sekuen fragmen gen terklon selanjutnya dianalisis lebih lanjut dengan program bioinformatika BLASTX.
HASIL DAN PEMBAHASAN
Isolasi RNA Total Bantalan Bunga Kakao
Isolasi RNA merupakan tahap awal untuk mendapatkan informasi yang berkaitan dengan ekspresi gen. Secara umum isolasi RNA lebih sulit dibandingkan dengan isolasi DNA karena RNA sangat mudah terdegradasi oleh RNAse (ribonuklease) sehingga membutuhkan perlakuan khusus seperti penggunaan DEPC untuk mengisolasinya. Penelitian ini menggunakan RNA total dari bantalan bunga kakao untuk mendapatkan gen LEAFY dari jaringan tersebut. Hal ini dikarenakan bunga kakao muncul dari bantalan bunganya. Bantalan bunga merupakan jaringan spesifik sehingga materi genetik yang diperoleh dari isolasi jaringan ini adalah RNA.
Isolasi RNA total bantalan bunga kakao dilakukan berdasarkan metode Chaidamsari et al. (2005). Setelah RNA berhasil diisolasi, selanjutnya dilakukan uji kuantitatif dan kualitatif RNA. Uji kuantitatif RNA
dilakukan dengan mengukur absorbansi RNA dengan spektrofotometer pada panjang gelombang 260 nm, 280 nm, dan 230 nm. Perbandingan nilai absorbansi 260 nm dengan 280 nm menunjukkan adanya kontaminan protein, sedangkan perbandingan nilai absorbansi 260 nm dengan 230 nm menunjukkan adanya kontaminan polisakarida. Nilai perbandingan yang menunjukkan tidak adanya pengotor berada pada kisaran 1.8-2.0 (Asif et al. 2000). Hasil pengukuran spektrometer terhadap RNA hasil isolasi pada penelitian ini menunjukkan angka 1.706 untuk nilai perbandingan absorbansi 260/280 nm, dan nilai 1.601 untuk nilai perbandingan absorbansi 260/230 nm. Nilai tersebut menandakan RNA masih memiliki pengotor protein dan polisakarida karena perolehan nilai absorban berada di bawah 1.8. Namun RNA masih dapat digunakan untuk penelitian selanjutnya. Hal ini ditunjang oleh hasil pengamatan kualitas RNA menggunakan elektroforesis gel agarosa 1% selama 1 jam dengan voltase 25 Volt (Gambar 6). Elektroforesis tidak menggunakan marker karena hanya bertujuan mengamati bahwa RNA hasil isolasi memiliki integritas yang baik dan tidak terdegradasi. Dua pita RNA yang terbentuk merupakan RNA ribosom (rRNA) dengan subunit 18s dan 28s. Perolehan dua pita RNA yang utuh menunjukkan bahwa isolasi RNA dari bantalan bunga kakao telah berhasil diisolasi.
Gambar 6 Hasil elektroforesis RNA total bantalan bunga kakao.
Sintesis Utas Pertama cDNA dan Amplifikasi Fragmen Gen LEAFY
RNA total hasil isolasi selanjutnya digunakan sebagai cetakan untuk mensintesis utas pertama cDNA. Konsentrasi RNA yang digunakan sebesar 250 ng/µL sehingga produk cDNA akan menghasilkan pendaran yang optimal saat divisualisasi dengan sinar UV setelah dilakukan elektroforesis gel agarosa 1%. Proses sintesis utas pertama cDNA dalam penelitian ini menggunakan kit dari Invitrogen. Selanjutnya utas pertama cDNA
28S
18S
hasil sintesis utas pertama digunakan sebagai cetakan untuk proses amplifikasi dengan teknik RT-PCR.
Ketepatan dalam perancangan primer akan menghasilkan amplifikasi yang optimal. Primer yang digunakan untuk mengenali gen LEAFY pada penelitian ini dirancang berdasarkan sekuen gen LEAFY pada Theobroma cacao L. di basis data bank gen. Primer yang digunakan dalam penelitian ini tersaji pada Lampiran 2. Amplifikasi fragmen gen LEAFY pada penelitian ini dilakukan dengan kombinasi pasangan-pasangan primer LEAFY-FI+TcLFY R971, LEAFY-F1+TcLFY
R260full, LEAFY-F1+TcLFY R380full,
dgLFY-F1+ TcLFY R971, dgLFY-F1+ TcLFY
R260full, dan dgLFY-F1+ TcLFY R380full.
Produk PCR yang dihasilkan selanjutnya dikonfirmasi pada elektroforesis gel agarosa 1%. Hasil elektroforesis dapat dilihat pada Gambar 7. Terlihat bahwa fragmen gen
LEAFY dengan primer LEAFY-F1+TcLFY
R971 menghasilkan pita yang berukuran
sekitar 400 bp. Pasangan primer LEAFY-F1+TcLFY R380full menghasilkan pita
sebesar 500 bp sedangkan pasangan primer LEAFY-F1+TcLFY R260full menghasilkan
pita yang besarnya sekitar 600 bp. Primer forward dgLFY-F1 mengisolasi gen LEAFY dengan ukuran sekitar 1100 bp dengan primer reverse TcLFY R971 dan sekitar 1200
bp dengan primer reverse TcLFY R380full
serta TcLFY R260full. Ukuran-ukuran basa
yang didapatkan ini awalnya berasal saat proses perancangan primer untuk mengamplifikasi gen LEAFY target. Hasil PCR pada gel agarosa 1% ini menjadi dasar untuk memperkirakan hasil sekuensing ukuran gen LEAFY.
Gambar 7 Hasil PCR gen LEAFY pada elektroforesis gel agarosa 1%.
Amplifikasi utas pertama pada penelitian ini menggunakan kit dari Invitrogen dengan suhu annealing sebesar 58°C dan siklus PCR sebanyak 35 siklus. Pemilihan suhu 58°C didasarkan pada perhitungan nilai Tm pada saat perancangan primer, sedangkan pemilihan 35 siklus dalam amplifikasi diharapkan akan memberikan hasil yang optimal dalam mendapatkan produk PCR. Adapun program PCR yang digunakan tersaji pada Lampiran 3.
Kloning Fragmen Gen LEAFY
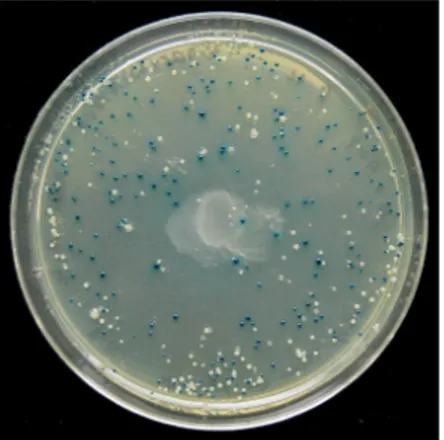
Kloning pada penelitian ini dilakukan dengan tujuan memperbanyak DNA yang tersisip pada sel kompeten E.coli. Tahapan kloning secara garis besar dapat dibagi menjadi tiga yaitu ligasi, transformasi, dan seleksi transforman. Pada pengujian selanjutnya fragmen gen LEAFY hasil pemurnian dengan kit ekstraksi gel dari Invitrogen diklon ke vektor kloning pGEM-T Easy dan kemudian ditransformasikan ke dalam bakteri E.coli XL-1 Blue. Rekombinasi vektor kloning pGEM-T Easy dengan fragmen gen LEAFY menghasilkan koloni biru putih.
Koloni putih dan koloni biru muncul pada permukaan agar pada cawan petri (Gambar 8). Koloni putih menunjukkan bahwa koloni membawa gen insert sebab akan menghambat sintesis α-peptidase sehingga enzim β-galaktosidase tidak aktif (Reece 2004). Selanjutnya seleksi plasmid rekombinan dilakukan terhadap koloni putih dengan metode PCR koloni mengunakan primer universal M13 forward dan M13 reverse kemudian dilanjutkan dengan uji kualitatif pada gel agarosa 1%.
Gambar 8 Koloni biru putih hasil kloning fragmen gen LEAFY.
M 971 380 260 971 380 260 LEAFY-F1 dgLFY-F1
400 bp 500 bp 600 bp
Gambar 9 Hasil elektroforesis PCR koloni primer LEAFY-F1.
Gambar 10 Hasil elektroforesis PCR koloni primer dgLFY-F1.
Gambar 9 dan Gambar 10 menunjukkan bahwa setelah mengalami PCR koloni, pita fragmen gen LEAFY dengan primer LEAFY-F1+TcLFY R971 mengalami
kenaikan dari 400 bp menjadi 600 bp, 500 bp menjadi 700 bp untuk fragmen gen LEAFY dengan pasangan primer LEAFY-F1+TcLFY R380full, 600 bp menjadi 800 bp
untuk fragmen gen LEAFY dengan pasangan primer LEAFY-F1+ TcLFY R260full, dan
1100 bp menjadi 1300 bp untuk fragmen gen
LEAFY dengan pasangan primer
dgLFY+TcLFY R971. Penambahan basa
sekitar 200 bp tersebut merupakan sebagian dari fragmen vektor kloning pGEM-T Easy yang ikut diamplifikasi pada saat proses PCR koloni yaitu masing-masing berjarak 100 bp sebelah kanan dan kiri dari fragmen gen LEAFY yang telah dikloning. Koloni-koloni positif yang menghasilkan pita tersebut selanjutnya dikultur dalam media NB cair + Ampisilin 100 ppm selama semalam pada 37 °C. Setelah itu dilakukan isolasi plasmid dari setiap kultur yang diperoleh menggunakan kit dari Roche. Uji kualitatif pada gel agarosa 1% selanjutnya dilakukan terhadap hasil isolasi plasmid dan terakhir plasmid dari masing-masing koloni dianalisis urutan basanya (sekuensing)
menggunakan primer universal M13 F dan M13 R. Sekuensing dilakukan terhadap fragmen gen LEAFY dengan pasangan primer LEAFY-F1+TcLFY R971,
LEAFY-F1+TcLFY R260 full, dan dgLFY-F1+ TcLFY
R971.
Analisis Urutan Basa Fragmen Gen Terklon
Urutan basa hasil sekuensing selanjutnya dianalisis dengan program bioinformatika. Untuk konfirmasi awal dapat menggunakan program BLASTX terhadap masing-masing hasil sekuensing sehingga dapat diketahui apakah gen target merupakan gen LEAFY atau bukan. Analisis awal BLASTX ini menunjukkan bahwa keenam hasil sekuensing menunjukkan tingkat homologi yang cukup tinggi dengan gen LEAFY pada basis data bank gen.
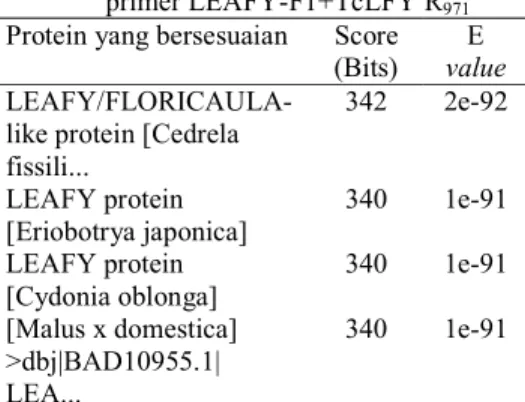
Tabel 1 menunjukkan analisis BLASTX hasil isolasi fragmen gen LEAFY menggunakan pasangan primer LEAFY-F1+TcLFY R971 untuk proses sekuensing
menggunakan primer universal M13F. Terlihat bahwa nilai skor (bits) hasil sekuensing tersebut melebihi angka 150 dan nilai E value lebih kecil dari 10-4 sehingga dapat dikatakan bahwa hasil sekuensing fragmen gen LEAFY memiliki tingkat homologi yang cukup tinggi dengan gen LEAFY yang terdapat pada basis data bank gen. Tingkat homologi dapat diketahui lebih jauh dengan melihat nilai skor (bits) dan E. value. Semakin tinggi skor (bits) maka tingkat homologinya semakin baik. Sebaliknya semakin rendah skor (bits) maka semakin buruk tingkat homologinya. Selain skor (bits), E value juga memberikan ukuran statistik yang cukup signifikan. Nilai E value yang lebih rendah menunjukkan tingkat homologi yang lebih baik atau sekuen gen lebih mirip dan data lebih dapat dipercaya. Di dalam dunia sekuen, untuk memastikan adanya homologi E value harus kurang dari e-04 (Claverie&Notredame 2003).
Setelah diketahui bahwa hasil sekuensing untuk masing-masing pasangan primer yang digunakan mempunyai tingkat homologi yang cukup tinggi dengan gen-gen referensi yang terdapat pada basis data, selanjutnya dilakukan analisis terhadap sekuen gen lengkap. Analisis dilakukan dengan cara manual serta menggunakan program bioedit dan BLASTX untuk analisis tingkat homologi. Sekuen gen yang digunakan untuk analisis sekuen gen lengkap adalah
1 2 3 4 5 M 6 7 8 9 10 11 12 1 3 1 4 1 5 LEAFY F1-971 LEAFY F1-380 LEAFY F1-260
M 1 2 3 4 5 M 6 7 8 9 10 M 11 1 2 1 3 1 4 15 dgLFY-260 dgLFYg-380 dgLFY-971
600 bp
700 bp 800 bp
sekuen gen yang menggunakan pasangan primer dgLFY-F1+TcLFY R971 dan sekuen
gen yang menggunakan pasangan primer LEAFY-F1+TcLFY R260full. Pemilihan
kedua hasil sekuensing gen ini merujuk pada panjang sekuen gen dengan pasangan primer dgLFYF1+TcLFY R971 yang mempunyai
panjang sekitar 1100 bp serta pertimbangan bahwa pada saat perancangan primer LEAFY-F1+TcLFY R260full terletak di luar
sekuen gen dengan pasangan primer dgLFY-F1+TcLFY+ R971 sehingga diharapkan akan
didapatkan sekuen gen lengkap LEAFY (full length).
Kontaminan vektor dihilangkan dengan cara manual untuk masing-masing hasil sekuensing fragmen gen LEAFY dengan pasangan primer dgLFY-F1+TcLFY R971
serta dicari masing-masing letak primer forward dan reverse serta dilakukan reverse complement dengan program bioedit dan selanjutnya dicari daerah overlap secara manual. Hal yang serupa juga dilakukan untuk hasil sekuensing fragmen gen LEAFY dengan pasangan primer LEAFY-F1+Tc LFY R260full. Selanjutnya dilakukan
penggabungan kedua sekuen gen yang telah dibersihkan dan dilakukan analisis dengan program BLASTX. Terlihat pada Tabel 2 bahwa gen target dengan gen-gen referensi mempunyai tingkat homologi yang sangat tinggi.
Hasil BLASTX terhadap sekuen gen LEAFY lengkap ini menghasilkan panjang basa sebesar 1188 bp. Tampilan grafik pada Lampiran 5 menunjukkan tingkat homologi sekuen gen target dengan sekuen gen LEAFY yang terdapat pada basis data bank gen. Masing-masing garis mewakili porsi homologi sekuen gen LEAFY yang ada di database dengan sekuen gen target. Garis merah menunjukkan tingkat homologi yang tinggi, garis hijau menunjukkan tingkat homologi sedang, garis biru kurang bagus, dan garis hitam paling buruk.
Protein dengan nomor akses ABA43523.1 FLORICAULA/LEAFY-like protein 3 dari Buddleja davidii mempunyai skor tertinggi yaitu 511 bits dan E.value terendah yaitu 7e-143. Hal ini menunjukkan bahwa protein tersebut mempunyai homologi paling tinggi dengan fragmen gen
LEAFY yang telah diisolasi.
FLORICAULA/LEAFY-like protein 3 merupakan protein yang berperan dalam proses pembentukan bunga yang berasal dari jaringan meristem (Adkins 2004). Data ini menunjukkan bahwa fragmen gen LEAFY
yang didapatkan merupakan LEAFY kakao dan fungsi berbagai protein tersebut berhubungan langsung dengan mekanisme pembungaan.
Tabel 1 Hasil analisis BLASTX fragmen gen LEAFY menggunakan pasangan primer LEAFY-F1+TcLFY R971
Protein yang bersesuaian Score (Bits)
E value
LEAFY/FLORICAULA-like protein [Cedrela fissili... 342 2e-92 LEAFY protein [Eriobotrya japonica] 340 1e-91 LEAFY protein [Cydonia oblonga] 340 1e-91 [Malus x domestica] >dbj|BAD10955.1| LEA... 340 1e-91
* hanya ditampilkan sebagian, secara lebih lengkap dapat dilihat pada lampiran 4
Tabel 2 Hasil analisis BLASTX fragmen gen LEAFY lengkap
Protein yang bersesuaian Score (bits)
E value
FLORICAULA/LEAFY-like protein 3 [Buddleja davi...
511 8e-143
FLORICAULA/LEAFY-like protein 1 isoform b [Bud...
510 1e-142
* hanya ditampilkan sebagian, secara lebih lengkap dapat dilihat pada lampiran 5
Karakterisasi Gen LEAFY pada Berbagai Jaringan
Karakterisasi dilakukan untuk mengetahui ekspresi gen target pada berbagai jaringan tanaman kakao. Pengamatan ekspresi gen LEAFY dilakukan pada jaringan bunga kuncup, bantalan bunga aktif, bantalan bunga pasif, dan daun. Tahapan yang dilakukan adalah isolasi RNA serta sintesis utas pertama beserta amplifikasinya. Tahap isolasi RNA dilakukan dengan metode Chaidamsari et al.(2005).
RNA beberapa jaringan tanaman kakao hasil isolasi mempunyai konsentrasi antara 24-1120 ng/µL dengan perbandingan nilai A260/230 sekitar 1.382-2.317 dan nilai A260/280 sekitar 1.075-2.151 (Tabel 3). Lampiran 6 menunjukkan bahwa RNA hasil
isolasi masih mengandung kontaminan berupa polisakarida dan protein. RNA hasil isolasi selanjutnya dikonfirmasi keberadaan RNAnya dengan elektroforesis gel agarosa 1% pada tegangan listrik 25 Volt selama 2 jam. Gambar 11 menunjukkan semua RNA hasil isolasi mempunyai 2 pita yang berarti RNA telah berhasil diisolasi yaitu adanya pita 18s dan 28s. Tahap berikutnya adalah sintesis utas pertama dan amplifikasi utas cDNA. Proses amplifikasi utas cDNA menggunakan cetakan hasil sintesis utas pertama. Proses amplifikasi ini menggunakan primer spesifik yang telah dipakai pada penelitian sebelumnya yaitu primer forward TcLFY-09F1 dan primer reverse TcLFY-GW Rev 4 (Lampiran 7).
Proses amplifikasi cDNA dilakukan pada suhu annealing 62ºC dan dengan variasi jumlah siklus. Pemilihan suhu annealing 62°C didasarkan pada hasil penelitian sebelumnya yang telah mendapatkan hasil bahwa suhu optimum penempelan primer spesifik yang digunakan adalah 62ºC. Variasi jumlah siklus dilakukan dengan tujuan memperoleh siklus yang optimum sehingga didapatkan hasil penampakan gen target yang optimal pada saat dilakukan running pada gel agarosa 1% dengan tegangan listrik 100 Volt selama 40 menit. Variasi siklus yang dilakukan adalah 30, 33, dan 35 (Gambar 12).
Proses amplifikasi dengan siklus 30 terlihat bahwa pita belum terbentuk pada semua amplikon jaringan tanaman kakao yang diisolasi. Amplifikasi selanjutnya dilakukan sebanyak 35 siklus dan hasil elektroforesis pada gel agarosa menunjukkan bahwa pita terbentuk pada semua jaringan yang diiisolasi.
Tabel 3 Hasil analisis kuantitatif RNA beberapa jaringan tanaman kakao Sampel A260/230 A260/280 [RNA] (ng/µL) Bunga kuncup 2.151 2.317 24 Bantalan bunga aktif 1.382 1.088 1052 Bantalan bunga pasif 1.426 1.075 1040 Daun 1.423 1.075 1120
* hanya ditampilkan sebagian, secara lebih lengkap dapat dilihat pada lampiran 6
Gambar 11 Hasil elektroforesis RNA berbagai jaringan tanaman kakao
Pita-pita yang terbentuk berukuran sekitar 400 bp dengan tingkat intensitas kecemerlangan yang berbeda. Untuk mengetahui optimalisasi siklus untuk mengamplifikasi fragmen gen LEAFY ini maka jumlah siklus diturunkan menjadi 33 siklus. Ternyata amplifikasi dengan 33 siklus ternyata sudah dapat mengamplifikasi fragmen gen LEAFY dengan baik. Pita terlihat pada semua jaringan yang diamplifikasi. Hasil ini memberikan informasi bahwa gen LEAFY ternyata juga diekspresikan pada jaringan-jaringan yang diisolasi yaitu bantalan bunga aktif, bantalan bunga pasif, bunga kuncup, daun, dan biji.
Gambar 12 Amplifikasi fragmen gen LEAFY pada berbagai jaringan tanaman kakao dengan siklus 30, 33, dan 35.
SIMPULAN DAN SARAN Simpulan
RNA dari bantalan bunga kakao berhasil diisolasi dengan menggunakan ketiga pasangan primer spesifik. Hal ini ditunjukkan dengan hasil BLASTX yang menunjukkan homologi yang sangat tinggi dengan gen LEAFY dari tanaman lain. Karakterisasi gen LEAFY melalui uji ekspresi pada berbagai jaringan tanaman kakao menunjukkan bahwa gen LEAFY terdapat pada semua jaringan yang diisolasi dengan tingkat ekspresi yang berbeda.
30 33 35 30 33 35 30 33 35 30 33 35 30 33 35 M BBA BBP BK Daun Biji
400bp Bunga BBA BBP Daun
28 S
Saran
Penelitian lanjutan perlu dilakukan untuk menyatukan sekuen gen yang diisolasi dengan cara amplifikasi menggunakan pasangan primer forward dgLFY-F1 dengan primer reverse TcLFY R260full. Penelitian
lanjutan perlu dilakukan untuk mengetahui peran dan fungsi gen LEAFY pada bantalan bunga pasif, bunga kuncup, daun, dan biji dari tanaman kakao.
DAFTAR PUSTAKA
Adkins JA. 2004. Characterization and
expression of
FLORICAULA/LEAFY
homologues in Buddeleja davidii. [tesis]. Raleigh: North Carolina University.
Asif et al. 2000. A simple prosedur for the isolation of high quality RNA from ripening banana fruit. Plant Molecular Biology Reporter 18: 109-115.
Chaidamsari T. 2005. Isolation and characterization of the MADS-box gens AGAMOUS and APETALA1 homologoes from cocoa. Di dalam: Chaidamsari T, editor. Biotechnology for cocoa pod borer resistance in cocoa [tesis]. Wageningen: Wageningen Universiteit. Chaidamsari et al. 2006. Isolation and
characterization of an AGAMOUS homologue from cocoa. Plant Sci 170: 968-975. Claverie JM, Notredame C. 2003.
Bioinformatics for Dummies. Indianapolis: Wiley.
[Dirjenbun] Direktorat Jendral Perkebunan. 2001. Statistik Perkebunan Indonesia. 1998-2001. Jakarta: Direktorat Jendral Perkebunan. Glick BR, Pasternak JJ. 1994. Molecular
Biotechnology Principle and Applications of Recombinant DNA. Washington: ASM Press. Henahan S. 1995. Flower power gene.
Nature 377: 495-499.
Innis MA, Gelfand, Sninsky. 1991. PCR Protocols. California: Cetus. James A. Sullivan, Ken Shirasu , Xing
Wang Deng. 2003. The diverse roles of ubiquitin and the 26S proteasome in the life of plants. Nature Reviews Genetics 4: 948-958.
Jusuf M. 2001. Genetika I Strukturdan Ekspresi Gen. Jakarta: Sagung Seto.
Liu et al. 1998. A method for isolation of total RNA from fruit tissues of banana. Plant Molecular Biology Reporter 16: 1-6.
Koolman J, KH Roehm. 2005. Color Atlas of Biochemistry. Edisi ke-2. New York: Thieme.
Newton CR. 1995. PCR Essential Data. Chichester: John Wiley&Sons. O’Connell J. 2002. Method in Molecular
Biology: RT-PCR Protocols.
Totowa: Humana Pr.
Pena et al. 2001. Constitutive expression of Arabidopsis LEAFY or APETALA1 genes in citrus reduces their generation time. Nature Biotech 19: 236-267. Promega. 2009. Technical Manual. Woods
Hollow Road: Promega.
Putri LD. 2004. Pemisahan dan pencirian pektin dari kulit buah kakao. [skripsi]. Bogor: Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Reece RJ. 2004. Analyses of Genes and Genomes. England: John Willey & Sons.
Saiki RK, David HG, Susanne S, Stephen JS, Russel H, Glenn TH, Kary BM, Henry AE. 1988. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science. 239: 487-491.
Sambrook J, Russell DW. 2001. Molecular Cloning a Laboratory Manual. New York: Cold Spring Harbor Laboratory.
Sartika D. 2006. Bioinformatika pada gen pembungaan: karakterisasi gen homolog pada Magnoliopsida dan Liliopsida. [tesis]. Bandung: Sekolah Ilmu dan Teknologi Hayati, Institut Teknologi Bandung.
Soltis DE et al. 2002. Trends in plant science. J Plant Sci 7 : 22-30. Thamrin E. 2001. Kloning gen penyandi
hidrolase dari sumber air panas rimbo panti, Sumatra Barat [tesis]. Bogor: Program Pascasarjana, Institut Pertanian Bogor.
Wardojo S. 1992. Major pest and diseases of cocoa in Indonesia In: Keane PJ and Putter CAJ (eds) Cocoa Pest and Disease Management in Southeast Asia and Australia FAO. Plant Production and Protection. 112: 63-67.
Wood GAR. 1975. Cocoa. London: Longman Group Limited.
Lampiran 1 Tahap penelitian
Sintesis
first strand
Elektroforesis gel agarosa 1%
RT-PCR
Analisis kuantitatif (spektrofotometer UV) dan kualitatif
(elektroforesis gel agarosa 1%) RNA total
Ekstraksi dan purifikasi
Elektroforesis gel agarosa 1%
Kloning
Ligasi
Transformasi
PCR koloni
Isolasi DNA plasmid
Sekuensing
Analisis hasil sekuensing
Lampiran 2 Primer-primer spesifik fragmen gen
LEAFY
Primer
Sekuen
Tm (ºC)
TcLFY Rev 260
full
5’- GGTGACCGCTTTAGAAGGCCATATGATCAG
74.6
TcLFY R971
5’- GCATCAATGTCCCAACCCTG
67.5
TcLFY R380 full 5’- GGTGACCGGCCATATGATCAGGTCCGCCGG
85.6
LEAFY-F1
5’- GAGAGACAGAGGGAGCACC
61.6
Lampiran 3 Program PCR untuk amplifikasi fragmen gen
LEAFY
Tahapan
T (°C)
Waktu
Siklus (X)
Predenaturasi
94
5’
1
Denaturasi
94
45’’
35
Penempelan primer
(annealing)
55
45’’
35
Pemanjangan
(extension)
72
2’
35
Pascapemanjangan
72
5’
1
Lampiran 4 Hasil analisis BLASTX fragmen gen
LEAFY
menggunakan pasangan
primer LEAFY F1+TcLFY R971
Score E
Sequences producing significant alignments: (Bits) Value
gb|AAT46474.1| LEAFY/FLORICAULA-like protein [Cedrela fissili... 342 2e-92
dbj|BAD10960.1| LEAFY protein [Eriobotrya japonica] 340 1e-91
dbj|BAD10958.1| LEAFY protein [Cydonia oblonga] 340 1e-91
dbj|BAB83097.1| AFL2 [Malus x domestica] >dbj|BAD10955.1| LEA... 340 1e-91
gb|EEF51685.1| Floricaula protein, putative [Ricinus communis] 338 4e-91
gb|AAS45976.1| floricaula [Leucocarpus perfoliatus] 338 5e-91
dbj|BAD10957.1| LEAFY protein [Pyrus communis] 338 5e-91
gb|ABI58284.1| leafy-like protein [Carya cathayensis] >gb|ABI... 338 6e-91
gb|ABF84009.1| LEAFY-like [Malus x domestica] 338 6e-91
dbj|BAD10959.1| LEAFY protein [Pseudocydonia sinensis] 338 6e-91
dbj|BAD10956.1| LEAFY protein [Pyrus pyrifolia] 338 6e-91
gb|ABB83126.1| leafy-like protein [Castanea mollissima] 337 8e-91
gb|ABP02007.1| LEAFY-like protein [Dimocarpus longan] >gb|ABC... 336 2e-90
gb|AAT57872.1| leafy/floricaula-like protein [Hevea brasilien... 333 1e-89
gb|AAS46002.1| floricaula [Verbena officinalis] 333 1e-89
gb|ABA43523.1| FLORICAULA/LEAFY-like protein 3 [Buddleja davi... 333 2e-89
gb|ABA43520.1| FLORICAULA/LEAFY-like protein 1 isoform b [Bud... 333 2e-89
gb|ABA43522.1| FLORICAULA/LEAFY-like protein 2 [Buddleja davi... 333 2e-89
gb|ABA43519.1| FLORICAULA/LEAFY-like protein 1 isoform a [Bud... 333 2e-89
emb|CAO68395.1| unnamed protein product [Vitis vinifera] 333 2e-89
gb|ABN11915.1| LEAFY-like protein [Vitis hybrid cultivar] 333 2e-89
gb|AAM46141.1|AF378126_1 LEAFY-like protein 1 [Vitis vinifera] 333 2e-89
gb|AAN14527.1|AF450278_1 floricaula/leafy-like protein [Vitis... 333 2e-89
gb|ABY91324.1| FLO/LFY-like protein [Chenopodium rubrum] 332 3e-89
gb|AAS46008.1| floricaula [Mimulus guttatus] 332 3e-89
gb|ABF61861.2| LEAFY [Clausena lansium] 332 4e-89
gb|AAF77610.1|AF106842_1 PlaraLFY protein [Platanus racemosa] 332 4e-89
gb|ABE02270.1| LEAFY-like protein [Glycine max] >gb|ABE02271.... 331 6e-89
gb|AAO73539.1| leafy/floricaula [Salix discolor] 331 6e-89
gb|AAF66099.1|AF197934_1 FLORICAULA/LEAFY-like protein [Lycop... 331 6e-89
gb|ABA43521.1| FLORICAULA/LEAFY-like protein 1 isoform c [Bud... 331 6e-89
gb|ABS18396.1| FLO/FLY protein [Capsicum annuum] 331 7e-89
gb|ABJ97281.1| LEAFY [Citrus unshiu] >gb|ABJ97282.1| LEAFY [C... 331 7e-89
gb|ABF61858.2| LEAFY [Fortunella crassifolia] 331 7e-89


![Tabel 3 Hasil analisis kuantitatif RNA beberapa jaringan tanaman kakao Sampel A 260/230 A 260/280 [RNA] (ng/µL) Bunga kuncup 2.151 2.317 24 Bantalan bunga aktif 1.382 1.088 1052 Bantalan bunga pasif 1.426 1.075 1040 Daun 1.423](https://thumb-ap.123doks.com/thumbv2/123dok/2307878.2733341/22.918.504.757.587.690/tabel-analisis-kuantitatif-jaringan-tanaman-sampel-bantalan-bantalan.webp)