SIMULASI REFOLDING PROTEIN 2PTL MENGGUNAKAN
NOT JUST ANOTHER MOLECULAR DYNAMICS PROGRAM
(NAMD)
NURIKA FITRIANI
DEPARTEMEN FISIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2016
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Simulasi Refolding Protein 2PTL Menggunakan Not Just Another Molecular Dynamics Program (NAMD) adalah benar karya saya dengan arahan dari dosen pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Agustus 2016
Nurika Fitriani NIM G74120046
ABSTRAK
NURIKA FITRIANI. Simulasi Refolding Protein 2PTL Menggunakan Not Just Another Molecular Dynamics Program (NAMD). Dibimbing oleh TONY IBNU SUMARYADA dan SETYANTO TRI WAHYUDI.
Memahami bagaimana proses unfolding dan refolding protein masih menjadi tantangan utama dalam ilmu kehidupan. Salah satu protein yang dapat diamati yaitu protein 2PTL. Protein 2PTL membantu Peptostreptococcus magnus menghindari pertahanan inang melalui sifat pengikatan protein melalui interaksi rantai ringan kappa. Pada penelitian ini dilakukan simulasi dinamika molekul untuk protein 2PTL pada temperatur 500K selama 15 ns untuk simulasi unfolding dan temperatur 200K selama 30 ns untuk simulasi refolding. Dari penelitian ini diperoleh proses unfolding terjadi pada waktu 6.5 ns. Secara geometri, hasil analisis RMSD pada 500K meningkat hingga 7.059Å dan pada 200K mengalami penurunan sebesar 2.683Å. Tingkat kemiripan asam amino proses unfolding dengan struktur native sebesar 36.78%. Setelah dilakukan proses refolding pada 200K dilakukan juga pengecekan tingkat kemiripan asam amino dengan struktur native sebesar 62.264%.
Kata kunci: NAMD, protein 2PTL, refolding, simulasi dinamika molekul, unfolding
ABSTRACT
NURIKA FITRIANI. Refolding Simulation of 2PTL Protein Using Not Just Another Molecular Dynamics Program (NAMD). Supervised by TONY IBNU SUMARYADA and SETYANTO TRI WAHYUDI.
Understanding how the process of unfolding and refolding proteins remains a major challenge in the life sciences. The example of protein that can be observed is protein 2PTL. Protein 2PTL helps Peptostreptococcus magnus to avoid host defenses by using the protein binding properties through kappa light chain interactions. In this research, molecular dynamics simulations were performed for protein 2PTL at 500K during 15 ns for unfolding simulation and at 200K during 30 ns for refolding simulation. The unfolding process occurs at 6.5 ns. In geometry result of RMSD analysis at 500K increased 7.059Å and decreased is about 2.683Å at 200K. The degree of amino acid compatibility with native structure in unfolding process is about 36.78%. After the refolding process at 200K do also checking the degree of amino acid compatibility with the native structure is about 62.264%.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Fisika
SIMULASI REFOLDING PROTEIN 2PTL MENGGUNAKAN
NOT JUST ANOTHER MOLECULAR DYNAMICS PROGRAM
(NAMD)
NURIKA FITRIANI
DEPARTEMEN FISIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2016
PRAKATA
Puji syukur penulis panjatkan kepada Allah subhanahu wa ta’ala dan shalawat serta salam semoga tercurah kepada Nabi Muhammad SAW. Alhamdulillah karena berkat rahmat dan karunia-Nya, penulis dapat menyelesaikan karya ilmiah yang berjudul “Simulasi Refolding Protein 2PTL Menggunakan Not Just Another Molecular Dynamics Program (NAMD)”.
Terimakasih penulis sampaikan kepada berbagai pihak yang telah terlibat dalam penelitian ini. Kepada Bapak Dr R Tony Ibnu Sumaryada dan Bapak Dr Setyanto Tri Wahyudi selaku pembimbing juga ibu Dr. Mersi Kurniati selaku dosen penguji yang selalu memberikan wawasan dan arahan serta bimbingannya kepada penulis. Tidak lupa penulis sampaikan ucapan terimakasih kepada kedua orang tua, keluarga, serta sahabat, atas segala doa, kasih sayang dan dukungannya.
Ucapan terimakasih juga penulis sampaikan kepada rekan penelitian dan rekan mahasiswa/i departemen fisika 49 yang selalu memberikan motivasi dan saran. Terimakasih penulis sampaikan kepada seluruh dosen dan staf Departemen Fisika FMIPA-IPB.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2016 Nurika Fitriani
DAFTAR ISI
DAFTAR TABEL vi DAFTAR GAMBAR vi DAFTAR LAMPIRAN vi PENDAHULUAN 1 Latar Belakang 1 Perumusan Masalah 2 Tujuan Penelitian 2 Manfaat Penelitian 2 TINJAUAN PUSTAKA 2 Protein 2PTL 2Unfolding dan Refolding Protein 3
Simulasi Dinamika Molekul 4
METODE 5
Tempat dan Waktu 5
Bahan 5
Alat 5
Prosedur Penelitian 5
HASIL DAN PEMBAHASAN 7
Analisis Struktur Sekunder 7
RMSD 9 Jari-jari Girasi 10 Energi Konformasi 11 SASA 12 Ikatan Hidrogen 13 RMSF 14
SIMPULAN DAN SARAN 16
Simpulan 16
Saran 16
DAFTAR PUSTAKA 16
LAMPIRAN 18
DAFTAR TABEL
1 Residu penyusun struktur sekunder protein 2PTL setelah pemotongan
coil 3
DAFTAR GAMBAR
1 Struktur protein 2PTL (a) sebelum pemotongan coil (b) setelah
pemotongan coil 3
2 Struktur Sekunder Protein 2PTL (a) unfolding 500 K selama 15 ns,
(b) refolding 200 K selama 30 ns 8
3 Perubahan konformasi struktur sekunder selama simulasi
unfolding 500 K selama 15 ns dan refolding 200 K selama 30 ns 9 4 RMSD hasil simulasi unfolding pada 500 K selama 6.5 ns dan refolding
pada 200 K selama 30 ns 10
5 Jari-jari girasi hasil simulasi (a) unfolding pada 500 K selama 6.5 ns
(b) refolding pada 200 K selama 30 ns 11
6 Energi konformasi hasil simulasi (a) unfolding pada 500 K selama 6.5 ns
(b) refolding pada 200 K selama 30 ns 12
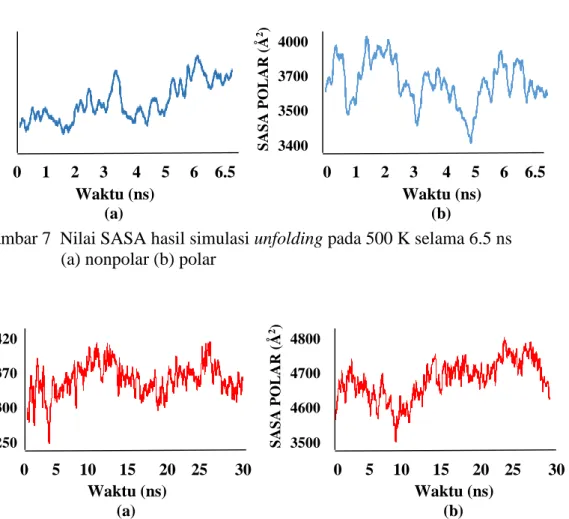
7 Nilai SASA hasil simulasi unfolding pada 500 K selama 6.5 ns
(a) nonpolar (b) polar 13 8 Nilai SASA hasil simulasi refolding pada 200 K selama 30 ns
(a) nonpolar (b) polar 13
9 Ikatan hidrogen protein to protein hasil simulasi (a) unfolding
pada 500 K selama 6.5 ns (b) refolding pada 200 K selama 30 ns 14 10 RMSF hasil simulasi (a) unfolding pada 500 K selama 6.5 ns
(b) refolding pada 200 K selama 30 ns 15
DAFTAR LAMPIRAN
1 Diagram alir penelitian 18
2 Persentase kesesuaian urutan residu penyusun antara struktur protein
native dengan hasil simulasi refolding 19 3 Karakteristik residu yang terdapat pada protein 2PTL 20
PENDAHULUAN
Latar Belakang
Protein merupakan senyawa organik kompleks dengan bobot molekul tinggi. Protein juga merupakan suatu polimer yang terdiri dari monomer-monomer asam amino yang dihubungkan dengan ikatan peptida. Protein memiliki banyak fungsi diantaranya sebagai enzim, hormon dan antibodi, namun protein hanya mampu berfungsi untuk suatu fungsi. Oleh karena itu agar suatu polipeptida yang baru dibentuk siap menjadi protein yang berfungsi secara biologis dan mampu mengkatalisis suatu reaksi metabolik, menggerakkan sel, atau makromolekul, polipeptida tersebut harus mengalami pelipatan membentuk susunan tiga dimensi tertentu atau konformasi.1
Perkembangan teknologi tentang metode penentuan protein selalu mengalami perkembangan, baik secara eksperimen maupun komputasi. Semua sistem kehidupan mengandung sejumlah protein yang berbeda dalam hal susunan asam amino, urutan asam amino, dan faktor yang mempengaruhi struktur molekul protein.2 Proses pelipatan protein mampu menjelaskan bagaimana protein dapat berfungsi. Proses yang dapat menentukan keadaan suatu protein dalam menjalankan fungsinya dintaranya proses unfolding dan refolding. Memahami bagaimana proses unfolding dan refolding protein tersebut masih menjadi salah satu tantangan utama dibidang ilmu kehidupan.3 Unfolding protein merupakan salah satu proses biokimia yang sangat fundamental dan hal tersebut sangat sulit dikarakterisasi menggunakan metode in vivo, begitu juga dengan proses refolding. Dengan demikian, metode lain yang dapat digunakan untuk memudahkan penelitian yang berhubungan dengan karakterisasi protein dapat menggunakan simulasi komputasi.
Skala waktu pelipatan protein yang berada dalam rentang mikro sampai milidetik masih belum benar-benar dapat diakses untuk studi simulasi secara langsung.4Pengamatan terhadap pelipatan suatu protein tentang bagaimana struktur protein tersebut berubah juga masih sulit dilakukan karena protein yang berada dalam bentuk mikromolekul. Salah satu metode yang dapat digunakan untuk mengamati proses tersebut ialah dengan menggunakan simulasi dinamika molekul. Simulasi dinamika molekul (MD) dapat memeriksa lintasan protein hingga puluhan nanodetik.4
Simulasi dinamika molekul adalah teknik suatu teknik dari sistem N partikel dengan integrasi numerik persamaan Newton tentang gerak, untuk potensial interatomik spesifik dengan kondisi awal dan kondisi batas tertentu.5 Salah satu protein yang yang dapat diamati yaitu protein 2PTL, karena protein tersebut memiliki kestabilan termal yang tinggi sehingga menarik untuk dimati. Oleh karena itu penelitian ini mensimulasikan keseluruhan protein 2PTL pada temperatur tertentu menggunakan simulasi dinamika molekul.
2
Perumusan Masalah
Permasalahan dalam penelitian ini adalah belum banyak dilakukan penelitian tentang proses pelipatan protein 2PTL terhadap pengaruh temperatur yang dapat membuatnya terdenaturasi serta bagaimana lintasan yang akan dilalui protein 2PTL dalam proses pelipatannya.
Tujuan Penelitian
Tujuan penelitian ini mempelajari pengaruh temperatur terhadap unfolding protein 2PTL pada temperatur 500 K dan proses refolding protein 2PTL pada temperatur 200 K.
Manfaat Penelitian
Penelitian ini diharapkan dapat memberikan pemahaman terhadap proses unfolding dan refolding protein 2PTL akibat pengaruh temperatur.
TINJAUAN PUSTAKA
Protein 2PTL
Protein L (kode PDB: 2PTL) merupakan protein mengikat-immunoglobulin yang awalnya berasal dari bakteri Peptostreptococcus magnus, namun kini diproduksi secara rekombinan. Protein L memiliki kemampuan unik untuk mengikat melalui interaksi rantai ringan kappa tanpa mengganggu situs antigen-antibodi yang mengikat itu. Hal ini memberikan Protein L kemampuan untuk mengikat lebih luas kelas dan subclass Ig dari protein antibodi mengikat lainnya. Protein L akan mengikat semua kelas Ig (IgG, IgM, IgA, IgE dan IgD).6
Struktur protein terdiri dari struktur primer, sekunder, tersier, dan kuartener. Struktur sekunder merupakan struktur tiga dimensi dari rangkaian asam amino pada protein yang distabilkan oleh ikatan hidrogen. Struktur sekunder ini terdiri dari alpha helix, beta sheet, turn, dan coil.7 Protein 2PTL terdiri dari 78 residu yang memiliki struktur utama 1 α-helix, 2 pasang β-sheet, 2 coil, dan 2 turn.
Pada penelitian ini dilakukan pengurangan jumlah residu dengan memotong coil pada sebagian protein 2PTL, dimulai dari indeks residu 1 sampai dengan 16 sehingga jumlah residu yang digunakan menjadi 62 residu. Pemotongan jumlah residu dilakukan untuk menyederhanakan struktur protein yang akan diamati pada penelitian ini.
3
No. Struktur Sekunder Index Residu Kode Residu
1 β-sheet β-sheet 1 18 s/d 26 V, T, I, K, A, N, L, I, F β-sheet 2 30 s/d 38 S, T, Q, T, A, E, F, K, G β-sheet 3 60 s/d 67 E, Y, T, V, D, V, A, D β-sheet 4 70 s/d 77 Y, T, L, N, I, K F, A 2 α-helix 40 s/d 54 F, E, K, A, T, S, E, A, Y, A, Y, A, D, T, L 3 turn 27 s/d 29 68 s/d 69 A, N, G K, G 4 coil 39 55 s/d 59 78 T K, K, D, N, G G
Unfolding dan Refolding Protein
Folded adalah suatu keadaan protein berbentuk lipatan-lipatan sehingga dapat melakukan fungsi biologisnya. Suatu protein akan kehilangan fungsinya jika mengalami denaturasi atau unfolding. Peningkatan temperatur yang diterapkan pada suatu protein merupakan salah satu kondisi yang dapat menyebabkan protein terdenaturasi. Unfolding mengakibatkan suatu protein kehilangan daya larutnya. Pada beberapa kasus, protein yang telah unfolded dapat kembali ke keadaan folded (reversible).
Unfolding dan refolding dapat direpresentasikan melalui 4 tahap diskret dan reversible yaitu
F↔H↔S↔U
F adalah keadaan folded, H adalah keadaan protein tanpa struktur sekunder namun dengan inti hidrofobik yang terbungkus, S adalah keadaan dengan inti yang terlarut sebagian, dan U adalah keadaan unfolded sempurna.
Tabel 1 Residu penyusun struktur sekunder protein 2PTL setelah pemotongan coil
(a) (b)
Gambar 1 Struktur protein 2PTL (a) sebelum pemotongan coil (b) setelah pemotongan coil
4
Proses unfolding dimulai dengan merusak ujung struktur sekunder secara cepat selama beberapa pikosekon. Selama proses ini berlangsung struktur sekunder protein tetap utuh dan inti hidrofobiknya tetap terbungkus dengan baik. Proses awal ini diakhiri dengan rusaknya struktur sekunder secara sempurna. Proses selanjutnya dimulai dengan rusaknya ikatan hidrogen namun inti hidrofobik tetap utuh. Setelah itu inti hidrofobik mulai rusak dan ditembus oleh beberapa molekul air dari lingkungan sekitar. Molekul air tersebut terperangkap diantara rantai samping selama beberapa pikosekon, sehingga inti hidrofobik dalam keadaan terlarut sebagian. Proses terakhir yaitu rusaknya inti hidrofobik secara sempurna sehingga protein dalam keadaan tanpa lipatan (unfolded state). Refolding merupakan kebalikan dari proses unfolding dimana pemanasan pada temperatur rendah diterapkan pada protein dalam keadaan unfolded.8
Simulasi Dinamika Molekul
Dinamika Molekul adalah metode untuk mengevaluasi gerakan yang dihasilkan dari interaksi atom dengan mengintegrasikan persamaan gerak. Secara umum molecular dynamics dapat digunakan untuk mengevaluasi gerak dari setiap sistem partikel. Pendekatan mudah adalah dengan mengintegrasikan persamaan gerak yang diperoleh dalam hukum kedua Newton :
𝐹
𝑥𝑡(𝑡) = 𝑚
𝑖.
𝑑2𝑥𝑡(𝑡)𝑑𝑡2
(1)
dimana 𝑥𝑖(t) dan 𝐹𝑥𝑡(𝑡) menunjukkan posisi dan gaya yang bekerja pada atom i
dalam arah x pada waktu t dan mi adalah massa atom i.9
Nilai energi berhubungan dengan temperatur melalui momentum partikel
∑
|𝑝𝑖|2 2𝑚𝑖 𝑁 𝑖=1=
𝑘𝑏𝑇 2(3𝑁 − 𝑁
𝑐)
(2)dimana 𝑁𝑐 adalah jumlah konstrain dan 3N-Nc = N df adalah jumlah derajat kebebasan. Temperatur rata-rata 〈𝑇〉 identik dengan temperatur makroskopik.10
Simulasi dilakukan menggunakan NVT ensemble (Jumlah molekul, Volume dan Temperatur tetap). Distribusi Boltzman untuk ensembel NVT kanonik dalam NAMD dihitung menggunakan persamaan Langevin
𝑚𝑣 = 𝐹(𝑟) − 𝛾𝑣 + √
2𝛾𝑘𝑏𝑇𝑚
𝑅(𝑡)
(3)
dimana m adalah massa, 𝑣 kecepatan, 𝐹 gaya, 𝑟 jarak, 𝛾 koefisien gesek. 𝑘b
5
METODE
Tempat dan Waktu
Penelitian ini dilakukan di Laboratorium Fisika Teori dan Komputasi, Departemen Fisika, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor dimulai pada November 2015 hingga Mei 2016.
Bahan
Bahan yang digunakan dalam simulasi dinamika molekul teknik in silico ini berupa data koordinat hasil NMR protein L, yang diunduh dari Protein Data Bank (http://www.rcsb.org/pdb/) dengan indeks 2PTL.
Alat
Penelitian ini menggunakan peralatan berupa alat tulis, perangkat keras dan perangkat lunak. Perangkat keras terdiri atas komputer dengan spesifikasi Quad Core Processor (Intel Core i7), RAM 12 GB, Graphic Card NVIDIA Ge Force GTS 9400, dan sistem operasi LINUX Ubuntu 12.04. Perangkat lunak yang digunakan untuk simulasi adalah NAMD (Not Just Another Molecular Dynamics Program) versi 2.10. Preparasi, analisis, dan menampilkan animasi molekul protein menggunakan VMD (Visual Molecular Dynamics Program) versi 1.9.1. NAMD dan VMD merupakan program simulasi dinamika molekuler yang dikembangkan oleh Theoretical and Computational Biophysics Group di University of Illiois, Urbana-Champaign. Perangkat lunak lain yang digunakan untuk pengolahan data antara lain CatDCD versi 4.0, Ms. Excel 2013, Gimp versi 2.6, dan Gnuplot 4.6.4.
Prosedur Penelitian Preparasi Sistem Simulasi
Struktur krital protein L (Kode PDB : 2PTL) yang digunakan pada simulasi diperoleh dari bank data Protein Data Bank (PDB), (http://www.rcsb.org/pdb/). Penentuan struktur kristal 2PTL dilakukan dengan metode NMR dengan jumlah residu penyusunnya 78 dan terdapat 21 frame sebagai model konformasinya.
Pada tahap preparasi molekul, program yang digunakan adalah VMD. Data 2PTL.pdb yang telah diunduh kemudian dipotong pada bagian coil nya sebanyak 16 residu dari residu 1 – 16, sehingga menyisakan 62 jumlah residu yang menjadi masukan awal simulasi. Data protein 2PTL.pdb yang memiliki 21 frame atau model konformasi selanjutnya diubah menjadi 1 frame dengan tujuan mempermudah pengamatan simulasi. Pemotongan pada bagian coil bertujuan menyederhanakan bentuk dari protein sehingga tidak terlalu besar.
Selanjutnya, atom hidrogen yang masih memiliki koordinat sembarang dihilangkan dari sistem molekul. Kemudian pusat koordinat digeser ke (0,0,0) yang bertujuan agar atom bersesuaian dengan atom-atom lainnya sehingga mempermudah dalam proses perhitungan. Data 2PTL.pdb tersebut tidak memuat
6
informasi spesifik bagaimana atom bisa saling berinteraksi, sehingga dibutuhkan data psf yang dibuat melalui automatic psf builder agar dapat digunakan untuk menerapkan medan gaya tertentu ke dalam sistem molekul. Medan gaya yang digunakan untuk mendefinisikan energi potensial struktur kristal adalah CHARMM22.
Tahap preparasi berikutnya adalah melarutkan molekul kedalam suatu pelarut, dalam penelitian ini pelarut yang digunakan adalah hidrogen melalui add solvation box. Kotak air berdimensi 74 Å x 59 Å x 59 Å dibuat sebagai wadah pelarut molekul 2PTL. Secara total, jumlah molekul air yang ditambahkan pada kotak adalah 22506 molekul air. Penambahan molekul air secara eksplisit dilakukan sebagai pendekatan terhadap keadaan molekul dalam pelarut, dimana interaksi dengan molekul pelarut ikut mempengaruhi konformasi molekul.
Tahap terakhir, karena beberapa residu protein memiliki muatan sehingga sistem perlu dinetralkan dengan menambahkan penetral sistem berupa NaCl dengan konsentrasi 0.15 mol/L, tahap ini dilakukan agar simulasi yang dilakukan hanya dipengaruhi oleh temperatur.
Simulasi Dinamika Molekul
Tahap simulasi dilakukan dengan menggunakan program NAMD. Simulasi yang dilakukan adalah unfolding dan refolding yang masing-masing simulasi di awali dengan proses minimisasi, pemanasan dan equilibrasi, serta production run. Simuasi dilakukan pada temperatur transisi 500 K untuk unfolding dan 200 K untuk refolding.
Keseluruhan simulasi menggunakan parameter integrasi waktu (time step) setiap 2 femto sekon (fs). Simulasi dilakukan dengan menggunakan metode periodic boundary condition (PBC) untuk menghilangkan efek tegangan permukaan dan untuk mencapai kondisi dengan kerapatan dan tekanan yang lebih seragam.12 Energi elektrostatik sistem dihitung secara menyeluruh menggunakan metode particle mesh Ewald (PME)13 sedangkan Interaksi van der Waals dihitung menggunakan potensial Lennard-Jones dengan cutoff masing-masing adalah 12 Å.14
Untuk mengontrol tahapan minimisasi, pemanasan dan production run dilakukan dengan menggunakan NVT ensemble (constant number, volume and temperature) pada temperatur yang diinginkan. Untuk mengontrol tahapan ekuilibrasi dijaga pada NPT Ensemble (constant number, pressure and temperature) oleh termostat Nose-Hoover.15
Unfolding
Proses unfolding diawali dengan tahap minimisasi, sebagai data masukan adalah data koordinat yang dihasilkan dari proses preparasi struktur native protein 2PTL. Minimisasi bertujuan untuk meminimalkan energi pada molekul agar berada di energi terendahnya (keadaan stabil) selama 100 ps.
Tahap selanjutnya adalah pemanasan dan equilibrasi. Proses pemanasan dilakukan secara bertahap dari temperatur 0 K sampai 500 K selama 20 ps dengan
7 kenaikan setiap 25 K. Selanjutnya dilakukan ekuilibrasi didalam sistem simulasi dengan protokol Langevin selama 40 ps.
Tahap terakhir adalah production run. Setelah molekul protein terekuilibrasi dengan baik maka tahap selanjutnya adalah tahap produksi (Production Run). Konstrain yang diterapkan pada proses ekuilibrasi kemudian dihilangkan pada proses production run sehingga molekul protein bebas bebas bergerak. Waktu simulasi yang diberikan pada tahap ini selama 15 ns.
Refolding
Setelah melakukan proses unfolding dilakukan proses refolding yaitu simulasi dinamika molekul yang berkebalikan dari proses unfolding, artinya setelah proses refolding protein dapat kembali melipat. Tahap yang dilakukan untuk proses refolding sama seperti tahap yang ada pada proses unfolding, namun sebagai data koordinat awal yang digunakan adalah data koordinat struktur protein setelah proses unfolding.
Data masukan awal refolding terlebih dahulu di preparasi dan minimisasi seperti proses unfolding selama 100 ps. Selanjutnya dilakukan pemanasan selama 20 ps dan equilibrasi dengan waktu 40 ps. Tahap terakhir yaitu production run pada temperatur dasar 200 K selama 30 ns.
Analisis data
Data yang diperoleh dari tahap simulasi dinamika molekul masih mengandung air, kemudian air dihilangkan terlebih dahulu. Dengan menggunakan program VMD, data tersebut diolah berdasarkan parameter struktur sekunder, RMSD, jari-jari girasi, energi, SASA, ikatan hidrogen, dan RMSF. Hasil pengolahan data menghasilkan grafik yang dibutuhkan untuk menganalisa dinamika protein 2PTL dan sebagai parameter pembanding antara trayektori dari proses unfolding dan refolding protein 2PTL.
HASIL DAN PEMBAHASAN
Analisis Struktur Sekunder
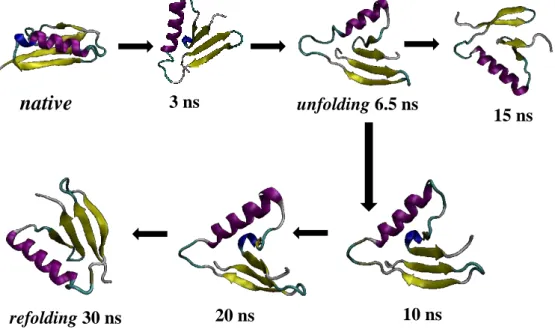
Simulasi dinamika molekul struktur native dapat dilakukan melalui proses unfolding dan refolding. Proses unfolding dan refolding dapat dilakukan dengan menggunakan temperatur ekstrem untuk mempercepat perubahan struktur sekunder pada protein. Dengan menganalisis struktur sekunder hasil simulasi unfolding dan refolding, dapat memberikan penjelasan mengenai perubahan struktur protein 2PTL dari keadaan awal (folded) atau native state menuju perubahan konformasi (unfolding-refolding) selama proses simulasi.
Proses unfolding merupakan proses perusakan pada protein, yang menyebabkan protein terdenaturasi dan kehilangan fungsinya. Data masukan awal
8
yang dijadikan koordinat untuk proses unfolding adalah koordinat dari struktur native.
Gambar 2(a) merupakan struktur unfolding protein 2PTL hasil simulasi selama 15 ns pada temperatur 500 K. Berdasarkan hasil tersebut, menunjukkan bahwa α-helix dan β-sheet mengalami kerusakan pada sebagian strukturnya begitu pula dengan turn dan coil. Kerusakan pada struktur protein menyebabkan protein kehilangan sebagian fungsinya. Meskipun perubahan struktur yang terjadi tidak signifikan, namun dari data tersebut dapat dianalisis bahwa protein 2PTL telah mengalami denaturasi atau unfolding parsial dengan persentase kemiripan anatara struktur native dengan struktur yang sudah unfold sebesar 36.78%.
Proses refolding merupakan simulasi yang bertujuan untuk mengembalikan struktur sekunder dan kestabilan termal protein yang telah mengalami unfolding. Sama halnya dengan proses unfolding, proses refolding ini dilakukan pada temperatur ekstrem (temperatur rendah). Data masukkan yang dijadikan koordinat awal pada proses ini merupakan koordinat dari hasil unfolding. Gambar 2(b) adalah struktur sekunder hasil proses simulasi refolding selama 30 ns pada temperatur 200 K. Terlihat bahwa alfa-helix secara perlahan terbentuk kembali dan beta-sheet mengalami penyempurnaan. Proses refolding sampai akhir memiliki kesesuaian urutan residu antara struktur protein native dengan hasil simulasi refolding sebesar 62.264% (lihat lampiran 2). Hal ini menunjukkan bahwa proses refolding mampu mengembalikan struktur awal protein sebelum dilakukan unfolding.
Pada gambar 2 dan 3 merupakan inti dari proses simulasi unfolding dan refolding protein 2PTL yang direpresentasikan melalui skema pembentukan
Waktu (ns) (a) Waktu (ns) (b) 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 0 2 4 6 8 10 12 14 16 18 20 22 24 26 28 30 R esid u 17 26 56 36 46 66 76 78
Gambar 2 Struktur Sekunder Protein 2PTL (a) unfolding pada 500 K selama 15 ns (b) refolding pada 200 K selama 30 ns
310-Helix
Alfa-helix Coil Turn Beta-sheet
9 struktur sekunder dari protein selama simulasi. Gambar 3(a) adalah strukutur protein 2PTL dalam keadaan native, masih memiliki struktur lengkap yaitu 1 alfa-helix, 2 pasang beta-sheet, 2 coil dan 2 turn dengan jarak antar residu yang lebih pendek. Gambar 3(b) menunjukkan struktur protein 2PTL yang mengalami unfolding pada waktu 6.5 ns. Pada simulasi ini terlihat perubahan struktur sekunder yaitu alfa-helix dan beta-sheet berkurang dari keadaan awalnya (native), dimana jarak antar residu cenderung lebih besar dari sebelumnya. Kemudian pada koordinat ini dijadikan masukan awal untuk proses refolding. Hasil proses refolding menunjukkan bahwa struktur sekunder muncul kembali setelah mengalami kerusakan selama proses unfolding. Terlihat pada Gambar 3(c) alfa-helix secara perlahan terbentuk kembali dan beta-sheet mengalami penyempurnaan pada waktu simulasi 30 ns.
RMSD
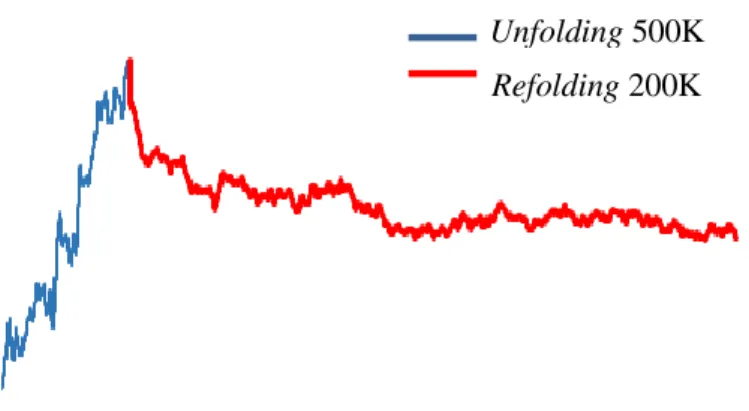
RMSD adalah jarak rata-rata antara konformasi dan struktur referensi dari dua buah atom.16 RMSD juga dapat dikatakan sebagai simpangan protein dari konformasi awalnya. Nilai RMSD dapat menjelaskan prosedur pelipatan protein dan mengetahui waktu perubahan konformasi dari struktur protein. Protein yang berada pada keadaan native memiliki nilai RMSD ≤ 2 Å, untuk nilai 2 Å ≤ RMSD ≤ 8 Å protein berada pada keadaan intermediate atau unfolding parsial, dan untuk nilai RMSD ≥ 8 Å protein tersebut telah mengalami unfolding sempurna.17
Grafik dibawah ini merupakan data yang diperhalus dengan moving average untuk setiap 50 frame. Simulasi yang dilakukan pada temperatur 500 K untuk protein native memiliki nilai RMSD yang cukup tinggi hingga mencapai 7.059 Å
refolding 30 ns 20 ns 10 ns 15 ns unfolding 6.5 ns 3 ns
re
fo
ld
ing
unfo
ld
in
g
Gambar 3 Perubahan konformasi struktur sekunder selama simulasi unfolding 500 K selama 15 ns dan refolding 200 K selama 30 ns
10
pada waktu simulasi 6.5 ns (Gambar 4), pada rentang skala tersebut protein sudah berada pada keadaan intermediet artinya kesetimbangan konformasi struktur protein sudah terganggu dengan jarak simpang dari keadaan awal terhadap keadaan akhir protein membesar sehingga dapat dikatakan terjadi unfolding. Sedangkan refolding dilakukan terhadap struktur protein yang telah mengalami unfolding, dengan waktu simulasi yang lebih lama yaitu 30 ns pada temperatur 200 K nilai RMSD pada proses ini mengalami penurunan sebesar 2.683 Å. Dengan kata lain protein mulai memiliki kesetimbangannya kembali dan telah berada pada kedaan native namun belum sempurna.
Protein dapat dikatakan kembali pada keadaan native sempurna apabila RMSD yang dicapai pada saat proses refolding mengalami penurunan hingga nilai akhir RMSD protein tersebut mencapai 2 Å sampai 3 Å. Protein 2PTL belum dapat kembali pada keadaan native sempurna hal ini dapat disebabkan waktu simulasi yang kurang panjang.
Jari-jari Girasi
Analisis terhadap jari-jari girasi selama simulasi merupakan salah satu parameter yang dapat mempelajari proses terjadinya unfolding. Kekompakan protein sering ditinjau dari nilai jari-jari girasi dan RMSD. Jari-jari girasi merupakan simpangan atom penyusun protein dari pusat massanya.18 Grafik dibawah ini merupakan data yang diperhalus dengan moving average untuk setiap 50 frame. Gambar 5(a) menunjukkan nilai jari-jari girasi pada temperatur 500 K selama waktu simulasi 6.5 ns memiliki nilai awal 11.834 Å dan akhir sebesar 12.183 Å. Selama simulasi protein mengalami fluktuasi hingga mencapai jari-jari tertinggi sebesar 12.439 Å pada waktu 1.916 ns. Perubahan ini menandakan bahwa protein sudah mulai kehilangan kestabilannya, dimana terjadi peningkatan sebesar 0.349 Å dari keadaan awal akibat pengaruh temperatur tinggi yang diberikan. Meningkatnya nilai jari-jari girasi dari keadaan awalnya digambarkan bahwa protein seperti bola yang membesar dari pusat massa bola tersebut.
Waktu (ns) 8 7 5 3 1 0 RM S D ( Å )
Gambar 4 RMSD hasil simulasi unfolding pada 500 K selama 6.5 ns dan refolding pada 200 K selama 30 ns
Unfolding 500K Refolding 200K
0 3 9 12 15 18 21 23 24 27 29 30 31 33 36.5
11 Begitu juga pada temperatur 200 K yang dipresentasikan pada Gambar 5(b), nilai jari-jari girasi menurun dari 12.203 Å menjadi 11.715 Å selama waktu simulasi 30 ns. Berdasarkan hasil simulasi refolding jari-jari girasi menurun sebesar 0.488 Å dari keadaan awalnya (unfolded). Penurunan jari-jari girasi pada proses refolding memberikan informasi bahwa pemberian temperatur rendah pada protein mampu mengembalikan kekompakan dari struktur protein tersebut meskipun protein belum sepenuhnya kembali ke keadaan native secara sempurna.
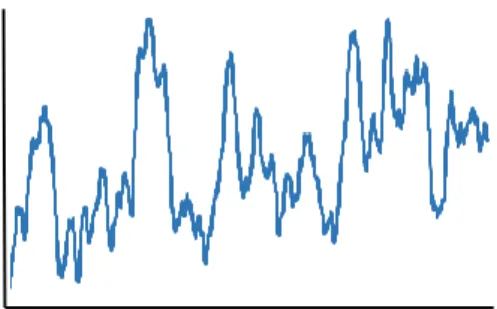
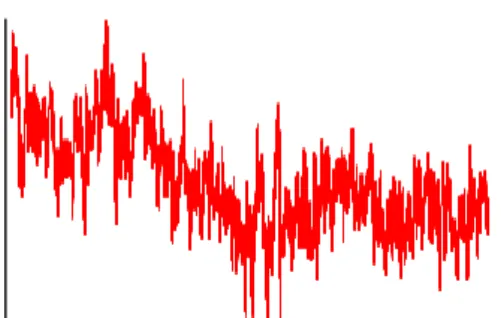
Energi Konformasi
Energi konformasi adalah energi yang dibutuhkan protein untuk mengubah konformasinya. Analisis terhadap perubahan energi selama simulasi juga dapat mempelajari proses terjadinya unfolding sehingga dapat menjelaskan stabilitas termal protein. Energi konformasi ini meliputi energi ikatan, sudut ikatan dan sudut dihedral, serta interaksi eksternal yaitu non-kovalen dan non-ikatan. Jenis interaksi non-ikatan adalah interaksi elektrostatik dan interaksi van der Waals.7
Grafik sudah diperhalus dengan moving average untuk setiap 50 frame. Sepanjang simulasi protein mengalami perubahan konformasi sehingga membutuhkan sejumlah energi. Dapat dilihat pada Gambar 6a, terjadi peningkatan energi sebesar 6703 kcal/mol pada temperatur 500 K selama waktu simulasi 6.5 ns dimana keadaan awal protein memiliki energi sebesar 1392.926 kcal/mol menjadi 1399,629 kcal/mol. Energi yang besar ini disebabkan pengaruh temperatur yang tinggi sehingga energi yang dibutuhkan untuk mengalami perubahan konformasi juga besar. Temperatur tinggi tersebut menyebabkan pergerakan protein dalam sistem lebih acak. Maka dapat dikatakan pada waktu simulasi 6.5 ns protein berada pada keadaan tidak stabil (unfolded).
Gambar 6b menunjukkan penurunan energi dari hasil simulasi refolding selama 30 ns. Penurunan energi sebesar 23.229 kcal/mol menjelaskan bahwa protein mengalami kestabilan kembali selama simulasi. Pemberian temperatur rendah membuat protein bergerak lebih stabil sehingga energi yang dibutuhkan protein untuk dapat kembali ke keadaan native tidak terlalu besar.
Waktu (ns) (a) 0 1 2 3 4 5 6 6.5 0 3 6 9 12 15 18 21 24 27 30 Waktu (ns) (b) J a ri -j a ri G ir a si (Å )
Gambar 5 Jari-jari girasi hasil simulasi (a) unfolding pada 500 K selama 6.5 ns (b) refolding pada 200 K selama 30 ns
12.5 12.3 12.1 12 11.8 12.5 12.3 12 11.7 11.5 J a ri -j a ri G ira si ( Å )
12
SASA
Analisis SASA dapat digunakan untuk menghitung luas area permukaan protein yang dapat dimasuki oleh molekul pelarut. Dalam penelitian ini molekul pelarut yang digunakan adalah air. Semakin besar nilai SASA menunjukkan semakin besar molekul protein berekspansi. Grafik pada Gambar 7 dan Gambar 8 ini diperhalus dengan moving average untuk setiap 50 frame.
Selama simulasi unfolding nilai SASA terus berfluktuasi dan mengalami peningkatan baik nilai SASA nonpolar ataupun nilai SASA polar seperti yang ditunjukkan pada gambar 7a dan 7b. Nilai SASA nonpolar mengalami peningkatan sebesar 420.353 Å2 dari nilai awalnya sebesar 1037.885 Å2 hingga 1458.238 Å2. Selain nilai SASA nonpolar, nilai dari SASA polar juga mengalami peningkatan sebesar 15.709 Å2 dari nilai awalnya 3651.208 Å2 menjadi 3666.91 Å2 pada nilai
akhirnya. Peningkatan nilai SASA menunjukkan bahwa luas permukaan yang bisa dijangkau oleh pelarut cukup besar sehingga inti hidrofobik protein mengalami kerusakan. Inti hidrofik yang rusak akibat pemberian temperatur tinggi membuat protein dalam keadaan tidak stabil atau unfolded.
Gambar 8a dan 8b mempresentasikan nilai SASA nonpolar dan polar yang mengalami penurunan dari nilai awalnya. Meskipun selama simulasi protein terus mengalami perubahan namun pada akhir simulasi protein menunjukkan penurunan nilai sebesar 127.118 Å2 untuk SASA nonpolar dan 38.389 Å2 untuk SASA polar. Penurunan nilai dari SASA nonpolar dan polar tersebut menunjukkan bahwa pada simuasi dengan temperatur rendah inti hidrofobik yang telah rusak mampu kembali ke struktur awalnya. 0 1 2 3 4 5 6 6.5 0 5 10 15 20 25 30 Waktu (ns) (a) Waktu (ns) (b)
Gambar 6 Energi konformasi hasil simulasi (a) unfolding pada 500 K selama 6.5 ns (b) refolding pada 200 K selama 30 ns
740 730 720 710 705 1415 1405 1395 1375 1365 E nergi K on for m as i (K ca l/ m o l) E nergi K on for m as i (K ca l/ m o l)
13
Ikatan Hidrogen
Ikatan hidrogen merupakan gaya tarik antarmolekul yang terjadi antara dua muatan listrik parsial dengan polaritas yang berlawanan. Kekuatannya dipengaruhi oleh perbedaan elektronegativitas antara atom-atom dalam molekul tersebut. Ikatan ini jauh lebih kecil dibandingkan ikatan kovalen dan ikatan ion tetapi merupakan salah satu ikatan penstabil protein.18
Analisis ikatan hidogen dapat dilakukan terhadap protein to protein dan protein to water. Analisis ikatan hidrogen protein to protein dilakukan untuk menghitung jumlah ikatan hidrogen pada permukaan dalam protein yang lebih bersifat hidrofobik sedangkan analisis protein to water digunakan untuk menghitung jumlah ikatan hidrogen diluar permukaan protein yang bersifat hidrofilik. Ikatan didefinisikan dengan jarak tertentu antara residu yang satu dengan residu atau molekul yang lainnya. Semakin tinggi jumlah ikatan hidrogen suatu protein maka dapat dikatakan ikatan hidrogen tersebut semakin kuat.
Gambar 9a dan 9b menunjukkan jumlah ikatan hidrogen protein to protein hasil simulasi unfolding 500 K selama 6.5 ns dan refolding 200 K selama 30 ns setelah diperhalus menggunakan moving avarage untuk setiap 50 frame.
Waktu (ns) (a) SASA NO N P O L AR (Å 2) 1600 1400 1200 1000 0 1 2 3 4 5 6 6.5 4000 3700 3500 3400 0 1 2 3 4 5 6 6.5 SASA P O L A R (Å 2) Waktu (ns) (b)
Gambar 7 Nilai SASA hasil simulasi unfolding pada 500 K selama 6.5 ns (a) nonpolar (b) polar
Waktu (ns) (a) 1420 1370 1300 1250 4800 4700 4600 3500 SASA P O L A R (Å 2) Waktu (ns) (b)
Gambar 8 Nilai SASA hasil simulasi refolding pada 200 K selama 30 ns (a) nonpolar (b) polar
0 5 10 15 20 25 30 0 5 10 15 20 25 30 SASA NO N P O L AR (Å 2)
14
Berdasarkan hasil analisis SASA didapatkan informasi bahwa dari hasil simulasi unfolding maupun refolding residu yang lebih berperan dalam proses pembentangan dan pelipatan kembali protein adalah residu yang bersifat nonpolar, sehingga diperlukan analisis ikatan hidrogen protein to protein untuk mendukung tingginya perubahan nilai pada analisis SASA nonpolar tersebut. Tingginya perubahan nilai SASA nonpolar akibat interaksi residu hidrofobik dengan molekul protein sehingga interaksi tersebut menyebabkan terbentangnya protein dan mempercepat proses unfolding. Hal yang sama terjadi saat proses refolding, perubahan nilai SASA nonpolar lebih tinggi dibanding nilai SASA polar.
Dari analisis ikatan hidrogen protein to protein, proses unfolding mencapai nilai 20 selama simulasi sedangkan untuk proses refolding nilai ikatan tertinggi mencapai 26. Nilai ikatan proses refolding lebih besar dibandingkan nilai ikatan saat unfolding menunjukkan bahwa saat proses refolding, inti hidrofobik lebih berperan kembali ke struktur awalnya. Meskipun interaksi inti hidrofobik dengan residu protein lebih besar saat refolding, protein belum mampu kembali ke struktur awalnya (native) untuk waktu singkat.
RMSF
RMSF adalah simpangan rata-rata masing-masing residu selama simulasi dinamika molekul dari konformasi awalnya. Nilai RMSF menggambarkan fleksibilitas pergerakan residu tersebut. Residu yang memiliki nilai RMSF tinggi memiliki arti bahwa residu tersebut terus berfluktuasi selama simulasi. Sedangkan residu yang memiliki nilai RMSF rendah artinya residu tersebut kaku (rigid).19
Grafik sudah diperhalus dengan moving average untuk setiap 50 frame. Gambar 10a menunjukkan kenaikan temperatur mampu mengubah fleksibilitas residu. Temperatur simulasi yang tinggi menyebabkan protein mendapatkan tambahan kalor sehingga residu bergerak lebih fleksibel dan nilai RMSF meningkat. Puncak-puncak yang muncul pada hasil RMSF simulasi 500 K mampu mencapai 12.828 Å. Sedangkan pada Gambar 10b memperlihatkan pada temperatur rendah 200 K nilai RMSF mengalami penurunan dan hanya mencapai 5.76 Å. Artinya pada temperatur tinggi residu cenderung bergerak aktif serta memiliki fleksibilitas yang
Waktu (ns) (a) 0 1 2 3 4 5 6 6.5 15 12 10 7 5 Juml ah Ika tan H idr ogen 12 10 8 6 4 Waktu (ns) (b) 0 5 10 15 20 25 30
Gambar 9 Ikatan hidrogen protein to protein hasil simulasi (a) unfolding pada 500 K selama 6.5 ns (b) refolding pada 200 K selama 30 ns
Juml ah Ika tan H idr ogen
15 tinggi sehingga mengalami unfolding sedangkan pada temperatur rendah residu memiliki pergerakan yang rendah dan membuat protein kembali ke keadaan folded. Pada temperatur tinggi hanya sebagian residu yang mampu memiliki fleksibilitas tinggi. Terlihat pada Gambar 10a puncak yang muncul dari hasil simulasi unfolding 500 K lebih sedikit dibanding puncak yang muncul pada simulasi 200 K. Puncak yang muncul saat proses unfolding berada pada struktur turn (A27, N28, G29) dan turn (K68, G69). Gambar 10b menunjukkan bahwa sebagian besar residu lebih kaku pada temperatur rendah dibandingkan pada saat temperatur tinggi. Namun, puncak yang muncul pada proses unfolding 500 K dan refolding 200 K sebagian besar berada pada struktur turn (A27, N28), coil (D57, N58), β-sheet (A77). 17 28 38 48 58 68 78 17 28 38 48 58 68 78 Residu (a) 14 12 10 8 4 9 7 5 3 1 RM S F ( Å )
Gambar 10 RMSF hasil simulasi (a) unfolding pada 500 K selama 6.5 ns (b) refolding pada 200 K selama 30 ns
RM S F ( Å ) (b) Residu
16
SIMPULAN DAN SARAN
Simpulan
Kestabilan termal dari protein 2PTL dianalisis menggunakan metode simulasi dinamika molekul. Dari penelitian ini diperoleh proses unfolding terjadi pada waktu 6.5 ns. Secara geometri hasil analisis RMSD unfolding pada 500K mencapai 7.059Å yang berada dalam keadaan unfolding parsial dalam range 2Å sampai 7Å dan untuk proses refolding terjadi penerunan sebesar 2.683Å. Protein yang sudah terdenaturasi mampu kembali ke keadaan native, dimana sebagian struktur sekunder yang hilang saat proses unfolding dapat muncul kembali saat proses refolding. Tingkat kemiripan asam amino proses unfolding dengan struktur native sebesar 36.78%. Setelah dilakukan proses refolding pada 200K dilakukan juga pengecekan tingkat kemiripan asam amino dengan struktur native sebesar 62.264% yang berarti protein belum melakukan refolding secara sempurna.
Saran
Simulasi refolding terhadap temperatur ekstrem masih belum sepenuhnya mengembalikan struktur sekunder protein 2PTL ke keadaan native. Simulasi harus dilanjutkan dengan waktu simulasi yang lebih panjang. Selain itu, variasi temperatur unfolding dan temperatur awal saat proses refolding juga patut dicoba untuk mendapatkan informasi yang lebih rinci mengenai dinamika protein. Selain itu, mekanisme pelipatan protein 2PTL masih belum dapat ditentukan model mekanisme yang seperti apa, juga bisa dilakukan mutasi pada residu yang memiliki fleksibilitas tinggi.
DAFTAR PUSTAKA
1. Murray, R. K., Graner, D. K., and Rodwell, V. W. Harper’s Ilustrated Biochemistry 28th edition. 2009.
2. Sari, Mayang. Identifikasi protein menggunakan fourier transform infrared. [skripsi]. Depok : Universitas Indonesia. 2011.
3. Shen T, Cao Y, Zuang S, Li H .Engineered Bi-Histidine metal chelation sites map the structure of the mechanical unfolding transition state of an elastomeric protein domain GB1. Biophysical Journal. 2012; 103: 807-816.
4. Jinhyuk Lee, Seokmin Shin. Understanding Betha Hairpin Formation by Molecular Dynamics Simulations of Unfolding. Biophysical Journal. 2001; 81: 2507–2516.
5. Li, Ju. Handbook of Materials Modeling. Department of Materials Science and Engineering, Ohio State University, USA. 2005.
6. N Meridian Road. Pierce Ig Binding Proteins (Protein A, G, A/G and L). Thermo Scientific. [diunduh 2015 Oktober 12].
17 7. Hati, Jellyta. Analisis kestabilan protein 1GB1 menggunakan simulasi dinamika
molekul. [skripsi]. Bogor : Institut Pertanian Bogor. 2014.
8. Pande, V. S., Rokhsar, D. S. Molecular dynamics simulations of unfolding and refolding of beta-hairpin fragment of protein G. Proc. Natl. Acad. Sci. USA. 1999; 96: 9062-9067.
9. Wolf, Maarten. Understanding amyloidogenesis through computer simulations. 2008.
10. Ruhle, Victor. Berendsen and Nose-Hoover thermostats. 2007.
11. Phillips, J. C., Braun, S., Wang, W., Gumbart, J., Tajkhorshid, E., Villa, E., Chipot, C., Skeel, R. D., Kale, L., Schulten, K., Scalable Molecular Dynamics with NAMD. Journal of Computational Chemistry. 2005; 26: 1781-1802. 12. Allen MP, Tildesley DJ. 1989. Computer Simulation of Liquids, Oxford
University Press, USA.
13. Darden T, York D, Pedersen L. 1993. Particle mesh Ewald: An N. log(N) method for Ewald sums in large systems. J. Chem. Phys. 98(12) : 10089-10092. 14. Bui JM, Gsponer J, Vendruscolo M, Dobson CM. 2009. Analysis of suband supra- motions in protein Gβ1 using molecular dynamics simulations. Biophyics Journal. 97: 2513-2520.
15. Martyna GJ, Tobias DJ, Klei ML. 1994. Constant pressure molecular dynamics algoritm. J. Chem. Phys. 101 (5): 4177-4189.
16. Micaelo, Nuno. Analysis of molecular simulation experiment [bahan presentasi] . Portugal: Universidade do Minho. 2010.
17. Sharma RD, Lynn AM, Sharma PK, Rajnee, Jawaid S. High temperature unfolding of Bacillus anthracis amidase-03 by molecular dynamics simulations. Bioinformation. 2009; 3(10):430-434.
18. Sawitri, Kania Nur. Pengaruh mutasi terhadap ketabilan termal protein 1GB1. [tesis]. Bogor : Institut Pertanian Bogor. 2014
19. Malau, Nya Daniaty. Kestabilan termal asam amino enzim lipase bacillus subtilis menggunakan simulasi dinamika molekul. [tesis]. Bogor : Institut Pertanian Bogor. 2015.
18
Lampiran 1 Diagram alir penelitian
Mulai
Persiapan Alat dan Bahan
Refolding Pemanasan Ekuilibrasi Production run Koordinat unfolded 2PTL Minimisasi Preparasi molekul protein Analisis Data Selesai Output : Data koordinat 2PTL hasil refolding Penyusunan laporan Unfolding Pemanasan Ekuilibrasi Production run Minimisasi Koordinat 2PTL.pdb Preparasi molekul protein Output : Data koordinat 2PTL hasil unfolding
19 Lampiran 2 Persentase kesesuaian urutan residu penyusun antara struktur protein
native dengan hasil simulasi refolding Struktur Sekunder Index Resitu Protein Native Index Residu Protein Hasil Refolding % kesesuaian Wild-type 500 K β-sheet β-sheet 1 18 s/d 26 20 s/d 26 62.264 % β-sheet 2 30 s/d 38 30 s/d 36 β-sheet 3 60 s/d 67 63 s/d 66 β-sheet 4 70 s/d 77 71 s/d 75 α-helix 40 s/d 54 44 s/d 58 turn 27 s/d 29 27 s/d 29 68 s/d 69 40 s/d 41 61 s/d 62 coil 17 17 s/d 19 39 37 s/d 38 55 s/d 59 43 59 s/d 60 70 78 76 s/d 78 Beta-Bridge - 42 310 - helix - 67 s/d 69
20
Lampiran 3 Karakteristik residu yang terdapat pada protein 2PTL
Struktur Sekunder Indeks Residu Kode Residu Jenis Residu
Coil 1 E Polar negatif 2 N Polar netral 3 K polar positif 4 E Polar negatif 5 E Polar negatif 6 T Polar netral 7 P Nonpolar 8 E Polar negatif 9 T Polar netral 10 P Nonpolar 11 E Polar negatif 12 T Polar netral 13 D Polar negatif 14 S Polar netral 15 E Polar negatif 16 E Polar negatif 17 E Polar negatif Betha sheet 18 V Nonpolar 19 T Polar netral 20 I Nonpolar 21 K polar positif 22 A Nonpolar 23 N Polar netral 24 L Nonpolar 25 I Nonpolar 26 F Aromatik Turn 27 A Nonpolar 28 N Polar netral 29 G Nonpolar Betha sheet 30 S Polar netral 31 T Polar netral 32 Q polar netral 33 T Polar netral 34 A Nonpolar 35 E Polar negatif 36 F Aromatik 37 K polar positif
21
38 Q polar netral
Coil 39 T Polar netral
Alpha helix 40 F Aromatik 41 E Polar negatif 42 K polar positif 43 A Nonpolar 44 T Polar netral 45 S Polar netral 46 E Polar negatif 47 A Nonpolar 48 Y Aromatik 49 A Nonpolar 50 Y Aromatik 51 A Nonpolar 52 D Polar negatif 53 T Polar netral 54 L Nonpolar Coil 55 K polar positif 56 K polar positif 57 D Polar negatif 58 N Polar netral 59 G Nonpolar Betha sheet 60 E Polar negatif 61 Y Aromatik 62 T Polar netral 63 V Nonpolar 64 D Polar negatif 65 V Nonpolar 66 A Nonpolar 67 D Polar negatif
Turn 68 K polar positif
69 G Nonpolar Betha sheet 70 Y Aromatik 71 T Polar netral 72 L Nonpolar 73 N Polar netral 74 I Nonpolar 75 K polar positif 76 F Aromatik 77 A Nonpolar Coil 78 G Nonpolar
22
RIWAYAT HIDUP
Penulis dilahirkan di Tangerang pada tanggal 08 Maret 1994 dari Ayah Sudrajat dan Ibu Dedeh Maryati. Penulis adalah seorang anak tunggal. Pada tahun 2012 penulis berhasil menyelesaikan studi di SMA Negeri 19 Kabupaten Tangerang dan pada tahun yang sama penulis lulus seleksi masuk Institut Pertanian Bogor (IPB) melalui seleksi nasional masuk perguruan tinggi negeri (SNMPTN) jalur undangan dan diterima di Departemen Fisika, Fakultas Matematika dan Ilmu Pengetahuan Alam.
Selama mengukuti perkuliahan, penulis aktif menjadi asisten praktikum Biofisika Umum di Departemen Fisika IPB. Penulis juga aktif sebagai sekretaris 1 di Green Power Club IPB selama periode 2014 s/d 2015. Selain itu pada tahun 2016, penulis dan team berhasil mendapatkan dana hibah dari DIKTI untuk Program Kreatifitas Mahasiswa bidang Penelitian Eksakta (PKM-PE).