KERAGAMAN GENETIK Shorea laevis Ridl.
DI KALIMANTAN BERDASARKAN PENANDA
MIKROSATELIT
ASEP MULYADIANA
DEPARTEMEN SILVIKULTUR
FAKULTAS KEHUTANAN
INSTITUT PERTANIAN BOGOR
KERAGAMAN GENETIK Shorea laevis Ridl.
DI KALIMANTAN BERDASARKAN PENANDA
MIKROSATELIT
ASEP MULYADIANA
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar Sarjana Kehutanan pada Fakultas Kehutanan
Institut Pertanian Bogor
DEPARTEMEN SILVIKULTUR
FAKULTAS KEHUTANAN
INSTITUT PERTANIAN BOGOR
Keragaman Genetik Shorea laevis Ridl. Di Kalimantan Berdasarkan Penanda Mikrosatelit
Oleh:
Asep Mulyadiana, Iskandar Z. Siregar dan Edje Djamhuri
Shorea laevis Ridl. adalah salah satu jenis komersial dari famili
Depterocarpaceae. Jenis ini merupakan salah satu jenis yang sangat penting ditinjau dari segi ekologi maupun ekonominya. Secara umum, S. laevis menyebar di Semenanjung Myanmar dan Thailand, Semenanjung Malaysia, Sumatera, Aceh, dan Pulau Kalimantan. Penelitian ini bertujuan menentukan cross spesies
amplification (seleksi primer) mikrosatelit dan keragaman genetik di dalam dan
antar populasi S. laevis yang tumbuh di Pulau Kalimantan. Pada penelitian ini menggunakan DNA inti mikrosatelit dan diamati alel serta lokus polimorfiknya. Hasil analisis menunjukan S. laevis memiliki nilai diferensiasi genetik (He) = 0,4443. Secara umum nilai diferensiasi genetik ini lebih kecil bila dibandingkan dengan jenis Shorea lainnya yang juga dianalisis dengan menggunakan metode mikrosatelit. Hubungan kekerabatan terdekat (jarak genetik terkecil) dimiliki oleh populasi Bukit Bangkirai dengan populasi PT. ITCIKU dan hubungan kekerabatan terjauh (jarak genetik terbesar) dimiliki oleh populasi Batu Ampar dan populasi Sari Bumi Kusuma.
Genetic Variation of Shorea laevis Ridl. In Kalimantan With Microsatellite Marker
By:
Asep Mulyadiana, Iskandar Z. Siregar and Edje Djamhuri
Shorea laevis Ridl. is one member of commercial species from
Dipterocarpaceae family. This species is also as one of the important trees in tropical rain forest, based on ecological and economic evaluation. In general, S.
laevis distributes naturally in Peninsular Myanmar and Thailand, Peninsular
Malaysia, Sumatera, Aceh, and Borneo Island. The objective of this study was to cross species amplify microsatellites and to estimate genetic variation within and among populations. This study used nuclear microsatellite analysis based on observed alleles and polymorphic loci. Results showed that S. laevis had moderate value of genetic variation within population (He) = 0,4443. In general, this value of genetic variation was smaller if compared to other Shorea species that also analysed by the same method. The closest genetic relationship (smallest distance) was possessed by S. laevis populations of Bukit Bangkirai and PT. ITCIKU, while the furthermost relationship (largest distance) was possessed populations of Batu Ampar and PT. Sari Bumi Kusuma.
PERNYATAAN
Dengan ini saya menyatakan bahwa skripsi yang berjudul “Keragaman
Genetik Shorea laevis Ridl. Di Kalimantan Berdasarkan Penanda Mikrosatelit” adalah benar-benar hasil karya sendiri dengan bimbingan dosen
pembimbing yang belum pernah digunakan sebagai karya pada perguruan tinggi atau lembaga manapun. Sumber informasi yang berasal atau dikutif dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Bogor, Februari 2010
Asep Mulyadiana NRP E44052375
6
Judul Skripsi : Keragaman Genetik Shorea laevis Ridl. Di Kalimantan Berdasarkan Penanda Mikrosatelit.
Nama : Asep Mulyadiana
NIM : E44052375
Menyetujui:
Pembimbing I, Pembimbing II,
(Dr. Ir. Iskandar Z. Siregar, M.For.Sc) (Ir. Edje Djamhuri) NIP: 19660320 199002 1 001 NIP: 19500215 197412 1 001
Mengetahui:
Ketua Departemen Silvikultur Fakultas Kehutanan IPB,
(Prof. Dr. Ir. Bambang Hero Saharjo, M.Agr) NIP:19641110 199002 1 001
KATA PENGANTAR
Segala puji dan syukur penulis panjatkan kehadirat Allah SWT yang mana atas segala rahmat dan karunia-Nya penulis dapat menyelesaikan skripsi ini. Shalawat dan salam semoga tercurah kepada baginda Rasulullah SAW, suri tauladan yang telah memberikan petunjuk kepada seluruh umat manusia.
Skripsi ini merupakan salah satu syarat dalam mendapatkan gelar Sarjana Kehutanan di Fakultas Kehutanan Institut Pertanian Bogor. Adapun judul Skripsi ini adalah Keragaman Genetik Shorea laevis Ridl. Di Kalimantan Berdasarkan Penanda Mikrosatelit. Shorea laevis Ridl. adalah salah satu jenis komersial dari famili Depterocarpaceae. Pada penelitian ini menggunakan DNA inti mikrosatelit dan mengamati alel serta lokus polimorfiknya. Mikrosatelit adalah sekuen sederhana yang berulang-ulang dan melimpah dalam genom suatu spesies. Mikrosatelit memiliki pengulangan sekuen yang berurutan dua sampai 4 motif sekuen nukleotida sebagai sekuen konservatif. Marka ini sangat berguna sebagai marka genetik karena bersifat kodominan, sehingga dapat mendeteksi keragaman alel pada level yang tinggi, mudah dan ekonomis dalam pengaplikasiannya karena menggunakan proses PCR.
Penulis menyadari bahwa skripsi ini masih banyak kekurangan. Oleh karena itu dengan kerendahan hati, penulis meminta maaf dan mengharapkan kritik serta saran yang membangun demi masukan dan perbaikan untuk penelitian yang akan datang. Semoga hasil dari penelitian ini dapat memberikan manfaat bagi penulis maupun semua pihak yang membutuhkan.
Bogor, Februari 2010
RIWAYAT HIDUP
Penulis lahir di Ciamis, Jawa Barat pada tanggal 8 April 1986 sebagai putra dari pasangan Drs. Muksin Samsudin dan I. Kusmiati. Penulis merupakan anak kedua dari dua bersaudara.
Pendidikan dasar diselesaikan pada tahun 1999 di SDN Selacai I. Pendidikan lanjutan menengah pertama diselesaikan pada tahun 2002 di MTsN Buniseuri I. Pada tahun 2005, Penulis lulus dari MAN 2 Ciamis dimana pada tahun yang sama lulus seleksi masuk IPB jalur Undangan Seleksi Masuk IPB (USMI). Penulis diterima sebagai mahasiswa di Departemen Silvikultur dengan sistem Mayor-Minor pada tahun 2005.
Selama menuntut ilmu di IPB, penulis aktif di beberapa organisasi kemahasiswaan yakni pada periode tahun 2007/2008 sebagai staf Scientific
Improvement 2007/2008, aktif di organisasi AFSA dan IFSA 2007/2008. Selain
itu penulis pernah menjadi asisten untuk program Sarjana pada mata kuliah Inventarisasi Sumberdaya Hutan tahun ajaran 2007/2008, mata kuliah Silvikultur dan Genetika Hutan tahun ajaran 2008/2009 dan 2009/2010.
Selama perkuliahan, penulis mengikuti Praktek Pengenalan Ekosistem Hutan (P2EH) yang dilaksanakan di Linggarjati-Indramayu. Praktek Pengelolaan Hutan dilaksanakan di Hutan Pendidikan Gunung Walat (HPGW) Sukabumi. Praktek Kerja Profesi (PKP) dilaksanakan di Dinas Kehutanan dan Perkebunan Ciamis dari bulan Maret sampai bulan Mei 2009. Untuk memperoleh gelar Sarjana Kehutanan IPB, penulis menyelesaikan skripsi dengan judul Keragaman Genetik Shorea laevis Ridl. Di Kalimantan Berdasarkan Penanda Mikrosatelit.
UCAPAN TERIMA KASIH
Puji dan syukur penulis panjatkan kehadirat Allah SWT karena berkat rahmat dan hidayah-Nya penulis dapat menyelesaikan skripsi ini. Selama melakukan penelitian dan penulisan skripsi ini, penulis banyak dibantu oleh berbagai pihak. Untuk itu penulis mengucapkan terima kasih kepada
1. Dr. Ir Iskandar Z. Siregar, M. For. Sc. Dan Ir. Edje Djamhuri atas bimbingan dan ilmu yang telah diberikan.
2. Dr. Ir. I Nyoman J. Wistara, M.Sc, Ir. Edi Sandra, M.Si dan Ir. Sudaryanto selaku dosen penguji.
3. Ayahanda tercinta Drs. Muksin Samsudin dan Ibunda tercinta I. Kusmiati, kakakku tersayang Nia Kurniasari dan Erwin Tedjasomantri, keponakan tercinta Edra Abiyyu Reswara serta seluruh keluarga besar yang telah menyemangati dan mendoakan penulis.
4. Tedi Yunanto S. Hut. M. Si yang telah membantu penulis selama penelitian dan penyusunan skripsi ini.
5. Teman-teman di Laboratorium Genetik Silvikultur (Fifi Gus Dwiyanti, Elviana, Utari O. Mahfira, Fitri Wulansari)
6. Sahabat-sahabat terbaik penulis di Silvikultur 42 terimakasih atas persahabatannya dan dukungannya.
7. Rekan-rekan Silvikultur angkatan 43, 44 dan 45 (Tree Grower Community)
8. Rekan-rekan di IFSA dan AFSA LC IPB.
9. Keluarga Besar Paguyuban Mahasiswa Galuh Ciamis (PMGC) 10. Civitas akademik FAHUTAN atas kekeluargaannya selama ini. 11. Seluruh civitas akademik Institut Pertanian Bogor.
Penulis berharap karya ini dapat bermanfaat dan berguna bagi semua pihak dan mohon maaf atas segala kekurangannya.
Bogor, Februari 2010
DAFTAR ISI
Halaman
KATA PENGANTAR ……….. i
UCAPAN TERIMA KASIH... ii
DAFTAR ISI ... iii
DAFTAR TABEL ... v
DAFTAR GAMBAR... vi
DAFTAR LAMPIRAN ... vii
BAB I PENDAHULUAN 1.1 Latar Belakang ... 1
1.2 Tujuan ………... 3
1.3 Hipotesis ………... 3
1.4 Manfaat Penelitian ……… 3
BAB II TINJAUAN PUSTAKA 2.1 Deskripsi Singkat Shorea laevis Ridl……… 4
2.2 Deoksiribonucleat Acid (DNA) ……….. 4
2.3 Keragaman atau Variasi Genetik Tanaman ……….. 5
2.4 Pengukuran Variasi Genetik ………. 6
2.4.1 Mengukur Variasi Genetik dalam Populasi ……... 6
2.4.2 Mengukur Variasi Genetik antar Populasi ………. 7
2.5 PCR ( Polymerase Chain Reaction) ………. 8
2.6 Mikrosatelit ……….. 10
BAB III METODE PENELITIAN 3.1 Tempat dan Waktu ………... 12
3.2 Bahan dan Alat ………. 12
3.2.1 Populasi Penelitian ………. 12
3.2.2 Alat dan Bahan Analisis Genetik ………... 13
3.3 Prosedur Penelitian ………... 15
3.3.2 Ekstraksi DNA ………... 15
3.3.3 Uji Kualitas dan Kuantitas DNA ……… 16
3.3.4 Polymerase Chain Reaction (PCR) Mikrosatelit ... 17
11
3.3.5 Pembuatan Gel Poliakrilamid ……… 19
3.3.5.1 Pencucian piringan kaca ……… 19
3.3.5.2 Pembuatan larutan akrilamid ………. 19
3.3.5.3 Loading sample ………. 19
3.3.5.4 Metode Pewarnaan ………. 20
3.4 Analisis Data………. 20
BAB IV HASIL DAN PEMBAHASAN 4.1 Ekstraksi DNA ………. 23
4.2 Seleksi Primer (Cross Spesies Amplification) ... 24
4.3 Interpretasi dan Analisis Data ……….. 27
4.3.1 Keragaman Genetik dalam Populasi ... 27
4.3.2 Keragaman Genetik antar Populasi ... 30
BAB V KESIMPULAN DAN SARAN... 34
5.1 Kesimpulan……… 34
5.2 Saran……….. 34
DAFTAR PUSTAKA……… 35
DAFTAR TABEL
No Judul Halaman
1. Koordinat lokasi populasi pengambilan daun pohon contoh
Shorea laevis……….... 12
2. Alat dan bahan untuk teknik analisis genetik……….. 14
3. Komposisi bahan untuk reaksi PCR Mikrosatelit………... 18
4. Primer spesifik pada jenis Shorea leprosula………... 19
5. Primer spesifik pada jenis Shorea curtisii………... 19
6. Tahapan-tahapan dalam proses PCR Mikrosatelit………... 20
7. Perbandingan ukuran pasangan basa (base pairs) ……….. 27
8 Peubah yang mencirikan keragaman genetik dalam populasi Shorea laevis……… 28
9 Keragaman genetik beberapa jenis meranti dan Jati……… 28
DAFTAR GAMBAR
No. Judul Halaman
1. Ilustrasi siklus PCR ………... 8
2. Peta lokasi populasi pengambilan daun pohon contoh Shorea laevis……….. 13
3. Bagan Alur Penelitian……… 15
4. Cara skoring DNA mikrosatelit……….. 22
5. Pola pita DNA hasil ekstraksi dan isolasi dari daun Shorea laevis……….. 23
6. Contoh besarnya pengenceran hasil ekstraksi DNA... 24
7. Foto hasil seleksi primer... 26
8 Hubungan antara jarak genetik dengan jarak fisik………. 31
DAFTAR LAMPIRAN
No. Judul Halaman
1. Instrumen/alat-alat penelitian……… 39 2. Hasil Skoring pita DNA daun Shorea laevis………. 40
BAB I
PENDAHULUAN
1.1 Latar Belakang
Indonesia merupakan salah satu negara yang memiliki hutan tropis terbesar di dunia. Kalimantan adalah salah satu pulau di Indonesia yang memiliki hutan tropis tersebut dimana hutan tropis ini banyak didominasi oleh famili Dipterocarpaceae. Famili Dipterocarpaceae memiliki tiga sub famili
Dipterocarpadeae, Pakaraimoideae, dan Monotoideae. Diantara sub famili
tersebut yang terpenting adalah Dipterocarpadeae karena mempunyai jumlah jenis yang banyak dan diantaranya dapat diperdagangkan (Anonim 1991).
Shorea laevis Ridl. atau bangkirai merupakan salah satu jenis komersial dari famili Dipterocarpaceae yang ada di Indonesia. Jenis ini sangat penting baik dari segi ekologi maupun ekonominya, terutama pada pemanfaatan kayunya. Kayu Bangkirai di daerah Kalimantan diguanakan sebagai kayu pertukangan pengganti kayu Ulin (Eusideroxylon zwageri), mengingat kayu Ulin sudah semakin jarang ditemukan di hutan alam. Secara umum, S. laevis menyebar di Semenanjung Myanmar dan Thailand, Semenanjung Malaysia, Sumatera, Aceh, dan P. Kalimantan. Jenis ini tersebar luas dan sering dijumpai dan bahkan berkelompok pada tanah-tanah tipis di punggung bukit di hutan Dipterocarpaceae perbukitan, khususnya pada ketinggian 200-1000 m, tetapi kadang lebih rendah antara (5-375 mdpl), adakalanya terpencar di sisi-sisi bukit (Prawira et al. 1973). Sampai saat ini, jenis S. laevis secara umum masih jarang diteliti baik dari segi populasi genetik maupun konservasi genetiknya.
Pengetahuan tentang keragaman genetik sangat penting karena akan memeberikan suatu informasi dasar dalam pengembangan tanaman selanjutnya. Menurut Wulandari (2008) keragaman genetik digunakan sebagai bahan seleksi genotipe yang dikehendaki. Salah satu pendekatan yang dilakukan untuk estimasi variabilitas genotipe adalah dengan menggunakan metode baru berdasarkan analisis molekuler. Pengembangan bidang molekuler dengan analisis DNA sudah sering digunakan untuk mengkarakterisasi variasi genetik dan kekerabatan dalam satu genus, spesies, kultivar atau aksesi.
Menurut Henry (1997) informasi yang dihasilkan dari analisis DNA berguna untuk penentuan hubungan kekerabatan dan filogenetik individu setelah mengalami evolusi karena pengaruh waktu, tempat dan varietas yang digunakan. Dewasa ini telah berkembang berbagai jenis penanda molekuler diantaranya adalah isoenzim, RAPD (Random Amplified Polymorfik DNA), RFLP (Restriction
Fragment Length Polymorphism), AFLP (Amplified Fragment Length Polymorphisms), SSR (Simple Sequence Repeat) atau mikrosatelit. Analisis
molekuler dapat memberikan perbedaan yang jelas dengan melihat perbedaan pola pita DNA.
Pendugaan ragam atau variasi genetik dalam beberapa penelitian umumnya menggunakan bahan tanaman berupa daun. Salah satu metode penanda genetik yang dapat diaplikasikan untuk analisis DNA adalah mikrosatelit. Mikrosatelit telah diidentifikasi pada DNA plastid (DNA kloroplas dan DNA mitokondria) dan juga pada DNA inti. Mikrosatelit inti biasanya bersifat ko-dominan. Mikrosatelit terutama sangat berguna untuk studi aliran gen dan sistem perkawinan dari suatu jenis, karena seringkali mikrosatelit menunjukkan variasi yang luas. Menurut Finkeldey (2005) banyak penelitian-penelitian yang telah dilakukan dengan menggunakan lokus mikrosatelit untuk beberapa genus pohon tropika, meliputi
Eucalyptus spp., Shorea spp. dan Dipterocarpaceae lainnya.
Mikrosatelit adalah sekuen sederhana yang berulang-ulang dan melimpah dalam genom suatu spesies. Mikrosatelit memiliki pengulangan sekuen yang berurutan dua sampai 4 motif sekuen nukleotida sebagai sekuen konservatif. Marka ini sangat berguna sebagai marka genetik karena bersifat kodominan, sehingga dapat mendeteksi keragaman alel pada level yang tinggi, mudah dan ekonomis dalam pengaplikasiannya karena menggunakan proses PCR. Bentuk pengulangan sekuen DNA sederhana yang berulang-ulang menjadikan marka mikrosatelit sering disebut simple sequence repeat (SSR), short tandem
repeats (STRs) atau simple sequence length polymorphisms (SSLPs) yang
sekarang menjadi salah satu marka paling banyak digunakan secara luas untuk pemetaan genetik, analisis keragaman genetik, dan studi evolusi. Marka atau penanda ini muncul sebagai marka yang sangat variatif dan mudah diulang, menjadikan sangat ideal untuk pemetaan genom. Mikrosatelit ini merupakan salah
3
satu tipe polimorfisme yang berulang-ulang, yang biasa dikelompokkan ke dalam
simple tandem repeat polymorphism (STRP), karena perbedaan genetik di antara
molekul-molekul DNA yang mengandung sejumlah kopi sekuen DNA pendek yang diulang beberapa kali. STRP yang memiliki pengulangan 2-9 pasang basa sering disebut mikrosatelit, sedangkan STRP dengan pengulangan 10-60 pasang basa sering disebut minisatelit atau variable number of tandem repeats (VNTR) (Prasetiyono et al 2004).
1.2 Tujuan
Tujuan dari penelitian ini adalah mengetahui keberhasilan cross species
amplification (seleksi primer) mikrosatelit untuk jenis S. laevis, mengetahui
keragaman genetik di dalam dan antar populasi S. laevis yang tumbuh di beberapa tempat di Pulau Kalimantan.
1.3 Hipotesis
Hipotesis yang diuji adalah adanya keragaman genetik di dalam dan antar populasi S. laevis di beberapa tempat di Pulau Kalimantan.
1.4 Manfaat
Manfaat yang diharapkan dari hasil penelitian ini adalah memberikan informasi dasar sebagai landasan ilmiah tentang pola keragaman genetik baik di dalam maupun antar populasi untuk kegiatan konservasi sumberdaya genetik dan pemuliaan tanaman S. laevis serta kegiatan perbaikan genetik pohon S. laevis di masa datang.
BAB II
TINJAUAN PUSTAKA
2.1 Deskripsi Singkat Shorea laevis Ridl.
Menurut Martawijaya et al (1981) Shorea laevis merupakan pohon yang memiliki tinggi pohon mencapai 50 m dengan panjang batang bebas cabang mencapai 35-45 m, diameter pohon mencapai 100 cm lebih, tinggi banir mencapai 2 m. kulit kayu berwarna kelabu, merah atau cokelat, kadang-kadang sampai merah tua, beralur dan mengelupas kecil-kecil, tipis, berdamar warna kuning tua. Kayu teras berwarna kunig-cokelat dan kayu gubal berwarna cokelat muda pucat kekuning-kuningan. Tekstur kayu halus sampai agak kasar. Arah serat berupa lurus atau berpadu. Permukaan kayu licin atau berganti-ganti antara licin dan kesat karena arah serat yang berpadu, dimana pemukaan kayu mengkilap.
Jenis ini tumbuh bersama-sama dengan jenis-jenis Dipterocarpaceae lainnya di dalam hutan tropis dengan tipe curah hujan A dan B, pada tanah pasir, basalt laterit tua dan podsolik, terutama pada tanah yang datar dan sering digenangi air tawar secara bermusim, dapat juga tumbuh di bukit-bukit secara berkelompok atau berpencar, pada ketinggian sampai 400 m dari permukaan laut. Pohon berbuah tidak menentu, sangat tergantung kepada keadaan iklim dan kadang- kadang berbuah banyak selang 3-7 tahun.
Kayu bangkirai banyak dipergunakan untuk kontruksi berat di bawah atap maupun di tempat terbuka, antara lain untuk bangunan jembatan, bantalan tiang listrik, lantai, bangunan maritim, perkapalan, karoseri dan perumahan. Kayu bangkirai ini memiliki kekuatan dan keawetan yang tinggi, dimana termasuk kelas awet I – II. Kayu bangkirai tidak baik untuk pembuatan venir dan kayu lapis, karena keras dan mempunyai berat jenis yang tinggi.
2.2 Deoksiribonucleat Acid (DNA)
Menurut Crowder (1986), DNA adalah bahan yang diwariskan dan merupakan unsur pokok kromosom yang disebut nuklein atau bahan yang ada hubungannya dengan nukleus. Sedangkan menurut Finkeldey (2005), DNA adalah material dasar dari hereditas dan makromolekul biologi untuk
5
penyimpanan informasi genetik. Elrod dan Stansfield (2002) mengatakan bahwa DNA adalah suatu polimer panjang yaitu sebuah makromolekul yang tersusun atas sejumlah sub-unit yang serupa atau identik (monomer) yang berikatan secara kovalen yang tersusun atas ribuan pasangan basa (base pair, bp) nukleotida. Molekul DNA terdiri atas nukleotida yang sangat panjang. Tiap nukleotida terdiri dari satu pentosa (satu gula deoksiribosa), satu fosfat, dan satu dari empat basa Adenin (A), Timin (T), Citosin (C) atau Guanin (G). Satu nukleotida diikat dengan nukleotida berikutnya dengan ikatan kovalen gula-fosfat. Informasi yang terkandung dalam rantai nukleotida tersebut tergantung pada sekuensi empat basa yaitu A, T, C, dan G yang dapat dipilih secara bebas. Pada tahap ini informasi yang dikandung pada rantai nukleotida dapat dibandingkan dengan bahasa yang ditulis hanya dengan empat huruf, yakni A, T, C, dan G. Sekuensi dari empat basa tersebut mengandung semua informasi yang dapat diturunkan (Finkeldey 2005).
DNA dapat dijumpai dalam inti sel dan pada beberapa organel lainnya. Pada tumbuhan tingkat tinggi DNA dijumpai hanya sebatas pada inti sel, mitokondria, dan kloroplas (Finkeldey 2005).
2.3 Keragaman atau Variasi Genetik Tanaman
Keragaman genetik dapat terjadi karena adanya perubahan nukleotida
penyusun DNA. Perubahan itu mungkin dapat mempengaruhi fenotipe suatu organisme yang dapat dipantau dengan mata telanjang atau mempengaruhi reaksi individu terhadap lingkungan tertentu. Secara umum keanekaragaman genetika suatu populasi dapat terjadi karena adanya mutasi dan rekombinasi. Di samping itu struktur genetika dari suatu populasi dipengaruhi oleh beberapa faktor seperti besarnya populasi, cara reproduksi dan seleksi (Finkeldey 2005).
Menurut Soerianegara dan Djamhuri (1979), dalam suatu jenis pohon dapat dijumpai beberapa variasi yaitu variasi geografis (antar provenansi), variasi lokal (antar tempat tumbuh, antar tapak), variasi antar pohon, dan variasi di dalam pohon. Ada dua sebab utama yang menimbulkan variasi, yaitu perbedaan lingkungan dan perbedaan struktur genetik. Variasi yang disebabkan oleh perbedaan keadaan tempat tumbuh, sifat-sifat tanah dan jarak tanam adalah variasi
6
yang disebabkan oleh lingkungan (environmental variation). Sedangkan variasi yang tidak dapat diterangkan dengan perbedaan tempat tumbuh, misalnya perbedaan bentuk batang, tebal cabang dan berat jenis kayu dari pohon-pohon dalam suatu tegakan adalah variasi yang banyak dipengaruhi oleh perbedaan genetik yang diturunkan dari tetua kepada keturunannya, dan disebut variasi genetik (genetic variation).
Variasi genetik dari suatu keturunan merupakan hasil dari perkembangbiakan seksual. Perkembangbiakan secara seksual terjadi dengan adanya sel reproduktif (gamet) jantan dan betina melalui proses meiosis dan selanjutnya terjadi proses reduksi jumlah kromosom dari diploid (2n) dalam sel tetua menjadi haploid (n) dalam gamet, mengikuti hukum segregasi secara bebas (Hukum Mendel 1). Selanjutnya pada saat perkawinan terjadi rekombinasi gamet secara acak menurut hukum Mendel 2. Selain itu pada saat meiosis, kromosom homolog juga akan mengalami pindah silang dan kadang-kadang terjadi perubahan susunan genetik karena mutasi, aliran gen dan migrasi, seleksi dan sistem perkawinan (Finkeldey 2005).
2.4 Pengukuran Keragaman Genetik
2.4.1 Mengukur Keragaman Genetik dalam populasi
Menurut Finkeldey (2005), dalam mengukur keragaman atau variasi genetik dalam populasi sering menggunakan parameter-parameter sebagai berikut:
(1) Presentase Lokus Polimorfik (PLP)
Suatu lokus gen dikatakan polimorfik jika sekurang-kurangnya ada dua varian yang berbeda (alel) dijumpai. Suatu lokus gen dikatakan monomorfik jika tidak memperlihatkan variasi genetik. Suatu lokus gen dikatakan polimorfik jika frekuensi dari alel yang paling sering ditemukan adalah kurang dari 95% menurut definisi yang secara luas digunakan yaitu "kriteria 95%".
(2) Multiplisitas genetik dan rata-rata jumlah alel per lokus (A/L)
Multiplisitas alelik dari populasi pada lokus tunggal adalah jumlah alel yang diamati tanpa memandang frekuensi alelnya. Jumlah rata-rata alel tiap lokus gen (A/L) dihitung dengan menjumlahkan semua alel yang diamati pada lokus gen dan membaginya dengan jumlah lokus (L).
7
(3) Keragaman genetik atau heterozigositas
Beberapa ukuran keragaman atau variasi genetik mempertimbangkan perbedaan frekuensi dari tipe-tipe genetik (alel dan genotipe) dalam suatu popolasi. Ukuran variasi yang sering digunakan adalah heterozigositas harapan (He). Frekuensi relatif individu heterozigot yang diamati (Ho), yaitu suatu ukuran berdasarkan struktur genetik juga digunakan untuk mengkarakterisasi variasi genetik dalam populasi.
2.4.2 Mengukur Keragaman Genetik antar Populasi
Menurut Finkeldey (2005), parameter - parameter yang digunakan untuk mengukur keragaman genetik antar populasi yaitu:
(1) Jarak genetik
Jarak genetik digunakan untuk mengukur perbedaan struktur genetik antar dua populasi pada suatu lokus gen tertentu. Sebagian besar tetapi tidak semua ukuran jarak genetik bervariasi antara 0 dan 1. Nilai minimum 0 diperoleh jika struktur genetik dari dua populasi identik, sedangkan nilai maksimum 1 dicapai jika dua populasi tidak membagi apapun tipe genetik (alel atau genotipe). Perbedaan genetik lebih dari dua populasi biasanya dianalisa oleh sebuah matrik dengan elemen-elemen berupa jarak genetik dengan pasangan kombinasinya yaitu populasi.
(2) Pembagian variasi genetik (FST dan GST)
FST atau GST adalah suatu ukuran diferensiasi relatif terhadap keseluruhan keragaman. Konsep pembagian variasi genetik dalam sebuah komponen dan satu komponen di antara populasi (FST) merupakan ukuran yang lebih luas digunakan apabila dibandingkan dengan perhitungan diferensiasi genetik.
(3) Analisis klaster/kelompok
Analisis klaster adalah metode untuk menggambarkan perbedaan genetik antar populasi secara geografis. Hal ini didasarkan atas perhitungan jarak genetik. Populasi dengan jarak genetik kecil, yaitu populasi yang secara genetik sama, bersatu pertama kali dan kemudian bersatu lagi dengan populasi yang secara genetik berbeda jarak. Dalam cara ini, pohon keturunan dapat digambarkan untuk mempermudah pengkajian pola diferensiasi genetik populasi-populasi. Populasi
8
tunggal atau OTUs (Original Taxonomic Units) biasanya berklaster menjadi beberapa cabang pohon. Analisis klaster dapat digunakan untuk mengetahui keberadaan hibrid (Finkeldey 2005).
Variasi genetik tanaman dapat dianalisis menggunakan teknik penanda genetik sebagai alat bantu mengidentifikasi genotipe suatu individu. Penanda genetik yang terpilih untuk diamati adalah penanda yang terpaut dengan sifat/karakter yang menjadi sasaran penelitian. Macam penanda genetik yang sering digunakan antara lain penanda morfologi, penanda biokimia atau penanda isoenzim dan penanda molekuler (Finkeldey 2005).
2.5 PCR ( Polymerase Chain Reaction)
Kary B. Mullis mengembangkan Polymerase Chain Reaction (PCR) pada tahun 1983. Pada mulanya metode untuk menghasilkan penggandaan rantai spesifik DNA membutuhkan banyak waktu dan mahal. Akan tetapi PCR dapat menghasilkan sejuta kopi dari gen tunggal atau beberapa potong DNA dengan cepat pada sebuah tabung reaksi kecil. PCR sangat spesifik, dimana rantai DNA target yang diperlukan dapat mencapai kurang dari seperjuta dari total contoh DNA. Ini berarti bahwa gen tunggal atau potongan kecil DNA termasuk seluruh gen manusia dapat diamplifikasi menggunakan PCR (Mader 2001).
9
Secara prinsip, PCR merupakan proses yang diulang-ulang antara 20–30 kali. Setiap siklus terdiri dari tiga tahap. Berikut adalah tiga tahap bekerjanya PCR dalam satu siklus, yaitu:
(1) Tahap peleburan atau denaturasi
Pada tahap ini (berlangsung pada suhu tinggi, 94–96°C) ikatan hidrogen DNA terputus (denaturasi) dan DNA menjadi berberkas tunggal. Biasanya pada tahap awal PCR tahap ini dilakukan agak lama (sampai 5 menit) untuk memastikan semua berkas DNA terpisah. Pemisahan ini menyebabkan DNA tidak stabil dan siap menjadi template ("patokan") bagi primer. Durasi tahap ini 1–2 menit.
(2) Tahap penempelan atau annealing
Primer menempel pada bagian DNA template (cetakan) yang komplementer urutan basanya. Ini dilakukan pada suhu antara 45–60°C. Penempelan ini bersifat spesifik. Suhu yang tidak tepat menyebabkan tidak terjadinya penempelan atau primer menempel di sembarang tempat. Durasi tahap ini 1–2 menit.
Menurut Promega (2003) suhu annealing ditentukan oleh persamaan sebagai berikut:
Tm = 81.5 + 16.6 (log M) + 0.41 (%GC) – (675/n) Keterangan:
Tm = Suhu annealing (dalam oC)
M = Konsentrasi garam dalam buffer (mM)
G = Banyaknya basa guanin dalam primer yang digunakan C = Banyaknya basa sitosin dalam primer yang digunakan n = Panjang primer (dalam bp)
(3) Tahap pemanjangan atau extension
Suhu untuk proses ini tergantung dari jenis DNA polimerase yang dipakai. Dengan Taq polimerase, proses ini biasanya dilakukan pada suhu 76°C. Durasi tahap ini biasanya 1 menit.
Teknik PCR dapat digunakan dengan metode mikrosatelit. Mikrosatelit terutama sangat berguna untuk studi aliran gen dan sistem perkawinan dari suatu jenis, karena seringkali mikrosatelit menunjukkan variasi yang luas (Lefort 1999 diacu dalam Finkeldey 2005).
10
2.6 Mikrosatelit
Mikrosatelit merupakan rangkaian pola nukleotida antara dua sampai enam pasang basa yang berulang secara berurutan. Mikrosatelit biasa digunakan sebagai penanda genetik untuk menguji kemurnian galur, studi filogenetik, lokus pengendali sifat kuantitatif dan forensik. Mikrosatelit diamplifikasi menggunakan teknik PCR dengan beberapa pasang mikrosatelit. Hasil PCR dideteksi menggunakan teknik elektroforesis gel poliakrilamida (PAGE) yang dilanjutkan dengan pewarnaan perak.
Mikrosatelit mempunyai karakteristik sebagai berikut: tingkat polimorfisme yang tinggi, kodominan dan diwariskan mengikuti hukum Mendel (Weising et al 2005). Bila satu primer yang spesifik telah didesain, lokus SSR dapat diamplifikasi dari sedikit sample DNA dengan PCR (Ujino et al. 1998 diacu dalam Zulfahmi, 2007). Mikrosatelit telah diaplikasikan untuk: 1. Identifikasi forensik, 2. Diagnosis dan identifikasi penyakit, 3. Studi populasi genetik dan efek leher botol (bottlenecks effect,) 4. Konservasi biologi, untuk mengamati perubahan dalam populasi, pengaruh fragmentasi dan interaksi populasi yang berbeda serta untuk identifikasi populasi yang baru terbentuk.
Ada beberapa permasalahan dalam penggunaan penanda mikrosatelit. Permasalahan ini dapat dikelompokkan kedalam problem teknis praktis dan problem data. Problem praktik meliputi: 1. Pemilihan primer untuk mikrosatelit, banyak jenis primer yang telah didesain untuk analisis mikrosatelit pada tanaman. Primer-primer itu perlu diskrining dan dioptimasi sebelum diaplikasikan pada jenis tanaman tertentu, karena setiap tanaman mempunyai karakteristik spesifik yang berbeda satu sama lain. 2. Slippage selama proses amplifikasi, termopolimerase dapat slip sehingga menghasilkan produk yang berbeda dalam ukurannya. 3. Ukuran produk amplifikasi berbeda dari ukuran produk sebenarnya. Ketidakakuratan dalam identifikasi alel mungkin juga disebabkan oleh Taq
polimerase yang menambah nukleotida adenosin sampai ujung 3’ produk
amplifikasi.
Homoplasi adalah salah satu problem data. Homoplasi didefinisikan ketika dua alel dalam keadaan sama, tetapi tidak sama secara keturunan. Homoplasi mungkin menyebabkan problem dalam analisis studi genetika populasi, dimana
11
dapat mempengaruhi pengukuran keragaman genetika, aliran gen, jarak genetika, ukuran neighbourhood, metode penetapan dan analisis filogenetika (Estoup et al. 2002).
BAB III
BAHAN DAN METODE
3.1 Tempat dan Waktu
Penelitian ini dilakukan di Ruang Analisis Genetik, Bagian Silvikultur, Departemen Silvikultur, Fakultas Kehutanan, Institut Pertanian Bogor. Penelitian dilakukan dari bulan Juni sampai bulan November 2009.
3.2 Bahan dan Alat 3.2.1 Populasi Penelitian
Bahan tanaman yang digunakan sebagai contoh uji adalah berupa daun S.
laevis yang diambil dari tujuh populasi berasal dari daerah di Kalimantan, yaitu
Bukit Bangkirai, Batu Ampar, Izin Usaha Pemanfaatan Hasil Hutan Kayu (IUPHHK) PT. Sari Bumi Kusuma (SBK), IUPHHK PT. ITCIKU, Berau, IUPHHK PT Suka Jaya Makmur, IUPHHK PT. Sarmiento Parakantja Timber (Sarpatim).
Untuk lebih lengkapnya rincian lokasi populasi pengambilan daun pohon contoh S. laevis disajikan pada Tabel 1.
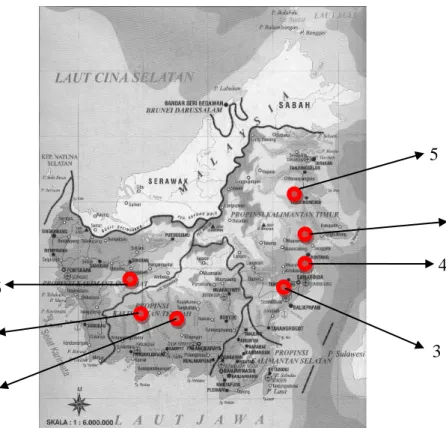
Tabel 1. Koordinat lokasi populasi pengambilan daun pohon contoh Shorea laevis
No Populasi/Lokasi Kabupaten Provinsi
Jumlah pohon contoh Letak geografis Arah Bujur Arah Lintang
1 Batu Ampar Kutai Timur Kalimantan
Timur 20
116048’ BT
00045’ LU 2 PT. Sari Bumi Kusuma Seruyan Kalimantan
Tengah 20 111018’ BT 01059’ LS 3 P.T. ITCIKU Penajam Paser Utara Kalimantan Timur 20 117 06’ BT 01018’ LS 4 Bukit Bangkirai Kutai
Kartanegara Kalimantan Timur 20 117032’ BT 01015’ LS
5 Berau Berau Kalimantan
Timur 20 117
o
24' BT 02o36'LS 6 PT. Suka Jaya Makmur Ketapang Kalimantan
Barat 20 110'49'54, 2" BT 01'31'07, 2" LS 7 PT. Sarmiento Parakantja
Timber (SARPATIM) Seruyan
Kalimantan Tengah 20 112'03'13, 7" BT 01'40'58, 3" LS
13
Keterangan: 1 = Batu Ampar, 2= PT. Sari Bumi Kusuma, 3= PT. ITCIKU, 4=Bukit Bangkirai, 5=Berau, 6= PT. Suka Jaya Makmur dan 7 =PT. Sarmiento Prakantja Timber
(Sarpatim)
Gambar 2 Peta lokasi populasi pengambilan daun pohon contoh Shorea laevis
3.2.2 Alat dan Bahan Analisis Genetik
Alat dan bahan yang digunakan untuk teknik analisis genetik dengan penanda Mikrosatelit terbagi dalam berbagai tahap pekerjaan, yaitu tahapan ekstraksi DNA, uji kualitas DNA, PCR dan visualisasi DNA serta analisis data. Alat dan bahan yang digunakan pada teknik analisis genetik disajikan pada Tabel 2. 2 5 1 4 3 6 7
14
Tabel 2 Alat dan bahan untuk teknik analisis genetik
Analisis
Tahapan Pekerjaan
Ekstraksi Uji kualitas DNA PCR Visualisasi DNA Analisis Data Mikrosatelit Alat : sarung tangan karet, gunting, tube 2 ml, alat tulis, mortar, pestel, sudip, mikropipet, tips, rak tube, vorteks, mesin sentrifugasi, waterbath, freezer,desikator. Alat : sarung tangan, timbangan analitik ,
gelas ukur, tabung erlenmeyer, bak/cetakan agar, microwave, mikropipet, mesin elektroforesis fisher scientific, bak EtBr, kamera/alat foto DNA, mesin UV Transiluminator, laptop. Alat : sarung tangan, tube 0.2 ml, rak mikrotube, spidol permanen, alat tulis, mikropipet, tips, mesin sentrifugasi, mesin PCR PTC - 100. Alat : sarung tangan, piringan kaca kecil, mesin elektroforesis fisher scientific, mikropipet, tips, magnetic stirrer, mesin sentrifugasi, kontainer/bak plastik, shaker, kamera, laptop. Alat : laptop, sofwer Popgene versi 1.31, NTSys versi 2.0. Bahan : buffer ekstrak, PVP 1 %, Chloroform IAA, Isopropanol dingin, NaCl, Etanol 95 %, buffer TE. Bahan : agarose, bufer TAE 1 x, DNA hasil ekstraksi, Blue juice 10x, EtBr. Bahan : DNA, primer spesifik forward dan reverse (Shc dan Sle), Green Go Taq Polymerase, nukleas free water Bahan : akrilamid, bisakrilamid, TEMED, APS, buffer TBE1x, bind slane,etanol, asam asetat, aquades, Silver nitrat, formaldehid, NaOH.
15
3.3 Prosedur Penelitian
Secara umum prosedur penelitian dengan metode mikrosatelit dapat dilihat pada Gambar 3.
Pewarnaan silver nitrat
PCR primer terbaik PCR seleksi primer Elektroforesis agarose 1 %
Ekstraksi DNA
Pewarnaan (staining)
Pembuatan gel poliakrilamid dan Elektroferesis
Gambar 3 Bagan Alur Penelitian Contoh uji daun
Tidak
Pewarnaan silver nitrat Elektroferesis gel poliakrilamid
Foto Tidak
Interpretasi dan Analisis Data
Popgene NTSys Deskriptif
16
3.3.1 Contoh daun
Contoh daun yang digunakan adalah daun Shorea laevis yang diambil dari setiap individu pohon sebanyak 20 individu pohon per lokasi, dengan jumlah lokasi sebanyak tujuh lokasi. Sehingga secara keseluruhan contoh daun yang digunakan pada penelitian ini berasal dari 140 individu pohon.
Adapun penentuan individu pohon untuk pengambilan contoh daun pada suatu lokasi dilakukan secara acak. Contoh daun S. laevis yang diambil dari setiap lokasi dimasukkan ke dalam plastik yang berisi silika gel. Fungsinya agar daun tetap terjaga kualitasnya sehingga daun menjadi tidak rusak. Selanjutnya daun disimpan di dalam freezer dan setelah dikeluarkan dari freezer maka daun harus segera dilakukan ekstraksi. Menurut Karsinah dalam Husnaeni (2008) hal ini dapat menyebabkan terjadinya pelepasan enzim nuklease yang dapat merusak DNA.
3.3.2 Ekstraksi DNA
Kegiatan ekstraksi DNA dari daun S. laevis dilakukan dengan menggunakan metode CTAB (Cetyl Trimethyl Ammonium Bromide) Doyle dan Doyle (1990) yang telah dimodifikasi untuk mendapatkan DNA yang cukup murni. Sampel daun 2 x 2 cm digerus dengan menggunakan nitrogen cair di dalam pestel yang bersih. Hasil gerusan selanjutnya dipindahkan ke dalam tube 1,5 ml, lalu hasil gerusan ditambahkan 500-700 mikroliter larutan buffer ekstrak (Tris-HCl, EDTA, NaCl, CTAB, Air, dll) dan 100 mikroliter PVP 1%. Fungsi buffer ekstrak dan PVP adalah mempercepat proses penghancuran.
Tahapan selanjutnya dilakukan proses inkubasi di dalam waterbath selama 45 menit-1 jam pada suhu 65o-70oC. Apabila proses inkubasi melebihi suhu optimal maka DNA yang ada dalam tube akan rusak. Jika proses inkubasi telah selesai maka tube diangkat dan didinginkan ± 15 menit. Untuk mengikat DNA ditambahkan kloroform 500 mikroliter, selanjutnya campuran tersebut dikocok agar menjadi homogen dan di sentrifuse pada kecepatan 13.000 rpm selama 2 menit. Proses sentrifugasi dilakukan untuk memisahkan bahan-bahan kimia atau fase organik dari fase air berupa supernatan. Yang digunakan untuk tahapan selanjutnya adalah fase air yang berisi benang-benang nukleat. Untuk itu fase air
17
dipisahkan dari fase organik dengan menggunakan mikro pipet lalu fase air dipindahkan ke dalam tube baru. Kegiatan di atas dilakukan sebanyak dua kali.
Selanjutnya ditambahkan isopropanol dingin 500 mikroliter dan NaCl 300 mikroliter, lalu disimpan dalam freezer selama 45 menit sampai 1 jam. Pemberian isopropanol dingin dan garam NaCl akan menyebabkan pengendapan DNA dan terbentuknya benang-benang asam nukleat yang halus berwarna putih.
Hasil pengendapan disentrifugasi pada kecepatan 13.000 rpm selama 2 menit dan cairan dalam tube dibuang. Pembuangan cairan ini harus dilakukan secara hati-hati agar pellet DNA yang mengendap tidak terbuang. Kegiatan selanjutnya adalah proses pencucian DNA dengan menambahkan etanol 95% sebanyak 300 mikroliter, lalu disentrifugasi pada kecepatan 13.000 rpm selama 2 menit dan cairan dalam tube dibuang dibuang kembali dan dilakukan secara hati-hati. Proses tersebut dilakuakan 2 kali.
Pellet DNA yang ada di tube dikeringkan dengan cara disimpan di dalam
desikator secara terbalik agar silikagel di dalam desikator dapat menyerap cairan yang ada dalam tube selama ± 15 menit dan setelah itu ditambahkan larutan buffer TE 20 mikroliter, lalu divortek dan disentrifugasi kembali.
3.3.3 Uji Kualitas dan Kuantitas DNA
Uji kualitas DNA dilakukan dari hasil ekstraksi DNA yang berupa pellet DNA yang telah ditambahkan buffer TE. Uji kualitas DNA ini dilakukan pada gel
agarose dengan konsentrasi sebesar 1 % (b/v), dimana 33 ml buffer TAE 1x
dicampurkan dengan 0,33 gram agarose (untuk cetakan 17-25 sumur). Campuran agar 1% tersebut dimasukkan ke erlenmeyer dan dipanaskan ke dalam microwave untuk selanjutnya dituangkan ke dalam cetakan dan ditunggu sampai menjadi padat, kemudian disimpan di dalam bak elektroforesis yang berisi buffer TAE.
Komposisi yang harus dimasukkan ke dalam lubang sumur adalah 3 mikro liter Blue juice 10x dan 4 mikro liter DNA. Elektroforesis dilakukan dengan menggunakan aliran listrik dengan tegangan 100 volt sekitar 30 menit. Proses elektroforesis yang dilakukan secara horizontal, dilihat dari posisi agar pada bak elektroforesisnya. Pada prinsipnya, proses elektroforesis ini dilakukan dengan memigrasikan DNA dalam gel agarose dari arus (-) ke arus (+). Hasil dari
18
elektroforesis ini dapat dilihat dengan menggunakan pewarnaan pada larutan
Ethidium Bromida (EtBr) dengan konsentrasi 0,01 % (v/v). Terlebih dahulu gel agarose direndam dalam larutan EtBr ini selama 15 menit dan selanjutnya pita
DNA hasil ekstraksi dilihat dengan menggunakan UV transiluminator dan didokumentasikan menggunakan kamera untuk difoto. Hasilnya dapat diinterpretasi dan dianalisis.
3.3.4 Polymerase Chain Reaction (PCR) Mikrosatelit
DNA hasil proses ekstraksi sebelum dilakukan amplifikasi PCR harus dilakukan pengenceran DNA. Pengenceran DNA dilakukan dengan menggunakan
aquabidest. Besarnya perbandingan antara DNA dengan aquabidest tergantung
dari tebal dan tipisnya DNA genomik hasil dari ekstraksi.
Prinsip proses PCR adalah suatu siklus berjangka pendek (30-60 detik) dengan tiga perubahan suhu yang berubah secara cepat. Komposisi bahan untuk reaksi PCR mikrosatelit telah ditentukan yang terdiri atas H2O, Green go taq, primer forward dan reverse, DNA isolasi. Reaksi PCR mikrosatelit tersebut dilakukan dengan menggunakan mesin PTC-100 Progammable Thermal Cycler (MJ Research, Massachussetts, USA). Komposisi bahan untuk reaksi PCR Mikrosatelit dapat dilihat pada Tabel 3.
Tabel 3 Komposisi bahan untuk reaksi PCR Mikrosatelit
No. Nama Bahan 1 sampel reaksi X sample reaksi
1 H2O 2 mikro liter X x 2 mikro liter
2 Green Go Taq 7.5 mikro liter X x 7.5 mikro liter 3 Primer Forward and
Reverse @
2 mikro liter X x @ 2 mikro liter
4 Cetakan DNA/DNA isolasi
2 mikro liter X x 2 mikro liter
Proses mikrosatelit pada daun S. laevis ini dilakukan dengan menggunakan 5 primer spesifik yang telah dipakai untuk jenis Shorea leprosula. Daftar nama-nama primer spesifik pada pada jenis Shorea leprosula dapat dilihat pada Tabel 4.
19
Tabel 4 Primer spesifik pada jenis Shorea leprosula (Ng 2008) No. Lokus Repeat PCR Primer (5’ to 3’) PCR
Product Length
Annealing Temp (0C) 1. Sle01 (AT)12 F:TCGTACTGATAATCGG
R:GTTTATAGGCTGATAATATGATTTA
179-188
45
2. Sle02 (AGC)9 F:GGAGGAGAGAAACGAAG
R:GTTTGAGGTAGGTAATAACGAGC
142-160
45
3. Sle05 (TA)9 F:ACTAATAATGCTTGTGGTAAT R:GTTTGTAACTAACCTCTAATGCCT
175-191
45
4. Sle07 (GAA)7 F:AGAAGAATATGGGTACGAACTG R:GTTTGAATCAACTGGCACCTCTTAT
175-190
45
5. Sle08 (AT)12 F:GGCTTCCTTTATATCCAATTT R:GTTTGAGTTGGCTGCATATGA
177-197
45
Primer spesifik lainnya yang digunakan untuk S. laevis adalah 5 primer telah digunakan pada jenis Shorea curtisii, tediri atas Shc01, Shc02, Shc03, Shc 04 dan Shc07. Adapun keterangan lengkap mengenai primer-primer ini dapat dilihat pada Tabel 5.
Tabel 5 Primer spesifik pada jenis Shorea curtisii (Ujino et al. 1998) No. Lokus Repeat PCR Primer (5’ to 3’) PCR
Product Length Annealing Temp (0C) 1. Shc01 (CT)8CT (CA),CTCA
F:GCTAT TGGCA AGGAT GTTCA R:CTTAT GAGAT CAATT TGACA
G
152 56
2. Shc02 (CT)2CA(CT)5 F:CACGC TTTCC CAATC TG R:TCAAGA GCAGA ATCCA G
149 54
3. Shc03 (CT)8 F:TTGAA GGGAA GGCTA TG R:CTTCT CAACT ACCTT ACC
124 54
20
R:TATTG ACGTG GAATC TG 5. Shc07 (CT)8CA(CT)5
CACCC(CTC A)3CT (CA)16
F:ATGTC CATGT TTGAG TG R:CATGG ACATA AGTGG AG
169 54
Proses PCR melalui 3 tahapan yaitu dennaturation, annealing dan extention. Pada proses PCR ini suhu yang digunakan berbeda-beda bergantung pada teknik, bahan kimia dan primer yang digunakan. Pengaturan suhu pada mesin PTC-100 untuk reaksi mikrosatelit disajikan pada Tabel 6, proses ini dilakukan atau diulang sebanyak 35 siklus.
Tabel 6 Tahapan-tahapan dalam proses PCR Mikrosatelit
Tahapan Suhu Waktu Jumlah Siklus
Pre-denaturation 950C 2 menit 1 Denaturation Annealing Extension 950C 450C-560C 2 menit 1 menit 2 menit 35
Final Extension 720C 5 menit 1
3.3.5 Pembuatan Gel Poliakrilamid
Beberapa persiapan yang dilakukan sebelum pembuatan gel poliakrilamid.
3.3.5.1 Pencucian piringan kaca
Piringan kaca kecil dicuci dengan 5 ml etanol menggunakan tissue. Kemudian kaca dikeringkan selama 2 menit dan diulang kembali. Kaca tersebut kemudian diberi perlakuan dengan menggunakan 15 µl
ɣ-methacryloxy-propyltrimethoxyselane (Bind Silane, M-6514, Sigma) dalam 4 ml etanol dan 1.25
ml asam asetat. Kemudian sisanya dibersihkan dengan tissue yang dilembabkan dengan etanol. Piringan kaca besar dicuci dengan 5 ml etanol menggunakan tissue kemudian dikeringkan selama 2 menit, dan diulangi kembali.
3.3.5.2 Pembuatan larutan akrilamid
Pembuatan larutan akrilamid dilakukan dengan menyiapkan larutan akrilamid 6%. Larutan ini didapatkan dengan menambahkan 5,7 g akrilamid, 0,3
21
g N (N-methylenbisacrylamida) pada 100 ml aquades. Pembuatan Gel 6% poliakrilamid; 8 M urea) dilakukan dengan mencampur 60 ml urea (larutan akrilamid dalam 10x TBE dengan 500 µl ammonium persulfat dan 50 µl TEMED (sigma). Larutan gel diletakkan pada lempengan gel (ketebalan 1.5 mm), selanjutnya gel didiamkan untuk berpolimerisasi selama 30 menit.
3.3.5.3 Loading sample
Hasil amplifikasi kemudian didenaturasi selama 2 menit pada suhu 92oC didalam thermocycler dan simpan di lemari es sebelum diaplikasikan ke gel dalam 5 µl yang mengandung volume yang hampir sama dengan larutan akhir (10 mM NaOH, 95% Formamid, 0.05% Bromopheneol Blue, 0.05% Xylene Cyanol). Kegiatan elektroforesis dilakukan pada 350 V, 40mA, 80 W selama 75 menit setelah pre-run gel selama 1 jam. Buffer untuk running (TBE 1x) mengandung 18mM Triz-HCl, 8.9 mM asam borat dan 2 mM Na2EDTA.
3.3.5.4 Metode Pewarnaan
Metode pewarnaan yang digunakan dalam penelitian ini berdasarkan pada metode penelitian Benbouza et al. (2006) yang dimodifikasi. Metode ini meliputi beberapa langkah atau tahapan. Setelah dilakukan elektroforesis, gel dicuci dalam 1000 ml air larutan (900 ml aquades, 100 ml etanol 95% dan 50 µl Asam asetat) dingin selama 5 menit. Tahap selanjutnya adalah mencuci gel dengan melakukan perendaman selama 6-7 menit pada suhu ruangan (22-24 oC) dalam 1000 ml larutan aquades. Impregnasi dengan perak nitrat (1000 ml aquades, 1.5 g Silver
nitrat, 1.5 ml formaldehid 37%) dilakukan pada ruangan dengan kondisi cahaya
penuh selama 20 – 60 menit.
Tahap selanjutnya adalah gel development yaitu dengan menggunakan larutan campuran dari 1000 ml aquades, 1.5% NaOH, dan 3 ml 37% HCOH sampai pita nampak pada intensitas rendah (3-5 menit). Jika intensitas yang diharapkan telah nampak pada intensitas rendah (3-5 menit), maka selanjutnya gel diimpregnasi dalam larutan akhir 1000 ml aquades, 10% etanol, dan 0.5% asam asetat selama 2 menit. Semua tahapan tersebut dilakukan dalam kontainer plastik. Kemudian lembaran gel selanjutnya digerakkan dalam shaker selama proses pewarnaan. Semua larutan disiapkan dengan menggunakan air ultra pure atau
22
aquades. Gel selanjutnya dikeringkan pada suhu ruangan dan pita DNA akan secara langsung terlihat dengan bantuan white light box, dan kemudian difoto.
3.8 Analisis Data
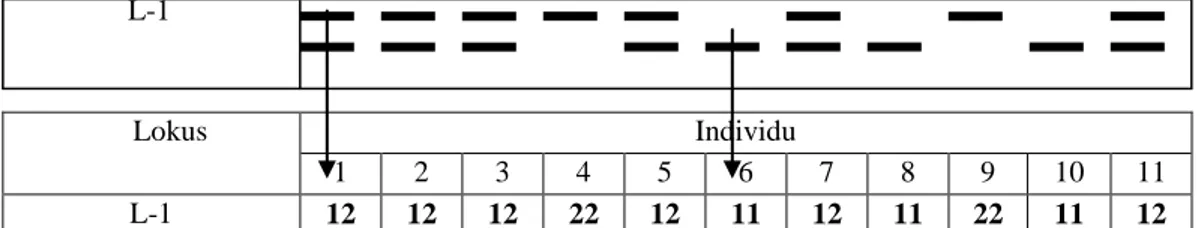
Hasil dari kegiatan teknik mikrosatelit pada daun selanjutnya difoto dan dianalisis dengan melakukan skoring pola pita yang muncul. Hasil interpretasi foto kemudian dianalisis dengan menggunakan software POPGENE 32 Versi 1.31 dan NTSYS Ver 2.0 (Rohlf 1998).
Lokus Individu 1 2 3 4 5 6 7 8 9 10 11 L-1 Lokus Individu 1 2 3 4 5 6 7 8 9 10 11 L-1 12 12 12 22 12 11 12 11 22 11 12
Gambar 4 Cara skoring DNA mikrosatelit
Parameter variasi genetik yang dicari dalam penelitian ini adalah sebagai berikut (Finkeldey 2005 dalam Yunanto 2006):
1. Persentase Lokus Polimorfik (PLP) =
(
)
( ) ) ( LM LP LP x 100% Keterangan :Σ(LP) : jumlah lokus polimorfik Σ(LM) : jumlah lokus monomorfik
2. Jumlah alel yang diamati (ne) =
Lokus lel A3. Jumlah alel yang efektif (na) =
i
pi2
1
Keterangan : pi = frekuensi genetik tipe ke i 4. Heterozigitas harapan (He) = 1-
i
pi2
BAB IV
HASIL DAN PEMBAHASAN
4.1 Ekstraksi DNA
Tahapan dan hasil dari kegiatan ekstraksi DNA menentukan langkah selanjutnya untuk tahapan PCR. Pada tahapan ekstraksi dilakukan uji kualitas DNA untuk dapat menentukan perbandingan pengenceran DNA untuk dapat dipakai sebagai salah satu bahan pada tahap selanjutnya, yaitu tahapan PCR.
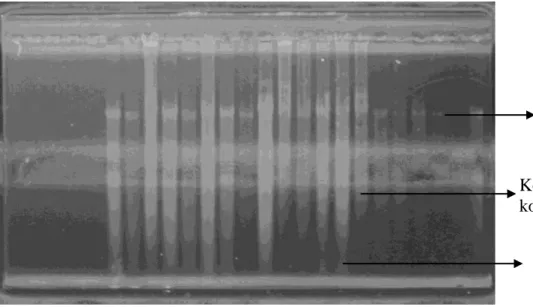
Kegiatan ekstraksi pada daun Shorea laevis yang dilakukan pada penelitian ini secara umum rata-rata memperlihatkan pola pita yang tebal (Gambar 5) yang mengindikasikan masih terkontaminasi (smear) oleh bahan-bahan kimia sisa proses ektraksi dan juga terkontaminasi oleh protein, polisakarida, dan RNA (Qiagen 2001).
Gambar 5 Pola pita DNA hasil ekstraksi dan isolasi dari daun Shorea laevis Untuk dapat melakukan analisis PCR diperlukan DNA dengan tingkat kemurnian dan berat molekul yang tinggi, akan tetapi ekstraksi DNA dari jaringan tanaman dengan tingkat kemurnian yang tinggi seringkali sulit diperoleh. Metode pemurnian yang tepat seringkali dibutuhkan terutama menyangkut kualitas DNA yang tinggi. Salah satu cara yang dapat digunakan untuk mendapatkan DNA hasil ekstraksi yang cukup murni adalah dengan melakukan proses pengenceran.
RNA DNA
Kotoran/ kontaminan
25
80x
40x 20x
10x Secara visual, pita DNA yang tebal (kotor) memerlukan perbandingan pengenceran yang lebih besar yaitu 100x (99 µL aquabidest: 1 µL DNA). Pengenceran selanjutnya mengikuti tingkatan ketebalan pita DNA. Pita DNA yang paling tipis menggunakan perbandingan pengenceran 10x (9 µL aquabidest: 1 µL DNA), karena kualitas DNA-nya termasuk bagus (tidak terlalu kotor) (Gambar 5).
Gambar 6 Contoh besarnya pengenceran hasil ekstraksi DNA
Oleh karena itu, setiap sampel DNA mempunyai perlakuan pengenceran yang berbeda-beda, tergantung dari kualitas DNA-nya. Pengenceran ini dimaksudkan agar pada tahap PCR primer dapat menempel pada pita DNA sehingga dapat teramplifikasi.
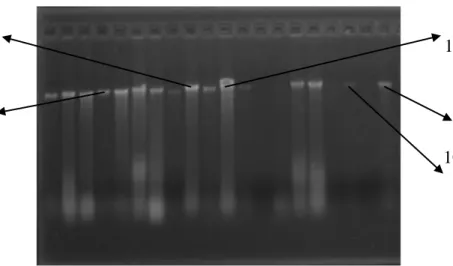
4.2 Seleksi Primer (Cross Spesies Amplification)
Seleksi primer dimaksudkan untuk mencari primer yang dapat menghasilkan amplifikasi. Keberhasilan amplifikasi dengan PCR didasarkan pada kesesuaian primer yang digunakan. Seleksi primer yang digunakan untuk kegiatan teknik mikrosatelit pada penelitian ini menggunakan primer dari hasil seleksi 10 primer spesifik untuk nuklear (DNA inti). Untuk analisis DNA inti pada daun dari 10 primer spesifik yang diseleksi/diuji hanya ada empat primer yang memperlihatkan pola pita polimorfik, yaitu Sle01, Sle07, Shc04 dan Shc 07, sedangkan pada primer spesifik lainnya yaitu primer Sle02, Sle05, Sle08, Shc01, Shc02 dan Shc 03 hanya memperlihatkan pola pita monomorfik. Proses cross
26
species amplification untuk kedua jenis primer Shc dan Sle tersebut juga sudah
pernah berhasil dilakukan pada jenis Shorea robusta (Panday dan Geburek 2009). Berdasarkan hasil seleksi primer tersebut, primer yang digunakan adalah primer yang teramplifikasi yang menghasilkan fragmen DNA polimorfik. Pada penelitian ini pemilihan primer yang diguanakan adalah sebanyak tiga primer yaitu Sle01, Sle07 dan Shc04. Hal ini berdasarkan pada keterwakilan jumlah primer polimorfik yang telah diuji / diseleksi. (Gambar 7).
(a) (b) 1 2 3 7 6 5 4 50 bp 50 bp 100 bp 150 bp 200 bp 250 bp 100 bp 150 bp 200 bp 250 bp
27
(c)
Keterangan: (a) 1 = Primer Sle08, 2 = Primer Sle02, 3= Primer Sle05; (b) 4= Primer Shc03, 5=Primer Shc02, 6=Primer Shc01 dan 7=Shc07; (c) 8=Primer Sle07, 9=Primer Shc04 dan 10=Primer Sle01.
Gambar 7 Foto hasil seleksi primer
Pada Gambar 7(a) menunjukkan bahwa adanya lokus dan jumlah alel yang sama (monomorfis), hal ini berhubungan dengan tipe pengulangan dari primer yang digunakan dan jumlah sampel yang digunakan. Sedangkan pada Gambar 7(c) menunjukkan adanya kecocokan dalam amplifikasi primer terhadap DNA dimana ditemukan posisi lokus pada alel yang berbeda, sehingga dapat dikatakan bahwa lokus tersebut polimorfik.
Hasil elektroforesis pada gel poliakrilamid mampu memisahkan DNA lebih sempurna. Penentuan ukuran dan jumlah alel yang muncul pada gel didasarkan pada asumsi bahwa semua pita DNA yang memiliki laju migrasi yang sama disebut homolog (Leung et al dalam Munarti 2005).
Berdasarkan amplifikasi tiga primer yang digunakan yaitu Sle01, Sle07 dan Shc04 dapat dilihat jumlah alel yang ditemukan adalah minimum 1 alel dan maksimum 2 alel dan menghasilkan lokus polimorfik. Pada Gambar 8 dapat dilihat bahwa DNA S.laevis menggunakan primer Shc04 memiliki fragmen DNA
8 10 9 50 bp 100 bp 150 bp 200 bp 250 bp
28
atau pita dengan berukuran antara 60-80 bp. Sedangkan untuk primer Sle01 dan Sle07 masing-masing memiliki fragmen DNA pada ukuran 150-200 bp dan 190 - 210 bp. Hal ini dapat dilihat perbandingan hasil produk PCR awal yang digunakan pada jenis Shorea leprosula dan Shorea curtisii dengan hasil produk PCR pada
jenis S.laevis. Adapun rinciannya disajikan pada Tabel 7.
Tabel 7 Perbandingan ukuran pasangan basa (base pairs)
Primer Hasil PCR awal (bp) Hasil PCR pada S. laevis (bp)
Keterangan
Sle01 179-188 150-200 Polimorfik
Sle07 175-190 190-210 Polimorfik
Shc04 96 60-80 Polimorfik
Pada Tabel 7 dapat dilihat bahwa primer Shc04 memiliki lokus awal (hasil PCR awal) 96 bp dan lokus pada S.laevis 60-80 bp, hal ini diduga adanya mutasi yang mengubah informasi genetik di dalam urutan DNA pada gen. Menurut Welsh (1990), mutasi dapat terjadi secara spontan dalam frekuensi tertentu yang tergantung pada keadaan lokus itu sendiri dan informasi genetik disekitar kromosom. Mutasi pada beberapa lokus dapat terjadi dengan mudah, sementara pada lokus - lokus lain kromosomnya sangat stabil. Tingkat kestabilan kromosom terhadap mutasi tergantung pada keadaan alel yang mengendalikan lokus.
4.3 Interpretasi dan Analisis Data
4.3.1 Keragaman Genetik dalam Populasi
Peubah yang digunakan untuk mencirikan keragaman genetik dalam populasi yaitu Presentase Lokus Polimorfik (PLP), jumlah alel yang diamati (na), jumlah alel efektif (ne) dan variasi genetik (He) (Finkeldey, 2005). Nilai peubah numerik hasil analisis berdasarkan mikrosatelit pada daun S. laevis disajikan pada Tabel 8, sedangkan hasil skoring pita DNA disajikan pada Lampiran 2.
29
Tabel 8 Peubah yang mencirikan keragaman genetik dalam populasi Shorea
laevis
No. Populasi N PLP(%) na ne He
1 Batu Ampar 20 100 2,3333 1,8384 0,4467
2 Berau 20 100 2,6667 1,7881 0,3958
3 Sarpatim 20 100 1,7802 0,6332 0,4200
4 Suka Jaya Makmur 20 100 2,6667 1,8229 0,4325
5 Bukit Bangkirai 20 100 2,3333 1,8826 0,4646
6 Sari Bumi Kusuma 20 100 2,0000 1,8235 0,4400
7 ITCIKU 20 100 2,3333 2,0444 0,5104
Rata-rata 20 100 2,3019 1,6904 0,4443
Keterangan :
N = Jumlah total individu; PLP = Persentase Lokus Polimorfik; na = Jumah alel yang diamati; ne = Jumah alel efektif (Kimura and Crow (1964); He = Diferensiasi genetik Nei
(1973)/Heterozigositas harapan
Berdasarkan Tabel 8 dapat dilihat bahwa nilai rata-rata PLP = 100 %, na = 2,3019, ne = 1,6904 dan He = 0,4443. Secara umum nilai variasi genetik ini lebih kecil bila dibandingkan dengan jenis Shorea lainnya yang juga dianalisis dengan menggunakan penanda mikrosatelit, dimana pada jenis Shorea cordifolia nilai He = 0,723 (Stacy et al 2001), Shorea curtisii nilai He = 0,639 (Ujino et al. 1998), S.
leprosula
(Lee et al. 2004) dan (Isoda et al. 2005) masing-masing nilai He = 0,622 dan 0,686 serta pada Jati (Tectona grandis) He= 0,601 (Tabel 9). Kemungkinan yang menyebabkan rendahnya nilai keragaman yang didapat pada penelitian ini adalah jumlah penanda/lokus yang digunakan sedikit.
Tabel 9 Keragaman genetik beberapa jenis Meranti dan Jati
Populasi S. laevis di PT. ITCIKU memiliki nilai keragaman genetik tertinggi dengan nilai He = 0,5104 (garis melingkar merah). Sedangkan populasi yang memiliki nilai rata-rata He paling kecil adalah S. laevis Berau, yaitu He = 0,3958 (garis melingkar hijau). Adanya nilai He tertinggi sebesar 0,5104 di lokasi PT ITCIKU, menjadikan bahwa keragaman genetik dari lokasi tersebut dapat mewakili keragaman secara keseluruhan dari enam lokasi/populasi lainnya.
No Jenis Metode He Sumber
1 S. cordifolia Mikrosatelit 0,723 Stacy et al. 2001
2 S. curtisii Mikrosatelit 0,639 Ujino et al. 1998
3 S. leprosula Mikrosatelit 0,622 Lee et al. 2004
4 S. leprosula Mikrosatelit 0,686 Isoda et al. 2005
30
Sedangkan pada lokasi Berau dengan nilai keragaman genetik rendah ini diindikasikan/ diduga lokasi ini memiliki kerapatan yang rendah. Nilai-nilai variasi genetik tersebut dapat menjadi bahan pertimbangan dalam merancang program pemuliaan dalam mempertahankan atau meningkatkan variasi sumberdaya genetik jenis S. laevis.
Hutan tropis adalah hutan yang memiliki keragaman yang tinggi, akan tetapi jumlah setiap jenisnya rendah (Finkeldey 2005). Jenis yang dijumpai dalam kerapatan yang rendah di hutan tropis kurang bervariasi dibandingkan dengan jenis-jenis yang dijumpai dalam populasi dengan kerapatan yang tinggi. Keragaman genetik disebabkan oleh perubahan pada struktur genetik dari suatu populasi. Perubahan struktur genetik suatu populasi disebabkan oleh mutasi, aliran gen dan migrasi, penghanyutan genetik, seleksi, dan juga sistem perkawinan.
Keragaman genetik merupakan landasan bagi pemulia untuk memulai suatu kegiatan perbaikan tanaman. Besarnya keragaman genetik dapat menjadi dasar untuk menduga keberhasilan perbaikan genetik didalam program pemuliaan. Keragaman genetik yang luas merupakan syarat berlangsungnya proses seleksi yang efektif karena memberikan keleluasaan dalam proses pemilihan suatu genotipe. Selain itu populasi dengan keragaman genetik yang lebih luas akan memberikan peluang yang lebih besar diperolehnya karakter-karakter yang diinginkan.
Adanya tingkat keragaman atau variasi yang tinggi pada populasi PT. ITCIKU dibandingkan dengan populsi lainnya menjadikan populasi ini dapat dijadikan bahan pertimbangan untuk kegiatan pemuliaan jenis S. laevis. Adanya keragaman genetik yang tinggi pada suatu populasi menjadikan syarat untuk dilakukannya seleksi tanaman. Seleksi tanaman ini merupakan proses dimana pemilihan individu-individu yang memiliki sifat tertentu dan lebih disukai untuk dikembangkan. Menurut Djamhuri (2009) Tujuan dari setiap program pemuliaan pohon adalah untuk memperoleh individu-individu pohon yang unggul untuk sifat-sifat tertentu. Individu – individu tersebut dipilih dan dikembangkan dari hasil seleksi terhadap suatu populasi tertentu.
31
4.3.2 Keragaman Genetik antar Populasi
Peubah yang digunakan untuk mencirikan variasi genetik antar populasi menurut Finkeldey (2005) yaitu jarak genetik, dan analisis klaster/kelompok. Salah satu peubah yang digunakan untuk mencirikan variasi genetik antar populasi adalah jarak genetik. Jarak genetik mengukur perbedaan struktur genetik antar dua populasi pada suatu lokus gen tertentu. Perbedaan genetik dari dua atau lebih populasi pada umumnya dianalisis dengan sebuah matrik dimana elemen-elemennya berupa jarak genetik dan pasangan kombinasi dari masing-masing populasi (Finkeldey, 2005). Adapun jarak genetik antar populasi S. laevis pada daun dapat dilihat pada Tabel 10.
Tabel 10 Jarak genetik Nei (1978) dan jarak fisik antar populasi Shorea laevis Populasi 1 2 3 4 5 6 7 1 0.0000 690.3 208.7 113.1 183.3 692.3 682.5 2 0.3726 0.0000 569.4 659.4 774.2 157.0 98.5 3 0.3733 0.1232 0.0000 114.1 382.2 624.0 475.8 4 0.4151 0.1151 0.0872 0.0000 292.5 686.4 561.6 5 0.1362 0.1524 0.1615 0.0745 0.0000 737.1 686.4 6 0.5229 0.1852 0.1822 0.0508 0.1599 0.0000 198.9 7 0.1181 0.1486 0.1372 0.0760 0.0072 0.1331 0.0000
Keterangan: 1= Batu Ampar, 2= Berau, 3= Sarpatim, 4=Suka Jaya Makmur, 5= Bukit Bangkirai, 6= Sari Bumi Kusuma, dan 7= ITCIKU, Sebelah kiri 0.0000 = ID/jarak genetik, sebelah kanan 0.0000 = jarak datar fisik (km)
Indeks ketidaksamaan (ID) / jarak genetik: maksimum dan minimum
Nilai dari Indeks ketidaksamaan menunjukkan nilai jarak genetik. Hasil analisis jarak genetik pada Tabel 10 menunjukkan bahwa populasi yang memiliki jarak genetik yang paling besar adalah antara populasi Batu Ampar dengan populasi Sari Bumi Kusuma dengan nilai jarak genetik yaitu 0,5229. Jarak genetik yang besar ini mengindikasikan bahwa hubungan kekerabatan kedua populasi ini cukup jauh. Sedangkan populasi dengan jarak genetik terdekat adalah antara populasi Bukit Bangkirai dengan populasi ITCIKU yaitu 0,0072.
Berdasarkan hasil analisis regresi antara jarak genetik dengan jarak fisik diperoleh persamaan yaitu jarak genetik = 0.200 - 0.00134 jarak fisik (km), dimana nilai koefisien determinasi (R-sq) = 0,5% dan jarak fisik tidak berkorelasi nyata terhadap jarak genetik (P = 0.754). Hasil analisis regresi disajikan pada Gambar 8.
0.0072 0.5229
32 jarak fisik Ja ra k g e n e ti k 800 700 600 500 400 300 200 100 0 0.5 0.4 0.3 0.2 0.1 0.0
Gambar 8 Hubungan antara jarak genetik dengan jarak fisik
Selain dari jarak serta nilai pembagian ragam genetik, peubah lain yang dapat digunakan untuk mencirikan ragam genetik antar populasi adalah analisis gerombol/kelompok atau dendogram jarak genetik antar populasi. Berdasarkan analisis nilai jarak genetik yang telah dihitung berdasarkan software Popgene versi 3.2 dan diolah menggunakan metode pemasangan kelompok aritmatika tidak berbobot (Unweighted Pair-Grouping Method with Aritmatic Averaging, UPGMA) dengan software Numerical Taxonomy and Mulivariate Analysis
System (NTSys) Versi 2.01., dihasilkan dendrogram jarak genetik antar populasi
seperti terlihat pada Gambar 9.