8
Perbedaan kodon bias tersebut didasarkan pada perhitungan jumlah efektif kodon (Nc) dengan rentang indeks 60-20. Jika nilainya 60 maka semua kodon (di luar stop kodon) digunakan dengan frekuensi yang sama, sedangkan jika nilainya 20 yaitu hanya satu kodon digunakan untuk setiap asam amino. Dimana nilai efektif nukleotida terbesar adalah 60 dan terkecil adalah 20. Komposisi kodon (Lampiran 6) diterjemahkan berdasarkan Vertebrate mitochondrial translation code yang terdapat dalam MEGA versi 3.0 (Kumar et al. 2004).
SIMPULAN DAN SARAN
Telah terjadi perubahan situs nukleotida pada Goura sp. sebesar 0.26%-7.07% dari 382 nukleotida namun yang berubah hanya 10 situs asam amino saja. Dari perubahan nukleotida yang ada didapat G. cristata lebih dekat kekerabatannya dengan G. scheepmakeri daripada G. victoria.
Untuk penelitian selanjutnya perlu dilakukan perunutan produk PCR dengan situs yang berbeda untuk mendapatkan data yang lebih akurat, informatif, variatif dari daerah geografis yang diperkirakan sebagai daerah pertemuan Goura sp.
DAFTAR PUSTAKA
Benstead P, Dutson G. 2005. Western Crowned-pigeon - BirdLife Species Factsheet.http://www.birdlife.org/datazone/ search/species_search.html?action=SpcHTM Details.asp&sid=2754&m=0.[29/11/2005]. Benstead P, Dutson G. 2005. Victoria Crowned- pigeon - BirdLife Species Factsheet.http://www.birdlife.org/datazone/ search/species_search.html?action=SpcHT MDetails.asp&sid=2755&m=0.[29/11/2005] Benstead P, Dutson G. 2005. Southern Crowned- pigeon - BirdLife Species Factsheet.http://www.birdlife.org/datazone/ search/species_search.html?action=SpcHT MDetails.asp&sid=2756&m=0.[29/11/2005] Bretagnolle V, Attie C, Pasquet E. 1998. Cytochrome b-evidence for
validity and phylogenetic relationship of pseudobulweria and Bulweria. The Auk 115(1); 188-195
Clayton DH, Bush SE, Goates BM, Johnson KP. 2003. Host defense reinforces host- parasite cospeciation. Proc. Natl. Acad. Sci. USA. 100 (26):15694-15699.
Desjardins, P and R. Morais. 1990. Sequence and gene organization of the chicken
mitochondrial genome. A novel gene order in higher vertebrates. J.Mol. Biol. 212(4): 599-634.
Duryadi D, 2002. Penentuan Sex burung Monomorf dengan Penanda Molekuler. Laporan Project Que Grant Tahun Ke-III. Jurusan Biologi.
Bogor. Institut Pertanian Bogor. Johnson KP, Clayton DH. 2000. Nuclear and
Mitochondrial Genes Contain Similar Phylogenetic Signal for Pigeons and Doves (Aves: Columbiformes). Mol. Phylogenet. Evol 14: 141-151.
Kilmaskossu A. 2001. Ekologi Persarangan, Musim Perkembangan dan Kajian Awall Keragamanan Morfogenetik Mambruk Polos (G.cristata). [tesis]. Bogor: Program Pascasarjana, Institut Pertanian Bogor. Kocher TD et al. 1989. Dynamics of
mitochondrial DNA evolution in animals: Amplifications and sequencing with conserved primers. J. Proc. NAtl. Acad. Sci. USA. 86:6196-6200
Kumar S, Tamura K, and Nei M. 2004. MEGA 3.0: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment. Bioinformatics 5:150-163.
Noerdjito M, Maryamto I. 2001. Jenis-jenis Hayati yang Dilindungi Perundang-undangan Indonesia. Cibinong. Balitbang Zoologi Puslitbang Biologi-Lipi dan The Nature Conservacy.
Perrins CM, Middleton ALA. 1985. The Encyclopedia of Birds. New York. Fact on File Inc.
Randi E. 1996. A Mitochondrial cytochrome b phylogeny of the Alectrois partridges [abstrak]. Di dalam. J.Mol. Phyl. Evol. 6 Shapiro B et al. 2002. Flight of the dodo.
Science 295 (5560): 1683.
Sibley CG, Ahlquist JE. 1991. Phylogeny and Classification of Bird: a study in molekular evolution. New York: Yale University.
Stanley SE and Harrison RG. 1999. Cytochrome b evolution in birds and mamals: An Evaluation of the avian constraint Hypotesis. Mol. Biol. Evol. 16(11): 1575-1585.
Sorenson MD. 2003. Avian mtDNA Primers. Boston University.
Thompson JD, Higgins DG, Gibson TJ. 1994. CLUSTAL W: Improving the sensitivity Of progressive multiple sequence aligment through sequence weighting. Position-specific gap penalties and weight matrix choice. Nucleid Acid Res 22:4673-4680.
Lampiran 1 Peta Penyebaran Goura sp. (http://www.birdlife.org)
1a. Daerah penyebaran Goura cristata (Western crowned-pigeon).
1c. Daerah penyebaran Goura victoria (Victoria crowned-pigeon)
Lampiran 2 Hasil perunutan nukleotida Sit b. Goura sp. hasil penelitian, G.victoria (Genbank ), G.cristata (Genbank) dan Gallus gallus sepanjang 382 nt
Gal_gallus
TGAAACACAG GAGTAATCCT CCTCCTCACA CTCATAGCCA CCGCCTTTGT GGGCTATGTT CTCCCATGGG GCCAAATATC ATTCTGAGGG GCCACCGTTA TCACAAACCT ATTCTCAGCA ATTCCCTACA TTGGACACAC CCTAGTAGAG TGAGCCTGAG GGGGATTTTC AGTCGACAAC CCAACCCTTA CCCGATTCTT CGCTTTACAC TTCCTCCTCC CCTTTGCAAT CGCAGGTATT ACTATCATCC ACCTCACCTT CCTACACGAA TCAGGCTCAA ACAACCCCCT AGGCATCTCA TCCGACTCTG ACAAAATTCC ATTTCACCCA TACTACTCCT TCAAAGACAT TCTGGGCTTA ACTCTCATAC TCACCCCATT CC
G.crist_GenB
TGAAACACAG GAGTCATCCT CCTACTAACC CTAATAGCAA CCGCCTTCGT AGGGTATGTC CTACCCTGAG GACAGATATC ATTTTGAGGT GCCACAGTTA TTACCAACCT ATTCTCCGCC ATCCCCTACA TCGGCCAAAC CATTGTCGAA TGAGCCTGAG GTGGCTTCTC AGTAGATAAT CCCACACTAA CACGATTCTT CACCCTCCAC TTCCTCCTCC CCTTCATAAT TGCTGGCCTC ACCATCATCC ACCTCACATT CTTACATGAA TCCGGCTCAA ACAATCCACT AGGCATCTCT TCCAACTGCG ACAAAATCCC ATTCCACCCC TACTTTTCCC TAAAAGACAT TCTTGGCTTC ATACTAATAC TCCTCCCCCT AA
G.crist_m3
TGAAACACAG GAGTCATCCT CCTACTAACC CTAATAGCAA CCGCCTTCGT AGGGTATGTC CTACCCTGAG GACAGATATC ATTTTGAGGT GCCACAGTTA TTACCAACCT ATTCTCCGCC ATCCCCTACA TCGGCCAAAC CATTGTCGAA TGAGCCTGAG GTGGCTTCTC AGTAGATAAT CCCACACTAA CACGATTCTT CACCCTCCAC TTCCTCCTCC CCTTCATAAT TGCTGGCCTC ACCATCATCC ACCTCACATT CTTACATGAA TCCGGCTCAA ACAATCCACT AGGCATCTCT TCCAACTGCG AAAAAATCCC ATTCCACCCC TACTTTTCCC TAAAAGACAT TCTTGGCTTC ATACTAATAC TCCTCCCCCT AA
G.vict_a1
TGAAATACAG GAGTCATCCT CCTACTAACC CTAATAGCAA CCGCCTTCGT AGGGTACGTC CTACCCTGAG GGCAGATATC ATTTTGAGGA GCCACAGTTA TTACCAACCT ATTCCCAGCT ATCCCCTACA TCGGCCAAAC CATTGTCGAA TGAGCCTGAG GCGGCTTCTC AGTAGACAAC CCCACACTAA CACGATTCTC CACCTTCCAT TTCCTCCTCC CCTTCATAAT CGCTGGCCTC ACCATCATCC ACCTCACATT CCTACACGAA TCCGGCTCAA ACAACCCATT AGGCATCTCC TCCAACTGGG ATAAAACCCC ATTCCACCCC TACTTTTCCT CAAAAGACAT CCTTGGCTTC ACACTGATAC TCCTCCCCCT AA
G.vict_GenB
TGAAACACAG GAGTCATCCT CCTACTAACC CTAATAGCAA CCGCCTTCGT AGGGTATGTC CTACCCTGAG GACAGATATC ATTCTGAGGC GCCACAGTTA TTACCAACCT ATTCTCCGCT ATCCCCTACA TCGGCCAAAC CATCGTCGAA TGGGCCTGAG GTGGCTTCTC AGTAGACAAC CCCACACTAA CACGATTCTT CACCCTCCAC TTCCTCCTCC CCTTCCTAAT TGCCGGCCTC GCCATCATCC ACCTCACATT CCTACACGAA TCCGGCTCAA ACAACCCATT AGGTATCTCT TCCAACTGCG ATAAAATCCC ATTCCACCCC TACTTCTCCC TAAAAGACAT CTTCGGCCTC ACACTAATAC TCCTTCCCCT AA
Lampiran 2 Lanjutan G.scheep_m7
TGAAACACAG GAGTCATCCT CCTACTAACC CTAATAGCAA CCGCCTTCGT AGGGTACGTC CTACCCTGAG GACAGATATC CTTTTGAGGA GCCACGGTTA TTACCAACCT ATTCTCCGCT ATCCCCTACA TCGGCCAAAC CATTGTCGAA TGAGCTTGAG GTGGCTTCTC AGTAGATAAT CCCACACTAA CACGATTCTT CACCCTCCAC TTCCTCCTCC CCTTCATAAT TGCCGGCCTC ACCATCATCC ACCTCACATT CTTACACGAA TCTGGCTCAA ACAACCCACT AGGCATCTCT TCCAACTGCG ATAAAATCCC ATTCCACCCC TACTTTTCCC TAAAAGACAT CTTTGGCTTC ATACTGATAC TCCTCCCCTT AA
Lampiran 1 Peta Penyebaran Goura sp. (
www.birdlife.org
)
G.victoria
G. cristata
1b Daerah penyebaran Goura cristata (Western crowned-pigeon). 1a Daerah penyebaran Goura victoria (Victoria crowned-pigeon)
Lampiran 1 Lanjutan
G. scheepmakeri
G. scheepmakeri
Lampiran 3 Matriks perbedaan nukleotida (diatas diagonal) dan jarak genetik (dibawah diagonal) dari Goura sp. hasil penelitian, G.victoria GenBank dan G.cristata(GenBank, spesies famili Columbidae dan Gallus gallus sebagai pembanding
1 2 3 4 5 6 7 8 9 10 11 12 1]Gal.gallus_net 2]G.crist_GenB 3]G.crist_m3 4]G.scheep_m7 5]G.vict_a1 6]G.vict_GenB 7]Did.strig 8]Alec.mdgac_Bnet 9]Pti.supbs_Bnet 10]Col_livia 11]Lep.mgal_Bnet 12]Lep.verful_Bnet 81 82 84 79 82 78 73 75 77 71 71 0.2120 1 15 25 23 42 40 37 44 36 36 0.2147 0.0026 15 25 23 42 41 38 44 37 37 0.2199 0.0393 0.0393 24 23 42 40 38 45 41 40 0.2068 0.0654 0.0654 0.0628 27 45 45 41 45 38 41 0.2147 0.0602 0.0602 0.0602 0.0707 45 42 44 43 42 40 0.2042 0.1099 0.1099 0.1099 0.1178 0.1178 40 44 44 47 47 0.1911 0.1047 0.1073 0.1047 0.1178 0.1099 0.1047 36 39 45 43 0.1963 0.0969 0.0995 0.0995 0.1073 0.1152 0.1152 0.0942 50 44 42 0.2016 0.1152 0.1152 0.1178 0.1178 0.1126 0.1152 0.1021 0.1309 38 42 0.1859 0.0942 0.0969 0.1073 0.0995 0.1099 0.1230 0.1178 0.1152 0.0995 23 0.1859 0.0942 0.0969 0.1047 0.1073 0.1047 0.1230 0.1126 0.1099 0.1099 0.0602
Gal.gallus_net TGA AAC ACA GGA GTA ATC CTC CTC CTC ACA CTC ATA GCC ACC GCC TTT GTG GGC TAT GTT CTC CCA TGG [ 69] G.crist_GenB ... ... ... ... ..C ... ... ..A ..A ..C ..A ... ..A ... ... ..C ..A ..G ... ..C ..A ..C ..A [ 69] G.crist_m3 ... ... ... ... ..C ... ... ..A ..A ..C ..A ... ..A ... ... ..C ..A ..G ... ..C ..A ..C ..A [ 69] G.scheep_m7 ... ... ... ... ..C ... ... ..A ..A ..C ..A ... ..A ... ... ..C ..A ..G ..C ..C ..A ..C ..A [ 69] G.vict_a1 ... ..T ... ... ..C ... ... ..A ..A ..C ..A ... ..A ... ... ..C ..A ..G ..C ..C ..A ..C ..A [ 69] G.vict_GenB ... ... ... ... ..C ... ... ..A ..A ..C ..A ... ..A ... ... ..C ..A ..G ... ..C ..A ..C ..A [ 69] Did.strig ... ... ... ... ..C ... ..A ..A ..A ..C ..A ... ..A ..T ... ..C ..A ..A ... ..C ..A ..C ..A [ 69] Alec.mdgac_Bnet ... ... ..G ... ..C ... ... ... ..A ..C ..A ... ..A ..T ... ..C ..C ..A ..C ..C ..A ..C ..A [ 69] Pti.supbs_Bnet ... ... ... ... A.C ..T ... ... ..G ..C ..A ... ..A ... ... ..C ..C ..A ..C ..C ..A ..C ..A [ 69] Col_livia ... ... ... ... ..C G.. ... ..A ..A ..C ..T ... ... ..T ..A ..C ..A ..A ... ..C ..A ..C ..A [ 69] Lep.mgal_Bnet ... ... ... ... ..C ... ... ..A ..A ..C ..A ... ..A ... ... ..C ..A ..A ..C ..C ..G ... ..A [ 69] Lep.verful_Bnet ... ... ... ... ..C ... ... ..A ..A ..C ..A ... ..A ... ... ..C ... ..G ..C ..C ..A ..C ..A [ 69] Gal.gallus_net GGC CAA ATA TCA TTC TGA GGG GCC ACC GTT ATC ACA AAC CTA TTC TCA GCA ATT CCC TAC ATT GGA CAC [138] G.crist_GenB ..A ..G ... ... ..T ... ..T ... ..A ... ..T ..C ... ... ... ..C ..C ..C ... ... ..C ..C ..A [138] G.crist_m3 ..A ..G ... ... ..T ... ..T ... ..A ... ..T ..C ... ... ... ..C ..C ..C ... ... ..C ..C ..A [138] G.scheep_m7 ..A ..G ... ..C ..T ... ..A ... ..G ... ..T ..C ... ... ... ..C ..T ..C ... ... ..C ..C ..A [138] G.vict_a1 ..G ..G ... ... ..T ... ..A ... ..A ... ..T ..C ... ... ... C.. ..T ..C ... ... ..C ..C ..A [138] G.vict_GenB ..A ..G ... ... ... ... ..C ... ..A ... ..T ..C ... ... ... ..C ..T ..C ... ... ..C ..C ..A [138] Did.strig ..G ... ... ... ... ... ..A ... ..T ..C ..T ..C ... ..C ... ..C ..C ..C ... ... ..C ..C ..G [138] Alec.mdgac_Bnet ..A ... ... ... ... ... ... ... ..A ..A ..T ..G ..T ... ... ..T ..T ..C ... ... ... ..T ..A [138] Pti.supbs_Bnet ..G ... ... ... ... ... ..A ... ..A ..A ... ..T ... ... ... ..C ..C G.A ..T ... ..C ..C ..A [138] Col_livia ..A ... ... ... ... ... ..A ..T ..A ..C ..T ..C ..T ... ... ... ..T G.C ... ... ... ..C ..A [138] Lep.mgal_Bnet ..A ... ... ... ... ... ..T ... ..A ..C ... ..C ... ..C ... ... ..C ..C ..T ..T ... ..C ..A [138] Lep.verful_Bnet ..A ... ... ... ... ... ..C ... ..A ..C ... ..C ... ..C ..T ..C ..T ... ..T ... ..C ... ..A [138]
Lanjutan Lampiran 4
Gal.gallus_net ACC CTA GTA GAG TGA GCC TGA GGG GGA TTT TCA GTC GAC AAC CCA ACC CTT ACC CGA TTC TTC GCT TTA [207] G.crist_GenB ... A.T ..C ..A ... ... ... ..T ..C ..C ... ..A ..T ..T ..C ..A ..A ..A ... ... ... A.C C.C [207] G.crist_m3 ... A.T ..C ..A ... ... ... ..T ..C ..C ... ..A ..T ..T ..C ..A ..A ..A ... ... ... A.C C.C [207] G.scheep_m7 ... A.T ..C ..A ... ..T ... ..T ..C ..C ... ..A ..T ..T ..C ..A ..A ..A ... ... ... A.C C.C [207] G.vict_a1 ... A.T ..C ..A ... ... ... ..C ..C ..C ... ..A ... ... ..C ..A ..A ..A ... ... .C. A.C ..C [207] G.vict_GenB ... A.C ..C ..A ..G ... ... ..T ..C ..C ... ..A ... ... ..C ..A ..A ..A ... ... ... A.C C.C [207] Did.strig ... A.C ..C ..A ... ..T ... ..A ... ... ... ..A ... ... ..C ..A ..C ..A ... ... ... A.C C.T [207] Alec.mdgac_Bnet ... A.T ..C ..A ... ..T ... ... ..G ... ... ..A ... ... ..C ..A T.A ..A ... ... ... A.C C.T [207] Pti.supbs_Bnet ... ..T ..C ..A ... ..T ... ... ..C ... ... ..A ... ... ..C ..A ..A ..A ... ... ... A.. C.C [207] Col_livia ... ..C ..T ..A ... ... ... ..C ... ... ..C ..A ..T ... ..T ..A T.A ..A ... ... ... A.C C.T [207] Lep.mgal_Bnet ... ... ..T ..A ... ... ... ..C ... ..C ..C ..A ..T ... ..C ..A ..A ..A ... ... ... A.C ..G [207] Lep.verful_Bnet ... ... ..C ..A ... ... ... ..T ... ... ..C ..A ..T ... ..C ..A ..A ..A ... ..T ... A.C ... [207] Gal.gallus_net CAC TTC CTC CTC CCC TTT GCA ATC GCA GGT ATT ACT ATC ATC CAC CTC ACC TTC CTA CAC GAA TCA GGC [276] G.crist_GenB ... ... ... ... ... ..C AT. ..T ..T ..C C.C ..C ... ... ... ... ..A ... T.. ..T ... ..C ... [276] G.crist_m3 ... ... ... ... ... ..C AT. ..T ..T ..C C.C ..C ... ... ... ... ..A ... T.. ..T ... ..C ... [276] G.scheep_m7 ... ... ... ... ... ..C AT. ..T ..C ..C C.C ..C ... ... ... ... ..A ... T.. ... ... ..T ... [276] G.vict_a1 ..T ... ... ... ... ..C AT. ... ..T ..C C.C ..C ... ... ... ... ..A ... ... ... ... ..C ... [276] G.vict_GenB ... ... ... ... ... ..C CT. ..T ..C ..C C.C G.C ... ... ... ... ..A ... ... ... ... ..C ... [276] Did.strig ... ... ... ..A ... ..C AT. ... ... ... C.. ... ... ... ... ..A ..A ... ... ..T ... ... ... [276] Alec.mdgac_Bnet ... ... ... ... ... ... AT. ... ... ... C.C ..C ... ... ... ..A ..A ... T.. ... ... ... ... [276] Pti.supbs_Bnet ... ... ... ... ... ..C AT. ... ..G ..C C.C ... ... ... ..T ... ..A ... ... ... ... ... ... [276] Col_livia ... ... ... ... ... ... AT. ... ... ..C C.C ..C ... ... ... ... ... ... ..G ... ... ... ... [276] Lep.mgal_Bnet ... ... ... ... ..A ..C .T. ... ... ..C C.C ..C ..T ... ... ... ... ... T.. ... ... ..C ... [276] Lep.verful_Bnet ... ... ... ... ..A ... AT. ... ... ..C C.C ..C ... ... ... ... ... ... T.. ..T ... ..C ... [276]
G.crist_GenB ... ... ..T ..A ... ... ... ..T ... A.. .GC ... ... ..C ... ..C ... ..C ... .TT ... C.A ... [345] G.crist_m3 ... ... ..T ..A ... ... ... ..T ... A.. .GC ..A ... ..C ... ..C ... ..C ... .TT ... C.A ... [345] G.scheep_m7 ... ... ... ..A ... ... ... ..T ... A.. .GC ..T ... ..C ... ..C ... ..C ... .TT ... C.A ... [345] G.vict_a1 ... ... ... ..A T.. ... ... ..C ... A.. .GG ..T ... ..C ... ..C ... ..C ... .TT ... ..A ... [345] G.vict_GenB ... ... ... ..A T.. ..T ... ..T ... A.. .GC ..T ... ..C ... ..C ... ..C ... .T. ... C.A ... [345] Did.strig ... ... ... ..A ... A.. ... ..C ... A.. .G. ..T ... ..C T.. ..C ... ..C .G. .T. ... C.A ... [345] Alec.mdgac_Bnet ... ... ... ... ... ... ... ..T ... A.. .G. ... ... ..C ... ..C ... ..C ... .T. ... C.A ... [345] Pti.supbs_Bnet ... ... ... ... ... ... ... ..T ... A.. .GC ... ... ..C ... ..C ... ..T ... .TT ..T C.A ... [345] Col_livia ... ... ... ..A ... ... ... A.C ... A.. .GC ..T ... ..C ... ..C ... ..C ... .T. ... C.A ... [345] Lep.mgal_Bnet ... ... ... ..A ... ... ... ..C ... A.. .GC ... ... ..C ... ..C ... ..C ... .T. ... C.A ... [345] Lep.verful_Bnet ... ... ... ..A ... ... ... ..C ... A.. .GC ... ... ..C ... ..C ... ..C ... .T. ..T C.A ... [345] Gal.gallus_net GAC ATT CTG GGC TTA ACT CTC ATA CTC ACC CCA TTC C [382]
G.crist_GenB ... ... ..T ... ..C .TA ..A ... ... CT. ..C C.A A [382] G.crist_m3 ... ... ..T ... ..C .TA ..A ... ... CT. ..C C.A A [382] G.scheep_m7 ... ..C T.T ... ..C .TA ..G ... ... CT. ..C ..A A [382] G.vict_a1 ... ..C ..T ... ..C ..A ..G ... ... CT. ..C C.A A [382] G.vict_GenB ... ..C T.C ... C.C ..A ..A ... ... CTT ..C C.A A [382] Did.strig ... ..C ..T ... ..C .TA T.A ... T.. CT. ..T ..A A [382] Alec.mdgac_Bnet ... ..C ..C ... ..C .TA ..A ... T.. CTA ..C C.G A [382] Pti.supbs_Bnet ... ..C ..T ... ..C .TA ..A ... T.. CT. ..C C.A A [382] Col_livia ... ..C ..C ... ..C .TG A.A ... ... CT. ..C C.A A [382] Lep.mgal_Bnet ... ..C ..T ... ..C ..A ... ... T.. CT. ..C C.A A [382] Lep.verful_Bnet ... ..C ..A ... ..C ..A ... ... T.. CT. ..C C.A A [382]
Lampiran 5 Penjajaran berganda asam amino dari Cyt b parsial (127 aa) G.cristata GenBank, G.victoria GenBank, Goura sp. hasil penelitian, spesies famili Columbidae dan Gallus gallus sebagai pembanding.
Gal.gallus_net WNTGVILLLT LMATAFVGYV LPWGQMSFWG ATVITNLFSA IPYIGHTLVE WAWGGFSVDN PTLTR [ 65]
G.crist_GenB ... ... ... ... ...Q.I.. ... ... [ 65] G.crist_m3 ... ... ... ... ...Q.I.. ... ... [ 65] G.scheep_m7 ... ... ... ... ...Q.I.. ... ... [ 65] G.vict_a1 ... ... ... ...P. ...Q.I.. ... ... [ 65] G.vict_GenB ... ... ... ... ...Q.I.. ... ... [ 65] Did.strig ... ... ... ... ...Q.I.. ... ... [ 65] Alec.mdgac_Bnet ... ... ... ... ...Q.I.. ... ... [ 65] Pti.supbs_Bnet ....I... ... ... ... V....Q.... ... ... [ 65] Col_livia ...V.... ... ... ... V....Q.... ... ... [ 65] Lep.mgal_Bnet ... ... ... ... ...Q.... ... ... [ 65] Lep.verful_Bnet ... ... ... ... ...Q.... ... ... [ 65]
Gal.gallus_net FFALHFLLPF AIAGITIIHL TFLHESGSNN PLGISSDSDK IPFHPYYSFK DILGLTLMLT PF [127] G.crist_GenB ..T... M...L... ... ...NC.. ...F.L. ....FM...L .L [127] G.crist_m3 ..T... M...L... ... ...NCE. ...F.L. ....FM...L .L [127] G.scheep_m7 ..T... M...L... ... ...NC.. ...F.L. ..F.FM...L .L [127] G.vict_a1 .STF... M...L... ... ...NW.. ...F.L. ....F....L .L [127] G.vict_GenB ..T... L...LA.... ... ...NC.. ...F.L. ..F...L .L [127] Did.strig ..T... M...L... ... ..S...NC.. .S...CF.L. ....FM..FL .L [127] Alec.mdgac_Bnet ..T... M...L... ... ...NC.. ...F.L. ....FM..FL .L [127] Pti.supbs_Bnet ..T... M...L... ... ...NC.. ...F.L. ....FM..FL .L [127] Col_livia ..T... M...L... ... ....T.NC.. ...F.L. ....FMM..L .L [127] Lep.mgal_Bnet ..T... V...L... ... ...NC.. ...F.L. ....F...FL .L [127] Lep.verful_Bnet ..T... M...L... ... ...NC.. ...F.L. ....F...FL .L [127]
UUC10.4(1.70) Leu UUA 1.4(0.42) UUG 0.0(0.00) UCC 4.2(3.15) UCA 2.8(2.10) UCG 0.0(0.00) UAC 2.4(1.60) * UAA 0.0(0.00) UAG 0.0(0.00) UGC 0.8(2.00) Trp UGA 4.8(1.85) UGG 0.4(0.15) Leu CUU 0.8(0.24) CUC 7.8(2.36) CUA 9.4(2.85) CUG 0.4(0.12) Pro CCU 0.0(0.00) CCC 6.0(2.93) CCA 2.2(1.07) CCG 0.0(0.00) His CAU 0.6(0.30) CAC 3.4(1.70) Gln CAA 1.0(1.00) CAG 1.0(1.00) Arg CGU 0.0(0.00) CGC 0.0(0.00) CGA 1.0(4.00) CGG 0.0(0.00) Ile AUU 3.0(0.55) AUC 8.0(1.45) Met AUA 4.4(2.00) AUG 0.0(0.00) Thr ACU 0.0(0.00) ACC 5.8(2.07) ACA 5.2(1.86) ACG 0.2(0.07) Asn AAU 1.2(0.40) AAC 4.8(1.60) Lys AAA 2.0(2.00) AAG 0.0(0.00) Ser AGU 0.0(0.00) AGC 0.0(0.00) * AGA 0.0(0.00) AGG 0.0(0.00) Val GUU 1.0(0.67) GUC 3.0(2.00) GUA 2.0(1.33) GUG 0.0(0.00) Ala GCU 1.4(0.90) GCC 3.8(2.45) GCA 1.0(0.65) GCG 0.0(0.00) Ala GAU 1.2(0.86) GAC 1.6(1.14) Glu GAA 2.2(2.00) GAG 0.0(0.00) Gly GGU 1.4(0.51) GGC 6.2(2.25) GGA 2.2(0.80) GGG 1.2(0.44) * Rata-rata kodon 127
angka cetak tebal: besar penggunaan kodon pada Goura sp.
(angka): Indeks pengunaan adalah perbandingan jumlah kodon yang digunakan terhadap rata-rata kodon sinonim.
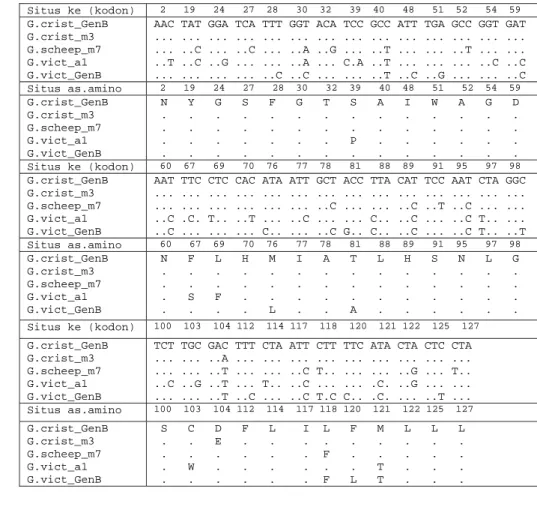
Tabel 3 Kodon penyandi dan asam amino yang disandikan yang mengalami mutasi pada Cyt b parsial dari G.cristata GenBank, G.victoria GenBank, dan Goura sp. hasil penelitian.
Situs ke (kodon) 2 19 24 27 28 30 32 39 40 48 51 52 54 59 G.crist_GenB G.crist_m3 G.scheep_m7 G.vict_a1 G.vict_GenB
AAC TAT GGA TCA TTT GGT ACA TCC GCC ATT TGA GCC GGT GAT ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ..C ... ..C ... ..A ..G ... ..T ... ... ..T ... ... ..T ..C ..G ... ... ..A ... C.A ..T ... ... ... ..C ..C ... ... ... ... ..C ..C ... ... ..T ..C ..G ... ... ..C Situs as.amino 2 19 24 27 28 30 32 39 40 48 51 52 54 59 G.crist_GenB G.crist_m3 G.scheep_m7 G.vict_a1 G.vict_GenB N Y G S F G T S A I W A G D . . . . . . . . . . . P . . . . . . . . Situs ke (kodon) 60 67 69 70 76 77 78 81 88 89 91 95 97 98 G.crist_GenB G.crist_m3 G.scheep_m7 G.vict_a1 G.vict_GenB
AAT TTC CTC CAC ATA ATT GCT ACC TTA CAT TCC AAT CTA GGC ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ..C ... ... ..C ..T ..C ... ... ..C .C. T.. ..T ... ..C ... ... C.. ..C ... ..C T.. ... ..C ... ... ... C.. ... ..C G.. C.. ..C ... ..C T.. ..T Situs as.amino 60 67 69 70 76 77 78 81 88 89 91 95 97 98 G.crist_GenB G.crist_m3 G.scheep_m7 G.vict_a1 G.vict_GenB N F L H M I A T L H S N L G . . . . . . . . . S F . . . . . . . . L . . A . . . . Situs ke (kodon) 100 103 104 112 114 117 118 120 121 122 125 127 G.crist_GenB G.crist_m3 G.scheep_m7 G.vict_a1 G.vict_GenB
TCT TGC GAC TTT CTA ATT CTT TTC ATA CTA CTC CTA ... ... ..A ... ... ... ... ... ... ... ... ... ... ... ..T ... ... ..C T.. ... ... ..G ... T.. ..C ..G ..T ... T.. ..C ... ... .C. ..G ... ... ... ... ..T ..C ... ..C T.C C.. .C. ... ..T ... Situs as.amino 100 103 104 112 114 117 118 120 121 122 125 127 G.crist_GenB G.crist_m3 G.scheep_m7 G.vict_a1 G.vict_GenB S C D F L I L F M L L L . . E . . . . . . . . . . . F . . . . . . W . . . . . . T . . . . . . . F L T . . .
Ordo : Columbiformes Famili : Phasianidae Spesies : Gallus gallus Nama Umum: Chicken
Ordo : Columbiformes Famili : Columbidae
Spesies : Alectroenas madagascarensis Nama umum: Madagascar blue-pigeon
Ordo : Columbiformes Famili : Columbidae
Spesies : Didunculus strigirostris Nama umum: Tooth-billed pigeon
Ordo : Columbiformes Famili : Columbidae Spesies : Columba livia Nama umum: Rock Pigeon/ Common pigeon
Lanjutan Lampiran 7
Ordo : Columbiformes Famili : Columbidae
Spesies : Leptotila verreauxi fulviventri
Nama umum: White-tipped doves
Ordo : Columbiformes Famili : Columbidae
Spesies : Ptilinopus superbus Nama umum: Fruit-doves
Ordo : Columbiformes Famili : Columbidae Spesies : Leptotila megalura Nama umum: White-faced dove