ISSN: 2089-9858 ® PS AGRONOMI PPsUNHALU
A
NALISIS
V
ARIASI
G
ENETIK
J
AMBU
M
ETE
(
A
NACARDIUM OCCIDENTALE
L.)
A
SAL
S
ULAWESI
T
ENGGARA
M
ENGGUNAKAN
M
ARKA
M
OLEKULER
AFLP
(
Amplified Fragment Length Polimorphism
)
Genetic Variation Analysis of Cashew Trees (
Anacardium occidentale
L.)
in Southeast Sulawesi using AFLP
(
Amplified Fragment Length Polymorphism
)
Oleh:
Richael Syam
1), Gusti Ray Sadimantara
2*), dan Muzuni
2).
1)Alumni S1 Agronomi Fakultas Pertanian Universitas Haluoleo 2)Dosen Fakultas Pertanian Universitas Haluoleo
*)Alamat surat-menyurat:gusti5@yahoo.com
ABSTRACT.The research was done to study the presence of genetic variation of cashew (Anacardium occidentale
L.) of four regencies of Southeast Sulawesi, i.e. Buton, Muna, Bombana and South Konawe regencies. The study was conducted in 2 places; i.e. in The Genetic Laboratory, Biology Department, Mathematics and Science Faculty of Haluoleo University for the DNA Isolation and in The Molecular Biology Laboratory of and Plant Cellular, Research Centre for Biotechnological Resources and Biotechnology (PPSHB) Bogor Agricultural University for AFLP stage. The research was conducted from November 2011 to June 2012. The research was conducted the first step for searching of excellent cashew by observing genetic variation using AFLP (Amplified Fragment Length Polymorphism) technique. The research was started by doing isolation of cashew genome, cutting DNA with two restriction enzymes (EcoRl danMse1), amplification selectively using four selective primer combinations, and doing amplicon on gel electrophoresis poliacrylamid. Total strand obtained was 322. Percentage average of polymorphisms obtained was 70,21% of four primer combinations showed the presence of genetic variation in each sample. Dendrogram analysis showed that there were two groups; first group consisted of the cashew from Buton, Muna and Bombana regencies and the second group was South Konawe regency that had a difference compared with three other samples, i.e Buton, Muna and Bombana regencies.
Key words:Amplified Fragment Length Polymorphism(AFLP), cashew tree, polymorphisms
ABSTRAK. Penelitian dilakukan untuk mempelajari adanya variasi genetik pada jambu mete ( Anacardium occidentale L) yang berasal dari empat kabupaten di Sulawesi Tenggara yaitu : Kabupaten Buton, Kabupaten Muna, Kabupaten Bombana dan Kabupaten Konawe Selatan. Penelitian dilaksanakan di dua tempat yakni di Laboratorium Genetika Jurusan Biologi Fakultas MIPA, Universitas Haluoleo untuk tahapan Isolasi DNA dan Laboratorium Biologi Molekuler dan Seluler Tanaman, Pusat Penelitian Sumber Daya Hayati dan Bioteknologi (PPSHB) Institut Pertanian Bogor untuk tahapan AFLP. Penelitian dilakukan mulai bulan November 2011 sampai dengan bulan Juli 2012. Penelitian dilakukan sebagai langkah awal pencarian jambu mete unggul dengan melihat variasi genetik menggunakan teknik AFLP (Amplified Fragment Length Polymorphism). Penelitian diawali dengan mengisolasi genom jambu mete, memotong DNA dengan dua enzim restriksi (EcoRI danMseI), mengamplifikasi secara selektif dengan 4 kombinasi primer selektif, dan menjalankan amplikon pada elektroforesis gel poliakrilamid. Jumlah pita yang didapatkan berjumlah 322 pita. Rata–rata persentase polimorfisme yang diperoleh adalah 70,21% dari 4 kombinasi primer yang menunjukkan adanya variasi genetik pada setiap sampel. Hasil dendrogram menunjukkan bahwa terdapat dua kelompok yaitu kelompok pertama terdiri dari sampel Kabupaten Buton, Kabupaten Muna dan Kabupaten Bombana dan kelompok kedua yaitu Kabupaten Konawe Selatan. Jambu mete asal Kabupaten Konawe Selatan memiliki perbedaan dibandingkan dengan ketiga sampel lainnya yaitu Kabupaten Buton, Kabupaten Muna dan Kabupaten Bombana.
Kata kunci:Amplified Fragment Length Polymorphism(AFLP), jambu mete, polimorfisme
PENDAHULUAN
Jambu mete (Anacardium occidentale L.) berasal dari Brasilia Tenggara, pertama kali dibawa
oleh pelaut Portugis ke Kepulauan Nusantara mela-lui Malabar, India pada abad ke-15 dengan demi-kian jambu mete telah lama dikenal di Indonesia, namun belum dibudidayakan dengan baik. Pada
umumnya (98%) jambu mete diusahakan oleh petani secara tercampur dengan tanaman industri dan tanaman buah-buahan lain dan hanya sebagian kecil saja (2 %) yang diusahakan oleh perkebunan negara dan perkebunan besar swasta (Kemal, 2000).
Jambu mete merupakan komoditas perke-bunan yang strategis, karena tanaman ini merupa-kan komoditas ekspor, yang mempunyai prospek pasar dalam negeri dan dapat meningkatkan pen-dapatan petani terutama di lahan-lahan marginal yang banyak terdapat di Kawasan Timur Indonesia seperti Sulawesi Tenggara, Sulawesi Selatan, NTB, NTT, Maluku dan Bali (Zaubir dan Suryadi, 2003).
Biji jambu mete mempunyai nilai ekonomi yang cukup baik, demikian pula cairan yang terkan-dung dalam kulit biji yang disebut CNSL (Cashew Nut Sheel Liquid) merupakan bahan ekspor non tradisional, sedangkan limbah yang diperoleh dari olahan cairan kulit biji merupakan bahan campuran pembuatanhardboard. Buah semunya dapat dibuat anggur dan sari buah. Kacang mete mengandung protein rata-rata 19 % dan lemak rata-rata 47 %, sedangkan sari buahnya mengandung vitamin A, B, dan C. Kandungan vitamin C ini 3 - 4 kali lipat dari kandungan sari buah jeruk (Van Eijnatten, 2011).
Komoditas jambu mete sebagai komoditas perkebunan rakyat di daerah Sulawesi Tenggara memiliki potensi yang besar untuk dikembangkan. Nilai ekonomis komoditas jambu mete di Sulawesi Tenggara dapat terlihat dari nilai dan volume per-dagangan antar pulau pada tahun 2002 dalam bentuk gelondongan sebesar 2.157,40 ton dengan nilai mencapai Rp. 8.689.410.000 sedangkan dalam bentuk kacang sebesar 26 ton, dengan nilai Rp. 49.000.000.000 dan ekspor gelondongan mencapai 5.375.700.000,- dengan volume sebesar 102 ton (BPMD Sultra, 2010).
Dalam bidang pemuliaan tanaman, pe-manfaatan jambu mete hingga saat ini masih ter-batas pada seleksi dan uji lapangan dengan meng-gunakan karakter morfologi dalam mendeskripsikan tanaman. Karakter morfologi telah banyak dipergu-nakan, namun karakter morfologi memiliki kendala yaitu adanya faktor lingkungan sehingga perbedaan antar spesies berkerabat dekat seringkali sulit di-amati. Kebanyakan karakter sulit dianalisis karena tidak memiliki sistem pengendalian genetik yang sederhana. Oleh karena itu, diperlukan adanya ana-lisis molekuler. Teknik molekuler memberikan pe-luang untuk mengembangkan dan mengidentifikasi peta genetik dari suatu kultivar jambu mete. Pende-katan genetika molekuler dengan menggunakan penanda DNA telah berhasil membentuk penanda molekuler yang mampu mendeteksi gen dan sifat-sifat tertentu dan mengevaluasi keragaman dan
evolusi pada tingkat genetik. Beberapa teknik penanda DNA tersebut adalah Random Amplified Polymorphic DNA (RAPD), Restriction Fragment Length Polymorphism (RFLP), Amplified Fragment Length Polymorphism (AFLP), Simple Sequence Repeat(SSR),Mikrosatelit(Hoon-Limet al.,1999).
Pemakaian penanda molekuler berdasar-kan pola pita DNA telah banyak digunaberdasar-kan untuk menyusun kekerabatan beberapa individu dalam spesies maupun kekerabatan antar spesies. Penggu-naan kekerabatan dapat dijadikan rujukan dalam pemuliaan persilangan untuk mendapatkan keane-karagaman yang tinggi dari hasil persilangan. Peng-gunaan penanda DNA dapat membantu pelaksa-naan pemilihan tetua persilangan yang memiliki perbedaan tinggi secara genetik (Correa et al.,
1999).
Variasi genetik jambu mete dilihat dari polimorfisme yang digambarkan dengan perbedaan pola pita yang dipisahkan berdasarkan ukuran berat molekul. Polimorfisme adalah variasi alel pada lokus DNA tertentu dari suatu populasi. Data polimor-fisme dapat digunakan untuk melihat variasi genetik pada populasi jambu mete. Variasi tersebut di-harapkan terekspresi sampai tingkat fenotip jambu mete. Salah satu teknik untuk mendeteksi adanya variasi genetik adalah AFLP (Amplified Fragment Length Polymorphism). Prinsip dasar teknik AFLP adalah mendeteksi perbedaan letak marka DNA di seluruh genom yang berupa urutan basa tertentu. Deteksi marka DNA tersebut dilakukan dengan amplifikasi secara selektif terhadap fragmen hasil digesti dua enzim restriksi. Enzim restriksi yang digunakan antara lain adalah EcoR1 (GAATTC) dan
Mse1 (TTAA). Amplifikasi dilakukan dengan primer selektif yang terdapat tambahan tiga basa pada ujung 3’. Primer dibagi menjadi dua, yaitu primer dengan ujung pemotonganEcoRI dan primer untuk ujung pemotonganMseI. Variasi genetik ditentukan dengan 4 kombinasi primer yang berasal dari ujung
EcoRI dan MseI. Hasil amplifikasi selektif adalah pita-pita DNA dengan berbagai ukuran yang dipi-sahkan oleh elektroforesis gel poliakrilamida. Anali-sis dilakukan untuk melihat jumlah dan keberadaan pita-pita yang mampu menunjukkan variasi genetik (Saunders, 2001).
Keunggulan teknik AFLP adalah dapat men-deteksi variasi genetik tanpa memerlukan informasi urutan basa genom. Selain itu, teknik AFLP memiliki tingkat reproduksi yang tinggi berdasarkan ampli-fikasi selektif fragmen hasil digesti genom (Mueller dan Wolfenbarger, 1999). Teknik AFLP mampu menganalisis genom secara menyeluruh sehingga dihasilkan informasi yang memadai untuk mengana-lisis variasi genetik (Mba dan Tohme, 2005).
Data hasil penelitian variasi genetik jambu mete diharapkan dapat menunjukkan perbedaan genetik jambu mete dan ekspresinya pada perbe-daan karakter fenotip. Perbeperbe-daan genetik tersebut dapat digunakan sebagai data awal untuk meleng-kapi penelitian terhadap jambu mete yang lebih kompleks.
Berdasarkan uraian di atas, maka peneli-tian ini dilakukan untuk mempelajari variasi genetik jambu mete yang ditanam pada empat lokasi yang berbeda di Sulawesi Tenggara menggunakan marka molekuler AFLP.
BAHAN DAN METODE
Penelitian ini dilakukan di dua tempat yaitu di Laboratorium Genetika Jurusan Biologi Fakultas MIPA, Universitas Haluoleo untuk tahapan Isolasi DNA dan di Laboratorium Biologi Molekuler dan Seluler Tanaman, Pusat Penelitian Sumber Daya Hayati dan Bioteknologi (PPSHB) Institut Pertanian Bogor untuk tahapan AFLP. Adapun waktu penelitian ini dilakukan mulai bulan Novem-ber 2011 sampai dengan bulan Juli 2012. Bahan penelitian adalah daun muda jambu mete yang diperoleh dari 4 lokasi yang berbeda di wilayah Sulawesi Tenggara yaitu: di Kabupaten Muna, Bombana, Buton, dan Konawe Selatan.
Bahan-bahan digunakan untuk isolasi DNA dan elektroforesis gel agarosa antara lain buffer TE (Tris HCL : EDTA), buffer ekstraksi (CTAB 2 % ; EDTA 0,02 M pH 8,0 ; Tris HCl 0,1 M pH 8,0 ;NaCl 1,26 M; H20 steril; PVP 3 %), kloroform isoamil (24 : 1),
nitrogen cair, etanol absolut, sodium asetat, enzim RNase, agarosa, buffer TBE 0,5 x, etidium bromida dan loading buffer. Bahan yang digunakan untuk proses AFLP adalah enzimEcoRI danMseI, 10 mM ATP, 5 x RL-Buffer, 1 unit T4 DNA ligase, Milliq H20,
5 mM dNTPs, 10 x super buffer, buffer TE, Taq-polymerase (5 unit/µl), 5 Primer masing–masing adalah P11-700 (GAC TGC GTA CAT GCA GAA), M48 (GAT GAG TCC TGA GTA AAC AC), M49 (GAT GAG TCC TGA GTA AAC AG), M50 (GAT GAG TCC TGA GTA AAC AT), M51 (GAT GAG TCC TGA GTA AAC CA), masing-masing dengan kombinasi yaitu P11-700 dengan M48, P11-P11-700 dengan M49, P11-P11-700 dengan M50, dan P11-700 dengan M51. Bahan yang digunakan untuk elektroforesis gel poliakri-lamid adalah loading buffer formamide (98% for-mamide, 10 mM EDTA pH 8,0 dan 0,1% bromo-phenol) gel poliakrilamid (50% long ranger, sanver-tech), urea 40 g, etanol absolute, asam asetat glacial,bind silane,repel silane, H2O, buffer TBE 10
x, ammonium persulfat 1,6% dan TEMED.
Alat yang digunakan adalah termos es, pipet mikro 0,1 -2 µl, 2-20 µl, 20-200 µl, 100-1.000
µl, tip 10 µl ,100 µl, 1.000 µl, freezer-20oC, lemari pendingin 4o C, mesin PCR, eppendorf 0,5 ml, 1,5
ml, dan 2,0 ml, pompa vakum, rak tabung, mesin sentrifugasi, timbangan, vorteks, inkubator, water-bath, oven, spatula, perangkat elektroforesis, dan lumpang. Alat gelas yang digunakan adalah gelas ukur, labu erlenmeyer, dan tabung penyimpanan bahan serta peralatan gelas yang umum digunakan di laboratorium.
Sampel yang digunakan berasal dari daun jambu mete dari empat lokasi yang berbeda. Adapun keempat lokasi pengambilan sampel, yaitu: (a) Desa Bombana Wulu, Kec. Gu Kab. Buton, (b) Desa Rarontole, Kec. Kabaena Kab. Bombana, (c) Desa Wapunto Kec. Duruka Kab. Muna, dan (d) Kebun Penelitian Onembute, Kab. Konawe Selatan. Pengambilan sampel dilakukan secara acak dengan mengambil daun yang masih muda atau pada pucuk pertama. Sampel daun yang telah dipilih kemudian dimasukkan dalam termos es untuk sementara waktu sebelum dimasukkan dalam freezer.
DNA jambu mete diekstraksi dari bagian daunnya dengan menggunakan metode CTAB (cetyltrimetyl ammonium bromide). Sebelum dilaku-kan ekstraksi, terlebih dahulu disiapdilaku-kan buffer eks-traksi, yang kebutuhannya tergantung dari jumlah sampel yang akan diekstraksi. Sampel yang akan diekstraksi di timbang (0,15 mg), lalu dipotong kecil-kecil, digerus menggunakan lumpang dengan ban-tuan nitrogen cair, dimasukkan dalam tabung eppendorf dan ditambahkan 1,5 ml buffer ekstraksi divortex dan dipanaskan pada waterbath selama 30 menit pada suhu 650 C (setiap 5 menit sekali di-keluarkan dan dibolak-balik) lalu disentrifuse pada 10.000 rpm selama 10 menit, suhu 4oC, mengambil supernatan lalu dimasukkan dalam eppendorf baru dan ditambahkan 1 x volume kloroform isoamil untuk melarutkan senyawa-senyawa organik.
Selanjutnya disentrifuse lagi pada 10.000 rpm selama 10 menit, suhu 4oC, kemudian
super-natan diambil dan ditambahkan 1 x volume sodium asetat dan 2 x volume etanol absolut dingin lalu diendapkan selama 2 jam dalam freezer. Setelah pengendapan kemudian disentrifuse lagi pada 10.000 rpm selama 20 menit, suhu 4oC, kemudian supernatan dibuang dan mengambil endapannya yang berada pada bagian bawah. Selanjutnya endapan tersebut ditambahkan dengan 500 μl ethanol 70% untuk membersihkan dari sodium asetat, dikocok sebentar lalu disentrifuse lagi pada 10.000 rpm selama 10 menit, suhu 4o C lalu endapannya diambil dengan membuang cairan bagian atasnya kemudian dikeringkan pada suhu 37o C di oven selama 20 menit hingga kering kemudian ditambahkan 20-30μl H2O, kocok hingga
freezer pada suhu -20oC. Selanjutnya larutan DNA ditambahkan enzim RNase hingga konsentrasi 100
μg/ml lalu diinkubasi pada suhu 37oC selama 1 jam. Selanjutnya larutan ditambahkan dengan 500 μl buffer TE, dikocok lalu ditambahkan kloroform isoamil alkohol (24 : 1).
Larutan dihomogenkan dengan vorteks, kemudian disentrifugasi dengan kecepatan 11.000 rpm selama 10 menit. Cairan bagian atas kemudian dipindahkan pada tabung baru. Sempel kemudian ditambahkan 0,1 volume sodium asetat 3 M, pH 5,2 dan 2 volume etanol absolut. Tabung dibolak balik perlahan lahan kemudian diinkubasi pada suhu -20o C selama 30 menit. Pelet dikeringkan dengan vakum selama 15 menit. Setelah pelet kering kemudian ditambahkan buffer TE sebanyak 20-30 µl, tabung dipanaskan pada suhu 50o C dengan heatblock sampai pelet larut.
Hasil isolasi DNA diuji dengan elektrofore-sis gel agarosa 0,8 % menurut Sambrook dan Russell (2001). Langkah pertama elektroforesis gel agarosa adalah bahan agarosa ditimbang sebanyak 0,2 g dan dilarutkan dengan buffer TBE 0,5 x sebanyak 25 ml di dalam labu erlenmeyer. Larutan agarosa kemudian dipanaskan selama 30 detik. Gel agarosa didiamkan pada suhu ruang selama 1 jam.
Gel agarosa yang telah dibekukan selama 1 jam siap untuk digunakan. Gel agarosa diletakkan pada chamber elektroforesis yang telah diisi dengan running buffer TBE 0,5 x. Loading buffer sebanyak 1 µl dicampur dengan akuabides seba-nyak 3 µl dan sampel sebaseba-nyak 2 µl. Percampuran dilakukan diatas kertas parafilm. Campuran terse-but kemudian dimasukkan kedalam sumur pada gel agarosa. Marka yang digunakan adalah marka DNA phageʎsebanyak 10 ng dan 30 ng. Elektroforesis dilakukan dengan voltase 100 volt selama 20 menit. Hasil elektroforesis kemudian dilihat di bawah sinar ultraviolet.
Hasil positif gel agarosa adalah munculnya pita yang berpendar jika gel dilihat di bawah sinar ultraviolet. Hasil negatif elektroforesis gel agarosa adalah tidak adanya pita yang berpendar jika gel agarosa dilihat di bawah sinar UV. Ketebalan dan intensitas pita DNA sampel dibandingkan dengan marka DNA phage ʎ yang telah diketahui konsen-trasinya. Hasil isolasi DNA jambu mete yang dida-patkan disimpan pada suhu 4oC, dan dapat digu-nakan untuk aplikasi selanjutnya.
Digesti genom dilakukan menurut invitro-gen (2003), digesti invitro-genom dilakukan dengan meng-gunakan enzim restriksi EcoRI/MseI. Sampel genom (500 ng dalam 5 µl), enzim EcoRI/MseI (0,5 µl), 5 x RL-buffer (10 µl), dan milliqH2O (30 µl) sampai
volume 50 µl dicampurkan di dalam tabung eppendorf 1,5 ml. Campuran kemudian dicampur
secara perlahan dan disentrifugasi singkat untuk menurunkan seluruh cairan dalam tabung. Sampel diinkubasi dalam inkubator selama 2 jam dengan suhu 37oC. Inkubasi sampel selama 15 menit pada suhu 70oC dilakukan untuk menginaktivasi enzim.
Tabung diletakkan dalam kotak es sampai tahap selanjutnya
Ligasi adapter dilakukan menurut invitro-gen (2003). Sampel yang telah didigesti ditambah-kan larutan ligase adapter (2 µl), enzim T4 DNA ligase (1 µl), 10 mM ATP, sampel dicampurkan perlahan, disentrifugasi, kemudian diinkubasi pada suhu 20oC ± 2oC selama 2 jam. Sampel diencerkan 10 kali dengan mengambil 10 µl sampel kemudian dipindahkan ke tabung eppendorf 1,5 ml dan dila-rutkan dengan 90 µl buffer TE. Sisa sampel disimpan pada suhu -20oC.
Preamplifikasi dilakukan menurut invitro-gen (2003) dengan modifikasi yaitu penambahan 5 mM dNTP. Preamplifikasi dilakukan dengan cara mencampurkan sampel DNA (5 µl), pre-amp primer mix (26 µl), 10 x super buffer (2 µl), 5 mM dNTP sebanyak 0,8 µl, dan enzim Taq-polymerase (5 unit/µl) sebanyak 0,08 µl didalam tabung eppendorf 200 µl. Larutan dicampur secara perlahan. Sampel dimasukkan ke dalam mesin PCR dengan 24 siklus seperti 94oC selama 30 detik, 56oC selama 30 detik, dan 72oC selama 60 detik dengan temperatur akhir
4oC. Hasil preamplifikasi disimpan pada suhu 20oC di freezer. Sebelum digunakan sebagai cetakan pada amplifikasi selektif, hasil preamplifikasi dien-cerkan dengan milliqH2O.
Amplifikasi selektif dilakukan menurut in-vitrogen (2003). Langkah yang dilakukan adalah membuat campuran dengan komposisi primerEcoRI sebanyak 0,5 µl dan primerMseI sebanyak 0,3 µl, 5 mM dNTPs (0,4 µl), 10 x super buffer (1,0 µl), milliq H2O sebanyak 2,8 µl, dan enzim Taq-polymerase 5
unit/µl sebanyak 0,04 µl. Reaksi amplifikasi dilaku-kan pada tabung mikrosentrifugasi 200 µl dengan komposisi DNA sampel hasil preamplifikasi yang telah diencerkan dan komposisi campuran. Larutan dicampur perlahan. Amplifikasi dilakukan dengan menggunakan mesin PCR. Program yang digunakan adalah sebagai berikut: Satu siklus 94oC selama 30 detik, 65o C selama 30 detik (penurunan 0,7 0C setiap siklus hingga mencapai 56 0C) dan 72o C
selama 60 detik untuk 12 siklus dan sisa 24 siklus dilakukan dengan suhu 94oC selama 30 detik, suhu 56o C selama 30 detik, dan suhu 72oC selama 60
detik dan diakhiri dengan suhu 10oC.
Hasil amplifikasi selektif kemudian dianali-sis dengan menggunakan elektroforedianali-sis gel poliakri-lamid menurut Sambrook dan Russell (2001). Lang-kah pertama adalah menyiapkan alat pencetak gel. Alat–alat pencetak gel terdiri atas dua buah
lempeng kaca, dua buah pemisah, dan dua buah penjepit kaca. Gel poliakrilamid 6% dapat dibuat dengan cara mencampurkan 13,3 ml polyacrilamide gel 45% (29 : 1), 41,4 ml H2O, 10 x TBE 10 ml, dan
urea 40 g. Larutan diinkubasi pada suhu 55o C
sampai seluruh urea larut. Ammonium persulfate 1,6% sebanyak 3,3 ml dan TEMED sebanyak 50 µl ditambahkan pada larutan gel dan diaduk selama 5 menit.
Pencetak gel disiapkan dengan cara, kaca panjang diberi campuran larutanbind silane, etanol absolut, asam asetat glasial sebanyak 1 ml dan disebar merata pada permukaan kaca dengan menggunakan tisu. Setelah 5 menit kaca diberi etanol absolut sebanyak 2 ml dan dilap dengan tisu. Pencucian dengan etanol dilakukan sebanyak 3 kali. Kaca pendek yang terhubung dengan tangki buffer diberi repel silane sebanyak 2 ml dan disebar merata pada seluruh permukaan kaca dengan tisu. Setelah 10 menit, kaca diberi H2O dan dilap dengan
tisu.
Pencetak gel dirancang dengan cara, mele-takkan pemisah setebal 0,4 mm dilemele-takkan diatas kaca pendek. Kaca panjang diletakkan diatas kaca pendek dan pemisah. Kedua lempeng kaca kemu-dian dijepit dengan penjepit pada kedua sisi, kemudian bagian bawah kaca ditahan dengan menggunakan karet silikon. Pencetak gel diletakkan
secara horizontal. Campuran gel dimasukkan ke pencetak gel dengan menggunakan syringe 60 ml. Gel kemudian didiamkan selama 30-60 menit. Sharktooth comb diangkat secara perlahan ketika gel sudah mengeras dan bagian comb yang tajam dimasukkan ke dalam gel 1 mm, sehingga terbentuk well yang rata.
Gel diletakkan pada tangki elektroforesis. Penampungan buffer atas diisi dengan 500 ml buffer 1 x TBE, sedangkan penampungan buffer bawah diisi dengan 350 ml buffer 1 x TBE. Sampel DNA dicampur dengan loading buffer formamide (20 µl). Sampel didenaturasi pada suhu 94oC selama lima menit kemudian langsung diletakkan. Sampel dimasukkan ke dalam well dengan tips dan mikro-pipet sebanyak 3µl. Elektroforesis dilakukan selama 3 jam 40 menit dengan daya 40 W. Setelah elek-troforesis selesai, buffer dipindahkan dari tempat penampungan. Pemisah dan kedua kaca dilepaskan lalu gel menempel pada kaca panjang.
Analisis pita AFLP dilakukan dengan membe-rikan angka 1 untuk keberadaan pita dan angka 0 untuk tidak adanya pita pada tabel data biner. Jumlah seluruh pita dan baris yang mengandung pita polimorfisme dihitung, kemudian persentase polimorfisme dihitung berdasarkan rumus sebagai berikut (Chenet al., 2004).
pita
seluruh
dengan
baris
Polymorfis
pita
dengan
baris
sme
Polymorphi
Persentase
_
_
_
_
_
_
_(%)
_
Pita polimorfis adalah pita yang tidak ter-dapat pada seluruh sampel sedangkan pita umum adalah pita yang terdapat pada seluruh sampel. Interpretasi pita dilakukan untuk mempermudah dalam melihat lokasi pita-pita spesifik. Data binari
seluruh primer yang mengandung pita polimorfis dimasukkan ke dalam program SPSS (Statistical Package for Social Science) versi 15.0 metode
Complete Linkage fungsi Phi (1,0). Analisis data biner menghasilkan gambar dendogram.
HASIL
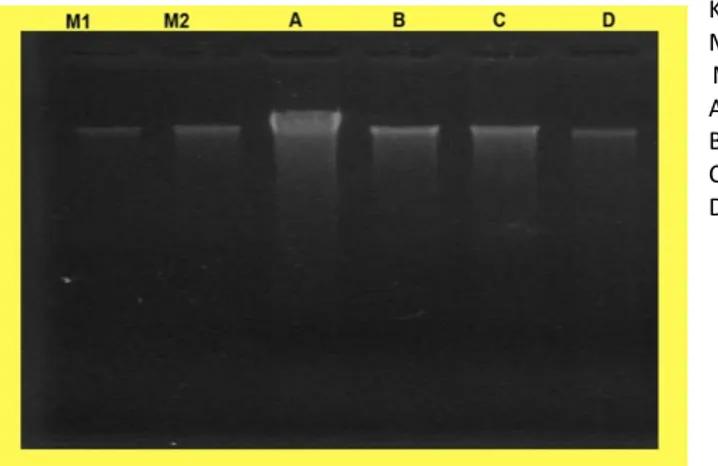
Keterangan :
M1. Marker DNA phageλ (10 ng)
M2. Marker DNA phageλ (30 ng)
A. Kab. Konsel (60 ng/µl) B. Kab. Bombana (20 ng/µl) C. Kab. Muna (20 ng/µl) D. Kab. Buton (5 ng/µl)
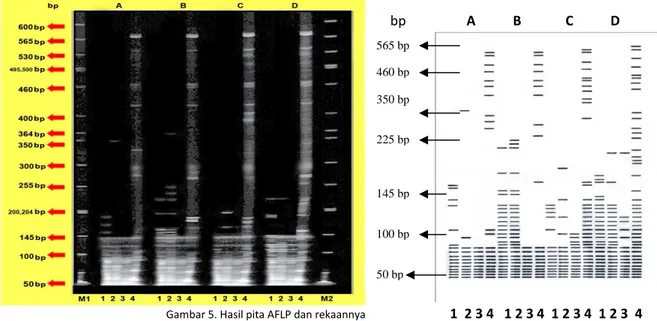
bp A B C D
Gambar 5. Hasil pita AFLP dan rekaannya 1 2 3 4 1 2 3 4 1 2 3 4 1 2 3 4 Keterangan: A, B, C, D = Buton, Muna, Bombana, Konsel; M1, M2 = Marker 1, Marker 2; Kombinasi P11 – 700 dan M48, B. Kombinasi
P11 – 700 dan M49, C. Kombinasi P11 – 700 dan M50, D. Kombinasi P11 – 700 dan M51
PEMBAHASAN
Genom jambu mete diisolasi dari daun jambu yang diambil dari daun muda. Genom meru-pakan seluruh materi DNA pada suatu organisme. Isolasi genom dilakukan dengan metode Bosquet (1990). Isolasi genom menggunakan buffer eks-traksi yang mengandung CTAB, merupakan deter-gen yang dapat melisis membran sel dan mampu mengendapkan polisakarida serta senyawa fenolik yang terdapat pada tanaman jambu mete. Kemam-puan CTAB mengendapkan polisakarida dan se-nyawa fenolik dipengaruhi oleh konsentrasi garam. Jika konsentrasi garam pada buffer lebih dari 0,5 M maka CTAB dapat mengendapkan polisakarida dan senyawa fenolik serta membentuk kompleks dengan DNA. Konsentrasi garam yang digunakan pada isolasi genom jambu mete adalah 1,26 M sehingga polisakarida dan senyawa fenolik pada jambu mete dapat diendapkan (Moore dan Dowhan, 2002).
Genom jambu mete dimurnikan dengan senyawa kloroform isoamilalkohol (24:1) untuk mengekstrasi protein dan RNase untuk melisiskan RNA. Isopropanol atau etanol absolut dapat digu-nakan untuk mengendapkan DNA dan etanol 70 % untuk memisahkan genom dari garam-garam mine-ral, serta melarutkan sisa CTAB. Pemurnian terse-but bertujuan menghilangkan senyawa-senyawa yang dapat menghambat reaksi enzimatis pada proses AFLP (Weisinget al., 1995).
Menurut Weisinget al. (1995), konsentrasi DNA dapat dihitung dengan membandingkan intensitas terang pita dan ketebalan pita DNA pada gel agarosa dengan pita DNA pada marka DNA yang
telah diketahui konsentrasinya. Pita DNA sampel jambu mete dibandingkan dengan pita DNA phage λ.
Menurut Moore dan Dowhan (2002), jum-lah total DNA yang diperoleh dapat diperbanyak dengan menaikkan konsentrasi NaCl pada buffer ekstraksi dan memodifikasi suhu serta waktu dalam tahap presipitasi DNA dengan etanol absolut.
Hasil isolasi DNA yang diperoleh menun-jukkan bahwa sampel jambu mete asal Kab. Konsel memiliki konsentrasi DNA yang tertinggi (120 ng/µl) dan jambu mete asal Kab. Buton (10 ng/µl) memiliki konsentrasi DNA yang terendah. Tinggi dan rendah-nya konsentrasi DNA yang diperoleh, hal ini mung-kin disebabkan oleh maksimum tidaknya kerja buffer ekstrasksi yang digunakan dalam melisis dinding sel, komposisi dinding sel, proses pengge-rusan dll. Konsentrasi DNA yang tinggi dapat meng-indikasikan bahwa jumlah pita-pita DNA yang di-hasilkan nantinya pada proses AFLP akan semakin banyak. Adapun isolasi DNA dilakukan untuk me-mastikan ada tidaknya DNA yang diperoleh sebelum dilakukan proses AFLP.
Pita–pita DNA dari seluruh primer diter-jemahkan ke dalam bentuk data biner yaitu dengan memberi angaka 1 bila terdapat pita dan angka 0 bila tidak terdapat pita. Berdasarkan data biner tersebut, jumlah seluruh pita dihitung. Ukuran pita AFLP yang diperoleh berkisar antara 50-700 pb dengan berbagai ukuran menunjukkan bahwa proses digesti dengan enzim restriksi, ligasi adapter, preamplifikasi dan amplifikasi selektif telah berhasil dilakukan.
Teknik AFLP dapat mendeteksi polimor-fisme pada sampel jambu mete dengan
mengana-460 bp 350 bp 100 bp 145 bp 225 bp 50 bp 565 bp
lisis seluruh genom. Menurut Omoto dan Lurquin (2004), polimorfisme yang dihasilkan menunjukkan adanya perbedaan letak marka AFLP (urutan basa pengenalan enzim restriksi EcoRI dan MseI dan primer selektif) sehingga dapat diperoleh perbeda-an informasi genetik pada setiap sampel. Perbe-daan ukuran fragmen DNA menghasilkan suatu pola pita DNA tertentu. Teknik AFLP diawali dengan memotong genom jambu mete dengan enzimEcoRI danMseI. Jambu mete termasuk organisme euka-riot yang memiliki basa adenin dan timin yang lebih tinggi daripada basa guanin dan sitosin. Analisis AFLP dengan enzimEcoRI danMseI bertujuan agar polimorfisme yang didapatkan lebih rinci karena komposisi basa adenin dan timin pada kedua enzim (EcoRI danMseI) lebih tinggi daripada basa guanin dan sitosin (Voset al., 1995).
Fragmen hasil digesti kemudian diligasi dengan adapter secara simultan. Adapter merupa-kan DNA untai ganda yang memiliki panjang sekitar 20 pb. Terdapat dua jenis adapter yaitu adapter ujung pemotongan EcoRI dengan kelebihan basa AATT pada ujung 5’ dan ujung pemotongan MseI dengan kelebihan basa TA pada ujung 5’. Proses ligasi dilakukan dengan bantuan enzim T4 DNA ligase yaitu membentuk ikatan fosfodiester antara ujung 5’ (ujung fosfat) dan ujung 3’ (ujung OH) pada untai DNA (Struhl, 1993). Adapter berfungsi menya-makan dua ujung pragmen hasil digesti dan sebagai tempat menempelnya primer untuk proses amplifi-kasi selanjutnya. Hasil positif ligasi dapat dilihat pada akhir proses AFLP karena urutan basa pada primer sehingga bila proses ligasi gagal maka proses amplifikasi tidak akan berjalan (Saunders et al., 2001).
Fragmen hasil ligasi kemudian diamplifikasi dengan teknik PCR. Amplifikasi pada AFLP dibagi menjadi dua tahap yaitu preamplifikasi dan ampli-fikasi selektif. Preampliampli-fikasi dilakukan dengan pri-mer selektif yaitu P11 - 700, M48, M49, M50, dan M51.
Menurut Voset al. (1995), preamplifikasi bertujuan mengurangi kompleksitas fragmen hasil digesti, sehingga tidak terjadi kesalahan penempe-lan primer pada amplifikasi selektif dan mengurangi hasil pita smear pada elektroforesis gel poliakri-lamid.
Amplifikasi selektif dilakukan dengan pri-mer selektif yang memiliki tambahan 3 basa pada ujung 3’. Primer selektif adalah primer yang ber-fungsi menyeleksi fragmen hasil digesti dengan adanya basa–basa selektif pada ujung 3’. Kombinasi primer digunakan untuk melihat polimorfisme (pola pita) secara lengkap. Masing–masing primer memi-liki komposisi basa–basa primer selektif. Basa-basa selektif pada primer melekat pada fragmen hasil
digesti yang memiliki basa–basa berkomplemen. Perbedaan basa–basa selektif pada setiap primer mengakibatkan perbedaan amplifikasi fragmen, sehingga menghasilkan perbedaan pita berdasar-kan ukuran pita yang dihasilberdasar-kan dari masing–masing sampel. Perbedaan ukuran pita menggambarkan polimorfisme dari sampel jambu mete (Saunderset al., 2001).
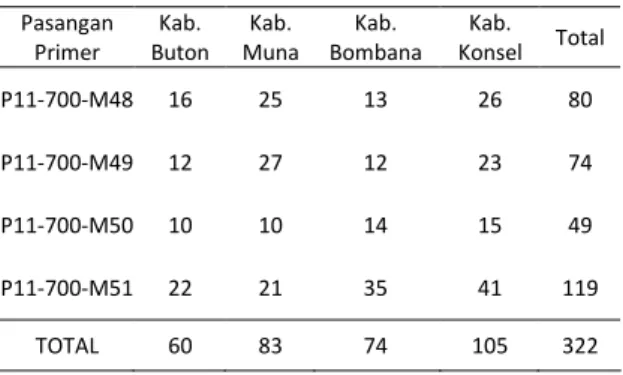
Hasil pita yang diperoleh dari empat kom-binasi primer dan empat sampel adalah sebanyak 322 pita (Tabel 1).
Tabel 1. Jumlah pita DNA berdasarkan sampel dan primer Pasangan
Primer ButonKab. MunaKab. BombanaKab. KonselKab. Total
P11-700-M48 16 25 13 26 80
P11-700-M49 12 27 12 23 74
P11-700-M50 10 10 14 15 49
P11-700-M51 22 21 35 41 119
TOTAL 60 83 74 105 322
Berdasarkan lokasi pita pada sampel jambu mete maka pita–pita AFLP dapat dibagi menjadi dua, yaitu pita umum (pita yang terdapat pada setiap sampel) dan pita polimorfis (pita yang hanya terdapat pada satu atau beberapa sampel). Poli-morfisme ditandai dengan ada dan tidak adanya pita pada suatu sampel yang disebabkan oleh per-bedaan ukuran pita yang dihasilkan oleh setiap sampel (Wang et al. 2003). Berdasarkan hasil pola pita–pita AFLP dapat disimpulkan bahwa terdapat polimorfisme pada 4 sampel jambu mete. Pita poli-morfisme AFLP yang dihasilkan tidak dapat diten-tukan sebagai alel atau lokus tertentu karena ana-lisis dilakukan pada seluruh genom. Pita polimor-fisme yang didapatkan tidak dapat dijadikan dasar perbandingan karakter fenotip secara langsung (Mba dan Tohme, 2005)
Primer selektif digunakan untuk menye-leksi fragmen berdasarkan komposisi basa–basa pada primer. Perbedaan komposisi basa pada pri-mer menghasilkan perbedaan fragmen yang teram-plifikasi. Perbedaan fragmen tersebut kemudian di-bedakan berdasarkan ukuran. Menurut Loh et al.
(2000), setiap kombinasi primer mampu menghasil-kan pola pita yang spesifik untuk setiap sampel sehingga dapat digunakan sebagai identitas sampel. Perbedaan pola pita dapat menggambarkan perbe-daan genetik pada setiap sampel jambu mete.
Perbedaan pola pita dapat ditunjukkan dalam perbedaan jumlah pita yang dihasilkan.
Jumlah pita yang dihasilkan oleh setiap 4 kombinasi primer berkisar antara 49–119 pita. Jumlah pita yang terbanyak dihasilkan oleh pasangan primer P11-700 dan M-51 yaitu 119 pita, sedangkan jumlah pita terendah dihasilkan oleh pasangan pri-mer P11-700 dan M-50 yaitu 49 pita. Berdasarkan jumlah pita yang diperoleh dapat disimpulkan bahwa frekuensi kombinasi primer P11-700 dan M-51 pada genom jambu mete lebih tinggi daripada kombinasi primer P11-700 dan M-50.
Menurut Restrepo et al. (1999), komposisi basa selektif pada primer akan mempengaruhi jumlah pita DNA hasil AFLP. Primer dengan basa sitosin atau guanin yang lebih banyak menghasilkan jumlah pita yang lebih sedikit daripada primer dengan komposisi basa adenin dan timin yang lebih banyak karena basa sitosin dan guanin lebih selektif dalam mengamplifikasi fragmen.
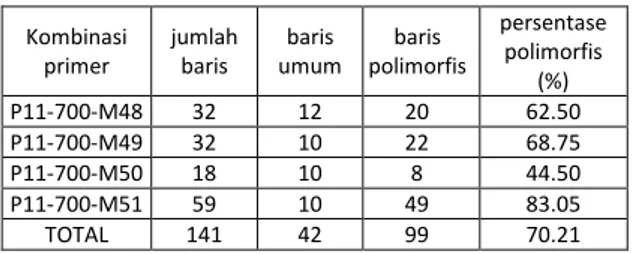
Jumlah seluruh baris pita–pita AFLP 4 kom-binasi primer adalah 141 baris. Dari seluruh baris tersebut terdapat 42 baris yang mengandung pita umum sedangkan baris pita polimorfis sebanyak 99 baris. Jumlah baris yang mengandung pita poli-morfis dihitung dan diperoleh kisaran persentase primer adalah 44,50–83,05%. Rata–rata persentase baris polimorfis seluruh pasangan primer adalah 70,21%. Persentase polimorfisme diperoleh berda-sarkan perbandingan jumlah baris yang mengan-dung pita polimorfis dengan jumlah baris yang mengandung pita umum kemudian dikalikan 100 %. Jumlah baris, baris umum, baris polimorfis dan per-sentase polimorfis dari 4 kombinasi primer dapat dilihat pada Tabel 2.
Tabel 2. Jumlah baris, baris yang umum, baris polimorfis dan persentase baris polimorfis dari 4 kombinasi primer
Kombinasi
primer jumlahbaris umumbaris polimorfisbaris
persentase polimorfis (%) P11-700-M48 32 12 20 62.50 P11-700-M49 32 10 22 68.75 P11-700-M50 18 10 8 44.50 P11-700-M51 59 10 49 83.05 TOTAL 141 42 99 70.21
Berdasarkan penelitian Chen et al. (2004), diperoleh persentase polimorfisme sebesar 70 % dari 30 kultivar Aglaonema dengan 6 pasang pri-mer. Hasil penelitian Chen et al. tersebut menun-jukkan bahwa ke 6 pasang primer yang digunakan dapat menggambarkan perbedaan genetik dengan persentase polimorfisme 70%. Berdasarkan hasil tersebut, Chenet al. berhasil membedakan secara identik masing–masing kultivarAglaonema.
Berdasarkan hasil rata–rata persentase polimorfisme jambu mete yang diperoleh yakni
sebesar 70,21%, maka dapat disimpulkan bahwa jambu mete dari setiap sampel dapat dibedakan secara genetik.
Penelitian dengan menggunakan marka AFLP untuk mendeteksi polimorfisme pada bebe-rapa spesies tanaman telah dilakukan. Aggarwalet al, mengidentifikasi 501 pita dari Oryza sativa L. dengan persentase polimorfisme 65%. Singh et al. menghasilkan 422 pita dari Azadiracht indica
dengan persentase polimorfisme 70%. Tomkins et al,menghasilkan persentase 75% padaHemerocallis spp.
Menurut Vergara dan Bughara (2003), tingginya tingkat polimorfisme menunjukkan tinggi-nya keragaman genetik. Oleh karena itu, hasil per-sentase polimorfisme empat sampel jambu mete yang diperoleh memiliki keragaman genetik yang tinggi. Keragaman genetik tersebut dapat digambar-kan dengan tingginya persentase polimorfisme.
Keunikan genetik jambu mete kemung-kinan disebabkan oleh mutasi DNA dan rekombi-nasi. Mutasi DNA dan rekombinasi secara seksual merupakan faktor utama terjadinya variasi genetik (Indrawanet al., 2007). Teknik AFLP tidak mengana-lisis gen–gen tertentu, tetapi hanya menganamengana-lisis seluruh genom jambu mete melalui pola pita yang terbentuk sehingga keunikan genetik yang diper-oleh tidak dapat langsung diterjemahkan ke dalam karakter fenotip. Menurut Griffiths et al. (2000), karakter fenotip dipengaruhi oleh faktor genetik dan lingkungan. Oleh karena itu, data variasi gene-tik jambu mete yang telah dilakukan dengan marka AFLP perlu dibandingkan dengan analisis faktor ling-kungan sehingga dapat digunakan untuk mengana-lisis karakter fenotip jambu mete.
Elektroforesis gel poliakrimida pada peneli-tian bertujuan memisahkan fragmen hasil amplifi-kasi selektif. Teknik AFLP menggunakan elektro-foresis gel poliakrilamida agar pita DNA dengan perbedaan satu basa pada setiap sampel dapat dianalisis. Gel poliakrilamid memiliki pori–pori yang lebih kecil dari pada sel agarosa sehingga dapat memisahkan pita DNA yang berukuran relatif kecil (5-500 pb) dan mampu memisahkan fragmen dengan perbedaan satu basa (Sambrook dan Russell, 2001). Pita–pita DNA dari seluruh primer diterjemahkan ke dalam bentuk data biner yaitu dengan memberi angka 1 bila terdapat pita dan angka 0 bila tidak terdapat pita. Data biner kemudian diolah dengan metode SPSS, sehingga didapatkan Proximity Matrix dan dendrogram seperti pada Tabel 3.
Dendogram merupakan topologi pohon filogenetik yang menggambarkan percabangan dan pengelompokkan (clustering) sampel yang berderet rata secara vertikal pada satu sisi pohon.
Berdasar-kan dendogram, maka dapat diketahui pola perca-bangan dengan pengelompokkan sampel berdasar-kan marka molekular AFLP. Software SPSS meng-analisis data biner menjadi dendogram dengan menggunakan perhitungan metode Complete Linkage.
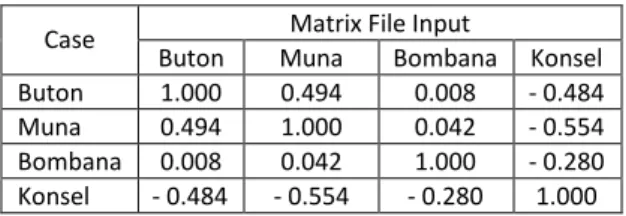
Tabel 3. Hasil Proximity Matrix dan dendogram jambu mete pada 4 lokasi berbeda
Matrix File Input Case
Buton Muna Bombana Konsel
Buton 1.000 0.494 0.008 - 0.484
Muna 0.494 1.000 0.042 - 0.554
Bombana 0.008 0.042 1.000 - 0.280
Konsel - 0.484 - 0.554 - 0.280 1.000
Hasil dendogram menunjukkan bahwa dari empat sampel jambu mete, terdapat dua kelompok yaitu kelompok A yang terdiri atas jambu mete asal Kab. Buton, Kab. Muna dan Kab. bombana sedang-kan kelompok B terdiri atas jambu mete asal Kab. Konsel. Pada kelompok A jambu mete asal Kab. Bombana berdiri sendiri dan dibedakan dengan jambu mete asal Kab. Muna dan Kab. Buton.
Pengelompokan sampel jambu mete ber-dasarkan marka AFLP menunjukkan perbedaan dan kemiripan genetik antar sampel. Perbedaan dan kemiripan genetik tersebut dihasilkan berdasarkan pola pita DNA (Mueller dan Wolfenbarger, 1999). Sampel yang tergolong dalam satu kelompok memi-liki pola pita yang mirip seperti pada jambu mete asal Kab. Muna, Kab. Buton dan Kab. Bombana, namun jambu mete asal Kab. Muna dan Kab. Buton memiliki kemiripan yang lebih dekat dibandingkan dengan Kab. Bombana.
Berdasarkan letak geografis, bahwa jambu mete asal Kab. Muna dan Kab. Buton berada pada wilayah kepulauan sedangkan jambu mete asal Kab. Bombana dan Kab. Konsel berada pada wilayah daratan. Adanya kemiripan genetik antara jambu mete asal Kab. Muna dan Kab. Buton mungkin disebabkan karena keduanya berada pada wilayah kepulauan yang saling berdekatan. Pada penelitian ini, jambu mete asal Kab. Konsel yang digunakan adalah jambu mete unggul yang berasal dari Kebun Penelitian Onembute. Sifat unggul yang dimiliki oleh jambu mete asal Kab. Konsel menyebabkan
jambu mete asal Kab. Konsel memiliki kelompok tersendiri dengan ketiga jambu mete lainnya. Adapun sifat unggul tersebut seperti gelendong berukuran panjang 2,5–3,0 cm dan lebar 2,0–2,25 cm, gelondong berbobot antara 7 g–10 gram per-butir dan rendemen kacang mete berkisar 22 %.
Jambu mete asal Kab. Bombana yang masuk satu kelompok dengan sampel Kab. Muna dan Kab. Buton hal ini mungkin disebabkan karena wilayah Kab. Bombana dulunya masuk dalam wilayah administratif/kecamatan dari Kab. Buton sehingga ada kemungkinan bahwa jambu mete yang ada di wilayah Kab. Bombana berasal dari bibit jambu mete yang berasal dari Kab. Buton.
Sebagai kesimpulan dari penelitian ini bahwa: (1) Terdapat variasi genetik pada empat asal sampel jambu mete yang digambarkan melalui poli-morfisme dengan menggunakan marka molekuler AFLP, (2) Polimorfisme dapat dilihat pada perbedaan pola pita AFLP dengan jumlah pita secara keseluruhan 322 pita dan rata-rata persentase polimorfisme sebesar 70,21% dari empat kombinasi primer selektif, dan (3) Hasil dendrogram menunjukkan bahwa terdapat dua kelompok yaitu kelompok pertama terdiri dari jambu mete asal Kab. Buton, Kab. Muna dan Kab. Bombana dan kelompok kedua yaitu jambu mete asal Kab. Konsel. Jambu mete asal Kab. Konsel memiliki perbe-daan dengan ketiga sampel jambu mete lainnya.
KEPUSTAKAAN
BPMD Provinsi Sulawesi Tenggara, 2010. Buku
po-tensi dan peluang investasi di daerah Sula-wesi Tenggara. BPS Sultra.
Chen, J.J., P.S. Devanand, D.J. Norman, R.J. Henny and C.T. Chao. 2004. Genetic relationship of Aglaonema species and cultivars infer-red from AFLP Markers. Annals of Botany 93: 157-166.
Correa, R.X., Ricardo V. A., Fabio G. F. Cosme D. C., Maurilio A. M., and Everaldo G. B. 1999.
Genetic distance in soybean based on RAPD Markers.
Griffiths, A.J.F., J.H. Miller, D.T. Suzuki, R.C. Lewontin and W.M. Gelbrat. 2000. An introduction to genetic analysis.7th ed.
W.H. Freeman, New York: xvii + 860 hlm. Hoon-Lim S, Peng Teng P. C., Lee Y. H., and Goh C. J.
1999.RAPD analysis of some species in the genus vanda (Orchidaceae). Annuals of Botany.
Indrawan, M., R.B. Primack dan J. Supriatna, 2007.
Biologi konservasi. rev. ed. Yayasan Obor Indonesia. Jakarta.
Kemal, P., 2000. Jambu mete. SIM Pembangunan Perdesaan. Bappenas. Jakarta.
Loh, J.P., R. Kiew, A. Kee, L.H. Gan and Y.Y. Gan. 1999.Amplified fragment length polymor-phism (AFLP) provides molecular markers for the identification of caladium bicolor cultivars. Annals of Botany 84: 155–161. Mba, C. dan J. Tohme, 2005.Use of AFLP markers in
surveys of plant diversity. Methods in enzy-mology.395: 177–201.
Moore, D.D. and D. Dowhan. 2002. Preparation and analysis of DNA.Dalam: Aususbel, F.M., R. Brent, R.E. Kingston, D.D Moore, J.G. Seidman, J.A. Smith dan K. Struhl, 1995.
Current protocol in molecular biology. Vol I. John Wiley & Sons, Inc.: xxiv + 9.17.3 hlm Mueller, U.G. dan L.L. Wolfenbarger, 1999. AFLP
genotyping and fingerprinting. Elsevier Science 14: 389-394
Restrepo, S., M. Duque, J. Tohme and V. Verdier, 1999. AFLP fingerprinting: an efficient technique for detecting genetic variation of Xanthomonas axonopodis pv. Manihotis. Microbiology 145: 107–114.
Sambrook, J. dan D.W. Russell, 2001. Molecular cloning: A laboratory Manual. 3rded. CSHL
Press, New York : xxvii + 18.136 + A.14.1 + R.22 + I44 hlm.
Saunders, J.A., S. Mischke, dan A.A. Hemeida, 2001.
The use of AFLP techniques for DNA finger-printing in plants. Beckman Coulter, Inc., Fullerton. 9 hlm.
Van Eijnatten, C.L.M., 1991.Anacardium occidentale
L. (http://proseanet.org/prosea.eprosea_ detail.php?frt=&id=1468) diakses tanggal 10 September 2011.
Vergara, G.V and S.S. Bughara, 2003.AFLP analyses of genetic diversity in bentgrass. Crop Science, 43: 2162–2171.
Wang, Z.S., A.J. Baker, G.E. Hill and S.V. Edwards, 2003. Reconciling actual and inferred population histories in the house finch (Carpodacus mexicanus) by AFLP analysis. Evolution57(12): 2852-2864.
Weising, K., Nybom, H., Wolff, K. and Meyer, W., 1995. DNA fingerprinting in plant and fungi.CRC Press. Boca Raton: 322 hlm. Zaubir, R. dan R. Suryadi, 2003.Kriteria kesesuaian
tanah dan iklim tanaman jambu mete. Litbang Jakarta.