i
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2010
POPULASI SAPI LOKAL INDONESIA BERDASARKAN
PENCIRI MOLEKULER DNA MIKROSATELIT KROMOSOM Y
DAN GEN CYTOCHROME B
ii
Dengan ini saya menyatakan bahwa disertasi berjudul Variasi Genetik dan Hubungan Filogenetik Populasi Sapi Lokal Indonesia Berdasarkan Penciri Molekuler DNA Mikrosatelit Kromosom Y dan Gen Cytochrome b, adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Bogor, 20 Januari 2010
iii
ARIS WINAYA. The Genetic Variations and Phylogenetic Relationship Between Indonesian Native Cattle Population Based on Molecular Marker of Y-Chromosome Mcrosatellite DNA and Cytochrome b Gene. Under direction of MULADNO, R. EDDIE GURNADI, and ASEP SAEFUDDIN.
Seven Y-chromosome specific microsatellites and cytochrome b (cyt b) of mitochondrial DNA (mtDNA) markers were assayed to get the genetic variation of 7 breeds of Indonesian native cattle, primary on male cattle. The analysis of Y-chromosome microsatellites variation was determinate from PCR products by using 7 primer pairs that flanking those microsatellites (INRA008, INRA057, INRA062, INRA124, INRA126, DYS 199, INRA 189). PCR products were separated on 10 % of polyacrylamide gel electrophoresis (PAGE), and then silver staining method was used to detect allele polymorphism at each locus by manually. The analysis of cyt b gene variation was carried out by analysis of nucleotide cyt b gene sequences based on sequencing results of PCR product from amplification of cyt b gene region of mtDNA genome.
From Y-chromosome microsatellite allelic polymorphism analysis showed only found limited allele (two alleles) by average 1.8. While, from heterozgousity level INRA 062 locus has higher value (h= 0.53) than others that found in Pesisir cattle population, also this locus has higher value of PIC (0.37). Because markers in Y chromosome, so it is a haplotype system. According to FAO, the specific allele must have minimum four distinct alleles per locus for proficient judgment of genetic differences between breeds. So, all locus in this research was not sufficient to categorized polymorphic allele. Nevertheless, locus INRA 062 could be considered in future study to get more information about polymorphism of this marker. Y-chromosome microsatellite in general has tend to specific in breed comparing with autosomal chromosome, because allele come from only male or Y sex chromosome and it contrary to autosomal chromosome where allele is contributed from male and female. The nucleotides variation of cyt b gene showed that in general all population of cattle only has 39.56 % nucleotide conserve side comparing with Banteng (Bos javanicus) mtDNA. These phenomena could be assumed that the Indonesian native cattle has more genetic introgession of Bos indicus and Bos taurus breeds. But, from phylogenetic relationship, Bali cattle (both from Bali and Lombok) and Madura cattle have closer relationship to Banteng. Its means that two breeds are still have more genetic composition of Banteng. For Aceh and Pesisir cattle were have opportunity to increase the genetic potential as a local breeds, where based on this study has been founded any specific nucleotide sequences of cyt b gene to those breeds comparing with Bos taurus or Bos Indicus.
For the next study we need more Y-chromosome microsatellite marker and also more mtDNA gene sequences to discriminate the Indonesian breeds related to tracking the genetic potential. Because the male cattle has roles in genetic spreading where could have an enormous impact on highly selected domestic animal populations and also in genetic conservations.
Key words : Y-chromosome microsatellite, cytochrome b, Bos javanicus, mitochondria
iv
ARIS WINAYA. Variasi Genetik dan Hubungan Filogenetik Populasi Sapi Lokal Indonesia Berdasarkan Penciri Molekuler DNA Mikrosatelit Kromosom Y dan Gen Cytochrome b Dibimbing oleh MULADNO, R. EDDIE GURNADI, dan ASEP SAEFUDDIN.
Karakterisasi genetik sapi lokal Indonesia sebenarnya telah lama dilakukan, dimulai sejak tahun 1974 hingga 1977 dan beberapa hasilnya telah dipublikasikan, diantaranya berdasarkan perbedaan golongan darah, protein, protein darah, enzim dan komposisi asam amino rantai β haemoglobin X. Hasilnya bahwa sapi Bali memiliki spesifikasi adanya alel HbX pada golongan darahnya yang tidak dimiliki sapi lokal lain. Tetapi, studi karakterisasi genetik di tingkat molekuler masih sangat kurang. Adanya pertimbangan perkembangan cepat sejumlah penciri genetik molekuler yang lebih diskriminatif dan akurat dibandingkan fenotipik, maka penggunaan marka ini akan sangat membantu dalam penanganan manajemen sistim seleksi pada ternak sapi potong.
Informasi tentang alel-alel spesifik (breed spesific allele) dari data molekuler sapi lokal Indonesia masih sangat terbatas. Beberapa hasil penelitian terdahulu menunjukkan adanya alel spesifik bangsa untuk sapi Bali pada lokus mikrosatelit INRA 023, HEL9 dan INRA 035 dibandingkan sapi Bos taurus (Simmental, Limousin, dan Brangus). Demikian pula hasil studi berdasarkan panel 16 lokus penciri mikrosatelit memberikan gambaran awal tentang hubungan genetik antara sapi Bali, Madura, PO dan Brangus. Namun hasil-hasil tersebut belum menunjukkan adanya hubungan antara penciri DNA mikrosatelit dengan sifat-sifat ekonomis sapi lokal, terutama terhadap kualitas daging.
Untuk pengukuran variasi genetik pada populasi (misal, breed-breed ternak domestikasi) masih diperlukan penciri mikrosatelit kromosom Y lebih banyak. Hal ini berbeda dengan tikus dan manusia yang telah tersedia banyak data sekuens DNA pada kromosom Y dibandingkan mamalia lain yang masih kurang. Tetapi, beberapa studi terakhir dilaporkan tentang filogenetik daerah kromosom Y pada spesies ternak domestikasi berdasarkan variasi keterpautan bagian spesifik jantan kromosom Y (male specific Y chromosome / MSY) dan mampu membedakan pula antara sapi keturunan Bos indicus dan Bos taurus.
Sampel sapi pada penelitian ini dikhususkan pada individu sapi jantan dengan pertimbangan bahwa penyebaran populasi sapi di Indonesia masih bertumpu pada teknologi Inseminasi Buatan (IB) untuk manajemen budidayanya. Sehingga peran pejantan sebagai sumber sperma menjadi sangat penting. Oleh karena itu, analisis variasi genetik menggunakan penciri molekuler DNA mikrosatelit spesifik kromosom Y dan DNA mitokondria (mtDNA) dari gen cytochrome b (cyt b) pada ternak jantan beberapa bangsa (breed) sapi lokal Indonesia menjadi hal yang penting. Sebagaimana diketahui bahwa penciri mtDNA berbeda dalam konteks segregasi secara genetis, yakni mtDNA diturunkan dari garis maternal (induk), sedangkan mikrosatelit pada kromosom Y melalui garis paternal (pejantan). Sehingga kedua penciri tersebut dapat dijadikan sebagai model pendekatan berbeda dalam segregasi genetiknya. Demikian pula penggunaan sapi jantan dalam studi ini dimaksudkan untuk lebih membuktikan sejauhmana kemungkinan terjadinya variasi di dalam mendapatkan hasil interpretasi dengan penggunaan penciri genetik yang berbeda.
v
yang spesifik untuk setiap populasi sapi tersebut; 3) mengetahui filogenetik populasi sapi lokal yang ada di Indonesia berdasarkan penciri DNA mikrosatelit kromosom Y dan gen cyt b; dan 4) mengetahui konsistensi filogenetik berbasis DNA mikrosatelit dan gen cyt b.
Kegunaan penelitian ini adalah sebagai salah satu landasan dalam menentukan program pemuliaan sapi lokal di Indonesia dan penciri DNA mikrosatelit dan gen cyt b diaplikasikan sebagai Marker Assisted Selection (MAS) atau Marker Pembantu Seleksi pada sifat-sifat produksi atau reproduksi sapi.
Ruang lingkup penelitian meliputi penggunaan sampel DNA yang diekstraksi dari beberapa jenis bangsa sapi yang sudah adapatip maupun endemik hidup di Indonesia, seperti sapi Aceh, Pesisir, Madura, Bali-Bali, Bali-Lombok, PO dan PFH; kemudian analisis DNA inti dan gen cyt b sapi-sapi tersebut; analisis data secara statistik yang meliputi analisis polimorfisme alel-alel mikrosatelit pada kromosom Y dan analisis variasi mutasi nukleotida pada gen cyt b. Perangkat lunak pendukung dalam analisis data diantaranya NTSYS dan MEGA ver. 4.
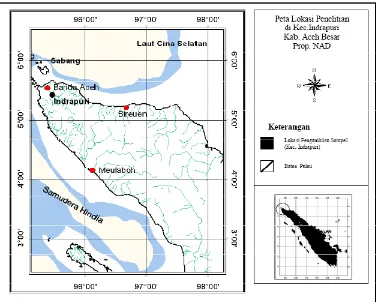
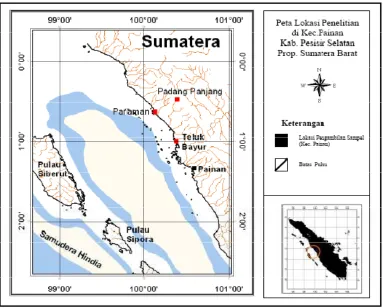
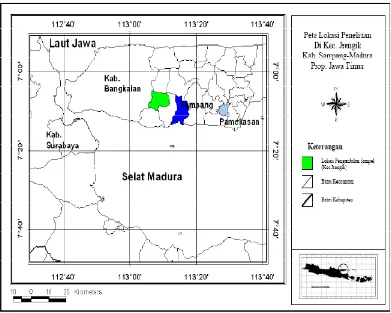
Dalam penelitian ini sampel digunakan sapi-sapi lokal atau adaptip Indonesia yang secara purposif seluruhnya ditentukan individu jantan, yaitu : 1) Sapi Aceh, berasal dari Balai Pembibitan Ternak Unggul (BPTU) sapi Aceh, berlokasi di Kecamatan Indrapuri, Kabupaten Aceh Besar, Propinsi Nangroe Aceh Darussalam (NAD); 2) Sapi Pesisir berasal dari sapi-sapi yang dipelihara pada Unit Pelaksana Teknis (UPT) Balai Pembibitan Ternak dan Hijauan Makanan Ternak (BPT-HMT) Dinas Peternakan Kabupaten Pesisir Selatan, Propinsi Sumatra Barat yang berada di kota Painan; 3) Sapi Madura, berasal dari peternakan rakyat berlokasi di Kecamatan Jrengik, Kabupaten Sampang – Madura, Propinsi Jawa Timur; 4) Sapi Bali di Bali (Bali-Bali), berasal dari peternakan rakyat berlokasi di Kecamatan Baturiti, Kabupaten Tabanan, Propinsi Bali; 5) Sapi Bali di Lombok (Bali-Lombok), berasal dari peternakan rakyat berlokasi di Kecamatan Lingsar dan Gunungsari, Kabupaten Lombok Barat, Propinsi Nusa Tenggara Barat; 6) Sapi Peranakan Ongole (PO), berasal dari peternakan rakyat berlokasi di Kecamatan Wongsorejo, Kabupaten Banyuwangi, Propinsi Jawa Timur; dan 7) Sapi Peranakan Fries Holland (PFH), berasal dari peternakan rakyat anggota KUD Sapi Perah KOPSAE di Kecamatan Pujon, Kabupaten Malang, Propinsi Jawa Timur.
Untuk pengambilan sampel darah, sel darah total sejumlah 10 mL diambil dari vena jugularis sapi, kemudian diawetkan dalam 10 % larutan EDTA yang dikoleksi pada beberapa lokasi dimana terdapat sapi-sapi lokal tersebut, yang selanjutnya diekstraksi dengan metode standart fenol-kloroform untuk digunakan sebagai sumber DNA genom.
vi
mikrosatelit, sedangkan variasi nukleotida gen cyt b pada mtDNA diperoleh dari hasil sekuensing daerah gen cyt b. Untuk menentukan jarak genetik (filogenetik) antara sapi-sapi lokal berdasarkan alel-alel DNA mikrosatelit dan variasi sekuens nukleotida gen cyt b ditetapkan dengan Metode Unweighted Pair-Group Method with Arithmetic Mean (UPGMA) (Sneath & Sokal 1973). Sedangkan konstruksi pohon genetik digunakan perangkat lunak NTSYS dan MEGA ( Molecular Evolutionary Genetics Analysis) version 4.0.
Dari variabel vital statistik, maka sapi PFH memiliki volume scrotum terbesar (+1.032 cc) dibandingkan bangsa sapi lainnya. Hal ini berarti bahwa sapi PFH memiliki kapasitas produksi sperma lebih banyak. Sebagaimana diketahui sapi PFH merupakan bangsa sapi keturunan spesies B. taurus, yang apabila dibandingkan dengan bangsa sapi Asia (B. indicus) secara umum karakteristik fenotipiknya lebih unggul, baik pada sapi perah maupun sapi potong. Jumlah alel secara keseluruhan rendah, tertinggi hanya 2 alel, sedangkan jumlah rata-rata alel pada seluruh populasi (tujuh bangsa sapi) adalah 1.8. Nilai heterosigositas (h) ke tujuh populasi adalah antara 0 % hingga 53 % dan nilai tertinggi h ditemukan pada lokus mikrosatelit INRA 062 (53 %) di populasi sapi Pesisir. Adanya fenomena sapi Pesisir lebih tinggi heterosigostasnya dibandingkan dengan sapi-sapi lainnya menunjukkan bahwa variasi genetik sapi-sapi Pesisir masih lebih tinggi dibandingkan sapi-sapi lainnya. Demikian pula lokus INRA 062 yang memiliki nilai PIC (37%) lebih tinggi dibanding lokus lain, maka perlu dilakukan studi lanjut sejenis agar nantinya dapat ditemukan alel-alel lebih banyak sehingga dapat pula ditemukan marker yang spesifik dan karena mikrosatelit Y merupakan sistem haploid, maka alel yang spesifik dapat pula merupakan haplogrup spesifik bagi penciri ini.
Menurut petunjuk FAO untuk justfikasi penilaian perbedaan atau variasi genetik antar breed minimum harus terdapat empat alel yang berbeda per lokus, sehingga dalam studi ini tidak ada satu lokuspun yang memenuhi kriteria tersebut. Jadi, lokus-lokus yang digunakan dalam studi ini belum dapat dikategorikan sebagai lokus polimorfik untuk uji evaluasi variasi genetik antar bangsa sapi lokal Indonesia. Untuk itu dapat dilakukan studi lanjut sejenis namun dengan menggunakan jumlah sampel yang memadai dan letak geografis yang lebih luas. Gambaran rata-rata alel yang rendah (1.8) dapat diasumsikan karena pola penyebaran sapi jantan kurang mencukupi untuk pejantan sapi betina yang ada, atau jumlah sampel yang masih belum optimal mewakili populasi yang ada serta diversitas letak geografis yang sangat jauh, sehingga memungkinkan terjadinya keragaman genetik akibat proses seleksi. Untuk lokus lain, yang pada studi ini memiliki nilai h diatas 50% selain lokus INRA 062, misal lokus INRA 124 (h=50 % dan 52%) pada sapi Bali dan PO, dapat dipertimbangkan pula untuk diuji kembali pada populasi sapi lokal Indonesia dengan populasi yang berbeda dan jumlah individu lebih banyak.
vii
Sisi variabel dan konservatif dari parsial gen cyt b ditetapkan dengan cara alignment (pensejajaran) terhadap sekuen konsensus (consensus sequence) dari sekeuens gen cyt b Banteng (Bos javanicus) yang diperoleh dari Genebank dengan nomor aksesi (accession number) NC_012706.1. Dalam penelitian ini panjang sekuens parsial gen cyt b adalah 230 bp, mulai posisi basa 14691 hingga 14920 dari sekuen komplit gen cyt b. Setiap perbedaan sekuen pada urutan basa tertentu berdasarkan sekuen konsensus (consensus sequence), maka merupakan sisi variabel dalam sekuen parsial. Dari hasil pensejajran (aligment) ditemukan bahwa sisi variabel lebih banyak yakni 139 buah (60.44 %) dibandingkan sisi konservatifnya yakni 91 buah (39.56 %). Hal ini menunjukkan bahwa dari gen cyt b sapi-sapi lokal di Indonesia hampir 60.44 % bukan berasal dari garis maternal induk Banteng (berdasarkan konsensus sekuen cyt b dari B. javanicus Genebank nomor akses NC_012706.1). Tetapi secara umum hasil penelitian ini menunjukkan bahwa garis maternal Banteng masih ditemukan meskipun mengalami penurunan jumlah sisi konservatifnya.
Hubungan genetik berdasarkan variasi sekuens cyt b menunjukkan sebagian besar sapi Aceh, Pesisir dan Madura berdekatan jaraknya dengan B. taurus dan B. indicus, sedangkan sebagian besar sapi Bali-Bali dan Bali-Lombok lebih berdekatan jaraknya dengan Banteng (B. javanicus). Hasil ini sesuai beberapa hasil studi sebelumnya bahwa sapi Madura dan Bali masih memiliki kedekatan genetik dengan Banteng. Sehingga hasil penelitian ini menunjukkan hal yang sama dengan hasil-hasil penelitian sebelumnya dengan menggunakan mtDNA, baik di daerah kontrol (CR) maupun cyt b.
Hasil penelitian ini juga membuktikan adanya aliran genetik sapi Bos taurus
maupun Bos indicus pada sapi-sapi lokal Indonesia, termasuk sapi Bali dan Madura sebagai sapi keturunan Banteng (Bos javanicus), yang merupakan salah satu tetua bangsa-bangsa sapi di dunia saat ini. Sapi Bali dan Madura masih memiliki proporsi genetik terbesar dari Banteng, maka kedua sapi ini masih dapat diakui eksistensinya sebagai sapi khas Indonesia. Bagi sapi Aceh dan Pesisir masih memiliki harapan untuk lebih ditingkatkan eksistensi sebagai sapi khas daerah tersebut berdasarkan titik mutasi tertentu pada gen cyt b, meskipun kedekatan genetik lebih ke arah Bos taurus dan Bos indicus.
Kelemahan penggunaan gen cyt b karena dugaan adanya pseudogen (gen mirip mtDNA dalam gen inti) namun dapat dihindari dengan sampel DNA yang memadai sebagai templat apabila digunakan primer universal. Sumber mtDNA sebaiknya jaringan aktif, misal otot skeletal atau organ-organ aktif lain seperti ginjal dan jantung. Sebab jumlah mtDNA jaringan darah tidak sebanyak DNA di jaringan aktif. Hal ini penting untuk meningkatkan akurasi hasil PCR.
Untuk meningkatkan eksistensi spesifikasi sapi-sapi lokal Indonesia, maka kegiatan penelitian yang berbasis molekuler harus diperbanyak. Sebab, hingga saat ini teknologi ini yang masih diakui dunia sebagai teknologi yang tepat untuk identifikasi genetik. Sehingga, di masa depan sapi-sapi lokal dapat diakui konservasi genetiknya secara internasional dengan data yang memadai. Termasuk dalam rangka pelestarian plasma nuftah tetua sapi asli Indonesia (Banteng) yang jumlahnya semakin berkurang akibat tekanan alam maupun manusia.
viii
© Hak Cipta milik IPB, tahun 2010
Hak Cipta Dilindungi Undang-undang
1. Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumber. a. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah. b. Pengutipan tersebut tidak merugikan kepentingan yang wajar IPB
ix
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2010
Disertasi
Sebagai salah satu syarat untuk memperoleh gelar
Doktor pada
Program Studi Ilmu Ternak
POPULASI SAPI LOKAL INDONESIA BERDASARKAN
PENCIRI MOLEKULER DNA MIKROSATELIT KROMOSOM Y
DAN GEN CYTOCHROME B
x
Kromosom Y dan Gen Cytochrome b Nama : Aris Winaya
NIM : D061050021
Disetujui Komisi Pembimbing
Prof. Dr. Muladno, MSA. Ketua
Prof. Dr. R. Eddie Gurnadi, M.Sc. Dr. Asep Saefuddin, M.Sc.
Anggota Anggota
Diketahui
Ketua Program Studi/Mayor Dekan Sekolah Pascasarjana
Dr. Ir. Rarah R.A. Maheswari, DEA Prof. Dr. Ir. Khairil A. Notodipuro, M.S.
xi
Penguji pada Ujian Tertutup : Prof. Dr. Cece Sumantri, M.Agr.Sc. Prof. (R). Dr. Kusuma Diwyanto, M.Sc. Penguji pada Ujian Terbuka : Prof. (R). Dr. Tjeppy D. Soedjana, M.Sc.
xii
Dengan hati yang tulus dan penuh rasa syukur kupersembahkan karya ini :
Kepada Mu Ya Allah SWT sebagai wujud syukur atas ilmu yang telah engkau limpahkan
Kepada kedua orang tua yang selalu memberikan motivasi, nasehat, kasih sayang serta do’a yang tiada hentinya
Kepada isteri dan anakku tercinta “Maftuchah” dan “Muhammad Syauqi Al Maghfirah” atas pengertian, perhatian, pengorbanan dan do’anya dalam mendukung suami dan bapaknya untuk menyelesaikan pendidikan S3 ini.
Kepada bangsa, negara dan agamaku
Kalamullah
“ Dan Dia telah menciptakan binatang ternak untuk kamu, padanya ada (bulu) yang menghangatkan dan berbagai-bagai manfaat, dan sebahagiannya kamu makan “ [An-Nahl (16) : 05].
xiii
Puji syukur kehadirat Allah SWT atas segala rahmat, taufiq, hidayah, serta inayah-Nya sehingga penulis dapat menyelesaikan penulisan disertasi ini dengan judul
“Variasi Genetik dan Hubungan Filogenetik Populasi Sapi Lokal Indonesia Berdasarkan Penciri Molekuler DNA Mikrosatelit Kromosom Y dan Gen
Cytochrome b ”, yang merupakan salah satu syarat penyelesaian Pendidikan Program Doktoral (S3) pada Program Studi Ilmu Ternak, Sekolah Pascasarjana Institut Pertanian Bogor. Shalawat dan salam semoga tercurahkan kepada Nabi Muhammad SAW, keluarga, para sahabat dan para pengikut yang diridhoi-Nya.
Penulis menyadari bahwa dalam penyelesaian penulisan disertasi ini adalah atas sumbangsih materi maupun pemikiran dari berbagai pihak. Oleh karena itu, dalam kesempatan ini penulis ingin menghaturkan terimakasih yang sebesar-besarnya dan penghargaan yang setinggi-tingginya kepada semua pihak yang telah membantu penulis, terutama kepada Yang Terhormat :
1. Bapak Prof. Dr. Muladno, MSA; Prof. Dr. R. Eddie Gurnadi, M.Sc., dan Dr. Asep Saefuddin, M.Sc., selaku Ketua dan Anggota Komisi Pembimbing yang telah memberikan bimbingan, arahan, motivasi dan saran selama ini, sejak penyusunan proposal hingga penyelesaian disertasi ini.
2. Ketua dan Sekretaris Program Studi Ilmu Ternak serta Koordinator Mayor ITP atas bantuan, perhatian dan motivasinya yang diberikan selama mengikuti pendidikan di PTK-IPB sehingga penulis dapat mengikuti pendidikan S3 dengan baik.
3. Rektor dan Dekan Sekolah Pascasarjana Institut Pertanian Bogor yang telah memberikan kesempatan kepada penulis untuk melanjutkan pendidikan S3 di Institut Pertanian Bogor.
4. Rektor dan Pembantu Rektor Universitas Muhammadiyah Malang yang telah memberikan ijin kepada penulis untuk melanjutkan pendidikan S3 di Institut Pertanian Bogor. Demikian pula kepada seluruh kolega di Fakultas Pertanian dan Peternakan Universitas Muhammadiyah Malang atas segala dukungan, motivasi, bantuan dan do’anya yang diberikan kepada penulis selama mengikuti pendidikan di IPB.
5. Para Kepala Dinas Peternakan terkait dengan pengambilan sampel penelitian, yakni Propinsi Nangroe Aceh Darussalam, Kabupaten Pesisir Selatan-Sumbar, Kabupaten Sampang-Madura, Kabupaten Tabanan-Bali, Kabupaten Lombok Barat, Kabupaten Malang, dan Kabupaten Banyuwangi, atas ijin yang diberikan kepada penulis untuk mengadakan penelitian dalam wilayahnya.
xiv
SPt, MP, dari Dinas Peternakan Propinsi NAD dan Ir. Achmadi, Kepala BPTU Sapi Aceh atas bantuan pendampingan koleksi sampel sapi Aceh; dan Ir. Suprio Guntoro atas bantuan pendampingan sampel sapi Bali-Bali dan diskusi mendalam tentang konsep peternakan organik. Juga rekan Dr.agr. M. Amin, dari FMIPA Universitas Negeri Malang atas bantuan analisis data menggunakan software MEGA dan juga diskusi mendalam tentang genetika molekuler hewan; sahabat Dr. Ir. Bram Brahmantiyo, M.Si dan Dr. Ir. Henny Nuraini, M.Si, atas dukungan moral maupun materi selama penulis tinggal di Bogor menempuh studi S-3; rekan Ir. J.L. Wahono, MT dan Hariyadi, S.Pi, M.Si. atas bantuan pembuatan peta digital wilayah penelitian serta sahabat Ir. Mulyoto Pangestu, MSc. PhD., Monash University Australia atas bantuan jurnal-jurnalnya.
7. Seluruh rekan-rekan di Program Studi Ilmu Ternak, terutama saat penulis menjadi Ketua Himpunan Mahasiswa Pascasarjana (HIWACANA) PTK periode 2005-2007 atas kebersamaannya dalam belajar dan berdiskusi selama mengikuti pendidikan di IPB.
8. Sembah sujud kepada kedua orang tua tercinta, Ayah Atang Soewarno (alm.) dan Ibu Ruth Sularsihati, yang telah membesarkan, mendidik, dan do’a-nya yang tiada putusnya untuk keberhasilan putranya. Demikian pula kepada mertua (alm.) atas pengertian, perhatian dan motivasinya selama penulis menempuh studi. Kepada saudara-saudara saya : Rr. Ratna Arum Widyati, Rr. Ari Widyarini, Rr. Ambar Widyasari dan R. Adi Wianjri Nugroho serta seluruh keluarga yang tidak dapat kami sebutkan satu persatu, atas bantuan dan do’a-nya selama ini.
9. Secara khusus kepada isteri tercinta “ Dr. Ir. Maftuchah, MP. “ dan anakku tersayang “ Muhammad Syauqi Al Maghfirah “, atas pengertian, perhatian dan pengorbanannya yang tulus dan semangat serta do’a-nya yang tiada putusnya diberikan kepada penulis sehingga penulis mampu menyelesaikan disertasi ini dengan lancar.
10. Kepada semua pihak yang tidak dapat kami sebutkan satu persatu, yang turut membantu penulis demi suksesnya penyelesaian studi Doktor di Institut Pertanian Bogor ini.
Kepada Allah SWT penulis berdo’a semoga amal kebaikan yang telah diberikan kepada penulis mendapatkan imbalan yang setimpal dari Allah SWT. “ Tiada gading yang tak retak “, penulis menyadari bahwa karya ilmiah ini masih jauh dari kesempurnaan, oleh karenanya kritik dan saran membangun akan selalu penulis harapkan demi kesempurnaan di masa mendatang. Akhirnya, semoga karya ilmiah ini dapat bermanfaat, khususnya bagi dunia peternakan dan pembaca umumnya. Amin.
Bogor, Januari 2010
xv
Penulis dilahirkan di Tangerang, Jawa Barat pada tanggal 14 Mei 1964 sebagai anak pertama dari lima bersaudara dari pasangan Drs. Atang Soewarno (alm.) dan Ruth Sularsihati BA. Pada tahun 1994 penulis menikah dengan Dr. Ir. Maftuchah, MP. dan dikarunia seorang putra bernama Muhammad Syauqi Al Maghfirah yang lahir pada tanggal 22 Desember 2001.
Pada tahun 1989 penulis menyelesaikan pendidikan Strata Satu (S1) di Fakultas Peternakan Universitas Jenderal Soedirman (UNSOED) Purwokerto dari Jurusan Produksi Ternak. Pada tahun 1997, penulis menyelesaikan pendidikan Strata Dua (S2) pada Program Studi Manajemen, Program Pascasarjana Universitas Muhammadiyah Malang (UMM) dan pada tahun 2001 penulis menyelesaikan pendidikan Strata Dua (S2) dari Program Studi Bioteknologi, Sekolah Pascasarjana Institut Pertanian Bogor melalui program penyelenggaraan Beasiswa Pendidikan Pascasarjana (BPPS) dari Departemen Pendidikan Nasional. Selama mengikuti program S3, penulis menjadi Ketua Himpunan Mahasiswa Pascasarjana (HIWACANA) Program Studi PTK periode 2005-2007.
Pada tahun 1990, penulis diangkat sebagai dosen tetap Kopertis Wilayah VII Jawa Timur dan dipekerjakan pada Fakultas Pertanian dan Peternakan Universitas Muhammadiyah Malang (UMM) sampai sekarang. Mulai tahun 2000 hingga saat ini penulis menduduki jabatan sebagai Koordinator Divisi Bioteknologi Hewan dan Sekretaris Pusat Pengembangan Bioteknologi UMM. Karya ilmiah terbaru yang telah ditulis dan merupakan bagian dari disertasi ini adalah : 1) Y Chromosomal Microsatellites Polymorphism in Madura Cattle (Bos javanicus). The 4th Indonesian Biotechnology Conference. Bogor, August, 5 – 7, 2008; 2) Genetic Variation of Indonesian Ongole Cattle Breed (Bos indicus) Based on Five Loci Y Chromosomal DNA Microsatellite. International Research Seminar and Exibition (IRSE) Universitas Muhammadiyah Malang. November, 7 – 8, 2008; 3) Y Chromosome Microsatellites Variation in Bali Cattle (Bos sondaicus)Population. Jurnal Animal Production Vol. 11, No. 3, September 2009, Fakultas Peternakan UNSOED. (Terakreditasi DIKTI No. 56/DIKTI/Kep./2008), dan 4)Contributions of Bos indicus Breed to Genetic Diversity Of Sumatra Native Cattle Based on Y-Chromosome Microsatellite Marker. International Seminar on Animal Industry, Faculty of Animal Science, Bogor Agricultural University. November, 23 – 24, 2009.
xvi
Halaman
DAFTAR ISI ………... xvi
DAFTAR TABEL ... xvii
DAFTAR GAMBAR ... xviii
DAFTAR LAMPIRAN ………..…… xx
PENDAHULUAN Latar Belakang ... 1
Perumusan Masalah ... 5
Tujuan Penelitian ... 7
Manfaat Penelitian ... 7
Ruang Lingkup Penelitian ... 7
TINJAUAN PUSTAKA Keberadaan Bangsa (Breed) Sapi Lokal Indonesia ………..….. 8
Keragaman Alel DNA dan Variasi Genetik Pada Ternak ... 12
DNA Mikrosatelit Spesifik Pada Kromosom Y ………..…... 15
DNA Mitokondria dan Gen Cytochrome b ……….... 18
BAHAN DAN METODE Bahan ………..… 23
Tahapan Penelitian ……….. 32
Metode ……….... 33
HASIL DAN PEMBAHASAN Parameter Sifat Kuantitatif Sapi Lokal ... 38
Isolasi DNA Genom dan Reaksi Amplifikasi PCR ... 42
Jenis dan Frekuensi Alel DNA Mikrosatelit Kromosom Y Genom Sapi 43
Nilai Heterosigositas (h) dan Indeks Polymorphic Information Content (PIC) Alel DNA Mikrosatelit Kromosom Y ... 49
Hubungan Kekerabatan Genetik (Filogenetik) Sapi Berdasarkan Alel DNA Mikrosatelit Kromosom Y ... 51
Reaksi Amplifikasi Gen Cytochrome b Sapi ... 53
Sisi Variabel (Variable Site) dan Konservatif (Conservative Variable) Gen Cytochrome b pada Genom Sapi ... 55
Hubungan Kekerabatan Genetik (Filogenetik) Sapi Berdasarkan Sekuens Gen Cytochrome b ... 74
PEMBAHASAN UMUM ... 79
KESIMPULAN DAN SARAN ... 82
DAFTAR PUSTAKA ... 84
xvii
Halaman
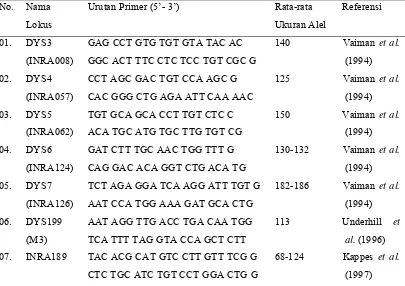
1. Nama lokus, urutan primer, rata-rata ukuran alel dan referensi mikrosatelit
pada Kromosom Y yang digunakan dalam penelitian ...…… 30 2. Rata-rata panjang badan, lingkar dada, tinggi badan dan volume scrotum
dari berbagai populasi sapi jantan penelitian ... 38 3. Tabulasi data lokus mikrosatelit Kromosom Y sapi penelitian ... 47 4. Nilai heterosigositas (h) dan polimorphic information content (PIC) alel
DNA mikrosatelit Kromosom Y pada populasi sapi penelitian ... 50
5. Pensejajaran (alignment) sekuens nukleotida gen cytb antara sapi Aceh
dengan Bos javanicus ... 55 6. Pensejajaran (alignment) sekuens nukleotida gen cyt b antara sapi Pesisir
dengan Bos javanicus ... 56 7. Pensejajaran (alignment) sekuens nukleotida gen cyt b antara sapi Madura
dengan Bos javanicus ... 58 8. Pensejajaran (alignment) sekuens nukleotida gen cyt b antara sapi Bali-Bali
dengan Bos javanicus ... 60 9. Pensejajaran (alignment) sekuens nukleotida gen cyt b antara sapi Bali-
Lombok dengan Bos javanicus ... 61 10. Pensejajaran (alignment) sekuens nukleotida gen cyt b antara sapi PO
dengan Bos javanicus ... 62 11. Pensejajaran (alignment) sekuens nukleotida gen cyt b antara sapi PFH
dengan Bos javanicus ... 64
12. Daftar insersi nukloetida yang terdapat pada sapi Aceh, Pesisir dan Bali-
Lombok ...……….…………... 67 13. Posisi dan jenis substitusi basa dari gen cytochrome b pada sapi penelitian . 69 14. Komposisi penggunaan kodon gen cytochrome b sapi penelitian …………. 72
xviii
Halaman
1. Perkembangan populasi sapi potong tahun 2000 - 2008 (X 1000 ekor) (Direktorat Jenderal Produksi Ternak dalam Badan Pusat Statistik [BPS]
2009) ... 9
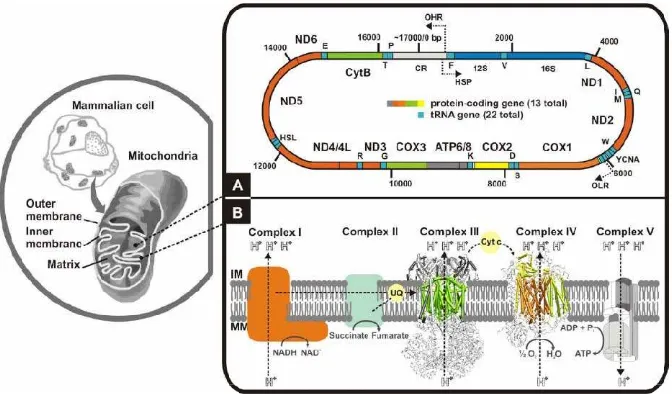
2. Peta genetik DNA mitokondria pada mamalia ... 20
3. Peta lokasi pengambilan sampel darah sapi Aceh ... 23
4. Peta lokasi pengambilan sampel darah sapi Pesisir ... 24
5. Peta lokasi pengambilan sampel darah sapi Madura... 25
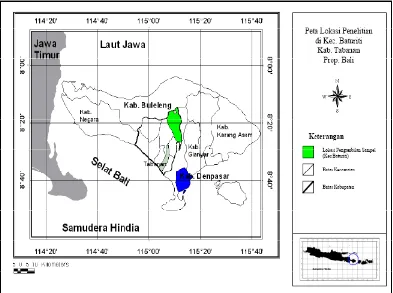
6. Peta lokasi pengambilan sampel darah sapi Bali-Bali ... 26
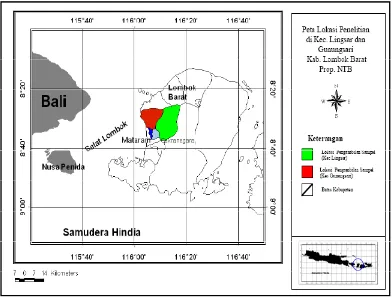
7. Peta lokasi pengambilan sampel darah sapi Bali-Lombok ... 27
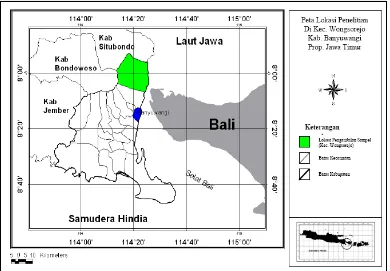
8. Peta lokasi pengambilan sampel darah sapi Peranakan Ongole (PO) ... 28
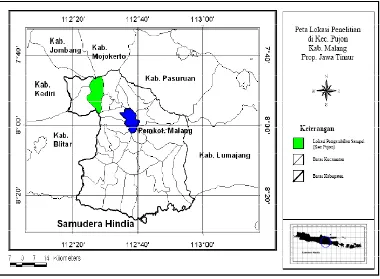
9. Peta lokasi pengambilan sampel darah sapi Peranakan Friesien Holland (PFH) ... 29
10. Daerah gen cytochrome b sebagai target amplifikasi PCR ... 31
11. Bangsa-bangsa sapi-sapi yang digunakan dalam penelitian dengan jenis kelamin jantan ... 41
12. Contoh hasil isolasi DNA genom sapi pada sapi PFH ... 42
13.a. Contoh produk PCR dengan pewarnaan perak (silver staining) dari lokus mikrosatelit INRA 126 genom sapi PO... 43
13.b. Contoh produk PCR dengan pewarnaan perak (silver staining) dari lokus mikrosatelit INRA 189 (hemizygous) genom sapi Bali-Bali ... 44
13.c. Contoh produk PCR dengan pewarnaan perak (silver staining) dari lokus mikrosatelit INRA 057 (multicopy event) genom sapi PFH ... 44
14. Diagram filogenetik sapi lokal Indonesia berdasarkanvariasi DNA mikrosatelit pada Kromosom Y ... 51
xix
17. Diagram pohon filogenetik sapi lokal Indonesia berdasarkan sekuen
xx
1. Haplotipe alel mikrosatelit pada Kromosom Y ... 98 2. Hasil sekuensing produk PCR gen cytochrome b ... 102
PENDAHULUAN
Latar Belakang
Dokumen ‘World Watch List for Domestic Animal Diversity‘ report (3rd ed.) melaporkan bahwa terdapat kurang lebih 6300 bangsa (breed) ternak di dunia dari sekitar 30 spesies hewan domestikasi dan hampir sebagian besar breed saat ini merupakan spesies lokal yang berasal dari negara-negara berkembang. Keragaman
genetik yang dimiliki breed lokal berperan besar dalam keberhasilan program pemuliaan ternak di negara-negara berkembang selama kurun waktu abad 19 hingga
20. Hal ini jelas menggambarkan bahwa spesies lokal merupakan sumber daya
genetik yang penting dan unik untuk antisipasi kebutuhan produksi ternak saat ini
maupun mendatang. Namun, kenyataannya saat ini banyak breed lokal yang mulai menurun potensinya, baik kuantitas maupun kualitas. Diperkirakan satu hingga dua
breed punah setiap minggunya (Schearf, 2003).
Dua spesies utama dari famili Bovidae yang telah didomestikasi adalah sapi
(Bos) dan kerbau (Buballus). Diantara spesies sapi yang didomestikasi dan masih eksis keberadaannya sebagai tetua bangsa sapi adalah Yak (Bos grunniens ), Gayal /
Mithan (Bos gaurus) dan sapi Bali (Bos javanicus), sedangkan Taurine (Bos taurus)
dan Indicine (Bos indicus) telah dijinakan sebagai ternak domestik (Lenstra &
Bradley 1999). Dua tipe bangsa sapi terakhir tersebut merupakan yang terbesar
jumlah populasinya di dunia dan tetua liar dari Bos taurus dan Bos indicus yaitu
Auroch (Bos primigenius) tersebar mulai dari Pasifik melalui Asia dan Eropa hingga
Atlantik, dan juga dari dataran tundra di belahan utara ke selatan hingga India dan
Afrika. Tetapi Bos primigenius ini diduga telah punah pada abad 14 SM di sekitar
Mesir, tetapi sisanya ditemukan hidup lebih lama di daerah lain, termasuk Eropa dan
diketahui spesies terakhir mati di tahun 1627 M (Epstein & Mason 1984).
Benua Asia memiliki kurang lebih tiga ratus juta sapi dan dua ratus juta
diantaranya terdapat di sub benua India. Sapi-sapi di Asia dan Afrika, umumnya
Indonesia dan Filipina. Oleh karena itu, peternak sapi di Asia telah dikenal
peranannya sangat penting dalam proses domestikasi maupun ekologi pertanian di
benua ini.
Di Indonesia, sapi Bali merupakan sapi lokal yang sangat populer terkait dengan
statusnya pada proses berkehidupan masyarakat petani di Indonesia, khususnya
sebagai hewan tarik dan penyedia daging. Selain sapi Bali, di Indonesia juga
terdapat jenis atau bangsa sapi lokal lain, baik yang karena status silsilahnya masih
terdapat hubungan darah dengan Banteng (seperti sapi Madura) maupun dari bangsa
sapi Bos taurus dan Bos indicus yang karena faktor ekonomi dan politik pada masa lampau (penjajahan maupun setelah merdeka) didatangkan ke Indonesia dan
akhirnya mampu beradaptasi dengan lingkungan sehingga menjadi bagian dari ternak
lokal yang ada. Oleh karenanya, studi genetik sapi lokal yang ada di Indonesia
menjadi menarik dikarenakan variasi genetiknya yang cukup besar. Hal ini penting
terkait dengan usaha perbaikan sifat maupun menjaga karakter genetiknya sehingga
ternak lokal ini tidak semakin menurun kualitas genetiknya bahkan punah dari negeri
Indonesia.
Bangsa sapi umumnya dibedakan secara morfologi dengan membedakan ada
(Bos indicus) dan tidak adanya (Bos taurus) gumba (Lenstra & Bradley 1999). Namun saat ini dengan kemajuan teknologi molekuler, maka pembedaan genus Bos
tersebut dapat didasarkan pada variasi urutan nukleotida pada DNA mitokondria
(Loftus et al. 1994), DNA mikrosatelit (MacHugh et al. 1997), DNA satelit (Nijman et al. 1999) dan kromosom Y (Kikkawa et al. 2003; Verkaar et al. 2003, Verkaar et al. 2004) yang kemudian digunakan untuk identifikasi introgesi genetik dan studi percampuran populasi. Data molekuler mengindikasikan adanya fenomena dikotomi
bahwa umumnya sapi Bos taurus adalah breed Eropa sedangkan sapi Bos indicus adalah breed Asia. Berdasarkan haplotipe DNA mitokondria yang diperoleh dari specimen arkeologi, juga telah dapat dirancang kembali tentang Auroch Eropa dan
Timur Dekat yang kemudian diinterpretasikan bahwa Timur Dekat merupakan asal
mulanya domestikasi sapi Bos taurus yang kemudian diikuti dengan introduksi
pejantan Auroch dari Eropa (Troy et al. 2001; Götherström et al. 2005; Edwards et
Banyak cara dan usaha yang telah dilakukan untuk karakterisasi genetik sapi
lokal Indonesia, dimana studi awal dimulai tahun 1974 hingga 1977 dan beberapa
hasilnya telah dipublikasikan oleh Namikawa et al. (1980) tentang karakterisasi sapi lokal di Indonesia berdasarkan golongan darah dan protein, kemudian Namikawa et al. (1982a) berdasarkan protein darah dan enzim, serta Namikawa et al. (1982b) berdasarkan komposisi asam amino rantai β dari haemoglobin X. Hasilnya antara
lain menunjukkan bahwa sapi Bali memiliki spesifikasi, seperti alel HbX pada golongan darahnya dan sapi lokal lain tidak, serta sapi Peranakan Ongole (PO)
sangat dominan karakter genetiknya dari sapi zebu India.
Studi karakterisasi genetik pada tingkat molekuler di Indonesia masih sangat
jarang dilakukan. Adanya pertimbangan bahwa perkembangan cepat sejumlah
penciri genetik molekuler lebih diskriminatif dan akurat dibandingkan fenotipiknya,
maka penggunaan marka ini akan sangat membantu dalam penanganan manajemen
sistim seleksi pada ternak sapi potong (Ge et al. 2002).
Penggunaan sekuen DNA pada studi genetika populasi telah berkembang
secara cepat pada ternak, terutama setelah ditemukannya sekuen DNA dengan
tingkat variabilitas dan polimorfis yang tinggi sebagai penanda genetik. Meskipun
penggunaan penanda genetik klasik seperti protein darah dan biokimia cukup
berperan penting dalam beberapa studi genetika populasi, namun salah satu masalah
yang membatasi penggunaan penanda ini adalah keterbatasan jumlah genotip pada
setiap lokus. Adanya penemuan lokus-lokus DNA dengan tingkat variabilitas tinggi,
misal DNA mikrosatelit menawarkan sejumlah solusi untuk memecahkan masalah
keterbatasan tersebut (Chakraborty & Jin 1993).
Informasi tentang alel-alel spesifik (breed spesific allele) dari data molekuler untuk sapi lokal Indonesia masih sangat terbatas. Hasil penelitian Muladno et al. (2000) dalam Kantor Menteri Negara Riset dan Teknologi [KMNRT] (2000) telah
menunjukkan adanya alel spesifik bangsa (breed spesific allele) untuk sapi Bali pada lokus mikrosatelit INRA 023. Hasil studi Noor et al. (2000) juga menunjukkan bahwa sapi Bali memiliki alel spesifik pada lokus mikrosatelit HEL9 dan INRA 035
dibandingkan sapi Bos taurus (Simmental, Limousin, dan Brangus), namun hasil ini masih merupakan gambaran awal tentang adanya fenomena alel spesifik pada ternak
Winaya et al. 2000), diketahui pula bahwa penciri molekuler DNA mikrosatelit mampu memberikan gambaran awal tentang hubungan genetik antara sapi Bali,
Madura, PO dan Brangus. Namun hasil ini belum mampu menunjukkan adanya
hubungan antara penciri DNA mikrosatelit dengan sifat-sifat ekonomis sapi,
terutama terhadap kualitas daging.
Sekuen DNA yang menawarkan sejumlah besar karakter memiliki pula
potensi homologi sehingga dapat dimanfaatkan bagi studi filogenetik. Pemahaman
ini akan menawarkan pula sejumlah besar potensi utama disamping kemungkinan
terdapatnya karakter yang menyesatkan dari indikasi homologi berdasarkan analisis
filogenetik. Sekuen DNA dapat sangat berbeda dari karakter morfologi. Adanya
perubahan karena mutasi juga menjadi penyebab perbedaan. Adanya perbedaan ini
memerlukan pula metode yang berbeda dalam analisis sekuen molekuler untuk
hubungan homologinya, yang akan membedakan dengan sifat morfologinya (Mindell
1991).
Sejak istilah homologi didefinisikan sebagai “ organ yang sama pada hewan
tetapi berbeda dalam berbagai variasi bentuk dan fungsi “ oleh Owen (1848) dan
telah banyak digunakan, namun hingga saat ini masih menjadi perdebatan (Scotland
1992). Homologi merupakan konsep yang menggambarkan suatu hipotesis yang
berkaitan dengan penampilan individu dan penggunaannya pada tingkat yang
berbeda dari organisasi biologinya (misal : fenotipik, genotipik). Sebagian besar
ilmuwan biologi evolusi, terutama bidang sistimatika organisme telah setuju bahwa
homologi menjelaskan hubungan antara tampilan-tampilan yang sama secara umum
pada organisme, yang kejadiannya dapat diturunkan. Demikian pula sebaliknya,
persamaan tersebut dapat pula terjadi karena adanya evolusi yang terpusat maupun
paralel. Oleh karena itu, identifikasi penampilan homologi merupakan suatu langkah
yang krusial dalam menentukan hubungan filogenetik (Mindell 1991).
Klasifikasi organisme juga telah menjadi suatu hal yang menarik bagi para
ilmuwan biologi sejak publikasi hasil-hasil penelitian awal dalam bidang biologi.
Misal, Aristoteles telah membuat suatu sistim untuk klasifikasi spesies hewan, yang
dimulai dengan membagi hewan ke dalam dua grup utama, yakni warna merah darah
seperti yang terdapat pada manusia atau vertebrata dan semakin berkurangnya warna
menjadi dua grup berdasarkan cara reproduksi, seperti siklus hidup, perkembangan
dalam telur, bentuk kepompong dan sebagainya. Oleh karenanya, klasifikasi
organisme merupakan suatu bagian esensial dari perkembangan pengetahuan
manusia mengenai dunia kehidupan (Dunn & Everitt 1982).
Ilmuwan biologi modern memberikan pula catatan bahwa metode awal dalam
klasifikasi hewan dan tanaman umumnya masih menggunakan pertimbangan yang
hanya menggambarkan permukaannya saja dan hanya bermanfaat untuk sarana
komunikasi. Secara umum masih belum menggambarkan sisi “ alamiah ” atau “
kenyataan “ yang sebenarnya. Untuk menjawab permasalahan tersebut, maka ide
tentang metode klasifikasi berdasarkan perbandingan asam amino atau urutan asam
nukleat dapat digunakan sebagai pendekatan metode untuk karakter klasifikasi pada
makluk hidup berdasarkan indikasi adanya homologi (Dunn & Everitt 1982).
Perkembangan dalam analisis genom dan genetika populasi pada manusia
mengarah pada haplotipe kromosom Y yang merupakan alat penting dalam
mempelajari populasi secara alami (Hurles & Jobling, 2001). Tanpa
mengesampingkan daerah pseudo-autosom, aksi kromosom Y secara keseluruhan
merupakan unit tunggal non rekombinan dan merupakan haploid spesifik jantan. Hal
ini dapat diyakinkan bahwa berbagai kombinasi pada kejadian mutasi sepanjang
garis keturunan jantan terkonservasi merupakan suatu haplotipe pautan tunggal yang
tidak meragukan (bias). Penciri kromosom Y diperlukan sebagai suatu analog jalur
jantan sebagaimana DNA mitokondria (mtDNA) pada jalur betina. Tingkat
polimorfisme penciri pada daerah non-rekombinan kromosom Y dimulai dari yang
paling rendah atau jarang, yakni pada kejadian bi-alel pada titik mutasi single nucleotide polymorphisms (SNPs) hingga yang paling sering ditemukan pada lokus penciri minisatelit atau mikrosatelit (short tandem repeat [STR]). Adapun polimorfisme SNPs pada kromosom Y sering ditemukan pada populasi yang spesifik
(Hammer et al. 1997).
Perumusan Masalah
Saat ini untuk pengukuran variasi genetik pada populasi (misal, breed-breed ternak domestikasi) masih memerlukan penciri mikrosatelit kromosom Y lebih
banyak data sekuens DNA pada kromosom Y, tetapi informasi untuk kromosom Y
pada mamalia lain masih kurang (Hurles & Jobling, 2001). Namun, akhir-akhir ini
telah dilaporkan beberapa survei tentang filogenetik daerah kromosom Y dari spesies
ternak domestikasi berdasarkan variasi keterpautan bagian spesifik jantan kromosom
Y (male specific Y chromosome / MSY). Dilaporkan pula variasi nukleotida yang rendah antar lokus MSY pada kuda (Lindgren et al. 2004), sapi (Hellborg and Ellegren 2004) dan domba (Meadows et al. 2004). Meskipun demikian, beberapa studi yang memanfaatkan variasi lokus MSY telah mampu mengangkat aspek-aspek
sejarah keberadaan populasi. Bradley et al. (1994) telah mengembangkan pelacak (probe) spesifik untuk lokus Y yang mampu membedakan subspecies Bos taurus (taurine) dan Bos indicus (zebu).
Penelitian ini merupakan analisis terhadap kromosom Y untuk mendukung
pengembangan metode analisis variasi genetik maupun kekerabatan individu pada
spesies sapi (bovidae) yang mengarah pada jenis kelamin jantan. Pertimbangan
dilakukan khusus pada individu sapi jantan adalah bahwa dalam konteks penyebaran
populasi sapi di Indonesia masih bertumpu pada teknologi Inseminasi Buatan (IB)
untuk keperluan manajemen budidaya, sehingga peran pejantan sebagai sumber
sperma menjadi sangat penting. Oleh karena itu, analisis menggunakan penciri
molekuler DNA mikrosatelit pada kromosom Y dan DNA mitokondria pada gen
cytochrome b (cyt b) yang diaplikasikan pada beberapa bangsa (breed) sapi lokal Indonesia menjadi hal yang penting. Sebagaimana diketahui bahwa penciri mtDNA
berbeda dalam konteks segregasi secara genetis, yakni mtDNA diturunkan dari garis
maternal (induk), sedangkan mikrosatelit pada kromosom Y melalui garis paternal
(pejantan), maka kedua penciri tersebut dapat dijadikan sebagai model pendekatan
berbeda dalam segregasi alel. Pada penelitian ini sampel breed merupakan sapi jantan dengan maksud untuk lebih membuktikan sejauhmana kemungkinan
terjadinya variasi di dalam mendapatkan hasil interpretasi penciri genetik yang
berbeda.
Berdasarkan hal tersebut diatas, maka penggunaan data molekuler dalam studi
genetik ternak, terutama ternak sapi-sapi lokal yang banyak terdapat di Indonesia
akan sangat berperan penting dalam penentuan status genetiknya. Pemanfaatan
membantu meningkatkan kembali keberadaan sapi lokal, terutama akibat kondisi
global, derasnya introduksi sapi-sapi impor lambat laun akan menghilangkan
keberadaan ternak lokal dan akan punah jika tidak diperhatikan secara serius.
Tujuan Penelitian
Penelitian ini secara khusus bertujuan untuk : 1) mendeteksi derajat polimorfisme
DNA mikrosatelit di kromosom Y dan gen cyt b pada populasi sapi Aceh, sapi Pesisir, sapi Madura, sapi Bali di Bali Bali), sapi Bali di Lombok
(Bali-Lombok), sapi PO dan sapi PFH; 2) mendapatkan alel-alel DNA mikrosatelit di
kromosom Y dan gen cyt b yang spesifik untuk setiap populasi sapi tersebut; 3) mengetahui filogenetik populasi sapi lokal yang ada di Indonesia berdasarkan penciri
DNA mikrosatelit kromosom Y dan gen cyt b; dan 4) mengetahui konsistensi filogenetik berbasis DNA mikrosatelit dan gen cyt b.
Manfaat Penelitian
Hasil penelitian ini dapat digunakan sebagai salah satu landasan dalam
menentukan program pemuliaan sapi lokal di Indonesia dan dapat menggunakan
penciri DNA mikrosatelit dan gen cyt b sebagai Marker Assisted Selection (MAS) atau Marker Pembantu Seleksi pada sifat-sifat produksi atau reproduksi sapi.
Ruang Lingkup Penelitian
Ruang lingkup penelitian meliputi penggunaan sampel DNA yang diekstraksi
dari beberapa jenis bangsa sapi yang sudah adapatip maupun endemik hidup di
Indonesia, seperti sapi Aceh, Pesisir, Madura, Bali-Bali, Bali-Lombok, PO dan PFH;
analisis DNA inti dan gen cyt b sapi-sapi tersebut; analisis data secara statistik yang meliputi analisis polimorfisme alel-alel mikrosatelit pada kromosom Y dan analisis
TINJAUAN PUST
AKAKeberadaan Bangsa (Breed) Sapi Lokal Indonesia
Indonesia merupakan negara kepulauan di Asia Tenggara, dan memiliki banyak
bangsa sapi sebagaimana hewan-hewan lainnya. Sapi Bali, sangat dipercaya sebagai
sapi lokal hasil domestikasi dari Banteng (Bos /Bibos banteng), yang merupakan hewan klas bovidae primitif dengan habitat di beberapa hutan wilayah Asia Tenggara
(Namikawa et al. 1980). Berdasarkan sejarah diketahui juga bahwa sapi zebu India secara berulangkali telah diintroduksi ke wilayah Indonesia pada awal abad 19.
Tampaknya komposisi genetik berbagai bangsa sapi yang ada di Indonesia menjadi
cukup sulit untuk diklarifikasi filogeninya. Namun, justru adanya variasi genetik
yang cukup besar pada sapi-sapi lokal Indonesia ini menjadi bahan studi yang
menarik, karena bisa jadi sapi-sapi lokal Indonesia menjadi salah satu kumpulan gen
terbesar terkait dengan asal usul sapi-sapi yang rumit tersebut dan deferensiasi
genetik menjadi masalah yang sangat penting untuk dikaji (Namikawa et al. 1980) Antara tahun 1870 – 1890, sapi Jawa merupakan sapi idaman untuk mengolah
lahan pertanian. Namun dilaporkan tahun 1907 – 1910 bahwa sapi Jawa menurun
kualitas genetiknya, seperti berkurangnya kemampuan konsumsi pakan dan tenaga.
Kemampuan reproduksinya juga menjadi rendah sehingga banyak yang diafkir.
Maka pemerintah yang berkuasa saat itu (Belanda) memutuskan tahun 1905 untuk
memasukkan sapi zebu ke Indonesia. Kegiatan ini berlangsung hingga tahun
1920-an. Kegiatan pemurnian genetik sapi-sapi lokal di Indonesia sebenarnya telah
dimulai sejak dikeluarkan Peraturan Pemerintah Belanda tahun 1913, yang
menetapkan pulau Madura sebagai tempat pemurnian sapi Madura dan pulau Bali
dan pulau Lombok sebagai tempat pemurnian sapi Bali, sedangkan sebelumnya
tahun 1912, pulau Sumba telah ditetapkan sebagai tempat pemurnian sapi Ongole
yang diimport dari Nellore, India dan kegiatan ini dilakukan dengan ketat di bawah
pengawasan pemerintah Belanda saat itu (Soeratmo 1923 dalam Talib 1988).
Diantara sapi-sapi lokal Indonesia, maka sapi Bali memang memiliki potensi
genetik yang besar (Ditjenak 2008), terutama sebagai ternak pedaging. Namun,
kenyataannya sampai saat ini teknologi yang tepat untuk sistem perkawinan ternak
sapi di Indonesia adalah teknologi Inseminasi Buatan (IB), maka sistem seleksi untuk
mendapatkan pejantan yang unggul, terutama untuk penyediaan sperma yang
berkualitas harus mendapatkan perhatian, sehingga akan mampu meningkatkan pula
kualitas sapi-sapi potong yang dihasilkan dari teknologi Inseminasi Buatan tersebut.
Bangsa-bangsa (breed) sapi yang ada di dunia saat ini sebenarnya terbentuk selama berabad-abad melalui proses seleksi alam maupun seleksi yang dilakukan
oleh manusia. Breed sapi diseleksi terutama untuk ketahanan terhadap kondisi lingkungan dan kebutuhan manusia (Maudet et al. 2002). Menurut beberapa buku catatan seleksi ternak, bahwa pada 200 tahun terakhir, seleksi mengarah pada isolasi
genetik beberapa breed sapi, dan seleksi yang ketat untuk produktivitas tinggi telah menyebabkan turunnya sejumlah populasi breed. Variasi genetik yang ditemukan pada breed dikembangkan ke arah karakter baru, seperti respon terhadap perubahan lingkungan, penyakit atau kondisi pasar. Namun, sapi-sapi lokal (indigenous) justru memiliki banyak kombinasi gen dan karakter spesifik untuk adaptasi terhadap
berbagai kondisi, seperti ketahanan penyakit, adaptasi kondisi pakan terbatas atau
kualitas pakan yang rendah dan sebagainya, yang tidak ditemukan pada breed-breed terseleksi. Namun, karena tidak adanya perhatian yang serius, maka beberapa breed lokal ini terancam punah (Maudet et al. 2002).
Upaya untuk mempertahankan keragaman genetik ternak-ternak lokal perlu
dijaga bahkan ditingkatkan, terutama untuk tujuan seleksi maupun pemanfaatan
gen-gen tertentu yang seringkali tidak dimiliki oleh breed modern hasil seleksi ketat. Sehingga, hal ini seolah-olah merupakan suatu tindakan kontroversi, antara upaya
meningkatkan produktivitas ternak (pola pemuliaan) yang berakibat pada turunnya
keragaman genetik dengan kepentingan pelestarian plasma nutfah yang
mengharapkan keragaman genetik. Jadi, konsep konservasi adalah pertimbangan
pembangunan masa kini jangan sampai merusak kemungkinan masa depan. Masih
banyak potensi ternak lokal yang belum tergali atau diketahui, sehingga konservasi
merupakan usaha yang rasional untuk menjaga mutu genetik ternak-ternak lokal
tersebut (Setiadi & Diwyanto 1997).
Contoh kasus pada sapi Bali, dalam sejarah panjang domestikasi Banteng
potensi genetik lebih baik jika dibandingkan sapi Bali saat ini. Hal ini berarti telah
terjadi penurunan mutu genetik (Darmadja 1980; Pane 1991). Demikian pula hasil
studi Noor et al. (2000), dengan menggunakan metode analisis lebih baru bahwa berdasarkan data hasil analisis polimorfisme iso-electrical focussing (IEF) protein darah, analisis DNA mikrosatelit, analisis struktur kromosom dan analisis struktur
bulu dengan menggunakan scanning electron microscope(SEM), diduga bahwa sapi Bali pejantan di Balai Inseminasi Buatan, Singosari, Malang, Jawa Timur dan pulau
Bali telah terkontaminasi oleh breed sapi lain. Meskipun hasil ini masih menggunakan sampel terbatas dan belum mengarah pada kesimpulan menyeluruh,
tetapi merupakan indikasi telah terjadi introduksi genetik pada sapi ini.
Pembahasan tentang kepentingan dan peluang pengembangan manajemen
sumber genetik ternak bagi kepentingan nasional maupun global dimulai di Amerika
Serikat tahun 1984 (CAST 1984; OTA 1987; NRC 1993). Secara umum diskusi
membahas tentang strategi mempertahankan breed-breed ternak yang populasinya telah langka, termasuk diskusi tentang masalah yang kritis dan peluang ke depan
serta program konservasi yang luas untuk breed-breed ternak langka. Untuk manajemen sumber genetik peternakan, difokuskan pada negara-negara sedang
berkembang dari sudut pandang kepentingan global. Karena, hampir sebagian besar
ternak domestikasi terdapat di wilayah negara-negara ini dan karakter genetik lokal
ternak di wilayah ini telah menurun akibat seleksi terarah yang sangat ketat (Mason
& Crawford 1993). Namun, peluang dan kesempatan untuk manajemen sumber
genetik ternak di negara-negara berkembang justru cenderung tidak diperhatikan.
Padahal saat ini variasi sumber genetik ternak diperlukan untuk keperluan
produktifitas ternak pada berbagai kondisi lingkungan, juga kemungkinan
mempertahankan sumber genetik ternak dan memberikan fasilitas yang cepat untuk
adaptasi terhadap perubahan tujuan pemuliaan ternak (Notter 1999).
Program manajemen sumber genetik ternak dalam jangka panjang sebaiknya
diarahkan untuk pencapaian potensi maksimal spesies dalam beradaptasi dengan
perubahan lingkungan. Jadi, tindakan saat ini jangan sampai mengurangi upaya kita
untuk mempertahankan tingkat kemampuan genetik dalam produksi maupun
performans ternak. Memang seringkali terjadi perbedaan informasi yang besar
punah. Studi umumnya difokuskan pada ukuran populasi dan struktur populasi yang
diperlukan untuk mencegah inbreeding dan menerimanya untuk tujuan menjaga tingkat heterosigositas dan potensi adaptasi, yang seolah-olah hal ini mencukupi
untuk menambah variasi genetik. Padahal sesuai dengan petunjuk manajemen
konservasi spesies langka, studi tidak hanya selalu tentang pemeliharaan variasi
genetik dalam breed itu sendiri tetapi lebih dari itu, yakni manajemen strategi yang sesuai untuk populasi secara keseluruhan (Notter 1999).
Keragaman Alel DNA dan Variasi Genetik pada Ternak
Manajemen yang menekankan pada pemeliharaan heterosigositas individu
mungkin sedikit berguna jika dibandingkan manajemen yang langsung bertujuan
untuk memelihara adanya keragaman alel-alel dalam genom suatu individu atau
spesies. Keragaman alel disini didefinisikan sebagai suatu nukleotida yang memiliki
urutan / sekuens unik yang dapat menghasilkan kemampuan individu dalam
produktivitas atau mampu mengeluarkan efek spesifik regulator lain dari sekuens
unik tersebut. (Notter 1999). Denniston (1977) menggambarkan bahwa ukuran
populasi yang efektif diperlukan untuk memelihara beberapa alel pada lokus dalam
suatu perkawinan tunggal secara random dan kepentingan ini lebih besar daripada
keperluan untuk mempertahankan nilai inbreeding yang telah ditetapkan pada populasi. Notter & Foose (1986) juga menunjukkan bahwa memelihara keragaman
alel dalam suatu spesies lebih efisien untuk pencapaian subdivisi spesies daripada
populasi utuh, sehingga subpopulasi sangat cukup untuk menjaga akumulasi efek
inbreeding atau menjaga kemungkinan untuk melakukan strategi crossing pada subpopulasi guna mendapatkan heterosigositas.
Diperkirakan jumlah breed ternak mamalia sekitar 4 000, sangat kecil dibandingkan seluruh spesies mamalia yang sudah dikenal, dan sekitar 1 000 spesies
dalam kondisi mengkawatirkan (Loftus & Scherf 1993). Sehingga, dalam jangka
panjang menjaga 4000 breed dengan suatu pendekatan ukuran populasi yang sesuai bagi spesies yang dilindungi menjadi tidak cocok dan tidak perlu untuk menjaga
potensi adaptif spesies tersebut. Akan tetapi, diperlukan dukungan untuk program
daftar FAO (1996) terdapat 783 breed sapi secara global. Pertanyaan yang harus diperhatikan adalah seberapa besar potensi nilai keragaman alel yang dimiliki dan
mungkinkah secara langsung memiliki nilai komersial. Jawaban dari pertanyaan ini
adalah bagaimana kita dapat menemukan alel-alel spesifik yang berkorelasi dengan
sifat-sifat kuantitatif tertentu yang memiliki nilai komersial atau lokus sifat
kuantitatif (quantitative trait loci/QTL). Oleh karenanya memelihara keragaman alel pada suatu spesies sapi yang memiliki potensi tertentu akan sangat berguna sebagai
suatu cara untuk memfasilitasi akses ke depan dalam memanfaatkan suatu peluang.
DNA genom eukariot yang jauh lebih panjang dibandingkan dengan DNA
genom prokariot, sebagian besar mengandung sekuen berulang. Walaupun fungsinya
belum diketahui secara pasti, sekuen tersebut diduga memiliki struktur yang berperan
dalam arsitek interfase nukleus, meiosis kromatin dan atau metafase kromosom.
Sejumlah besar informasi telah dikumpulkan mengenai elemen berulang dan
organisasinya dalam genom ternak. Konsekuensinya adalah sesuatu yang harus
dipertimbangkan berkenaan arti yang ada, yakni tentang kegunaan klasifikasi sekuen
berulang berdasarkan struktur, distribusi dan frekuensi pengulangannya. Beberapa
genom eukariotik mengandung beberapa macam repetitif DNA, mulai dari yang
sederhana berupa suatu nukleotida tunggal hingga tingkatan-tingkatan satelit DNA
(Tautz et al. 1984; Ellegren et al. 1992), dan beberapa repetitif DNA ini memiliki polimorfik tinggi.
Beberapa studi menunjukkan bahwa sekuen berulang DNA (mikrosatelit) dapat
digunakan untuk mengidentifikasi asal usul individu dari suatu populasi (Bradley et al. 1998; Edwards et al. 2000; Maudet et al. 2002). Analisis variasi genetik menggunakan penciri mikrosatelit adalah analog dengan metode lama yakni
elektroforesis protein. Sejumlah mikrosatelit diaplikasikan, setiap penciri dideteksi
sejumlah alternatif alel pada lokus genetik spesifik. Alel-alel individu mencerminkan
frekuensi yang berbeda antar populasi yang berbeda. Perbedaan yang besar pada alel
ini memungkinkan ketersediaan data dasar untuk pendugaan jarak genetik (Bradley
et al. 1998). Bagaimanapun juga penciri mikrosatelit berbeda dengan protein. Mikrosatelit lebih variatif (lebih banyak alel per lokus) dan tingkat informasinya
menjadi lebih bervariasi dalam hubungan evolusinya antar alel dalam lokus yang
alelnya. Gambaran ini dijelaskan hasil studi bahwa distribusi ukuran alel yang
berbeda sering ditemukan antar populasi Bos indicus dan Bos taurus. Oleh karena alel breed zebu juga terdapat pada hewan yang tetuanya campuran, maka frekuensi alelnya dapat digunakan untuk mengestimasi kontribusi relatif dari breed zebu dan Bos taurus dalam genom inti keturunannya.
Mitokondria DNA (mtDNA) merupakan satu diantara penciri DNA yang
sangat popular digunakan sebagai penciri genetik pada populasi hewan (Avise et al., 1987). Alasannya, karena penciri ini memiliki tipikal tingkatan variabilitas tinggi,
merupakan jalur turunan maternal (induk), dan lebih mendekati model evolusi netral.
Dalam suatu populasi yang mengalami mutasi atau penyimpangan genetik setimbang
(equilibrium), maka tingkat dugaan variasi genetik dari suatu lokus netral adalah proporsional terhadap ukuran populasi efektif (dan tingkat laju mutasi Wright 1953).
Variasi mtDNA secara tipikal juga dapat menggambarkan pengaruh demografi,
seperti variasi ukuran populasi antar spesies maupun antar populasi sehingga
menjadikan mtDNA alat yang populer untuk tujuan konservasi genetik (Harrison
1989; Roman & Palumbi 2003).
DNA mitokondria (mtDNA) telah menjadi sarana yang sangat penting dalam
memahami asal usul bangsa dan proses domestikasi sapi. Studi awal oleh Loftus et al. (1994) dan Bradley et al. (1996) menunjukkan bahwa terdapat dua kelompok utama haplotipe berdasarkan mtDNA, yang mewakili garis keturunan dari Bos indicus (zebu) dan Bos taurus (taurine). Identifikasi proses domestikasi independen pada kedua breed sapi tersebut menunjukkan pula bahwa sapi zebu didomestikasi di benua Asia, sedangkan taurine berasal dari suatu pemisahan kelompok spesies
aurochs di Timur Dekat dan Afrika (Loftus et al. 1994a). Studi lebih lanjut pada bangsa sapi taurine di Eropa, Afrika dan Timur Dekat menunjukkan bahwa hampir
semua sekuens mtDNA memiliki satu akar dari empat tetua utama haplotipe, salah
satu dari tetua merupakan haplotipe predominan di Afrika sedangkan lainnya di
Eropa dan Timur Dekat. Setiap lokasi geografi sampel sapi menghasilkan suatu titik
sebagai pusat filogeni dari salah satu dari dua haplotipe, yang dibedakan hanya pada
transisi basa di 16050, 16113 dan 16255 dari d-loop mitokondria. Hal ini juga menggambarkan bahwa domestikasi sapi Afrika terpisah dari sapi taurine. Hasil studi
(MacHugh et al. 1997; Loftus et al. 1999; Hanotte et al. 2002), yang disimpulkan bahwa sapi Afrika berawal dari benua tersebut dan adanya introduksi genetik dari
sapi Eropa dan Timur Dekat.
DNA mitokondria hewan berukuran kecil (sekitar 15-20 kb) berupa molekul
sirkuler, terdiri atas sekitar 37 gen yang mengkode 22 tRNA, dua rRNA dan 13
mRNA, yang terakhir gen yang mengkode protein utama termasuk transport elektron
dan fosforilasi oksidatif dari mitokondria. Genom mitokondria tersusun sangat
efisien; tanpa intron, memiliki daerah antara (spacer) intergenik yang kecil dimana posisi reading frames kadang-kadang overlap. Daerah kontrol (control region) merupakan daerah non koding utama, dan bertanggung jawab untuk regulasi
transkripsi untai berat / heavy (H) and ringan/light (L) dan replikasi untai H.
Terdapat banyak molekul mtDNA pada suatu mitokondrion yang menjadikan
mtDNA secara alamiah memiliki sumber variasi genetik. Hal ini menjadikan pula
mtDNA lebih cepat perbaikan inefisiensi replikasinya (Brown et al. 1982) dibandingkan dengan DNA inti (Clayton, 1991). mtDNA pada awalnya dilaporkan
tidak mengalami rekombinasi (Hayashi et al. 1985), tetapi beberapa bukti telah menunjukkan adanya kejadian rekombinasi (Hagelberg et al. 1999). Tingkat kejadian mutasi yang relatif cukup tinggi dibandingkan dengan gen-gen inti telah mengarah
pada suatu hasil kumpulan informasi tentang adanya titik-titik mutasi yang
memungkinkan dalam diskriminasi spesies hewan yang berdekatan. Hal ini
menjadikan catatan pula bahwa mtDNA menunjukkan adanya derajat variabilitas
intraspesifik tinggi sehingga dapat pula dijadikan referensi ketika kita mempelajari
perbedaan genetik antar organisme yang didasarkan pada polimorfisme (Chow &
Inogue 1993).
DNA Mikrosatelit Spesifik pada Kromosom Y
Pada dekade terakhir ini beberapa studi telah difokuskan pada analisis
kromosom Y mamalia, diantaranya studi kesehatan masyarakat (Jobling &
Tyler-Smith 2000) dan evolusi pembentukan jenis kelamin/sex (Graves 2002). Kromosom
Y hanya merupakan bagian dari genom mamalia yang secara eksklusif diturunkan
terhadap sejarah evolusi suatu spesies, sebagai contoh penciri genetik yang terkait
dengan kromosom Y telah memberikan kontribusi sebagai bahan pertimbangan
dalam sejarah filogeografi manusia. (Hammer et al. 1997). Kelangkaan marker pada kromosom Y dan informasi sekuens-nya (Petit et al. 2002) serta level variasinya yang rendah (Hellborg & Ellegren 2004; Meadows et al. 2004; Queney et al. 2001) menyebabkan aplikasi data kromosom Y pada populasi genetik non primata, seperti
kuda, sapi dan domba masih sangat jarang ditemukan.
DNA mikrosatelit spesifik pada kromosom Y, sebenarnya telah dikembangkan
pada manusia untuk diskriminasi populasi yang berdekatan maupun untuk
menetapkan hubungan genetik antar populasi yang berbeda (Hammer et at. 1997; Pérez-Lezaun et al. 1997; Seielstad et al. 1999). Tanpa mengesampingkan daerah pseudoautosomal, aksi kromosom Y secara keseluruhan merupakan unit tunggal non
rekombinasi, yang merupakan sifat spesifik jantan dan haploid efektif. Hal ini
membuktikan bahwa awal kejadian kombinasi mutasi dari garis keturunan jantan
dipertahankan secara pautan tunggal oleh haplotipe. Variasi genetik pada haplotipe
yang tersusun antar populasi telah dikembangkan pada ternak dan telah
meningkatkan keberhasilan reproduksi jantan (Nowak 1991).
Pada ternak domestikasi yang memiliki indikasi adanya variasi individu
dalam populasi berdasarkan penciri molekuler mikrosatelit spesifik pada kromosom
Y telah ditunjukkan pula adanya hibridisasi breed dan pola migrasinya (MacHugh et al. 1997; Bradley et al. 1998). Adanya polimorfisme pada mikrosatelit spesifik kromosom Y dapat dijadikan titik awal dalam menyediakan informasi tentang studi
variasi genetik paternal pada sapi maupun spesies yang terkait. Indikasi
ditemukannya pseudogen hormon pertumbuhan (GH) spesifik jantan pada hewan
domestikasi, menunjukkan bahwa kromosom sex dapat mempengaruhi proses
diferensiasi individu. Peran kromosom sex terpaut sifat jantan pada hewan penting
untuk diketahui secara lebih mendalam misal kemampuan dalam reproduksi.
Banyak studi penciri molekuler DNA mikrosatelit dilakukan pada bagian
autosom dan jarang pada kromosom sex, terutama kromosom Y. Hasil studi Hanotte
et al. (2000) dengan menggunakan penciri mikrosatelit spesifikasi pada kromosom Y INRA23 menunjukkan adanya introgesi pejantan zebu di sapi lokal yang ada di
Mozambique, dimana pada masa lalu teluk Mozambique merupakan daerah
perdagangan internasional, termasuk perdagangan sapi, sehingga kemungkinan
terjadinya introgesi bangsa sapi dari luar sangat besar, termasuk adanya alel pada
bangsa sapi Bos taurus yang toleran terhadap penyakit tripanosoma.
Hasil penelitian Edwards et al. (2000) dengan menggunakan 4 buah penciri DNA mikrosatelit (INRA124, INRA126, INRA189 dan BM861) menggambarkan
dugaan adanya aliran gen pejantan dari bangsa taurine maupun zebu pada populasi
hibrid beberapa spesies bovida. Hal yang sama ditemukan pula pada kerbau Afrika,
penggunaan penciri DNA mikrosatelit pada kromosom Y mampu menunjukkan
adanya perbedaan yang cukup tinggi antara kerbau di daerah Afrika Tengah dengan
Afrika Barat Daya.
Studi tentang invasi dan sejarah migrasi populasi manusia modern saat ini
juga difokuskan pada variasi daerah spesifik jantan pada kromsom Y (male-spesific region of the Y chromosome /MSY). Observasi alel-alel yang diketahui haploid dan tidak terikut dalam rekombinasi X-Y memungkinkan adanya haplotipe-haplotipe
yang dapat diidentifikasi untuk menentukan garis keturunan dari jantan. Hal ini
terbukti efektifitasnya untuk studi penyebaran populasi global (Kayser et al. 2000, 2001; Deng et al. 2004) serta untuk penentuan waktu terjadinya ekspansi populasi manusia (Shen et al. 2000).
Uji garis keturunan jantan juga menjadi bagian penting dalam studi
domestikasi spesies, karena ternyata perkawinan terkontrol telah menyebabkan
beberapa pejantan kontribusinya menjadi tidak seimbang pada sejumlah
keturunannya pada generasi yang berbeda. Meskipun masih jarang, beberapa studi
filogenetik yang melibatkan kromosom Y telah dilaporkan pada spesies domestikasi
dikaitkan variasi MSY. Nilai variasi nukleotida yang sangat rendah antar MSY dilaporkan pada kuda (Lindgren et al. 2004), sapi (Hellborg & Ellegren 2004) dan domba (Meadows et al. 2004). Hal ini terjadi karena daerah kromosom Y umumnya bukan daerah rekombinasi sehingga memiliki lokus tunggal (haplotipe), sebaliknya
situasinya sangat berbeda pada autosome (secara umum) dan kromosom X, yaitu
kejadian rekombinasi mampu menghasilkan urutan nukleotida hasil penggabungan