Bab IV Hasil dan Pembahasan
Lumbrokinase merupakan enzim fibrinolitik yang berasal dari cacing tanah L. rubellus. Enzim ini dapat digunakan dalam pengobatan penyakit stroke. Penelitian mengenai lumbrokinase, telah dilakukan sebelumnya oleh peneliti dari Cina dan Jepang (Jin et al., 2000; Mihara et al., 1991), menggunakan cacing tanah L. rubellus dan juga spesies cacing lain yang termasuk dalam famili Lumbricidae.
Pada penelitian ini dilakukan studi pendahuluan untuk mengisolasi gen pengkode lumbrokinase yang berasal dari cacing tanah L. rubellus galur lokal dengan pendekatan melalui isolasi RNA total, mRNA, dan DNA kromosom, yang selanjutnya digunakan sebagai templat dalam proses RT-PCR dan PCR untuk menghasilkan cDNA dan DNA pengkode gen lumbrokinase. Kemudian, dilakukan juga penentuan urutan nukleotida yang mengkode gen lumbrokinase.
Adapun tahap awal yang dilakukan adalah proses penanaman cacing tanah L. rubellus. Selama proses perkembangbiakkan cacing tanah ini, dilakukan pula perancangan primer yang menggunakan program komputer, sehingga diperoleh primer ZN1 dan ZN2. Selain itu, pada proses PCR dan RT-PCR digunakan juga primer FL1 dan FL2 (Wulan, 2006).
IV.1 Penanaman Cacing
Cacing tanah yang digunakan dalam penelitian ini, yaitu cacing tanah spesies L. rubellus galur lokal, yang diperoleh dari peternakan cacing tanah di daerah Sekeloa, Bandung.
Media yang digunakan dalam perkembangbiakkan cacing, yaitu serbuk gergaji, yang sebelumnya telah mendapat perlakuan sehingga memiliki tekstur mirip dengan tanah. Adapun perlakuan yang dilakukan yaitu memasukkan serbuk gergaji ke dalam karung, diamkan selama kurang lebih 1–1,5 minggu dengan setiap hari disirami dengan air. Setelah kurang lebih 1 minggu, media sudah dapat
digunakan untuk mengembangbiakkan cacing tanah. Media pertumbuhan cacing tanah diganti setiap 2 minggu sekali, sedangkan media bekas pertumbuhan cacing tersebut (Gambar IV.1) dapat digunakan sebagai pupuk (kascing).
Gambar IV.1. Perkembangbiakkan cacing tanah.
Budidaya cacing tanah diketahui dapat mengurangi masalah limbah organik, minimal dapat digunakan dalam penanggulangan masalah sampah organik rumah tangga. Cacing tanah sendiri meggunakan bahan organik tersebut, sebagai sumber bahan makanan. Cacing tanah memiliki kemampuan konsumsi yang tinggi, yaitu dapat mengkonsumsi bahan makanan sebesar berat tubuhnya. Dalam penelitian ini, cacing tanah diberi makan setiap 2–3 hari sekali, berupa potongan sayuran sisa memasak, makanan sisa, kotoran burung, dan juga ampas tahu. Potongan sayuran dan makanan sisa biasanya direbus dahulu kemudian ditiriskan, selanjutnya dicampur dengan kotoran burung atau dengan kotoran sapi kemudian diberikan kepada cacing. Sedangkan ampas tahu, dapat pula dicampur dengan makanan tersebut atau hanya diberikan berupa ampas tahu saja, asalkan jangan terlalu basah. Pemberian makanan pada cacing tanah biasanya dilakukan pada bagian permukaan media yang terdapat cacing tanah atau dapat pula dengan memasukkan ke dalam media yang mengandung cacing dan ditutup kembali dengan media.
Cacing tanah L. rubellus yang digunakan pada penelitian ini, yaitu cacing tanah yang telah berusia 1 bulan, kurang lebih memiliki berat 1-2 gram dengan panjang tubuh 8 sampai 12 cm.
IV.2 Perancangan Primer
Perancangan primer dilakukan menggunakan database yang diperoleh dari website NCBI, urutan nukleotida gen pengode lumbrokinase asal cacing tanah, yang telah dilakukan oleh peneliti sebelumnya (Lee et al., 2000; Sugimoto et al., 2000). Perancangan menggunakan program Clustal X, DNA Star, dan Gene Doc Program Primer Select. Adapun yang digunakan gen nomor kode akses AF304199, ABO45719, ABO45720, dan LRAJ3152 (NCBI Protein Data Bank).
Program Clustal X digunakan untuk melakukan penjajaran urutan nukleotida, dalam hal ini, urutan data nukleotida berada dalam bentuk fasta, sehingga dapat dioperasikan dalam program ini. Hasil penjajaran akan memberikan daerah lestari, yaitu daerah yang sama (Gambar IV.2). Hasil penjajaran secara lengkap terlampir (Lampiran A). Primer dibuat berdasarkan daerah lestari tersebut.
Hasil penjajaran menunjukkan bahwa gen nomor kode akses LRAJ3152 terdapat banyak daerah yang berbeda bila dibandingkan dengan tiga nomor kode gen yang lain. Oleh karena itu, urutan gen yang digunakan, yaitu AF304199, ABO45719, dan ABO45720 (NCBI Protein Data Bank). Adapun urutan primer yang dihasilkan, yaitu: primer majuZN1 (GCG GAG GTA GCA TCA TCA ACG) dan primer mundur ZN2 (GMG TAR ACT CTG GAT AGC).
* 180 * 200 * gi|2334519 : TGAAGCCAGACCATACGAGTTCCCATGGCAGGTGTCCGTCCGAAGGAAGTCTTC : 200
gi|1353713 : TGAAGCCAGACCATACGAGTTCCCATGGCAGGTGTCCGTCCGAAGGAAGTCTTC : 198 gi|1353713 : TGAAGCCAGACCATACGAGTTCCCATGGCAGGTGTCCGTCCGAAGGAAGTCTTC : 195 gi|2707925 : TGAAGCGAGAGCGCACGAGTTTCCATGGACGGTGTCAGTCCGAAGGAGGTCAAG : 210 TGAAGCcAGAcCatACGAGTTcCCATGGcaGGTGTCcGTCCGAAGGAaGTCttc 220 * 240 * 260 * gi|2334519 : CGATTCCCATTTCTGCGGAGGTAGCATCATCAACGATCGTTGGGTTGTCTGCGC : 254 gi|1353713 : CGATTCCCATTTCTGCGGAGGTAGCATCATCAACGATCGTTGGGTTGTCTGCGC : 252 gi|1353713 : TGATTCCCATTTCTGCGGAGGTAGCATCATCAACGATCGTTGGGTTGTCTGCGC : 249 gi|2707925 : CGATTCTCATTTCTGCGGAGGCAGCATCATCAACGATCGTTGGATAATCACCGC : 264 cGATTCcCATTTCTGCGGAGGtAGCATCATCAACGATCGTTGGgTtgTCtgCGC
280 * 300 * 320 gi|2334519 : TGCTCACTGCATGCAGGGAGAGAGCCCTGCCCTGGTTTCATTGGTCGTCGGTGA : 308
gi|1353713 : TGCTCACTGCATGCAGGGAGAGGCCCCCGCTCTGGTTTCATTGGTCGTGGGTGA : 306 gi|1353713 : TGCTCACTGCATGCAGGGAGAGAGCCCTGCCCTGGTCTCATTGGTCGTCGGCGA : 303 gi|2707925 : TGCTCACTGCATGGTCGGAGAGAGCCCGGCTGGTGTTTCGATCGTCGTAGGCGA : 318 TGCTCACTGCATGcagGGAGAGagCCCGCctgGTtTCatTgGTCGTGGGA
760 * 780 * 800 * gi|2334519 : CAGCCTCATTGGTATTGTGTCTTGGGGAATCGGTTGCGCATCTGGCTATCCAGG : 791 gi|1353713 : CAGCCTGATTGGTATTGTGTCTTGGGGAATTGGTTGCGCTTCTGGCTATCCAGG : 786 gi|1353713 : CAGTCTGGGTGGCATTGTGTCTTGGGGAATTGGTTGCGCCTCTGGCTATCCAGG : 783 gi|2707925 : CAGTCTGATTGGAATCGTCTCTTGGGGAATTGCGTGCGCTTCTGGATATCCAGG : 801 CAGCTgatTGGATtGTgTCTTGGGGAATtGgtTGCGCTCTGGcTATCCAGG
820 * 840 * 860 gi|2334519 : AGTCTACGCCCGCGTCGGATCCCAAACTGGATGGATCACAGACATTATTACCAA : 845
gi|1353713 : AGTCTACTCCCGCGTCGGATTCCATGCTGCATGGATCACCGACATCATCACCAA : 840 gi|1353713 : AGTTTACTCCCGCGTCGGATTTCATGCTGGATGGATCACCGACACGATCACCAA : 837 gi|2707925 : AGTCTACTCTCGCGTCACCTACAACATTGATTGGATCACCACCACCATCGCAAA : 855 AGTcTACtCcCGCGTCggaTccAcTGaTGGATCACcgaCAATcaCcAA
* 880 * 900 * 9 gi|2334519 : CAACTAG--- : 852 gi|1353713 : CAACTAAACCGGAGTTATCCAGTCGACTATAACTGGAACGACTTTACCATCACG : 894 gi|1353713 : CAACTAAACCGACGATGGCCAGTCAACTATAACGGACTCTTATTACCTGCAGTT : 891 gi|2707925 : CAACTAAGCCT--- : 866
CAACTAa cc
Gambar IV.2. Daerah perancangan primer bagian maju dan mundur. Daerah yang berwarna hitam menunjukkan daerah lestari sedangkan daerah yang berwarna abu-abu dan putih menunjukkan daerah yang berbeda. Bagian (1) Daerah perancangan primer maju, Bagian (2) Daerah perancangan primer mundur.
IV.3 RNA Total L. rubellus
Isolasi RNA total asal cacing tanah L. rubellus dilakukan berdasarkan metode Schmitt et al. (1990) yang telah dimodifikasi dengan menggunakan cacing yang telah dihaluskan sebanyak 50 mg kemudian dimurnikan menggunakan phenol : chloroform.
Hasil elektroforesis (Gambar IV.3) menunjukkan RNA total hasil isolasi berupa 1 pita yang melebar. Hal ini disebabkan RNA total terdiri dari tRNA, rRNA, dan mRNA. Dalam penelitian ini yang menjadi target berikutnya adalah mRNA, yang (2)
pada umumnya berukuran pendek dan pada elektroforegram biasanya berupa pita yang terdapat pada bagian bawah elektroforesis.
Gambar IV.3. Hasil isolasi RNA total menggunakan penanda pUC19/Hinf1. Jalur 1: Isolasi RNA total, Jalur 2: penanda pUC19/Hinf1
RNA total hasil isolasi berukuran sekitar 200 sampai 500 pb (kurva kalibrasi lihat Lampiran B). Berdasarkan hasil pengukuran absorbansi pada panjang gelombang 260 dan 280 nm, berturut- turut 0,124 dan 0,066. Perbandingan A260/280 menunjukkan nilai perbandingan 1,87 dengan konsentrasi 4,85 µg/µl. Hal ini menunjukkan hasil isolasi memiliki kemurnian yang baik karena tingkat kemurnian yang baik berkisar antara 1,8–2,0 (Sambrook and Russel, 2001).
IV.4 Isolasi mRNA L. rubellus
Isolasi mRNA dilakukan dengan menggunakan mRNA isolation kit (data tidak ditampilkan). Perbandingan absorbansi pada panjang gelombang 260 nm dan 280 nm, adalah 1,8 yang menunjukkan kualitas mRNA yang baik. mRNA hasil isolasi ini digunakan sebagai templat dalam proses RT-PCR.
IV.5 DNA Kromosom L. rubellus
Isolasi DNA kromosom dilakukan menurut metode isolasi DNA kromosom pada ekor tikus (Sambrook and Russell, 2001) yang telah dimodifikasi. Proses isolasi
1 2
1419 pb
517 pb
214 pb 394 pb
RNA total ini menggunakan cacing yang telah dihaluskan sebanyak 50 mg kemudian dimurnikan menggunakan phenol : chloroform : isoamilalkohol. DNA kromosom pada cacing tanah L. Rubellus berada di sekitar 21.226 pb (Wulan, 2006)(Gambar IV.4).
Gambar IV.4. Hasil elektroforesis isolasi DNA kromosom menggunakan penanda DNAλ/ Hind III. Bagian 1: Hasil elektroforesis isolasi DNA kromosom setelah lisis, Jalur 1, 2, 3 : isolasi DNA kromosom, Jalur 4: penanda DNAλ/ Hind III, Bagian 2: Hasil elektroforesis isolasi DNA kromosom setelah pemurnian menggunakan phenol: chloroform: isoamilalkohol, Jalur 1, 2, 3: isolasi DNA kromosom, Jalur 4: penanda DNAλ/ Hind III.
Elektroforesis dilakukan setelah hasil isolasi dimurnikan terlebih dahulu. Namun untuk mengamati apakah setelah tahap lisis terdapat DNA kromosom, maka dilakukan pencuplikan sampel untuk pengujian elektroforesis terlebih dahulu, selanjutnya sisa sampel yang lain dimurnikan. Setelah elektroforesis tahap awal dilakukan menunjukkan bahwa terdapat DNA kromosom, yang siap untuk dimurnikan. Berdasarkan hasil pengukuran absorbansi pada panjang gelombang 260 dan 280 nm, berturut- turut 0,501 dan 0,227. Apabila dibandingkan antara absorbansi A260/280 menunjukkan nilai perbandingan 2,207. Hal ini menunjukkan hasil isolasi memiliki kemurnian yang baik karena tingkat kemurnian yang baik berkisar antara 1,8–2,0 (Sambrook and Russel, 2001).
IV.6 Amplifikasi Gen Lumbrokinase dengan Berbagai Templat
23.130 pb 9.416 pb 6557 pb 4361 pb 2.322 pb 564 pb DNA kromosom 2 3 4 1 RNA total 1 2 3 2.042 pb 4 1 2
Seperti telah diulas sebelumnya, untuk mengetahui fragmen gen pengkode lumbrokinase asal cacing tanah L. rubellus dilakukan dengan berbagai pendekatan, diantaranya dengan melakukan proses RT-PCR menggunakan templat RNA total dan mRNA, yang menunjukkan hasil elektroforesis bahwa pita berada di daerah 322 pb (Gambar IV.5) (kurva kalibrasi lihat Lampiran B). RT-PCR menggunakan enzim AMV reverse transcriptase yang bekerja pada sistem RT untuk menghasilkan cDNA dan Taq polimerase bekerja pada sistem PCR. Enzim AMV reverse transcriptase bekerja pada rentang suhu 42 sampai 60 oC.
Gambar IV.5. Elektroforesis amplifikasi gen pengode lumbrokinase dengan menggunakan templat RNA total dan mRNA. Jalur 1: marker DNAλ/ Hind III, jalur 2: PCR templat RNA total dengan primer ZN1 dan ZN2, jalur 3: PCR templat RNA total dengan primer FL1 dan FL2 (Wulan, 2006), jalur 4: RT-PCR templat mRNA dengan primer ZN1 dan ZN2.
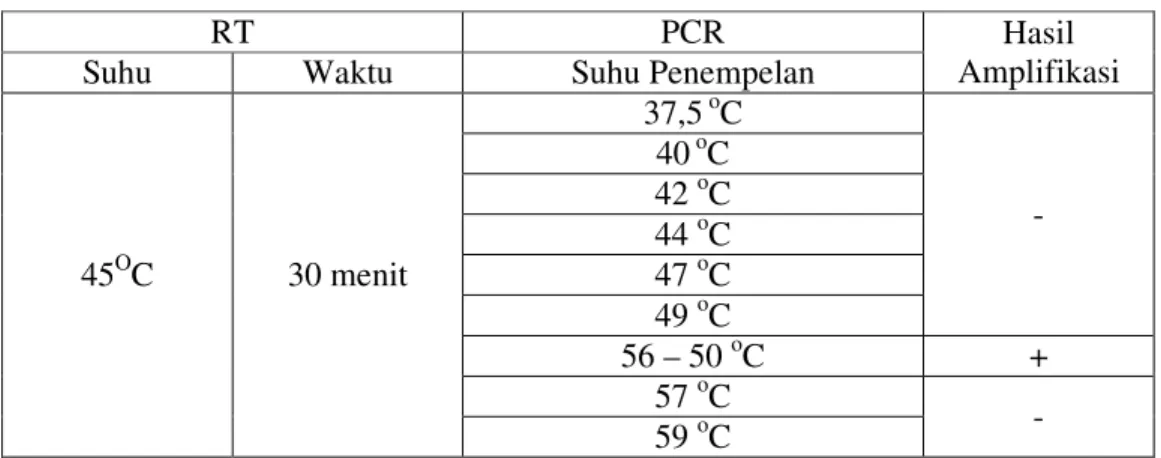
Pada penelitian ini dilakukan berbagai variasi kondisi RT-PCR, yang diawali dengan menggunakan suhu penempelan 37,5 oC dan suhu RT 45 oC, namun hasil elektroforesis tidak menunjukkan adanya pita. Kemudian dilakukan berbagai variasi suhu RT-PCR (Tabel. IV.1). Proses amplifikasi menggunakan templat RNA total dan mRNA dengan RT-PCR meliputi proses transkripsi balik untuk memproduksi cDNA, yang selanjutnya akan digunakan sebagai templat dalam proses PCR. Dalam penelitian ini digunakan metode touchdown PCR, yaitu suhu penempelan menurun secara bertahap. Pada penelitian diperoleh suhu penempelan (56–50)oC dengan menggunakan primer ZN1 dan ZN2. Sedangkan pada
1 2 3 4 23.130 pb 6557 pb 9.416 pb 4361 pb 2.027 pb 564 pb 2322 pb
penggunaan primer FL1 dan FL2 memiliki suhu penempelan 65 sampai 59 oC dengan suhu RT pada 45 oC dan 55 oC (Wulan, 2006).
Tabel IV.1 Variasi Suhu RT-PCR
RT PCR Hasil
Amplifikasi
Suhu Waktu Suhu Penempelan
45OC 30 menit 37,5 oC - 40 oC 42 oC 44 oC 47 oC 49 oC 56 – 50 oC + 57 oC - 59 oC
Hasil amplifikasi yang sangat rendah diduga karena primer tidak menempel secara spesifik, sehingga pita yang dihasilkan tidak lengkap. Amplifikasi dengan menggunakan templat mRNA menunjukkan pita yang sangat tipis, berada di daerah ~300 pb, diduga karena konsentrasi mRNA sangat rendah, akibat mRNA sudah terdegradasi dan mRNA memiliki waktu paruh yang sangat pendek.
Selain menggunakan templat RNA total dan mRNA, proses amplifikasi gen lumbrokinase juga dilakukan dengan menggunakan templat DNA kromosom, yang diharapkan dapat menghasilkan pita yang berukuran lebih tinggi, karena proses amplifikasi dengan menggunakan templat DNA kromosom masih memiliki ekson dan intron. Hasil PCR dengan menggunakan templat DNA kromosom yang telah dielektroforesis menunjukkan pita yang berada di daerah 305 pb (Gambar IV.6). Pita yang smear dan kurang jelas ini diduga karena produk dan templat yang tidak spesifik dan terlalu banyak.
PCR menggnakan primer FL1 dan FL2 (Wulan, 2006) digunakan sebagai pembanding. Penelitian sebelumnya, menunjukkan suhu penempelan pada 59OC pada PCR menggunakan templat DNA kromosom (Gambar IV. 6). Hasil yang serupa juga diperoleh dari amplifikasi dengan menggunakan primer ZN1 dan ZN2
(Gambar IV.7). Dalam penelitian ini, digunakan MgCl2 2mM dan 3 mM dengan suhu penempelan 50 oC.
Gambar IV.6. Elektroforesis amplifikasi gen pengode lumbrokinase dengan menggunakan templat DNA kromosom primer FL1. Jalur 1: penanda pUC19/Hinf1, jalur 2: PCR templat DNA kromosom dengan primer FL1 dan FL2 (Wulan, 2006), jalur 3: PCR ulang templat DNA kromosom dengan primer FL1 dan FL2 (Wulan, 2006).
Pada tahap awal, digunakan berbagai variasi kondisi suhu penempelan, yang dimulai pada suhu 37,5OC, namun hasil elektroforesis tidak menunjukkan adanya pita. Kemudian dilakukan variasi suhu PCR (Tabel IV.2).
Tabel IV.2 Variasi Suhu PCR
Suhu Penempelan Hasil Amplifikasi
37,5 oC - 40 oC 42 oC 44 oC 46 oC 48 oC 50 oC + 52 oC - 54 oC
Amplifikasi dengan menggunakan primer ZN1 dan ZN2 menunjukkan pita sebesar 338 pb (kurva kalibrasi lihat Lampiran B). Berdasarkan hasil tersebut, diduga akibat konsentrasi templat yang terlalu rendah dan mungkin juga akibat primer yang tidak menempel secara spesifik.
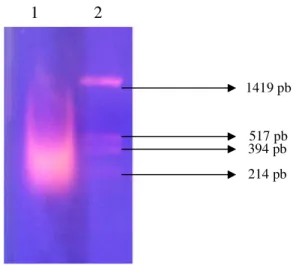
517 pb 394 pb 214 pb 1419 pb
Gambar IV.7. Elektroforesis amplifikasi gen pengkode lumbrokinase dengan menggunakan templat DNA kromosom primer ZN1. Jalur 1: DNAλ/ Hind III, jalur 2: PCR templat DNA kromosom dengan primer ZN1 dan ZN2 konsentrasi MgCl2 2mM, jalur 3: PCR templat DNA kromosom dengan primer ZN1 dan ZN2 konsentrasi MgCl2 3mM.
Dalam penelitian ini, dilakukan amplifikasi gen lumbrokinase dengan berbagai templat menunjukkan nilai yang berbeda, namun secara keseluruhan ketiganya memiliki ukuran yang sama yaitu berkisar 300 pb (kurva kalibrasi lihat Lampiran B).
IV.7 Penentuan Urutan Hasil Amplifikasi Gen Lumbrokinase
Penentuan urutan nukleotida dilakukan pada hasil amplifikasi yang menggunakan templat RNA total primer ZN1 dan ZN2 serta FL1 dan FL2. Urutan nukleotida mengunakan templat RNA total primer ZN1 (Lampiran C) dan primer FL1 (Lampiran D) menunjukkan urutan nukleotida yang dihasilkan tidak sesuai dengan urutan nukleotida gen pengkode lumbrokinase yang terdapat pada database Genebank Hal ini diduga karena hasil amplifikasi merupakan campuran dan primer tidak menempel secara spesifik.
Apabila ditinjau dari taksonomi, cacing tanah L. rubellus termasuk dalam kelompok famili Lumbricidae. Dimana dalam famili Lumbricidae ini terdapat berbagai jenis spesies cacing, meliputi Allolobophora sp., Aporrectodea caliginosa, Aporrectodea trapezoids, Aporrectodea tuberculata, Aporrectodea
1 2 3 23.130 pb 9.416 pb 6557 pb 2.322 pb 4361 pb 564 pb 2.027 pb
rubidus tenuis , Eisenia andrei, Eisenia fetida, dan Eisenia japonica (Blakemore, 2003).
Bab V Kesimpulan dan Saran