Korelasi Jarak Genetik Berbasis Marka Mikrosatelit Inbrida Jagung
dengan Bobot Biji F1
M.B. Pabendon1, M.J. Mejaya2, J. Koswara3, dan H. Aswidinnoor3 1Balai Penelitian Tanaman Serealia
Jl. Dr. Ratulangi Nomor 274, Maros 90514
2Balai Penelitian Tanaman Kacang-Kacangan dan Umbi-Umbian Jl. Kendalpayak, Kotak Pos 66 Malang, Jawa Timur
3Institut Pertanian Bogor (IPB) Jl. Dramaga, Bogor
ABSTRACT. Correlation between Genetic Distances based on Microsatellite Marker in Maize Inbred with Seed Weight of F1. This study was aimed to determine: (a) the correlation between the value of microsatellite marker-based genetic distances with maize seed yield of F1 testcross, and (b) the effectiveness of Mr14 and Mr4 inbred as testers on maize phenotypic seed weight performances. The trial was conducted in two stages. The first stage was the formation of two sets of F1 single crosses, one set using Mr4 inbred tester and another set using Mr14 inbred tester, each tester was crossed with 32 maize lines. Each set contained 32 F1 testcrosses. The values of genetic distances of the F1 testcrosses were obtained by DNA-based characterization using the microsatellite markers. The second stage was to test yield performances of the F1 testcrosses of each set derived from Mr4 and Mr14. Results of the trial showed that one F1 testcross with Mr4 tester and one F1 testcross with Mr14 tester produced seed weights per plant significantly higher than that of cultivar Bisma 1 (control variety). The two genotypes of the F1 testcrosses were P5/GM26-9 x Mr4 and Bisma-3-1 x Mr14, each produced 179.1 and 178.5 g seed weight/plant, respectively. The genetic distances of both F1 testcrosses were 0.82 and 0.84, respectively, where as in the Bima1 hybrid was 0.65. The two F1 testcrosses indicate potential as new testers replacing the Mr14 and Mr4 inbred testers. The coefficient of correlations between genetic distances and seed weights of P5/GM26-9 x Mr4 and Bisma-3-1 x Mr14 inbred were 0.81 and 0.76, respectively. These values are quite high, suggesting that the microsatellite markers can be used to test maize genotypes to obtain better maize testers than Mr4 testers and Mr14. Keywords: Microsatellite marker-based genetic distances, maize
inbred testers, yield potentials
ABSTRAK. Penelitian ini bertujuan untuk mengetahui: (a) korelasi antara nilai jarak genetik berbasis marka mikrosatelit dengan bobot biji F1 jagung inbrida hasil silang uji (testcross), dan (b) efektivitas inbrida Mr4 dan Mr14 sebagai inbrida penguji (tester) terhadap penampilan fenotipik (bobot biji). Penelitian dilakukan di lapangan dalam dua tahap. Tahap pertama adalah pembentukan dua set F1 silang tunggal, masing-masing dengan inbrida penguji Mr4 dan Mr14 yang disilangkan dengan 32 galur jagung, sehingga masing-masing set materi menghasilkan 32 genotipe F1 silang tunggal. Nilai jarak genetik diperoleh dengan melakukan karakterisasi DNA berbasis marka mikrosatelit. Tahap kedua adalah uji daya hasil jagung silang tunggal dari masing-masing set (Mr4 dan Mr14). Hasil penelitian menunjukkan bahwa satu genotipe silang tunggal F1 dari hasil persilangan dengan inbrida Mr4 dan satu genotipe F1 dari hasil persilangan dengan inbrida Mr14 memiliki bobot biji per tanaman yang secara nyata lebih tinggi dibanding dengan bobot biji pada varietas pembanding Bima1. Kedua genotipe silang tunggal F1 tersebut adalah P5/GM26-9 x Mr4 dan Bisma-3-1 x Mr14, dengan bobot biji masing-masing 179,1 g/tanaman dan 178,5 g/tanaman.
Nilai jarak genetik kedua genotipe silang tunggal F1 adalah masing-masing 0,82 dan 0,84, sedangkan jarak genetik hibrida Bima1 adalah 0,65. Kedua genotype inbrida tersebut berpotensi sebagai inbrida penguji baru, masing-masing menggantikan inbrida penguji Mr14 dan Mr4. Nilai korelasi antara jarak genetik dengan bobot biji genotipe F1 hasil silang uji adalah 0,81 dan 0,76, masing masing pada persilangan 32 inbrida dengan inbrida penguji Mr4 dan Mr14. Nilai ini tergolong tinggi, sehingga marka mikrosatelit dapat digunakan untuk menguji inbrida jagung lain guna mendapatkan inbrida yang lebih potensial daripada inbrida penguji Mr4 dan Mr14.
Kata kunci: Jarak genetik, inbrida penguji, potensi hasil
P
emuliaan jagung hibrida termasuk pembentukan galur dan evaluasi kemampuan daya gabung hasil persilangan yang dibentuk berdasarkan karakter yang ingin dikembangkan. Identifikasi kombinasi tetua yang menghasilkan hibrida superior merupakan tahapan yang sangat penting dalam pengembangan hibrida. Kegiatan ini merupakan tahapan yang paling mahal dan membutuhkan banyak waktu, seperti penyilangan inbrida yang satu dengan lainnya dan mengevaluasi hibrida secara ekstensif di lapangan. Penampilan galur inbrida jagung tidak menggambarkan hasil biji hibrida jagung (Hallauer and Miranda 1988). Oleh karena itu, kemampuan memprediksi nilai hibrida silang tunggal atau heterosis antara tetua galur inbrida berperan penting dalam meningkatkan efisiensi pemuliaan hibrida, terutama jika dapat dilakukan sebelum persilangan.Permintaan jagung akan terus meningkat sehingga potensi hasil tinggi dari calon varietas selalu menjadi prioritas utama. Oleh sebab itu, jika seleksi tetua menggunakan metode silang uji maka materi penguji harus mempunyai kemampuan daya gabung yang tinggi terhadap berbagai karakter. Dalam beberapa studi, efek daya gabung umum (DGU) untuk tetua dan efek daya gabung khusus (DGK) untuk persilangan telah diestimasi pada jagung (Dehghanpour et al. 1996; San-Vicente et
al. 1998; Konak et al. 1999; Chaudhary et al. 2000; Araujo et al. 2001; Kalla et al. 2001). Derajat heritabilitas
bervariasi dari rendah sampai sedang untuk hasil biji (Deghanpour et al. 1996; Singh et al. 1998).
Efek heterosis yang besar ditentukan oleh penampilan kedua tetua dan hibrida. Penampilan tetua akan berbeda antarlingkungan, dan besarnya heterosis kemungkinan akan memperlihatkan variasi yang sama. El-Haddad (1975) dan Uddin et al. (1992) menyarikan hasil studi pendahuluan yang mengindikasikan terjadinya heterosis nyata pada heterosis rata-rata tetua dan heterosis tetua tertinggi untuk hasil dan beberapa karakter agronomi.
Jumlah pengujian molekuler yang tersedia untuk studi keragaman tanaman meningkat secara dramatis, masing-masing metode berbeda dalam prinsip, aplikasi, tipe dan jumlah polimorfisme yang terdeteksi, termasuk biaya dan waktu yang dibutuhkan (Tanksley and McCouch 1997). Menurut Warburton et al. (2005), alat bantu marka molekuler bermanfaat dalam menyaring kelompok heterotik dan menyeleksi materi penyaji representatif yang akan digunakan dalam program pemuliaan hibrida. Single Sequence Repeats (SSRs) atau biasa disebut mikrosatelit merupakan unit pengulangan 1-6 pasang basa DNA dengan variasi yang tinggi (Gupta
et al. 1996; Senior et al. 1998). Primer SSR dibentuk
berdasarkan daerah pengapit konservatif (conserved
flanking region) lokus SSR (Akkaya et al. 1992), yang bisa
menghasilkan amplifikasi PCR (Polymerase Chain
Reaction) pada lokus SSR tersebut. Hasil produk PCR
dapat dielektroforesis yang dibedakan menurut jumlah unit pengulangan DNA dalam alel-alel SSR yang muncul dan menghasilkan polimorfisme yang tinggi antarspesies (Senior and Heun 1993; Taramino and Tingey 1996; Senior et al. 1998), dan yang lebih penting adalah antarindividu dalam spesies dan populasi (Gupta
et al. 1996; Chen et al. 1997). Modifikasi jarak Roger telah
digunakan dalam menentukan jarak genetik antarsejumlah galur inbrida jagung dataran rendah tropis (Xia et al. 2004), serta galur jagung inbrida dan populasi CIMMYT (Warburton et al. 2002).
Tujuan penelitian ini adalah untuk mengetahui (a) posisi inbrida Mr4 dan Mr14 sebagai inbrida penguji terhadap hasil (bobot biji) berdasarkan marka mikrosatelit, dan (b) kegunaan marka molekuler melalui korelasi antara nilai jarak genetik berbasis marka mikrosatelit dengan bobot biji genotipe jagung hasil silang uji.
BAHAN DAN METODE
Materi Genetik
Untuk pembuatan materi silang uji digunakan 32 inbrida dan dua materi penguji (Mr4 dan Mr14). Uji daya hasil silang uji terdiri atas dua set genotipe hasil silang uji yaitu 32 genotipe silang tunggal dengan inbrida penguji Mr4
dan 32 genotipe silang tunggal dengan inbrida penguji Mr14. Masing-masing set materi menggunakan tiga varietas pembanding yaitu Bima 1, Bisi 2, dan Semar 10.
Karakterisasi Genetik untuk Penentuan Nilai Jarak Genetik
Protokol karakterisasi genetik berbasis marka SSR meliputi ekstraksi DNA, amplifikasi, elektroforesis, dan visualisasi gel mengikuti prosedur yang digunakan oleh George et al. (2004) dengan sedikit modifikasi, yaitu mengganti penggunaan cairan nitrogen dengan buffer CTAB (Khan et al. 2004). Proses ini lebih sederhana dan lebih murah tanpa mengurangi kualitas DNA yang diperoleh. Proses amplifikasi menggunakan mesin PTC-100 Programmable Thermal Controller (MJ Research, Waltham, Mass). Primer dibeli dari Research Genetics (Huntsville, Ala). Fenotipe SSR diskoring sebagai data biner yaitu 1 jika ada pita dan 0 jika tidak ada pita, atau 9 jika pita yang muncul meragukan. Jika pita yang muncul meragukan maka diskoring sebagai missing data. Data biner akan digunakan dalam analisis data molekuler untuk mengestimasi nilai jarak genetik.
Pembentukan Hibrida F1
Pembentukan dua set hibrida F1 dilaksanakan di KP Maros, Balitsereal, dilakukan dengan metode silang uji, menggunakan dua penguji (tester) atau sebagai tetua jantan, yaitu inbrida Mr4 dan Mr14. Materi genetik pasangan persilangan (tetua betina) terdiri atas 32 inbrida harapan. Benih ditanam dengan jarak 75 cm x 20 cm dan panjang baris 5 m. Setiap nomor ditanam dua biji per lubang tanam. Sebagai penguji yang akan menjadi sumber tepung sari 32 inbrida, kedua genotipe masing ditanam dalam dua petak, masing-masing terdiri atas empat baris. Sebagai tetua betina (32 inbrida), setiap nomor ditanam dua baris dengan jarak tanam 75 cm x 20 cm dan panjang baris 5 m.
Takaran pupuk yang diberikan 350 kg urea, 200 kg SP36, dan 100 kg KCl/ha. Pemupukan dilakukan dua kali, pertama pada saat tanam, yaitu setengah takaran pupuk urea dan seluruh pupuk SP36 dan KCl. Pemupukan kedua dilakukan pada umur 30 hari setelah tanam (HST), yaitu setengah takaran pupuk urea yang tersisa. Penjarangan tanaman dilakukan pada umur 7-10 HST dengan meninggalkan satu tanaman per lubang tanam. Pemeliharaan tanaman sesuai dengan rekomendasi dan kondisi di lapangan, antara lain penyiangan, pengairan, dan pembumbunan.
Sebelum keluar bunga jantan dan bunga betina, kantong penutup untuk masing-masing bunga sudah dipersiapkan. Untuk menghindari kontaminasi bunga betina, pada saat tongkol sudah muncul segera
Tingkat kemiripan genetik (GS) diestimasi dari data jumlah alel dengan menggunakan koefisien Jaccard (Rohlf 2000) dengan formula:
(
n
u
)
m
S
+
=
, di manam = jumlah pita (alel) DNA yang sama posisinya, n = total pita (alel) DNA, dan
u = jumlah pita (alel) DNA yang tidak sama posisinya. Kemiripan genetik dianalisis dengan menggunakan program komputer NTSYS-pc versi 2.1 (Rohlf 2000). Analisis matriks jarak genetik diperoleh dari hasil analisis kemiripan genetik (Lee 1998) dengan formula:
S = 1 – GS di mana
S = jarak genetik,
GS = Kemiripan genetik (Genetic Similarity).
Principal Coordinate Analysis (PCoA) digunakan
untuk mengetahui posisi relatif dari masing-masing hibrida dan tetuanya. Komponen utama dari peubah data asal diperoleh dari matriks varians-kovarians peubah asalnya (Dillon and Goldstein 1984).
Analisis data lapangan menggunakan program IRRI-stat. Koefisien korelasi (r) antara jarak genetik berdasarkan marka mikrosatelit dan bobot biji menggunakan analisis korelasi sederhana.
Analisis data lapangan mengikuti rancangan alpha latis, sedangkan analisis korelasi sederhana dilakukan untuk melihat hubungan antara nilai jarak genetik berbasis marka SSR dengan data fenotipik hibrida F1 hasil biji, rendemen biji, dan bobot 1.000 biji. Jika nilai r positif berarti data molekuler didukung oleh data fenotipik, namun jika nilai r negatif maka data molekuler tidak didukung oleh data fenotipik.
HASIL DAN PEMBAHASAN
Nilai jarak genetik antara masing-masing galur dengan materi penguji dalam dua set persilangan disajikan pada Tabel 1. Pada set genotipe F1 hasil silang uji dengan Mr4, nilai jarak genetik berkisar antara 0,62-0,89. Nilai jarak genetik terendah adalah pada persilangan P5/GM26-22xMr4 yang menghasilkan bobot biji 106,8 g/tanaman, sedangkan tertinggi pada persilangan MKB24xMr4 dengan bobot biji 169,2 g/tanaman. Bobot biji terendah adalah 96,0 g/tanaman dengan jarak genetik 0,70, dan bobot biji tertinggi 179,1 g/tanaman dengan jarak genetik 0,82. Dalam hal ini terdapat 11 genotipe F1 yang berbeda nyata dengan varietas pembanding Semar 10 dan Bima 1. disungkup dengan kantong sebelum rambut tongkol
keluar untuk menghindari benangsari yang tidak diinginkan jatuh pada rambut tongkol. Untuk menghindari kontaminasi pada bunga jantan (malai), pada saat malai sudah siap disilangkan segera disungkup dengan kantong kertas yang sudah disiapkan, maksimal 24 jam sebelum disilangkan.
Pada saat panen, masing-masing set persilangan dipanen secara terpisah, dikeringkan, kemudian disimpan dalam kantong kertas untuk digunakan pada musim berikutnya. Hal ini bertujuan untuk melihat penampilan fenotipik masing-masing genotipe yang akan dikorelasikan dengan nilai jarak genetik masing-masing pasangan.
Uji Daya Hasil Pendahuluan F1 Hasil Silang Uji
Pengujian dilakukan di KP Bajeng, Balitsereal, Sulawesi Selatan. Jumlah perlakuan sebanyak 35 genotipe terdiri atas 32 hibrida silang tunggal dan tiga kultivar pembanding, yaitu Semar 10, Bisi 2, dan Bima 1. Tata letak di lapangan menggunakan rancangan alpha latis 7 x 6 diulang dua kali. Jarak tanam 75 cm x 20 cm, panjang baris 5 m, masing-masing genotipe terdiri atas dua baris. Pupuk yang digunakan pada semua kegiatan di lapangan sesuai dengan yang direkomendasikan untuk jagung, yaitu 300 kg urea, 200 kg SP36, dan 100 kg KCl/ha. Pemberian urea dilakukan dua kali, pertama pada umur satu minggu setelah tanam (150 kg urea/ha), bersamaan dengan semua pupuk SP36 dan KCl. Pemupukan urea kedua (150 kg/ha) pada umur 30 HST.
Data yang dikumpulkan adalah umur 50% berbunga, tinggi tanaman, tinggi letak tongkol, skoring hama dan penyakit yang menyerang, skoring penampilan tanaman dan tongkol, kadar air biji saat panen, jumlah tongkol panen per petak, bobot tongkol per petak, rendemen biji, dan komponen hasil (panjang tongkol, diameter tongkol, jumlah baris per tongkol, jumlah biji per baris, dan bobot 1.000 biji).
Analisis Data
Analisis data molekuler dilakukan berdasarkan hasil skoring pita DNA yang muncul pada plate. Skoring dilakukan dengan cara:jika ada pita diberi skor satu (1) dan jika tidak ada pita diberi skor nol (0). Hasil skoring dalam bentuk data biner. Tingkat polimorfisme (PIC) dari primer yang digunakan dihitung untuk masing-masing marka SSR (Smith et al. 1997), dengan formula:
∑
−
=
nf
iPIC
1 21
i = 1, 2, 3,………n,kecil jika pengujinya inbrida Mr4 dan Mr14. Oleh sebab itu, jika potensi hasil merupakan tujuan utama maka perlu dipertimbangkan untuk mencari inbrida baru dengan potensi daya gabung yang lebih tinggi dari inbrida penguji Mr4 dan Mr14.
Dalam penelitian ini hasil ketiga varietas pembanding yang masing-masing adalah 9 t/ha untuk Semar 10, 12 t/ha untuk Bisi 2, dan 9 t/ha untuk Bima 1. Semar 10 adalah varietas hibrida silang tiga jalur, sedangkan Bisi 2 dan Bima 1 merupakan hibrida silang tunggal.
Pada set genotipe F1 hasil silang uji dengan Mr14, nilai jarak genetik berkisar antara 0,52-0,87. Nilai jarak genetik tersebut diperoleh dari hasil persilangan Bisma137xMr14 yang menghasilkan bobot biji 76,0 g/ tanaman, sedangkan nilai jarak genetik tertinggi pada persilangan P5/GM25-42xMr14 menghasilkan bobot biji 146,0 g/tanaman. Bobot biji terendah adalah 76,0 g/ tanaman dengan jarak genetik 0,52, dan bobot biji tertinggi adalah 178,5 g/tanaman dengan jarak genetik 0,84. Dalam hal ini terdapat sembilan genotipe F1 yang berbeda nyata dengan pembanding Semar 10 dan Bima 1.
Pada kedua set persilangan, pasangan persilangan dengan bobot biji tertinggi terdapat pada P5/GM26-9xMr4 dan Bisma-3-1xMr14, berbeda nyata dengan pembanding Bima 1. Tidak terdapat genotipe F1 yang berbeda nyata dengan pembanding Bisi 2, baik pada hibrida hasil silang uji dengan inbrida penguji Mr4 maupun pada hibrida hasil silang uji dengan inbrida penguji Mr14.
Tidak ditemukan genotipe yang berbeda nyata dengan Bisi 2, baik pada hibrida hasil silang tunggal dengan inbrida Mr4 maupun dengan inbrida Mr14. Hal ini wajar karena inbrida penguji Mr4 dan Mr14 diperoleh hanya melalui seleksi fenotipik. Genotipe F1 yang berbeda nyata dengan Bima1 juga sangat rendah, hanya satu pada masing-masing set persilangan. Berdasarkan hasil pengujian secara fenotipik selama lima tahun, inbrida Mr4 dan Mr14 mempunyai kemampuan daya gabung yang tinggi, sehingga dipilih sebagai tetua penguji (Mejaya et al. 2005). Namun pengujian tersebut dilakukan lebih dari 10 tahun yang lalu dan sampai sekarang belum ada inbrida penguji lain yang digunakan. Di lain pihak, dengan tingginya permintaan, banyak varietas-varietas baru yang dilepas oleh berbagai perusahaan perbenihan, terutama dari swasta dengan potensi hasil yang sangat tinggi berkisar antara 12-13 t/ha.
Nilai jarak genetik genotipe hasil silang uji dengan Mr4 yang berbeda nyata dengan varietas pembanding adalah 0,80 dengan bobot biji rata-rata 158,61 g/ tanaman, sedangkan nilai jarak genetik genotipe hasil silang tunggal yang tidak berbeda nyata dengan varietas Inbrida penguji Mr4 dan Mr14 adalah tetua Bima 1.
Dari karakterisasi marka SSR diperoleh nilai jarak genetik Mr4 dengan Mr14 sebesar 0,65 yang tergolong sedang. Dengan demikian, peluang untuk memperoleh hibrida yang lebih baik dari varietas hibrida pembanding relatif
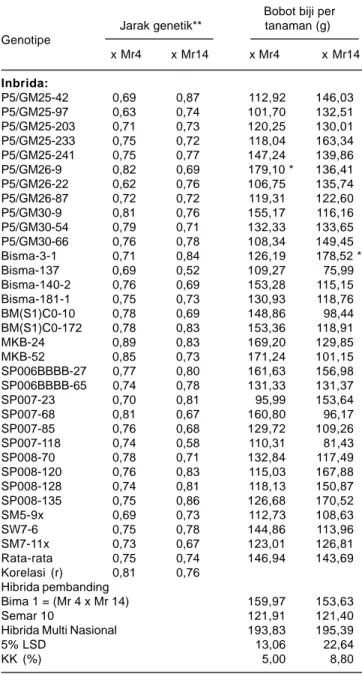
Tabel 1. Jarak genetik dan bobot biji per tanaman hibrida F1 hasil silang uji 32 inbrida dengan penguji inbrida Mr4 dan Mr14, KP. Bajeng, Sulawesi Selatan 2006
Bobot biji per Jarak genetik** tanaman (g) Genotipe x Mr4 x Mr14 x Mr4 x Mr14 Inbrida: P5/GM25-42 0,69 0,87 112,92 146,03 P5/GM25-97 0,63 0,74 101,70 132,51 P5/GM25-203 0,71 0,73 120,25 130,01 P5/GM25-233 0,75 0,72 118,04 163,34 P5/GM25-241 0,75 0,77 147,24 139,86 P5/GM26-9 0,82 0,69 179,10 * 136,41 P5/GM26-22 0,62 0,76 106,75 135,74 P5/GM26-87 0,72 0,72 119,31 122,60 P5/GM30-9 0,81 0,76 155,17 116,16 P5/GM30-54 0,79 0,71 132,33 133,65 P5/GM30-66 0,76 0,78 108,34 149,45 Bisma-3-1 0,71 0,84 126,19 178,52 * Bisma-137 0,69 0,52 109,27 75,99 Bisma-140-2 0,76 0,69 153,28 115,15 Bisma-181-1 0,75 0,73 130,93 118,76 BM(S1)C0-10 0,78 0,69 148,86 98,44 BM(S1)C0-172 0,78 0,83 153,36 118,91 MKB-24 0,89 0,83 169,20 129,85 MKB-52 0,85 0,73 171,24 101,15 SP006BBBB-27 0,77 0,80 161,63 156,98 SP006BBBB-65 0,74 0,78 131,33 131,37 SP007-23 0,70 0,81 95,99 153,64 SP007-68 0,81 0,67 160,80 96,17 SP007-85 0,76 0,68 129,72 109,26 SP007-118 0,74 0,58 110,31 81,43 SP008-70 0,78 0,71 132,84 117,49 SP008-120 0,76 0,83 115,03 167,88 SP008-128 0,74 0,81 118,13 150,87 SP008-135 0,75 0,86 126,68 170,52 SM5-9x 0,69 0,73 112,73 108,63 SW7-6 0,75 0,78 144,86 113,96 SM7-11x 0,73 0,67 123,01 126,81 Rata-rata 0,75 0,74 146,94 143,69 Korelasi (r) 0,81 0,76 Hibrida pembanding Bima 1 = (Mr 4 x Mr 14) 159,97 153,63 Semar 10 121,91 121,40
Hibrida Multi Nasional 193,83 195,39
5% LSD 13,06 22,64
KK (%) 5,00 8,80
Angka bobot biji per tanaman yang diikuti notasi * berarti berbeda nyata lebih tinggi terhadap hibrida pembanding Bima1, sedangkan yang tidak mempunyai notasi berarti lebih kecil atau tidak berbeda nyata terhadap pembanding Bima1; r adalah korelasi antara jarak genetik dan bobot biji F1 hasil silang uji dengan Mr4 dan Mr14. Nilai jarak genetik anatara Mr4 dan Mr14 sebesar 0,70. **Sumber:Laboratorium Biologi Molekuler, Balitsereal (2007).
lebih kecil, yaitu 0,76 dengan bobot biji rata-rata 120,4 g/ tanaman. Nilai jarak genetik genotipe hasil silang uji dengan Mr14 yang berbeda nyata dengan varietas pembanding adalah 0,81 dengan bobot biji rata-rata 159,7 g/tanaman, sedangkan nilai jarak genetik genotipe hasil silang uji yang tidak berbeda nyata dengan varietas pembanding juga lebih kecil, yaitu 0,72 dengan bobot biji rata-rata 111,3 g/tanaman (Tabel 2).
Bobot biji tertinggi yang diperoleh dari kedua set silang uji yaitu P5/GM26-9xMr4 (179,10 g/tanaman) dan Bisma-3-1xMr14 masing-masing adalah 179,1 g dan 178,5 g/tanaman, berbeda nyata dengan pembanding Bima 1. Jika bobot biji yang diperoleh kedua hibrida tersebut diekstrapolasi ke dalam ton/ha dengan populasi 66.000 tanaman/ha maka diperoleh hasil masing-masing 10,8 dan 10,7 t/ha. Nilai jarak genetik hibrida Bima 1 (Mr4xMr14) adalah 0,70 dengan bobot biji antara set-1 dan set-2 rata-rata 156,8 g/tanaman. Jika bobot biji varietas Bima1 diekstrapolasi ke dalam t/ha dengan populasi 66.000 tanaman/ha maka diperoleh hasil 10,4 t/ha. Hasil ekstrapolasi Bima 1 lebih rendah dari kedua hibrida terdahulu. Dengan demikian kedua inbrida tersebut mempunyai peluang untuk diuji lebih lanjut sebagai inbrida penguji yang baru, sebagai alternatif untuk memperbaiki atau meningkatkan kualitas inbrida penguji. Selain itu perlu dilakukan eksplorasi yang lebih jauh untuk mencari genotipe yang mampu bersaing dengan varietas unggul baru berpotensi hasil tinggi.
Salah satu harapan dari penelitian ini adalah mendapatkan konfirmasi yang tegas bahwa dengan nilai jarak genetik jauh akan diperoleh hasil yang tinggi dan sebaliknya. Ada beberapa data yang tidak konsisten. Misalnya pada kedua set silang uji, bobot tertinggi tidak diperoleh dari pasangan persilangan dengan nilai jarak genetik tertinggi. Namun secara umum menunjukkan kecenderungan bahwa nilai jarak genetik rendah
menghasilkan bobot biji yang rendah dan nilai jarak genetik tinggi menghasilkan bobot biji yang tinggi (Tabel 2). Bobot biji genotipe F1 yang berbeda nyata dengan varietas pembanding mempunyai nilai jarak genetik yang lebih tinggi dibandingkan bobot biji genotipe F1 yang tidak berbeda nyata dengan varietas pembanding. Nilai korelasi (r) antara jarak genetik vs bobot biji F1 hasil silang uji dengan Mr4 dan Mr14 masing-masing 0,81 dan 0,76, tergolong sedang sampai tinggi. Nilai korelasi rata-rata dari kedua set genotipe silang tunggal tergolong sedang sampai tinggi, artinya kadang-kadang ditemukan nilai jarak genetik tinggi tetapi bobot biji lebih rendah. Sebaliknya, nilai jarak genetik sedang tetapi bobot biji tinggi, namun secara umum dari nilai jarak genetik sedang sampai tinggi diperoleh bobot biji sedang sampai tinggi. Nilai jarak genetik yang rendah (< 0,70) menghasilkan bobot biji yang rendah, yang tidak bisa bersaing dengan varietas hibrida lain. Gambar 1 menunjukkan nilai regresi jarak genetik terhadap bobot biji dan nilai korelasi antara jarak genetik dan bobot biji per tanaman.
Perlu diketahui bahwa faktor lingkungan seperti lokasi atau musim turut berpengaruh sehingga nilai korelasi antara jarak genetik dan penampilan fenotipik seperti bobot biji tidak akan maksimum. Parentoni et al. (2001) menggunakan marka RAPD pada jagung, di mana filogeny yang diperoleh sesuai dengan data pedigree. Walaupun korelasi antara jarak genetik dan DGK positif nyata, tetapi sangat lemah. Barbosa et al. (2003) menganalisis gerombol untuk membentuk kelompok heterotik inbrida jagung, dan memperoleh korelasi yang signifikan antara jarak genetik dan hasil. Lanza et al. (1997) tidak menemukan korelasi antara jarak genetik
Gambar 1. Hasil regresi jarak genetik berdasarkan marka SSR terhadap bobot biji per tanaman (g) hibrida F1 hasil silang uji dengan inbrida Mr4 dan Mr14. Nilai r adalah hasil analisis korelasi antara jarak genetik dan bobot biji per tanaman (g) hibrida F1 hasil silang uji dengan inbrida Mr4 dan Mr14. y = 247,54x - 55,281 R2 = 0,5793 r = 0,76 y = 323,55x - 110,38 R2 = 0,6608 r = 0,81 50 60 70 80 90 100 110 120 130 140 150 160 170 180 190 0,40 0,45 0,50 0,55 0,60 0,65 0,70 0,75 0,80 0,85 0,90 0,95 Jarak genetik B o b o t b iji p e r t anam a n (g )
Hibrida F1 hasil silang uji dengan galur Mr4 Hibrida F1 hasil silang uji dengan galur Mr14
y = 247,54x - 55,281 R2 = 0,5793 r = 0,76 y = 323,55x - 110,38 R2 = 0,6608 r = 0,81 50 60 70 80 90 100 110 120 130 140 150 160 170 180 190 0,40 0,45 0,50 0,55 0,60 0,65 0,70 0,75 0,80 0,85 0,90 0,95 Jarak genetik B o b o t b iji p e r t anam a n (g )
Hibrida F1 hasil silang uji dengan galur Mr4 Hibrida F1 hasil silang uji dengan galur Mr14
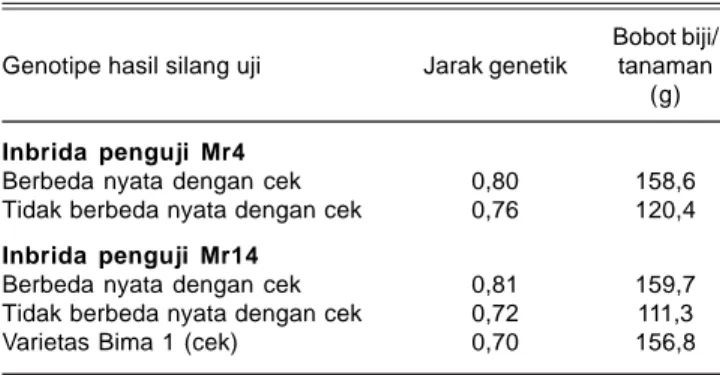
Tabel 2. Nilai rata-rata jarak genetik dan bobot biji dua set genotipe hasil silang uji.
Bobot biji/
Genotipe hasil silang uji Jarak genetik tanaman
(g) Inbrida penguji Mr4
Berbeda nyata dengan cek 0,80 158,6
Tidak berbeda nyata dengan cek 0,76 120,4
Inbrida penguji Mr14
Berbeda nyata dengan cek 0,81 159,7
Tidak berbeda nyata dengan cek 0,72 111,3
Varietas Bima 1 (cek) 0,70 156,8
Tidak berbeda nyata atau berbeda nyata terhadap kultivar pembanding Bima 1 dan Semar 10.
dan hasil secara umum, namun korelasi menjadi nyata jika menggunakan analisis antargerombol. Menurut Dias
et al. (2004), perbedaan genetik yang kontras dan
heterosis tidak selalu berhubungan secara linier. Sant et
al. (1999) menggambarkan bahwa hubungan nonlinier
yang terjadi antara jarak genetik dan hasil yang tidak menentu dari sejumlah hasil penelitian disebabkan oleh pengaruh lingkungan.
Dari hasil penelitian ini dapat dikemukakan bahwa nilai jarak genetik berdasarkan marka molekuler bermanfaat dalam menyaring sejumlah besar inbrida berdasarkan nilai jarak genetik antara inbrida yang diuji dengan inbrida pengujinya, sehingga dapat mengurangi materi pengujian. Jadi peluang untuk mendapatkan hibrida bisa hanya melalui metode silang uji, tanpa harus dilanjutkan ke persilangan dialel, namun inbrida penguji harus mempunyai potensi penggabung yang besar.
KESIMPULAN
1. Jarak genetik antartetua hibrida yang rendah (<0,70) menghasilkan hibrida dengan bobot biji yang rendah, sedangkan jarak genetik antartetua hibrida sedang sampai tinggi (>0,70) menghasilkan hibrida dengan bobot biji tinggi.
2. Nilai jarak genetik dapat digunakan sebagai alat prediksi awal untuk menyeleksi atau menyaring sejumlah besar inbrida, sehingga peluang untuk memperoleh kandidat hibrida potensial melalui metode silang uji akan lebih cepat dan akurat. 3. Inbrida P5/GM26-9 dan Bisma-3-1 mempunyai
peluang sebagai inbrida penguji yang baru, masing-masing menggantikan inbrida penguji Mr4 dan Mr14.
DAFTAR PUSTAKA
Akkaya, M.S., A.A. Bhagwat, P.B. Cregan. 1992. Length polymorphisms of simple sequence repeat DNA in soybean. Genetics 132: 1131-1134.
Araujo, P.M. and J.B. Miranda. 2001. Analysis of diallel cross for evaluation of maize populations across environments. Crop Breed. Appl. Biotech. 1: 255-262.
Barbosa, A.M.M., I.O. Gerald, L.L. Benchimol, A.A.F. Garcia, Jr. Souza, and A.P. Souza. 2003. Relatioship of intra- and interpopulation tropical maize single cross hybrid performance and genetic distances computed from AFLP and SSR markers. Euphytica 130: 87-99.
Chaudhary, A.K., L.B. Chaudhar y, and K.C. Sharma. 2000. Combining ability estimates of early generation inbred lines derived from two maize populations. Ind. J. Genet. and Plant Breeding 60: 55-61.
Chen, X., S. Temnykh, Y. Xu, Y.G. Cho, and S.R. McCouch. 1997. Development of a microsatellite framework map providing genome-wide coverage in rice (Oryza sativa L.). Theor. Appl. Genet. 95: 553-657.
Deghanpour, Z., B. Ehdaie, and M. Moghaddam. 1996. Diallel analysis of agronomic characters in white endosperm maize. J. Genet. and Breeding 50: 357-365.
Dias, L.A.S., E.A.T. Picolt, R.B. Roca, and A.C. Alfenas. 2004. A priori choise of hybrid parents in plants. Genet. Mol. Res. 3(3): 356-368.
El-Haddad, M.M. 1975. Genetical analysis of diallel crosses in spring wheat. Egypt J. Genet. Cytol. 4: 174-188.
George, M.L.C., E. Regalado, M. Warburton, S. Vasal, and D. Hoisington. 2004. Genetic diversity of maize inbred lines in relation to downy mildew. Euphytica 135: 145-155.
Gupta, P.K., H.S Balyan, P.C. Sharma, and B. Ramesh. 1996. Microsatellites in plants: A new class of molecular markers. Current Sci. 7 (1): 45-54.
Hallauer, A.R., and J.B. Miranda. 1988. Quantitative genetics in maize breeding. Second Edition. Iowa State University Press/ Ames. Iowa. p. 337-368.
Kalla, V., R. Kumar, and A.K. Basandrai. 2001. Combining ability analysis and gene action estimates of yield and yield contributing characters in maize. Crop Res. Hisar. 22: 102-106.
Khan, I.A., F.S. Awan, A. Ahmad, and A.A. Khan. 2004. A modified mini-prep method for economical and rapid extraction of genomic DNA in plants. Plant Molecular Biology Reporter 22:89a-89e.
Konak, C., A. Ünay, E. Serter, and H. Baal. 1999. Estimation of combining ability effects, heterosis and heterobeltiosis by line x tester method in maize. Turk J. of Field Crops 4:1-9. Lanza, L.L.B., C.L. de Souza, L.M.M. Ottoboni, M.L.C. Vieira, and
A.P. de Souza. 1997. Genetik distance of inbred lines and prediction of maize single-cross performance using RAPD markers. Theor. Appl. Genet. 94:1023-1030.
Mejaya, M.J., M.B. Pabendon, dan M. Dahlan. 2005. Pola heterosis dalam pembentukan varietas unggul jagung bersari bebas dan hibrida. Makalah disampaikan pada Seminar Puslitbangtan. Bogor, 12 Mei 2004.
Parentoni, S.N., J.V. Magalhaes, C.A.P. Pacheco, M.X. Santos, T. Abadie, and E.E.G. Gama, P.E.O. Guimaraes, W.F. Meirelles, M.A. Lopes, M.J.V. Vasconcelos, E. Paiva. 2001. Heterotic groups based on yield specific combining ability data and phylogenetic relationship determined by RAPD markers for 28 tropicalmaize open pollinated varieties. Euphytica 121: 197-208.
Rohlf, F.J. 2000. NTSYSpc numerical taxonomy and multivariate analysis system version 2.1. Applied Biostatistics Inc. San-Vicente, F.M., A. Bejarano, C. Marin, and J. Crossa. 1998.
Analysis of diallel crosses among improved tropical white endosperm maize populations. Maydica, 43: 147-153. Sant, V.J., A.G. Patankar, N.D. Sarode, L.B. Mhase, M.N. Sainani,
R.B. Deshmukh, P.K. Rajenkar, and V.S. Gupta. 1999. Potential of DNA markers in detecting divergence and in analyzing heterosis in Indian elite chickpea cultivars. Theor. Appl. Genet. 98: 1217-1225.
Senior, M.L. and M. Heun. 1993. Mapping maize microsatellites and polymerase chain reaction confirmation of the targeted repeats using a CT primer. Genome 36: 884-889.
Senior, M.L., J.P. Murphy, M.M. Goodman, and C.W. Stuber. 1998. Utility of SSR for determining genetic similarities and relationships in maize using an agarose gel system. Crop Sci. 38: 1088-1098.
Singh, A.K., J.P. Shai, and J.K. Singh. 1998. Heritability and genetic advance for maturity and yield attributes in maize. J. Appl. Biol. 8:42-45.
Tanksley, S.D. and S.R. McCouch. 1997. Seed banks and molecular maps: unlocking genetic potential from the wild. Science 277: 1063-1066.
Taramino, G. and S. Tingey. 1996. Simple sequence repeats for germplasm analysis and mapping in maize. Genome 39: 277-288.
Uddin, M.N., F.W. Ellison, L. O’Brian, and B.D.H. Latter. 1992. Heterosis in F1 hybrids derived from crosses of adapted Australian Wheats. Aust. J. Agric. Res. 43:907-919. Warburton, M., J.M. Ribaut, J. Franco, J. Crossa, P. Dubreuil, and
F.J. Betran. 2005. Genetic characterization of 218 elite CIMMYT maize inbred lines using RFLP markers. Euphytica 142:97-106.
Warburton, M.L., X.Xianchun, J. Crossa, J. Franco, A.E. Melchinger, M. Frisch, M. Bohn, and D. Hoisington. 2002. Genetic characteristization of CIMMYT inbred maize lines and open-pollinated population using large-scale fingerprinting methods. Crop Sci. 42:1822-1840.
Xia, X., J. C. Reif, D.A. Hoisington, A.E. Melchinger, M. Frich, and M.L. Warburton, 2004. Genetic diversity among CIMMYT maize inbred lines investigated with SSR markers: I. lowlanf tropical maize. Crop Sci. 44:2230-2237.