KLASIFIKASI FRAGMEN METAGENOM MENGGUNAKAN
FITUR SPACED N-MERS DAN K-NEAREST NEIGHBOUR
FITRIA ELLIYANA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2014
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Klasifikasi Fragmen Metagenom menggunakan Fitur Spaced N-Mers dan K-Nearest Neighbour adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Januari 2014
Fitria Elliyana
ABSTRAK
FITRIA ELLIYANA. Klasifikasi Fragmen Metagenom Menggunakan Fitur
Spaced N-Mers dan K-Nearest Neighbour. Dibimbing oleh WISNU ANANTA
KUSUMA.
Metagenom merupakan studi DNA total dari sumber lingkungan yang diisolasi secara langsung. Studi tersebut dilakukan dengan membaca seluruh DNA dari suatu ekosistem lengkap (bukan hanya satu organisme). Metagenom mengacu pada kandungan genomik dari ekosistem mikroba lengkap. Karena sampel mikroba yang diambil dari ekosistem mengandung bermacam-macam organisme maka perlu dilakukan proses
binning untuk klasifikasi. Pada penelitian ini digunakan algoritme k-nearest neighbour
(KNN) untuk mengklasifikasi fragmen metagenom dan spaced n-mers untuk ekstraksi fitur. Penelitian dilakukan pada dua kelompok dataset yaitu organisme latih dan organisme uji dengan panjang fragmen 500 bp, 1 kbp, 5 kbp, dan 10 kbp. Hasil akurasi terbaik yang diperoleh dari dataset organisme latih mencapai 99.75% pada pengujian fragmen dengan panjang 10 kbp dan nilai k = 3. Nilai sensitivitas dan spesifisitas tertinggi juga diperoleh dari dataset organisme yang sama yaitu 99.71% dan 99.85%.
Kata kunci: DNA, k-nearest neighbour, metagenom, oligonucleotide frequency, sensitivitas, spaced n-mers, spesifisitas.
ABSTRACT
FITRIA ELLIYANA. Metagenome Fragment Classification using Spaced N-Mers Features and K-Nearest Neighbour. Supervised by WISNU ANANTA KUSUMA.
Metagenome is a study of total DNA from some environmental sources that are directly isolated. The study is conducted by reading the entire DNA of a complete ecosystem (not just one organism). Metagenome refers to the genomic content of complete microbial ecosystems. Since the samples taken from the ecosystems may contain a variety of organisms, it requires a binning process to classify. In this research, k-nearest neighbour (KNN) algorithm was used to classify metagenome fragments and spaced n-mers was used for feature extraction. The research was conducted on two groups of datasets, namely the training organisms and testing organisms with fragment length of 500 bp, 1 kbp, 5 kbp, and 10 kbp. The best accuracy obtained from the training organism dataset reached 99.75% on the fragment test with a length of 10 kbp and k = 3. The highest value of its sensitivity and specificity was also obtained from the same organism dataset, 99.71% and 99.85% respectively.
Keywords: DNA, k-nearest neighbour, metagenome, oligonucleotide frequency, sensitivity, spaced n-mers, specificity.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer
pada
Departemen Ilmu Komputer
KLASIFIKASI FRAGMEN METAGENOM MENGGUNAKAN
FITUR SPACED N-MERS DAN K-NEAREST NEIGHBOUR
FITRIA ELLIYANA
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2014
Penguji:
1 Dr Irman Hermadi, SKom MT 2 Toto Haryanto, SKom MSi
Judul Skripsi : Klasifikasi Fragmen Metagenom menggunakan Fitur Spaced
N-Mers dan K-Nearest Neighbour
Nama : Fitria Elliyana NIM : G64104020
Disetujui oleh
Dr Wisnu Ananta Kusuma, ST MT Pembimbing
Diketahui oleh
Dr Ir Agus Buono, MSi MKom Ketua Departemen
Judul Skripsi: Klasifikasi Fragmen Metagenom menggunakan Fitur Spaced N Mel's dan K-Nearest Neighbour
Nama : Fitria Elliyana
NJM: : G641 04020 Disetujui oleh usuma Dr Wisnu Ananta ST MT Pembim ing Ketua Departemen Tanggal Lulus:
2
9
JAN 2
,
014
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah dengan judul Klasifikasi Fragmen Metagenom menggunakan Fitur Spaced N-Mers dan K-Nearest Neighbour ini berhasil diselesaikan.
Adapun penulis mengucapkan terima kasih kepada:
1 Kedua orangtua serta seluruh keluarga yang telah memberikan dukungan, perhatian, dan doa sehingga penulis dapat menyelesaikan penelitian ini.
2 Bapak Dr Wisnu Ananta Kusuma, ST MT selaku pembimbing yang telah banyak memberi saran.
3 Dosen penguji, Bapak Dr Irman Hermadi, SKom MS dan Bapak Toto Haryanto, SKom MSi atas saran dan bimbingannya.
4 Teman-teman satu bimbingan Agung, Fariz, Haris, Bernita, dan Galih, terima kasih atas kerja samanya.
5 Teman-teman Ekstensi Ilkom angkatan 5 atas kerja samanya selama penelitian. 6 Guru, sahabat, sekaligus temanku Bayu Widodo yang bersedia mendengarkan
keluh kesah dan selalu memberikan dukungan.
7 Semua pihak yang telah memberikan bantuan selama pengerjaan penelitian ini yang tidak dapat penulis tuliskan satu per satu.
Semoga penelitian ini bermanfaat bagi semua pihak yang membutuhkan.
Bogor, Januari 2014
DAFTAR ISI
DAFTAR TABEL vii
DAFTAR GAMBAR vii
DAFTAR LAMPIRAN viii
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
METODE 3
Penyiapan Data 4
Ekstraksi Fitur 4
F-Fold Cross Validation 5
Klasifikasi 5
Pengujian 6
Evaluasi dan Analisis Hasil 6
Lingkungan Implementasi 7
HASIL DAN PEMBAHASAN 7
Penyiapan Data 7
Percobaan 1 : Dataset Organisme Latih dengan Panjang Fragmen 500 bp 9 Percobaan 2 : Dataset Organisme Latih dengan Panjang Fragmen 1 kbp 9 Percobaan 3 : Dataset Organisme Latih dengan Panjang Fragmen 5 kbp 10 Percobaan 4 : Dataset Organisme Latih dengan Panjang Fragmen 10 kbp 11 Percobaan 5 : Dataset Organisme Uji dengan Panjang Fragmen 500 bp 13 Percobaan 6 : Dataset Organisme Uji dengan Panjang Fragmen 1 kbp 14 Percobaan 7 : Dataset Organisme Uji dengan Panjang Fragmen 5 kbp 14 Percobaan 8 : Dataset Organisme Uji dengan Panjang Fragmen 10 kbp 15 Perbandingan Akurasi, Sensitivitas, dan Spesifisitas antara Dataset
Organisme Latih dengan Organisme Uji 16
SIMPULAN DAN SARAN 20
Simpulan 20
DAFTAR PUSTAKA 20
LAMPIRAN 22
DAFTAR TABEL
1 Confusion matrix 6
2 Hasil akurasi pengujian dataset organisme latih dengan panjang
fragmen 500 bp 9
3 Hasil akurasi pengujian dataset organisme latih dengan panjang
fragmen 1 kbp 10
4 Hasil akurasi pengujian dataset organisme latih dengan panjang
fragmen 5 kbp 10
5 Hasil akurasi pengujian dataset organisme latih dengan panjang
fragmen 10 kbp 11
6 Hasil akurasi fold tertinggi pada pengujian dataset organisme latih 11
7 Hasil sensitivitas organisme latih 12
8 Hasil spesifisitas organisme latih 12
9 Perbandingan akurasi, sensitivitas, dan spesifisitas organisme latih 13
10 Confusion matrix dataset organisme uji dengan panjang fragmen 500 bp 13
11 Confusion matrix datset organisme uji dengan panjang fragmen 1 kbp 14
12 Confusion matrix dataset organisme uji dengan panjang fragmen 5 kbp 15
13 Confusion matrix dataset organisme uji dengan panjang fragmen 10 kbp 15
14 Hasil akurasi pada pengujian dataset organisme uji 16
15 Hasil sensitivitas dan spesifisitas organisme uji 16
16 Perbandingan akurasi, sensitivitas, dan spesifisitas organisme uji 16
17 Perbandingan akurasi, sensitivitas, dan spesifisitas organisme latih dan
uji 16
18 Hasil sensitivitas organisme latih 17
19 Hasil sensitivitas organisme uji 18
20 Hasil spesifisitas organisme latih 18
21 Hasil spesifisitas organisme uji 18
22 Perbandingan hasil akurasi penelitian terkait 19
DAFTAR GAMBAR
1 Skema metode penelitian 3
2 Fitur spaced n-mers 4
DAFTAR LAMPIRAN
1 Pembagian subset untuk panjang fragmen 500 bp 21
2 Pembagian subset untuk panjang fragmen 1 kbp 21
3 Pembagian subset untuk panjang fragmen 5 kbp 21
4 Pembagian subset untuk panjang fragmen 10 kbp 21
5 Dataset organisme latih 21
6 Dataset organisme uji 21
7 Confusion matrix percobaan 1 pada dataset organisme latih dengan
panjang fragmen 500 bp 21
8 Confusion matrix percobaan 1 pada panjang fragmen 500 bp dengan
nilai fold terbaik 21
9 Confusion matrix percobaan 2 pada dataset organisme latih dengan
panjang fragmen 1 kbp 21
10 Confusion matrix percobaan 2 pada panjang fragmen 1 kbp dengan nilai
fold terbaik 21
11 Confusion matrix percobaan 3 pada dataset organisme latih dengan
panjang fragmen 5 kbp 21
12 Confusion matrix percobaan 3 pada panjang fragmen 5 kbp dengan nilai
fold terbaik 21
13 Confusion matrix percobaan 4 pada dataset organisme latih dengan
panjang fragmen 10 kbp 21
14 Confusion matrix percobaan 4 pada panjang fragmen 10 kbp dengan
PENDAHULUAN
Latar Belakang
Para peneliti di dunia ilmiah telah banyak menghasilkan perkembangan baru dalam sains dan teknologi, tak terkecuali di bidang biologi. Metagenom merupakan studi DNA total dari sumber lingkungan yang diisolasi secara langsung (Fanani 2011). Penggunaan pendekatan metagenom untuk eksplorasi gen dari DNA total yang berasal dari sampel lingkungan memberikan beberapa kelebihan di antaranya yaitu dapat diperoleh gen yang berasal dari mikroorganisme yang tidak dapat dikulturkan, mikroorganisme yang hidup di daerah ekstrim, misalnya kadar garam tinggi, temperatur panas, temperatur rendah, dan lingkungan yang sangat asam-basa. Namun dari sekian banyak mikroorganisme yang terdapat di dunia, hanya sekitar 1% yang dapat dikulturkan dengan menggunakan metode standar. Sisanya harus diambil langsung dari ekosistemnya. Sampel yang diambil langsung dari ekosistem ini mengandung bermacam-macam organisme sehingga perlu dilakukan proses klasifikasi.
Genom adalah set lengkap molekul DNA dalam setiap sel dari organisme hidup yang diturunkan dari satu generasi ke generasi berikutnya. Deoxyribo
nucleic acid (DNA) merupakan pembawa informasi genetik dari makhluk hidup.
DNA merupakan rantai ganda dari molekul sederhana (nukleotida) yang diikat bersama-sama dalam struktur helix yang dikenal dengan double helix. Nukleotida tersebut tersusun atas empat basa nitrogen yaitu adenin, timin, guanin, dan sitosin yang dapat direpresentasikan dalam abjad A, T, G, dan C (de Carvalho 2003).
Sekuens DNA suatu organisme berasal dari sekuens generasi yang telah ada sebelumnya. Jika ditemukan sekuens baru yang belum diketahui sebelumnya, maka sekuens baru dibandingkan dengan sekuens yang sudah ada sehingga dapat diperoleh informasi genetik dari sekuens yang baru. Klasifikasi adalah satu cara yang digunakan untuk menentukan suatu fragmen DNA termasuk ke dalam tingkat taksonomi tertentu. Hal ini diperlukan karena dimungkinkan terdapat DNA yang memiliki perbedaan dalam fragmennya namun ternyata masih termasuk dalam satu genus yang sama.
Metode yang umum digunakan untuk mengekstraksi fitur fragmen metagenom adalah dengan menghitung frekuensi n-mers. Wu (2008) menggunakan principal component analysis (PCA) dengan oligonucleotide frekuensi n-mers untuk melakukan ekstraksi fitur. Hasil penelitian dengan empat jenis pengklasifikasi yaitu klasifikasi linear, klasifikasi kuadrat, k-nearest
neighbour, dan decision tree menunjukkan bahwa PCA dengan n-mers mampu
menangkap karakteristik intrinsik dari fragmen DNA sehingga memadai sebagai fitur klasifikasi. PCA dengan n-mers cenderung lebih efektif dan stabil ketika panjang fragmen DNA meningkat. Pengklasifikasi linear sederhana dapat mencapai akurasi yang tinggi untuk klasifikasi fragmen metagenom pada tingkat berbagai taksonomi, bahkan pada tingkat spesifik seperti spesies.
Oligonucleotide frekuensi n-mers telah banyak digunakan untuk prediksi
gen, konstruksi pohon filogenetik, dan klasifikasi metagenom. Namun, penggunaan n-mers akan mengakibatkan dimensi fitur yang tinggi bahkan untuk nilai n kecil. Untuk mereduksi dimensi fitur yang tinggi pada n-mers, Wu (2008) menerapkan PCA pada penelitiannya. Sebagai contoh, untuk nilai n = 5 pada
n-2
mers, maka akan diperoleh dimensi sebesar 45 yaitu 1024 fitur. Apabila
dibandingkan dengan spaced n-mers dengan nilai n = 3 akan diperoleh dimensi sebesar 43 + 43 + 43 yaitu 192. Spaced 3-mers menghasilkan nilai 192 karena nilai
don’t care yang dilambangkan dengan simbol (*) tidak dihitung, sehingga
menghasilkan dimensi yang lebih sedikit namun lebih kaya fitur.
Pada penelitian yang dilakukan oleh Wu (2008) diperoleh rata-rata tingkat akurasi klasifikasi tertinggi dicapai oleh pengklasifikasi yang lebih sederhana, seperti; pengklasifikasi linear sederhana, kuadrat, dan 3-NN. Klasifikasi linear sederhana mampu mencapai tingkat akurasi di atas 85% untuk mengklasifikaskan fragmen DNA pada tingkat yang lebih spesifik. 3-NN mencapai akurasi 92% untuk level taksonomi genus.
Kusuma dan Akiyama (2011) juga melakukan penelitian klasifikasi fragmen metagenom menggunakan algoritme SVM berbasis characterization vector. Penelitian tersebut dilakukan pada dua kelompok dataset yang merepresentasikan organisme latih dan organisme uji dengan panjang fragmen 500 bp, 1 kbp, 5 kbp, dan 10 kbp. Akurasi yang dicapai dari penelitian tersebut yaitu 78% untuk panjang fragmen 500 bp, 80% untuk panjang fragmen 1 kbp, 86% untuk panjang fragmen 5 kbp, dan 87% untuk panjang fragmen 10 kbp.
Oleh karena itu pada penelitian ini akan dilakukan klasifikasi fragmen metagenom menggunakan algoritme k-nearest neighbour (KNN) dengan spaced
n-mers sebagai ekstraksi fitur dan merujuk pada data penelitian Kusuma dan
Akiyama (2011), untuk kemudian hasilnya akan dibandingkan.
Tujuan Penelitian
Tujuan penelitian ini adalah untuk melakukan klasifikasi fragmen metagenom pada tingkat genus dengan fitur spaced n-mers dan algoritme KNN serta membandingkan tingkat akurasi dengan penelitian sebelumnya (Kusuma dan Akiyama 2011).
Manfaat Penelitian
Penelitian ini diharapkan dapat membantu para peneliti dalam mengidentifikasi dan mengklasifikasi fragmen metagenom sesuai dengan tingkat taksonomi.
Ruang Lingkup Penelitian
Ruang lingkup penelitian ini yaitu:
1 Data yang digunakan merujuk referensi data dari penelitian Kusuma dan Akiyama (2011). Data terdiri dari dua kelompok dataset yang dibangkitkan oleh perangkat lunak MetaSim, dengan format FASTA, yang merepresentasikan organisme latih dan organisme uji.
2 Panjang fragmen untuk dataset organisme latih dan organisme uji meliputi 500 bp, 1 kbp, 5 kbp, dan 10 kbp. Dataset tersebut dipilih dari genus
3
3 Sekuens DNA direpresentasikan sebagai empat karakter A, T, G, dan C yang mewakili basa nitrogen adenin, timin, guanin, dan sitosin. Sekuens DNA diasumsikan bebas error yaitu tidak ada karakter huruf lain selain A, T, G, dan C.
METODE
Penelitian ini dilakukan dengan beberapa tahapan proses, yaitu penyiapan data fragmen metagenom, ekstraksi fitur fragmen metagenom dengan spaced
n-mers, pembagian data uji dengan f-fold cross validation, dan klasifikasi fragmen
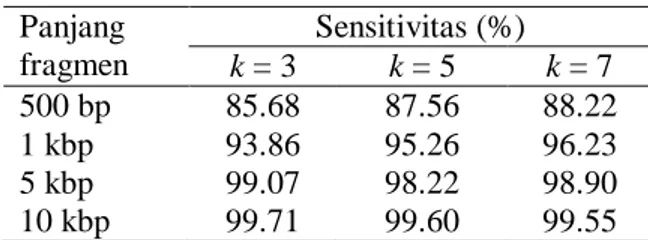
metagenom dengan menggunakan algoritme KNN, dan analisis hasil akurasi, sensitivitas, dan spesifisitas. Skema metode penelitian dapat dilihat pada Gambar 1. Penyiapan Data Mulai Spaced N-Mers Organisme Uji Pengujian Analisis Selesai K-Nearest Neighbor
F-Fold Cross Validation Organisme Uji
Data Latih Data Uji
4
Penyiapan Data
Data yang digunakan berupa dua kelompok dataset yang dibangkitkan oleh MetaSim, dengan format FASTA. MetaSim merupakan sebuah perangkat lunak yang berfungsi sebagai simulator penderetan untuk genomik dan metagenomik. MetaSim memungkinkan pengguna untuk mensimulasikan pembacaan dataset individu yang dapat digunakan sebagai skenario pengujian standar untuk perencanaan proyek sekuensing atau untuk sebagai tolok ukur perangkat lunak metagenomik. Dataset terdiri dari dataset organisme latih dan dataset organisme uji. Dataset organisme latih terdiri atas 10 spesies. Sedangkan dataset organisme uji terdiri atas sembilan spesies. Panjang fragmen untuk setiap dataset adalah tetap yaitu terdiri dari 500 bp, 1 kbp, 5 kbp, dan 10 kbp. Jumlah fragmen untuk dataset organisme latih adalah 10 000 dan organisme uji 5000.
Ekstraksi Fitur
Tahapan ini dilakukan untuk mendapatkan fitur yang akan digunakan dalam klasifikasi dan pengujian. Metode ekstraksi fitur yang digunakan dalam penelitian ini adalah spaced n-mers frequency. Spaced n-mers frequency digunakan untuk mencari sequence-composition-spaced pada fragmen DNA (Kusuma 2012).
Spaced n-mers perlu didefinisikan 𝜛𝜛𝑑𝑑𝑤𝑤 sebagai pola spaced, dimana w
mendefinisikan bobot sequence-composition-spaced dan merepresentasikan jumlah posisi yang cocok atau match, disimbolkan dengan 1, dan d merepresentasikan jumlah posisi don’t care, disimbolkan dengan (*). Pola 𝜛𝜛𝑑𝑑𝑤𝑤 digunakan untuk mendaftarkan pola nukleotida seperti {AA, AT, AG, AC, …, GG, A*A, A*T, …}.
Ekstraksi fitur spaced n-mers yang digunakan adalah pola 𝜛𝜛𝑑𝑑=0,1,2𝑤𝑤=3 yang direpresentasikan dengan {111 1*11 1**11} sehingga diperoleh pola fitur {AAA, AAT, AAG, AAC, …, CCC, A*AA, A*AT, …, C*CC, A**AA, A**AT, …, C**CC}. Dimensi yang didapat sebesar 43 + 43 + 43 yaitu 192. Spaced n-mers menghasilkan nilai 192 karena nilai don’t care yang dilambangkan dengan simbol (*) tidak dihitung. Frekuensi kemunculan tiap fragmen DNA dihitung dengan pola fitur tersebut. Spaced n-mers dengan 𝜛𝜛𝑑𝑑=0,1,2𝑤𝑤=3 dan pola {111 1*11 1**11} dengan sekuens ATGCTTACGTAGCATG, maka diperoleh fitur seperti pada Gambar 2.
AAA AAT AAG AAC ATA ATT ATG ... CCC
0 0 0 0 0 0 2 0
A*AA A*AT A*AG A*AC A*TA A*TT A*TG ... C*CC
0 0 0 0 0 0 0 0
A**AA A**AT A**AG A**AC A**TA A**TT A**TG ... C**CC
0 1 0 0 1 0 0 0
5
F-Fold Cross Validation
F-fold cross validation merupakan metode untuk melakukan pembagian
data menjadi f subset dengan ukuran yang sama antara subset satu dengan subset lainnya. Perulangan dilakukan sebanyak f kali, dimana salah satu subset dijadikan data uji dan f-1 subset lainnya dijadikan data latih. Tingkat akurasi dihitung dengan membagi jumlah keseluruhan klasifikasi yang benar dengan jumlah semua data uji.
Jumlah fragmen metagenom yang merepresentasikan organisme latih, yang terdiri dari 10 000 fragmen, akan dibagi menjadi 5 (lima) subset untuk masing-masing panjang fragmen. Pembagian subset untuk setiap panjang fragmen dapat dilihat pada lampiran.
Klasifikasi
Metode klasifikasi yang digunakan pada penelitian ini yaitu KNN. KNN banyak diterapkan dalam pengenalan pola dan data mining untuk klasifikasi. KNN merupakan algoritme supervised dalam klasifikasi dimana hasil dari kueri
instance yang baru diklasifikasikan berdasarkan mayoritas kategori pada k
tetangga terdekat. KNN mengklasifikasi objek baru berdasarkan atribut dan
training samples (Larose 2005).
Konsep dasar dari KNN adalah mencari jarak terdekat antara data yang akan dievaluasi dengan k tetangga terdekatnya. Nilai dari jarak antara data uji dengan data latih diurutkan dari nilai terendah. Kelas dari nilai dengan jarak terendah diperiksa. Kelas yang memiliki nilai vote tertinggi menjadi kelas dari data uji tersebut.
Jarak antara dua titik dalam ruang fitur dapat didefiniskan dengan banyak cara, salah satunya menggunakan jarak Euclid. Hasil dari perhitungan jarak euclides digunakan untuk menentukan kemiripan antara data latih dan data uji. Kecocokan dilihat dari nilai (jarak) yang paling minimum. Jarak Euclid diperoleh dengan menggunakan Persamaan 1.
dist(p,q) = � � (pi-qi)2 n
i=1
(1)
dengan : dist(p,q) = jarak sampel pi = data sampel ke-i
qi = data input ke-i
n = jumlah sampel
Tahapan algoritme KNN adalah sebagai berikut (Song et al. 2007): 1 Menentukan nilai k, dengan k merupakan jumlah tetangga terdekat.
2 Menghitung jarak data pada setiap data latih dengan menggunakan jarak Euclid.
6
Pengujian
Pengujian dilakukan dengan melakukan klasifikasi pada fragmen uji. Hasil prediksi tersebut kemudian dibandingkan dengan kelas aktual yang telah diketahui sebelumnya, apakah fragmen uji benar diklasifikasi atau salah diklasifikasi.
Evaluasi dan Analisis Hasil
Analisis merupakan tahapan yang bertujuan memperoleh informasi yang terdapat pada hasil klasifikasi. Alat ukur klasifikasi yang digunakan yaitu
confusion matrix. Confusion matrix adalah sebuah matriks yang menunjukkan
nilai aktual dan nilai prediksi dari klasifikasi (Kohavi dan Provost 1998). Hasil penelitian diukur dengan menghitung tingkat akurasi dari fragmen metagenom yang diuji dan diamati. Akurasi dihitung dengan Persamaan 2. Melalui confusion
matrix selain dapat diketahui tingkat akurasi juga dapat diketahui nilai sensitivitas
dan spesifisitas. Sensitivitas dan spesifisitas masing-masing akan dihitung dengan Persamaan 3 dan Persamaan 4.
Akurasi menghitung proporsi dari data uji yang diklasifikasikan dengan benar dari keseluruhan data uji. Sensitivitas dapat didefinisikan sebagai nilai true
positive (Kohavi dan Provost 1998). Sensitivitas merupakan kemampuan suatu classifier untuk menunjukkan hasil positif pada genus yang sedang diamati,
sedangkan spesifisitas merupakan kemampuan suatu classifier untuk menunjukkan hasil yang negatif pada genus yang sedang diamati. Sensitivitas memberi informasi seberapa baik sebuah classifier untuk mengidentifikasi hasil prediksi positif jika diberikan sampel aktual positif. Sensitivitas tidak memberi informasi tentang sampel aktual negatif namun diprediksi sebagai positif (Akobeng 2007). Spesifisitas merupakan nilai true negative (Kohavi dan Provost 1998). Spesifisitas hanya memberi informasi proporsi dari hasil prediksi negatif jika diberikan sampel aktual negatif. Spesifisitas tidak memberi informasi tentang sampel aktual positif namun diprediksi negatif (Akobeng 2007).
Dengan menggunakan Tabel 1 dapat diketahui nilai akurasi, sensitivitas, dan spesifisitas dari suatu hasil klasifikasi. Untuk mendapatkan nilai sensitivitas dapat dihitung dengan menggunakan Persamaan 2, dengan true positive (TP) berarti banyaknya data dari kelas aktual yang benar dan berhasil diprediksi oleh classifier dengan benar. false negative (FN) terjadi jika data yang secara aktual benar diprediksi menjadi kelas yang salah. Spesifisitas dihitung dengan menggunakan Persamaan 3, dengan true negative (TN) didefinisikan banyaknya data dari kelas aktual yang salah dan berhasil diprediksi sebagai kelas yang salah. False positive (FP) adalah ketika data yang secara aktual salah namun diprediksi sebagai kelas yang benar.
Tabel 1 Confusion matrix Kelas
aktual
Kelas prediksi
A ¬ A
7
¬ A FP TN
Tes yang ideal ialah selalu positif apabila kelas aktualnya positif (sensitivitas 100%) dan selalu negatif apabila kelas aktualnya negatif (spesifisitas 100%). Sensitivitas mengukur efektivitas suatu classifier untuk mengidentifikasi label positif. Spesifisitas mengukur efektivitas suatu classifier untuk mengidentifikasi label negatif.
Akurasi= TP + TN TP + TN + FP + FN × 100% (2) Sensitivitas = TP TP + FN × 100% (3) Spesifisitas = TN TN + FP × 100% (4) Lingkungan Implementasi
Lingkungan implementasi penelitian ini menggunakan perangkat keras dan perangkat lunak sebagai berikut:
1 Perangkat keras berupa notebook:
• Intel Core i3 CPU M370 @ 2.40 GHz • Memori 2 GB
• Harddisk kapasitas 500 GB
• Monitor dengan resolusi 1366 × 768 piksel 2 Perangkat lunak:
• Sistem operasi Microsoft Windows 7 Professional • Simulator metagenom MetaSim versi 0.9.1
• Bloodshed Dev-C++ • Matlab 7.7 (R2008b)
HASIL DAN PEMBAHASAN
Penyiapan Data
Pada penelitian ini digunakan fragmen metagenom dari 3 genus yaitu
Agrobacterium, Bacillus, dan Staphylococcus. Fragmen metagenom tersebut
terbagi menjadi dua yaitu fragmen metagenom organisme latih dan fragmen metagenom organisme uji. Total jumlah fragmen untuk pelatihan dan pengujian masing-masing adalah 10 000 dan 5000. Fragmen metagenom yang digunakan dapat dilihat pada Lampiran 1 dan Lampiran 2. Data dibangkitkan menggunakan MetaSim dan berformat FASTA. Contoh data dengan format FASTA dapat dilihat pada Gambar 3. Data dengan format FASTA ini kemudian akan diekstraksi dan
8
dihitung frekuensi kemunculan fiturnya menggunakan spaced n-mers. Fitur
spaced n-mers dengan n = 3 akan menghasilkan fitur sebanyak 192. Sehingga
akan dihasilkan matriks dataset organisme latih berukuran 10 000 × 192 dan dataset organisme uji berukuran 5000 × 192.
Penelitian ini terdiri dari 8 percobaan dengan menggunakan fragmen dengan panjang dan nilai k bervariasi 3, 5, dan 7. Percobaan 1 sampai Percobaan 4 dilakukan dengan menggunakan dataset organisme latih sedangkan Percobaan 5 hingga Percobaan 8 menggunakan dataset organisme uji. Percobaan 1 dan 5 menggunakan panjang fragmen 500 bp. Percobaan 2 dan 6 menggunakan dataset dengan panjang fragmen 1 kbp. Percobaan 3 dan 7 akan menggunakan fragmen dengan panjang 5 kbp sedangakan Percobaan 4 dan 8 dengan panjang fragmen 10 kbp. Daftar fragmen metagenom yang digunakan dapat dilihat pada Lampiran 5 dan 6.
Klasifikasi
Sebuah himpunan fragmen metagenom latih dengan klasifikasi label yang akurat harus diketahui pada awal algoritme. Kemudian untuk fragmen metagenom yang belum diketahui labelnya dihitung jaraknya. Setelah hasil perhitungan jarak diurutkan, keputusan label kelas dapat dibuat sesuai dengan label k terdekat dalam himpunan fragmen metagenom latih.
Fragmen metagenom latih maupun fragmen metagenom uji diproses dengan
spaced n-mers frequency terlebih dahulu. Kemudian proses klasifikasi data
dilakukan dengan menggunakan metode KNN dengan jarak Euclid sebagai metode pengukuran jarak kemiripan. Sebelum proses klasifikasi dilakukan, nilai k, yaitu jumlah tetangga terdekat yang akan dilihat kelasnya untuk menentukan kelas terbanyak yang merupakan kelas dari titik baru, harus ditentukan terlebih dahulu.
Pembagian data akan dibagi menjadi kelompok data menggunakan 5-fold
cross validation. Setiap fold akan diujicobakan dengan kombinasi panjang
fragmen 500 bp, 1 kbp, 5 kbp, dan 10 kbp serta nilai untuk KNN dengan k = 3, 5, dan 7 yang akan diuraikan pada percobaan 1, 2, 3, dan 4. Sehingga untuk setiap data latih akan terbentuk matriks berukuran 2000 × 192 dan data uji berukuran 8000 × 192. Jumlah kelas dalam klasifikasi adalah 3 sesuai dengan jumlah genus. Setelah semua dataset organisme latih diklasifikasi kemudian dataset tersebut akan digunakan untuk proses pengujian dataset organisme uji.
>gi|38232642|ref|NC_002935.2| Corynebacterium diphtheriae NCTC 13129 chromosome, complete genome GTGTCGGAAACGCCATCCGTGTGGAACGAGACG TGGAATGAGATCACCAATGAACTCATTCAGCTA TCTCGCGAACCCGAAAGCGAGATTCCACGAATC ACTGCTGAACAACGCGCTTATCTCAAACTCGTC CGACCTGCGGCTTTTGTCGAAGGCATCGCCGTT TTACGGGTACCGCACTCCCGCGCCAAGGAGACG ATTGAAACCCATTTGGGGCAAGCGATAACCTCC GTGCTCTCCCGTCGTATGGGACGCCCCTTTACT GTGGCAGTCACCGTCGACCCCACGTTGGACGTC
9
Percobaan 1: Dataset Organisme Latih dengan Panjang Fragmen 500 bp
Pada percobaan 1 ini dataset yang digunakan adalah dataset organisme latih dengan panjang fragmen 500 bp. Hasil akurasi yang didapatkan pada percobaan ini dilihat pada Tabel 2 untuk panjang fragmen 500 bp.
Hasil dari percobaan 1 pada pengujian dataset organisme latih dengan panjang fragmen 500 bp diperoleh fold dengan nilai akurasi tertinggi untuk masing-masing nilai k tetangga terdekat. Untuk panjang fragmen 500 bp didapatkan fold 5 dengan nilai akurasi tertinggi pada semua nilai k. Pada nilai k = 3 diperoleh akurasi 87.15%, k = 5 sebesar 89.20%, dan k = 7 sebesar 89.95%. Nilai fold ini akan digunakan sebagai data latih untuk pengujian dataset organisme uji. Rata-rata akurasi maksimum sebesar 86.19% pada k = 7.
Pada fold 1 dan 2 terjadi penurunan akurasi pada k = 7 menjadi 77.45% dan 87.65%. Pada fold 1 terjadi bias yang menyebabkan banyak fragmen yang berasal dari genus Bacillus diprediksi sebagai genus Agrobacterium. Pada fold 2 terjadi bias yang menyebabkan banyak fragmen yang berasal dari genus Bacillus diprediksi sebagai genus Staphylococcus dan sebaliknya dari genus
Staphylococcus diprediksi sebagai genus Bacillus. Untuk mengetahui record yang
salah diklasifikasi dapat dilihat pada confusion matrix yang dilampirkan pada Lampiran 7.
Percobaan 2: Dataset Organisme Latih dengan Panjang Fragmen 1 kbp
Percobaan 2 ini menggunakan dataset organisme latih dengan panjang fragmen 1 kbp. Untuk panjang fragmen 1 kbp didapatkan fold 5 dengan nilai akurasi tertinggi pada semua nilai k. Pada nilai k = 3 diperoleh akurasi 94.50%, pada k = 5 sebesar 95.80%, dan k = 7 sebesar 96.75%. Rata-rata akurasi maksimum dari diperoleh sebesar 93.53% pada k = 7. Hasil akurasi yang didapatkan pada percobaan ini dilihat pada Tabel 3 untuk panjang fragmen 1 kbp.
Pada fold 1 terjadi penurunan akurasi pada k = 7 dari 92.25% menjadi 91.25%. Fold 1 terjadi bias yang menyebabkan banyak fragmen yang berasal dari genus Bacillus diprediksi sebagai genus Agrobacterium. Untuk mengetahui
record yang salah diklasifikasi dapat dilihat pada confusion matrix yang
dilampirkan pada Lampiran 9.
Tabel 2 Hasil akurasi pengujian dataset organisme latih dengan panjang fragmen 500 bp Fold Akurasi (%) k = 3 k = 5 k = 7 1 78.80 77.85 77.45 2 86.25 88.00 87.65 3 85.95 86.15 87.50 4 85.45 86.85 88.40 5 87.15 89.20 89.95 Rata-rata 84.72 85.61 86.19
10
Percobaan 3: Dataset Organisme Latih dengan Panjang Fragmen 5 kbp
Hasil dari percobaan 3 pada pengujian dataset organisme latih dengan panjang fragmen 5 kbp didapatkan 2 nilai fold dengan akurasi tertinggi. Pada nilai
k = 3 dan k = 5 dipilih dari fold 1. Sedangkan pada nilai k = 7 dipilih dari fold 5.
Dengan fold 1 diperoleh nilai akurasi dari k = 3 sebesar 99.20% dan k = 5 sebesar 99.25%. Sedangkan k = 7 diperoleh nilai akurasi sebesar 99.05% dari fold 5. Rata-rata akurasi maksimum sebesar 98.84% pada k = 3.
Pada fold 1 terjadi penurunan akurasi pada k = 7 menjadi 98.75. Hal ini terjadi karena banyak fragmen yang berasal dari genus Bacillus diprediksi sebagai genus Agrobacterium dan genus Bacillus diprediksi sebagai genus Staphylococcus dan sebaliknya. Pada fold 2 terjadi penurunan namun tidak terlalu signifikan dari 98.40% menjadi 98.35%. Ini terjadi karena ada penambahan satu fragmen yang awalnya berasal dari genus Staphylococcus namun diprediksi sebagai genus
Bacillus. Untuk mengetahui record yang salah diklasifikasi dapat dilihat pada confusion matrix yang dilampirkan pada Lampiran 11.
Tabel 4 Hasil akurasi pengujian dataset organisme latih dengan panjang fragmen 5 kbp Fold Akurasi (%) k = 3 k = 5 k = 7 1 99.20 99.25 98.75 2 98.40 98.35 98.50 3 98.75 98.70 98.75 4 98.75 98.70 98.55 5 99.10 99.15 99.05 Rata-rata 98.84 98.83 98.72 Tabel 3 Hasil akurasi pengujian dataset
organisme latih dengan panjang fragmen 1 kbp Fold Akurasi (%) k = 3 k = 5 k = 7 1 92.20 92.25 91.25 2 91.25 92.15 92.80 3 92.75 92.90 93.65 4 92.10 92.80 93.20 5 94.50 95.80 96.75 Rata-rata 92.56 93.18 93.53
11
Percobaan 4: Dataset Organisme Latih dengan Panjang Fragmen 10 kbp
Pada percobaan 4 ini dataset yang digunakan adalah dataset organisme latih dengan panjang fragmen 10 kbp. Hasil akurasi yang didapatkan pada percobaan ini dilihat pada Tabel 5 untuk panjang fragmen 10 kbp. Dari percobaan 4 didapatkan 2 nilai fold dengan akurasi tertinggi. Pada nilai k = 3 dan k = 5 dipilih dari fold 3. Sedangkan pada nilai k = 7 dipilih dari fold 5. Dengan fold 3 diperoleh nilai akurasi dari k = 3 sebesar 99.75% dan k = 5 sebesar 99.65%. Sedangkan k = 7 diperoleh nilai akurasi sebesar 99.55% dari fold 5. Rata-rata akurasi maksimum sebesar 99.59% pada k = 3.
Pada fold 1 terjadi penurunan akurasi pada k = 5 dari 99.55% menjadi 99.40%. Fold 1 terjadi bias yang menyebabkan banyak fragmen yang berasal dari genus Staphylococcus diprediksi sebagai genus Bacillus. Fold 2 dan fold 3 juga mengalami penurunan akurasi pada k = 5 dan k = 7. Pada fold 2 dan fold 3 terjadi bias yang menyebabkan fragmen dari genus Bacillus diprediksi menjadi genus
Staphylococcus dan sebaliknya dari genus Staphylococcus menjadi genus Bacillus.
Tabel 5 Hasil akurasi pengujian dataset organisme latih dengan panjang fragmen 10 kbp Fold Akurasi (%) k = 3 k = 5 k = 7 1 99.55 99.40 99.40 2 99.75 99.55 99.50 3 99.75 99.65 99.50 4 99.50 99.35 99.40 5 99.40 99.50 99.55 Rata-rata 99.59 99.49 99.47
Untuk mengetahui record yang salah diklasifikasi dapat dilihat pada
confusion matrix yang dilampirkan pada Lampiran 13. Fold dengan akurasi
tertinggi dari setiap panjang fragmen akan digunakan sebagai data latih untuk pengujian dataset organisme uji. Hasil akurasi tertinggi dari setiap percobaan dengan bermacam panjang fragmen yang akan digunakan sebagai dataset organisme latih dapat dilihat pada Tabel 6.
Dari pengujian dataset organisme latih terlihat bahwa semakin panjang fragmen yang diklasifikasi, nilai akurasi, sensitiviy, dan spesifisitas semakin meningkat. Namun hal ini tidak berlaku jika diterapkan nilai k yang beragam. Terdapat penurunan tingkat akurasi, sensitivitas dan spesifisitas pada percobaan 4 menggunakan data organisme latih dengan panjang fragmen 10 kbp dengan nilai k = 5 dan k = 7. Sebelumnya akurasi bernilai 99.75% pada nilai k = 3, kemudian menurun pada nilai k = 5 menjadi 99.65% dan kembali turun pada k = 7 hingga 99.55%. Begitu juga dengan nilai sensitivitas dan spesifisitas. Hal ini disebabkan karena pada proses pengujian dengan menggunakan klasifikasi KNN, akurasi sangat dipengaruhi oleh jumlah k tetangga terdekat. Pada proses pengujian dataset organisme latih banyak terdapat fragmen yang salah prediksi dan diidentifikasi ke kelas yang tidak sesuai. Sensitivitas dan spesifisitas tidak mampu memberikan informasi mengenai fragmen yang salah prediksi.
Tabel 6 Hasil akurasi fold tertinggi pada pengujian dataset organisme latih Panjang fragmen Akurasi (%) k = 3 k = 5 k = 7 500 bp 87.15 89.20 89.95 1 kbp 94.50 95.80 96.75 5 kbp 99.20 99.25 99.05 10 kbp 99.75 99.65 99.55
12
Sensitivitas dan spesifisitas organisme latih dihitung dari fold pengujian dataset organisme latih yang memperoleh nilai akurasi terbaik. Pada percobaan 1 dengan panjang fragmen 500 bp dan percobaan 2 dengan panjang fragmen 1 kbp dipilih fold 5. Pada percobaan 3 dengan panjang fragmen 5 kbp ambil dari 2 fold yaitu fold 1 untuk k = 1 dan k = 3 dan fold 5 untuk k = 7. Percobaan 4 dengan panjang fragmen 10 kbp juga dipilih dari 2 fold yaitu fold 3 untuk k = 1 dan k = 3 dan fold 5 untuk k = 7. Hasil sensitivitas dan spesifisitas dari pengujian dataset organisme latih bisa dilihat pada Tabel 7 dan Tabel 8.
Penurunan sensitivitas dan spesifisitas terjadi pada percobaan 3 dengan panjang fragmen 5 kbp dengan k = 5 namun kembali mengalami peningkatan pada
k = 7. Penurunan ini disebabkan karena ada salah prediksi fragmen dari genus Bacillus sebagai genus Agrobacterium. Selain itu juga banyak terdapat fragmen
dari genus Bacillus yang diprediksi sebagai genus Staphylococcus dan sebaliknya dari genus Staphylococcus diprediksi sebagai genus Bacillus. Peningkatan kembali terjadi pada k = 7 karena tidak lagi ada fragmen dari genus Bacillus yang diprediksi sebagai genus Agrobacterium.
Penurunan sensitivitas dan spesifisitas juga terjadi pada panjang fragmen 10
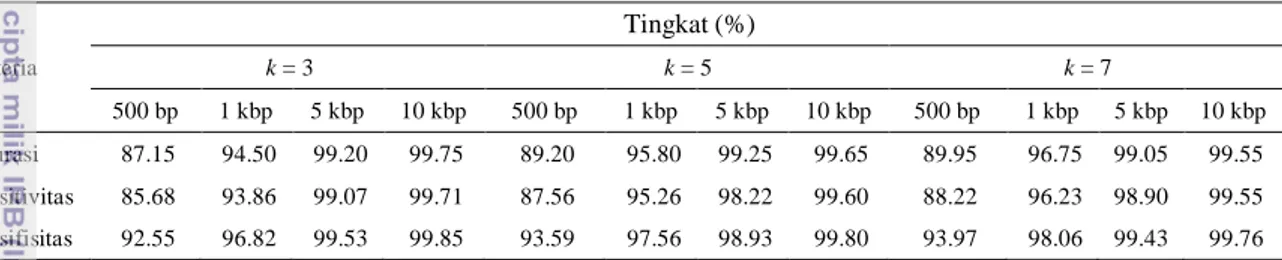
Tabel 7 Hasil sensitivitas organisme latih Panjang fragmen Sensitivitas (%) k = 3 k = 5 k = 7 500 bp 85.68 87.56 88.22 1 kbp 93.86 95.26 96.23 5 kbp 99.07 98.22 98.90 10 kbp 99.71 99.60 99.55
Tabel 8 Hasil spesifisitas organisme latih Panjang fragmen Spesifisitas (%) k = 3 k = 5 k = 7 500 bp 92.55 93.59 93.97 1 kbp 96.82 97.56 98.06 5 kbp 99.53 98.93 99.43 10 kbp 99.85 99.80 99.76
13
kbp dengan nilai k = 5 dan k = 7. Hal ini disebabkan karena masih terdapat kesalahan prediksi fragmen yang berasal dari genus Bacillus menjadi genus
Staphylococcus. Pada k = 7 terdapat satu kesalahan prediksi genus Agrobacterium
menjadi genus Bacillus.
Namun demikian nilai sensitivitas dan spesifisitas terbaik diperoleh dari percobaan 4 dengan panjang fragmen 10 kbp dengan nilai k = 3. Nilai sensitivitas dan spesifisitas yang diperoleh berturut-turut yaitu 99.71% dan 99.85%. Hasil akurasi tertinggi dari percobaan 4 dengan menggunakan nilai k = 3 sebesar 99.75%. Perbandingan tingkat akurasi, sensitivitas, dan spesifisitas dari dataset organisme latih ditunjukkan pada Tabel 9.
Percobaan 5: Dataset Organisme Uji dengan Panjang Fragmen 500 bp
Percobaan 5 dilakukan dengan menguji dataset organisme uji pada panjang fragmen 500 bp. Nilai k atau jumlah tetangga terdekat yang digunakan yaitu 3, 5, dan 7. Dari pengujian dataset organisme latih diperoleh fold dengan akurasi nilai tertinggi. Fold dengan akurasi nilai tertinggi tersebut kemudian digunakan sebagai data latih dalam pengujian dataset organisme uji. Sehingga, diperoleh data latih sebanyak 8000 data dan data uji sebanyak 5000 data.
Hasil akurasi organisme uji mengalami peningkatan dengan bertambahnya nilai k tetangga terdekat. Pada k = 3 diperoleh akurasi 85.64%. Untuk nilai k = 5 dan k = 7 masing-masing diperoleh akurasi sebesar 86.18% dan 86.52%. Hal serupa juga terjadi pada hasil sensitivitas dan spesifisitas yang mengalami peningkatan dengan bertambahnya nilai k tetangga terdekat. Sensitivitas dan spesifisitas pada k = 3 sebesar 84.28 dan 91.61%. Sensitivitas pada k = 5 dan k = 7 masing-masing sebesar 84.65% dan 84.89%. Sedangkan spesifisitas pada k = 5 dan k = 7 masng-masing sebesar 91.85% dan 92%.
Peningkatan sensitivitas dan spesifisitas terjadi karena tidak ada fragmen dari genus Staphylococcus yang salah diprediksi menjadi genus Agrobacterium. Selain itu juga terjadi penurunan jumlah fragmen yang salah prediksi dari genus
Tabel 9 Perbandingan akurasi, sensitivitas, dan spesifisitas organisme latih
Kriteria Tingkat (%) k = 3 k = 5 k = 7 500 bp 1 kbp 5 kbp 10 kbp 500 bp 1 kbp 5 kbp 10 kbp 500 bp 1 kbp 5 kbp 10 kbp Akurasi 87.15 94.50 99.20 99.75 89.20 95.80 99.25 99.65 89.95 96.75 99.05 99.55 Sensitivitas 85.68 93.86 99.07 99.71 87.56 95.26 98.22 99.60 88.22 96.23 98.90 99.55 Spesifisitas 92.55 96.82 99.53 99.85 93.59 97.56 98.93 99.80 93.97 98.06 99.43 99.76
Tabel 10 Confusion matrix dataset organisme uji dengan panjang fragmen 500 bp Kelas aktual
Kelas prediksi
k = 3 k = 5 k = 7
Agr Bac Sta Agr Bac Sta Agr Bac Sta Agrobacterium 1257 27 0 1260 24 0 1262 22 0
Bacillus 38 2178 168 34 2206 144 27 2223 134
14
Agrobacterium menjadi genus Bacillus dan sebaliknya, serta salah prediksi untuk
fragmen Bacillus menjadi Staphylococcus. Untuk mengetahui fragmen yang salah diklasifikasi dapat dilihat pada confusion matrix pada Tabel 10.
Percobaan 6: Dataset Organisme Uji dengan Panjang Fragmen 1 kbp
Percobaan 6 dilakukan dengan menguji dataset organisme uji pada panjang fragmen 1 kbp. Hasil akurasi organisme uji ini juga mengalami peningkatan dengan bertambahnya nilai k tetangga terdekat. Pada k = 3 diperoleh akurasi 91.34%. Untuk nilai k = 5 dan k = 7 masing-masing diperoleh akurasi sebesar 91.92% dan 92.04%. Peningkatan juga terjadi pada hasil sensitivitas dan spesifisitas dengan bertambahnya nilai k tetangga terdekat. Sensitivitas dan spesifisitas pada k = 3 sebesar 90.69% dan 95.04%. Sensitivitas pada k = 5 dan k = 7 masing-masing sebesar 91.30% dan 91.32%. Sedangkan spesifisitas pada k = 5 dan k = 7 masng-masing sebesar 95.37% dan 95.40%.
Fragmen dari genus Staphylococcus tidak ada yang salah diprediksi menjadi genus Agrobacterium. Namun, ada genus Agrobacterium yang diprediksi menjadi genus Staphylococcus. Peningkatan sensitivitas dan spesifisitas terjadi karena terdapat penurunan jumlah fragmen yang salah prediksi dari genus Bacillus menjadi genus Agrobacterium. Serta penurunan salah prediksi untuk fragmen
Bacillus menjadi Staphylococcus. Hasil akurasi, sensitivitas, dan spesifisitas yang
didapatkan pada percobaan ini masing-masing dapat dilihat pada Tabel 14 dan Tabel 15. Untuk mengetahui fragmen yang salah diklasifikasi dapat dilihat pada
confusion matrix pada Tabel 11.
Percobaan 7: Dataset Organisme Uji dengan Panjang Fragmen 5 kbp
Percobaan 7 menguji dataset organisme uji pada panjang fragmen 5 kbp. Hasil akurasi organisme uji mengalami penurunan dengan bertambahnya nilai k tetangga terdekat. Pada k = 3 diperoleh akurasi 96.78%. Untuk nilai k = 5 dan k = 7 masing-masing diperoleh akurasi sebesar 96.60% dan 96.42%. Hal serupa juga terjadi pada hasil sensitivitas dan spesifisitas yang mengalami penurunan. Pada k = 3 diperoleh sensitivitas 96.40%. Pada k = 5 dan k = 7 terjadi penurunan menjadi 96.17% dan 95.92%. Pada k = 3 diperoleh spesifisitas sebesar 98.08%. Pada k = 5 dan k = 7 terjadi penurunan menjadi 97.96% dan 97.84%.
Fragmen dari genus Staphylococcus tidak ada yang salah diprediksi menjadi genus Agrobacterium begitu juga sebaliknya. Pada k = 5 dan k = 7 terjadi peningkatan fragmen yang salah prediksi dari genus Agrobacterium menjadi
Bacillus dan Staphylococcus menjadi Bacillus. Hasil akurasi, sensitivitas, dan
Tabel 11 Confusion matrix datset organisme uji dengan panjang fragmen 1 kbp Kelas aktual
Kelas prediksi
k = 3 k = 5 k = 7
Agr Bac Sta Agr Bac Sta Agr Bac Sta Agrobacterium 1317 10 1 1314 13 1 1315 12 1
Bacillus 8 2157 99 7 2165 92 3 2179 82
Staphylococcus 0 315 1093 0 291 1117 0 300 1108
15
spesifisitas yang didapatkan pada percobaan ini masing-masing dapat dilihat pada Tabel 14 dan Tabel 15. Hasil prediksi mengalami peningkatan ketika melakukan klasifikasi pada genus Bacillus. Untuk mengetahui fragmen yang salah diklasifikasi dapat dilihat pada confusion matrix pada Tabel 12.
Percobaan 8: Dataset Organisme Uji dengan Panjang Fragmen 10 kbp
Pada percobaan 8 digunakan dataset organisme uji dengan panjang fragmen 10 kbp. Hasil akurasi organisme uji juga mengalami penurunan dengan bertambahnya nilai k tetangga terdekat. Pada k = 3 diperoleh akurasi 98.28%. Untuk nilai k = 5 dan k = 7 masing-masing diperoleh akurasi sebesar 98.02% dan 97.58%. Penurunan juga terjadi pada hasil sensitivitas dan spesifisitas dengan bertambahnya nilai k tetangga terdekat. Sensitivitas dan spesifisitas pada k = 3 sebesar 98.06% dan 98.95%. Sensitivitas pada k = 5 dan k = 7 masing-masing sebesar 97.77% dan 97.27%. Sedangkan spesifisitas pada k = 5 dan k = 7 masing-masing sebesar 98.80% dan 98.53%.
Fragmen dari genus Agrobacterium tidak ada yang salah diprediksi menjadi genus Bacillus maupun Staphylococcus begitu juga sebaliknya. Fragmen dari genus Bacillus dan Staphylococcus tidak ada yang salah prediksi menjadi genus
Agrobacterium. Pada k = 5 dan k = 7 terjadi peningkatan fragmen yang salah
prediksi dari genus Bacillus menjadi Staphylococcus dan sebaliknya dari
Staphylococcus menjadi Bacillus. Hasil akurasi, sensitivitas, dan spesifisitas yang
didapatkan pada percobaan ini masing-masing dapat dilihat pada Tabel 14 dan Tabel 15. Untuk mengetahui fragmen yang salah diklasifikasi dapat dilihat pada
confusion matrix pada Tabel 13.
Pada pengujian dataset organisme uji diperoleh nilai akurasi yang semakin meningkat berdasarkan jumlah fragmen yang diuji. Hal yang serupa juga ditunjukkan oleh sensitivity dan specificity. Hasil akurasi terbaik diperoleh dari
Tabel 12 Confusion matrix dataset organisme uji dengan panjang fragmen 5 kbp Kelas aktual
Kelas prediksi
k = 3 k = 5 k = 7
Agr Bac Sta Agr Bac Sta Agr Bac Sta Agrobacterium 1285 0 0 1283 2 0 1279 6 0
Bacillus 5 2281 17 5 2282 16 0 2286 17
Staphylococcus 0 139 1273 0 147 1265 0 156 1256
Tabel 13 Confusion matrix dataset organisme uji dengan panjang fragmen 10 kbp Kelas aktual
Kelas prediksi
k = 3 k = 5 k = 7
Agr Bac Sta Agr Bac Sta Agr Bac Sta Agrobacterium 1258 0 0 1258 0 0 1258 0 0
Bacillus 0 2288 4 0 2286 6 0 2286 6
Staphylococcus 0 82 1368 0 93 1357 0 115 1335
16
Percobaan 1 dengan nilai k = 3 mencapai 98.28%. Sensitivitas dan spesifisitas dengan nilai terbaik juga didapat dari percobaan yang sama. Nilai sensitivitas dan spesifisitas yang diperoleh berturut-turut yaitu 98.06% dan 98.95% sebagaimana dilihat dari Tabel 16.
Perbandingan Akurasi, Sensitivitas, dan Spesifisitas antara Dataset Organisme Latih dengan Organisme Uji
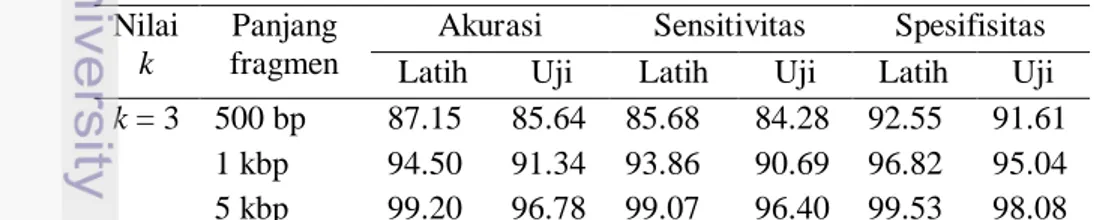
Perbandingan akurasi, sensitivitas, dan spesifisitas dari dataset organisme latih dan organisme uji ditunjukkan pada Tabel 17. Dari tabel tersebut tampak bahwa hasil akurasi, sensitivitas, dan spesifisitas antara organisme latih lebih tinggi daripada organisme uji. Ini mungkin disebabkan karena ada tumpang tindih urutan antara fragmen yang berbeda dalam spesies atau antar spesies dalam genus yang sama. Sehingga menyebabkan salah klasifikasi dengan menetapkan fragmen ke genus yang berbeda.
Tabel 14 Hasil akurasi pada pengujian dataset organisme uji Panjang fragmen Akurasi (%) k = 3 k = 5 k = 7 500 bp 85.64 86.18 86.52 1 kbp 91.34 91.92 92.04 5 kbp 96.78 96.60 96.42 10 kbp 98.28 98.02 97.58
Tabel 15 Hasil sensitivitas dan spesifisitas organisme uji Panjang fragmen Sensitivitas (%) Spesifisitas (%) k = 3 k = 5 k = 7 k = 3 k = 5 k = 7 500 bp 84.28 84.65 84.89 91.61 91.85 92.00 1 kbp 90.69 91.30 91.32 95.04 95.37 95.40 5 kbp 96.40 96.17 95.92 98.08 97.96 97.84 10 kbp 98.06 97.77 97.27 98.95 98.80 98.53
Tabel 16 Perbandingan akurasi, sensitivitas, dan spesifisitas organisme uji
Kriteria Tingkat (%) k = 3 k = 5 k = 7 500 bp 1 kbp 5 kb 10 kbp 500 bp 1 kbp 5 kbp 10 kbp 500 bp 1 kbp 5 kbp 10 kbp Akurasi 85.64 91.34 96.78 98.28 86.18 91.92 96.60 98.02 86.52 92.04 96.42 97.58 Sensitivitas 84.28 90.69 96.40 98.06 84.65 91.30 96.17 97.77 84.89 91.32 95.92 97.27 Spesifisitas 91.61 95.04 98.08 98.95 91.85 95.37 97.96 98.80 92.00 95.40 97.84 98.53
Tabel 17 Perbandingan akurasi, sensitivitas, dan spesifisitas organisme latih dan uji
Nilai
k
Panjang fragmen
Akurasi Sensitivitas Spesifisitas Latih Uji Latih Uji Latih Uji
k = 3 500 bp 87.15 85.64 85.68 84.28 92.55 91.61 1 kbp 94.50 91.34 93.86 90.69 96.82 95.04 5 kbp 99.20 96.78 99.07 96.40 99.53 98.08
17
Pengujian Dataset Organisme Latih dan Organisme Uji Berdasarkan Genus
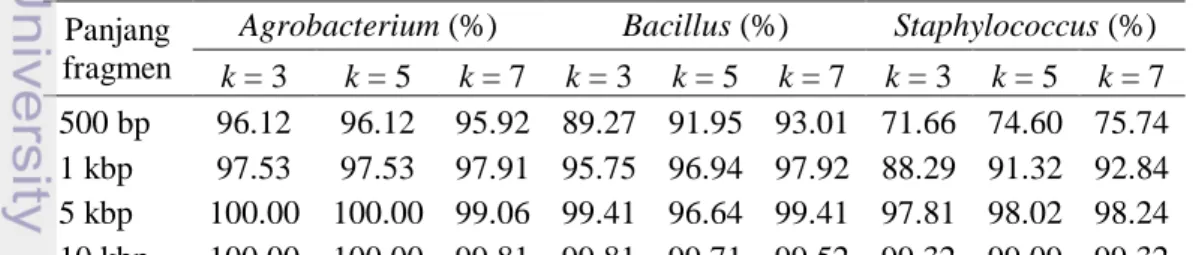
Tabel 18 dan Tabel 19 menunjukkan nilai sensitivitas dari setiap genus dengan panjang fragmen berbeda masing-masing dari organisme latih dan organisme uji. Sensitivitas dari penelitian ini sangat tinggi ketika menggunakan spesies dari genus Agrobacterium, bahkan ketika hanya menggunakan fragmen dengan panjang 500 bp. Kinerja algoritme meningkat dengan meningkatnya panjang fragmen. Untuk organisme latih, sensitivitas dengan panjang fragmen 500 bp rata-rata mencapai 96.05%. Kecenderungan ini juga diperlihatkan oleh dataset yang mewakili organisme uji yang mencapai rata-rata sensitivitas 98.10%. Ini berarti pengujian yang dilakukan berhasil mengenali fragmen dari spesies yang berasal dari genus Agrobacterium sekitar 96% dengan benar.
Namun, sensitivitas dari penelitian ini hanya mencapai rata-rata 74% untuk organisme latih dan 63.34% untuk organisme uji ketika mengklasifikasi fragmen spesies yang termasuk dalam genus Staphylococcus dengan panjang fragmen 500 bp. Penyebab hal tersebut mungkin karena terdapat banyak fragmen dari genus
Staphylococcus yang diprediksi ke dalam Bacillus. Hal ini dimungkinkan karena Staphylococcus dan Bacillus berasal dari satu ordo yang sama yaitu Bacilalles.
Kesalahan klasifikasi fragmen mungkin terjadi karena terdapat overlap sekuens antara strain yang berbeda dalam spesies di ordo yang sama. Sehingga perlu dipertimbangkan untuk memperluas penelitian ini untuk mengklasifikasikan fragmen ke tingkat taksonomi yang lebih tinggi.
Tabel 18 Hasil sensitivitas organisme latih Panjang
fragmen
Agrobacterium (%) Bacillus (%) Staphylococcus (%) k = 3 k = 5 k = 7 k = 3 k = 5 k = 7 k = 3 k = 5 k = 7
500 bp 96.12 96.12 95.92 89.27 91.95 93.01 71.66 74.60 75.74 1 kbp 97.53 97.53 97.91 95.75 96.94 97.92 88.29 91.32 92.84 5 kbp 100.00 100.00 99.06 99.41 96.64 99.41 97.81 98.02 98.24 10 kbp 100.00 100.00 99.81 99.81 99.71 99.52 99.32 99.09 99.32
18
Tabel 20 dan Tabel 21 menunjukkan nilai spesifisitas dari setiap genus dengan panjang fragmen berbeda masing-masing dari organisme latih dan organisme uji. Hasil spesifisitas dari penelitian ini juga sangat tinggi ketika menggunakan spesies dari genus Agrobacterium, walaupun hanya memakai fragmen dengan panjang 500 bp. Kinerja algoritme meningkat dengan meningkatnya panjang fragmen. Untuk organisme latih, spesifisitas dengan panjang fragmen 500 bp rata-rata mencapai 99.26%. Kecenderungan ini juga diperlihatkan oleh dataset yang mewakili organisme uji yang mencapai rata-rata akurasi 99.10%.
Spesifisitas yang didapat dari genus Bacillus dengan panjang fragmen 500 bp hanya mencapai rata-rata 85.91% untuk organisme latih dan 80.42% untuk organisme uji. Hal tersebut mungkin disebabkan karena terdapat banyak fragmen dari genus Bacillus yang diprediksi ke dalam Staphylococcus. Hal ini dimungkinkan karena Bacillus dan Staphylococcus berasal dari satu ordo yang sama yaitu Bacilalles.
Tabel 19 Hasil sensitivitas organisme uji Panjang
fragmen
Agrobacterium (%) Bacillus (%) Staphylococcus (%) k = 3 k = 5 k = 7 k = 3 k = 5 k = 7 k = 3 k = 5 k = 7
500 bp 97.90 98.13 98.29 91.36 92.53 93.25 63.59 63.29 63.14 1 kbp 99.17 98.95 99.02 95.27 95.63 96.25 77.63 79.33 78.69 5 kbp 100.00 99.84 99.53 99.04 99.09 99.26 90.16 89.59 88.95 10 kbp 100.00 100.00 100.00 99.83 99.74 99.74 94.34 93.59 92.07
Tabel 20 Hasil spesifisitas organisme latih Panjang
fragmen
Agrobacterium (%) Bacillus (%) Staphylococcus (%) k = 3 k = 5 k = 7 k = 3 k = 5 k = 7 k = 3 k = 5 k = 7
500 bp 99.06 99.33 99.39 84.94 86.19 86.61 93.65 95.25 95.89 1 kbp 99.93 99.93 100.00 93.32 94.84 95.55 97.21 97.92 98.64 5 kbp 99.66 98.66 100.00 98.99 99.09 98.68 99.94 99.05 99.61 10 kbp 100.00 100.00 100.00 99.69 99.59 99.59 99.87 99.81 99.68
Tabel 21 Hasil spesifisitas organisme uji Panjang
fragmen
Agrobacterium (%) Bacillus (%) Staphylococcus (%) k = 3 k = 5 k = 7 k = 3 k = 5 k = 7 k = 3 k = 5 k = 7
500 bp 98.95 99.09 99.27 80.47 80.39 80.39 95.42 96.07 96.35 1 kbp 99.78 99.81 99.92 88.12 88.89 88.60 97.22 97.41 97.69 5 kbp 99.87 99.87 100.00 94.85 94.48 93.99 99.53 99.55 99.53 10 kbp 100.00 100.00 100.00 96.97 96.57 95.75 99.89 99.83 99.83
19
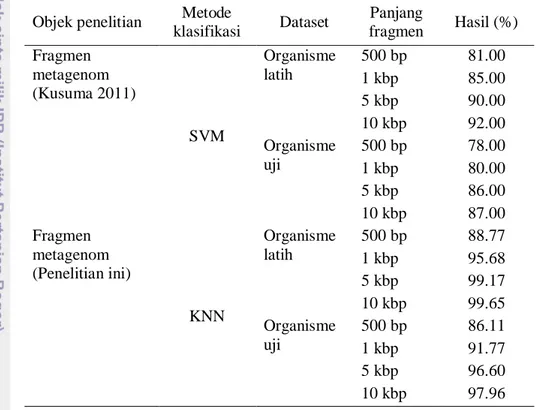
Dari Tabel 24 dapat dilihat bahwa penelitian ini memiliki kesamaan objek penelitian dengan penelitian Kusuma dan Akiyama (2011) yaitu fragmen metagenom. Penelitian Kusuma dan Akiyama (2011) menerapkan algoritme
characterization vector dalam ekstraksi fitur dan mengimplementasikan SVM
sebagai classifier menghasilkan akurasi terbaik sebesar 92% pada Percobaan 4 untuk fragmen latih dengan panjang 10 kbp. Penelitian ini merujuk pada data yang sama (Kusuma dan Akiyama 2011) dengan menerapkan spaced n-mers sebagai ekstraksi fitur dan KNN sebagai classifier dan mendapatkan akurasi tertinggi pada percobaan yang sama mencapai rata-rata sebesar 99.65%.
Tabel 22 Perbandingan hasil akurasi penelitian terkait Objek penelitian Metode
klasifikasi Dataset Panjang fragmen Hasil (%) Fragmen metagenom (Kusuma 2011) SVM Organisme latih 500 bp 81.00 1 kbp 85.00 5 kbp 90.00 10 kbp 92.00 Organisme uji 500 bp 78.00 1 kbp 80.00 5 kbp 86.00 10 kbp 87.00 Fragmen metagenom (Penelitian ini) KNN Organisme latih 500 bp 88.77 1 kbp 95.68 5 kbp 99.17 10 kbp 99.65 Organisme uji 500 bp 86.11 1 kbp 91.77 5 kbp 96.60 10 kbp 97.96
20
SIMPULAN DAN SARAN
Simpulan
Berdasarkan penelitian yang telah dilakukan dapat disimpulkan bahwa: 1 Ekstraksi fitur spaced n-mers dan algoritme KNN dapat diimplementasikan
dalam klasifikasi fragmen metagenom pada tingkat genus.
2 Klasifikasi KNN dengan data latih yang memiliki panjang 10 kbp merupakan dataset dengan akurasi terbaik dan hasil akurasi tersebut lebih tinggi daripada penelitian Kusuma dan Akiyama (2011).
3 Nilai akurasi maksimal yang dicapai yaitu 99.75% pada dataset organisme latih dengan panjang fragmen 10 kbp dengan nilai k = 3, dan nilai akurasi minimum 85.64% pada dataset organisme latih dengan panjang fragmen 500 bp yaitu pada nilai k = 3.
4 Nilai sensitivitas dan spesifisitas maksimal yang diperoleh masing-masing yaitu 99.71% dan 99.85%, untuk dataset organisme latih 10 kbp serta nilai sensitivitas dan spesifisitas minimum yaitu 84.28% dan 91.61% dari dataset organisme uji 500 bp.
5 Penelitian ini mencapai sensitivitas dan spesifisitas 100% ketika mengidentifikasi spesies yang berasal dari genus Agrobacterium. Hal ini disebabkan karena genus Bacillus dan Staphylococcus berasal dari ordo yang sama yaitu Bacilalles sehingga memiliki kemiripan yang lebih tinggi.
Saran
Beberapa hal yang dapat dikembangkan lebih lanjut dari penelitian ini antara lain:
1 Melakukan klasifikasi dengan tingkat taksonomi yang lebih tinggi karena pada penelitian ini belum dapat menghasilkan akurasi yang baik untuk klasifikasi genus jika ordonya sama.
2 Menambah jenis spesies dan genus agar fragmen yang diidentifikasi lebih bervariasi dan merepresentasikan data pada lingkungan ekosistem yang sebenarnya.
3 Melakukan penelitian dengan menggunakan fragmen yang memiliki panjang tidak seragam karena penelitian ini hanya menggunakan data yang memiliki panjang fragmen tetap yaitu 500 bp, 1 kbp, 5 kbp, dan 10 kbp.
DAFTAR PUSTAKA
Akobeng AK. 2007. Understanding diagnostic tests 1: sensitivity, specificity, and predictive values. Acta Paediatrica. 96(3):338-341.
de Carvalho Jr SA. 2003. Sequence alignment algorithms [disertasi]. London (GB): University for London.
21
Fanani MZ. 2011. Eksplorasi novel gen glikosida hidrolase untuk degradasi biomassa: pendekatan secara metagenomik [Internet]. [diunduh 2013 Nov 10]. Tersedia pada: http://mazfanani.wordpress.com/2011/06/14/ eksplorasi- novel-gen-glikosida-hidrolase-untuk-degradasi-biomassa-pendekatan-secara-metagenomik/.
Kohavi R, Provost F. 1998. On applied research in machine learning. Machine
Learning. 30(2):127-132.
Kusuma WA, Akiyama Y. 2011. Metagenome fragments classification based on characterization vectors. Di dalam: Proceedings of International Conference
on Bioinformatics and Biomedical Technology; 2011 Mar; Sanya, China.
hlm 50-54.
Kusuma WA. 2012. Combine approaches for improving the performance of de novo DNA sequence assembly & metagenomic classification of short fragments from next generation sequences [disertasi]. Tokyo (JP): Tokyo Institute of Technology.
Larose DT. 2005. Discovering Knowledge in Data: An Introduction to Data
Mining.New Jersey (US): Wiley.
Song Y, Huang J, Zhou D, Zha H, Giles CL. 2007. IKNN: Informative k-nearest neighbor pattern classification. Di dalam: Knowledge Discovery in
Databases: PKDD 2007. hlm 248-264.
Wu H. 2008. PCA-based linear combinations of oligonucleotide frequencies for metagenomic DNA fragment binning. Di dalam: Computational Intelligence
22
Lampiran 1 Pembagian subset untuk panjang fragmen 500 bp
Subset Data latih (indeks) Data uji (indeks)
Fold 1 517-2579, 3623-7795, 8237-10 000 1-516, 2580-3622, 7796-8236
Fold 2 1-516 & 1033-2579, 2580-3622 & 4666-7795, 7796-8236 & 8678-10 000
517-1032, 3623-4665, 8237-8677
Fold 3 1-1032 & 1549-2579, 2580-4665 & 5709-7795, 7796-8677 & 9119-10 000
1033-1548, 4666-5708, 8678-9118
Fold 4 1-1548 & 2065-2579, 2580-5708 & 6752-7795, 7796-9118 & 9560-10 000
1549-2064, 5709-6751, 9119-9559
Fold 5 1-2064, 2580-6751, 7796-9559 2065-2579, 6752-7795, 9560-10 000
Lampiran 2 Pembagian subset untuk panjang fragmen 1 kbp
Subset Data latih (indeks) Data uji (indeks)
Fold 1 529-2639, 3652-7699, 8160-10 000 1-528, 2640-3651, 7700-8159
Fold 2 1-528 & 1057-2639, 2640-3651 & 4664-7699, 7700-8159 & 8620-10 000
529-1056, 3652-4663, 8160-8619
Fold 3 1-1056 & 1585-2639, 2640-4663 & 5676-7699, 7700-8619 & 9080-10 000
1057-1584, 4664-5675, 8620-9079
Fold 4 1-1056 & 2113-2639, 2640-5675 & 6688-7699, 7700-9079 & 9080-10 000
1585-2112, 5676-6687, 9080-9539
Fold 5 1-2112, 2640-6687, 7700-9539 2113-2639, 6688-7699, 9540-10 000
Lampiran 3 Pembagian subset untuk panjang fragmen 5 kbp
Subset Data latih (indeks) Data uji (indeks)
Fold 1 533-2661, 3674-7724, 8181-10000 1-532, 2662-3673, 7725-8180
Fold 2 1-532 & 1065-2661, 2662-3673 & 4687-7724, 7725-8180 & 8636-10 000
533-1064, 3674-,4686, 8181-8635
Fold 3 1-1064 & 1597-2661, 2662-4686 & 5700-7724, 7725-8635 & 9091-10 000
1065-1596, 4687-5699, 8636-9090
Fold 4 1-1064 & 2129-2661, 2662-5699 & 6713-7724, 7725-9090 & 9546-10 000
1597-2128, 5700-6712, 9091-9545
Fold 5 1-2128, 2662-6712, 7725-9545 2129-2661, 6713-7699, 9546-10 000
23
Lampiran 4 Pembagian subset untuk panjang fragmen 10 kbp
Subset Data latih (indeks) Data uji (indeks)
Fold 1 527-2630, 3666-7804, 8244-10 000 1-526, 2631-3665, 7805-8243
Fold 2 1-526 & 1053-2630, 2631-3665 & 4701-7804, 7805-8243 & 8683-10 000
527-1052, 3666-4700, 8244-8682
Fold 3 1-1052 & 1579-2630, 2631-4700 & 5736-7804, 7805-8682 & 9122-10 000
1053-1578, 4701-5735, 8683-9121
Fold 4 1-1578 & 2105-2630, 2631-5735 & 6771-7804, 7805-9121 & 9561-10 000
1579-2104, 5736-6770, 9122-9560
Fold 5 1-2104, 2631-6770, 7805-9560 2105-2630, 6771-, 9561-10 000
Lampiran 5 Dataset organisme latih
Spesies Genus
Agrobacterium radiobacter K48 chromosome 2 Agrobacterium Agrobacterium tumefaciens str. C58 chromosome circular
Agrobacterium vitis S4 chromosome 1
Bacillus amyloliquefaciens FZB42 Bacillus Bacillus anthracis str. ‘Ames Ancestor’
Bacillus cereus 03BB102
Bacillus pseudofirmus OF4 chromosome
Staphylococcus aureus subsp. Sureus JH1 Staphylococcus Staphylococcus epidermidis 1228 chromosome
Staphylococcus haemolyticus JCSC1435 chromosome
Lampiran 6 Dataset organisme uji
Spesies Genus
Agrobacterium radiobacter K48 chromosome 1 Agrobacterium Agrobacterium tumefaciens str. C58 chromosome linear
Agrobacterium vitis S4 chromosome 2
Bacillus pumilus SAFR-032 Bacillus
Bacillus subtilis subsp. subtilis str. 16B chromosome Bacillus thuringiensis str. Al Hakam chromosome
Staphylococcus carnosus subsp. carnosus TM300 chromosome
Staphylococcus Staphylococcus lugdunensis HKU09-01 chromosome
Staphylococcus saprophyticus subsp. saprophyticus ATCC
24
Keterangan :
• agr = Agrobacterium • bac = Bacillus • sta = Staphylococcus
Lampiran 7 Confusion matrix percobaan 1 pada dataset organisme latih dengan panjang fragmen 500 bp
genus
fold 1 fold 2
k = 3 k = 5 k = 7 k = 3 k = 5 k = 7 agr bac sta agr bac sta agr bac sta agr bac sta agr bac sta agr bac sta agr 515 1 0 515 1 0 515 1 0 507 9 0 511 5 0 508 8 0 bac 286 737 20 307 719 17 322 705 16 1 879 163 1 896 146 1 897 145 sta 0 117 324 0 118 323 0 112 329 0 102 339 0 88 353 0 93 348 genus fold 3 fold 4 k = 3 k = 5 k = 7 k = 3 k = 5 k = 7 agr bac sta agr bac sta agr bac sta agr bac sta agr bac sta agr bac sta agr 504 12 0 503 13 0 503 13 0 488 27 1 488 27 1 487 29 0 bac 1 885 157 1 893 149 1 907 135 1 893 149 1 905 137 1 924 118 sta 0 111 330 1 113 327 1 100 340 0 113 328 0 97 344 0 84 357 genus fold 5 k = 3 k = 5 k = 7 agr bac sta agr bac sta agr bac sta agr 495 20 0 495 20 0 494 21 0
bac 13 932 99 10 960 74 9 971 64
sta 1 124 316 0 112 329 0 107 334
Lampiran 8 Confusion matrix percobaan 1 pada panjang fragmen 500 bp dengan nilai fold terbaik
Genus k = 3 k = 5 k = 7
Agr Bac Sta Agr Bac Sta Agr Bac Sta Agrobacterium 495 20 0 495 20 0 494 21 0
Bacillus 13 932 99 10 960 74 9 971 64
25
Lampiran 9 Confusion matrix percobaan 2 pada dataset organisme latih dengan panjang fragmen 1 kbp
genus
fold 1 fold 2
k = 3 k = 5 k = 7 k = 3 k = 5 k = 7 agr bac sta agr bac sta agr bac sta agr bac sta agr bac sta agr bac sta agr 528 0 0 528 0 0 528 0 0 528 0 0 525 3 0 525 3 0 bac 77 925 10 80 924 8 96 906 10 96 906 10 2 910 100 2 918 92 sta 0 69 391 0 69 393 0 69 391 0 69 391 0 52 408 0 47 413 genus fold 3 fold 4 k = 3 k = 5 k = 7 k = 3 k = 5 k = 7 agr bac sta agr bac sta agr bac sta agr bac sta agr bac sta agr bac sta agr 524 4 0 523 5 0 524 4 0 515 13 0 514 14 0 513 15 0 bac 0 908 104 0 908 104 0 914 98 2 905 105 1 919 92 1 927 84 sta 0 37 423 0 33 427 0 25 435 0 38 422 0 37 423 0 36 424 genus fold 5 k = 3 k = 5 k = 7 agr bac sta agr bac sta agr bac sta agr 514 12 1 514 11 2 516 11 0 bac 1 969 42 1 981 30 0 991 21 sta 0 54 407 0 40 421 0 33 428
Lampiran 10 Confusion matrix percobaan 2 pada panjang fragmen 1 kbp dengan nilai fold terbaik
Genus k = 3 k = 5 k = 7
Agr Bac Sta Agr Bac Sta Agr Bac Sta Agrobacterium 514 12 1 514 11 2 516 11 0
Bacillus 1 969 42 1 981 30 0 991 21
Staphylococcus 0 54 407 0 40 421 0 33 428