HUBUNGAN KUANTITATIF STRUKTUR AKTIVITAS OBAT ANTIBIOTIK,
ANALGETIK, DAN ANTI KANKER
HUBUNGAN KUANTITATIF STRUKTUR AKTIVITAS
OBAT ANTI KANKER
STUDI QSAR DERIVAT 3- INDOLYL-3-HYDROXY
OXINDOLE
SEBAGAI ANTI-LEUKEMIA
PENDAHULUAN
3-substituted-3-hydroxyoxindole
C3 functionalised oxindole is featured heterocyclic nucleus in a number of
natural products as well as medicinally relevant compounds.
In particular 3-substituted-3-
hydroxyoxindole is an emerging new
scaffold for drug discovery with a broad
spectrum of biological activities including
antiviral, antibacterial, antitubercular, anti-
inflammatory, antiangiogenic, antifungal,
anticonvulsant and new targets for cancer
chemotherapy.
3-substituted-3-hydroxyoxindole
Several pharmacologically active alkaloids such as Maremycin A and B,
Flustramnol, Arundaphine, Donaxaridine, CPC-1,
Welwitindoline C in
addition to several others
contain 3-hydroxyoxindole.
3-indolyl-3-hydroxy oxindole
The combination of indole and oxindole motif with a hydroxybearing C3
substitution were envisioned to generate biologically
attractive architectures.
Struktur Induk
3-indolyl-3-hydroxy oxindole derivatives
In vitro cytotoxic activity
Human cancer cell lines such as leukemia (U937, THP-1), lung (A549) and breast
cancer (MCF7) cell lines.
Positive control ➔ Etoposide
Result ➔ IC 50 data
Slide Title
STUDI QSAR
Tahapan QSAR
Gambar struktur derivat
(ChemDraw Ultra)
Optimasi geometri (HyperChem)
Pemilihan, penentuan nilai deskriptor, dan cross validation, 12 deskriptor
(MOE)
Penentuan regresi linear deskriptor, 9
deskritor (SPSS)
Validasi deskriptor (metode LOO) Penentuan regresi
linear kombinasi 2, 3, 4, dst. dari
deskriptor Validasi kombinasi yang
dipilih, 5 deskriptor (metode LOO) Persamaan
QSAR
Desain molekulbaru (lakukan tahap 1,2,
dan 3)
Penentuan IC50 prediksi molekul baru
(dengan persamaan QSAR)
Docking (Autodock)
ChemDraw Ultra
HyperChem
MOE
Deskriptor awal
No Deskriptor Simbol umum Simbol pada software MOE
1. Momen dipol total μ AM1_dipole
2. Energi Total ETot AM1_E
3. Energi Elektronik EEle AM1_Eele
4. Energi HOMO EHOMO AM1_HOMO
5. Energi LUMO ELUMO AM1_LOMO
6. Panas pembentukan HF AM1_HF
7. Luas permukaan hidrofobik A ASA_H
8. Globularitas GLOB glob
9. Koefisien partisi logP, (logP)2 logP(o/w), (logP(o/w))2
10. Logaritma kelarutan (solubility) S, logS S, logS
11. Refraktivitas molar MR mr
12. Volume van der Waals Vw vol
Deskriptor awal
Parameter Deskriptor Simbol umum Simbol pada Software MOE
Hidrofobik
Luas Permukaaan hidrofobik A ASA_H
Koefisien Partisi Log P / (log P)² Log P (o/w), (log P (o/w))²
Logaritma Kelarutan S, log S S, log S
Sterik
Globularitas GLOB glob
Refraktivitas molar MR Mr
Volume van der waals Vw Vol
Elektronik
Momen dipol total µ AM 1_dipole
Energi Total ETot AM 1_E
Energi Elektronik EEle AM 1_ Eele
Energi HOMO EHOMO AM 1_HOMO
Energi LUMO ELUMO AM 1_LUMO
Panas Pembentukan HF AM 1_ HF
SPSS
Autodock
HASIL
Mol Sebelum Optimasi Setelah Optimasi Mol Sebelum Optimasi Setelah Optimasi
7 15
8 16
9 19
14 24
H
Cl N H
O OH H N
H
H
F3CO
H
N
H O OH H N
H
H
H
H N
O OH H N
H
H
Cl
H
N
H O OH H N
H
Br
Br
H N H
O OH H N
H
Br
I
H N
H O OH H N
H
H
H N
O OH H N
H
Br
OCH3 N
O I
OH N
H H
H H
H
Cl
N H
O
N O
H H
H
H H
H H H
H
H N
H O O N H
H
H
O F
F F
H H
H H H H
H
H
N O O
N H
H H
H H
H H
H H H H
H
H H
H H
Cl
H N
H O O N
H
H Br
H
H H
H H H
Br
H N
H O O N
H
H Br
H
H H
H H H
H
H N
O O
N H
H
Br
H H
H H
H H H H
H
H H
H H
I
H N
H O O N
H H
H
H H
H H H
H H
H
N O
I O
HN H
H H
O
H
H H
H H H
H H
H
Mol Sebelum Optimasi Setelah Optimasi Mol Sebelum Optimasi Setelah Optimasi
25 33
26 34
31 35
32 36
N O F
OH N
H H
H H
Br
N O I
OH N
H H
H H
F
Cl
H N
O OH H N
H
Br
Br
H
H N
O OH H N
H
Br
Br
H
H N
O OH H N
H
Br
Cl
Cl
H N
O OH H N
H
Br
Cl
Cl
H N
O OH H N
H
Br
Cl
H N
O OH
N H
H
H
N O F
O HN
H
H H
Br
H
H H
H H H
N O
I O
HN H
H H
F
H
H H
H H H
Cl
H N
O O
N
H
H Br
Br H
H
H H
H H H H
H
H H
H
H
H
N O O
N
H
H Br
H Br H
H H
H H H H
H
H H
H
H
H
N O O
N
H
H Br
H Cl H
H H
H H H H
H
H H
H
Cl
H N
O O
N
H
H Br
H Cl H
H H
H H H H
H
H H
H
Cl
H N
O O
N
H H
Br
H H
H H
H H H H
H
H H
H H
Cl
H
N O O
N
H H
H
H H
H H
H H H H
H
H H
H H
Mol Sebelum Optimasi Setelah Optimasi 37
38
40
41
H
H
N
O OH
N H
H
H
Cl
Cl
H
N
O OH H N
H
H
Cl
H
H
N
O OH
H N
H
O
Cl
Cl
H
N
O OH
N H
H
O
Cl
H
H
N
O O
N H
H H
H Cl
H
H H
H
H H H
H
H H
H
Cl
H
N
O O
N
H
H H
H Cl
H
H H
H
H H H
H
H H
H
H
H
N
O O
N H
H
O
H Cl
H
H H
H H
H H
H H
H
H
H H
H
Cl
H
N
O O
N
H
H O
H Cl
H
H H
H
H H H
H H
H
H
H H
H
Nilai deskriptor masing-masing molekul
mol AM1_dipoleAM1_E AM1_Eele AM1_HF AM1_HOMOAM1_LUMOASA_H glob logP(o/w) logS mr vol 7 4,444983 -83631,5 -542077 15,49977 -8,67403 -0,1653 483,4416 0,08718 3,126 -4,29892 8,040628 262,625 8 3,310871 -118942 -765207 -178,435 -8,73847 -0,51603 513,6326 0,078778 3,97176 -4,75396 8,285371 280,125 9 3,89138 -97883,2 -768968 54,78258 -8,57523 0,04668 592,9229 0,093887 4,521 -5,22654 10,51146 349,375 14 4,786722 -91462,9 -584232 20,56008 -8,87705 -0,33847 520,712 0,083787 4 -5,38931 8,776903 289 15 4,689982 -90990,4 -582275 32,25204 -8,88754 -0,39019 535,6302 0,08438 4,206 -5,74541 8,995439 301,875 16 3,271004 -86615,1 -581061 31,03269 -8,62362 -0,27871 534,0525 0,076499 4,098 -5,03007 9,330148 296,375 19 5,352083 -105714 -818519 61,40577 -8,77719 -0,16509 627,9674 0,095227 5,356 -6,31693 11,26298 372,75 24 4,31698 -93993,2 -629796 1,91129 -8,47614 -0,2619 550,6915 0,06451 3,756 -4,60653 9,524138 304,5 25 4,622634 -94029,2 -590175 -17,0999 -8,88592 -0,35092 500,1594 0,082499 3,561 -4,95 8,345777 278,5 26 4,881792 -93891,5 -588888 -6,2884 -8,85766 -0,37737 508,7875 0,084955 3,953 -4,85113 8,934822 280,5 31 5,350748 -121850 -920167 58,23035 -8,90826 -0,36071 691,8878 0,096292 6,783 -8,14161 12,48101 415,5 32 6,051395 -113543 -879986 68,1432 -8,74596 -0,14336 646,7033 0,090219 6,152 -7,40732 11,9857 399,125 33 5,910593 -114017 -882925 55,08483 -8,74735 -0,1372 636,6988 0,08857 5,946 -7,05122 11,76717 388,375 34 5,019662 -122321 -938673 48,14453 -8,81821 -0,24581 660,9938 0,089624 6,575 -7,78551 12,23502 401,875 35 4,418902 -114019 -872402 52,92743 -8,85752 -0,28484 652,339 0,094653 5,985 -7,05122 11,76343 390,875 36 3,129496 -106187 -821681 48,19671 -8,64548 -0,1742 615,3097 0,095876 5,15 -5,96083 11,01324 364,375 37 4,40035 -106185 -831693 50,14769 -8,54223 -0,00088 596,7639 0,088794 5,111 -5,96083 11,01698 362,25 38 3,694886 -114489 -886327 43,1403 -8,6177 -0,1302 622,9453 0,089074 5,74 -6,69512 11,48623 374,875 40 4,827447 -117157 -938217 13,67499 -8,35281 0,0159 639,5245 0,072303 5,104 -6,01121 11,66838 387,125 41 4,29514 -125462 -995053 6,64348 -8,41779 -0,10931 664,7637 0,07265 5,733 -6,7455 12,14021 403,125
Cross validation: ZSCORE
mol $PRED $RES $Z-SCORE $XPRED $XRES $XZ-SCORE 7 2,069236 -0,13403 0,802134 2,102764 -0,16756 0,997661 8 1,162501 -0,02515 0,150498 1,194522 -0,05717 0,333897 9 1,339831 0,469864 2,811979 1,219459 0,590236 4,852724 14 1,887616 0,105908 0,633826 1,871156 0,122369 0,722223 15 1,897019 0,038035 0,227625 1,891104 0,04395 0,256751 16 1,939156 0,000912 0,005459 1,938974 0,001094 0,006379 19 1,138568 0,012802 0,076617 1,137239 0,014131 0,082443 24 1,744086 0,095894 0,573895 1,733863 0,106118 0,624719 25 1,849 0,031528 0,188683 1,843276 0,037252 0,217523 26 1,853646 -0,05846 0,349874 1,864304 -0,06912 0,404648 31 0,725065 0,057407 0,343564 0,715339 0,067134 0,392959 32 0,905676 -0,06494 0,38866 0,911354 -0,07062 0,413644 33 0,893654 -0,02619 0,156715 0,895955 -0,02849 0,166285 34 0,671719 0,115032 0,688428 0,653147 0,133605 0,790285 35 0,921587 -0,06911 0,413582 0,927108 -0,07463 0,437341 36 1,125963 -0,4244 2,539864 1,168213 -0,46664 3,38838 37 1,099387 -0,25615 1,532994 1,131294 -0,28806 1,803688 38 0,88041 -0,03469 0,207621 0,883505 -0,03779 0,220674 40 0,718852 0,029336 0,175564 0,714559 0,033629 0,196334 41 0,494029 0,136399 0,816302 0,465534 0,164893 0,981821
MODEL SUMMARY
R Square
Change F Change df1 df2
Sig. F Change
1 .992a ,985 ,959 ,10856268908 ,985 38,388 12 7 ,000
2 .992b ,985 ,964 ,10209171704 ,000 ,075 1 7 ,792
3 .992c ,985 ,968 ,09688636800 ,000 ,106 1 8 ,754
4 .991d ,982 ,965 ,10047580621 -,003 1,755 1 9 ,218
a. Predictors: (Constant), vol, AM1_HOMO, AM1_dipole, AM1_HF, glob, AM1_LUMO, logS, AM1_E, logP(o/w), ASA_H, AM1_Eele, mrb. Predictors: (Constant), vol, AM1_HOMO, AM1_dipole, AM1_HF, glob, AM1_LUMO, AM1_E, logP(o/w), ASA_H, AM1_Eele, mr
c. Predictors: (Constant), vol, AM1_HOMO, AM1_dipole, AM1_HF, glob, AM1_LUMO, AM1_E, logP(o/w), ASA_H, AM1_Eele d. Predictors: (Constant), vol, AM1_HOMO, AM1_dipole, AM1_HF, glob, AM1_LUMO, logP(o/w), ASA_H, AM1_Eele
Model Summary
Model R R Square
Adjusted R Square
Std. Error of the Estimate
Change Statistics
Validasi 9 deskriptor (metode LOO)
Model R R Square Adjusted R
Square
Std. Error of the Estimate
Sum of
Squares df Mean
Square F hitung Sig. F tabel F hitung/
Ftabel q2
vol, glob, AM1_LUMO, AM1_HF, LogP, E_ele, LogS, ASA_H, mr
0,991 0,982 0,965 0,100 5,411 9 0,601 59,552 0 2,714356 5,213486 0,8633
• R > 0,9
• R 2 > 0,8
• Fhit / Ftab > 1
• q 2 > 0,5
Kombinasi 2 deskriptor
Kombinasi 3 deskriptor
Kombinasi 4 deskriptor
Kombinasi 5 deskriptor
Validasi 5 deskriptor (metode LOO)
• R > 0,9
• R 2 > 0,8
• Fhit / Ftab > 1
• q 2 > 0,5
R R Square Adjusted R Square
Std. Error of the Estimate
Sum of Squares df Mean
Square F hitung Sig. F tabel F hitung/
Ftabel q2
.990 0,979497 0,970955 0,091475 4,797128847 5 0,959426 114,6585 .000b 3,105875 36,91665 0,905428
Persamaan QSAR
Standardized Coefficients
B Std. Error Beta
(Constant)
2,715 ,602 4,510 ,001
AM1_dipol
e ,001 ,037 ,002 ,039 ,969
AM1_Eele 4,996E-06 ,000 1,475 5,406 ,000
AM1_HF -,002 ,001 -,158 -1,186 ,259
AM1_LUM
O ,244 ,310 ,064 ,788 ,446
ASA_H ,004 ,002 ,535 1,857 ,088
a. Dependent Variable: Log IC50
Coefficientsa
Model
Unstandardized Coefficients
t Sig.
1
Persamaan QSAR
Log IC50 = 2.7148 + (0.0014 x AM1_dipole) + (4.9955 x 10-6 x AM1_Eele) + (- 0.0015 x AM1_HF) + (0.2441 + AM1_LUMO) + (0.0042 x ASA_H) Agar IC 50 rendah maka:
• AM1_dipole, AM1_Eele, AM1_LUMO, dan ASA_H harus kecil
• AM1_HF harus besar
y = 0,9795x + 0,026 R² = 0,9795
0 0,5 1 1,5 2 2,5
0 0,5 1 1,5 2 2,5
Log IC50 Experiment
Log IC50 Prediksi KURVA LOG IC50
Series1 Linear (Series1)
Pengaruh deskriptor
• AM1_dipole↓ = Polaritas↓ = energi attraktif ↓ a. Untuk molekul-molekul yang sejenis:
E = - (2μ 4 /3r 6 )(1/(k B T)
b. Untuk molekul-molekul yang berbeda:
E = - (2μ A 2 .μ B 2 /3r 6 )(1/(k B T)
• AM1_Eele ↓ = molekul dengan ikatan σ
Energi ikatan σ < π < n
Pengaruh deskriptor
• AM1_LUMO ↓ = tingkat energi terendah elektron yang tidak berpasangan ↓.
➔konfigurasi elektron ➔ jumlah elektron ↓
• ASA_H ↓ = permukaan hidrofobik ↓ = polaritas ↑
• AM1_HF ↑ = ∆H ↑ = ∆G ↑= reaksi kurang spontan
➔Dibutuhkannya gugus yang dapat menyebabkan
penyerapan kalor pada sistem (endoterm).
Perbandingan IC
50eksperimen dan IC
50prediksi
Mol IC
50Eksperimen IC
50Prediksi Mol IC
50Eksperimen IC
50Prediksi
7 86,14 95,5003 31 6,06 7,1649
8 13,72 15,8900 32 6,93 8,0362
14 98,52 75,2434 33 7,37 7,4060
15 86,11 82,8498 34 6,12 4,7420
16 87,11 88,0594 35 7,12 8,9738
19 14,17 13,7210 37 6,97 9,9180
24 69,18 66,2661 38 7,01 6,4804
25 75,95 65,2655 40 5,6 5,0619
26 62,4 68,3537 41 4,27 3,2050
Desain molekul baru
OCH3
41A N
O Cl
OH
N H H
H
Cl
R1
R2
OCH3
41B N
O Cl
OH
N H H
H
Cl
R1
R2
Struktur induk
Desain molekul baru
IC 50 prediksi molekul baru
Mol IC
50pred. Mol IC
50pred. Mol IC
50pred. Mol IC
50pred.
1A 1,123453435 9A 1,362101138 1B 1,214725567 9B 1,398453242
2A 0,983293244 10A 1,059087546 2B 1,087756294 10B 1,225660855
3A 1,209022771 11A 1,777006471 3B 1,452575924 11B 1,152763398
4A 0,803259988 12A 0,951419864 4B 0,893046271 12B 0,940023136
5A 1,00179975 13A 0,728348843 5B 1,019883466 13B 0,955230394
6A 0,942063195 14A 0,636698942 6B 0,953531488 14B 0,801235689
7A 1,045419741 15A 0,788349655 76 1,137382358 15B 1,104486355
8A 0,710227098 16A 0,522430521 8B 0,736081192 16B 0,588740169
Docking
• 41A16
• 41B16
• 41A14
Validasi docking
Ligan Etoposide
• Valid, if RMSD<2
Kesimpulan
1. Log IC50 = 2.7148 + (0.0014 x AM1_dipole) +(4.9955 x 10-6 x AM1_Eele) + (-0.0015 x AM1_HF) + (0.2441 + AM1_LUMO) + (0.0042 x
ASA_H)
2. Dari senyawa hasil eksperimen in-vitro, berdasarkan
hasil QSAR yang terbaik adalah senyawa no. 41
• Dari turunan senyawa 41 berdasarkan hasil docking diperoleh senyawa yang mirip dengan control
(etoposide) adalah senyawa 41A14
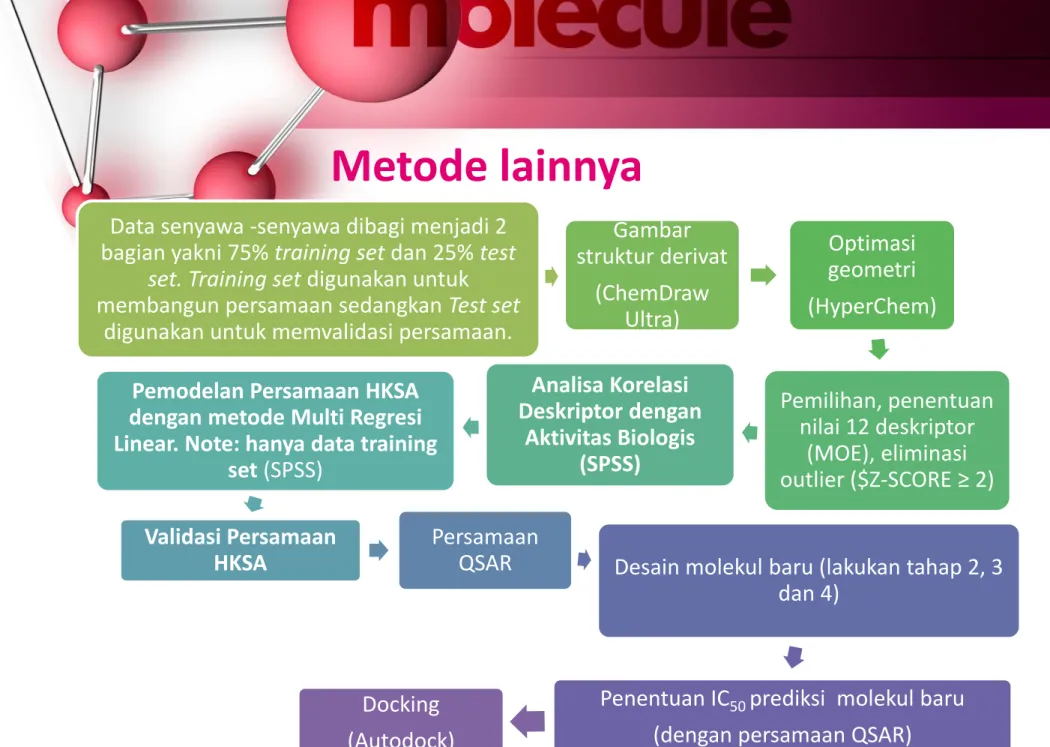
Data senyawa -senyawa dibagi menjadi 2 bagian yakni 75% training set dan 25% test
set. Training set digunakan untuk
membangun persamaan sedangkan Test set
digunakan untuk memvalidasi persamaan.
Gambar struktur derivat
(ChemDraw Ultra)
Optimasi geometri (HyperChem)
Pemilihan, penentuan nilai 12 deskriptor
(MOE), eliminasi outlier ($Z-SCORE ≥ 2) Analisa Korelasi
Deskriptor dengan Aktivitas Biologis
(SPSS) Pemodelan Persamaan HKSA
dengan metode Multi Regresi Linear. Note: hanya data training
set (SPSS)
Validasi Persamaan HKSA
Persamaan
QSAR Desain molekul baru (lakukan tahap 2, 3 dan 4)
Penentuan IC
50prediksi molekul baru (dengan persamaan QSAR)
Docking (Autodock)
Metode lainnya
Contoh penentuan training set dan test set untuk data 21 senyawa
• 75% training set dan 25% test set.
• Training set digunakan untuk
membangun model persamaan HKSA yang dipilih berdasarkan aktivitas
tertinggi, sedang, dan rendah.
• Test set digunakan untuk memvalidasi persamaan yang dibangun dari
training set .
Contoh: Pemodelan Persamaan HKSA
dengan metode Multi Regresi Linear (SPSS)
Model Deskriptor yang digunakan r r² SE Fhit Ftab Fhit/Ftab
1 Vol, AM1_HOMO, glob,
AM1_LUMO,AM1_HF,AM1_dipole,logS,AM1_E,A SA_H, logP(o/w), AM1_Eele
0.9914 0.9829 0.0962 15.7611 9 2
2
vol, AM1_HOMO, glob, AM1_LUMO, AM1_dipole, logS, AM1_E, ASA_H, logP(o/w), AM1_Eele
0.9914 0.9829 0.0834 23.0864 6 4
3
vol, AM1_HOMO, glob, AM1_LUMO, AM1_dipole, logS, ASA_H, logP(o/w), AM1_Eele
0.9909 0.9820 0.0766 30.3409 5 6
4 vol, AM1_HOMO, glob, AM1_LUMO, AM1_dipole, ASA_H, logP(o/w), AM1_Eele
0.9872 0.9746 0.0831 28.8396 4 7
5 vol, AM1_HOMO, glob, AM1_LUMO,ASA_H, logP(o/w), AM1_Eele
0.9855 0.9713 0.0817 33.9295 4 9
6 vol, AM1_HOMO, glob, ASA_H, logP(o/w), AM1_Eele
0.9803 0.9611 0.0890 33.0132 4 9
Contoh: Pemodelan Persamaan HKSA dengan metode Multi Regresi Linear (SPSS)
Model Bentuk Persamaan
1
Log 1/ IC 50 = 15.339 - 0.214 x AM1_ dipole + 3.911 E-5 x AM1_E + 1.638 E-5 x AM1_Eele - 0.004 x AM1 HF + 0.902 x AM1_Homo + 4.727 x AM1_Lumo - 0.051 x ASA_H + 2.242 x glob - 1.054 x Log P - 0.401 x Log S + 0.100 x vol
2
Log 1/ IC 50 = 13.573- 0.232 x AM1_ dipole + 3.438 E-5 x AM1_E + 1.449 E-5 x AM1_Eele + 0.736 x AM1_Homo + 4.498 x AM1_Lumo - 0.049 x ASA_H + 2.143 x glob - 1.176 x Log P - 0.431 x Log S + 0.098 x vol
3 Log 1/ IC 50 = 16.390 - 0.188 x AM1_ dipole + 9.136 E-6 x AM1_Eele + 0.944 x AM1_Homo + 3.334 x AM1_Lumo - 0.048 x ASA_H + 1.827 x glob - 1.149 x Log P - 0.410 x Log S + 0.091 x vol
4 Log 1/IC 50 = 21.514 - 0.098 x AM1_ dipole + 9.554 E-6 x AM1_Eele + 1.569 x AM1_Homo + 2.886 x AM1_Lumo - 0.052 x ASA_H + 2.171 x glob - 0.786 x Log P + 0.100 x vol
5 Log 1/ IC 50 = 21.674 + 9.765 E-6 x AM1_Eele + 1.576 x AM1_Homo + 2.592 x AM1_Lumo - 0.057 x ASA_H + 1.809 x glob - 0.840 x Log P + 0.108 x vol
6 Log 1/ IC 50 = 19.282 + 1.581 E-5 x AM1_Eele + 1.537 x AM1_Homo - 0.059 x ASA_H + 1.715 x glob - 1.278 x Log P + 0.137 x vol
Contoh: Validasi Persamaan HKSA
• Validasi yang dilakukan pada penelitian ini adalah PRESS (Prediction Residual Sum of Square) dan RMSD (Root Mean Square Deviation).
• Validasi PRESS menggunakan data Training set yang digunakan untuk mengetahui kualitas dan kemampuan memprediksi dari setiap model persamaan.
PRESS = ∑{(Log 1/ MIC eksperimen) – ( Log 1/ MIC prediksi)}²
• Persamaan yang memiliki PRESS ≤ 1 dipilih sebagai
persamaan yang terbaik
Contoh: Data nilai PRESS
Kode Log IC 50 Eksperimen Log IC 50 Prediksi
model 1 model 2 model 3 model 4 model 5 model 6
4l -0.6989 -0.6990 -0.6991 -0.6992 -0.6990 -0.6808 -0.6512
4h 0.2041 0.1922 0.1930 0.1974 0.2112 0.1736 0.0943
4m 0.3617 0.4715 0.4705 0.4592 0.4787 0.4789 0.4762
4t 0.4771 0.5161 0.5165 0.5193 0.4963 0.4825 0.4717
4i 0.4914 0.5180 0.5190 0.5205 0.5823 0.5832 0.6285
4a 0.5185 0.5209 0.5219 0.5499 0.5269 0.5224 0.5446
4e 0.5682 0.5681 0.5684 0.5600 0.5754 0.5717 0.5730
4b 0.5682 0.5511 0.5475 0.5444 0.5186 0.5113 0.5150
4g 0.5798 0.5791 0.5792 0.5825 0.5739 0.5965 0.5949
4o 0.5798 0.5389 0.5409 0.5365 0.5220 0.5050 0.5628
4k 0.5911 0.4888 0.4876 0.4771 0.4965 0.4976 0.5003
4p 0.6232 0.6271 0.6243 0.6375 0.6385 0.6555 0.6486
4c 0.6335 0.6497 0.6513 0.6370 0.5710 0.5862 0.5731
4j 0.6628 0.6370 0.6387 0.6405 0.6662 0.7021 0.6459
4u 0.6721 0.6731 0.6727 0.6700 0.6741 0.6469 0.6550
PRESS 0.0278 0.0278 0.0294 0.0414 0.0468 0.0635
Contoh: Validasi Persamaan HKSA
• Validasi RMSD dilakukan untuk memvalidasi
kemampuan persamaan tersebut dapat digunakan untuk memprediksikan aktifitas.
• RMSD dihitungan dengan cara membandingkan nilai aktivitas eksperimen dengan nilai aktivitas prediksi dan nilai RMSD terbaik ≤1.
RMSD = ∑{(𝑳𝒐𝒈
𝟏𝑴𝑰𝑪 𝒆𝒌𝒔𝒑𝒆𝒓𝒊𝒎𝒆𝒏
) – ( Log 1/ MIC prediksi)}²
Contoh: Data nilai RMSD
Kode Log IC 50 Eksperimen
Log IC 50 Prediksi
model 1 model 2 model 3 model 4 model 5 model 6
4d 0.5682 1.4162 1.3906 1.3415 1.3895 1.4109 1.0441
4n 0.5441 -0.0967 -0.1137 -0.0752 -0.0722 -0.0901 0.0268
4q 0.5911 0.5103 0.5254 0.5562 0.5085 0.5638 0.5699
4r 0.4914 0.5862 0.5887 0.5872 0.5705 0.5595 0.6190
4s 0.5441 -0.5445 -0.5623 -0.5407 -0.4884 -0.3934 -0.2694
RMSD 0.6827 0.6851 0.6586 0.6532 0.6319 0.4843