1
1 PENDAHULUAN
Ikan belida (Chitala lopis) merupakan salah satu jenis ikan air tawar yang memiliki nilai ekonomis tinggi di Indonesia. Persebarannya meliputi daerah Jawa, Riau, Sumatra Selatan, Jambi, Bengkulu, Lampung dan Kalimantan (Kottelat et al. 1993). Ikan belida merupakan ikan konsumsi dan ikan hias karena tampilannya unik. Harga ikan belida konsumsi di pasar berkisar Rp. 50.000 ‐140.000/kg dan ikan belida hias berawal dari Rp. 20.000/ekor, tergantung umur, ukuran dan corak tubuh. Berdasarkan kelebihannya, ikan belida sudah banyak ditangkap baik dari ukuran besar untuk konsumsi, dan ukuran kecil untuk ikan hias. Adanya aktivitas penangkapan (overfishing) dan perubahan lingkungan maupun kerusakan lingkungan perairan, dapat menyebabkan kelestarian ikan ini terancam punah. Ikan belida termasuk dalam kategori ikan air tawar yang dilindungi menurut peraturan pemerintah no. 7 tahun 1999. Pada tahun 2018 ada 4 jenis ikan belida yang dilindungi pemerintah, yaitu belida Borneo (Chitala borneensis), belida Sumatra (Chitala hypselonotus), belida lopis (Chitala lopis), dan belida Jawa (Notopterus notopterus) (Men. LHK 2018). Conservation Assessment and Management Plan (CAMP) mengkategorikan Chitala spp. terutama jenis Chitala lopis sebagai spesies langka (Sarkar et al. 2008).
Dalam menjaga kelestarian ikan belida, perlu adanya upaya kebijakan pengelolaan dan konservasi untuk menjaga populasi ikan belida. Sebagai dasar konservasi, ada 3 prinsip konservasi yang tertuang dalam peraturan pemerintah, yaitu konservasi ekosistem, konservasi jenis, dan konservasi genetik (UU. RI No.31/2004). Upaya untuk menjaga populasi ikan belida sudah dilakukan dengan berbagai kajian, seperti karakter morfologis (Sunarno et al. 2007, Nugroho et al. 2019), keragaman genetik (Wibowo et al. 2010, Wibowo 2012, Wibowo & Marson 2012), biologi reproduksi (Adjie et al. 1999, Santoso 2009, Sunarno & Syamsunarno 2015), ekobiologi (Wibowo & Subagja 2014). Dari beberapa penelitian tersebut, penelitian ini dapat membantu menjaga ekosistem dan kelestarian ikan belida dengan informasi ilmiah tentang kerakteristik dan keragaman populasi ikan belida berdasarkan DNA mitokondria di wilayah Sumatra dan Kalimantan.
Analisis gen mtDNA dapat memberikan informasi dalam melakukan identifikasi spesies, mengklarifikasi taksonomi dan menentukan sebaran populasi hewan yang diamati (Hebert et al. 2003). Penggunaan gen mtDNA sering digunakan untuk memperoleh informasi keragaman genetik karena berevolusi sangat cepat dan dapat digunakan untuk membedakan interspesies dan intraspesies hewan yang berkerabat dekat (Solihin 1994).
Beberapa tahun terakhir penggunaan gen mitokondria yang paling populer adalah gen Cytochrome Oxidase subunit I (COI) dan Cytochrome b (Cyt b) untuk menganalisis taksa dari tingkat species hingga tingkat family (Kartavtsev & Lee 2006, Page & Hughes 2010). Gen COI merupakan salah satu marka molekular yang terdapat di DNA mitokondria. Gen ini banyak digunakan sebagai DNA barcoding, identifikasi spesies, karakterisasi dan pengelompokan spesies dari berbagai taksa hewan (Solihin 1994, Hebert et al. 2003). Penggunaan gen CO1 sebagai DNA Barcoding, mampu untuk mengidentifikasi ikan pada berbagai stadia ikan, maupun
2
status ikan cryptic species (Ward et al. 2005, Bhattacharya et al. 2016, Linh et al. 2018).
Informasi keragaman genetik dapat diperoleh melalui analisis marka DNA mitokondria (mtDNA). Gen COI mampu melihat keragaman genetik dan verifikasi spesies ikan belida adalah Chitala lopis ;Bleeker 1851, maka penggunaan gen ini sangat penting untuk barcoding species. Pada beberapa kasus, penggunaan gen COI mampu membedakan asal-usul spesies tersebut (Artamonova et al. 2018). Namun dari beberapa literatur yang ada, pemilihan gen untuk melihat sebaran populasi menggunakan marker yang paling efektif yaitu gen Cyt b (Peng et al. 2004, Habib et al. 2011). gen ini mampu melihat perbedaan pada intrapopulasi dengan sebaran geografik yang berbeda. Gen Cyt b bersifat conserve/lestari dalam spesiesnya dan juga harus menunjukkan kemungkinan pembeda/variabel bagi antar populasinya (Gordeeva & Volkov 2016). Penggunaan gen ini banyak digunakan pada spesies ikan, terutama untuk merekonstruksi pohon filogeni, species origin, maupun sebaran populasi (Page & Hughes 2010, Kartavtsev 2011, Esa et al. 2012). Dalam studi ini, penggunaan gen COI pada ikan Belida diharapkan mampu mengkarakteristik genetik di wilayah Sumatra dan Kalimantan.
Tujuan Penelitian
Penelitian ini bertujuan untuk mengkaji karakteristik genetik ikan Belida (Chitala lopis) dari sebaran pulau Sumatra dan Kalimantan berdasarkan gen COI DNA Mitokondria.
Manfaat Penelitian
Penelitian ini diharapkan dapat memberikan informasi dasar mengenai karakteristik genetik ikan Belida di wilayah Sumatra dan Kalimantan berdasarkan gen COI DNA Mitokondria untuk upaya konservasi berkelanjutan.
2 TINJAUAN PUSTAKA
Ikan Belida
Di Indonesia ikan belida dikenal dengan nama belido (Sumatra) atau pipih (Kalimantan). Salah satu genus dari famili ini adalah genus Chitala yang terdiri atas 6 spesies yaitu C. blanci (Indochina Featherback) C. borneensis (Indonesian Featherback) C. chitala (Clown Knifefish) C. Hypselonotus, C. lopis (Giant Featherback) dan C. ornata (Clown Featherback) (Kottelat et al. 1993). Nama ilmiah Chitala telah mengalami perbaikan dan nama yang sah atau valid adalah: Chitala lopis. Ikan Belida memiliki sinonim Notopterus chitala dengan nama
internasional giant featherback (Fishbase.org 2017).
Klasifikasi ikan belida (Chitala lopis, Bleeker 1851) adalah sebagai berikut (Fishbase.org, 2017):
3 Kingdom : Animalia Phylum : Chordata Class : Actinopterygii Order : Osteoglossiformes Family : Notopteridae Genus : Chitala Species : C. Lopis
Sinonim : Notopterus chitala
Nama lokal : Pangaju (Jawa), Lopis (Jawa Barat), Belidah/Blidah (Kalimantan Barat), Pipih (Kalimantan Selatan) (Schuster & Djajadiredja, 1952)
Nama Umum : Giant featherback
Gambar 1 Ikan Belida (Chitala lopis)
Ikan belida (Chitala lopis) merupakan spesies ikan air tawar yang menghuni perairan umum di Indonesia. Ikan belida tergolong ikan purba dengan bentuk tubuh yang unik. Ikan ini merupakan ikan yang bersifat predator dan nokturnal, pada siang hari mereka bersembunyi di antara vegetasi (Kottelat et al. 1993).
Ciri morfologis ikan belida, berdasarkan Weber & deBeaufort (1913); Kottelat et al. (1993), memiliki badan pipih dan memanjang dengan bagian punggung yang tampak membesar. Bentuk kepala dekat punggung cekung, rahang semakin panjang sesuai dengan meningkatnya umur sampai jauh melampaui batas belakang mata pada spesimen yang besar. Bagian perut berduri ganda dengan bagian ekor yang juga memanjang. Ukuran sisik kecil, berbentuk sikloid, pada samping badan membentuk gurat sisi. Bukaan mulut lebar, dibatasi rahang atas depan dan rahang atas. Sirip punggung kecil, terletak kira-kira direntang pertengahan sirip dubur yang bersatu dengan sirip ekor. Sirip perut yang bersatu pada dasarnya kecil (rudiment). Selaput insang bersatu pada bagian dasarnya dan bebas dari isthmus dengan jari-jari selaput insang berjumlah 7-9. Saringan insang tidak banyak, kuat, ada serangkaian tonjolan pada bagian dalam lengkung insang yang pertama.
Daur hidup ikan belida hampir secara keseluruhan hidup di perairan air tawar (Adjie et al. 1999), sebagian lagi populasi ikan belida dapat hidup di perairan rawa banjiran yang memiliki kualitas air yang kurang baik, khususnya kadar oksigen terlarut rendah, oleh karena itu ikan belida memiliki alat bantu pernapasan berupa labirin (Wibowo & Sunarno 2006).
Pola migrasi lokal ikan belida yaitu pada saat debit air kecil di musim kemarau sebagian besar ikan belida cenderung tinggal di perairan sungai dan sebagian lagi di tempat-tempat terdalam yang tergenang air, sedangkan pada saat air melimpah di musim hujan mereka menyebar ke rawa banjiran dan persawahan
4
baik untuk memijah maupun untuk mencari makan. Ikan belida memijah dilokasi yang banyak terdapat kayu, perakaran yang terendam air. Induk ikan belida menempelkan telurnya pada substrat (akar akar kayu) yang terendam dalam air sebagai tempat pemijahan. Induk ikan belida menempelkan telur-telurnya pada benda-benda yang berada 1,5-2 m di bawah permukaan air (Adjie & Utomo 1994).
DNA Mitokondria
Mitokondria merupakan organel sel penghasil energi yang terdapat di dalam sitoplasma. DNA mitokondria (mtDNA) merupakan DNA utas ganda yang berbentuk sirkuler. Panjang basa dari mtDNA hewan berkisar antara 14.000 – 39.000 yang terdiri dari 13 gen yang menyandi protein, 2 gen ribosomal RNA (12S dan 16S), 22 gen transfer RNA (tRNA), dan sebuah daerah yang tidak menyandi informasi genetik, bagian ini dikenal sebagai control region (displacement-loop/d-loop) (Solihin 1994). Ketiga belas gen penyandi protein meliputi tujuh subunit (ND1, ND2, ND3, ND4, ND4L, ND5 dan ND6) dari kompleks I rantai respirasi, satu subunit (Cytochrome B) dari kompleks III, tiga subunit (Cytochrome C oxidase subunit I,
Cytochrome C oxidase subunit II, Cytochrome C oxidase subunit III) dari kompleks IV
dan dua subunit (ATP ase 6 dan ATP ase 8) dari kompleks V. 22 gen tRNA terdiri dari prolin (tRNAPro), fenil alanin (tRNAPhe), valin (tRNAVal), leusin (tRNALeu),
isoleusin (tRNAIle), asparagin (tRNAAsn), glutamin (tRNAGln), metionin (tRNAMet),
triptofan (tRNATrp), alanin (tRNAAla), sistein (tRNACys), tirosin (tRNATyr), serin
(tRNASer), asam aspartat (tRNAAsp), lisin (tRNALys), glisin (tRNAGly), arginin
(tRNAArg), histidin (tRNAHis), serin (tRNASer), leusin (tRNALeu) asam glutamat
(tRNAGlu), dan treonin (tRNAThr) (Pereira 2000; Broughton et al. 2001).
5 DNA mitokondria (mtDNA) banyak digunakan untuk mengidentifikasi keragaman genetik dan dinamika populasi karena mempunyai beberapa kelebihan. DNA mitokondria terdapat dalam jumlah kopi yang tinggi. Jumlah kopi yang tinggi ini menjadikannya mudah diisolasi dan dipurifikasi untuk berbagai keperluan analisis genom. Memiliki ukuran yang kompak dan relatif kecil (14.000-39.000 pasang basa) sehingga dapat dipelajari sebagai satu kesatuan yang utuh meskipun tidak sekompleks DNA inti. DNA mitokondria hanya diturunkan dari induk betina atau maternal saja tanpa ada genom dari jantan karena hanya sel telur yang menyumbangkan material mitokondria. Genom mtDNA berevolusi dengan kecepatan berbeda-beda (Solihin 1994).
Tingkat evolusi dari suatu gen atau bagian DNA yang berbeda merupakan faktor penting yang menentukan penggunaan penanda DNA dalam studi sistematika dan biogeografi. Umumnya, gen-gen yang terkonservasi dengan baik (berevolusi lambat) dapat dijadikan dasar penelusuran asal-usul atau filogeni. Sebaliknya, gen-gen yang tidak terkonservasi dengan baik (berevolusi cepat) dapat digunakan untuk perbandingan galur-galur baru (Solihin 1994).
Gen Cytochrome Oxidase Subunit I (COI) dan Cytochrome b (Cyt b)
DNA mitokondria memiliki banyak gen diantaranya gen Cytochrome Oxidase subunit I (COI) dan Cytochrome b (Cyt b). Dalam studi molekuler, gen COI digunakan sebagai penanda genetik untuk mempelajari karakteristik genetik antar spesies. Gen COI merupakan salah satu gen yang digunakan dalam ”DNA Barcode” pada organisme hewan (Hebert et al. 2003). Gen COI merupakan salah satu dari 13 gen yang mengkode protein di dalam DNA mitokondria dan termasuk kelompok kompleks IV. Menurut Hebert et al. (2003), penggunaan gen ini merupakan suatu standar sistem bioidentifikasi global untuk hewan, karena sekuens gen tunggal dalam DNA mitokondria dapat membedakan sebagian besar spesies hewan. Kelebihan dari DNA Barcoding ini dapat mengidentifikasi berbagai stadia ikan, dari telur hingga ikan dewasa. Material DNA yang digunakan bisa berasal dari ikan utuh, potongan daging, sirip, sisik, juvenile, larva atau telur. Potongan daging atau sirip sering digunakan analisis molekuler karena ciri-ciri morfologi yang digunakan untuk identifikasi spesies umumnya tidak ada. Identifikasi telur dan larva juga sulit karena karakteristik yang membedakan seringkali tidak ada atau tidak terdeskripsikan (Ward et al. 2009). Bahkan studi yang telah dilakukan Smith et al. (2008) dapat mengidentifikasi menggunakan produk olahan ikan. Banyak studi yang telah dilakukan menggunakan gen ini pada ikan untuk mengidentifikasi spesies (Ward et al. 2005, Fields et al. 2015, Bhattacharya et al. 2016, Bingpeng et al. 2018, Linh et al. 2018).
Salah satu penanda DNA mitokondria yang sering digunakan dalam mengamati variasi genetik antarpopulasi adalah gen Cytochrome b (Cyt b). Gen ini terletak diantara 2 gen tRNA, yaitu tRNAGlu dan tRNAThr dengan Panjang 1141 pb.
Gen Cyt b dapat digunakan sebagai alat yang efektif untuk identifikasi spesies, rekonstruksi pohon filogeni interspesies ataupun intraspesies, filogeografi dan studi genetika populasi ikan (Johnson & Jordan 2000, Peng et al. 2004, Goordeva & Volkov 2016). Pada beberapa studi, penggunaan gen ini mampu menentukan distribusi dan pola variasi genetik pada Channa marulius (Habib et al. 2011),
6
variasi geografi pada Oryzias latipes (Takehana et al. 2003), merekonstruksi pohon filogeni dari taksa cyprinid (Esa et al. 2012), dan variasi genetik pada populasi Chitala chitala (Mandal et al. 2012).
3 METODE
Waktu dan Tempat Penelitian
Penelitian ini dilaksanakan pada 2018 – 2019. Koleksi sampel ikan belida berasal dari Sumatra dan Kalimantan yang terbagi menjadi 4 lokasi (Tabel 1), Riau (1 sampel), Lampung (3 sampel), Kalimantan Barat (3 sampel), dan Kalimantan Selatan (3 sampel). Sampel Riau diperoleh dari Balai Riset Perikanan dan Perairan Umum Palembang (BRPPU Palembang). Sampel Kalimantan Selatan diperoleh dari Instalasi Penelitian dan Pengembangan Teknologi Lingkungan dan Toksikologi Perikanan Budidaya Air Tawar Cibalagung, Bogor. Analisis genetik dilaksanakan di Laboratorium Konservasi Genetik, Pusat Penelitian Sumberdaya Hayati dan Bioteknologi (PPSHB) IPB Bogor.
Tabel 1 Sumber sampel ikan belida yang dianalisis gen COI
Preservasi Sampel
Sampel yang digunakan berasal dari darah dan sirip. Sampel darah diambil sebanyak ± 0.5 ml menggunakan spuit dengan Teknik Puncturing the Caudal Vessel (Pembuluh darah Bagian Caudal) (MUAWC 2008). Darah yang diperoleh lalu dimasukkan ke dalam tube eppendorf 1.5 ml yang berisi buffer EDTA 10% dengan perbandingan ± darah 7:3 EDTA dan diberi label.
Sampel sirip diambil dari sirip dada (pectoral fin). Sirip diambil dalam potongan kecil (±2cm) menggunakan gunting diseksi lalu dimasukkan ke dalam tube eppendorf 1.5 ml yang berisi buffer DET hingga sampel sirip terendam seutuhnya dan diberi label. Sampel yang diperoleh dari koleksi BRPPU Palembang
No. Kode Sampel Lokasi Sampel Bentuk Sampel Sumber 1 BKS 1 Kalimantan Selatan Darah / Sirip Koleksi Balai Cibalagung 2 BKS 2 Kalimantan Selatan Darah / Sirip
3 BKS 3 Kalimantan Selatan Darah / Sirip 4 BKB 1 Kalimantan Barat Darah / Sirip
Pedagang 5 BKB 2 Kalimantan Barat Darah / Sirip
6 BKB 3 Kalimantan Barat Darah / Sirip
7 BL 1 Lampung Darah / Sirip Penduduk
Lokal
8 BL 2 Lampung Darah / Sirip
9 BL 3 Lampung Darah / Sirip
7 menggunakan pengawet ethanol absolut. Sampel darah dan sirip selanjutnya dibawa ke Laboratorium Konservasi Genetik, lalu disimpan dalam freezer.
Isolasi DNA Total
Ekstraksi DNA total dari sampel darah dan sirip yang menggunakan pengawet ethanol absolut harus dicuci terlebih dahulu dengan aquades atau buffer LOW TE sebanyak 3-6 kali yang bertujuan untuk membebaskan ethanol dari sampel, selanjutnya dapat langsung menggunakan metode ektraksi dari Dneasy® Blood dan Tissue Kit (Qiagen). Ekstraksi DNA total dari sampel darah dan sirip yang menggunakan buffer EDTA dan DET dapat diproses langsung menggunakan Dneasy® Blood dan Tissue Kit (Qiagen 2003) dengan beberapa modifikasi untuk meningkatkan hasil dan kualitas DNA yang diektraksi.
Amplifikasi dan Sekuensing Produk PCR
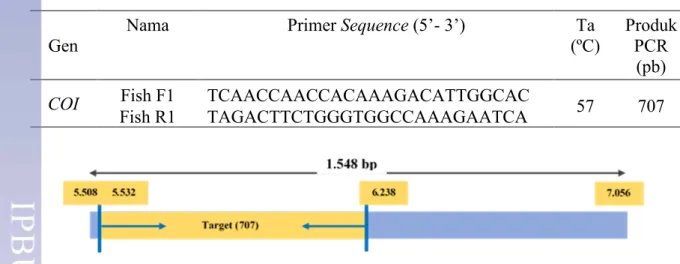
DNA total hasil purifikasi digunakan sebagai DNA template untuk proses amplifikasi dengan teknik Polymerase Chain Reaction (PCR). Primer yang digunakan pada gen COI adalah COI Fish F1 dan COI Fish R1 (Ward et al. 2005) pada Tabel 2. Primer tersebut diperoleh dari koleksi Dr Ir Dedy Duryadi Solihin, DEA. Target dari primer gen COI menghasilkan produk PCR sepanjang 707 pb. Total volume pereaksi PCR yang diperoleh adalah 25 µl, dengan komposisi DDH2O: 10,8 µl, 5x Buffer Q5: 4 µl, 5x Enhancer Q5: 5 µl, dNTP: 1 µl, Primer COI Fish F1: 1 µl, Primer COI Fish R1: 1 µl, DNA total: 2 µl, dan Taq DNA Polymerase: 0,2 µl untuk gen COI. PCR dilakukan menggunakan mesin PCR Sensoquest Labcycler 48.
Kondisi PCR yang digunakan untuk gen CO1 meliputi tahapan pradenaturasi 94 ºC selama 3 menit, denaturasi 94 ºC selama 45 detik, annealing 57 ºC selama 45 detik, elongasi 72 ºC selama 1 menit sebanyak 35 siklus, elongasi akhir (final extension) pada suhu 72 °C selama 5 menit, dan pendinginan 20 ºC selama 10 menit. Tabel 2 Runutan Primer Gen COI
Gambar 3 Skema posisi penempelan Primer Gen COI
Gen Nama Primer Sequence (5’- 3’) (ºC) Ta Produk PCR (pb) COI Fish R1 Fish F1 TCAACCAACCACAAAGACATTGGCAC TAGACTTCTGGGTGGCCAAAGAATCA 57 707
8
Visualisasi DNA Total dan Produk PCR
Hasil dari isoslasi DNA total dan produk PCR diimigrasikan pada gel agarose 1.2% (gel agarosa 0.6 mg, TBE 1x 50 ml, dan EtBr 2.5 μl) dalam larutan 1xTBE (Tris-borate-EDTA) menggunakan alat Submarine Electrophoresis Hoefer, USA. Elektroforesis dijalankan selama 60 menit dengan daya sebesar 85 volt. Setelah dielektroforesis, gel agarose divisualisasi menggunakan alat gel uvitec Cambridge.
DNA Sequencing
Produk PCR yang didapatkan kemudian disekuen untuk melihat runutan susunan basa-basa nukleotida. Sekuensing dilakukan menggunakan metode Sanger Sequencing di perusahaan 1st Base Malaysia melalui perusahaan jasa pelayanan sekuensing di Indonesia yaitu PT. Genetika Science, Jakarta.
Analisis Data
Hasil sekuensing diperiksa dahulu untuk memastikan tidak adanya error sekuensing. Hasil dari tiap sekuens, disejajarkan berdasarkan sekuens forward dan reverse untuk mendapatkan sekuen utuh. Sekuen utuh tersebut kemudian dikoreksi dengan primer forward dan reverse dari gen COI agar diperoleh sekuen sesuai target dari primer yang digunakan. Pensejajaran sekuen menggunakan program MEGA (Molecular Evolutionary Genetics Analysis) versi 7.0 (Kumar et al. 2016). Hasil koreksian tersebut kemudian dilakukan BLAST-n (Basic Local Alignment Search Tool-nucleotide) di situs National Center for Biotechnology Information (NCBI). Jarak genetik dianalisis berdasarkan model Kimura 2-parameter dengan nilai boostrap 1000 kali. Rekonstruksi pohon filogenetik menggunakan metode Neighbor-joining dengan model Kimura 2- parameter, serta bootstraps sebanyak 1000 kali pengulangan (Kumar et al. 2016). Selanjutnya sekuen dari GenBank Chitala lopis (AP008922.1 dan KM213054.1) ditambahkan sebagai ingroup dan Notopterus notopterus (AP008924.1) sebagai outgroup.
4 HASIL
Pita elektroforesis dan ukuran gen COI parsial DNA Mitokondria
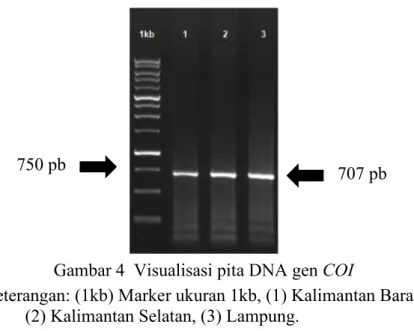
Hasil produk PCR di elektroforesis dan diperoleh pita elektroforesis yang teramplifikasi berukuran 707 pb untuk semua sampel (Gambar 4). Jumlah sampel yang digunakan untuk gen COI berjumlah 10 sampel, yang terdiri dari 3 sampel Lampung, 3 sampel Kalimantan Selatan, 3 sampel Kalimantan Barat, dan 1 sampel Riau. Berdasarkan runutan gen COI utuh Chitala lopis berukuran 1.548 pb, posisi gen COI parsial ikan Belida menggunakan primer COI Fish F1 teramplifikasi mulai pada basa ke 24 dan primer COI Fish R1 hingga pada basa 731 sehingga ukurannya sepanjang 707 pb (Gambar 3).
9
Identifikasi spesies berdasarkan gen COI menggunakan BLAST-n
Identifikasi spesies ikan belida dilakukan dengan mencocokkan sekuen gen COI dengan ikan sejenis yang terdapat pada GenBank. Hasil yang diperoleh dari 10 sampel ikan belida menunjukan kemiripan (Percent Identity) dengan spesies ikan belida Chitala lopis dengan kode akses AP008922.1. Nilai yang diperoleh berkisar dari 97.60 - 99.15% dengan query cover sebesar 100% pada semua sampel (Tabel 3). Hasil terendah diperoleh pada sampel yang berasal dari Kalimantan Selatan dengan nilai 97.60%. Tingkat kemiripan tertinggi didapat pada sampel yang berasal dari Sumatra (Lampung dan Riau) dengan nilai 99.15%.
Tabel 3 Hasil analisis BLAST-n menggunakan gen COI ikan belida
Keterangan: BKS: Belida Kalimantan Selatan; BKB: Belida Kalimantan Barat; BL: Belida Lampung; BR: Belida Riau.
No Sampel Terdekat Spesies Query Cover (%) Percent Identity (%) Kode akses
1 BKS1 Chitala lopis 100 97.60 AP008922.1
2 BKS 2 Chitala lopis 100 97.60 AP008922.1
3 BKS 3 Chitala lopis 100 97.60 AP008922.1
4 BKB 1 Chitala lopis 100 98.59 AP008922.1
5 BKB 2 Chitala lopis 100 98.59 AP008922.1
6 BKB 3 Chitala lopis 100 98.59 AP008922.1
7 BL 1 Chitala lopis 100 99.15 AP008922.1
8 BL 2 Chitala lopis 100 99.15 AP008922.1
9 BL 3 Chitala lopis 100 99.15 AP008922.1
10 BR 1 Chitala lopis 100 99.15 AP008922.1
Gambar 4 Visualisasi pita DNA gen COI
Keterangan: (1kb) Marker ukuran 1kb, (1) Kalimantan Barat, (2) Kalimantan Selatan, (3) Lampung.
707 pb 750 pb
10
Karakteristik genetik molekuler
Karakterisasi gen COI pada spesies Chitala lopis berdasarkan analisis MEGA 7.0 dengan metode multiple alignment dengan perbandingan sekuens Chitala lopis (AP008922.1) dari Genbank menunjukan hasil situs konservatif sebesar 687 pb (97.17%), situs variasi 20 pb (2.83%), situs parsimoni 17 pb (2.40%), dan singleton 2 pb (0.28%) (Tabel 4). Komposisi basa nukleotida gen COI menunjukan bahwa rata-rata tertinggi adalah Adenosin (28.5 %) dan diikuti oleh basa Sitosin (27.5%), Timin (27.2%) dan paling rendah adalah Guanin (16.8%) (Tabel 4). Pada penelitian ini diperoleh komposisi basa AT sebesar 55.7% dan komposisi basa GC 44.3%. Pada penelitian ini diperoleh hasil nilai analisis statistik frekuensi nukelotida subtitusi adalah ii = 700, si = 6, sv = 2, R = 3.4.
Tabel 4 Perbandingan karakteristik nukleotida dengan gen COI Chitala lopis (AP008922.1)
*Situs parsimoni: ditemukan minimal dua jenis nukleotida, setiap jenis nukleotida dimiliki oleh minimal 2 runutan, **Situs singleton: nukleotida yang berbeda hanya ditemukan pada satu runutan.
Polimorfisme interpopulasi Chitala lopis
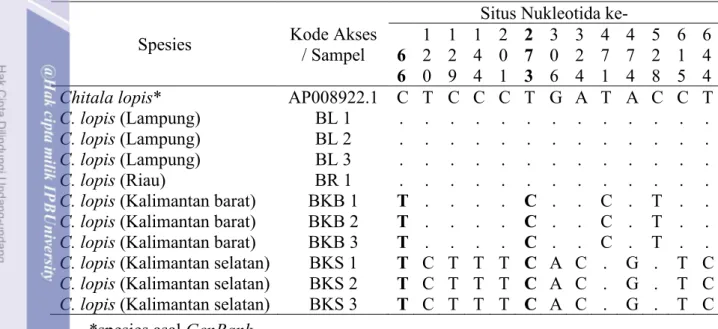
Pada hasil penelitian ini ditemukan 13 situs polimorfisme bervariasi pada populasi Sumatra dan Kalimantan. Perbedaan pada sampel Kalimantan dengan Sumatra terletak pada nukleotida spesifik geografik situs ke 66 dan 273. Nukleotida spesifik pada sampel Kalimantan dapat membedakan antar lokasi. Pada sampel Kalimantan Selatan menunjukan nukleotida spesifik terdapat pada situs 120, 129, 144, 201, 306, 324, 474, 615 dan 644. Pada sampel Kalimantan Barat situs nukleotida spesifik ditunjukan pada situs 471 dan 528. Berbeda dengan Kalimantan, pada sampel Sumatra cenderung menunjukan nukleotida yang sama dengan sampel pembanding dari GenBank (Chitala lopis AP008922.1). Berdasarkan hasil yang diperoleh, variasi situs nukleotida pada populasi Kalimantan lebih tinggi dibandingkan populasi Sumatra. Perbedaan situs nukleotida antar sampel, hal ini dapat dilihat pada Tabel 5.
No Karakter Gen COI
1 Individu Sampel 1 Genbank dan 10 sampel
2 Situs Konservatif (%) 687 (97.17)
3 Situs Variasi (%) 20 pb (2.83)
4 Situs Parsimoni* (%) 17 pb (2.40)
5 Situs Singleton** (%) 2 pb (0.28)
6 Persentase Adenin (A) 28.5
7 Persentase Guanin (G) 16.8
8 Persentase Sitosin (C) 27.5
9 Persentase Timin (T) 27.2
10 Komposisi basa AT 55.7
11 Tabel 5 Polimorfisme sekuen nukleotida Chitala lopis berdasarkan gen COI
*spesies asal GenBank.
Jarak genetik intra dan inter populasi
Berdasarkan hasil yang diperoleh, Perbedaan jarak genetik interpopulasi antara Kalimantan Selatan dan Kalimantan Barat dengan nilai 1.63%. Pada perbedaan jarak genetik interpopulasi Sumatra dan Kalimantan didapat nilai sebesar 0.006 - 0.016 dengan rata-rata 0.011 (1.11%). Persentase perbedaan jarak genetik intraspesies pada sampel ini dengan Chitala lopis dari GenBank (AP008922.1) sebesar 0.70% - 2.30% dengan rata-rata 1.36%. Andaikata seluruh sampel dibandingkan dengan Chitala lopis dari GenBank (KM213054.1) maka terdapat perbedaan sebesar 0.90% - 2.50 dengan rata-rata 1.53%. Jarak genetik interspesies dengan outgroup Notopterus notopterus (AP008924.1) 12.50 - 13.19% (Tabel 6) dengan rata-rata 12.97%.
Spesies Kode Akses / Sampel
Situs Nukleotida ke-
1 1 1 2 2 3 3 4 4 5 6 6 6 2 2 4 0 7 0 2 7 7 2 1 4 6 0 9 4 1 3 6 4 1 4 8 5 4
Chitala lopis* AP008922.1 C T C C C T G A T A C C T C. lopis (Lampung) BL 1 . . . . C. lopis (Lampung) C. lopis (Lampung) C. lopis (Riau) BL 2 BL 3 BR 1 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . C. lopis (Kalimantan barat) BKB 1 T . . . . C . . C . T . .
C. lopis (Kalimantan barat) BKB 2 T . . . . C . . C . T . .
C. lopis (Kalimantan barat) BKB 3 T . . . . C . . C . T . .
C. lopis (Kalimantan selatan) BKS 1 T C T T T C A C . G . T C
C. lopis (Kalimantan selatan) BKS 2 T C T T T C A C . G . T C
C. lopis (Kalimantan selatan) BKS 3 T C T T T C A C . G . T C
Tabel 6 Rata-rata jarak genetik intra dan inter spesies berdasarkan gen COI (%)
Lokasi [1] [2] [3] [4] [5] [6] [7] [1] BL 10.00* [2] BR 10.00 10.00* [3] BKS 11.63 11.63 10.00* [4] BKB 10.60 10.60 11.63 10.00* [5] C. lopis1 10.70 10.70 12.30 11.30 10.00* [6] C. lopis2 10.90 10.90 12.50 11.40 10.40 10.00* [7]N. notopterus3 13.20 13.20 13.00 12.80 12.50 12.70 10.00* * jarak genetik antar individu dalam populasi (intrapopulasi). 1 Spesies Asal GenBank (AP008922.1). 2 Spesies Asal GenBank (KM213054.1). 3 Spesies Asal GenBank (AP008924.1). Keterangan: BL: Belida Lampung ; BR: Belida Riau ; BKS: Belida Kalimantan Selatan ; BKB: Belida Kalimantan Barat.
12
Hubungan kekerabatan
Rekonstruksi pohon filogenetik pada intraspesies Chitala lopis dan ditambah 3 data pembanding dari Genbank (2 ingroup) yaitu Chitala lopis (AP008922.1 dan KM213054.1) dan dari data outgroup yaitu Notopterus notopterus (AP008924.1) dilakukan berdasarkan model Kimura 2-parameter dengan metode neighbour joining dan menggunakan nilai bootstrap 1000 kali pengulangan. Berdasarkan hasil analisis pohon filogenetik gen COI menunjukan adanya dua group utama, terdiri dari group I yang berisi sampel pada penelitian ini dan dua data spesies pembanding (ingroup) dan group II berisi outgroup Notopterus notopterus (AP008924.1) (Gambar 5). Pada group I terbagi lagi menjadi dua group, yaitu group Ia dengan nilai bootstrap 54 dan group Ib yang terdiri dari sampel asal Kalimantan selatan dengan nilai bootstrap 100. Pada group Ia terbagi menjadi beberapa sub-group, yaitu sub-group Ia11 (Lampung dan Riau), sub-group Ia12 (ingroup) dan sub-group Ia2 (Kalimantan Barat).
Pembahasan
Penyejajaran pada 10 sampel ikan Belida dan spesies pembanding, diperoleh runutan nukleotida sepanjang 707 pb pada gen parsial COI spesies Chitala lopis. Pada runutan gen parsial COI terdapat 2.83% situs bervariasi dan 97.17% situs konservatif. Dari hasil tersebut menunjukan bahwa tidak terdapat nukleotida yang mengalami insersi dan delesi, namun ditemukannya nukleotida yang mengalami subtitusi. Pada gen COI sangat jarang terjadinya insersi, delesi dan variasi, dikarenakan gen ini bersifat konservatif dan digunakan sebagai standar DNA Barkoding (Hebert et al. 2003).
Persentase komposisi basa nukleotida GC pada ikan belida adalah 44.3%. Hal ini tidak berbeda jauh pada beberapa takson, komposisi basa GC berada dibawah 50%. Seperti pada spesies C. chitala 43.5% & N. notopterus 43.85% (Sengupta & Gambar 5 Rekonstruksi pohon filogenetik Chitala lopis berdasarkan metode bootstrap neighboor-Joining (NJ) 1000 kali pengulangan dengan model Kimura 2- parameter berdasarkan marka gen COI.
G roup I a G roup I b Sub-Group Ia11 Sub-Group Ia12 Sub-Group Ia2 Sub-Group Ib I II
13 Homechaudhuri 2013), genus Kryptopterus 46.40% (Jusmaldi et al. 2014), kelompok Teleostei 47.06% (Ward et al. 2005), kelompok Elasmobranchs 41.78% & kelompok Actinopterygii 47.12% (Ward & Holmes 2007).
Karakteristik nukleotida pada gen COI ikan belida, terdapat nukleotida yang mengalami subtitusi transisi dan transversi. Dilihat dari subtitusi nukleotidanya, frekuensi perubahan yang paling sering terjadi pada posisi kodon ketiga (94.4%) dibandingkan dengan posisi kodon pertama ataupun kodon kedua (5.6%). Menurut Bofkin & Goldman (2007) Subtitusi nukleotida yang terjadi pada posisi kodon ketiga banyak yang bersifat sinonim, yaitu subtitusi yang tidak menyebabkan perubahan aktivitas pada produk yang dikode oleh gen. Sebaliknya dengan subtitusi pada kodon ketiga, subtitusi pada kodon pertama dan kodon kedua bersifat non-sinonim. Hasil ini menunjukan bahwa frekuensi subtitusi sinonim pada gen COI ikan belida lebih banyak yang bersifat sinonim, hal ini didukung juga oleh penelitian Ward & Holmes (2007) bahwa subtitusi sinonim lebih tinggi dibanding subtitusi non-sinonim pada gen parsial COI dengan panjang 655 pb.
Berdasarkan hasil yang diperoleh, subtitusi nukleotida yang terjadi pada gen parsial COI lebih banyak terjadi subtitusi transisi dibandingkan transversi. Jumlah basa nukleotida yang mengalami subtitusi transisi dengan nilai si = 6, sv = 2, R = 3.4 Hasil ini didukung penelitian Sengupta & Homechaudhuri (2013), yaitu hasil subtitusi nukleotida transisi lebih banyak terjadi dibanding subtitusi nukleotida transversi. Hasil tersebut terjadi pada famili Notopteridae, dimana subtitusi transisi yang diperoleh yaitu, si = 28, dan sv = 11 dengan nilai R = 2.6.
Hasil penyejajaran runutan gen COI parsial (707 pb), ditemukan adanya situs nukleotida spesifik lokasi berjumlah 13 situs sebagai pembeda pada interpopulasi. Kalimantan Selatan memiliki 9 situs nukleotida spesifik lokasi dan Kalimantan Barat memiliki 2 situs nukleotida spesifik lokasi. Sumatra (Lampung dan Riau) cenderung memiliki kesamaan nukleotida dengan spesies pembanding. Diduga spesies pembanding yang berada di GenBank diperoleh dari Sumatra (Indonesia). Wibowo (2014) melaporkan berdasarkan gen COI bahwa C. lopis asal Indonesia memiliki 14 situs nukleotida spesifik lokasi, yang dimana tiap populasi memiliki ciri situs nukleotida spesifik lokasi tersendiri.
Persentase rata-rata jarak genetik pada interpopulasi asal Kalimantan dan Sumatra sebesar 1.12%. Jarak genetik rata-rata intraspesies dengan Chitala lopis (AP008922.1) sebesar 1.36% dan Chitala lopis (KM213054.1) sebesar 1.53%, yang berarti sampel pada penelitian ini dengan ingroup masih dalam satu spesies yang sama. Pada sistem identifikasi COI, suatu spesies dapat dibedakan jenisnya apabila perbedaan jarak genetiknya melebihi ambang batas yang sudah ditentukan, yaitu 3% (Hebert et al. 2003), dan 3.5% pada ikan (Ward et al. 2009). Perbedaan ditunjukan pada spesies pembanding N. notopterus, dengan jarak sebesar 12.83% - 13.19% pada sampel asal Sumatra dan Kalimantan, dan 12.50% pada spesies Chitala lopis (AP008922.1) sebagai ingroup. Pada hasil penelitian Sengupta & Homechaudhuri (2013), spesies C. chitala dan N. notopterus memliki jarak genetik intraspesies 0% dan interspesies 11%.
Berdasarkan hasil jarak genetik populasi belida Sumatra dan belida Kalimantan Barat memiliki perbedaan jarak genetik yang dekat (0.60%). Hal ini disebabkan karena pada zaman Pleistocene, pulau Sumatra, Kalimantan dan Asia merupakan suatu daratan besar yang disebut paparan Sunda. Aliran sungai besar Sunda Utara mengalir dari sungai Indragiri, Musi (utara – timur Sumatra) hingga
14
menuju sungai Kapuas (Kalimantan Barat) (Voris 2000) sehingga memiliki pengaruh pada penyebaran ikan air tawar (Inger & Chin, 1962). Hal ini dapat dilihat dari populasi ikan Botia (Chromobotia macracanthus), bahwa populasi ikan Botia di Kalimantan dan Sumatra memiliki keragaman genetik dan morfologi yang mirip (Sudarto & Rizal 2007; Sudarto et al. 2008). Begitu pula dengan sampel ikan Belida dari populasi Riau dan Lampung, menunjukan jarak genetik 0%, dikarenakan Sungai Kampar terhubung dengan sistem aliran sungai Sunda Utara (Voris 2000). Perbedaan jarak genetik ditunjukan antara populasi Kalimantan Selatan dengan Kalimantan Barat sebesar 1.63%. Hal ini diduga bahwa aliran sungai di Kalimantan Selatan tidak terhubung dengan sungai Sunda Utara. Hasil ini diperkuat dari hasil penelitian Sudarto & Rizal (2007), bahwa morfologi ikan Botia dari Kalimantan Selatan berbeda dengan ikan Botia yang berasal dari Sumatra.
Berdasarkan hasil rekonstruksi pohon filogeni ikan belida, terdapat 2 group utama. Masing-masing populasi mengelompok berdasarkan lokasi. Sampel yang berasal dari Sumatra dan Kalimantan Barat mengelompok dengan spesies pembanding Chitala lopis (AP008922.1 dan KM213054.1) pada satu group. Terlihat pada group Ib, spesies Chitala lopis (AP008922.1) berada satu sub-group dengan Chitala lopis (KM213054.1) yang berasal dari sungai Kampar, Riau. Berdasarkan perhitungan jarak genetik, perbedaan antara sampel yang berasal dari Sumatra dengan Chitala lopis (AP008922.1) tidak berbeda jauh dengan nilai sebesar 0.71%. Begitu pula dengan hasil analisis jarak genetik antara Chitala lopis (AP008922.1) dan Chitala lopis (KM213054.1) yang hanya memiliki perbedaan sebesar 0.40%. Berdasarkan hasil tersebut, spesies pembanding Chitala lopis (AP008922.1) diduga berasal dari Indonesia, khususnya populasi Sumatra. Pada group Ia, populasi Kalimantan Barat memiliki kekerabatan lebih dekat dengan populasi Sumatra. Hasil ini ditunjang oleh nilai Jarak genetik perbedaan populasi Kalimantan Barat dengan Sumatra sebesar 0.60% dan sejarah aliran sungai Sunda besar pada zaman Pleistocene (Voris 2000). Pada spesies outgroup, terlihat memiliki klaster terpisah yang berarti spesies tersebut berbeda atau memiliki kekerabatan yang jauh yaitu rata-rata jarak genetik sebesar 12.97%. Namun demikian pada penelitian Wibowo (2014), C. lopis yang berasal dari sungai Indragiri dan sungai Mahakam dalam rekonstruksi pohon filogeninya berada pada klaster yang berbeda. Berdasarkan hasil penelitian ini dan hasil penelitian lain rekonstruksi pohon filogeni berdasarkan gen COI mampu memisahkan kelompok populasi ikan belida dengan memiliki jumlah situs spesifik bervariasi. Hal ini menunjukkan bahwa hubungan kekerabatan berdasarkan runutan gen COI DNA mitokondria memiliki kepercayaan yang tinggi dalam pengelompokkan populasi intraspesies. Cabang pada pohon filogenetik mewakili hubungan antar unit yang menggambarkan hubungan keturunan dengan leluhurnya, sedangkan panjang cabang menggambarkan jumlah perubahan evolusioner yang terjadi antara dua nodus (Li & Graur 2000). Menurut Kottelat et al. (1993) di Indonesia memiliki 4 jenis ikan belida, namun demikian dari hasil penelitian ini jarak genetik antara Sumatra dengan Kalimantan Barat sebesar 0.60%, sedangkan perbandingan dengan Kalimantan Selatan sebesar 1.63%. Hal ini belum memberikan bukti adanya 4 spesies tersebut karena perbedaan jarak genetik tidak melebihi ambang batas yang telah ditentukan sebesar >3% (Hebert et al. 2003; Ward et al. 2009).
15
5 SIMPULAN DAN SARAN
Simpulan
Populasi Sumatra dan Kalimantan terpisahkan secara baik dan jelas dengan jarak genetik rata-rata sebesar 1,12%. Ikan Belida asal Sumatra dan Kalimantan memiliki 13 situs nukleotida polimorfisme sedangkan situs nukleotida spesifik geografik hanya 2 situs (66 dan 273). Sampel yang berasal dari populasi Kalimantan Selatan memiliki 9 situs nukleotida spesifik. Hasil ini juga ditunjang dengan data dari hasil analisis jarak genetik dan rekonstruksi pohon filogenetik. Belida Kalimantan Barat memiliki kekerabatan lebih dekat dengan Belida Sumatra (0.60%) dibandingkan dengan belida Kalimantan Selatan (1.63%).
Saran
Perlunya penambahan sampel dari berbagai daerah di Indonesia untuk memberikan informasi dasar mengenai karakteristik genetik ikan Belida. Penambahan gen mitokondria seperti gen Cytochrome b (Cyt b) dan D-loop (Control Region), diharapkan mampu berkontribusi untuk menambah informasi ilmiah karakter genetik serta memetakan populasi ikan Belida di Indonesia. Berdasarkan hasil penelitian ini, populasi belida Kalimantan Barat mampu menunjuang upaya konservasi pada populasi Sumatra maupun sebaliknya dikarenakan kedekatan jarak genetiknya.
16
DAFTAR PUSTAKA
Adjie S. Husnah. Gaffar AK. 1999. Studi biologi ikan belida (Notopterus chitala) di daerah aliran Sungai Batanghari. Provinsi Jambi. Jurnal Penelitian Perikanan Indonesia. 1: 38-43.
Adjie S. Utomo AD. 1994. Aspek Biologi Ikan Belida (Notopterus chitala) di Sungai Lempuing. Sumatra Selatan. Prosiding Seminar PPEHP Perikanan Perairan Umum Palembang: 174-177.
Artamonova VS. Kolmakova OV. Kirillova EA. Makhrov AA. 2018. Phylogeny of Salmonoid Fishes (Salmonoidei) Based on mtDNA COI Gene Sequences (Barcoding). Contemporary Problems of Ecology. 11 (3): 271-285.
Bhattacharya M. Sharma AR. Patra BC. Sharma G. Seo EM. Nam JS. Chakraborty C. Lee SS. 2016. DNA Barcoding to Fishes: Current Status and Future Directions. Mitochondrial DNA. 27: 2744-2752.
Bingpeng X. Heshan L. Zhilan Z. Chunguang W. Yanguo W. Jianjun W. 2018. DNA barcoding for identification of fish species in the Taiwan Strait. PLoS ONE. 13(6).
Bofkin L. Goldman N. 2007. Variation in evolutionary processes at different codon positions. Molecular Biology and Evolution. 24 (2): 513-521.
Broughton RE. Milam JE. Roe BA. 2001. The Complete Sequence of the Zebrafish (Danio rerio) Mitochondrial Genome and Evolutionary Patterns in Vertebrate Mitochondrial DNA. Genome Res 11:1958-1967.
Esa YB. Japning JRR. Rahim KAA. Siraj SS. Daud SK. Tan SG. Sungan S. 2012. Phylogenetic Relationships among Several Freshwater Fishes (Family: Cyprinidae) in Malaysia Inferred from Partial Sequencing of the Cytochrome b Mitochondrial DNA (mtDNA) Gene. Pertanika Journal of Tropical Agricultural Science. 35 (2): 307-318.
Fields AT. Abercrombie DL. Eng R. Feldheim K. Chapman DD. 2015. A Novel Mini-DNA Barcoding Assay to Identify Processed Fins from Internationally Protected Shark Species. PLoS ONE. 10(2).
Gordeeva NV. Volkov AA. 2016. A New Primer Set for Amplification of the Cytochrome b Gene in Lantern Fishes (Myctophidae). Journal of Ichthyology. 56: 899-903.
Habib M. Lakra WS. Vindhya M. Khare P. Barman AS. Singh A. Lal KK. Punia P. Khan AA. 2011. Evaluation of cytochrome b mtDNA sequences in genetic diversity studies of Channa marulius (Channidae: Perciformes). Molecular Biology Reports. 38: 841-846.
Hebert PDN. Cywinska A. Ball SL. deWard JR. 2003. Biological identifications through DNA barcodes. Proceedings of the Royal Society B. 270: 313-322. Inger RF. Chin PK. 1962. The fresh-water fishes of North Borneo. Fieldiana
Zoology. 45: 1-268.
Inoue JG. Kumazawa Y. Miya M. Nishida M. 2009. The historical biogeography of the freshwater knifefishes using mitogenomic approaches: A Mesozoic origin of the Asian notopterids (Actinopterygii: Osteoglossomorpha). Molecular Phylogenetics and Evolution. 51: 486-499.
17 Johnson JB. Jordan S. 2000. Phylogenetic divergence in leatherside chub (Gila copei) inferred from mitochondrial cytochrome b sequences. Molecular Ecology. 9: 1029-1035.
Jusmaldi. Solihin DD. Affandi R. Rahardjo MF. Gustiano R. 2014. Kode batang DNA ikan lais genus Kryptopterus asal Sungai Mahakam Kalimantan Timur. Jurnal Iktiologi Indonesia. 14(3):191-199.
Kartavtsev YP. Lee JS. 2006. Analysis of Nucleotide Diversity at the Cytochrome b and Cytochrome Oxidase 1 Genes at the Population. Species. and Genus Levels. Genetika. 42:437-461.
Kartavtsev YP. Park T-J. Vinnikov KA. Ivankov VN. Sharina SN. Lee J-S. 2007. Cytochrome b (Cyt-b) gene sequence analysis in six flatfish species (Teleostei. Pleuronectidae). with phylogenetic and taxonomic insights. Marine Biology. 152: 757-773.
Kottelat M. Whitten JA. Kartikasari N. Wiryoatmojo S. 1993. Freshwater fishes of Western Indonesia and Sulawesi. Jakarta (ID): Periplus Edition and Emdi Project Indonesia. p 5.
Kumar S. Stecher G. Tamura K. 2016. MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets. Mol Biol Evol. 33(7):1870-1874. Li WH. Graur D. 2000. Fundamental of molecular evolution. Second edition.
Sinauer Associates Inc. Sunderland (US). 481p.
Linh NM. Thu PT. Quan NV. Chien PV. Ly DH. Nhan DV. Len DT. 2018. DNA Barcoding Application of Mitochondrial COI Gene to Identify Some Fish Species of Family Gobiidae in Vietnam. Journal of Marine Science and Technology. 18 (4): 433-451.
Mandal A. Mohindra V. Singh RK. Punia P. Singh AK. Lal KK. 2012. Mitochondrial DNA variation in natural populations of endangered Indian Feather-Back Fish. Chitala chitala. Molecular Biology Reports. 39:1765-1775. Menteri LHK. 2018. Peraturan Lingkungan Hidup dan Kehutanan Republik
Indonesia. Nomor: P.20/ MENLHK/SETJEN/KUM.1/6/2018 tentang jenis tumbuhan dan satwa yang dilindungi.
MUAWC. 2008. Blood collection guidelines. Monash University Animal Welfare Committee (MUAWC). 1-10.
Nugroho E. Dewi RRSPN. Subagja J. Priono B. 2019. Keragaman Genetik dan Karakter Biometrik Ikan Belida (Chitala lopis. Bleeker 1851) Budidaya Asal Sungai Kampar. Riau. Jurnal Riset Akuakultur. 14 (1): 1-8.
Page TJ. Hughes JM. 2010. Comparing the Performance of Multiple Mitochondrial Genes in the Analysis of Australian Freshwater Fishes. Journal of Fish Biology. 77: 2093-2122.
Pemerintah Republik Indonesia. 2004. Undang-Undang Republik Indonesia Nomor 31 Tahun 2004 tentang Perikanan. Jakarta (ID): Sekretariat Negara.
Peng Z. He S. Zhang Y. 2004. Phylogenetic relationships of glyptosternoid fishes (Siluriformes: Sisoridae) inferred from mitochondrial cytochrome b gene sequences. Molecular Phylogenetics and Evolution. 31: 979-987.
Pereira SL. 2000. Mitochondrial Genome Organization and Vertebrate Phylogenetics. Gen Mol Biol 23:745-752.
Santoso L. 2009. Biologi Reproduksi Ikan Belida (Chitala lopis) Di Sungai Tulang Bawang. Lampung. Berkala Perikanan Terubuk. 31 (1): 38-46.
18
Sarkar UK. Negi RS. Deepak PK. Lakra WS. and Paul SK. 2008. Biological parameters of the endangered fish Chitala chitala (Osteoglossiformes: Notopteridae) from some Indian Rivers. Fisheries Research. 90: 170-177. Schuster WH. Djajadiredja RR. 1952. Local common name of Indonesian fishes .
W.Van Hoeve. Bandung. p. 237.
Sengupta S. Homechaudhuri S. 2013. DNA barcodes of some threatened freshwater indigenous fishes in India. Current Science. 105 (1): 84-90.
Smith PJ. McVeagh SM. Steinke D. 2008. DNA barcoding for the identification of smoked fish products. Journal of Fish Biology. 72: 464-471.
Solihin DD. 1994. Peran DNA Mitokondria (mtDNA) dalam Studi Keragaman Genetik dan Biologi Populasi pada Hewan. Hayati. 1:1-4.
Sudarto. Pouyaud L. Kusuma RV. 2008. Struktur Populasi dan Sejarah Kolonisasi Ikan Botia (Chromobotia Macracanthus Bleeker) Asal Sumatera dan Kalimantan Berdasarkan Sekuen Intron dari Gen Aldolase-B. Jurnal Perikanan. X (2): 203-212.
Sudarto. Rizal M. 2007. Variasi Morfometri Ikan Botia (Botia macracanthus Bleeker) dari Perairan Sumatera dan Kalimantan. Jurnal Perikanan. IX (2): 214-219.
Sunarno MTD. Syamsunarno MB. 2015. Pengaruh Naungan Terhadap Pematangan Gonad dan Pemijahan Ikan Belida (Chitala lopis) di Kolam Rawa. Jurnal Ilmu Pertanian dan Perikanan. 4 (1): 71-77.
Sunarno MTD. Wibowo A. Subagja. 2007. Identifikasi Tiga Kelompok Ikan Belida (Chitala lopis) di Sungai Tulang Bawang. Kampar. dan Kapuas dengan Pendekatan Biometrik. Jurnal Penelitian Perikanan Indonesia. 13 (2): 87-94. Takehana Y. Nagai N. Matsuda M. Tsuchiya K. Sakaizumi K. 2003. Geographic
Variation and Diversity of the Cytochrome b Gene in Japanese Wild Populations of Medaka. Oryzias latipes. Zoological Science. 20: 1279-1291.
Voris HK. 2000. Maps of Pleistocene sea-levels in South East Asia: shorelines, river systems, time durations. Journal of Biogeography. 27: 1153-1167.
Ward RD. Hanner R. Hebert PDN. 2009. The campaign to DNA barcode all fishes. FISH-BOL. Journal of Fish Biology. 74: 329-356.
Ward RD. Holmes BH. 2007. An analysis of nucleotide and amino acid variability in the barcode region of cytochrome c oxidase I (cox1) in fishes. Molecular Ecology Notes. 7:899-907.
Ward RD. Zemlak TS. Innes BH. Last PR. Hebert PDN. 2005. DNA Barcoding Australia’s Fish Species. Philosophical Transactions of the Royal Society B. 360: 1887-1857.
Weber M. de Beaufort LF. 1916. The Fishes of The Indo-Australian Archipelago. Vol VIII. E.J. Brill Ltd.. Leiden. 456 p.
Wibowo A. 2012. Struktur Genetik Populasi Ikan Belida (Chitala lopis. Bleeker 1851) di Waduk Kutopanjang. BAWAL. 4 (1): 53-58.
Wibowo A. 2014. Barcoding Ikan Belida (Chitala Lopis) Berdasarkan Gen Cytochrome Oxidase Subunit I (COI) DNA Mitokondria. Prosiding Seminar Nasional Tahunan XI Hasil Penelitian Perikanan dan Kelautan Tahun 2014 Jilid II. Yogyakarta (ID): Jurusan Perikanan dan Kelautan Fakultas Pertanian UGM. 227-235 hlm.
Wibowo A. Affandi R. Soewardi K. Sudarto. 2010. Genetic Differentiation of the Kampar River's Giant Featherback (Chitala lopis Bleeker 1851) Base on
19 Mitochondrial Dna Analysis. Indonesian Fisheries Research Journal. 16 (2): 49-58.
Wibowo A. Marson. 2012. Fenomena Plastisitas Fenotipik Ikan Belida (Chitala lopis) di Sungai Kampar. Riau. BAWAL. 4 (3): 195-204.
Wibowo A. Subagja. 2014. Penilaian Indeks Kualitas Lingkungan Untuk Menentukan Wilayah Konservasi Ikan Belida (Chitala lopis) di Sungai Kampar. Riau. BAWAL. 6 (1): 1-9.
www.fishbase.com. Chitala lopis. [terhubung berkala]. http://www.fishbase.se/summary/Chitala-lopis.html [17 Jan 2017].
22
RIWAYAT HIDUP
Penulis dilahirkan di kota Bogor, Jawa Barat pada tanggal 26 Agustus 1993 dari ayah Zainal Abidin Arief dan ibu Nurani Nurulaini. Penulis adalah putra kedua dari tiga bersaudara. Penulis memulai pendidikannya di TK Kuncup Harapan, Bogor, kemudian melanjutkan Pendidikan di SDN Bantarjati 5, Bogor. Menyelesaikan Pendidikan di SMP PGRI 5, Bogor pada tahun 2008 dan melanjutkan ke SMA PGRI 4, Bogor. Mulai menempuh Pendidikan di perguruan tinggi pada tahun 2011 di Universitas Pakuan program studi Biologi, FMIPA dan lulus mendapatkan gelar sarjana pada tahun 2015. Penulis sempat magang di Instalasi Penelitian dan Pengembangan Teknologi Lingkungan dan Toksikologi Perikanan Budidaya Air Tawar Cibalagung, Bogor dengan mengerjakan beberapa proyek, salah satunya Aquaponik (Yumina Bumina) bersama FAFI dan FAO sebagai teknisi lapangan. Pada tahun 2016, penulis melanjutkan pendidikan magister di IPB University program studi Biosains Hewan, FMIPA. Selama menempuh studi, penulis pernah menjadi asisten praktikum mata kuliah Ekologi Hewan di Sekolah Vokasi IPB University pada T.A 2016/2017 dan 2017/2018. Selama menempuh studi Magister, penulis mempublikasikan beberapa artikel pada jurnal Nasional yaitu, “Perilaku Harian Pachliopta aristolochiae Betina di Museum Serangga dan Taman Kupu Taman Mini Indonesia Indah” pada Jurnal Sumberdaya HAYATI, sebagai syarat lulus mata kuliah Perilaku Hewan dan “Karakteristik genetik ikan belida (Chitala lopis, Bleeker 1851) asal pulau Sumatra dan Kalimantan berdasarkan gen COI“ pada Jurnal Iktiologi Indonesia yang merupakan hasil penelitian dari tesis ini. Pada tahun 2020, penulis serta mengikuti pelatihan yang diselenggarakan oleh LIPI. Pelatihan tersebut merupakan pelatihan penanganan Virus SARS-coV-2 yang dimana pada tahun 2020 terjadi pandemi COVID-19 dengan program yang bernama “Indonesia Memanggil“.