AMPLIFIKASI GEN PENYANDI CARBOHYDRATE BINDING MODULE
(CBM) GbtXyl43B DARI Geobacillus thermoleovorans IT-08
SKRIPSI
IKA QURROTUL AFIFAH
PROGRAM STUDI KIMIA DEPARTEMEN KIMIA
FAKULTAS SAINS DAN TEKNOLOGI UNIVERSITAS AIRLANGGA
i
AMPLIFIKASI GEN PENYANDI CARBOHYDRATE BINDING MODULE
(CBM) GbtXyl43B DARI Geobacillus thermoleovorans IT-08
SKRIPSI
IKA QURROTUL AFIFAH
PROGRAM STUDI KIMIA DEPARTEMEN KIMIA
FAKULTAS SAINS DAN TEKNOLOGI UNIVERSITAS AIRLANGGA
AMPLIFIKASI GEN PENYANDI CARBOHYDRATE BINDING MODULE
(CBM) GbtXyl43B DARI Geobacillus thermoleovorans IT-08
SKRIPSI
Sebagai Salah Satu Syarat untuk Memperoleh Gelar Sarjana Sains Bidang Kimia pada Fakultas Sains dan Teknologi
Universitas Airlangga
Oleh:
IKA QURROTUL AFIFAH NIM 081015063
Tanggal Lulus : 22 Agustus 2014
Disetujui oleh:
Pembimbing I, Pembimbing II,
iii
LEMBAR PENGESAHAN NASKAH SKRIPSI
Judul : Amplifikasi Gen Penyandi Carbohydrate Binding Module (CBM) GbtXyl43B dari Geobacillus thermoleovorans IT-08 Penyusun : Ika Qurrotul Afifah
NIM : 081015063
Pembimbing I : Prof. Dr. Ni Nyoman Tri P., M.Si. Pembimbing II : Dr. Purkan, S.Si., M.Si.
Tanggal seminar : 22 Agustus 2014
Disetujui oleh:
Pembimbing I, Pembimbing II,
Prof. Dr. Ni Nyoman Tri P., M.Si. Dr. Purkan, S.Si., M.Si. NIP. 19630615 198701 2 001 NIP. 19721116 199702 1 001
Mengetahui: Ketua Departemen Kimia, Fakultas Sains dan Teknologi
Universitas Airlangga
PEDOMAN PENGGUNAAN SKRIPSI
Skripsi ini tidak dipublikasikan, namun tersedia di perpustakaan dalam lingkungan Universitas Airlangga, diperkenankan untuk dipakai sebagai referensi kepustakaan, tetapi pengutipan harus seijin penyusun dan harus menyebutkan sumbernya sesuai kebiasaan ilmiah.
v
Afifah, Ika Qurrotul, 2014, Amplifikasi Gen Penyandi Carbohydrate Binding Module (CBM) GbtXyl43B dari Geobacillus thermoleovorans IT-08, Skripsi di bawah bimbingan Prof. Dr. Ni Nyoman Tri P., M.Si. dan Dr. Purkan, S.Si., M.Si., Departemen Kimia, Fakultas Sains dan Teknologi, Universitas Airlangga
ABSTRAK
Carbohydrate Binding Module (CBM) merupakan sisi non katalitik yang berperan untuk meningkatkan efisiensi katalitik enzim melalui pengikatan terhadap substrat polisakarida tak larut. Penelitian ini bertujuan untuk mengamplifikasi gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B beserta linkernya dari Geobacillus thermoleovorans IT-08 dan mengetahui urutan basa nukleotidanya. Amplifikasi dengan teknik PCR dilakukan menggunakan sepasang primer forward dan reverse yang diberi nama FCBM1 dan RCBM1. Primer didesain berdasarkan urutan Carbohydrate Binding Module (CBM) GbtXyl43B beserta linkernya pada urutan asam amino 1-156 gen penyandi exo-xilanase putative yang telah terdeposit dalam GenBank No. DQ387047. Hasil analisis menggunakan elektroforesis membuktikan bahwa amplikon memiliki ukuran gen sekitar 0,4 kb. Ukuran tersebut dikonfirmasi dengan hasil sekuensing menggunakan metode Sanger. Dari hasil pensejajaran dengan Basic Local Alignment Search Tool (BLAST), sekuen gen hasil amplifikasi terbukti sama dengan sekuen Carbohydrate Binding Module (CBM) GbtXyl43B target.
Afifah, Ika Qurrotul, 2014, Amplification of Gene Encoding Carbohydrate Binding Module (CBM) GbtXyl43B from Geobacillus thermoleovorans IT-08, This Project is under guidance by Prof. Dr. Ni Nyoman Tri P., M.Si. dan Dr. Purkan, S.Si., M.Si., Chemistry Department, Faculty of Science and Geobacillus thermoleovorans IT-08 and determine the nucleotide sequences of the gene. Amplification was performed by PCR using forward and reverse primer namely FCBM1 and RCBM1. Primer was designed based on the sequence of Carbohydrate Binding Module (CBM) along GbtXyl43B with it’s linker on the 1-156 amino acid of putative exo-xilanase sequence that has been deposited in GenBank No. DQ387047. The analysis using electrophoresis showed that the gene size of amplicon approximately 0,4 kb. The gene size is confirmed by the result of sequencing using the Sanger method. From the results of alignment with the Basic Local Alignment Search Tool (BLAST), the nucleotide sequence positif that the gene is Carbohydrate Binding Module (CBM) GbtXyl43B.
vii
KATA PENGANTAR
Puji syukur ke hadirat Allah SWT atas segala rahmat, karunia serta hidayah-Nya, sehingga penyusun dapat menyelesaikan skripsi dengan judul
“Amplifikasi Gen Penyandi Carbohydrate Binding Module (CBM) GbtXyl43B dari Geobacillus thermoleovorans IT-08”. Naskah skripsi ini disusun untuk memenuhi syarat kelulusan dalam menempuh pendidikan S1 di bidang kimia Fakultas Sains dan Teknologi, Universitas Airlangga.
Penyusunan naskah skripsi ini tidak lepas dari bantuan berbagai pihak. Oleh karena itu, dalam kesempatan ini penyusun mengucapkan terima kasih kepada:
1. Ibu Prof. Dr. Ni Nyoman Tri P., M.Si. selaku pembimbing I yang selalu memberikan arahan dan masukan serta meluangkan waktu bagi penyusun untuk berkonsultasi.
2. Bapak Dr. Purkan, S.Si., M.Si. selaku pembimbing II yang juga selalu memberikan arahan dan masukan serta meluangkan waktu bagi penyusun untuk berkonsultasi.
3. Ibu Dr. Sri Sumarsih, M.Si. selaku dosen penguji I yang telah memberikan saran, nasehat, dan masukan dalam menyelesaikan penyusunan skripsi ini. 4. Ibu Dra. Usreg Sri Handajani, M.Si. selaku dosen penguji II yang senantiasa
memberikan bimbingan dan nasehat selama penyusunan skripsi ini
5. Ibu Siti Wafiroh, S.Si, M.Si. selaku dosen wali yang senantiasa memberikan saran, nasehat, dan motivasi selama ini.
6. Seluruh tenaga pendidik dan tenaga kependidikan Departemen Kimia Fakultas Sains dan Teknologi Universitas Airlangga atas ilmu yang telah diberikan. 7. Bapak, Ibu, dan keluarga atas dukungan dan semangat demi terselesaikannya
skripsi ini.
No. 177 (MU177) yang selalu memberi dukungan dan motivasi penuh kepada penyusun untuk menyelesaikan skripsi ini.
Penyusun menyadari bahwa naskah skripsi ini masih jauh dari sempurna. Oleh karena itu, kritik dan saran yang bersifat membangun sangat diharapkan sehingga naskah skripsi ini dapat disempurnakan. Akhirnya penyusun berharap semoga skripsi ini bermanfaat bagi semua. Amin.
Surabaya, 11 Agustus 2014
Penyusun,
ix
PEDOMAN PENGGUNAAN SKRIPSI ... iv
ABSTRAK ... v
1.1 Latar Belakang Permasalahan ... 1
1.2 Rumusan Masalah... 3
1.3 Tujuan Penelitian ... 4
1.4 Manfaat Penelitian ... 4
BAB II: KERANGKA KONSEPTUAL ... 5
BAB III: METODE PENELITIAN……… 10
3.1 Tempat dan Waktu Penelitian ... 10
3.5.1.1 Pembuatan media cair untuk pemeliharaan E.coli pembawa plasmid pET-GbtXyl43B ... 13
3.5.1.2 Pembuatan media padat untuk E.coli pembawa plasmid pET-GbtXyl43B ... 13
3.5.2 Peremajaan isolat bakteri ... 13
3.5.3 Teknik DNA rekombinan ... 14
3.5.3.1 Isolasi DNA plasmid E.coli pembawa plasmid pET-GbtXyl43B ... 14
3.5.3.2 Amplifikasi gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B menggunakan teknik PCR ... 15
3.5.3.4 Pemurnian Produk PCR ... 16
3.5.3.5 Sekuensing dengan metode Sanger ... 16
BAB IV: HASIL DAN PEMBAHASAN ... 17
4.1 Isolasi DNA plasmid pET-GbtXyl43B ... 17
4.2 Amplifikasi gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B menggunakan teknik PCR ... 18
4.3 Analisis urutan basa nukleotida gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B ... 25
BAB V: KESIMPULAN DAN SARAN ... 31
5.1 Kesimpulan ... 31
5.2 Saran ... 31
DAFTAR PUSTAKA ... 32 LAMPIRAN
xi
DAFTAR GAMBAR
Nomor Judul Halaman
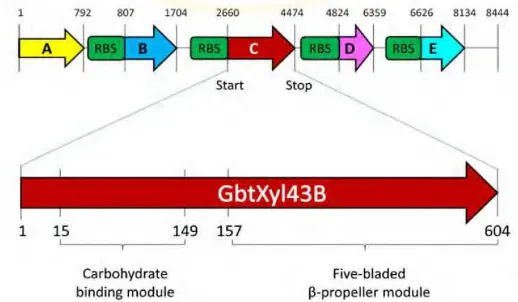
2.1 Topologi GbtXyl43B dalam klaster gen xilanase 5
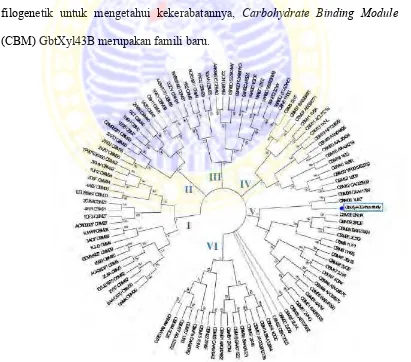
2.2 Pohon filogenetik dari famili CBM 6
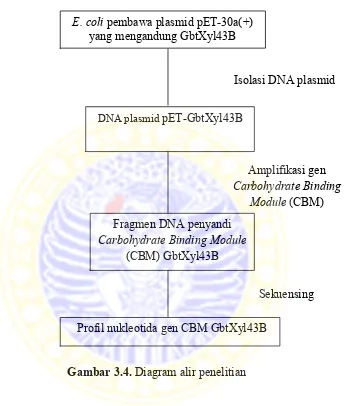
3.4 Diagram alir penelitian 12
4.1 Elektroforegram DNA plasmid pET-GbtXyl43B 18 4.2a Mekanisme pembentukan ikatan fosfodiester dari primer dan 19
dNTP oleh DNA polimerase pada sisi aktifnya
4.2b Parameter primer FCBM1 yang dihasilkan dari program Clone 21 Manager
4.2c Parameter primer RCBM1 yang dihasilkan dari program Clone 22 Manager
4.2d Siklus yang terjadi dalam PCR 23
4.2e Kondisi reaksi PCR 24
4.2f Elektroforegram hasil amplifikasi gen Carbohydrate Binding Module (CBM) GbtXyl43B beserta linkernya dengan
menggunakan primer FCBM1 dan RCBM1 24 4.2g Elektroforegram hasil pemurnian gen Carbohydrate
Binding Module (CBM) GbtXyl43B beserta linkernya 25 4.3 Proses sekuensing berdasarkan metode dideoksi Sanger yang
DAFTAR TABEL
Nomor Judul Halaman
xiii
DAFTAR LAMPIRAN
Nomor Judul
1.Komposisi Buffer PCR 2.Komposisi Buffer TAE
BAB I PENDAHULUAN
1.1. Latar Belakang Permasalahan
Limbah sektor pertanian, perkebunan dan kehutanan di Indonesia yang cukup melimpah belum dimanfaatkan secara optimal dan bahkan menimbulkan masalah lingkungan dan kesehatan karena dibiarkan menumpuk tanpa ada penanganan yang baik. Kandungan hemiselulosa yang cukup tinggi dalam limbah biomassa tersebut dapat dimanfaatkan menjadi monomer xilosa yang berpotensi untuk bahan baku industri.
Senyawa utama penyusun dinding tanaman adalah selulosa, hemiselulosa, dan lignin. Hemiselulosa yang terletak di dinding sel sekunder merupakan biopolimer bercabang heterogen yang mengandung gula pentosa (β-D-xilosa, α-L-arabinosa), gula heksosa (β-D-manosa, β-D-glukosa, α-D-galaktosa) dan atau asam urgonat (glukuronat, 4-O-metil-galakturonat dan asam α-D-galakturonat) (Girio et al., 2010;Mood et al., 2013).
Komposisi lignoselulosa menentukan jenis enzim yang diperlukan untuk degradasi sempurna substrat tersebut menjadi monomernya. Degradasi xilan memerlukan kompleks enzim xilanolitik yang meliputi endoxilanase (β-D-xilanase), eksoxilanase (β-D-xilosidase), dan α-L-arabinofuranosidase (Ratanakhanokchai et al., 1999;Fan et al., 2008).
Xilanase telah diisolasi dan dikarakterisasi dari berbagai macam mikroorganisme seperti jamur, bakteri dan ragi, dan beberapa telah dikloning dan diekspresikan pada Eschericia coli (Huang et al., 2006). Enzim xilanolitik telah diisolasi dari bakteri termofilik Geobacillus thermoleovorans IT-08 dari kawah panas Gunung Pancar Bogor Jawa Barat, Indonesia (Tan, 1999). Pada penelitian selanjutnya diperoleh gen klaster kelompok pengkode enzim xilanolitik dari Geobacillus thermoleovorans IT-08 melalui proses shotgun cloning. Gen klaster
yang terisolasi mempunyai panjang 8444 bp nukleotida, yang mengandung 5 gen meliputi gen transposase, ABC permease, GbtXyl43A (β-D-xylosidase gene, xyl43A GenBank No. DQ345777), arabinofuranosidase (abfa51 GenBank
No.DQ387046) dan ekso-xilanase putative (GenBank No. DQ387047) (Puspaningsih, 2004).
Ratnadewi (2013) mengkarakterisasi β-xilosidase kedua dari Geobacillus thermoleovorans IT-08 yang dinamakan GbtXyl43B. Hasil karakterisasi
menunjukkan bahwa GbtXyl43B termasuk kelompok famili GH43 yang memiliki dua domain pada strukturnya yaitu Carbohydrate Binding Module (CBM) dan five bladed β-propeller fold (Catalytic Module/CM). β-xilosidase menujukkan potensi
pulp dan industri kertas (Sunna and Antranikian, 1997;Teng et al., 2011). Pohon filogenetik menunjukkan bahwa CBM dari GbtXyl43B merupakan kelompok baru.
Carbohydrate Binding Module (CBM) bertanggung jawab untuk mendekatkan sisi katalitik enzim pada permukaan polisakarida yang tak larut sehingga meningkatkan aktivitas enzim (Obembe et al., 2007;Yin et al., 2011). Aplikasi domain non katalitik pengikat karbohidrat ini penting dalam berbagai macam industri. Carbohydrate Binding Module (CBM) difusikan ke beberapa enzim seperti kutinase yang digunakan dalam proses penghilangan kutikula di industri kapas dan lakase dalam proses pemutihan kertas.
Dalam penelitian ini akan dilakukan amplifikasi gen penyandi Carbohydrate Binding Module (CBM) pET-GbtXyl43B hasil penelitian
Ratnadewi (2013). Gen penyandi Carbohydrate Binding Module (CBM) diamplifikasi dengan teknik Polymerase Chain Reaction (PCR) menggunakan konsensus sepasang primer forward dan reverse yang telah didesain berdasarkan urutan basa nukleotida ekso-xilanase putative yang terdeposit dalam GenBank No. DQ387047. Hasil amplifikasi ini kemudian disekuensing untuk menentukan urutan basa nukleotidanya.
1.2. Rumusan Masalah
2. Bagaimanakah urutan basa nukleotida gen Carbohydrate Binding Module (CBM) GbtXyl43B dari Geobacillus thermoleovorans IT-08?
1.3. Tujuan Penelitian
1. Mengisolasi gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B dari Geobacillus thermoleovorans IT-08 dengan PCR menggunakan primer forward dan primer reverse yang telah didesain berdasarkan urutan basa nukleotida gen ekso-xilanase putative yang terdeposit dalam GenBank No. DQ387047.
2. Menentukan urutan basa nukleotida gen Carbohydrate Binding Module (CBM) GbtXyl43B dari Geobacillus thermoleovorans IT-08.
1.4. Manfaat Penelitian
Carbohydrate Binding Module (CBM) yang berhasil dipisahkan dari
BAB II
KERANGKA KONSEPTUAL
-Xilosidase (EC 3.2.1.37) merupakan salah satu enzim dalam kelompok xilanolitik yang mengkatalisis hidrolisis ikatan 1,4-β-D-xilo-oligosakarida dengan cara menghilangkan residu D-xilosa dari ujung non pereduksi (Subramaniyan and Prema, 2002). Sinergisitas -xilosidase dengan enzim 1,4-β-endoxilanase, α-L-arabinofuranosidase, α-D-glukuronidase, asetil xilan esterase dan asam fenolat (asam ferulat dan asam koumarat) esterase dapat mendegradasi sempurna substrat xilan (Collins et al., 2005).
Ratnadewi (2013) telah melakukan karakterisasi terhadap β-xilosidase kedua dari Geobacillus thermoleovorans IT-08 yang dinamakan GbtXyl43B. Hasil karakterisasi menunjukkan bahwa GbtXyl43B termasuk kelompok famili GH43 yang memiliki dua modul pada strukturnya yaitu Carbohydrate Binding Module (CBM) pada urutan asam amino 15-149 dan five bladed β-propeller fold (Catalytic Module/CM) pada urutan asam amino 157-604.
β-Xilosidase termasuk ke dalam kelompok enzim glikosida hidrolase yang dapat menghidrolisis ikatan glikosidik. Glikosida hidrolase melibatkan modul katalitik dan modul non katalitik dalam menghidrolisis polisakarida. Modul pengenalan polisakarida non katalitik dari glikosida hidrolase disebut sebagai CBD (Cellulose Binding Domains) karena contoh pertama dari domain protein ini mengikat selulosa kristalin sebagai ligan primernya (Gilkes et al., 1988; Boraston et al., 2004). Selanjutnya istilah yang lebih inklusif adalah CBM (Carbohydrate
Binding Module) yang menunjukkan spesifitas ligan beragam dari modul ini
(Boraston et al., 2004;Christiansen et al., 2009). Dari analisa menggunakan pohon filogenetik untuk mengetahui kekerabatannya, Carbohydrate Binding Module (CBM) GbtXyl43B merupakan famili baru.
Carbohydrate Binding Module (CBM) dapat mengikat polisakarida tak larut baik komponen dinding sel tanaman seperti selulosa, kitin, xilan, glukan, galaktan maupun polisakarida simpanan seperti kanji dan glikogen. Banyak CBM yang telah diidentifikasi secara eksperimental dan beberapa ratus CBM putative diidentifikasi berdasarkan kesamaan asam amino seperti modul katalitik glikosida hidrolase (Boraston et al., 2004).
Berdasarkan kesamaan struktur dan fungsinya Carbohydrate Binding Module (CBM) dikelompokkan menjadi tiga tipe yaitu CBM suface-binding (tipe
A), CBM glycan-chain-binding (tipe B), dan CBM smallsugar-binding (tipe C) (Boraston et al., 2004). CBM tipe A mengikat substrat tak larut seperti selulosa kristalin dan kitin. Permukaan hidrofob CBM tipe A tersusun atas tiga residu aromatis yang mengikat permukaan tak larut mikrofibil selulosa (Gilbert et al., 2013). CBM tipe B memiliki sisi pengikatan yang lebih sesuai untuk interaksi dengan rantai glikan. Seperti pada CBM tipe A, residu aromatis juga memiliki peranan penting dalam pengikatan substrat dan orientasi asam amino merupakan kunci penentu spesifitasnya. Rantai samping dari triptofan, tirosin dan sebagian kecil fenilalanin membentuk sisi pengikatan CBM yang hidrofobik. Perbedaannya dengan tipe A adalah ikatan hidrogen langsung juga menentukan afinitas dan spesifitas substrat. CBM tipe C mengikat substrat ligan kecil seperti mono-, di- atau trisakarida. Peranan ikatan hidrogen antara protein dengan karbohidrat pada tipe ini lebih luas dibandingkan pada tipe B (Boraston et al., 2004).
penelitian sebelumnya menunjukkan bahwa dua jenis kutinase Thermobifida fusca yang telah difusikan CBM mampu menurunkan kutikula dari serat kapas dengan efisiensi yang relatif tinggi dibandingkan dengan kutinase. Carbohydrate Binding Module (CBM) direkatkan dengan C-terminal dari kutinase untuk meningkatkan efisiensi katalitik (Zhang et al., 2010). Lakase dari Pycnoporus cinnabarinus telah berhasil difusikan dengan linker dan Carbohydrate Binding Module (CBM) famili 1 dari Aspergillus niger CBHB. Pemberian Carbohydrate Binding Module (CBM) juga dapat meningkatkan kemampuan pengikatan lakase terhadap substrat lignoselulosa seperti pulp kertas. Adanya modul non katalitik ini mengurangi jumlah lakase yang dibutuhkan untuk efek katalitik yang sama (Ravalason et al., 2009).
Dalam aplikasi lain dua domain pengikat selulosa dari Clostridum cellulovorans difusikan untuk membentuk Cellulose Crosslinking Protein (CCP).
Rekombinan selulosa-pengikat protein diekspresikan dalam E. coli kemudian diaplikasikan pada kertas saring selulosa Whatman. Dari hasil pengujian proses fusi ini dapat meningkatkan sifat mekanik kertas (Levy et al., 2002). Strategi fusi CBM juga telah digunakan untuk purifikasi protein berdasarkan afinitas terhadap selulosa. Endoglukanase (CenA) dari Cellulomonas fimi memiliki domain pengikat selulosa pada N-terminal dan domain katalitik pada C-terminal. CenA telah dikloning dalam E.coli dan produknya dimurnikan dari ekstrak sel menggunakan kromatografi afinitas terhadap selulosa (Greenwood et al., 1988).
thermoleovorans IT-08. Amplifikasi adalah proses pengambilan gen tertentu dari
suatu DNA yang dilakukan secara in vitro melalui teknik PCR (Brown, 2010). Hasil BLASTP penelitian Ratnadewi (2013) menemukan bahwa Carbohydrate Binding Module (CBM) GbtXyl43B dari Geobacillus thermoleovorans IT-08 terletak pada residu asam amino ke 15-149. Gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B beserta linkernya diamplifikasi dengan mesin
Polymerase Chain Reaction (PCR) menggunakan primer yang spesifik. Primer
didesain berdasarkan urutan basa nukleotida gen ekso-xilanase putative yang telah terdeposit dalam GenBank No. DQ387047 pada urutan asam amino 1-156.
Carbohydrate Binding Module (CBM) GbtXyl43B yang dihasilkan dari proses amplifikasi selanjutnya disekuensing untuk menentukan urutan basa nukleotidanya. Urutan basa nukleotida dapat digunakan untuk mengkarakterisasi Carbohydrate Binding Module (CBM) GbtXyl43B dari Geobacillus thermoleovorans IT-08 famili baru melalui pemodelan struktur dan mendukung
10 BAB III
METODE PENELITIAN
3.1 Tempat dan Waktu Penelitian
Penelitian ini dilaksanakan di Laboratorium Proteomik, Lembaga Penyakit Tropis (LPT), Universitas Airlangga, Surabaya. Penelitian ini dimulai pada bulan Februari 2014 sampai bulan Juni 2014.
3.2 Sampel Penelitian
Sampel yang digunakan dalam penelitian ini adalah isolat E. coli pembawa pET-30a(+) yang mengandung GbXyl43B (pET-GbtXyl43B). Isolat ini merupakan koleksi dari Laboratorium Proteomik, Lembaga Penyakit Tropis (LPT), Universitas Airlangga, Surabaya.
3.3 Bahan dan Alat Penelitian 3.3.1 Bahan penelitian
Bahan yang digunakan dalam penelitian ini antara lain: kit Qiagen (QIAprep Miniprep), QIAquick Gel Extraction Kit, dNTP, Taq DNA polimerase, primer forward FCBM1, primer reverse RCBM1, buffer PCR dengan MgSO4, ddH2O, agarosa, tripton, yeast extract, bacto agar, NaCl, buffer TAE, etidium bromida, dan kanamisin.
3.2.2 Alat penelitian
Alat-alat yang digunakan adalah autoklaf (OSK 6508 Steam pressure apparatus Ogawa Seiki Co., LTD), laminair air flow cabinet (Kottermann 8580),
3.4 Diagram Alir Penelitian
Gambar 3.4. Diagram alir penelitian E. coli pembawa plasmid pET-30a(+)
yang mengandung GbtXyl43B
Isolasi DNA plasmid
DNA plasmid pET-GbtXyl43B
Amplifikasi gen Carbohydrate Binding
Module (CBM) GbtXyl43B Fragmen DNA penyandi
Carbohydrate Binding Module (CBM) GbtXyl43B
Profil nukleotida gen CBM GbtXyl43B
3.5 Prosedur Penelitian 3.5.1 Pembuatan media
3.5.1.1 Pembuatan media cair untuk pemeliharaan E.coli pembawa plasmid pET-GbtXyl43B
Media cair yang digunakan adalah media Luria Bertani (LB) yang mengandung kanamisin. Media ini dibuat dengan melarutkan 1% (b/v) tripton, 1% (b/v) NaCl, dan 0,5% (b/v) yeast extract dalam akuades kemudian disterilkan dengan autoklaf suhu 1210C selama 15 menit. Media steril yang telah suam-suam kuku ditambah kanamisin 50 µg/mL.
3.5.1.2 Pembuatan media padat untuk E.coli pembawa plasmid pET-GbtXyl43B
Media yang digunakan merupakan media Luria Bertania (LB) yang mengandung kanamisin. Bacto agar 2% (b/v), tripton 1% (b/v), NaCl 1% (b/v), dan yeast extract 0,5% (b/v) dilarutkan dalam aquades dan disterilkan dengan autoklaf suhu 1210C selama 15 menit. Media steril yang telah suam-suam kuku ditambah dengan kanamisin 50 µg/mL, dihomogenkan kemudian dituang ke dalam cawan petri.
3.5.2 Peremajaan isolat bakteri
3.5.3 Teknik DNA rekombinan
Teknik DNA rekombinan dilakukan berdasarkan metode dalam Sambrook (1989).
3.5.3.1 Isolasi DNA plasmid E.coli pembawa plasmid pET-GbtXyl43B
Miniprep dipindahkan ke tabung Eppendorf 1,5 mL yang baru. Selanjutnya buffer
PB sebanyak 25 μL ditambahkan, didiamkan selama 1 menit dan disentrifugasi selama 1 menit untuk mengelusi DNA. Tahapan ini dilakukan sebanyak 2 x sehingga diperoleh volume akhir 50 μL. Keseluruhan tahap sentrifugasi dilakukan pada suhu 4°C dengan kecepatan 10.000 rpm.
3.5.3.2 Amplifikasi gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B menggunakan teknik PCR
Proses amplifikasi gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B menggunakan teknik PCR. Komponen reaksi PCR yang digunakan adalah 5 ng DNA template, 1x pfu buffer MgSO4, 0,2 mM dNTP mix, 0,002 u/mL enzim Taq DNA polimerase, 0,5 M primer FCBM1, 0,5 M primer RCBM1 dan milliQ water. Proses amplifikasi dilakukan pada kondisi denaturasi dengan suhu 95oC selama 30 s, annealing pada variasi suhu (sesuai dengan hasil optimasi PCR gradien) selama 30 s dan extension pada suhu 72oC selama 56 s dengan jumlah siklus 30 kali. Proses amplifikasi diawali dengan predenaturasi pada suhu 95oC selama 3 menit dan diakhiri dengan post extension yang dilakukan pada suhu 72oC selama 7 menit.
3.5.3.3 Elektroforesis gel agarosa
Elektroforesis dihentikan ketika loading dye telah bermigrasi kira-kira 2/3 dari panjang gel. Gel agarosa kemudian direndam dalam larutan EtBr 0,5 μg/mL selama 15-30 menit dan selanjutnya direndam dalam bufer akuades selama 10 menit. Pita-pita DNA diamati dengan sinar UV.
3.5.3.4 Pemurnian Produk PCR
Pemurnian produk PCR dilakukan sesuai dengan prosedur yang ada dalam manual QIAquick Gel Extraction Kit. Dalam penelitian ini pemurnian tidak diawali dengan elektroforesis karena ukuran produk PCR kecil. Produk PCR ditambah dengan isopropanol dingin sebanyak 2x volume kemudian didiamkan. Sampel dipindahkan ke kolom QIAquick yang diletakkan dalam tube 2 mL dan disentrifugasi selama 1 menit. Larutan yang dapat melewati kolom dibuang. Buffer PE sebanyak 0,75 mL ditambahkan ke kolom QIAquick, didiamkan selama 5 menit dan disentrifugasi selama 1 menit. Larutan yang dapat melewati kolom dibuang dan kolom disentrifugasi kembali selama 5 menit. Kolom QIAquick selanjutnya dipindahkan ke tabung Eppendorf 1,5 mL yang baru. Buffer EB (10 mM Tris-Cl pH 8) sebanyak 10 μL ditambahkan ke pusat membran dan disentrifugasi selama 1 menit untuk mengelusi DNA. Tahapan ini dilakukan sebanyak 2 x sehingga dihasilkan volume akhir 20 μL. Keseluruhan tahap sentrifugasi dilakukan pada suhu 4°C dengan kecepatan 10.000 rpm.
3.5.3.5 Sekuensing dengan metode Sanger
HASIL DAN PEMBAHASAN
4.1. Isolasi DNA plasmid pET-GbtXyl43B
DNA plasmid pET-GbtXyl43B digunakan sebagai cetakan dalam amplifikasi gen Carbohydrate Binding Module (CBM) GbtXyl43B dengan PCR. Isolasi DNA plasmid dilakukan berdasarkan metode alkali dengan menggunakan kit dari Qiagen. Pada pH 12,0-12,5, DNA non-supercoiled terdenaturasi sedangkan DNA plasmid yang memiliki konformasi supercoiled tidak. Ikatan hidrogen dari DNA non-supercoiled putus sehingga menyebabkan konformasi untai ganda terbuka dan dua rantai polinukleotida terpisah. Penurunan pH dapat menyebabkan untai DNA yang terdenaturasi beragregasi. DNA plasmid dapat dipisahkan dengan DNA yang terdenaturasi dengan sentrifugasi (Brown, 2010). DNA yang terdenaturasi membentuk pelet yang tidak larut dan DNA plasmid larut dalam supernatan.
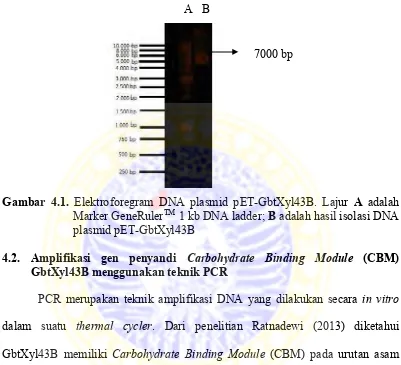
plasmid diukur dengan menggunakan Spektrofotometer NanoDrop. Kadar DNA plasmid pET-GbtXyl43B hasil isolasi adalah 205,8 ng/μL.
A B
Gambar 4.1. Elektroforegram DNA plasmid pET-GbtXyl43B. Lajur A adalah Marker GeneRulerTM 1 kb DNA ladder; B adalah hasil isolasi DNA plasmid pET-GbtXyl43B
4.2. Amplifikasi gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B menggunakan teknik PCR
PCR merupakan teknik amplifikasi DNA yang dilakukan secara in vitro dalam suatu thermal cycler. Dari penelitian Ratnadewi (2013) diketahui GbtXyl43B memiliki Carbohydrate Binding Module (CBM) pada urutan asam amino 15-149. Amplifikasi dilakukan terhadap CBM beserta linkernya pada urutan asam amino 1-156. Campuran reaksi yang diperlukan dalam PCR adalah cetakan DNA berupa plasmid pET-GbtXyl43B sebagai sumber gen Carbohydrate Binding Module (CBM) yang akan diamplifikasi, sepasang primer,
deoksinukleosida trifosfat (dNTP), bufer PCR yang mengandung MgSO4, dan enzim termostabil Taq DNA polimerase. Proses amplifikasi secara enzimatis membutuhkan primer yang merupakan sejumlah kecil oligonukleotida yang komplemen dengan ujung-ujung sekuens DNA cetakan (Howe, 2007). Sisi
cara menambahkan deoksinukleosida trifosfat (dATP, dGTP, dCTP dan dTTP) yang komplemen dengan DNA template. Dalam proses amplifikasi digunakan enzim DNA polimerase I dari organisme yang hidup dalam air panas Thermus aquaticus sehingga biasa disebut Taq polimerase (Brown, 2010). Taq polimerase
tidak memiliki aktivitas proof-reading (3’-5’ eksonuklease) sehingga sekitar 1 nukleotida dalam 104 satuan mengalami kesalahan. Taq polimerase tidak sepenuhnya termostabil karena memiliki waktu paruh sekitar 40 menit pada suhu 95°C. Hal ini menyebabkan penurunan aktivitas Taq polimerase dalam beberapa siklus PCR (Howe, 2007). DNA polimerase membutuhkan kofaktor berupa kation Mg2+ sehingga diperlukan penambahan larutan Mg2+ (Sambrook and Russel, 2001). Pada sisi aktif DNA polimerase terdapat dua kation Mg2+ yang terikat. Saat reaksi polimerisasi terjadi satu kation mengikat gugus hidroksil pada ujung 3’ primer, sedangkan kation lain mengikat dNTP. Kation yang terikat pada primer mengaktifkan gugus hidroksil 3’ untuk menyerang gugus α-fosforil dari dNTP. Adanya penyerangan tersebut menyebabkan terbentuknya ikatan O-P yang baru, sehingga jika reaksi polimerisasi berlangsung terus menerus akan terbentuk suatu rantai DNA (Berg et al., 2010).
teknik PCR adalah desain primer. Primer yang pendek memberikan spesifitas yang tinggi pada amplifikasi menggunakan template pendek seperti DNA plasmid (Howe, 2007). Sedangkan primer yang terlalu pendek dapat menyebabkan hibridisasi ke sisi non target sehingga menghasilkan produk amplifikasi yang tidak diinginkan (Brown, 2010). Persyaratan desain primer dalam Sambrook and Russel (2001) adalah sebagai berikut: (1) mengandung G+C sekitar 40-60%, (2) pajang nukleotida antara 18-25, (3) tidak terdapat repeat sequence atau self-complementary sequence lebih dari 3 bp agar tidak terjadi hairpin, (4) ujung 3’
dari primer forward dan reverse tidak saling berikatan agar tiak terjadi dimer, (5) perbedaan Tm antara primer tidak melebihi 5° C, (6) ujung 3’ dari primer berupa basa G atau C jika memungkinkan, (7) terdapat sekurang-kurangnya 3 basa sebelum sisi restriksi.
Dalam penelitian ini primer didesain berdasarkan urutan Carbohydrate Binding Module (CBM) GbtXyl43B berserta linkernya pada urutan asam amino
untuk primer reverse. Berikut hasil desain primer menggunakan program Clone Manager.
Tabel 4.2. Hasil desain primer menggunakan program Clone Manager
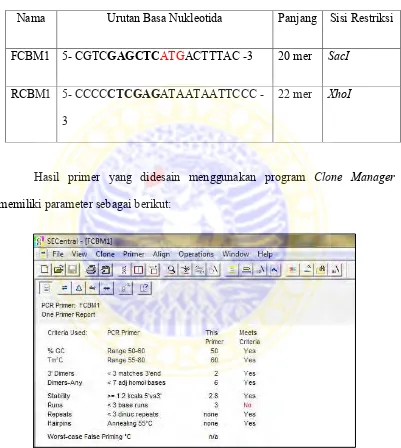
Hasil primer yang didesain menggunakan program Clone Manager memiliki parameter sebagai berikut:
Gambar 4.2b. Parameter primer FCBM1 yang dihasilkan dari program Clone Manager
Nama Urutan Basa Nukleotida Panjang Sisi Restriksi
FCBM1 5- CGTCGAGCTCATGACTTTAC -3 20 mer SacI
RCBM1 5 CCCCCTCGAGATAATAATTCCC -3
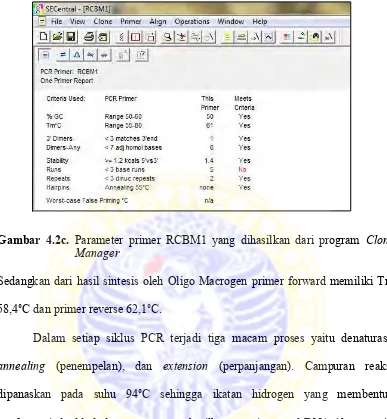
Gambar 4.2c. Parameter primer RCBM1 yang dihasilkan dari program Clone Manager
Sedangkan dari hasil sintesis oleh Oligo Macrogen primer forward memiliki Tm 58,4°C dan primer reverse 62,1°C.
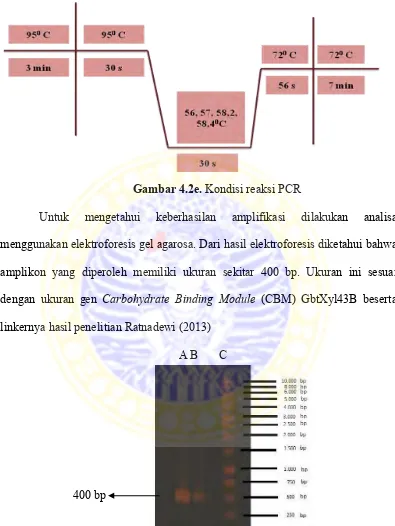
Dalam setiap siklus PCR terjadi tiga macam proses yaitu denaturasi, annealing (penempelan), dan extension (perpanjangan). Campuran reaksi
dipanaskan pada suhu 94°C sehingga ikatan hidrogen yang membentuk konformasi double helix terputus menghasilkan rantai tunggal DNA (denaturasi). Suhu kemudian diturunkan menjadi 50-60 °C sehingga memungkinkan terjadinya annealing (penempelan) primer pada sisi yang spesifik (Brown, 2010). Annealing
74 °C sesuai dengan suhu optimum enzim Taq DNA polimerase (Brown, 2010).
Gambar 4.2d. Siklus yang terjadi dalam PCR (Brown, 2010)
Dalam penelitian ini amplifikasi dilakukan dengan menggunakan alat thermal cycler gradien. Thermal cycler gradien memungkinkan untuk evaluasi
Gambar 4.2e. Kondisi reaksi PCR
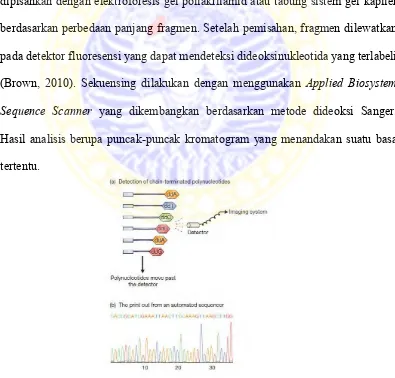
Untuk mengetahui keberhasilan amplifikasi dilakukan analisa menggunakan elektroforesis gel agarosa. Dari hasil elektroforesis diketahui bahwa amplikon yang diperoleh memiliki ukuran sekitar 400 bp. Ukuran ini sesuai dengan ukuran gen Carbohydrate Binding Module (CBM) GbtXyl43B beserta linkernya hasil penelitian Ratnadewi (2013)
A B C
menghilangkan sisa-sisa zat dari reagen PCR sehingga hanya tertinggal DNA saja. Pemurnian DNA dilakukan dengan menggunakan reagen dari QIAquick Gel Extraction Kit. Kadar amplikon diukur dengan menggunakan Spektrofotometer
NanoDrop. Kadar DNA Carbohydrate Binding Module (CBM) GbtXyl43B hasil amplifikasi adalah 482,8 ng/μL.
A B
Gambar 4.2g. Elektroforegram hasil pemurnian gen Carbohydrate Binding Module (CBM) GbtXyl43B beserta linkernya. Lajur A adalah Marker GeneRulerTM 1 kb DNA ladder; B adalah hasil pemurnian amplikon
4.3. Analisis urutan basa nukleotida gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B
Pada penelitian ini sekuensing dilakukan dengan menggunakan metode dideoksi terminasi rantai Sanger yang banyak diadopsi untuk prosedur sekuensing. Metode ini dimulai dengan sintesis untai yang komplementer dengan ssDNA menggunakan enzim E.coli polimerase I fragmen klenow (atau lebih sering modifikasi T7 DNA polimerase) dan campuran dNTP (dATP, dTTP, dGTP, dCTP). Sintesis yang dikatalisis oleh enzim DNA polimerase juga membutuhkan primer untuk inisiasi. Dalam proses sintesis, nukleotida (dNTP)
terdapat 2’, 3'-dideoksiribonukleosida trifosfat (ddNTP) yang tidak memiliki gugus OH bebas di posisi karbon 3’ dalam campuran reaksi terambil, polimerisasi akan berakhir karena tidak dapat terhubung dengan nukleotida selanjutnya (Wong, 2006).
Deoksinukleotida terdapat dalam jumlah yang lebih besar dari dideoksinukleotida sehingga sintesis tidak selalu berakhir didekat primer. Hasil dari reaksi polimerisasi adalah satu set fragmen baru dengan panjang yang berbeda-beda dan berakhir di dideoksinukleotida. Fragmen yang terbentuk dipisahkan dengan elektroforesis gel poliakrilamid atau tabung sistem gel kapiler berdasarkan perbedaan panjang fragmen. Setelah pemisahan, fragmen dilewatkan pada detektor fluoresensi yang dapat mendeteksi dideoksinukleotida yang terlabeli (Brown, 2010). Sekuensing dilakukan dengan menggunakan Applied Biosystem Sequence Scanner yang dikembangkan berdasarkan metode dideoksi Sanger.
Hasil analisis berupa puncak-puncak kromatogram yang menandakan suatu basa tertentu.
Data hasil sekuensing menggunakan primer forward adalah sebagai
Sedangkan data hasil sekuensing menggunakan primer reverse adalah sebagai berikut.
Sebelum disejajarkan, data sekuensing menggunakan primer reverse dibuat komplemennya dan dibalik dengan menggunakan software reverse complement yang terdapat di www.bioinformatics.org sehingga dihasilkan untai
AAGGGAAACACTTTTGTTAATGACGTAGTTTGTCATTACGACGATGAA AAAAAGGTTATCTCATCAAATGAAGT
Selanjutnya data hasil sekuensing disejajarkan dengan sekuens target Carbohydrate Binding Module (CBM) GbtXyl43B beserta linkernya pada basa
1-468. Berikut hasil pensejajaran dengan menggunakan program Clone Manager.
digabungkan untuk mendapatkan sekuens CBM yang utuh.
tgactttac agacgaataa aaaatcaaat tatatacttt gttataccag actaccaaaa
gaagatatca tttattcggc caaattagct tatagtatgc atcttgctta cagtaatgat
ggcataaatt ttgaaccgtt aaatcacaat tctggaattt tatttgcaaa ggctactgaa
aatgagaacg gttcccttaa cgcaaagagt ttaaaaaatc catatatatt ccatttgaaa
gacgggaatt tcggagtcat agctgttcgg actaaaccag atggtacaaa cgatgaggag
agtaaaggaa aagtccttat tttttcatcc ccggatttat tgcaatataa agaaattgga
ttgctagact tgaagggaaa cacttttgtt aatgacgtag tttgtcatta cgacgatgaa
aaaaaggttt atctcatcaa atggagtgat ggtttgggga attattat
KESIMPULAN DAN SARAN
5.1 Kesimpulan
Dalam penelitian ini dapat disimpulkan bahwa:
1. Gen penyandi Carbohydrate Binding Module (CBM) GbtXyl43B dari Geobacillus thermoleovorans IT-08 dapat diamplifikasi dengan PCR
menggunakan primer forward FCBM1 dan primer reverse RCBM1 2. Gen Carbohydrate Binding Module (CBM) GbtXyl43B yang diperoleh
dari proses amplifikasi dengan PCR memiliki ukuran 400 bp. 5.2 Saran
32
Berg, J. M., Tymockzo, J. L., and Stryer, L., 2010, Biochemistry, 7th Edition, W. H Freeman and Company, New York.
Boraston, Alisdair B., David N. Bolam, Harry J. Gilbert, and Gideon J. Davies, 2004, Carbohydrate-Binding Modules: Fine-Tuning Polysaccharide Recognition, Biochem. J, 382:769–781.
Brown, T. A., 2010, Gene Cloning and DNA Analysis, 6th Edition, Wiley Blackwell, UK.
Christiansen, Camilia, Maher Abou Hachem, Stefan Janecek, Anders Vikso Nielsen, Andreas Blennow, and Birte Svensson, 2009, The Carbohydrate-Binding Module Family 20-Diversity, Structure, and Function, FEBS Journal, 276:5006-5029.
Collins T., Gerday C., and Feller G., 2005, Xylanases, Xylanase Families and Extremophilic Xylanases, FEMS Microbiol Rev, 29:3-23.
Dieffenbach, C. W., T. M. J. Lowe, and G. S. Dveksler, 1993, General Consepts for PCR Primer Design, Genome Res., 3:S30-S37.
Fan, Zhanmin, Kurt Wagschal, Charles C. Lee, Que Kong, Katherine A. Shen, Indu B. Maiti, and Ling Yuan, 2008, The Construction and Characterization of Two Xylan-Degrading Chimeric Enzymes, Biotechnol. and Bioeng., 102:684-692.
Gilbert, Harry J., J. Paul Knox, and Alisdair B. Boraston, 2013, Advances in Understanding the Molecular Basis of Plant Cell Wall Polysaccharide Recognition by Carbohydrate-Binding Modules, Curr. Opinion in Strct. Biol., 23:669-677.
Polysaccharides, In: Higuchi, T. (Ed.), Biosynthesis and Biodegradation of Wood Components, Academic Press Inc., Orlando, FL, 109–135.
Howe, Christopher, 2007, Gene Cloning and Manipulation, Cambridge University Press, New York.
Huang, Junli, Guixue Wang and Li Xiao, 2006, Cloning, Sequencing and Expression of the Xylanase Gene from a Bacillus subtilis Strain B10 in
Escherichia coli, Bioresource Technol., 97:802–808.
Levy, Ilan, Amos Nussinovitch, Etai Shpigel, and Oded Shoseyov, 2002, Recombinant Cellulose Crosslinking Protein: A Novel Paper-Modification Biomaterial, Cellulose, 9:91–98.
Mood, Sohrab Haghighi, Amir Hossein Golfeshan, Meisam Tabatabaei, Gholamreza SalehiJouzani, Gholam Hassan Najafi, Mehdi Gholami, and Mehdi Ardjmand, 2013, Lignosellulosic Biomass to Bioethanol, a Comprehensive Review with a Focus on Pretreatment, Renew.and Sust. Review, 27:77-93.
Obembe, Olawole O., Evert Jacobsen, Jaap Timmers, Harry Gilbert, Anthony W. Blake, J. Paul Knok, Richard G. F. Visser, and Jean Paul Vincken, 2007, Promiscuous, Non-Catalytic, Tandem Carbohydrate-Binding Modules Modulate the Cell-Wall Structure and Development of Transgenic Tobacco (Nicotina tabacum) Plants, J. Plasnt Res., 120:605-617.
Puspaningsih, Ni Nyoman Tri, 2004, Pencirian Enzim Xilanolitik dan Kloning Gen Penyandi Xilosidase dari Bacillus thermoleovorans IT-08, Disertasi, Institut Pertanian Bogor, Bogor.
Ratanakhanokchai, Khanok, Khin Lay Kyu, and Morakot Tanticharoen, 1999, Purification and Properties of a Xylan Binding Endoxylanase from Alkaliphilic Bacillus sp. Strain K-1, Applied and Environ. Microbiol., 65(2):694–697.
Ratnadewi, Anak Agung Istri, Muchzainal Fanani, Sari Dewi Kurniasih, Makiko Sakka, Eddy Bagus Wasito, Kazuo Sakka, Zeily Nurachman, and Ni Nyoman Tri Puspaningsih, 2013, β-D-Xylosidase from Geobacillus thermoleovorans IT-08: Biochemical Characterization and Bioinformatics of the Enzyme, Appl Biochem Biotechnol, 170:1950– 1964.
Biobleaching, Journal of Biotechnol., 142:220–226.
Sambrook, J., and Russel, David W., 2001, Molecular Cloning, A Laboratory Manual, 3th Edition, Volume 2, Cold Spring Harbor Laboratory Press,
United States of America.
Subramaniyan, S., and P. Prema, 2002, Biotechnology of Microbial Xylanases: Enzymology, Molecular Biology and Application, Critical Rev. in Biol., 22(1):33-64.
Sunna, A., and G. Antranikian, 1997, Xylanolytic Enzymes from Fungi and Bacteria, Critical Rev. in Biotechnol., 17(1):39-67.
Tan, I., 1999, Characterization of Thermophilic Bacterium Producing Xylanolitic Enzyme from Hot Spring Gunung Pancar Bogor, Master Thesis, Institut Pertanian Bogor, Indonesia.
Teng, Chao, Huiyong Jia, Qiaojuan Yan, Peng Zhou, and Zhengqiang Jiang, 2011, High-Level Expression of Extracellular Secretion of A β-Xylosidase Gene from Paecilomyces thermophila in Escherichia coli, Journal of Bio. Technol., 102:1822–1830.
Wagschal, Kurt, Chamroeun Heng, Charles C. Lee, George H. Robertson, William J. Orts and Dominic W. S. Wong, 2009, Purification and Characterization of a Glycoside Hydrolase Family 43 β-xylosidase from Geobacillus thermoleovorans IT-08, Appl Biochem Biotechnol, 155:304–313.
Wong, Dominic W.S., 2006, The ABCs of Gene Cloning, Second Edition, Springer, USA.
Yin, Qiuyu, Yigang Teng, Ming Ding, and Fukun Zhao, 2011, Site-Directed Mutagenesis of Aromatic Residues in the Carbohyfrate-Binding Module of Bacillus endoglucanase EGA Decreases Enzym Thermostability, Biothecnol Lett, 33:2209-2216.
Komposisi 10x Pfu Buffer dengan 20 mM MgSO4: 200 mM Tris-HCl (pH 8,8 pada suhu 25°C)
100 mM (NH4)2SO4 100 mM KCl
1 mg/mL Bovine Serum Albumin (BSA)
1% (v/v) Triton X-100
Larutan Stok TAE 50X: 242 g Tris-Base
57,1 mL asam asetat glasial 100 mL EDTA 0,5 M pH 8 Konsentrasi larutan kerja (1x): 0,04 Tris-base