i
SAINS, TEKNOLOGI DAN REKAYASA
LAPORAN AKHIR
PENELITIAN PERCEPATAN PROFESOR
IDENTIFIKASI MOLEKULER ISOLAT BAKTERI
PENDEGRADASI INULIN DARI RIZOSFER
UMBI TANAMAN DAHLIA
Dr. Minda Azhar, M.Si.
NIDN 0024116406
Dibiayai oleh DIPA Universitas Negeri Padang melalui PNBP Fakultas MIPA
Surat Perjanjian Pelaksanaan Penelitian Nomor : 372/UN35.1/PG/2015
Tanggal 10 September 2015
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
UNIVERSITAS NEGERI PADANG
iv
ABSTRAK
Bakteri pendegradasi inulin dari rizosfer umbi dahlia merupakan sumber potensial gen dan enzim inulinase yang digunakan untuk pembuatan fruktosa dari inulin. Isolat bakteri pendegradasi inulin telah ditemukan dari rizosfer umbi tanaman dahlia yang tumbuh di Padang Panjang tetapi belum diidentifikasi secara molekuler. Tujuan utama penelitian adalah ditemukan nama genus dan spesies isolat bakteri pendegradasi inulin melalui identifikasi molekuler gen 16S rRNA. Metoda penelitian yang digunakan untuk mengidentifikasi isolat bakteri pendegradasi inulin secara molekuler adalah dengan mengkarakterisasi gen 16S rRNA melalui langkah mengisolasi DNA genom bakteri, mengamplifikasi gen 16S rRNA dengan metoda PCR dan sekuesing gen 16S rRNA menggunakan metoda dideoxy-Sanger. Data sekuens gen 16S rRNA bakteri dianalisis dengan program BLASTn pada National Center for Biotechnology Information (NCBI). Berdasarkan identifikasi secara molekuler melalui sekuens parsial gen 16S rRNA, isolat bakteri RZ-01 memiliki kekerabatan terdekat dengan kelompok genus Klebsiella dan spesies Klebsiella pneumoniae.
.
1
BAB I
PENDAHULUAN
1.1 Latar Belakang
Tanaman dahlia tumbuh subur di dataran tinggi Sumatera Barat seperti Solok,
Bukittinggi dan Padang Panjang. Umbi tanaman dahlia merupakan sumber inulin,
sedangkan rizosfer umbi dahlia terdapat bakteri pendegradasi inulin. Bakteri
pendegradasi inulin dari rizosfer umbi dahlia merupakan sumber potensial gen dan
enzim inulinase. Enzim inulinase adalah enzim yang penting dalam pangan. Enzim ini
sangat potensial digunakan sebagai biokatalis reaksi hidrolisis inulin menjadi fruktosa
dan fructooligosaccharide (FOS). Fruktosa dan FOS merupakan dua senyawa yang
sangat penting pada industri makanan, minuman, dan farmasi (Tohamy, 2006;
Sirisansaneeyakul et al., 2007).
Pembuatan fruktosa dari raw material inulin jauh lebih efektif dan efisien
dibandingkan dari pati. Pembuatan fruktosa dari pati melibatkan tiga enzim, yaitu amilolisis pati dengan katalis α-amilase dan amiloglukosidase, diikuti dengan pengubahan glukosa ke fruktosa yang dikatalisis oleh glukosa isomerase. Proses ini
menghasilkan maksimal hanya sekitar 42% fruktosa, sisanya 50% glukosa dan 8%
oligosakarida (Zittan, 1981). Sebaliknya, pembuatan fruktosa dari inulin hanya
melibatkan satu enzim yaitu inulinase.
Isolasi dan karakterisasi bakteri yang mempunyai aktivitas inulinase dari sampel
tanah telah dilaporkan pertamakali oleh Allais et al., pada tahun 1986. Kebanyakan
bakteri yang ditemukan adalah Flavobacterium multivorum (Allais et al., 1986).
Bakteri pendegradasi inulin telah disolasi dari beberapa tempat ekstrim seperti sumber
air panas, lokasi dengan pH tinggi dan lokasi di dasar laut dalam. Actinomycete
Nicardiopsis sp.DN-K15 diisolasi dari sedimen laut Jiaozhou Bay China
mengekspresikan inulinase alkalitoleran yang mempunyai aktivitas optimum pada
suhu 60ºC, pH 8 dan mempunyai range aktivitas yang baik dari pH 5-11 (Lu et al.,
2013). Bakteri pendegradasi inulin yang berasal dari sumber air panas Bukik Kili di
Solok Sumatera Barat telah diidentifikasi sebagai Bacillus licheniformis (Azhar et al.,
2013). Beberapa bakteri potensial pendegradasi inulin untuk pembuatan fruktosa telah
dieksplorasi dan diskrining dari sumber air panas Batu Bajanjang, Bukik Gadang dan
2
bawah di Solok (Azhar, et al, 2014). Bakteri pendegradasi inulin juga telah diskrining
dari rizosfer umbi tanaman dahlia yang tumbuh di Padang Panjang. Uji pendahuluan
secara kualitatif pada bakteri tersebut menunjukan aktivitas yang besar pada substrat
inulin, tetapi bakteri tersebut belum diidentifikasi secara molekuler. Identifikasi
molekuler ini penting untuk riset selanjutnya yaitu mendesain primer yang digunakan
untuk mengisolasi gen pengkode enzim inulinase.
1.2 Perumusan Masalah
Enzim pendegradasi inulin dari bakteri paling tepat digunakan sebagai katalis reaksi
hidrolisis inulin menghasilkan fruktosa dan FOS. Fruktosa dan FOS dari inulin sangat
potensial dikembangkan di daerah Sumatera Barat karena tanaman dahlia tumbuh
subur di dataran tinggi Sumatera Barat seperti Solok, Bukittinggi, dan Padang
Panjang. Pada umbi tanaman dahlia terdapat inulin dalam jumlah besar, sedangkan
pada rizosfer umbi tanaman dahlia kemungkinan besar ditemukan bakteri
pendegradasi inulin. Oleh sebab itu, bakteri pendegradasi inulin diisolasi dari rizosfer
umbi tanman dahlia. Isolasi bakteri dari rizosfer umbi dahlia yang tumbuh di Padang
3
BAB 2
TINJAUAN PUSTAKA
2.1 Inulin
Inulin adalah polimer alami kelompok karbohidrat dengan monomer fruktosa.
Antara monomer fruktosa pada inulin dihubungkan oleh ikatan (21) residu
-D-fructofuranosyl (Kulminskaya et al., 2003). Tiap ujung pereduksi untai polimer inulin
dapat hadir glukosa (Franck, 2003). Dengan demikian, hidrolisis sempurna inulin
menghasilkan fruktosa dan sedikit glukosa, jika diasumsikan tiap ujung molekul inulin
terikat satu residu glukosa.
Gambar 1. Struktur Inulin
Inulin terdapat pada umbi tanaman dahlia, akar chicory, dan umbi Jerusalem
artichoke dalam jumlah besar (Franck, 2003; Marchessault et al., 1980). Tanaman
chicory, Jerusalem artichoke tumbuh baik di Amerika Utara, sedangkan tanaman dahlia
dapat tumbuh baik di dataran tinggi Indonesia. Inulin sukar larut dalam air dingin dan
pelarut organik, sebaliknya inulin mudah larut dalam air panas. Kelarutan inulin dalam
air tergantung pada cara bagaimana inulin tersebut direkristalisasi. Inulin yang
direkristalisasi dengan etanol lebih besar kelarutannya dibandingkan dengan inulin yang
direkristalisasi dengan air (Phelps, 1965).
2.2 Definisi Spesies pada Bakteri
Sel bakteri berukuran kecil dibandingkan sel eukariot dan mempunyai keragaman
morfologis yang rendah (Madigan dan Martinko, 2006). Oleh sebab itu, metoda
4
pengklasifikasian bakteri. Pendekatan dengan metoda fenotifik meliputi ekspressi sifat
diantaranya warna dan bentuk koloni, pewarnaan gram serta uji biokimia.
Pendekatan genomik untuk pengklasifikasian mikroba dapat digunakan DNA total
atau urutan basa nukleotida suatu gen (Vandamme et al., 1996). Urutan basa nukleotida
suatu gen seperti gen 16S rRNA paling sering digunakan untuk pengklasifikasian atau
identifikasi bakteri. Gen ini telah digunakan sebagai penanda molekuler untuk
penentuan spesies bakteri pada saat ini. Kemiripan urutan basa nukleotida gen 16S
rRNA dapat digunakan sebagai “gold standard” untuk mengidentifikasi bakteri sampai
pada tingkat spesies (Armougom et al., 2009).
Molekul 16S rRNA merupakan salah satu molekul rRNA penyusun ribosom sub
unit kecil pada prokariot. Molekul 16S rRNA mempunyai fungsi yang identik pada
seluruh organisme, terdistribusi secara universal dan bersifat sangat lestari (Madigan
dan Martinko, 2006). Molekul rRNA lainnya pada prokariot adalah 5S rRNA, dan 23S
rRNA. Ukuran gen 5S rRNA adalah sekitar 120 basa, 23S rRNA sekitar 2900 basa,
sedangkan 16S rRNA sekitar 1500 basa. Ukuran gen 16S rRNA cukup memadai dan
memudahkan dalam proses amplifikasi gen tersebut dan dalam proses sekuensing.
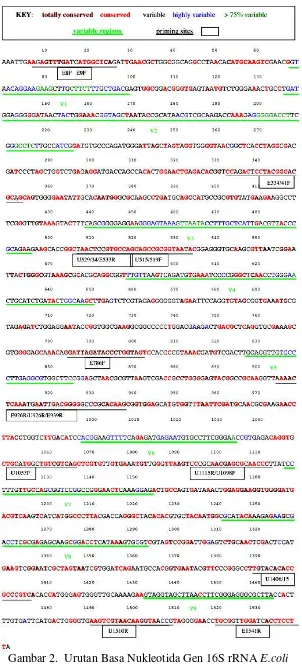
Gen 16S rRNA memiliki daerah-daerah yang secara universal bersifat lestari. Pada
beberapa bagian lain terdapat daerah yang bersifat semi-lestari, dan variabel. Pada gen
16S rRNA terdapat 9 daerah variabel yang ditandai dengan V1 sampai V9 (Gambar 2)
(Baker et al., 2003). Daerah-daerah variabel memungkinkan untuk membedakan
organisme dalam genus, bahkan spesies namun tidak antar strain dalam spesies yang
sama (Amann et al., 1995). Pada daerah yang sangat lestari (absolutely conserved)
dapat dijadikan primer universal untuk amplifikasi gen 16S rRNA bakteri secara PCR.
Analisis kesamaan urutan basa nukleotida gen 16S rRNA praktis untuk definisi
spesies. Derajat kesamaan urutan basa nukleotida gen 16S rRNA > 97% sering
dipertimbangkan sebagai kelompok spesies yang sama (Stackebrandt et al, 1994).
Penentuan derajat kesamaan urutan basa nukleotida suatu gen 16S rRNA dengan urutan
basa nukleotida gen 16S rRNA lainnya pada GenBank digunakan program BLASTn
pada http://www.ncbi.nlm.nih.gov. Analisis perbandingan urutan basa nukleotida dari
gen-gen 16S rRNA digunakan untuk mengkonstruksi pohon filogenetik dan dapat
dijadikan sebagai pengklasifikasian makhluk hidup. Dengan demikian, penggunaan
analisis gen 16S rRNA sebagai pendekatan untuk definisi spesies secara molekuler pada
5
6
Amplifikasi gen 16S rRNA bakteri dengan metoda PCR dapat digunakan primer
universal dan primer spesifik untuk spesies bakteri tertentu. Primer universal gen 16S
rRNA bakteri adalah primer yang komplemen dengan suatu urutan nukleotida yang
umum banyak terdapat dalam gen 16S rRNA dari bermacam-macam bakteri yang
berbeda. Daerah ini merupakan daerah yang paling lestari pada gen 16S rRNA bakteri.
Primer universal yang dipakai untuk amplifikasi gen 16S rRNA bakteri diantaranya
adalah primer 27F (GAGAGTTTGATCCTGGTCCAG), 765R (CTGTTT GCTCCCC
ACGCTTC) dan 1495R (CTACGGCTACCTTGTTACGA).
Langkah yang digunakan untuk mengidentifikasi suatu isolat murni bakteri secara
molekular melalui gen 16S rRNA adalah (1) mengisolasi DNA genom bakteri, (2) DNA
genom tersebut dijadikan template untuk mengisolasi gen 16S rRNA dengan teknik
PCR menggunakan primer universal, kemudian produk PCR dimurnikan dan dilakukan
sekuensing, (3) Basa nukleotida hasil sekuensing dibandingkan dengan basa nukleotida
sekuens gen 16S rRNA bakteri lain yang terdapat pada basis data GenBank dengan cara
menjajarkan. Proses penjajaran basa nukleotida tersebut dapat digunakan program
BLASTn yang terdapat pada GenBank. Derajat kesamaan urutan basa nukleotida gen
16S rRNA > 97% sering dipertimbangkan sebagai kelompok spesies yang sama. (4).
Hasil penjajaran dapat dibuat pohon phylogenetic.
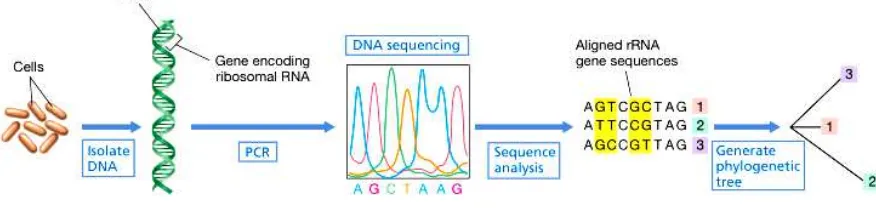
Pohon phylogenetik dapat dibuat menggunakan program MEGA5. Penjajaran
sekuens DNA yang mengkode 16S rRNA pada Gambar 3 dapat ditafsirkan bahwa
organisme 1 dan 2 terdapat tiga perbedaan basa, sedangkan organisme 1 dan 3
mempunyai 2 perbedaan basa. Organisme 2 dan 3 terdapat empat perbedaan basa.
Dengan demikian, organisme 1 dan 3 relatif paling dekat dibandingkan organisme 2 dan
3 atau 1 dan 2.
7
2.3 Bakteri Pendegradasi Inulin
Sekarang telah diketahui bahwa bakteri pendegradasi inulin mengekpresikan
inulinase dengan tipe exo- atau endo-. Beberapa bakteri pendegradasi inulin mampu
tumbuh pada suhu tinggi. Allais et al. telah mengisolasi dan mengkarakterisasi strain
bakteri yang mempunyai aktivitas inulinase dari sampel tanah yang ternyata
diidentifikasi sebagai Flavobacterium multivorum (Allais et al., 1986). Bacillus
polymyxa merupakan bakteri mesofilik dari tanah yang mengekspresikan exoinulinase
optimal aktif pada suhu 35ºC dan pH 7 (Kwon et al., 2003). Paenbacillus polymyxa
ZJ-9 diisolasi dari sampel tanah. Bakteri ini mengeskpresikan exoinulinase yang aktiv
maksimum pada suhu 25ºC, pH 6 (Gao et al., 2014).
Bakteri termofilik pertama, Geobacillus stearothermophilis KP1289 yang gen
pengkode exoinulinase telah diisolasi dari DNA genomnya dilaporkan pada tahun 2003
(Tsujimoto et al., 2003). Gen inuA (exoinulinase) bakteri ini terdiri dari 1482 pb yang
mengkode protein dengan 493 residu asam amino. Enzim ini aktif antara suhu 30C dan
75C dengan suhu optimum 60C. Inulinase termostabil dari Actinomycete nicardiopsis
sp.DN-K15 yang diisolasi dari sedimen laut Jiaozhou Bay China merupakan alkali
toleran yang mempunyai aktivitas optimum pada suhu 60ºC, pH 8 dan mempunyai
range aktivitas yang baik dari pH 5-11 (Lu et al., 2013). Bakteri pendegradasi inulin
dari sumber air panas Bukik Kili Solok Sumatera Barat diindentifikasi secara molekuler
melalui gen 16S rRNA merupakan Bacillus licheniformis (Azhar et al., 2013),
sedangkan dari sumber air panas Padang Balimbiang di Solok diidentifikasi sebagai
Bacillus subtilis (Azhar et al, 2011). Beberapa bakteri potensial pendegradasi inulin
untuk pembuatan fruktosa telah dieksplorasi dari sumber air panas: Batu Bajanjang,
Bukik Gadang dan Sapan Maluluang, serta rizosfer umbi dahlia (Azhar, et al, 2014).
Sampai saat ini, bakteri pendegradasi inulin yang mengekspresikan exoinulinase
yang gennya telah ditemukan adalah Geobacillus stearothermophilis, Paenibacillus
polymyxa, Pseudomonas mucidolens, Vibrio coralliilyticus ATCC BAA-450, Bacillus
subtilis, Bacillus sp snu-7 dan Paenibacillus sp. Aloe-11, Bacillus licheniformis,
Burkhoderia sp, Arthobacter siccitolerans (http://www.ncbi.nlm.gov/, pada 26 Maret
2015). Bakteri pendegradasi inulin yang mengekspresikan endoinulinase yang gennya
telah ditemukan adalah Arthrobacter sp. S37, Rhodopirellula baltica SH 1,
Paenibacillus mucilaginosus KNP414, Paennibacillus elgii B69, Pseudomonas
8
2.4 Studi Pendahuluan yang Telah Dilakukan
Penelitian aktivitas inulinase yang diekstrak dari umbi dahlia telah diteliti dengan judul “Aktivitas enzim inulinase umbi dahlia hasil fraksinasi dengan etanol” (Dana HED-JICA, 2006) dan telah dipublikasi pada Jurnal Sainsteks (terakreditasi) Vol.x,
No.1, 2007. Inulinase tumbuhan sangat sulit diisolasi dalam jumlah cukup. Oleh sebab
itu, mikroorganisme adalah pilihan untuk mengisolasi enzim dalam jumlah banyak.
Enzim ini dapat diperoleh dari bakteri pendegradasi inulin. Bakteri termofilik
pendegradasi inulin Bacillus licheniformis UBCT-007 dan Bacillus subtilis UBCT-030
yang masing-masing telah diskrining dari sumber air panas Bukik Kili dan Padang
Balimbiang di Solok (Azhar et al, 2013; Azhar dkk, 2011). Aktivitas enzim
ekstraseluler pada substrat inulin kecil. Gen pengkode enzim pendegradasi inulin yang
ditemukan pada B. licheniformis UBCT-007 adalah gen pengkode levanase (Azhar,
2013). Beberapa bakteri potensial pendegradasi inulin untuk pembuatan fruktosa telah
dieksplorasi dan diskrining dari sumber air panas Batu Bajanjang, Bukik Gadang dan
Sapan Maluluang Solok dan dari rizosfer umbi dahlia danau atas dan danau bawah
(Azhar, et al, 2014). Bakteri pendegradasi inulin juga telah diisolasi dari rizosfer umbi
tanaman dahlia yang tumbuh di Padang Panjang.
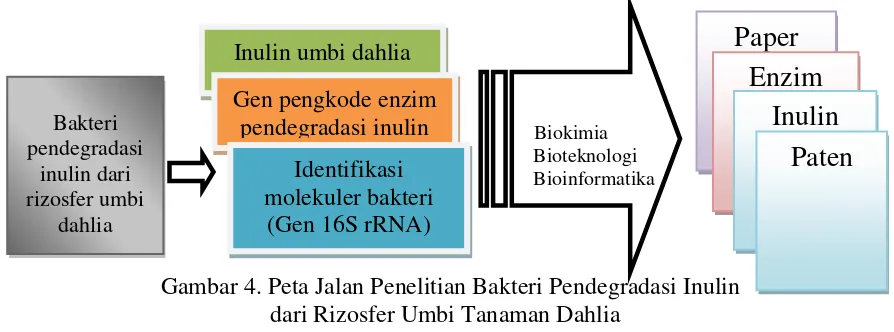
2.5 Peta Jalan Penelitian Bakteri Pendegradasi Inulin
Riset ini memanfaatkan potensi lokal Sumatera Barat yaitu bakteri lokal sebagai
sumber enzim dan gen serta umbi tanaman dahlia lokal sebagai sumber inulin. Inulin
diekstrak dari umbi dahlia yang tumbuh subur di Bukittinggi, sedangkan bakteri berasal
dari rizosfer umbi dahlia yang tumbuh di Padang Panjang. Enzim dan gen pendegradasi
inulin yang berasal dari bakteri sejalan dengan minat riset kelompok bidang studi
biokimia. Peta jalan penelitian bakteri pendegradasi inulin dimuat pada Gambar 4.
9
BAB 3
TUJUAN DAN MANFAAT PENELITIAN
3.1 Tujuan Penelitian
Tujuan utama penelitian adalah mengidentifikasi secara molekuler gen 16S rRNA
isolat bakteri pendegradasi inulin dari rizosfer umbi tanaman dahlia yang tumbuh di
Padang Panjang. Tujuan khusus penelitian adalah
a. Mengisolasi DNA genomik
b. Mengamplifikasi gen 16S rRNA secara in vitro menggunakan teknik PCR
c. Mengsekuens gen 16S rRNA
d. Menganalisis sekuens gen 16S rRNA dengan program BLASTn sehingga dapat
ditentukan kelompok genus dan spesiesnya
3.2 Luaran Penelitian
a. Diperoleh bakteri pendegradasi inulin dari rizosfer umbi tanaman dahlia asal
Padang Panjang yang telah teridentifikasi genus dan spesiesnya.
b. Artikel riset untuk dipublikasi pada jurnal
3.3 Kontribusi Penelitian
Penelitian berkontribusi pada pengembangan ilmu biokimia khususnya pada
10
BAB 4
METODE PENELITIAN
4.1 Jenis Penelitian
Penelitian ini merupakan penelitian eksperimen yang dilakukan di Laboratorium
Biokimia, Laboratorium Mikrobiologi FMIPA UNP serta Laboratorium Biokimia ITB.
4.2 Alat Penelitian
Peralatan yang digunakan pada penelitian ini merupakan peralatan standar pada
biologi molekuler. Peralatan gelas yang digunakan adalah cawan petri sebagai wadah
media padat untuk pembiakan sel bakteri. Erlenmeyer sebagai wadah pembiakan bakteri
pada media cair. Gelas ukur dan labu ukur digunakan untuk pembuatan larutan. Tabung
mikro yang digunakan berukuran 1,5 mL dan 2 mL. Tip pipet mikro dan pipet mikro
untuk pengambilan cairan skala mikro (Socorex, Swiss).
Peralatan lain yang digunakan adalah pH-meter. Autoklaf untuk sterilisasi peralatan
dan media. Kultivasi bakteri secara aerobik menggunakan shaking inkubator. Stok
gliserol mikroorganisme dan DNA disimpan di freezer -20ºC. Mikrosentrifuga untuk
pengendapan bakteri.
4.3 Bahan Penelitian
Bahan yang digunakan meliputi reagen kimia, bakteri, DNA, inulin.
4.3.1. Reagen kimia
Reagen kimia yang digunakan adalah reagen yang lazim dipakai dalam
laboratorium kimia dan biologi molekuler. Semua bahan yang digunakan memiliki
derajat kemurnian pro-analysis (pa) kecuali jika disebutkan lain. Bahan yang digunakan
untuk membuat media pertumbuhan bakteri pendegradasi inulin adalah inulin,
bakto-agar, (NH4)2SO4, KH2PO4, K2HPO4.3H2O,MgSO4.7H2O, trisodium sitrat.
Bahan untuk ekstraksi dan analisis DNA antara lain EDTA, etanol, tris-base,
gliserol, asam asetat glasial, isopropanol, ethidium bromida, agarose, buffer PCR 10x,
dNTPs, ddH2O, MgCl2. Enzim yang digunakan pada penelitian ini meliputi lisozim,
11
4.3.2 DNA
DNA yang digunakan pada penelitian ini adalah DNA kromosom bakteri
pendegradasi inulin sebagai templat untuk amplifikasi gen 16S rRNA dengan metoda
PCR. Oligonukleotida sebagai primer PCR. DNA 1 kb ladder sebagai marker pada
elektroforesis gel agarosa. Primer yang digunakan untuk amplifikasi gen 16S rRNA
adalah BactF1, UniB1 dan 519F.
4.3.3 Bakteri
Bakteri pada penelitian ini adalah isolat bakteri yang berasal dari rizosfer umbi
tanaman dahlia yang tumbuh di Padang Panjang.
4.3.4 Inulin
Inulin diekstraksi dari umbi tanaman dahlia.
4.4 Metode Penelitian
Metode pada penelitian ini secara keseluruhan adalah (1) Peremajaan bakteri
pendegradasi inulin, (2) Identifikasi bakteri pendegradasi inulin secara molekuler
dilakukan dengan langkah sebagai berikut, isolasi DNA genom bakteri, amplifikasi gen
16S rRNA menggunakan metoda PCR, pengecekan amplikon pada gel agarosa,
pemurnian amplikon, sekuensing amplikon dan analisis sekuens gen 16S rRNA
menggunakan program BLASTn.
4.4.1 Peremajaan Bakteri Pendegradasi Inulin dan Pembuatan Stok Gliserol
Bakteri pendegradasi inulin ditumbuhkan pada media padat yang mengandung
inulin sebagai satu-satunya sumber karbon (Castro et al., 1995). Koloni tunggal isolat
ditumbukan pada media cair yang mengandung inulin sebagai satu-satunya sumber
karbon. Sebanyak 850 mL kultur cair ditambahkan 15 mL gliserol steril pada tabung
mikro steril, kemudian dihomogenkan, disimpan pada suhu -20ºC.
4.4.2 Identifikasi Bakteri
Identifikasi bakteri secara molekuler dilakukan dengan langkah sebagai berikut (1).
Isolasi DNA genom bakteri. Isolasi dan pemurnian DNA genom bakteri dilakukan
sesuai prosedur pada Wizard Genomic DNA Purificarion Kit (Promega); (2).
12
rRNA diamplifikasi dengan metoda PCR. Komposisi master mix sebagai berikut: 2,5
µL 10x Dream Taq bufer, 2,5 µL dNTP mix 2mM, 1 µL MgCl2 2,5mM, 0,5 µL primer
BactF1 20 µM, 0,5 µL primer UniB1 20 µM, sampel 0,4 µL (150ng/µL), 0,125 µL
Dream Taq polymerase, ddH2O ditambahkan sampai volume 25 µL. Proses PCR
dilakukan pada denaturasi awal 94C selama 2 menit, denaturasi 94C selama 0,3 menit,
annealing 48C selama 30 detik, elongasi 72C selama 1,5 menit, elongasi akhir 72C
selama 5 menit. Siklus PCR dilakukan 29 kali. Amplikon dielektroforesis pada gel
agarosa. (3). Pengecekan amplikon pada gel agarosa. Amplikon dielektroforesis
menggunakan gel agarosa 0,8%. Agarosa dilarutkan dengan bufer TAE 1x dan
dipanaskan sampai mendidih. Setelah larutan agarosa agak dingin (40C) ditambahkan
EtBr, dituang dalam tray dan dipasang sisir. Setelah gel membeku tray berisi gel
diletakkan pada bejana elektroforesis yang telah berisi bufer TAE 1x, kemudian sisir
dicabut dengan pelan. Sampel DNA dicampur dengan loading buffer bromphenol blue
(Fermentas) dan dimasukkan pada sumur gel. Sebagai penanda ukuran dan penentuan
perkiraan konsentrasi DNA digunakan DNA marker 1 kb ladder (Fermentas).
Elektroforesis dilakukan pada tegangan 80 volt selama 35 menit. Hasil elektroforesis
diamati di bawah sinar UV.; (4). Pemurnian amplikon PCR. Amplikon dimurnikan
sesuai prosedur pada Gel/PCR DNA Fragments Extraction Kit (Geneaid).
(5).Sekuensing. Amplikon gen 16S rRNA murni 25 ng/µ L, primer BactF1 dan UniB1
masing-masing dengan konsentrasi 10 µM dikirim ke Macrogen di Korea. Amplikon
gen 16S rRNA disekuensing menggunakan primer BactF1, UniB1, dan 519F.
4.4.3 Bioinformatika
13
BAB 5
HASIL DAN PEMBAHASAN
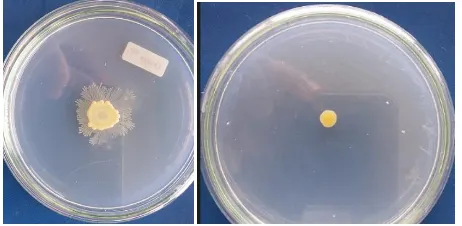
5.1. Peremajaan Isolat Bakteri Pendegradasi Inulin
Isolat bakteri pendegradasi inulin yang ditemukan pada rizosfer umbi dahlia ada
dua yaitu berwarna coklat dan putih. Isolat ini ditumbuhkan pada media padat yang
mengandung inulin sebagai satu-satunya sumber karbon (Gambar 5). Media ini
merupakan media selektif untuk menskrining bakteri pendegradasi inulin.
Gambar 5. Isolat Bakteri Pendegradasi Inulin putih (kiri), coklat (kanan)
5.2 Isolasi DNA Genom Bakteri dan Amplifikasi Gen 16S rRNA
Isolat bakteri pendegradasi inulin dari rizosfer umbi dahlia yang tumbuh di Padang
Panjang dua buah yaitu isolat warna koloni putih dengan kode isolat RZ-01 dan isolat
warna koloni agak kecoklatan dengan kode isolat RZ-02. Identifikasi bakteri isolat RZ-01
telah dilakukan secara molekuler. Identifikasi molekuler gen 16S rRNA bakteri isolat
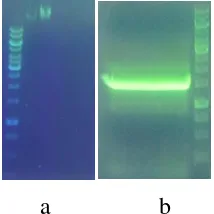
RZ-01 diawali dengan mengisolasi DNA genom. DNA genom bakteri hasil isolasi dimuat
pada Gambar (6a). DNA genom ini dijadikan template untuk mengamplifikasi gen 16S
rRNA menggunakan metoda PCR. Gen 16S rRNA dari isolat RZ-01 telah berhasil
diamplifikasi secara PCR. Amplikon sejajar dengan marker DNA 1 kb ladder ukuran
1500 bp (Gambar 6b). Ukuran amplikon sekitar 1500 bp diperoleh karena posisi primer
BacF1 adalah basa 8 sampai 27, sedangkan posisi primer UniB1 adalah basa
ke-1510 sampai ke-1492 posisi pada gen 16S rRNA E.coli (Baker et al., 2003). ). Dengan
demikian, ukuran amplikon yang diperkirakan 1500 bp tepatnya adalah 1485 bp pada
pada gen 16S rRNA E.coli. Konsentrasi amplikon diperkirakan 100 ng/µL. Gen 16S
rRNA terdapat dalam multi kopi dalam kromosom bakteri ini. Gen 16S rRNA di dalam
kromosom bakteri terdapat 1 sampai 15 kopi. Sekitar 62% bakteri mempunyai lebih dari
14
a b
Gambar 6. a. Elektroforesis DNA genomik bakteri dari isolat RZ-01 (lajur 1, marker DNA 1 kb, lajur 2 dan 3, DNA genomik) b. Fragmen Gen 16S rRNA Hasil PCR.
(lajur 1, amplikon, lajur 2, marker DNA 1 kb)
5.3 Sekuens Gen 16S rRNA Bakteri Isolat RZ-01
Amplikon murni fragmen gen 16S rRNA dari isolat bakteri RZ-01 digunakan
sebagai template reaksi sekuensing dengan primer BactF1, UniB1 dan 519F. Hasil
sekuensing parsial gen 16S rRNA isolat bakteri RZ-01 menggunakan primer BactF1
adalah 800 bp (Gambar 7), sedangkan hasil sekuensing menggunakan primer 519F adalah
410 bp (Gambar 8). Hasil sekuensing parsial gen 16S rRNA isolat bakteri RZ-01
menggunakan primer UniB1 adalah 920 bp (Gambar 9). Fragmen gen 16S rRNA isolat
bakteri RZ-01 yang berhasil ditemukan adalah 1337 pb (Gambar 10).
TGCAGTCGAG CGGTAGCACA GAGAGCTTGC TCTCGGGTGA CGAGCGGCGG 50
ACGGGTGAGT AATGTCTGGG AAACTGCCTG ATGGAGGGGG ATAACTACTG 100
GAAACGGTAG CTAATACCGC ATAACGTCGC AAGACCAAAG TGGGGGACCT 150
TCGGGCCTCA TGCCATCAGA TGTGCCCAGA TGGGATTAGC TGGTAGGTGG 200
GGTAACGGCT CACCTAGGCG ACGATCCCTA GCTGGTCTGA GAGGATGACC 250
AGCCACACTG GAACTGAGAC ACGGTCCAGA CTCCTACGGG AGGCAGCAGT 300
GGGGAATATT GCACAATGGG CGCAAGCCTG ATGCAGCCAT GCCGCGTGTG 350
TGAAGAAGGC CTTCGGGTTG TAAAGCACTT TCAGCGAGGA GGAAGGCGTT 400
AAGGTTAATA ACCTTGTCGA TTGACGTTAC TCGCAGAAGA AGCACCGGCT 450
AACTCCGTGC CAGCAGCCGC GGTAATACGG AGGGTGCAAG CGTTAATCGG 500
AATTACTGGG CGTAAAGCGC ACGCAGGCGG TCTGTCAAGT CGGATGTGAA 550
ATCCCCGGGC TCAACCTGGG AACTGCATTC GAAACTGGCA GGCTAGAGTC 600
TTGTAGAGGG GGGTAGAATT CCAGGTGTAG CGGTGAAATG CGTAGAGATC 650
TGGAGGAATA CCGGTGGCGA AGGCGGCCCC CTGGACAAAG ACTGACGCTC 700
AGGTGCGAAA GCGTGGGGAG CAAACAGGAT TAGATACCCT GGTAGTCCAC 750
GCTGTAAACG ATGTCGACTT GGAGGTTGTG CCCTTGAGGC GTGGCTTCCG 800
15
ACAACCCGAA GGCCTTCTTC ACACACGCGG CATGGCTGCA TCAGGCTTGC 50
GCCCATTGTG CAATATTCCC CACTGCTGCC TCCCGTAGGA GTCTGGACCG 100
TGTCTCAGTT CCAGTGTGGC TGGTCATCCT CTCAGACCAG CTAGGGATCG 150
TCGCCTAGGT GAGCCGTTAC CCCACCTACC AGCTAATCCC ATCTGGGCAC 200
ATCTGATGGC ATGAGGCCCG AAGGTCCCCC ACTTTGGTCT TGCGACGTTA 250
TGCGGTATTA GCTACCGTTT CCAGTAGTTA TCCCCCTCCA TCAGGCAGTT 300
TCCCAGACAT TACTCACCCG TCCGCCGCTC GTCACCCGAG AGCAAGCTCT 350
CTGTGCTACC GCTCGACTTG CATGTGTTAG GCCTGCCGCC AGCGTTCAAT 400
CTGAGCCTGA
Gambar 8. Hasil sekuensing parsial gen 16S rRNA isolat RZ-01 menggunakan primer 519F
CGATTGACGT TACTCGCAGA AGAAGCACCG GCTAACTCCG TGCCAGCAGC 50
CGCGGTAATA CGGAGGGTGC AAGCGTTAAT CGGAATTACT GGGCGTAAAG 100
CGCACGCAGG CGGTCTGTCA AGTCGGATGT GAAATCCCCG GGCTCAACCT 150
16
Hasil sekuensing parsial gen 16S rRNA menggunakan primer BactF1
disambungkan dengan hasil sekuensing parsial gen 16S rRNA primer UniB1. Kedua
sekuens tersebut tumpang tindih sebanyak 383 bp. Sekuans gen 16S rRNA hasil
gabungan dimuat pada Gambar l0. Dengan demikian, urutan basa nukleotida fragmen
gen 16S rRNA isolat bakteri RZ-01 yang berhasil ditemukan adalah 1337 pb.
TGCAGTCGAG CGGTAGCACA GAGAGCTTGC TCTCGGGTGA CGAGCGGCGG 50
GAGCTAACGC GTTAAATCGA CCGCCTGGGG AGTACGGCCG CAAGGTTAAA 850
ACTCAAATGA ATTGACGGGG GCCCGCACAA GCGGTGGAGC ATGTGGTTTA 900
17
5.4. Identifikasi Molekuler Isolat RZ-01 Bakteri Pendegradasi Inulin
Identifikasi bakteri secara molekuler dilakukan dengan membandingkan urutan
basa nukleotida parsial gen 16S rRNA dari isolat RZ-01 dengan urutan basa nukleotida
gen 16S rRNA dari berbagai bakteri yang dimuat pada basis data GenBank menggunakan
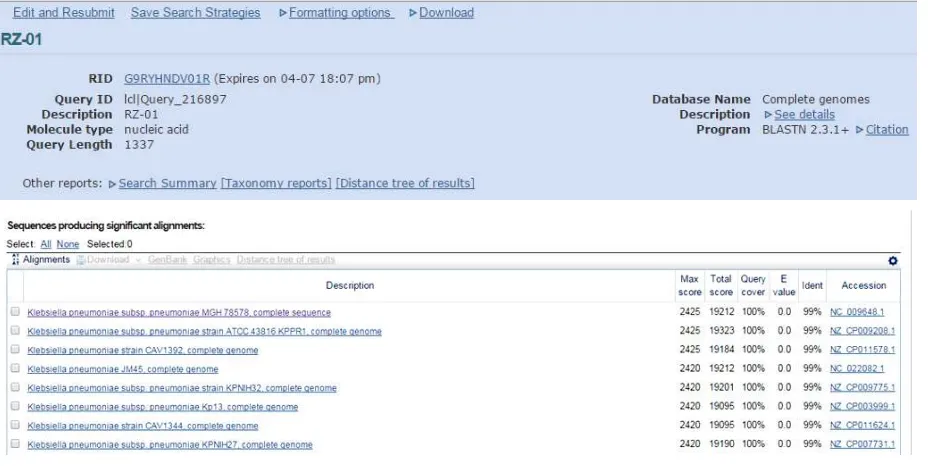
program BLASTn (http://www.ncbi.nlm.nih.gov pada 06 April 2016). Hasil BLASTn
dimuat pada Gambar 11. Fragmen gen 16S rRNA isolat RZ-01 mempunyai kemiripan
yang tinggi dengan 100 buah gen 16S rRNA bakteri pada basis data GeneBank.
Kebanyakan bakteri tersebut adalah Klebsiella. Dengan demikian dapat disimpulkan
bahwa isolat RZ-01 termasuk kelompok genus Klebsiella dengan spesies Klebsiella
pneumoniae. Klebsiella pneumoniae merupakan bakteri gram negatif. Metoda
nonstaining menggunakan larutan KOH 3% pada isolat RZ-01 juga menunjukkan bakteri
gram negatif. Elektroferogram parsial gen 16S RNA isolat RZ-01 menggunakan primer
BactF1 dimuat pada Lampiran 2.
18
BAB 6
KESIMPULAN DAN SARAN
6.1 Kesimpulan
Isolat RZ-01 adalah salah satu isolat bakteri pendegradasi inulin yang telah
ditemukan dari rizosfer umbi tanaman dahlia yang tumbuh di Padang Panjang.
Berdasarkan identifikasi secara molekuler melalui sekuens parsial gen 16S rRNA, isolat
RZ-01 memiliki kekerabatan terdekat dengan kelompok genus Klebsiella dan spesies
Klebsiella pneumoniae.
6.2 Saran
Identifikasi isolat RZ-01 berdasarkan parsial gen 16S rRNA adalah sebagai
kelompok genus Klebsiella dan spesies Klebsiella pneumoniae. Sekuens genom
Klebsiella pneumoniae tersedia pada basis data GenBank. Alur penelitian selanjutnya
yang dapat dilakukan adalah melacak gen penderadasi inulin pada isolat RZ-01. Selain itu
diperlukan uji biokimia isolat RZ-01.
19
DAFTAR PUSTAKA
Allais, JJ; Kammoun,S; Blanc, P; Girard, C; Baratti, JC. (1986). Isolation and Characteriztion of Bacterial Strain with Inulinase Activity. Applied and Enviromental Microbiology. Vol.52.No5. 1086-1090.
Amann, RI; Ludwig,W; Schleifer, KH (1995). Phylogenetic identification and in situ detection of individual microbial cells without cultivation. Microbiology Reviews, 59:143-169.
Armougom, F; Raoult, D (2009). Exploring microbial diversity using 16S rRNA high-throughput methods. JCSB, 2:074-092’
Azhar, M; Oktavia, B (2014). Eksplorasi bakteri pendegradasi inulin yang potensial untuk pembuatan fruktosa dari sumber air panas di Solok dan rizosfer umbi dahlia. Laporan Penelitian. Universitas Negeri Padang.
Azhar, M; Syukur, S; Natalia, D; Vovien; Jamsari; Munaf, E (2013) Characterization of extracellular enzyme and identification of inulin degrading bacteria from hot spring West Sumatra. International Journal of Chemistry
Azhar, M; Syukur, S; Natalia, D; Vovien; Jamsari (2011). Skrining dan identifikasi bakteri pendegradasi inulin dari sumber air panas Padang Balimbiang di Solok.
Jurnal Riset Kimia. vol.5.no.1: 32-39.
Baker, GC; Smith, JJ; Cowan, DA (2003) Review and re-analysis of domain-specific 16S primers. Journal of Microbiological Methods, 55:541-555
Case RJ, Boucher Y, Dahllof I, Holmstrom C, Doolittle WF, Kjelleberg S (2007). Use 16S rRNA and rpoB genes as molecular markers for microbial ecology studies.
Applied and Environmental Microbiology, 73:278-288.
Castro GR, Baigori MD, Sineriz, F (1995) A plate technique for screening of inulin degrading microorganisms. Journal of Microbiological Methods, 22:51-56.
Franck, A; Leenheer, LD (2003). “Inulin”. Email: [email protected]. Diakses 25 Maret 2004
Gao, J; Xu, YY; Yang, HM; Xu, H; Xue, F; Li, S; Feng, XH (2014). Gene Cloning, Expression, and Characterization of an Exo-inulinase from Paenbaccillus polymyxa
ZJ-9. App. Biochem Biotechnol.
Kwon HJ, Jeon SJ, You DJ, Kim KH, Jeong YK, Kim YH, Kim YM, Kim BW (2003). Cloning and Characterization of exoinulinase from Bacillus polymyxa.
Biotechnology Letter 25:155-159.
Kulminskaya,AA; Arand,M; Eneyskaya, EV; Ivanen, DR; Shabalin, KA; Shishlyannikov, SM; Saveliev, AN; Korneeva, OS; Neustroev, KN.(2003). Biochemical Characterization of Aspergillus awamori Exoinulinase: Substrate Binding Characteristics and Regio Selectivity of Hydrolysis. Biochimica et Biophysica Acta
1650.22-29.
Li, AX; Guo, LZ; Fu, Q; Lu, WD (2013). A simple and rapid plate assay for screening of inulin-degrading microorganisms using Lugo’s iodine solution. African Journal of Biotechnology. Vol.10(46); 9518-9521.
Lu, WD; Li, AX; Guo, QL (2013). Production of novel alkalitolerant and thermostable inulinase from marine actinomycete Nocardiopsis sp DN-K15 and inulin hydrolysis by the enzyme.
Madigan, MT; Martinko, JM (2006). Brock biology of microorganism. Pearson Education Inc.USA.
20
Phelps,CF (1965). The Physical Properties of Inulin Solution. Biochem.J 95:41-47. Sirisansaneeyakul, S; Worawuthiyanan, N; Vanichsriratana, W; Srinophakum,P; Chisti,
Y (2007). Production of Fructose from Inulin Mixed Inulinases from Aspergllus niger and Candida guilliermondii. Word J Microbial Biotechnol 23:543-552.
Stackebrandt, E; Goebel, BM (1994). Taxonomic note: A place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present spesies definition in bacteriology. International Journal of Systematic Bacteriology, 44:846-849.
Tsujimoto,Y; Watanabe, A; Nakano,K; Watanabe,K; Matsui,H; Tsuji,K; Tsukihara,T; Suzuki, Y. (2003). Gene Cloning, Expression, and Crystallization of a Thermostable Exo-inulinase from Geobacillus stearothermophilus KP1289. Appl Microbiol Biotechnol 62:180-185.
Tohamy, EY (2006). Purification and characterization of exoinulinase enzyme from
Sterptomyces grisenus. Pakistan Journal of Biological Sciences, 9:911-916.
21
Lampiran 1. Daftar Riwayat Hidup Peneliti
A. Identitas Diri
1 Nama Lengkap dengan Gelar Dr. Minda Azhar, M.Si
2 Jenis Kelamin Perempuan
3 Jabatan Fungsional / Gol Lektor kepala/ IV-c 4 NIP. 196411241991122001 5 NIDN 00-2411-6406
6 Tempat dan Tanggal Lahir Bukittinggi, 24 November 1964
7 E-mail [email protected] ; [email protected] 8 No HP 081267225154
9 Alamat Kantor Jurusan Kimia FMIPAUniversitas Negeri Padang Jl.Prof.Dr.Hamka Air Tawar Padang
10 No. Telepon 0751 7057420
11 Lulusan yang telah dihasilkan S1= +40 , S2= 0, S3=0 12 Matakuliah yang diampu 1. Biokimia (S1 dan S2)
2. Bioteknologi (S1) 3. Praktikum Biokimia (S1) 4. Kapita Selekta Biokimia (S1)
5. Seminar Literatur Kimia (S1) dan Kimia Dasar (S1)
B. Riwayat Pendidikan
S1 S2 S3
1.Nama PT IKIP Padang ITB Bandung Universitas Andalas 2.Bidang Ilmu Pendidikan Kimia Biokimia/Bioteknologi Biokimia/Bioteknologi 3.Tahun Masuk 1984 (Pra S2 :1992-1993)
1993
2008
4.Tahun lulus September 1990 Februari 1996 01 Februari 2013 5.Judul
C. Pengalaman Penelitian (3 terakhir)
No Tahun Judul penelitian Pendanaan
Sumber Jumlah (Rp) 1 2014 Eksplorasi bakteri pendegradasi inulin yang potensial
untuk pembuatan fruktosa dari sumber air panas di Solok dan rizosfer umbi dahlia (Ketua).
Dikti 50 juta
2 2011 Penentuan kadar RBB pada dye-inulin secara HPLC melalui pembentukan senyawa dye-inulin (Anggota)
DIPA UNP
7,5 juta
3 2010 Kloning dan sekuensing fragmen gen exo-inulinase bakteri termotoleran isolat Sumatera Barat (Mandiri)
22 D. Pengalaman Pengabdian kepada Masyarakat (2 terakhir)
No Tahun Judul pengabdian kepada mayarakat Pendanaan
Sumber Jumlah (Rp) 1 2015 Pelatihan pengembangan media pembelajaran berbasis
ICT dengan pola penyajian penalaran induktif bagi guru-guru kimia di SMA Payakumbuh (Ketua)
DIPA UNP
15 juta
2 2014 Penerapan bioteknologi sederhana pada pengolahan buah kelapa menjadi VCO dan nata de Coco bagi masyarakat Kelurahan Bulakan Balai Kandi Kecamatan Payakumbuh Barat (Ketua)
DIPA UNP
10 juta
E.Pengalaman Penulisan Artikel Ilmiah dalam Jurnal Nasioanal dan Internasional (3 tahun terakhir)
No Judul Artikel Ilmiah Vol./ No./Th
Nama jurnal
1 The gene fragments that encodes inulin hydrolysis enzyme from geno-mic Bacillus licheniformis: isolation by PCR technique using new primers (Minda Azhar, Sumaryati Syukur, Dessy Natalia, Vovien, Jamsari)
2015 accepted
International Journal of Biological Chemistry
2 Perkiraan massa molekul enzim pendegradasi inulin dari bakteri termofilik Bacillus licheniformis dan aktivitas enzim pada granula inulin (Minda Azhar, Sumaryati Syukur, Dessy Natalia, Vovien, Jamsari)
F. Pemakalah / Penyajian Poster (2 tahun terakhir)
No Nama Pertemuan Ilmiah/Seminar
Judul Artikel Ilmiah/Poster Waktu dan tempat
1 Seminar Nasional
Himpunan Kimia Indonesia cabang SUMBAR
Identifikasi gen 16S rRNA bakteri termofilik yang memperlihatkan aktivitas enzim penghidrolisis inulin tipe exo- dari sumber air panas Rimbo Panti (Minda Azhar,
Sumaryati syukur, Dessy Natalia, Mardaleni Fitri, VovienVionica, Jamsari)
2013 Universitas Negeri Padang
G. Pengalaman Penulisan Buku (2 tahun terakhir)
1 2015 Biomolekul sel : Karbohidrat, Protein dan Enzim (Minda Azhar) (dalam proses cetak) ISBN 978-602-1178-12-6
Penerbit UNP Press
H. Penghargaan yang pernah diraih (10 tahun terakhir)
No Jenis Penghargaan Institusi Pemberi Penghargaan Tahun 1 Beasiswa BPPS Dikti 2008-2012 2 Dosen Berprestasi FMIPA UNP 2008
Semua data yang saya isikan dan tercantum dalam biodata ini adalah benar dan dapat dipertanggungjawabkan secara hukum. Apabila dikemudian hari ternyata dijumpai ketidak-sesuaian dengan kenyataan, saya sanggup menerima resikonya. Demikian biodata ini saya buat dengan sebenarnya.
Padang, Desember 2015 Peneliti,