IDENTIFIKASI MORFOLOGI DAN MOLEKULER FUNGI (ISOLAT M)
YANG DAPAT MENDEGRADASI PEWARNA SINTETIK
SKRIPSI
Disusun untuk Memenuhi Sebagian dari Syarat Memperoleh Gelar Sarjana
Sains Program Studi Biologi Jurusan Pendidikan Biologi
Oleh:
HANA GARDENIA MAHBUBAH
0902088
PROGRAM STUDI BIOLOGI
JURUSAN PENDIDIKAN BIOLOGI
FAKULTAS PENDIDIKAN MATEMATIKA DAN ILMU PENGETAHUAN ALAM
UNIVERSITAS PENDIDIKAN INDONESIA
IDENTIFIKASI MORFOLOGI DAN MOLEKULER FUNGI (ISOLAT M)
YANG DAPAT MENDEGRADASI PEWARNA SINTETIK
Oleh:
Hana Gardenia Mahbubah
0902088
Diajukan untuk Memenuhi Sebagian dari Syarat
Memperoleh Gelar Sarjana Sains Program Studi Biologi
© Hana Gardenia Mahbubah 2013
Universitas Pendidikan Indonesia
Oktober 2013
Hak Cipta dilindungi undang-undang.
Skripsi ini tidak boleh diperbanyak seluruhya atau sebagian,
LEMBAR PENGESAHAN
IDENTIFIKASI MORFOLOGI DAN MOLEKULER FUNGI (ISOLAT M) YANG
DAPAT MENDEGRADASI PEWARNA SINTETIK
Oleh
Hana Gardenia Mahbubah
0902088
DISETUJUI DAN DISAHKAN OLEH:
Pembimbing I
Dr. Topik Hidayat, M.Si
NIP. 1970044101997021001
Pembimbing II
Kusnadi, S.Pd., M.Si
NIP. 196805091994031001
Mengetahui,
Ketua Jurusan Pendidikan Biologi FPMIPA UPI
Dr. H. Riandi, M.Si
IDENTIFIKASI MORFOLOGI DAN MOLEKULER FUNGI (ISOLAT M) YANG DAPAT MENDEGRADASI PEWARNA SINTETIK
ABSTRAK
Pewarna sintetik saat ini telah banyak digunakan dalam proses pewarnaan di berbagai industri. Limbah industri yang mengandung pewarna sintetik sering memberikan masalah bagi ekosistem perairan. Berdasarkan penelitian yang telah dilakukan sebelumnya, ditemukan lima isolat fungi yang diketahui dapat mendegradasi pewarna sintetik. Penelitian ini bertujuan untuk mengidentifikasi fungi (isolat M) secara morfologi dan molekuler yang dapat mendegradasi pewarna sintetik. Karakteristik morfologi yang diamati meliputi warna dan bentuk koloni fungi, sedangkan identifikasi hifa dan bentuk spora dilakukan bedasarkan pengamatan pada slide culture menggunakan mikroskop. Berdasarkan identifikasi morfologi, empat isolat yang diidentifikasi termasuk ke dalam genus Trichoderma yaitu isolat M04, M06, M10, dan M17, sedangkan satu isolat dengan kode isolat M15 termasuk ke dalam genus Absidia. Identifikasi molekuler dilakukan menggunakan sikuen gen 18S rRNA dengan menggunakan teknik PCR, dari hasil amplifikasi menghasilkan fragmen DNA berukuran sekitar 1800 pb. Selanjutnya dibuat pohon filogenetik untuk mengetahui hubungan kekerabatan lima isolat fungi M. Berdasarkan pohon filogenetik yang terbentuk, isolat M04, M06, M10, dan M17 memiliki hubungan kekerabatan terdekat dengan Trichoderma harzianum, di dukung dengan nilai bootstrap sebesar 100%, sedangkan isolat sampel M15 memiliki hubungan kekerabatan terdekat dengan Absidia spinosa di dukung dengan nilai bootstrap 100%.
MORPHOLOGICAL AND MOLECULAR IDENTIFICATION OF FUNGI
(M ISOLATE ) THAT CAPABLE TO DEGRADE SYNTHETIC DYE
ABSTRACT
Large number of chemicals, including dyes are used in textile processing. Recently, environmental contamination has been pointed as one of the greatest problem of modern society. Textile industry produce a large amount of effluent which can cause serious environmental problems. There is five isolate of fungi that known to be able to degrade synthetic dye. This research purpose is to identify fungi (M isolate) according to their morphology and molecular characteristic. Morphological characteristic that is being observed include color and colony form of fungi, whereas hyphae identification and form of spora is observed based on slide culture identification using microscope. Based on morphology identification, four isolates are Trichoderma, that is M04, M06, M10, and M17, whereas one other isolate is Absidia (M15). Molecular identification is done using 18S rRNA gene amplification. The amplification result is DNA fragmen which size is 1800 bp. Furthermore phylogenetic tree were construct to know genetic relationship between five isolate of M fungi. Based on phylogenetic tree, isolates M04, M06, M10, and M17 have a close relationship with Trichoderma harzianum which a bootstrap value is 100%, whereas isolate of M15 as a close phylogenetic relationship with Absidia spinosa which a bootstrap value is 100%.
DAFTAR ISI
HALAMAN
ABSTRAK ... i
KATA PENGANTAR ... ii
DAFTAR ISI ... iv
DAFTAR TABEL ... vii
DAFTAR GAMBAR ... viii
DAFTAR LAMPIRAN ... ix
BAB I PENDAHULUAN A. Latar Belakang Masalah ... 1
B. Rumusan Masalah ... 4
C. Batasan Masalah ... 4
D. Tujuan ... 4
E. Manfaat Penelitian ... 4
BAB II FUNGI, PEWARNA SINTETIK, IDENTIFIKASI MORFOLOGI DAN MOLEKULER A.Fungi ... 6
B.Pewarna Sintetik ... 10
C.Metode Degradasi dan Dekolorisasi Pewarna Sintetik………... 12
D.Degradasi dan Dekolorisasi Pewarna Sintetik oleh Fungi ... 13
E. Identifikasi Fungi Secara Morfologi dan Molekuler ... 16
F. PCR (Polymerase Chain Reaction) ... 18
G.Elektroforesis DNA ... 20
I. Analisis Filogenetik Molekuler ... 23
BAB III METODE PENELITIAN A.Jenis Penelitian ... 26
B.Populasi dan Sampel ... 26
C.Waktu dan Lokasi Penelitian ... 26
D.Alat dan Bahan Penelitian ... 26
E. Prosedur Penelitian ... 30
1. Persiapan Penelitian ... 30
a. Pembuatan medium ... 30
b. Kultivasi Fungi M ... 30
2. Tahap Penelitian ... 30
1) Identifikasi Morfologi Fungi M ... 30
a. Pembuatan Slide Culture ... 30
b. Identifikasi Morfologi Fungi M ... 31
2) Identifikasi Molekuler Fungi M ... 31
a. Ekstraksi DNA Fungi ... 31
b. Elektroforesis Gel Agarose ... 32
c. Polymerase Chain Reaction(PCR) ... 33
d. Elektroforesis Gel Agarose ... 35
e. Sikuensing DNA... 35
f. Analisis Data Bioinformatika ... 35
3. Alur Penelitian ... 36
BAB IV HASIL DAN PEMBAHASAN A.Identifikasi Morfologi Fungi Isolat M ... 37
1. Karakteristik Morfologi Fungi secara Makroskopis ... 37
2. Karakteristik Morfologi Fungi Secara Mikroskopis ... 38
B. Identifikasi Molekuler Fungi Isolat M ... 46
2. Amplifikasi Gen 18s rRNA ... 50
3. Amplifikasi Bioinformatika Sikuen Gen 18s rRNA ... 52
BAB V SIMPULAN DAN SARAN A. Simpulan ... 57
B. Saran ... 57
DAFTAR PUSTAKA ... 58
LAMPIRAN ... 66
DAFTAR TABEL
Tabel HALAMAN
2.1 Golongan Pewarna Sintetik ... 10
2.2 Metode Degradasi dan Dekolorisasi Pewarna Sintetik ... 12
2.3 Mekanisme Degradasi dan Dekolorisasi Pewarna ... 16
2.4 Karakteristik Gel Agarosa ... 21
3.1 Alat-alat Penelitian ... 27
3.2 Bahan-bahan Penelitian ... 29
3.3 Komponen Reaksi PCR ... 34
3.4 Tahapan Reaksi PCR... 34
4.1 Pengamatan Morfologi Fungi Makroskopis... 37
4.2 Pengamatan Morfologi Fungi Mikroskopis ... 38
DAFTAR GAMBAR
Gambar HALAMAN
2.1 Jenis Spora Aseksual Fungi ... 8
2.2 Jenis Spora Seksual Fungi ... 9
2.3 Beberapa Struktur Kimia Pewarna Sintetik ... 11
2.4 Tahapan Pembuatan Slide Culture ... 17
2.5 Tahapan Proses PCR ... 19
2.6 Proses Elektroforesis ... 21
2.7 Struktur gen penanda 18S rRNA... 23
2.8 Pohon kekerabatan dan polarisasi karakter analisis filogenetika ... 24
3.1 Profile PCR Gen 18S rRNA ... 35
3.2 Alur Penelitian ... 36
4.1 (a) Koloni isolat M04 dalam medium PDA, (b) reverse of colony isolat M04 dalam medium PDA, (c) morfologi mikroskopis isolat M04 ... 39
4.2 (a) Koloni isolat M06 dalam medium PDA, (b) reverse of colony isolat M06 dalam medium PDA, (c) morfologi mikroskopis isolat M06 ... 40
4.3 (a) Koloni isolat M10 dalam medium PDA, (b) reverse of colony isolat M10 dalam medium PDA, (c) morfologi mikroskopis isolat M10 ... 41
4.4 (a) Koloni isolat M15 dalam medium PDA, (b) reverse of colony isolat M15 dalam medium PDA, (c) morfologi mikroskopis isolat M15 ... 42
4.5 (a) Koloni isolat M17 dalam medium PDA, (b) reverse of colony isolat M17 dalam medium PDA, (c) morfologi mikroskopis isolat M17 ... 43
4.6 Foto gel hasil elektroforesis isolasi DNA fungi isolat M ... 48
4.7 Foto gel hasil elektroforesis amplikon gen 18S rRNA ... 52
DAFTAR LAMPIRAN
Lampiran HALAMAN
1 Protokol Pembuatan Reagen dan Medium yang digunakan
1 BAB I
PENDAHULUAN
A. Latar Belakang
Limbah-limbah industri menjadi semakin bertambah seiring dengan pesatnya perkembangan industri, baik volume maupun jenisnya. Limbah industri khususnya limbah industri tekstil, kertas, kosmetik, makanan, obat-obatan, dan lain-lain, merupakan salah satu penyebab masalah lingkungan akibat dari buangan limbah tersebut yang mencemari lingkungan. Akibatnya beban pencemaran lingkungan semakin berat, sedangkan kemampuan alam untuk menerima beban limbah terbatas. Jenis limbah industri banyak macamnya, tergantung bahan baku dan proses yang digunakan masing-masing industri. Salah satu masalah yang paling mengganggu dari limbah industri tersebut adalah kandungan zat warna (Pratiwi, 2010).
Industri tekstil merupakan salah satu jenis industri di Indonesia. Industri tekstil menghasilkan limbah cair berwarna, zat warna dalam industri tekstil merupakan salah satu bahan baku utama, sekitar 10-15% dari zat warna yang sudah digunakan tidak dapat dipakai ulang dan harus dibuang (Selvam et al., 2003). Zat warna yang dikandung limbah industri tekstil dapat mengganggu kesehatan, misalnya iritasi kulit dan iritasi mata hingga menyebabkan kanker. Selain itu, zat warna juga dapat menyebabkan terjadinya mutagen (Mathur, 2005). Zat warna juga dapat memberikan efek terhadap organisme akuatik akibat berkurangnya intensitas cahaya matahari dan dapat bersifat toksik bagi fauna dan flora karena mengandung senyawa aromatik, logam, khlorida, dll. (Dhaneshvar et al., 2007).
2
banyak digunakan karena mudah ditemukan dan memiliki variasi warna yang lebih banyak dibandingkan dengan pewarna alami (Pandey et al., 2007). Zat warna azo disintesis untuk tidak mudah rusak oleh perlakuan kimia maupun perlakuan fotolitik. Untuk itu, bila dibuang ke perairan akan mengganggu estetika dan meracuni biota air di dalam badan air tersebut. Hal ini dikarenakan berkurangnya oksigen yang dihasilkan selama proses fotosintesis akibat terhalangnya sinar matahari untuk masuk ke dalam badan air akibat keberadaan limbah zat warna. Selain itu perombakan zat warna azo secara aerobik pada dasar perairan menghasilkan senyawa amina aromatik yang kemungkinan lebih toksik dibandingkan dengan zat warna azo itu sendiri (Van der Zee, 2002).
3
bakteri dan fungi. Kelemahan perombakan menggunakan bakteri adalah bekerja pada substrat yang spesifik sehingga aktivitasnya pada spektrum yang spesifik (Van der Zee, 2002).
Penggunaan fungi semakin intensif setelah ditemukannya beberapa jenis fungi seperti Coriolus versicolor (Zahida et al., 2007), dan Trametes versicolor (Adosinda et al., 2003). Fungi tersebut sangat efektif digunakan merombak senyawa xenobiotik termasuk zat warna azo. Kemampuan fungi merombak zat warna tekstil disebabkan enzim lignolitik ekstraseluler seperti mangan peroksidase (MNP), lignin peroksidase (LIP), dan laccase (Boran & Yesilada,
2011). Potensi strategis penggunaan enzim lignolitik ini adalah proses perombakannya sampai pada mineralisasi menghasilkan zat tidak toksik, bersifat nonspesifik sehingga aktivitasnya pada spektrum luas (Katia et al., 2005). Berdasarkan penelitian oleh Singh & Singh (2010) diketahui bahwa beberapa jenis fungi seperti Trichoderma harzianum, Phanerocheate chrysosporium, Aspergillus flavus, Gliocladium virens dan Trametes versicolor
dapat mendegradasi pewarna sintetik, khususnya pewarna dari golongan azo.
Berdasarkan penelitian oleh Hadibarata (2011) ditemukan beberapa isolat fungi yang ditumbuhkan pada limbah pewarna sintetik dan dapat tumbuh dengan optimum dengan kondisi lingkungan tersebut, dan fungi tersebut diketahui dapat mendegradasi pewarna sintetik golongan azo diantaranya yaitu Anilin yellow, Methyl red, Methyl orange, Bismark brown, dan congo red.
4
karena organisme yang berkerabat jauh dapat memiliki morfologi yang serupa. Penggunaan data molekuler digunakan sebagai penunjang data morfologi dan diharapkan dapat menjadi dasar yang lebih kuat dalam penentuan klasifikasi dan identifikasi fungi (Simpson, 2011).
B. Rumusan Masalah
Berdasarkan latar belakang yang telah diuraikan di atas diperoleh rumusan masalah penelitian, yaitu bagaimanakah karakteristik morfologi dan molekuler fungi (isolat M) yang dapat mendegradasi pewarna sintetik ? serta bagaimanakah hubungan kekerabatan antara beberapa fungi (isolat M) ?
C. Batasan Masalah
1. Isolat fungi yang diamati sebanyak lima isolat fungi yaitu isolat M04, M06, M10, M15 dan M17
2. Isolat fungi yang digunakan berasal dari hutan Samarinda, Kalimantan Timur
3. Primer yang digunakan untuk amplifikasi gen 18S rRNA adalah primer forward NSI dan primer reverse NS8 (Marchesi et al., 1998).
D. Tujuan Penelitian
Tujuan dilaksanakannya penelitian ini adalah untuk mengidentifikasi fungi (isolat M) yang dapat mendegradasi pewarna sintetik secara morfologi dan molekuler, serta mengetahui hubungan kekerabatan fungi yang dapat mendegradasi pewarna sintetik menggunakan software bioinformatika.
E. Manfaat Penelitian
Manfaat dari penelitian ini adalah:
5
2. Fungi yang diidentifikasi tersebut diharapkan dapat digunakan sebagai agen biologi dalam pengolahan limbah pewarna sintetik
26 BAB III
METODE PENELITIAN
A. Jenis Penelitian
Jenis penelitian yang digunakan yaitu penelitian deskriptif. Menurut Hartoto (2009) penelitian deskriptif merupakan metode penelitian yang berusaha menggambarkan dan menginterpretasi objek sesuai dengan apa adanya. Penelitian deskriptif pada umumnya dilakukan dengan tujuan utama, yaitu menggambarkan secara sistematis fakta dan karakteristik objek dan subjek yang diteliti secara tepat.
B. Populasi dan Sampel
Populasi dan sampel yang digunakan dalam penelitian ini adalah sebanyak lima isolat fungi dengan kode isolat M. Sampel yang digunakan dalam penelitian ini adalah isolat fungi yang ditumbuhkan dari limbah pewarna sintetik. Sampel fungi diperoleh dari hutan Samarinda, Kalimantan Timur yang merupakan hasil isolasi dan diketahui dapat mendegradasi pewarna sintetik (Hadibarata, 2011). Hutan tempat pengambilan sampel fungi tersebut merupakan hutan yang dekat dengan pemukiman masyarakat dan dekat dengan limbah industri tekstil.
C. Waktu dan Lokasi Penelitian
Penelitian ini dimulai pada bulan Januari 2013 yang dilaksanakan di Laboratorium Mikrobiologi dan Laboratorium Struktur tumbuhan Jurusan Pendidikan Biologi Fakultas Pendidikan Matematika dan Ilmu Pengetahuan Alam Universitas Pendidikan Indonesia, Jalan Dr. Setiabudhi No. 299 Bandung.
D. Alat dan Bahan Penelitian
27
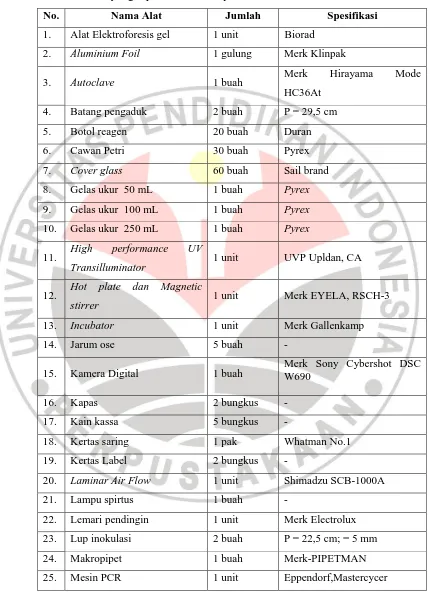
Tabel 3.1 Alat yang diperlukan dalam penelitian
No. Nama Alat Jumlah Spesifikasi
1. Alat Elektroforesis gel 1 unit Biorad
2. Aluminium Foil 1 gulung Merk Klinpak
3. Autoclave 1 buah Merk Hirayama Mode
HC36At
4. Batang pengaduk 2 buah P = 29,5 cm
5. Botol reagen 20 buah Duran
6. Cawan Petri 30 buah Pyrex
7. Cover glass 60 buah Sail brand
8. Gelas ukur 50 mL 1 buah Pyrex
9. Gelas ukur 100 mL 1 buah Pyrex
10. Gelas ukur 250 mL 1 buah Pyrex
11. High performance UV
Transilluminator 1 unit UVP Upldan, CA
12. Hot plate dan Magnetic
stirrer 1 unit Merk EYELA, RSCH-3
13. Incubator 1 unit Merk Gallenkamp
14. Jarum ose 5 buah -
15. Kamera Digital 1 buah Merk Sony Cybershot DSC W690
16. Kapas 2 bungkus -
17. Kain kassa 5 bungkus -
18. Kertas saring 1 pak Whatman No.1
19. Kertas Label 2 bungkus -
20. Laminar Air Flow 1 unit Shimadzu SCB-1000A
21. Lampu spirtus 1 buah -
22. Lemari pendingin 1 unit Merk Electrolux
23. Lup inokulasi 2 buah P = 22,5 cm; = 5 mm
24. Makropipet 1 buah Merk-PIPETMAN
28
No. Nama Alat Jumlah Spesifikasi
26. Microwave 1 unit Merk Electrolux
27. Mikropipet 1 µl 1 buah Merk Socorex
28. Mikropipet 10 µl 1 buah Merk Socorex
29. Mikropipet 5 µl 1 buah Merk Socorex
30. Mikroskop binokuler 1 unit Merk Shimadzu
31. Mortar dan alu 5 buah -
32. Object glass 30 buah Sail brand
33. Pipet 2 buah -
34. Plastik Tahan Panas 5 pak Merk Diamond
35. Plastik wrap 1 gulung Merk Klinpak
36. Rak tabung 3 buah -
37. Spatula 5 buah
38. Spektrofotometer 1 unit Genesis,Thermo scientific
39. Spidol Marker 2 buah Merk Snowman
40. Tabung mikrosentrifugasi 75 buah -
41. Tabung reaksi 50 buah Pyrex
42. Timbangan Analitik 1 unit Merk AND, HF 300
43. Tips 1 µl Secukupnya Extragene
44. Tips 10 µl Secukupnya Extragene
45. Tips 5 µl Secukupnya Extragene
46. Tissue 1 pak Merk Paseo
47. Vorteks 1 unit Merk SIBATA
48. Waterbath shaker 1 unit UNI Thermoshaker NTS-1300
49. Shaker 1 unit Merk EYELA
50. Gelas Arloji kecil 1 buah -
51. Corong Buhner 5 buah -
52. Aspirator 1 buah -
53. Sentrifuge 1 unit Merk Hettich
54. Termos air panas 1 buah -
29
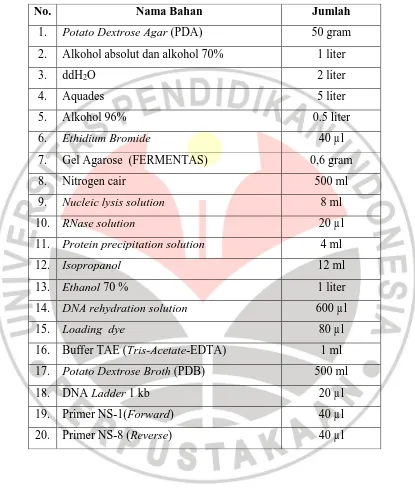
Tabel 3.2 Bahan yang dibutuhkan dalam penelitian
No. Nama Bahan Jumlah
1. Potato Dextrose Agar (PDA) 50 gram
2. Alkohol absolut dan alkohol 70% 1 liter
3. ddH2O 2 liter
4. Aquades 5 liter
5. Alkohol 96% 0.5 liter
6. Ethidium Bromide 40 µl
7. Gel Agarose (FERMENTAS) 0,6 gram
8. Nitrogen cair 500 ml
9. Nucleic lysis solution 8 ml
10. RNase solution 20 µl
11. Protein precipitation solution 4 ml
12. Isopropanol 12 ml
13. Ethanol 70 % 1 liter
14. DNA rehydration solution 600 µl
15. Loading dye 80 µl
16. Buffer TAE (Tris-Acetate-EDTA) 1 ml
17. Potato Dextrose Broth (PDB) 500 ml
18. DNA Ladder 1 kb 20 µl
19. Primer NS-1(Forward) 40 µl
30
E. Prosedur Penelitian
1. Persiapan Penelitian
a. Pembuatan medium sebagai Media Tumbuh Fungi Isolat M
Medium yang digunakan adalah medium PDA (Potato Dextrose Agar) dan PDB (Potato Dextrose Broth). Alat-alat yang terbuat dari kaca dan plastik yang akan digunakan dicuci terlebih dahulu dan dikeringkan. Alat kemudian dibungkus dengan kertas pembungkus setelah dimasukan ke dalam plastik untuk dilakukan sterilisasi panas lembab dengan cara dimasukan kedalam autoclave selama 15-20 menit pada suhu 121° C dan tekanan 1.5 atm.
b. Kultivasi Fungi Isolat M
Semua alat dan bahan yang akan digunakan disimpan dalam laminar air flow dengan disinari sinar UV terlebih dahulu selama 15 menit. Kemudian
sebanyak lima isolat fungi yang didapat di subkultur dan ditumbuhkan kembali dalam medium PDA miring dan PDA pada cawan Petri, setiap isolat fungi diinkubasi pada suhu ruang.
Untuk isolasi DNA fungi isolat M, Sampel fungi di inokulasi pada medium PDB kemudian diinkubasi pada suhu kamar dan di shaker selama lima hari untuk pertumbuhan optimal. Kultur fungi tersebut kemudian digunakan untuk ekstraksi DNA fungi(Hidayah, 2012).
2. Tahap Penelitian
Identifikasi fungi yang dapat mendegradasi pewarna sintetik (isolat M) dilakukan secara morfologi dan molekuler.
1) Identifikasi Morfologi Fungi Isolat M
a. Pembuatan Slide Culture
31
glass steril. Tahap dalam pembuatan slide culture yaitu, medium PDA yang
telah dibuat, dituangkan ke dalam cawan Petri kosong dengan ketebalan agar satu sentimeter, kemudian menunggu hingga agar benar-benar menjadi padat, setelah agar padat blok agar steril kira-kira berukuran satu sentimeter kuadrat dipotong dari medium PDA dalam cawan Petri dan diletakkan di atas object glass dengan menggunakan pisau steril atau dengan spatula. Inokulasi fungi
dilakukan pada agar yang disimpan di atas object glass, agar yang telah diinokulasi tersebut ditutup dengan cover glass, lalu menuangkan aquades secukupnya pada kertas saring yang berbentuk bundar. Slide culture tersebut kemudian diinkubasi pada suhu ruang, setelah beberapa hari diinkubasi slide culture tersebut dapat diamati dengan menggunakan mikroskop pada
perbesaran 400x, kemudian diidentifikasi (Hamdiyati, 2010). b. Identifikasi Morfologi Fungi
Identifikasi lima isolat fungi M dilakukan melalui dua tahap. Tahap pertama yaitu, pengamatan fungi secara makroskopis meliputi pengamatan terhadap warna dan bentuk koloni. Tahap kedua yaitu, pengamatan secara mikroskopis yang dilakukan setelah fungi ditumbuhkan pada slide culture dalam suhu ruang meliputi pengamatan terhadap hifa atau miselium dilihat dari spora, bentuk hifa, dan ada tidaknya sekat pada hifa (Hamdiyati, 2010). Identifikasi morfologi dilakukan sampai tingkat taksa genus menggunakan mikroskop binokuler. Lima isolat fungi M yang didadapatkan dibandingkan dengan menggunakan buku panduan identifikasi Ilustrated Genera of Imperfect Fungi (Barnett, 1960), dan A Manual of Soil Fungi (Gilman, 1975),
serta sumber lain yang mendukung.
2) Identifikasi Molekuler Fungi Isolat M
a. Ekstraksi DNA Fungi
Genom DNA fungi di ekstraksi menggunakan Wizard Genomic DNA Purification Kit (PROMEGA). Sebanyak 400 µl nucleic lysis solution
32
telah berisi kultur fungi dan di tumbuk hingga menjadi bubuk. Kemudian bubuk tersebut dipindahkan ke dalam tabung mikrosentrifugasi 1,5 ml yang telah berisi larutan nucleic lysis solution dan di vortex untuk membasahkan jaringan, kemudian tabung diinkubasi pada waterbath dengan suhu 650C selama 15 menit. Sebanyak 3 µl RNase solution ditambahkan untuk melisiskan dinding sel dan dihomogenkan dengan cara dibolak-balik sebanyak 3-5 kali, kemudian setelah homogen, tabung tersebut diinkubasi pada suhu 370C selama 15 menit. Tabung mikrosentrifugasi tersebut di simpan pada suhu ruang selama 5 menit, selanjutnya menambahkan protein precipitation solution sebanyak 200 µl dan di vortex dengan kecepatan tinggi selama 20 detik. Kemudian di sentrifugasi selama 3 menit dengan kecepatan 13000 rpm.
Setelah di sentrifugasi pada tabung ependorf akan terbentuk dua fasa, yaitu fasa atas dan bawah. Kemudian fasa atas (supernatan yang mengandung DNA) diambil dengan ujung tips yang steril, lalu dipindahkan ke tabung ependorf yang baru yang mengandung 600 µl isopropanol. Setelah larutan tercampur akan menghasilkan untai benang DNA yang membentuk suatu massa yang terlihat. Larutan tersebut di sentrifugasi kembali dengan kecepatan 13000 rpm selama 1 menit pada temperatur ruang. Selanjutnya supernatan dibuang, dan pellet (DNA) yang dihasilkan ditambahkan 600µl ethanol 70% dan dihomogenkan dengan cara dibolak balik untuk mencuci DNA. Kemudian di sentrifugasi kembali selama 1 menit dengan kecepatan 13000 rpm. Ethanol kemudian dibuang menggunakan pipet. Tabung mikrosentrifugasi dibalik dan disimpan diatas tissue untuk mengeringkan pellet selama 15 menit. Sebanyak 30 µl DNA rehydration solution ditambahkan dan diinkubasi pada suhu 650C selama 30 menit, kemudian disimpan pada suhu 2-80C (Hidayah, 2012).
b. Elektroforesis Gel Agarose
33
waterpass. Gel agarose dibuat dengan konsentrasi 1% dibuat dari 0,25 g
bubuk agarose dilarutkan dalam 1 × TAE (Tris Asetat-EDTA) sebagai larutan penyangga sebanyak 25 ml hingga larut. Kemudian, larutan dipanaskan dalam microwave selama 1 menit untuk melarutkan bubuk agarose. Kemudian gel
agarose didiamkan hingga hangat-hangat kuku lalu dituangkan ke dalam
cetakan yang dilengkapi dengan sisir (comb). Sisir dipasang dengan posisi tegak dan berjarak 0,5-1 mm dari dasar cetakan. Selanjutnya gel dibiarkan mengeras pada suhu ruang.
Gel dan cetakan direndam pada buffer TAE 1X pada kolom elektroforesis. Larutan sampel dari freeze diambil sebanyak 3-5 µl, kemudian dicampurkan dengan 2 µl loading dye. Sampel dimasukkan ke dalam sumur yang terdapat dalam gel pada kolom elektroforesis. Setelah sampel dimasukkan, kemudian dielektroforesis pada tegangan 100 volt selama 30 menit (Mahuku, 2004). Gel berisi DNA hasil elektroforesis diwarnai menggunakan larutan Ethidium Bromide (EtBr) dengan konsentrasi 0,5 µg/ml selama 5 menit, kemudian dibilas dengan deion water steril untuk membuang kelebihan EtBr. Gel hasil elektroforesis dilihat dengan alat sinar UV (UV transluminator), pada panjang gelombang 312 nm dan difoto dengan
menggunakan kamera digital merek Sony CyberShot DSC-W690.
c. Polymerase Chain Reaction (PCR)
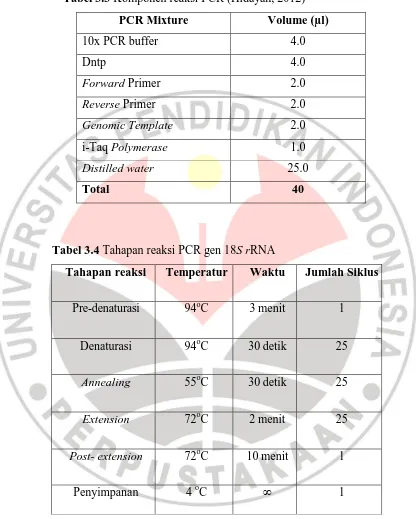
PCR didasarkan pada kemampuan polimerase DNA untuk mensintesis untai DNA komplementer baru dengan template strand. PCR memungkinkan untuk menghasilkan jutaan salinan dari urutan DNA tertentu. Proses PCR digunakan untuk amplifikasi DNA fungi. Ekstraksi genom DNA fungi di amplifikasi menggunakan primer universal 18S rRNA. Primer tersebut yaitu NS1 dan NS8, Noor Hidayah dalam Hadibarata et al., 2012. Primer NS1 (5’ -GTA GTC ATATGC TTG TCT C-3) dengan berat molekul 5785 dan NS8 (5’ -TCC GCA CGT TCA CCT ACG GA-3) dengan berat molekul 6078.
34
Tabel 3.3 Komponen reaksi PCR (Hidayah, 2012)
PCR Mixture Volume (µl)
10x PCR buffer 4.0
Dntp 4.0
Forward Primer 2.0
Reverse Primer 2.0
Genomic Template 2.0
i-Taq Polymerase 1.0
Distilled water 25.0
Total 40
Tabel 3.4 Tahapan reaksi PCR gen 18S rRNA
Tahapan reaksi Temperatur Waktu Jumlah Siklus
Pre-denaturasi 94oC 3 menit 1
Denaturasi 94oC 30 detik 25
Annealing 55oC 30 detik 25
Extension 72oC 2 menit 25
Post- extension 72oC 10 menit 1
Penyimpanan 4 oC ∞ 1
35
Gambar 3.1 Profile PCR gen 18S rRNA (Hidayah, 2012)
d. Elektroforesis Gel Agorose
Gel agarose hasil PCR yang di dapat kemudian dielektroforesis kembali
dengan cara yang sama seperti sebelumnya.
e. Sequencing DNA
Proses sequencing DNA dilakukan dengan menggunakan mesin squencer BigDye Applied Biosystem model 3730 yang dilakukan di Macrogen inc.,
Korea.
f. Analisis Data Bioinformatika
Isolat fungi yang telah di subkultur, didentifikasi sampai tingkat taksa Genus melalui analisis sikuen gen 18S rRNA menggunakan metode bioinformatika (BLAST) secara online. Setiap sikuen gen fungi yang didapat dari proses sequencing diterjemahkan terlebih dahulu menjadi kode nukleotida. Kemudian setiap sikuen nukleotida tersebut disejajarkan dan dibandingkan dengan data sikuen gen isolat fungi yang terdapat pada database Bank Gen NCBI (National Center for Biotechnology Information) di alamat website http://www.ncbi.nml.nih.gov/BLAST/Blast.cgi. Proses alignment dari setiap sikuen nukleotida gen isolat fungi menggunakan program tool multiple-sikuense alignment dari software Clustal X dan software MEGA (version 5) untuk menganalisis filogenetik fungi tersebut melalui pohon filogenetik yang dihasilkan.
940C
’
940C
’’
720C
’
720C
’
Post extension extension
denaturasi
Pre denaturasi 550C
’’ 40C
∞
36
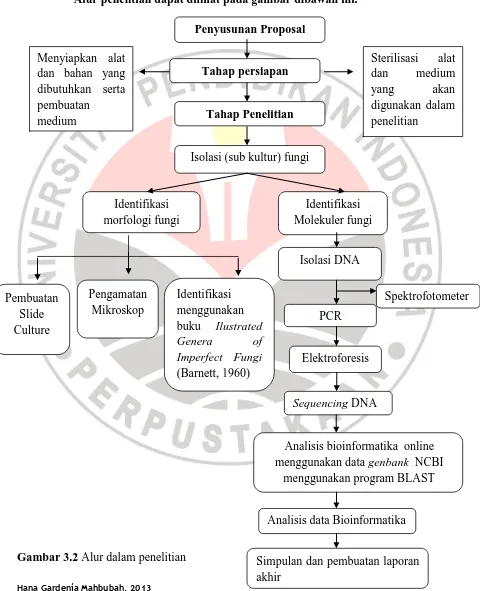
3. ALUR PENELITIAN
Alur penelitian dapat dilihat pada gambar dibawah ini.
Gambar 3.2 Alur dalam penelitian
Penyusunan Proposal
Menyiapkan alat dan bahan yang dibutuhkan serta pembuatan
medium
Sterilisasi alat dan medium
yang akan
digunakan dalam penelitian
Tahap persiapan
Tahap Penelitian
Isolasi (sub kultur) fungi
Identifikasi morfologi fungi Identifikasi Molekuler fungi Isolasi DNA Pengamatan Mikroskop Pembuatan Slide Culture Identifikasi menggunakan buku Ilustrated
Genera of
Imperfect Fungi (Barnett, 1960)
Spektrofotometer PCR
Elektroforesis
Sequencing DNA
Analisis bioinformatika online menggunakan data genbank NCBI
menggunakan program BLAST
Analisis data Bioinformatika
57 BAB V
SIMPULAN DAN SARAN
A. Simpulan
Berdasarkan identifikasi morfologi lima isolat fungi M, empat isolat yang diidentifikasi termasuk ke dalam genus Trichoderma yaitu isolat M04, M06, M10, dan M17, sedangkan satu isolat dengan kode isolat M15 termasuk ke dalam genus Absidia. Dari empat isolat yang teridentifikasi sebagai Trichoderma memiliki ciri yaitu warna koloni putih kehijauan, bentuk koloni
tidak beraturan dengan permukaan koloni halus seperti kapas, berbutir-butir, hifa bersekat, memiliki konidia hialin berbentuk bulat dan fialida berbentuk seperti botol. Sedangkan Absidia memiliki ciri yaitu warna koloni putih kemerahan, bentuk koloni tidak beraturan dengan permukaan koloni halus berserabut, hifa tidak bersekat, menghasilkan sporangia berbentuk seperti buah pir. Hasil identifikasi secara molekuler dengan menggunakan sikuen gen 18S rRNA menyatakan bahwa isolat M04, M06, M10, dan M17 memiliki
hubungan kekerabatan terdekat dengan Trichoderma harzianum, di dukung dengan nilai bootstrap sebesar 100%, sedangkan isolat sampel M15 memiliki hubungan kekerabatan terdekat dengan Absidia spinosa di dukung dengan nilai bootstrap 100%.
B. Saran
1. Studi lanjutan mengenai karakteristik morfologi fungi yang dapat mendegradasi pewarna sintetik ini perlu ditindaklanjuti agar dapat dimanfaatkan oleh industri sebagai agen biologi dalam pengolahan limbah pewarna sintetik.
58
DAFTAR PUSTAKA
Achmad, Herliyana E.N., and Agustian F.R. (2009). “Hubungan Kekerabatan Jamur Pelapuk Putih Pleurotus spp. dengan Analisis Isoenzim”. Jurnal AgroBiogen. 5, (2), 78-83.
Adosinda, M., Nelson L., Armando J.D.Silcestre., and Queiroz M.J. (2003), “Comparative studies of fungal degradation of single or mixed bioaccessible reactive azo dyes”. Institute of Biotechnology and Fine Chemistry (IBQF). Chemosphere. 52, 967–977.
Alexopoulus, C. J., Blackwell M., and Mims C.W. (1996). Introducy Micology. 4th Ed. New York: John Wiley & Sons, Inc: hal.30.
Ali P. & Muhammad S.K. (2008). “Biodecolorization of Acid Violet 19 by Alternaria solani”. African Journal of Biotechnology.7, 831-833.
Arifin. (2008). Teknologi Pengendalian Limbah Cair (Spec. Tekstil) .[online]. Tersedia: http://smk3ae.wordpress.com/2008/06/30/teknologi-pengendalian-limbah-cair-spec-tekstil [28 Januari 2013]
Awaluddin R., Darah S., Ibrahim C.D., and Uyub A.M. (2001). “Decolorization of commercially available synthetic dyes by the white rot fungus Phanerochaete chrysosporium”. J Fungi and Bactery. 62, 55-63.
Bagewadi Z.K., Vernekar A.G., Patil A.Y., Limaye A.A., and Jain V.M. (2011). “Biodegradation of Industrially Important Textile Dyes by Actinomycetes
Isolated From Activated Sludge”. Department of
Biotechnology,BV.Bhoomaradi college of engineering and technology. 60, 67-80.
Baldwin B.G., Sanderson M.J., Porter J.M., Wojciechowski M.F., Campbell C.S., and Donoghue M.J. (1995). “The ITS region of nuclear ribosomal DNA: A valuable sources of evidence on Angiospermae phylogeny”. Ann.Missouri Bot. Gard, 247-277.
Barnett, I. (1960). Ilustrated Genera of Imperfect and Fungi. Department of Plant Phatology, Bacteriology, Entomology West Virginia University. 2nd Edition. Morgantown West Virginia: Burgess Publishing Company.
59
Berkhout. (1923). Absidia spp. [Online]. Tersedia: http://www.doctorfungus.org/the fungi/absidia.html [01 Maret 2013].
Boran, F. & Yesilada O. (2011). “Enhanced Production Of Laccase By Fungi Under Solid Substrate Fermentation Condition”. Bioresources. 6, (4), 4404-4416.
Brinkman, F. & Leipe D. (2001). “Phylogenetic Analysis, A Practical Guide to the Analisys of Gene and Protein”. Baxevanis. 5, 323-358.
Brock, T.D. & Madigan M.T. (1991). Biology of Microorganisms. 7th Ed. New Jersey : Prentice-Hall International, Inc.
Budiyanto. (2013). Ciri-ciri fungi. [online]. Tersedia http: // budisma .web .id /materi /sma / biologi-kelas-x/ciri-ciri-fungi [20 Januari 2013].
Cahyadi, W. (2008). Analisis dan Aspek Kesehatan Bahan Tambahan Pangan. Jakarta: Bumi Aksara.
Campbell N.A., Reece B.J., and Mitchell L.G. (2002). Biologi (Ed. Terjemahan). Jakarta: Erlangga
Campbell N.A., Reece B.J., and Mitchell L.G. (2011). Biology. California: The Benjamin/Cummings Publishing Company Inc.
Chang, S.T. & Quimio H. (1982). Tropical Mushrooms: Biological Nature and Cultivation Methods. Hongkong: Te Chinese University Press.
Christian V., Rshrivastava, Sukla D., Modi M.A., & Vyas B.R.M. (2005). “Degradation of Xenobiotic Compounds by Lignin-degradibg White-rot fungi: Enzymology and
Mechanism Involved”. Indian Journal of Experimental Biology. 43, 301-312.
Daneshvar N, Ayazloo M, and Khatae AR. (2007). “Biological Decolourization of Dye Solution Containing Malacite Green by Microalgae Cosmarium sp”. Bioresource Technology. 2, 29 1-7.
Davis, L., Kuehl M., and Battey J. (1994). Basic methods: Molecular biology. Norwegia: Appleton & Lange.
Dewi, C.L.H. (2012). Analisis Biomolekuler Gen Internal Transcribed Spacer (ITS) dalam studi Filogenetik Zingiber loerzingii Valeton (ZINGIBERACEAE). Skripsi. Departemen Biokimia FMIPA. Institut Pertanian Bogor.
60
Eschmid. (2001). Gel Elechtrophoresis .[Online]. Tersedia: http:// classroom.sdmesa.edu/eschmid/lab.html [ 03 Februari 2013].
Faddin, J.F. (1985). Media for isolation cultivation identification maintenance of medical bacteria. Baltimore: Williams & Wilkins.
Fairbank D.J & Andersen W.R. (1999). Genetics: The continuity of life. London: academic press.
Fatchiyah. (2005). Gel Elektroforesis. Malang: Laboratorium Sentral Biologi Molekuler dan Seluler Universitas Brawijaya.
Fatchiyah. (2005). PCR: Dasar teknik Amplifikasi DNA dan Applikasinya. [Online]. Tersedia: http://fatchiyah.lecture.ub.ac.id [ 03 Maret 2013].
Gams W., Van der Aa H.A., Samson R.A., and Stalpers J.A. (1987). Cbs Course of Mycology. Institute Of The Royal Netherlands Academy Of Arts And Science. Netherlands: Centraalbureau Voor Schimmelcultures.
Gandjar, I., Samson R.A., van den Tweel, Vermeulen, A. Oetari and Santoso I. (1999). Pengenalan kapang tropik umum. Jakarta: Yayasan Obor Indonesia
Gomes EA., Kasaya M.C., DeBarros E.G., Borgs A.C., and Araujo E.F. (2002). “Polymorphism in the internal transcribed spacer (ITS) of the ribosomal DNA of 26 isolates of ectomycorrhizal fungi”. Genet Mol Biol. 25, (4), 477-48 Gupta M. & Manisha K. (2012). “Biodegradation of Textile Dye Congo Red by
Fungus”. Indian Journal of Fundamental ang Applied Life Sciences ISSN : 2231-6345.
Hadibarata, T., Yusoff, A. R, Aris A, Salmiati, Hidayat T, and Kristanti R.A., (2011). “Decolorization of Azo, Triphenylmethane, and Anthraquinone Dyes by Laccase of a Newly Isolated Armillaria sp. F022. Water Air Pollution”.
Hakala, T.K. (2007). Characterization 0f The Lignin-Modifying Enzymes of The Selective White-Rot Fungus Physisporinus Rivulosus. Disertasi Doktor pada Department of Applied Chemistry and Microbiology University of Helsinki. Hamdiyati, Yanti. (2010). Cara Membuat Slide Culture. [Online]. Tersedia:
61
Handoyo, Darmo. (2001). Prinsip Umum dan Pelaksanaan Polymerase Chain Reaction (PCR).[Online]. Tersedia: http://repository.ubaya.ac.id/35/1/AR T002 .pdf [05 Februari 2013].
Hartoto. (2009). Penelitian Deskriptif. [online].Tersedia: http://www.penalaran-unm.org/index.php/artikel-nalar/penelitian/163-penelitian-deskriptif.html [15 Desember 2013].
Hattaka A. (1994). “Lignin Modifying Enzyme From Selected White-Rot Fungi:
Production And Role In Lignin Degdradation”. FEMS Microbial. 13, 125-135.
Hesseltine, C.W. & Ellis J.J. (1966). “Species of Absidia with Ovoid Sporangiospores”. Mycologia. 58, 761-785.
Hidayah, Noor. (2012). Molecular Identification of Fungus That Capable to Degrade Synthetic Dye. Skripsi pada Universitas Teknologi Malaysia.1-50.
Hidayat, T., & Pancoro, A. (2006). Sistematika dan Filogenetika Molekuler. [Online]. Tersedia:http://file.upi.edu/Direktori/FPMIPA/JUR._PEND._BIOLOGI/19700410 1997021/TOPIK_HIDAYAT/Makalah_Filogenetik_Molekuler.pdf. [10 Juni 2013].
Hidayat, T., & Pancoro, A. (2008). Kajian Filogenetika Molekuler dan Peranannya dalam Menyediakan Informasi Dasar untuk Meningkatkan Kualitas Sumber GenetikAnggrek.[Online].Tersedia:http://biogen.litbang .deptan.go.id/terbitan/pdf/ agrobiogen [10 Juni 2013].
Hofrichter M. (2002). “Lignin Conversion by Manganese Peroxidase (MnP)”. Enzyme Microbiol. Technol. 30, 454-466.
Hudson., Flemming., and Rand. (2004). BALF. Total protein, albumin, pro-inflammatory cytokine (IL-1, TNF-α) and LDH responses significantly different (time and dose dependently) by fungal species (S. chartarum vs C. cladosporioides) and fungal strain. WHO guidelines for indoor air quality : dampness and mould. Jerman: World Health Organization.
Huffmann, Kerstin., Discher, S., and Voight, K. (1997). Revision of the genus Absidia (Mucorales, Zygomycetes) based on physiological, phylogenetic, and morphological characters; thermotolerant Absidia spp. form a coherent group,Mycocladiaceae fam.nov. [Online]. Tersedia: http://www.sciencedirect.com [20 Februari 2013].
62
Katia M.G.M., Luciana C.A.A., Rubio O.M., Luiz H.R., and Mercia H.S. (2006). “Biodegradation of reactive textile dyes by Basidiomycetous fungi from Brazilian ecosystems”. Brazilian J.Microbiol. 37, 481-487.
Kirk T. K., Jeffries T. W., and Leatha G. F. (1990). “Biotechnology: applications and implications for the pulp and paper industry”. The Journal of the Technical Association of the Pulp and Paper Industry. 66, (5), 100-130.
Lafontaine D.L.J & Tollervey D. (2001). “Conserved organization of the pre-rRNA”. Nature Reviews Molecular Cell Biology. 2, 514-520.
Li W., & Graur D. (1991). Fundamental of Molecular Evolution. Sunderland: Sinauer Associates, Inc.
Li W., Pearl D. and Doss, H. (1999). Phylogenetic tree construction using Markov Chain Monte Carlo. Fred Hutchinson Cancer Research Center Washington. [Online]. Tersedia: http://www.stat.ohio-state.edu/~doss/Research/mctrees.pdf. [20 Agustus 2013].
Mahuku, G. S., (2004). “A Simple Extraction Method Suitable for PCR Based Analysis of Plant, Fungal, and Bacterial DNA”. International Society for Plant Molecular Biology. 22, 71-81.
Marchesi J.R., Sato T., Weightman A.J., Martin T.A., Fry J.C., Hiom S.J., Wde W.G.,
and Wade W.G. (1998). “Design and Evaluation of Useful Bacterium Specific
PCR Primers that Amplify Genes Coding for Bacterial 16S Rrna”. Application
Environmental Microbiology. 64, 795-799.
Mathur, N., P. Bhatnagar, and P. Bakre. (2005). “Assessing Mutagenicity of Textile Dyes From Pali (Rajasthan) Using Ames Bioassay”. Applied ecology and environmental research .4, (1), 111-118.
Mc.Kane., & Kandel Judy. (1995). Microbiology: Essentials and Applications (scnd ed.) London: McGraw-Hill Companies.
Meyer A., Todt C., Mikkelsen N.T., and Lieb B. (2010). “Fast evolving 18S rRNA sikuences from Solenogastres (Mollusca) resist standard PCR amplification and give new insights into mollusk substitution rate heterogeneity”. BMC Evolutionary Biology 2011. 5, 715-810.
63
Mohan G., Logambal K., and Ravikumar R. (2011). “Investigation on The Removal of Direct Red Dye Using Aspergiluus Niger and Aspergillus Flavus Under Static and Shaking Conditions with modeling”. Internasional Jurnal of Science, Environment and Technology. 1, (5), 144-153.
Moore, D. (1998). Fungal Morphogenesis. A volume in the Development and cell biology series. New York: Cambridge University Press.
Moorthi S., Selvam P., Sasikavaleni A., Murugesan K., and Kalaichelvan P.T. (2007). “Decolorization of Textile Dyes and their Effluents using White Rot Fungi”. African Journal of Biotechnology. 6, (4), 424-429.
Muladno. (2002). Teknologi Rekayasa Genetika, Bogor: Pustaka Wirausaha Muda. Nyoman, Sukarta I. (2009). Pemanfaatan Fungi Pelapuk Kayu Jenis Pleurotus sp untuk
Mendegradasi Zat Warna Tekstil Jenis Azo. [online].Tersedia: www.undiksha.ac.id. [03 Januari 2013].
Pandey A, Singh P, and Iyengar L. Biodegrad. (2007). 59, 73-84
Pavko, A. (2011). “Fungal Decolourization and Degradation of Synthetic Dyes some Chemical Engineering Aspects”. Intechopen. 4, 65-70.
Pelczar, M.J, & Chan E. C. S. (1986). Dasar-dasar Mikrobiologi 1. Jakarta : UI-Press. Pratiwi, Yuli. (2010). Penentuan Tingkat Pencemaran Limbah Industri Tekstil
Berdasarkan Nutrition Value Coeficient Bioindikator.[Online]. Tersedia : http://jurtek.akprind.ac.id/sites/default/files/129_137_yul_pratiwii.pdf. [21 Januari 2013].
Rifai M.A. (1969). “Revision of The Genus Trichoderma”. Mycology paper. 116, 1-11.
Sambrook, J. R, & David, W. (1989). Molecular Cloning: A Laboratory Manual, (secnd ed.). Australia: Cold Spring Harbour Laboratory Press.
Sathiya M., Periyar S., Sasikalaveni, Murugesan, and Kalaichelvan. (2007). “Decolorization of textile dyes and their effluents using white rot fungi”. African Journal of Biotechnology. 6, (4), 510-550.
64
Setyowati. (2003). “Penurunan Penyakit Busuk dan Akar Pertumbuhan Gulma Selada
yang Dipupuk Mikroba”. Jurnal-Jurnal Pertanian Indonesia ISSN: 1411-0067. Simpson. (2011). Plant Systematics. Canada: Elsevier Academic Press.
Singh L & Singh VP. (2010). “Microbial degradation and decolourization of dyes in semi-solid medium by the fungus-Trichoderma harzianum”. International Journal of Science & Technology. 5, (3), 147-153.
Siswanto, Suharyanto, and Fitria R. (2007). “Produksi dan Karakteristik Lakase Omphilina sp.”. Menara Perkebunan.75, 107-110.
Socaciu, C. (2008). Analysis of synthetic food colorants. New York: CRC Press.
Soepardi, G. (1983). Sifat dan Ciri Tanah. Skripsi pada Fakultas Pertanian Institut Pertanian Bogor. Bogor.
Swamy, J & Ramsay, J.A. (1999). “The evaluation of white-rot fungi in the decolourization of textile dyes”. Enzyme and Microbial Technology. 24, 130-137. Syafarudin & Santoso, T.J. (2011). “Optimasi Teknik Isolasi dan Purifikasi DNA yang
Efisien dan Efektif Pada Kemiri Sunan (Reutalis trispema (Blanco) Airy Shaw)”. Jurnal Litri. 17, (1), 11-17.
Tanksley, S.D. (1983). “Molecular Markers in Plant Breeding”. Plant Mol. Bio. Report. 1, 3-8.
Toha, A.H.A. (2001). Deoxyribo Nucleic Acid. Keanekaragaman, Ekspresi, Rekayasa, dan Efek Pemanfatannya. Bandung: Alfabeta.
Tortora, G.J., Berdell R. F., and Christine L. C. (2001). Microbiology : an introduction. (sevn ed) . San Francisco : Benjamin Cummings.
Van der Zee. (2002). Anaerobic azo dye reduction. Thesis pada Wageningen University. Netherlands.
Windiastika, Gati. (2012). Metode Uji Kualitatif DNA dengan Elektroforesis Gel agarosa. [Online].Tersedia: http:// ditjenbun.deptan.go.id/ bbp2tpsur/images /stories/perbenihan/ ujikualitatifdna.pdf [15 April 2013].
65
Yuri. (2012). Slide Culture Technique. [Online]. Tersedia: http://thunderhouse4-yuri.blogspot.com/2011/11/slide-culture-technique.html. [20 Agustus 2013]. Yuwono T. (2005). Biologi Molekuler. Jakarta: Erlangga.