37
Mukhtasyam Zukhrullah, Muhammad Aswad, dan Subehan
Fakultas Farmasi Universitas Hasanuddin, Makassar
ABSTRAK
Dewasa ini telah ditemukan banyak senyawa antiinflamasi dari bahan alam. Penelitian ini telah dilakukan untuk memprediksi mekanisme kerja senyawa-senyawa anti-inflamasi dari bahan alam pada tingkat molekuler sebagai inhibitor COX-2 dengan menggunakan metode Molecular Docking Simulation. Target yang dievaluasi adalah interaksi ikatan hydrogen antara protein and ligan. Simulasi docking ini menggunakan program PLANTS (Protein Ligand Ant System) dengan menggunakan 10 konformasi ligan. Hasil penelitian dari 22 senyawa bahan alam dapat mengikat COX-2 ditunjukkan dengan energi dan interaksi ikatan hidrogen (ΔG). Hasil menunjukkan bahwa ikatan hidrogen (ΔG) terbaik yaitu senyawa barbaloin (-17,8976 kkal/mol), Interaksi antara barbaloin dengan COX-2 ditandai dengan adanya 11 ikatan hidrogen yang terjadi dengan residu-residu yang ada didalam binding site dalam hal ini adalah 2 ikatan hidrogen pada Arg120 (gugus yang terikat adalah Hidroksil {OH} dan gugus karbonil {=O}, 3 ikatan hidrogen pada Tyr355 (gugus hidroksil), 2 ikatan hidrogen pada Met522 (gugus hidroksil), 1 ikatan hidrogen pada Val523 (gugus hidroksil), 1 ikatan hidrogen pada Ala527 (gugus hidroksil), dan 2 ikatan hidrogen pada Ser530 (gugus hidroksil) yang berarti bahwa barbaloin mampu mengikat daerah katalitik residu pada COX-2. Dari hasil ini dapat disimpulkan bahwa senyawa barbaloin memiliki potensi yang baik untuk menghambat COX-2.
Kata kunci : antiinflamasi, docking, inhibitor COX-2, in silico
PENDAHULUAN
Obat analgesik antipiretik serta obat anti-inflamasi non steroid (AINS) merupakan salah satu kelompok obat yang banyak diresepkan dan juga digunakan tanpa resep dokter. Obat-obat ini meru-pakan suatu kelompok obat yang heterogen, se-cara kimia. Walaupun demikian obat-obat ini ter-nyata memiliki banyak persamaan dalam efek terapi maupun efek samping. (1)
Golongan obat AINS menghambat enzim siklooksigenase sehingga konversi asam arakidon-at menjadi PGG2 terganggu. Setiap obarakidon-at AINS menghambat siklooksigenase dengan kekuatan dan selektivitas yang berbeda. Obat-obat kelom-pok AINS ini termasuk rofekoksib, selekoksib, eterokoksib, lumirakoksib, dan meloksikam. (1)
Enzim siklooksigenase merupakan enzim yang mengkatalisis pembentukan prostaglandin, suatu mediator inflamasi, dan produk metabolisme asam arakidonat. Enzim COX terdiri dari 2 iso-enzim yaitu COX-1 dan COX-2. Enzim COX-1 ber-sifat konstitutif untuk memelihara fisiologi normal dan homeostasis, sedangkan COX-2 merupakan enzim yang terinduksi pada sel yang mengalami inflamasi oleh sitokin, endotoksin, dan faktor per-tumbuhan (growth factors). COX-2 juga berperan dalam proliferasi sel kanker. Ekspresi berlebihan COX-2 ditemukan pada kebanyakan tumor (2).
Penemuan isoform COX-2 membuka lem-baran baru penelitian yang didasarkan pada
asum-si bahwa patologis prostaglandin (PG) diprodukasum-si oleh induktif yang isoform, sedangkan fisiologis prostaglandin diproduksi oleh konstitutif COX-1 (3). Mengingat besarnya peran COX-2 dalam proses inflamasi dan kanker, maka perlu dilakukan pencarian ikatan obat-obat AINS yang sesuai de-ngan reseptor tertentu yang berikatan dede-ngan en-zim COX-2. Oleh karena itu, diperlukan suatu studi mengenai senyawa-senyawa yang dapat meng-hambat COX-2, yang dengan teknologi komputasi hal ini dapat dilakukan dengan lebih mudah. Studi komputasi dikenal dengan terminologi in silico me-rupakan analog in vivo dan in vitro, yang meng-gunakan aplikasi komputer sehingga waktu dan biaya menjadi lebih efisien (4,5).
Terminologi in silico di antaranya dikenal sebagai penapisan virtual. Untuk melakukan pena-pisan senyawa biologis terhadap milyaran senya-wa masih sangat sulit, oleh karena itu pendekatan secara virtual menjadi alternatif. Karena metode ini relatif lebih cepat bahkan mampu menangani ribu-an senyawa dalam satu hari dribu-an bergribu-antung pada senyawa yang diuji dan kecepatan komputer. Kini, penapisan virtual telah mencapai status sebagai teknologi yang dinamis dan menguntungkan dalam penemuan senyawa obat (6,7).
Proses penapisan virtual digunakan untuk membantu menemukan senyawa senyawa yang kemungkinan besar berpotensi sebagai obat, de-ngan membutuhkan waktu yang relatif singkat. Jika target telah diketahui, algoritma docking dapat
di-gunakan untuk menempatkan kandidat obat ke da-lam sisi aktif dari target seperti enzim atau resep-tor. Kemudian interaksi senyawa-senyawa yang te-lah diikatkan kemudian diurutkan berdasarkan ha-sil analisis secara komputasi komponen sterik dan elektrostatiknya. (8,9,10,11)
Dewasa ini telah banyak ditemukan senya-wa antiinflamasi dari bahan alam. Tetapi, belum diketahui senyawa-senyawa yang selektif meng-hambat COX-2.
Berdasarkan uraian di atas, maka dilaku-kan penelitian yang secara khusus memodeldilaku-kan interaksi beberapa senyawa yang diketahui diketa-hui memiliki aktivitas antiinflamasi melalui peng-hambatan COX-2.
Metode yang digunakan dalam penelitian ini adalah studi interaksi beberapa senyawa bahan alam yang diketahui dapat menghambat COX-2 dengan menggunakan aplikasi komputer secara in silico pada tingkat molekuler atau yang dikenal dengan metode Simulasi Penambatan Molekul
(molecular docking simulation) dengan
meng-gunakan informasi dari struktur target maupun sifat fisikokimia ligan dapat dilakukan skrining melalui uji interaksi senyawa-senyawa yang diketahui dapat menghambat COX-2 pada sisi aktifnya.
Tujuan penelitian ini adalah memprediksi mekanisme kerja senyawa-senyawa antiinflamasi dari bahan alam pada tingkat molekuler, sebagai inhibitor COX-2 dengan menggunakan metode
Molecular Docking Simulation.
Manfaat penelitian ini adalah memperlihat-kan interaksi senyawa-senyawa antiinflamasi dari bahan alam yang diketahui selektif menghambat COX-2, sehingga mekanisme kerja senyawa terse-but dapat diprediksi sebagai penghambat COX-2 pada tingkat molekuler.
METODE PENELITIAN Data yang digunakan
Untuk melaksanakan penelitian, digunakan data struktur COX-2 yang diperoleh dari Protein
Data Bank (PDB) dengan PDB ID : 6COX, dari
hasil penelitian sebelumnya (11), seperti yang ter-lihat pada gambar 1. Selain itu, diperlukan juga data struktur senyawa-senyawa bahan alam yang geometrinya telah dioptimasi, seperti yang disaji-kan pada tabel 1.
Alat yang digunakan
Perangkat yang digunakan pada penelitian ini adalah AMD Athlon (tm) Neo X2 Dual Core Pro-cessor L335 1.66 GHZ, 2.0 RAM, Sistem Operasi Linux Ubuntu Versi 10.10, PLANTS (Protein
Ligand ANT System) versi 1.2. MarvinSketch 5.6.2
dari YASARA versi 6. MMV (Molegro Molecular Viewer) versi 2.0, dan VMD (Visual Molecular Dinamic) versi 1.9
Gambar 1. Struktur Enzim COX-2 dengan PDB ID : 6COX. Sumber : Kurumbail, R.G.,et al. Cyclooxygenase-2 (Prostaglandin Synthase-Cyclooxygenase-2 Complexed with a Selective Inhibittor), SC-558 IN I222 Space Goup.1996. Available from:http://www.pdb.org/pdb/explore/explore.do?structur eId=6COX. Accessed at may, 16,2011 (11)
Tabel 1. Data senyawa-senyawa bahan alam yang telah dioptimasi
No Senyawa Nama Rumus umum Golongan senyawa Tanaman Asal 1 Androgafolid C20H30O5 Diterpenoid Andrographis paniculata 2 Kurkumin C21H20O6 Kurkuminoid Curcuma longa 3 Alizarin C14H8O4 Kuinon Rubia cordifolia L.
4 Serumbon C15H22O Seskuiterpen Zingiber zerumbet
5 Luteolin C15H10O6 Flavonoid Lindera erythrocarpa
6 Diosmin C28H32O15 Flavonoid Citrus aurantifolia
7 Ortosiponin C12H12O5
Glikosida saponin
Orthosiphon stamineus 8 Kaemferol C15H10O6 Flavonoid Glycyrrhiza glabra L.
9 Barbaloin C21H22O9 Antrakuinon Aloe vera Linn.
10 Kuersetin C15H10O7 Flavonoid
Ruta angustifolia (L.)Pers. 11 Pisalin C28H30O10 Steroid Physalis angulata L.
12 Lucidone C24H34O5 Diterpen Lindera erythrocarpa
13 Diosgenin C27H42O3 Steroid sapogenin Solanum nigrum L 14 Mollugin C17H16O4 Xantopurpurin Rubia cordifolia L. 15 Chrysin C15H10O4 Flavonoid Lindera erythrocarpa
16 Sedanolid C12H18O2 Lakton Apium graveolens L.
17 Isoliquiritin apiosid C21H22O9 Flavonoid Glycyrrhiza glabra L.
18 Mangostin-α C24H26O6 Xantonoid Garcinia mangostana
19 Mangostin-β C25H28O6 Xantonoid Garcinia mangostana 20 Mangostin-γ C23H24O6 Xantonoid Garcinia mangostana 21 Gertanin C23H24O6 Xantonoid Garcinia mangostana 22 Emodin C15H10O5 Antrakuinon Garcinia mangostana
Pengambilan Data
Dalam penelitian ini, data struktur protein target diambil melalui Protein Data Bank (PDB) dengan PDB ID: 6COX dan digunakan sebagai tar-get docking. Data ini merupakan hasil teknik bio-fisika seperti kristalografi X-ray maupun spektro-skopi NMR dari COX-2 yang meliputi struktur, sisi aktif dan sekuens.
Pemodelan Molekul Senyawa Antiinflamasi dari Bahan Alam
Data struktur semua sampel dari bahan alam digambar dengan mengunakan MarvinSketch
v. 5.6 (disimpan dalam format file ligand_2D.mrv). Kemudian setiap senyawa di protonasi pada pH = 7.4, kemudian dilanjutkan mencari konformasi de-ngan menggunakan program yang sama untuk yang diperlihatkan dalam bentuk 3-dimensi. Kemu-dian berkas disimpan dalam bentuk mol2.
Preparasi Protein Target
Protein target dipreparasi dengan meng-gunakan YASARA, yaitu berkas protein dalam format #.pdb, kemudian dihapus salah satu rantai protein (rantai B) untuk meminimisasi wilayah
docking, dilanjutkan dengan menghapus NAG dan
air apabila diperlukan sehingga hanya tersisa mo-lekul asam amino. Hidrogen ditambahkan, hasilnya disimpan dalam bentuk berkas mol2 yang kemudi-an digunakkemudi-an sebagai protokol docking untuk pe-napisan virtual senyawa COX-2.
Simulasi dan Validasi Docking
File input ligan dan file protein target di-simpan bersama dalam sebuah folder bersama
PLANTS versi 1.2, kemudian ditentukan pose
kon-figurasi dan konkon-figurasi docking (plantsconfig) yang merupakan modifikasi dari konfigurasi stan-dar stan-dari PLANTS. Definisi konfigurasi diubah men-jadi 5Ǻ dari koordinat lokasi SC558 terikat pada COX-2. Pose yang memiliki nilai skoring terbaik di-pilih sebagai sebagai tempat sampel diperkirakan berikatan. Kemudian docking ligan asli (SC558), setelah itu dilakukan pehitungan RMSD (Root
Mean Square Deviance) dengan menggunakan
YASARA (RMSD dinyatakan valid apabila lebih
rendah dari 2Å). Apabila nilainya kurang dari 2Ǻ, maka dapat digunakan sebagai protokol docking
untuk skrining virtual sampel berikutnya.
Analisis Data
Dilakukan analisis data nilai skoring dan pose pada sisi binding pocket serta analisis ikatan hidrogen ligand-sekuens asam amino dengan menggunakan MMV. Molekul dengan nilai skoring terendah menunjukkan afinitas kestabilan yang baik, setelah itu visualisasi interaksi dengan meng-gunakan program VMD.
HASIL DAN PEMBAHASAN
Dalam penelitian ini dipelajari interaksi se-kelompok senyawa-senyawa antiinflamasi dari be-berapa golongan senyawa yang telah diisolasi dan diidentifikasi oleh para peneliti terdahulu dari ber-bagai referensi.
Metode yang digunakan dalam mempela-jari interaksi tersebut yakni molecular docking.
Docking merupakan suatu perlakuan dimana suatu
senyawa lainnya dan pada saat yang bersamaan dilakukan perhitungan energi interaksi dari bebera-pa orientasi yang sama dari keduannya. Suatu prosedur docking digunakan sebagai acuan untuk menentukan orientasi yang baik dari senyawa ter-hadap senyawa lainnya yang bersifat relatif.
Senyawa target atau reseptor yaitu siklo-oksigenase-2 (COX-2). Sebelum docking dilaku-kan, senyawa target harus dipreparasi terlebh dahulu. Senyawa yang telah diunduh dari situs
RCSB PDB ditampilkan dalam jendela YASARA,
senyawa-senyawa air sebaiknya dihilangkan se-hingga dipastikan yang berantaraksi adalah senya-wa uji dan senyasenya-wa target. Selanjutnya dilakukan protonasi 3 dimensi untuk menambahkan muatan atom dan hidrogen pada senyawa. Untuk memas-tikan telah dilakukan protonasi 3 dimensi dilakukan pengecekan hingga tiap atom dipastikan memiliki nilai muatan (q). Data disimpan dalam file format mol2 (12).
Seperti halnya senyawa target, senyawa uji juga harus dipreparasi terlegih dahulu dengan cara membuat struktur 2 dimensi dari senyawa-senyawa tersebut dengan marvinsketch. Dalam membuat model senyawa harus dipastikan struktur yang dibuat adalah benar dan tidak terdapat ke-salahan sama sekali dengan melakukan penge-cekan pada marvinsketch pada perintah check structure. Seperti halnya pada reseptor senyawa uji juga dilakukan protonasi 3 dimensi untuk menambahkan muatan atom dan hidrogen pada senyawa uji. Kemudian dilakukan pencarian kon-formasi senyawa yang telah melewati proses minimisasi energi dengan metode AM1 dengan gradien 0,01. Dalam hal ini didapatkan 10 konfor-masi senyawa terbaik. Model senyawa ini disimpan dalam file format mol2.
Docking (redocking) dilakukan dengan
me-lakukan docking native ligand pada reseptor.
Native ligand dibuat seperti halnya pada preparasi
senyawa uji di atas.
Validasi metode docking yang dilakukan dengan re-docking native ligand pada binding site. Kemudian didapatkan nilai RMSD (root mean
square deviation) sebesar 1,0410 yang berarti
me-tode tersebut memiliki nilai validitas yang tinggi yang dibuktikan dengan nilai RMSD < 2, artinya posisi ligand copy mirip dengan native ligand. Dalam hal ini diperlihatkan pada gambar 2.
Proses re-docking native ligand terhadap COX-2 dapat digunakan untuk mengidentifikasi
binding site dari enzim tersebut. Binding site tersebut teridentifikasi berupa residu-residu asam
amino yang berada pada jarak 5 Å dari native
ligand. Binding site ini ditentukan dengan
menentukan koordinat dimana letak native ligand
berada yaitu pada radius 5 Å dengan koordinat x=23.6651, y=23.3126, z= 47.8265 dan pada
binding radius=11.3323.
Gambar 2. Pose native ligand S558 dan pose prediksi docking dari simulasi docking. Native ligand (atom karbon diperlihatkan dengan warna biru muda), pose prediksi docking dari simulasi docking (atom karbon diperlihatkan dengan warna ungu). Atom Nitrogen diperlihatkan dengan warna biru, atom oksigen diperlihatkan pada warna merah ,atom Sulfur diperlihatkan dengan warna hijau dan atom Hidrogen diperlihatkan dengan warna putih. Dengan nilai RMSD = 1.0410 Å
Sekuens-sekuens asam amino yang ter-dapat pada binding site yang telah dipreparasi adalah Pro86, Asn87, Val89, His90, Leu93, Thr94, Met113, Val116, Leu117, Arg120, Gln192, Ser194, Phe198, Val344, Ile345, Glu346, Asp347, Tyr348, Val349, Gln350, His351, Leu352, Ser353, Gly354, Tyr355, His356, Phe357, Leu359, Phe381, Leu384, Tyr385, Trp387, Val434, Ala435, Leu507, Arg513, Ala516, Ile517, Phe518, Gly519, Glu520, Thr521, Met522, Val523, Hlu524, Leu525, Gly526, Ala527, Pro528, Phe529, Ser530, Leu531, Lys532, Lu534. Dengan yang teridentifikasi sebagai
binding pocket adalah His90, Leu117, Val349,
Leu352, Ser353, Trp387, Ala516, Phe518, Met522, Val523, Gly526, Ala527, Ser530, dan Leu534. Serta Asam amino yang membentuk ikat-an hidrogen yaitu Arg120, Leu352 dan dengan Arg513 yang memiliki energi yang rendah dalam
binding pocket dari nativeligand. Dengan demikian binding site yang telah dilakukan preparasi memenuhi syarat untuk dilakukan docking untuk senyawa-senyawa uji selanjutnya (13).
Hasil docking terhadap COX-2 menunjuk-kan terjadinya interaksi dengan binding pocket dari model enzim tersebut. Hal ini dapat dibuktikan dengan melihat adanya interaksi oleh hampir semua senyawa uji yang ditandai dengan adanya ikatan hidrogen dengan salah satu residu asam amino yang terdapat pada binding pocket. Hasil
docking ini diperlihatkan pada tabel 2 dan tabel 3. Gambaran interaksi hasil re-docking SC558 diper-lihatkan pada gambar 3.
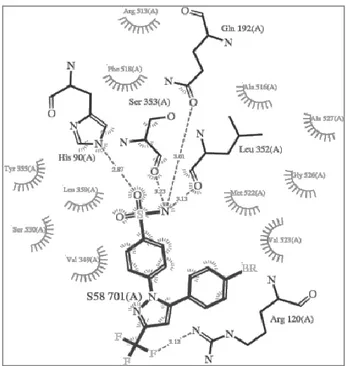
Gambar 3. Interaksi SC558 (bola dan batang) dengan residu siklooksigenase-2 Ser353 dan His90 (batang)
Hasil menunjukkan bahwa SC558 dapat mengikat 2 katalitik residu dari siklooksigenase-2, yakni His90 dan Ser353 melalui ikatan hidrogen. Hasil redocking SC558 dibandingkan dengan interaksi yang terjadi antara native ligand dengan COX-2 (gambar 3) meskipun memiliki struktur yang berbeda dengan native ligand, tetapi ikatan hidrogen yang terjadi antara re-docking SC558 dengan COX-2 juga terjadi pada interaksi native
ligand dengan COX-2. Hal ini menunjukkan bahwa
re-docking SC558 mampu menempati posisi yang
sama dengan native ligand.
Gambar 3. Interaksi native ligand (SC558) dengan residu siklooksigenase-2 (PDB ID=6COX) (sumber : Kurumbail, R.G, et all. Ligand S58-1-phenylsulfonamide-3-trifluoromethyl-5-parabromophenylpyrazole.1996. From:http://www.ebi.ac.uk/thorntonsrv/databases/cgibin/ pdbsum/GetPage.pl?pdbcode=6cox&template=ligands.h tml&l=3.1)
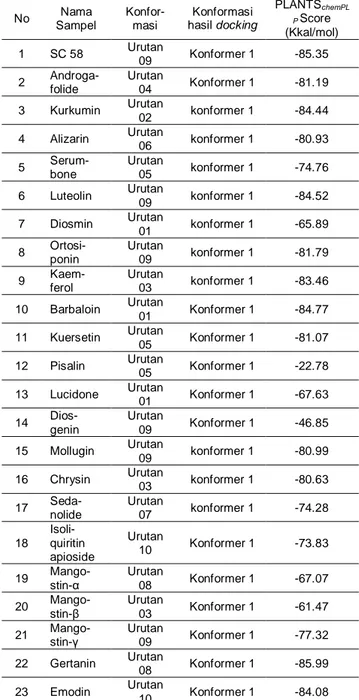
Tabel 2. Hasil docking (konformasi tebaik dengan skoring docking) No Nama Sampel Konfor-masi Konformasi hasil docking PLANTSchemPL P Score (Kkal/mol) 1 SC 58 Urutan 09 Konformer 1 -85.35
2 Androga-folide Urutan 04 Konformer 1 -81.19
3 Kurkumin Urutan
02 konformer 1 -84.44
4 Alizarin Urutan
06 konformer 1 -80.93
5 Serum-bone Urutan 05 konformer 1 -74.76
6 Luteolin Urutan
09 konformer 1 -84.52
7 Diosmin Urutan
01 konformer 1 -65.89
8 Ortosi-ponin Urutan 09 konformer 1 -81.79
9 Kaem-ferol Urutan 03 konformer 1 -83.46 10 Barbaloin Urutan 01 Konformer 1 -84.77 11 Kuersetin Urutan 05 Konformer 1 -81.07 12 Pisalin Urutan 05 Konformer 1 -22.78 13 Lucidone Urutan 01 Konformer 1 -67.63 14 Dios-genin Urutan 09 Konformer 1 -46.85 15 Mollugin Urutan 09 konformer 1 -80.99 16 Chrysin Urutan 03 konformer 1 -80.63 17 Seda-nolide Urutan 07 konformer 1 -74.28 18 Isoli-quiritin apioside Urutan 10 Konformer 1 -73.83
19 Mango-stin-α Urutan
08 Konformer 1 -67.07
20 Mango-stin-β Urutan
03 Konformer 1 -61.47
21 Mango-stin-γ Urutan
09 Konformer 1 -77.32
22 Gertanin Urutan
08 Konformer 1 -85.99
23 Emodin Urutan
10 Konformer 1 -84.08
Interaksi yang terjadi antara senyawa-senyawa antiinflamasi dengan enzim siklooksigen-ase-2 ditandai dengan docking score dan interaksi pose ikatan. Dalam hal ini diambil parameter hasil pengikatan energi Gibbs (ΔG) sebagai acuan, dan
docking score hanyalah digunakan sebagai para-meter untuk menilai pose pada konformasi yang memiliki nilai energi Gibbs yang terbaik (14). Da-lam hal ini diambil nilai terendah yaitu barbaloin dengan nilai energi gibs (ΔG=-17.8976 kkal/mol). Interaksi antara barbaloin dengan COX-2 ditandai dengan adanya 11 ikatan hidrogen yang terjadi dengan residu-residu yang ada didalam binding site dalam hal ini adalah 2 ikatan hidrogen pada Arg120 (gugus yang terikat adalah Hidroksil {OH} dan gugus karbonil {=O}, 3 ikatan hidrogen pada Tyr355 (gugus hidroksil), 2 ikatan hidrogen pada Met522 (gugus hidroksil), 1 ikatan hidrogen pada
Val523 (gugus hidroksil), 1 ikatan hidrogen pada Ala527 (gugus hidroksil), dan 2 ikatan hidrogen pada Ser530 (gugus hidroksil). Yang berarti bahwa barbaloin mampu mengikat daerah katalitik residu seperti yang ditampilkan pada gambar 27 di atas. Dengan ini dapat diambil kesimpulan bahwa se-nyawa barbaloin mempunyai kemungkinan memi-liki aktivitas penghambatan COX-2 yang lebih baik dari yang lainnya. Interaksi antara barbaloin de-ngan residu-residu asam amino pada COX-2 diper-lihatkan pada gambar 4.
Gambar 4. Ikatan hidrogen antara barbaloin (bola dan batang) dengan asam amino Arg120, Tyr355, Ser530 dan Met522 (batang)
KESIMPULAN
Hasil penelitian dari 22 senyawa bahan alam dapat mengikat COX-2 ditunjukkan dengan energi dan interaksi ikatan hidrogen (ΔG). Hasil menunjukkan bahwa ikatan hidrogen (ΔG) terbaik yaitu senyawa barbaloin (-17,8976 kkal/mol), Inter-aksi antara barbaloin dengan COX-2 ditandai ngan adanya 11 ikatan hidrogen yang terjadi de-ngan residu-residu yang ada didalam binding site
dalam hal ini adalah 2 ikatan hidrogen pada Arg120 (gugus yang terikat adalah Hidroksil {OH} dan gugus karbonil {=O}, 3 ikatan hidrogen pada Tyr355 (gugus hidroksil), 2 ikatan hidrogen pada Met522 (gugus hidroksil), 1 ikatan hidrogen pada Val523 (gugus hidroksil), 1 ikatan hidrogen pada Ala527 (gugus hidroksil), dan 2 ikatan hidrogen pada Ser530 (gugus hidroksil) yang berarti bahwa barbaloin mampu mengikat daerah katalitik residu pada COX-2. Dari hasil ini dapat disimpulkan bahwa senyawa barbaloin memiliki potensi yang baik terhadap penghambatan COX-2.
SARAN
Docking protein-ligan dengan
mengguna-kan PLANTS dapat dilanjutkan dengan membatasi
docking pada asam amino yang penting dalam
interaksi dengan cara “chemPLP_hb_constraints” pada atom NH 7808 6 pada Arg513, di mana Arg513 mempunyai peranan penting dalam peng-hambatan COX-2.
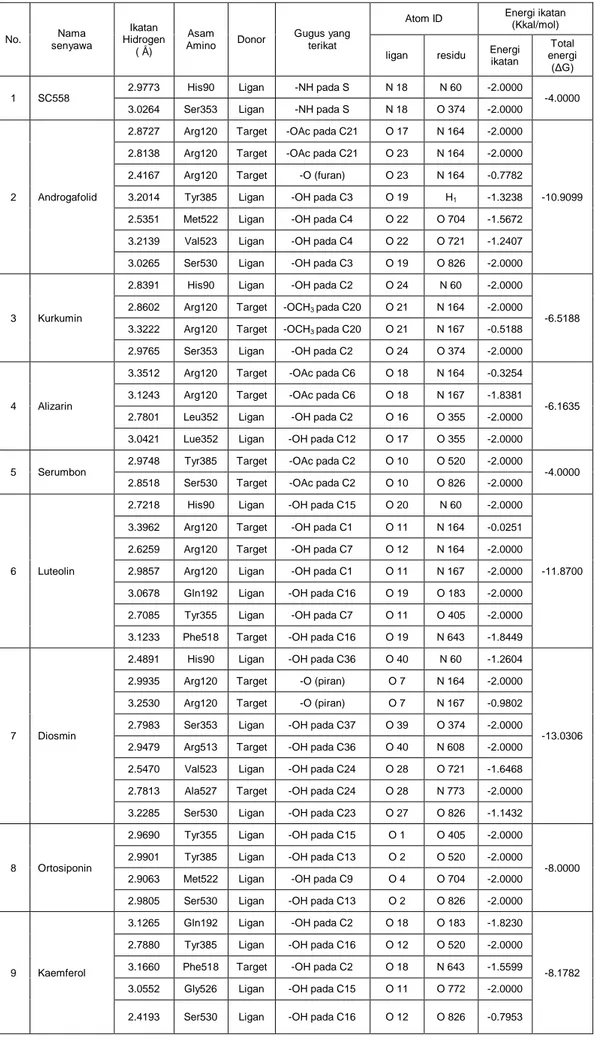
Tabel 3. Data analisis hasil docking konformasi terbaik senyawa-senyawa menggunakanMMV 2.0, pilihan PLANTS Score.
No. Nama senyawa Ikatan Hidrogen ( Å) Asam Amino Donor Gugus yang terikat
Atom ID Energi ikatan (Kkal/mol)
ligan residu Energi ikatan
Total energi (ΔG) 1 SC558
2.9773 His90 Ligan -NH pada S N 18 N 60 -2.0000
-4.0000 3.0264 Ser353 Ligan -NH pada S N 18 O 374 -2.0000
2 Androgafolid
2.8727 Arg120 Target -OAc pada C21 O 17 N 164 -2.0000
-10.9099 2.8138 Arg120 Target -OAc pada C21 O 23 N 164 -2.0000
2.4167 Arg120 Target -O (furan) O 23 N 164 -0.7782
3.2014 Tyr385 Ligan -OH pada C3 O 19 H1 -1.3238
2.5351 Met522 Ligan -OH pada C4 O 22 O 704 -1.5672
3.2139 Val523 Ligan -OH pada C4 O 22 O 721 -1.2407
3.0265 Ser530 Ligan -OH pada C3 O 19 O 826 -2.0000
3 Kurkumin
2.8391 His90 Ligan -OH pada C2 O 24 N 60 -2.0000
-6.5188 2.8602 Arg120 Target -OCH3 pada C20 O 21 N 164 -2.0000
3.3222 Arg120 Target -OCH3 pada C20 O 21 N 167 -0.5188
2.9765 Ser353 Ligan -OH pada C2 O 24 O 374 -2.0000
4 Alizarin
3.3512 Arg120 Target -OAc pada C6 O 18 N 164 -0.3254
-6.1635 3.1243 Arg120 Target -OAc pada C6 O 18 N 167 -1.8381
2.7801 Leu352 Ligan -OH pada C2 O 16 O 355 -2.0000
3.0421 Lue352 Ligan -OH pada C12 O 17 O 355 -2.0000
5 Serumbon
2.9748 Tyr385 Target -OAc pada C2 O 10 O 520 -2.0000
-4.0000 2.8518 Ser530 Target -OAc pada C2 O 10 O 826 -2.0000
6 Luteolin
2.7218 His90 Ligan -OH pada C15 O 20 N 60 -2.0000
-11.8700 3.3962 Arg120 Target -OH pada C1 O 11 N 164 -0.0251
2.6259 Arg120 Target -OH pada C7 O 12 N 164 -2.0000
2.9857 Arg120 Ligan -OH pada C1 O 11 N 167 -2.0000
3.0678 Gln192 Ligan -OH pada C16 O 19 O 183 -2.0000
2.7085 Tyr355 Ligan -OH pada C7 O 11 O 405 -2.0000
3.1233 Phe518 Target -OH pada C16 O 19 N 643 -1.8449
7 Diosmin
2.4891 His90 Ligan -OH pada C36 O 40 N 60 -1.2604
-13.0306 2.9935 Arg120 Target -O (piran) O 7 N 164 -2.0000
3.2530 Arg120 Target -O (piran) O 7 N 167 -0.9802
2.7983 Ser353 Ligan -OH pada C37 O 39 O 374 -2.0000
2.9479 Arg513 Target -OH pada C36 O 40 N 608 -2.0000 2.5470 Val523 Ligan -OH pada C24 O 28 O 721 -1.6468
2.7813 Ala527 Target -OH pada C24 O 28 N 773 -2.0000
3.2285 Ser530 Ligan -OH pada C23 O 27 O 826 -1.1432
8 Ortosiponin
2.9690 Tyr355 Ligan -OH pada C15 O 1 O 405 -2.0000
-8.0000 2.9901 Tyr385 Ligan -OH pada C13 O 2 O 520 -2.0000
2.9063 Met522 Ligan -OH pada C9 O 4 O 704 -2.0000
2.9805 Ser530 Ligan -OH pada C13 O 2 O 826 -2.0000
9 Kaemferol
3.1265 Gln192 Ligan -OH pada C2 O 18 O 183 -1.8230
-8.1782 2.7880 Tyr385 Ligan -OH pada C16 O 12 O 520 -2.0000
3.1660 Phe518 Target -OH pada C2 O 18 N 643 -1.5599
3.0552 Gly526 Ligan -OH pada C15 O 11 O 772 -2.0000
No. Nama senyawa Ikatan Hidrogen ( Å) Asam Amino Donor Gugus yang terikat
Atom ID Energi ikatan (Kkal/mol)
ligan residu Energi ikatan
Total energi (ΔG)
10 Barbaloin
2.6116 Arg120 Target -OH pada C16 O 6 N 164 -2.0000
-17.8976 3.1943 Arg120 Target -OAc pada C10 O 5 N 167 -1.3714
2.9986 Tyr355 Ligan -OH pada C4 O 5 O 405 -2.0000
2.8859 Tyr355 Ligan -OH pada C4 O 7 O 405 -2.0000
2.8556 Tyr385 Ligan -OH pada C20 O 4 O 520 -2.0000
2.5011 Met522 Ligan -OH pada C22 O 1 O 704 -1.3410
3.1267 Met522 Ligan -OH pada C19 O 3 O 704 -1.8217
2.3077 Val523 Ligan -OH pada C23 O 1 O 721 -0.0513
3.2032 Ala527 Target -OH pada C23 O 1 N 773 -1.3122
2.9566 Ser530 Ligan -OH pada C20 O 4 O 826 -2.0000
2.7006 Ser530 Ligan -OH pada C12 O 8 O 826 -2.0000
11 Kuersetin
2.7808 His90 Ligan -OH pada C13 O 21 N 60 -2.0000
-7.8450 3.0721 Gln192 Ligan -OH pada C12 O 20 O 183 -2.0000
3.1232 Phe518 Target -OH pada C12 O 20 N 38 -1.8450
3.1232 Phe518 Target -OH pada C12 O 20 N 38 -1.8450
2.9675 Ser530 Ligan -OH pada C8 O 17 O 826 -2.0000
12 Pisalin
3.1329 Arg120 Target -OH pada C24 O 9 N 164 -1.7809
-6.9411 2.8452 Arg120 Target -OH pada C17 O 9 N 167 -2.0000
2.9987 Tyr355 Target -O (piran) O 5 N 773 -2.0000
3.2260 Arg513 Ligan -OAc pada C12 H 59 O 826 -1.1602
13 Lucidon
2.9253 Val349 Ligan -OH pada C10 O (8) O 305 -2.0000
-4.6087 3.3087 Ser353 Target -OAc pada C1 O (8) N 369 -0.6087
2.8084 Met522 Ligan -OH pada C21 O 1 N 704 -2.0000
14 Diosgenin
3.0007 Arg120 Target -O (piran) O 1 N 164 -2.0000
-4.2468 3.3630 Arg120 Target -O (piran) O 1 N 167 -0.2468
3.0299 Trp387 Target -OH pada C27 O 2 N 534 -2.0000
15 Mollugin
2.8370 Tyr355 target -OH pada C1 O 18 O 405 -2.0000
-2.4602 3.3310 Arg120 either -OH pada C1 O 15 N 164 -0.4602
16 Chrysin
3.0460 Tyr385 Target -OH pada C13 O 17 O 520 -2.0000
-4.0000 3.0049 Ser530 Target -OH pada C13 O 17 O 826 -2.0000
17 Sedanoline 2.7552 Tyr385 Target -OAc pada C8 O 9 O 520 -2.0000 -4.0000 2.8314 Ser530 Target -OAc pada C8 O 9 O 826 -2.0000
18 Isoliquiritin apioside
2.8514 Arg120 Target -OH pada C11 O 26 N 164 -2.0000
-9.1890 2.8487 Tyr385 Ligan -OH pada C31 O 36 O 520 -2.0000
2.9057 Met522 Ligan -OH pada C21 O 34 O 704 -2.0000
3.2217 Ala527 Ligan -OH pada C12 O 25 O 778 -1.1886
3.0105 Ser530 Ligan -OH pada C31 O 36 O 826 -2.0000
19 Mangostin-α
3.2278 Arg120 Target -OH pada C12 O 14 N 164 -1.1478
-9.1478 2.9190 Arg120 Target -OH pada C12 O 14 N 167 -2.0000
2.8204 Tyr355 Ligan -OH pada C12 O 14 O 405 -2.0000
3.0768 Tyr385 Ligan -OH pada C8 O 22 O 520 -2.0000
3.0037 Ser530 Ligan -OH pada C8 O 22 O 826 -2.0000
20 Mangostin-β
2.5462 Tyr355 Ligan -OH pada C1 O 21 O 405 -1.6410
-3.0427 3.1898 Met522 Ligan -OH pada C14 O 20 O 704 -1.4017
DAFTAR PUSTAKA
1. Ganiswarna, S.G., 2007, Farmakologi dan
Terapi. ed. 7. Universitas Indonesia Press.
Jakarta, hal. 230
2. Zullies, I., Agung, E.N., dan Widyah, A., 2006.
Penekanan Ekspresi Enzim COX-2 pada Kultur Sel Raji oleh Ekstrak Kloroform Daun Cang-kring (Erythrinafusca Lour.), Fakultas Farmasi Universitas Gadjah Mada. Yogyakarta. 17 (2). hal. 85-90
3. Gehan, H.H., Ghaneya, S.H., Nahla, A.F., and Amal, Y., 2011, Design, Synthesis, and Docking Studies of Novel Diarylpyrazoline and Diaryl-soxazoline Derivatives of Expected Antiinflam-matory, and Analgesic Activities. Life Science
Journal. Vol 8(1).pp.487-496
4. Quinion, M., 2011, World Wide Words : In Silico. [Serial on the internet]. 2006. Available from:http://www.worldwidewords.org/weirdword s/ww-ins1.html.accessed at 1 May 2011. 5. Kroemer R.T., 2003, Molecular Modelling
Prob-es : Docking and Scoring. J. Biochemical Socie-ty, 31(5). pp.980-981.
6. Shoichet, B.K., 2004, Virtual screening of Che-mical Libraries. Nature. Vol.432. pp. 862-865 7. Schapira, M., et al., 2003, Making virtual
screening a reality. PNAS. Vol. 100.pp.7354– 7359
8. Bissantz, C., Folkers, G., Rognan, D., 2000, Protein-based virtual screening of chemical databases. Evaluation of different docking/ scoring combination. J. Med. Chem. Vol. 43. pp.4759-4767
9. Barnard, M.J., and Drowns, M.G., 1998, Model-ling scene illumination colour for computer visi-on and image reproductivisi-on: A survey of com-putational approach. J. Chem. Inf. Comput. Sci. Vol. 38. pp.983-996
10. Bender, A., and Glen, C.R., 2005, A discussion of measure of enrichment in virtual screening: Comparing the information content of descript-ors with increasing levels of sophistication. J.
Chem. Inf. Model. 2005. Vol 45. pp. 1369-1375
11. Kurumbail, R.G., et al.,1996, Cyclooxygenase-2 (Prostaglandin Synthase-2 Complexed with a Selective Inhibittor), SC-558 IN I222 Space Goup.1996. Available from: http://www.pdb.org/ pdb/explore/explore.do?structureId=6COX Accessed at 1 may 2011
12. Prasojo, S.L., et al., 2010, Docking of 1-phenil- sulfonamide-3-trifluoromethyl-5-parabromo-phenyl-pyrazole to cyclooxygenase-2 using PLANTS. Indo J. chem. Vol 10 (3). pp 348-351 13. Jutti, L., As’ari N., Abdul, M., and Slamet, I.,
2010, Andrograpolide Inhibits COX-2 Express-ion in Human Fibroblast Cell Due to its Inter-action with Arginine and Histidine in Cyclo-oxygenase Site. Asian Network For Scientific
Information. Indonesia. Vol. 10.pp.1481-1484
14. Bissantz, C., et al., 2010, A Medicinal Chemist’s Guide to Molecular Interactions. J. Med. Chem. 2010. XXXX, XXX, 000–000 No. Nama senyawa Ikatan Hidrogen ( Å) Asam Amino Donor Gugus yang terikat
Atom ID Energi ikatan (Kkal/mol)
ligan residu Energi ikatan
Total energi (ΔG)
21 Mangostin-γ
3.2124 Arg120 Target -OH pada C11 O 1 O 164 -1.2572
-9.2572 2.8601 Arg120 Target -OH pada C11 O 1 O 167 -2.0000
2.8046 Tyr355 Ligan -OH pada C11 O 1 O 405 -2.0000
2.9155 Tyr385 Ligan -OH pada C3 O 5 O 520 -2.0000
3.0314 Ser530 Ligan -OH pada C3 O 5 O 826 -2.0000
22 Gertanin
3.0948 Arg120 Target -OH pada C4 O 23 N 164 -2.0000
-5.9344 2.5092 Tyr355 Ligan -OAc pada C8 O 21 O 405 -1.3944
2.9180 Ser530 Ligan -OH pada C1 O 28 O 826 -2.0000
23 Emodin
2.9789 Arg120 Target -OH pada C1 O 17 N 164 -2.0000
-5.8025 3.3054 Arg120 Target -OAc pada C7 O 16 N 167 -0.6305
2.4757 Leu352 Ligan -OH pada C13 O 14 O 355 -1.1715