Analisis Keragaman Genetik 41 Genotipe Cabai
(Capsicum annuum L.) Berdasarkan Marka SSR
(Genetic Diversity Analysis of 41 Chili Pepper Genotypes
[Capsicum annuum L.] Based on SSR Markers)
Wartono1,2, Suryo Wiyono2, Muhamad Syukur3, Giyanto2, Kristianto Nugroho1, dan Puji Lestari1 1Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian, Jl. Tentara Pelajar 3A, Bogor 16111 Indonesia
Telp. (0251) 8622833; Faks. (0251) 8622833; *E-mail: wartono72@pertanian.go.id, war.tono@yahoo.com 2
Departemen Proteksi Tanaman, Fakultas Pertanian, Institut Pertanian Bogor, Jl. Kamper, Kampus IPB Dramaga, Bogor 16680 Indonesia 3Departemen Agronomi dan Hortikultura, Fakultas Pertanian, Institut Pertanian Bogor, Jl. Meranti, Kampus IPB Dramaga, Bogor 16680 Indonesia
Diajukan: 2 Agustus 2019; Direvisi: 19 Oktober 2019; Diterima: 31 Oktober 2019
ABSTRACT
Chili (Capsicum annuum L.) is an important horticultural crop which possesses high genetic diversity. Their genetic diversity needs to be assessed through molecular marker-based analysis to facilitate chili breeding scheme. The objective of the study was to analyze the genetic diversity of 41 chili genotypes using Simple Sequence Repeat (SSR) markers. Eleven SSR markers were used to amplify all genotypes through PCR technique, visualized on Polyacrylamide Gel Electrophoresis (PAGE). The data were analyzed using NTSYSpc version 2.1 and PowerMarker version 3.25 softwares. Results showed that the average of SSR alleles was 7.9 with a range of 411 alleles per marker. All SSR markers showed PIC value >0.5, indicating their suitable use for chili genetic studies. Cluster analysis of 41 genotypes generated three main groups according to coefficient similarity of 77.4%. The genetic clusters appeared as reflection of the genetic background of each genotype. In addition, the SSR markers used can identify potential parent as indicated by the genetic distance between genotypes. The highest genetic diversity (0.698) was demonstrated by combinations of Yuni × Ayesha, Yuni × Ayesha 2, and Yuni × Namira. The estimated genetic diversity produced by SSR markers in this study should be useful as guidance for chili breeders to select suitable parents for new cultivar development for high productivity and resistance to diseases.
Keywords: Capsicum annuum, chili, genetic diversity, SSR markers, plant breeding.
ABSTRAK
Cabai (Capsicum annuum L.) merupakan tanaman hortikultura penting yang mempunyai keanekaragaman genetik tinggi. Keragaman genetik cabaiperlu dievaluasi melalui analisis berbasis marka molekuler untuk memfasilitasi program pemuliaan cabai. Penelitian ini bertujuan menganalisis diversitas genetik 41 genotipe cabaidengan marka Simple Sequence Repeat (SSR). Sebelas marka SSR digunakan untuk mengamplifikasi semua genotipe dengan teknik PCR, produk PCR kemudian divisualisasikan dengan menggunakan Polyacrylamide Gel Electrophoresis (PAGE). Data dianalisis dengan perangkat lunak NTSYSpc versi 2.1 dan PowerMarker versi 3.25. Hasil penelitian menunjukkan bahwa jumlah alel rata-rata yang terdeteksi adalah 7,9 dengan kisaran 411 alel per marka. Semua marka SSR menunjukkan nilai PIC >0,5, mengindikasikan bahwa marka tersebut sesuai untuk analisis diversitas genetik cabai. Analisis klaster menunjukkan bahwa 41 genotipe cabai terbagi menjadi tiga kelompok utama berdasarkan koefisien kesamaan genetik 77,4%. Kelompok genetik ini merupakan refleksi dari latar belakang genetik setiap genotipe. Selain itu, marka SSR yang digunakan juga dapat mengidentifikasi tetua potensial yang ditunjukkan dengan diidentifikasinya genotipe dengan jarak genetik jauh. Nilai jarak genetik terjauh (0,698) terdapat pada kombinasi Yuni × Ayesha, Yuni × Ayesha 2, dan Yuni × Namira. Estimasi diversitas genetik yang dihasilkan oleh marka SSR pada penelitian ini dapat digunakan sebagai panduan bagi pemulia cabai untuk memilih tetua yang sesuai untuk perakitan varietas baru dengan produktivitas tinggi dan tahan penyakit.
Kata kunci:Capsicum annuum, cabai, keragaman genetik, marka SSR, pemuliaan tanaman.
Hak Cipta © 2019, BB Biogen
PENDAHULUAN
Cabai (Capsicum annuum L.) merupakan
komo-ditas sayuran penting di dunia, dengan luas tanam total 3,7 juta hektar dan produksi 33,4 juta ton per tahun (FAOSTAT 2011). Di Indonesia, luas panen cabai hingga tahun 2018 mencapai 136,857 hektar dengan produksi 1.067.373 ton (Direktorat Jenderal Hortikultura 2019).
Pengembangan varietas unggul baru (VUB) ca-bai produktivitas tinggi masih terus dilakukan meng-ingat produktivitas cabai nasional masih tergolong rendah, berkisar antara 8,4–8,8 t/ha di bawah potensi hasilnya yang dapat mencapai 20 t/ha (Syukur et al.
2010). Selain itu, diperlukan juga varietas yang tahan terhadap penyakit utama cabai yang disebabkan oleh
Phytophthora sp. (Gómez-Rodríguez et al. 2017) untuk mengimbangi dinamika perubahan virulensi patogen yang sangat tinggi. Ketersediaan sumber daya genetik (SDG) dan metode pemuliaan yang tepat merupakan hal penting dalam merakit VUB (Sutjahjo et al. 2015). SDG cabai dengan karakter unggul dapat diperoleh dari varietas lokal, varietas unggul nasional, galur-galur percobaan, genotipe introduksi, dan tanaman liar sekerabat. Karakter
unggul tersebut dapat saling dikombinasikan
antargenotipe melalui program pemuliaan terstruktur sehingga diperoleh genotipe dengan gabungan karakter unggul.
Analisis keragaman genetik dan kekerabatan, baik antarspesies maupun antargenotipe, mempunyai andil besar dalam mendukung keberhasilan program pemuliaan tanaman karena dapat mengelompokkan SDG berdasarkan kesamaan genetiknya dan dapat digunakan untuk menentukan tetua persilangan (Mahatma et al. 2009). Penentuan kombinasi tetua persilangan dapat dilakukan melalui analisis kera-gaman genetik menggunakan marka morfologi dan molekuler. Penggunaan marka molekuler (DNA) mendukung marka morfologi karena marka DNA tidak dipengaruhi oleh faktor lingkungan (Collard et al. 2005; Wang et al. 2006).
Berbagai jenis marka DNA telah banyak diguna-kan untuk analisis diversitas genetik cabai, seperti
Restriction Fragment Length Polymorphisms (RFLPs),
Random Amplified Polymorphic DNA (RAPD),
Amplified Fragment Length Polymorphism (AFLP), dan Simple Sequence Repeat (SSR) (Jang et al. 2004; Lee et al. 2004). SSR merupakan salah satu marka yang cukup sederhana dengan tingkat polimorfisme yang tinggi, reprodusibel, dan bersifat kodominan se-hingga sesuai untuk analisis kekerabatan (Becher et al. 2000). Informasi genetik yang dihasilkannya dapat
digunakan sebagai petunjuk dalam program pemulia-an (Asmono et al. 2005). Penggunapemulia-an marka SSR untuk mengkaji keragaman genetik cabai dari bera-gam spesies telah dilaporkan oleh peneliti sebelum-nya, di antaranya Dhaliwal et al. (2014), Sharmin et al. (2018), dan Terryana et al. (2018). Sementara, Khan et al. (2015) menggunakan marka SSR untuk menyeleksi ketahanan varietas padi terhadap penya-kit hawar daun bakteri. Tasma et al. (2018) menggu-nakan marka SSR untuk menganalisis diversitas gene-tik aksesi kedelai Indonesia dan introduksi dari Amerika Serikat pada program pemuliaan tanaman kedelai produktivitas tinggi dengan karakter juvenil panjang (long juvenile character). Selain itu, marka SSR juga digunakan untuk menentukan tetua persi-langan tanaman padi (Wang et al. 2006). Tujuan penelitian ini ialah menganalisis diversitas genetik 41 genotipe cabaidengan marka SSR.
BAHAN DAN METODE
Penelitian dilaksanakan pada bulan Desember 2018 sampai dengan Februari 2019. Persiapan tanam-an dtanam-an tanam-analisis molekuler masing-masing dilakuktanam-an di rumah kaca dan Laboratorium Biologi Molekuler, Balai Besar Penelitian dan Pengembangan Biotek-nologi dan Sumber Daya Genetik Pertanian.
Materi Genetik
Genotipe cabai yang digunakan untuk analisis keragaman genetik sebanyak 41 genotipe, terdiri atas 38 genotipe asal Indonesia dan 3 genotipe introduksi (Tabel 1). CM334 dan Jalapeno masing-masing meru-pakan cabai introduksi dari Meksiko yang sering di-gunakan sebagai cek tahan dan cek rentan dalam uji ketahanan terhadap P. capsici (Candole et al. 2012). Sementara itu, Giant A merupakan cabai introduksi dari Argentina dengan bentuk buah yang besar, tetapi belum diketahui ketahanannya terhadap P. capsici. Tipe cabai yang digunakan dalam pengujian ini terdiri atas cabai rawit, rawit hias, cabai besar, dan cabai keriting.
Isolasi DNA Genomik dan Pengukuran Kuantitatif dan Kualitatif DNA
Isolasi DNA genomik dilakukan dengan meng-gunakan metode Doyle dan Doyle (1990) yang dimo-difikasi dengan penambahan polyvinylpyrrolidone
(PVP) dan natrium disulfit. Sebanyak 0,5 g potongan daun cabai digerus dalam tabung mikro 2 ml meng-gunakan blue pestle steril.
Selanjutnya, dilakukan penambahan bufer ekstraksi sebanyak 800 μl yang mengandung Tris-HCl
100 mM (pH 8,0), NaCl 1,4 M, EDTA 20 mM (pH 8,0),
cetyltrimethylammonium bromide (CTAB) 2% (w/v), PVP 2% (w/v), dan natrium disulfit 0,38% (w/v). Sampel diinkubasi pada 65ºC selama 15 menit dan di-homogenkan dengan cara dibolak-balik setiap 5 me-nit. Sampel kemudian diekstraksi dengan 800 μl larut-an kloroform:isoamil alkohol (24:1) dlarut-an disentrifugasi dengan kecepatan 12.000 rpm selama 10 menit. Se-banyak 600 μl supernatan yang terbentuk selanjutnya dipindahkan ke tabung mikro 1,5 ml yang baru, diikuti dengan penambahan natrium asetat 3 M (pH 5,2) dan isopropanol dingin masing-masing sebanyak 1/10 dan 1 kali volume supernatan. Larutan campuran ke-mudian diinkubasi pada -20ºC selama 1 jam. Setelah itu, sampel disentrifugasi kembali dengan kecepatan 12.000 rpm selama 10 menit. Supernatan kemudian dibuang dan pelet yang terbentuk dibilas dengan 200
μl etanol 70% untuk menghilangkan sisa-sisa garam. Sampel disentrifugasi kembali dengan kecepatan 12.000 rpm selama 5 menit. Pelet yang telah bersih di-keringanginkan selama semalam dan dilarutkan dalam 100 μl larutan TE (Tris 10 mM [pH 8,0], EDTA 1 mM) dan ditambah 2 μl RNase (10 mg/ml). Larutan stok DNA tersebut kemudian diinkubasi pada 37ºC selama 1 jam.
Pengukuran kuantitatif larutan stok DNA
genomik dilakukan dengan Nanodrop™ 2000
Spectrophotometer (Thermo Scientific, AS). Semen-tara, pengukuran kualitatifnya dilakukan dengan teknik elektroforesis pada gel agarosa 1%.
PCR, Elektroforesis, dan Deteksi Pita Marka SSR
PCR dilakukan dengan menggunakan sebelas marka SSR yang berasal dari beberapa referensi (Tabel 2) (Lee et al. 2004; Minamiyama et al. 2007; Nagy et al. 2007). Reaksi PCR dilakukan dengan volume total 10 µl yang terdiri atas DNA template (10 ng/μl) sebanyak 2 μl, MyTaq™ HS Ready Mix (Bioline, UK) sebanyak 5 μl, primer forward dan reverse (10
μM) masing-masing sebanyak 0,5 μl, dan ddH2O steril.
Proses reaksi PCR dilakukan pada mesin T1
Thermocycler (Biometra, Jerman) dengan profil PCR sebagai berikut: denaturasi awal pada 95ºC selama 5 menit, diikuti 35 siklus yang terdiri atas denaturasi pada 94ºC selama 30 detik, annealing pada 55ºC selama 1 menit, dan ekstensi pada 72ºC selama 1 menit. Reaksi PCR diakhiri dengan tahap ekstensi akhir pada 72ºC selama 7 menit.
Hasil amplifikasi PCR selanjutnya dielektrofore-sis pada gel poliakrilamida 6% yang dibuat dengan mencampurkan 50 ml acrylamide:bis-acrylamide 6% (19:1) dengan 500 μl amoniumpersulfat (APS) dan 50
μl TEMED. Elektroforesis dilakukan dengan tegangan 90 volt pada tangki berisi bufer Tris-borate-EDTA
(TBE) 1 selama 2 jam.
Untuk mendeteksi pita marka SSR, hasil elektroforesis sebelas marka SSR dan DNA genomik 41 genotipe cabai kemudian diwarnai dengan etidium bromida dan divisualisasi dengan ChemiDoc EQ™ System(Bio-Rad, AS).
Tabel 1. Genotipe cabai (C. annuum) yang digunakan pada penelitian ini.
No. Genotipe Tipe cabai Status Negara asal No. Genotipe Tipe cabai Status Negara asal 1. Ayesha Rawit hias Varietas Indonesia 22. Violeta 1 Rawit hias Varietas Indonesia 2. Laris Keriting Varietas Indonesia 23. Giant A Besar Varietas Argentina 3. Bara Rawit Varietas Indonesia 24. C5 Besar Varietas Indonesia 4. CM334 Besar Landrace Meksiko 25. Ayesha Ungu Rawit hias Varietas Indonesia 5. Violeta Rawit hias Varietas Indonesia 26. F5 Syak × 320-202-2-2 Rawit hias Galur Indonesia 6. Lembayung Rawit hias Varietas Indonesia 27. F5 Syak × 320-202-2-1 Rawit hias Galur Indonesia 7. SSP Keriting Varietas Indonesia 28. F5 Syak × 320-201-1-1 Rawit hias Galur Indonesia 8. Jalapeno Besar Varietas Meksiko 29. Ayesha 2 Rawit hias Varietas Indonesia 9. Genie Rawit Varietas Indonesia 30. Namira Rawit hias Varietas Indonesia 10. F6 145291-10-7-1-1-1 Rawit Galur Indonesia 31. Lokal Lembang Keriting Varietas Indonesia 11. F8 145318-1-1-1-1-3-1 Rawit hias Galur Indonesia 32. Tanjung-2 Besar Varietas Indonesia 12. F8 145291-115-8-1-1 Rawit Galur Indonesia 33. Vitra Keriting Varietas Indonesia 13. F8 160291-9-4-3-2-1-1 Rawit Galur Indonesia 34. Lingga Besar Varietas Indonesia 14. F8 160291-3-12-5-4-51-1 Rawit Galur Indonesia 35. Kencana Keriting Varietas Indonesia 15. Landung Besar Varietas Indonesia 36. Cakri Andalas Keriting Varietas Indonesia 16. Anies Keriting Varietas Indonesia 37. Lembang 1 Keriting Varietas Indonesia 17. Nazla Rawit hias Varietas Indonesia 38. Ciko Besar Varietas Indonesia 18. Seloka-5 Besar Varietas Indonesia 39. CL-4 Rawit Varietas Indonesia 19. Yuni Keriting Varietas Indonesia 40. Sempurna Besar Varietas Indonesia 20. Ungara Rawit hias Varietas Indonesia 41. Rama Rawit Varietas Indonesia 21. Viola Rawit hias Varietas Indonesia
Analisis Data
Pita marka SSR yang muncul pada gel dianggap sebagai satu alel. Setiap pita yang muncul diberi nilai 1 dan yang tidak muncul diberi nilai 0 sehingga hasil skoring pita berupa data biner. Nilai Polymorphic Information Content (PIC) dihitung berdasarkan per-samaan:
dengan PICj adalah nilai PIC pada marka ke-j, Pi adalah frekuensi alel ke-i pada marka ke-j, dan n adalah jumlah alel pada marka ke-j (Hildebrand et al. 1992).
Data hasil skoring dianalisis dengan prosedur
Unweighted Pair-Group Method with Arithmetic
-Sequential Agglomerative Hierarchical and Nested
(UPGMA-SAHN) menggunakan perangkat lunak
NTSYSpc versi 2.1, ditampilkan dalam bentuk dendrogram dan tabel matriks jarak genetik (Rohlf 2000). Untuk mengetahui nilai frekuensi alel utama, diversitas genetik, dan nilai PIC, data hasil skoring dianalisis dengan perangkat lunak PowerMarker versi 3.25 (Liu dan Muse 2005).
HASIL DAN PEMBAHASAN
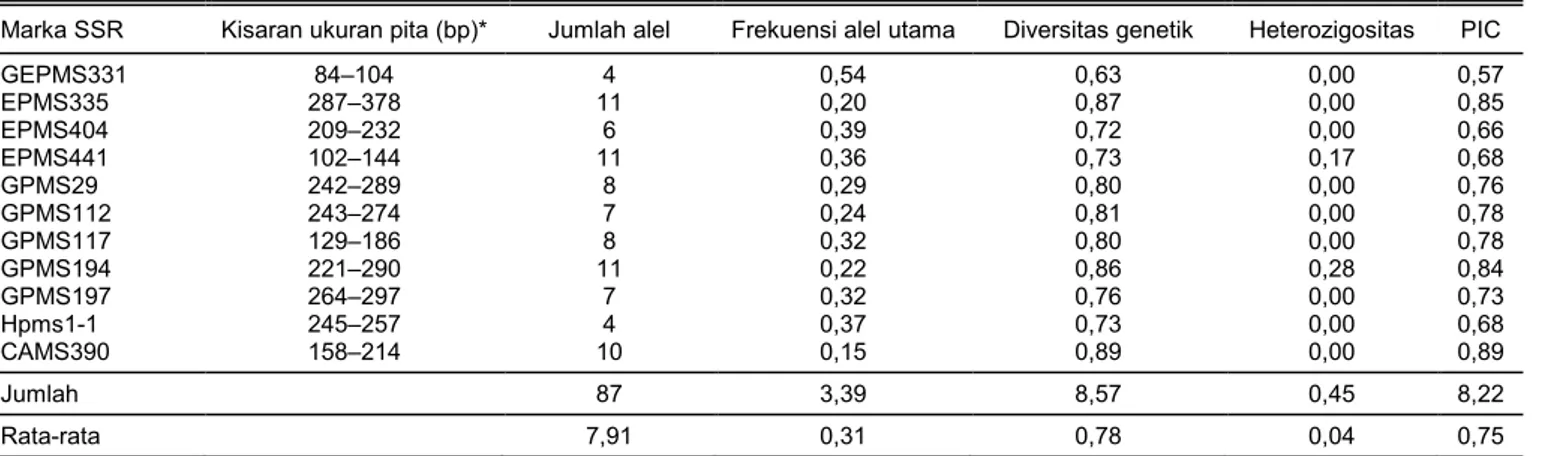
Polimorfisme Marka SSR pada 41 Genotipe Cabai
Sebanyak sebelas marka SSR yang digunakan pada penelitian ini menghasilkan pita polimorfik pada
41 genotipe cabai yang dianalisis. Contoh pola pita dan distribusi alel per lokus SSR ditunjukkan pada
elektroforegram marka EPMS441 (Gambar 1).
Sementara, karakteristik sebelas marka SSR hasil uji
genotyping ditampilkan pada Tabel 3. Sebanyak 87 alel terdeteksi, berkisar antara 4–11 alel per lokus SSR dengan rata-rata 7,9 alel per lokus. Jumlah alel yang terdeteksi pada penelitian cukup tinggi dan lebih tinggi dibanding dengan hasil penelitian Carvalho et al. (2015) yang mendeteksi 55 alel berdasarkan 19 marka SSR yang diuji pada 4 genotipe cabai. Namun, jumlah alel tersebut lebih rendah daripada hasil penelitian Terryana et al. (2018) dengan total 179 alel berdasarkan 24 marka SSR yang diuji pada 27 genotipe cabai. Frekuensi alel utama rata-rata yang diperoleh pada penelitian ini sebesar 31% dengan nilai tertinggi 53% (EPMS331) dan terendah 14% (CAMS390). Nilai tersebut lebih rendah dibanding dengan hasil penelitian Oh et al. (2012) dan Terryana et al. (2018) yang masing-masing sebesar 32% pada analisis menggunakan 22 marka SSR dan 24 marka SSR yang diuji pada 61 genotipe cabai dan 27 genotipe cabai.
Dari sebelas marka yang digunakan, terdapat dua marka yang dapat membedakan alel heterozigot di antara 41 genotipe cabai yang diuji dengan nilai heterozigositas berturut-turut 0,17 (EPMS441) dan 0,28 (GPMS194). Adanya alel heterozigot ini dapat di-sebabkan oleh karakteristik cabai yang termasuk tanaman menyerbuk silang yang memengaruhi level heterozigositasnya. Sementara, nilai diversitas gen
Tabel 2. Sebelas marka SSR dan urutan primer yang digunakan pada penelitian ini.
Marka SSR Sekuen 5’3’ Ta (ºC) Referensi
EPMS331 F: AACCCAATCCCCTTATCCAC R: GCATTAGCAGAAGCCATTTG 53 Nagy et al. (2007) EPMS335 F: ATGCAGAGATTGTCGAAGCC R: GCAGAGAAGACTCACCAGTCC 53 Nagy et al. (2007) EPMS404 F: TCTCTCTCTACATCTCTCCGTTG R: TGTCGTTCGTCGACGTACTC 50 Nagy et al. (2007) EPMS441 F: GCACGAGGAAAGAGAGAGACATAG R: TCAACGGATTCAGTCTTCCC 53 Nagy et al. (2007) GPMS29 F: CAGGCAATACGGAGCATC R: TGTGTTGCTTCTTGGACGAC 53 Nagy et al. (2007) GPMS112 F: TCCCTCAGCAGCAACAATTT R: GTCGGGCTCTTTGATTGTGT 50 Nagy et al. (2007) GPMS117 F: GATGTTAGGTCCGTGCTTCG R: AAGCCCCATGGAAGTTATCC 53 Nagy et al. (2007) GPMS194 F: AGGTGGCAGTTGAGGCTAAG R: GTTCTAGGTCTTTGCCCTGG 50 Nagy et al. (2007) GPMS197 F: GCAGAGAAAATAAAATTCTCGG R: CAATGGAAATTTCATCGACG 53 Nagy et al. (2007) Hpms1-1 F: TCAACCCAATATTAAGGTCACTTCC R: CCAGGCGGGGATTGTAGATG 53 Lee et al. (2004) CAMS390 F: CTGTTCTCCTCCCTCCCTCT R: TGAAGCAAGAAACTGAACAATCA 53 Minamiyama et al. (2007) Ta = annealing temperature pasangan primer.
n i 1 2Pi
1
jPIC
rata-rata yang diperoleh sebesar 0,78 dengan nilai
terendah 0,63 (EPMS331) dan tertinggi 0,90
(CAMS390). Nilai rata-rata ini lebih tinggi dibanding dengan hasil penelitian Oh et al. (2012) yang sebesar 0,15. Adanya perbedaan jumlah alel, frekuensi alel, dan diversitas gen kemungkinan terjadi karena jumlah dan jenis primer SSR serta genotipe cabai yang digunakan pada penelitian ini berbeda diban-ding dengan penelitian sebelumnya. Varietas unggul dan galur-galur cabai hasil persilangan yang berasal dari Indonesia dan hasil introduksi dari negara lain diduga menjadi salah satu faktor yang menentukan tingginya diversitas gen yang dihasilkan pada peneliti-an ini.
Seluruh marka SSR yang digunakan menghasil-kan nilai PIC yang berkisar antara 0,57–0,89 dengan rata-rata 0,75. Hal ini menunjukkan bahwa seluruh marka SSR yang digunakan pada penelitian ini sangat
informatif. Menurut Botstein et al. (1980), marka sangat informatif bila nilai PIC >0,5, sedang bila 0,5> PIC >0,25, dan kurang informatif bila PIC <0,25. Nilai PIC yang diperoleh pada penelitian ini tidak jauh berbeda dengan hasil penelitian Terryana et al. (2018) yang menghasilkan 76% polimorfisme dengan 24 marka SSR pada 27 genotipe cabai. Namun demikian, penelitian ini menghasilkan nilai PIC lebih tinggi daripada penelitian sebelumnya. Sebagai contoh, Oh et al. (2012) menghasilkan 33% polimorfisme dengan 22 marka SSR pada 66 genotipe cabai dan Sharmin et al. (2018) menghasilkan 37% polimorfisme dengan 20 marka SSR pada 20 genotipe cabai.
Nilai PIC marka DNA dapat digunakan sebagai dasar dalam memilih marka yang dapat membeda-kan antargenotipe cabai. Nilai PIC tidak hanya di-tentukan oleh jumlah alel, tetapi juga frekuensi tiap alel dalam set genotipe yang diuji (Sharmin et al.
Gambar 1. Contoh pola pita marka EPMS441hasil elektroforesis pada gel poliakrilamida 6%. M = 100 bp ladder, 1 = Ayesha, 2 = Laris, 3 = Bara, 4 = CM334, 5 = Violeta, 6 = Lembayung, 7 = SSP, 8 = Jalapeno, 9 = Genie, 10 = F6 145291-10-7-1-1-1, 11 = F8 145318-1-1-1-1-3-1, 12 = F8 145291-115-8-1-1, 13 = F8 160291-9-4-3-2-1-1, 14 = F8 160291-3-12-5-4-51-1, 15 = Landung, 16 = Lokal Lembang (keriting), 17 = Tanjung-2, 18 = Vitra, 19 = Lingga, 20 = Kencana, 21 = Cakri Andalas, 22 = Lembang 1, 23 = Ciko, 24 = CL-4, 25 = Sempurna, 26 = Rama, 27 = Anies, 28 = Nazla, 29 = Seloka-5, 30 = Yuni, 31 = Ungara, 32 = Viola, 33 = Violeta 1, 34 = Giant A, 35 = C5, 36 = Ayesha Ungu, 37 = F5 Syak × 320-202-2-2, 38 = F5 Syak × 320-202-2-1, 39 = F5 Syak × 320-201-1-1, 40 = Ayesha 2, 41 = Namira.
Tabel 3. Ringkasan statistik marka SSR hasil analisis genotyping 41 genotipe cabai.
Marka SSR Kisaran ukuran pita (bp)* Jumlah alel Frekuensi alel utama Diversitas genetik Heterozigositas PIC
GEPMS331 84–104 4 0,54 0,63 0,00 0,57 EPMS335 287–378 11 0,20 0,87 0,00 0,85 EPMS404 209–232 6 0,39 0,72 0,00 0,66 EPMS441 102–144 11 0,36 0,73 0,17 0,68 GPMS29 242–289 8 0,29 0,80 0,00 0,76 GPMS112 243–274 7 0,24 0,81 0,00 0,78 GPMS117 129–186 8 0,32 0,80 0,00 0,78 GPMS194 221–290 11 0,22 0,86 0,28 0,84 GPMS197 264–297 7 0,32 0,76 0,00 0,73 Hpms1-1 245–257 4 0,37 0,73 0,00 0,68 CAMS390 158–214 10 0,15 0,89 0,00 0,89 Jumlah 87 3,39 8,57 0,45 8,22 Rata-rata 7,91 0,31 0,78 0,04 0,75
*Ukuran produk pita SSR yang dihasilkan. PIC = Polymorphism Information Content.
2018). Diversitas gen dan nilai PIC merepresentasikan jumlah alel dan distribusinya pada total genotipe cabai. Semakin banyak alel yang terdeteksi, semakin tinggi nilai PIC. Hal ini sejalan dengan hasil penelitian Yu et al. (2003). Tingginya nilai PIC rata-rata pada penelitian ini menunjukkan tingginya keragaman ge-netik genotipe cabai yang diuji. Berdasarkan hasil yang diperoleh, marka SSR yang paling potensial un-tuk menganalisis 41 genotipe cabai adalah CAMS390 dengan nilai PIC sebesar 0,89, diikuti EPMS335 dan GPMS194 masing-masing sebesar 0,85 dan 0,84. Hasil penelitian ini menunjukkan bahwa marka SSR yang tersebar dalam genom cabai terbukti mampu meng-estimasi keragaman genetik yang tinggi pada 41 genotipe yang diuji, relevan dengan hasil penelitian Zhang et al. (2016).
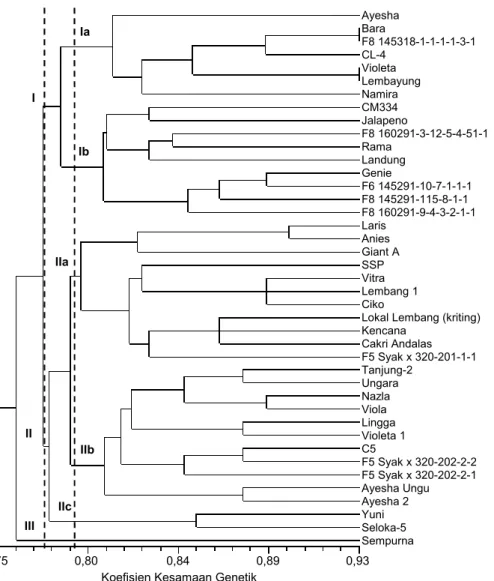
Hasil Analisis Filogenetik dan Jarak Genetik
Analisis filogenetik 38 genotipe cabaiIndonesia dan 3 genotipe introduksi berdasarkan sebelas marka SSR menghasilkan koefisien kesamaan genetik antara 77–93% (Gambar 2). Hal ini menunjukkan bahwa 41 genotipe yang dianalisis memiliki hubungan kekera-batan yang tinggi sebagai satu spesies cabai. Hasil pe-nelitian terdahulu menunjukkan bahwa jarak kemirip-an dikatakkemirip-an jauh apabila tingkat kemiripkemirip-annya ku-rang dari 60% (Trimanto 2012). Pada tingkat kesama-an 77,4% terbentuk tiga grup utama, yaitu grup I, grup II, dan grup III, yang masing-masing terdiri atas 16 genotipe, 24 genotipe, dan 1 genotipe.
Pada dendrogram terlihat bahwa genotipe cabai tidak mengelompok menurut tipe cabai. Tiap grup meliputi kombinasi genotipe dengan tipe cabai yang berbeda. Berdasarkan tipenya, genotipe cabai pada grup I terdiri atas 9 tipe cabai rawit, 4 rawit hias, dan 3
Gambar 2. Dendrogram kekerabatan 41 genotipe cabai berdasarkan hasil analisis menggunakan sebelas marka SSR. Ayesha Bara F8 145318-1-1-1-1-3-1 CL-4 Violeta Lembayung Namira CM334 Jalapeno F8 160291-3-12-5-4-51-1 Rama Landung Genie F6 145291-10-7-1-1-1 F8 145291-115-8-1-1 F8 160291-9-4-3-2-1-1 Laris Anies Giant A SSP Vitra Lembang 1 Ciko
Lokal Lembang (kriting) Kencana Cakri Andalas F5 Syak x 320-201-1-1 Tanjung-2 Ungara Nazla Viola Lingga Violeta 1 C5 F5 Syak x 320-202-2-2 F5 Syak x 320-202-2-1 Ayesha Ungu Ayesha 2 Yuni Seloka-5 Sempurna I II III Ia Ib IIa IIb IIc 0,75 0,80 0,84 0,89 0,93
cabai besar. Grup II terdiri atas 9 rawit hias, 10 cabai keriting, dan 5 cabai besar. Sementara, pada grup III hanya terdapat 1 genotipe cabai besar. Hasil peneliti-an menunjukkpeneliti-an bahwa pada koefisien kesamapeneliti-an genetik 77,4% tidak terlihat pengelompokan berdasar-kan tipe cabai. Namun, pada koefisien kesamaan ge-netik 79,3% pengelompokan cenderung berdasarkan kemiripan karakternya, seperti pada subgrup IIa yang didominasi oleh tipe cabai keriting (Laris, Anies, SSP, Vitra, Lembang 1, Lokal Lembang, Kencana, dan Cakri Andalas). Selain itu, subgrup Ib juga didominasi satu tipe cabai rawit (Genie, F6 145291-10-7-1-1-1, F8 145291-115-8-1-1, F8 160291-9-4-3-2-1-1, Rama, dan F8 160291-3-12-5-4-51-1). Keberadaan tipe cabai lain, CM334 dan Jalapeno pada subgrup Ib dan Giant A dan F5 Syak 320-201-1-1 pada subgrup IIa, kemung-kinan karena adanya kemiripan pada karakter lain yang perlu dianalisis lebih lanjut, seperti kandungan
capsaicin dan bentuk buah yang mengerut (keriting),
yang belum dapat dibedakan oleh marka SSR. Hasil penelitian ini menunjukkan bahwa meskipun secara morfologi berkerabat dekat, yaitu sama-sama C. annuum, genotipe tidak selalu mengelompok dalam satu klaster berdasarkan marka SSR. Hasil penelitian lain menunjukkan bahwa pengelompokan varietas kedelai berdasarkan marka SSR tidak mencerminkan karakter morfologi (Widaningsih et al. 2014). Ke-banyakan marka SSR mengukur variasi genetik dari urutan non-coding, hal inilah yang memungkinkan marka SSR tidak berdampak terhadap morfologi genotipe (Kwon et al. 2005). Secara umum, hasil penelitian ini menunjukkan bahwa marka SSR sangat aplikatif dan efektif dalam membedakan genotipe dalam spesies yang sama, C. annuum. Hasil ini didu-kung oleh pemanfaatan marka SSR pada koleksi genotipe C. annuum yang tidak hanya dari aspek keragaman genetik, tetapi juga struktur populasinya (Pacheco-Olvera et al. 2012; Nicolai et al. 2013).
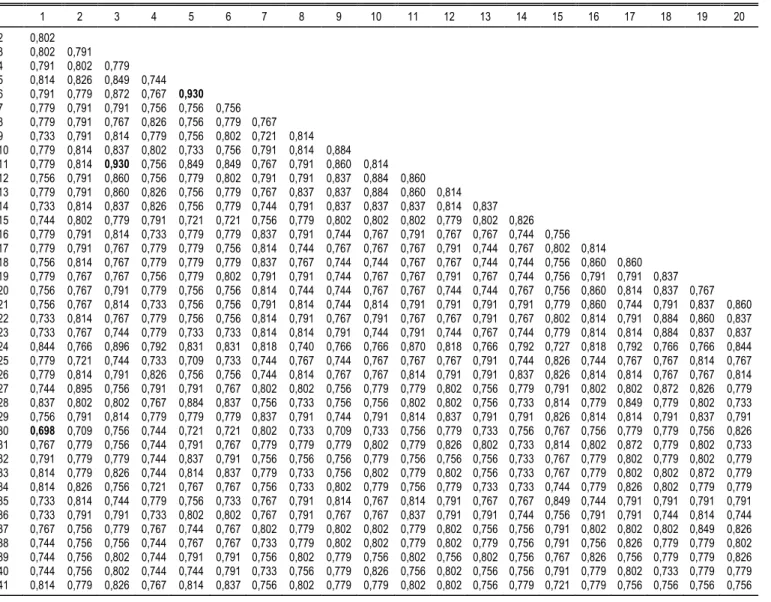
Tabel 4. Jarak genetik 41 genotipe cabai hasil analisis menggunakan sebelas marka SSR.
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 2 0,802 3 0,802 0,791 4 0,791 0,802 0,779 5 0,814 0,826 0,849 0,744 6 0,791 0,779 0,872 0,767 0,930 7 0,779 0,791 0,791 0,756 0,756 0,756 8 0,779 0,791 0,767 0,826 0,756 0,779 0,767 9 0,733 0,791 0,814 0,779 0,756 0,802 0,721 0,814 10 0,779 0,814 0,837 0,802 0,733 0,756 0,791 0,814 0,884 11 0,779 0,814 0,930 0,756 0,849 0,849 0,767 0,791 0,860 0,814 12 0,756 0,791 0,860 0,756 0,779 0,802 0,791 0,791 0,837 0,884 0,860 13 0,779 0,791 0,860 0,826 0,756 0,779 0,767 0,837 0,837 0,884 0,860 0,814 14 0,733 0,814 0,837 0,826 0,756 0,779 0,744 0,791 0,837 0,837 0,837 0,814 0,837 15 0,744 0,802 0,779 0,791 0,721 0,721 0,756 0,779 0,802 0,802 0,802 0,779 0,802 0,826 16 0,779 0,791 0,814 0,733 0,779 0,779 0,837 0,791 0,744 0,767 0,791 0,767 0,767 0,744 0,756 17 0,779 0,791 0,767 0,779 0,779 0,756 0,814 0,744 0,767 0,767 0,767 0,791 0,744 0,767 0,802 0,814 18 0,756 0,814 0,767 0,779 0,779 0,779 0,837 0,767 0,744 0,744 0,767 0,767 0,744 0,744 0,756 0,860 0,860 19 0,779 0,767 0,767 0,756 0,779 0,802 0,791 0,791 0,744 0,767 0,767 0,791 0,767 0,744 0,756 0,791 0,791 0,837 20 0,756 0,767 0,791 0,779 0,756 0,756 0,814 0,744 0,744 0,767 0,767 0,744 0,744 0,767 0,756 0,860 0,814 0,837 0,767 21 0,756 0,767 0,814 0,733 0,756 0,756 0,791 0,814 0,744 0,814 0,791 0,791 0,791 0,791 0,779 0,860 0,744 0,791 0,837 0,860 22 0,733 0,814 0,767 0,779 0,756 0,756 0,814 0,791 0,767 0,791 0,767 0,767 0,791 0,767 0,802 0,814 0,791 0,884 0,860 0,837 23 0,733 0,767 0,744 0,779 0,733 0,733 0,814 0,814 0,791 0,744 0,791 0,744 0,767 0,744 0,779 0,814 0,814 0,884 0,837 0,837 24 0,844 0,766 0,896 0,792 0,831 0,831 0,818 0,740 0,766 0,766 0,870 0,818 0,766 0,792 0,727 0,818 0,792 0,766 0,766 0,844 25 0,779 0,721 0,744 0,733 0,709 0,733 0,744 0,767 0,744 0,767 0,767 0,767 0,791 0,744 0,826 0,744 0,767 0,767 0,814 0,767 26 0,779 0,814 0,791 0,826 0,756 0,756 0,744 0,814 0,767 0,767 0,814 0,791 0,791 0,837 0,826 0,814 0,814 0,767 0,767 0,814 27 0,744 0,895 0,756 0,791 0,791 0,767 0,802 0,802 0,756 0,779 0,779 0,802 0,756 0,779 0,791 0,802 0,802 0,872 0,826 0,779 28 0,837 0,802 0,802 0,767 0,884 0,837 0,756 0,733 0,756 0,756 0,802 0,802 0,756 0,733 0,814 0,779 0,849 0,779 0,802 0,733 29 0,756 0,791 0,814 0,779 0,779 0,779 0,837 0,791 0,744 0,791 0,814 0,837 0,791 0,791 0,826 0,814 0,814 0,791 0,837 0,791 30 0,698 0,709 0,756 0,744 0,721 0,721 0,802 0,733 0,709 0,733 0,756 0,779 0,733 0,756 0,767 0,756 0,779 0,779 0,756 0,826 31 0,767 0,779 0,756 0,744 0,791 0,767 0,779 0,779 0,779 0,802 0,779 0,826 0,802 0,733 0,814 0,802 0,872 0,779 0,802 0,733 32 0,791 0,779 0,779 0,744 0,837 0,791 0,756 0,756 0,756 0,779 0,756 0,756 0,756 0,733 0,767 0,779 0,802 0,779 0,802 0,779 33 0,814 0,779 0,826 0,744 0,814 0,837 0,779 0,733 0,756 0,802 0,779 0,802 0,756 0,733 0,767 0,779 0,802 0,802 0,872 0,779 34 0,814 0,826 0,756 0,721 0,767 0,767 0,756 0,733 0,802 0,779 0,756 0,779 0,733 0,733 0,744 0,779 0,826 0,802 0,779 0,779 35 0,733 0,814 0,744 0,779 0,756 0,733 0,767 0,791 0,814 0,767 0,814 0,791 0,767 0,767 0,849 0,744 0,791 0,791 0,791 0,791 36 0,733 0,791 0,791 0,733 0,802 0,802 0,767 0,791 0,767 0,767 0,837 0,791 0,791 0,744 0,756 0,791 0,791 0,744 0,814 0,744 37 0,767 0,756 0,779 0,767 0,744 0,767 0,802 0,779 0,802 0,802 0,779 0,802 0,756 0,756 0,791 0,802 0,802 0,802 0,849 0,826 38 0,744 0,756 0,756 0,744 0,767 0,767 0,733 0,779 0,802 0,802 0,779 0,802 0,779 0,756 0,791 0,756 0,826 0,779 0,779 0,802 39 0,744 0,756 0,802 0,744 0,791 0,791 0,756 0,802 0,779 0,756 0,802 0,756 0,802 0,756 0,767 0,826 0,756 0,779 0,779 0,826 40 0,744 0,756 0,802 0,744 0,744 0,791 0,733 0,756 0,779 0,826 0,756 0,802 0,756 0,756 0,791 0,779 0,802 0,733 0,779 0,779 41 0,814 0,779 0,826 0,767 0,814 0,837 0,756 0,802 0,779 0,779 0,802 0,802 0,756 0,779 0,721 0,779 0,756 0,756 0,756 0,756
Selain melalui koefisien kesamaan genetik, ke-kerabatan antargenotipe juga dapat dilihat dari nilai jarak genetik. Menurut Kuswandi et al. (2014), hasil analisis berupa dendrogram dan matriks jarak genetik dapat menjelaskan persentase kemiripan dan jarak genetik antarkultivar. Kedekatan genetik antargeno-tipe cabai berdasarkan matriks jarak genetik ditunjuk-kan pada Tabel 4. Genotipe yang mempunyai jarak genetik terdekat adalah Bara (rawit) F8 145318-1111-3-1 (rawit) dan Violeta (rawit hias) Lembayung (rawit hias) dengan nilai kesamaan genetik sebesar 0,93. Hal ini kemungkinan karena genotipe yang di-bandingkan mempunyai kedekatan karakter fenotipe, yaitu sama-sama bertipe rawit. Sementara, genotipe dengan jarak genetik terjauh adalah Yuni (keriting) Ayesha (rawit hias), Yuni (keriting) Ayesha 2 (rawit hias), dan Yuni (keriting) Namira (rawit hias) dengan nilai sebesar 0,698. Ketiga kombinasi geno-tipe tersebut mempunyai jarak genetik terjauh karena berasal dari tipe cabai yang berbeda.
Informasi jarak genetik yang diperoleh dapat di-manfaatkan untuk tujuan pemuliaan, misalnya untuk ketahanan terhadap hama dan penyakit, pro-duktivitas tinggi, adaptasi cekaman lingkungan, dan kualitas tanaman yang diinginkan lainnya (Haydar et al. 2007). Salah satu cara mendapatkan cabai dengan sifat yang diinginkan adalah dengan melakukan per-silangan buatan (hand pollination). Salah satu faktor yang menentukan keberhasilan persilangan adalah nilai koefisien kesamaan genetik dan jarak genetik.
Menurut Sukartini (2008), semakin kecil nilai koefi-sien kesamaan genetik atau semakin besar jarak ge-netik, semakin tinggi tingkat keberhasilan persilangan untuk mendapatkan progeni superior, demikian pula sebaliknya. Karena itu, penelusuran sumber tetua persilangan perlu dilakukan untuk mengetahui kontri-busi varietas yang akan digunakan dalam memben-tuk keragaman genetik sehingga dapat memberikan informasi lebih lengkap terhadap persilangan ke depannya.
Pada penelitian lain diketahui bahwa dari semua genotipe cabai lokal yang diuji, terdapat enam geno-tipe yang bereaksi tahan terhadap P. capsici isolat CpnsCK1, yaitu Ungara, Violeta, Ayesha, Violeta 1, Sempurna, dan Ayesha Ungu (Wartono et al., data belum dipublikasikan). Keenam genotipe tersebut dapat dimanfaatkan sebagai donor genetik untuk memperbaiki sifat genotipe cabai lainnya. Genotipe cabai tahan, seperti Ayesha dan Ungara, dapat diman-faatkan sebagai sumber tetua untuk disilangkan dengan genotipe yang berproduksi tinggi tetapi rentan terhadap P. capsici,seperti Tanjung-2, Ciko, dan Yuni. Dari hasil persilangan tersebut diharapkan akan di-peroleh progeni yang tahan terhadap P. capsici dan berproduksi tinggi. Persilangan antargenotipe dengan jarak genetik yang jauh akan menghasilkan progeni dengan keragaman yang tinggi (Santoso et al. 2014). Selain itu, nilai jarak genetik yang tinggi antartetua persilangan dapat memunculkan efek heterosis pada keturunannya (Izzah dan Reflinur 2018). Hasil
pene-Tabel 4. Lanjutan. 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 22 0,837 23 0,791 0,884 24 0,792 0,740 0,766 25 0,814 0,791 0,767 0,740 26 0,814 0,744 0,791 0,818 0,791 27 0,826 0,849 0,802 0,753 0,733 0,802 28 0,733 0,756 0,756 0,779 0,756 0,779 0,814 29 0,814 0,791 0,767 0,792 0,791 0,837 0,826 0,802 30 0,779 0,779 0,802 0,779 0,733 0,756 0,744 0,744 0,849 31 0,756 0,779 0,779 0,753 0,779 0,802 0,814 0,860 0,826 0,721 32 0,779 0,779 0,802 0,753 0,709 0,733 0,814 0,884 0,756 0,767 0,860 33 0,779 0,779 0,779 0,805 0,733 0,756 0,814 0,860 0,779 0,744 0,814 0,860 34 0,756 0,779 0,779 0,753 0,733 0,756 0,814 0,791 0,756 0,721 0,791 0,791 0,791 35 0,791 0,837 0,837 0,740 0,767 0,791 0,849 0,802 0,814 0,779 0,802 0,826 0,802 0,779 36 0,767 0,744 0,744 0,779 0,791 0,791 0,779 0,779 0,814 0,709 0,849 0,756 0,779 0,733 0,767 37 0,826 0,849 0,849 0,766 0,733 0,733 0,791 0,791 0,779 0,791 0,767 0,837 0,860 0,767 0,872 0,756 38 0,779 0,802 0,802 0,727 0,756 0,756 0,767 0,791 0,733 0,744 0,860 0,814 0,814 0,791 0,826 0,802 0,860 39 0,826 0,826 0,826 0,753 0,756 0,756 0,767 0,767 0,756 0,744 0,814 0,837 0,791 0,791 0,802 0,802 0,814 0,837 40 0,779 0,756 0,733 0,753 0,779 0,756 0,744 0,791 0,756 0,698 0,837 0,814 0,860 0,744 0,779 0,872 0,837 0,860 0,814 41 0,756 0,733 0,733 0,831 0,709 0,779 0,744 0,767 0,756 0,698 0,791 0,791 0,767 0,767 0,733 0,802 0,767 0,767 0,767 0,814
1 = Ayesha, 2 = Laris, 3 = Bara, 4 = CM334, 5 = Violeta, 6 = Lembayung, 7 = SSP, 8 = Jalapeno, 9 = Genie, 10 = F6 145291-10-7-1-1-1, 11 = F8 145318-1-1-1-1-3-1, 12 = F8 145291-115-8-1-1, 13 = F8 160291-9-4-3-2-1-1, 14 = F8 160291-3-12-5-4-51-1, 15 = Landung, 16 = Lokal Lembang (keriting), 17 = Tanjung-2, 18 = Vitra, 19 = Lingga, 20 = Kencana, 21 = Cakri Andalas, 22 = Lembang 1, 23 = Ciko, 24 = CL-4, 25 = Sempurna, 26 = Rama, 27 = Anies, 28 = Nazla, 29 = Seloka-5, 30 = Yuni, 31 = Ungara, 32 = Viola, 33 = Violeta 1, 34 = Giant A, 35 = C5, 36 = Ayesha Ungu, 37 = F5 Syak × 320-202-2-2, 38 = F5 Syak × 320-202-2-1, 39 = F5 Syak × 320-201-1-1, 40 = Ayesha 2, 41 = Namira.
litian ini menyediakan informasi dasar mengenai keragaman genetik beberapa tipe cabai berdasarkan marka SSR yang dapat dimanfaatkan untuk memilih tetua persilangan.
KESIMPULAN
Keragaman genetik yang tinggi ditemukan pada 38 genotipe cabai Indonesia yang berbeda dengan tiga genotipe cabai introduksi seperti direfleksikan dengan nilai PIC rata-rata dan diversitas gen lebih dari 0,7. Sebelas marka SSR berhasil mengestimasi kera-gaman genetik 41 genotipe cabai dan memberi infor-masi jarak genetik kandidat kombinasi tetua per-silangan. Berdasarkan jarak genetik, diperoleh tiga kombinasi tetua ideal untuk persilangan cabai yang berasal dari spesies yang berbeda, yaitu Yuni (keriting) × Ayesha (rawit hias), Yuni (keriting) × Ayesha 2 (rawit hias), dan Yuni (keriting) × Namira (rawit hias). Marka SSR dengan nilai PIC >0,5 pada penelitian ini sangat informatif dan sangat potensial untuk digunakan pada analisis diversitas genetik ber-bagai aksesi SDG cabai.
UCAPAN TERIMA KASIH
Penelitian ini didanai melalui DIPA Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian dan Proyek SMARTD, Balitbangtan, Kementan. Penulis menyampaikan terima kasih kepada Tim Genom BB Biogen yang telah membantu kegiatan penelitian ini. Ucapan terima kasih juga disampaikan kepada Rerenstradika T. Terryana untuk konsultasi pengolahan data dan Ade Ahmad yang telah membantu pelaksanaan kegiatan penelitian di rumah kaca.
KONTRIBUTOR PENULISAN
WTN: kontributor utama, penanggung jawab penelitian, pengujian rumah kaca, analisis data. SW: kontributor anggota, perencanaan penelitian. MS: kontributor anggota, perencanaan penelitian. GYT: kontributor anggota, perencanaan penelitian. KN: kontributor anggota, analisis genotyping. PL: kontributor anggota, perencanaan penelitian.
DAFTAR PUSTAKA
Asmono, D., Purba, A.R., Yenni, Y., Kobar, M., Zaelani, H., Liwang, T. & Beng, A.B. (2005) Peta dan prospek pemuliaan dalam industri perbenihan kelapa sawit di Indonesia. Simposium Nasional dan Kongres V PERIPI. Puwokerto, Perhimpunan Ilmu Pemuliaan Indonesia, hlm. 25–27.
Becher, S.A., Steinmetz, K., Weising, K., Boury, S., Peltier, D., Renou, J.P., Kahl, G. & Wolff, K. (2000) Microsatellites for cultivar identification in Pelargonium. Theoretical and Applied Genetics, 101, 643–651. Botstein, D., Whie, R.L., Skolnick, M. & Davis, R.W. (1980)
Construction of a genetic linkage map in man using restriction fragment length polymorphism. American Journal of Human Genetics, 32, 314–331.
Candole, B.L., Conner, P.J. & Ji, P. (2012) Evaluation of phytophthora root rot-resistant Capsicum annuum accessions for resistance to phytophthora foliar blight and phytophthora stem blight. Agricultural Sciences, 3 (5), 732–737.
Carvalho, S.I.C., Ragassi, C.F., Oliveira, L.B., Amaral, Z.P.S., Reifschneider, F.J.B., Faleiro, F.G. & Buso, G.S.C. (2015) Transferability of microsatellite markers of Capsicum annuum L. to C. frutescens L. and C. chinense Jacq. Genetics and Molecular Research, 14 (3), 7937–7946.
Collard, B.C.Y., Jahufer, M.Z.Z., Brouwer, J.B. & Pang, E.C.K. (2005) An introduction to markers, quantitative trait loci (QTL) mapping and marker-assisted selection for crop improvement: The basic concepts. Euphytica, 142, 169–196.
Dhaliwal, M.S., Yadav, A. & Jindal, S.K. (2014) Molecular characterization and diversity analysis in chilli pepper using simple sequence repeats (SSR) markers. African Journal of Biotechnology, 13 (31), 3137–3143.
Direktorat Jenderal Hortikultura (2019) Data produksi sayuran. [Online] Tersedia pada: http:// hortikultura2.pertanian.go.id [Diakses 2 Januari 2019]. Doyle, J.J. & Doyle, J.L. (1990) Isolation of plant DNA from
fresh tissue. Focus, 12, 13–15.
FAOSTAT (2011) Food and Agriculture Organization corporate statistical database. [Online] Available from: http://faostat.fao.org/site/339/default.aspx [Accessed 20 Desember 2018].
Gómez-Rodríguez, O., Corona-Torres, T. & Aguilar-Rincón, V.H. (2017) Differential response of pepper (Capsicum annuum L.) lines to Phytophthora capsici and root-knot nematodes. Crop Protection, 92, 148–152.
Haydar, A., Ahmed, M.B., Hannan, M.M., Razvy, M.A., Mandal, M.A., Salahin, M., Karim, R. & Hossain, M. (2007) Analysis of genetic diversity in some potato varieties grown in Bangladesh. Middle East Journal of Scientific Research, 2 (3–4), 143–145.
Hildebrand, E., Torney, D.C. & Wagner, R.P. (1992) Informativeness of polymorphic DNA markers. Los Alamos Science, 20, 100–102.
Izzah, N.K. & Reflinur (2018) Pemilihan tetua persilangan pada kubis (Brassica oleracea var. capitata) melalui analisis keragaman genetik. Jurnal Hortikultura, 28 (1), 33–40.
Jang, I.O., Moon, J.H., Yoon, J.B., Yoo, J.H. & Yang, T.J. (2004) Application of RAPD and SCAR markers for
purity testing of F1 hybrid seed in chili pepper
(Capsicum annuum). Molecular Cell, 18, 295–299. Khan, T.H., Evamoni, F.Z., Rubel, M.H., Nasiruddin, K.M. &
Mahbubur Rahman, M. (2015) Screening of rice varieties for bacterial leaf blight resistance by using SSR markers. Journal of Bioscience and Agriculture Research, 3 (1), 45–58.
Kuswandi, Sobir & Suwarno, W.B. (2014) Keragaman genetik plasma nutfah rambutan di Indonesia berdasarkan karakter morfologi. Jurnal Hortikultura, 24 (4), 289–298.
Kwon, Y.S., Lee, J.M., Yi, G.B., Yi, S.I., Kim, K.M., Soh, E.H., Bae, K.M., Park, E.K., Song, I.H. & Kim, B.D. (2005) Use of SSR markers to complement tests of distinctiveness, uniformity, and stability (DUS) of pepper (Capsicum annuum L.) varieties. Molecular Cell, 19, 428–435.
Lee, J.M., Nahm, S.H., Kim, Y.M. & Kim, B.D. (2004) Characterization and molecular genetic mapping of microsatellite loci in pepper. Theoretical and Applied Genetics, 108, 619–627.
Liu, K. & Muse, S.V. (2005) PowerMarker: An integrated analysis environment for genetic marker analysis. Bioinformatics, 21 (9), 2128–2129.
Mahatmam, K., Khandelwal. V., Jha, S.K., Kumar, V. & Shah, R.R. (2009) Genetic diversity analysis of elite parental lines of cotton using RAPD, ISSR, and isozyme markers. Indian Journal of Plant Physiology, 14 (2), 105–110.
Nicolai, M., Cantet, M., Lefebvre, V., Sage-Palloix, A.M. & Palloix, A. (2013) Genotyping a large collection of pepper (Capsicum spp.) with SSR loci brings new evidence for the wild origin of cultivated C. annuum and the structuring of genetic diversity by human selection of cultivar types. Genetic Resources and Crop Evolution, 60, 2375–2390.
Oh, S., Song, J., Lee, J., Lee, G., Ko, H., Stoilova, T., Krasteva, L., Kim, Y., Rhee, J., Gwag, J., Ro, N., Hur, O. & Lee, M. (2012) Evaluation of genetic diversity of red pepper landraces (Capsicum annuum L.) from Bulgaria using SSR markers. Korean Journal of Environmental Agriculture, 24 (5), 547–556.
Pacheco-Olvera, A., Hernandez-Verdugo, S., Rocha-Ramírez, V., Gonzalez-Rodríguez, A. & Oyama, K. (2012) Genetic diversity and structure of pepper (Capsicum annuum L.) from northwestern Mexico analyzed by microsatellite markers. Crop Science, 52, 231–241.
Rohlf, F.J. (2000) NTSYS-pc: Numerical taxonomy and multivariate analysis system, version 2.1. Exeter Software, Setauket, New York, USA.
Santoso, P.J., Djamas, N., Rebin & Pancoro, A. (2014) Analisis diversitas dan paternitas progeni F1 hasil
persilangan Arumanis 143 Mangga Merah menggunakan marka mikrosatelit. Jurnal Hortikultura, 24 (3), 210–219.
Sharmin, A., Hoque, Md.E., Haque, Md.M. & Khatun, F. (2018) Molecular diversity analysis of some chilli (Capsicum spp.) genotypes using SSR markers. American Journal of Plant Sciences, 9, 368–379. Sukartini (2008) Analisis jarak genetik dan kekerabatan
aksesi-aksesi pisang berdasarkan primer random amplified polymorphic DNA. Jurnal Hortikultura, 18 (3), 261–266.
Sutjahjo, S., Herison, C., Sulastrini, I. & Marwiyah, S. (2015) Pendugaan keragaman genetik beberapa karakter pertumbuhan dan hasil pada 30 genotipe tomat lokal. Jurnal Hortikultura, 25 (4), 304–310.
Syukur, M., Sujiprihati, S., Yunianti, R. & Kusumah, D.A. (2010) Evaluasi daya hasil cabai hibrida dan daya adaptasinya di empat lokasi dalam dua tahun. Jurnal Agronomi Indonesia, 38 (1), 43–51.
Tasma, I.M., Yani, N.P.M.G., Purwaningdyah, R., Satyawan, D., Nugroho, K., Lestari, P., Trijatmiko, K.R. & Mastur (2018) Genetic diversity analysis and F2 population
development for breeding of long juvenile trait in soybean. Jurnal AgroBiogen, 14 (1), 11–24.
Terryana, R.T., Nugroho, K., Rijzaani, H. & Lestari, P. (2018) Genetic diversity characterization of 27 chili pepper genotypes based on SSR (simple sequence repeat) markers. Berita Biologi, 17 (2), 183–194. Trimanto (2012) Karakterisasi dan jarak kemiripan uwi
(Dioscorea alata L.) berdasarkan penanda morfologi umbi. Buletin Kebun Raya, 15 (1), 46–55.
Wang, S., Lu, Z. & Wan, J. (2006) Genetic diversity among parents of hybrid rice based on cluster analysis of morphological traits and simple sequence repeat markers. Rice Science, 13 (3), 155–160.
Widaningsih, N.A., Purwanto, E., Nandariyah & Reflinur (2014) The use of DNA microsatellite markers for genetic diversity identification of soybean (Glycine max [L.] Merrill.) as a supplementary method in reference collections management. Indonesian Journal of Biotechnology, 19 (2), 136–145.
Yu, S.B., Xu, W.J., Vijayakumar, Ali, J., Fu, B.Y., Xu, J.L., Jiang, Y.Z., Maghirang, R., Domingo, J. & Aquino, C. (2003) Molecular diversity and multilocus organization of the parental lines used in the international rice molecular breeding program. Theoretical and Applied Genetics, 108 (1), 131–140.
Zhang, X.M., Zhang, Z.H., Gu, X.Z., Mao, S.L., Li, X.X., Chadœuf, J.P.A., Wang, L.H. & Zhang, B.X. (2016) Genetic diversity of pepper (Capsicum spp.) germplasm resources in China reflects selection for cultivar types and spatial distribution. Journal of Integrative Agriculture, 15 (9), 1991–2001.