perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
i
STABILITAS KONFORMASI
QUADRUPLE
MUTAN p53
M133L/V203A/N239Y/N268D DAN PENGARUH
PRIMA-1
TERHADAP
WILD TYPE
P53
Disusun oleh :
ANITA KUSUMA DEWI
M0303018
SKRIPSI
Diajukan untuk memenuhi sebagian
persyaratan mendapatkan gelar Sarjana Sains Kimia
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN
ALAM
UNIVERSITAS SEBELAS MARET
SURAKARTA
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
iii
PERNYATAAN
Dengan ini saya menyatakan bahwa skripsi saya yang berjudul ―STABILITAS
KONFORMASI QUADRUPLE MUTAN p53 M133L/V203A/N239Y/N268D DAN PENGARUH PRIMA-1 TERHADAP WILD TYPE P53 ‖ belum pernah
diajukan untuk memperoleh gelar kesarjanaan di suatu perguruan tinggi, dan sepanjang pengetahuan saya juga belum pernah ditulis atau dipublikasikan oleh orang lain, kecuali yang secara tertulis diacu dalam naskah ini dan disebutkan dalam daftar pustaka.
Surakarta, Februari 2011
perpustakaan.uns.ac.id digilib.uns.ac.id
Jurusan Kimia. Fakultas MIPA. Universitas Sebelas Maret.
ABSTRAK
Quadruple mutan p53 M133L/V203A/N239Y/N268D mampu
meningkatkan stabilitas termodinamika ikatan residu-residu yang mengalami mutasi [Joerger, A. C., Allen, M.D., and Fersht, A. R., 2004, J. Biochem., 279: pp.
1291-1296]. Selain itu, struktur kristalografi sinar-x quadruple mutan ini mirip dengan struktur wild type p53 ( kode PDB : wt-p53). Kemiripan struktur ini
melandasi studi dinamika konformasinya. Studi tersebut telah dilakukan dengan menggunakan simulasi dinamika molekuler. Dinamika konformasi quadruple
mutan (kode PDB : QMT) mengungkap adanya pergeseran struktur yang ditunjukkan oleh perubahan nilai RMSD selama simulasi 5ns berlangsung.
Pergeseran ini terutama disebabkan oleh fluktuasi pada loop L2 dan loop S7-S8.
Fluktuasi pada loop S7-S8 lebih signifikan dibanding dengan loop L2. Selain itu,
dinamika konformasi residu-residu penting p53 untuk berikatan dengan DNA
(yaitu lysin 120 dan arginin 248) tidak dapat membedakan quadruple mutan dan
wild type p53. Simulasi dinamika molekuler ini juga dapat menunjukkan pengaruh
PRIMA-1 terhadap wild type p53. Berdasarkan hasil dockingnya, PRIMA-1 tidak
cukup spesifik berinteraksi pada wild type p53. PRIMA-1 cukup stabil menempel
pada wild type p53 di area β-sandwich. Keberadaan PRIMA-1 memberikan
dinamika konformasi yang berbeda antara wild type p53 kompleks dan
tunggalnya. Perbedaannya terletak pada residu histidin 116, glysin 245, dan arginin 248. Menariknya lagi residu 186 yang terletak jauh dari penempelan
PRIMA-1 juga mengalami pergeseran.
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
v
QUADRUPLE MUTAN P53 AND THE EFFECTS OF PRIMA-1
AGAINST WILD TYPE P53
ANITA KUSUMA DEWI
Department of Chemistry. Faculty of Mathematics and Natural Sciences. Sebelas Maret University.
ABSTRACT
M133L/V203A/N239Y/N268D quadruple mutant p53 was able to
increase the thermodynamic stability of mutated residue bonds [Joerger, A. C., Allen, M.D., and Fersht, A. R., 2004, J. Biochem., 279: pp. 1291-1296]. In
additions, x-ray crystallography structure of quadruple mutant similar to the wild type structure ( PDB code: wt-p53). This similarity was underlying to study its conformational dynamics. The study was done by means molecular dynamics simulations. Conformational dynamics of quadruple mutant (PDB code: QMT)
reveal a shifted structure which was demonstrated by RMSD value changes during 5ns of simulation. This shift was mainly caused fluctuations in L2 loops and S7-S8 loops. the last was showing more significant fluctuations. In additions, conformational dynamics of p53 important residues for DNA binding (i.e lysine
120 and arginine 248) could not distinguish quadruplemutant and wild typep53. This molecular dynamic simulation can also show effects of PRIMA-1 against wild type p53. According to its docking results, PRIMA-1 was not specific enough to interact in wild type p53. PRIMA-1 was quite stable against wild type p53 in β-sandwich area. The existence of PRIMA-1distinguished conformational
dynamics between un-complex and complex wild type p53. The discrepancy was located at residues of histidine 116, glycine 245, and arginine 248. Interestingly, residue of 186 which is far from PRIMA-1 binding site, was also shifted due to the bind.
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
vi MOTTO
Setiap kenikmatan akan lebih terasa manakala kita tahu bagaimana rasa pahit. Untuk itu syukuri setiap pahit yang terasa hari ini untuk nikmat esok hari.
(Anita Kusuma Dewi)
Sebuah kegagalan bisa jadi sebuah keberhasilan menemukan hal baru asal kita mampu melihat sisi positifnya. (Fajar R. Wibowo)
Sabar dan Syukurlah niscaya Allah akan menambah nikmatmu setiap hari (Suami tercinta)
Kebenaran bukan untuk dipaksakan tetapi diakui keberadaannya (Achdiat Kartamihardja)
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
vii
PERSEMBAHAN
Untuk Allah Yang Maha Cerdas, Maha Bijaksana, dan Maha Segalanya
Untuk Bapak dan Ibu yan paling pengertian
Untuk adik-adikku tersayang
Untuk suami tercinta yang selalu sabar dan support
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
viii
KATA PENGANTAR
Segala puji dan segenap syukur bagi Allah SWT yang telah menunjukkan jalan yang indah bagi penulis sehingga skripsi ini dapat penulis selesaikan dengan baik sebagai salah satu persyaratan dalam memperoleh gelar sarjana sains Jurusan Kimia Fakultas Matematika dan Ilmu Pengetahuan Alam Universitas Sebelas Maret Surakarta. Atas segala karuniaNya pulalah penulis menyadari bahwa segala sesuatu memiliki proses dan waktunya masing-masing.
Dalam menyusun skripsi ini penulis menemui berbagai hambatan dan permasalahan yang beragam. Namun, atas bimbingan, kritikan, saran, dan dorongan semangat yang bermanfaat dari berbagai pihak, semua hambatan dan permasalahan tersebut dapat penulis atasi dengan baik. Oleh karena itu, penulis ingin menyampaikan terima kasih kepada pihak-pihak yang telah membantu penulis, yaitu sebagai berikut.
1. Prof. Drs. Sutarno, M.Sc. Ph.D. selaku dekan Fakultas Matematika dan Ilmu Pengetahuan Alam Universitas Sebelas Maret Surakarta.
2. Prof. Drs. Sentot Budi Rahardjo, Ph.D., selaku ketua Jurusan Kimia Fakultas Matematika dan Ilmu Pengetahuan Alam Universitas Sebelas Maret Surakarta. 3. Soerya Dewi Marliyana, M.Si., selaku pembimbing akademik yang dengan
sabar telah membimbing penulis dalam penyelesaian studi di Jurusan Kimia. 4. Dr. rer. nat. Fajar R. Wibowo, M.Si., selaku dosen pembimbing I, yang
dengan penuh kesabaran dan ketulusan membimbing penulis dari titik nol, membuka mata penulis bahwa segala sesuatu itu memiliki berbagai kemungkinan dengan alasannya masing-masing.
5. Yuniawan Hidayat, M.Si., selaku dosen pembimbing II, yang dengan ketulusan membimbing penulis mengenai cara penulisan yang baik dan sesuai aturan. Atas bimbingan beliau pulalah penulis mendapatkan dorongan semangat untuk menyelesaikan skripsi ini dengan efektif.
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
ix
7. Bapak Ibu dosen dan seluruh staf jurusan Kimia yang telah memberikan fasilitas dan pelayanan yang baik bagi penulis.
8. Teman-teman Kimia berbagai generasi yang menjadi kawan di medan juang. 9. Semua pihak yang tidak dapat penulis tuliskan satu per satu yang telah
memberikan bantuannya.
Penulis menyadari bahwa penelitian dan penyusunan skripsi yang penulis lakukan masih jauh dari sempurna sehingga membutuhkan saran dan kritik yang membangun dari para pembaca. Namun, lepas dari semua itu, semoga para pembaca mendapatkan manfaat setelah membaca skripsi ini.
Surakarta, Februari 2011
perpustakaan.uns.ac.id digilib.uns.ac.id
HALAMAN PERSEMBAHAN ... vii
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
xi
7. Simulasi Dinamika Molekuler (DM) ... 12
8. AMBER7 (Assisted Model Building withEnergyRefinement) ... 14
a. Antechamber ... 14
b. Parmchk. ... 14
c. LEaP. ... 14
d. Sander (Simulated Annealing with NMR-derived Energy Restraints) ... 15
e. Ptraj dan Carnal ... 15
9. RootMeanSquareDeviation (RMSD) ... 15
10.B-factor ... 16
11.Struktur Protein dan Sudut Dihedral Backbone Protein ... 16
B. Kerangka Pemikiran ... 19
C.Hipotesis ... 20
BAB III METODOLOGI PENELITIAN... 21
A.Waktu dan Tempat Penelitian ... 21
2. Penentuan Koordinat Awal Sistem ... 22
3. Minimisasi Sistem ... 22
4. Equilibrasi Sistem ... 22
5. Simulasi Sistem ... 23
D.Teknik Pengumpulan dan Analisis Data ... 23
BAB IV HASIL DAN PEMBAHASAN ... 24
A.Parameterisasi PRIMA-1... 24
B. Hasil Simulasi ... 24
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
xii
a. Residu Lisin 120 dan Arginin 248 ... 27
b. Residu Arginin 181, Asam Aspartat 186, dan Glysin 226 .. 30
2. Perilaku PRIMA-1 pada wild type p53 (wt-p53) ... 34
BAB V KESIMPULAN DAN SARAN ... 46
DAFTAR PUSTAKA. ... 47
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
xiii
DAFTAR TABEL
Tabel 1. Ringkasan Karakteristik Pergerakan dalam Protein. ... 13 Tabel 2. Kode Atom, Tipe Atom, dan Muatan PRIMA-1 yang Diperoleh
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
xiv
DAFTAR GAMBAR
Gambar 1. Skema struktur p53 ... 6
Gambar 2. Distribusi elemen dan skema ribbon struktur sekunder p53 ... 7
Gambar 3. Siklus sel ... 8
Gambar 4. Superposisi bentuk stereo dari backbone wt-p53 tanpa DNA, wt-p53 dengan DNA dan quadruple mutan p53 ... 9
Gambar 14. Grafik fluktuasi sudut dihedral Psi (ψ) dan Phi (Ф) residu 118 -123 (melibatkan residu lisin 120) dan perbedaan posisi sudut dihedral selama simulasi. ... 28
Gambar 15. Grafik fluktuasi sudut dihedral Psi (ψ) dan Phi (Ф) residu 244 -248 (melibatkan residu arginin -248) dan perbedaan posisi sudut dihedral selama simulasi ... 30
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
xv
Gambar 17. Profil sudut dihedral Psi (ψ) dan Phi (Ф) residu 179-181, residu 184-188, dan residu 225-230 ... 33 Gambar 18. Perubahan sudut dihedral selama simulasi backbone residu asam
amino 185-187 ... 34 Gambar 19. Perbandingan profil RMSD (Å) terhadap waktu (ps) hasil
simulasi DM untuk wild type p53 wt-p53 dan kompleks p53 dengan PRIMA_1 ... 35
Gambar 20. Perbandingan profil B-factor (Å2) terhadap nomor residu hasil
simulasi DM untuk wild type p53 wt-p53 dan kompleks wt-p53 dengan PRIMA-1 ... 36
Gambar 21. Perbandingan posisi PRIMA-1 pada p53 selama simulasi ... 37
Gambar 22. Posisi PRIMA-1 terhadap dua residu yang diperkirakan mampu
membentuk ikatan hidrogen antara keduanya berturut-turut ... 38 Gambar 23. Profil sudut dihedral Psi (ψ) dan Phi (Ф) residu 114-118
komplekss p53 dengan PRIMA-1 dan perbedaan posisinya selama simulasi ... 39 Gambar 24. Posisi PRIMA-1 terhadap wt-p53 dengan beberapa residu
terdekatnya valin 178, ileusin 157, arginin 248, dan glysin 245. ... 40 Gambar 25. Dinamika perubahan sudut torsi terhadap waktu antara
komplekss p53 dengan PRIMA-1 dengan pembanding wt-p53
sudut Ф dan ψ untuk rentang residu 242-246 yang memuat residu
245 dan rentang residu 244-248 ... 41 Gambar 26. Dinamika posisi kompleks p53 dengan PRIMA-1 dihimpitkan
dengan wt-p53 sebagai untuk residu 244-249 selama simulasi ... 42 Gambar 27. Dinamika perubahan sudut torsi terhadap waktu antara
komplekss p53 dengan PRIMA-1 dengan pembanding wt-p53 sudut Ф dan ψ untuk rentang residu 183-187 ... 43 Gambar 28. Dinamika posisi komplekss p53 dengan PRIMA-1 dihimpitkan
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
xvi
Gambar 29. Gambar keadaan sistem secara umum pada saat penentuan koordinat awal, minimisasi, equilibrasi, dan simulasi. A, B, C, dan D berturut-turut adalah keadaan sistem pada saat awal, setelah disimulasi, setelah diequilibrasi, dan setelah simulasi berjalan. Warna hitam adalah sistem yaitu protein p53 dan warna biru adalah molekul air. ... 51 Gambar 30. Densitas, volume, energi total, dan temperatur sistem selama
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
xvii
DAFTAR LAMPIRAN
Lampiran 1. Gambaran Sistem Secara Umum pada Saat Penentuan Koordinat Awal, Minimisasi, Equilibrasi, dan Simulasi ... 51 Lampiran 2. Densitas, Volume, Energi Total, dan Temperatur Sistem Selama
perpustakaan.uns.ac.id digilib.uns.ac.id
Kanker telah lama menjadi masalah utama dalam bidang kedokteran dan menjadi fokus penelitian untuk mencari penyebab, mekanisme, sampai cara pengobatannya. Sofyan (2002) menyebutkan ―kebanyakan kanker bisa disebabkan oleh salah satu atau lebih dari tiga kategori gen; onkogen, gen yang mengatur
replikasi atau perbaikan DNA , dan gen suppressor tumor. Salah satu produk gen
suppressor tumor dikenal dengan nama p53.
Protein p53 bekerja mencegah replikasi DNA yang rusak dengan
memperbaiki kerusakan yang ada. Protein ini mampu menginduksi kematian sel terprogram (apoptosis) jika upaya perbaikan tidak dapat dilakukan (Murray et al.,
1998). Adanya mutasi p53 dapat mengakibatkan disfungsi p53. Mutasi p53 menurut Vousden dan Lu (2002) terjadi 95% pada domain inti. Sebanyak 40% diantaranya ditemukan pada enam hotspot (Arg 175, Gly 245, Arg 248, Arg 249,
Arg 273, dan Arg 282 (Wright dan Lim, 2007).
Banyak studi struktur dasar mutasi p53 dilakukan untuk mengetahui pengaruh mutasi terhadap stabilitas molekul secara keseluruhan dan perubahan struktur lokal p53 berikatan dengan DNA. Kebanyakan studi ini melibatkan
quadruple mutan (QMT) p53 M133L/V203A/N239Y/N268D (Joerger et al.,
2006). Joerger dkk (2004) menyatakan quadruple mutan ini sangat stabil
(superstable mutan) pada sistem kristalnya, sedangkan kebanyakan mutasi lain
meyebabkan penurunan stabilitas termodinamika ikatan masing-masing residu yang mengalami mutasi. Selain itu, kristalografi sinar-x menunjukkan perpaduan QMT p53 ini hanya mengubah sedikit struktur lokalnya tanpa merubah struktur global p53 (inti β-Sandwich dan surface ikatan dengan DNA). Melalui fakta diatas
QMT p53 ini dapat dikatakan memiliki kemiripan struktur dengan p53 normalmya (wild type p53).
Kemiripan struktur QMT p53 dengan wild type p53 (wt-p53) sejalan
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
2
(2002b). Penelitian tersebut menemukan cara pengembalian fungsi p53 termutasi dengan mengatur konformasi mutan p53 menyerupai wild typenya. Pengaturan
konformasi p53 dilakukan dengan menempelkan molekul kecil atau peptida pendek pada mutan p53. Peptida pendek yang digunakan adalah PRIMA-1 (p53
Reactivation and Induction Massive Apoptosis). Menindaklanjuti penelitian
Bykov dkk (2002a), Warsino (2008) melakukan docking (penempelan ligan pada
makromolekul protein) antara PRIMA-1 dengan QMT p53 dan wt-p53. Hasil
docking menunjukkan situs potensial PRIMA-1 menempel pada QMT p53 dan
wt-p53. Posisi PRIMA-1 yang paling potensial ditemukan pada area yang sama yaitu
terletak pada daerah loop. Kedua posisi tersebut menghasilkan besar energi
docking yang relatif sama. PRIMA-1 dikatakan tidak cukup spesifik berinteraksi
pada wt-p53 dan QMT p53 karena beda energi saat dilakukan docking awal dan
lanjutan kurang dari 2 kkal/mol sebagai batas residual error autodock (Morris et
al., 1998).
Kemiripan struktur QMT p53 dan wt-p53, serta ketidakspesifikan
PRIMA-1 menempel pada wt-p53 mendorong studi dinamika molekul QMT p53
dan wt-p53 serta dinamika molekul PRIMA-1 menempel pada wt-p53. Sebuah
metode yang sangat tepat untuk mengamati dinamika molekul level atomik adalah simulasi dinamika molekuler (DM). Simulasi DM cukup baik dalam menentukan kontribusi dominan terjadinya fluktuasi atomik suatu protein (Pikkemaat et al., 2002). Ada beberapa karakter dinamika molekul yang dapat diamati melalui simulasi DM diantaranya adalah; fleksibilitas docking ligan, alur difusi temporal,
konformasi sisi aktif, spesifitas ikatan, transisi allosteric, dll. Tambahan informasi hasil simulasi DM ini diharapkan dapat menunjang studi karakteristik dinamika molekul QMT p53 dan wt-p53.
B. Perumusan Masalah
1. Identifikasi Masalah
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
3
273, dan 282 (Wright, 2007). Banyak studi tentang struktur dasar mutasi p53 yang melibatkan quadruple mutan M133L/V203A/N239Y/N268D (Joerger et al., 2006)
karena mutan ini dikatakan sangat stabil (superstable mutan) pada kondisi kristal
(Joerger etal., 2004). Selain itu, struktur kristal QMT p53ini memiliki kemiripan
dengan wt-p53.
Kemiripan struktur QMT dan wt-p53 sejalan dengan hasil docking
PRIMA-1 pada wt-p53 dan QMT. PRIMA-1 menempel pada posisi yang sama
dengan energi yang relatif sama. Namun pengamatan terhadap dinamika molekul QMT p53 dan wt-p53 serta pengaruh PRIMA-1 terhadap keduanya belum
diketahui. Oleh karena itu simulasi DM dapat dilakukan untuk mengamati lebih lanjut dinamika molekul QMT dan wt-p53 serta pengaruh PRIMA-1 terhadap
dinamika molekul keduanya.
Ada beberapa karakter dinamika molekul yang dapat dipilih. Beberapa diantaranya adalah; fleksibilitas docking ligan, alur difusi temporal, konformasi
sisi aktif, spesifitas ikatan, transisi allosteric, dll. Selain pemilihan karakter langkah berikutnya adalah memilih program simulasi Dinamika Molekuler (DM) yang sesuai. Berbagai program simulasi DM yang populer seperti AMBER
(Assisted Model Building with Energy Refinement), CHARMM (Chemistry at
HARvard Macromolecular Mechanics), Tinker, GROMOS (Groningen Molecular
Simulation), dan NAMD (NAnoscale Molecular Dynamics) dapat digunakan untuk
perbaikan molekul (Esposito et al., 2006).
2. Batasan Masalah
Simulasi Dinamika Molekuler (DM) terhadap karakter dinamika konformasi dilakukan untuk tiga sistem. Ketiga sistem tersebut antara lain; mutan p53 yang memuat quadruple mutan M133L/V203A/N239Y/N268D, wild type
p53, serta kompleks PRIMA-1 dengan wild type p53. Pemilihan kompleks hanya
untuk PRIMA-1 dengan wild type p53 saja terutama karena kemiripan struktur
yang dimiliki oleh wt-p53 dan QMT p53, serta posisi interaksi PRIMA-1 yang
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
4
Simulasi Dinamika Molekuler dilakukan dengan menggunakan program AMBER7 karena program ini cukup baik untuk simulasi biomolekul protein dan memiliki kemampuan untuk menggabungkan lebih dari satu force field. Simulasi
dilakukan dalam eksplisit solven dengan sistem periodik jangka waktu 9ns untuk
wild type p53 serta 5ns untuk quadruple mutan p53 dan kompleks PRIMA-1
dengan wt-p53.
3. Rumusan Masalah
a. Bagaimana perbandingan dinamika konformasi wild type p53 dan quadruple
mutan M133L/V203A/N239Y/N268D tanpa ligan PRIMA-1?
b. Apakah terdapat kesamaan karakteristik umum dinamika konformasi wt-p53 dan QMT p53?
c. Bagaimana dinamika konformasi kompleks PRIMA-1 dengan wild type p53?
C. Tujuan Penelitian
1. Mengetahui perbandingan dinamika konformasi wild type p53 pada manusia
dan quadruple mutan M133L/V203A/N239Y/N268D.
2. Mengetahui apakah terdapat kesamaan umum dinamika konformasi wt-p53 dan QMT p53.
3. Mengetahui dinamika konformasi kompleks PRIMA-1 dengan wild type p53.
D. Manfaat Penelitian
1. Memberikan informasi tentang dinamika konformasi molekul quadruple mutan
p53 M133L/V203A/N239Y/N268D dengan dinamika konformasi molekul
wt-p53 wt-p53 sebagai pembandingnya.
2. Dengan mengetahui perbandingan dinamika konformasi kompleks PRIMA-1 dengan wt-p53 dan QMT p53 akan memberikan sumbangan bagi ilmu
kesehatan dalam terapi kanker berkaitan dengan selektifitas PRIMA-1 terhadap
target mutasi diluar hotspot.
perpustakaan.uns.ac.id digilib.uns.ac.id
Kanker merupakan penyakit yang ditandai dengan pembelahan sel yang tidak terkendali menjadi sel-sel yang mampu menyerang jaringan biologis lainnya, baik yang bersebelahan (invasi), atau dengan migrasi sel ke tempat yang
jauh (metastase) (Murray et al., 1998). Mengenai penyebab kanker, Sofyan (2002) menyebutkan ―kebanyakan kanker bisa disebabkan oleh salah satu atau lebih dari
tiga kategori gen; onkogen, gen yang mengatur replikasi atau perbaikan DNA , dan
gen suppressor tumor―.
Pertumbuhan kanker diawali dengan mutasi berangkai. Mutasi tersebut terjadi pada gen penekan tumor dilanjutkan dengan mutasi pada gen yang berfungsi untuk memperbaiki kerusakan DNA (DNA repair gene). Mutasi pada
protoonkogen dapat mengaktifkan onkogen dan menonaktifkan gen penekan
tumor. Beberapa faktor hereditas dapat meningkatkan perubahan mutasi penyebab kanker, mencakup aktivasi onkogen atau penghambat gen penekan tumor. Fungsi
berbagai gen penekan tumor dan onkogen dapat diganggu pada tahapan berbeda
pertumbuhan tumor (Hadi dan Nurlalila, 2008).
2. Gen Supressor Tumor - p53
Dari ribuan jumlah gen dalam tubuh manusia, secara umum dibagi menjadi dua kelompok utama yakni onkogen dan gen penekan tumor atau gen
supressor tumor (Syaifudin, 2007). Onkogen adalah versi mutan dari gen normal,
yang memicu pertumbuhan sel. Gen pada sel normal yang dapat berubah menjadi
onkogen aktif akibat mutasi, disebut protoonkogen.
Gen supressor tumor merupakan gen penekan keganasan kanker. Gen ini
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
6
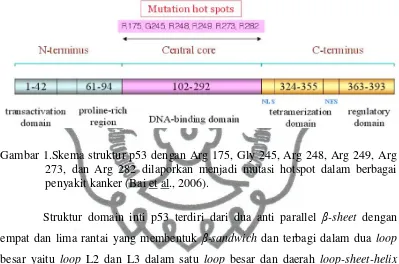
sebagai domain terminal amino yang dibutuhkan untuk aktivitas transaktivasi dan residu 61-94 yang kaya prolin, domain inti residu 102-292 yang dibutuhkan untuk berikatan dengan DNA, terminal C residu 301-393 terdiri dari domain
oligomerisasi (residu 342-355) dan domain regulator terminal karboksil (residu 363-393), yang disajikan dalam gambar 1 dibawah.
Gambar 1.Skema struktur p53 dengan Arg 175, Gly 245, Arg 248, Arg 249, Arg 273, dan Arg 282 dilaporkan menjadi mutasi hotspot dalam berbagai penyakit kanker (Bai et al., 2006).
Struktur domain inti p53 terdiri dari dua anti parallel β-sheet dengan
empat dan lima rantai yang membentuk β-sandwich dan terbagi dalam dua loop
besar yaitu loop L2 dan L3 dalam satu loop besar dan daerah loop-sheet-helix
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
7
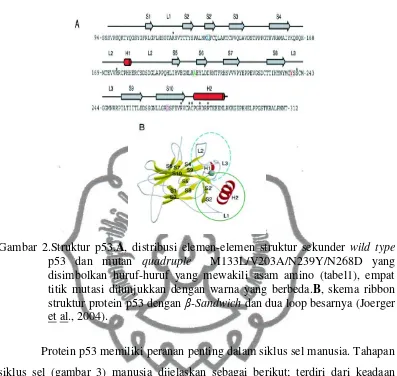
Gambar 2.Struktur p53.A, distribusi elemen-elemen struktur sekunder wild type
p53 dan mutan quadruple M133L/V203A/N239Y/N268D yang
disimbolkan huruf-huruf yang mewakili asam amino (tabel1), empat titik mutasi ditunjukkan dengan warna yang berbeda.B, skema ribbon struktur protein p53 dengan β-Sandwich dan dua loop besarnya (Joerger
etal., 2004).
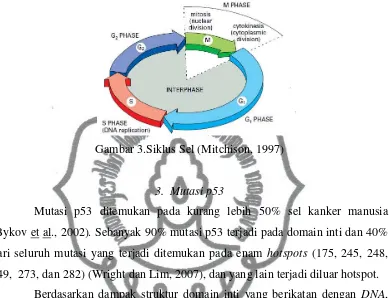
Protein p53 memiliki peranan penting dalam siklus sel manusia. Tahapan siklus sel (gambar 3) manusia dijelaskan sebagai berikut; terdiri dari keadaan istirahat (fase G0), pertumbuhan sel dan persiapan kromosom untuk replikasi (fase G1). Siklus dilanjutkan dengan sintesis DNA (fase S) dan diikuti dengan
persiapan pemisahan sel (fase G2). Siklus disempurnakan dengan mitosis (fase M) hingga dihasilkan sel-sel belahan yang baru (Enten et al., 2005).
Pada tahapan siklus sel tersebut protein p53 memiliki tiga fungsi utama yaitu bekerja sebagai aktivator transkripsional dengan mengatur gen tertentu yang terlibat dalam pembelahan sel, bekerja sebagai kontrol checkpoint G1 siklus sel
bagi kerusakan DNA. Saat terjadi kerusakan yang berlebih dapat meningkatkan
aktivitas dengan menghambat pembelahan sel dan memberikan waktu untuk perbaikan agar tidak terjadi replikasi DNA yang rusak. Perbaikan DNA dilakukan
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
8
Apoptosis merupakan bentuk kematian sel terprogram yang diperlukan dalam perkembangan sel, dan dikendalikan oleh berbagai gen (Kresno, 2002).
Gambar 3.Siklus Sel (Mitchison, 1997)
3. Mutasi p53
Mutasi p53 ditemukan pada kurang lebih 50% sel kanker manusia (Bykov et al., 2002). Sebanyak 90% mutasi p53 terjadi pada domain inti dan 40% dari seluruh mutasi yang terjadi ditemukan pada enam hotspots (175, 245, 248,
249, 273, dan 282) (Wright dan Lim, 2007), dan yang lain terjadi diluar hotspot. Berdasarkan dampak struktur domain inti yang berikatan dengan DNA,
Hainaut et al (1997) mengelompokkan mutasi p53 dalam tiga kelas yaitu mutasi kelas I mempengaruhi residu yang berikatan dengan DNA (Arg 248 dan Arg 273)
dengan menggangu titik kontak residu-residu tersebut untuk berikatan dengan
DNA. Mutasi kelas II domain inti mempengaruhi residu-residu yang penting untuk
orientasi permukaan ikatan DNA (Arg 175, Gly 245, Arg 249, dan Arg 282 yang
terdapat dalam area penghubung scaffold dan permukaan ikatan DNA) terhadap
fleksibilatas protein p53, sedangkan mutasi kelas III mempengaruhi struktur tersier domain inti dan memberikan sifat fungsional protein yang berbeda.
Pengembalian fungsi p53 termutasi tidak dapat dilakukan dengan penambahan konsentrasi p53 melainkan dengan mengatur konformasi dari p53 termutasi sehingga menyerupai wild type p53 (Protein p53 yang tidak mengalami
PRIMA-perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
9
1 (p53 Reaktivation and Induction Massive Apoptosis ) (Bykov et al., 2002a). Studi tentang pemahaman struktur dasar mutasi domain inti p53 yang dilakukan
oleh Joerger et al (2006) selalu disertai mutan quadruple
M133L/V203A/N239Y/N268D.
4. Mutan Quadruple M133L/V203A/N239Y/N268D
Hasil kristalografi sinar-X dalam penelitian Joerger et al (2004) menunjukkan bahwa mutan ini dikategorikan sebagai superstable mutan dengan
hanya sedikit perubahan struktur lokal tanpa merubah struktur global (inti β
-Sandwich dan permukaan yang berikatan dengan DNA) protein p53 sehingga
menyerupai bentuk wild type-nya sebagaimana ditunjukkan gambar 4. Studi ini
menunjukkan salah satu dampak struktural mutasi yaitu terbentuknya celah besar untuk akses air atau celah internal hidrofobik tanpa perubahan struktur tapi menyebabkan penurunan stabilitas termodinamika.
Gambar 4. Superposisi bentuk stereo dari backbone wt-p53 tanpa DNA(rantai
A,biru), wt-p53 dengan DNA(rantai B, jingga) dan quadruple mutan
p53(hitam) (Joerger et al., 2004).
Studi reaktivasi p53 dengan ligan PRIMA-1 telah dilakukan oleh Warsino (2008) menggunakan metode Docking. Metode Docking dilakukan pada
wild type p53 (wt-p53) manusia (kode PDB:wt-p53) dan tikus, serta beberapa
mutan p53 yakni mutasi pada hotspot 245, mutasi pada 273, serta mutasi diluar
hotspot (kode PDB:QMT). Molekul QMT adalah p53 termutasi melibatkan
quadruple mutan M133L/V203A/N239Y/N268D.
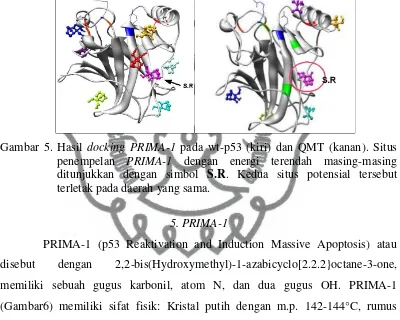
Pada gambar 5 ditunjukkan situs potensial PRIMA-1 menempel pada
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
10
pada daerah inti β-sandwich dengan energi docking yang relatif sama sebesar
-6,24 kkal/mol untuk wt-p53 dan -5,92 kkal/mol untuk QMT. PRIMA-1 dikatakan tidak cukup spesifik berinteraksi pada wt-p53 dan QMT karena beda energi saat dilakukan docking awal dan lanjutan kurang dari 2 kkal/mol (dibawah residual error autodock) (Morris et al., 1998).
Gambar 5. Hasil docking PRIMA-1 pada wt-p53 (kiri) dan QMT (kanan). Situs
penempelan PRIMA-1 dengan energi terendah masing-masing
ditunjukkan dengan simbol S.R. Kedua situs potensial tersebut terletak pada daerah yang sama.
5. PRIMA-1
PRIMA-1 (p53 Reaktivation and Induction Massive Apoptosis) atau disebut dengan 2,2-bis(Hydroxymethyl)-1-azabicyclo[2.2.2}octane-3-one, memiliki sebuah gugus karbonil, atom N, dan dua gugus OH. PRIMA-1 (Gambar6) memiliki sifat fisik: Kristal putih dengan m.p. 142-144°C, rumus molekul C9H15NO, dan berat molekul 185.2 (Bykov et al., 2002b).
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
11
6. Simulasi Kimia
Sebelum adanya komputer, percobaan untuk melihat interaksi antar partikel dilakukan dengan model fisika sederhana yang dilakukan oleh Bernal pada tahun 1962 yaitu dengan mengikat beberapa bola karet menggunakan pengikat dengan variasi panjang pengikat, kemudian mengamati interaksi yang terjadi antar bola karet setiap 5 menit sekali. Beberapa percobaan terus dikembangkan hingga tahun 1998, penghargaan nobel diberikan kepada Walter Kohn dan John Pople karena mengaplikasikan mekanika kuantum dalam memecahkan masalah struktur dan reaksi kimia dari molekul kecil (Becker et al., 2001). Mulai dari eksperimen sederhana, studi simulasi kimia menggunakan bantuan komputer, yang disebut dengan teknik simulasi kimia terus dikembangkan sampai saat ini.
Metode simulasi komputer memudahkan kita untuk mempelajari beberapa sistem dan memprediksikan sifat-sifatnya dengan penggunaan teknik yang mempertimbangkan replikasi yang kecil dari sistem makroskopik dengan sejumlah atom atau molekul yang dapat diatur. Simulasi menghasilkan suatu konfigurasi yang representatif dari replikasi yang kecil ini dalam beberapa cara yang nilai akurat dari sifat-sifat struktural dan termodinamiknya dapat diperoleh dengan sejumlah komputasi yang mungkin mudah dikerjakan. Teknik simulasi juga memungkinkan perilaku bergantung-waktu dari sistem atomik dan molekuler untuk didekati, menyediakan suatu gambaran yang detail dari cara di mana sistem berubah dari satu konformasi atau konfigurasi ke yang lain. Teknik simulasi juga digunakan secara luas dalam beberapa prosedur eksperimental, seperti pendekatan struktur protein dari kristalografi sinar X (Leach, 2001).
Menurut Leach (2001) ada dua jenis teknik simulasi yang umum dalam pemodelan molekuler adalah metode Dinamika Molekuler(DM) dan Monte Carlo
(MC). Simulasi DM dan Monte Carlo berbeda dalam berbagai hal. Perbedaan
signifikan adalah dalam hal DM menyediakan informasi mengenai ketergantungan waktu sifat-sifat sistem sedangkan konfigurasi successive Monte
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
12
Dalam simulasi Monte Carlo pengeluaran dari tiap-tiap percobaan
pergerakan hanya tergantung pada immediate predecessor, sedangkan DM
memungkinkan untuk memprediksikan konfigurasi sistem di setiap waktu di waktu yang akan datang atau waktu-waktu yang sudah terlewati.
DM memiliki kontribusi energi kinetik terhadap total energi sedangkan dalam simulasi Monte Carlo total energi ditentukan secara langsung dari fungsi
energi potensial (Leach, 2001). Simulasi DM dari beberapa makromolekul biologi yang menarik seperti DNA dan kompleks DNA-protein telah terbukti menjadi cara
tepat memahami lebih dalam struktur dan sifat-sifat dinamikanya (Wibowo et al., 2005).
7. Simulasi Dinamika Molekuler (DM)
Simulasi dinamika molekuler merupakan metodologi untuk model mikroskopis secara detil dalam skala atomic dengan mengamati proses ketergantungan waktu sistem molekuler dan secara numeric memecahkan persamaan gerak hukum Newton. Hasilnya adalah suatu trajektori yang menspesifikkan bagaimana posisi dan kecepatan partikel di dalam sistem bervariasi sesuai waktu. Trajektori dihasilkan dengan menyelesaikan persamaan diferensial yang diwujudkan dalam Hukum Newton 2 (F = ma):
(2.7.1) Persamaan tersebut menggambarkan pergerakan partikel yang bermassa mi
sepanjang satu koordinat (xi) dengan Fxi merupakan gaya pada partikel dalam arah
tersebut (Leach, 2001).
Terdapat empat tahap utama dalam simulasi DM. Pertama penentuan koordinat awal berkaitan dengan penggunaan solven dan pemilihan kotak simulasi. Penggunaan solven dalam simulasi DM dapat dilakukan dengan eksplisit solven maupun implisit solven. Partikel eksplisit solven seperti TIP3P umum digunakan dalam simulasi biomolekuler (Becker dan Watanabe., 2001). Setelah menemukan konfigurasi awal sistem, fase penyeimbangan dilakukan untuk memperoleh sistem yang stabil. Atom-atom makromolekul dan pelarut di sekitarnya yang mengalami fase relaksasi biasanya menghabiskan 10 atau 100 ps
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
13
sebelum sistem mencapai keadaan stasioner.
Sifat-sifat termodinamik seperti temperatur, energi, dan densitas dipantau sampai nilainya stabil. Segmen non-stasioner awal dari trajektori akan dibuang dalam penghitungan sifat-sifat kesetimbangan. Sebelum melakukan simulasi DM, sistem harus diseimbangkan dengan kontrol volume, tekanan, dan temperatur untuk menyesuaikan misalnya densitas pelarut untuk nilai eksperimental dan temperatur sistem untuk temperatur yang dipilih.
Setelah penyeimbangan, fase produksi dimulai, yang akan memproduksi hasil simulasi aktual dengan simulasi DM berdurasi sekitar 1 ns. Pada dasarnya, protokol yang sama seperti pada saat tahap akhir penyeimbangan dapat digunakan. Simulasi DM dapat diteruskan sampai diperoleh konfigurasi molekuler yang memuaskan. Jalannya produksi DM ditampilkan berada pada kondisi jumlah partikel (N), volume (V), dan energi (E) konstan yang mewakili ensembel mikrokanonikal NVE dan memungkinkan pengamatan molekul yang berinteraksi dengan lingkungannya selama interval waktu yang telah ditentukan sebelumnya, biasanya dalam orde nanosekon (Molinelli, 2004).
Makromolekul (protein) memiliki range karakteristik pergerakan yang berbeda-beda.Tipe pergerakan makromolekul menggunakan metode DM menurut Becker Watanabe (2001) dapat dilihat pada tabel dibawah ini:
Tabel 1. Ringkasan Karakteristik Pergerakan dalam Protein
No Tipe Pergerakan Contoh aplikasi Skala waktu dan
perpustakaan.uns.ac.id digilib.uns.ac.id
8. Assisted Model Building with Energy Refinement (AMBER7)
(Case et al., 2002)
DM memiliki beberapa software utama, antara lain AMBER, CHARMM, dan GROMOS. AMBER (Assisted Model Building and Energy Refinement) merupakan kelompok medan gaya untuk biomolekul DM. Paket program
AMBER7 terdiri dari 60 program yang beberapa di antaranya dideskripsikan
sebagai berikut:
a. Antechamber
Antechamber merupakan program yang mengotomatisasi proses
pengembangan deskriptor-deskriptor force field khususnya untuk
molekul-molekul organik. Antechamber dihidupkan dari masing-masing arsip PDB
(format PDB), arsip (‗prepin’) baru dengan format yang dapat dibaca dalam
LEaP untuk digunakan dalam pemodelan molekuler. Deskripsi force field yang
dibuat dirancang untuk sesuai dengan forcefieldAmber yang biasa.
b. Parmchk
Parmchk dibaca dalam suatu arsip ‗ac’ atau arsip input ‗prep’
sebagaimana suatu arsip force field. Parameter menuliskan arsip ‗frcmod’
perpustakaan.uns.ac.id digilib.uns.ac.id
parameter/topologi. Program tersebut meliputi editor molekuler yang memungkinkan pembuatan residu dan memanipulasi molekul.
d. Sander (Simulated Annealing with NMR-derived Energy Restraints)
Sander adalah program utama yang digunakan untuk simulasi DM.
Program ini merelaksasi struktur dengan memindahkan atom-atom secara iteratif menurunkan gradien energi sampai gradien rata-rata yang cukup diperoleh. Porsi DM membentuk konfigurasi sistem dengan menggabungkan persamaan Newtonian tentang gerak. DM akan melakukan sampling ruang
konfigurasional yang lebih banyak daripada minimisasi dan akan memungkinkan struktur untuk melewati halangan energi potensial yang kecil. Konfigurasi dapat disimpan pada interval tetap selama simulasi untuk analisis lebih lanjut, dan perhitungan energi bebas dasar menggunakan integrasi termodinamik dapat dilaksanakan.
e. Ptraj dan Carnal
Ptraj dan Carnal merupakan program-program untuk menganalisa
trajektori-trajektori DM, menghitung (misalnya Root Mean Square deviation
dari struktur referen), analisis ikatan hidrogen, fungsi korelasi waktu, perilaku difusional, dan sebagainya (Molinelli, 2004).
1) RMSD (Root Mean Square Deviation)
Pengukuran kesamaan diperlukan untuk perbandingan kuantitatif suatu struktur dengan lainnya. Kesamaan struktur biasanya diukur dengan
root mean square deviation (RMSD) antara dua konformasi (Becker dan
Watanabe., 2001).
RMSD menyediakan informasi apakah konformasi telah mencapai
suatu keadaan yang stasioner. Deviasi masing-masing frame terhadap
frame pertama dalam trajektori dihitung. Harga ini sangat berguna dalam
perpustakaan.uns.ac.id digilib.uns.ac.id
koordinat Cartesian dari atom k dalam konformasi i dan j. Harga minimum
dari persamaan di atas diperoleh dengan superposisi optimal dari dua struktur (Becker, 2001).
2) B-factor
B-factor adalah ukuran termal dari ketidaktentuan (luasan densitas
elektron) untuk struktur sebagai factor fluktuasi suatu molekul. B-factor ini
ditetapkan terhadap tiap-tiap atom dan dapat dihitung untuk tiap-tiap residu asam amino. Pergerakan termal paling besar biasanya ditemukan pada rantai samping dan loop (Esposito et al., 2006).
B-factor kristalografik dapat digunakan sebagai indikator mobilitas
konformasional atau fleksibilitas protein. Analisis distribusi B-factor telah
digunakan lebih awal untuk menganalisa karakteristik struktural dan fungsional protein (Kumar et al., 2009)
Fluktuasi atomik simulasi dapat diperkirakan dengan B-factor yang
persamaannya sebagai berikut.
𝐵𝑖 = 8𝜋
2
3 <∆𝑟𝑖 >
2 (2.8.2)
Di mana Δri adalah akar pangkat dua fluktuasi posisional atom (Karjiban,
etal., 2009).
3) Struktur Protein dan Sudut Dihedral Backbone Protein
David (2001) dalam buku Lehninger Principles of Biochemistry
menjelaskan tentang protein. Protein adalah molekul besar yang komplekss, yang terdapat dalam semua sel. Pada dasarnya protein disusun oleh suatu rangkaian unit asam amino dengan struktur umum sebagai
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
17
Gambar 7. Struktur umum Asam Amino dengan gugus R berbeda untuk masing-masing asam amino (David et al., 2001)
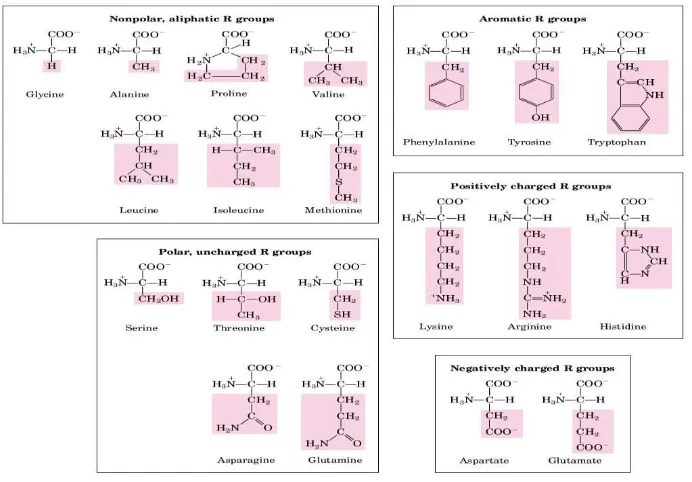
Dua puluh macam asam amino telah teridentifikasi dan dikelompokkan
sesuai dengan gugus R-nya sebagaimana ditunjukkan gambar 8.
Gambar 8. 20 asam amino protein yang dikelompokkan menurut gugus fungsinya (David et al., 2001)
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
18
Gambar 9. Beberapa struktur protein berturut-turut dari kiri ke kanan adalah struktur primer, sekunder, tertier, dan quartener (Jeremy et al., 2007).
Struktur primer protein adalah beberapa asam amino yang dihubungkan oleh ikatan peptida membentuk rantai polipepetida. Struktur sekunder protein yaitu rantai polipeptida yang dapat membentuk lipatan beberapa struktur regular seperti alpha helix, beta sheet, serta turn dan loop.
Struktur tertier protein sendiri umumnya protein yang larut dalam air dan melipat dalam struktur yang padat dengan inti nonpolar. Struktur quartener disebut juga dengan protein multisubunit.
Dalam masalah prediksi struktur sekunder protein, inputnya adalah urutan dan outputnya adalah struktur yang diprediksikan (yang juga disebut konformasi, yang merupakan kombinasi dari alfa heliks, beta
sheet, dan loop). Suatu protein yang khusus mengandung sekitar 32% alfa
heliks, 21% beta sheet, dan 47% loop atau struktur non regular (Branden
dan Tooze, 1991).
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
19
Gambar 10. Sudut dihedral psi (ψ) dan phi (Ф) pada backbone protein
(Arjunan et al., 2001).
Phi adalah sudut rotasi di sekitar ikatan N–C sedangkan psi merupakan sudut rotasi di sekitar ikatan C–C. Rotasi-rotasi menentukan masing-masing struktur protein (seperti alfa heliks, beta sheet, atau loop).
Asam-asam amino yang berada di bagian dalam molekul protein adalah asam-asam amino golongan hidrofobik sedangkan asam-asam amino yang bersifat polar berada di permukaan protein dan biasanya memiliki urutan dan konformasi asam-asam amino yang sama, sehingga memiliki fungsi dan sifat-sifat yang sama pula (Arjunan et al., 2001).
Konfigurasi backbone protein sepenuhnya ditentukan oleh
spesifikasi sudut dihedral Ф dan ψ. Korelasi sudut dihedral dalam protein asli dan terdenaturasi sangat penting karena mengandung sumber utama informasi dalam folding dan stabilitas protein (Keskin, 2004).
B. Kerangka Pemikiran
Studi tentang struktur dasar mutasi hotspot seringkali menyertakan
quadruple mutan M133L/V203A/N239Y/N268D. Struktur QMT p53 sering
digunakan karena dinyatakan sebagai superstable mutan dan memiliki kemiripan
dengan struktur normal p53. Berdasarkan kemiripan struktur dan stabilitas struktur yang tinggi, kedua molekul diduga memiliki dinamika konformasi yang sama. Hal ini berkaitan erat dengan fungsi p53 normal dalam berikatan dengan
DNA. Permasalahan yang muncul disini adalah apakah benar kedua molekul
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
20
Studi tentang penempelan PRIMA-1 pada wt-p53 dan QMT (Docking)
telah dilakukan oleh Warsino (2008). Cara ini terbukti dapat merubah konformasi mutan p53 menyerupai p53 normal. Namun docking yang telah dilakukan
terhadap wt-p53 dan QMT p53 dikatakan tidak spesifik. Ketidakspesifikan penempelan PRIMA-1 ditandai dengan ditemukannya situs penempelan yang
sama antara keduanya dan menghasilkan energi docking yang hampir sama antara
keduanya sebelum dan sesudah docking lanjutan. Permasalahan yang muncul
disini adalah belum ada pengamatan dinamika konformasi penempelan PRIMA-1
pada wt-p53 dan QMT p53 untuk menunjukkan kespesifikan penempelan
PRIMA-1.
Permasalahan ada tidaknya perbedaan yang nyata antara molekul wt-p53 dan QMT p53, serta karakter kespesifikan PRIMA-1 menempel pada wt-p53 dan
QMT p53 dapat diselesaikan dengan melihat dinamika konformasi wt-p53, QMT p53, dan kompleks PRIMA-1 dengan wt-p53 saja dengan asumsi ada kemiripan
struktur antara wt-p53 dengan QMT p53. Pengamatan dinamika konformasi ketiga molekul tersebut dilakukan dengan simulasi dinamika molekuler (DM) program AMBER7 kurun waktu 5-9 ns.
C. Hipotesis
1. Dinamika konformasi QMT p53 tidak berbeda nyata dengan wt-p53 dengan asumsi adanya kemiripan struktur QMT p53 dan wt-p53.
2. Keberadaan PRIMA-1 pada wt-p53 di situs energi terendah memberikan
perpustakaan.uns.ac.id digilib.uns.ac.id bertempat di Laboratorium Kimia Dasar bagian Komputasi Kimia jurusan Kimia FMIPA UNS.
B. Alat dan Bahan yang Dibutuhkan
1. Alat
Seperangkat komputer dengan spesifikasi : CPU berprosesor AMD Athlon
(tm) 64 X2 Dual Core Processor 5200+, 2.60 GHz, RAM 4 GB, dan harddisk
2x250 GB. Perangkat lunak berupa program AMBER7 (Case, et al., 2002),
program Molden (Klinsky et al., 2002), MATLAB7 (MathWorks, 2004),
CHIMERA (Pettersen etal., 2004), dan VMD (Humphrey et al., 1996).
2. Bahan
Struktur p53 wild type pada manusia kode PDB = 1GZH (Derbyshire etal.,
2002), quadruple mutan M133L/V203A/N239Y/N268D kode PDB = 1UOL
(Joerger etal., 2004), dan PRIMA-1.
C. Prosedur Penelitian
1. Parameterisasi PRIMA-1
Parameterisasi PRIMA-1 dilakukan dengan mengambil terlebih dahulu
struktur PRIMA-1 teroptimasi dari hasil penelitian Warsino (2008). Densitas
elektron (Electrostatic Potensial/ESP) dihitung dengan GAUSSIAN98 (Frisch et
al., 1995) metode ab initio pada level teori HF dan basis set 6-31G*. Populasi
elektron dihitung dengan metode Mulliken. Arsip log yang dihasilkan kemudian
diolah dengan program Antechamber dan parmchk dalam AMBER7 di mana di
dalamnya terdapat RESP untuk metode penghitungannya. Hasilnya berupa arsip
prep dan arsip frcmod sebagai template dan parameter ligan PRIMA-1 yang akan
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
22
2. Penentuan Koordinat Awal Sistem
Simulasi dilakukan terhadap tiga sistem yaitu wt-p53, QMT, dan kompleks
PRIMA-1 dengan wt-p53. Sistem QMT dilakukan sebanyak dua kali untuk
memperoleh probabilitas kondisi yang lain. Pada semua sistem ion Cl- sebagai
counterion ditambahkan menggunakan modul XLEAP dalam AMBER7. Sistem
kemudian disolvasi dengan penambahan eksplisit solvent berupa model air TIP3P
(Jorgensen, et al., 1983) yang berupa sekumpulan molekul air yang berbentuk kotak yang melingkupi sistem dengan jarak antara sistem dan model air sebesar
12 Ǻ. Setelah itu, sistem tersebut disimpan dalam format arsip pdb (urutan atom
dan posisinya), arsip prmtop (topologi sistem), dan arsip prmcrd (parameter sistem) yang nantinya akan digunakan dalam proses minimisasi, penyeimbangan, dan simulasi.
3. Minimisasi Sistem
Agar proses solvasi sempurna (yaitu jarak model air dekat dengan sistem), maka dilakukan minimisasi. Minimisasi sistem dilakukan sebanyak 500 step di
mana tiap 100 step besarnya penahanan harmonik pada makromolekul dan
counterion diubah. Pada 100 step pertama, besarnya penahanan harmonik pada
makromolekul dan counterion adalah sama-sama sebesar 25 kcal/mol-1A-2. Pada
100 step 2, besarnya penahanan harmonik pada makromolekul tetap 25 kcal/mol
-1A-2 dan pada
counterion hanya sebesar 20 kcal/mol-1A-2. Pada 100 step ketiga,
besarnya penahanan harmonik pada makromolekul adalah 20 kcal/mol-1A-2 dan pada counterion hanya 15 kcal/mol-1A-2. Pada step keempat, besarnya penahanan
harmonik pada makromolekul adalah 15 kcal/mol-1A-2 dan pada counterion hanya
sebesar 10 kcal/mol-1A-2. Pada step kelima, besarnya penahanan harmonik pada
makromolekul adalah 10 kcal/mol-1A-2 dan pada
counterion hanya sebesar 5
kcal/mol-1A-2. Minimisasi ini akhirnya dilakukan tanpa adanya restraints.
4. Equilibrasi Sistem
Penyeimbangan dilakukan dengan pemanasan bertahap 50-300 K (sesuai suhu sistem yang sebenarnya) selama 200 ps di mana makromolekul dan posisi-posisi ion dijaga konstan dengan penahanan harmonik (harmonic restraint) 25
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
23
25 ps berikutnya, hingga akhirnya kesetimbangan akan berlangsung tanpa adanya penahanan. Untuk meningkatkan dan menjaga temperatur sistem pada 300 K digunakan algoritma Berendsen (Berendsen etal., 1984).
5. Simulasi Sistem
Simulasi dijalankan pada temperatur konstan 300 K, tekanan 1 atm, 2 fs
time step, SHAKE constraints 0,00005 Ǻ (mengabaikan vibrasi yang melibatkan
atom hidrogen), nonbonded cutoff9 Ǻ, dan 0,00001 untuk prosedur particle-mesh
Ewald (PME) (Kawata et al., 2001) yang digunakan untuk menangani interaksi
elektrostatik yang jangkauannya jauh (long-range electrostatic interactions).
Informasi struktural dikumpulkan setiap 500 step (1 ps).
D. Teknik Pengumpulan dan Analisis Data
Data yang berupa trajektori hasil simulasi DM diolah dengan perangkat analisis yang terdapat dalam program AMBER7 (ptraj) dan program MATLAB7.
Pengamatan awal dilakukan terhadap perubahan nilai RMSDnya. Faktor fluktuasi
penyebab perubahan nilai RMSD dapat dicari melalui profil B-factornya.
Program CHIMERA dan VMD digunakan untuk menampilkan data secara visual.
perpustakaan.uns.ac.id digilib.uns.ac.id
Fungsi parameterisasi PRIMA-1 adalah mendapatkan
parameter-parameter PRIMA-1 yang diperlukan dalam proses minimisasi, equilibrasi, dan
simulasi. Struktur PRIMA-1 diambil dari hasil penelitian Warsino (2008)
sedangkan koordinat atom wt-p53 diambil dari data pdb (kode pdb : wt-p53). Koordinat hidrogen ditambahkan dengan program XLEAP dalam AMBER7.
Muatan digambarkan sebagai RESP (Restrained ElectroStatic Potensial) dihitung
dengan GAUSSIAN98 pada level teori HF/6-31G*. Hasil parameterisasi PRIMA-1
disajikan pada gambar 11 dan tabel 2 berikut.
Gambar 11. Struktur PRIMA-1
Terparameterisasi
Tabel 2. Kode atom, tipe atom, dan muatan PRIMA-1 yang
1. Perbandingan perilaku wild type p53 (wt-p53) dan quadruple mutan
M133L/V203A/N239Y/N268D (QMT)
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
25
dilakukan adalah pengamatan pergesaran posisi rata-rata molekul tiap waktu terhadap posisi awalnya yang disebut dengan RMSD (Root Mean Square
Deviation).
Hasil simulasi DM berupa profil RMSD total wt-p53, QMT_1 (simulasi
QMT yang pertama), dan QMT_2 (simulasi QMT yang kedua) ditunjukkan oleh gambar 12. Gambar tersebut menujukkan ketiga sistem sama-sama bergeser naik dari 1Å ke kisaran 2,3Å diawal simulasi hingga 800 ps. Hal ini menunjukkan ketiga sistem tersebut masih mengalami tahap equilibrasi artinya masih mencari posisi stabil sebelum simulasi. Pada saat 1-2 ns perbedaan pergeseran posisi mulai terjadi antara wt-p53 dan QMT_2 dengan QMT_1. Baik wt-p53 dan QMT_2 keduanya turun dulu baru naik, sedangkan QMT_1 naik dulu baru turun. Gambaran ini menunjukkan posisi wt-p53 pada saat tersebut bisa dicapai oleh QMT_2 tetapi QMT_1 tidak bisa.
Rentang waktu berikutnya antara 2-3 ns ketiga sistem bertemu pada jarak yang sama. Setelah 3 ns QMT_1 sempat naik lagi tapi kembali turun dan stabil pada jarak 2,5Å hampir sama dengan profil wt-p53 yang naik dulu tapi kembali lagi dan stabil pada jarak 2,5Å.
Profil QMT_2 relatif terus naik perlahan hingga akhir simulasi namun jika dibandingkan dengan profil p53 kenaikan QMT_2 masih mendekati wt-p53 diarea 2,5Å. Pada pertengahan hingga akhir simulasi profil RMSD ketiga
sistem stabil pada jarak yang sama, meski pada rentang waktu 1-2 ns ada perbedaan pergeseran posisi ketiganya. Perbedaan posisi yang terjadi pada ketiga sistem berkaitan dengan faktor fluktuasi atomik rata-rata ketiga sistem tersebut. Oleh karena itu dilakukan analisis ptraj B-factor ketiga sistem untuk menjelaskan
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
26
Gambar 12.Perbandingan profil RMSD (Å) terhadap waktu (ps) hasil simulasi DM untuk quadruple mutan QMT_1 (garis merah), QMT_2(garis
biru), dan wild type p53 wt-p53 (garis hitam).
Hasil simulasi DM yang telah dilakukan untuk QMT dan wt-p53 menunjukkan profil B-factor yang relatif sama meski masih ada fluktuasi yang
lebih tingi untuk QMT. Jika dibandingkan dengan wt-p53 fluktuasi QMT lebih tinggi pada beberapa residu yang memang lebih fluktuatif dibanding yang lain. Pada gambar 13 dibawah nilai B-factor QMT hasil simulasi DM menunjukkan
nilai yang lebih tinggi pada daerah tertentu (residu 120, 181, 186, 226, dan 248).
Gambar 13.Perbandingan profil B-factor (Å2) terhadap nomor residu untuk
quadruple mutan QMT_1 (garis merah), QMT_2 (garis biru), rerata QMT_1 dan QMT_2 (orange) dan wild type p53 wt-p53 (garis hitam).
Perulangan simulasi DM untuk QMT dilakukan untuk memastikan fluktuasiQMT. Hasil simulasi ternyata menunjukkan profil B-factor yang berbeda
120
181 186
248 226
RMSD (Å)
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
27
meski dilakukan pada rentang waktu yang sama yaitu 5ns. B-factor QMT_2 yang
ditunjukkan gambar 13 secara keseluruhan tampak lebih mendekati wt-p53 dari pada B-factor QMT_1 meskipun masih ada beberapa residu yang lebih fluktuatif
(120, 186, dan 248).
Perbedaan profil fluktuasi QMT ini hanya menunjukkan probabilitas dinamika molekulnya dimana suatu keadaan bisa dicapai seperti QMT_1 sedangkan keadaan yang lain bisa saja dicapai seperti QMT_2. Bahkan hasil rata-rata B-factor keduanya tidak jauh berbeda dan sangat mendekati wt-p53
sebagaimana ditunjukkan oleh garis orange pada gambar 13. Oleh karena itu untuk mengamati lebih dalam perbedaan profil B-factor yang ada berbeda nyata
atau tidak dilakukan analisis sudut dihedral Ф (phi) dan ψ (psi) backbone residu
yang penting untuk berikatan dengan DNA dan residu lain yang berbeda cukup
signifikan.
a. Residu Lisin 120 dan Arginin 248
Baik QMT_1 maupun QMT_2 keduanya menunjukkan fluktuasi pada residu 120 dan 248 yang penting untuk berikatan dengan DNA. Residu 120
adalah asam amino lisin yang merupakan bagian dari loop L1 (Wong etal.,1999)
dan berikatan hidrogen dengan O6 dan N7 dari basa Gua 8 dalam major groove
(lekukan besar) DNA, sedangkan arginin 248 berikatan dengan minor groove DNA
(Wright, 2007).
Pada residu lisin 120 profil B-factor QMT tampak sedikit lebih fluktuatif
dari wt-p53. Faktor fluktuasi berasal dari kontribusi fluktuatif sudut torsi phi (Ф4)
yaitu backbone C120:N121:CA121:C121 dari residu lisin 120 dan serin 121 baik
untuk QMT_1 maupun QMT_2. Ada fluktuasi sudut sebesar 50° pada kisaran 1,5ns hingga akhir simulasi namun masih dominan pada sudut -50° yaitu posisi yang sama dengan wt-p53. Selain itu ada juga perubahan sudut torsi ψ5 backbone
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
28
rantai samping wt-p53 masih dominan dapat dicapai oleh QMT_1 atau QMT_2. Posisi yang sama kita lihat dapat terjadi saat 1,5 ns; 2,5 ns; dan 4 ns.
A
B
Gambar 14. Profil perbandingan fluktuasi sudut dihedral Psi (ψ) pada baris bawah
dan Phi (Ф) pada baris atas residu 118-123 (melibatkan residu lisin
120) antara wt-p53, QMT_1, dan QMT_2 berturut-turut ditunjukkan dengan warna hitam, merah, dan biru, A. Grafik fluktuasi sudut dihedral terhadap waktu yang fluktuatif pada Ф4 memuat residu 120
dan 121 dan ψ5 yang memuat residu 121 dan 122. B. Posisi sudut
dihedral selama simulasi dengan backbone residu lisin 120 dan serin
121 digambarkan sebagai stik dan bola sedangkan rantai samping lisin 120 digambarkan sebagai stik.
Lain halnya dengan residu 248, baik wt-p53 dan QMT keduanya memiliki tingkat fluktuasi yang hampir sama berdasarkan profil B-factornya
namun terdapat perbedaan yang signifikan pada profil sudut torsinya.Perbedaan
terjadi pada sudut dihedral Ф3 yaitu backbone C246:N247:CA247:C247 dari
residu 246 (metionin) dan 247 (asparagin) serta ψ4 backbone
N247:CA247:C247:N248 yang memuat residu 247 (asparagin) dan 248 (arginin) masing-masing sebesar 100°. Meski berbeda tapi menunjukkan garis yang stabil dari awal hingga akhir simulasi. Perbedaan ini menjelaskan satu kondisi yang
0 ns 1,5ns
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
29
berbeda dapat dicapai wt-p53 dan QMT stabil dari awal hingga akhir simulasi 5 ns sehingga dapat dikatakan stabil secara termodinamika sebagaimana ditunjukkan gambar 15A.
Pada gambar 15B kita melihat adanya perubahan cukup signifikan saat 1 dan 2 ns. Backbone wt-p53 residu 244-246 berubah dari loop menjadi sheet
sedangkan QMT tetap pada struktur loop. Saat 3 ns sheet wt-p53 244-245 berubah
menjadi loop kembali tak lama kemudian berubah kembali saat 4 ns. Gambar 14B
memang menunjukkan fluktuasi konformasi backbone 244-245 tetapi profil grafik
torsi Ф1, Ф2, ψ1, dan ψ2 tampak stabil. Hal ini menunjukkan konformasi sheet
protein sama dengan konformasi loopnya.
Pada backbone residu 246-248 gambar 15A tampak adanya perubahan
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
30
A
B
Gambar 15. Profil perbandingan fluktuasi sudut dihedral Psi (ψ) dan Phi (Ф)
residu 244-248 (melibatkan residu arginin 248) antara wt-p53, QMT_1, dan QMT_2 berturut-turut ditunjukkan dengan warna hitam, merah, dan biru, A. Grafik fluktuasi sudut dihedral terhadap waktu yang fluktuatif
pada Ф3 memuat residu 246 dan 247 serta ψ4 yang memuat residu 247
dan 248. B. Perubahan sudut dihedral selama simulasi dengan backbone
residu 244-249 digambarkan sebagai ribbon sedangkan rantai samping
arginin 248 digambarkan sebagai stik.
Berdasarkan hasil simulasi kedua residu tersebut dapat disimpulkan
quadruple mutan ini tidak berpengaruh signifikan terhadap residu lisin 120 dan
arginin 248. Quadruple mutan ini bisa membuat residu lisin 120 lebih fluktuatif
namun masih dominan pada posisi yang sama dengan wt-p53. Pada residu 248
quadruple mutan ini memang memberikan perubahan signifikan untuk rantai
utamanya namun tidak signifikan karena masih pada posisi yang berdekatan dengan wt-p53.
b. Residu Arginin 181, Asam Aspartat 186, dan Glysin 226
Pengamatan berikutnya dilakukan pada residu-residu yang lebih fluktuatif ditandai dengan nilai puncak B-factor yang lebih tinggi dibanding yang
0 ns 1 ns 2 ns 3 ns 4 ns
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
31
lain, yaitu daerah residu 181,186, dan 226. Pada dasarnya daerah yang sangat fluktuatif dari hasil simulasi DM sama dengan hasil kristalografi sinar-x nya yaitu adanya perbedaan struktur pada daerah loop L1, loop L2, loop L3, dan paling signifikan terjadi pada loop putaran strand S7 dan S8 (gambar 16); dimana, residu 181 dan 186 pada loop L2, serta residu 226 terdapat pada loop putaran S7-S8.
Gambar 16.Perbandingan struktur hasil kristalografi sinar-x wt-p53 dan QMT dalam bentuk stereo dari backbone wild type-p53 tanpa DNA (rantai
A,biru), wild type p53 dengan DNA(rantai B, jingga) dan quadruple
mutan p53(hitam) setelah dihimpitkan (Joerger,et al, 2004).
Sudut dihedral psi(ψ) dan phi (Ф) untuk backbone residu-residu
fluktuatif pada gambar 17 menunjukkan adanya banyak kontribusi perubahan sudut dihedral terhadap nilai B-factor residu-residu tersebut. Pada gambar 17A
kita melihat perbedaan profil Ф1 backbone C179:N180:CA180:C180 untuk
QMT_1 dan QMT_2 dibandingkan dengan wt-p53. Meskipun torsi Ф1 dari QMT_1 dan QMT_2 saling berbeda satu sama lain namun masing-masing kondisi masih dapat dicapai oleh wt-p53 pada satu waktu tertentu. Sehingga perbedaan yang ditunjukkan pada dasarnya tidak cukup nyata antara wt-p53 dan QMT.
Lain halnya dengan rentang residu 184-188 pada gambar 17B. Profil torsi QMT_1 dan QMT_2 untuk Ф2 backbone C184:N185:CA185:C185 dan ψ3
backbone N185:CA185:C185:N186 nampak berbeda dibandingkan dengan
wt-p53. Profil torsi Ф2 untuk QMT_2 saat kisaran 4 ns masih bisa dicapai oleh
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
32
Perubahan yang cukup signifikan terjadi pada ψ3 residu serin 185 dan
asam aspartat 186. Backbone kedua residu tersebut mengalami perubahan sudut
hampir 200○ sejak 500 ps hingga 4500 ps. Backbone tersebut sempat kembali
pada kondisi wt-p53 sejenak lalu berubah lagi hingga akhir simulasi. Perubahan sudut backbone tampak jelas pada gambar 18. Gambar 18 menunjukkan ada
tekukan besar residu serin 185 dan asam aspartat 186 untuk QMT_2 berangsur-angsur selama simulasi. Sedangkan untuk QMT_1 sendiri sempat ada perubahan mendekati arah perubahan QMT_2 namun masih dominan pada kondisi wt-p53 yaitu pada sudut 100○.
Residu 226 adalah residu yang sangat fluktuatif ditandai dengan nilai
B-factor paling tinggi dibanding yang lain. Residu ini terletak pada loop putaran S7
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
33
A
Ф1 Ф2 Ф3
Ф4
Ψ1 Ψ2 Ψ3 Ψ4
B
Ф1 Ф2 Ф3
Ф4
Ψ1 Ψ2 Ψ3 Ψ4
C
Ф1 Ф2 Ф3 Ф4 Ф5
Ψ1 Ψ2 Ψ3 Ψ4 Ψ5
Gambar17. Profil sudut dihedral Psi (ψ) dan Phi (Ф), A. Residu 179-181, B. Residu 184-188, C. Residu 225-230
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
34
sama secara umum artinya kondisi wt-p53 sebagai pembanding masih dapat dicapai oleh quadruple mutan meski ada perbedaan namun secara umum dominan
sama dengan wt-p53.
0ns 1ns 2ns
3ns 4ns 5ns
Gambar18. Perubahan sudut dihedral selama simulasi 0-5ns dengan backbone
residu asam amino 185-187 berturut-turut dari atas ke bawah digambarkan sebagai ribbon sedangkan rantai sampingnya
digambarkan sebagai stik.
Dengan asumsi quadruple mutan tidak berpengaruh cukup signifikan
terhadap residu penting p53 berikatan DNA (lisin 120, glysin 245, arginin 248,
dan arginin 273) dan adanya kesamaan ketiga karakter dinamika konformasi molekul residu fluktuatif, maka dapat dikatakan quadruple mutan
M133L/V203A/N239Y/N268D QMT tidak berbeda nyata dengan wild type p53
wt-p53. Akan tetapi perlu adanya studi lebih lanjut mengenai interaksi quadruple mutan M133L/V203A/N239Y/N268D dengan DNA khususnya pada residu 120,
245 dan 248.
2. Perilaku PRIMA-1 pada wild type p53 (wt-p53)
perpustakaan.uns.ac.id digilib.uns.ac.id
commit to user
35
Setelah parameterisasi selesai diperoleh, dilakukan penempelan PRIMA-1 pada
wt-p53 dengan XLEAP AMBER7 kemudian minimisasi, equilibrasi dan
selanjutnya simulasi selama 5ns sebagaimana molekul wt-p53 dan QMT tanpa
PRIMA-1 sebelumnya.
Hasil simulasi kemudian diolah dengan analisis ptraj. Analisis yang
pertama dilakukan adalah pengamatan perubahan posisi RMSD wt-p53 dengan
kompleks. Penambahan PRIMA-1 pada wt-p53 memberikan efek perubahan posisi RMSD jika dibandingkan dengan wt-p53 tunggal. Hal ini terjadi karena ada
penambahan berat molekul PRIMA-1. Meski demikian jika diamati lebih lanjut
dapat dilihat bahwa karakter perubahan posisi yang terjadi antara wt-p53 tunggal dan kompleks relatif sama sebagaimana ditunjukkan gambar 19 dibawah. Jika
RMSD wt-p53 mulai stabil pada 2.5Å setelah 3 ns, pada interaksinya dengan
PRIMA-1 profil RMSD juga mulai stabil pada posisi 2.5Å setelah 4500ps. Untuk
itu perlu adanya simulasi lanjutan untuk kedua molekul tersebut diatas 5 ns guna mengamati lebih lanjut perubahan posisi kedua molekul tersebut hingga mencapai posisi stabilnya.
Gambar 19.Perbandingan profil RMSD (Å) terhadap waktu (ps) hasil simulasi DM untuk wild type p53 wt-p53 (garis hitam) dan kompleks p53
dengan PRIMA-1.
Pengamatan terhadap salah satu karakter dinamika molekul yaitu fluktuasi molekul dapat dilihat dari profil B-factor nya sebagaimana ditunjukkan
gambar 20 dibawah. Adanya PRIMA-1 tampak meningkatkan nilai B-factor
beberapa residu asam amino wt-p53 menjadi lebih fluktuatif sedangkan pada RMSD
(Å)