ANALISIS VARIASI GENETIK AMORPHOPHALLUS MUELLERI BLUME DARI BERBAGAI POPULASI DI JAWA TIMUR BERDASARKAN SEKUEN INTRON trnL
Erta Puri Rosidiani*), Estri Laras Arumingtyas & Rodliyati Azrianingsih
Program Studi Magister Biologi, Fakultas MIPA, Universitas Brawijaya Malang
*) e-mail: [email protected]
Erta Puri Rosidiani, Estri Laras Arumingtyas & Rodliyati Azrianingsih. 2013. Analysis of Genetic Variation of Amorphophallus muelleri Blume from Several Populations in East Jawa Based on Sequence Intron trnL.
Floribunda 4(6): 129–137. — The objective of this study is to determine the genetic variability of porang at
different populations based on analysis of trnL intron sequences. Research was conducted in East Jawa, which is porang population at Jember (Mayang: MY), Malang (Lawang: LW, Kalipare: KP), Blitar (Brongkos: BL), Nganjuk (KPH tritik: TR), Madiun (Klangon: KL, KPH Saradan: MD), Bojonegoro (Klino: KN). Exploration conducted in 8 populations to take samples of young leaves as many as three sheets at two different individuals of each population. Then extract the DNA, and amplify the trnL intron using universal primers 'c' and 'd', then were purified and sequenced. The results of sequencing were analyzed the genetic variation and relationship based on Maximum Parsimony tree by MEGA5 software. Sequencing of 16 indi-viduals porang, produce 13 indiindi-viduals which have a clear chart chromatography which is resulting in the proper sequence and can be used in subsequent analyzes. The sequences showed the differences in nucleo-tide base composition of each individual. Six region is also known as conserved region, 52 sequences nu-cleotide variability, and 8 deletion. The phylogeny tree showed four clade Major. Porang were clustered ran-domly, not based on the population to which the individual is taken. Thus, the origin of porang at each study site cannot be identified based on the geographical analysis.
Keywords: Amorphophallus muelleri, genetic variability, sequences, trnL intron, East Jawa.
Erta Puri Rosidiani, Estri Laras Arumingtyas & Rodliyati Azrianingsih. 2013. Analisis Variasi Genetik
Amorphophallus muelleri Blume dari Berbagai Populasi di Jawa Timur Berdasarkan Sekuen Intron trnL. Floribunda 4(6): 129–137. — Penelitian ini bertujuan mengetahui variasi genetik porang pada populasi yang
berbeda berdasarkan analisis sekuen intron trnL. Penelitian ini dilakukan di Jawa Timur, yaitu di Jember (Mayang: MY), Malang (Lawang: LW, Kalipare: KP), Blitar (Brongkos: BL), Nganjuk (KPH tritik: TR), Madiun (Klangon: KL, KPH Saradan: MD), dan Bojonegoro (Klino: KN). Eksplorasi di delapan populasi bertujuan untuk mengambil sampel daun muda sebanyak tiga lembar pada dua individu berbeda di masing-masing populasi. Selanjutnya DNA diekstrak, diamplifikasi menggunakan primer universal ‘c’ dan ‘d’, lalu dipurifikasi dan disekuensing. Hasil sekuensing dianalisis variasi genetik dan kekerabatan berdasarkan pohon Maksimum Parsimony menggunakan program MEGA5. Berdasarkan sekuensing 16 individu porang diperoleh 13 individu dengan grafik kromatografi yang jelas, menghasilkan pembacaan sekuen yang tepat sehingga dapat digunakan dalam analisis selanjutnya. Sekuen yang dihasilkan menunjukkan perbedaan komposisi basa nukleotida pada masing-masing individu. Enam region diketahui sebagai ”conserved region”, perubahan basa nukleotida sebanyak 52 sekuen, dan delesi pada 8 sekuen. Pohon filogeni menunjukkan adanya empat klad besar. Individu porang mengelompok secara acak, tidak berdasarkan lokasi populasi pengambilan sampel. Sehingga, asal porang pada masing-masing populasi tidak dapat diketahui berdasarkan analisis geografi.
Kata kunci: Amorphophallus muelleri, intron trnL, porang, populasi, sekuen, variasi genetik, Jawa
Timur.
Amorphophallus muelleri mempunyai
per-sebaran dari Thailand tengah ke selatan melalui Sumatera, Jawa hingga ke Kepulauan Sunda Kecil. Porang (A. muelleri Blume) merupakan satu dari 27 jenis Amorphophallus yang ada di Indonesia dan dari 170 jenis yang dikenal di dunia. Porang merupakan tanaman sumber karbohidrat alternatif
yang mengandung glukomanan tertinggi di antara jenis Amorphophallus lainnya di Indonesia (Jansen et al. 1996, Sumarwoto 2005). Berdasarkan pene-litian yang dilakukan oleh Poerba & Martanti (2008) pada 50 aksesi di Jawa Timur melalui metode RAPD, diketahui adanya variasi genetik yang ditunjukkan pada perbedaan pola pita DNA,
yaitu 69.05% merupakan polimorfik. Hasil pene-litian tersebut juga mengungkapkan adanya pemi-sahan aksesi ke dalam dua klaster utama, dengan nilai keanekaragaman genetik tertinggi ada pada populasi Saradan. Selain informasi keanekara-gaman genetik, Azrianingsih et al. (2008) mela-porkan adanya ciri morfologi yang paling menon-jol, yaitu warna dan corak tangkai daun yang ditemukan pada sembilan sampel porang, dari sembilan lokasi di Jawa Timur. Kombinasi keane-karagaman morfologi dan molekular diperlukan untuk identifikasi dan seleksi sumberdaya genetik porang sebagai materi dasar pemuliaan tanaman.
Penelitian sumberdaya genetik yang dilaku-kan Poerba & Martanti (2008) terhadap porang pa-da level infraspesies memiliki kelemahan papa-da metode yang digunakan. Metode random amplified polymorphic DNA (RAPD) yang digunakan Poer-ba & Martanti (2008), memiliki kelemahan dalam konsistensi hasil amplifikasi. Ketika dilakukan pe-ngulangan amplifikasi dengan primer dan sampel yang sama maka hasil yang didapatkan berbeda dengan hasil amplifikasi awal, sehingga akan mempengaruhi hasil analisis. Hal ini terjadi karena ukuran sekuen primer yang pendek, sehingga dapat menempel pada sekuen yang berbeda dari ampli-fikasi awal (Jones et al. 1997). Oleh karena itu perlu digunakan penanda molekular lain yang dapat memberikan hasil amplifikasi yang konsis-ten. Penanda molekular yang telah digunakan dalam analisis filogenetik dan memiliki hasil amplifikasi yang konsisten adalah sekuen intron DNA kloroplas seperti matK, rbcL, trnK dan trnL (Cabrera et al. 2008; Sedayu et al. 2010 dan Grob et al. 2004).
Intron kloroplas matK, rbcL dan trnL meru-pakan marka molekular yang sering digunakan dalam analisis filogenetik pada tumbuhan. Ketiga marka molekular tersebut memiliki tingkat subs-titusi yang tinggi, cepat berevolusi dan memiliki variasi genetik yang cukup tinggi, sehingga tepat jika digunakan dalam analisis filogenetik dan variasi genetik (Selvaraj et al. 2008). Analisis filogenetik Amorphophallus dengan menggunakan sekuen intron rbcL dan matK mengungkapkan posisi Amorphophallus dan Araceae lainnya seba-gai klad monofiletik, yaitu berada dalam satu kelompok evolusi (Tamura et al. 2004).
Marka molekular menggunakan intron matK, rbcL dan trnL telah berhasil digunakan dalam analisis filogenetik, namun kemampuan marka matK dan rbcL dalam mengungkap hu-bungan kekerabatan terbatas pada tingkat taksa famili dan genus (Selvaraj et al. 2008). Hal ini
berbeda dengan trnL yang mampu mengungkapkan hubungan kekerabatan di tingkat infra spesies. Taberlet et al. (2007) mengungkapkan bahwa in-tron trnL memiliki resolusi lebih tinggi pada konteks yang spesifik, misalnya individu spesies yang berasal dari ekosistem tunggal. Selain itu primer yang digunakan sangat ”conserved” dan sistem amplifikasinya sangat baik (”robust”). Sehingga trnL menjadi kandidat marka molekular untuk analisis variasi genetik pada infraspesies. Pengetahuan tentang keanekaragaman ge-netik penting untuk program pemuliaan tanaman dan menjadi dasar dalam menentukan langkah perbaikan kualitas dan kuantitas porang. Sejauh ini, studi keanekaragaman genetik pada porang infra spesies di Jawa Timur masih terbatas pada pembuktian ada atau tidaknya variasi genetik me-nggunakan metode RAPD, belum pada variasi genetik pada basa nukleotidanya. Untuk menyedia-kan informasi tersebut perlu dilakumenyedia-kan serangkaian penelitian yang terdiri dari studi variasi genetik pada porang infra spesies beda populasi di Jawa Timur dengan menggunakan marka molekular intron trnL, serta melakukan pengamatan variasi morfologi sebagai dasar identifikasi spesies. Dengan informasi variasi genetik yang diperoleh, diharapkan dapat membantu upaya pemuliaan dengan mengembangkan plasma nutfah, sedangkan kekerabatan yang diketahui, diharapkan dapat menjadi data dasar dalam usaha pelestarian porang (A. muelleri) di Jawa Timur.
BAHAN DAN METODE
Pengambilan sampel dilakukan secara eksplorasi di hutan jati Kabupaten Jember (Desa Mayang), Malang (Kecamatan Lawang dan Keca-matan Kalipare), Blitar (Desa Brongkos), Nganjuk (KPH Tritik desa Bendoasri), Madiun (Desa Kla-ngon, dan KPH Saradan), Bojonegoro (Kecamatan Klino), Jawa Timur. Ekstraksi dan analisis kekera-batan dilakukan di Laboratorium Fisiologi Tum-buhan, Kultur Jaringan dan Mikroteknik (FKM) dan Laboratorium Biologi Molekuler, Fakultas Matematika dan Ilmu Pengetahuan Alam, Univer-sitas Brawijaya, Malang. Produk PCR dikirim ke Macrogen Inc. Korea untuk mendapatkan hasil sekuensing.
Pengambilan sampel dilakukan secara acak, yaitu memilih dua individu A. muelleri dalam satu populasi dengan jarak 5–10 meter dan memiliki corak tangkai yang berbeda dan terdapat bulbil pada percabangannya. Individu yang terpilih se-lanjutnya diambil tiga daun muda yang sehat.
Ekstraksi DNA dilakukan dengan mengikuti me-tode ekstraksi menggunakan CTAB (Doyle & Doyle 1987, Taberlet et al. 1991).
Amplifikasi dilakukan menggunakan Master
Cycler Gradient Eppendorf dan mengamplifikasi
sebanyak 30 mL mix reaction yang terdiri dari 6 mL air milliQ, 15 mL PCR Intron Master Mix, 3mL Primer Forward (‘c’), 3 mL Primer Reverse (‘d’) dan 3 mL DNA sampel. Siklus amplifikasi terdiri dari 35 siklus dengan fase denaturasi awal pada 95°C selama 1 menit, denaturasi pada 95°C selama 45 detik, annealing pada 60.2–61.7°C sela-ma 45 detik dan fase ekstensi pada 72°C selasela-ma 45 detik dan ekstensi terakhir 10 menit pada suhu 72° C. Amplikon hasil PCR yang selanjutnya dipuri-fikasi dan disekuensing di perusahaan Macrogen, Inc. Korea.
”Alignment” dilakukan secara langsung
de-ngan menjajarkan sekuen menggunakan program Bioedit untuk Microsoft Windows dan dilakukan BLAST untuk mengkonfirmasi identitas gen. Se-kuen yang telah disejajarkan kemudian diamati setiap basa nukleotidanya. Analisis ini bertujuan untuk mengetahui adanya perubahan basa nukleo-tida, mengetahui ”conserved region” dan penduga-an jenis perubahpenduga-an (mutasi) ypenduga-ang terjadi. Selpenduga-an- Selan-jutnya dibuat pohon filogeni menggunakan metode Maximum Parsimony dengan program MEGA5 (Molecular Evolutionary Genetics Analysis) (Ta-mura et al. 2011). Analisis ini bertujuan untuk me-ngetahui pola pengelompokkan individu porang.
HASIL
Analisis Keragaman Genetik Berdasarkan Sekuen Intron trnL
Sekuensing dari 16 individu porang meng-hasilkan 13 individu porang dengan hasil sekuen yang baik, yaitu memiliki grafik kromatografi yang jelas sehingga menghasilkan pembacaan sekuen yang tepat yang dapat digunakan dalam analisis selanjutnya. Tiga sekuen individu porang yang tidak dapat digunakan yaitu LW1, KP2, dan BL2, karena sekuen tersebut memiliki grafik kroma-tografi yang tidak dapat dibaca dengan jelas, sehingga menghasilkan pembacaan sekuen yang rancu. Ketiga sekuen tersebut tidak digunakan dalam analisis selanjutnya.
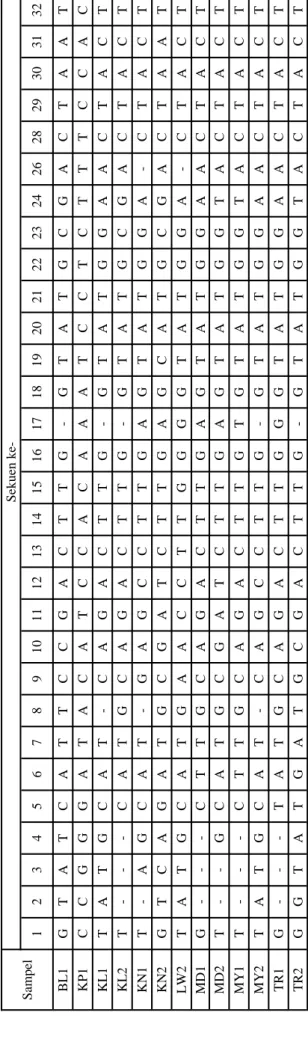
Berdasarkan sekuen dari 13 individu porang, diketahui sebanyak 6 daerah yang tidak me-ngalami perubahan basa. Keenam region ini diduga merupakan “conserved region”. Keenam “conser-ved region” tersebut adalah pada posisi basa ke: 54–151; 153–177; 179–285; 287–302; 337–430;
dan 432–458. “Conserved region” terdiri atas basa yang berulang dan memiliki kemiripan basa nu-kleotidanya. Selain “conserved region”, diketahui juga adanya daerah yang mengalami perubahan basa. Daerah ini diduga merupakan “variable re-gion” dan menjadi “site of mutation”. “Site of mu-tation” ini diketahui terjadi pada 52 daerah, yaitu pada basa ke 1–53;152; 178–179; 286; 456–570. Jenis basa yang berubah pada masing-masing “site of mutation” dapat di lihat pada Tabel 1.
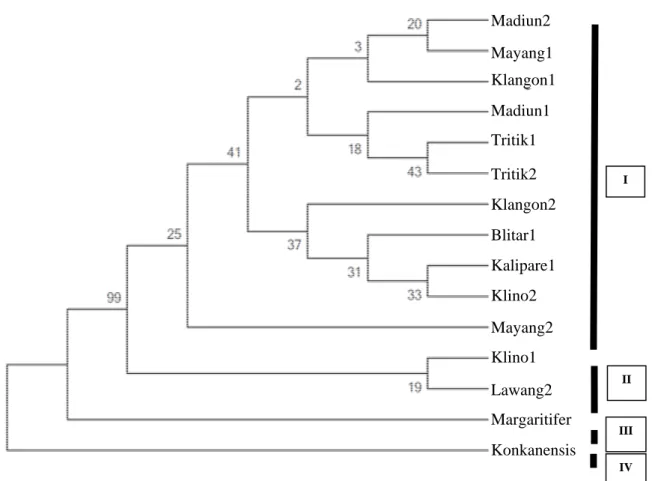
Nilai yang muncul pada titik (node) perca-bangan merupakan nilai bootstrap. Bootstrap meru-pakan nilai yang menyatakan peluang terjadinya perubahan susunan klad dan “sister klad” pada pohon filogenetik. Jika nilai bootstrap di antara 70–100 maka peluang terjadinya perubahan susu-nan klad adalah rendah, dengan demikian ketika dilakukan analisis, percabangan dan pohon yang dibentuk mencapai konsistensinya dan tidak akan berubah. Sebaliknya, jika nilai bootstrap kurang dari 70 maka peluang terjadinya perubahaan susu-nan klad adalah tinggi, sehingga ketika dilakukan analisis, percabangan dan pohon yang dibentuk masih dapat berubah-ubah (Simpson 2006). Pada gambar 1, nilai bootstrap klad I kurang dari 70 sehingga susunan “sister klad” dan anggotanya masih dapat berubah, demikian pula pada klad II, susunan “sister klad” dan anggotanya masih dapat berubah. Hal ini berbeda dengan nilai bootstrap antara klad I dan II yang mencapai lebih dari 70 yaitu 99, yang menunjukkan bahwa susunan kedua klad tersebut telah mencapai konsisten-sinya, peluang terjadinya perubahan susunan ada-lah rendah, sehingga ketika dilakukan analisis ulang akan tetap menghasilkan dua percabangan besar (2 “Major Klad”) pada individu porang terse-but.
Pohon filogeni yang terbentuk (Gambar 1), menunjukkan 4 klad besar, yaitu klad I, II, III, dan IV. Klad I terdiri atas 3 sub-klad yaitu sub-klad pertama terbagi menjadi dua “sister klad”. “Sister klad” pertama terdiri dari individu Madiun2, Ma-yang1 dan Klangon1, “sister klad” kedua terdiri dari Madiun1, Tritik1, dan Tritik2. Sub-klad kedua terdiri dari Klangon2, Blitar1, Kalipare1 dan Kli-no2. Sub-klad ketiga terdiri dari individu Ma-yang2. Klad II terdiri dari individu Klino1 dan La-wang2. Klad III dan IV terdiri dari outgrup Marga-ritifer (A. margaMarga-ritifer) dan Konkanensis (A.
kon-kanensis).
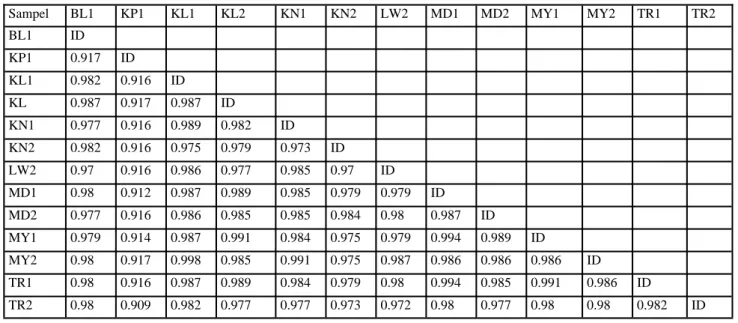
Analisis menggunakan Maximum Parsi-mony menghasilkan hipotesis hubungan kekera-batan infraspesies A. muelleri di Jawa Timur ber-dasarkan similaritas sekuen intron trnL (Tabel 2).
Sa mpe l Sekuen ke- 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 26 28 29 30 31 32 BL1 G T A T C A T T C C G A C T T G - G T A T G C G A C T A A T KP1 C C G G G A T A C A T C C A C A A A T C C T C T T T C C A C KL1 T A T G C A T - C A G A C T T G - G T A T G G A A C T A C T KL2 T - - - C A T G C A G A C T T G - G T A T G C G A C T A C T KN1 T - A G C A T - G A G C C T T G A G T A T G G A - C T A C T KN2 G T C A G A T G C G A T C T T G A G C A T G C G A C T A A T LW2 T A T G C A T G A A C C T T G G G G T A T G G A - C T A C T MD1 G - - - C T T G C A G A C T T G A G T A T G G A A C T A C T MD2 T - - G C A T G C G A T C T T G A G T A T G G T A C T A C T MY1 T - - - C T T G C A G A C T T G T G T A T G G T A C T A C T MY2 T A T G C A T - C A G C C T T G - G T A T G G A A C T A C T TR1 G - - - T A T G C A G A C T T G G G T A T G G A A C T A C T TR2 G G T A T G A T G C G A C T T G - G T A T G G T A C T A C T Sa mpe l Sekuen ke- 34 35 38 41 49 53 152 286 474 476 483 490 494 499 502 504 509 521 532 543 551 556 563 570 BL1 A G G A A A T T G A G T A A T T A G T T C G T T KP1 G A C - - C C C A A A C T C G C C A A C T A G A KL1 A G G A A A T T G G G T A A T T A G T T C G T T KL2 A G G A A A T T G A G T A A T T A G T T C G T T KN1 A G G A A A T T G G G T A A T T A G T T C G T T KN2 A G G A A A T T G G G T A A T T A G T T C G T T LW2 A G G A A A T T G G G T A A T T A G T T C G T T MD1 A G G A A A T T G G G T A A T T A G T T C G T T MD2 A G G A A A T T G G G T A A T T A G T T C G T T MY1 A G G A A A T T G G G T A A T T A G T T C G T T MY2 A G G A A A T T G G G T A A T T A G T T C G T T TR1 A G G A A A T T G G G T A A T T A G T T C G T T TR2 A G G A A A T T G G G T A A T T A G T T C G T T
Tabel 1. Perbedaan jenis basa pada urutan
sekuen intron trnL
Perbedaan genetik pada “sister klad” pertama yaitu individu Madiun2 dan Mayang1 adalah sebesar 2%, hal ini menunjukkan bahwa kedua individu tersebut memiliki kemiripan dalam susunan basa nukleotida. Perbedaan genetik Madiun2, Mayang1 terhadap Klangon1 adalah sebesar 2%, hal ini menunjukkan bahwa ketiga individu tersebut memiliki kemiripan susunan basa nukleotida, se-hingga dikelompokkan ke dalam satu klad.
Perbe-daan genetik pada “sister klad” kedua yaitu indi-vidu Tritik1 dan Tritik2 adalah sebesar 2%, hal ini menunjukkan bahwa kedua individu tersebut me-miliki kemiripan dalam susunan basa nukleotida. Perbedaan genetik Tritik1, Tritik2 terhadap Madi-un1 adalah sebesar 1–2%, hal ini menunjukkan bahwa ketiga individu tersebut memiliki kemiripan susunan basa nukleotida, sehingga dikelompokkan ke dalam satu klad.
Analisis Kekerabatan Berdasarkan Sekuen Intron trnL
Gambar 1. Pohon Maximum Parsimony dari 13 Individu Porang dan outgroup Perbedaan genetik pada sub-klad kedua
yaitu individu Klino2 terhadap Kalipare1 adalah sebesar 8%, hal ini menunjukkan bahwa kedua individu tersebut memiliki kemiripan dalam susun-an basa nukleotida ysusun-ang rendah, meskipun terletak dalam satu titik percabangan yang sama (Gambar 1). Intron trnL berevolusi lebih cepat, sehingga memiliki variasi genetik yang tinggi dan memiliki perbedaan antar individu dalam satu spesies (Kuhsel et al. 1990). Sedangkan Klino2, Kalipare1 terhadap Blitar1 dan Klangon2 adalah sebesar 2%, hal ini menunjukkan bahwa keempat individu
tersebut memiliki kemiripan susunan basa nukleo-tida, sehingga dikelompokkan ke dalam satu klad. Perbedaan genetik sub-klad ketiga yaitu individu Mayang2 terhadap sub klad pertama dan kedua adalah sebesar 2–8% , hal ini menunjukkan bahwa individu tersebut memiliki kemiripan dalam susun-an basa nukleotida, sehingga berada pada klad yang sama. Perbedaan genetik pada klad II yaitu individu Klino1 terhadap Lawang2 sebesar 2%, hal ini menunjukkan bahwa individu tersebut memiliki kemiripan dalam susunan basa nukleotida, sehing-ga berada pada klad yang sama.
I II III IV Madiun2 Mayang1 Klangon1 Madiun1 Tritik1 Tritik2 Klangon2 Blitar1 Kalipare1 Klino2 Mayang2 Klino1 Lawang2 Margaritifer Konkanensis
Sampel BL1 KP1 KL1 KL2 KN1 KN2 LW2 MD1 MD2 MY1 MY2 TR1 TR2 BL1 ID KP1 0.917 ID KL1 0.982 0.916 ID KL 0.987 0.917 0.987 ID KN1 0.977 0.916 0.989 0.982 ID KN2 0.982 0.916 0.975 0.979 0.973 ID LW2 0.97 0.916 0.986 0.977 0.985 0.97 ID MD1 0.98 0.912 0.987 0.989 0.985 0.979 0.979 ID MD2 0.977 0.916 0.986 0.985 0.985 0.984 0.98 0.987 ID MY1 0.979 0.914 0.987 0.991 0.984 0.975 0.979 0.994 0.989 ID MY2 0.98 0.917 0.998 0.985 0.991 0.975 0.987 0.986 0.986 0.986 ID TR1 0.98 0.916 0.987 0.989 0.984 0.979 0.98 0.994 0.985 0.991 0.986 ID TR2 0.98 0.909 0.982 0.977 0.977 0.973 0.972 0.98 0.977 0.98 0.98 0.982 ID
Tabel 2. Nilai similaritas sekuen intron trnL pada 13 individu porang
PEMBAHASAN
Variasi Genetik Berdasarkan Sekuen Intron trnL
Analisis perubahan basa yang terjadi pada masing-masing ”site of mutation” didasarkan atas perubahan basa tunggal terhadap basa dominan. Sebagai contoh, pada sekuen ke 34, basa Adenosin merupakan basa yang selalu ada pada tiap individu porang, dibandingkan dengan basa Guanin yang hanya ada pada salah satu individu, yaitu individu KP1. Dengan demikian, maka individu KP1 dinya-takan mengalami perubahan pada sekuen ke 34.
Berdasarkan hal tersebut, maka dapat dike-tahui daerah “site of mutation” dan jenis basa yang berubah pada masing-masing individu po-rang. Individu BL1 mengalami perubahan basa pada sekuen ke 1, 2, 3, 4, 8, 10, 23, 24, 31, dan 476, serta mengalami delesi pada sekuen ke 17. Individu KP1 mengalami perubahan basa pada sekuen ke 1, 2, 3, 5, 8, 11, 12, 14, 15, 16, 18, 20, 21, 22, 23, 24, 26, 28, 34, 35, 38, 53, 152, 286, 474, 476, 483, 490, 494, 499, 502, 504, 509, 521, 532, 543, 551, 556, 563 dan 570, serta terjadi delesi pada sekuen ke 41 dan 49. Individu KL1 tidak ditemukan terjadinya perubahan basa, tetapi terdapat delesi pada sekuen ke 8 dan 17. Individu KL2 mengalami perubahan basa pada sekuen ke 32, 24 dan 476, serta terjadi delesi pada sekuen ke 2, 3, 4 dan 17. Individu KN1 mengalami perubah-an basa pada sekuen ke 3, 9 dperubah-an 12, serta terjadi delesi pada sekuen ke 2, 8 dan 26. Individu KN2 mengalami perubahan basa pada sekuen ke 1, 2, 3, 4, 5, 10, 11, 12, 19, 23, 24 dan 31, tetapi tidak dite-mukan adanya delesi. Individu LW2 mengalami
perubahan basa pada sekuen ke 9, 11, 12, 13, 15 dan 17, serta mengalami delesi pada sekuen ke 26. Individu MD1 mengalami perubahan basa pada sekuen ke 1 dan 6 serta mengalami delesi pada se-kuen ke 2, 3 dan 4. Individu MD2 mengalami pe-rubahan basa pada sekuen ke 10, 11, 12 dan 24, serta mengalami delesi pada sekuen ke 2 dan 3. Individu MY1 mengalami perubahan basa pada sekuen ke 6, 17 dan 24, serta mengalami delesi pada sekuen ke 2, 3 dan 4. Individu MY2 menga-lami perubahan basa pada sekuen ke 12 dan me-ngalami delesi pada sekuen ke 8 dan 17. Individu TR1 mengalami perubahan basa pada sekuen ke 1, 5 dan 17, serta mengalami delesi pada sekuen ke 2, 3 dan 4. Individu TR2 mengalami perubahan basa pada sekuen ke 1, 2, 4, 5, 6, 7, 8, 9, 10 dan 24, ser-ta mengalami delesi pada sekuen ke 17.
Pada analisis di atas, diketahui individu-individu yang mengalami perubahan basa nukleo-tida paling sering dan jarang. Adapun individu dengan perubahan basa nukleotida paling sering sampai jarang, berturut-turut adalah KP1, KN2, BL1 dan TR2, LW2, MD2,KL2, KN1, MY1, TR1, MD1, MY2 dan KL1. Sedangkan delesi paling banyak dialami oleh individu KL2, KN1, MY1, TR1, KP1, KL1, MD2, MY2, BL1, LW2, TR2 dan KN2.
Perubahan basa yang diamati pada ketiga-belas individu porang menunjukkan bahwa indivi-du KP1 mengalami banyak perubahan basa, jika dibandingkan dengan individu lain. Adapun jumlah perubahan yang terjadi pada individu KP1 adalah sebanyak 45 perubahan. Delesi paling banyak ter-jadi pada individu KL2 yaitu sebanyak 4 delesi. Perubahan basa yang terjadi merupakan substitusi,
yaitu perubahan basa purin menjadi basa subseku-ennya, yaitu pirimidin. Perubahan basa dan delesi pada sekuen tersebut mempengaruhi variasi gene-tik, sehingga mempengaruhi hubungan kekerabat-an tkekerabat-anamkekerabat-an porkekerabat-ang. Adkekerabat-anya delesi pada suatu sekuen menyebabkan proses translasi yang meng-hasilkan protein yang berbeda dari protein yang seharusnya. Perubahan protein yang drastis ini dapat menyebabkan perubahan morfologi (Yuwo-no 2005).
Pada penelitian ini, tidak tampak adanya pengaruh perubahan basa dan delesi terhadap va-riasi morfologi. Hal ini ditunjukkan oleh individu KP1 dan KL2. Individu KP1 diketahui memiliki perubahan basa terbanyak jika dibandingkan de-ngan individu lain, tetapi ciri morfologi yang menonjol, yaitu corak tangkai daun yang tidak berbeda jauh dengan individu lainnya (LW2, BL2, TR2 dan KN2). Sedangkan KL2 yang diketahui memiliki jumlah delesi sekuen terbanyak, juga memiliki ciri morfologi yang sama dengan MD1 dan KP2. Jika sekuen antara KP1 disejajarkan dengan LW2, TR2 dan KN2 menunjukkan tidak adanya korelasi antara variasi genetik dengan kesamaan karakter morfologi yang dimiliki. Hal tersebut juga terjadi pada pensejajaran sekuen KL2 terhadap MD1 dan KP2. Hal ini ditunjukkan juga pada pohon Maximum Parsimony (Gambar 1), yaitu individu-individu yang memiliki kesamaan ciri morfologi tidak mengelompok ke dalam satu titik percabangan yang sama. LW2, KP1, BL2, TR2 dan KN2 yang secara morfologi memiliki kesamaan corak tangkai daun tidak mengelompok dalam satu titik percabangan. Demikian juga pada KL2 dan MD1 yang tidak berada dalam satu titik percabangan yang sama. Dengan demikian dapat dibuat kesimpulan bahwa variasi genetik intron trnL pada ketigabelas individu porang tidak mem-pengaruhi perubahan morfologi.
Gen trnL (UAA) merupakan gen yang berperan dalam pembentukan protein Leusin, tidak pada pembentukan corak tangkai daun. Lebih lanjut, variasi genetik pada intron trnL tidak mempengaruhi variasi morfologi, karena sifat dan peranan alami intron trnL tersebut yang tidak mengkode protein tertentu dan mengalami ”spli-cing” saat proses sintesis protein. Intron trnL merupakan intron Group 1 yang diketahui sebagai intron tertua (ancient) yang mampu mengkatalisis pemotongan intronnya sendiri tanpa ”spliceo-some”, sehingga mengalami ”self splicing” saat proses translasi mRNA (Costa 2004, Yuwono 2005). Sebuah studi terbaru melakukan uji coba menghilangkan empat sekuen ”conserved region”
dari non-coding DNA (intron) pada mencit dan diketahui tidak mengalami perubahan signifikan pada karakter fenotipnya (Ahituv et al. 2007). Dengan demikian, intron trnL tidak berpengaruh dan berperan dalam pembentukan corak tangkai daun Amorphophallus, karena mengalami ”spli-cing”, tidak ditranslasi dan tidak diekspresikan sebagai protein.
Kekerabatan Porang Berdasarkan Sekuen Intron trnL
Pohon filogeni yang terbentuk dari hasil analisis menggunakan Maximum Parsimony me-nunjukkan adanya monofiletik pada setiap kelom-pok taksa. Monofiletik memiliki arti bahwa setiap kelompok taksa berasal dari satu nenek moyang (ancestor) yang sama. Monofiletik menjadi para-meter penting dalam analisis filogenetik, karena dalam analisis evolusi, jumlah ancestor yang lebih dari satu akan menimbulkan kerancuan dalam menentukan perubahan karakter yang terjadi sela-ma evolusi. Kerancuan tersebut terjadi akibat adanya ketidakpastian ancestor mana yang menu-runkan karakter yang dimilikinya kepada taksa keturunannya (Simpson 2006).
Perbedaan genetik pada masing-masing individu dalam klad menunjukkan tingkat kemi-ripan basa nukleotida yang dimiliki. Jika nilai perbedaan genetik adalah 1–2%, hal ini menun-jukkan individu yang dibandingkan memiliki kemiripan susunan sekuen basa nukleotida dan diduga merupakan individu yang identik atau mi-rip. Sebaliknya, jika nilai perbedaan genetik lebih dari 2%, maka individu yang dibandingkan tidak memiliki kemiripan susunan sekuen basa nukleo-tida dan diduga merupakan individu yang berbeda atau spesies yang berbeda (Simpson 2006). Perbedaan basa tersebut menjadi ciri khusus yang dimiliki masing-masing individu. Dengan kata lain, menjadi karakter apomorphy.
Nilai yang muncul pada node percabangan merupakan nilai bootstrap. Pada Gambar 1, nilai bootstrap klad I kurang dari 70 sehingga susunan “sister klad” dan anggotanya masih dapat berubah, demikian pula pada klad II, susunan “sister klad” dan anggotanya masih dapat berubah. Hal ini berbeda dengan nilai bootstrap antara klad I dan II yang mencapai lebih dari 70 yaitu 99. Hal ini me-nunjukkan bahwa susunan kedua klad tersebut telah mencapai konsistensinya dan peluang terja-dinya perubahan adalah rendah. Nilai bootstrap ini juga menunjukkan kedua klad tersebut merupakan spesies yang sama tetapi diduga berbeda varian. Hal ini dikarenakan nilai bootsrap yang tergolong
tinggi yaitu 99 yang menunjukkan kepastian bahwa individu-individu dalam klad besar tersebut akan selalu terbagi menjadi dua.
Bootstrap merupakan nilai yang menya-takan peluang terjadinya perubahan susunan klad dan “sister klad” pada pohon fiogenetik. Jika nilai bootstrap diantara 70–100 maka peluang terjadinya perubahan susunan klad adalah rendah, dengan demikian ketika dilakukan analisis, percabangan dan pohon yang dibentuk mencapai konsistensinya dan tidak akan berubah. Sebaliknya, jika nilai bootstrap kurang dari 70 maka peluang terjadinya perubahaan susunan klad adalah tinggi, sehingga ketika dilakukan analisis, percabangan dan pohon yang dibentuk masih dapat berubah-ubah (Simpson 2006).
Klad I dan II berada pada kelompok mono-filetik, dengan demikian kedua klad besar tersebut memiliki ancestor yang sama dan diduga berasal dari daerah yang sama. Kedua klad tersebut monofiletik terhadap klad III yang merupakan outgroup, dengan demikian individu-individu yang berada pada klad I dan II merupakan spesies yang sama tetapi berbeda asal, dan memiliki hubungan kekerabatan yang dekat dengan outgroup karena memiliki ancestor yang sama. Penelitian tentang DNA porang pada analisis ITS dan analisis gen trn-F dan rbcL, menunjukkan bahwa porang yang berasal dari Madiun, Nganjuk, Bojonegoro dan Blitar adalah A. muelleri (Indriyani 2011).
Analisis lebih lanjut menghasilkan dugaan asal usul pada masing-masing individu porang. Berdasarkan pada pola pengelompokkan individu yang acak, masing-masing individu tidak me-ngelompok berdasarkan populasi asal diambilnya individu tersebut. Pada gambar 1 tampak bahwa pada anggota “sister klad” pertama merupakan individu yang berasal dari Madiun, Mayang dan Klangon. Jika dilihat berdasarkan pohon yang terbentuk, individu Madiun2 dan Mayang1 berasal dari induk yang sama yang di duga berkerabat dengan Klangon1. Pada “sister klad” kedua indi-vidu Tritik1 dan Tritik2 berasal dari induk yang sama dan diduga berkerabat dekat dengan Ma-diun1. Kedua “sister klad” ini membentuk mono-filetik dan dengan demikian diduga bahwa asal individu Madiun2, Mayang1, Klangon1, Madiun 1, Tritik1 dan Tritik2 berasal dari satu induk yang sama. Pada sub-klad kedua diketahui bahwa indi-vidu Kalipare1 dan Klino2 berasal dari induk yang sama yang diduga berkerabat dengan Blitar1. Dengan demikian induk dari Blitar1 dan Kalipare1 dengan Klino2 adalah sama yang diduga berkera-bat dengan Klangon2. Berdasarkan penjelasan
diatas, maka dapat ditarik kesimpulan bahwa indi-vidu Madiun2, Mayang1, Klangon1, Madiun1, Tri-tik1, Tritik2, Klangon2, Blitar1, Kalipare1, dan Klino2 berasal dari satu induk yang sama.
Analisis lebih lanjut menghasilkan dugaan bahwa induk dari sepuluh individu yang telah dise-butkan di atas diduga berasal dari induk yang sama dengan individu Mayang2. Selanjutnya, induk in-dividu Klino1 dan Lawang2 diduga juga berasal dari induk yang sama dengan induk antara Ma-yang2 dan sepuluh individu sebelumnya. Dengan demikian dapat disimpulkan bahwa seluruh indivi-du yang diteliti berasal dari satu inindivi-duk yang sama. Asal induk tersebut belum diketahui secara pasti, sebab jika ditelaah berdasarkan kondisi geografis, individu-individu yang terletak pada geografis yang sama yaitu seperti Madiun, Klangon, Klino, Tritik tidak mengelompok menjadi satu yang menunjukkan adanya hubungan kekerabatan de-ngan lokasi geografis. Dede-ngan demikian, asal po-rang di masing-masing lokasi penelitian tidak da-pat diketahui berdasarkan analisis secara geogra-fis, sehingga perlu dilakukan penelitian lebih lanjut terkait analisis sosial budaya porang, untuk men-dapatkan informasi originalitas porang di area studi.
KESIMPULAN
Variasi genetik intron trnL dipengaruhi oleh perubahan basa nukleotida dan delesi pada masing-masing individu.Intron trnL individu po-rang pada studi ini memiliki “conserved region” pada basa ke: 54–151; 153–177; 179–285; 287–302; 337–430; dan 432–458. Perubahan basa nukleotida terjadi pada 52 “site of mutation” yaitu antara basa ke 1–53;152; 178–179; 286; 456– 570.Individu dengan perubahan basa nukleotida paling sering sampai jarang, berturut-turut adalah KP1, KN2, BL1 dan TR2, LW2, MD2,KL2, KN1, MY1, TR1, MD1, MY2 dan KL1. Sedangkan de-lesi paling banyak dialami oleh individu KL2, lalu KN1, MY1, TR1, KP1, KL1, MD2, MY2, BL1, LW2, TR2 dan KN2.
Seluruh individu yang diteliti berasal dari satu induk yang sama. Asal induk tersebut be-lum diketahui secara pasti, sebab jika ditelaah berdasarkan kondisi geografis, individu-individu yang terletak pada geografis yang sama tidak me-ngelompok menjadi satu yang menunjukkan adanya hubungan kekerabatan akibat lokasi geo-grafis yang sama.
PUSTAKA ACUAN
Ahituv N, Zhu Y, Visel A, Holt A, Afzal V, Pe-nnacchio LA & Rubin EM. 2007. Deletion of ultraconserved elements yields viable mice. PLos Biol. 5: 9.
Azrianingsih R, Ekowati G & Wahono T . 2008. Seleksi varian porang (Amorphophalls
on-cophyllus) berdasarkan struktur tanaman,
kadar glukomannan dan Ca-oksalat umbi-nya. Laporan Hasil Penelitian Program Re-search Grant I-MHERE. Universitas Brawi-jaya. Malang.
Cabrera LI, Salazar GA, Chase MW, Mayo SJ, Bogner J & Dávila P. 2008. Phylogenetic relationships of aroids and duckweeds
(Ara-ceae) inferred from coding and noncoding
plastid DNA. Amer. J. Bot. 95(9): 1153– 1163.
Costa JL. 2004. The tRNA Leu (UAA) Intron of
Cyanobacteria. Towards understanding a
genetic marker. Dissertation. Faculty of Sci-ence and Technology. Uppsala University. Swedia.
Doyle JJ & Doyle. 1987. A rapid DNA isolation procedure from small quantities of fresh leaf tissues. Phytochem. Bull. 19: 11–15.
Grob GBJ, Gravendeel B & Eurlings MCM. 2004. Potential phylogenetic utility of the nuclear FLORICAULA/LEAFY second intron: comparison with three chloroplast DNA regions in Amorphophallus (Araceae). Mol.
Phyl. Evol.30: 13–23.
Indriyani S. 2011. Pola pertumbuhan porang (Amorphophallus muelleri Blume) dan pe-ngaruh lingkungan terhadap kandungan ok-salat dan glukomanan umbi. Disertasi Pro-gram Pascasarjana, Universitas Airlangga. Surabaya.
Jansen PCM, Westphal E & Wulijarni-Soetjipto N. 1996. Plant Resources of South-East Asia 9: Plants Yielding Non-Seed Carbohydrates. Prosea Foundation. Bogor.
Jones CJ, Edwards KJ, Castagiole S, Winfield, F Sala MO, Wiel C van del, Bredemeijer G, Vosman B, Matthes M, Daly A, Bretts-shneider R, Bettini P, Buiatti M, Maestri E, Malcevschi A, Marmiroli N, Aert R, Vol-ckaert G, Rueda J, Linacero R, Vasquez A & Karp A. 1997. A reproducibility testing of
RAPD, AFLP and SSR markers in plants by a network of European laboratories.
Molecu-lar Breeding 3(5): 382–390.
Kuhsel MG, Strickland R & Palmer JD. 1990. An ancient group I intron shared by Eubacteria and chloroplast. Science 250: 1570–1573. Poerba YS & Martanti D. 2008. Keragaman
ge-netik berdasarkan marka random amplified polymorphic DNA pada Amorphophallus
muelleri Blume di Jawa. Technology 9: 245–
249.
Sedayu A, Eurlings MCM, Gravendeel B & Wil-bert LA. 2010. Morphological character evo-lution of Amorphophallus (Araceae) based on a combined phylogenetic analysis of trnL, rbcL and LEAFY second intron se-quences. Botanical Studies 51: 473–490. Selvaraj D, Sarmadan RK & Sathiskumar R. 2008.
Phylogenetic analysis of chloroplast matK gene from Zingiberaceae for plant DNA barcoding. Bioinformation 3(1): 24–27. Simpson MG. 2006. Plant Systematics. Elsevier
Academic Press. San Diego, California. Sumarwoto. 2005. Iles-iles (Amorphophallus
mu-elleri Blume) deskripsi dan sifat-sifat
lain-nya. Biodiversitas 6(3): 185–190.
Taberlet P, Coissac E, Pompanon F, Gielly L, Miquel C, Valentini A, Vermat T, Corthier G, Brochmann C & Willerslev E. 2007. Power and limitations of the chloroplast trnL (UAA) intron for plant DNA barcoding.
Nucleic acids research 35(3): 14.
Taberlet P, Gielly L, Pautou G & Bouvet J. 1991. Universal primers for amplification of three non-coding regions of chloroplast DNA.
Plant Mol. Biol. 17: 1105–1109.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M & Kumar S. 2011. MEGA5: Molecular evolutionary genetics analysis using maxi-mum likelihood, evolutionary distance, and maximum parsimony methods. Mol. Biol.
Evol. 28(10): 2731–2739.
Tamura MN, Yamashita J, Fuse S & Haraguchi M. 2004. Molecular phylogeny of Monocotyle-dons inferred from combined analysis of plastid matK and rbcL gene sequences. J.
Plant. Res. 117: 109–120.
Yuwono T. 2005. Biologi Molekuler. Penerbit Er-langga. Jakarta.