IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RELEASING HORMONE
(GHRH) PADA KERBAU LOKAL
(
Bubalus bubalis
) DENGAN METODE PCR-RFLP
SKRIPSI
ALMIRA PRIMASARI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
ALMIRA PRIMASARI. 2009. Identifikasi Keragaman Gen Growth Hormone Releasing Hormone (GHRH) pada Kerbau Lokal (Bubalus bubalis) dengan Metode PCR-RFLP. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Dr. Ir. Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Dr. Ir. Achmad Farajallah, M.Si.
Salah satu usaha yang dapat dilakukan untuk mendapatkan ternak lokal yang memiliki produktivitas tinggi adalah dengan perbaikan manajemen pemeliharaan dan perbaikan genetik. Perbaikan secara genetik dapat dilakukan dengan mengetahui karakteristik genetik ternak yang berpengaruh terhadap sifat pertumbuhan yang dikontrol oleh banyak gen dan sebagian besar aksinya bersifat aditif. Growth Hormone Releasing Hormone (GHRH) merupakan salah satu faktor pertumbuhan yang berperan menstimulasi sintesis dan sekresi Growth Hormone yang berpengaruh secara aditif terhadap pertumbuhan. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen Growth Hormone Releasing Hormone (GHRH) pada kerbau lokal di Indonesia. Penelitian ini dilaksanakan di Laboratorium Zoologi Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Sampel darah kerbau yang digunakan berjumlah 320 sampel yang berasal dari empat daerah, yaitu Semarang (Jawa Tengah), Mataram (Nusa Tenggara Barat), Medan (Sumatera Utara) dan Banten. Amplifikasi gen GHRH dilakukan dengan teknik PCR, sedangkan untuk identifikasi keragaman dilakukan menggunakan metode PCR-RFLP dengan menggunakan enzim restriksi HaeIII. Gen GHRH kerbau yang berhasil diamplifikasi berukuran 451 pb terletak di sebagian ekson 2, intron 2 dan sebagian ekson 3. Pendeteksian keragaman gen GHRH kerbau lokal Indonesia dengan metode PCR-RFLP menghasilkan dua tipe alel yaitu alel A (18%) dan alel B (82%). Genotipe yang diperoleh yaitu AA (0%), AB (36%) dan BB (64%) dengan nilai heterozigositas sebesar 46%. Frekuensi Alel A pada setiap populasi yaitu Semarang (15%), Mataram (19%), Medan (2%) dan Banten (45%). Alel B memiliki frekuensi lebih besar dibandingkan alel A pada semua populasi ternak kerbau dari empat daerah di Indonesia. Frekuensi alel B pada setiap populasi yaitu Semarang (85%), Mataram (81%), Medan (98%) dan Banten (48%). Nilai frekuensi alel B terbesar terdapat pada populasi kerbau Medan (98%) dengan nilai heterozigositas 4%. Nilai heterozigositas tertinggi terdapat pada populasi kerbau di Banten yaitu 48%. Nilai indeks fiksasi gen GHRH menunjukkan bahwa dari keempat populasi kerbau lokal tidak terdapat gen yang terfiksasi dengan nilai indeks fiksasi tidak sama dengan nol. Nilai jarak genetik gen GHRH terkecil yaitu antara populasi kerbau lokal Semarang dan kerbau lokal Mataram (0,001) dan terbesar antara populasi kerbau lokal Medan dan Banten (0,202).
ABSTRACT
Identification of Growth Hormone Releasing Hormone Gene in Local Buffalo (Bubalus bubalis) Using PCR-RFLP
Primasari, A., C. Sumantri and A. Farajallah
GHRH is a hypothalamic hormone which stimulates growth hormone secretion in the pituitary gland. The objective of this study was to identify polymorphisms Growth Hormone Releasing Hormone (GHRH) gene of Indonesian buffalo’s. A total of 320 blood samples from Indonesian buffalo were used to determined polymorphism using PCR-RFLP method. The polymorphism of GHRH gene that spanned within exon 2 and exon 3 was amplified, and their mutation was detected using endonuclease HaeIII. In this study, there were found to be three GHRH/HaeIII genotype (AA genotype 0%, AB genotype 36% and BB genotype 64%) determined by two alleles, A (18%) and B (82%). The frequency of A allele was found 15% for Semarang population, 19% for Mataram population, 2% for Medan population and 40% for Banten population. The frequency of B allele was found 85% for Semarang population, 81% for Mataram population, 98% for Medan population and 60% for Banten population. The observed heterozygosis value were different among populations. The highest heterozygosis ( ) 0,485 for Banten population and the lowest was 0.037 for Medan population and the average heterozygosis for all populations ( ) detected was 0.270. Index fixation value of GHRH gene showed there was not fixed into one gene type (Fski 0). The smallest genetic distance value
of GHRH gene was found between Semarang and Mataram population (0.001) and highest between Medan and Banten population (0.202).
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RELEASING HORMONE
(GHRH) PADA KERBAU LOKAL
(
Bubalus bubalis
) DENGAN METODE PCR-RFLP
ALMIRA PRIMASARI D14051318
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RELEASING HORMONE
(GHRH) PADA KERBAU LOKAL
(
Bubalus bubalis
) DENGAN METODE PCR-RFLP
Oleh
ALMIRA PRIMASARI D14051318
Skripsi ini telah disetujui dan disidangkan di hadapan Komisi Ujian Lisan pada tanggal 02 April 2009
Pembimbing Utama Pembimbing Anggota
Dr. Ir. Cece Sumantri, M.Agr.Sc. Dr. Ir. Achmad Farajallah, M.Si.
Dekan Ketua Departemen
Fakultas Peternakan Ilmu Produksi dan Teknologi Peternakan Institut Pertanian Bogor Fakultas Peternakan
Institut Pertanian Bogor
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 17 September 1988 di Sukabumi. Penulis
adalah anak pertama dari tiga bersaudara dari pasangan H.Tejo Sriwijoyo, SKM.,
M.MKes dan Hj.Henny Liswara.
Pendidikan dasar diselesaikan pada tahun 1999 di SD Negeri Cisande I,
Sukabumi. Pendidikan lanjutan tingkat pertama diselesaikan pada tahun 2002 di
SLTP Negeri 1 Cibadak, Sukabumi dan pendidikan lanjutan menengah atas
diselesaikan pada tahun 2005 di SMA Negeri 1 Sukabumi.
Penulis diterima sebagai mahasiswa pada departemen Ilmu Produksi dan
Teknologi Peternakan, Fakultas Peternakan Institut Pertanian Bogor melalui jalur
Undangan Seleksi Masuk IPB (USMI) pada tahun 2005.
Selama mengikuti pendidikan, penulis aktif di beberapa organisasi
kemahasiswaan diantaranya Badan Eksekutif Mahasisa Fakultas Peternakan
(2006-2007 dan (2006-2007-2008), Kelompok Pecinta Alam Fakultas Peternakan (2006-(2006-2007 dan
2007-2008), Ikatan Mahasiswa Sukabumi (2006-2007 dan 2007-2008) dan UKM
Tae Kwon Do (2006-2007). Skripsi dengan judul Identifikasi Keragaman Gen
KATA PENGANTAR
Puji dan syukur penulis panjatkan ke hadirat Allah SWT karena hanya
dengan rahmat dan karunia-Nya lah penulis dapat menyelesaikan studi, penelitian,
dan penulisan skripsi dengan judul Identifikasi Keragaman Gen Growth Hormone Releasing Hormone (GHRH) Pada Kerbau Lokal (Bubalus bubalis) dengan Metode PCR-RFLP.
Beberapa hal yang mendasari dilakukannya penelitian ini diantaranya adalah
1) kerbau di Indonesia merupakan salah satu ternak lokal dengan beberapa kelebihan
yang dimilikinya sehingga berpotensi untuk dikembangkan menjadi ternak penghasil
daging 2) masih rendahnya perkembangan populasi, produksi dan produktivitas
kerbau di Indonesia, serta 3) informasi molekuler kerbau lokal masih terbatas
terutama gen yang mempengaruhi pertumbuhan.
Penelitian ini bertujuan untuk mengidentifikasi keragaman gen Growth Hormone Releasing Hormone (GHRH) pada kerbau lokal dari empat daerah di Indonesia. Hasil yang diperoleh dari penelitian ini ditujukan sebagai informasi awal
mengenai keragaman gen GHRH kerbau lokal Indonesia untuk penelitian
selanjutnya, guna mendapatkan ternak kerbau dengan produktivitas yang lebih baik.
Semoga penulisan skripsi ini dapat bermanfaat dan memberikan sumbangan yang
berarti bagi kemajuan peternakan Indonesia. Amin
Bogor, April 2009
DAFTAR ISI
Restriction Fragment Length Polymorphism (RFLP) ... 11
Elektroforesis ... 11
Restriction Fragment Length Polymorphism (RFLP) .... 14
Elektroforesis ... 14
Halaman
Pendeteksian Keragaman DNA ... 15
HASIL DAN PEMBAHASAN ... 16
Amplifikasi Gen GHRH ... 16
Pendeteksian Keragaman Gen GHRH dengan Metode PCR RFLP ... 17
Keragaman Gen GHRH Kerbau Lokal ... 20
Nilai Heterozigositas ... 22
Indeks Fiksasi ... 24
Jarak Genetik dan Pohon Genetik ... 24
KESIMPULAN DAN SARAN ... 27
Kesimpulan ... 27
Saran ... 27
UCAPAN TERIMAKASIH ... 28
DAFTAR PUSTAKA ... 29
DAFTAR TABEL
Nomor Halaman
1. Beberapa Informasi Sifat Biologis Ternak Kerbau dengan Pola
Pemeliharaan Ekstensif ... 5
2. Keragaman Gen GHRH Menurut Beberapa Penelitian pada
Beberapa Jenis Ternak ... 10
3. Nilai Frekuensi Genotipe dan Frekuensi Alel Gen GHRH-HaeIII
Kerbau Lokal ... 21
4. Nilai Heterozigositas ( ) dan Rataan Heterozigositas ( ) Gen
GHRHKerbau Lokal ... 23
5. Nilai Indeks Fiksasi Gen GHRHKerbau Lokal ... 24
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RELEASING HORMONE
(GHRH) PADA KERBAU LOKAL
(
Bubalus bubalis
) DENGAN METODE PCR-RFLP
SKRIPSI
ALMIRA PRIMASARI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
ALMIRA PRIMASARI. 2009. Identifikasi Keragaman Gen Growth Hormone Releasing Hormone (GHRH) pada Kerbau Lokal (Bubalus bubalis) dengan Metode PCR-RFLP. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Dr. Ir. Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Dr. Ir. Achmad Farajallah, M.Si.
Salah satu usaha yang dapat dilakukan untuk mendapatkan ternak lokal yang memiliki produktivitas tinggi adalah dengan perbaikan manajemen pemeliharaan dan perbaikan genetik. Perbaikan secara genetik dapat dilakukan dengan mengetahui karakteristik genetik ternak yang berpengaruh terhadap sifat pertumbuhan yang dikontrol oleh banyak gen dan sebagian besar aksinya bersifat aditif. Growth Hormone Releasing Hormone (GHRH) merupakan salah satu faktor pertumbuhan yang berperan menstimulasi sintesis dan sekresi Growth Hormone yang berpengaruh secara aditif terhadap pertumbuhan. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen Growth Hormone Releasing Hormone (GHRH) pada kerbau lokal di Indonesia. Penelitian ini dilaksanakan di Laboratorium Zoologi Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Sampel darah kerbau yang digunakan berjumlah 320 sampel yang berasal dari empat daerah, yaitu Semarang (Jawa Tengah), Mataram (Nusa Tenggara Barat), Medan (Sumatera Utara) dan Banten. Amplifikasi gen GHRH dilakukan dengan teknik PCR, sedangkan untuk identifikasi keragaman dilakukan menggunakan metode PCR-RFLP dengan menggunakan enzim restriksi HaeIII. Gen GHRH kerbau yang berhasil diamplifikasi berukuran 451 pb terletak di sebagian ekson 2, intron 2 dan sebagian ekson 3. Pendeteksian keragaman gen GHRH kerbau lokal Indonesia dengan metode PCR-RFLP menghasilkan dua tipe alel yaitu alel A (18%) dan alel B (82%). Genotipe yang diperoleh yaitu AA (0%), AB (36%) dan BB (64%) dengan nilai heterozigositas sebesar 46%. Frekuensi Alel A pada setiap populasi yaitu Semarang (15%), Mataram (19%), Medan (2%) dan Banten (45%). Alel B memiliki frekuensi lebih besar dibandingkan alel A pada semua populasi ternak kerbau dari empat daerah di Indonesia. Frekuensi alel B pada setiap populasi yaitu Semarang (85%), Mataram (81%), Medan (98%) dan Banten (48%). Nilai frekuensi alel B terbesar terdapat pada populasi kerbau Medan (98%) dengan nilai heterozigositas 4%. Nilai heterozigositas tertinggi terdapat pada populasi kerbau di Banten yaitu 48%. Nilai indeks fiksasi gen GHRH menunjukkan bahwa dari keempat populasi kerbau lokal tidak terdapat gen yang terfiksasi dengan nilai indeks fiksasi tidak sama dengan nol. Nilai jarak genetik gen GHRH terkecil yaitu antara populasi kerbau lokal Semarang dan kerbau lokal Mataram (0,001) dan terbesar antara populasi kerbau lokal Medan dan Banten (0,202).
ABSTRACT
Identification of Growth Hormone Releasing Hormone Gene in Local Buffalo (Bubalus bubalis) Using PCR-RFLP
Primasari, A., C. Sumantri and A. Farajallah
GHRH is a hypothalamic hormone which stimulates growth hormone secretion in the pituitary gland. The objective of this study was to identify polymorphisms Growth Hormone Releasing Hormone (GHRH) gene of Indonesian buffalo’s. A total of 320 blood samples from Indonesian buffalo were used to determined polymorphism using PCR-RFLP method. The polymorphism of GHRH gene that spanned within exon 2 and exon 3 was amplified, and their mutation was detected using endonuclease HaeIII. In this study, there were found to be three GHRH/HaeIII genotype (AA genotype 0%, AB genotype 36% and BB genotype 64%) determined by two alleles, A (18%) and B (82%). The frequency of A allele was found 15% for Semarang population, 19% for Mataram population, 2% for Medan population and 40% for Banten population. The frequency of B allele was found 85% for Semarang population, 81% for Mataram population, 98% for Medan population and 60% for Banten population. The observed heterozygosis value were different among populations. The highest heterozygosis ( ) 0,485 for Banten population and the lowest was 0.037 for Medan population and the average heterozygosis for all populations ( ) detected was 0.270. Index fixation value of GHRH gene showed there was not fixed into one gene type (Fski 0). The smallest genetic distance value
of GHRH gene was found between Semarang and Mataram population (0.001) and highest between Medan and Banten population (0.202).
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RELEASING HORMONE
(GHRH) PADA KERBAU LOKAL
(
Bubalus bubalis
) DENGAN METODE PCR-RFLP
ALMIRA PRIMASARI D14051318
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RELEASING HORMONE
(GHRH) PADA KERBAU LOKAL
(
Bubalus bubalis
) DENGAN METODE PCR-RFLP
Oleh
ALMIRA PRIMASARI D14051318
Skripsi ini telah disetujui dan disidangkan di hadapan Komisi Ujian Lisan pada tanggal 02 April 2009
Pembimbing Utama Pembimbing Anggota
Dr. Ir. Cece Sumantri, M.Agr.Sc. Dr. Ir. Achmad Farajallah, M.Si.
Dekan Ketua Departemen
Fakultas Peternakan Ilmu Produksi dan Teknologi Peternakan Institut Pertanian Bogor Fakultas Peternakan
Institut Pertanian Bogor
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 17 September 1988 di Sukabumi. Penulis
adalah anak pertama dari tiga bersaudara dari pasangan H.Tejo Sriwijoyo, SKM.,
M.MKes dan Hj.Henny Liswara.
Pendidikan dasar diselesaikan pada tahun 1999 di SD Negeri Cisande I,
Sukabumi. Pendidikan lanjutan tingkat pertama diselesaikan pada tahun 2002 di
SLTP Negeri 1 Cibadak, Sukabumi dan pendidikan lanjutan menengah atas
diselesaikan pada tahun 2005 di SMA Negeri 1 Sukabumi.
Penulis diterima sebagai mahasiswa pada departemen Ilmu Produksi dan
Teknologi Peternakan, Fakultas Peternakan Institut Pertanian Bogor melalui jalur
Undangan Seleksi Masuk IPB (USMI) pada tahun 2005.
Selama mengikuti pendidikan, penulis aktif di beberapa organisasi
kemahasiswaan diantaranya Badan Eksekutif Mahasisa Fakultas Peternakan
(2006-2007 dan (2006-2007-2008), Kelompok Pecinta Alam Fakultas Peternakan (2006-(2006-2007 dan
2007-2008), Ikatan Mahasiswa Sukabumi (2006-2007 dan 2007-2008) dan UKM
Tae Kwon Do (2006-2007). Skripsi dengan judul Identifikasi Keragaman Gen
KATA PENGANTAR
Puji dan syukur penulis panjatkan ke hadirat Allah SWT karena hanya
dengan rahmat dan karunia-Nya lah penulis dapat menyelesaikan studi, penelitian,
dan penulisan skripsi dengan judul Identifikasi Keragaman Gen Growth Hormone Releasing Hormone (GHRH) Pada Kerbau Lokal (Bubalus bubalis) dengan Metode PCR-RFLP.
Beberapa hal yang mendasari dilakukannya penelitian ini diantaranya adalah
1) kerbau di Indonesia merupakan salah satu ternak lokal dengan beberapa kelebihan
yang dimilikinya sehingga berpotensi untuk dikembangkan menjadi ternak penghasil
daging 2) masih rendahnya perkembangan populasi, produksi dan produktivitas
kerbau di Indonesia, serta 3) informasi molekuler kerbau lokal masih terbatas
terutama gen yang mempengaruhi pertumbuhan.
Penelitian ini bertujuan untuk mengidentifikasi keragaman gen Growth Hormone Releasing Hormone (GHRH) pada kerbau lokal dari empat daerah di Indonesia. Hasil yang diperoleh dari penelitian ini ditujukan sebagai informasi awal
mengenai keragaman gen GHRH kerbau lokal Indonesia untuk penelitian
selanjutnya, guna mendapatkan ternak kerbau dengan produktivitas yang lebih baik.
Semoga penulisan skripsi ini dapat bermanfaat dan memberikan sumbangan yang
berarti bagi kemajuan peternakan Indonesia. Amin
Bogor, April 2009
DAFTAR ISI
Restriction Fragment Length Polymorphism (RFLP) ... 11
Elektroforesis ... 11
Restriction Fragment Length Polymorphism (RFLP) .... 14
Elektroforesis ... 14
Halaman
Pendeteksian Keragaman DNA ... 15
HASIL DAN PEMBAHASAN ... 16
Amplifikasi Gen GHRH ... 16
Pendeteksian Keragaman Gen GHRH dengan Metode PCR RFLP ... 17
Keragaman Gen GHRH Kerbau Lokal ... 20
Nilai Heterozigositas ... 22
Indeks Fiksasi ... 24
Jarak Genetik dan Pohon Genetik ... 24
KESIMPULAN DAN SARAN ... 27
Kesimpulan ... 27
Saran ... 27
UCAPAN TERIMAKASIH ... 28
DAFTAR PUSTAKA ... 29
DAFTAR TABEL
Nomor Halaman
1. Beberapa Informasi Sifat Biologis Ternak Kerbau dengan Pola
Pemeliharaan Ekstensif ... 5
2. Keragaman Gen GHRH Menurut Beberapa Penelitian pada
Beberapa Jenis Ternak ... 10
3. Nilai Frekuensi Genotipe dan Frekuensi Alel Gen GHRH-HaeIII
Kerbau Lokal ... 21
4. Nilai Heterozigositas ( ) dan Rataan Heterozigositas ( ) Gen
GHRHKerbau Lokal ... 23
5. Nilai Indeks Fiksasi Gen GHRHKerbau Lokal ... 24
DAFTAR GAMBAR
Nomor Halaman
1. Hasil Amlifikasi Gen GHRH Menggunakan Metode PCR pada
Gel Poliakrilamida 6% ... 16
2. Posisi Penempelan Primer dan Situs Pemotongan Enzim Restriksi HaeIII didasarkan pada sekuens gen GHRH pada sapi di GenBank
(no akses GenBank AF242855) (Zhou et al., 2000) ... 17 3. Hasil Pemotongan Fragmen Gen GHRH Menggunakan Metode
PCR-RFLP dengan Enzim Pemotong HaeIII pada Gel
Poliakrilamida 6% ... 18
4. Diagram Elektroforesis Hasil Pemotongan Gen GHRH/HaeIII ... 19 5. Dendogram Pohon Genetik Berdasarkan Gen GHRH pada Empat
Populasi Kerbau Lokal Indonesia ... 25
DAFTAR LAMPIRAN
Nomor Halaman
1. Modifikasi Metode Isolasi DNA Menggunakan Genomic DNA
PENDAHULUAN
Latar Belakang
Kerbau merupakan salah satu ternak ruminansia besar yang dapat
dimanfaatkan sebagai penghasil daging, susu dan kerja. Secara umum pemeliharaan
ternak kerbau di Indonesia bukan ditujukan untuk diambil dagingnya. Peternak
kerbau di Indonesia umumnya hanya memelihara kerbau sebagai ternak kerja untuk
mengolah lahan pertanian dan sebagai tabungan hidup.
Daging kerbau dapat menjadi komplemen bahkan substitusi daging sapi
sehingga pengembangan kerbau perlu mendapat perhatian agar kerbau dapat
berkontribusi lebih besar terhadap program kecukupandaging nasional. Revitalisasi
peternakan kerbau harus dilakukan dalam upaya mendukung program kecukupan
daging sapi tahun 2010. Diharapkan pada tahun tersebut ketergantungan impor
daging dapat dikurangi secara signifikan (10%). Saat ini impor daging untuk
memenuhi permintaan daging di Indonesia masih sekitar 30% (Riady, 2006).
Perkembangan populasi, produksi dan produktivitas kerbau di Indonesia
masih kurang optimal. Populasi kerbau di Indonesia terus mengalami penurunan dari
tahun ke tahun. Populasi ternak kerbau di Indonesia dari 3,065 juta ekor pada tahun
1997 menjadi 2,201 juta ekor pada tahun 2006 (Badan Pusat Statistik, 2006).
Penurunan tersebut disebabkan oleh peralihan fungsi kerbau sebagai ternak kerja
karena adanya mesin atau traktor dan makin sempitnya areal lahan untuk
penggembalaan serta kurang adanya usaha – usaha perbaikan mutu genetik dan
teknik budidaya.
Perbaikan mutu genetik kerbau masih tertinggal jauh dari ternak lainnya.
Perbaikan dengan mempertimbangkan penanda genetik dapat dijadikan sebagai salah
satu alternatif dalam melakukan seleksi. Salah satu metode seleksi yang sedang
berkembang saat ini adalah metode MAS (Marker Assisted Selection) yaitu seleksi berdasarkan penciri DNA yang mengontrol sifat-sifat ekonomis.
Growth Hormone Releasing Hormone merupakan hormon yang menstimulasi sintesis dan sekresi Growth Hormone yang mempengaruhi pertumbuhan. Oleh karena itu, gen GHRH merupakan penciri genetik yang dapat dijadikan sebagai salah
Tujuan
Penelitian ini bertujuan untuk mengidentifikasi keragaman gen growth hormone releasing hormone (GHRH) dengan metode PCR-RFLP pada kerbau lokal dari empat daerah di Indoesia yaitu Semarang (Jawa Tengah), Mataram (Nusa
TINJAUANPUSTAKA
Kerbau
Tetua kerbau domestik (Bubalus bubalis) berasal dari kerbau liar Asia (Wild Asian buffalo – Bubalus bubalis). Kerbau domestik satu dengan lainnya agak berbeda, menunjukkan bahwa tetua mereka berasal dari beberapa subspesies yang
dapat dijumpai di beberapa bagian dunia. Kebanyakan kerbau domestik dijumpai
pada wilayah yang beriklim panas dan basah dimana padi dihasilkan. Sekitar 95%
dari kerbau domestik terdapat di Asia, dan sekitar 2,5% terdapat di Afrika,
khususnya Mesir (FAO, 2000).
Kerbau merupakan ternak yang multifungsi yaitu sebagai penghasil daging,
susu dan kerja yang potensial untuk mengolah lahan pertanian. Selain itu, kerbau
berfungsi sebagai sumber pupuk dan mempunyai fungsi sosial budaya di beberapa
daerah di Indonesia. Kerbau mempunyai keistimewaan tersendiri dibandingkan sapi,
karena ternak ini mampu hidup di kawasan yang relatif sulit terutama bila pakan
yang tersedia berkualitas sangat rendah. Pertumbuhan kerbau dapat menyamai atau
justru lebih baik dibandingkan sapi dan masih dapat berkembang biak dalam kondisi
kualitas pakan yang tersedia relatif kurang baik. Kerbau memiliki beberapa
keunggulan tetapi juga tidak terlepas dari adanya kelemahan. Salah satu kelemahan
kerbau adalah ketidaktahanannya terhadap udara yang panas. Oleh sebab itu untuk
melangsungkan proses faali hidupnya memerlukan waktu untuk merendam diri di
lumpur (berkubang) (Diwyanto dan Handiwirawan, 2006).
Umumnya semua tipe kerbau domestik (Bubalus bubalis) dibagi menjadi dua kelompok yaitu kerbau sungai (riverine buffalo) dan kerbau rawa atau kerbau lumpur (swamp buffalo). Kromosom kerbau liar Asia maupun kerbau domestik (kerbau rawa) adalah 2n = 48, sedangkan kerbau sungai (riverine buffalo) adalah 2n =50 (FAO, 2000). Kedua kelompok kerbau ini mempunyai sifat biologis yang berbeda.
Kerbau sungai menunjukkan kesenangan terhadap air mengalir yang bersih,
sedangkan kerbau rawa suka berkubang dalam lumpur, rawa-rawa dan air
menggenang (Hasinah dan Handiwirawan, 2006).
Populasi kerbau di Indonesia sebagian besar merupakan kerbau rawa dan
hanya sedikit kerbau sungai di Sumatera Utara yaitu kerbau Murrah yang dipelihara
biasa digunakan sebagai ternak kerja, untuk nantinya dipotong sebagi penghasil
daging dan jarang digunakan sebagai penghasil susu. Hanya sedikit sekali kerbau
rawa yang dimanfaatkan susunya, karena produksi susunya sangat rendah yaitu
hanya 1-1,5 l/hari, dibandingkan dengan tipe sungai yang mampu menghasilkan susu
sebanyak 6-7 l/hari (Hasinah dan Handiwirawan, 2006).
Ciri – ciri Fisik Kerbau
Ciri - ciri fisik kerbau sungai yaitu memiliki tanduk melingkar ke bawah atau
lurus memanjang dan memiliki kulit berwarna hitam atau abu-abu agak gelap.
Sedangkan kerbau rawa atau kerbau lumpur umumnya memiliki tanduk melengkung
ke atas dan memiliki kulit berwarna abu – abu terang. Hasil penelitian yang
dilakukan oleh Erdiansyah (2008) di Kabupaten Dompu Nusa Tenggara Barat
menunjukkan bahwa kerbau yang terdapat di daerah tersebut merupakan kerbau rawa
dengan jenis tanduk melingkar ke atas sebesar 98%. Hal tersebut sama dengan
penelitian yang dilakukan oleh Asoen (2008) yang melakukan pengamatan terhadap
kerbau rawa yang menghasilkan 96,2% dari jumlah kerbau memiliki bentuk tanduk
normal yang memanjang ke belakang lalu melengkung ke atas. Kerbau rawa yang
diamati memiliki warna abu – abu terang (36,5%), abu – abu gelap (29,5%) coklat
dan merah masing – masing 11% dan 19%. Sifat khas warna kulit berkaitan dengan
hasil pengukuran morfometrik tubuh kerbau dimana kerbau yang memiliki warna
kulit merah dan coklat memiliki ukuran relatif lebih besar (Erdiansyah, 2008).
Garis kalung (chevron) merupakan ciri spesifik kerbau rawa. Berdasarkan
hasil penelitian Sitorus (2008) ditemukan lima variasi garis kalung pada kerbau rawa
yaitu tunggal di bagian atas, tunggal di bagian bawah, tunggal di bagian bawah dan
bercabang, double yaitu di leher bagian atas dan bawah, serta double dengan bagian bawah yang bercabang. Erdiansyah (2008) menyebutkan terdapat juga sebanyak
1,5% kerbau lokal dalam penelitiannya di Nusa Tenggara Barat yang tidak memiliki
chevron dan terdapat pula kerbau lokal yang memiliki chevron tunggal yang memiliki persentase cukup besar yaitu 18,5%. Kaki kerbau lokal umumnya berwarna
terang Sitorus (2008) menyebutkan bahwa terdapat dua variasi warna kaki kerbau
rawa yaitu 94,12% berwarna abu – abu muda dan hanya 5,88% berwarna abu-abu.
Warna hitam pada kaki ditemukan hanya 4% dari kerbau lokal yang diamati. Kerbau
mengolah lahan pertanian karena kemampuannya menekan keras ke bawah
(Erdiansyah, 2008).
Penelitian yang dilakukan Hidayat (2007) menunjukkan bahwa antara kerbau
Banten dan Sumatera utara mempunyai ukuran tubuh yang berbeda, kerbau Sumatera
Utara mempunyai tinggi pundak, dan lingkar dada yang lebih besar dibandingkan
kerbau Banten. Rataan tinggi pundak kerbau Banten yaitu 120 cm, Sumaera Utara
126 cm. Rataan lingkar dada kerbau Banten yaitu 170 cm dan Sumatra Utara 182 cm.
Rataan panjang badan kerbau Banten 121 cm dan rataan panjang badan kerbau
Sumatera Utara yaitu 118 cm.
Produktivitas Kerbau
Produktivitas kerbau dalam beberapa hal lebih rendah dibandingkan sapi
terkait dengan sifat-sifat biologis yang dimilikinya. Dewasa kelamin kerbau relatif
lebih lambat, calving interval sekitar dua tahun dan persentase karkas relatif lebih kecil dibandingkan sapi (<50%). Pertambahan bobot badan kerbau sekitar 0,3-0,9 kg
per hari (Tabel 1).
Tabel 1. Beberapa Informasi Sifat Biologis Ternak Kerbau dengan Pola Pemeliharaan Ekstensif
Sifat biologis Keterangan
Umur beranak pertama 3,5-4 tahun Lama kebuntingan 11-12 bulan
Jarak beranak 20-24 bulan
Pertambahan bobot badan 0,3-0,9 kg per hari Persentase karkas <50%
Senang berkubang Perlu tempat berkubang
Estrus Tanda-tanda lemah dan relatif tenang (silent heat)
Anestrus Bermusim
Postpartum unestrus Panjang
Posisi vagina Bagian depan lebih rendah dibanding belakang, sewaktu berahi cairan tidak keluar Libido pejantan di musim kemarau Menurun drastis
Jumlah pejantan yang dipelihara peternak Terkadang terlalu banyak, sehingga tidak efisien, sering berkelahi dan kawin beberapa kali (>3 kali)
Perkawinan tidak terkontrol Meningkatnya inbreeding
Populasi Kerbau di Indonesia
Populasi kerbau di Indonesia berdasarkan data yang diperoleh dari Badan
Pusat Statistik (2006) menunjukkan bahwa jumlah populasi kerbau di Indonesia terus
mengalami penurunan dari tahun ke tahun, populasi ternak kerbau di Indonesia pada
tahun 1997, 1999, 2000, 2001, 2005 berturut – turut adalah sebanyak 3,065 juta
ekor, 2,504 juta ekor, 2,405 juta ekor, 2,333 juta ekor, 2,403 juta ekor, 2,428 juta
ekor dan pada tahun 2006 menjadi 2,201 juta ekor yang menyebar hampir di seluruh
propinsi tetapi tidak merata jumlahnya. Populasi kerbau terbanyak terdapat di
sepuluh propinsi di Indonesia yaitu propinsi Nangroe Aceh Darussalam, Sumatera
Utara, Sumatera Barat, Jawa Barat, Nusa Tenggara Barat, Banten, Nusa Tenggara
Timur, Sulawesi Selatan, Jawa Tengah dan Sumatera Selatan yang masing-masing
berjumlah 340.031, 261.308, 211.008, 156.570, 156.568, 145.439, 141.236, 128.502,
123.826 dan 103.577 ekor.Rataan pertumbuhan populasi kerbau di Indonesia adalah
sekitar 3,41% per tahun.
Potensi Ternak Kerbau
Kerbau lokal mempunyai potensi yang besar untuk dapat dikembangkan
sebagai ternak penghasil daging karena mudah menyesuaikan diri, mempunyai bobot
karkas yang relatif lebih tinggi dibandingkan sapi lokal serta telah biasa dipelihara di
perdesaan (Hasinah dan Handiwirawan, 2006). Ternak kerbau belum digunakan
sebagi ternak penghasil daging walaupun dari segi bobot badan cukup potensial.
(Diwyanto dan Handiwirawan, 2006).
Kerbau dapat berkembang dalam rentang kondisi agroekosistem yang sangat
luas dari daerah dengan kondisi yang basah sampai dengan kondisi yang kering.
Melihat kemampuan adaptasi kerbau tersebut, pengembangan dan penyebaran kerbau
dapat dilakukan di banyak daerah di Indonesia dengan memperhatikan jenis kerbau
dan daya adaptasinya (Diwyanto dan Handiwirawan, 2006).
Daging kerbau lebih merah dibanding daging sapi karena mempunyai
pigmentasi yang lebih banyak dan kurang lemak intramuskuler. Kondisi ini
menyebabkan daging kerbau relatif lebih keras dibanding sapi, tetapi justru disukai
sebagian konsumen yang memiliki resep masakan tradisional yang unik. Secara
umum harga daging dan kerbau hidup lebih rendah dibandingkan sapi, kecuali di
rendah maka pasar bagi daging kerbau menjadi lebih luas, banyak konsumen yang
mampu untuk membelinya sehingga peluang pengembangannya menjadi lebih
terbuka (Diwyanto dan Handiwirawan, 2006).
Analisis Keragaman DNA
Polimerase chain reaction (PCR) adalah suatu reaksi in vitro untuk menggandakan molekul DNA pada target tertentu dengan cara mensintesa molekul
DNA baru yang berkomplemen dengan molekul DNA tersebut dengan enzim
polymerase dan oligonukleotida pendek sebagai primer dalam mesin thermocycler Metode ini berjalan secara enzimatik melalui mekanisme perubahan suhu. Proses
yang terjadi dalam mesin PCR melipti tiga tahap utama yaitu denaturasi (pemisahan
untai ganda DNA), annealing (penempelan primer) dan ekstensi (pemanjangan primer). Proses dari mulai denaturasi, penempelan dan ektensi disebut sebagai satu
siklus. Produk PCR dapat langsung divisualisasikan melalui proses elektroforesis dan
dapat digunakan untuk analisis lebih lanjut (Muladno, 2002).
Restriction Fragment Length Polymorphism (RFLP) merupakan metode analisis lanjutan dari produk PCR. RFLP adalah profil DNA berupa
fragmen-fragmen DNA hasil pemotongan enzim endonuklease atau enzim restriksi yang
mengenali situs pemotongan empat dan enam basa. RFLP memiliki kemampuan
untuk mendeteksi keragaman di tingkat alel didasarkan pada polimorfisme yang
muncul karena adanya basa yang mengalami substitusi, penambahan, pengurangan
dan perpindahan (translokasi) pada genom DNA. Perubahan tersebut menyebabkan
perbedaan ukuran dari fragmen restriksi yang dicerna oleh enzim restriksi tertentu.
RFLP hanya mendeteksi perbedaan-perbedaan dari fragmen tersebut, dimana satu
dengan lainnya berhubungan dengan homolognya dari probe molekuler yang
digunakan dalam hibridisasi. Probe molekuler yang digunakan untuk RFLP antara
lain probe genom DNA (yang diturunkan dari genom DNA) dan probe cDNA (yang
diturunkan dari mRNA). Kelebihan dari RFLP adalah dapat mendeteksi sifat
kodominan, artinya dapat membedakan antara yang homozigot dan heterozigot.
Selain itu kelebihan yang lain adalah diharapkan didapatkan homologi polimorfik
(Gupta et al., 2002).
Metode lain yang dapat digunakan untuk menganalisis keragaman pada
Conformation Polymorphism atau PCR-SSCP yang merupakan salah satu metode analisis lebih lanjut yang memanfaatkan produk PCR. Metode ini didasarkan pada
asumsi bahwa perubahan yang terjadi pada fragmen DNA akan mempengaruhi
bentuk dari fragmen DNA untai tunggalnya yang terlihat dari perubahan pola migrasi
pada gel poliakrilamida nondenaturasi. Metode SSCP dapat mendeteksi adanya mutasi paa fragmen DNA akan tetapi tidak dapat memberikan informasi tentang
posisi dimana terjadinya mutasi pada fragmen DNA dan memiliki keterbatasan
dalam menentukan jumlah alel (Barroso et al., 1999).
Temperature-Gradient Gel Electrophoresis (TGGE) merupakan salah satu metode analisis keragaman yang mendeteksi adanya mutasi menggunakan gel yang
memiliki perbedaan suhu (Jasik dan Reichert, 2006). Denaturing-Gradient Gel Electrophoresis (DGGE) merupakan salah satu metode yang dapat membedakan adanya mutasi berdasarkan berat molekul fragmen DNA pada gel yang memiliki
perbedaan konsentrasi bahan untuk menyamakan berat molekul (denaturing) (Liu et al., 2008).
Analisis keragaman molekul DNA juga dapat dilakukan dengan metode
sekuensing yang merupakan satu dari terobosan utama dalam genetika molekuler.
Sekuensing merupakan proses penentuan urutan nukleotida pada suatu fragmen DNA
atau RNA. Sekuensing menghasilkan penggambaran linear simbolik yang disebut
sekuens yang meringkas sebagian besar struktur tingkat atom atas molekul yang
disekuensing. SekuensingDNA akan menghasilkan sekuens DNA yang digambarkan
sebagai untaian abjad lambang nukleotida-nukleotida penyusun DNA (Muladno,
2002). Tipe polimorfisme yang dideteksi analisis sekuen yaitu pertukaran satu basa
dan menghasilkan informasi sekuens yang dibutuhkan. Proses sekuensing dapat
dilakukan secara cepat dan hasil yang diperoleh diperoleh tinggi akan tetapi biaya
yang dibutuhkan cukup besar (Gupta et al., 2002).
Keragaman Gen Growth Hormone Releasing Hormone (GHRH) Growth Hormone Releasing Hormone (GHRH) dikenal juga dengan nama Growth Releasing Factor (GRF) atau Growth Hormone Releasing Factor (GHRF) atau somatocrinin terdiri dari 44 peptida asam amino dengan berat molekul 12447 Da
pituitari dan sel hipothalamus. Berdasarkan fungsi protein, somatoliberin sama
seperti jenis glukagon, sekretin dan VIP (Vasoactive Intestinal Peptide). GHRH
terikat pada spesifik reseptor (GHRH-R) yang terdapat dalam sel pituitari anterior
dan menstimulasi sintesis dan sekresi hormon pertumbuhan (Growth Hormone).
Growth Hormone dibutuhkan untuk pertumbuhan jaringan, metabolisme lemak dan berperan penting untuk reproduksi, laktasi, dan pertumbuhan tubuh (Etherton, 1998).
Beberapa penelitian telah dilakukan pada ternak sapi dan diketahui bahwa
somatotropin, somatoliberin dan sejenis sintesisnya meningkatkan produksi baik
pada ternak perah yaitu meningkatkan produksi dan lemak susu (Bonneau dan
Laarveld, 1999) maupun ternak pedaging (Achtung et al., 2001) yang dapat meningkatkan laju pertumbuhan sehingga mempersingkat waktu yang dibutuhkan
untuk mencapai bobot potong. Cheong et al. (2006) menambahkan berdasarkan hasil penelitiannya bahwa gen GHRH merupakan salah satu penanda genetik untuk
produksi daging.
Gen GHRH pada sapi terletak pada kromosom nomor 13 (Barendse et al., 1994) terdiri dari lima ekson dan empat intron (Zhou et al., 2000). Menurut Zhou et al. (2000) panjang gen GHRH pada sapi adalah 9356 pb (pasang basa).
Moody et al. (1995) melaporkan adanya keragaman gen GHRH pada sapi dengan metode PCR-RFLP menggunakan primer GHRH forward 5’-GTA AGG ATG CCA GCT CTG GGT3’ dan GHRH reverse 5’-TGC CTG CTC ATG ATG TCC TGG A-3’ serta enzim restriksi HaeIII yang menghasilkan dua alel yaitu 317,83, 55 pb (alel A) dan 196, 121, 83, 55 pb (alel B). Keragaman ditemukan pada
intron pertama gen GHRH pada sapi. Situs keragaman untuk GHRH/HaeIII pada sapi Polish Black and White menurut Dybus dan Grzesiak (2006) terletak menutupi bagian ekson 2, seluruh intron 2, dan sebagian dari ekson 3; analisis keragaman
terletak di intron 2 (no. Akses GenBank AF242855). Kmiéc et al. (2007) melakukan penelitian terhadap keragaman GHRH/HaeIII dan hubungannya dengan sifat produksi susu pada sapi Polish Red-and-White (salah satu varietas bangsa Fresian Holstein). Produk PCR teramplifikasi sepanjang 296 pb, menghasilkan tiga genotipe
yang memiliki pola pemotongan fragmen 194, 55 dan 48 pb (GHRHB /GHRHB) ;
242, 194, 55 dan 48 pb (GHRHA/GHRHB) dan 242, 55 dan 48pb (GHRHA/GHRHA).
GHRHA/GHRHB 37% dan genotipe GHRHB/GHRHB sebesar 53,4%. Frekuensi alel
GHRHA sebesar 28,1% dan Frekuensi alel GHRHB sebesar 79,1%.
Cheong et al. (2006) dalam penelitiannya menemukan hubungan yang nyata antara keragaman gen GHRH dengan sifat karkas yang meliputi bobot karkas dan
perototan bagian longisimus pada ternak pedaging asli Korea (Hanwoo) dengan
metode analisis sekuen DNA, enam lokasi keragaman dipilih dari 12 keragaman
single nukleotida yang ditemukan untuk kemudian ditetapkan genotipe dari ternak
tersebut dan ditemukan lima penanda haplotipe (frekuensi > 0,1).
Pierzchala et al. (2003) menyatakan bahwa keragaman gen GHRH berhubungan dengan laju pertumbuhan dan kualitas karkas pada ternak babi dengan
enzim restriksi AluI yang menghasilkan Frekuensi genotipe AA (8,8%), AB (61,8%) dan BB (27,97%). Beberapa penelitian mengenai keragaman gen GHRH pada
beberapa ternak disajikan dalam Tabel 2.
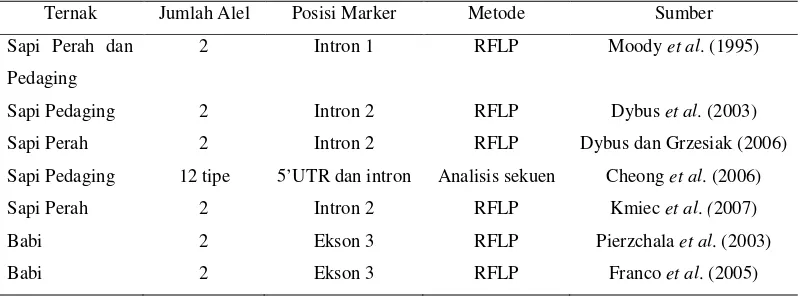
Tabel 2. Keragaman Gen GHRH Menurut Beberapa Penelitian pada Beberapa Jenis Ternak
Ternak Jumlah Alel Posisi Marker Metode Sumber
Sapi Perah dan Pedaging
2 Intron 1 RFLP Moody et al. (1995)
Sapi Pedaging 2 Intron 2 RFLP Dybus et al. (2003)
Sapi Perah 2 Intron 2 RFLP Dybus dan Grzesiak (2006)
Sapi Pedaging 12 tipe 5’UTR dan intron Analisis sekuen Cheong et al. (2006)
Sapi Perah 2 Intron 2 RFLP Kmiec et al. (2007)
Babi 2 Ekson 3 RFLP Pierzchala et al. (2003)
Babi 2 Ekson 3 RFLP Franco et al. (2005)
METODE
Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboratorium Zoologi Departemen Biologi,
Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Penelitian ini dilaksanakan mulai bulan September 2008 sampai dengan bulan
Desember 2008.
Materi
Sampel Darah dan Isolasi DNA
Sampel darah kerbau yang digunakan berjumlah 320 sampel yang diambil
dari empat daerah, yaitu 75 dari Semarang (Jawa Tengah), 103 dari Mataram (Nusa
Tenggara Barat), 65 dari Siborong-borong (Sumatera Utara), dan 77 dari Banten.
Sampling darah dilakukan menggunakan vaccutainer 10 ml berheparin yang
dilengkapi dengan jarum no. 18. Ekstraksi DNA dilakukan menggunakan Genomic DNA mini kit (Geneaid).
PCR (Amplifikasi DNA)
Primer yang digunakan dalam penelitian ini adalah berdasarkan primer yang
digunakan Moody et al. (1995), yaitu GHRH forward 5’-GTA AGG ATG CCA GCT CTG GGT3’ dan GHRH reverse 5’-TGC CTG CTC ATG ATG TCC TGG A-3’, enzim Taq polymerase, dNTP, 10x buffer dan air destilata
Alat – alat yang digunakan antara lain tabung PCR, pipet mikro dengan
tipsnya, alat sentrifugasi, vortex dan mesin thermocycler (TaKaRa PCR Thermal Cycler).
Restriction Fragment Length Polymorphism (RFLP)
Bahan – bahan yang digunakan adalah Produk PCR, enzim restriksi (RE) HaeIII dengan buffernya dan air destilata.
Alat – alat yang digunakan antara lain tabung PCR, pipet mikro dengan
Elektroforesis
Bahan – bahan yang digunakan adalah air destilata steril, akrilamida 30%, 5 x
TBE, TEMED (tetramethylendiamine), APS (ammonium persulfat) 10%, loading dye, dan marker.
Alat – alat yang digunakan yaitu gelas ukur, tabung reaksi, dua buah kaca
untuk cetakan gel, sisir khusus untuk sumur, pipet berskala, pipet mikro 2 µl dengan
tipsnya dan power supply 500VA.
Pewarnaan Perak
Bahan – bahan yang digunakan adalah air destilata, CTAB (cetyltrimetil
ammonium bromide), NH4OH, AgNO3, NaOH, Na2CO3, formaldehida dan asam
asetat glacial.
Alat – alat yang digunakan antar lain nampan, gelas ukur, tabung Erlenmeyer, dan water-bath shaker.
Rancangan
Keragaman genotipe tiap-tiap individu dapat ditentukan dari pita-pita DNA
yang ditemukan. Masing-masing sampel dibandingkan berdasarkan ukuran (marker)
yang sama dan dihitung frekuensi alelnya. Frekuensi alel dihitung berdasarkan rumus
Nei (1987) :
Keterangan :
Xi = Frekuensi alel i
nii = Jumlah individu bergenotip ii
nij = Jumlah individu bergenotip ij
n = Jumlah individu sampel
Derajat heterozigositas ( ) dihitung berdasarkan frekuensi alel pada tiap lokus
DNAmenggunakan rumus Nei (1987) :
Keterangan :
Xi = Frekuensi alel i
n = Jumlah individu sampel
Ragam heterozigositas (Vsl( )) pada tiap populasi dapat dihitung dengan
rumus sebagai berikut :
! " # $ %
Standar error (SE) diperoleh dari akar ragam heterozigositas.
Rataan heterozigositas ( ) dihitung dengan rumus sebagai berikut :
& '() ) '*+
Keterangan :
j= derajat heterozigositas untuk lokus ke-j
r = jumlah lokus yang diuji
= rataan heterozigositas
Indeks fiksasi pada masing – masing populasi diperoleh dari persamaan :
,-./ / /
/ 0 /
Keterangan :
Xkii = Frekuensi genotipe homozigot alel i pada populasi ke-k
Xki = Frekuensi alel i
J
arak genetik (D) dihitung berdasarkan rumus :Pix = Frekuensi alel ke i pada populasi X
Piy = Frekuensi alel ke i pada populasi Y
Prosedur
Penanganan dan Pengambilan Sampel
Sampel darah diambil melalui vena jugularis kerbau menggunakan vaccutainer berheparin. Darah yang diperoleh kemudian disimpan dalam alkohol 70% dalam 1mM EDTA. Darah dibawa ke laboratorium untuk dikerjakan lebih
lanjut.
Ekstraksi DNA dari Sampel Darah
Ekstraksi DNA dilakukan menggunakan Genomic DNA mini kit Geneaid yang dimodifikasi untuk darah yang disimpan dalam alkohol (lampiran 1).
Amplifikasi Gen GHRH
Proses amplifikasi DNA secara in vitro menggunakan pereaksi yang terdiri dari 2 µl sampel DNA dimasukkan kedalam tabung PCR, kemudian ditambah 2 µl
primer. Larutan dalam tabung kemudian ditambah dengan premix yang terdiri dari
17,85 µl air destilata, 10 x bufer 2,5 µl, 2 mM MgCl2, 0,24 mM dNTP, dan 0,75 unit
enzim Taq polymerase. Campuran tersebut kemudian diinkubasi pada mesin thermocycler dengan suhu pradenaturasi 940C selama 5 menit, 30 siklus yang terdiri dari denaturasi 940C selama 1 menit, annealing 600C selama 1 menit dan elongasi 720C selama 2 menit. Elongasi akhir 720C selama 5 menit.
Restriction Fragment Length Polymorphism (RFLP)
Produk PCR kemudian dipotong dengan enzim restriksi HaeIII yang mengenali situs pemotongan empat basa dan memotong pada posisi GG|CC. Produk
PCR sebanyak 5 µl dipindahkan ke dalam tabung baru 0,5 ml dan ditambahkan
dengan enzim restriksi HaeIII dan buffernya mengikuti petunjuk produsen. Campuran tersebut diinkubasi dalam inkubator pada suhu 37°C selama 12 jam.
Elektroforesis
Fragmen DNA produk PCR yang telah dipotong divisualisasikan dengan
teknik elektroforesis gel polyakrilamida 6%. Gel dibuat dengan cara mencampurkan
12 ml air destilata, 4 ml 5 x TBE, 4 ml akrilamida 30%, 15 µl TEMED, dan 160 µl
dilakukan selama 40 menit pada tagangan konstan 220 v atau sampai pewarna
bromtimol blue mencapai bagian bawah gel. Setelah elektroforesis selesai, gel diambil untuk dilakukan pewarnaan perak.
Pewarnaan perak
Pewarnaan perak (silver staining) dilakukan dengan langkah sebagai berikut:
gel dimasukan kedalam larutan CTAB 0,2 gram /200 ml air destilata selama delapan
menit sambil digoyang, kemudian dicuci dengan air destilata selama 2 x 2 menit. Air
tersebut dibuang dan ditambahkan larutan NH4OH selama 6 menit sambil digoyang.
Kemudian dilanjutkan dengan larutan perak nitrat (AgNO3) selama 10 menit sambil
digoyang. Gel dicuci kembali dengan air destilata 2 x 2 menit. Untuk memunculkan
pita, gel direndam dalam larutan Na2CO3 dan formaldehid. Setelah pita muncul,
ditambahkan larutan asam asetat untuk menghentikan aktifitas oksidasi perak oleh
formaldehid.
Pendeteksian keragaman DNA
Setelah pewarnaan perak, akan muncul pita – pita DNA. Setiap pita DNA
yang muncul dibandingkan dengan marker untuk mengetahui panjangnya. Setiap pita
DNA dari setiap sampel dibandingkan untuk menentukan genotip pita DNA. Satu
HASIL DAN PEMBAHASAN
Amplifikasi Gen GHRH
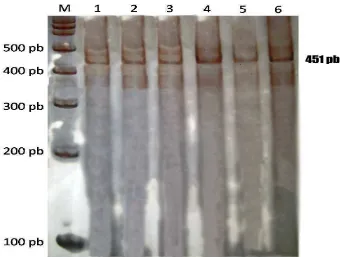
Gen GHRH pada kerbau lokal dari empat populasi di Indonesia berhasil
diamplifikasi menggunakan metode PCR (Polymerase Chain Reaction) dengan primer yang mengikuti Moody et al. (1995) pada ternak sapi. Gen GHRH kerbau lokal yang berhasil diamplifikasi berukuran 451 pb (gambar 1) dan sesuai dengan
sekuen DNA yang menjadi acuan (Gambar 2).
Gambar 1. Hasil Amplifikasi Gen GHRH Menggunakan Metode PCR pada Gel Poliakrilamida 6%
Ruas gen GHRH yang diamplifikasi pada penelitian ini terletak di sebagian
ekson 2, intron 2 dan sebagian ekson 3. Panjang fragmen gen GHRH hasil
Gambar 2. Posisi penempelan primer dan situs pemotongan Enzim Restriksi HaeIIIdidasarkan pada sekuens gen GHRH pada sapi di GenBank (no akses GenBank AF242855) (Zhou et al., 2000).
Keberhasilan amplifikasi gen GHRH sangat ditentukan oleh bahan pereaksi
PCR , kondisi penempelan primer pada DNA genom (gen target) dan kondisi mesin
PCR. Menurut Viljoen et al. (2005), keberhasilan mengamplifikasi DNA bergantung pada interaksi komponen PCR dalam konsentrasi yang tepat. Keberhasilan
amplifikasi DNA juga dipengaruhi oleh adanya inhibitor, yaitu haemoglobin yang
dapat menghambat kerja enzim taq polymerase. Upaya yang umum dilakukan untuk optimasi PCR diantaranya yaitu suhu penempelan primer, konsentrasi primer,
konsentrasi DNA target dan konsentrasi Mg2+.
Pendeteksian Keragaman Gen GHRH dengan metode PCR-RFLP
Pendeteksian keragaman gen GHRH pada kerbau lokal Semarang, Mataram,
Medan dan Banten dilakukan dengan metode RFLP. Enzim restriksi yang digunakan
dalam penelitian ini yaitu HaeIII yang mengenali situs pemotongan empat basa dan memotong pada posisi GG|CC. Berdasarkan sekuen gen GHRH sapi (no akses
GenBank AF242855) ditemukan tiga titik potong enzim restriksi HaeIII, yaitu pada nukleotida ke-4472, 4666 dan 4760 dari sekuen gen GHRH sapi sepanjang 9356 pb
atau pada posisi basa ke-118, 312 dan 406 dari produk PCR (Gambar 2 dan
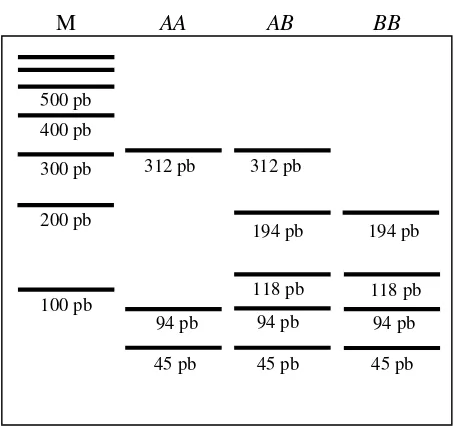
Lampiran 2). Pemotongan tersebut menghasilkan empat fragmen yang panjangnya
adalah 118, 194, 94 dan 45 pb. Keempat pita tersebut menunjukkan alel B. Jika
terdapat mutasi satu basa dari keempat basa sebagai pengenal oleh enzim restriksi
4321 cctgtctgtc atttcccagg taccagcaca ggggtgaagg atgctgctct gggtgttctt
Primer Forward
4381 cctcgtgacc ctcaccctca gcagcggctc ccacggttcc ctgccttccc agcctctcag
4441 gtaagcagtt ctgagaagag aagcaagaga gg|ccctttga ggatgcgact cgagctggtc
4501 cccagctggg tcctcaggca gcctcccttg ctcatctctg ggagggtggc agactgagcc
4561 ccagagaggt caccacccag ccctggttcc agccctctct ggggacgagc agggcaagag
4621 gcgacagaaa gacctcacag agaccaagtg agcacagtcc cctggg|cctc ccaccccacc
4681 ctttgacctc tgactccttc tactaggatt ccacggtacg cagatgccat cttcactaac
4741 agctaccgga aggttctggg|ccagctgtct gcccgcaagc tactccagga tatcatgaac
Primer Reverse
HaeIII maka situs pemoton
ternak tersebut memiliki k
pemotongan fragmen gen G
3 dan Gambar 4.
Gambar 3. Hasil P PCR-R Poliakr
tongan tersebut tidak dikenali lagi sehingga frag
an fragmen tersebut menunjukkan alel A.
katakan memiliki genotipe AA apabila terdapat tig
jang 312 pb, 94 pb dan 45 pb. Genotipe AB d
ng memiliki panjang 312 pb, 194 pb, 118 pb, 94
kkan dengan terdapatnya empat fragmen yaitu 19
DNA dengan ukuran 45 pb tidak dapat ditampilk
ntrasi 6% kurang tepat digunakan untuk memisa
ari 60 pb (Muladno, 2002). Ternak kerbau denga
BB menunjukkan bahwa kedua tetua menyumba
kan kerbau dengan genotipe heterozigot AB me
i kombinasi gen yang berbeda dari kedua tetua
GHRH/HaeIII pada kerbau lokal ditunjukkan pad
M AA AB BB
Gambar 4. Diagram Elektroforesis Hasil Pemotongan Gen GHRH/HaeIII
Bagian struktural gen terdiri dari bagian yang dapat mengkode protein
(kodon) yang disebut ekson dan bagian yang tidak dapat mengkode protein (non
kodon) yang disebut intron atau interving sequences (IVS). Ekson berisi informasi kode asam amino yang akan di produksi oleh gen, sedangkan non kodon berperan
sebagai penyambung dalam proses produksi asam amino (Nei, 1987). Situs
keragaman yang diperoleh pada penelitian ini terletak di intron 2 sehingga mutasi
terjadi pada sekuen yang tidak ditranskripsikan.
Pola pemotongan fragmen DNA pada penelitian ini berbeda dengan
penelitian – penelitian yang telah dilakukan pada beberapa jenis ternak. Hal ini
dikarenakan susunan basa pada setiap individu berbeda sehingga daerah pengenalan
dan pemotongan enzim restriksinya pun berbeda pula, sehingga pita hasil
pemotongannya dapat berbeda ukuran.
Identifikasi keragaman gen GHRH pada sapi telah dilakukan dengan metode
PCR-RFLP menggunakan enzim restriksi HaeIII oleh Moody et al. (1995) yang melaporkan adanya dua jenis alel yaitu alel A dan alel B dengan tiga jenis genotipe
yaitu AA, AB dan BB. Ternak sapi dikatakan mempunyai genotipe AA apabila pola
pemotongan enzim restriksi menghasilkan tiga fragmen DNA dengan panjang 317
menghasilkan lima fragmen DNA dengan panjang 317 pb, 196 pb, 121 pb, 83 pb dan
55 pb. Genotipe BB apabila fragmen DNA terpotong di 196bp, 121bp, 83bp, 55bp,
menghasilkan empat pola pemotongan fragmen DNA.
Kmiéc et al. (2007) melaporkan adanya keragaman gen GHRH/HaeIII pada ternak sapi Limousin dengan metode PCR-RFLP. Produk PCR sepanjang 296 pb
dapat terpotong dan menghasilkan dua jenis alel yaitu alel A dan alel B serta
menghasilkan tiga genotipe yang memiliki pola pemotongan fragmen 194, 55 dan 48
bp (BB); 242, 194, 55 dan 48 bp (AB) dan 242, 55 dan 48bp (AA). Sedangkan
Franco et al. (2005) melaporkan terdapat keragaman gen GHRH pada babi Landrace yaitu alel A dengan keragaman fragmen pada 250 pb dan alel B dengan keragaman
fragmen pada 230 pb.
Keragaman Gen GHRH Kerbau Lokal
Hasil penelitian ini menunjukkan bahwa gen GHRH pada kerbau lokal
bersifat polimorfik (beragam) pada semua populasi dari empat daerah di Indonesia.
Suatu alel dikatakan polimorfik jika memiliki frekuensi alel sama dengan atau
kurang dari 0,99 (99%). Keragaman genetik terjadi apabila terdapat dua alel atau
lebih dalam suatu populasi (biasanya lebih dari 1%) (Nei, 1987).
Persentase keberhasilan pendeteksian keragaman Gen GHRH dengan metode
PCR-RFLP dalam penelitian ini sebesar 76,56%, yaitu dari 320 sampel dapat
diidentifikasi keragaman gen GHRH sebanyak 245 sampel. Nilai frekuensi genotipe
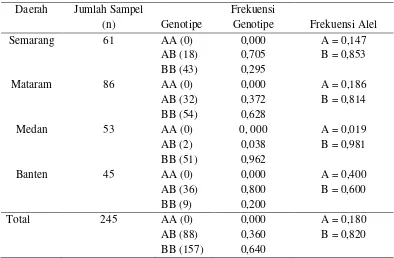
dan frekuensi alel gen GHRH kerbau lokal disajikan pada Tabel 3.
Genotipe yang ditemukan pada penelitian ini yaitu AA (0%), AB (36%) dan
BB (64%). Nilai frekuensi genotipe AB dan BB pada populasi kerbau Semarang
(Jawa Tengah) yaitu 30% dan 70% dengan frekuensi alel A (15%) dan alel B (85%),
frekuensi genotipe AB dan BB kerbau Mataram yaitu 37% dan 63% dengan
frekuensi alel A (19%) dan alel B (81%), populasi kerbau Medan mempunyai
frekuensi genotipe AB dan BB sebesar 3% dan 97% dengan frekuensi alel A (2%)
dan alel B (98%), sedangkan untuk kerbau pada populasi daerah Banten memiliki
nilai frekuensi genotipe AB dan BB sebesar 80% dan 20% dengan frekuensi alel A
(4%) dan alel B (6%). Nilai frekuensi alel B terbesar terdapat pada populasi kerbau
Tabel 3. Nilai Frekuensi Genotipe dan Frekuensi Alel Gen GHRH-HaeIII
Nilai frekuensi genotipe tertinggi kerbau lokal dalam penelitian ini adalah
genotipe BB yaitu sebesar 64%, sedangkan genotipe AA tidak ditemukan dari 320
sampel yang berasal dari empat daerah di Indonesia. Kerbau lokal yang berasal dari
empat daerah di Indonesia dalam penelitian ini memiliki frekuensi alel B yang lebih
tinggi dibandingkan alel A pada semua populasi. Hasil penelitian ini serupa dengan
beberapa penelitian yang dilakukan sebelumnya yang menyebutkan bahwa genotipe
AA jarang ditemukan pada gen GHRH (Moody et al.,1995). Penelitian yang dilakukan oleh Dybus et al. (2003) membuktikan bahwa sapi Limousin yang dilahirkan dengan genotipe homozigot GHRHA/GHRHA lebih rendah (p 0,01)
daripada yang dilahirkan dengan genotipe GHRHA/GHRHB dan GHRHB/GHRHB.
Menurut Moody et al. (1995) GHRHA merupakan alel yang berpengaruh terhadap persentase lemak dan dalam penelitiannya membuktikan bahwa adanya
hubungan antara keragaman GHRH/HaeIIIdan sifat produksi susu pada ternak sapi Fresian Holstein (FH). Individu yang memiliki genotipe GHRHA/GHRHA secara
signifikan mempunyai persentase dan produksi lemak susu yang lebih tinggi. Hasil
yang sama dilaporkan Dybus dan Grzesiak (2006) yang menyebutkan bahwa
yang memiliki satu atau dua alel GHRHA dapat menghasilkan lebih banyak lemak
dalam susunya. Kmiec et al (2007) melaporkan dalam penelitiannya pada ternak sapi perah diperoleh frekuensi genotipe GHRHA/GHRHA sebesar 9,6%, genotipe
GHRHA/GHRHB 37% dan genotipe GHRHB/GHRHB sebesar 53,4%. Frekuensi alel
GHRHA sebesar 28,1% dan Frekuensi alel GHRHB sebesar 79,1%. Penelitian
tersebut menunjukkkan adanya keragaman sekuen gen GHRH/HaeIII dan menghasilkan nilai yang lebih tinggi untuk analisis sifat produksi susu yang meliputi
produksi susu (kg), produksi lemak susu (kg), protein susu (kg), juga total lemak dan
protein (kg) serta kandungan lemak dan protein (%) pada sapi dengan genotipe
GHRHA/GHRHA.
Pierzchala et al. (2003) melaporkan keragaman gen GHRH dengan enzim restriksi AluI dan hubungannya dengan laju pertumbuhan dan kualitas karkas pada ternak babi. Keragaman gen GHRH/aluI menghasilkan Frekuensi genotipe AA (8,8%), AB (61,8%) dan BB (27,97%). Ketebalan lemak pundak berhubungan
dengan genotipe AA. Ternak babi dengan genotipe AB menunjukkan berat lemak
babi yang lebih rendah tetapi lebih tinggi kandungan dagingnya dibandingkan
genotipe AA.
Menurut Cheong et al. (2006) Gen GHRH merupakan salah satu penanda genetik untuk produksi daging. Hasil yang diperoleh dalam penelitiannya
menunjukkan terdapat hubungan yang nyata antara keragaman gen GHRH dengan
sifat karkas yang meliputi bobot karkas dan perototan bagian longisimus pada ternak
pedaging asli Korea (Hanwoo). Hasil tersebut menunjukkan bahwa keragaman
GHRH dapat menjadi faktor penting yang berpengaruh terhadap produksi karkas
pada ternak pedaging.
Nilai Heterozigositas
Heterozigositas disebut juga sebagai keragaman genetik. Nilai heterozigositas
merupakan cara yang paling akurat untuk mengukur keragaman genetik suatu
populasi (Nei, 1987). Nilai heterozigositas dipengaruhi oleh jumlah sampel, jumlah
alel dan frekuensi alel. Nilai heterozigositas kerbau lokal pada penelitian ini berkisar
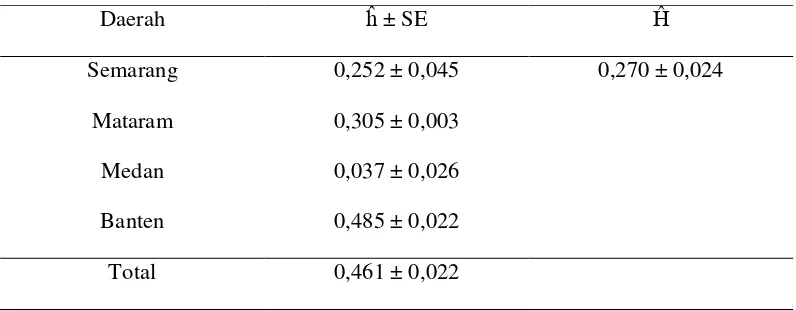
Tabel 4. Nilai heterozigositas ( ) dan Rataan Heterozigositas ( ) Gen GHRH Kerbau Lokal
Daerah ± SE
Semarang 0,252 ± 0,045 0,270 ± 0,024
Mataram 0,305 ± 0,003
Medan 0,037 ± 0,026
Banten 0,485 ± 0,022
Total 0,461 ± 0,022
Populasi kerbau lokal di daerah Medan mempunyai nilai heterozigositas
paling rendah yaitu 0,037. Rendahnya nilai heterozigositas ini kemungkinan besar
diakibatkan oleh rendahnya frekuensi salah satu alel dalam populasi kerbau Medan.
Nilai heterozigositas tertinggi terdapat pada populasi kerbau lokal di daerah Banten
yaitu sebesar 0,485. Hal tersebut menunjukkan bahwa kerbau lokal pada populasi
Banten memiliki keragaman gen GHRH paling tinggi dari keempat populasi dalam
penelitian ini. Nilai heterozigositas total dari empat populasi kerbau lokal di
Indonesia yaitu sebesar 0,461, sedangkan nilai heterozigositas rata – rata dari
keempat populasi yang diamati adalah sebesar 0,270. Nilai heterozigositas kerbau
lokal dalam penelitian ini dapat dikatakan rendah jika dibandingkan nilai keragaman
genetik yang telah ditemukan pada beberapa jenis ternak lain. Sumantri et al. (2006) melaporkan dalam penelitiannya pada DNA mikrosatelit sapi perah FH bahwa nilai
heterozigositas per lokus ( ) paling rendah 0,6151 untuk lokus BM415 dan tertinggi
0,7301 untuk lokus BM888 dengan rataan heterozigositas ( ) untuk kedelapan lokus
sebesar 0,6768. Nilai rataan heterozigositas dalam penelitian Hidayat (2004) pada
DNA mikrosatelit lokus CSSM018 dan IDVGA-30 domba garut yaitu 0,6899.
Sedangkan Fauzi (2006) menyebutkan bahwa rataan nilai heterozigositas DNA
mikrosatelit pada ternak domba lokal lokus CSSM018, ILSTS054 dan IDVGA-30
Indeks Fiksasi
Indeks fiksasi dapat digunakan untuk mengetahui pola perkawinan dan
seleksi yang dilakukan dalam populasi. Indeks fiksasi bisa bernilai positif maupun
negatif, hal tersebut dipengaruhi oleh adanya inbreeding, seleksi dan kawin tidak acak. Nilai indeks fiksasi gen GHRH kerbau lokal dari empat populasi di Indonesia
ditunjukkan pada Tabel 5.
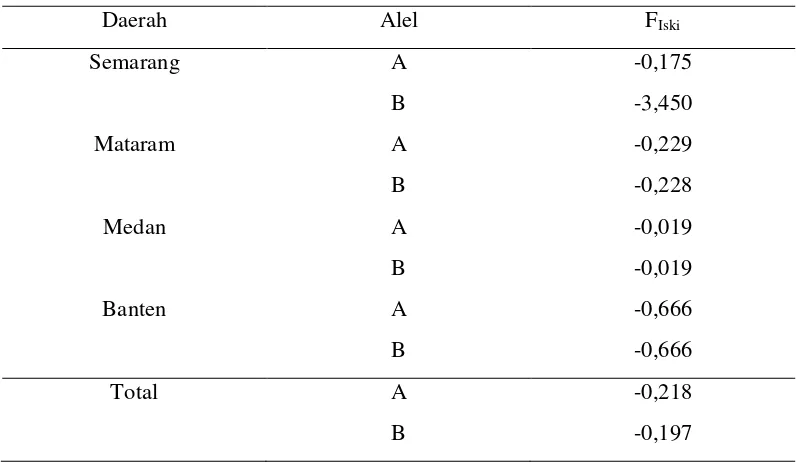
Indeks fiksasi gen GHRH pada keempat populasi kerbau lokal dalam
penelitian ini tidak sama dengan nol. Hal tersebut menunjukkan bahwa pada keempat
populasi kerbau lokal di Indonesia tidak terjadi fiksasi gen GHRH.
Tabel 5. Nilai Indeks Fiksasi Gen GHRH Kerbau Lokal
Daerah Alel FIski
Semarang A -0,175
B -3,450
Mataram A -0,229
B -0,228
Medan A -0,019
B -0,019
Banten A -0,666
B -0,666
Total A -0,218
B -0,197
Jarak Genetik dan Pohon Genetik Gen GHRH
Jarak genetik adalah ukuran perbedaan material genetik diantara individu
yang berbeda spesies atau pun individu dengan spesies sama. Hubungan kekerabatan
dari beberapa kerbau lokal berdasarkan gen GHRH dapat diketahui dengan
menghitung jarak genetik menggunakan rumus Nei (1987). Semakin kecil nilai jarak
genetik antar populasi semakin dekat hubungan kekerabatan antar populasi tersebut,
sebaliknya semakin besar nilai jarak genetik antar populasi maka semakin jauh
berdasarkan gen GHRH antara empat populasi kerbau lokal di Indonesia disajikan
pada Tabel 6.
Tabel 6. Nilai Jarak Genetik Gen GHRHKerbau Lokal
Daerah Daerah
Semarang Mataram Medan Banten Semarang 0,000
Mataram 0,001 0,000
Medan 0,012 0,023 0,000
Banten 0,089 0,064 0,171 0,000
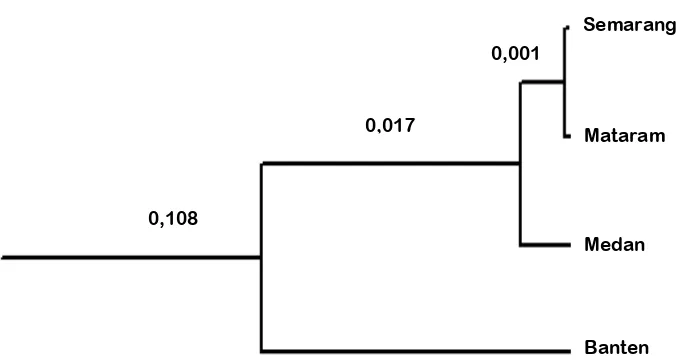
Jarak genetik dapat digunakan untuk membuat dendogram (pohon filogeni)
yang dapat digunakan untuk menunjukkan hubungan kekerabatan antara populasi.
Menurut Nei (1987) hubungan kekerabatan antar populasi dapat diketahui dengan
menggunakan metode yang paling sederhana rata – rata jarak genetik UPGMA
(Unweighted Pair-Group Methode with Arithmetic mean). Hubungan kekerabatan
antara empat populasi kerbau lokal ditunjukkan pada Gambar 5.
Nilai jarak genetik terkecil adalah kerbau lokal Mataram dengan kerbau lokal
Semarang, yaitu 0,001 yang menunjukkan bahwa antara kedua populasi tersebut
memiliki hubungan kekerabatan yang dekat. Hal ini diduga terjadi karena adanya
penyebaran dan pola migrasi. Sedangkan nilai jarak genetik terbesar yaitu antara
kerbau lokal Medan dengan kerbau lokal Banten sebesar 0,202, yang menunjukkan
bahwa hubungan kekerabatan antara kedua populasi kerbau lokal tersebut jauh.
Penelitian ini merupakan penelitian pendahuluan yang dilakukan untuk
mengetahui keragaman gen GHRH pada kerbau lokal di Indonesia sehingga
informasi yang diperoleh masih sangat sedikit untuk dijadikan sebagai bahan
perbandingan. Data yang diperoleh dalam penelitian ini dapat digunakan untuk
penelitian selanjutnya agar dapat menganalisis hubungan antar tipe maupun genotipe
gen GHRH dengan sifat – sifat kuantitatif kerbau lokal Indonesia, sehingga dapat
KESIMPULAN DAN SARAN
Kesimpulan
Gen GHRH pada populasi kerbau lokal di Indonesia bersifat polimorfik
(beragam). Identifikasi keragaman gen GHRH pada kerbau lokal Indonesia dengan
teknik PCR-RFLP menghasilkan dua alel, yaitu alel A (18%) dan alel B (82%)
Genotipe yang diperoleh yaitu AA (0%), AB (36%) dan BB (64%) dengan nilai
heterozigositas sebesar 46%. Alel B memiliki frekuensi lebih besar dibandingkan alel
A pada semua populasi ternak kerbau dari empat daerah di Indonesia. Frekuensi Alel
A pada setiap populasi yaitu Semarang (15%), Mataram (19%), Medan (2%) dan
Banten (45%). Alel B memiliki frekuensi lebih besar dibandingkan alel A pada
semua populasi ternak kerbau dari empat daerah di Indonesia. Frekuensi alel B pada
setiap populasi yaitu Semarang (85%), Mataram (81%), Medan (98%) dan Banten
(48%). Nilai frekuensi alel B terbesar terdapat pada populasi kerbau Medan (98%)
dengan nilai heterozigositas 4%. Nilai heterozigositas tertinggi terdapat pada
populasi kerbau di Banten yaitu 48%. Nilai indeks fiksasi gen GHRH menunjukkan
bahwa dari keempat populasi kerbau lokal tidak terdapat gen yang terfiksasi dengan
nilai indeks fiksasi tidak sama dengan nol. Nilai jarak genetik gen GHRH terkecil
yaitu antara populasi kerbau lokal Semarang dan kerbau lokal Mataram (0,001) dan
terbesar antara populasi kerbau lokal Medan dan Banten (0,202).
Saran
Perlu dilakukan penelitian lanjutan untuk melakukan analisis hubungan
antara keragaman gen GHRH dengan sifat-sifat kuantitatif kerbau lokal Indonesia.
Data yang diperoleh selanjutnya dapat dijadikan sebagi acuan dalam melakukan