RINGKASAN
Febynia Mutiara Zainatha. D14080220. 2012. Identifikasi Keragaman Gen Growth Hormone Receptor Exon 8 (GHR|SSpI) pada Sapi Friesian Holstein dengan Metode PCR-RFLP. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Ir. Anneke Anggraeni, M.Si, Ph.D
Growth Hormone Receptor (GHR) merupakan suatu protein transmembran yang mengikat GH dengan afinitas dan spesifitas yang tinggi. Gen Growth Hormone membutuhkan suatu reseptor dalam mekanisme ekspresinya ke target jaringan, yaitu gen Growth Hormone Receptor (GHR). Gen GHR memiliki fungsi sebagai mediasi gen pertumbuhan yang mempengaruhi sifat pertumbuhan karkas pada sapi pedaging dan sifat produksi susu pada sapi perah. Gen GHR diharapkan dapat menjadi informasi sebagai gen yang mempengaruhi produktifitas ternak melalui analisis dari keragaman gen tersebut. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen Growth Hormone Receptor (GHR) exon 8 dengan menggunakan enzim restriksi SSpI pada sapi Friesian Holstein dengan metode PCR-RFLP pada lima daerah yang berbeda, yaitu KPSBU Pasir Kemis, KPSBU Cilumber, BPPT SP Cikole, BIB Lembang, dan BBIB Singosari.
Materi penelitian berupa sampel darah sebanyak 302 sampel dan semen sapi FH sebanyak 10 sampel dengan total sebanyak 312 ekor yang terdiri dari sapi pejantan yang berasal dari BIB Lembang (10 ekor) dan BBIB Singosari (32 ekor) serta sapi betina yang berasal dari BPPT SP Cikole (81 ekor), KPSBU Pasir Kemis (95 ekor), dan KPSBU Cilumber (94 ekor). Amplifikasi gen GHR|SSpI dilakukan dengan metode Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP). Hasil produk amplifikasi adalah sepanjang 230 bp. Genotipe yang ditemukan pada gen GHR|SSpI adalah AA (230 BP), AT (230 bp, 200 bp, 30 bp), dan TT (200 bp, 30 bp) dengan dua macam alel yaitu alel A dan alel T.
Hasil analisis menunjukkan bahwa frekuensi genotipe tertinggi yang terdapat di semua populasi adalah genotipe TT dan frekuensi genotipe terendah adalah genotipe AA. Nilai frekuensi alel total untuk semua populasi memperlihatkan bahwa frekuensi alel tertinggi sebesar 0,728 untuk alel T dan terendah sebesar 0,272 untuk alel A. Sapi FH di BPPT SP Cikole, KPSBU Cilumber dan KPSBU Pasir Kemis berada pada keadaan keseimbangan Hardy-Weinberg (χ2 hitung > χ2 0,05). Hasil analisis heterozigositas menunjukkan bahwa sapi FH di BPPT SP Cikole dan KPSBU Pasir Kemis memiliki nilai heterozigositas pengamatan (Ho) yang lebih rendah dari nilai heterozigositas harapan (He), sedangkan nilai heterozigositas pengamatan (Ho) pada sapi FH di KPSBU Cilumber, BBIB Singosari, dan BIB Lembang lebih tinggi dari nilai heterozigositas harapannya (He). Kesimpulan yang dapat diambil dari hasil analisis adalah gen GHR|SSpI pada BPPT SP Cikole, KPSBU Pasir Kemis, KPSBU Cilumber, BIB Lembang dan BBIB Singosari bersifat polimorfik.
ABSTRACT
Identification of the Growth Hormone Receptor Exon 8 (GHR|SSpI) Gene Polymorphism in Holstein Friesian with PCR-RFLP Method
Zainatha, F. M., C. Sumatri, and A. Anggraeni
PENDAHULUAN
Latar Belakang
Bangsa sapi Friesian Holstein (FH) merupakan salah satu bangsa sapi penghasil susu tertinggi dibandingkan bangsa sapi perah lainnya di daerah tropis maupun sub tropis. Produksi susu sapi FH di Indonesia rata-rata 10 liter/hari. Namun demikian, produksi susu sapi FH belum dapat memenuhi kebutuhan susu di dalam negeri. Perbaikan mutu genetik sapi FH di Indonesia dapat dikatakan sangat lambat, walaupun sudah dilakukan inseminasi buatan. Hal ini dikarenakan belum adanya recording yang baik pada peternakan-peternakan di Indonesia. Salah satu cara untuk memperbaiki mutu genetik adalah dengan melakukan seleksi pada sapi FH itu sendiri. Perkembangan teknologi molekuler saat ini dapat lebih mempermudah dalam melakukan seleksi dibandingkan dengan cara lainnya. Seleksi melalui molekuler dapat dilakukan dengan melihat keragaman gen yang mempengaruhi produksi susu sapi FH.
Salah satu teknologi molekuler yang dapat digunakan untuk menganalisis keragaman gen adalah PCR-RFLP (Polymerase Chain Reaction-Restriction Fragment Lenght Polymorphism). Metode PCR memanfaatkan perbedaan pola pemotongan enzim restriksi atau enzim pemotong yang berbeda-beda pada setiap mikroorganisme. Analisis RFLP biasa digunakan untuk mendeteksi adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu (Sumantri et al., 2007).
Hormon pertumbuhan (Growth Hormone) merupakan salah satu hormon yang mempengaruhi produksi susu pada sapi perah. Hormon pertumbuhan ini diekspresikan oleh gen-gen. Gen Growth Hormone memiliki berat molekul yang tinggi sehingga sulit memasuki sel-sel yang harus melewati membran sel. Oleh karena itu, gen GH membutuhkan gen Growth Hormone Receptor (GHR) sebagai reseptor yang terdapat dalam membran sel, agar gen GH dapat memberikan efeknya ke target sel tanpa merusak membrannya (Djojosoebagio, 1996).
memiliki panjang 25.688 pb (Lucy et al., 1998, Jiang dan Lucy, 2001). Menurut Blott et al. (2003) terdapat beberapa exon yang diduga mempengaruhi produksi susu sapi perah, yaitu exon 3. 8, 9, dan10. Exon 8 merupakan exon yang diduga sebagai kandidat yang paling mempengaruhi produksi susu sapi perah. Keragaman gen GHR di exon 8 ini dapat dianalisis dengan menggunakan enzim rekstriksi SSpI. Oleh karena itu diperlukan penelitian mengenai keragaman gen GHR di exon 8 dan pengaruhnya terhadap produktifitas sapi perah. Keragaman gen GHR exon 8 ini diharapkan dapat dijadikan sebagai informasi untuk seleksi dalam hal peningkatan mutu genetic sapi FH di Indonesia
Tujuan
Penelitian ini bertujuan untuk mengidentifikasi keragaman gen Growth Hormone Receptor (GHR) exon 8 dengan menggunakan enzim restriksi SSpI pada sapi Friesian Holstein dengan metode PCR-RFLP pada lima daerah yang berbeda, yaitu KPSBU Pasir Kemis, KPSBU Cilumber, BPPT SP Cikole, BIB Lembang, dan BBIB Singosari.
TINJAUAN PUSTAKA
Sapi Perah Friesian Holstein
Sapi Friesian Holstein (FH) merupakan bangsa sapi yang paling banyak terdapat di Amerika Serikat, sekitar 80-90% dari seluruh sapi perah yang berada di sana. Sapi ini berasal dari Belanda yaitu di provinsi North Holand dan West Friesland yang memiliki padang rumput yang sangat luas. Sapi FH mempunyai beberapa keunggulan, salah satunya yaitu jinak, tidak tahan panas tetapi sapi ini mudah menyesuaikan diri dengan keadaan lingkungan. Ciri-ciri sapi Friesian Holstein yang baik adalah memiliki tubuh luas ke belakang, sistem dan bentuk perambingan baik, puting simetris, dan efisiensi pakan tinggi yang dialihkan menjadi produksi susu (Blakely dan Bade, 1998). Sapi ini tubuhnya memiliki pola warna hitam dan putih dan ada pula yang memiliki pola warna merah dan putih. Gambar sapi FH dapat dilihat pada Gambar 1.
Ukuran badan, kecepatan pertumbuhan serta karkasnya yang bagus menyebabkan sapi ini sangat disukai pula untuk tujuan produksi daging serta pedet untuk dipotong selain untuk produksi susu (Blakely dan Bade, 1998). Bangsa Friesian Holstein sangat menonjol dibandingkan bangsa lain karena memiliki jumlah produksi susu yang banyak dengan kadar lemak yang rendah. Sifat seperti ini sangat dibutuhkan oleh peternak sapi perah saat ini.
Bangsa sapi FH merupakan penghasil susu tertinggi dibandingkan bangsa sapi perah lainnya di daerah tropis maupun sub tropis dengan kadar lemak yang rendah. Populasi sapi perah di Indonesia diperkirakan sebanyak 487.000 ekor yang sebagian besar adalah bangsa sapi Friesian Holstein (FH) (Direktorat Jenderal Peternakan, 2009). Susu dihasilkan di sentra-sentra produksi sapi perah seperti di provinsi Jawa Barat, Jawa Tengah, dan Jawa Timur.
Gambar 1. Sapi Friesian Holstein Jantan Sumber : http://dompi.co.id
Gen Growth Hormone (GH)
Pertumbuhan merupakan salah satu sifat utama dan penting dari makhluk hidup. Secara umum pertumbuhan memiliki aspek yang luas seperti pertumbuhan sel, organ, fetus, tulang dan beberapa aspek lain yang terkait dengan pertumbuhan individu (Zulkharnaim et al., 2010). Pertumbuhan suatu individu dipengaruhi oleh hormon pertumbuhan. Hormon pertumbuhan merupakan hormon anabolik yang disintesis dan disekresikan oleh sel somatotrof pada lobus anterior pituitary (Ayuk dan Sheppard, 2006). Hormon pertumbuhan diekspresikan oleh gen-gen. Gen Growth Hormone adalah salah satu gen yang penting yang diperlukan untuk pertumbuhan jaringan, metabolisme lemak, dan pertumbuhan badan normal. Oleh karena itu, gen GH sering dijadikan kandidat untuk program Marker Assisted Selection pada sapi (Beauchemin et al., 2006). Gen GH mempunyai panjang 2800 pb dengan 5 ekson dan 4 intron dan terletak pada kromosom ke 19 (Ardiyanti et al., 2009).
Fungsi dari gen GH khususnya pada ternak menjadi sangat penting karena gen GH mengatur sifat-sifat yang bernilai ekonomi yang tinggi. Menurut Sumantran et al. (1992) gen GH terbukti menjadi pengatur utama pada pertumbuhan pasca
kelahiran, metabolisme pada mamalia, kecepatan pertumbuhan, susunan tubuh, kesehatan. Selanjutnya, gen GH merupakan kandidat gen dalam pengaturan produksi susu, karkas dan respon immun (Ge et al., 2003).
Gen Growth Hormone Receptor (GHR) menjadi kandidat gen untuk pertumbuhan pada ternak disebabkan gen GH membutuhkan reseptor dalam mekanisme ekspresinya ke target jaringan (Zulkharnaim et al., 2010). Zhou dan
Jiang (2005) menyatakan bahwa pada tingkatan jaringan, gen GH dimediasi oleh gen GHR.
Gen Growth Hormone Receptor (GHR)
Menurut Di Stasio et al. (2005), Growth Hormone Receptor (GHR) merupakan suatu protein transmembran yang mengikat GH dengan afinitas dan spesifitas yang tinggi. Gen Growth Hormone memiliki berat molekul yang tinggi sehingga sulit memasuki sel-sel yang harus melewati membran sel. Oleh karena itu, gen GH membutuhkan reseptor yang terdapat dalam membran sel, agar gen GH dapat memberikan efeknya ke target sel tanpa merusak membrannya (Djojosoebagio, 1996).
Gen GHR pada sapi dipetakan sebagai gen tunggal yang terletak pada kromosom 20 (Moody et al., 1995), terdiri atas 10 ekson dan 9 intron dan memiliki panjang 25.688 pb (Lucy et al., 1998, Jiang dan Lucy, 2001). Gambar rekonstruksi gen GHR dapat dilihat pada Gambar 2. Proses transkripsi gen GHR pada sapi di inisiasikan oleh tiga promotor exon utama 1A, 1B, dan 1C (Jiang dan Lucy, 2001).
Gambar 2. Rekonstruksi Struktur Gen Growth Hormone Receptor Berdasarkan Sekuen Gen GHR di GenBank (Kode Akses. EF207442)
Hasil produk PCR pada gen GHR exon 8 adalah sepanjang 230 bp. Hasil pemotongan fragmen GHR oleh SspI menghasilkan tiga genotipe yaitu AA (230 bp), AT (230 bp, 200 bp, 30 bp), dan TT (200 bp, 30 bp). Genotipe TT adalah genotipe yang mempunyai karakteristik produksi susu paling rendah dibandingkan dengan genotipe AT dan AA (Khatib et al., 2009). Menurut Komisarek et al. (2010) genotipe AA merupakan genotipe yang mempunyai karakteristik produksi susu paling tinggi.
Keragaman Genetik
Menurut Kirby (1990) keragaman genetik atau polimorfisme genetik adalah terdapatnya lebih dari satu bentuk atau macam genotipe di dalam populasi. Keragaman genetik adalah perbedaan di dalam runutan DNA antar individu, kelompok, atau suatu populasi. Sumber keragaman ini dapat disebabkan oleh adanya pengulangan urutan sekuen, insersi, delesi, dan rekombinasi.
Keragaman genetik dapat digunakan untuk mengetahui dan melestarikan bangsa-bangsa dalam suatu populasi yang erat kaitannya dengan penciri atau suatu sifat khusus (Blott et al., 1998). Suatu populasi yang alami memiliki tingkat keragaman yang tinggi. Frekuensi genotipe dan alel merupakan suatu parameter dasar untuk mempelajari proses terjadinya evolusi karena perubahan genetik pada sebuah populasi yang biasanya digambarkan dengan adanya perubahan pada frekuensi alel (Nei dan Kumar, 2000).
Tingkat keragaman dalam suatu populasi dapat dilihat dari frekuensi alelnya. Frekuensi alel adalah rasio relatif suatu alel terhadap keseluruhan alel yang ditemukan dalam suatu populasi (Nei dan Kumar, 2000). Menurut Falconer dan Mackay (1996), suatu populasi dinilai beragam apabila memiliki dua atau lebih alel dalam satu lokus dengan frekuensi yang cukup. Suatu alel dikatakan polimorfik jika memiliki frekuensi alel sama atau kurang dari 0,99.
Hukum Hardy-Weinberg dapat menggambarkan keseimbangan suatu lokus dalam populasi yang telah mengalami kawin acak dan bebas dari faktor yang berpengaruh terhadap terjadinya proses evolusi seperti mutasi, migrasi, dan pergeseran genetik (Gillespie, 1998). Noor (2000) menyatakan bahwa hukum ini memperlihatkan frekuensi gen dominan dan resesif pada suatu populasi yang cukup besar tidak akan berubah dari satu generasi ke generasi berikutnya jika tidak
terjadinya seleksi, migrasi, mutasi, dan genetic drift. Keadaan populasi yang demikian disebut dalam keadaan equilibrium (seimbang).
Ukuran tinggi rendahnya keragaman genetik dalam suatu kelompok atau populasi dapat dilihat berdasarkan nilai heterozigositas. Semakin tinggi derajat heterozigositas suatu populasi maka daya tahan hidup populasi tersebut akan semakin tinggi (Avise, 1994). Menurut Nei (1987), derajat heterozigositas adalah rataan presentase lokus heterozigositas tiap individu atau rataan presentase individu heterozigot dalam populasi.
Analisis Keragaman DNA
Analisis keragaman DNA bisa dilakukan dengan berbagai cara, antara lain RFLP, SSCP, DGGE, dan sequencing. Polimerase Chain Reaction (PCR) adalah suatu reaksi in vitro untuk menggandakan molekul DNA pada target yang telah ditentukan dengan cara mensintesa molekul DNA baru yang berkomplemen dengan molekul DNA tersebut dengan enzim polimerase dan oligonukleotida pendek. Proses yang terjadi dalam PCR meliputi tiga tahap yang penting yaitu denaturasi (pemisahan untai ganda DNA), annealing (penempelan primer), dan ekstensi (pemanjangan primer) (Muladno, 2002). Analisis RFLP sering digunakan untuk mendeteksi lokasi genetik dalam kromosom yang menyandikan penyakit yang diturunkan (Orita et al., 1989) ataupun untuk mendeteksi keragaman gen yang berhubungan dengan sifat-sifat ekonomis ternak seperti produksi dan kualitas susu (Sumantri et al., 2007).
Selain PCR-RFLP, PCR-SSCP juga dapat digunakan untuk analisis keragaman DNA. PCR-SSCP merupakan metode analisis lebih lanjut yang memanfaatkan produk PCR. Metode PCR-SSCP merupakan metode yang handal dalam mendeteksi adanya mutasi secara cepat (Hayashi, 1991). Asumsi yang mendasari metode analisis SSCP adalah bahwa perubahan yang terjadi pada nukleotida meskipun terjadi hanya pada satu basa, akan mempengaruhi bentuk (conformation) dari fragmen DNA pada kondisi untai tunggal (Bastos et al., 2001). Perbedaan konformasi molekul akan menyebabkan perbedaan migrasinya dalam gel poliakrilamid pada saat elektroforesis (Montaldo et al., 1998). Metode PCR-SSCP dapat mendeteksi perubahan pada satu basa tetapi tidak dapat diketahui basa mana yang berubah.
Polymerase Chain Reaction-Restriction Fragment Length Polymorphism
(PCR-RFLP)
Polymerase Chain Reaction-Restriction Fragment Length Polymorphism
(PCR-RFLP) adalah suatu metode analisis lanjutan dari produk PCR. Metode PCR memanfaatkan perbedaan pola pemotongan enzim restriksi atau enzim pemotong yang berbeda-beda pada setiap mikroorganisme (Orita et al., 1989). Prinsip kerja RFLP adalah menghilangkan semua mutasi untuk menciptakan sekuen rekognisi baru. Metode ini bisa mendeteksi mutasi jika situs restriksi mengalami perubahan susunan basa. Apabila mutasi terjadi diluar situs restriksi, maka mutasi tersebut tidak dapat dideteksi. Menurut Nei dan Kumar (2000), atas dasar terpotong atau tidaknya fragmen DNA dengan enzim pemotong, hasil fragmen potongan DNA tersebut dapat divisualisasikan melalui teknik elektroforesis yang hasilnya menunjukan ada tidaknya polimorfisme pada suatu individu atau populasi. Analisis RFLP biasa digunakan untuk mendeteksi adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu (Sumantri et al., 2007).
METODE
Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboratorium Pemuliaan dan Genetika Molekuler, Bagian Pemuliaan dan Genetik Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian ini dilaksanakan selama enam bulan, yaitu dari September 2011 sampai dengan Maret 2012.
Materi
Ternak
Sampel darah dan semen yang digunakan merupakan koleksi sampel Laboratorium Genetika Molekuler tahun 2008-2010. Jumlah sampel yang digunakan adalah 312 sampel berasal dari lima daerah yang berbeda (Tabel 1).
Tabel 1. Jumlah Sampel DNA yang Digunakan dan Asal Pengambilan Sampel Darah.
Asal Sampel Jenis Kelamin Asal DNA Jumlah Sampel
KPSBU Pasir Kemis Betina Darah 95
KPSBU Cilumber Betina Darah 94
BPPT SP Cikole Betina Darah 81
BBIB Singosari Jantan Darah 32
BIB Lembang Jantan Semen 10
Jumlah 312
Bahan dan Alat
Bahan yang digunakan adalah alkohol, es, dan kapas. Alat yang digunakan antara lain jarum vacutainer, tabung vacum 10 ml dan termos.
Ekstraksi DNA
EDTA). Alat yang digunakan antara lain autoclave, satu set pipet mikro dan tipnya, vortexmixer, alat sentrifugasi, refrigerator, dan freezer.
Primer
Primer yang digunakan dalam penelitian untuk mengamplifikasi gen Growth Hormone Receptor (GHR) menurut Khatib et al. (2009) adalah
forward: 5´-CTT TGG AAT ACT TGG GCT AGC AGT GAC A”A”T AT-3´; reverse: 5´ -GTC TCT CTG TGG ACA CAA CA-3‟
Amplifikasi DNA dengan PCR-RFLP
Bahan yang digunakan adalah air bebas ion (steril), sampel DNA, buffer, MgCl2, pasangan primer, enzim taq dan dNTP, enzim retriksi SSpI dan buffer G. Alat yang digunakan adalah satu set pipet mikro dan tipnya, alat sentrifugasi, tabung PCR, mesin PCR, vortex, lemari es.
Elektroforesis
Bahan yang digunakan adalah air destilasi, agarose, 0,5x TBE, EtBr, loading dye (0,01% Xylene Cyanol, 0,01% Bromtimolblue, 50% gliserol), dan marker 100 bp. Alat yang digunakan antara lain satu set alat pencetak gel, power supply 100 volt, pipet mikro, tip dan alat gelas.
Genotyping
Bahan-bahan yang digunakan yaitu loading dye (bromthymol blue 0,01%, Xylene cyanol 0,01% dan gliserol 50%) dan untuk membuat 1 lembar gel agarose 2% adalah sebagai berikut: agarose 0,6 g, 0,5 x TBE 30 ml, dan 2,5 μl EtBr. Alat-alat yang digunakan antara lain adalah microwive, stirer, magnetic stirer, gelas ukur, tabung erlenmeyer, gel tray, pencetak untuk sumur (comb), power supply 100 volt, gelas ukur, tip, pipet makro dan mikro.
Prosedur
Pengambilan Sampel
Sampel yang digunakan terdiri dari sampel darah dan semen. Pengambilan sampel darah dilakukan pada empat tempat berbeda, yaitu KSPBU Pasir Kemis, KPSBU Cilumber, BPPT SP Cikole, dan BBIB Singosari. Sampel darah diambil
melalui vena jugularis menggunakan jarum vacutainer tabung vacum yang ditambahkan alkohol 70%. Sampel disimpan dalam termos es atau lemari es sampai akan digunakan lebih lanjut. Sampel semen didapatkan dengan cara membeli ke BIB Lembang. Sampel kemudian ditambahkan dengan alcohol 70 % dan disimpan pada suhu ruang.
Ekstraksi DNA
Sampel darah dan semen diambil sebanyak 200 µl, kemudian ditambahkan 1.000 μl DW untuk sampel darah dan 1000 µl untuk sampel semen. Sampel divortex lalu didiamkan selama 5 menit. Setelah itu disentrifuse pada kecepatan 8.000 rpm selama 5 menit, lalu supernatan dibuang, dan diulangi seperti proses sebelumnya, kemudian ditambahkan 10 µl proteinase-K yang berfungsi untuk menghancurkan protein, 350 µl 1xSTE (sodium tris-EDTA) dan 40 µl 10% SDS (sodium dodesil sulfat) yang berfungsi untuk melisiskan membran sel. Campuran tersebut kemudian diinkubasi pada suhu 55oC selama 2 jam sambil dikocok pelan menggunakan alat pemutar (tilting).
Molekul DNA kemudian dimurnikan dengan metode fenol-chloroform, yaitu dengan menambahkan 40 µl 5M NaCl, 400 µl larutan fenol dan CIAA (chloroform iso amil alcohol), lalu dikocok pelan (tilting) pada suhu ruang selama 1 jam. Molekul DNA yang larut dalam fase air dipisahkan dari fase fenol dengan alat sentrifugasi pada kecepatan 12.000 rpm selama 5 menit. Setelah terbentuk fase DNA, diambil sebanyak 400 µl pada fase DNA untuk dipindahkan ke tabung baru 1,5 ml. Kemudian ditambahkan 5M NaCl sebanyak 40 µl dan etanol absolut sebanyak 800 µl. Molekul DNA kemudian disimpan selama semalam (over night) pada suhu -20oC.
Molekul DNA kemudian dipisahkan dari etanol absolut dengan cara sentrifugasi pada kecepatan 12.000 rpm selama 5 menit, kemudian supernatan yang diperoleh dibuang. Endapan tersebut didiamkan sampai kering. Lalu endapan DNA disuspensikan dalam 100 µl 80% buffer TE (tris EDTA).
Amplifikasi DNA
Amplifikasi gen GHR dilakukan dengan menggunakan campuran yang terdiri dari 1 µl sampel DNA yang sudah diekstraksi, 10,85 µl DW, 0,3 µl primer, 0,05 µl taq polymerase, 1,5 µl buffer, 0,3 µl dNTP dan 1 µl MgCl2. Campuran tersebut
kemudian dimasukan ke dalam mesin PCR dengan kondisi suhu pradenaturasi 95oC selama 5 menit, 35 siklus (denaturasi 95oC selama 45 detik, annealing 60oC selama 45 detik dan elongasi 72oC selama 1 menit) dan elongasi akhir 72oC selama 5 menit.
Elektroforesis
Elektroforesis produk PCR dilakukan menggunakan 5 μl produk PCR pada gel agarose 1,5% dengan tegangan 100 volt selama 30 menit. Gel dibuat dengan cara memanaskan agarose 0,45 g yang dilarutkan dalam larutan 0,5xTBE 30 ml serta menambahkan 2,5 μl EtBr pada saat distearer sampai didapatkan larutan jernih. Larutan yang masih cair dituangkan ke dalam pencetak gel serta menempatkan sisir di dekat tepian gel dan gel dibiarkan mengeras. Apabila gel sudah mengeras, sisir dicabut sehingga akan terbentuk sumur-sumur yang digunakan untuk menempatkan sebanyak 5μl produk PCR dicampur dengan loading dye (bromthymol blue 0,01%, Xylene cyanol 0,01% dan gliserol 50%). Gel ditempatkan ke dalam gel tray elektroforesis yang sudah terisi larutan buffer dan dialiri listrik, molekul DNA yang bermuatan negatif pada pH netral akan bergerak (migrasi) ke arah positif (anode). Setelah elektroforesis selesai gel agarose diambil untuk dilihat panjang pita DNA dengan menggunakan sinar Ultraviolet yaitu dengan menarik garis lurus antara posisi pita dari masing-masing sampel DNA yang ingin diukur dengan posisi pita DNA marker, kita dapat mengestimasi ukuran sampel DNA karena ukuran DNA pengukur telah diketahui.
Genotyping
Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP) dilakukan dengan menggunakan sebanyak 2 μl produk PCR yang dipindahkan ke dalam tabung 0,5 ml dan ditambahkan 1 μl DW, 0,3 μl enzim restriksi SspI dan 0,7 μl buffer G. Campuran tersebut diinkubasikan dalam inkubator pada suhu 37°C selama 16 jam. Sampel DNA yang telah dipotong dengan enzim restriksi kemudian dielektroforesis pada gel agarose 2% dengan tegangan 100 volt selama 30 menit. Setelah elektroforesis selesai gel agarose diambil untuk dilihat panjang pita DNA dengan menggunakan sinar Ultraviolet dan dibandingkan dengan marker untuk mengetahui panjangnya. Setiap pita DNA dari setiap sampel diperbandingkan untuk menentukan genotipe pita DNA.
Satu posisi migrasi yang sama dianggap sebagai satu tipe atau alel. Menurut Khatib et al. (2009), alel yang dihasilkan dari genotyping adalah alel A sepanjang 230 bp dan alel T sepanjang 200 bp dan 30 bp. Hasil pemotongan fragmen GHR oleh SSpI menghasilkan tiga genotipe yaitu AA (230 bp), AT (230 bp, 200 bp, 30 bp), dan TT (200 bp, 30 bp) (Khatib et al., 2009).
Rancangan dan Analisa Data
Frekuensi Genotipe dan Alel
Keragaman genotipe pada masing-masing sampel dapat dilihat dari pita-pita yang ditemukan. Frekuensi genotipe dapat diperkirakan dengan menghitung perbandingan jumlah genotipe pada populasi bedasarkan rumus Nei dan Kumar (2000) :
Keterangan:
= frekuensi genotipe ke-ii = jumlah individu bergenotipe ii = jumlah individu sampel
Frekuensi alel merupakan rasio suatu alel terhadap keseluruhan alel pada suatu lokus dalam populasi. Frekuensi alel masing-masing alel setiap lokus dihitung berdasarkan rumus Nei (1987) :
Keterangan:
Xi = frekuensi alel ke-i
nii = jumlah individu bergenotipe ii
nij = jumlah individu bergenotipe ij
n = jumlah individu sampel
Keseimbangan Hardy-Weinberg
Keseimbangan Hardy-Weinberg diuji dengan Chi-Kuadrat (Hartl and Clark, 1997):
Keterangan:
x2 = uji Chi-Kuadrat (obs-exs)2
obs = jumlah pengamatan genotipe ke-ii
exp = jumlah harapan genotipe ke-ii
Derajat bebas (db) dihitung untuk mendapatkan nilai x2 tabel. Nilai derajat bebas dihitung berdasarkan Allendorf dan Luikart (2007) dengan menggunakan rumus :
Db = (Genotipe-1) – (Alel-1)
Heterozigositas
Keragaman genetik (genetic variability) dilakukan melalui estimasi frekuensi heterozigositas pegamatan (H0), heterozigositas harapan (He) (Weir, 1996) :
Keterangan :
Ho = nilai heterozigositas pengamatan Nij = jumlah individu heterozigositas N = jumlah individu yang dianalisis
Keterangan :
He = nilai heterozigositas harapan P1i = frekuensi alel homozigot n = jumlah alel
HASIL DAN PEMBAHASAN
Amplifikasi Gen GHR|SSpI
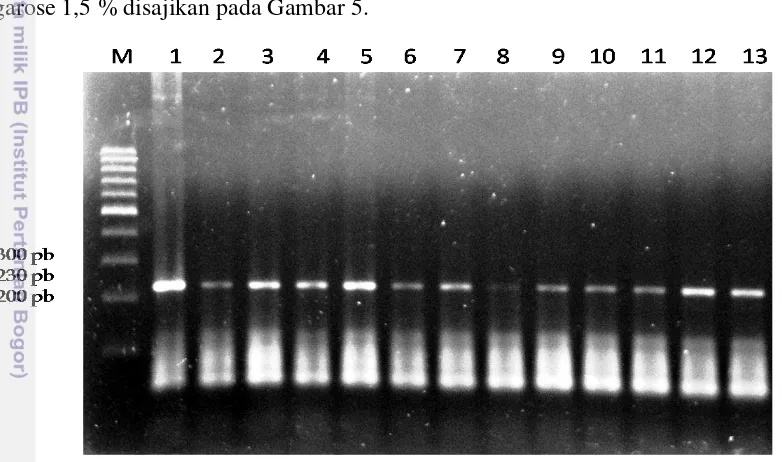
Proses amplifikasi gen GHR|SSpI pada sapi FH di KPSBU Pasir Kemis, KPSBU Cilumber, BPPT SP Cikole, BIB Lembang, dan BBIB Singosari dilakukan dengan metode Polymerase Chain Reaction (PCR). Produk amplifikasi gen GHR|SSpI yang didapatkan adalah sepanjang 230 bp. Hal ini sesuai dengan Khatib et al. (2009) yang menyatakan bahwa produk hasil amplifikasi gen GHR|SSpI exon 8 adalah sepanjang 230 bp. Hasil amplifikasi gen GHR|SSpI sepanjang 230 bp pada gel agarose 1,5 % disajikan pada Gambar 5.
Gambar 5. Visualisasi Hasil Amplifikasi Gen GHR|SSpI Sepanjang 230 bp pada Gel Agarose 1,5 %. M (Marker) dan 1-13 (Sampel)
Forward
1983 ttccagtttc catggttctt aattattatc tttggaatac ttgggctagc agtgacaat|a
2043 tttttactca tattttctaa acagcaaagg taagtgtgat ataacctact ctgatatgtt
3003 ttgccagtta tttagcaaat gtccatgttt ccattttttg tttgatgttt tcttttgtga
3063 atcctgagtg aagtgtttca tcaacccagt gaaacgttat cgctctacat ttacatcttt 4023 gttgtgtcca cagagagaca acacaggtct cagttttatc tggaaagttg cataggatgt
reverse
Alel T: 5’---agtgacAAT|ATTttta----3’ Alel A: 5’---agtgacAATATAttta----3’
Keterangan : Alel T mempunyai basa T pada posisi basa ke 2044 Alel A mempunyai basa A pada posisi basa ke 2044
Gambar 6. Posisi Penempelan Primer (cetak tebal) pada Sekuen Gen Growth Hormone Receptor Terjadi Mutasi pada Situs Pemotongan SspI (aat|att)
Pendeteksian keragaman gen GHR|SSpI exon 8 pada sapi FH menggunakan metode PCR-RFLP (Polymerase Chain Reaction-Restriction Fragment Length Polymorphism). PCR-RFLP merupakan salah satu metode analisis lanjutan dari produk PCR. Analisis RFLP biasa digunakan untuk mendeteksi adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu (Sumantri et al., 2007). Pemotongan fragmen tersebut dilakukan dengan menggunakan enzim restriksi SSpI dengan buffer G. Suhu inkubasi enzim ini adalah 37o C. Enzim ini memotong situs aat|att yang pada produk PCR berada pada basa ke 2042. Enzim SSpI tidak akan mengenali situs pemotongan apabila terjadi mutasi pada sekuen gen. Khatib et al. (2009) menyatakan bahwa terjadi mutasi pada basa ke- 2044 di sekuen gen GHR|SSpI. Mutasi adalah suatu perubahan struktur kimia gen yang berakibat perubahan fungsi gen. Mutasi yang terjadi adalah perubahan asam amino phenylalanine yang mengkodekan alel T menjadi asam amino tyrosine yang mengkodekan alel A (Komisarek et al., 2010). Mutasi yang terjadi merupakan mutasi substitusi transversi dimana terjadi perubahan basa pirimidin (T-C) berubah menjadi basa purin (A-G). Mutasi ini bersifat non-synonimus atau mutasi yang menyebabkan perubahan asam amino. Mutasi ini menyebabkan produk amplifikasi tidak akan dikenali oleh enzim dan tidak akan terpotong. Mutasi pada gen GHR telah diasosiasikan sebagai Larontype dwarfism pada manusia (Godowski et al., 1989),
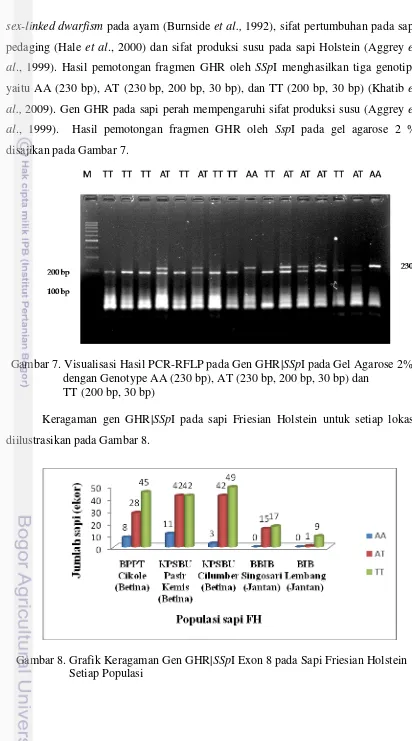
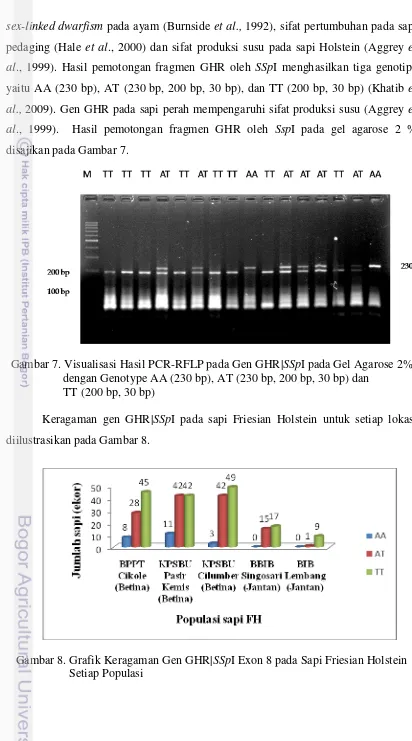
sex-linked dwarfism pada ayam (Burnside et al., 1992), sifat pertumbuhan pada sapi pedaging (Hale et al., 2000) dan sifat produksi susu pada sapi Holstein (Aggrey et al., 1999). Hasil pemotongan fragmen GHR oleh SSpI menghasilkan tiga genotipe yaitu AA (230 bp), AT (230 bp, 200 bp, 30 bp), dan TT (200 bp, 30 bp) (Khatib et al., 2009). Gen GHR pada sapi perah mempengaruhi sifat produksi susu (Aggrey et al., 1999). Hasil pemotongan fragmen GHR oleh SspI pada gel agarose 2 % disajikan pada Gambar 7.
Gambar 7. Visualisasi Hasil PCR-RFLP pada Gen GHR|SSpI pada Gel Agarose 2% dengan Genotype AA (230 bp), AT (230 bp, 200 bp, 30 bp) dan
TT (200 bp, 30 bp)
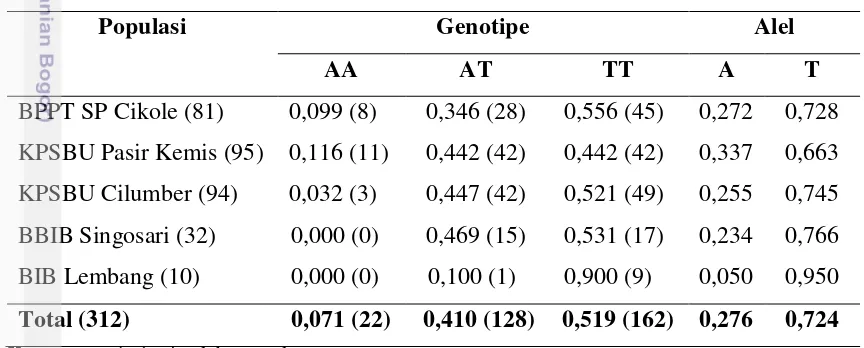
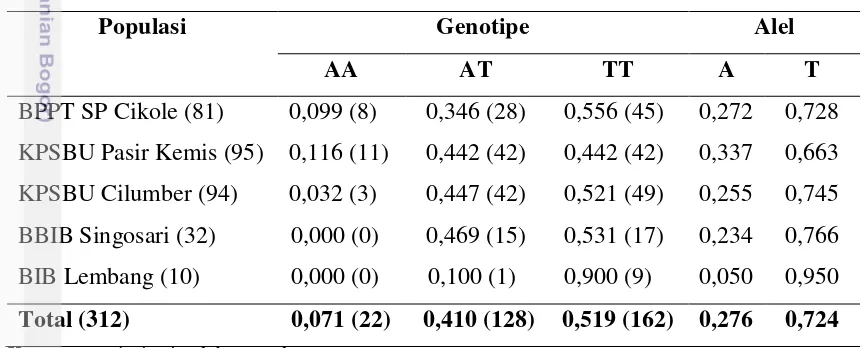
Keragaman gen GHR|SSpI pada sapi Friesian Holstein untuk setiap lokasi diilustrasikan pada Gambar 8.
Gambar 8. Grafik Keragaman Gen GHR|SSpI Exon 8 pada Sapi Friesian Holstein Setiap Populasi
Genotipe yang banyak terdapat pada sapi FH di BPPT SP Cikole adalah genotipe TT (45/81) yang diikuti dengan genotipe AT (28/81) dan terakhir adalah genotipe AA (8/81). Sapi FH di daerah KPSBU Pasir Kemis memiliki jumlah genotipe TT dan AT yang sama yaitu (42/95) kemudian genotipe paling sedikit adalah genotipe AA (11/95). Genotipe terbanyak pada daerah KPSBU Cilumber adalah genotipe AT (49/94) kemudian diikuti dengan genotipe TT (42/94) dan terakhir adalah genotipe AA (3/94). Genotipe TT pada sapi FH di BBIB Singosari merupakan genotipe terbanyak (17/32) kemudian diikuti genotipe AT (15/32) dan tidak terdapat genotipe AA. Sapi FH di BIB Lembang memiliki genotipe TT sebanyak 9 dan hanya memiliki 1 genotipe AT dari jumlah 10 sampel.
Frekuensi Genotipe dan Alel Gen GHR|SSpI
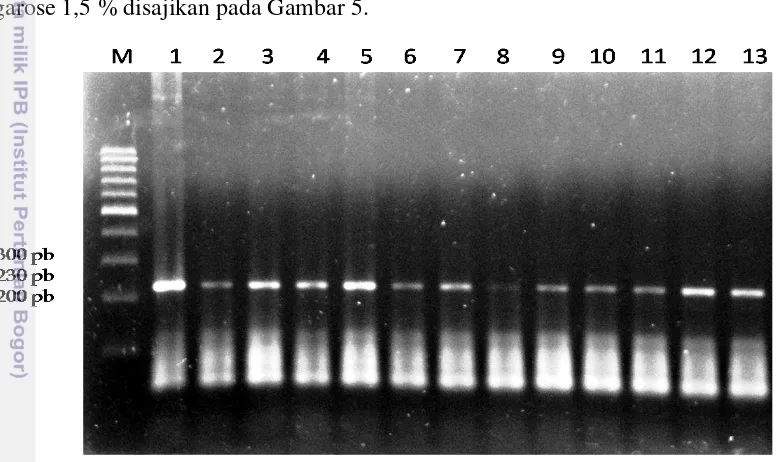
Hasil analisis nilai frekuensi genotipe dan alel gen GHR|SSpI pada sapi FH di setiap populasi dapat disajikan pada Tabel 2.
Tabel 2. Frekuensi Genotipe dan Alel pada Sapi FH di Setiap Populasi
Populasi Genotipe Alel
AA AT TT A T
BPPT SP Cikole (81) 0,099 (8) 0,346 (28) 0,556 (45) 0,272 0,728 KPSBU Pasir Kemis (95) 0,116 (11) 0,442 (42) 0,442 (42) 0,337 0,663 KPSBU Cilumber (94) 0,032 (3) 0,447 (42) 0,521 (49) 0,255 0,745 BBIB Singosari (32) 0,000 (0) 0,469 (15) 0,531 (17) 0,234 0,766 BIB Lembang (10) 0,000 (0) 0,100 (1) 0,900 (9) 0,050 0,950
Total (312) 0,071 (22) 0,410 (128) 0,519 (162) 0,276 0,724
Keterangan : (…) = jumlah sampel
Frekuensi genotipe merupakan rasio dari jumlah genotipe yang muncul pada suatu populasi. Frekuensi genotipe dan alel merupakan parameter dasar untuk mempelajari proses terjadinya evolusi karena perubahan genetik pada sebuah populasi biasanya digambarkan dengan adanya perubahan pada frekuensi alel (Nei dan Kumar, 2000). Tabel 2. menunjukkan bahwa frekuensi genotipe total dari sapi FH di semua populasi sebesar 0,071 untuk genotipe AA, 0,410 untuk genotipe AT, dan 0,519 untuk genotipe TT. Berdasarkan hasil tersebut dapat dilihat bahwa genotipe tertinggi yang terdapat di semua populasi adalah genotipe TT dan genotipe
terendah adalah genotipe AA. Nilai frekuensi genotipe tertinggi pada sapi FH di BPPT Cikole sebesar 0,556 yaitu genotipe TT. Begitu pula dengan sapi FH di KPSBU Cilumber yang memiliki nilai frekuensi genotipe tertinggi untuk genotipe TT yaitu sebesar 0,521. Nilai frekuensi genotipe TT dan AT pada sapi FH di KPSBU Pasir Kemis memiliki proporsi yang sama yaitu 0,442. Sapi FH pejantan yang berada pada BBIB Singosari dan BIB Lembang memiliki frekuensi genotipe tertinggi untuk genotipe TT yaitu sebesar 0,531 dan 0,9. Hal ini menunjukkan hasil yang sama dengan penelitian yang dilakukan oleh Komisarek et al. (2010) pada sapi Jersey yaitu nilai frekuensi tertinggi adalah genotipe TT sebesar 0,74 dan frekuensi terendah adalah genotipe AA sebesar 0,04. Begitu pula dengan hasil penelitian Fontanesi et al. (2007) pada sapi Italian Holstein Friesian, Italian Brown, Jersey, Italian Simmental, Reggiana, Rendena, Modenese yang menunjukkan bahwa nilai frekuensi tertinggi adalah genotipe TT dengan nilai berkisar 0,509-0,894 dan frekuensi terendah adalah genotipe AA dengan nilai berkisar 0,000-0,056. Menurut hasil penelitian Rahmatalla et al. (2011) nilai frekuensi genotipe tertinggi adalah genotipe TT dengan nilai 0,691 dan terendah adalah genotipe AA dengan nilai 0,0021.
Frekuensi alel adalah frekuensi relatif dari suatu alel dalam populasi atau jumlah suatu alel terhadap jumlah total alel yang terdapat dalam suatu populasi (Nei dan Kumar, 2000). Nilai frekuensi alel total untuk semua populasi memperlihatkan bahwa frekuensi alel tertinggi sebesar 0,724 untuk alel T dan terendah sebesar 0,276 untuk alel A. Frekuensi alel pada sapi FH betina di BPPT SP Cikole, KPSBU Pasir Kemis, dan Cilumber menunjukkan persamaan untuk nilai frekuensi alel tertinggi yaitu alel T dengan nilai berkisar antara 0,663-0,745. Begitu pula dengan nilai frekuensi pada sapi FH pejantan di BBIB Singosari dan BIB Lembang yang memiliki frekuensi alel tertinggi untuk alel T sebesar 0,766 dan 0,950. Hal ini sesuai dengan penelitian yang dilakukan oleh Komisarek et al. (2010) pada sapi Jersey dimana frekuensi alel T lebih besar dibandingkan alel A dengan nilai sebesar 0,85 untuk alel T dan 0,15 untuk alel A. Menurut hasil penelitian Viitala (2006) pada sapi Finnish Ayrshire frekuensi alel T lebih besar dibandingkan alel A dengan nilai sebesar 0,89 untuk alel T dan 0,11 untuk alel A. Hasil penelitian Fontanesi et al. (2007) menunjukkan bahwa pada sapi Italian Holstein Friesian, Italian Brown, Jersey, Italian Simmental, Reggiana, Rendena, Modenese memiliki nilai frekuensi alel T
yang lebih besar dibandingkan dengan alel A dengan nilai berkisar antara 0,727-0,947. Menurut hasil penelitian Rahmatalla et al. (2011) pada sapi Germany Holstein menunjukkan nilai frekuensi pada alel T lebih besar dibandingkan alel A dengan nilai 0,835 untuk alel T dan 0,165 untuk alel A.
Nei (1987) menyatakan tingkat keragaman dalam populasi digambarkan dari frekuensi alel. Suatu alel dikatakan polimorfik jika memiliki frekuensi alel sama atau kurang dari 0.99. Menurut Falconer dan Mackay (1996), suatu populasi dinilai beragam apabila memiliki dua atau lebih alel dalam satu lokus dengan frekuensi yang cukup. Berdasarkan hasil analisis frekuensi alel dapat dikatakan bahwa gen GHR|SSpI bersifat polimorfik pada sapi FH di BPPT SP Cikole, KPSBU Pasir Kemis, KPSBU Cilumber, BBIB Singosari, dan BIB Lembang.
Keseimbangan Hardy-Weinberg Gen GHR|SSpI
Hukum Hardy-Weinberg menggambarkan keseimbangan suatu lokus dalam populasi yang telah mengalami kawin acak dan bebas dari faktor yang berpengaruh terhadap terjadinya proses evolusi seperti mutasi, migrasi, dan pergeseran genetik (Gillespie, 1998). Noor (2000) menyatakan bahwa hukum ini memperlihatkan frekuensi gen dominan dan resesif pada suatu populasi yang cukup besar tidak akan berubah dari satu generasi ke generasi berikutnya jika tidak terjadinya seleksi, migrasi, mutasi, dan genetic drift. Keadaan populasi yang demikian disebut dalam keadaan equilibrium (seimbang). Hasil pengujian keseimbangan Hardy-Weinberg gen GHR|SSpI pada sapi FH di setiap populasi dengan Chi-Kuadrat (χ2) disajikan pada Tabel 3.
Hasil analisis menunjukkan bahwa pada sapi FH di BPPT SP Cikole, KPSBU Cilumber dan KPSBU Pasir Kemis berada pada keadaan keseimbangan Hardy-Weinberg (χ2 hitung < χ2
0,05). Suatu populasi dinyatakan dalam keadaan keseimbangan Hardy-Weinberg, jika frekuensi genotipe dan frekuensi alel tetap dari generasi ke generasi karena akibat penggabungan gamet yang terjadi secara acak ke dalam populasi yang besar (Vasconcellos et al., 2003). Hal ini menunjukkan tidak adanya seleksi, migrasi, mutasi ataupun genetic drift pada ketiga daerah tersebut seperti yang dikatakan oleh Noor (2010), ketidakseimbangan dapat diakibatkan karena terjadinya seleksi, migrasi, mutasi, dan genetic drift.
Sapi FH di BBIB Singosari dan BIB Lembang tidak dapat dianalisis dengan menggunakan uji Chi-Kuadrat karena populasi tersebut tidak memenuhi syarat untuk pengujian. Kedua populasi tersebut hanya memiliki dua macam genotipe dan dua macam alel sehingga derjat bebasnya bernilai nol. Allendorf dan Luikart (2007) menyatakan bahwa derajat bebas merupakan hasil pengurangan antara jumlah genotipe dengan jumlah alel.
Pendugaan Nilai Heterozigositas Gen GHR|SSpI
Hartl dan Carlk (1997) menyatakan bahwa nilai heterozigositas pengamatan dan nilai heterozigositas harapan juga dapat digunakan sebagai salah satu cara untuk menduga nilai koefisien biak dalam (inbreeding) pada suatu kelompok ternak. Hasil analisis heterozigositas pada sapi FH di setiap populasi disajikan pada Tabel 4.
Tabel 4. Pendugaan Nilai Heterozigositas Pengamatan (Ho) dan Nilai Hetezigositas Harapan (He) Gen GHR|SSpI
Keterangan : Ho = heterozigositas pengamatan He = heterozigositas harapan
Hasil analisis heterozigositas pada Tabel 4 menunjukkan bahwa pada sapi FH di BPPT SP Cikole dan KPSBU Pasir Kemis memiliki nilai heterozigositas
pengamatan (Ho) yang lebih rendah dari nilai heterozigositas harapan (He). Tambasco et al. (2003) menyatakan bahwa apabila nilai heterozigositas pengamatan (Ho) lebih rendah dari heterozigositas harapan (He) dapat mengindikasikan adanya derajat endogami atau perkawinan dalam kelompok.
Nilai heterozigositas pengamatan (Ho) pada sapi FH di KPSBU Cilumber, BBIB Singosari, dan BIB Lembang lebih tinggi dari nilai heterozigositas harapannya (He). Hal ini mengindikasikan bahwa tidak terjadi perkawinan dalam kelompok di populasi tersebut. Sapi FH pada BIB Lembang dan BBIB Singosari menyediakan semen beku dimana sapi FH yang digunakan berasal dari luar Indonesia sehingga tidak ada perkawinan dalam populasi. Nilai heterozigositas pengamatan pada semua populasi berkisar antara 0,100-0,469 dimana nilai tersebut lebih rendah dari 0,5. Javanmard et al. (2005) menyatakan bahwa nilai heterozigositas di bawah 0,5 mengindikasikan rendahnya variasi suatu gen dalam populasi. Hal ini menunjukkan bahwa pada sapi FH di semua lokasi memliki variasi gen yang rendah.
KESIMPULAN DAN SARAN
Kesimpulan
Gen GHR|SSpI pada sapi FH di BPPT SP Cikole, KPSBU Cilumber, KPSBU Pasir Kemis, BIB Lembang, dan BBIB Singosari bersifat polimorfik. Genotipe yang ditemukan yaitu AA, AT, dan TT dengan alel A dan T. Sapi FH pada BPPT Cikole, KPSBU Cilumber dan KPSBU Pasir Kemis berada pada keseimbangan Hardy-Weinberg. Nilai heterozigositas pengamatan (Ho) pada sapi FH yang berasal dari BPPT SP Cikole dan KPSBU Pasir Kemis lebih rendah dari nilai heterozigositas harapan (He), sedangkan nilai heterozigositas pengamatan (Ho) pada sapi FH di KPSBU Cilumber, BBIB Singosari, dan BIB Lembang lebih tinggi dari nilai heterozigositas harapannya (He).
Saran
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RECEPTOR
EXON 8 (GHR|
SSp
I) PADA SAPI FRIESIAN
HOLSTEIN DENGAN METODE PCR-RFLP
SKRIPSI
FEBYNIA MUTIARA ZAINATHA
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RECEPTOR
EXON 8 (GHR|
SSp
I) PADA SAPI FRIESIAN
HOLSTEIN DENGAN METODE PCR-RFLP
SKRIPSI
FEBYNIA MUTIARA ZAINATHA
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
Febynia Mutiara Zainatha. D14080220. 2012. Identifikasi Keragaman Gen Growth Hormone Receptor Exon 8 (GHR|SSpI) pada Sapi Friesian Holstein dengan Metode PCR-RFLP. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Ir. Anneke Anggraeni, M.Si, Ph.D
Growth Hormone Receptor (GHR) merupakan suatu protein transmembran yang mengikat GH dengan afinitas dan spesifitas yang tinggi. Gen Growth Hormone membutuhkan suatu reseptor dalam mekanisme ekspresinya ke target jaringan, yaitu gen Growth Hormone Receptor (GHR). Gen GHR memiliki fungsi sebagai mediasi gen pertumbuhan yang mempengaruhi sifat pertumbuhan karkas pada sapi pedaging dan sifat produksi susu pada sapi perah. Gen GHR diharapkan dapat menjadi informasi sebagai gen yang mempengaruhi produktifitas ternak melalui analisis dari keragaman gen tersebut. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen Growth Hormone Receptor (GHR) exon 8 dengan menggunakan enzim restriksi SSpI pada sapi Friesian Holstein dengan metode PCR-RFLP pada lima daerah yang berbeda, yaitu KPSBU Pasir Kemis, KPSBU Cilumber, BPPT SP Cikole, BIB Lembang, dan BBIB Singosari.
Materi penelitian berupa sampel darah sebanyak 302 sampel dan semen sapi FH sebanyak 10 sampel dengan total sebanyak 312 ekor yang terdiri dari sapi pejantan yang berasal dari BIB Lembang (10 ekor) dan BBIB Singosari (32 ekor) serta sapi betina yang berasal dari BPPT SP Cikole (81 ekor), KPSBU Pasir Kemis (95 ekor), dan KPSBU Cilumber (94 ekor). Amplifikasi gen GHR|SSpI dilakukan dengan metode Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP). Hasil produk amplifikasi adalah sepanjang 230 bp. Genotipe yang ditemukan pada gen GHR|SSpI adalah AA (230 BP), AT (230 bp, 200 bp, 30 bp), dan TT (200 bp, 30 bp) dengan dua macam alel yaitu alel A dan alel T.
Hasil analisis menunjukkan bahwa frekuensi genotipe tertinggi yang terdapat di semua populasi adalah genotipe TT dan frekuensi genotipe terendah adalah genotipe AA. Nilai frekuensi alel total untuk semua populasi memperlihatkan bahwa frekuensi alel tertinggi sebesar 0,728 untuk alel T dan terendah sebesar 0,272 untuk alel A. Sapi FH di BPPT SP Cikole, KPSBU Cilumber dan KPSBU Pasir Kemis berada pada keadaan keseimbangan Hardy-Weinberg (χ2 hitung > χ2 0,05). Hasil analisis heterozigositas menunjukkan bahwa sapi FH di BPPT SP Cikole dan KPSBU Pasir Kemis memiliki nilai heterozigositas pengamatan (Ho) yang lebih rendah dari nilai heterozigositas harapan (He), sedangkan nilai heterozigositas pengamatan (Ho) pada sapi FH di KPSBU Cilumber, BBIB Singosari, dan BIB Lembang lebih tinggi dari nilai heterozigositas harapannya (He). Kesimpulan yang dapat diambil dari hasil analisis adalah gen GHR|SSpI pada BPPT SP Cikole, KPSBU Pasir Kemis, KPSBU Cilumber, BIB Lembang dan BBIB Singosari bersifat polimorfik.
ABSTRACT
Identification of the Growth Hormone Receptor Exon 8 (GHR|SSpI) Gene Polymorphism in Holstein Friesian with PCR-RFLP Method
Zainatha, F. M., C. Sumatri, and A. Anggraeni
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RECEPTOR
EXON 8 (GHR|
SSp
I) PADA SAPI FRIESIAN
HOLSTEIN DENGAN METODE PCR-RFLP
FEBYNIA MUTIARA ZAINATHA
D14080220
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
Judul : Identifikasi Keragaman Gen Growth Hormone Receptor Exon 8 (GHR|SspI) pada Sapi Friesian Holstein dengan Metode PCR-RFLP Nama : Febynia Mutiara Zainatha
NIM : D14080220
Menyetujui,
Mengetahui, Ketua Departemen,
Ilmu Produksi dan Teknologi Peternakan
Tanggal Ujian: 25 Juni 2012 Tanggal Lulus: Pembimbing Utama,
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc ) NIP. 19591212 198603 1 004
Pembimbing Anggota,
(Ir. Anneke Anggraeni, M.Si, Ph.D) NIP. 19630924 199803 2 001
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 10 Januari 1990 di Jakarta. Penulis adalah anak pertama dari empat bersaudara pasangan Bapak Chandra Mahadewa dan Ibu Indira Komala.
Penulis mengawali pendidikan dasar tahun 1995 di TK Tunas Kasih, Cimanggis. Kemudian Penulis melanjutkan pendidikan ke SD Kartika XI-3 Jakarta pada tahun 1996 hingga kemudian pindah ke Madrasah Ibtidaiyah Muhammadiyah Bogor pada tahun keempat. Setelah itu penulis pindah kembali ke SDN Batu Ampar 04 Jakarta hingga menyelesaikan pendidikan pada tahun 2002. Pendidikan lanjutan menengah pertama dimulai pada tahun 2002 dan diselesaikan pada tahun 2005 di SMP Negeri 20 Jakarta. Penulis melanjutkan pendidikan di Sekolah Menengah Atas pada tahun 2005 dan diselesaikan pada tahun 2008 di SMA N 14 Jakarta Timur.
KATA PENGANTAR
Puji syukur penulis panjatkan kepada Tuhan Yang Maha Esa atas rahmat dan karunia yang dilimpahkan sehingga penulis dapat menyelesaikan penelitian dan penulisan skripsi ini. Salawat serta salam semoga senantiasa terlimpah dan tercurah kepada Rasulullah SAW, serta para sahabat, keluarga dan pengikutnya. Skripsi yang berjudul “Identifikasi Keragaman Gen Growth Hormone Receptor Exon 8 (GHR|SSpI) pada Sapi Friesian Holstein dengan Metode PCR-RFLP” merupakan salah satu syarat untuk memperoleh gelar sarjana peternakan di Fakultas Peternakan, Institut Pertanian Bogor.
Produktivitas sapi FH di Indonesia masih belum banyak berkembang, sehingga belum dapat memenuhi kebutuhan susu di dalam negeri. Perbaikan mutu genetik sapi perah di Indonesia dapat dikatakan sangat lambat. Perkembangan teknologi molekuler saat ini dapat lebih mempermudah dalam melakukan seleksi dibandingkan dengan cara lainnya.
Gen Growth Hormone Receptor (GHR) memiliki fungsi sebagai mediasi gen pertumbuhan yang mempengaruhi sifat pertumbuhan karkas pada sapi pedaging dan sifat produksi susu pada sapi perah. Gen GHR diharapkan dapat menjadi informasi sebagai gen yang mempengaruhi produktifitas ternak melalui analisis dari keragaman gen tersebut. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen Growth Hormone Receptor (GHR) exon 8 dengan metode PCR-RFLP pada sapi Friesian Holstein yang dipelihara di KPSBU Pasir Kemis, KPSBU Cilumber, BPPT SP Cikole, BIB Lembang, dan BBIB Singosari.
Penulis menyadari bahwa dalam penulisan skripsi ini masih banyak terdapat kekurangan, oleh karena itu kririk dan saran yang membangun sangat diharapkan demi kesempurnaan tulisan ini. Semoga tulisan ini dapat bermanfaat bagi pembaca dan memberikan kontribusi bagi kemajuan ilmu pengetahuan.
Bogor, Juli 2012
Analisis Data……….. 13 Frekuensi Genotipe dan Alel..……….. 13 Keseimbangan Hardy-Weinberg…….………. 13 Heterozigositas………. 14 HASIL DAN PEMBAHASAN……….. 15 Amplifikasi Gen GHR|SspI……….... 15 Frekuensi Genotipe dan Alel Gen GHR|SspI………. 18 Keseimbangan Hardy-Weinberg Gen GHR|SspI……… 20 Pendugaan Nilai Heterozigositas Gen GHR|SspI……… 21
KESIMPULAN DAN SARAN……….. 23
Kesimpulan………. 23
Saran……… 23
UCAPAN TERIMA KASIH……….. 24 DAFTAR PUSTAKA………. 25 LAMPIRAN……… 29
DAFTAR TABEL
Nomor Halaman
DAFTAR GAMBAR
Nomor Halaman
1. Sapi Perah Friesian Holstein………. 4 2. Rekonstruksi Struktur Gen GHR Berdasarkan Sekuen di Genbank
(Kode Akses. EF207442)………... 5 3. Visualisasi Hasil Amplifikasi Gen GHR|SSpI……….. 15
4. Posisi Penempelan Primer pada Sekuen Gen GHR………... 16 5. Visualisasi Hasil PCR-RFLP pada Gen GHR|SSpI…………... 17
DAFTAR LAMPIRAN
Nomor Halaman
1. Sekuens Gen Growth Hormone Receptor yang Diakses dari
GenBank dengan Kode Akses DQ168862.1………. 30 2. Sekuens Gen Growth Hormone Receptor yang Diakses dari
GenBank dengan Kode Akses EF207442………..
PENDAHULUAN
Latar Belakang
Bangsa sapi Friesian Holstein (FH) merupakan salah satu bangsa sapi penghasil susu tertinggi dibandingkan bangsa sapi perah lainnya di daerah tropis maupun sub tropis. Produksi susu sapi FH di Indonesia rata-rata 10 liter/hari. Namun demikian, produksi susu sapi FH belum dapat memenuhi kebutuhan susu di dalam negeri. Perbaikan mutu genetik sapi FH di Indonesia dapat dikatakan sangat lambat, walaupun sudah dilakukan inseminasi buatan. Hal ini dikarenakan belum adanya recording yang baik pada peternakan-peternakan di Indonesia. Salah satu cara untuk memperbaiki mutu genetik adalah dengan melakukan seleksi pada sapi FH itu sendiri. Perkembangan teknologi molekuler saat ini dapat lebih mempermudah dalam melakukan seleksi dibandingkan dengan cara lainnya. Seleksi melalui molekuler dapat dilakukan dengan melihat keragaman gen yang mempengaruhi produksi susu sapi FH.
Salah satu teknologi molekuler yang dapat digunakan untuk menganalisis keragaman gen adalah PCR-RFLP (Polymerase Chain Reaction-Restriction Fragment Lenght Polymorphism). Metode PCR memanfaatkan perbedaan pola pemotongan enzim restriksi atau enzim pemotong yang berbeda-beda pada setiap mikroorganisme. Analisis RFLP biasa digunakan untuk mendeteksi adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu (Sumantri et al., 2007).
Hormon pertumbuhan (Growth Hormone) merupakan salah satu hormon yang mempengaruhi produksi susu pada sapi perah. Hormon pertumbuhan ini diekspresikan oleh gen-gen. Gen Growth Hormone memiliki berat molekul yang tinggi sehingga sulit memasuki sel-sel yang harus melewati membran sel. Oleh karena itu, gen GH membutuhkan gen Growth Hormone Receptor (GHR) sebagai reseptor yang terdapat dalam membran sel, agar gen GH dapat memberikan efeknya ke target sel tanpa merusak membrannya (Djojosoebagio, 1996).
memiliki panjang 25.688 pb (Lucy et al., 1998, Jiang dan Lucy, 2001). Menurut Blott et al. (2003) terdapat beberapa exon yang diduga mempengaruhi produksi susu sapi perah, yaitu exon 3. 8, 9, dan10. Exon 8 merupakan exon yang diduga sebagai kandidat yang paling mempengaruhi produksi susu sapi perah. Keragaman gen GHR di exon 8 ini dapat dianalisis dengan menggunakan enzim rekstriksi SSpI. Oleh karena itu diperlukan penelitian mengenai keragaman gen GHR di exon 8 dan pengaruhnya terhadap produktifitas sapi perah. Keragaman gen GHR exon 8 ini diharapkan dapat dijadikan sebagai informasi untuk seleksi dalam hal peningkatan mutu genetic sapi FH di Indonesia
Tujuan
Penelitian ini bertujuan untuk mengidentifikasi keragaman gen Growth Hormone Receptor (GHR) exon 8 dengan menggunakan enzim restriksi SSpI pada sapi Friesian Holstein dengan metode PCR-RFLP pada lima daerah yang berbeda, yaitu KPSBU Pasir Kemis, KPSBU Cilumber, BPPT SP Cikole, BIB Lembang, dan BBIB Singosari.
TINJAUAN PUSTAKA
Sapi Perah Friesian Holstein
Sapi Friesian Holstein (FH) merupakan bangsa sapi yang paling banyak terdapat di Amerika Serikat, sekitar 80-90% dari seluruh sapi perah yang berada di sana. Sapi ini berasal dari Belanda yaitu di provinsi North Holand dan West Friesland yang memiliki padang rumput yang sangat luas. Sapi FH mempunyai beberapa keunggulan, salah satunya yaitu jinak, tidak tahan panas tetapi sapi ini mudah menyesuaikan diri dengan keadaan lingkungan. Ciri-ciri sapi Friesian Holstein yang baik adalah memiliki tubuh luas ke belakang, sistem dan bentuk perambingan baik, puting simetris, dan efisiensi pakan tinggi yang dialihkan menjadi produksi susu (Blakely dan Bade, 1998). Sapi ini tubuhnya memiliki pola warna hitam dan putih dan ada pula yang memiliki pola warna merah dan putih. Gambar sapi FH dapat dilihat pada Gambar 1.
Ukuran badan, kecepatan pertumbuhan serta karkasnya yang bagus menyebabkan sapi ini sangat disukai pula untuk tujuan produksi daging serta pedet untuk dipotong selain untuk produksi susu (Blakely dan Bade, 1998). Bangsa Friesian Holstein sangat menonjol dibandingkan bangsa lain karena memiliki jumlah produksi susu yang banyak dengan kadar lemak yang rendah. Sifat seperti ini sangat dibutuhkan oleh peternak sapi perah saat ini.
Bangsa sapi FH merupakan penghasil susu tertinggi dibandingkan bangsa sapi perah lainnya di daerah tropis maupun sub tropis dengan kadar lemak yang rendah. Populasi sapi perah di Indonesia diperkirakan sebanyak 487.000 ekor yang sebagian besar adalah bangsa sapi Friesian Holstein (FH) (Direktorat Jenderal Peternakan, 2009). Susu dihasilkan di sentra-sentra produksi sapi perah seperti di provinsi Jawa Barat, Jawa Tengah, dan Jawa Timur.
Gambar 1. Sapi Friesian Holstein Jantan Sumber : http://dompi.co.id
Gen Growth Hormone (GH)
Pertumbuhan merupakan salah satu sifat utama dan penting dari makhluk hidup. Secara umum pertumbuhan memiliki aspek yang luas seperti pertumbuhan sel, organ, fetus, tulang dan beberapa aspek lain yang terkait dengan pertumbuhan individu (Zulkharnaim et al., 2010). Pertumbuhan suatu individu dipengaruhi oleh hormon pertumbuhan. Hormon pertumbuhan merupakan hormon anabolik yang disintesis dan disekresikan oleh sel somatotrof pada lobus anterior pituitary (Ayuk dan Sheppard, 2006). Hormon pertumbuhan diekspresikan oleh gen-gen. Gen Growth Hormone adalah salah satu gen yang penting yang diperlukan untuk pertumbuhan jaringan, metabolisme lemak, dan pertumbuhan badan normal. Oleh karena itu, gen GH sering dijadikan kandidat untuk program Marker Assisted Selection pada sapi (Beauchemin et al., 2006). Gen GH mempunyai panjang 2800 pb dengan 5 ekson dan 4 intron dan terletak pada kromosom ke 19 (Ardiyanti et al., 2009).
Fungsi dari gen GH khususnya pada ternak menjadi sangat penting karena gen GH mengatur sifat-sifat yang bernilai ekonomi yang tinggi. Menurut Sumantran et al. (1992) gen GH terbukti menjadi pengatur utama pada pertumbuhan pasca
kelahiran, metabolisme pada mamalia, kecepatan pertumbuhan, susunan tubuh, kesehatan. Selanjutnya, gen GH merupakan kandidat gen dalam pengaturan produksi susu, karkas dan respon immun (Ge et al., 2003).
Gen Growth Hormone Receptor (GHR) menjadi kandidat gen untuk pertumbuhan pada ternak disebabkan gen GH membutuhkan reseptor dalam mekanisme ekspresinya ke target jaringan (Zulkharnaim et al., 2010). Zhou dan
Jiang (2005) menyatakan bahwa pada tingkatan jaringan, gen GH dimediasi oleh gen GHR.
Gen Growth Hormone Receptor (GHR)
Menurut Di Stasio et al. (2005), Growth Hormone Receptor (GHR) merupakan suatu protein transmembran yang mengikat GH dengan afinitas dan spesifitas yang tinggi. Gen Growth Hormone memiliki berat molekul yang tinggi sehingga sulit memasuki sel-sel yang harus melewati membran sel. Oleh karena itu, gen GH membutuhkan reseptor yang terdapat dalam membran sel, agar gen GH dapat memberikan efeknya ke target sel tanpa merusak membrannya (Djojosoebagio, 1996).
Gen GHR pada sapi dipetakan sebagai gen tunggal yang terletak pada kromosom 20 (Moody et al., 1995), terdiri atas 10 ekson dan 9 intron dan memiliki panjang 25.688 pb (Lucy et al., 1998, Jiang dan Lucy, 2001). Gambar rekonstruksi gen GHR dapat dilihat pada Gambar 2. Proses transkripsi gen GHR pada sapi di inisiasikan oleh tiga promotor exon utama 1A, 1B, dan 1C (Jiang dan Lucy, 2001).
Gambar 2. Rekonstruksi Struktur Gen Growth Hormone Receptor Berdasarkan Sekuen Gen GHR di GenBank (Kode Akses. EF207442)
Hasil produk PCR pada gen GHR exon 8 adalah sepanjang 230 bp. Hasil pemotongan fragmen GHR oleh SspI menghasilkan tiga genotipe yaitu AA (230 bp), AT (230 bp, 200 bp, 30 bp), dan TT (200 bp, 30 bp). Genotipe TT adalah genotipe yang mempunyai karakteristik produksi susu paling rendah dibandingkan dengan genotipe AT dan AA (Khatib et al., 2009). Menurut Komisarek et al. (2010) genotipe AA merupakan genotipe yang mempunyai karakteristik produksi susu paling tinggi.
Keragaman Genetik
Menurut Kirby (1990) keragaman genetik atau polimorfisme genetik adalah terdapatnya lebih dari satu bentuk atau macam genotipe di dalam populasi. Keragaman genetik adalah perbedaan di dalam runutan DNA antar individu, kelompok, atau suatu populasi. Sumber keragaman ini dapat disebabkan oleh adanya pengulangan urutan sekuen, insersi, delesi, dan rekombinasi.
Keragaman genetik dapat digunakan untuk mengetahui dan melestarikan bangsa-bangsa dalam suatu populasi yang erat kaitannya dengan penciri atau suatu sifat khusus (Blott et al., 1998). Suatu populasi yang alami memiliki tingkat keragaman yang tinggi. Frekuensi genotipe dan alel merupakan suatu parameter dasar untuk mempelajari proses terjadinya evolusi karena perubahan genetik pada sebuah populasi yang biasanya digambarkan dengan adanya perubahan pada frekuensi alel (Nei dan Kumar, 2000).
Tingkat keragaman dalam suatu populasi dapat dilihat dari frekuensi alelnya. Frekuensi alel adalah rasio relatif suatu alel terhadap keseluruhan alel yang ditemukan dalam suatu populasi (Nei dan Kumar, 2000). Menurut Falconer dan Mackay (1996), suatu populasi dinilai beragam apabila memiliki dua atau lebih alel dalam satu lokus dengan frekuensi yang cukup. Suatu alel dikatakan polimorfik jika memiliki frekuensi alel sama atau kurang dari 0,99.
Hukum Hardy-Weinberg dapat menggambarkan keseimbangan suatu lokus dalam populasi yang telah mengalami kawin acak dan bebas dari faktor yang berpengaruh terhadap terjadinya proses evolusi seperti mutasi, migrasi, dan pergeseran genetik (Gillespie, 1998). Noor (2000) menyatakan bahwa hukum ini memperlihatkan frekuensi gen dominan dan resesif pada suatu populasi yang cukup besar tidak akan berubah dari satu generasi ke generasi berikutnya jika tidak
terjadinya seleksi, migrasi, mutasi, dan genetic drift. Keadaan populasi yang demikian disebut dalam keadaan equilibrium (seimbang).
Ukuran tinggi rendahnya keragaman genetik dalam suatu kelompok atau populasi dapat dilihat berdasarkan nilai heterozigositas. Semakin tinggi derajat heterozigositas suatu populasi maka daya tahan hidup populasi tersebut akan semakin tinggi (Avise, 1994). Menurut Nei (1987), derajat heterozigositas adalah rataan presentase lokus heterozigositas tiap individu atau rataan presentase individu heterozigot dalam populasi.
Analisis Keragaman DNA
Analisis keragaman DNA bisa dilakukan dengan berbagai cara, antara lain RFLP, SSCP, DGGE, dan sequencing. Polimerase Chain Reaction (PCR) adalah suatu reaksi in vitro untuk menggandakan molekul DNA pada target yang telah ditentukan dengan cara mensintesa molekul DNA baru yang berkomplemen dengan molekul DNA tersebut dengan enzim polimerase dan oligonukleotida pendek. Proses yang terjadi dalam PCR meliputi tiga tahap yang penting yaitu denaturasi (pemisahan untai ganda DNA), annealing (penempelan primer), dan ekstensi (pemanjangan primer) (Muladno, 2002). Analisis RFLP sering digunakan untuk mendeteksi lokasi genetik dalam kromosom yang menyandikan penyakit yang diturunkan (Orita et al., 1989) ataupun untuk mendeteksi keragaman gen yang berhubungan dengan sifat-sifat ekonomis ternak seperti produksi dan kualitas susu (Sumantri et al., 2007).
Selain PCR-RFLP, PCR-SSCP juga dapat digunakan untuk analisis keragaman DNA. PCR-SSCP merupakan metode analisis lebih lanjut yang memanfaatkan produk PCR. Metode PCR-SSCP merupakan metode yang handal dalam mendeteksi adanya mutasi secara cepat (Hayashi, 1991). Asumsi yang mendasari metode analisis SSCP adalah bahwa perubahan yang terjadi pada nukleotida meskipun terjadi hanya pada satu basa, akan mempengaruhi bentuk (conformation) dari fragmen DNA pada kondisi untai tunggal (Bastos et al., 2001). Perbedaan konformasi molekul akan menyebabkan perbedaan migrasinya dalam gel poliakrilamid pada saat elektroforesis (Montaldo et al., 1998). Metode PCR-SSCP dapat mendeteksi perubahan pada satu basa tetapi tidak dapat diketahui basa mana yang berubah.
Polymerase Chain Reaction-Restriction Fragment Length Polymorphism
(PCR-RFLP)
Polymerase Chain Reaction-Restriction Fragment Length Polymorphism
(PCR-RFLP) adalah suatu metode analisis lanjutan dari produk PCR. Metode PCR memanfaatkan perbedaan pola pemotongan enzim restriksi atau enzim pemotong yang berbeda-beda pada setiap mikroorganisme (Orita et al., 1989). Prinsip kerja RFLP adalah menghilangkan semua mutasi untuk menciptakan sekuen rekognisi baru. Metode ini bisa mendeteksi mutasi jika situs restriksi mengalami perubahan susunan basa. Apabila mutasi terjadi diluar situs restriksi, maka mutasi tersebut tidak dapat dideteksi. Menurut Nei dan Kumar (2000), atas dasar terpotong atau tidaknya fragmen DNA dengan enzim pemotong, hasil fragmen potongan DNA tersebut dapat divisualisasikan melalui teknik elektroforesis yang hasilnya menunjukan ada tidaknya polimorfisme pada suatu individu atau populasi. Analisis RFLP biasa digunakan untuk mendeteksi adanya keragaman pada gen yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu (Sumantri et al., 2007).
METODE
Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboratorium Pemuliaan dan Genetika Molekuler, Bagian Pemuliaan dan Genetik Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian ini dilaksanakan selama enam bulan, yaitu dari September 2011 sampai dengan Maret 2012.
Materi
Ternak
Sampel darah dan semen yang digunakan merupakan koleksi sampel Laboratorium Genetika Molekuler tahun 2008-2010. Jumlah sampel yang digunakan adalah 312 sampel berasal dari lima daerah yang berbeda (Tabel 1).
Tabel 1. Jumlah Sampel DNA yang Digunakan dan Asal Pengambilan Sampel Darah.
Asal Sampel Jenis Kelamin Asal DNA Jumlah Sampel
KPSBU Pasir Kemis Betina Darah 95
KPSBU Cilumber Betina Darah 94
BPPT SP Cikole Betina Darah 81
BBIB Singosari Jantan Darah 32
BIB Lembang Jantan Semen 10
Jumlah 312
Bahan dan Alat
Bahan yang digunakan adalah alkohol, es, dan kapas. Alat yang digunakan antara lain jarum vacutainer, tabung vacum 10 ml dan termos.
Ekstraksi DNA
EDTA). Alat yang digunakan antara lain autoclave, satu set pipet mikro dan tipnya, vortexmixer, alat sentrifugasi, refrigerator, dan freezer.
Primer
Primer yang digunakan dalam penelitian untuk mengamplifikasi gen Growth Hormone Receptor (GHR) menurut Khatib et al. (2009) adalah
forward: 5´-CTT TGG AAT ACT TGG GCT AGC AGT GAC A”A”T AT-3´; reverse: 5´ -GTC TCT CTG TGG ACA CAA CA-3‟
Amplifikasi DNA dengan PCR-RFLP
Bahan yang digunakan adalah air bebas ion (steril), sampel DNA, buffer, MgCl2, pasangan primer, enzim taq dan dNTP, enzim retriksi SSpI dan buffer G. Alat yang digunakan adalah satu set pipet mikro dan tipnya, alat sentrifugasi, tabung PCR, mesin PCR, vortex, lemari es.
Elektroforesis
Bahan yang digunakan adalah air destilasi, agarose, 0,5x TBE, EtBr, loading dye (0,01% Xylene Cyanol, 0,01% Bromtimolblue, 50% gliserol), dan marker 100 bp. Alat yang digunakan antara lain satu set alat pencetak gel, power supply 100 volt, pipet mikro, tip dan alat gelas.
Genotyping
Bahan-bahan yang digunakan yaitu loading dye (bromthymol blue 0,01%, Xylene cyanol 0,01% dan gliserol 50%) dan untuk membuat 1 lembar gel agarose 2% adalah sebagai berikut: agarose 0,6 g, 0,5 x TBE 30 ml, dan 2,5 μl EtBr. Alat-alat yang digunakan antara lain adalah microwive, stirer, magnetic stirer, gelas ukur, tabung erlenmeyer, gel tray, pencetak untuk sumur (comb), power supply 100 volt, gelas ukur, tip, pipet makro dan mikro.
Prosedur
Pengambilan Sampel
Sampel yang digunakan terdiri dari sampel darah dan semen. Pengambilan sampel darah dilakukan pada empat tempat berbeda, yaitu KSPBU Pasir Kemis, KPSBU Cilumber, BPPT SP Cikole, dan BBIB Singosari. Sampel darah diambil
melalui vena jugularis menggunakan jarum vacutainer tabung vacum yang ditambahkan alkohol 70%. Sampel disimpan dalam termos es atau lemari es sampai akan digunakan lebih lanjut. Sampel semen didapatkan dengan cara membeli ke BIB Lembang. Sampel kemudian ditambahkan dengan alcohol 70 % dan disimpan pada suhu ruang.
Ekstraksi DNA
Sampel darah dan semen diambil sebanyak 200 µl, kemudian ditambahkan 1.000 μl DW untuk sampel darah dan 1000 µl untuk sampel semen. Sampel divortex lalu didiamkan selama 5 menit. Setelah itu disentrifuse pada kecepatan 8.000 rpm selama 5 menit, lalu supernatan dibuang, dan diulangi seperti proses sebelumnya, kemudian ditambahkan 10 µl proteinase-K yang berfungsi untuk menghancurkan protein, 350 µl 1xSTE (sodium tris-EDTA) dan 40 µl 10% SDS (sodium dodesil sulfat) yang berfungsi untuk melisiskan membran sel. Campuran tersebut kemudian diinkubasi pada suhu 55oC selama 2 jam sambil dikocok pelan menggunakan alat pemutar (tilting).
Molekul DNA kemudian dimurnikan dengan metode fenol-chloroform, yaitu dengan menambahkan 40 µl 5M NaCl, 400 µl larutan fenol dan CIAA (chloroform iso amil alcohol), lalu dikocok pelan (tilting) pada suhu ruang selama 1 jam. Molekul DNA yang larut dalam fase air dipisahkan dari fase fenol dengan alat sentrifugasi pada kecepatan 12.000 rpm selama 5 menit. Setelah terbentuk fase DNA, diambil sebanyak 400 µl pada fase DNA untuk dipindahkan ke tabung baru 1,5 ml. Kemudian ditambahkan 5M NaCl sebanyak 40 µl dan etanol absolut sebanyak 800 µl. Molekul DNA kemudian disimpan selama semalam (over night) pada suhu -20oC.
Molekul DNA kemudian dipisahkan dari etanol absolut dengan cara sentrifugasi pada kecepatan 12.000 rpm selama 5 menit, kemudian supernatan yang diperoleh dibuang. Endapan tersebut didiamkan sampai kering. Lalu endapan DNA disuspensikan dalam 100 µl 80% buffer TE (tris EDTA).
Amplifikasi DNA
Amplifikasi gen GHR dilakukan dengan menggunakan campuran yang terdiri dari 1 µl sampel DNA yang sudah diekstraksi, 10,85 µl DW, 0,3 µl primer, 0,05 µl taq polymerase, 1,5 µl buffer, 0,3 µl dNTP dan 1 µl MgCl2. Campuran tersebut